Method Article
Гель seq: Метод для подготовки библиотека одновременных секвенирования ДНК и РНК с помощью матрицы Гидрогель
В этой статье
Резюме
Гель seq позволяет исследователям одновременно готовить библиотек для обоих ДНК - и РНК seq в незначительной добавленной стоимости, начиная от 100-1000 ячеек с помощью простой гидрогеля устройства. Этот документ представляет детальный подход для изготовления устройства, а также биологические протокол для создания парных библиотек.
Аннотация
В за последние пять лет был достигнут только возможность усилить и последовательности ДНК или РНК от небольшой начальный образцов. К сожалению стандартные протоколы для генерации геномной или транскриптомики библиотеки являются несовместимыми и исследователи должны выбрать, следует ли последовательность ДНК или РНК для конкретной выборки. Гель seq решает эту проблему, позволяя исследователей подготовить одновременно библиотек для ДНК и РНК, начиная с 100-1000 ячеек с помощью простой гидрогеля устройства. Этот документ представляет детальный подход для изготовления устройства, а также биологические протокол для создания парных библиотек. Мы разработали гель seq, так что он легко может осуществляться другими исследователями; Многие лаборатории генетики уже есть необходимое оборудование, чтобы воспроизвести изготовление гель seq устройства. Наш протокол использует часто используемые наборы для обоих целом Стенограмма амплификации (WTA) и подготовка библиотеки, которые также могут быть знакомы исследователей уже разбираются в генерации геномных и транскриптомики библиотеки. Наш подход позволяет исследователям привнести власть ДНК и РНК последовательности на одном образце без разделения и с незначительной добавленной стоимости.
Введение
Следующее поколение виртуализации (НГС) оказывает глубокое влияние на способ исследования генетики. Там, где исследователи раз сосредоточено на Секвенирование генома всего видов, это теперь возможно последовательности генома единый опухоли или даже одну ячейку в одном эксперименте. 1 NGS также сделал экономически эффективным для последовательности РНК стенограммы, нашли в ячейке, сбор данных, известный как транскриптом. В за последние пять лет был достигнут только возможность усилить и последовательности ДНК или РНК от небольшой начальный образцов. 2 , 3 , К сожалению, 4 стандартные протоколы несовместимы и исследователи должны выбрать, следует ли последовательность ДНК или РНК для данного образца. Когда образец начала достаточно велик, он может разделить пополам. В меньших масштабах однако потери материала за счет расщепления образцов может повлиять на качество библиотеки, и объединения образцов может среднем, интересные вариации между ячейками. 5 Кроме того, исследователи все больше заинтересованы в изучении образцов, которые нельзя разделить, например отдельные ячейки или биопсии малых гетерогенных опухоли. 6
Для решения этой проблемы, три были недавно разработаны протоколы для последовательностей ДНК и РНК из того же образца начала: гель seq7,8G и T-seq и DR-seq9. Эта статья представляет подробный протокол для гель seq, который может использоваться для одновременно создавать библиотеки ДНК и РНК из всего лишь 100 клеток в незначительной добавленной стоимости. Роман аспект гель seq является возможность отделить ДНК и РНК, исключительно на основе размера с использованием недорогих гидрогеля матрицы. Основные инновации гель-Seq протокола является физическое разделение ДНК из РНК. Это разделение достигается электрофорезно используя комбинацию полиакриламида мембран, которые используют преимущества размер различия между этими молекулами. Положить эти различия размер в контексте, рассмотрим как отражаются ДНК и РНК: в то время как ДНК существует на шкале микрон и могут быть просмотрены с помощью традиционных Микроскопы, РНК существует в нанометровом масштабе и должны отражаться с использованием сложных методов например крио электрон микроскопия. 10
Подход к отделять ДНК и РНК в этот протокол показан на рисунке 1. Левая панель показывает, что ДНК и РНК Свободный плавающий в растворе вблизи мембраны. Когда электрическое поле применяется, как показано в правой панели, ДНК и РНК испытывают электрофоретические сила, которая побуждает миграции через мембрану. Путем настройки свойства мембраны, мы создали полупроницаемую мембрану, разделяющую ДНК от РНК. Молекулы ДНК толкнул мембраны, но запутаться на границе из-за их больших размеров. Малые молекулы РНК, с другой стороны, можно перенастроить и ткут свой путь через мембрану. Этот процесс, известный как reptation, подобно тому, как змея движется через траву. В конечном итоге эти молекулы РНК остановлены мембраной второй, высокой плотности, который является слишком сложным для даже меньше полимеров (> 200 пар оснований) чтобы пробираться через. После того, как физически отделены, ДНК и РНК можно восстановить и обработаны для получения информации о геноме и транскриптом. Хотя мы можем отделить ДНК и РНК, мы нашли, что лучшие результаты получаются, если РНК обратной транскрипции cDNA перед разлукой. Гибридов cDNA/РНК более стабильны, чем РНК только и может еще пройти через low-density мембраны.

Рисунок 1 . Гель seq принцип. Основной принцип, используемый для физически отделить ДНК и РНК. В прикладной электрического поля малых молекул РНК мигрируют через low-density мембраны, но большие молекулы ДНК оказываются на поверхности. Этот показатель был воспроизведен из ссылка 7 с разрешения Королевского общества химии. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Этот документ подробно описывает, как изготовление гель seq устройства и биологических протокол для создания паре библиотеки ДНК и РНК. Обзор как показано на рисунке 2. Устройство изготавливается путем наслаивать три разные плотности полиакриламидных гелей друг на друга в процесса, аналогичного процессу создания стандартных гели для укладки. 11 биологического протокола начинается с 100-1000 ячеек приостановлено в PBS. Клетки лизированы и РНК преобразуется в cDNA прежде чем устройство используется для разделения геномной ДНК от гибридов cDNA/РНК. После разделения и восстановления, геномных и транскриптомики библиотеки готовятся с использованием процесса, который внимательно следит за протокол комплект подготовки стандартной библиотеки всего генома. Более подробно о разработке и проверке гель seq можно прочитать в лаборатории на публикацию чип «гель seq: всего генома и транскриптом последовательности путем одновременной низкозатратных, ДНК и РНК подготовки библиотеки с использованием полупроницаемой гидрогеля барьеры ." 7

Рисунок 2 . Гель seq протокол. Обзор шагов для изготовления гель seq устройства и протокол для генерации парных библиотеки ДНК и РНК. Части этой фигуры были воспроизведены из ссылка 7 с разрешения Королевского общества химии. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Для создания библиотеки ДНК и РНК из одной клетки, исследователи следует рассмотреть возможность использования G & T-seq или DR-след G и T-Seq, как гель seq, опирается на физическое разделение РНК от геномной ДНК. Этот подход основывается на матричная РНК (мРНК) 3′ polyadenylated хвост как выпадающее цель. МРНК захватывается на магнитный шарик, используя праймер биотинилированным oligo-dT. После того, как был захвачен мРНК бисер удерживаются на месте с магнитом и супернатанта, содержащий геномной ДНК могут быть удалены и переведен в другой трубки. После завершения этого физического разделения отдельных библиотек могут создаваться мРНК и ДНК. 8 этот подход работает хорошо, если РНК интерес polyadenylated, однако он не может использоваться для изучения стенограммы non-polyadenylated, таких как рибосомная РНК, tRNA, или РНК от прокариот.
DR-seq зависит от предварительного усиления шаг, где cDNA полученных от РНК и ДНК размножаются в том же трубе. Этот образец затем надвое и обрабатываются параллельно подготовить ДНК и РНК seq библиотек. Чтобы различать геномной ДНК и cDNA, производный от РНК, DR-seq занимает вычислительной подход. Последовательности, где присутствуют только экзонов вычислительно подавляются в геномной ДНК-данных, как те могли происходить от ДНК или РНК. 9 преимуществом этого подхода является, что ДНК и РНК/cDNA нужно не быть разделены физически как это делается в гель seq и G и T-seq. Недостатком, однако, является, что DR-seq требует априорных знаний о геноме и транскриптом (то есть, экзонов против интронов) и не может быть идеально подходит для приложений, таких как последовательности ядер, в которых многие стенограммы еще не полностью сращивания и по-прежнему содержать интроны. 12
Роман аспект гель seq является возможность отделить ДНК и РНК в сотни клеток, исключительно на основе размера. Этот метод требует априори знания геном или транскриптом, устойчива против неполной сплайсинга и не ограничивается стенограммы поли adenylated. Для приложений, где исследователь может начать с по крайней мере 100 клеток гель seq обеспечивает простой подход с использованием материалов, дешево и широко доступны.
протокол
1. химический раствор подготовка
Примечание: Следующие шаги являются для приготовления химических растворов, необходимых в последующих шагах. Это можно сделать навалом и хранятся в течение нескольких месяцев.
- Чтобы начать, подготовьте 50 мл очищенной и обессоленной воды, стерилизация в 254 Нм УФ сшивки духовку на 15 мин (15 МДж/см2 общее воздействие) для нейтрализации любых загрязняющих ДНК. Нагрейте до 37 ° C для использования в следующих шагов.
- Сделать 10 мл 40% всего (T) и 3,3% сшивателя (C) (29: 1) раствор полиакриламида прекурсоров. Вес 3.867 г мономера акриламида и 0,133 g бис акриламид мономера. Комбинат и довести объем до 10 мл с теплой очищенной воды и вихревой до растворения. Магазин, защищенном от света при комнатной температуре.
Примечание: Готовые 40% T, C (29: 1) 3,3% полиакриламида решения могут быть приобретены коммерчески. - Сделайте 10 мл 50% T, гель 5% C решения. Весят 4.750 g на мономера акриламида и 0,250 g бис акриламид мономера. Комбинат и довести объем до 10 мл, с использованием теплой очищенной воды и вихревой до растворения. Магазин, защищенном от света при комнатной температуре.
- Сделайте 10 мл раствора сахарозы 50% (w/v). 5 г сахарозы в мензурку и добавить теплой очищенной воды до общего объема 10 мл. Вихревой пока растворится и хранить при комнатной температуре.
- Сделать 10 мл 10% APS (w/v). Добавьте 1 g Персульфат аммония (APS) в мерный цилиндр. Добавить холодной (~ 4° C) очищенной воды до общего объема по 10 мл и вихревой до растворения. Немедленно заморозить в аликвоты 200 мкл.
2. гель seq Изготовление кассеты
Примечание: Гель seq был первоначально разработан с вертикально кассеты (см. Таблицу материалов для получения дополнительной информации); Однако этот протокол может быть адаптирована для работы с любой стандартной гель электрофорез кассеты.
- Подготовка геля прекурсоров в трех отдельных пластиковых труб путем добавления реагентов, как показано ниже в таблице 1 . Не добавить APS или Tetramethylethylenediamine (TEMED) до тех пор, пока направлены в следующих шагах. Вихревой ингредиенты тщательно перемешать.
| Наполнитель гель прекурсоров | Высокая плотность геля прекурсоров | Низкой плотности геля прекурсоров | |||
| 40 %T, 3.3%C акриламида Bisacrylamide решения | 1.6 mL | 50 %T, 5 %C акриламида раствор Bisacrylamide | 2.4 mL | 40 %T, 3.3%C акриламида Bisacrylamide решения | 0,6 мл |
| Деионизированная вода | 10.2 мл | Деионизированная вода | 1,0 мл | Деионизированная вода | 4,8 мл |
| Раствора сахарозы (50% w/v) | 2,6 мл | Раствора сахарозы (50% w/v) | 0,6 мл | ||
| 10 X трис Борат ЭДТА | 1.6 mL | 10 X трис Борат ЭДТА | 0,6 мл | ||
| Персульфат аммония (10% w/v) | 104.0 МКЛ | Персульфат аммония (10% w/v) | 50.0 МКЛ | Персульфат аммония (10% w/v) | 39.0 МКЛ |
| TEMED | 6.0 МКЛ | TEMED | 1.0 МКЛ | TEMED | 2.2 МКЛ |
| Общий объем | 16.1 мл | Общий объем | 4.1 мл | Общий объем | 6.0 мл |
Таблицы 1. Гель синтез реагентов. Геля полиакриламида прекурсоров реагенты достаточно для изготовления 2 кассеты.
- Де газ решения гелем мономера, вставляя иглу через крышку трубки и подключения этой иглы к линии вакуумной дом. Погружать этой Ассамблеей в ультразвуковых ванн до максимума и ждать, пока пузырьки остановить, возникающих от жидкости перед переходом к следующему шагу (~ 60 секунд / tube).
Примечание: Качество вакуума и мощности в ультразвуковой ванне важно не до тех пор, как пузыри можно увидеть новые решения. - Добавьте APS и TEMED наполнитель гель прекурсоров и вихревой кратко. Сразу же добавьте 6 мл наполнитель гель прекурсора каждая кассета гель, закупорить решения в верхней части кассеты. Используйте 1 мл микропипеткой для добавления прекурсоров в шести шагом, чтобы избежать разлив. Общее время для этого шага должна быть не более 3 минут.
- Убедитесь, что жидкость является уровень позиционирования кассеты вертикально на уровне таблицы. На оставшуюся часть кассеты с дегазацию деионизированной водой. Пипетка воды, снова с шагом 1 мл, медленно в центре кассета для сведения к минимуму перемешивания. Разрешить полимер для лечения по крайней мере одного часа, до для ночевки.
- После отверждения полимеров, Инвертируйте гель кассеты над раковиной для удаления воды оверлея. Пневматический пистолет может использоваться для Осторожно высушить интерфейс продувкой воздуха через верхнее отверстие кассеты с расстояния 6 дюймов.
- Добавьте APS и TEMED плотностью геля прекурсоров и вихревой кратко. Сразу же мкл 320 высокой плотности прекурсор к кассете, снова с помощью пипетки 1 мл. Убедитесь, что жидкость равномерно пальто гель слоя наполнителя, тряся кассету обратно примерно в 3 раза. Этот шаг должен принять менее трех минут.
- На оставшуюся часть кассеты с дегазацию деионизированной водой. Пипетка медленно с пипетки 1 мл в центре кассета для сведения к минимуму перемешивания. Разрешить полимер для лечения для, по крайней мере пятнадцать минут, желательно через час.
- Опять же Инвертируйте кассеты гель для удаления воды оверлея. Сжатый воздух может использоваться для Осторожно высушить интерфейс. Добавьте APS и TEMED low-density гель прекурсоров и вихревой кратко. Сразу же заполнить оставшуюся часть кассеты (~1.65 мл) с низкой плотностью геля прекурсоров и вставить гребень геля.
- Пипетка избыток заповедник прекурсоров в верхней части гребня, как он будет поглощена в процессе полимеризации. Разрешить полимер для лечения по крайней мере 4 часов, желательно на ночь. Гели могут храниться в течение недели или больше в буфер Tris-Борат-Этилендиаминтетрауксусная кислота (ЭДТА) (КЭ буфера).
3. Подготовка и обратить вспять транскрипции
- Начиная с суспензию клеток интерес и в полимеразной цепной реакции (ПЦР) Ламинарный шкаф, использовать Горяева или автоматический клетки счетчик для расчета концентрации клеток. Разбавьте клетки к концентрации 100 до 1000 клеток / мкл в фосфат амортизированное saline (PBS).
Примечание: Этот протокол была проверена на диапазон ячеек, включая PC3 и НеЬа клетки мыши печени. - С помощью реагентов в WTA комплекта (см. Таблицу материалы), смесь 19 мкл буфера lysis и 1 мкл АБС битор РНКазы подготовить 10 X запасов решение реакции буфера. Создайте лизис мастер смесь достаточно тома, содержащего 0,5 мкл буфера реакции и 2,75 мкл нуклеиназы бесплатный воды для каждого образца.
- Пипетка суспензию клеток вверх и вниз 5 раз вновь приостановить поселились клетки и затем Пипетка 1 мкл пример в трубу свободного газа нуклеиназы 200 мкл, стерилизована УФ. Повторите при необходимости в зависимости от количество выборок. Не забудьте включить отрицательный контроль дозирования 1 мкл нуклеиназы свободной воды вместо клетки для одного реакции. Далее 3.25 мкл лизис мастер смеси для каждой выборки и смешайте нежно закупорить вверх и вниз 5 раз.
- Разогрейте тепловая велосипедист (с подогревом крышкой) до 72 ° C. Добавьте 1 мкл RT грунтовки и 1 мкл 20 мкм случайных hexamer с WTA адаптер (5 ' AAGCAGTGGTAT-CAACGCAGAGTAC-NNNNNN-3′) для каждого образца. Резерв по крайней мере одна трубка как положительный контроль подготовки библиотека геномная ДНК и 2 мкл воды вместо грунтовки.
Примечание: Случайные hexamer с WTA адаптер является необязательным и оказывает минимальное влияние на результаты секвенирования. - Проинкубируйте образцы на 72 ° C в разогретой тепловой циклователь на 3 минуты для Лизируйте клетки. Удаление ячеек из тепловая велосипедист и место на льду на 2 минуты. Сохранять положительный контроль при температуре 4 ° C до шаг 5.
- В то время как лизировать клетки, создать достаточный объем обратной транскрипции мастер смеси для всех образцов РНК, содержащие следующие соотношения реагентов: 2 мкл буфера первой пряди, 0.5 мкл шаблон переключатель олигонуклеотида (ОПС), 0,25 мкл битор РНКазы и 1 мкл обратной транскриптазы (100 U/мкл).
- Разогрейте тепловая велосипедист до 42 ° C. 3.75 мкл обратной транскрипции мастер смеси для оставшихся образцов, достигло общей выборки объем 10 мкл. микс от закупорить вверх и вниз 5 раз.
- Выполнение обратной транскрипции, немедленно поставив образцов в разогретой тепловая велосипедист. Запустите следующую программу: 42 ° C 90 мин, 70 ° C на 10 мин., 4 ° C навсегда. Это точка безопасной остановки.
4. гель разделения и восстановления
- Тщательно очистите камеру электрофореза геля с помощью ДНК удаления продукта. Применить несколько мл жидкого агента очистки бесплатно протрите одноразовой Линт и протрите все поверхности камеры, а затем заполнить камеры с чистой 0,5 x TBE. Для получения оптимальных результатов место весь аппарат в 254 Нм УФ сшивки печи и стерилизовать 15 минут (15 МДж/см2).
- Вставьте кассету гель seq в зале электрофореза геля и зафиксируйте его на месте. Медленно снимите гребень для геля, потянув вверх. Двигаться медленно, чтобы избежать разрывов геля или копирования любого оружия.
- Сохранение образцов из шага 3 на льду, зарезервировать по крайней мере один образец как позитивный элемент управления для поколения кДНК библиотеки. Храните этот элемент управления на 4 ° C до шага 6. 2 мкл 6 X загрузки краситель для оставшихся образцов, результате чего общий объем ~ 12 мкл. тщательно перемешать образцы, закупорить вверх и вниз 5 раз.
- 1 мкл лестница ДНК в сочетании с 2 мкл 6 x загрузки красителя и 7 мкл воды. Пипетка эту смесь в майну 1 гель seq кассеты как элемент электрофореза. Пипетка образцы из предыдущего шага в отдельные полосы гель seq кассеты. Будьте осторожны, чтобы не допустить загрязнения между скважинами, вставив пипеткой полностью в каждой скважине и удаление его с помощью только вертикальное движение.
- Использование стандартного геля электрофорез питания, применить электрического поля 250 V через гель seq кассету для 30 минут, чтобы отделить геномная ДНК от гибридов cDNA/РНК. После отделения, извлеките кассету гель seq из камеры электрофореза геля и открыть две половинки кассету, выломав края с соскоб инструмент.
- С помощью скальпеля, пополам геля чуть ниже плотности слоя. Отказаться от половины, содержащий наполнитель гель, подбирая с рукой в перчатке. Аккуратно чистить остатки геля офф кассеты соскабливания с скребок краской или другие аналогичные средства. Поместите этот раздел геля в блюдо, содержащие ~ 30 мл 0,5 x TBE с 3 мкл пятно гель.
- Обложка контейнер, чтобы свести к минимуму Фотообесцвечивание и замочить гель во время нежно тряска контейнер для 5 минут. Поместите гель на полиэтиленовой пленкой и УФ образ с помощью системы документации геля (для дальнейшей детализации см 13 ссылка). 30 второй экспозиции обычно создает четкие изображения. Убедитесь, что произошло разделение.
- Перенести гель УФ transilluminator для облегчения визуализации нуклеиновых кислот. Носить соответствующие очки УФ, подтверждают результаты от гель системы документации. Геномная ДНК должен быть расположен в начале плотностью геля и cDNA на стыке регионов низкой плотностью и высокой плотности.
- Использовать скальпель, вырезать из регионов гель, содержащий и геномная ДНК cDNA. Лучшие образцы восстанавливаются путем разрезания 4 мм х 10 мм прямоугольного сечения геля; Однако точный геометрия будет зависеть от используемой электрофореза геля. Не забудьте также сократить полосу загружен с отрицательного контроля.
- Место каждого вырезан секции геля в трубку газа, используя пинцет тупой конец. Будьте осторожны, чтобы не применять слишком много сил или гель будет разделить на несколько частей. Следует это произойдет, просто забрать каждый кусок и добавить его к трубе.
- Измельчения геля в каждой тюбике с помощью кончика пипетки (200 мкл пипеткой советы работают хорошо), перемещая наконечник пипетки в циркуляре моды против нижней части трубки. Добавьте нуклеиназы свободной воды (40 мкл в геномная ДНК образцов и 80 мкл в cDNA образцы) в каждую пробирку перед удалением наконечник пипетки, используется для измельчения геля для сведения к минимуму потерь образца.
- Поместите трубки газа в вихревой смеситель внутри инкубатора 37 ° C и встряхнуть для 8-12 часов. Это позволяет нуклеиновые кислоты для диффузного из геля и является естественным остановочный пункт для этот день multi протокола.
- Пипетка образцы в 8 мкм сетка фильтра плиты и спина пластину на 2600 x g 5 минут штамм фрагментов геля. Поднимите пластину сетки фильтра от корпусным диском и Пипетка воды гель бесплатные образцы в новой трубки газа 200 мкл.
- 1 мкл протеазы (0,9 AU/мл) в каждый образец, содержащий геномная ДНК, смесь хорошо, закупорить вверх и вниз и Инкубируйте на 50 ° C 15 мин, следуют тепловой инактивации при 70 ° C на 15 мин. Этот шаг имеет решающее значение для разрушающих нуклеосом и делает доступным для последующей реакции шаги геномная ДНК.
- С помощью 18 иглы, тыкать отверстия в шапках всех образцов труб. Поместите образцы в vacufuge для уменьшения объема жидкости. Геномная ДНК образцов следует уменьшить до 5 мкл и cDNA образцов, уменьшен до 10 мкл.
- В зависимости от vacufuge и количество образцов суммарного испарения время будет варьироваться между 30 и 60 минут. Если объем образца падает ниже целевого тома, просто добавьте нуклеиназы свободной воды для увеличения объема выборки.
5. геномная ДНК библиотеки подготовка
- С помощью флуориметр или аналогичные технологии, количественного определения концентрации ДНК в каждом геномная ДНК образцов из шага 4, а также положительный контроль из шага 3. Для подробного протокола увидеть флуориметр справочное руководство. 14
- Развести образцы для 0.2 нг/мкл ДНК. В зависимости от начальной ячейки тип и качество результатов требуется более низкие концентрации может все еще производят жизнеспособный библиотек. Потребуются некоторые эксперименты; Однако авторы имели успех с библиотеками, как низко как 0.1 нг/мкл.
- Завершить подготовку библиотека геномная ДНК, следуя библиотека подготовки половина реакции объем протокол на шаге 7.
6. кДНК библиотеки подготовка
- Начиная с 10 мкл cDNA образцов и позитивного управления из шага 4, 2 X qPCR микс, 0.5 мкл cDNA PCR праймер и 2 мкл нуклеиназы свободной воды 12,5 мкл.
- Выполнять ПЦР в реальном времени Термоциклер, используя следующий протокол: горячий старт при 95 ° C в течение 3 мин, затем 20-30 циклов 98 ° c 10 s, 65 ° C для 30 s и 72 ° C для объем общей выборки 3 мин 25 мкл. контролировать реакцию кривых и остановить усиление до го e реакции покинуть экспоненциальная фаза (линейный сигнал увеличение против номер цикла) во избежание ПЦР артефакты из-за overamplification. Более подробно о избегая overamplification в разделе обсуждения этот документ, а также ссылка 15.
- После усиления очистите продукт с помощью твердой фазы реверсивные иммобилизации (ОСПР) бусы после протокол на шаге 8. После завершения, перейдите к следующему шагу.
- С помощью флуориметр или аналогичные технологии, количественного определения концентрации ДНК в каждой пробе cDNA, а также положительный контроль из шага 4. Развести образцы, если необходимо содержать примерно 0,2 нг/мкл ДНК. Несколько более низкие концентрации все еще производят жизнеспособный библиотек. Некоторые эксперименты потребуются, однако авторы имел успех с библиотеками, как низко как 0.1 нг/мкл.
- Необязательный шаг: выполнить разделение электрофорез стандартного геля полиакриламида на 1-2 мкл каждого продукта ПЦР для проверки реакции поколения библиотека работал. Пример изображения успешного результата показана на рисунке 4. Для подробного протокола о том, как проводить электрофорез геля полиакриламида, ссылка 16 см.
- Завершить подготовку кДНК библиотеки, следуя протокол библиотеки подготовка комплекта половины объема реакции на шаге 7.
7. Библиотека подготовка с половины объема реакции
- Подготовка библиотеки следующим библиотеки подготовка комплекта протокола с использованием половину объема реакции. 17 все реагенты, ссылки в этом разделе протокола являются из библиотеки подготовка комплекта (см. Таблицу материалы). Начните с УФ стерилизации достаточное количество газа трубы для количество выборок для обработки.
- Выполните Транспозаза реакции, добавив 5 мкл Транспозаза буфер для каждой полосы трубы для использования в assay. Затем добавьте 2.5 мкл ввода ДНК на 0.2 нг/мкл (всего 0,5 нг) следуют 2,5 мкл Транспозаза. Смешайте закупорить вверх и вниз 5 раз.
- Инкубируйте на 55 ° C за 5 минут, а затем провести на 10 ° C. После того, как образец достигает 10 ° C, удалите его из Термоциклер и сразу же добавить 2,5 мкл Транспозаза остановка буфера для каждого образца. Держите образцов при комнатной температуре за 5 минут.
- Подготовьте реакции PCR усиливает образец транспонировать лечение путем добавления 7,5 мкл библиотека приготовительный ПЦР микс, 2.5 мкл грунтовка индекс 1 и 2,5 мкл индекс 2 праймера. Эти грунты являются собственностью и поставляются заводом-изготовителем комплекта подготовки библиотеки. Смешайте хорошо закупорить вверх и вниз 5 раз.
Примечание: Убедитесь, что уникальный грунтовка комбинаций, которые используются для каждого образца. Есть 12 различных индекс 1 грунты и 8 различных индекс 2 грунтовки, что дает возможность однозначно ярлык до 96 различных образцов. Выберите уникальное сочетание праймеров для каждого образца. - Выполняйте ПЦР, используя следующую программу на Термоциклер. Объем выборки составляет 25 мкл.
72 ° C 3 минуты
95 ° C на 30 секунд
12 циклов:
95 ° C в течение 10 секунд
72 ° C на 30 секунд
55 ° C за 30 секунд
72 ° C за 5 минут
Держите на 10 ° C - Чистите подготовлены библиотек с использованием Спри бусы после протокол на шаге 8. Библиотеки должны проверяться с помощью электрофореза геля или аналогичного анализа. Обратитесь к руководству комплект подготовки библиотека для подробной информации о том, как проверить библиотеки. 17
8. твердой фазы реверсивные иммобилизации шарик библиотека очистка
- Твердой фазы бусы реверсивные иммобилизации (ОСПР) должны храниться в 1,5 мл аликвоты и должны быть доведены до комнатной температуры перед каждым использованием. Также рекомендуется подготовить свежие 80% этанола для каждого эксперимента. Следующие шаги основаны Спри шарик протокол в руководстве комплект WTA. 18
- 1 мкл буфера lysis комплект WTA, для каждого продукта PCR. Вихревой Спри бусы до тех пор, пока равномерно смешанные, затем 50 мкл Спри бусы для каждого образца. Смешать, закупорить образец вверх и вниз в 10 раз и затем Проинкубируйте образцы при комнатной температуре за 8 минут.
- Кратко спина образцы для сбора жидкости со стороны трубы. Поместите образцы на устройстве магнитной сепарации ~ 5 минут до тех пор, пока жидкость появляется совершенно ясно.
- Хотя образцы на устройстве магнитной сепарации, медленно Пипетка супернатант и отменить - будьте осторожны, чтобы не нарушить кольцо из бисера на трубе. Далее добавьте 200 мкл 80% этанола для каждого образца не нарушая бисер. Подождите 30 секунд и затем тщательно Пипетка покинуть супернатант. Повторите этот шаг (этанол мыть) один раз.
- Разрешить образцы для просушки на 30 сек - 1 мин. Не чрезмерно сухой образцы как большие фрагменты будут стать постоянно привязан к бисер.
Примечание: Краткое время просушки рекомендуется убедиться, что удалены все следы этанола. Следовые количества этанола останется позади они могут слегка препятствуют течению реакций. - Удалить образцы из устройства магнитной сепарации и 15 мкл воды для каждого образца для элюировать ДНК из бисера. Пипетка вверх и вниз и убедитесь, что бусины, удаляются из стороны трубы. Инкубации при комнатной температуре в течение 2 минут. Кратко спина образцы и затем разместить их на устройство магнитной сепарации для ~ 1 минуту, пока не появится четкое решение.
- Хотя образцы на устройстве магнитной сепарации, медленно Пипетка вверх супернатант и перенести его на чистые трубы. Будьте осторожны, чтобы не нарушить кольцо из бисера на трубе. Отказаться от трубы с бисером.
Результаты
Физическое разделение геномная ДНК и cDNA/RNA гибридов в гель seq устройства могут быть визуализированы через флуоресцентные гель изображений; Представитель результат показан на рисунке 3. Группа A показывает сфабрикованные гель seq устройства; ложный цвет был добавлен отличить различные гель регионов. Группа B показывает закрыть вверх четыре различных отделений, используемых для проверки. Третий Лейн, отрицательный контроль, представляет фон и показывает, что не autoflourescence геля на интерфейсы. Мы погрузили первой и второй полосы с ДНК лестницы. Эти полосы показывают только темные полосы на стыке между низкой и высокой плотностью мембраны, показывая, что небольшие фрагменты могут пройти через плотностью геля. Четвертый Лейн показывает поведение биологического образца интерес: 500 клеток PC3. Мы погрузили переулок 4, как описано в шаге 3 протокола. На рисунке показано разделение геномной ДНК и гибридов cDNA/РНК. Темная полоса в верхней части low-density мембраны то megabase шкала геномной ДНК cDNA/RNA гибридов укладываются на стыке регионов низкой и высокой плотности. В отличие от полосы, загружен с лестницей есть также несколько полос, присутствующие в регионе с высокой плотностью геля. Эти фрагменты, меньше, чем 100 bp, пробить продукты, образующиеся грунтовка олигонуклеотиды во время обратной транскрипции. Группа C показывает образ представителя всего гель seq устройства от успешный эксперимент. Полос, помечены РНК/cDNA были обработаны с гель seq протокол, в то время как полосы, помечены РНК показывают разделение просто геномная ДНК и РНК. Группа D показывает неудавшийся эксперимент с черными полосами в верхней части каждого Лейн low-density мембраны. Это было вызвано буфер электрофореза, загрязненные фрагментированных ДНК. На этом шаге исследователи должны искать чистый негативный контроль и два отдельных черные полосы, указывающих на наличие разлученных геномная ДНК и РНК/cDNA гибридов.

Рисунок 3 . Гель Seq разделения результатов. Гель seq устройства (A) и флуоресцентного изображения показаны разделение ДНК и РНК/cDNA гибриды (B). Ложный цвет был добавлен к более легко различать между различными регионами плотности в гель. (C и D) Представитель флуоресцентного изображения всего гель seq устройства от успешной (C) и (D) неудавшийся эксперимент. NTC = нет шаблона элемента управления. Части этой фигуры были воспроизведены из ссылка 7 с разрешения Королевского общества химии. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
После того, как ДНК и РНК/cDNA гибриды были разделены, и остальная часть геля seq протокола завершена, можно создавать последовательности библиотек. Чтобы проверить подготовленные библиотек, мы запускаем либо стандартный гель электрофорез эксперимент (Рисунок 4) или bioanalyzer. По результатам библиотек, созданных из 500 PC3 клетки и 750 клетки HeLa на рисунке 4 . На рисунке показан фрагмент распределений для соответствующих библиотек, образующиеся гель seq (с пометкой «Гель») по сравнению с непревзойденной образцы, созданные со стандартными протоколами (с пометкой «Труба»). Размер фрагмента для гель seq появляются между 200 и 800 basepairs при подготовке библиотек с помощью стандартной библиотеки всего генома подготовки комплекта. Если библиотека фрагментов не появляются в диапазоне правильный размер в этот шаг, Библиотека подготовка произошел сбой.
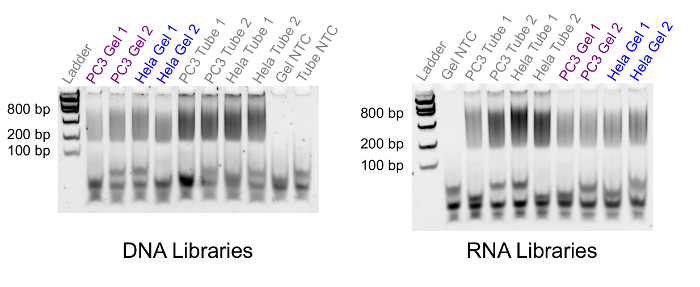
Рисунок 4 . Сравнение размера фрагмента библиотеки. Флуоресцентный гель-электрофорез изображение сравнения библиотека размер распределение между гель seq (гель) и стандартные элементы управления (труба). Левой полосе содержит низкой массы ДНК лестница с фрагмент размеров 100, 200, 400, 800, 1200 и 2000 basepairs. Размер фрагмента для всех библиотек попадают между 200 и 800 basepairs, как ожидается для библиотек, подготовленных с помощью этого комплекта. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Конечная проверка гель seq протокола на основе анализа результатов секвенирования. Мы выбрали PC3 клетки для наших экспериментов проверки, как эти клетки имеют однородные выражение профилей, которые позволяют образцов быть разделены и обрабатываются с помощью гель seq и традиционных методов; Смотрите Рисунок 5. Сравнение между геномной ДНК для PC3 клеток показана в цифры 5A и 5B. Рисунок 5 A показывает сравнение копии генома общесистемной номер вариации (CNV) профилей из PC3 с помощью гель seq или Стандартная библиотека всего генома подготовительные реакции (контроль труб). Каждая точка является среднее нормализованных бин Счетчик; контейнеры определяются из справочных данных генома, таким образом, что каждый бин имеет равные ожидаемого количества здоровых диплоидных клеток, то есть, плоская линия, представляющая равных копий для каждого региона всех аутосомно-(исключая X и Y) хромосом. PC3 содержит несколько копий тех же регионов, которые показывают вверх как пики выше фонового копирования количество два. Гель seq дает качественно подобного профиля ЦНВ стандартной трубки реакции. Соглашение между двумя участками могут быть оценены количественно линейной регрессии, как показано в группе B. Корреляции Пирсона R = 0.90 указывает, что геномных данных, собранных из либо метода функционально эквивалентны.

Рисунок 5 . Библиотека проверки. Гель seq проверки для геномной (A и B) и транскриптомики (C, D и E) данные, полученные от PC3 и НеЬа клетки. Группа A показывает сравнение генома общесистемной CNV профилей из PC3 с помощью гель seq (гель seq) или стандартной реакции (трубки). MAPD = средний абсолютный попарных разница. Группа B является линейной регрессии между двумя образцами в группе A, R = 0.90 указанием геномных данных являются функционально эквивалентными. Осей показывают, что рассчитывает нормализованные бин log2. Панели, C и D сравнение данных транскриптомики от PC3 клеток, с каждой точки показаны счетчик в стенограммы за kilobase на миллион (TPM). Осей Показать сравнение между двумя образцами как log2 нормированный Стенограмма графов. Группа C показывает сравнение технических реплицирует, созданная с помощью гель seq, и Группа D показывает, что сравнение между гель seq и традиционных РНК след группа E показывает, что гель seq может разрешить тип ячейки, основанные на РНК выражения, используя анализ главных компонентов. Ось x показывает, что первый основной компонент приходится 91,6% вариации между образцами, а ось y показывает, что второй основной компонент приходится только 6,5% дисперсии. Части этой фигуры были воспроизведены из ссылка 7 с разрешения Королевского общества химии. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Аналогичным образом мы сравнили транскриптомики данных от нашей гель seq протокола к стандартному протоколу смарт-Seq в трубку. Рисунок 5 показывает взаимосвязь между техническим реплицирует оба гель seq (рис. 5C) и гель seq и стандартный метод (рис. 5D). Каждая точка является подсчет в стенограммы за kilobase на миллион (TPM) для каждого гена, обнаруженных в TPM > 5 в обоих наборах данных. Линейной регрессии отображаются в виде красных линий, и коэффициент корреляции Пирсона отображается в левом верхнем углу. Технические реплицирует от гель seq согласен (R ∼ 0,8), но коррелирует менее хорошо с стандартным методом (R < 0,7). Это свидетельствует о том, что гель seq вводит уклоном в ген графов. К счастью эта предвзятость систематический и, как видно, анализ главных компонентов в Рисунок 5E, значимые выводы можно сделать по-прежнему между различных биологических образцов.
Обсуждение
Есть несколько критических шагов, связанных с гель seq устройства изготовления, а также сам протокол. Во время изготовления мы рекомендуем, начиная с толщины предписанные слоя для различных регионов геля. Мы потратили значительное время тестирования изготовление различных вариантов и протокол, описанные здесь производит лучшие устройства для кассет, перечисленных в таблице материалы и реагенты. Если исследователи используют систему альтернативного кассеты, они могут счесть необходимым для настройки томов, используемых при создании устройства. Главной задачей в изготовлении, что, если в регионе с высокой плотностью геля является слишком большим, он может расслаиваться из краев кассету и создавать воздушные карманы на интерьер кассеты, которые будут нарушать электрофореза. На кастинг несколько кассет с нескольких слоев различных томов, исследователи должны иметь возможность быстро определить оптимальную конфигурацию для их конкретного оборудования.
Гель seq протокол также имеет несколько важных шагов, которые могут быть проверены, прежде чем протокол является полным. Один из потенциальной точкой отказа является разделение геномная ДНК и РНК/cDNA гибридов. Это можно проверить с помощью визуализации гель seq устройства после разделения (см. Рисунок 3B). В одном наборе экспериментов, мы обнаружили, что наши лаборатории буфера быть загрязнены с ДНК и вызывает существенные autoflourescence в нашем устройстве (см. Рисунок 3D). Это было трудно определить, если разделение имело место. Флуоресцентных изображений помогли нам выявить и исправить эту проблему, перед использованием каких-либо дорогостоящих реагентов для создания последовательности библиотек.
Другой критической точки — шаг 6.2, ПЦР амплификация cDNA после разделения. Исследователи должны уделять пристальное внимание не на overamplify в этот шаг, как он уменьшит качество данных РНК seq. Это рассмотрение не является уникальной для гель seq, но является общим аспектом низкозатратных РНК seq библиотека подготовки. Амплификации PCR во время виртуализации библиотеки подготовка часто является необходимым, но он может ввести последовательность ошибок и предубеждения. Необходимое количество циклов для ПЦР зависит от количества образца и сложности. Обычно рекомендуется ограничить номер цикла PCR до минимума обязаны принести достаточно кластеризации, когда библиотеки виртуализируются. В теории протокол могут быть оптимизированы для определения точного цикла номер, который дает достаточно скопировать номер без введения чрезмерных артефактов. На практике однако, несоответствия в качестве образца, загрузки или обработки в начале протокол может значительно повлиять на распределение молекулярного шаблонов, доступных для библиотеки prep PCR, который в свою очередь влияет на оптимальный номер цикла PCR. Наиболее общее решение мы нашли следить за ходом реакций амплификации, с использованием флуоресцентных красителей, запустите реакции на реальном масштабе времени PCR Термоциклер и остановить реакции в экспоненциальное (линейная против номер цикла) фаза. По нашему опыту мониторинг в реальном времени особенно актуально при разработке, адаптации или принятия нового протокола.
Последний важный шаг создание геномных и транскриптомики библиотеки. Ключ к этот шаг должен установить Начальная концентрация образца для подготовительные реакции библиотеки ДНК как можно ближе 0,2 нг/мкл (всего 0,5 нг) как можно скорее. Это относительно простая задача для ПЦР усиленный cDNA как обычно существует избыток cDNA, но она может быть более сложным для геномная ДНК образцов. Мы нашли тщательное внимание к vacufuge шагу не требуется, в то время как образцы сконцентрированы. Как и ожидалось, в экспериментах с 1000 клеток, vacufuge шаг может быть остановлен гораздо раньше, чем эксперименты с 100 клеток. Количество образцов в vacufuge также влияет скорость испарения в наших экспериментах. Мы обнаружили, что с помощью flourometer для проверки ДНК, на полпути через концентрацию шаг может быть полезно при выполнении протокола с незнакомой образцы контента. К счастью, если воды исследователи над концентрат пробу, нуклеиназы бесплатно могут быть добавлены для разбавления образца. Теоретически можно использовать vacufuge для сухой ДНК и затем Ресуспензируйте его в желаемый объем; Однако мы рекомендуем избегать полного испарения.
Мы рассматриваем три текущие методы для генерации одновременных библиотеки ДНК и РНК, гель seq7,8G и T-seq и DR-seq9, как бесплатный. Гель seq является идеальным для образцов в диапазоне 100-1000 ячеек и не требует выпадающее цели или априорное знание генома. Два других способа лучше подходят для одной ячейки приложений. Одна из наших целей в развитии гель seq заключалась в создании протокол, который может быть легко реализован другими исследователями. Поэтому мы решили изготовить устройства в пределах стандартного форм-фактор кассеты геля полиакриламида. Хотя техника, которую мы использовали для определения наших различных оболочек () — роман, большинство лабораторий генетика уже есть все необходимое оборудование для изготовления геля seq устройства. Кроме того, стоимость устройства является тривиальным - всего $5,25 для устройства, которое может обрабатывать 12 образцов. Что сказал, как с любой библиотеки подготовка протокола с использованием коммерческих реактивов, Общая стоимость для создания библиотек остается высоким. Стоимость наших реагента на сэмпл было $50 для всего стенограмма амплификации и $28 для подготовки библиотеки ДНК и РНК. К счастью само устройство гель seq является независимым от протокола. Например во время разработки мы успешно апробировано устройство с использованием клеток от культуры и старые РНК библиотека амплификации протокол19, хотя мы обнаружили, что это не подходит для образцов тканей от мышей. Глядя в будущее, как дешевле альтернатив для приготовления Библиотека, наш протокол может быть адаптирована для работы с этими новыми методами. Мы считаем, что исследователи будут найти его несложно реализовать гель seq в собственных лабораториях. Мы надеемся, что это будет способствовать быстрому принятию технологии.
Раскрытие информации
«KZ» является соучредителем и научным советником Singlera геномики Inc.
Благодарности
Финансирование для этой работы, предусмотренных в университете Сан-Диего, национальной науки выпускник исследовательских стипендий программы Фонда, низ предоставляют R01-HG007836 и корейского министерства науки, ИКТ и будущего планирования.
Более ранние версии несколько фигур были впервые опубликованы в «Hoople, г. и др. Гель seq: всего генома и транскриптом последовательности путем одновременной низкозатратных, ДНК и РНК подготовки библиотеки с использованием полупроницаемой гидрогеля барьеров. Лаборатории на чип 17, 2619-2630, doi:10.1039 / c7lc00430c (2017).» Лаборатории на чипе санкционировало повторное использование фигур в этой публикации.
Материалы
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Acrylamide Monomer | Sigma Aldrich | A8887-100G | |
| Ammonium Persulfate | Sigma Aldrich | A3678-25G | |
| Ampure XP Beads | Beckman Coulter | A63880 | Referred to in the text as solid phase reversible immobilization (SPRI) beads |
| DNA Gel Loading Dye (6x) | ThermoFisher Scientific | R0611 | Referred to in the text as 6X loading dye |
| Ethyl alcohol | Sigma Aldrich | E7023-500ML | |
| KAPA SYBR FAST One-Step qRT-PCR Kits | Kapa BioSystems | 7959613001 | Referred to in the text as 2X qPCR mix |
| N,N′-Methylenebis(acrylamide) | Sigma Aldrich | 146072-100G | Also known as bis-acrylamide |
| NexteraXT DNA Library Preparation Kit (referred to in the text as library preparation kit) | Illumina | FC-131-1024 | Includes: TD (referred to in the text as transposase buffer), ATM (referred to in the text as transposase), NT (referred to in the text as transposase stop buffer), and NPM (Referred to in the text as library prep PCR mix) |
| Nuclease Free Water | Millipore | 3098 | |
| Protease | Qiagen | 19155 | |
| SMART-Seq v4 Kit (referred to in the text as whole-transcript amplification (WTA) kit) | Takara/Clontech | 634888 | Includes: Lysis buffer, RNase inhibitor, 3’ SMART-Seq CDS Primer II A (referred to in the text as RT primer), 5X Ultra Low First Strand Buffer (referred to in the text as first strand buffer), SMART-Seq v4 Oligonucleotide (referred to in the text as template switch oligonucleotide (TSO)), SMART-Scribe Reverse Transcriptase (referred to in the text as reverse transcriptase), and PCR Primer II A (referred to in the text as cDNA PCR primer) |
| Random hexamer with WTA adapter | IDT | n/a | 5′-AAGCAGTGGTATCAACGCAGAGTAC-NNNNNN-3′ |
| Sucrose | Sigma Aldrich | S0389-500G | |
| TEMED | Sigma Aldrich | T9281-25ML | |
| DNA AWAY Surface Decontaminant | ThermoFisher Scientific | 7010PK | Referred to in the text as DNA removal product |
| Tris-Borate-EDTA buffer (10X concentration) | Sigma Aldrich | T4415-1L | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher Scientific | S11494 | Referred to in the text as gel stain |
| Equipment | |||
| BD Precisionglide syringe needles, gauge 18 | Sigma Aldrich | Z192554 | Any equivalent hardware is acceptable |
| Branson CPX series ultrasonic bath | Sigma Aldrich | Z769363 | Any equivalent hardware is acceptable |
| Empty Gel Cassettes, mini, 1.0 mm | ThermoFisher Scientific | NC2010 | Any equivalent hardware is acceptable |
| Mesh Filter Plate - Corning HTS Transwell 96 well permeable supports - 8.0 µm pore size | Sigma Aldrich | CLS3374 | Referred to in the text as 8 um mesh filter plate |
| PowerPac HC Power Supply | Bio-Rad | 1645052 | Any equivalent hardware is acceptable |
| Qubit Fluorometer | ThermoFisher Scientific | Q33216 | Any equivalent hardware is acceptable |
| Vacufuge Concentrator | Eppendorf | 22822993 | Any equivalent hardware is acceptable |
| XCell SureLock Mini-Cell system | ThermoFisher Scientific | EI0001 | Any equivalent hardware is acceptable |
| Bio-Rad CFX96 Touch Real-Time PCR Detection System | Bio-Rad | 1855195 | Any equivalent hardware is acceptable |
| Amersham UVC 500 Ultraviolet Crosslinker | GE Healthcare Life Sciences | UVC500-115V | Discontinued, any equivalent hardware is acceptable |
| Gel Doc XR+ Gel Documentation System | Bio-Rad | 1708195 | Referred to in the text as gel imager |
| Dark Reader Transilluminator | Clare Chemical Research | DR89 | Referred to in the text as UV transilluminator |
| Ultrasonic Bath | Bransonic | 1207K35 | Any equivalent ultrasonic bath is acceptable. |
Ссылки
- Mawy, T. Single-cell sequencing. Nat Methods. 11 (1), 18(2014).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nat Biotech. 31 (12), 1126-1132 (2013).
- Sasagawa, Y., et al. Quartz-Seq: a highly reproducible and sensitive single-cell RNA-Seq reveals non-genetic gene expression heterogeneity. Genome Biol. 14 (4), 31(2013).
- Ramsköld, D., et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat Biotechnol. 30 (8), 777-782 (2012).
- Shapiro, E., Biezuner, T., Linnarsson, S. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet. 14 (9), 618-630 (2013).
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Hoople, G. D., et al. Gel-seq: whole-genome and transcriptome sequencing by simultaneous low-input DNA and RNA library preparation using semi-permeable hydrogel barriers. Lab Chip. 17, 2619-2630 (2017).
- Macaulay, I. C., et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nat Methods. 12 (6), 519-522 (2015).
- Dey, S. S., Kester, L., Spanjaard, B., Bienko, M., van Oudenaarden, A. Integrated genome and transcriptome sequencing of the same cell. Nat Biotechnol. 33 (3), 285-289 (2015).
- Gopal, A., Zhou, Z. H., Knobler, C. M., Gelbart, W. M. Visualizing large RNA molecules in solution. RNA. 18 (2), 284-299 (2012).
- SDS-PAGE Gel. Cold Spring Harb Protoc. 2015 (7), 087908(2015).
- Lake, B. B., et al. Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science. 352 (6293), at http://science.sciencemag.org/content/352/6293/1586 (2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. -Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923(2012).
- Qubit 3.0 Fluorometer Manual. MAN0010866. Life Technologies. , Available from: https://tools.thermofisher.com/content/sfs/manuals/qubit_3_fluorometer_man.pd (2017).
- Vitak, S. A., et al. Sequencing thousands of single-cell genomes with combinatorial indexing. Nat Methods. 14 (3), 302-308 (2017).
- A Guide to Polyacrylamide Gel Electrophoresis and Detection. Bulletin 6040 Rev B. Bio Rad. , Available from: http://www.bio-rad.com/webroot/web/pdf/lsr/literature/Bulletin_6040.pdf (2017).
- Illumina. Nextera ® XT DNA Library Preparation Kit. , (2017).
- Takara Bio USA Inc. SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing User Manual. , (2016).
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Saitou, M. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis. Nat Protoc. 2 (3), 739-752 (2007).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены