Method Article
DamID seq: Adenin-metillenmiş DNA Fragments Yüksek Verimli Dizileme tarafından Protein-DNA Etkileşimleri genom Haritalama
Bu Makalede
Özet
Biz yüksek verimlilik sıralama (DamID seq) DNA adenin metiltransferaz kimlik (DamID) bağlanması burada bir tahlil açıklar. Bu geliştirilmiş bir yöntem daha yüksek çözünürlüğü ve daha geniş bir dinamik aralığı ve karşılaştırın vb ChIP-SEQ RNA-SEQ gibi diğer yüksek verimli sekanslama verileri ile birlikte DamID seq verileri analiz
Özet
The DNA adenine methyltransferase identification (DamID) assay is a powerful method to detect protein-DNA interactions both locally and genome-wide. It is an alternative approach to chromatin immunoprecipitation (ChIP). An expressed fusion protein consisting of the protein of interest and the E. coli DNA adenine methyltransferase can methylate the adenine base in GATC motifs near the sites of protein-DNA interactions. Adenine-methylated DNA fragments can then be specifically amplified and detected. The original DamID assay detects the genomic locations of methylated DNA fragments by hybridization to DNA microarrays, which is limited by the availability of microarrays and the density of predetermined probes. In this paper, we report the detailed protocol of integrating high throughput DNA sequencing into DamID (DamID-seq). The large number of short reads generated from DamID-seq enables detecting and localizing protein-DNA interactions genome-wide with high precision and sensitivity. We have used the DamID-seq assay to study genome-nuclear lamina (NL) interactions in mammalian cells, and have noticed that DamID-seq provides a high resolution and a wide dynamic range in detecting genome-NL interactions. The DamID-seq approach enables probing NL associations within gene structures and allows comparing genome-NL interaction maps with other functional genomic data, such as ChIP-seq and RNA-seq.
Giriş
DNA adenin metiltransferaz tanımlaması (DamID) 1,2 in vivo protein-DNA etkileşimlerini tespit etmek için bir yöntem olup, kromatin, immüno-çökeltme (chip) 3 için alternatif bir yöntemdir. Hücrelerin nispeten düşük bir miktarda kullanılır ve DNA ya da yüksek ölçüde spesifik bir antikor, proteinin kimyasal olarak çapraz-bağlanmasını gerektirmez. Hedef protein gevşek veya dolaylı olarak DNA ile ilişkili olduğunda ikincisi özellikle yararlıdır. DamID başarılı bir şekilde çekirdek zarı proteinlerinin 4-10, kromatin ilişkili proteinler 11-13, kromatin değiştirici enzimler 14, transkripsiyon faktörleri ve 15-18 ko-faktörler ve RNAi makine 19 de dahil olmak üzere bir çok protein bağlanma sitesi eşlemek için kullanılmıştır. Yöntem S. dahil olmak üzere birden organizmalarda uygulanabilir cerevisiae 13, S. pombe 7, C. elegans 9,17, D. melanogaster 5,11,18,20, A. 21,22 yanı sıra, fare ve insan hücre hatları 6,8,10,23,24 thaliana.
DamID deneyinin geliştirilmesi endojen adenin metilasyonu 2 eksikliği ökaryotik hücrelerde adenin-metillenmiş DNA fragmanlarının spesifik olarak saptanması dayanıyordu. Ilgi ve E, DNA bağlama proteini aşağıdakilerden oluşan bir füzyon proteini, coli DNA adenin metiltransferaz (Baraj), mekansal yakınlık (en önemlisi 1 kb içinde ve yukarı yaklaşık 5 kb) olan genom 2 protein bağlanma sitelerine GAİK dizileri adenin baz metillenmesi olabilir. Tadil edilmiş DNA fragmanları, özellikle ilgi 1,25,26 proteinin genomik bağlanma bölgelerini tespit etmek için microarrays yükseltilir ve hibridize edilebilir. Bu orijinal DamID yöntemi mikrodizilerinin kullanılabilirliği ve önceden belirlenmiş prob yoğunluğu ile sınırlı kalmıştır. Bu nedenle, yüksek verimli sekanslamayı entegre etmişDamID 10 ve DamID-SEQ olarak yöntem belirlenmiş. Kısacası çok sayıda DamID-seq oluşturulan okur genom protein-DNA etkileşimleri hassas yerelleştirme sağlar. Biz DamID seq genom nükleer lamina (NL) dernekleri 10 çalışmak için mikrodizi tarafından DamID daha yüksek çözünürlük ve daha geniş bir dinamik aralık sağlanan bulundu. Bu geliştirilmiş bir yöntem gen yapıları 10 içinde NL dernek sondalama izin verir ve bu ChIP-seq ve RNA-seq gibi diğer yüksek verim sıralama verileri ile karşılaştırmalar kolaylaştırır.
Burada anlatılan DamID seq protokol başlangıçta haritalama genom NL dernekler 10 için geliştirilmiştir. Bu E. fare ya da insan Lamin B1 hayvan zinciri ile bir füzyon proteini oluşturulur coli DNA adenin metiltransferaz ve C2C12 fare (veri yayınlanmadı) 10 ve IMR90 insan fetal akciğer fibroblastlar miyoblastların, 3T3 fare embriyonik fibroblast protokolü test. Bu protokol, biz c ile başlayanvektörleri onstructing ve memeli hücrelerinde 24 lentiviral enfeksiyon Barajı-gergin füzyon proteinleri ifade. Sonra, adenin-metillenmiş DNA fragmanları amplifiye ve diğer organizmalarda uygulanabilir olmalı dizileme kitaplıkları hazırlama ayrıntılı protokolleri açıklar.
Protokol
1. Nesil ve Fusion Proteinler ve Serbest Barajı Proteinlerin İfadesi
- DamID vektörüne ilgi klon proteini.
- Üreticinin protokolüne göre (POI) protein istenen yüksek doğrulukta bir DNA polimerazı kullanılarak, uygun primerler ait cDNA yı çoğaltmak. Deneysel insertlerin düzgün amplifikasyonu sağlamak için uygun amplifikasyon koşulları belirlemek.
- Bir Agaroz ve üreticinin protokolüne uygun olarak jel ekstraksiyon kiti ile özel hedefler amplifıye cDNA arındırmak.
- Üreticinin protokolüne göre bipolar klonaz II kullanılarak pDONR201 vektörüne POI klon cDNA.
- POI füzyon istenen yönüne bağlı olarak, üreticinin protokolüne uygun olarak LR klonaz II kullanılarak PLGW-RFC1-V5-EcoDam hedef vektör veya PLGW-EcoDam-V5-RFC1 hedef vektör 27 donör vektöründen POI cDNA klonu N-ucuna ya da inciE. e C-terminali E. coli DNA adenin metiltransferaz (EcoDam) 27.
- Klonlanan cDNA'nın çerçeve füzyonu EcoDam doğru bir dizisini ve formları vardır serilendirme ile kontrol edin.
- Lentiviral stokları oluşturun.
- Lentiviral ifade sistemleri kullanılarak Barajı-V5-POI ve (PLGW-V5-EcoDam vektörü 27) V5-Dam ifade lentiviral stokları oluşturun. Üreticinin protokolüne uygun olarak ileriye transfeksiyon prosedürü kullanarak.
- Transfeksiyon için, üreticinin protokolüne uygun olarak lentiviral stoklarını oluşturmak üzere 293T hücreleri ve lipofeksiyon kullanın.
- 0.45 mikron PVDF filtre aşamasını içerir.
- Lentiviral ifade sistemleri kullanılarak Barajı-V5-POI ve (PLGW-V5-EcoDam vektörü 27) V5-Dam ifade lentiviral stokları oluşturun. Üreticinin protokolüne uygun olarak ileriye transfeksiyon prosedürü kullanarak.
- Lentivirüs hücreleri enfekte.
- Enfeksiyon (Gün 0) bir gün önce, antibiyotik olmadan aynı büyüme ortamı kullanılarak 6 oyuklu doku kültürü plakası, bu hücre tipi için uygun bir büyüme ortamı içinde kültürlendi yapışmış olan hücrelerin geçmesienfeksiyonun günde 50% konfluansta elde. 37 ° C kuluçka makinesi içinde yer hücreleri.
- Enfeksiyon (Gün 1) gününde, bir Dam-V5-POI ve V5-Baraj, hem lentiviral süpernatanların 2 cryovials kaldırmak -80 C derin dondurucuda ve yere ° çözülmeye 37 ° C su banyosunda.
- Hücrelerden büyüme ortamı çıkarın ve antibiyotik içermeyen taze büyüme ortamı 0,5 ml ile değiştirin.
- Her iyi (V5-Barajı ile 2 kuyu ve Baraj-V5-POI 2 kuyular) çözülmüş lentivirüs 1 ml ekleyin. Geri kalan 2 antibiyotikler olmadan büyüme ortamının 1 ml ilave edilir kuyu-Bu, negatif kontrol olarak hizmet edecektir. Yavaşça karıştırın ve bir 37 ° C inkübatör O / N geri yerleştirmek için 6-plaka sallayın.
- Enfeksiyon (Gün 2) gün sonra, hücrelerin viral süspansiyonlar kaldırmak ve antibiyotik olmadan 2 ml büyüme ortamı ile değiştirin. Lütfen 48 saat süreyle 37 ° C kuluçka makinesi içinde yer hücreleri.
- GDNA izole edin.
- Her iyi ve de aspire medya250 ul% 0.05 tripsin-EDTA kullanılarak taşikardi hücreleri. 2 dakika boyunca 37 ° C'de inkübe edilir.
- 1.5 ml mikrosantrifüj tüpe her kuyudan 1 ml büyüme ortamı ve pipet hücreleri ile plaka kapalı hücreleri yıkayın. Oda sıcaklığında 5 dakika boyunca 200 x g'de santrifüj hücreleri Pelet.
- Oda sıcaklığında 2 dakika boyunca 200 x g'de topaklanır 500 ul PBS ile hücreler ve santrifüj yıkayın.
- 200 ul PBS pelet hücreleri tekrar süspansiyon.
- Üreticinin protokolüne uygun olarak, kan ve doku kiti ile gDNA'sından izole edin. Elute 200 ul tampon AE gDNA ve bir spektrofotometre kullanılarak OD 260 ölçerek konsantrasyonunu belirlemek.
Not: gDNA enfekte edilmemiş veya sahte enfekte edilmiş hücreler, negatif kontroller olarak izole edilebilir. Uzun süreli depolama için yüksek konsantrasyonlara gDNA'sından çöktürün.- % 100 etanol içinde 3 hacim ve 3 M sodyum asetat (pH 5.5) 0.1 hacim ekleyin ve boruları 4-6 kez ters yüz edilerek karıştırılır.
- -20 ° CO / N saklayınız.
- 16.000 x Santrifüj4 ° C'de 15 dakika boyunca örn.
- Dikkatle süpernatant kaldırmak. 4 ° C'de 5 dakika boyunca 16,000 x g'de 70% (hacim / hacim) etanol ve santrifüj ile pelet yıkayın.
- Dikkatlice oda sıcaklığında 3 dakika boyunca hava ile kuruması için topaklar etanolün uzaklaştırılması ve izin verir.
- / Ml, yaklaşık 1 ug T 10 E 0.1 (pH 7.5) içinde gDNA'sından eritin. Her deney numune veya negatif kontrol ve konsantrasyonu ölçmek dan Havuz gDNA. -20 ° C'de saklayın.
2. Genişletme Adenin-metillenmiş DNA fragmanları
- Sadece adenin-metillenmiş GATCs kesen DpnI ile Digest gDNA.
- 2,5 ug gDNA, 1 ul 10x tampon, 0.5 ul DpnI (20 U / ul) ile buz üzerinde bir tepki kurun ve 10 ul bir toplam hacme H2O ile doldurun. Ve ikisi (w 0,5 ul H2O ile DpnI yerine, "hayır DpnI") DpnI olmadan tek - Her bir gDNA numune için üç reaksiyonları hazırlanması("DpnI ile") ith DpnI.
- Özet O / 37 ° C'de, N ve 20 dakika boyunca 80 ° C 'de DpnI etkisiz hale getirirler.
- DamID adaptörleri Arter.
- DamID adaptörleri hazırlayın.
- 100 uM nihai konsantrasyona kadar H2O iki DamID adaptörü oligos 24 her biri yeniden süspanse edin.
- İki DamID adaptörü oligo eşit hacimlerde birleştirin karıştırın ve sıkıca kapatılmış bir tüpe yerleştirin. Parafilm ile tüp Seal, 90 ° C'de su ile dolu bir beher içinde, bir raf ve bir yerde otur. Tüpün kapağı altındaki su seviyesini tutun ama oligo karışımı yüzeyi üzerinde (tüp içine alma suyu önlemek için).
- Böylece adaptörleri tavlama yavaş oda sıcaklığına kadar soğumasını bekleyin su.
- -20 ° C'de tavlanmış adaptörler (50 uM) ve mağaza alikotu.
- Buz üzerinde bir tepki ayarlayın. 2.1.2 Her tüp, 6.2 ul H2O, 2 ul 10x ligasyon tamponu, 0.8 ul 50 uM DamID reklam eklemekaptors (buz üzerinde eritildi) ve 1 ul T4 DNA ligazı (5 U / ul). Toplam hacmi 20 ul olduğunu. . Tüpleri "DpnI ile" iki birinde, 1 ul H 2 O ("hayır ligaz") ile ligaz yerine her gDNA örnek iki negatif kontroller olduğunu unutmayın - "hayır DpnI" ve "hayır ligaz".
- Arter O / 16 ° C'de, N ve 10 dakika boyunca 65 ° C 'de ligazı etkisiz hale getirirler.
- DamID adaptörleri hazırlayın.
- DpnII ile Digest DNA unmethylated GATCs ihtiva parçaları yok etmek.
- Buz üzerinde bir tepki ayarlayın. 2.2.3 Her bir tüp içinde, 24 ul H2O, 5 ul 10x DpnII tamponu ve 1 ul DpnII (10 U / ul) ilave edin. Toplam hacim 50 ml.
- 2-3 saat boyunca 37 ° C'de Digest ve 20 dakika boyunca 65 ° C 'de DpnII etkisiz hale getirirler.
- Adenin-metillenmiş DNA fragmanının amplifiye.
- 2.3.2 den, 5 u sindirmek DpnII 5 ul buz üzerinde bir tepki kurmal 10x PCR tamponu, 12.5 ul 5 mcM DamID PCR ul 10 mM dNTP mix astar 24, 4, 1 ul 50x polimeraz karışımı ve 22.5 ul H 2 O Toplam hacim 50 ml.
- Aşağıdaki gibi PCR çalıştırın: 68 ° C, 10 dakika boyunca; 1 dakika için 94 ° C, 5 dakika için 65 ° C, 15 dakika boyunca 68 ° C; 1 dakikada, 1 dakika boyunca 65 ° C, 10 dakika boyunca 68 ° C sıcaklıkta 94 ° C 4 döngü; 1 dakikada, 1 dakika, 2 dakika boyunca 68 ° C, 65 ° C, 94 ° C 17 döngü.
- Bir% 1 agaroz jeli üzerinde Her reaksiyondan 5 ul PCR ürünleri analiz edin. PCR ürünleri, 0.2 ila 2 kb (Şekil 2) arasında değişen bir yayma olarak görünmelidir. "Hiçbir DpnI" ve "hayır ligaz" kontroller ya da açıkça daha az amplifikasyon gerekir.
- Aşama 2.4.3 elde edilen sonuç tatmin edici ise, deney numunesi için iki ya da üç reaksiyonlar ve iki negatif kontrollerin her biri için tek bir reaksiyon ile adımları 2.4.1-2.4.3 tekrarlayın.
- Havuz aynı PCR ürünlerini arındırmak veüreticinin protokolüne uygun olarak PCR saflaştırma kitleri ya da katı faz immobilizasyon Döner (SPRI) boncuklar kullanılarak, deneysel Örnek. "Hiçbir DpnI" ya da "hayır ligaz" denetimleri arındırmak etmeyin. Tampon EB ile DNA Zehir.
- Etrafında 0.1 ug / ul veya daha yüksek olmalıdır spektrofotometre kullanılarak OD 260 ölçerek saflaştırılmış DNA konsantrasyonu ölçün. Her bir numune için 10 ug DNA, en az toplayın. PCR saflaştırma kitleri kullanılarak ise, bir sütun her 50 ul PCR ürünleri arındırmak 30 ul tampon EB Zehir ve eluatları havuz.
- DpnII ile Digest DNA dizilenmiş edilmesini DamID PCR primerlerini bilmek.
- Bir 2.4.6 5 ug saflaştırılmış DNA ile buz üzerinde tepkime 5 ul 10x DpnII tamponu, 1 ul DpnII (10 U / ul) ayarlamak ve 50 ul bir toplam hacme H2O ile doldurun. Her numune için iki ya da üç reaksiyonları hazırlayın.
- 2-3 saat boyunca 37 ° C'de Digestve 20 dakika boyunca 65 ° C 'de DpnII etkisiz hale getirirler.
- Havuz üreticinin protokolüne uygun olarak PCR saflaştırma kiti ya SPRI boncuklar ile aynı örnekten digests arındırmak ve. Tampon EB ile DNA Zehir.
- Etrafında 0.06 ug / ul veya daha yüksek olmalıdır saflaştırılmış DNA konsantrasyonu ölçün. Her numune için 6 ug DNA, en az toplayın. PCR saflaştırma kitleri kullanılarak ise 30 ul tampon EB Zehir, her 50 ul bir sütun ile sindirmek arındırmak ve eluatları havuz.
Yüksek verim Dizileme için 3. kütüphane hazırlanması
- Parçası DNA
- Deneysel dsDNA Fragmentase her yeni parti için uygun sindirim süresini belirler. Enzim aktivitesi zamanla azalabilir gibi, yeni bir deneme yapmadan önce testi tekrarlayın. Gerçek parçalanma 2.5.4 DNA kaydetmek için, önceki experime dan 2.4.6 veya ekstra DNA saflaştırılmış metil PCR ürünleri kullanmayınBu aşamada NTS.
- 6 ug DNA ile ana karışımı, 12 ul 10x Fragmentase tampon kurun ve 114 ul toplam hacmine H2O ile doldurun.
- Vortex 3 sn Fragmentase stok flakon, usta karışımı içine 6 ul ekleyin ve 3 saniye boyunca ana karışımı vorteks. Toplam hacmi 120 ul.
- Kısım 5, yeni tüplerin her ana karışımı 20 ul. 10 dakikalık bir artışında 5 dakika 55 için 37 ° C sıcaklıkta 6 inkübe edin. Reaksiyonu durdurmak üzere 5 ul 0.5 M EDTA ekleyin.
- 12,5 ul her bir reaksiyonun (0.5 ug DNA) ve bir agaroz jeli üzerinde 0,5 ug sindirilmemiş DNA (Şekil 3) sindirmek analiz edin. Etrafında 0,2 kb smear çoğunluğunu sindirmek için gerekli olan asgari süre (T 0.2kb) belirleyin. Gerçek parçalanma eşit artışlarla (5 dakika ve T 0.2kb dahil) 5 dakika ile T 0.2kb arasında 6 zaman süreleri seçin.
- Gerçek fragmen kurmakyon olarak 3.1.1.1-3.1.1.3 tarif edilmiştir. 3.1.1.4 belirlenen zaman süreleri için, 37 ° C 'de reaksiyonlar inkübe edin.
- Havuz 6 reaksiyonları ve PCR saflaştırma kitleri veya üreticinin protokolüne göre SPRI boncuklar ile digests arındırmak. 51 ul tampon EB DNA Zehir. Nihai yıkama sıvısı, ~ 50 ul.
- Deneysel dsDNA Fragmentase her yeni parti için uygun sindirim süresini belirler. Enzim aktivitesi zamanla azalabilir gibi, yeni bir deneme yapmadan önce testi tekrarlayın. Gerçek parçalanma 2.5.4 DNA kaydetmek için, önceki experime dan 2.4.6 veya ekstra DNA saflaştırılmış metil PCR ürünleri kullanmayınBu aşamada NTS.
- Onarım parçalanmış DNA biter
- 3.1.3 ayrılan malzeme, 25 ul H ile buz üzerinde bir kimyasal reaksiyon ayarlama 2, O, 10 ul 10 mM ATP 10x T4 DNA ligaz tampon maddesi, 4 ul 10 mM dNTP karışımı, 5 ul T4 DNA polimeraz (3 U / ul) , 1 ul Klenow DNA polimerazı (5 U / ul) ve 5 ul T4 polinükleotid kinaz olabilir. Toplam hacim 100 ul olduğunu. Pipet ile iyice karıştırın. Köpük ve kabarcıkları kaçının.
- 30 dakika boyunca 20 ° C'de inkübe edin.
- PCR saflaştırma kitleri veya üreticinin protokolüne göre SPRI boncuklar ile DNA saflaştırılır. 33 ul tampon EB DNA Zehir. Nihai Elüat ~ 32 & # ise181, l.
- "A" çıkıntılar ekle
- Bir 3.2.3 ayrılan malzeme ile buz üzerinde tepkime 5 ul 10x Klenow tampon maddesi, 10 ul 1 mM dNTP ve 3 ul Klenow (3 '→ 5' ekso-) (5 U / ul) ayarlayın. Toplam hacim 50 ml. Pipet ile iyice karıştırın. Köpük ve kabarcıkları kaçının.
- 30 dakika boyunca 37 ° C'de inkübe edilir.
- PCR saflaştırma kitleri veya üreticinin protokolüne göre SPRI boncuklar ile DNA saflaştırılır. 22 ul tampon EB DNA Zehir. Nihai yıkama sıvısı, ~ 21 ul.
- Ligatı sıralama adaptörleri
- Sıralama adaptörleri 28 hazırlayın.
- 10 mM Tris-Cl (pH 7.8), 0.1 mM EDTA (pH 8.0) ve 50 mM NaCl 100 uM'lik bir konsantrasyonda, her bağdaştırıcı oligo yeniden süspanse edin.
- Evrensel adaptör 28 ve dizinlenmiş adaptörün 28 eşit hacimlerde karıştırın.
- Aşağıdaki ile bir termal döngü içinde adaptörleri tavlamaProgram: 2 dakika boyunca 95 ° C; 30 saniye 95 ° C'de başlayan ve 0.5 ° C, her döngü tarafından azaltılması için 140 döngü; 4 ° C'de tutun.
- Kısım adaptörleri ve mağaza -20 ° C'de.
- 3.4.1.4 ve 2.5 | il T4 DNA ligaz ile ilgili (buz üzerinde eritildi) 3.3.3 ayrılan malzeme ile buz üzerinde bir reaksiyon, 25 ul 2x ligasyon tamponu, sıralama adaptörleri tavlanmış 1.5 ul 50 uM kadar ayarlayın. Pipet ile iyice karıştırın. Köpük ve kabarcıkları kaçının. Mültipleks dizme isteniyorsa, her bir örnek için, farklı bir endeksli adaptör.
- 1 saat boyunca oda sıcaklığında inkübe edin.
- PCR saflaştırma kitleri veya üreticinin protokolüne göre SPRI boncuklar ile DNA saflaştırılır. 24 ul tampon EB DNA Zehir. Nihai yıkama sıvısı, ~ 23 ul.
- Sıralama adaptörleri 28 hazırlayın.
- DNA fragmanı 28 boyutları belirlenmesi doğru etkinleştirmek için dsDNA'nın Y-şekilli adaptörleri dönüştürme
- 3.4.4 ayrılan malzeme, 12,5 ul H2O ile buz üzerinde bir kimyasal reaksiyon ayarlama, 1 ul 25 mM oncul 1 28, 1 ul 25 mM oncul 2 28, 1.5 ul 10 mM dNTP, 10 ul 5x PCR Tampon, 1 ul DNA polimeraz (1 U / ul).
- Aşağıdaki gibi PCR çalıştırın: 95 ° C, 3 dakika boyunca; 15 saniye, 30 saniye için 63 ° C, 30 sn için 72 ° C sıcaklıkta 98 ° C 5 döngü; 1 dakika için 72 ° C; Beklemeye 4 ° C.
- PCR saflaştırma kitleri veya üreticinin protokolüne göre SPRI boncuklar ile DNA saflaştırılır. 11 ul tampon EB DNA Zehir. Nihai yıkama sıvısı, ~ 10 ul.
- Boyut kitaplığı seçin
- 1x TAE tamponu ile,% 2 agaroz jel hazırlayın. Etidyum bromid (Dikkat gösterilmelidir!) Ekleyin 500 ng / ml'lik nihai bir konsantrasyona kadar erimiş TAE-agaroz çözelti, etidiyum bromür solunması soğuduğunda. Tüm örneklerde, DNA merdiveni ve boş şeritleri için yeterli şerit olduğundan emin olun.
- Bölüm 3.5.3'te ayrılan malzeme 8 ul 6x yükleme boya ekleyin.
- 1 kb artı 1 bir oranda bir DNA merdiveni (1,0 ug / ul), 6x yükleme boyası ve H2O karıştırılarak, DNA merdiveni hazırlayın: 1: 4.
- DNA merdiveni ul yükleme 6, ayrı bir şeritte ve bitişik örnek / merdivenler en az bir boş şeritli 3.6.2 örnekler ve başka bir 6 ul DNA merdiveni, her biri.
- 60 dakika boyunca 120 V jel çalıştırın.
- UV transilluminator üzerine jel görüntüle (UV maruz kalma süresini en aza indirmek). Koruyucu gözlük ve yüz maskesi kullanılmalıdır. Emin DNA merdivenleri en az biri 300 bp ve 400 bp bantları arasında (3 jel dilimleri eksize için yeterli) uygun aralıkla iyi çalışmasını sağlamak. Geniş aralık eksize jel dilimlerinin hacmini artırırken dar aralık, birden jel dilim eksize zorluklar artar.
- Yeni neşter veya her kulvar için jilet kullanın. Tüketim 300 ve her kulvarın 400 bp arasında üç ince jel dilimleri (Şekil 4) ve bir mikrosantrifüj tüp bunları her koyun. Her jel s hacmini tutunmümkün (<100 ul) gibi düşük bitler.
- Her bir jel dilimi (1 ul jel, yaklaşık 1 mg ağırlığındadır) hacminin ölçün QG tampon 6x birimler eklemek ve 50 ° C'de inkübe edin.
- Jel dilimi tamamen çözülene kadar QG-jel karışımı, her 2-3 dakikada vorteksleyin. Izopropanol 2x jel hacimleri ilave et ve karıştır.
- Bu adımdaki, jel ekstraksiyon kitleri protokolü izleyin. 51 ul tampon EB DNA Zehir. Nihai yıkama sıvısı, ~ 50 ul.
- Dizileme adaptörü tadil edilmiş DNA fragmanları zenginleştirmek
- PCR döngüleri 29 sayısını optimize edin.
- 3.6.10 1 ul sıyırma sıvısı, 1 ul 25 mM oncul 1 28, 1 ul 25 mM oncul 2 28, 7 ul H2O ve 10 ul SYBR Green Supermix ile buz üzerinde bir kimyasal reaksiyon ayarlayın. Toplam hacmi 20 ul olduğunu.
- Şöyle qPCR çalıştırın: 95 ° C, 3 dakika boyunca; 30 saniye için 95 ° C 20 döngü, 30 saniye için 63 ° C, 730 saniye için 2 ° C, levha okuyun.
- Bir qPCR analiz yazılımı kullanılarak verileri analiz ve üreticinin protokolünü kullanarak her bir numune için Kantitasyonu Döngüsü (Cq) veya Eşik Döngüsü (Ct) belirler. Nihai PCR döngüsü sayısı (N PCR) olarak (bir sonraki yüksek tam sayıya yuvarlanır) Cq / Ct ≤ 14. kullanın sahip numuneler maksimum Cq (Cq 0) eksi 1 ile devam edin.
- Farklı örnekler PCR döngüleri aynı sayıda çalıştırdıktan sonra yaklaşık olarak eşit miktarlarda amplifiye edilecek ve böylece DNA şablonları miktarlarda ayarlayın. En yüksek Cq (Cq 0) ile numune için 8 ul şablonu kullanın ve aşağıdaki formüle sahip diğer örneklerin şablon hacmini hesaplamak:
Vol i = 8 x 1.8 Cq i 0 -Cq
- 1 ul 25 mM astar 1 28 1 ul 25 mM astar 2 28 1.5: 3.7.1.4 hesaplanan şablon hacimleri ile buz üzerinde PCR reaksiyonları kurmak| il 10 mM dNTP, 10 ul 5x tamponu, 1 ul DNA polimeraz (1 U / | il) ve 50 ul bir toplam hacme H2O ile doldurun.
- Aşağıdaki gibi PCR çalıştırın: 95 ° C 45 saniye boyunca; N PCR döngüsü 15 saniye, 30 saniye için 63 ° C, 30 sn için 72 ° C sıcaklıkta 98 ° C (3.7.1.3 belirlenen); 1 dakika için 72 ° C; Beklemeye 4 ° C.
- % 2 agaroz jel (Şekil 5A) içinde 5 ul PCR ürünleri analiz edin. Açık bir "tek bir" bant Çoğaltılan DNA fragmanları, dar bir uzunluk aralığında olan ve DNA kütüphanesi daha analize tabi olabilir olduğuna işaret etmektedir.
- Seçilen örnekler için tek reaksiyonu tekrarlayın ve aynı örnekten büyütülmüş DNA kütüphaneleri havuz.
- Dizileme için seçilen DNA kütüphanelerinin arıtın.
- Astar / adaptör dimerleri 3.7.4 görünür değilse, PCR saflaştırma kitleri veya üreticinin protokolüne göre SPRI boncuklar ile DNA kütüphanelerinin arındırmak. 21 ul tampon EB DNA Zehir.Nihai yıkama sıvısı, ~ 20 ul. Kullanıcının sıralama tesisi elüsyon tamponu, son hacim, vb özel talimatlar varsa, buna uygun numuneleri hazırlamak.
- Astar / adaptör dimerleri 3.7.4 açıkça görülebilir iseniz aşağıdaki gibi, kütüphaneler arındırmak.
- % 2 agaroz jeli içinde prekast 3.7.5 Yük DNA ve DNA merdiveni. Hacmini azaltmak 3.7.6.1 tarif edilen veya birden fazla kuyu içine yüklenebilir olarak Numune saflaştırılabilir. Uygun bir güç sisteminde agaroz jel yerleştirin. Jel 15 dakika çalışmasına izin verin.
- Uygun bir transilluminator'de kullanarak, her bir örnek için temiz bir neşter / ustura ile istenilen bant kesip ve 1.5 ml mikrosantrifüj tüp jel dilim yerleştirin.
- Iki değişiklik, üreticinin protokolüne uygun olarak jel özütleme kitleri kullanılarak DNA izole: 37 ° C 'de, bir termo-mikser içinde tampon QG jel karışımı inkübe edildi ve 30 dakika için 1000 rpm ve izopropanol 2x jel hacimleri ekleyin.
- PCR döngüleri 29 sayısını optimize edin.
- . Bir sıralama tesisine saflaştırılmış DNA kütüphanelerinin gönderin tüm tesis yönergeleri izleyin.
Not: Bioanalyzer (Şekil 5B) tarafından yapılan bir kalitesi analizi kesin boyut aralığı ve bir DNA kütüphanesinde konsantrasyonunu belirlemek amacıyla, önceki sekanslama yapılmalıdır.
Sonuçlar
Baraj V5-LmnB1 füzyon proteini, immünofloresan boyama endojen lamin B proteini (Şekil 1) ile birlikte lokalize olduğu teyid edildi.
Adenin-metillenmiş DNA fragmanlarının PCR amplifikasyonu, başarılı DamID-SEQ için önemli bir adımdır. No-veya açıkça daha az büyütme (Şekil 2) yol açmalıdır (ligaz olmadan ya da PCR şablon olmadan, DpnI olmadan) negatif kontrol ederken 2 kb - deney örnekleri 0.2 bir karalama yükseltmek gerekir.
Bir NGS kütüphane için arzu edilen uç boyutu 200 ila 300 bp ise metillenmiş DNA fragmanları, 0.2 2 kb arasında aralığındadır. Bu nedenle, uygun bir boyut aralığına metil PCR ürünleri parçalara esastır. Bununla birlikte, aynı anda bir uygun boyutta aşağı daha geniş DNA fragmanlarını kırmak için pratik olduğu bulunmuşturs ve tek bir parçalanma süresi sağlam küçük DNA parçalarının çoğunluğu tutun. Bu nedenle, zaman süreçli deneyler, 200 bp (Şekil 3) merkezlenen bir lekelenmesine parçası 1 ug DNA için gereken minimum süre (T 0.2kb) belirlemek için yapıldı. Sonra eşit artışlarla 6 sürelerde gerçek parçalanma 5 dakika ile T 0.2kb arasında seçildi. Çift iplikli DNA'nın Fragmentase enzimatik aktivitesi, partiden partiye değişebilir ve zamanla azalabilir, bu nedenle Fragmentase yeni bir parti ya da belirli bir süre depolandıktan sonra bu adımı tekrar tavsiye edilir.
İstenen uç boyutlu agaroz jeli üzerinde 300 ve (121 bp dizi adaptörleri de dahil olmak üzere), 400 bp arasında, DNA fragmanlarına karşılık gelen 200 ve 300 bp. Bu aralıkta üç ince dilim bir kütüphane boyutu aralığını daraltmak ve olasılığını artırmak için her deney örnek kesilmiştirEn az bir nitelikli sıralama kitaplığı (Şekil 4) elde.
Her bir amplifiye DNA kütüphanesinin 5 ul'lik bir tümböleni sekanslama için uygun olabilir, ki kütüphane belirlemek için agaroz jeli üzerinde analiz edilmiştir. Şekil 5A'da gösterildiği gibi, kesilmiş jel dilimine aynı boyutta açık bir tek bant agaroz jel (aşama 3.7.4) görünür olmalıdır. Daha sonra, seçilen kütüphaneleri dizileme önce kesin boyut aralığı ve konsantrasyonlarını belirlemek için bir Bioanalyzer (Şekil 5B) ile incelenmiştir. Arzu edildiği takdirde, amplifiye DNA kütüphaneleri ile doğrudan jel analizi olmaksızın Bioanalyzer ile incelenebilir. Birden kütüphaneler kaliteli olduğunda, benzer büyüklükteki kütüphaneleri örnekleri (V5-Baraj ifade eden hücreler) deneysel (Baraj-V5-POI salgılayan hücrelerin) ve kontrol bir çift aralıkları sıralamak için tavsiye edilir.
Kısacası tarafından oluşturulan okursıralama sistemleri ilk olarak geri gelen genom haritası çizilmiştir bulundu. Benzersiz daha sonraki analizlere geçirildi okur hizalanmış. Kısa işlemek için bir boru hattı, bir genom-NL etkileşim haritası oluşturmak ve gen-NL dernekler önceki çalışmaları 10 ayrıntılı olarak tarif edilmiştir analiz okur. Örnek sonuçlar Şekil 6'da gösterilmektedir.

Immünofloresan boyama ile füzyon proteinlerinin çekirdek içi lokalizasyonu doğrulama Şekil 1.. IMR90, insan akciğer fibroblast hücreleri, geçici olarak Dam-V5-LmnB1 eksprese eden bir plazmid ile transfekte edildi ve anti-lamin B (A, kırmızı) ve anti-V5 (B ile boyandı yeşil). (C) A ve B görüntüleri birleştirme t büyük halini görmek için tıklayınızOnun rakam.

Adenin-metillenmiş DNA fragmanları amplifiye Şekil 2. PCR. Her bir PCR reaksiyonundan kaynaklanan, 5 ul bir alikosu, bir% 1 agaroz jeli üzerinde analiz edilmiştir. 0.2 2 kb arasında değişen bir smear her deney numuneden amplifiye edildi, ancak hiçbir amplifikasyon negatif kontroller gözlendi (adım 2.1 hiçbir DpnI, adım 2.2 veya hiçbir PCR şablonunda hiçbir Ligaz). Tıklayınız daha büyük bir versiyonunu görmek için bu rakamın.
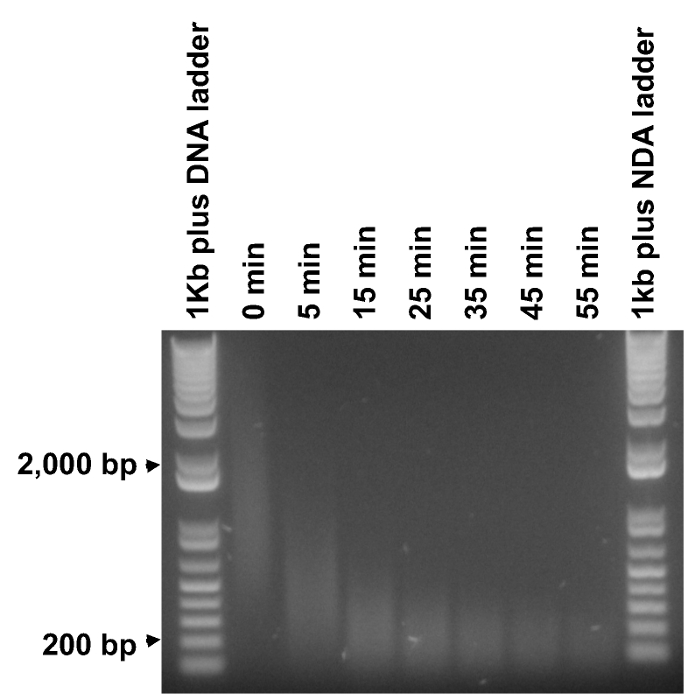
Şekil 3. Optimal parçalanma süreleri belirlenmesi. Arıtılmış metil PCR ürünleri 10 dakikalık bir artışında 5 ila 55 dakika zaman süreleri için parçalanma tabi tutulmuştur (sindirilmemiş DNA "0 dak") olarak etiketlenmiş. 1 DNA merdiveni ug ve her bir parçalanmış DNA örneği 0.5 ug bir agaroz jeli üzerinde analiz edilmiştir. Yaklaşık 200 bp DNA smear çoğunluğu sindirmek için çok az zaman 5 ile 35 dakika (5 ve 35 dk dahil) arasında altı eşit aralıklı sürelerde gerçek parçalanma gerçekleştirmek için seçildi nedenle yaklaşık 35 dakika, olduğu belirlendi. Lütfen Bu rakamın büyük halini görmek için buraya tıklayın.

Şekil 4. Boyut adımı 3.6% 2 agaroz jel üzerinde yürütülmüştür dan. Baraj-V5-LmnB1 ve V5-Baraj DNA örnekleri DNA kütüphanelerinin seçme ve üç eşit büyüklükteki jel dilimleri (L, M ve H düşüğe gelen orta sarı çizgilerle gösterildiği gibi) boyutu yüksek 300 ve 400 bp arasında çıkarılmıştır. om / files / ftp_upload / 53620 / 53620fig4large.jpg "target =" _ blank "> bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
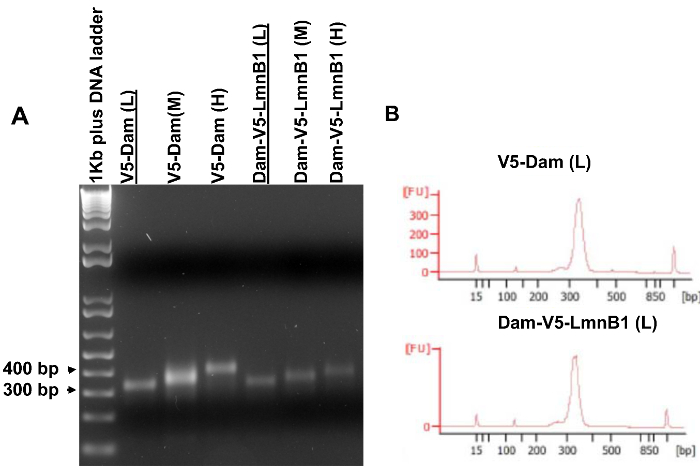
Bir agaroz jeli üzerinde analiz Şekil yüksek sekanslama 5. Kuvvetlendirilmiş DNA kütüphaneleri (A). Amplifiye DNA kütüphaneleri. PCR şablonları Şekil 4 üzerinde gösterilen jel dilimlerinden saflaştırılmıştır. (B) V5-Baraj (L) numune ve (A) 'de vurgulanmıştır Dam-V5-LmnB1 (L) numunenin Bioanalyzer sonuçlanır. Bu iki kütüphaneler benzer dar boyut aralıkları vardı ve yüksek verimlilik sıralaması dolayısıyla nitelikli. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
jpg "/>
UCSC genom tarayıcısı. Fare kromozom 1 görüntülenen Şekil 6. NGS veri örnek olarak gösterilmektedir. Dizi verileri, fare C2C12 miyoblastların 10 elde edildi. Sırasıyla Barajı-V5-LmnB1 ve V5-Barajı'nı salgılayan hücrelerin verilerine karşılık gelen İzleri "MB.LmnB1.w2k" ve "MB.Dam.w2k", arsa normalize okuma yoğunlukları (milyon benzersiz okur, ya da RPKM eşlenen başına kilobaz başına okur ) kromozom boyunca örtüşmeyen ardışık 2 kb pencerelerde. Parça "MB.log2FC.w2k" araziler genom NL dernekler, yani log2 2 kb çözünürlükte Barajı üzerinde LmnB1 ve RPKM oranları. Sıralama tabanlı Lamina İlişkili Alanlar (sLADs, istatistiksel öneme sahip Barajı üzerinde LmnB1 yüksek okuma yoğunlukları var yani genomik bölgeleri) beyaz, gri ve belirsiz bölgelerde siyah, sigara sLADs boyar "MB.sLADs" izleyin. Için tıklayınız Yazılımı yükleBu dosyayı d.
Tartışmalar
Whether Dam-tagged proteins retain the functions of endogenous proteins should be examined before a DamID-seq experiment. The subcellular localization of Dam-tagged nuclear envelope proteins should always be determined and compared with that of the endogenous proteins. For studying transcription factors, it is suggested to examine whether the Dam-fusion protein can rescue the functions of the endogenous protein in regulating gene expression. This functional test can be performed in organisms in which knockout mutants of endogenous DNA-binding proteins are available. Because advances in genome engineering have potentially allowed knocking out any endogenous gene of interest, functions of Dam-tagged DNA-binding proteins can be examined in cultured mammalian cells.
The critical step in this protocol is to successfully fragment the DpnII-digested DamID PCR products to around 200 bp. This step is designed to render the amplified adenine-methylated fragments to a narrow size range for sequencing and to randomize the starting nucleotides of the DNA fragments in a sequencing library. Inefficient fragmentation will leave the majority of the DNA fragments starting with GATC (the 5'-overhang from the second DpnII digestion), and will result in a much lower performance and yield or even a failure in Illumina sequencing. Other DNA fragmentation methods may be used as an alternative approach.
The resolution of DamID (and DamID-seq described here) is limited by the frequency of GATCs in the genome to be studied. Moreover, even with high throughput sequencing, the genomic localizations of a DNA-binding protein can only be mapped within two consecutive GATCs rather than to the actual DNA-binding sites.
Despite its limitation, the DamID assay has important advantages. Because DamID does not require highly-specific antibodies, it can be used to detect a subset of nuclear proteins that could be difficult to assay by ChIP (such as the nuclear envelope proteins). To study how these proteins regulate genome functions, it is important to integrate and cross-analyze their genome-wide localization data with the current epigenomic mapping data (such as data from the ENCODE and NIH Roadmap Epigenomics Projects 30,31). The DamID-seq approach provides both higher resolution and higher sensitivity than DamID by microarray and enables detecting differential NL-associations within gene structures 10. A combinatorial analysis of DamID-seq data, ChIP-seq data 32 and gene expression data has identified a class of NL-associated genes with distinct epigenetic and transcriptional features (data not published).
Another advantage of DamID is that it only requires a small number of cells. In recent years, there has been an explosion in single cell analysis of gene regulation 33,34. Although genome sequence 35, genome-wide gene expression 36 and chromatin conformation 37 can be assayed in a single cell, there has not been an available approach for detecting protein-DNA interactions genome-wide in a single cell. DamID-seq is a highly promising approach for this goal, and may complement the single cell imaging approach in detecting the dynamics of genome-NL interactions 38. One complication is that because the Dam-fusion protein is expressed at a much lower level than the endogenous protein in the DamID assay, it is possible that the Dam-fusion protein may only occupy a subset of genomic binding sites as compared to the endogenous protein.
DamID assay has mostly been used in cultured animal cells to detect protein-DNA interactions. Notably, developmental biologists have applied this assay in detecting protein-DNA interactions in specific cell types in vivo. For example, Dam-tagged RNA polymerase II was expressed specifically in Drosophila neural stem cells to detect their genome-wide occupancy without cell isolation 39. DamID-seq will be highly useful to study the genome-wide localizations of nuclear envelope proteins, transcription factors and chromatin regulators during development in animal models.
Açıklamalar
The authors have nothing to disclose.
Teşekkürler
We thank Dr. Bas van Steensel for providing the DamID mammalian expression vectors. We thank Yale Center for Genome Analysis and the Genomics Core in Yale Stem Cell Center for advice on preparing NGS libraries and implementing high throughput DNA sequencing. This work was supported by the startup funding from Yale School of Medicine, a Scientist Development Grant from American Heart Association (12SDG11630031) and a Seed Grant from Connecticut Innovations, Inc. (13-SCA-YALE-15).
Malzemeler
| Name | Company | Catalog Number | Comments |
| ViraPower Lentiviral Expression Systems | Life Technologies | K4950-00, K4960-00, K4970-00, K4975-00, K4980-00, K4985-00, K4990-00, K367-20, K370-20, and K371-20 | |
| Gateway BP Clonase II Enzyme Mix | Life Technologies | 11789-020 | |
| Gateway LR Clonase II Enzyme Mix | Life Technologies | 11791-020 | |
| DNeasy Blood & Tissue Kit (250) | QIAGEN | 69506 or 69504 | |
| Gateway pDONR 201 | Life Technologies | 11798-014 | |
| 293T cells | American Type Culture Collection | CRL-11268 | |
| Trypsin-EDTA (0.05%), phenol red | Life Technologies | 25300-054 | |
| DMEM, high glucose, pyruvate | Life Technologies | 11995-065 | |
| Fetal Bovine Serum | Sigma | F4135 | |
| Tris | brand not critical | ||
| EDTA | brand not critical | ||
| 200 Proof EtOH | brand not critical | ||
| Isopropanol | brand not critical | ||
| Sodium Acetate | brand not critical | ||
| DpnI | New England Biolabs | R0176 | supplied with buffer |
| DamID adaptors "AdRt" and "AdRb" | Integrated DNA Technologies | sequences available in ref. 24; no phosphorylation of the 5' or 3' end to prevent self-ligation. | |
| T4 DNA Ligase | Roche Life Science | 10481220001 | supplied with buffer |
| DpnII | New England Biolabs | R0543 | supplied with buffer |
| DamID PCR primer "AdR_PCR" | Integrated DNA Technologies | sequences available in ref. 24 | |
| Deoxynucleotide (dNTP) Solution Set | New England Biolabs | N0446 | 100 mM each of dATP, dCTP, dGTP and dTTP |
| Advantage 2 Polymerase Mix | Clontech | 639201 | supplied with buffer |
| 1Kb Plus DNA Ladder | Life Technologies | 10787018 | 1.0 µg/µl |
| QIAquick PCR Purification Kit | QIAGEN | 28104 or 28106 | |
| MinElute PCR Purification Kit | QIAGEN | 28004 or 28006 | for an elution volume of less than 30 µl |
| SPRI beads / Agencourt AMPure XP | Beckman Coulter | A63880 | apply extra mixing and more elution time if less than 40 µl elution buffer is used |
| Buffer EB | QIAGEN | 19086 | |
| NEBNext dsDNA Fragmentase | New England Biolabs | M0348 | supplied with buffer |
| T4 DNA Ligase Reaction Buffer | New England Biolabs | B0202 | |
| T4 DNA Polymerase | New England Biolabs | M0203 | |
| DNA Polymerase I, Large (Klenow) Fragment | New England Biolabs | M0210 | |
| T4 Polynuleotide Kinase | New England Biolabs | M0201 | |
| Klenow Fragment (3’ → 5’ exo-) | New England Biolabs | M0212 | supplied with buffer |
| sequencing adaptors | Integrated DNA Technologies | sequences available in ref. 28 | |
| Quick Ligation Kit | New England Biolabs | M2200 | used in 3.4.3; supplied with Quick Ligation Reaction Buffer and Quick T4 DNA Ligase |
| sequencing primer 1 and 2 | Integrated DNA Technologies | sequences available in ref. 28 | |
| KAPA HiFi PCR Kit | Kapa Biosystems | KK2101 or KK2102 | supplied with KAPA HiFi DNA Polymerase, 5x KAPA HiFi Fidelity Buffer and 10 mM dNTP mix |
| agarose | Sigma Aldrich | A4679 | |
| ethidium bromide | Sigma Aldrich | E1510-10ML | 10 mg/ml |
| QIAquick Gel Extraction Kit | QIAGEN | 28704 or 28706 | |
| iTaq Universal SYBR Green Supermix | Bio-Rad Laboratories | 1725121 or 1725122 | |
| Spectrophotometer | brand not critical | ||
| 0.45 μm PVDF Filter | brand not critical | ||
| 25 ml Seringe | brand not critical | ||
| 10 cm Tissue Culture Plates | brand not critical | ||
| 6-well Tissue Culture Plates | brand not critical | ||
| S1000 Thermal Cycler | Bio-Rad Laboratories | ||
| C1000 Touch Thermal Cycler | Bio-Rad Laboratories | for qPCR | |
| Vortex Mixer | brand not critical | ||
| Dry Block Heater or Thermomixer | brand not critical | ||
| Microcentrifuge | brand not critical | ||
| Gel electrophoresis system with power supply | brand not critical | ||
| Magnet stand | for purification of DNA with SPRI beads; should hold 1.5-2 ml tubes; brand not critical | ||
| UV transilluminator | brand not critical | ||
| E-gel electrophoresis system | Life Technologies | G6400, G6500, G6512ST |
Referanslar
- van Steensel, B., Delrow, J., Henikoff, S. Chromatin profiling using targeted DNA adenine methyltransferase. Nat Genet. 27, 304-308 (2001).
- van Steensel, B., Henikoff, S. Identification of in vivo DNA targets of chromatin proteins using tethered dam methyltransferase. Nat Biotechnol. 18, 424-428 (2000).
- Fu, A. Q., Adryan, B. Scoring overlapping and adjacent signals from genome-wide ChIP and DamID assays. Mol Biosyst. 5, 1429-1438 (2009).
- Guelen, L. Domain organization of human chromosomes revealed by mapping of nuclear lamina interactions. Nature. 453, 948-951 (2008).
- Kalverda, B., Pickersgill, H., Shloma, V. V., Fornerod, M. Nucleoporins directly stimulate expression of developmental and cell-cycle genes inside the nucleoplasm. Cell. 140, 360-371 (2010).
- Kubben, N. Mapping of lamin A- and progerin-interacting genome regions. Chromosoma. 121, 447-464 (2012).
- Steglich, B., Filion, G. J., van Steensel, B., Ekwall, K. The inner nuclear membrane proteins Man1 and Ima1 link to two different types of chromatin at the nuclear periphery in S. pombe. Nucleus. 3, 77-87 (2012).
- Harr, J. C. Directed targeting of chromatin to the nuclear lamina is mediated by chromatin state and A-type lamins. J Cell Biol. 208, 33-52 (2015).
- Gonzalez-Aguilera, C. Genome-wide analysis links emerin to neuromuscular junction activity in Caenorhabditis elegans. Genome Biol. 15, R21(2014).
- Wu, F., Yao, J. Spatial compartmentalization at the nuclear periphery characterized by genome-wide mapping. BMC Genomics. 14, 591(2013).
- Filion, G. J. Systematic protein location mapping reveals five principal chromatin types in Drosophila cells. Cell. 143, 212-224 (2010).
- Vogel, M. J. Human heterochromatin proteins form large domains containing KRAB-ZNF genes. Genome Res. 16, 1493-1504 (2006).
- Venkatasubrahmanyam, S., Hwang, W. W., Meneghini, M. D., Tong, A. H., Madhani, H. D. Genome-wide, as opposed to local, antisilencing is mediated redundantly by the euchromatic factors Set1 and H2A.Z. Proc Natl Acad Sci U S A. 104, 16609-16614 (2007).
- Shimbo, T. MBD3 localizes at promoters, gene bodies and enhancers of active genes. PLoS Genet. 9, e1004028(2013).
- Orian, A. Genomic binding by the Drosophila Myc, Max, Mad/Mnt transcription factor network. Genes Dev. 17, 1101-1114 (2003).
- Artegiani, B. Tox: a multifunctional transcription factor and novel regulator of mammalian corticogenesis. EMBO J. , (2014).
- Schuster, E. DamID in C. elegans reveals longevity-associated targets of DAF-16/FoxO. Mol Syst Biol. 6, 399(2010).
- Bianchi-Frias, D. Hairy transcriptional repression targets and cofactor recruitment in Drosophila. PLoS Biol. 2, e178(2004).
- Woolcock, K. J., Gaidatzis, D., Punga, T., Buhler, M. Dicer associates with chromatin to repress genome activity in Schizosaccharomyces pombe. Nat Struct Mol Biol. 18, 94-99 (2011).
- Luo, S. D., Shi, G. W., Baker, B. S. Direct targets of the D. melanogaster DSXF protein and the evolution of sexual development. Development. 138, 2761-2771 (2011).
- Germann, S., Gaudin, V. Mapping in vivo protein-DNA interactions in plants by DamID, a DNA adenine methylation-based method. Methods Mol Biol. 754, 307-321 (2011).
- Zhang, X. The Arabidopsis LHP1 protein colocalizes with histone H3 Lys27 trimethylation. Nat Struct Mol Biol. 14, 869-871 (2007).
- Orian, A. Chromatin profiling, DamID and the emerging landscape of gene expression. Curr Opin Genet Dev. 16, 157-164 (2006).
- Vogel, M. J., Peric-Hupkes, D., van Steensel, B. Detection of in vivo protein-DNA interactions using DamID in mammalian cells. Nat Protoc. 2, 1467-1478 (2007).
- Greil, F., Moorman, C., van Steensel, B. DamID: mapping of in vivo protein-genome interactions using tethered DNA adenine methyltransferase. Methods Enzymol. 410, 342-359 (2006).
- de Wit, E., Greil, F., van Steensel, B. Genome-wide HP1 binding in Drosophila: developmental plasticity and genomic targeting signals. Genome Res. 15, 1265-1273 (2005).
- DamID mammalian vectors. , Available from: http://research.nki.nl/vansteensellab/Mammalian_plasmids.htm (2015).
- Illumina TruSeq adaptors & PCR primers. , Available from: https://ethanomics.wordpress.com/chip-seq-library-construction-using-the-illumina-truseq-adapters/ (2015).
- Optimization of PCR cycles for NGS. , Available from: https://ethanomics.wordpress.com/ngs-pcr-cycle-quantitation-protocol/ (2015).
- Bernstein, B. E. The NIH Roadmap Epigenomics Mapping Consortium. Nat Biotechnol. 28, 1045-1048 (2010).
- Encode Project Consortium. A user's guide to the encyclopedia of DNA elements (ENCODE). PLoS Biol. 9, e1001046(2011).
- Asp, P. Genome-wide remodeling of the epigenetic landscape during myogenic differentiation. Proc Natl Acad Sci U S A. 108, E149-E158 (2011).
- Hoppe, P. S., Coutu, D. L., Schroeder, T. Single-cell technologies sharpen up mammalian stem cell research. Nat Cell Biol. 16, 919-927 (2014).
- Avital, G., Hashimshony, T., Yanai, I. Seeing is believing: new methods for in situ single-cell transcriptomics. Genome Biol. 15, 110(2014).
- Navin, N. E. Cancer genomics: one cell at a time. Genome Biol. 15, 452(2014).
- Saliba, A. E., Westermann, A. J., Gorski, S. A., Vogel, J. Single-cell RNA-seq: advances and future challenges. Nucleic Acids Res. 42, 8845-8860 (2014).
- Nagano, T. Single-cell Hi-C reveals cell-to-cell variability in chromosome structure. Nature. 502, 59-64 (2013).
- Kind, J. Single-cell dynamics of genome-nuclear lamina interactions. Cell. 153, 178-192 (2013).
- Southall, T. D. Cell-type-specific profiling of gene expression and chromatin binding without cell isolation: assaying RNA Pol II occupancy in neural stem cells. Dev Cell. 26, 101-112 (2013).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır