Method Article
DamID-seq: Genome-wide Mappatura delle interazioni proteina-DNA da High Throughput Sequencing di Adenina-metilato frammenti di DNA
In questo articolo
Riepilogo
Descriviamo qui un test con l'accoppiamento di identificazione del DNA adenina metiltransferasi (DamID) ad alto rendimento sequenziamento (DamID-ss). Questo metodo fornisce una migliore risoluzione più elevata e una gamma dinamica più ampia, e permette di analizzare i dati DamID-Seq in combinazione con altri dati di sequenziamento ad alto rendimento, come ChIP-seq, RNA-Seq, etc.
Abstract
The DNA adenine methyltransferase identification (DamID) assay is a powerful method to detect protein-DNA interactions both locally and genome-wide. It is an alternative approach to chromatin immunoprecipitation (ChIP). An expressed fusion protein consisting of the protein of interest and the E. coli DNA adenine methyltransferase can methylate the adenine base in GATC motifs near the sites of protein-DNA interactions. Adenine-methylated DNA fragments can then be specifically amplified and detected. The original DamID assay detects the genomic locations of methylated DNA fragments by hybridization to DNA microarrays, which is limited by the availability of microarrays and the density of predetermined probes. In this paper, we report the detailed protocol of integrating high throughput DNA sequencing into DamID (DamID-seq). The large number of short reads generated from DamID-seq enables detecting and localizing protein-DNA interactions genome-wide with high precision and sensitivity. We have used the DamID-seq assay to study genome-nuclear lamina (NL) interactions in mammalian cells, and have noticed that DamID-seq provides a high resolution and a wide dynamic range in detecting genome-NL interactions. The DamID-seq approach enables probing NL associations within gene structures and allows comparing genome-NL interaction maps with other functional genomic data, such as ChIP-seq and RNA-seq.
Introduzione
DNA identificazione adenina metiltransferasi (DamID) 1,2 è un metodo per rilevare le interazioni proteina-DNA in vivo ed è un approccio alternativo per immunoprecipitazione della cromatina (ChIP) 3. Esso utilizza una quantità relativamente bassa di cellule e non richiede reticolazione chimica della proteina con DNA o un anticorpo altamente specifico. Quest'ultima è particolarmente utile quando la proteina bersaglio è liberamente o indirettamente associati con il DNA. DamID è stato utilizzato con successo per mappare i siti di legame di una varietà di proteine tra cui proteine dell'involucro nucleare 4-10, cromatina proteine associate 11-13, cromatina modifica enzimi 14, fattori di trascrizione e co-fattori 15-18 e RNAi macchinari 19. Il metodo è applicabile in diversi organismi compreso S. cerevisiae 13, S. pombe 7, C. elegans 9,17, D. melanogaster 5,11,18,20, A. thaliana 21,22 così come topo e cellule umane linee 6,8,10,23,24.
Lo sviluppo del test DamID si è basata sulla rilevazione specifica di adenina-metilato frammenti di DNA nelle cellule eucariotiche che la mancanza adenina endogena metilazione 2. Una proteina di fusione espressa, consistente della proteina legante il DNA di interesse e E. DNA coli adenina metiltransferasi (Dam), può metilare la base adenina in sequenze GATC che sono in prossimità spaziale (la maggior parte in modo significativo entro 1 kb e fino a circa 5 kb) per i siti di legame della proteina nel genoma 2. I frammenti di DNA modificati possono essere specificamente amplificati e ibridati microarray per rilevare i siti di legame genomici della proteina di interesse 1,25,26. Questo metodo originale DamID era limitata dalla disponibilità di microarrays e la densità di sonde predeterminati. Abbiamo quindi integrata high throughput sequencing ina DamID 10 e designato il metodo DamID-ss. Il gran numero di breve letture generata da DamID-ss permette la localizzazione precisa delle interazioni proteina-DNA del genoma-larghi. Abbiamo scoperto che DamID-ss fornito una risoluzione più elevata e una gamma dinamica più ampia rispetto DamID mediante microarray per lo studio del genoma nucleare lamina (NL) 10 associazioni. Questo metodo permette una migliore sondaggio associazioni NL all'interno di strutture geniche 10 e facilita il confronto con altri dati di sequenziamento ad alto rendimento, come ChIP-seq e RNA-Seq.
Il protocollo DamID-ss qui descritto è stato inizialmente sviluppato per la mappatura del genoma-NL associazioni 10. Abbiamo generato una proteina di fusione per legare il mouse o Lamin B1 umana a E. DNA metiltransferasi coli adenina e testato il protocollo di 3T3 fibroblasti embrionali di topo, topo C2C12 mioblasti 10 e IMR90 fibroblasti polmonari fetali umane (dati non pubblicati). In questo protocollo, iniziamo con constructing vettori ed esprimere proteine di fusione Dam-tethered di infezione lentivirale in cellule di mammifero 24. Successivamente, si descrivono i protocolli dettagliati di amplificare frammenti di DNA adenina-metilato e preparare le librerie di sequenziamento che dovrebbero essere applicabili in altri organismi.
Protocollo
1. Generazione ed espressione di proteine di fusione e Free Dam proteine
- Clone proteina di interesse nel vettore DamID.
- Amplifica cDNA di proteina di interesse (POI) utilizzando l'alta fedeltà DNA polimerasi desiderato e primer appropriati secondo il protocollo del produttore. Sperimentalmente determinare le condizioni ottimali di amplificazione per garantire la corretta amplificazione inserti.
- Eseguire un gel e purificare amplificato cDNA POI dal kit di estrazione gel secondo il protocollo del produttore.
- CDNA clone del PDI nel vettore pDONR201 utilizzando BP Clonase II secondo il protocollo del produttore.
- Clonare il cDNA del POI dal vettore donatore nella destinazione vettore pLGW-RFC1-V5-EcoDam o pLGW-EcoDam-V5-RFC1 destinazione vettore 27 con LR Clonase II secondo il protocollo del produttore, a seconda della direzione desiderata di fusione POI al N-terminale o the C-terminale della E. DNA coli adenina metiltransferasi (EcoDam) 27.
- Verificare mediante sequenziamento che il cDNA clonato ha una sequenza corretta e forme in-frame fusione a EcoDam.
- Generare scorte lentivirali.
- Generare scorte lentivirali che esprimono Dam-V5-POI e V5-Dam (dal vettore pLGW-V5-EcoDam 27) utilizzando sistemi di espressione lentivirali. Utilizzare la procedura di trasfezione in avanti secondo il protocollo del produttore.
- Utilizzare le cellule 293T e lipofezione per generare scorte lentivirali secondo il protocollo del produttore per la trasfezione.
- Includere il passo filtro da 0,45 micron PVDF.
- Generare scorte lentivirali che esprimono Dam-V5-POI e V5-Dam (dal vettore pLGW-V5-EcoDam 27) utilizzando sistemi di espressione lentivirali. Utilizzare la procedura di trasfezione in avanti secondo il protocollo del produttore.
- Infettare le cellule con lentivirus.
- Il giorno prima dell'infezione (Giorno 0), passare cellule aderenti coltivate in terreni di crescita appropriati per questo tipo di cellule di un tessuto di piastra di coltura 6-bene usando lo stesso terreno di crescita senza antibiotici perraggiungere il 50% di confluenza il giorno di infezione. Cellule posto in un incubatore a 37 °.
- Il giorno di infezione (giorno 1), rimuovere le 2 cryovials sia Dam-V5-POI e V5-Dam supernatanti lentivirali da un -80 ° C congelatore e posto in un bagno d'acqua a 37 ° per scongelare.
- Rimuovere i supporti di crescita dalle cellule e sostituirlo con 0,5 ml di terreni di crescita fresco senza antibiotici.
- Aggiungere 1 ml di lentivirus scongelato in ogni pozzetto (2 pozzi con V5-Dam e 2 pozzi con Dam-V5-POI). Aggiungere 1 ml di terreni di crescita, senza antibiotici per i restanti 2 pozzi questo servirà come controllo negativo. Agitare delicatamente il 6 pozzetti per mescolare e mettere di nuovo a 37 ° C incubatore O / N.
- Il giorno dopo l'infezione (Day 2), rimuovere le sospensioni virali dalle cellule e sostituire con 2 ml terreni di crescita senza antibiotici. Cellule posto torna in un incubatore a 37 ° per 48 ore.
- Isolare gDNA.
- Aspirare i media di ogni bene e dicellule tachimetrici utilizzando 250 microlitri 0,05% tripsina-EDTA. Incubare a 37 ° C per 2 min.
- Lavare le cellule la piastra con 1 ml terreni di crescita e cellule pipetta di ogni bene in una provetta da 1,5 ml microcentrifuga. Agglomerare cellule per centrifugazione a 200 xg per 5 minuti a temperatura ambiente.
- Lavare le cellule pellet con 500 microlitri di PBS e centrifugare a 200 xg per 2 minuti a temperatura ambiente.
- Risospendere le cellule pellet in 200 l di PBS.
- Isolare gDNA da sangue e tessuti kit secondo il protocollo del produttore. Eluire gDNA in 200 microlitri tampone AE e determinare la concentrazione misurando OD 260 utilizzando uno spettrofotometro.
Nota: gDNA da cellule infette non infettate o finti può essere isolato come controlli negativi. Precipitare gDNA a concentrazioni più elevate per la conservazione a lungo termine.- Aggiungere 3 volumi di etanolo 100% e 0,1 volume di 3 M acetato di sodio (pH 5,5), e mescolare invertendo tubi 4-6 volte.
- Conservare a -20 ° CO / N.
- Centrifugare a 16.000 xg per 15 minuti a 4 ° C.
- Rimuovere con attenzione il surnatante. Lavare pellet con 70% (volume / volume) di etanolo e centrifugare a 16.000 xg per 5 minuti a 4 ° C.
- Rimuovere con cautela l'etanolo e lasciare pellet asciugare all'aria per 3 minuti a temperatura ambiente.
- Sciogliere gDNA in T 10 E 0.1 (pH 7.5) a circa 1 mg / mL. Piscina gDNA da ogni campione sperimentale o il controllo negativo e misurare la concentrazione. Conservare a -20 ° C.
2. Amplify Adenina-metilati frammenti di DNA
- Digest gDNA con DpnI che taglia solo GATCs adenina-metilato.
- Impostare una reazione su ghiaccio con 2,5 mg gDNA, 1 ml 10x tampone, 0,5 microlitri DpnI (20 U / ml) e riempire con H 2 O per un volume totale di 10 ml. Per ogni campione gDNA, preparare tre reazioni - uno senza DpnI ("no DpnI", sostituire DpnI con 0,5 ml H 2 O) e due wesimo DpnI ("con DpnI").
- Digest O / N a 37 ° C e inattivare DpnI a 80 ° C per 20 min.
- Legare gli adattatori DamID.
- Preparare gli adattatori DamID.
- Risospendere ciascuno dei due adattatori oligo DamID 24 in H 2 O a una concentrazione finale di 100 mM.
- Combinare uguali volumi dei due adattatori oligo DamID, mescolare e mettere in un tubo ben chiuso. Sigillare il tubo con Parafilm, sedersi in un rack e posto in un bicchiere pieno d'acqua a 90 ° C. Mantenere il livello dell'acqua sotto il tappo del tubo (per evitare infiltrazioni di acqua nel tubo), ma al di sopra della superficie del mix oligo.
- Lasciare raffreddare l'acqua a RT in modo che il adattatori ricottura lentamente.
- Aliquota gli adattatori ricotto (50 micron) e conservare a -20 ° C.
- Impostare una reazione su ghiaccio. In ogni tubo da 2.1.2, aggiungere 6,2 ml H 2 O, 2 microlitri 10x tampone legatura, 0,8 ml 50 micron DamID annuncioaptors (scongelati su ghiaccio) e 1 ml DNA ligasi T4 (5 U / ml). Il volume totale è di 20 microlitri. In uno dei due "con DpnI" tubi, sostituire ligasi con 1 ml H 2 O ("no ligasi") Si noti che ogni campione gDNA ha due controlli negativi -. "No DpnI" e "no ligasi".
- Legare O / N a 16 ° C e inattivare ligasi a 65 ° C per 10 min.
- Preparare gli adattatori DamID.
- Digest DNA con DpnII di distruggere frammenti che contengono GATCs unmethylated.
- Impostare una reazione su ghiaccio. In ogni tubo da 2.2.3, aggiungere 24 ml H 2 O, buffer di 5 ml 10x DpnII e 1 ml DpnII (10 U / ml). Il volume totale è di 50 microlitri.
- Digest a 37 ° C per 2-3 ore e inattivare DpnII a 65 ° C per 20 min.
- Amplifica frammenti di DNA di adenina-metilato.
- Impostare una reazione su ghiaccio con 5 ml DpnII digerire da 2.3.2, 5 μl 10x PCR buffer, 12,5 ml 5 micron DamID PCR Primer 24, 4 microlitri 10 mM dNTP mix, 1 ml 50x mix polimerasi e 22,5 ml H 2 O. Il volume totale è di 50 microlitri.
- Eseguire la PCR come segue: 68 ° C per 10 min; 94 ° C per 1 min, 65 ° C per 5 min, 68 ° C per 15 min; 4 cicli di 94 ° C per 1 min, 65 ° C per 1 min, 68 ° C per 10 min; 17 cicli di 94 ° C per 1 min, 65 ° C per 1 min, 68 ° C per 2 min.
- Analizzare 5 microlitri PCR prodotti da ciascuna reazione su un gel di agarosio 1%. Prodotti di PCR dovrebbero apparire come una macchia variabile da 0,2 a 2 kb (Figura 2). I comandi "no DpnI" e "no" ligasi non dovrebbero avere o meno chiaramente di amplificazione.
- Se il risultato del punto 2.4.3 è soddisfacente, ripetere i passaggi 2.4.1-2.4.3 con due o tre reazioni per il campione sperimentale e uno di reazione per ciascuna delle due controlli negativi.
- Piscina e purificare prodotti di PCR dalla stessacampione sperimentale utilizzando i kit di purificazione PCR o in fase solida reversibile immobilizzazione (SPRI) perline secondo il protocollo del produttore. Non purificare "no DpnI" o controlli "no ligasi". Eluire il DNA con tampone EB.
- Misurare la concentrazione del DNA purificato misurando OD 260 utilizzando uno spettrofotometro, che dovrebbe essere di circa 0,1 mg / mL o superiore. Raccogliere un minimo di 10 ug di DNA per ciascun campione. Se utilizzando kit di purificazione PCR, purificare ogni 50 microlitri PCR prodotti con una colonna, eluire in 30 microlitri tampone EB e riunire gli eluati.
- Digest DNA con DpnII per evitare primer DamID PCR di essere sequenziato.
- Impostare una reazione su ghiaccio con 5 mg DNA purificato da 2.4.6, 5 ml 10x tampone DpnII, 1 ml DpnII (10 U / mL) e si riempiono di H 2 O per un volume totale di 50 ml. Preparare due o tre reazioni per ciascun campione.
- Digest a 37 ° C per 2-3 oree inattivare DpnII a 65 ° C per 20 min.
- Piscina e purificare i digest dallo stesso campione con i kit di purificazione PCR o SPRI perline secondo il protocollo del produttore. Eluire il DNA con tampone EB.
- Misurare la concentrazione del DNA purificato che dovrebbe essere di circa 0,06 mg / mL o superiore. Raccogliere un minimo di 6 mg di DNA per ciascun campione. Se utilizzando kit di purificazione di PCR, purificare ogni 50 ml digerire con una colonna, eluire in 30 microlitri tampone EB e riunire gli eluati.
3. Biblioteca Preparazione per High-throughput Sequencing
- DNA Fragment
- Sperimentalmente determinare il tempo di digestione appropriata per ogni nuovo lotto di dsDNA Fragmentase. Poiché l'attività enzimatica può diminuire nel tempo, ripetere il test prima di eseguire un nuovo esperimento. Per salvare il DNA da 2.5.4 per la frammentazione attuale, utilizzare prodotti di PCR metile purificati da 2.4.6 o DNA extra da experime precedententi di questo passo.
- Impostare un master mix con 6 mg di DNA, 12 microlitri di buffer 10x Fragmentase e riempire con H 2 O per un volume totale di 114 microlitri.
- Vortex lo stock fiala Fragmentase per 3 secondi, aggiungere 6 microlitri nel mix master e Vortex il master mix per 3 secondi. Il volume totale è di 120 microlitri.
- Aliquotare 20 microlitri della master mix per ciascuno dei 5 nuovi tubi. Incubare tutte le provette a 6 ° C per 37 5 a 55 min con un incremento di 10 min. Aggiungere 5 microlitri 0,5 M EDTA per bloccare la reazione.
- Analizzare 12,5 microlitri digest (0,5 ug di DNA) di ciascuna reazione e 0,5 mg di DNA digerito su gel di agarosio (Figura 3). Determinare il tempo minimo (T 0.2kb) necessario per digerire la maggioranza della striscio di circa 0,2 kb. Selezionare 6 durate tra 5 minuti e T 0.2kb (di cui 5 min e T 0.2kb) con incrementi uguali per la frammentazione attuale.
- Impostare i frammentazione attualezione come descritto in 3.1.1.1-3.1.1.3. Incubare le reazioni a 37 ° C per le durate di tempo determinato in 3.1.1.4.
- Pool 6 reazioni e purificare i digest con kit di purificazione PCR o SPRI perline secondo il protocollo del produttore. Eluire il DNA in tampone 51 microlitri EB. L'eluato finale è ~ 50 ml.
- Sperimentalmente determinare il tempo di digestione appropriata per ogni nuovo lotto di dsDNA Fragmentase. Poiché l'attività enzimatica può diminuire nel tempo, ripetere il test prima di eseguire un nuovo esperimento. Per salvare il DNA da 2.5.4 per la frammentazione attuale, utilizzare prodotti di PCR metile purificati da 2.4.6 o DNA extra da experime precedententi di questo passo.
- Riparazione estremità del DNA frammentato
- Impostare una reazione su ghiaccio con l'eluato da 3.1.3, 25 ml H 2 O, 10 microlitri 10x T4 DNA ligasi tampone con l'ATP 10 mm, 4 pl 10 mM dNTP mix, 5 microlitri DNA polimerasi T4 (3 U / ml) , 1 ml DNA polimerasi Klenow (5 U / ml), e 5 microlitri T4 polinucleotide chinasi. Il volume totale è di 100 microlitri. Mescolare bene con la pipetta. Evitare la schiuma e bolle.
- Incubare a 20 ° C per 30 min.
- Purificare il DNA con kit di purificazione PCR o SPRI perline secondo il protocollo del produttore. Eluire il DNA in tampone 33 microlitri EB. L'eluato finale è ~ 32 & #181; l.
- Aggiungere sbalzi "A"
- Impostare una reazione su ghiaccio con l'eluato da 3.2.3, 5 ml di buffer 10x Klenow, 10 microlitri 1 mM dNTP, e 3 microlitri Klenow (3 '→ 5' eso) (5 U / ml). Il volume totale è di 50 microlitri. Mescolare bene con la pipetta. Evitare la schiuma e bolle.
- Incubare a 37 ° C per 30 min.
- Purificare il DNA con kit di purificazione PCR o SPRI perline secondo il protocollo del produttore. Eluire il DNA in tampone 22 microlitri EB. L'eluato finale è ~ 21 microlitri.
- Adattatori sequenziamento legare
- Preparare gli adattatori sequenziamento 28.
- Risospendere ogni oligo adattatore ad una concentrazione di 100 mM a 10 mM Tris-Cl (pH 7,8), 0,1 mM EDTA (pH 8,0) e 50 mM NaCl.
- Miscelare volumi uguali della adattatore universale 28 e un adattatore indicizzato 28.
- Anneal gli adattatori in un termociclatore con il seguenteProgramma: 95 ° C per 2 min; 140 cicli per 30 sec a partire da 95 ° C e diminuendo di 0,5 ° C ogni ciclo; tenere a 4 ° C.
- Adattatori aliquote e conservare a -20 ° C.
- Impostare una reazione su ghiaccio con l'eluato da 3.3.3, 25 microlitri 2x tampone legatura, 1,5 ml 50 micron ricotto adattatori sequenziamento (scongelati su ghiaccio) da 3.4.1.4 e 2,5 microlitri DNA ligasi T4. Mescolare bene con la pipetta. Evitare la schiuma e bolle. Se il sequenziamento multiplex si desidera, utilizzare un adattatore indicizzato diverso per ogni campione.
- Incubare a temperatura ambiente per 1 ora.
- Purificare il DNA con kit di purificazione PCR o SPRI perline secondo il protocollo del produttore. Eluire il DNA in tampone 24 microlitri EB. L'eluato finale è ~ 23 microlitri.
- Preparare gli adattatori sequenziamento 28.
- Convertire adattatori a Y per dsDNA per consentire precisione determinare frammento di DNA di dimensioni 28
- Impostare una reazione su ghiaccio con l'eluato da 3.4.4, 12.5 ml H 2 O, 1 ml 25 micron di primer 1 28, 1 ml 25 micron di primer 2 28, 1,5 microlitri 10mM dNTP, 10 microlitri 5x PCR Buffer e 1 ml DNA polimerasi (1 U / ml).
- Eseguire la PCR come segue: 95 ° C per 3 min; 5 cicli di 98 ° C per 15 sec, 63 ° C per 30 sec, 72 ° C per 30 sec; 72 ° C per 1 min; 4 ° C in attesa.
- Purificare il DNA con kit di purificazione PCR o SPRI perline secondo il protocollo del produttore. Eluire il DNA in tampone 11 microlitri EB. L'eluato finale è ~ 10 ml.
- Dimensioni selezionare la libreria
- Preparare un gel di agarosio al 2% con 1x tampone TAE. Aggiungere etidio bromuro (ATTENZIONE!) Ad una concentrazione finale di 500 ng / ml quando la soluzione TAE-agarosio fuso è raffreddato per evitare l'inalazione di etidio bromuro. Assicuratevi di avere abbastanza corsie per tutti i campioni, scala del DNA e corsie vuote.
- Aggiungere 8 ml 6x carico colorante per l'eluato da 3.5.3.
- Preparare DNA ladder miscelando 1 kb più DNA ladder (1,0 mg / mL), loading dye 6x e H 2 O in un rapporto di 1: 1: 4.
- Carico 6 ml scala del DNA, i campioni di 3.6.2 e un'altra scala del DNA 6 microlitri, ciascuno in una corsia separata e con almeno una corsia vuota dalle adiacenti campioni / scale.
- Attivare il gel a 120 V per 60 min.
- Mostra il gel su un transilluminatore UV (minimizzare il tempo di esposizione ai raggi UV). Indossare occhiali protettivi e una visiera. Assicurarsi che almeno una delle scale di DNA ben gestito con spaziatura appropriata (sufficiente per asportare 3 fette di gel) tra i 300 bp e 400 bp bande. Narrow spaziatura aumenta le difficoltà di asportare più fette di gel, mentre un'ampia spaziatura aumenta il volume di fette di gel escisse.
- Utilizzare un nuovo bisturi o una lama di rasoio per ogni corsia. Excise tre fette sottili di gel tra 300 e 400 bp da ciascuna corsia (Figura 4) e ogni luogo in una provetta. Mantenere il volume di ogni gel spidocchi più basso possibile (<100 ml).
- Misurare il volume di ogni fetta gel (1 ml gel pesa circa 1 mg), aggiungere i volumi di tampone 6x QG e incubare a 50 ° C.
- Vortex la miscela QG-gel ogni 2-3 minuti fino a quando la fetta gel è completamente dissolto. Aggiungere volumi di gel 2x di isopropanolo e mescolare.
- Da questo punto, seguire il protocollo da kit di estrazione gel. Eluire il DNA in tampone 51 microlitri EB. L'eluato finale è ~ 50 ml.
- Arricchisci sequenziamento adattatore modificato frammenti di DNA
- Ottimizzare il numero di cicli di PCR 29.
- Impostare una reazione su ghiaccio con 1 ml eluato dal 3.6.10, 1 ml 25 micron di primer 1 28, 1 ml 25 micron di primer 2 28, 7 ml H 2 O e 10 microlitri SYBR Green Supermix. Il volume totale è di 20 microlitri.
- Eseguire il qPCR come segue: 95 ° C per 3 min; 20 cicli di 95 ° C per 30 sec, 63 ° C per 30 sec, 72 ° C per 30 sec, piastra di lettura.
- Analizzare i dati utilizzando un software di analisi qPCR e determinare il ciclo di Quantificazione (Cq) o ciclo soglia (Ct) per ogni campione utilizzando il protocollo del produttore. Continuare con i campioni che hanno Cq / Ct ≤ 14. Utilizzare la massima Cq (CQ 0) meno 1 (arrotondato al numero intero più alto), come il numero di cicli PCR finale (N PCR).
- Regolare le quantità di modelli di DNA in modo che diversi campioni verranno amplificati a quantità approssimativamente uguali dopo aver eseguito lo stesso numero di cicli di PCR. Utilizzare template 8 microlitri del campione con il più alto Cq (Cq 0), e calcolare il volume template di altri campioni con la seguente formula:
Vol i = 8 x 1.8 Cq i -Cq 0
- Impostare reazioni PCR in ghiaccio con i volumi dei modelli calcolati da 3.7.1.4: 1 ml 25 micron di primer 1 28, 1 ml 25 micron di primer 2 28, 1.5microlitri 10 mM dNTP, 10 microlitri tampone 5x, 1 microlitri DNA polimerasi (1 U / ml) e riempire con H 2 O per un volume totale di 50 microlitri.
- Eseguire la PCR come segue: 95 ° C per 45 sec; N cicli di PCR (determinati in 3.7.1.3) di 98 ° C per 15 sec, 63 ° C per 30 sec, 72 ° C per 30 sec; 72 ° C per 1 min; 4 ° C in attesa.
- Analizzare 5 microlitri prodotti di PCR in un gel di agarosio al 2% (Figura 5A). Un chiaro banda di "single" indica che i frammenti di DNA amplificati sono all'interno di un intervallo di lunghezza stretto e che la biblioteca DNA può essere oggetto di ulteriori analisi.
- Ripetere una reazione per i campioni selezionati e riunire le librerie di DNA amplificati dallo stesso campione.
- Purificare librerie DNA selezionati per il sequenziamento.
- Se dimeri di primer / adattatori non sono visibili in 3.7.4, purificare le librerie di DNA con kit di purificazione PCR o SPRI perline secondo il protocollo del produttore. Eluire il DNA in tampone 21 microlitri EB. Ileluato finale è ~ 20 l. Se impianto sequencing dell'utente ha istruzioni specifiche sul tampone di eluizione, il volume finale, ecc, preparare campioni di conseguenza.
- Se dimeri di primer / adattatori sono chiaramente visibili in 3.7.4, purificare le biblioteche come segue.
- Carico DNA da 3.7.5 e DNA Ladder su gel di agarosio 2% prefabbricato. I campioni possono essere purificati come descritto in 3.7.6.1 per ridurre il volume o può essere caricato in diversi pozzetti. Porre il gel di agarosio in un sistema di alimentazione appropriato. Lasciare che il gel a correre per 15 minuti.
- Utilizzando un transilluminatore adeguata, tagliare la banda desiderata con un pulito bisturi / rasoio per ogni campione e posizionare la fetta gel in una provetta da 1,5 ml microcentrifuga.
- Isolare DNA utilizzando kit di estrazione gel secondo il protocollo del produttore con due modifiche: incubare tampone miscela QG-gel in un termo-mixer a 37 ° C e 1.000 rpm per 30 min, e aggiungere volumi gel 2x di isopropanolo.
- Ottimizzare il numero di cicli di PCR 29.
- Invia le biblioteche del DNA purificato in un impianto di sequenziamento. Seguire tutte le linee guida della funzione.
Nota: Un analisi della qualità da un Bioanalyzer (Figura 5B) deve essere eseguita prima del sequenziamento per determinare la gamma esatta dimensione e la concentrazione di una libreria di DNA.
Risultati
La proteina di fusione Dam-V5-LmnB1 è stato verificato per essere co-localizzato con il endogena Lamin B proteine mediante immunofluorescenza (Figura 1).
Il successo di amplificazione PCR di frammenti di DNA adenina-metilato è un passo fondamentale per DamID-ss. I campioni sperimentali dovrebbero amplificare una macchia di 0,2 - 2 kb, mentre i controlli negativi (senza DpnI, senza ligasi o senza mascherina PCR) dovrebbe portare a non-o chiaramente meno-amplificazione (Figura 2).
I frammenti di DNA metilati sono nell'intervallo da 0,2 a 2 kb, mentre la dimensione dell'inserto desiderata per una libreria NGS è da 200 a 300 bp. Pertanto, è essenziale per frammentare i prodotti di PCR di metile nella gamma formato adatto. Tuttavia, è stato trovato per essere poco pratico per rompere contemporaneamente frammenti di DNA più grandi a dimensioni adattes e mantenere la maggior parte dei piccoli frammenti di DNA intatto in una singola durata frammentazione. Pertanto, gli esperimenti sono stati eseguiti corso di tempo per determinare il tempo minimo (T 0.2kb) necessario per frammento di 1 ug di DNA ad uno striscio centrato a 200 bp (Figura 3). Poi sono stati selezionati 6 durate di tempo in incrementi uguali tra 5 minuti e T 0.2kb per la frammentazione attuale. L'attività enzimatica della doppia elica di DNA Fragmentase può variare da lotto a lotto e può diminuire nel tempo, quindi si consiglia di ripetere questa operazione per un nuovo lotto di Fragmentase o dopo conservazione per un periodo di tempo.
La dimensione inserto desiderato è compreso tra 200 e 300 bp corrispondente a frammenti di DNA tra 300 e 400 bp (compresi 121 bp adattatori sequenziamento) sul gel di agarosio. Tre fette sottili all'interno di questa gamma sono stati asportati da ciascun campione sperimentale per restringere la gamma di dimensioni di una biblioteca e di aumentare la possibilitàdi ottenere almeno una libreria sequenziamento qualificato (Figura 4).
Un'aliquota di 5 ml di ogni biblioteca DNA amplificato è stato analizzato il gel per determinare quale biblioteca possono qualificarsi per il sequenziamento. Come mostrato in Figura 5A, una chiara singola banda della stessa dimensione della fetta gel escisse dovrebbe essere visibile sul gel di agarosio (fase 3.7.4). Avanti, librerie selezionate sono stati esaminati da un Bioanalyzer (Figura 5B) per determinare la gamma esatta dimensione e concentrazione prima di sequenziamento. Se lo si desidera, le biblioteche di DNA amplificati possono essere esaminati direttamente da un Bioanalyzer senza l'analisi di gel. Quando più librerie sono di buona qualità, si consiglia di sequenziare librerie di dimensioni simili gamme per un paio di sperimentali (cellule che esprimono Dam-V5-POI) e di controllo (cellule che esprimono V5-Dam) campioni.
Il corto si legge generato dasistemi di sequenziamento sono stati mappati torna alla corrispondente genoma. Unicamente allineato letture venivano poi passati alle analisi successive. Una conduttura di elaborare a breve legge, costruire un'interazione mappa del genoma-NL e analizzare le associazioni gene-NL sono stati descritti in dettaglio nel nostro precedente lavoro 10. Risultati rappresentativi sono mostrati in Figura 6.

Figura 1. Convalida localizzazioni subnucleari di proteine di fusione da immunofluorescenza. IMR90 cellule di fibroblasti polmonari umane sono state transitoriamente trasfettate con un plasmide che esprime Dam-V5-LmnB1 e sono state colorate da anti-Lamin B (A, rossa) e anti-V5 (B, verde). (C) Unire di immagini in A e B. Si prega di cliccare qui per vedere una versione più grande di tla sua figura.

Figura 2. PCR amplificare frammenti di DNA adenina-metilato. Un'aliquota di 5 microlitri da ogni reazione PCR è stata analizzata su gel di agarosio all'1%. Uno striscio che vanno 0,2-2 kb è stato amplificato da ogni campione sperimentale, ma nessuna amplificazione è stata osservata nei controlli negativi (nessun DpnI al punto 2.1, non Ligase al punto 2.2, o nessun modello PCR). Cliccate qui per vedere una versione più grande di questa figura.
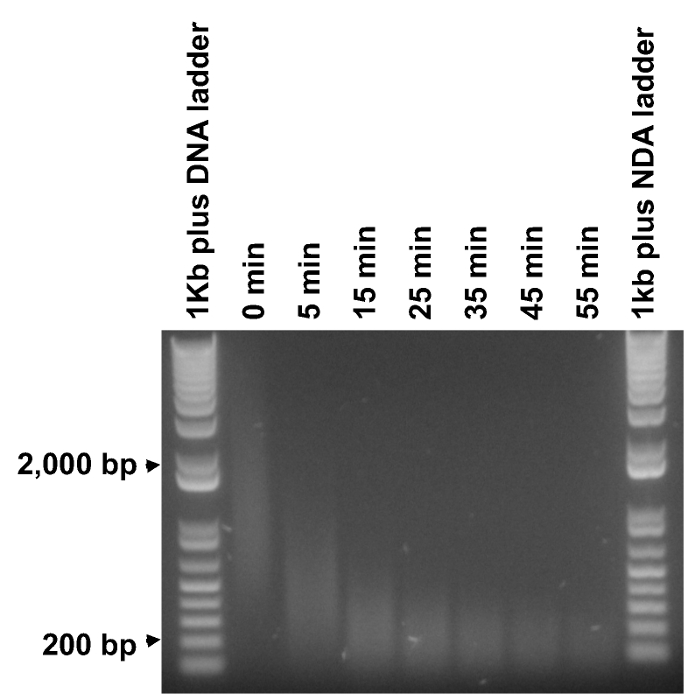
Figura 3. Determinazione durate ottimali di frammentazione. I prodotti di PCR purificati metile sono stati sottoposti a frammentazione per durate di tempo 5-55 min a un incremento di 10 min (DNA non digerito , Etichettato come "0 min"). 1 pg di DNA ladder e 0,5 mg di ciascun campione di DNA frammentato sono stati analizzati su un gel di agarosio. Il tempo minimo per digerire la maggioranza della striscio DNA di circa 200 bp è stato determinato in circa 35 minuti, quindi sei durate di tempo equidistanti tra 5 e 35 min (5 e 35 min incluse) sono stati selezionati per eseguire la frammentazione attuale. Favore clicca qui per vedere una versione più grande di questa figura.

Figura 4. Formato selezionando le librerie di DNA. Campioni di DNA di Dam-V5-LmnB1 e V5-Dam dal punto 3.6 sono stati eseguiti su un gel di agarosio al 2%, e tre fette di gel uguali dimensioni (L, M e H corrispondente a bassa, media ed alta dimensioni) venivano asportate tra 300 e 400 bp come mostrato dalle linee gialle. om / files / ftp_upload / 53620 / 53620fig4large.jpg "target =" _ blank "> Clicca qui per vedere una versione più grande di questa figura.
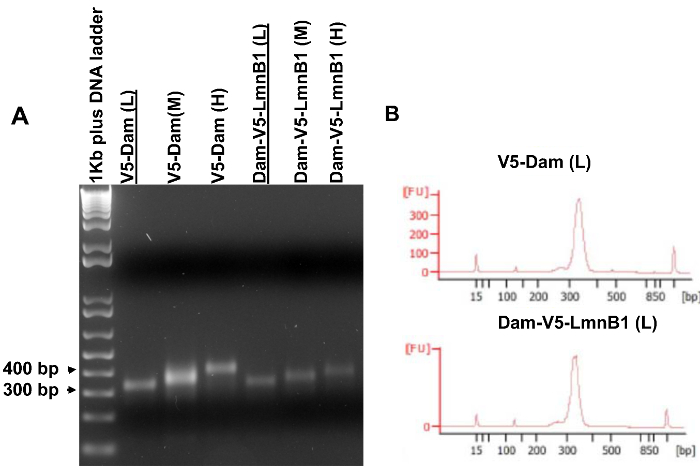
Figura 5. librerie di DNA amplificato per alta sequenziamento rendimento. (A) amplificata librerie di DNA analizzati su un gel di agarosio. Modelli di PCR sono stati purificati da fette di gel mostrato in figura 4. (B) Bioanalyzer risultati del campione V5-Dam (L) e il campione Dam-V5-LmnB1 (L) che sono stati sottolineato in (A). Questi due biblioteche avevano gli intervalli di dimensioni simili stretto e quindi qualificato per alta sequenziamento rendimento. Clicca qui per vedere una versione più grande di questa figura.
jpg "/>
Figura 6. dati NGS visualizzati nel UCSC browser genomico. Cromosoma mouse 1 Di seguito riportiamo come esempio. Dati della sequenza sono state prodotte da mioblasti topo C2C12 10. Tracce "MB.LmnB1.w2k" e "MB.Dam.w2k", corrispondenti ai dati provenienti da cellule che esprimono rispettivamente Dam-V5-LmnB1 e V5-Dam, trama normalizzato densità di lettura (legge per kilobase per milione mappata univocamente legge, o RPKM ) a non sovrapposte consecutivi finestre 2 kb lungo il cromosoma. Tieni traccia trame "MB.log2FC.w2k" associazioni genoma-NL, cioè log2 RPKM rapporti di LmnB1 oltre Dam, a 2 risoluzione kb. Traccia "MB.sLADs" dipinge Lamina Associated Domini basato sequenziamento-(slads, ossia regioni genomiche che hanno densità di lettura più elevate di LmnB1 su Dam con significatività statistica) in nero, non slads nelle regioni grigie e indeterminati in bianco. Cliccate qui per download questo file.
Discussione
Whether Dam-tagged proteins retain the functions of endogenous proteins should be examined before a DamID-seq experiment. The subcellular localization of Dam-tagged nuclear envelope proteins should always be determined and compared with that of the endogenous proteins. For studying transcription factors, it is suggested to examine whether the Dam-fusion protein can rescue the functions of the endogenous protein in regulating gene expression. This functional test can be performed in organisms in which knockout mutants of endogenous DNA-binding proteins are available. Because advances in genome engineering have potentially allowed knocking out any endogenous gene of interest, functions of Dam-tagged DNA-binding proteins can be examined in cultured mammalian cells.
The critical step in this protocol is to successfully fragment the DpnII-digested DamID PCR products to around 200 bp. This step is designed to render the amplified adenine-methylated fragments to a narrow size range for sequencing and to randomize the starting nucleotides of the DNA fragments in a sequencing library. Inefficient fragmentation will leave the majority of the DNA fragments starting with GATC (the 5'-overhang from the second DpnII digestion), and will result in a much lower performance and yield or even a failure in Illumina sequencing. Other DNA fragmentation methods may be used as an alternative approach.
The resolution of DamID (and DamID-seq described here) is limited by the frequency of GATCs in the genome to be studied. Moreover, even with high throughput sequencing, the genomic localizations of a DNA-binding protein can only be mapped within two consecutive GATCs rather than to the actual DNA-binding sites.
Despite its limitation, the DamID assay has important advantages. Because DamID does not require highly-specific antibodies, it can be used to detect a subset of nuclear proteins that could be difficult to assay by ChIP (such as the nuclear envelope proteins). To study how these proteins regulate genome functions, it is important to integrate and cross-analyze their genome-wide localization data with the current epigenomic mapping data (such as data from the ENCODE and NIH Roadmap Epigenomics Projects 30,31). The DamID-seq approach provides both higher resolution and higher sensitivity than DamID by microarray and enables detecting differential NL-associations within gene structures 10. A combinatorial analysis of DamID-seq data, ChIP-seq data 32 and gene expression data has identified a class of NL-associated genes with distinct epigenetic and transcriptional features (data not published).
Another advantage of DamID is that it only requires a small number of cells. In recent years, there has been an explosion in single cell analysis of gene regulation 33,34. Although genome sequence 35, genome-wide gene expression 36 and chromatin conformation 37 can be assayed in a single cell, there has not been an available approach for detecting protein-DNA interactions genome-wide in a single cell. DamID-seq is a highly promising approach for this goal, and may complement the single cell imaging approach in detecting the dynamics of genome-NL interactions 38. One complication is that because the Dam-fusion protein is expressed at a much lower level than the endogenous protein in the DamID assay, it is possible that the Dam-fusion protein may only occupy a subset of genomic binding sites as compared to the endogenous protein.
DamID assay has mostly been used in cultured animal cells to detect protein-DNA interactions. Notably, developmental biologists have applied this assay in detecting protein-DNA interactions in specific cell types in vivo. For example, Dam-tagged RNA polymerase II was expressed specifically in Drosophila neural stem cells to detect their genome-wide occupancy without cell isolation 39. DamID-seq will be highly useful to study the genome-wide localizations of nuclear envelope proteins, transcription factors and chromatin regulators during development in animal models.
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
We thank Dr. Bas van Steensel for providing the DamID mammalian expression vectors. We thank Yale Center for Genome Analysis and the Genomics Core in Yale Stem Cell Center for advice on preparing NGS libraries and implementing high throughput DNA sequencing. This work was supported by the startup funding from Yale School of Medicine, a Scientist Development Grant from American Heart Association (12SDG11630031) and a Seed Grant from Connecticut Innovations, Inc. (13-SCA-YALE-15).
Materiali
| Name | Company | Catalog Number | Comments |
| ViraPower Lentiviral Expression Systems | Life Technologies | K4950-00, K4960-00, K4970-00, K4975-00, K4980-00, K4985-00, K4990-00, K367-20, K370-20, and K371-20 | |
| Gateway BP Clonase II Enzyme Mix | Life Technologies | 11789-020 | |
| Gateway LR Clonase II Enzyme Mix | Life Technologies | 11791-020 | |
| DNeasy Blood & Tissue Kit (250) | QIAGEN | 69506 or 69504 | |
| Gateway pDONR 201 | Life Technologies | 11798-014 | |
| 293T cells | American Type Culture Collection | CRL-11268 | |
| Trypsin-EDTA (0.05%), phenol red | Life Technologies | 25300-054 | |
| DMEM, high glucose, pyruvate | Life Technologies | 11995-065 | |
| Fetal Bovine Serum | Sigma | F4135 | |
| Tris | brand not critical | ||
| EDTA | brand not critical | ||
| 200 Proof EtOH | brand not critical | ||
| Isopropanol | brand not critical | ||
| Sodium Acetate | brand not critical | ||
| DpnI | New England Biolabs | R0176 | supplied with buffer |
| DamID adaptors "AdRt" and "AdRb" | Integrated DNA Technologies | sequences available in ref. 24; no phosphorylation of the 5' or 3' end to prevent self-ligation. | |
| T4 DNA Ligase | Roche Life Science | 10481220001 | supplied with buffer |
| DpnII | New England Biolabs | R0543 | supplied with buffer |
| DamID PCR primer "AdR_PCR" | Integrated DNA Technologies | sequences available in ref. 24 | |
| Deoxynucleotide (dNTP) Solution Set | New England Biolabs | N0446 | 100 mM each of dATP, dCTP, dGTP and dTTP |
| Advantage 2 Polymerase Mix | Clontech | 639201 | supplied with buffer |
| 1Kb Plus DNA Ladder | Life Technologies | 10787018 | 1.0 µg/µl |
| QIAquick PCR Purification Kit | QIAGEN | 28104 or 28106 | |
| MinElute PCR Purification Kit | QIAGEN | 28004 or 28006 | for an elution volume of less than 30 µl |
| SPRI beads / Agencourt AMPure XP | Beckman Coulter | A63880 | apply extra mixing and more elution time if less than 40 µl elution buffer is used |
| Buffer EB | QIAGEN | 19086 | |
| NEBNext dsDNA Fragmentase | New England Biolabs | M0348 | supplied with buffer |
| T4 DNA Ligase Reaction Buffer | New England Biolabs | B0202 | |
| T4 DNA Polymerase | New England Biolabs | M0203 | |
| DNA Polymerase I, Large (Klenow) Fragment | New England Biolabs | M0210 | |
| T4 Polynuleotide Kinase | New England Biolabs | M0201 | |
| Klenow Fragment (3’ → 5’ exo-) | New England Biolabs | M0212 | supplied with buffer |
| sequencing adaptors | Integrated DNA Technologies | sequences available in ref. 28 | |
| Quick Ligation Kit | New England Biolabs | M2200 | used in 3.4.3; supplied with Quick Ligation Reaction Buffer and Quick T4 DNA Ligase |
| sequencing primer 1 and 2 | Integrated DNA Technologies | sequences available in ref. 28 | |
| KAPA HiFi PCR Kit | Kapa Biosystems | KK2101 or KK2102 | supplied with KAPA HiFi DNA Polymerase, 5x KAPA HiFi Fidelity Buffer and 10 mM dNTP mix |
| agarose | Sigma Aldrich | A4679 | |
| ethidium bromide | Sigma Aldrich | E1510-10ML | 10 mg/ml |
| QIAquick Gel Extraction Kit | QIAGEN | 28704 or 28706 | |
| iTaq Universal SYBR Green Supermix | Bio-Rad Laboratories | 1725121 or 1725122 | |
| Spectrophotometer | brand not critical | ||
| 0.45 μm PVDF Filter | brand not critical | ||
| 25 ml Seringe | brand not critical | ||
| 10 cm Tissue Culture Plates | brand not critical | ||
| 6-well Tissue Culture Plates | brand not critical | ||
| S1000 Thermal Cycler | Bio-Rad Laboratories | ||
| C1000 Touch Thermal Cycler | Bio-Rad Laboratories | for qPCR | |
| Vortex Mixer | brand not critical | ||
| Dry Block Heater or Thermomixer | brand not critical | ||
| Microcentrifuge | brand not critical | ||
| Gel electrophoresis system with power supply | brand not critical | ||
| Magnet stand | for purification of DNA with SPRI beads; should hold 1.5-2 ml tubes; brand not critical | ||
| UV transilluminator | brand not critical | ||
| E-gel electrophoresis system | Life Technologies | G6400, G6500, G6512ST |
Riferimenti
- van Steensel, B., Delrow, J., Henikoff, S. Chromatin profiling using targeted DNA adenine methyltransferase. Nat Genet. 27, 304-308 (2001).
- van Steensel, B., Henikoff, S. Identification of in vivo DNA targets of chromatin proteins using tethered dam methyltransferase. Nat Biotechnol. 18, 424-428 (2000).
- Fu, A. Q., Adryan, B. Scoring overlapping and adjacent signals from genome-wide ChIP and DamID assays. Mol Biosyst. 5, 1429-1438 (2009).
- Guelen, L. Domain organization of human chromosomes revealed by mapping of nuclear lamina interactions. Nature. 453, 948-951 (2008).
- Kalverda, B., Pickersgill, H., Shloma, V. V., Fornerod, M. Nucleoporins directly stimulate expression of developmental and cell-cycle genes inside the nucleoplasm. Cell. 140, 360-371 (2010).
- Kubben, N. Mapping of lamin A- and progerin-interacting genome regions. Chromosoma. 121, 447-464 (2012).
- Steglich, B., Filion, G. J., van Steensel, B., Ekwall, K. The inner nuclear membrane proteins Man1 and Ima1 link to two different types of chromatin at the nuclear periphery in S. pombe. Nucleus. 3, 77-87 (2012).
- Harr, J. C. Directed targeting of chromatin to the nuclear lamina is mediated by chromatin state and A-type lamins. J Cell Biol. 208, 33-52 (2015).
- Gonzalez-Aguilera, C. Genome-wide analysis links emerin to neuromuscular junction activity in Caenorhabditis elegans. Genome Biol. 15, R21(2014).
- Wu, F., Yao, J. Spatial compartmentalization at the nuclear periphery characterized by genome-wide mapping. BMC Genomics. 14, 591(2013).
- Filion, G. J. Systematic protein location mapping reveals five principal chromatin types in Drosophila cells. Cell. 143, 212-224 (2010).
- Vogel, M. J. Human heterochromatin proteins form large domains containing KRAB-ZNF genes. Genome Res. 16, 1493-1504 (2006).
- Venkatasubrahmanyam, S., Hwang, W. W., Meneghini, M. D., Tong, A. H., Madhani, H. D. Genome-wide, as opposed to local, antisilencing is mediated redundantly by the euchromatic factors Set1 and H2A.Z. Proc Natl Acad Sci U S A. 104, 16609-16614 (2007).
- Shimbo, T. MBD3 localizes at promoters, gene bodies and enhancers of active genes. PLoS Genet. 9, e1004028(2013).
- Orian, A. Genomic binding by the Drosophila Myc, Max, Mad/Mnt transcription factor network. Genes Dev. 17, 1101-1114 (2003).
- Artegiani, B. Tox: a multifunctional transcription factor and novel regulator of mammalian corticogenesis. EMBO J. , (2014).
- Schuster, E. DamID in C. elegans reveals longevity-associated targets of DAF-16/FoxO. Mol Syst Biol. 6, 399(2010).
- Bianchi-Frias, D. Hairy transcriptional repression targets and cofactor recruitment in Drosophila. PLoS Biol. 2, e178(2004).
- Woolcock, K. J., Gaidatzis, D., Punga, T., Buhler, M. Dicer associates with chromatin to repress genome activity in Schizosaccharomyces pombe. Nat Struct Mol Biol. 18, 94-99 (2011).
- Luo, S. D., Shi, G. W., Baker, B. S. Direct targets of the D. melanogaster DSXF protein and the evolution of sexual development. Development. 138, 2761-2771 (2011).
- Germann, S., Gaudin, V. Mapping in vivo protein-DNA interactions in plants by DamID, a DNA adenine methylation-based method. Methods Mol Biol. 754, 307-321 (2011).
- Zhang, X. The Arabidopsis LHP1 protein colocalizes with histone H3 Lys27 trimethylation. Nat Struct Mol Biol. 14, 869-871 (2007).
- Orian, A. Chromatin profiling, DamID and the emerging landscape of gene expression. Curr Opin Genet Dev. 16, 157-164 (2006).
- Vogel, M. J., Peric-Hupkes, D., van Steensel, B. Detection of in vivo protein-DNA interactions using DamID in mammalian cells. Nat Protoc. 2, 1467-1478 (2007).
- Greil, F., Moorman, C., van Steensel, B. DamID: mapping of in vivo protein-genome interactions using tethered DNA adenine methyltransferase. Methods Enzymol. 410, 342-359 (2006).
- de Wit, E., Greil, F., van Steensel, B. Genome-wide HP1 binding in Drosophila: developmental plasticity and genomic targeting signals. Genome Res. 15, 1265-1273 (2005).
- DamID mammalian vectors. , Available from: http://research.nki.nl/vansteensellab/Mammalian_plasmids.htm (2015).
- Illumina TruSeq adaptors & PCR primers. , Available from: https://ethanomics.wordpress.com/chip-seq-library-construction-using-the-illumina-truseq-adapters/ (2015).
- Optimization of PCR cycles for NGS. , Available from: https://ethanomics.wordpress.com/ngs-pcr-cycle-quantitation-protocol/ (2015).
- Bernstein, B. E. The NIH Roadmap Epigenomics Mapping Consortium. Nat Biotechnol. 28, 1045-1048 (2010).
- Encode Project Consortium. A user's guide to the encyclopedia of DNA elements (ENCODE). PLoS Biol. 9, e1001046(2011).
- Asp, P. Genome-wide remodeling of the epigenetic landscape during myogenic differentiation. Proc Natl Acad Sci U S A. 108, E149-E158 (2011).
- Hoppe, P. S., Coutu, D. L., Schroeder, T. Single-cell technologies sharpen up mammalian stem cell research. Nat Cell Biol. 16, 919-927 (2014).
- Avital, G., Hashimshony, T., Yanai, I. Seeing is believing: new methods for in situ single-cell transcriptomics. Genome Biol. 15, 110(2014).
- Navin, N. E. Cancer genomics: one cell at a time. Genome Biol. 15, 452(2014).
- Saliba, A. E., Westermann, A. J., Gorski, S. A., Vogel, J. Single-cell RNA-seq: advances and future challenges. Nucleic Acids Res. 42, 8845-8860 (2014).
- Nagano, T. Single-cell Hi-C reveals cell-to-cell variability in chromosome structure. Nature. 502, 59-64 (2013).
- Kind, J. Single-cell dynamics of genome-nuclear lamina interactions. Cell. 153, 178-192 (2013).
- Southall, T. D. Cell-type-specific profiling of gene expression and chromatin binding without cell isolation: assaying RNA Pol II occupancy in neural stem cells. Dev Cell. 26, 101-112 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon