Method Article
-DamID siguientes: Genoma de toda la cartografía de la proteína-ADN Interacciones de alto rendimiento de secuenciación de fragmentos de ADN adenina-metilado
En este artículo
Resumen
Se describe en este documento un ensayo mediante el acoplamiento de identificación de ADN metiltransferasa adenina (DamID) a alto rendimiento de secuenciación (DamID-ss). Este método mejorado proporciona una mayor resolución y un rango dinámico más amplio, y permite el análisis de datos DamID-Seq en conjunción con otros datos de secuenciación de alto rendimiento tales como chip-ss, RNA-Seq, etc.
Resumen
The DNA adenine methyltransferase identification (DamID) assay is a powerful method to detect protein-DNA interactions both locally and genome-wide. It is an alternative approach to chromatin immunoprecipitation (ChIP). An expressed fusion protein consisting of the protein of interest and the E. coli DNA adenine methyltransferase can methylate the adenine base in GATC motifs near the sites of protein-DNA interactions. Adenine-methylated DNA fragments can then be specifically amplified and detected. The original DamID assay detects the genomic locations of methylated DNA fragments by hybridization to DNA microarrays, which is limited by the availability of microarrays and the density of predetermined probes. In this paper, we report the detailed protocol of integrating high throughput DNA sequencing into DamID (DamID-seq). The large number of short reads generated from DamID-seq enables detecting and localizing protein-DNA interactions genome-wide with high precision and sensitivity. We have used the DamID-seq assay to study genome-nuclear lamina (NL) interactions in mammalian cells, and have noticed that DamID-seq provides a high resolution and a wide dynamic range in detecting genome-NL interactions. The DamID-seq approach enables probing NL associations within gene structures and allows comparing genome-NL interaction maps with other functional genomic data, such as ChIP-seq and RNA-seq.
Introducción
ADN metiltransferasa identificación adenina (DamID) 1,2 es un método para detectar la proteína-DNA in vivo y es un enfoque alternativo a la cromatina immunoprecipitation (CHIP) 3. Se utiliza una cantidad relativamente baja de células y no requiere la reticulación química de la proteína con el ADN o un anticuerpo altamente específico. Este último es particularmente útil cuando la proteína diana está vagamente o indirectamente asociada con el ADN. DamID ha sido utilizado con éxito para mapear los sitios de unión de una variedad de proteínas, incluyendo proteínas de la envoltura nuclear, la cromatina asociada 4-10 11-13 proteínas, enzimas modificadoras de la cromatina 14, factores de transcripción y co-factores 15-18 y maquinarias RNAi 19. El método es aplicable en múltiples organismos incluyendo S. cerevisiae 13, S. pombe 7, C. elegans 9,17, D. melanogaster 5,11,18,20, A. thaliana 21,22, así como de ratón y células humanas líneas 6,8,10,23,24.
El desarrollo del ensayo DamID se basó en la detección específica de fragmentos de ADN adenina-metilado en células eucariotas que carecen de adenina endógena metilación 2. Una proteína de fusión expresada, que consiste en la proteína de unión a ADN de interés y E. ADN metiltransferasa coli adenina (Dam), puede metilar la base adenina en las secuencias GATC que están en proximidad espacial (más significativamente dentro de 1 kb y hasta aproximadamente 5 kb) a los sitios de unión de la proteína en el genoma 2. Los fragmentos de ADN modificados se pueden amplificar específicamente y se hibridaron a microarrays para detectar los sitios de unión genómicas de la proteína de interés 1,25,26. Este método original DamID estaba limitada por la disponibilidad de microarrays y la densidad de sondas predeterminados. Por ello, hemos integrado secuenciación de alto rendimiento ena DamID 10 y designado el método DamID-ss. El gran número de cortos lee genera a partir DamID-ss permite la localización precisa de la proteína-DNA del genoma de ancho. Encontramos que DamID-ss proporciona una mayor resolución y un rango dinámico más amplio que DamID de microarrays para el estudio de la lámina genoma nuclear (NL) 10 asociaciones. Este método mejorado permite sondear asociaciones NL dentro de las estructuras de genes 10 y facilita las comparaciones con otros datos de secuenciación de alto rendimiento, tales como chip-ss y ARN-ss.
El protocolo DamID-ss aquí descrito fue desarrollado inicialmente para el mapeo de asociaciones del genoma NL 10. Hemos generado una proteína de fusión mediante la inmovilización del ratón o Lamin B1 humano a E. coli DNA metiltransferasa adenina y se prueba el protocolo en 3T3 fibroblastos embrionarios de ratón, ratón C2C12 mioblastos 10 y IMR90 fibroblastos de pulmón fetal humano (datos no publicados). En este protocolo, empezamos con constructing vectores y expresar proteínas de fusión Dam-atados por la infección lentiviral en células de mamíferos 24. A continuación, se describen los protocolos detallados de amplificación de fragmentos de ADN adenina-metilado y la preparación de bibliotecas de secuenciación que deberían ser aplicables en otros organismos.
Protocolo
1. Generación y expresión de proteínas de fusión y Dam gratuito Proteínas
- Clone proteína de interés en el vector DamID.
- Amplificar cDNA de proteína de interés (POI) utilizando la ADN polimerasa de alta fidelidad deseada y cebadores apropiados de acuerdo con el protocolo del fabricante. Experimentalmente determinar las condiciones óptimas de amplificación para asegurar la amplificación adecuada de insertos.
- Ejecutar un gel de agarosa y purificar cDNA amplificado de POI por el kit de extracción de gel de acuerdo con el protocolo del fabricante.
- CDNA clon del PDI en el vector pDONR201 usando BP Clonase II de acuerdo con el protocolo del fabricante.
- Clonar el ADNc de PDI de la vector donante en el vector de destino pLGW-RFC1-V5-EcoDam o el destino vector pLGW-EcoDam-V5-RFC1 27 usando LR Clonase II de acuerdo con el protocolo del fabricante, dependiendo de la dirección deseada de la fusión de POI a la N-terminal o THe C-terminal de la E. ADN metiltransferasa coli adenina (EcoDam) 27.
- Verificar mediante secuenciación que el cDNA clonado tiene una secuencia correcta y formas en el marco de fusión para EcoDam.
- Generar las existencias lentiviral.
- Generar las existencias de lentivirus que expresan Dam-V5-PDI y V5-Dam (a partir del vector pLGW-V5-EcoDam 27), utilizando sistemas de expresión lentiviral. Utilice el procedimiento de transfección con interés, según el protocolo del fabricante.
- Utilice células 293T y lipofección para generar stocks lentiviral de acuerdo con el protocolo del fabricante para la transfección.
- Incluya el paso filtro de 0,45 micras PVDF.
- Generar las existencias de lentivirus que expresan Dam-V5-PDI y V5-Dam (a partir del vector pLGW-V5-EcoDam 27), utilizando sistemas de expresión lentiviral. Utilice el procedimiento de transfección con interés, según el protocolo del fabricante.
- Infectar las células con lentivirus.
- El día antes de la infección (día 0), pase células adherentes cultivadas en medios de cultivo apropiados para este tipo de células a una placa de cultivo tisular de 6 pocillos utilizando el mismo medio de crecimiento sin antibióticos aalcanzar 50% de confluencia en el día de la infección. Colocar las células en una incubadora a 37 °.
- En el día de la infección (día 1), retire 2 crioviales tanto Dam-V5-PDI y V5-Dam sobrenadantes lentivirales de un congelador a -80ºC y el lugar en un baño de agua a 37 ° C para descongelar.
- Retire el medio de crecimiento de las células y reemplazar con 0,5 ml de medio de crecimiento fresco sin antibióticos.
- Añadir 1 ml de la lentivirus descongelado a cada pocillo (2 pozos con V5-Dam y 2 pozos con Dam-V5-POI). Añadir 1 ml de medio de crecimiento sin antibióticos a las 2 restantes pozos de esto servirá como un control negativo. Agite suavemente la placa de 6 pocillos para mezclar y colocar de nuevo en una incubadora a 37 ° O / N.
- El día después de la infección (día 2), retire las suspensiones virales de las células y reemplazar con 2 ml de medio de crecimiento sin antibióticos. Coloque las células de espalda en una incubadora a 37 ° durante 48 horas.
- Aislar gDNA.
- Aspirar los medios de cada pocillo y decélulas tacómetro utilizando 250 l 0,05% de tripsina-EDTA. Incubar a 37 ° C durante 2 min.
- Lavar las células de la placa con 1 ml de medio de crecimiento y células de pipeta de cada pocillo en un tubo de microcentrífuga de 1,5 ml. Se precipitan las células por centrifugación a 200 xg durante 5 min a TA.
- Lavar las células sedimentadas con 500 l de PBS y centrifugar a 200 xg durante 2 min a TA.
- Resuspender las células sedimentadas en 200 l de PBS.
- Aislar gDNA por la sangre y el tejido kit de acuerdo con el protocolo del fabricante. Eluir gDNA en 200 l de tampón AE y determinar la concentración mediante la medición de OD 260 utilizando un espectrofotómetro.
Nota: gDNA de las células infectadas o no infectadas simuladas se puede aislar como controles negativos. Precipitar gDNA a concentraciones más altas para el almacenamiento a largo plazo.- Añadir 3 volúmenes de etanol 100% y 0,1 volúmenes de acetato de sodio 3 M (pH 5,5), y se mezcla por inversión de los tubos 4-6 veces.
- Conservar a -20 ° CO / N.
- Se centrifuga a 16.000 xg durante 15 min a 4 ° C.
- Retire con cuidado el sobrenadante. Lavar pellets con 70% (volumen / volumen) de etanol y se centrifuga a 16.000 xg durante 5 min a 4 ° C.
- Retire cuidadosamente etanol y permitir pellets de secar al aire durante 3 min a TA.
- Disolver gDNA en T 10 E 0,1 (pH 7,5) hasta aproximadamente 1 g / l. Piscina gDNA de cada muestra experimental o el control negativo y medir la concentración. Almacenar a -20 ° C.
-Metilados Adenina 2. amplificar fragmentos de ADN
- Digesto gDNA con DpnI que sólo corta GATCs adenina-metilado.
- Configurar una reacción en hielo con 2,5 mg gDNA, 1 l de 10x tampón, 0,5 l DpnI (20 U / l) y se llenan de H 2 O hasta un volumen total de 10 l. Para cada muestra gDNA, preparar tres reacciones - uno sin DpnI ("no DpnI", reemplace DpnI con 0,5 l H 2 O) y dos wITH DpnI ("con DpnI").
- Recopilación de O / N a 37 ° C e inactivar DpnI a 80 ° C durante 20 minutos.
- Ligar adaptadores DamID.
- Preparar adaptadores DamID.
- Resuspender cada uno de los dos oligos adaptadores DamID 24 en H 2 O a una concentración final de 100 mM.
- Combinar volúmenes iguales de los dos oligos adaptadores DamID, mezclar y colocar en un tubo cerrado herméticamente. Sellar el tubo con Parafilm, sentarse en un estante y colóquelo en un vaso lleno de agua a 90 ° C. Mantenga el nivel de agua por debajo de la tapa del tubo (para evitar el agua entrar en el tubo) pero por encima de la superficie de la mezcla de oligo.
- Deje enfriar el agua a temperatura ambiente por lo que el recocido adaptadores lentamente.
- Alícuota los adaptadores recocidos (50 m) y almacenar a -20 ° C.
- Configurar una reacción en hielo. En cada tubo de 2.1.2, añadir 6,2 l H 2 O, 2 l de 10x tampón de ligación, 0,8 l 50 mM DamID adaptors (descongelados en hielo) y 1 l de ADN ligasa de T4 (5 U / l). El volumen total es de 20 l. En uno de los dos "con DPNI" tubos, reemplace ligasa con 1 l H2O ("no ligasa") Tenga en cuenta que cada muestra gDNA tiene dos controles negativos -. "No DpnI" y "no ligasa".
- Ligar O / N a 16 ° C y inactivar la ligasa a 65 ° C durante 10 minutos.
- Preparar adaptadores DamID.
- Digerir el ADN con DpnII para destruir fragmentos que contienen GATCs no metilados.
- Configurar una reacción en hielo. En cada tubo de 2.2.3, añadir 24 l de H2O, tampón 5 l 10x DpnII y 1 l DpnII (10 U / l). El volumen total es de 50 l.
- Digesto a 37 ° C durante 2-3 horas e inactivar DpnII a 65 ° C durante 20 minutos.
- Amplificar fragmentos de ADN adenina-metilado.
- Configurar una reacción en hielo con 5 l DpnII digerir desde 2.3.2, 5 μl 10x tampón de PCR, 12,5 l 5 M DamID PCR imprimación 24, 4 l 10 mM dNTP mix, 1 l 50x mezcla polimerasa y 22,5 l H2O El volumen total es de 50 l.
- Ejecutar la PCR de la siguiente manera: 68 ° C durante 10 min; 94 ° C durante 1 min, 65 ° C durante 5 min, 68 ° C durante 15 min; 4 ciclos de 94 ° C durante 1 min, 65 ° C durante 1 min, 68 ° C durante 10 min; 17 ciclos de 94 ° C durante 1 min, 65 ° C durante 1 min, 68 ° C durante 2 min.
- Analizar 5 productos l de cada reacción de PCR en un gel de agarosa al 1%. Productos de PCR deberían aparecer como una mancha que van desde 0,2 a 2 kb (Figura 2). Los controles "sin DpnI" y "no ligase" no deben tener o claramente menos amplificación.
- Si el resultado del paso 2.4.3 es satisfactoria, repetir los pasos 2.4.1-2.4.3 con dos o tres reacciones para la muestra experimental y una reacción para cada uno de los dos controles negativos.
- Piscina y purificar los productos de PCR de la mismamuestra experimental utilizando kits de purificación de PCR o fase sólida Inmovilización Reversible (SPRI) perlas de acuerdo con el protocolo del fabricante. No purificar "no DpnI" o controles "sin ligasa". Eluir el ADN con tampón EB.
- Medir la concentración del ADN purificado mediante la medición de OD 260 utilizando un espectrofotómetro, que debe ser alrededor de 0,1 g / l o superior. Recoger un mínimo de 10 g de ADN para cada muestra. Si el uso de kits de purificación de PCR, purifica cada 50 l productos de PCR con una columna, eluir en 30 l de tampón EB y poner en común los eluidos.
- Digerir el ADN con DpnII para prevenir cebadores de PCR DamID de ser secuenciado.
- Configurar una reacción en hielo con 5 g de ADN purificado a partir de 2.4.6, 5 l de 10x tampón DpnII, 1 l DpnII (10 U / l) y se llenan de H 2 O hasta un volumen total de 50 l. Preparar dos o tres reacciones para cada muestra.
- Digerir a 37 ° C durante 2-3 horase inactivar DpnII a 65 ° C durante 20 min.
- Piscina y purificar los digestos de la misma muestra con los kits de purificación de PCR o perlas SPRI de acuerdo con el protocolo del fabricante. Eluir el ADN con tampón EB.
- Medir la concentración del ADN purificado que debe estar alrededor de 0,06 g / l o superior. Recoger un mínimo de 6 g de ADN para cada muestra. Si el uso de kits de purificación de PCR, purifica cada 50 l digerir con una columna, eluir en 30 l de tampón EB y poner en común los eluidos.
3. Preparación Biblioteca de secuenciación de alto rendimiento
- Fragmento de ADN
- Experimentalmente determinar el tiempo de digestión apropiada para cada nuevo lote de ADN de doble cadena Fragmentase. A medida que la actividad de la enzima puede disminuir con el tiempo, repetir la prueba antes de realizar un nuevo experimento. Para guardar ADN a partir de 2.5.4 para la fragmentación real, utilizar productos de PCR de metilo purificado a partir de 2.4.6 o ADN extra de EXPERIME anteriornoches en este paso.
- Configurar un maestro de la mezcla con el ADN 6 mg, 12 l de 10x tampón Fragmentase y rellenar con H 2 O hasta un volumen total de 114 l.
- Vortex la Fragmentase Stock vial durante 3 segundos, añadir 6 l en la mezcla maestra y agitar la mezcla maestra durante 3 s. El volumen total es de 120 l.
- Alícuota de 20 l de la mezcla maestra a cada uno de 5 tubos nuevos. Incubar los 6 tubos a 37 ° C durante 5 a 55 min a un incremento de 10 min. Añadir 5 l 0,5 M EDTA para detener la reacción.
- Analizar 12,5 l de digerir (0,5 g de ADN) de cada reacción, así como 0,5 g de ADN no digerido en un gel de agarosa (Figura 3). Determinar el tiempo mínimo (T 0.2kb) necesaria para digerir la mayoría de la mancha a alrededor de 0,2 kb. Seleccione 6 duraciones de tiempo entre 5 min y T 0.2kb (incluyendo 5 min y T 0.2kb) con incrementos iguales para la fragmentación real.
- Configure los fragmen realestación como se describe en 3.1.1.1-3.1.1.3. Incubar reacciones en 37 ° C para las duraciones de tiempo determinados en 3.1.1.4.
- Piscina 6 reacciones y purificar los digiere con kits de purificación de PCR o perlas SPRI acuerdo con el protocolo del fabricante. Eluir el ADN en tampón EB 51 l. El eluato final es ~ 50 l.
- Experimentalmente determinar el tiempo de digestión apropiada para cada nuevo lote de ADN de doble cadena Fragmentase. A medida que la actividad de la enzima puede disminuir con el tiempo, repetir la prueba antes de realizar un nuevo experimento. Para guardar ADN a partir de 2.5.4 para la fragmentación real, utilizar productos de PCR de metilo purificado a partir de 2.4.6 o ADN extra de EXPERIME anteriornoches en este paso.
- Reparación extremos del ADN fragmentado
- Configurar una reacción en hielo con el eluato a partir de 3.1.3, 25 l H 2 O, 10 l de 10x tampón de ligasa T4 DNA con ATP 10 mM, 4 l 10 mM dNTP mix, 5 l de ADN polimerasa de T4 (3 U / l) , 1 l de ADN polimerasa de Klenow (5 U / l), y 5 l de polinucleótido quinasa de T4. El volumen total es de 100 microlitros. Mezclar bien con la pipeta. Evite espuma y burbujas.
- Incubar a 20 ° C durante 30 min.
- Se purifica el ADN con kits de purificación de PCR o perlas SPRI de acuerdo con el protocolo del fabricante. Eluir el ADN en tampón EB 33 l. El eluato final es ~ 32 & #181; l.
- Añadir voladizos "A"
- Configurar una reacción en hielo con el eluato de 3.2.3, 5 l 10x tampón Klenow, 10 l 1 mM dNTP, y 3 l Klenow (3 '→ 5' exo) (5 U / l). El volumen total es de 50 l. Mezclar bien con la pipeta. Evite espuma y burbujas.
- Incubar a 37 ° C durante 30 min.
- Se purifica el ADN con kits de purificación de PCR o perlas SPRI de acuerdo con el protocolo del fabricante. Eluir el ADN en tampón EB 22 l. El eluato final es ~ 21 l.
- Se ligan los adaptadores de secuenciación
- Preparar adaptadores de secuenciación 28.
- Resuspender cada oligo adaptador a una concentración de 100 mM en 10 mM Tris-Cl (pH 7,8), EDTA 0,1 mM (pH 8,0) y NaCl 50 mM.
- Mezclar volúmenes iguales de el adaptador universal 28 y un adaptador indexada 28.
- Recocido de los adaptadores en un ciclador térmico con el siguienteprograma: 95 ° C durante 2 min; 140 ciclos durante 30 segundos a partir de 95 ° C y la disminución de 0,5 ° C por cada ciclo; mantener a 4 ° C.
- Adaptadores alícuotas y almacenar a -20 ° C.
- Configurar una reacción en hielo con el eluato de 3.3.3, 25 l 2x tampón de ligación, 1,5 l de 50 mM recocidos adaptadores de secuenciación (descongelado en el hielo) de 3.4.1.4 y 2.5 l de ADN ligasa de T4. Mezclar bien con la pipeta. Evite espuma y burbujas. Si se desea secuenciación multiplex, utilice un adaptador indexada diferente para cada muestra.
- Incubar a temperatura ambiente durante 1 hr.
- Se purifica el ADN con kits de purificación de PCR o perlas SPRI de acuerdo con el protocolo del fabricante. Eluir el ADN en tampón EB 24 l. El eluato final es ~ 23 l.
- Preparar adaptadores de secuenciación 28.
- Convertir adaptadores en forma de Y a dsDNA para permitir determinar con precisión el tamaño de fragmento de ADN 28
- Configurar una reacción en hielo con el eluato de 3.4.4, 12.5 l H 2 O, 1 l 25 mM imprimación 1 28, 1 l 25 mM imprimación 2 28, 1,5 l de 10 mM dNTP, 10 l 5x PCR Buffer y 1 l de ADN polimerasa (1 U / l).
- Ejecutar la PCR de la siguiente manera: 95 ° C durante 3 min; 5 ciclos de 98 ° C durante 15 seg, 63 ° C durante 30 seg, 72 ° C durante 30 s; 72 ° C durante 1 min; 4 ° C en espera.
- Se purifica el ADN con kits de purificación de PCR o perlas SPRI de acuerdo con el protocolo del fabricante. Eluir el ADN en tampón EB 11 l. El eluato final es ~ 10 l.
- Tamaño seleccionar la biblioteca
- Preparar un gel de agarosa al 2% con tampón TAE 1x. Añadir bromuro de etidio (PRECAUCIÓN!) A una concentración final de 500 ng / ml cuando la solución TAE-agarosa derretida se ha enfriado para evitar la inhalación de bromuro de etidio. Asegúrese de tener suficientes carriles para todas las muestras, escalera de ADN y carriles vacíos.
- Añadir 8 l de tinte 6x carga hasta el eluato de 3.5.3.
- Prepare escalera de ADN por mezcla de 1 kb además de escalera de ADN (1,0 mg / l), colorante de carga 6x y H 2 O en una proporción de 1: 1: 4.
- Cargar 6 l escalera de ADN, las muestras de 3.6.2 y otra escalera de ADN 6 l, cada uno en un carril separado y con al menos un carril vacío de las muestras adyacentes / escaleras.
- Correr el gel a 120 V durante 60 minutos.
- Ver el gel en un transiluminador UV (minimizar el tiempo de exposición a UV). Use gafas de seguridad y una careta. Asegúrese de que al menos una de las escaleras de ADN corren bien con espacios apropiados (suficiente para extirpar 3 rodajas de gel) entre 300 pb y 400 pb bandas. Estrecho espaciamiento aumenta las dificultades en la escisión de múltiples cortes de gel, mientras amplio espaciamiento aumenta el volumen de rodajas de gel extirpados.
- Use un nuevo bisturí o una hoja de afeitar para cada carril. Impuestos Especiales tres rodajas de gel finas entre 300 y 400 pb de cada carril (Figura 4) y colocarlos cada uno en un tubo de microcentrífuga. Mantenga el volumen de cada s gelpiojos lo más bajo posible (<100 l).
- Medir el volumen de cada rodaja de gel (1 l gel pesa aproximadamente 1 mg), agregar volúmenes de tampón QG 6x y se incuba a 50 ° C.
- Vortex la mezcla QG-gel cada 2-3 minutos hasta que el corte de gel se haya disuelto completamente. Añadir los volúmenes de gel 2x de isopropanol y mezclar.
- A partir de este paso, siga el protocolo a partir de kits de extracción de gel. Eluir el ADN en tampón EB 51 l. El eluato final es ~ 50 l.
- Enriquecer adaptador modificado fragmentos de ADN de secuenciación
- Optimizar el número de ciclos de PCR 29.
- Configurar una reacción en hielo con 1 l eluato de 03.06.10, 1 l 25 mM imprimación 1 28, 1 l 25 mM imprimación 2 28, 7 l H 2 O y 10 l SYBR Green Supermix. El volumen total es de 20 l.
- Ejecutar la qPCR de la siguiente manera: 95 ° C durante 3 min; 20 ciclos de 95 ° C durante 30 segundos, 63 ° C durante 30 segundos, 72 ° C durante 30 segundos, la placa de leer.
- Analizar los datos utilizando un software de análisis de qPCR y determinar el ciclo de cuantificación (Cq) o ciclo umbral (Ct) para cada muestra utilizando el protocolo del fabricante. Continuar con las muestras que tienen Cq / Ct ≤ 14. Utilice el Cq máxima (CQ 0) menos 1 (redondeado al siguiente número entero superior) como el número final de ciclo de PCR (N PCR).
- Ajustar las cantidades de plantillas de ADN de manera que diferentes muestras se amplificaron a cantidades aproximadamente iguales después de ejecutar el mismo número de ciclos de PCR. Utilice 8 l plantilla para la muestra con la más alta Cq (Cq 0), y calcular el volumen de plantilla de otras muestras con la siguiente fórmula:
Vol i = 8 x 1,8 Cq i -Cq 0
- Establecer las reacciones de PCR en el hielo con los volúmenes de plantilla calculados a partir 3.7.1.4: 1 l 25 mM imprimación 1 28, 1 l 25 mM imprimación 2 28, 1.5l 10 mM dNTP, 10 l de 5x tampón, 1 l de ADN polimerasa (1 U / l) y rellenar con H 2 O hasta un volumen total de 50 l.
- Ejecutar la PCR de la siguiente manera: 95 ° C durante 45 seg; N ciclos de PCR (determinado en 3.7.1.3) de 98 ° C durante 15 seg, 63 ° C durante 30 seg, 72 ° C durante 30 s; 72 ° C durante 1 min; 4 ° C en espera.
- Analizar 5 productos l de PCR en un gel de agarosa al 2% (Figura 5A). Una banda clara "single" indica que los fragmentos de ADN amplificados están dentro de un rango de longitud limitada y de que la biblioteca de ADN puede ser objeto de un análisis adicional.
- Repetir una reacción para muestras seleccionadas y agrupar los bancos de ADN amplificados a partir de la misma muestra.
- Purificar bibliotecas de ADN seleccionados para la secuenciación.
- Si dímeros de cebadores / adaptador no son visibles en 3.7.4, purificar las bibliotecas de ADN con kits de purificación de PCR o perlas SPRI acuerdo con el protocolo del fabricante. Eluir el ADN en tampón EB 21 l. loseluato final es ~ 20 l. Si las instalaciones de secuenciación del usuario tiene instrucciones específicas sobre el tampón de elución, el volumen final, etc., preparar muestras en consecuencia.
- Si dímeros de cebadores / adaptadores son claramente visibles en 3.7.4, purificar las bibliotecas de la siguiente manera.
- ADN Cargar desde 3.7.5 y el ADN de escalera en un gel de agarosa al 2% prefabricados. Las muestras se pueden purificar como se describe en 3.7.6.1 para reducir el volumen o puede ser cargado en múltiples pozos. Colocar el gel de agarosa en un sistema de alimentación adecuado. Deje que el gel se ejecute durante 15 min.
- El uso de un transiluminador apropiada, cortar la banda deseada con un limpio bisturí / navaja para cada muestra y colocar la porción de gel en un tubo de microcentrífuga de 1,5 ml.
- Aislar el ADN usando kits de extracción de gel de acuerdo con el protocolo del fabricante con dos modificaciones: incubar mezcla tampón QG-gel en un termo-mezclador a 37 ° C y 1000 rpm durante 30 min, y añadir volúmenes de gel 2x de isopropanol.
- Optimizar el número de ciclos de PCR 29.
- Presentar bibliotecas de ADN purificados a un centro de secuenciación. Siga todas las normas de su institución.
Nota: Un análisis de la calidad por un Bioanalyzer (Figura 5B) se debe realizar antes de la secuenciación con el fin de determinar el rango de tamaño exacto y la concentración de una biblioteca de ADN.
Resultados
La proteína de fusión Dam-V5-LMNB1 se verificó a ser co-localizada con la proteína Lamin B endógeno mediante tinción de inmunofluorescencia (Figura 1).
La amplificación por PCR con éxito de fragmentos de ADN adenina-metilado es un paso clave para DamID-ss. Las muestras experimentales deberían amplificar un frotis de 0,2 - 2 kb mientras que los controles negativos (sin DpnI, sin ligasa o sin plantilla PCR) debería resultar en no-o-claramente menos de amplificación (Figura 2).
Los fragmentos de ADN metilado están en el intervalo de 0,2 a 2 kb, mientras que el tamaño del inserto deseado para una biblioteca de NGS es de 200 a 300 pb. Por lo tanto, es esencial para fragmentar los productos de PCR de metilo en el rango de tamaño adecuado. Sin embargo, se encontró que era poco práctico para romper simultáneamente fragmentos de ADN más grandes a un tamaño adecuados y mantener la mayoría de los fragmentos más pequeños de ADN intactos en una sola duración de fragmentación. Por lo tanto, se realizaron experimentos de tiempo para determinar el tiempo mínimo (T 0.2kb) necesarios para fragmento de 1 g de ADN a un frotis de centrado a 200 pb (Figura 3). Entonces 6 duraciones de tiempo en incrementos iguales fueron seleccionados entre 5 min y T 0.2kb para la fragmentación real. La actividad enzimática de la doble cadena de ADN Fragmentase puede variar de un lote a otro y puede disminuir con el tiempo, por lo que se recomienda repetir este paso para un nuevo lote de Fragmentase o después del almacenamiento durante un período de tiempo.
El tamaño del inserto deseado es entre 200 y 300 pb correspondiente a los fragmentos de ADN entre 300 y 400 pb (incluyendo 121 adaptadores de secuenciación pb) en el gel de agarosa. Tres rebanadas delgadas dentro de este rango fueron extirpados de cada muestra experimental para reducir el rango de tamaño de una biblioteca y aumentar la posibilidadla obtención de al menos una biblioteca de secuenciación cualificado (Figura 4).
Una alícuota de 5 l de cada biblioteca de ADN amplificado se analizó en el gel de agarosa para determinar qué biblioteca puede calificar para la secuenciación. Como se muestra en la Figura 5A, una banda única clara del mismo tamaño que la porción de gel escindido debe ser visible en el gel de agarosa (etapa 3.7.4). A continuación, las bibliotecas seleccionadas fueron examinados por un Bioanalyzer (Figura 5B) para determinar el rango de tamaño exacto y las concentraciones antes de la secuenciación. Si se desea, las bibliotecas de ADN amplificados pueden ser examinados directamente por un Bioanalyzer sin análisis en gel. Cuando varias bibliotecas son de buena calidad, se recomienda para secuenciar las bibliotecas de tamaño similar rangos para un par de experimentales (las células que expresan Dam-V5-POI) y control (células expresando V5-Dam) muestras.
El corto lee generada porsistemas de secuenciación fueron asignadas primera vuelta al genoma correspondiente. Únicamente alineado lecturas fueron luego se pasa a los análisis posteriores. Una tubería para procesar corta lee, construir un mapa interacción genoma NL y analizar las asociaciones de genes NL fueron descritos en detalle en nuestro trabajo anterior 10. Los resultados representativos se muestran en la Figura 6.

Figura 1. Validación de subnuclear localizaciones de las proteínas de fusión por la tinción de inmunofluorescencia. IMR90 células de fibroblastos de pulmón humano se transfectaron transitoriamente con un plásmido que expresa Dam-V5-LMNB1 y se tiñeron por anti-Lamin B (A, rojo) y anti-V5 (B, verde). (C) Combinar de imágenes en A y B. Haga clic aquí para ver una versión más grande de tsu figura.

Figura 2. PCR amplificar fragmentos de ADN adenina-metilado. Una alícuota de 5 l de cada reacción de PCR se analizó en un gel de agarosa al 1%. Una mancha que van de 0,2 a 2 kb se amplificó a partir de cada muestra experimental, pero no se observó amplificación en los controles negativos (sin DpnI en el paso 2.1, sin ligasa en el paso 2.2, o ningún molde de PCR). Haga clic aquí para ver una versión más grande de esta cifra.
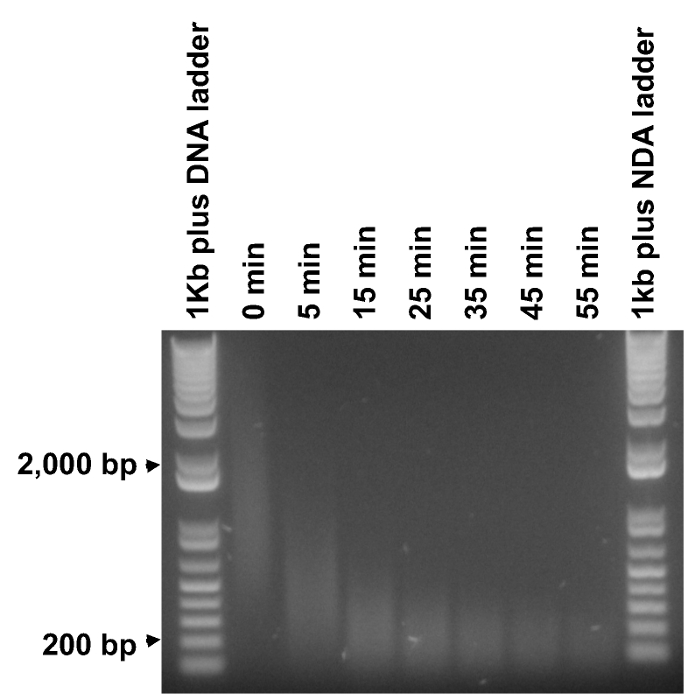
Figura 3. Determinación de duraciones óptimas de fragmentación. Los productos de PCR de metilo purificado estaban sujetos a la fragmentación para duraciones de tiempo a partir de 5 a 55 min en un incremento de 10 min (ADN sin digerir , Etiquetado como "0 min"). 1 g de escalera de ADN y 0,5 g de cada muestra de ADN fragmentado se analizaron en un gel de agarosa. El tiempo mínimo de digerir la mayoría de la frotis de ADN a alrededor de 200 pb se determinó que era alrededor de 35 min, por lo tanto, se seleccionaron seis duraciones de tiempo espaciados uniformemente entre 5 y 35 min (5 y 35 min incluido) para llevar a cabo la fragmentación real. Por favor haga clic aquí para ver una versión más grande de esta figura.

Figura 4. Tamaño de seleccionar las bibliotecas de ADN. Muestras de ADN de Dam-V5-LMNB1 y V5-Dam desde el paso 3.6 se corrieron en un gel de agarosa al 2%, y tres rodajas de gel de igual tamaño (L, M y H que corresponde a baja, media y alta en tamaño) fueron extirpados entre 300 y 400 pb como se muestra por las líneas amarillas. om / archivos / ftp_upload / 53620 / 53620fig4large.jpg "target =" _ blank "> Haga clic aquí para ver una versión más grande de esta figura.
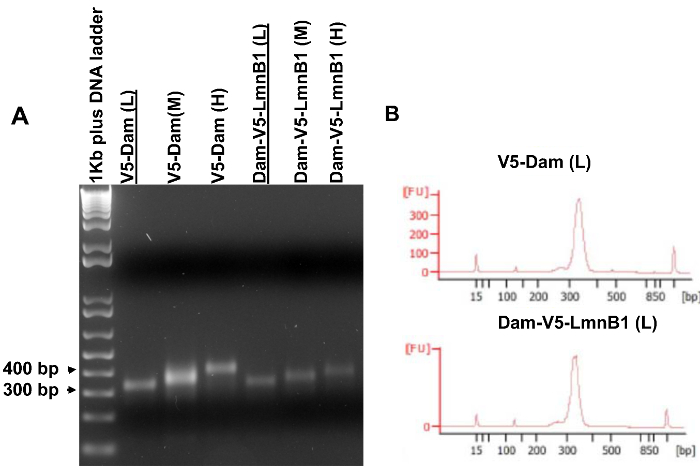
Figura 5. bibliotecas de ADN amplificado para secuenciación de alto rendimiento. (A) Amplified bibliotecas de ADN analizadas en un gel de agarosa. Plantillas de PCR se purificaron a partir de rodajas de gel mostrados en la Figura 4. (B) Bioanalyzer resultados de la muestra V5-Dam (L) y la muestra Dam-V5-LMNB1 (L) que se subraya en (A). Estas dos bibliotecas tenían los rangos de tamaño estrecha similares y, por tanto cualificado para alta secuenciación rendimiento. Por favor haga clic aquí para ver una versión más grande de esta figura.
jpg "/>
Figura 6. Los datos NGS se muestran en la UCSC genoma navegador. Cromosoma 1 del ratón se muestra como un ejemplo. Los datos de secuencia se producen a partir de mioblastos C2C12 de ratón 10. Tracks "MB.LmnB1.w2k" y "MB.Dam.w2k", que corresponde a los datos de las células que expresan Dam-V5-LMNB1 y V5-Dam, respectivamente, de la trama normalizado densidades de lectura (lee por kilobase por millón asignada únicamente lee, o RPKM ) en consecutivos que no se superponen las ventanas 2 kb a lo largo del cromosoma. Seguir parcelas "MB.log2FC.w2k" asociaciones genoma-NL, es decir log2 RPKM proporciones de LMNB1 más de la presa, en una resolución de 2 kb. Pista "MB.sLADs" pinta Lamina Asociados dominios basados en la secuenciación (slads, es decir, las regiones genómicas que tienen mayores densidades de lectura de LMNB1 sobre la presa con significación estadística) en, no slads negros en las regiones grises y indeterminados en blanco. Haga clic aquí para download este archivo.
Discusión
Whether Dam-tagged proteins retain the functions of endogenous proteins should be examined before a DamID-seq experiment. The subcellular localization of Dam-tagged nuclear envelope proteins should always be determined and compared with that of the endogenous proteins. For studying transcription factors, it is suggested to examine whether the Dam-fusion protein can rescue the functions of the endogenous protein in regulating gene expression. This functional test can be performed in organisms in which knockout mutants of endogenous DNA-binding proteins are available. Because advances in genome engineering have potentially allowed knocking out any endogenous gene of interest, functions of Dam-tagged DNA-binding proteins can be examined in cultured mammalian cells.
The critical step in this protocol is to successfully fragment the DpnII-digested DamID PCR products to around 200 bp. This step is designed to render the amplified adenine-methylated fragments to a narrow size range for sequencing and to randomize the starting nucleotides of the DNA fragments in a sequencing library. Inefficient fragmentation will leave the majority of the DNA fragments starting with GATC (the 5'-overhang from the second DpnII digestion), and will result in a much lower performance and yield or even a failure in Illumina sequencing. Other DNA fragmentation methods may be used as an alternative approach.
The resolution of DamID (and DamID-seq described here) is limited by the frequency of GATCs in the genome to be studied. Moreover, even with high throughput sequencing, the genomic localizations of a DNA-binding protein can only be mapped within two consecutive GATCs rather than to the actual DNA-binding sites.
Despite its limitation, the DamID assay has important advantages. Because DamID does not require highly-specific antibodies, it can be used to detect a subset of nuclear proteins that could be difficult to assay by ChIP (such as the nuclear envelope proteins). To study how these proteins regulate genome functions, it is important to integrate and cross-analyze their genome-wide localization data with the current epigenomic mapping data (such as data from the ENCODE and NIH Roadmap Epigenomics Projects 30,31). The DamID-seq approach provides both higher resolution and higher sensitivity than DamID by microarray and enables detecting differential NL-associations within gene structures 10. A combinatorial analysis of DamID-seq data, ChIP-seq data 32 and gene expression data has identified a class of NL-associated genes with distinct epigenetic and transcriptional features (data not published).
Another advantage of DamID is that it only requires a small number of cells. In recent years, there has been an explosion in single cell analysis of gene regulation 33,34. Although genome sequence 35, genome-wide gene expression 36 and chromatin conformation 37 can be assayed in a single cell, there has not been an available approach for detecting protein-DNA interactions genome-wide in a single cell. DamID-seq is a highly promising approach for this goal, and may complement the single cell imaging approach in detecting the dynamics of genome-NL interactions 38. One complication is that because the Dam-fusion protein is expressed at a much lower level than the endogenous protein in the DamID assay, it is possible that the Dam-fusion protein may only occupy a subset of genomic binding sites as compared to the endogenous protein.
DamID assay has mostly been used in cultured animal cells to detect protein-DNA interactions. Notably, developmental biologists have applied this assay in detecting protein-DNA interactions in specific cell types in vivo. For example, Dam-tagged RNA polymerase II was expressed specifically in Drosophila neural stem cells to detect their genome-wide occupancy without cell isolation 39. DamID-seq will be highly useful to study the genome-wide localizations of nuclear envelope proteins, transcription factors and chromatin regulators during development in animal models.
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
We thank Dr. Bas van Steensel for providing the DamID mammalian expression vectors. We thank Yale Center for Genome Analysis and the Genomics Core in Yale Stem Cell Center for advice on preparing NGS libraries and implementing high throughput DNA sequencing. This work was supported by the startup funding from Yale School of Medicine, a Scientist Development Grant from American Heart Association (12SDG11630031) and a Seed Grant from Connecticut Innovations, Inc. (13-SCA-YALE-15).
Materiales
| Name | Company | Catalog Number | Comments |
| ViraPower Lentiviral Expression Systems | Life Technologies | K4950-00, K4960-00, K4970-00, K4975-00, K4980-00, K4985-00, K4990-00, K367-20, K370-20, and K371-20 | |
| Gateway BP Clonase II Enzyme Mix | Life Technologies | 11789-020 | |
| Gateway LR Clonase II Enzyme Mix | Life Technologies | 11791-020 | |
| DNeasy Blood & Tissue Kit (250) | QIAGEN | 69506 or 69504 | |
| Gateway pDONR 201 | Life Technologies | 11798-014 | |
| 293T cells | American Type Culture Collection | CRL-11268 | |
| Trypsin-EDTA (0.05%), phenol red | Life Technologies | 25300-054 | |
| DMEM, high glucose, pyruvate | Life Technologies | 11995-065 | |
| Fetal Bovine Serum | Sigma | F4135 | |
| Tris | brand not critical | ||
| EDTA | brand not critical | ||
| 200 Proof EtOH | brand not critical | ||
| Isopropanol | brand not critical | ||
| Sodium Acetate | brand not critical | ||
| DpnI | New England Biolabs | R0176 | supplied with buffer |
| DamID adaptors "AdRt" and "AdRb" | Integrated DNA Technologies | sequences available in ref. 24; no phosphorylation of the 5' or 3' end to prevent self-ligation. | |
| T4 DNA Ligase | Roche Life Science | 10481220001 | supplied with buffer |
| DpnII | New England Biolabs | R0543 | supplied with buffer |
| DamID PCR primer "AdR_PCR" | Integrated DNA Technologies | sequences available in ref. 24 | |
| Deoxynucleotide (dNTP) Solution Set | New England Biolabs | N0446 | 100 mM each of dATP, dCTP, dGTP and dTTP |
| Advantage 2 Polymerase Mix | Clontech | 639201 | supplied with buffer |
| 1Kb Plus DNA Ladder | Life Technologies | 10787018 | 1.0 µg/µl |
| QIAquick PCR Purification Kit | QIAGEN | 28104 or 28106 | |
| MinElute PCR Purification Kit | QIAGEN | 28004 or 28006 | for an elution volume of less than 30 µl |
| SPRI beads / Agencourt AMPure XP | Beckman Coulter | A63880 | apply extra mixing and more elution time if less than 40 µl elution buffer is used |
| Buffer EB | QIAGEN | 19086 | |
| NEBNext dsDNA Fragmentase | New England Biolabs | M0348 | supplied with buffer |
| T4 DNA Ligase Reaction Buffer | New England Biolabs | B0202 | |
| T4 DNA Polymerase | New England Biolabs | M0203 | |
| DNA Polymerase I, Large (Klenow) Fragment | New England Biolabs | M0210 | |
| T4 Polynuleotide Kinase | New England Biolabs | M0201 | |
| Klenow Fragment (3’ → 5’ exo-) | New England Biolabs | M0212 | supplied with buffer |
| sequencing adaptors | Integrated DNA Technologies | sequences available in ref. 28 | |
| Quick Ligation Kit | New England Biolabs | M2200 | used in 3.4.3; supplied with Quick Ligation Reaction Buffer and Quick T4 DNA Ligase |
| sequencing primer 1 and 2 | Integrated DNA Technologies | sequences available in ref. 28 | |
| KAPA HiFi PCR Kit | Kapa Biosystems | KK2101 or KK2102 | supplied with KAPA HiFi DNA Polymerase, 5x KAPA HiFi Fidelity Buffer and 10 mM dNTP mix |
| agarose | Sigma Aldrich | A4679 | |
| ethidium bromide | Sigma Aldrich | E1510-10ML | 10 mg/ml |
| QIAquick Gel Extraction Kit | QIAGEN | 28704 or 28706 | |
| iTaq Universal SYBR Green Supermix | Bio-Rad Laboratories | 1725121 or 1725122 | |
| Spectrophotometer | brand not critical | ||
| 0.45 μm PVDF Filter | brand not critical | ||
| 25 ml Seringe | brand not critical | ||
| 10 cm Tissue Culture Plates | brand not critical | ||
| 6-well Tissue Culture Plates | brand not critical | ||
| S1000 Thermal Cycler | Bio-Rad Laboratories | ||
| C1000 Touch Thermal Cycler | Bio-Rad Laboratories | for qPCR | |
| Vortex Mixer | brand not critical | ||
| Dry Block Heater or Thermomixer | brand not critical | ||
| Microcentrifuge | brand not critical | ||
| Gel electrophoresis system with power supply | brand not critical | ||
| Magnet stand | for purification of DNA with SPRI beads; should hold 1.5-2 ml tubes; brand not critical | ||
| UV transilluminator | brand not critical | ||
| E-gel electrophoresis system | Life Technologies | G6400, G6500, G6512ST |
Referencias
- van Steensel, B., Delrow, J., Henikoff, S. Chromatin profiling using targeted DNA adenine methyltransferase. Nat Genet. 27, 304-308 (2001).
- van Steensel, B., Henikoff, S. Identification of in vivo DNA targets of chromatin proteins using tethered dam methyltransferase. Nat Biotechnol. 18, 424-428 (2000).
- Fu, A. Q., Adryan, B. Scoring overlapping and adjacent signals from genome-wide ChIP and DamID assays. Mol Biosyst. 5, 1429-1438 (2009).
- Guelen, L. Domain organization of human chromosomes revealed by mapping of nuclear lamina interactions. Nature. 453, 948-951 (2008).
- Kalverda, B., Pickersgill, H., Shloma, V. V., Fornerod, M. Nucleoporins directly stimulate expression of developmental and cell-cycle genes inside the nucleoplasm. Cell. 140, 360-371 (2010).
- Kubben, N. Mapping of lamin A- and progerin-interacting genome regions. Chromosoma. 121, 447-464 (2012).
- Steglich, B., Filion, G. J., van Steensel, B., Ekwall, K. The inner nuclear membrane proteins Man1 and Ima1 link to two different types of chromatin at the nuclear periphery in S. pombe. Nucleus. 3, 77-87 (2012).
- Harr, J. C. Directed targeting of chromatin to the nuclear lamina is mediated by chromatin state and A-type lamins. J Cell Biol. 208, 33-52 (2015).
- Gonzalez-Aguilera, C. Genome-wide analysis links emerin to neuromuscular junction activity in Caenorhabditis elegans. Genome Biol. 15, R21(2014).
- Wu, F., Yao, J. Spatial compartmentalization at the nuclear periphery characterized by genome-wide mapping. BMC Genomics. 14, 591(2013).
- Filion, G. J. Systematic protein location mapping reveals five principal chromatin types in Drosophila cells. Cell. 143, 212-224 (2010).
- Vogel, M. J. Human heterochromatin proteins form large domains containing KRAB-ZNF genes. Genome Res. 16, 1493-1504 (2006).
- Venkatasubrahmanyam, S., Hwang, W. W., Meneghini, M. D., Tong, A. H., Madhani, H. D. Genome-wide, as opposed to local, antisilencing is mediated redundantly by the euchromatic factors Set1 and H2A.Z. Proc Natl Acad Sci U S A. 104, 16609-16614 (2007).
- Shimbo, T. MBD3 localizes at promoters, gene bodies and enhancers of active genes. PLoS Genet. 9, e1004028(2013).
- Orian, A. Genomic binding by the Drosophila Myc, Max, Mad/Mnt transcription factor network. Genes Dev. 17, 1101-1114 (2003).
- Artegiani, B. Tox: a multifunctional transcription factor and novel regulator of mammalian corticogenesis. EMBO J. , (2014).
- Schuster, E. DamID in C. elegans reveals longevity-associated targets of DAF-16/FoxO. Mol Syst Biol. 6, 399(2010).
- Bianchi-Frias, D. Hairy transcriptional repression targets and cofactor recruitment in Drosophila. PLoS Biol. 2, e178(2004).
- Woolcock, K. J., Gaidatzis, D., Punga, T., Buhler, M. Dicer associates with chromatin to repress genome activity in Schizosaccharomyces pombe. Nat Struct Mol Biol. 18, 94-99 (2011).
- Luo, S. D., Shi, G. W., Baker, B. S. Direct targets of the D. melanogaster DSXF protein and the evolution of sexual development. Development. 138, 2761-2771 (2011).
- Germann, S., Gaudin, V. Mapping in vivo protein-DNA interactions in plants by DamID, a DNA adenine methylation-based method. Methods Mol Biol. 754, 307-321 (2011).
- Zhang, X. The Arabidopsis LHP1 protein colocalizes with histone H3 Lys27 trimethylation. Nat Struct Mol Biol. 14, 869-871 (2007).
- Orian, A. Chromatin profiling, DamID and the emerging landscape of gene expression. Curr Opin Genet Dev. 16, 157-164 (2006).
- Vogel, M. J., Peric-Hupkes, D., van Steensel, B. Detection of in vivo protein-DNA interactions using DamID in mammalian cells. Nat Protoc. 2, 1467-1478 (2007).
- Greil, F., Moorman, C., van Steensel, B. DamID: mapping of in vivo protein-genome interactions using tethered DNA adenine methyltransferase. Methods Enzymol. 410, 342-359 (2006).
- de Wit, E., Greil, F., van Steensel, B. Genome-wide HP1 binding in Drosophila: developmental plasticity and genomic targeting signals. Genome Res. 15, 1265-1273 (2005).
- DamID mammalian vectors. , Available from: http://research.nki.nl/vansteensellab/Mammalian_plasmids.htm (2015).
- Illumina TruSeq adaptors & PCR primers. , Available from: https://ethanomics.wordpress.com/chip-seq-library-construction-using-the-illumina-truseq-adapters/ (2015).
- Optimization of PCR cycles for NGS. , Available from: https://ethanomics.wordpress.com/ngs-pcr-cycle-quantitation-protocol/ (2015).
- Bernstein, B. E. The NIH Roadmap Epigenomics Mapping Consortium. Nat Biotechnol. 28, 1045-1048 (2010).
- Encode Project Consortium. A user's guide to the encyclopedia of DNA elements (ENCODE). PLoS Biol. 9, e1001046(2011).
- Asp, P. Genome-wide remodeling of the epigenetic landscape during myogenic differentiation. Proc Natl Acad Sci U S A. 108, E149-E158 (2011).
- Hoppe, P. S., Coutu, D. L., Schroeder, T. Single-cell technologies sharpen up mammalian stem cell research. Nat Cell Biol. 16, 919-927 (2014).
- Avital, G., Hashimshony, T., Yanai, I. Seeing is believing: new methods for in situ single-cell transcriptomics. Genome Biol. 15, 110(2014).
- Navin, N. E. Cancer genomics: one cell at a time. Genome Biol. 15, 452(2014).
- Saliba, A. E., Westermann, A. J., Gorski, S. A., Vogel, J. Single-cell RNA-seq: advances and future challenges. Nucleic Acids Res. 42, 8845-8860 (2014).
- Nagano, T. Single-cell Hi-C reveals cell-to-cell variability in chromosome structure. Nature. 502, 59-64 (2013).
- Kind, J. Single-cell dynamics of genome-nuclear lamina interactions. Cell. 153, 178-192 (2013).
- Southall, T. D. Cell-type-specific profiling of gene expression and chromatin binding without cell isolation: assaying RNA Pol II occupancy in neural stem cells. Dev Cell. 26, 101-112 (2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados