Method Article
DamID-seq: Genoma-wide Mapeamento das Interações Proteína-DNA por High Throughput Sequencing de fragmentos de DNA metilado-Adenina
Neste Artigo
Resumo
Descrevemos aqui um ensaio por acoplamento de identificação do DNA adenina metiltransferase (DamID) para sequenciamento de alta taxa de transferência (DamID-seq). Este método melhorado fornece uma resolução mais alta e uma maior gama dinâmica, e permite analisar os dados DamID-seq em conjunto com outros dados de sequenciação de alto rendimento tal como Chip-SEQ, ARN-SEQ, etc.
Resumo
The DNA adenine methyltransferase identification (DamID) assay is a powerful method to detect protein-DNA interactions both locally and genome-wide. It is an alternative approach to chromatin immunoprecipitation (ChIP). An expressed fusion protein consisting of the protein of interest and the E. coli DNA adenine methyltransferase can methylate the adenine base in GATC motifs near the sites of protein-DNA interactions. Adenine-methylated DNA fragments can then be specifically amplified and detected. The original DamID assay detects the genomic locations of methylated DNA fragments by hybridization to DNA microarrays, which is limited by the availability of microarrays and the density of predetermined probes. In this paper, we report the detailed protocol of integrating high throughput DNA sequencing into DamID (DamID-seq). The large number of short reads generated from DamID-seq enables detecting and localizing protein-DNA interactions genome-wide with high precision and sensitivity. We have used the DamID-seq assay to study genome-nuclear lamina (NL) interactions in mammalian cells, and have noticed that DamID-seq provides a high resolution and a wide dynamic range in detecting genome-NL interactions. The DamID-seq approach enables probing NL associations within gene structures and allows comparing genome-NL interaction maps with other functional genomic data, such as ChIP-seq and RNA-seq.
Introdução
ADN identificação adenina metiltransferase (DamID) 1,2 é um método para detectar interacções proteína-ADN in vivo e é uma abordagem alternativa para a imunoprecipitação da cromatina (ChIP) 3. Ele usa uma quantidade relativamente baixa de células e não necessita de reticulação química de proteína com ADN ou um anticorpo altamente específico. O último é particularmente útil quando a proteína alvo é frouxamente ou indirectamente associada com o ADN. DamID tem sido utilizado com êxito para mapear locais de ligação de uma variedade de proteínas, incluindo as proteínas do envelope nuclear 4-10, proteínas de cromatina associada cromatina 11-13, enzimas modificadoras 14, factores de transcrição e co-factores 15-18 e maquinarias RNAi 19. O método é aplicável em vários organismos, incluindo S. 13 cerevisiae, S. pombe 7, C. elegans 9,17, D. melanogaster 5,11,18,20, A. thaliana 21,22, bem como de ratinho e células humanas linhas 6,8,10,23,24.
O desenvolvimento do ensaio DamID foi baseado na detecção específica de fragmentos de ADN metilado-adenina em células eucarióticas que não possuem adenina endógenos metilação 2. Uma proteína de fusão expressa, que consiste da proteína de ligação de ADN de interesse e E. coli ADN metiltransferase adenina (DAM), pode metilar a base de adenina nas sequências GATC que estão em proximidade espacial (mais significativamente dentro de 1 kb e até cerca de 5 kb) para os locais de ligação da proteína no genoma 2. Os fragmentos de ADN podem ser modificados especificamente amplificado e hibridado com microarrays para detectar os locais de ligação genómicos da proteína de interesse 1,25,26. Este método DamID original é limitada pela disponibilidade de microarranjos e a densidade de sondas predeterminados. Temos, portanto, integrada alto throughput sequenciação empara DamID 10 e designado como o método DamID-SEQ. O grande número de curto geradas a partir de leituras DamID-SEQ permite localização precisa de interacções proteína-ADN a nível genoma. Descobrimos que DamID-seq fornecida uma resolução maior e uma gama dinâmica mais ampla do que DamID por microarray para estudar lâmina genoma nuclear (NL) 10 associações. Este método permite uma melhor sondagem associações NL dentro das estruturas do gene 10 e facilita comparações com outros dados de seqüenciamento de alto rendimento, como ChIP-seq e RNA-seq.
O protocolo DamID-seq aqui descrito foi desenvolvido inicialmente para mapeamento do genoma-NL 10 associações. Geramos uma proteína de fusão amarrados mouse ou Lamin B1 humana para E. coli DNA metiltransferase adenina e testou o protocolo em 3T3 fibroblastos de rato embrionárias, C2C12 mioblastos rato 10 e IMR90 fibroblastos de pulmão fetal humano (dados não publicados). Neste protocolo, começamos com constructing vetores e expressando proteínas de fusão Dam-tethered por infecção lentivírus em células de mamíferos 24. A seguir, descrevemos os protocolos detalhados de amplificar fragmentos de DNA metilado-adenina e preparar bibliotecas de seqüenciamento que deveriam ser aplicável em outros organismos.
Protocolo
1. Geração e Expressão de Proteínas de Fusão e Proteínas gratuito Dam
- O clone da proteína de interesse no vector DamID.
- Amplificar ADNc de proteína de interesse (POI), utilizando a ADN-polimerase de alta fidelidade desejado e iniciadores apropriados de acordo com o protocolo do fabricante. Determinar experimentalmente as condições de amplificação óptimas para garantir uma amplificação apropriada de inserções.
- Executar um gel de agarose e amplificado ADNc de purificar PI pelo kit de extracção de gel de acordo com o protocolo do fabricante.
- O clone de ADNc de PI no vector pDONR201 usando BP clonase II de acordo com o protocolo do fabricante.
- Clonar o ADNc de PI a partir do vector dador no vector de destino pLGW-RFC1-V5-EcoDam ou o destino vector pLGW-EcoDam-V5-RFC1 27 usando LR clonase II de acordo com o protocolo do fabricante, dependendo da direcção desejada de fusão PI para o terminal N ou the C-terminal da E. coli ADN metiltransferase adenina (EcoDam) 27.
- Verifique por sequenciação de que o cDNA clonado tem uma seqüência correta e formas em-frame de fusão para EcoDam.
- Gerar stocks de lentivírus.
- Gerar stocks de lentivírus que expressam Dam-V5-POI e V5-Dam (a partir do vector pLGW-V5-EcoDam 27) utilizando sistemas de expressão de lentivírus. Use o procedimento de transfecção para a frente segundo o protocolo do fabricante.
- Use células 293T e lipofecção para gerar stocks lentivirais de acordo com o protocolo do fabricante para a transfecção.
- Incluem o passo de filtro de PVDF de 0,45 um.
- Gerar stocks de lentivírus que expressam Dam-V5-POI e V5-Dam (a partir do vector pLGW-V5-EcoDam 27) utilizando sistemas de expressão de lentivírus. Use o procedimento de transfecção para a frente segundo o protocolo do fabricante.
- Infectar as células com lentivírus.
- O dia antes da infecção (dia 0), passar células aderentes cultivadas em meio de crescimento apropriado para este tipo de célula para uma placa de cultura de tecido de 6 poços utilizando o mesmo meio de crescimento sem a antibióticosalcançar 50% de confluência no dia da infecção. Células lugar em um 37 ° C incubadora.
- No dia da infecção (dia 1), retire 2 cryovials de ambos Dam-V5-PI e V5-Dam sobrenadantes de lentivírus de um freezer -80 ° C e coloque em um banho de 37 ° C água para descongelar.
- Remover o meio de crescimento a partir de células e substituir com 0,5 ml de meio de crescimento fresco, sem antibióticos.
- Adicionar 1 ml do lentivírus descongeladas a cada poço (2 poços com V5-Dam e 2 poços com Dam-V5-POI). Adicionar 1 ml de meio de crescimento sem os antibióticos para os restantes poços de 2-isso vai servir como um controlo negativo. Agite suavemente a placa de 6 poços para misturar e colocar de volta em um 37 ° C incubadora O / N.
- O dia após a infecção (dia 2), remova suspensões virais a partir de células e substituir com 2 ml de meio de crescimento sem antibióticos. Células lugar de volta em uma incubadora de 37 ° C durante 48 horas.
- Isolar gDNA.
- Meios aspirado de cada poço e decélulas tach usando 250 ul 0,05% tripsina-EDTA. Incubar a 37 ° C durante 2 min.
- Lave as células da placa com 1 ml de meio de crescimento e células de cada cavidade de pipeta para um tubo de microcentrífuga de 1,5 ml. Peletizar as células por centrifugação a 200 xg durante 5 min à temperatura ambiente.
- Lavar as células sedimentadas com 500 ul de PBS e centrifugar a 200 xg durante 2 min à temperatura ambiente.
- Ressuspender as células peletizadas em 200 ul de PBS.
- Isolar ADNg pelo kit de sangue e de tecido de acordo com o protocolo do fabricante. Eluir ADNg em 200 ul de tampão AE e determinar a concentração medindo a OD 260 utilizando um espectrofotómetro.
Nota: ADNg partir de células infectadas ou não infectadas simuladas pode ser isolado como controlos negativos. Precipitar ADNg para concentrações mais elevadas para o armazenamento a longo prazo.- Adicionar 3 volumes de etanol a 100% e 0,1 volume de acetato de sódio 3M (pH 5,5), e misturar por inversão tubos 4-6 vezes.
- Armazenar a -20 ° CO / N.
- Centrifugar a 16.000 xg durante 15 min a 4 ° C.
- Remova cuidadosamente o sobrenadante. Lavar peletes com 70% (volume / volume) de etanol e centrifuga-se a 16.000 xg durante 5 min a 4 ° C.
- Cuidadosamente remover etanol e permitir peletes secar ao ar durante 3 minutos à temperatura ambiente.
- Dissolver ADNg em T 10 E 0,1 (pH 7,5) a cerca de 1 ug / uL. Piscina gDNA de cada amostra experimental ou o controlo negativo e medir a concentração. Armazenar a -20 ° C.
Fragmentos de DNA 2. Amplify Adenina-metilados
- Digest ADNg com Dpnl, que corta apenas GATCs metilado-adenina.
- Defina-se uma reacção em gelo com 2,5 ug ADNg, 1 ul de tampão 10x, 0,5 ul DpnI (20 U / ul) e encher com H 2 O para um volume total de 10 ul. Para cada amostra de ADNg, preparar três reacções - um sem Dpnl ("não Dpnl", substituir Dpnl com 0,5 ul de H2O) e dois wom DpnI ("com DpnI").
- Digest O / N a 37 ° C e Dpnl inactivar a 80 ° C durante 20 min.
- Ligadura adaptadores DamID.
- Prepare adaptadores DamID.
- Ressuspender cada um dos dois oligonucleótidos adaptadores DamID 24 em H 2 O para uma concentração final de 100 uM.
- Combine volumes iguais dos dois oligos adaptador DamID, misture e coloque em um tubo bem fechado. Selar o tubo com Parafilm, sentar-se em um rack e coloque em um copo cheio de água a 90 ° C. Manter o nível da água abaixo da tampa do tubo (para evitar a entrada de água no tubo), mas acima da superfície da mistura de oligo.
- Deixe a água fresca para RT de modo que o recozimento adaptadores lentamente.
- Alíquota os adaptadores recozidos (50 mm) e armazenar a -20 ° C.
- Configurar uma reacção em gelo. Em cada tubo de 2.1.2, adicionar 6,2 mL de H2O, 2 uL de tampão de ligação 10x, 0,8 ad 50 uL uM DamIDaptors (descongeladas em gelo) e 1 ul de DNA ligase de T4 (5 U / uL). O volume total é 20 jil. Em um dos dois "tubos" com DpnI, substitua com 1 ul de ligase H2O ("não-ligase") Note-se que cada amostra ADNg tem dois controlos negativos -. "Dpnl nenhuma" e "não-ligase".
- Ligadura O / N a 16 ° C e inactivar a ligase a 65 ° C durante 10 min.
- Prepare adaptadores DamID.
- ADN digerido com DpnII para destruir os fragmentos que contêm GATCs não metilados.
- Configurar uma reacção em gelo. Em cada tubo de 2.2.3, adicionar 24 ul de H2O, 5 mL de tampão 10x e 1 ul DpnII DpnII (10 U / ul). O volume total é 50 jil.
- Digest a 37 ° C durante 2-3 horas e inactivar DpnII a 65 ° C durante 20 min.
- Amplificar fragmentos de ADN metilado-adenina.
- Configurar uma reação no gelo com 5 ul DpnII digerir a partir de 2.3.2, 5 μl tampão de PCR 10x, 5 jil 12,5 uM DamID iniciador de PCR de 24, 4 ul de dNTP 10 mM de misturar, 1 ul de polimerase 50x mistura e 22,5 jil de H2O O volume total é 50 jil.
- Executar a PCR como se segue: 68 ° C durante 10 min; 94 ° C durante 1 min, 65 ° C durante 5 min, 68 ° C durante 15 min; 4 ciclos de 94 ° C durante 1 min, 65 ° C durante 1 min, 68 ° C durante 10 min; 17 ciclos de 94 ° C durante 1 min, 65 ° C durante 1 min, 68 ° C durante 2 min.
- Analise 5 ul produtos de PCR de cada reacção sobre um gel de agarose a 1%. Os produtos de PCR deve aparecer como uma mancha que varia de 0,2 para 2 kb (Figura 2). Os controles "não DpnI" e "não ligase" não deve ter nenhum ou menos claramente amplificação.
- Se o resultado do passo 2.4.3 é satisfatória, repetir os passos 2.4.1-2.4.3 com duas ou três reacções para a amostra experimental e uma reacção para cada um dos dois controlos negativos.
- Piscina e purificar produtos de PCR a partir da mesmaamostra experimental utilizando kits de purificação de PCR ou de fase sólida reversível Imobilização (SPRI) esferas de acordo com o protocolo do fabricante. Não purificar "não DpnI" ou controles "não ligase". Eluir DNA com tampão de EB.
- Medir a concentração de DNA medindo a OD purificada 260 utilizando um espectrofotómetro, que deve ser cerca de 0,1 mg / mL ou superior. Recolha um mínimo de 10 ug de ADN para cada amostra. Se estiver usando kits de purificação de PCR, purificar cada 50 ul produtos de PCR com uma coluna, eluir em 30 ul de tampão EB e reunir os eluatos.
- ADN digerido com DpnII para evitar que os iniciadores de PCR a partir de DamID sendo sequenciados.
- Defina-se uma reacção em gelo com 5 ug de ADN purificado a partir de 2.4.6, 5 ul de tampão 10x DpnII, 1 ul DpnII (10 U / ul) e encher com H 2 O para um volume total de 50 ul. Preparar dois ou três reacções para cada amostra.
- Digerir a 37 ° C durante 2-3 hre inactivar DpnII a 65 ° C durante 20 min.
- Piscina e purificar os fluidos de digestão a partir da mesma amostra com kits de purificação de PCR ou grânulos SPRI de acordo com o protocolo do fabricante. Eluir DNA com tampão de EB.
- Medir a concentração de ADN purificado, que deve ser cerca de 0,06 mg / mL ou superior. Recolha um mínimo de 6 ug de DNA de cada amostra. Se estiver usando kits de purificação de PCR, purificar cada 50 ul digerir com uma coluna, eluir em 30 ul de tampão EB e reunir os eluatos.
3. Preparação para a Biblioteca de alta capacidade Sequencing
- Fragmento de ADN
- Experimentalmente determinar o tempo de digestão apropriados para cada novo lote de ADNcd Fragmentase. À medida que a actividade da enzima pode diminuir ao longo do tempo, repetir o teste antes de executar uma nova experiência. Para guardar o DNA de 2.5.4 para a fragmentação actual, utilizam produtos de PCR purificados a partir de metilo ou 2.4.6 DNA extra de experime anteriornts nesta etapa.
- Configurar uma mistura principal com DNA 6 mg, 12 ul 10x tampão Fragmentase e encher com H 2 O para um volume total de 114 mL.
- Vortex do frasco estoque Fragmentase por 3 s, adicionar 6 ul na mistura principal e vortex a mistura principal por 3 s. O volume total é de 120 l.
- Alíquota de 20 ul da mistura principal para cada um dos 5 novos tubos. Incubar todas as 6 tubos a 37 ° C durante 5 a 55 min a um incremento de 10 minutos. Adicionar 5 uL de EDTA 0,5 M para parar a reacção.
- Analise 12,5 ul digerir (0,5 ug de ADN) de cada uma das reacções, bem como 0,5 ug de ADN digerido num gel de agarose (Figura 3). Determine o tempo mínimo (t 0.2kb) necessária para digerir a maioria da mancha de cerca de 0,2 kb. Selecione 6 durações de tempo entre 5 min e T 0.2kb (incluindo 5 min e T 0.2kb) com incrementos iguais para a fragmentação actual.
- Configure os Fragmen reaisção como descrito no 3.1.1.1-3.1.1.3. Incubar as reacções a 37 ° C, para as durações de tempo determinado em 3.1.1.4.
- Piscina 6 reacções e purificar as digestões com kits de purificação de PCR ou grânulos SPRI de acordo com o protocolo do fabricante. Elui-se o ADN em tampão 51 ul de EB. O eluato final é ~ 50 ul.
- Experimentalmente determinar o tempo de digestão apropriados para cada novo lote de ADNcd Fragmentase. À medida que a actividade da enzima pode diminuir ao longo do tempo, repetir o teste antes de executar uma nova experiência. Para guardar o DNA de 2.5.4 para a fragmentação actual, utilizam produtos de PCR purificados a partir de metilo ou 2.4.6 DNA extra de experime anteriornts nesta etapa.
- Reparação termina do DNA fragmentado
- Defina-se uma reacção em gelo com o eluato de 3.1.3, 25 ul de H2O, 10 pi de tampão ligase 10x de ADN de T4 com ATP 10 mM, 4 ul de mistura de dNTP 10 mM, 5 jil de polimerase do DNA de T4 (3 U / ul) , 1 ul de ADN polimerase de Klenow (5 U / uL), e 5 ul de polinucleotídeo cinase de T4. O volume total é de 100 uL. Misturar bem com pipeta. Evite espuma e bolhas.
- Incubar a 20 ° C durante 30 min.
- Purifica-se o ADN com kits de purificação de PCR ou grânulos SPRI de acordo com o protocolo do fabricante. Elui-se o ADN em tampão 33 ul de EB. O eluato final é ~ 32 & #181; l.
- Adicionar saliências "A"
- Defina-se uma reacção em gelo com o eluato de 3.2.3, 5 ul de tampão Klenow 10x, 10 ul de dNTPs 1 mM, e 3 ul de Klenow (3 '→ 5' exo) (5 U / uL). O volume total é 50 jil. Misturar bem com pipeta. Evite espuma e bolhas.
- Incubar a 37 ° C durante 30 min.
- Purifica-se o ADN com kits de purificação de PCR ou grânulos SPRI de acordo com o protocolo do fabricante. Elui-se o ADN em tampão 22 ul de EB. O eluato final é ~ 21 ul.
- Adaptadores de sequenciamento ligadura
- Prepare adaptadores de sequenciação 28.
- Ressuspender cada oligo adaptador a uma concentração de 100 ^ M em Tris 10 mM-Cl (pH 7,8), EDTA 0,1 mM (pH 8,0) e NaCl 50 mM.
- Misturar volumes iguais de o adaptador universal 28 e um adaptador indexada 28.
- Emparelhar as placas num termociclador com a seguinteprograma: 95 ° C durante 2 min; 140 ciclos durante 30 segundos a partir de 95 ° C e uma diminuição de 0,5 ° C a cada ciclo; manter a 4 ° C.
- Adaptadores de alíquotas e armazenar a -20 ° C.
- Configurar uma reacção em gelo com o fluido a partir de 3.3.3, 25 ul 2x tampão de ligação, 1,5 ul 50 mM recozidos adaptadores de seqüenciamento (descongeladas em gelo) do 3.4.1.4 e 2,5 ul DNA ligase de T4. Misturar bem com pipeta. Evite espuma e bolhas. Se seqüenciamento multiplex é desejada, use um adaptador indexada diferente para cada amostra.
- Incubar à temperatura ambiente durante 1 h.
- Purifica-se o ADN com kits de purificação de PCR ou grânulos SPRI de acordo com o protocolo do fabricante. Elui-se o ADN em tampão 24 ul de EB. O eluato final é ~ 23 ul.
- Prepare adaptadores de sequenciação 28.
- Converter adaptadores em forma de Y para dsDNA para permitir determinar com precisão fragmento de DNA de tamanhos 28
- Configurar uma reacção em gelo com o fluido a partir de 3.4.4, 12,5 ul H2O, 1 ul 25 uM iniciador 1 28, 1 25 ul de 2 28 uM de iniciadores, 1,5 ul de dNTP 10 mM, 10 ul de tampão de PCR 5x e 1 ul de DNA polimerase (1 U / ul).
- Executar a PCR como se segue: 95 ° C durante 3 min; 5 ciclos de 98 ° C durante 15 seg, 63 ° C durante 30 seg, 72 ° C durante 30 seg; 72 ° C durante 1 min; 4 ° C em espera.
- Purifica-se o ADN com kits de purificação de PCR ou grânulos SPRI de acordo com o protocolo do fabricante. Elui-se o ADN em tampão 11 ul de EB. O eluato final é ~ 10 ul.
- Tamanho selecione a biblioteca
- Preparar um gel de agarose a 2% em tampão TAE 1X. Adicionar brometo de etídio (CUIDADO!) A uma concentração final de 500 ng / ml quando a solução de TAE-agarose fundida foi arrefecida para evitar a inalação de brometo de etídio. Certifique-se de ter pistas suficientes para todas as amostras, escada de DNA e pistas vazias.
- Adicionar corante 6x carregamento 8 ul a partir do eluato 3.5.3.
- Prepare a escada de ADN por mistura de 1 kb, mais escada de ADN (1,0 ug / uL), corante de carga de 6x e H2O numa proporção de 1: 1: 4.
- Carga 6 ul escada de DNA, as amostras a partir de 3.6.2 e outra escada de ADN 6 ul, cada um em uma pista separada e com pelo menos uma fila vazia a partir das amostras / escadas adjacentes.
- Submeter o gel a 120 V durante 60 min.
- Ver o gel num transiluminador de UV (minimizar o tempo de exposição ao UV). Usar óculos de protecção e uma viseira. Certifique-se de pelo menos uma das escadas de DNA correr bem com espaçamento adequado (o suficiente para extirpar 3 fatias de gel) entre 300 pb e 400 pb bandas. Espaçamento estreito aumenta as dificuldades de excisão múltiplas fatias de gel, enquanto o espaçamento de largura aumenta o volume de fatias de gel excisado.
- Use uma nova bisturi ou uma lâmina de barbear para cada pista. Excise três fatias finas de gel entre 300 e 400 pb a partir de cada pista (Figura 4) e colocá-los em cada um tubo de microcentrífuga. Mantenha o volume de cada gel spiolhos tão baixo quanto possível (<100 uL).
- Medir o volume de cada fatia de gel (gel de 1 ul pesa aproximadamente 1 mg), adicionar volumes de tampão QG 6x e incubar a 50 ° C.
- Vortex a mistura QG-gel cada 2-3 min, até que a fatia de gel foi completamente dissolvido. Adicionar volumes de gel 2x de isopropanol e misture.
- A partir desta etapa, siga o protocolo de kits de extração de gel. Elui-se o ADN em tampão 51 ul de EB. O eluato final é ~ 50 ul.
- Enriqueça fragmentos de DNA adaptador modificado seqüenciamento
- Otimizar o número de ciclos de PCR 29.
- Defina-se uma reacção em gelo com 1 ml de eluato 3.6.10, 1 ul 25 uM iniciador 1 28, 1 25 ul de 2 28 uM de iniciadores, 7 ul de H2O e 10 ul de SYBR Green Supermix. O volume total é 20 jil.
- Executar o qPCR como se segue: 95 ° C durante 3 min; 20 ciclos de 95 ° C durante 30 seg, 63 ° C durante 30 seg, 72 ° C durante 30 seg, a placa de leitura.
- Analisar os dados utilizando um software de análise de qPCR e determinar o ciclo de Quantificação (Cq) ou o ciclo limiar (Ct) para cada amostra utilizando o protocolo do fabricante. Continue com as amostras que têm Cq / Ct ≤ 14. Use o máximo Cq (CQ 0) menos 1 (arredondado para o próximo inteiro para cima) como o número de ciclos PCR final (N PCR).
- Ajustar as quantidades de moldes de ADN de modo a que diferentes amostras será amplificado a quantidades aproximadamente iguais depois de executar o mesmo número de ciclos de PCR. Use 8 ul molde para a amostra com a mais elevada Cq (Cq 0), e calcular o volume do molde de outras amostras com a seguinte fórmula:
Vol I = 8 x 1,8 cq i -Cq 0
- Configure reações de PCR em gelo com os volumes do modelo calculados a partir 3.7.1.4: 1 pi 25 um iniciador 1 28, 1 ul 25 um iniciador 2 28, 1.5uL dNTP 10 mM, 10 ul de tampão 5x, 1 ul de DNA polimerase (1 U / ul) e encher com H 2 O para um volume total de 50 ul.
- Executar a PCR como se segue: 95 ° C durante 45 seg; N ciclos de PCR (determinado em 3.7.1.3) de 98 ° C durante 15 seg, 63 ° C durante 30 seg, 72 ° C durante 30 seg; 72 ° C durante 1 min; 4 ° C em espera.
- Analise 5 ul produtos de PCR num gel de agarose a 2% (Figura 5A). Uma banda clara "única" indica que os fragmentos de ADN amplificados estão dentro de uma gama estreita de comprimento e de que a biblioteca de ADN pode ser sujeito a mais análises.
- Repetir uma reacção para amostras seleccionadas e reunir as bibliotecas de DNA amplificados a partir da mesma amostra.
- Purificar bibliotecas de DNA seleccionados para sequenciamento.
- Se dímeros primer / adaptador não são visíveis no 3.7.4, purificar bibliotecas de DNA com kits de purificação de PCR ou contas SPRI de acordo com o protocolo do fabricante. Elui-se o ADN em tampão 21 ul de EB. oeluato final é ~ 20 ul. Se serviço de sequenciação do usuário tem instruções específicas sobre o tampão de eluição, o volume final, etc., preparar amostras em conformidade.
- Se dímeros de iniciador / adaptador são claramente visíveis em 3.7.4, purificar bibliotecas como se segue.
- Carga de ADN a partir de 3.7.5 e escada de DNA em gel de agarose a 2% pré-moldado. As amostras podem ser purificados tal como descrito em 3.7.6.1 para reduzir o volume ou pode ser colocado em várias cavidades. Coloque o gel de agarose em um sistema de alimentação adequada. Deixar o gel correr durante 15 min.
- Usando um transilluminator disso, cortar a banda desejada com um bisturi limpo / navalha para cada amostra e colocar a fatia de gel em um tubo de microcentrífuga de 1,5 ml.
- Isolar o ADN utilizando kits de extracção de gel de acordo com o protocolo do fabricante, com duas modificações: incubar tampão mistura QG-gel num termo-misturador a 37 ° C e 1000 rpm durante 30 min, e adicionar volumes de gel 2x de isopropanol.
- Otimizar o número de ciclos de PCR 29.
- Enviar bibliotecas de DNA purificado de uma academia de seqüenciamento. Siga todas as orientações facilidade.
Nota: Uma análise da qualidade por um Bioanalyzer (Figura 5B) deve ser realizada antes da sequenciação, a fim de determinar a gama de dimensão exacta e a concentração de uma biblioteca de ADN.
Resultados
Verificou-se a proteína de fusão Dam-V5-LmnB1 para ser co-localizada com a proteína endógena lamina B por coloração de imunofluorescência (Figura 1).
A amplificação bem sucedida PCR de fragmentos de DNA metilado-adenina é um passo fundamental para DamID-seq. As amostras experimentais devem amplificar um esfregaço de 0,2 - 2 KB, enquanto que os controlos negativos (sem Dpnl, sem ligase ou sem molde de PCR) deve resultar em nenhuma ou-menos claramente-amplificação (Figura 2).
Os fragmentos de ADN são metilados no intervalo de 0,2 a 2 kb, enquanto que o tamanho da inserção desejada para uma biblioteca de NGS é de 200 a 300 pb. Portanto, é essencial para fragmentar os produtos de PCR de metilo para a gama de tamanho apropriado. No entanto, verificou-se ser impraticável para romper simultaneamente os fragmentos de ADN maiores para baixo para o tamanho adequados e manter a maioria dos fragmentos de ADN menores intactas num período de fragmentação. Portanto, as experiências foram realizadas decurso de tempo para determinar o tempo mínimo (t 0.2kb) necessária para fragmento de 1 ug de ADN para um esfregaço centrado em 200 pb (Figura 3). Em seguida, foram selecionados 6 durações de tempo em incrementos iguais entre 5 min e T 0.2kb para a fragmentação actual. A actividade enzimática da dupla cadeia de ADN Fragmentase pode variar de lote para lote e pode diminuir ao longo do tempo, por isso, recomenda-se repetir este passo para um novo lote de Fragmentase ou após armazenagem durante um período de tempo.
O tamanho da inserção desejada situa-se entre 200 e 300 pb, correspondente aos fragmentos de ADN entre 300 e 400 pb (incluindo 121 pb adaptadores de sequenciação) em gel de agarose. Três fatias finas dentro desta gama foram excisados a partir de cada amostra experimental para limitar o tamanho de uma biblioteca e aumentar a possibilidadea obtenção de pelo menos uma biblioteca de sequenciação qualificado (Figura 4).
Uma alíquota de 5 ul de cada biblioteca de ADN amplificado foi analisado em gel de agarose para determinar qual biblioteca podem qualificar para sequenciação. Como mostrado na Figura 5A, uma banda única clara do mesmo tamanho que a fatia de gel excisado deve ser visível no gel de agarose (passo 3.7.4). Em seguida, as bibliotecas seleccionadas foram examinados por um Bioanalyzer (Figura 5B) para determinar a gama de concentrações e tamanho exacto antes da sequenciação. Se desejado, as bibliotecas de ADN amplificadas pode ser directamente analisada por uma análise em gel sem Bioanalyzer. Quando várias bibliotecas são de boa qualidade, recomenda-se para sequenciar bibliotecas de tamanho similar varia de um par de experimentais (células que expressam Dam-V5-POI) e controle (células expressando V5-DAM) amostras.
A curto lê gerado porsistemas de sequenciamento foram mapeados pela primeira vez ao genoma correspondente. Excepcionalmente alinhado leituras foram então passadas para análises subsequentes. Um gasoduto para processar curto lê, construir uma interação mapa do genoma-NL e analisar associações de genes-NL foram descritos em detalhes em nosso trabalho anterior 10. Os resultados representativos são apresentados na Figura 6.

Figura 1. A validação subnucleares localizações de proteínas de fusão por imunofluorescência. As células IMR90 de fibroblastos de pulmão humano, foram transfectadas transitoriamente com um plasmídeo que expressa Dam-V5-LmnB1 e foram coradas por anti-lamina B (A, vermelho) e anti-V5 (B, verde). (C) Mesclar de imagens em A e B. Por favor, clique aqui para ver uma versão ampliada da tsua figura.

Figura 2. amplificar por PCR fragmentos de ADN metilado-adenina. Uma alíquota de 5 ul de cada reacção de PCR foi analisado num gel de agarose a 1%. Uma mancha que varia 0,2-2 kb foi amplificado a partir de cada amostra experimental, mas nenhuma amplificação foi observada em controlos negativos (sem DpnI no passo 2.1, não Ligase na etapa 2.2, ou nenhum modelo de PCR). Por favor clique aqui para ver uma versão maior desta figura.
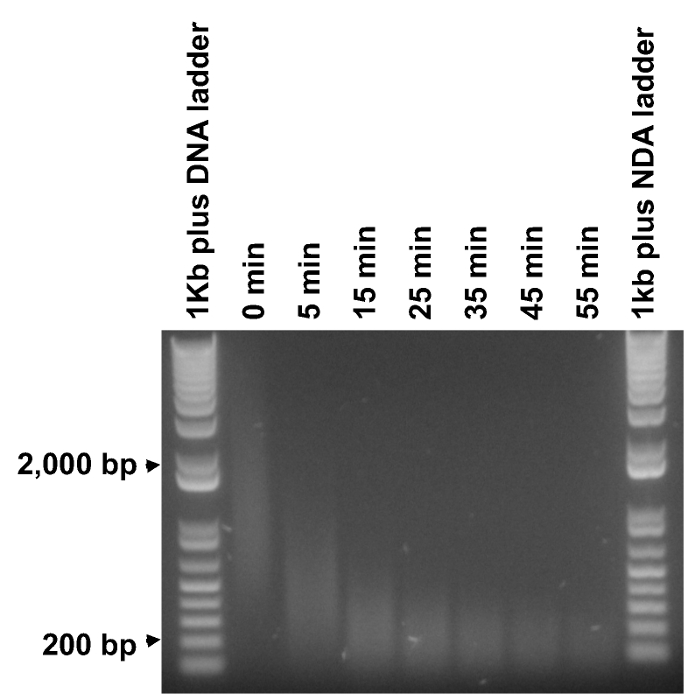
Figura 3. Determinar durações óptimas de fragmentação. Os produtos de PCR purificados foram sujeitos metil à fragmentação por períodos de tempo a partir de 5 a 55 min a um incremento de 10 min (ADN digerido , Rotulado como "0 minutos"). 1 ug de ADN de escada e 0,5 ug de cada amostra de ADN fragmentado foram analisados num gel de agarose. O tempo mínimo para digerir a maioria da mancha de ADN de cerca de 200 pb foi determinada como sendo cerca de 35 min, por conseguinte, seis durações de tempo espaçados uniformemente entre 5 e 35 min (5 e 35 min incluídos) foram seleccionados para realizar a fragmentação real. Por favor clique aqui para ver uma versão maior desta figura.

Figura 4. Tamanho seleccionando as bibliotecas de ADN. As amostras de ADN de Dam-V5 e V5-LmnB1-Dam do passo 3.6 foram corridos num gel de agarose a 2%, e três pedaços de gel de tamanho igual (G, H e H correspondente a baixa, média e alta em tamanho) foram excisadas entre 300 e 400 pb, como mostrado pelas linhas amarelas. om / files / ftp_upload / 53620 / "target =" _ blank 53620fig4large.jpg "> Clique aqui para ver uma versão maior desta figura.
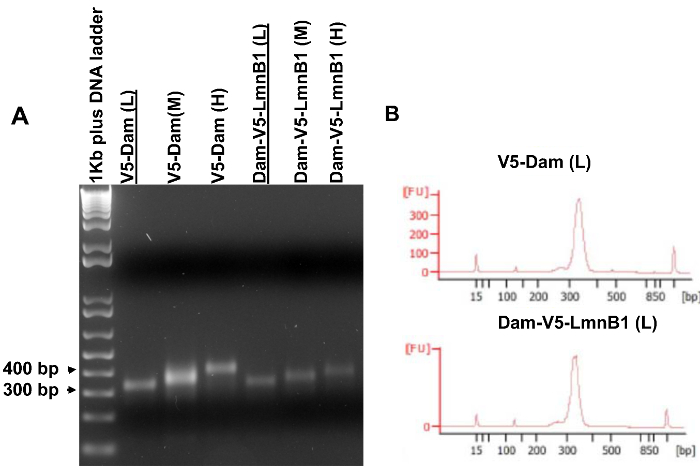
Figura 5. bibliotecas de ADN amplificado para sequenciação de alto rendimento. (A) ADN amplificado bibliotecas analisadas num gel de agarose. Modelos de PCR foram purificados a partir de fatias de gel mostrados na Figura 4. (B) Os resultados Bioanalyzer da amostra V5-Dam (L) e a amostra Dam-V5-LmnB1 (L) que foram sublinhados em (A). Estas duas bibliotecas tinha as faixas de tamanho estreita semelhantes e, portanto, qualificado para sequenciamento alta taxa de transferência. Por favor clique aqui para ver uma versão maior desta figura.
jpg "/>
Figura 6. Os dados apresentados no NGS navegador genoma UCSC. Mouse cromossoma 1 é mostrado como um exemplo. Os dados da seqüência foram produzidos a partir de mioblastos C2C12 rato 10. Faixas "MB.LmnB1.w2k" e "MB.Dam.w2k", correspondente aos dados de células que expressam Dam-V5-LmnB1 e V5-Dam, respectivamente, enredo normalizada densidades de leitura (leituras por kilobase por milhão mapeados exclusivamente lê, ou RPKM ) em que não se sobrepõem consecutivos janelas 2 kb ao longo do cromossoma. Faixa "MB.log2FC.w2k" terrenos associações genoma-NL, ou seja, log2 RPKM rácios de LmnB1 mais de Dam, na resolução de 2 kb. Faixa "MB.sLADs" pinta Lamina Associated Domínios baseados em seqüenciamento (slads, ou seja, regiões genômicas que têm densidades de leitura mais altas de LmnB1 sobre Dam com significância estatística) em preto, não-slads em regiões de cinza e indeterminados em branco. Por favor clique aqui para download este arquivo.
Discussão
Whether Dam-tagged proteins retain the functions of endogenous proteins should be examined before a DamID-seq experiment. The subcellular localization of Dam-tagged nuclear envelope proteins should always be determined and compared with that of the endogenous proteins. For studying transcription factors, it is suggested to examine whether the Dam-fusion protein can rescue the functions of the endogenous protein in regulating gene expression. This functional test can be performed in organisms in which knockout mutants of endogenous DNA-binding proteins are available. Because advances in genome engineering have potentially allowed knocking out any endogenous gene of interest, functions of Dam-tagged DNA-binding proteins can be examined in cultured mammalian cells.
The critical step in this protocol is to successfully fragment the DpnII-digested DamID PCR products to around 200 bp. This step is designed to render the amplified adenine-methylated fragments to a narrow size range for sequencing and to randomize the starting nucleotides of the DNA fragments in a sequencing library. Inefficient fragmentation will leave the majority of the DNA fragments starting with GATC (the 5'-overhang from the second DpnII digestion), and will result in a much lower performance and yield or even a failure in Illumina sequencing. Other DNA fragmentation methods may be used as an alternative approach.
The resolution of DamID (and DamID-seq described here) is limited by the frequency of GATCs in the genome to be studied. Moreover, even with high throughput sequencing, the genomic localizations of a DNA-binding protein can only be mapped within two consecutive GATCs rather than to the actual DNA-binding sites.
Despite its limitation, the DamID assay has important advantages. Because DamID does not require highly-specific antibodies, it can be used to detect a subset of nuclear proteins that could be difficult to assay by ChIP (such as the nuclear envelope proteins). To study how these proteins regulate genome functions, it is important to integrate and cross-analyze their genome-wide localization data with the current epigenomic mapping data (such as data from the ENCODE and NIH Roadmap Epigenomics Projects 30,31). The DamID-seq approach provides both higher resolution and higher sensitivity than DamID by microarray and enables detecting differential NL-associations within gene structures 10. A combinatorial analysis of DamID-seq data, ChIP-seq data 32 and gene expression data has identified a class of NL-associated genes with distinct epigenetic and transcriptional features (data not published).
Another advantage of DamID is that it only requires a small number of cells. In recent years, there has been an explosion in single cell analysis of gene regulation 33,34. Although genome sequence 35, genome-wide gene expression 36 and chromatin conformation 37 can be assayed in a single cell, there has not been an available approach for detecting protein-DNA interactions genome-wide in a single cell. DamID-seq is a highly promising approach for this goal, and may complement the single cell imaging approach in detecting the dynamics of genome-NL interactions 38. One complication is that because the Dam-fusion protein is expressed at a much lower level than the endogenous protein in the DamID assay, it is possible that the Dam-fusion protein may only occupy a subset of genomic binding sites as compared to the endogenous protein.
DamID assay has mostly been used in cultured animal cells to detect protein-DNA interactions. Notably, developmental biologists have applied this assay in detecting protein-DNA interactions in specific cell types in vivo. For example, Dam-tagged RNA polymerase II was expressed specifically in Drosophila neural stem cells to detect their genome-wide occupancy without cell isolation 39. DamID-seq will be highly useful to study the genome-wide localizations of nuclear envelope proteins, transcription factors and chromatin regulators during development in animal models.
Divulgações
The authors have nothing to disclose.
Agradecimentos
We thank Dr. Bas van Steensel for providing the DamID mammalian expression vectors. We thank Yale Center for Genome Analysis and the Genomics Core in Yale Stem Cell Center for advice on preparing NGS libraries and implementing high throughput DNA sequencing. This work was supported by the startup funding from Yale School of Medicine, a Scientist Development Grant from American Heart Association (12SDG11630031) and a Seed Grant from Connecticut Innovations, Inc. (13-SCA-YALE-15).
Materiais
| Name | Company | Catalog Number | Comments |
| ViraPower Lentiviral Expression Systems | Life Technologies | K4950-00, K4960-00, K4970-00, K4975-00, K4980-00, K4985-00, K4990-00, K367-20, K370-20, and K371-20 | |
| Gateway BP Clonase II Enzyme Mix | Life Technologies | 11789-020 | |
| Gateway LR Clonase II Enzyme Mix | Life Technologies | 11791-020 | |
| DNeasy Blood & Tissue Kit (250) | QIAGEN | 69506 or 69504 | |
| Gateway pDONR 201 | Life Technologies | 11798-014 | |
| 293T cells | American Type Culture Collection | CRL-11268 | |
| Trypsin-EDTA (0.05%), phenol red | Life Technologies | 25300-054 | |
| DMEM, high glucose, pyruvate | Life Technologies | 11995-065 | |
| Fetal Bovine Serum | Sigma | F4135 | |
| Tris | brand not critical | ||
| EDTA | brand not critical | ||
| 200 Proof EtOH | brand not critical | ||
| Isopropanol | brand not critical | ||
| Sodium Acetate | brand not critical | ||
| DpnI | New England Biolabs | R0176 | supplied with buffer |
| DamID adaptors "AdRt" and "AdRb" | Integrated DNA Technologies | sequences available in ref. 24; no phosphorylation of the 5' or 3' end to prevent self-ligation. | |
| T4 DNA Ligase | Roche Life Science | 10481220001 | supplied with buffer |
| DpnII | New England Biolabs | R0543 | supplied with buffer |
| DamID PCR primer "AdR_PCR" | Integrated DNA Technologies | sequences available in ref. 24 | |
| Deoxynucleotide (dNTP) Solution Set | New England Biolabs | N0446 | 100 mM each of dATP, dCTP, dGTP and dTTP |
| Advantage 2 Polymerase Mix | Clontech | 639201 | supplied with buffer |
| 1Kb Plus DNA Ladder | Life Technologies | 10787018 | 1.0 µg/µl |
| QIAquick PCR Purification Kit | QIAGEN | 28104 or 28106 | |
| MinElute PCR Purification Kit | QIAGEN | 28004 or 28006 | for an elution volume of less than 30 µl |
| SPRI beads / Agencourt AMPure XP | Beckman Coulter | A63880 | apply extra mixing and more elution time if less than 40 µl elution buffer is used |
| Buffer EB | QIAGEN | 19086 | |
| NEBNext dsDNA Fragmentase | New England Biolabs | M0348 | supplied with buffer |
| T4 DNA Ligase Reaction Buffer | New England Biolabs | B0202 | |
| T4 DNA Polymerase | New England Biolabs | M0203 | |
| DNA Polymerase I, Large (Klenow) Fragment | New England Biolabs | M0210 | |
| T4 Polynuleotide Kinase | New England Biolabs | M0201 | |
| Klenow Fragment (3’ → 5’ exo-) | New England Biolabs | M0212 | supplied with buffer |
| sequencing adaptors | Integrated DNA Technologies | sequences available in ref. 28 | |
| Quick Ligation Kit | New England Biolabs | M2200 | used in 3.4.3; supplied with Quick Ligation Reaction Buffer and Quick T4 DNA Ligase |
| sequencing primer 1 and 2 | Integrated DNA Technologies | sequences available in ref. 28 | |
| KAPA HiFi PCR Kit | Kapa Biosystems | KK2101 or KK2102 | supplied with KAPA HiFi DNA Polymerase, 5x KAPA HiFi Fidelity Buffer and 10 mM dNTP mix |
| agarose | Sigma Aldrich | A4679 | |
| ethidium bromide | Sigma Aldrich | E1510-10ML | 10 mg/ml |
| QIAquick Gel Extraction Kit | QIAGEN | 28704 or 28706 | |
| iTaq Universal SYBR Green Supermix | Bio-Rad Laboratories | 1725121 or 1725122 | |
| Spectrophotometer | brand not critical | ||
| 0.45 μm PVDF Filter | brand not critical | ||
| 25 ml Seringe | brand not critical | ||
| 10 cm Tissue Culture Plates | brand not critical | ||
| 6-well Tissue Culture Plates | brand not critical | ||
| S1000 Thermal Cycler | Bio-Rad Laboratories | ||
| C1000 Touch Thermal Cycler | Bio-Rad Laboratories | for qPCR | |
| Vortex Mixer | brand not critical | ||
| Dry Block Heater or Thermomixer | brand not critical | ||
| Microcentrifuge | brand not critical | ||
| Gel electrophoresis system with power supply | brand not critical | ||
| Magnet stand | for purification of DNA with SPRI beads; should hold 1.5-2 ml tubes; brand not critical | ||
| UV transilluminator | brand not critical | ||
| E-gel electrophoresis system | Life Technologies | G6400, G6500, G6512ST |
Referências
- van Steensel, B., Delrow, J., Henikoff, S. Chromatin profiling using targeted DNA adenine methyltransferase. Nat Genet. 27, 304-308 (2001).
- van Steensel, B., Henikoff, S. Identification of in vivo DNA targets of chromatin proteins using tethered dam methyltransferase. Nat Biotechnol. 18, 424-428 (2000).
- Fu, A. Q., Adryan, B. Scoring overlapping and adjacent signals from genome-wide ChIP and DamID assays. Mol Biosyst. 5, 1429-1438 (2009).
- Guelen, L. Domain organization of human chromosomes revealed by mapping of nuclear lamina interactions. Nature. 453, 948-951 (2008).
- Kalverda, B., Pickersgill, H., Shloma, V. V., Fornerod, M. Nucleoporins directly stimulate expression of developmental and cell-cycle genes inside the nucleoplasm. Cell. 140, 360-371 (2010).
- Kubben, N. Mapping of lamin A- and progerin-interacting genome regions. Chromosoma. 121, 447-464 (2012).
- Steglich, B., Filion, G. J., van Steensel, B., Ekwall, K. The inner nuclear membrane proteins Man1 and Ima1 link to two different types of chromatin at the nuclear periphery in S. pombe. Nucleus. 3, 77-87 (2012).
- Harr, J. C. Directed targeting of chromatin to the nuclear lamina is mediated by chromatin state and A-type lamins. J Cell Biol. 208, 33-52 (2015).
- Gonzalez-Aguilera, C. Genome-wide analysis links emerin to neuromuscular junction activity in Caenorhabditis elegans. Genome Biol. 15, R21(2014).
- Wu, F., Yao, J. Spatial compartmentalization at the nuclear periphery characterized by genome-wide mapping. BMC Genomics. 14, 591(2013).
- Filion, G. J. Systematic protein location mapping reveals five principal chromatin types in Drosophila cells. Cell. 143, 212-224 (2010).
- Vogel, M. J. Human heterochromatin proteins form large domains containing KRAB-ZNF genes. Genome Res. 16, 1493-1504 (2006).
- Venkatasubrahmanyam, S., Hwang, W. W., Meneghini, M. D., Tong, A. H., Madhani, H. D. Genome-wide, as opposed to local, antisilencing is mediated redundantly by the euchromatic factors Set1 and H2A.Z. Proc Natl Acad Sci U S A. 104, 16609-16614 (2007).
- Shimbo, T. MBD3 localizes at promoters, gene bodies and enhancers of active genes. PLoS Genet. 9, e1004028(2013).
- Orian, A. Genomic binding by the Drosophila Myc, Max, Mad/Mnt transcription factor network. Genes Dev. 17, 1101-1114 (2003).
- Artegiani, B. Tox: a multifunctional transcription factor and novel regulator of mammalian corticogenesis. EMBO J. , (2014).
- Schuster, E. DamID in C. elegans reveals longevity-associated targets of DAF-16/FoxO. Mol Syst Biol. 6, 399(2010).
- Bianchi-Frias, D. Hairy transcriptional repression targets and cofactor recruitment in Drosophila. PLoS Biol. 2, e178(2004).
- Woolcock, K. J., Gaidatzis, D., Punga, T., Buhler, M. Dicer associates with chromatin to repress genome activity in Schizosaccharomyces pombe. Nat Struct Mol Biol. 18, 94-99 (2011).
- Luo, S. D., Shi, G. W., Baker, B. S. Direct targets of the D. melanogaster DSXF protein and the evolution of sexual development. Development. 138, 2761-2771 (2011).
- Germann, S., Gaudin, V. Mapping in vivo protein-DNA interactions in plants by DamID, a DNA adenine methylation-based method. Methods Mol Biol. 754, 307-321 (2011).
- Zhang, X. The Arabidopsis LHP1 protein colocalizes with histone H3 Lys27 trimethylation. Nat Struct Mol Biol. 14, 869-871 (2007).
- Orian, A. Chromatin profiling, DamID and the emerging landscape of gene expression. Curr Opin Genet Dev. 16, 157-164 (2006).
- Vogel, M. J., Peric-Hupkes, D., van Steensel, B. Detection of in vivo protein-DNA interactions using DamID in mammalian cells. Nat Protoc. 2, 1467-1478 (2007).
- Greil, F., Moorman, C., van Steensel, B. DamID: mapping of in vivo protein-genome interactions using tethered DNA adenine methyltransferase. Methods Enzymol. 410, 342-359 (2006).
- de Wit, E., Greil, F., van Steensel, B. Genome-wide HP1 binding in Drosophila: developmental plasticity and genomic targeting signals. Genome Res. 15, 1265-1273 (2005).
- DamID mammalian vectors. , Available from: http://research.nki.nl/vansteensellab/Mammalian_plasmids.htm (2015).
- Illumina TruSeq adaptors & PCR primers. , Available from: https://ethanomics.wordpress.com/chip-seq-library-construction-using-the-illumina-truseq-adapters/ (2015).
- Optimization of PCR cycles for NGS. , Available from: https://ethanomics.wordpress.com/ngs-pcr-cycle-quantitation-protocol/ (2015).
- Bernstein, B. E. The NIH Roadmap Epigenomics Mapping Consortium. Nat Biotechnol. 28, 1045-1048 (2010).
- Encode Project Consortium. A user's guide to the encyclopedia of DNA elements (ENCODE). PLoS Biol. 9, e1001046(2011).
- Asp, P. Genome-wide remodeling of the epigenetic landscape during myogenic differentiation. Proc Natl Acad Sci U S A. 108, E149-E158 (2011).
- Hoppe, P. S., Coutu, D. L., Schroeder, T. Single-cell technologies sharpen up mammalian stem cell research. Nat Cell Biol. 16, 919-927 (2014).
- Avital, G., Hashimshony, T., Yanai, I. Seeing is believing: new methods for in situ single-cell transcriptomics. Genome Biol. 15, 110(2014).
- Navin, N. E. Cancer genomics: one cell at a time. Genome Biol. 15, 452(2014).
- Saliba, A. E., Westermann, A. J., Gorski, S. A., Vogel, J. Single-cell RNA-seq: advances and future challenges. Nucleic Acids Res. 42, 8845-8860 (2014).
- Nagano, T. Single-cell Hi-C reveals cell-to-cell variability in chromosome structure. Nature. 502, 59-64 (2013).
- Kind, J. Single-cell dynamics of genome-nuclear lamina interactions. Cell. 153, 178-192 (2013).
- Southall, T. D. Cell-type-specific profiling of gene expression and chromatin binding without cell isolation: assaying RNA Pol II occupancy in neural stem cells. Dev Cell. 26, 101-112 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados