Method Article
DAMiD-seq: Genomweite Mapping von Protein-DNA-Wechselwirkungen von Hochdurchsatz-Sequenzierung von Adenin-methylierten DNA-Fragmenten
In diesem Artikel
Zusammenfassung
Wir beschreiben hier einen Test durch Kopplung DNA-Adenin-Methyltransferase-Identifikation (DAMiD), um Hochdurchsatz-Sequenzierung (DAMiD-seq). Dieses verbesserte Verfahren bietet eine höhere Auflösung und einen größeren Dynamikbereich und erlaubt die Analyse DAMiD-seq Daten in Verbindung mit anderen Hochdurchsatz-Sequenzierung Daten wie ChIP-seq, RNA-seq usw.
Zusammenfassung
The DNA adenine methyltransferase identification (DamID) assay is a powerful method to detect protein-DNA interactions both locally and genome-wide. It is an alternative approach to chromatin immunoprecipitation (ChIP). An expressed fusion protein consisting of the protein of interest and the E. coli DNA adenine methyltransferase can methylate the adenine base in GATC motifs near the sites of protein-DNA interactions. Adenine-methylated DNA fragments can then be specifically amplified and detected. The original DamID assay detects the genomic locations of methylated DNA fragments by hybridization to DNA microarrays, which is limited by the availability of microarrays and the density of predetermined probes. In this paper, we report the detailed protocol of integrating high throughput DNA sequencing into DamID (DamID-seq). The large number of short reads generated from DamID-seq enables detecting and localizing protein-DNA interactions genome-wide with high precision and sensitivity. We have used the DamID-seq assay to study genome-nuclear lamina (NL) interactions in mammalian cells, and have noticed that DamID-seq provides a high resolution and a wide dynamic range in detecting genome-NL interactions. The DamID-seq approach enables probing NL associations within gene structures and allows comparing genome-NL interaction maps with other functional genomic data, such as ChIP-seq and RNA-seq.
Einleitung
DNA-Methyltransferase-Adenin-Identifikation (DAMiD) 1,2 ist eine Methode, um Protein-DNA-Wechselwirkungen in vivo zu detektieren und ist ein alternativer Ansatz zur Chromatin-Immunopräzipitation (ChIP) 3. Es verwendet eine relativ geringe Menge an Zellen und keine chemische Vernetzung des Proteins mit der DNA oder einem hochspezifischen Antikörpers erforderlich. Letzteres ist insbesondere dann hilfreich, wenn das Zielprotein ist lose oder indirekt mit DNA verbunden. DAMiD wurde erfolgreich verwendet, um die Bindungsstellen aus einer Vielzahl von Proteinen, einschließlich Kernhüllenproteine 4-10, Chromatin-assoziierte Proteine 11-13, Chromatin modifizierender Enzyme 14, Transkriptionsfaktoren und Co-Faktoren und 15-18 RNAi Maschinen 19 abzubilden. Das Verfahren ist in mehreren Organismen, einschließlich S. erhoben cerevisiae 13, S. pombe 7, C. elegans 9,17, D. melanogaster 5,11,18,20, A. thaliana 21,22 sowie Maus und humanen Zelllinien 6,8,10,23,24.
Die Entwicklung des DAMiD Assay wurde auf den spezifischen Nachweis von Adenin-methylierten DNA-Fragmente in eukaryontischen Zellen, die endogene Adenin-Methylierung 2 fehlt basiert. Ein exprimiertes Fusionsprotein, bestehend aus der DNA-bindenden Proteins von Interesse und E. coli DNA-Adenin-methyltransferase (Dam), können die Adeninbase in GATC-Sequenzen, die in räumlicher Nähe befinden (am deutlichsten in 1 kb und bis zu etwa 5 kb) an die Bindungsstellen des Proteins in das Genom 2 methylieren. Die modifizierten DNA-Fragmenten, spezifisch amplifiziert und hybridisiert werden, um Mikroarrays, um die genomische Bindungsstellen des Proteins von Interesse 1,25,26 zu erfassen. Diese ursprüngliche DAMiD Verfahren wurde durch die Verfügbarkeit von Mikroarrays und der Dichte des vorbestimmten Sonden beschränkt. Daher haben wir in integrierten Hochdurchsatz-Sequenzierungum DAMiD 10 und bezeichnet das Verfahren, wie DAMiD-seq. Die große Anzahl von kurzen liest aus DAMiD-seq generiert ermöglicht eine präzise Lokalisierung von Protein-DNA-Interaktionen Genom-weiten. Wir fanden, dass DAMiD-seq vorgesehen eine höhere Auflösung und einen breiteren Dynamikbereich als DAMiD von Mikroarray für die Untersuchung genom Kernlamina (NL) 10 Verbände. Dieses verbesserte Verfahren ermöglicht Sondieren NL Assoziationen innerhalb Genstrukturen 10 und erleichtert den Vergleich mit anderen Hochdurchsatz-Daten, wie Chip-seq und RNA-seq.
Die hier beschriebene DAMiD-seq-Protokoll wurde ursprünglich für Mapping-Genom-NL Verbände 10 entwickelt. Wir erzielten ein Fusionsprotein angebunden Maus oder Mensch Lamin B1 bis E. coli DNA-Adenin-Methyltransferase und getestet, das Protokoll in 3T3 embryonalen Maus-Fibroblasten, Myoblasten C2C12 Maus 10 und IMR90 menschlichen fötalen Lungenfibroblasten (Daten nicht veröffentlicht). In diesem Protokoll, beginnen wir mit constructing Vektoren und ausdrücken Dam-gebundenen Fusionsproteine durch lentivirale Infektion in Säugetierzellen 24. Als nächstes beschreiben wir die ausführliche Protokolle zum Verstärken Adenin-methylierten DNA-Fragmente und Sequenzierung vorbereitet Bibliotheken, die in anderen Organismen anwendbar sein sollte.
Protokoll
1. Erzeugung und Expression von Fusionsproteinen und Free Dam Proteine
- Klon Protein von Interesse in das DAMiD Vektor.
- Amplifizieren cDNA des Proteins von Interesse (POI) mit dem gewünschten High Fidelity DNA-Polymerase und geeignete Primer gemäß dem Protokoll des Herstellers. Optimale Verstärkung Bedingungen experimentell zu bestimmen, um eine ordnungsgemäße Verstärkung der Einsätze zu gewährleisten.
- Führen Sie ein Agarose-Gel und zu reinigen, amplifizierte cDNA der POI von der Gelextraktionskit nach dem Protokoll des Herstellers.
- Clone cDNA des POI in die pDONR201 Vektor unter Verwendung BP Clonase II nach dem Protokoll des Herstellers.
- Klonen Sie die cDNA von POI aus dem Donor-Vektor in die PLGW-RFC1-V5-EcoDam Zielvektor oder der PLGW-EcoDam-V5-RFC1 Zielvektor 27 mit LR Clonase II nach dem Protokoll des Herstellers, abhängig von der gewünschten Richtung der Verschmelzung POI an den N-Terminus oder the C-Terminus des E. coli DNA-Adenin-Methyltransferase (EcoDam) 27.
- Überprüfen, die durch Sequenzierung der klonierten cDNA hat einen richtigen Reihenfolge und Formen in-frame Fusion an EcoDam.
- Generieren lentiviralen Aktien.
- Generieren lentiviralen Aktien zum Ausdruck Dam-V5-POI und V5-Dam (vom PLGW-V5-EcoDam Vektor 27) unter Verwendung von lentiviralen Expressionssysteme. Verwenden Sie den Vorwärts Transfektionsverfahren nach dem Protokoll des Herstellers.
- Verwenden 293T Zellen und Lipofektion lentivirale Bestände nach dem Protokoll des Herstellers für die Transfektion zu erzeugen.
- Fügen Sie die 0,45 um PVDF Filterstufe.
- Generieren lentiviralen Aktien zum Ausdruck Dam-V5-POI und V5-Dam (vom PLGW-V5-EcoDam Vektor 27) unter Verwendung von lentiviralen Expressionssysteme. Verwenden Sie den Vorwärts Transfektionsverfahren nach dem Protokoll des Herstellers.
- Infizieren Zellen mit Lentivirus.
- Am Tag vor der Infektion (Tag 0), vorbei an adhärenten Zellen in geeigneten Nährmedien kultiviert diesen Zelltyp zu einer 6-Well-Gewebekulturplatte unter Verwendung der gleichen Wachstumsmedium ohne Antibiotikaerreichen 50% Konfluenz am Tag der Infektion. Ortszellen in einem 37 ° C Inkubator.
- Am Tag der Infektion (Tag 1), entfernen 2 Kryoröhrchen von dem Dam-V5-POI und V5-Dam lentiviralen Stände aus einem -80 ° C Gefrierschrank und in einem 37 ° C Wasserbad auftauen.
- Entfernen Wachstumsmedien von Zellen und ersetzen Sie sie durch 0,5 ml frisches Wachstumsmedium ohne Antibiotika.
- 1 ml der aufgetauten Lentivirus in jede Vertiefung (2 Wells mit V5-Dam und 2 Brunnen mit Dam-V5-POI). 1 ml Wachstumsmedium ohne Antibiotika auf die verbleibenden 2 Vertiefungen-Dies wird als Negativkontrolle zu dienen. Schütteln Sie die Platte mit 6 Vertiefungen zu mischen und platzieren Sie zurück in einem 37 ° C Inkubator O / N.
- Der Tag nach der Infektion (Tag 2), entfernen Sie Virus-Suspensionen von Zellen und ersetzen mit 2 ml Wachstumsmedium ohne Antibiotika. Ortszellen wieder in einem 37 ° C Inkubator für 48 Stunden.
- Isolieren gDNA.
- Saugen Sie Medien aus jeder Vertiefung und detach Zellen unter Verwendung von 250 ul 0,05% Trypsin-EDTA. Inkubieren bei 37 ° C für 2 min.
- Wash-Zellen von der Platte mit 1 ml Wachstumsmedium und Pipetten Zellen aus jedem Well in ein 1,5 ml Mikrozentrifugenröhrchen. Pellet-Zellen durch Zentrifugieren bei 200 xg für 5 Minuten bei RT.
- Waschen pelletierten Zellen mit 500 ul PBS und Zentrifugation bei 200 × g für 2 min bei RT.
- Sedimentierte Zellen in 200 ul PBS.
- Isolieren gDNA von Blut und Gewebe-Kit nach Vorschrift des Herstellers. Eluieren gDNA in 200 ul Puffer AE und bestimmen die Konzentration durch Messung der OD 260 unter Verwendung eines Spektrophotometers.
Hinweis: gDNA von nicht infizierten oder mock-infizierten Zellen können als negative Kontrollen isoliert werden. Auszufällen gDNA zu höheren Konzentrationen für langfristige Lagerung.- Werden 3 Volumina 100% Ethanol und 0,1 Volumen 3 M Natriumacetat (pH 5,5) und durch Umschütteln Rohre 4-6 mal.
- Lagerung bei -20 ° CO / N.
- Zentrifuge bei 16.000 xg für 15 min bei 4 ° C.
- Den Überstand vorsichtig entfernen. Waschen Pellets mit 70% (Volumen / Volumen) Ethanol und Zentrifugieren bei 16.000 xg für 5 Minuten bei 4 ° C.
- Ethanol Entfernen Sie vorsichtig und lassen Pellets an der Luft trocknen für 3 min bei RT.
- Aufzulösen gDNA in T 10 E 0,1 (pH 7,5) auf ungefähr 1 ug / ul. Pool gDNA aus jeder Versuchsprobe oder der negativen Kontrolle und Messung der Konzentration. Lagerung bei -20 ° C.
2. Amplify Adenin-methylierten DNA-Fragmenten
- Digest gDNA mit DpnI, die nur schneidet Adenin-methylierten GATCs.
- Eine Reaktion auf Eis mit 2,5 ug gDNA, 1 ul 10x-Puffer, 0,5 & mgr; l DpnI (20 U / ul) und Fülleigenschaften mit H 2 O auf ein Gesamtvolumen von 10 ul. Für jede Probe gDNA, bereiten drei Reaktionen - eine ohne DpnI ("no DpnI", ersetzen Sie DpnI mit 0,5 & mgr; l H 2 O) und zwei with DpnI ("mit DpnI").
- Digest O / N bei 37 ° C inaktiviert DpnI bei 80 ° C für 20 min.
- Ligieren DAMiD Adapter.
- Bereiten DAMiD Adapter.
- Resuspendieren jeder der beiden DAMiD Adapter Oligos 24 in H 2 O zu einer Endkonzentration von 100 uM.
- Kombinieren Sie gleiche Volumina der beiden DAMiD Adapter Oligos, mischen und in einem dicht geschlossenen Rohr. Verschließen Sie die Röhrchen mit Parafilm, sitzen in einem Gestell und in ein Becherglas mit Wasser bei 90 ° C gefüllt. Halten der Wasserstand unter dem Deckel des Rohres (Wasser vermeidet, in der Röhre), aber über der Oberfläche der Oligo-Mix.
- Lassen Sie das Wasser auf RT abkühlen, so dass die Adapter Glühen langsam.
- Aliquot der geglühten Adaptern (50 uM) und bei -20 ° C.
- Eine Reaktion auf Eis gesetzt. In jedes Röhrchen aus 2.1.2, fügen 6,2 & mgr; l H 2 O, 2 ul 10x Ligationspuffer, 0,8 ul 50 uM DAMiD adaptors (auf Eis aufgetaut) und 1 ul T4 DNA Ligase (5 U / ul). Das Gesamtvolumen beträgt 20 & mgr; l. In einem der beiden "mit DpnI" Rohre ersetzen Ligase mit 1 & mgr; l H 2 O ("no-Ligase") Beachten Sie, dass jedes gDNA Probe hat zwei negative Kontrollen -. "Keine DpnI" und "nicht-Ligase".
- Ligat O / N bei 16 ° C und inaktivieren Ligase bei 65 ° C für 10 min.
- Bereiten DAMiD Adapter.
- Digest-DNA mit DpnII um Fragmente, die nicht methylierten GATCs enthalten zerstören.
- Eine Reaktion auf Eis gesetzt. In jeder Röhre von 2.2.3, fügen 24 ul H & sub2; O, 5 ul 10x DpnII-Puffer und 1 ul DpnII (10 U / ul). Das Gesamtvolumen beträgt 50 & mgr; l.
- Verdau bei 37 ° C für 2-3 h, und inaktivieren DpnII bei 65 ° C für 20 min.
- Verstärken Adenin-methylierten DNA-Fragmente.
- Einrichten einer Reaktions auf Eis mit 5 ul DpnII von 2.3.2, 5 μ verdauenl 10x PCR-Puffer, 12,5 ul 5 uM DAMiD PCR-Primer 24, 4 & mgr; l 10 mM dNTP Mix, 1 ul 50x Polymerase-Mix und 22,5 & mgr; l H 2 O. Das Gesamtvolumen beträgt 50 & mgr; l.
- Führen Sie die PCR wie folgt: 68 ° C für 10 min; 94 ° C für 1 min, 65 ° C für 5 min, 68 ° C für 15 min; 4 Zyklen bei 94 ° C für 1 min, 65ºC für 1 min, 68 ° C für 10 min; 17 Zyklen von 94ºC für 1 min, 65ºC für 1 min, 68 ° C für 2 min.
- Analyse 5 & mgr; l PCR-Produkte aus jeder Reaktion auf einem 1% Agarosegel. PCR-Produkte sollten als Abstrich von 0,2 bis 2 kb (Abbildung 2) angezeigt. Die "no DpnI" und "nein Ligase" Kontrollen sollten keine oder deutlich weniger Verstärkung.
- Wenn das Ergebnis aus Schritt 2.4.3 zufriedenstellend ist, wiederholen Sie die Schritte 2.4.1-2.4.3 mit zwei oder drei Reaktionen für die Versuchsprobe und eine Reaktion für jede der beiden negativen Kontrollen.
- Pool und Reinigung PCR Produkte aus derselbenVersuchsprobe unter Verwendung der PCR-Reinigungs-Kits oder festen Phase reversible Immobilisierung (SPRI) Perlen gemäß dem Protokoll des Herstellers. Reinigen Sie nicht "keine DpnI" oder "nein Ligase" Kontrollen. Eluieren der DNA mit Puffer EB.
- Messung der Konzentration der gereinigten DNA durch Messung der OD 260 unter Verwendung eines Spektrophotometers, das etwa 0,1 ug / ul oder höher sein sollte. Sammeln von mindestens 10 & mgr; g DNA für jede Probe. Bei der Verwendung von PCR-Reinigungs-Kits, reinigen, je 50 & mgr; l PCR-Produkte mit einer Säule, eluiert in 30 ul Puffer EB und Pool der Eluate.
- Digest-DNA mit DpnII zu DAMiD PCR-Primer aus sequenziert verhindern.
- Bis eine Reaktion auf Eis mit 5 ug gereinigte DNA von 2.4.6, 5 ul 10x DpnII Puffer, 1 ul DpnII (10 U / ul) und Fülleigenschaften mit H 2 O auf ein Gesamtvolumen von 50 ul. Vorbereitung zwei oder drei Reaktionen für jede Probe.
- Verdauen bei 37 ° C für 2-3 Stdund inaktivieren DpnII bei 65 ° C für 20 min.
- Pool und Reinigung der Verdaus aus der gleichen Probe mit der PCR-Reinigungs-Kits oder SPRI Perlen nach dem Protokoll des Herstellers. Eluieren der DNA mit Puffer EB.
- Messung der Konzentration der gereinigten DNA, das um 0,06 & mgr; g / & mgr; l oder höher sein sollte. Sammeln von mindestens 6 & mgr; g DNA für jede Probe. Bei der Verwendung von PCR-Reinigungs-Kits, reinigen jeweils 50 ul mit einer Spalte zu verdauen, zu eluieren in 30 ul Puffer EB und Pool der Eluate.
3. Bibliothek Vorbereitung zum Hochdurchsatz-Sequenzierung
- DNA-Fragment,
- Experimentell die geeignete Aufschlusszeit für jede neue Charge von dsDNA Fragmentase. Da die Enzymaktivität über die Zeit zu verringern, wiederholen Sie den Test, bevor Sie ein neues Experiment. DNA von 2.5.4 für die tatsächliche Fragmentierung zu speichern, verwenden gereinigte Methyl PCR-Produkte von 2.4.6 oder zusätzliche DNA aus früheren Experiments bei diesem Schritt.
- Einen Master-Mix mit 6 ug DNA, 12 ul 10x Fragmentase Puffersatz füllen und mit H 2 O auf ein Gesamtvolumen von 114 & mgr; l.
- Vortex die Fragmentase Vorratsröhrchen für 3 Sekunden, fügen Sie 6 & mgr; l in die Master-Mix und Vortex den Master-Mix für 3 Sekunden. Das Gesamtvolumen beträgt 120 ul.
- Aliquot 20 ul des Master-Mix zu jedem der 5 neuen Röhren. Alle 6-Röhrchen bei 37 ° C für 5 bis 55 min bei einer Schrittweite von 10 min inkubiert. Werden 5 ul 0,5 M EDTA zum Stoppen der Reaktion.
- Analyse 12,5 ul verdauen (0,5 ug DNA) jeder Reaktion sowie 0,5 ug unverdaute DNA auf einem Agarosegel (Abbildung 3). Bestimmen Sie die minimale Zeit (T 0.2kb) benötigt, um den Großteil der Abstrich auf etwa 0,2 kb zu verdauen. Wählen Sie 6 Zeitdauern zwischen 5 min und T 0.2kb (einschließlich 5 min und T 0.2kb) mit gleichen Schritten für die tatsächliche Fragmentierung.
- Richten Sie die tatsächlichen Fragmentierungtation wie in 3.1.1.1-3.1.1.3 beschrieben. Inkubieren Reaktionen bei 37 ° C für die Zeitdauern in 3.1.1.4 ermittelt.
- Pool 6 Reaktionen und Reinigung der Verdauungsprodukte mit PCR-Reinigungs-Kits oder SPRI Perlen nach dem Protokoll des Herstellers. Eluieren der DNA in 51 ul Puffer EB. Der letzte Eluat ~ 50 & mgr; l.
- Experimentell die geeignete Aufschlusszeit für jede neue Charge von dsDNA Fragmentase. Da die Enzymaktivität über die Zeit zu verringern, wiederholen Sie den Test, bevor Sie ein neues Experiment. DNA von 2.5.4 für die tatsächliche Fragmentierung zu speichern, verwenden gereinigte Methyl PCR-Produkte von 2.4.6 oder zusätzliche DNA aus früheren Experiments bei diesem Schritt.
- Reparatur endet der fragmentierte DNA
- Eine Reaktion auf Eis mit dem Eluat aus 3.1.3, 25 & mgr; l H 2 O eingestellt, 10 ul 10x T4 DNA-Ligase-Puffer mit 10 mM ATP, 4 ul 10 mM dNTP-Mix, 5 ul T4-DNA-Polymerase (3 U / ul) 1 ul Klenow-DNA-Polymerase (5 U / ul) und 5 ul T4-Polynukleotid-Kinase. Das Gesamtvolumen beträgt 100 ul. Gut mischen mit Pipette. Schaum und Blasen vermeiden.
- Inkubieren bei 20 ° C für 30 min.
- Reinigen der DNA mit PCR-Reinigungs-Kits oder SPRI Perlen nach dem Protokoll des Herstellers. Eluieren der DNA in 33 ul Puffer EB. Der letzte Eluat ~ 32 & #181; l.
- In "A" Überhänge
- Bis eine Reaktion auf Eis mit dem Eluat aus 3.2.3 5 ul 10x Klenow-Puffer, 10 ul 1 mM dNTP, und 3 & mgr; l Klenow (3 '→ 5'-Exo-) (5 U / ul) eingestellt. Das Gesamtvolumen beträgt 50 & mgr; l. Gut mischen mit Pipette. Schaum und Blasen vermeiden.
- Inkubieren bei 37 ° C für 30 min.
- Reinigen der DNA mit PCR-Reinigungs-Kits oder SPRI Perlen nach dem Protokoll des Herstellers. Eluieren der DNA in 22 ul Puffer EB. Der letzte Eluat ~ 21 & mgr; l.
- Ligat Sequenzierung Adapter
- Bereiten Sequenzierung Adapter 28.
- Resuspendieren jeden Adapter Oligo auf eine Konzentration von 100 uM in 10 mM Tris-Cl (pH 7,8), 0,1 mM EDTA (pH 8,0) und 50 mM NaCl.
- Mischen Sie gleiche Volumina der Universal-Adapter 28 und einem indizierten Adapter 28.
- Annealing der Adapter in einem Thermocycler mit folgendemProgramm: 95 ° C für 2 min; 140 Zyklen für 30 Sekunden beginnend bei 95 ° C und verringert sich um 0,5 ° C pro Zyklus; Halten bei 4 ° C.
- Aliquot Adaptern und bei -20 ° C.
- Eine Reaktion auf Eis mit dem Eluat aus 3.3.3, 25 ul 2x Ligationspuffer, 1,5 ul 50 uM geglüht Sequenzierung Adaptern (auf Eis aufgetaut) von 3.4.1.4 und 2,5 ul T4 DNA-Ligase gesetzt. Gut mischen mit Pipette. Schaum und Blasen vermeiden. Wenn Multiplex-Sequenzierung gewünscht wird, verwenden Sie einen anderen indizierten Adapter für jede Probe.
- Inkubieren bei Raumtemperatur für 1 Std.
- Reinigen der DNA mit PCR-Reinigungs-Kits oder SPRI Perlen nach dem Protokoll des Herstellers. Eluieren der DNA in 24 ul Puffer EB. Der letzte Eluat ~ 23 & mgr; l.
- Bereiten Sequenzierung Adapter 28.
- Konvertieren Y-Adapter, um dsDNA genau zu aktivieren Bestimmung DNA-Fragment, NG 28
- Einrichten einer Reaktions auf Eis mit dem Eluat aus 3.4.4, 12,5 ul H 2 O, 1 ul 25 uM Primer 1 28, 1 ul 25 uM Primer 2 28, 1,5 ul 10 mM dNTP, 10 ul 5x PCR-Puffer und 1 ul DNA-Polymerase (1 U / ul).
- Führen Sie die PCR wie folgt: 95 ° C für 3 min; 5 Zyklen von 98 ° C für 15 sec, 63 ° C für 30 sec, 72 ° C für 30 sec; 72 ° C für 1 min; 4 ° C gehalten.
- Reinigen der DNA mit PCR-Reinigungs-Kits oder SPRI Perlen nach dem Protokoll des Herstellers. Eluieren der DNA in 11 ul Puffer EB. Der letzte Eluat ~ 10 & mgr; l.
- Größe wählen Sie die Bibliothek
- Vorbereitung einer 2% Agarose-Gel mit 1 x TAE-Puffer. Hinzufügen Ethidiumbromid (Vorsicht!) Zu einer Endkonzentration von 500 ng / ml, wenn das geschmolzene TAE-Agarose-Lösung wurde gekühlt, um die Inhalation von Ethidiumbromid zu vermeiden. Stellen Sie sicher, genügend Fahrspuren für alle Proben, DNA-Leiter und leere Gassen haben.
- Hinzufügen 8 ul 6x Ladefarbstoff zu dem Eluat aus 3.5.3.
- Vorbereiten DNA-Leiter durch Mischen von 1 kb DNA-Leiter und (1,0 ug / ul), 6x Ladefarbstoff und H 2 O in einem Verhältnis von 1: 1: 4.
- Last 6 & mgr; l DNA-Leiter, die Proben aus 3.6.2 und weitere 6 & mgr; l DNA-Leiter, die jeweils in einer separaten Spur und mit mindestens einer leeren Spur von den benachbarten Proben / Leitern.
- Führen Sie das Gel bei 120 V für 60 min.
- Sehen Sie das Gel auf einem UV-Transilluminator (minimieren, die Belichtungszeit auf UV). Eine Schutzbrille und einen Gesichtsschutz. So dass sie mindestens eines der DNA-Leitern mit den entsprechenden Abstand (genug zum Ausschneiden 3 Gelscheiben) zwischen 300 bp und 400 bp Banden gut laufen. Schmalabstand erhöht die Schwierigkeiten bei der Herausschneiden mehrerer Gelscheiben, während weiten Abstand erhöht das Volumen der ausgeschnitten Gelscheiben.
- Verwenden Sie ein neues Skalpell oder einer Rasierklinge für jede Spur. Verbrauch drei dünne Gelscheiben zwischen 300 und 400 bp von jeder Spur (Abbildung 4) und legen Sie sie jeweils in einem Mikrozentrifugenröhrchen. Halten Sie die Lautstärke der einzelnen Gel sLäuse möglichst gering (<100 & mgr; l).
- Messen Sie die Lautstärke der einzelnen Gelstück (1 ul Gel wiegt etwa 1 mg), fügen 6x Mengen QG-Puffer und Inkubation bei 50 ° C.
- Mischen Sie den QG-Gel-Mischung alle 2-3 Minuten, bis das Gelstück vollständig gelöst hat. In 2x Gel Volumina Isopropanol und mischen.
- Von diesem Schritt folgen das Protokoll von Gel Extraction Kits. Eluieren der DNA in 51 ul Puffer EB. Der letzte Eluat ~ 50 & mgr; l.
- Bereichern Sequenzierung Adapter modifizierten DNA-Fragmente
- Optimieren Sie die Anzahl der PCR-Zyklen 29.
- Eine Reaktion auf Eis mit 1 ul Eluat von 3.6.10, 1 ul 25 uM Primer 1 28, 1 ul 25 uM Primer 2 28, 7 & mgr; l H 2 O und 10 ul SYBR Green Supermix eingestellt. Das Gesamtvolumen beträgt 20 & mgr; l.
- Führen Sie die qPCR wie folgt: 95 ° C für 3 min; 20 Zyklen von 95 ° C für 30 sec, 63 ° C für 30 sec, 72 ° C für 30 sec, gelesen Platte.
- Analyse von Daten unter Verwendung eines qPCR-Analyse-Software und bestimmen die Quantifizierung Zyklus (CC) oder Schwellenzyklus (Ct) für jede Probe unter Verwendung des Protokolls des Herstellers. Fahren Sie mit den Proben, die Cq / Ct ≤ 14. Verwenden Sie haben die maximale Cq (CQ 0) minus 1 (bis auf die nächsthöhere ganze Zahl gerundet) als die endgültige PCR-Zyklenzahl (N PCR).
- Einstellen der Mengen an DNA-Matrizen, so dass verschiedene Proben werden nach der Ausführung die gleiche Anzahl von PCR-Zyklen auf etwa gleiche Mengen amplifiziert werden. Verwenden 8 ul template für die Probe mit der höchsten Cq (Cq 0), und die Berechnung der Vorlage Lautstärke anderer Proben mit der folgenden Formel:
Vol i = 8 x 1,8 Cq i -cq 0
- 1 ul 25 uM Primer 1 28, 1 ul 25 uM Primer 2 28, 1,5: bis PCR-Reaktionen mit den von 3.7.1.4 berechneten Vorlage Volumes auf Eisul 10 mM dNTP, 10 ul 5x Puffer, 1 ul DNA-Polymerase (1 E / ul) und füllen sich mit H 2 O auf ein Gesamtvolumen von 50 ul.
- Führen Sie die PCR wie folgt: 95 ° C für 45 sec; N PCR Zyklen (in 3.7.1.3 bestimmt) von 98 ° C für 15 sec, 63 ° C für 30 sec, 72 ° C für 30 sec; 72 ° C für 1 min; 4 ° C gehalten.
- Analyse 5 & mgr; l PCR-Produkte in einem 2% igen Agarosegel (5A). Ein klares "single" Band zeigt, dass die amplifizierte DNA-Fragmente sind in einem engen Längenbereich und dass die DNA-Bibliothek kann vorbehaltlich weiterer Analysen sein.
- Wiederholen einer Reaktion für ausgewählte Proben mit Schwimmbad Das amplifizierte DNA-Bibliotheken, die aus der gleichen Probe.
- Reinige ausgewählten DNA-Bibliotheken für die Sequenzierung.
- Wenn Primer / Adapter Dimere sind in 3.7.4 sichtbar reinigen DNA-Bibliotheken mit PCR-Reinigungs-Kits oder SPRI Perlen nach dem Protokoll des Herstellers. Eluieren der DNA in 21 ul Puffer EB. dasAbschluss Eluat ~ 20 & mgr; l. Wenn Sequenzierung Anlage des Benutzers hat spezifische Anweisungen auf dem Elutionspuffer, der endgültige Volumen etc., bereiten Proben entsprechend.
- Wenn Primer / Adapter Dimere sind deutlich in 3.7.4 sichtbar, reinigen Bibliotheken wie folgt.
- Last DNA aus 3.7.5 und DNA Ladder in einer 2% Fertig Agarosegel. Proben können wie in 3.7.6.1 beschrieben, um das Volumen zu reduzieren oder sie können in mehreren Mulden eingelegt werden, gereinigt werden. Legen Sie die Agarose-Gel in eine geeignete Stromsystem. Lassen Sie das Gel für 15 Minuten laufen.
- Mit Hilfe eines geeigneten Transilluminator, schneiden Sie den gewünschten Wellenbereich mit einem sauberen Skalpell / razor für jede Probe und legen Sie das Gelstück in einem 1,5 ml Mikrozentrifugenröhrchen.
- Isolieren Sie DNA Gelextraktionskits nach dem Protokoll des Herstellers mit zwei Modifikationen: inkubieren Puffers QG-Gel-Mischung in einem Thermomixer bei 37 ° C und 1000 rpm für 30 min, und fügen 2x Gelvolumen Isopropanol.
- Optimieren Sie die Anzahl der PCR-Zyklen 29.
- Senden gereinigten DNA-Bibliotheken, um eine Sequenzierung Möglichkeiten. Befolgen Sie alle Anlage-Richtlinien.
Hinweis: Ein Qualitätsanalyse von einem Bioanalyzer (5B) sollte vor der Sequenzierung, um die genaue Größenbereich und die Konzentration einer DNA-Bibliothek bestimmen, durchgeführt werden.
Ergebnisse
Dam-V5-LmnB1 Fusionsprotein wurde überprüft, um mit dem endogenen Lamin B-Protein durch Immunfluoreszenzfärbung (Abbildung 1) co-lokalisiert werden.
Die erfolgreiche PCR-Amplifikation von Adenin-methylierten DNA-Fragmente ist ein wichtiger Schritt für DAMiD-seq. Die Versuchsproben sollten einen Abstrich von 0,2 zu verstärken - 2 kb, während der Negativkontrollen (ohne DpnI ohne Ligase oder ohne PCR-Vorlage) sollte in keiner oder eindeutig weniger Verstärkung (Abbildung 2) zur Folge haben.
Das methylierte DNA-Fragmente im Bereich von 0,2 bis 2 kb, während die gewünschte Plattengröße für eine NGS Bibliothek 200-300 bp. Daher ist es wichtig, die Methyl PCR-Produkte in den geeigneten Grßenbereich fragmentieren. Dennoch wurde festgestellt, nicht praktikabel, gleichzeitig brechen größere DNA-Fragmente auf geeignete Größe seins und halten die Mehrheit der kleineren DNA-Fragmente intakter in einem einzigen Fragmentierung Dauer. Daher wurden Zeitverlaufsexperimente durchgeführt, um die minimale Zeit (T 0.2kb) bis 1 ug DNA-Fragment zu einem Abstrich bei 200 bp (3) zentriert ist notwendig zu bestimmen. Dann wurden 6 Zeitdauern in gleichen Schritten zwischen 5 min und T 0.2kb für die tatsächliche Fragmentierung ausgewählt. Die enzymatische Aktivität der Doppelstrang-DNA Fragmentase kann von Charge zu Charge variieren und kann im Laufe der Zeit zu verringern, so empfiehlt es sich, diesen Schritt für eine neue Charge von Fragmentase oder nach Lagerung über einen Zeitraum zu wiederholen.
Die gewünschte Plattengröße zwischen 200 und 300 bp entsprechen, DNA-Fragmente zwischen 300 und 400 bp (einschließlich 121 bp Sequenzierung Adaptern) auf dem Agarosegel. Drei dünne Scheiben in diesem Bereich wurden von jeder Versuchsprobe herausgeschnitten, um den Größenbereich von einer Bibliothek einzugrenzen und erhöhen die Möglichkeit,erhalten mindestens einen qualifizierten Sequenzierung Bibliothek (Abbildung 4).
Ein Aliquot von 5 ul jeder amplifizierten DNA-Bibliothek wurde auf dem Agarosegel analysiert, um zu bestimmen, welche Bibliothek für die Sequenzierung in Betracht kommen. Wie in 5A gezeigt ist, sollte eine klare einzelne Bande mit der gleichen Größe wie der ausgeschnittenen Gelscheibe auf dem Agarosegel (Schritt 3.7.4) sichtbar. Als nächstes wurden die ausgewählten Bibliotheken von einem Bioanalyzer (5B) untersucht, um die genaue Größenbereich und Konzentrationen vor der Sequenzierung zu bestimmen. Falls gewünscht, amplifizierte DNA-Bibliotheken können direkt von einem Bioanalyzer ohne Gel-Analyse untersucht werden. Wenn mehrere Bibliotheken sind von guter Qualität, ist es empfehlenswert, zu sequenzieren Bibliotheken ähnlicher Größe reicht für ein Paar von experimentellen (Zellen, die Dam-V5-POI) und Kontrolle (Zellen, die V5-Dam) Proben.
Die kurze Lesevorgänge erzeugtSequenzierungssysteme wurden zum ersten Mal wieder auf die entsprechende Genom abgebildet. Eindeutig ausgerichtet liest wurden dann in nachfolgenden Analysen übergeben. Eine Pipeline zu verarbeiten kurzen liest, errichten ein Genom-NL Interaktionskarte und zu analysieren Gen-NL Verbände wurden im Detail in unserer bisherigen Arbeit 10 beschrieben. Repräsentative Ergebnisse sind in 6 gezeigt.

Abbildung 1 Validieren subnuclear Lokalisierungen von Fusionsproteinen durch Immunfluoreszenzfärbung. IMR90 menschlichen Lungenfibroblasten-Zellen wurden transient mit einem Plasmid, Dam-V5-LmnB1 transfiziert und wurden durch anti-Lamin B (A, rot) und Anti-V5 (B gefärbt, grün). (C) Zusammenführen von Bildern in A und B. Bitte klicken Sie hier, um eine größere Version zu sehen tseine Figur.

Abbildung 2. PCR Amplifikation Adenin-methylierten DNA-Fragmente. Ein aliquoter Teil von 5 & mgr; l von jeder PCR-Reaktion wurde auf einem 1% Agarosegel analysiert. Ein Abstrich von 0,2 bis 2 kb wurde aus jeder Versuchsprobe verstärkt, aber keine Amplifikation wurde in Negativkontrollen beobachtet (kein DpnI in Schritt 2.1, keine Ligase in Schritt 2.2, oder kein PCR-Vorlage). Zu Bitte klicken Sie hier eine größere Ansicht dieser Figur.
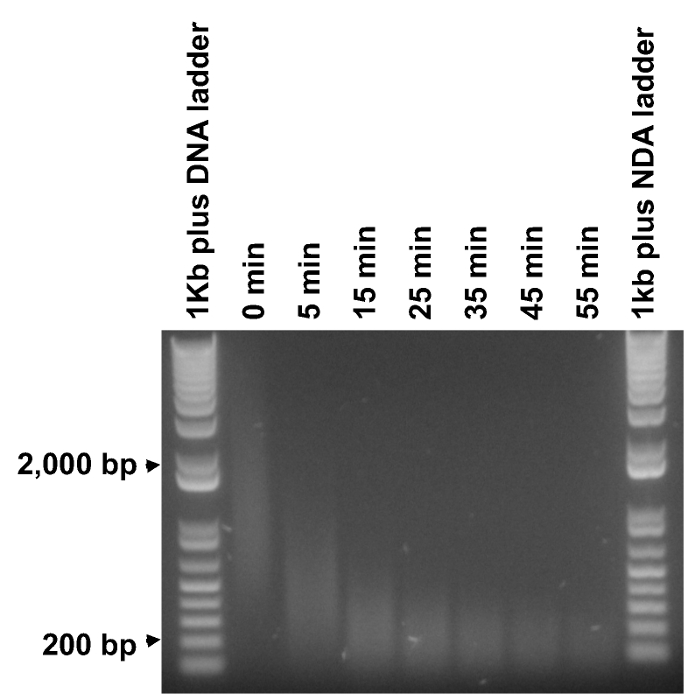
Abbildung 3. Bestimmen der optimalen Dauer der Fragmentierung. Gereinigtes methyl PCR-Produkte wurden unter Fragmentierung für Zeitdauern von 5 bis 55 min bei einer Schrittweite von 10 min (unverdaute DNA Als "0 min") markiert. 1 ug DNA-Leiter und 0,5 ug jedes fragmentierten DNA-Probe wurden auf einem Agarosegel analysiert. Die minimale Zeit, um den Großteil der DNA-Abstrich auf etwa 200 bp verdaut wurde bestimmt auf etwa 35 min, also sechs gleichmäßig beabstandeten Zeitdauern zwischen 5 und 35 min (5 und 35 min enthalten) wurden ausgewählt, um die tatsächliche Fragmentierung durchzuführen. Bitte Klicken Sie hier, um eine größere Version dieser Figur zu sehen.

Abbildung 4. Größe der Auswahl der DNA-Bibliotheken. DNA-Proben der Dam-V5-LmnB1 und V5-Dam von Schritt 3.6 wurden auf einem 2% Agarosegel laufen gelassen, und drei gleich große Gelscheiben (L, M und H entsprechend niedrig, mittel und hoch in der Größe) wurden zwischen 300 und 400 bp ausgeschnitten, wie durch die gelben Linien dargestellt. om / files / ftp_upload / 53.620 / 53620fig4large.jpg "target =" _ blank "> Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
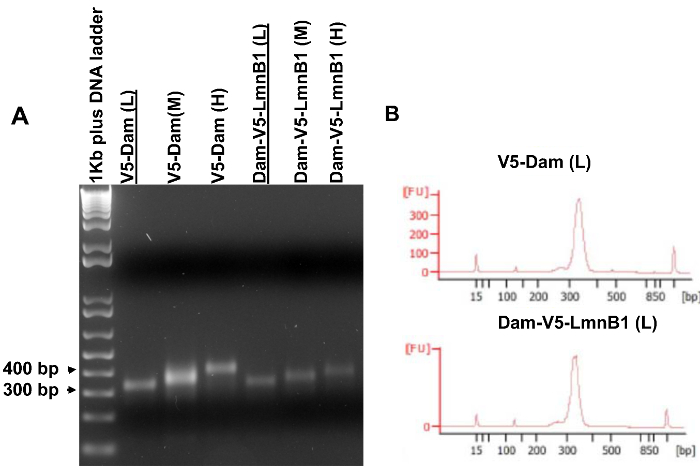
Figur 5. Amplified DNA-Bibliotheken für Hochdurchsatz-Sequenzierung. (A) Die amplifizierten DNA-Bibliotheken auf einem Agarosegel analysiert. PCR-Templates wurden von über 4 gezeigten Gelscheiben gereinigt. (B) Bioanalyzer Ergebnisse der V5-Dam (L) Probe und dem Dam-V5-LmnB1 (L) Probe, die in (A) unterstrichen wurden. Diese beiden Bibliotheken hatten die ähnliche enge Größenbereiche und damit qualifiziert für Hochdurchsatz-Sequenzierung. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
jpg "/>
Abbildung 6. NGS Daten im UCSC Genome Browser. Maus-Chromosom 1 angezeigt wird, als ein Beispiel gezeigt. Sequenzdaten wurden aus Maus-C2C12 Myoblasten 10 produziert. Tracks "MB.LmnB1.w2k" und "MB.Dam.w2k", entsprechend Daten von Zellen, die Dam-V5-LmnB1 und V5-Dam bzw. Grundstück normalisiert Lesedichten (Lesevorgänge pro Kilobase pro Million eindeutig zugeordnet liest oder RPKM ) in nicht überlappenden aufeinanderfolgenden 2 kb Fenster entlang des Chromosoms. Verfolgen "MB.log2FC.w2k" Grundstücke Genom-NL Verbände, dh log2 RPKM Verhältnisse LmnB1 über Dam, bei 2 kb Auflösung. Verfolgen "MB.sLADs" malt Sequenzierung basierende Lamina verbundenen Domänen (sLADs, dh genomische Regionen, die höhere Lesedichten LmnB1 über Dam mit statistischer Signifikanz haben) in Schwarz, nicht-sLADs in grau und unbestimmten Regionen in weiß. Bitte klicken Sie hier, um downloaD Diese Datei.
Diskussion
Whether Dam-tagged proteins retain the functions of endogenous proteins should be examined before a DamID-seq experiment. The subcellular localization of Dam-tagged nuclear envelope proteins should always be determined and compared with that of the endogenous proteins. For studying transcription factors, it is suggested to examine whether the Dam-fusion protein can rescue the functions of the endogenous protein in regulating gene expression. This functional test can be performed in organisms in which knockout mutants of endogenous DNA-binding proteins are available. Because advances in genome engineering have potentially allowed knocking out any endogenous gene of interest, functions of Dam-tagged DNA-binding proteins can be examined in cultured mammalian cells.
The critical step in this protocol is to successfully fragment the DpnII-digested DamID PCR products to around 200 bp. This step is designed to render the amplified adenine-methylated fragments to a narrow size range for sequencing and to randomize the starting nucleotides of the DNA fragments in a sequencing library. Inefficient fragmentation will leave the majority of the DNA fragments starting with GATC (the 5'-overhang from the second DpnII digestion), and will result in a much lower performance and yield or even a failure in Illumina sequencing. Other DNA fragmentation methods may be used as an alternative approach.
The resolution of DamID (and DamID-seq described here) is limited by the frequency of GATCs in the genome to be studied. Moreover, even with high throughput sequencing, the genomic localizations of a DNA-binding protein can only be mapped within two consecutive GATCs rather than to the actual DNA-binding sites.
Despite its limitation, the DamID assay has important advantages. Because DamID does not require highly-specific antibodies, it can be used to detect a subset of nuclear proteins that could be difficult to assay by ChIP (such as the nuclear envelope proteins). To study how these proteins regulate genome functions, it is important to integrate and cross-analyze their genome-wide localization data with the current epigenomic mapping data (such as data from the ENCODE and NIH Roadmap Epigenomics Projects 30,31). The DamID-seq approach provides both higher resolution and higher sensitivity than DamID by microarray and enables detecting differential NL-associations within gene structures 10. A combinatorial analysis of DamID-seq data, ChIP-seq data 32 and gene expression data has identified a class of NL-associated genes with distinct epigenetic and transcriptional features (data not published).
Another advantage of DamID is that it only requires a small number of cells. In recent years, there has been an explosion in single cell analysis of gene regulation 33,34. Although genome sequence 35, genome-wide gene expression 36 and chromatin conformation 37 can be assayed in a single cell, there has not been an available approach for detecting protein-DNA interactions genome-wide in a single cell. DamID-seq is a highly promising approach for this goal, and may complement the single cell imaging approach in detecting the dynamics of genome-NL interactions 38. One complication is that because the Dam-fusion protein is expressed at a much lower level than the endogenous protein in the DamID assay, it is possible that the Dam-fusion protein may only occupy a subset of genomic binding sites as compared to the endogenous protein.
DamID assay has mostly been used in cultured animal cells to detect protein-DNA interactions. Notably, developmental biologists have applied this assay in detecting protein-DNA interactions in specific cell types in vivo. For example, Dam-tagged RNA polymerase II was expressed specifically in Drosophila neural stem cells to detect their genome-wide occupancy without cell isolation 39. DamID-seq will be highly useful to study the genome-wide localizations of nuclear envelope proteins, transcription factors and chromatin regulators during development in animal models.
Offenlegungen
The authors have nothing to disclose.
Danksagungen
We thank Dr. Bas van Steensel for providing the DamID mammalian expression vectors. We thank Yale Center for Genome Analysis and the Genomics Core in Yale Stem Cell Center for advice on preparing NGS libraries and implementing high throughput DNA sequencing. This work was supported by the startup funding from Yale School of Medicine, a Scientist Development Grant from American Heart Association (12SDG11630031) and a Seed Grant from Connecticut Innovations, Inc. (13-SCA-YALE-15).
Materialien
| Name | Company | Catalog Number | Comments |
| ViraPower Lentiviral Expression Systems | Life Technologies | K4950-00, K4960-00, K4970-00, K4975-00, K4980-00, K4985-00, K4990-00, K367-20, K370-20, and K371-20 | |
| Gateway BP Clonase II Enzyme Mix | Life Technologies | 11789-020 | |
| Gateway LR Clonase II Enzyme Mix | Life Technologies | 11791-020 | |
| DNeasy Blood & Tissue Kit (250) | QIAGEN | 69506 or 69504 | |
| Gateway pDONR 201 | Life Technologies | 11798-014 | |
| 293T cells | American Type Culture Collection | CRL-11268 | |
| Trypsin-EDTA (0.05%), phenol red | Life Technologies | 25300-054 | |
| DMEM, high glucose, pyruvate | Life Technologies | 11995-065 | |
| Fetal Bovine Serum | Sigma | F4135 | |
| Tris | brand not critical | ||
| EDTA | brand not critical | ||
| 200 Proof EtOH | brand not critical | ||
| Isopropanol | brand not critical | ||
| Sodium Acetate | brand not critical | ||
| DpnI | New England Biolabs | R0176 | supplied with buffer |
| DamID adaptors "AdRt" and "AdRb" | Integrated DNA Technologies | sequences available in ref. 24; no phosphorylation of the 5' or 3' end to prevent self-ligation. | |
| T4 DNA Ligase | Roche Life Science | 10481220001 | supplied with buffer |
| DpnII | New England Biolabs | R0543 | supplied with buffer |
| DamID PCR primer "AdR_PCR" | Integrated DNA Technologies | sequences available in ref. 24 | |
| Deoxynucleotide (dNTP) Solution Set | New England Biolabs | N0446 | 100 mM each of dATP, dCTP, dGTP and dTTP |
| Advantage 2 Polymerase Mix | Clontech | 639201 | supplied with buffer |
| 1Kb Plus DNA Ladder | Life Technologies | 10787018 | 1.0 µg/µl |
| QIAquick PCR Purification Kit | QIAGEN | 28104 or 28106 | |
| MinElute PCR Purification Kit | QIAGEN | 28004 or 28006 | for an elution volume of less than 30 µl |
| SPRI beads / Agencourt AMPure XP | Beckman Coulter | A63880 | apply extra mixing and more elution time if less than 40 µl elution buffer is used |
| Buffer EB | QIAGEN | 19086 | |
| NEBNext dsDNA Fragmentase | New England Biolabs | M0348 | supplied with buffer |
| T4 DNA Ligase Reaction Buffer | New England Biolabs | B0202 | |
| T4 DNA Polymerase | New England Biolabs | M0203 | |
| DNA Polymerase I, Large (Klenow) Fragment | New England Biolabs | M0210 | |
| T4 Polynuleotide Kinase | New England Biolabs | M0201 | |
| Klenow Fragment (3’ → 5’ exo-) | New England Biolabs | M0212 | supplied with buffer |
| sequencing adaptors | Integrated DNA Technologies | sequences available in ref. 28 | |
| Quick Ligation Kit | New England Biolabs | M2200 | used in 3.4.3; supplied with Quick Ligation Reaction Buffer and Quick T4 DNA Ligase |
| sequencing primer 1 and 2 | Integrated DNA Technologies | sequences available in ref. 28 | |
| KAPA HiFi PCR Kit | Kapa Biosystems | KK2101 or KK2102 | supplied with KAPA HiFi DNA Polymerase, 5x KAPA HiFi Fidelity Buffer and 10 mM dNTP mix |
| agarose | Sigma Aldrich | A4679 | |
| ethidium bromide | Sigma Aldrich | E1510-10ML | 10 mg/ml |
| QIAquick Gel Extraction Kit | QIAGEN | 28704 or 28706 | |
| iTaq Universal SYBR Green Supermix | Bio-Rad Laboratories | 1725121 or 1725122 | |
| Spectrophotometer | brand not critical | ||
| 0.45 μm PVDF Filter | brand not critical | ||
| 25 ml Seringe | brand not critical | ||
| 10 cm Tissue Culture Plates | brand not critical | ||
| 6-well Tissue Culture Plates | brand not critical | ||
| S1000 Thermal Cycler | Bio-Rad Laboratories | ||
| C1000 Touch Thermal Cycler | Bio-Rad Laboratories | for qPCR | |
| Vortex Mixer | brand not critical | ||
| Dry Block Heater or Thermomixer | brand not critical | ||
| Microcentrifuge | brand not critical | ||
| Gel electrophoresis system with power supply | brand not critical | ||
| Magnet stand | for purification of DNA with SPRI beads; should hold 1.5-2 ml tubes; brand not critical | ||
| UV transilluminator | brand not critical | ||
| E-gel electrophoresis system | Life Technologies | G6400, G6500, G6512ST |
Referenzen
- van Steensel, B., Delrow, J., Henikoff, S. Chromatin profiling using targeted DNA adenine methyltransferase. Nat Genet. 27, 304-308 (2001).
- van Steensel, B., Henikoff, S. Identification of in vivo DNA targets of chromatin proteins using tethered dam methyltransferase. Nat Biotechnol. 18, 424-428 (2000).
- Fu, A. Q., Adryan, B. Scoring overlapping and adjacent signals from genome-wide ChIP and DamID assays. Mol Biosyst. 5, 1429-1438 (2009).
- Guelen, L. Domain organization of human chromosomes revealed by mapping of nuclear lamina interactions. Nature. 453, 948-951 (2008).
- Kalverda, B., Pickersgill, H., Shloma, V. V., Fornerod, M. Nucleoporins directly stimulate expression of developmental and cell-cycle genes inside the nucleoplasm. Cell. 140, 360-371 (2010).
- Kubben, N. Mapping of lamin A- and progerin-interacting genome regions. Chromosoma. 121, 447-464 (2012).
- Steglich, B., Filion, G. J., van Steensel, B., Ekwall, K. The inner nuclear membrane proteins Man1 and Ima1 link to two different types of chromatin at the nuclear periphery in S. pombe. Nucleus. 3, 77-87 (2012).
- Harr, J. C. Directed targeting of chromatin to the nuclear lamina is mediated by chromatin state and A-type lamins. J Cell Biol. 208, 33-52 (2015).
- Gonzalez-Aguilera, C. Genome-wide analysis links emerin to neuromuscular junction activity in Caenorhabditis elegans. Genome Biol. 15, R21(2014).
- Wu, F., Yao, J. Spatial compartmentalization at the nuclear periphery characterized by genome-wide mapping. BMC Genomics. 14, 591(2013).
- Filion, G. J. Systematic protein location mapping reveals five principal chromatin types in Drosophila cells. Cell. 143, 212-224 (2010).
- Vogel, M. J. Human heterochromatin proteins form large domains containing KRAB-ZNF genes. Genome Res. 16, 1493-1504 (2006).
- Venkatasubrahmanyam, S., Hwang, W. W., Meneghini, M. D., Tong, A. H., Madhani, H. D. Genome-wide, as opposed to local, antisilencing is mediated redundantly by the euchromatic factors Set1 and H2A.Z. Proc Natl Acad Sci U S A. 104, 16609-16614 (2007).
- Shimbo, T. MBD3 localizes at promoters, gene bodies and enhancers of active genes. PLoS Genet. 9, e1004028(2013).
- Orian, A. Genomic binding by the Drosophila Myc, Max, Mad/Mnt transcription factor network. Genes Dev. 17, 1101-1114 (2003).
- Artegiani, B. Tox: a multifunctional transcription factor and novel regulator of mammalian corticogenesis. EMBO J. , (2014).
- Schuster, E. DamID in C. elegans reveals longevity-associated targets of DAF-16/FoxO. Mol Syst Biol. 6, 399(2010).
- Bianchi-Frias, D. Hairy transcriptional repression targets and cofactor recruitment in Drosophila. PLoS Biol. 2, e178(2004).
- Woolcock, K. J., Gaidatzis, D., Punga, T., Buhler, M. Dicer associates with chromatin to repress genome activity in Schizosaccharomyces pombe. Nat Struct Mol Biol. 18, 94-99 (2011).
- Luo, S. D., Shi, G. W., Baker, B. S. Direct targets of the D. melanogaster DSXF protein and the evolution of sexual development. Development. 138, 2761-2771 (2011).
- Germann, S., Gaudin, V. Mapping in vivo protein-DNA interactions in plants by DamID, a DNA adenine methylation-based method. Methods Mol Biol. 754, 307-321 (2011).
- Zhang, X. The Arabidopsis LHP1 protein colocalizes with histone H3 Lys27 trimethylation. Nat Struct Mol Biol. 14, 869-871 (2007).
- Orian, A. Chromatin profiling, DamID and the emerging landscape of gene expression. Curr Opin Genet Dev. 16, 157-164 (2006).
- Vogel, M. J., Peric-Hupkes, D., van Steensel, B. Detection of in vivo protein-DNA interactions using DamID in mammalian cells. Nat Protoc. 2, 1467-1478 (2007).
- Greil, F., Moorman, C., van Steensel, B. DamID: mapping of in vivo protein-genome interactions using tethered DNA adenine methyltransferase. Methods Enzymol. 410, 342-359 (2006).
- de Wit, E., Greil, F., van Steensel, B. Genome-wide HP1 binding in Drosophila: developmental plasticity and genomic targeting signals. Genome Res. 15, 1265-1273 (2005).
- DamID mammalian vectors. , Available from: http://research.nki.nl/vansteensellab/Mammalian_plasmids.htm (2015).
- Illumina TruSeq adaptors & PCR primers. , Available from: https://ethanomics.wordpress.com/chip-seq-library-construction-using-the-illumina-truseq-adapters/ (2015).
- Optimization of PCR cycles for NGS. , Available from: https://ethanomics.wordpress.com/ngs-pcr-cycle-quantitation-protocol/ (2015).
- Bernstein, B. E. The NIH Roadmap Epigenomics Mapping Consortium. Nat Biotechnol. 28, 1045-1048 (2010).
- Encode Project Consortium. A user's guide to the encyclopedia of DNA elements (ENCODE). PLoS Biol. 9, e1001046(2011).
- Asp, P. Genome-wide remodeling of the epigenetic landscape during myogenic differentiation. Proc Natl Acad Sci U S A. 108, E149-E158 (2011).
- Hoppe, P. S., Coutu, D. L., Schroeder, T. Single-cell technologies sharpen up mammalian stem cell research. Nat Cell Biol. 16, 919-927 (2014).
- Avital, G., Hashimshony, T., Yanai, I. Seeing is believing: new methods for in situ single-cell transcriptomics. Genome Biol. 15, 110(2014).
- Navin, N. E. Cancer genomics: one cell at a time. Genome Biol. 15, 452(2014).
- Saliba, A. E., Westermann, A. J., Gorski, S. A., Vogel, J. Single-cell RNA-seq: advances and future challenges. Nucleic Acids Res. 42, 8845-8860 (2014).
- Nagano, T. Single-cell Hi-C reveals cell-to-cell variability in chromosome structure. Nature. 502, 59-64 (2013).
- Kind, J. Single-cell dynamics of genome-nuclear lamina interactions. Cell. 153, 178-192 (2013).
- Southall, T. D. Cell-type-specific profiling of gene expression and chromatin binding without cell isolation: assaying RNA Pol II occupancy in neural stem cells. Dev Cell. 26, 101-112 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten