Method Article
DamID-seq: מיפוי הגנום של אינטראקציות החלבון-DNA על ידי תפוקה גבוהה רצף של קטעי DNA-מפוגל אדנין
In This Article
Summary
אנו מתארים זאת assay על ידי צימוד זיהוי methyltransferase אדנין DNA (DamID) לרצף תפוקה גבוה (DamID-seq). שיטה משופרת זה מספקת רזולוציה גבוהה יותר וטווח דינמי רחב יותר, ומאפשרת ניתוח נתונים DamID-seq בשיתוף עם נתונים רצף אחרים תפוקה גבוהה כגון שבב seq, RNA-seq, וכו '
Abstract
The DNA adenine methyltransferase identification (DamID) assay is a powerful method to detect protein-DNA interactions both locally and genome-wide. It is an alternative approach to chromatin immunoprecipitation (ChIP). An expressed fusion protein consisting of the protein of interest and the E. coli DNA adenine methyltransferase can methylate the adenine base in GATC motifs near the sites of protein-DNA interactions. Adenine-methylated DNA fragments can then be specifically amplified and detected. The original DamID assay detects the genomic locations of methylated DNA fragments by hybridization to DNA microarrays, which is limited by the availability of microarrays and the density of predetermined probes. In this paper, we report the detailed protocol of integrating high throughput DNA sequencing into DamID (DamID-seq). The large number of short reads generated from DamID-seq enables detecting and localizing protein-DNA interactions genome-wide with high precision and sensitivity. We have used the DamID-seq assay to study genome-nuclear lamina (NL) interactions in mammalian cells, and have noticed that DamID-seq provides a high resolution and a wide dynamic range in detecting genome-NL interactions. The DamID-seq approach enables probing NL associations within gene structures and allows comparing genome-NL interaction maps with other functional genomic data, such as ChIP-seq and RNA-seq.
Introduction
זיהוי DNA methyltransferase אדנין (DamID) 1,2 הוא שיטה לגילוי אינטראקציות החלבון-דנ"א in vivo והוא גישה חלופית לimmunoprecipitation הכרומטין (שבב) 3. היא משתמשת בכמות נמוכה יחסית של תאים ואינו דורשת קישור צולב כימי של חלבון עם DNA או נוגדן ספציפי מאוד. האחרון הוא מועיל במיוחד כאשר חלבון המטרה הוא רופף או בעקיפין קשור ל- DNA. DamID שמש בהצלחה כדי למפות את אתרי הקישור של מגוון רחב של חלבונים כוללים חלבוני מעטפת גרעין 4-10, חלבוני הכרומטין קשור 11-13, אנזימי הכרומטין שינוי 14, גורמי שעתוק ושיתוף גורמים 15-18 ומנגנוני RNAi 19. השיטה היא ישימה באורגניזמים רבים, כולל ס 13 cerevisiae, ס 7 pombe, ג 9,17 elegans, ד ' melanogaster 5,11,18,20, א thaliana 21,22 כמו גם קווי עכבר ותא אנושי 6,8,10,23,24.
הפיתוח של assay DamID היה מבוסס על זיהוי הספציפי של שברי ה- DNA מפוגל אדנין בתאים האיקריוטים שחסרים אדנין אנדוגני מתילציה 2. חלבון היתוך מתבטא, בהיקף של החלבון קושר DNA של עניין וא methyltransferase coli DNA אדנין (Dam), יכול methylate בסיס אדנין ברצפי GATC שנמצאים בקרבת מרחבי (באופן משמעותי ביותר בתוך 1 קילו ועד בערך 5 קילו) לאתרי הקישור של החלבון בגנום 2. קטעי דנ"א שונה יכולים להיות מוגבר במיוחד והכלאה לmicroarrays כדי לזהות את אתרי הקישור הגנומי של החלבון של עניין 1,25,26. שיטת DamID מקורית זה הייתה מוגבל על ידי הזמינות של microarrays והצפיפות של בדיקות שנקבעו מראש. לכן יש לנו משולב רצף תפוקה גבוהה בלDamID 10 ומיועד השיטה כDamID-seq. המספר הגדול של כניסות קצרות שנוצר מDamID-seq מאפשר לוקליזציה מדויקת של אינטראקציות החלבון-דנ"א הגנום. מצאנו שDamID-seq סיפק ברזולוציה גבוהה יותר וטווח דינמי רחב יותר מאשר DamID ידי microarray ללימוד lamina הגנום גרעיני עמותות (NL) 10. שיטה משופרת זו מאפשרת חיטוט עמותות NL בתוך מבני גן 10 ומקלה על השוואות עם נתונים אחרים תפוקה גבוהה רצף, כגון שבב seq ו- RNA-seq.
פרוטוקול DamID-seq המתואר כאן פותח בתחילה לעמותות הגנום-NL מיפוי 10. אנחנו שנוצרנו חלבון היתוך על ידי קשירת עכבר או B1 Lamin אדם לE. methyltransferase אדנין coli DNA ובדק את הפרוטוקול ב3T3 fibroblasts העוברי של העכבר, עכבר C2C12 myoblasts 10 וIMR90 fibroblasts ריאות העובר אנושי (נתונים לא פורסמו). בפרוטוקול זה, אנחנו מתחילים עם גonstructing וקטורים ולהביע חלבוני היתוך דאם-קשורים בזיהום lentiviral בתאי יונקים 24. לאחר מכן, אנו מתארים את הפרוטוקולים מפורטים של הגברת קטעי DNA-מפוגל אדנין והכנת ספריות רצף שצריכה להיות מיושמים באורגניזמים אחרים.
Protocol
1. דור וביטוי של חלבונים היתוך וחלבונים סכר חינם
- חלבון שיבוט של עניין לתוך וקטור DamID.
- להגביר cDNA של חלבון של העניין (POI) באמצעות DNA פולימרז איכות גבוהה הרצוי ופריימרים מתאימים על פי הפרוטוקול של היצרן. בניסוי לקבוע תנאי הגברה אופטימליים כדי להבטיח הגברה נכונה של מוסיף.
- הפעל agarose ג'ל ולטהר cDNA המוגבר של נקודות עניין על ידי ערכת חילוץ ג'ל על פי הפרוטוקול של היצרן.
- השיבוט cDNA של נקודות עניין לתוך וקטור pDONR201 באמצעות BP Clonase השני על פי הפרוטוקול של היצרן.
- לשכפל cDNA של נקודות עניין מוקטור התורם לוקטור pLGW-RFC1-V5-EcoDam יעד או וקטור יעד pLGW-EcoDam-V5-RFC1 27 באמצעות LR Clonase השני על פי הפרוטוקול של היצרן, בהתאם לכיוון הרצוי של פיוזינג POI לN-סופית או ההדואר C-סופית של א ' methyltransferase coli DNA אדנין (EcoDam) 27.
- ודא ידי רצף שיש cDNA המשובט רצף נכון וצורות במסגרת היתוך EcoDam.
- צור מניות lentiviral.
- צור מניות lentiviral להביע דאם-V5-POI וV5-דאם (מוקטור pLGW-V5-EcoDam 27) שימוש במערכות ביטוי lentiviral. השתמש בהליך transfection קדימה על פי הפרוטוקול של היצרן.
- להשתמש בתאי 293T וlipofection ליצור מניות lentiviral פי הפרוטוקול של היצרן עבור transfection.
- כלול את צעד מסנן 0.45 מיקרומטר PVDF.
- צור מניות lentiviral להביע דאם-V5-POI וV5-דאם (מוקטור pLGW-V5-EcoDam 27) שימוש במערכות ביטוי lentiviral. השתמש בהליך transfection קדימה על פי הפרוטוקול של היצרן.
- להדביק תאים עם lentivirus.
- היום לפני זיהום (יום 0), עובר תאים חסיד תרבותיים בתקשורת צמיחה המתאימה לסוג תא זה לצלחת תרבית רקמה 6-גם שימוש באותה תקשורת צמיחה ללא אנטיביוטיקה ללהשיג מפגש 50% ביום של זיהום. תאי מקום בחממה 37 ° C.
- ביום של זיהום (יום 1), להסיר 2 cryovials של supernatants lentiviral שני דאם-V5-POI וV5-דאם ממקפיא -80 מעלות צלזיוס ומקום במים 37 מעלות צלזיוס אמבטיה להפשיר.
- הסר תקשורת צמיחת תאים ולהחליף עם 0.5 מיליליטר של תקשורת צמיחה טרי ללא אנטיביוטיקה.
- הוסף 1 מיליליטר של lentivirus המופשר היטב כל אחד (2 בארות עם V5-דאם ו -2 בארות עם הסכר-V5-POI). הוסף 1 מיליליטר של תקשורת צמיחה ללא אנטיביוטיקה לנותרו 2 בארות-זה ישמש כביקורת שלילית. נער בעדינות את הצלחת 6-גם לערבב ולמקם בחזרה בחממה 37 ° C O / N.
- היום לאחר הדבקה (יום 2), להסיר השעיות נגיפיות מהתאים ולהחליף עם 2 מיליליטר תקשורת צמיחה ללא אנטיביוטיקה. תאי מקום בחזרה בחממה 37 ° C למשך 48 שעות.
- לבודד gDNA.
- תקשורת לשאוב מכל הטוב ודהתאי Tach באמצעות 250 μl 0.05% טריפסין- EDTA. לדגור על 37 מעלות צלזיוס למשך 2 דקות.
- לשטוף את התאים מהצלחת עם 1 מיליליטר תקשורת צמיחה ותאי פיפטה מכל טוב לתוך צינור microcentrifuge 1.5 מיליליטר. גלולה תאים על ידי צנטריפוגה XG 200 במשך 5 דקות על RT.
- לשטוף את תאי pelleted עם 500 μl PBS ו צנטריפוגות ב XG 200 למשך 2 דקות על RT.
- Resuspend תאי pelleted ב200 μl PBS.
- לבודד gDNA ידי ערכת דם ורקמות על פי הפרוטוקול של היצרן. Elute gDNA ב200 חיץ μl AE ולקבוע את הריכוז על ידי מדידת OD 260 באמצעות ספקטרופוטומטר.
הערה: gDNA מהתאים נגועים נגוע או מדומים יכולה להיות מבודדת כפקדים שליליים. לזרז gDNA לריכוזים גבוהים יותר עבור אחסון לטווח ארוך.- הוסף 3 כרכים של 100% אתנול ו 0.1 נפח של 3 M נתרן אצטט (pH 5.5), ומערבבים על ידי היפוך צינורות 4-6 פעמים.
- חנות ב -20 ° CO / N.
- צנטריפוגה ב 16,000 xגרם במשך 15 דקות ב 4 מעלות צלזיוס.
- מוציא בזהירות את supernatant. לשטוף כדורים עם 70% אתנול (נפח / נפח) וצנטריפוגות ב 16,000 XG במשך 5 דקות על 4 מעלות צלזיוס.
- מוציאים בזהירות אתנול ולאפשר כדורים לייבוש באוויר במשך 3 דקות ב RT.
- ממיסים gDNA בT 10 E 0.1 (pH 7.5) בכ 1 מיקרוגרם / μl. הבריכה gDNA מכל מדגם ניסיוני או הביקורת השלילית ולמדוד את הריכוז. חנות ב -20 מעלות צלזיוס.
שברי DNA מפוגלים-אדנין 2. להגביר
- התקציר gDNA עם DpnI שרק חותך GATCs-מפוגל אדנין.
- הגדר את תגובה על קרח עם 2.5 מיקרוגרם gDNA, 1 μl 10x חיץ, 0.5 (U / μl 20) DpnI μl ולמלא עם H 2 O להיקף כולל של 10 μl. עבור כל דגימת gDNA, להכין שלוש תגובות - אחד ללא DpnI ("אין DpnI", להחליף DpnI עם 0.5 μl H 2 O) ושני wה- i DpnI ("עם DpnI").
- תקציר O / N ב 37 מעלות צלזיוס ולהשבית DpnI על 80 מעלות צלזיוס למשך 20 דקות.
- ולקשור מתאמי DamID.
- הכן מתאמי DamID.
- Resuspend כל אחד משני oligos מתאם DamID 24 בH 2 O לריכוז סופי של 100 מיקרומטר.
- לשלב כמויות שווה של שני oligos מתאם DamID, לערבב ולהניח בצינור סגור היטב. לאטום הצינור עם Parafilm, לשבת במעמד ומקום בכוס מלאה במים על 90 מעלות צלזיוס. שמרו את מפלס המים מתחת לכובע של הצינור (כדי למנוע מים נכנסים לצינור) אבל מעל פני השטח של תערובת אוליגו.
- בואו מגניב מים לRT כך לחשל מתאמים לאט.
- Aliquot מתאמי מורפה (50 מיקרומטר) ולאחסן ב -20 ° C.
- הגדר את תגובה על קרח. בצינור אחד מ2.1.2, להוסיף 6.2 0.8 מודעת μl 50 מיקרומטר DamID H 2 O, 2 μl 10x חיץ קשירה, μlaptors (מופשר על קרח) וμl 1 האנזים T4 DNA (5 U / μl). הנפח הכולל הוא 20 μl. באחד משני "עם DpnI" צינורות, להחליף האנזים עם μl 1 H 2 O ("אין האנזים") שים לב שלכל מדגם gDNA שני פקדים שליליים -. "לא DpnI" ו- "אין האנזים".
- O לקשור / N על 16 מעלות צלזיוס ולהשבית האנזים ב 65 מעלות צלזיוס למשך 10 דקות.
- הכן מתאמי DamID.
- תקציר DNA עם DpnII להרוס ברים המכילים GATCs unmethylated.
- הגדר את תגובה על קרח. בצינור אחד מ2.2.3, להוסיף 24 μl H 2 O, 5 μl 10x DpnII חיץ וμl 1 DpnII (10 U / μl). הנפח הכולל הוא 50 μl.
- תקציר על 37 מעלות צלזיוס במשך 2-3 שעות ולהשבית DpnII על 65 מעלות צלזיוס למשך 20 דקות.
- להגביר ברי ה- DNA מפוגל אדנין.
- הגדר את תגובה על קרח עם 5 μl DpnII לעכל מ2.3.2, 5 μl 10x חיץ PCR, פריימר 24, 4 μl 10 מ"מ dNTP לערבב, 1 μl H 50x לערבב פולימראז ו22.5 μl 2 א '12.5 μl 5 מיקרומטר DamID PCR הנפח הכולל הוא 50 μl.
- הפעל את ה- PCR כדלקמן: 68 מעלות צלזיוס במשך 10 דקות; 94 מעלות צלזיוס במשך דקות 1, 65 מעלות צלזיוס למשך 5 דקות, 68 מעלות צלזיוס במשך 15 דקות; 4 מחזורים של 94 מעלות צלזיוס במשך דקות 1, 65 מעלות צלזיוס במשך דקות 1, 68 מעלות צלזיוס במשך 10 דקות; 17 מחזורים של 94 מעלות צלזיוס במשך דקות 1, 65 מעלות צלזיוס במשך דקות 1, 68 מעלות צלזיוס למשך 2 דקות.
- לנתח 5 מוצרי μl PCR מכל תגובה על 1% agarose ג'ל. מוצרי ה- PCR אמורים להופיע ככתם החל 0.2-2 קילו (איור 2). בקרות "אין DpnI" ו- "אין האנזים" צריכה שום או באופן ברור פחות הגברה.
- אם התוצאה מהצעד 2.4.3 היא משביעת רצון, חזור על שלבי 2.4.1-2.4.3 עם שתיים או שלוש תגובות למדגם הניסיוני ותגובה אחת לכל אחד משני הפקדים השליליים.
- בריכה ולטהר מוצרי PCR מאותומדגם ניסיוני באמצעות ערכות טיהור PCR או חרוזים שלב מוצק הפיך קיבוע (SPRI) על פי הפרוטוקול של היצרן. אל לטהר "אין DpnI" או בקרות "אין האנזים". Elute DNA עם חיץ EB.
- למדוד את הריכוז של ה- DNA המטוהר על ידי מדידת OD 260 באמצעות ספקטרופוטומטר, שאמורה להיות סביב 0.1 מיקרוגרם / μl או גבוה יותר. לאסוף מינימום של 10 מיקרוגרם DNA עבור כל דגימה. אם משתמש בערכות טיהור PCR, לטהר כל מוצרי ה- PCR 50 μl עם עמודה אחת, elute ב 30 חיץ μl EB וברכת eluates.
- תקציר DNA עם DpnII למנוע פריימרים DamID PCR מלהיות רצף.
- הגדר את תגובה על קרח עם DNA המטוהר 5 מיקרוגרם מ2.4.6, 5 μl 10x חיץ DpnII, (10 U / μl) μl 1 DpnII ולמלא עם H 2 O להיקף כולל של 50 μl. הכן שתיים או שלוש תגובות לכל דגימה.
- לעכל על 37 מעלות צלזיוס במשך 2-3 שעותולהשבית DpnII על 65 מעלות צלזיוס למשך 20 דקות.
- בריכה ולטהר את מעכלת מהמדגם זהה עם ערכות טיהור PCR או חרוזים SPRI פי הפרוטוקול של היצרן. Elute DNA עם חיץ EB.
- למדוד את הריכוז של ה- DNA המטוהר שצריכה להיות סביב 0.06 מיקרוגרם / μl או גבוה יותר. לאסוף מינימום של DNA מיקרוגרם 6 עבור כל דגימה. אם משתמש בערכות טיהור PCR, לטהר כל 50 μl לעכל עם עמודה אחת, elute ב 30 חיץ μl EB וברכת eluates.
3. הספרייה הכנה לרצף תפוקה גבוהה
- הקטע DNA
- בניסוי לקבוע את זמן העיכול המתאים לכל קבוצה חדשה של dsDNA Fragmentase. כפעילות האנזים עלולה להפחית לאורך זמן, לחזור על הבדיקה לפני ביצוע ניסוי חדש. כדי לשמור DNA מ2.5.4 לפיצול בפועל, להשתמש במוצרי PCR התיל מטוהרים מ2.4.6 או DNA נוסף מexperime הקודםNTS בשלב זה.
- הגדר את תערובת הורים עם DNA 6 מיקרוגרם, 12 μl 10x חיץ Fragmentase ולמלא עם H 2 O להיקף כולל של 114 μl.
- מערבולת בקבוקון מניית Fragmentase במשך 3 שניות, להוסיף 6 μl לתערובת ההורים ומערבולת תערובת האמן במשך 3 שניות. הנפח הכולל הוא 120 μl.
- Aliquot 20 μl של מיקס מאסטר לכל אחד 5 צינורות חדשים. דגירה כל 6 הצינורות ב C ° 37 במשך 5 עד 55 דקות בתוספת של 10 דקות. הוסף 5 μl 0.5 M EDTA כדי לעצור את התגובה.
- לנתח 12.5 μl לעכל (0.5 מיקרוגרם DNA) של כל תגובה, כמו גם 0.5 מיקרוגרם DNA מעוכל על agarose ג'ל (איור 3). קבע את הזמן המינימלי (0.2kb T) צריך לעכל את רוב למרוח סביב 0.2 קילו. בחר 6 משכי זמן בין 5 דקות ו0.2kb T (כולל 5 דקות ו0.2kb T) עם מרווחים שווים לפיצול בפועל.
- הגדרת fragmen בפועלtation כפי שתואר ב3.1.1.1-3.1.1.3. דגירה תגובות על 37 מעלות צלזיוס למשך משכי הזמן שנקבעו ב3.1.1.4.
- בריכה 6 תגובות ולטהר את מעכל עם ערכות טיהור PCR או חרוזים SPRI פי הפרוטוקול של היצרן. Elute DNA במאגר 51 μl EB. Eluate הסופי הוא ~ 50 μl.
- בניסוי לקבוע את זמן העיכול המתאים לכל קבוצה חדשה של dsDNA Fragmentase. כפעילות האנזים עלולה להפחית לאורך זמן, לחזור על הבדיקה לפני ביצוע ניסוי חדש. כדי לשמור DNA מ2.5.4 לפיצול בפועל, להשתמש במוצרי PCR התיל מטוהרים מ2.4.6 או DNA נוסף מexperime הקודםNTS בשלב זה.
- תיקון מסתיים של DNA המקוטע
- הגדר את תגובה על קרח עם eluate מ3.1.3, 25 H μl 2 O, 10 μl 10x חיץ האנזים T4 DNA עם 10 מ"מ ATP, 4 μl 10 מ"מ שילוב dNTP, DNA פולימראז T4 5 μl (3 U / μl) , Μl 1 פולימראז Klenow DNA (5 U / μl), וקינאז polynucleotide T4 5 μl. הנפח הכולל הוא 100 μl. מערבבים היטב עם פיפטה. הימנע קצף ובועות.
- לדגור על 20 מעלות צלזיוס למשך 30 דקות.
- לטהר את ה- DNA עם ערכות טיהור PCR או חרוזים SPRI פי הפרוטוקול של היצרן. Elute DNA במאגר 33 μl EB. Eluate הסופי הוא ~ 32 & #181; l.
- להוסיף הסככות ""
- הגדירו את תגובה על קרח עם eluate מ3.2.3, 5 μl 10x חיץ Klenow, 10 μl 1 מ"מ dNTP, ו -3 μl Klenow (3 '→ 5' exo-) (5 U / μl). הנפח הכולל הוא 50 μl. מערבבים היטב עם פיפטה. הימנע קצף ובועות.
- לדגור על 37 מעלות צלזיוס למשך 30 דקות.
- לטהר את ה- DNA עם ערכות טיהור PCR או חרוזים SPRI פי הפרוטוקול של היצרן. Elute DNA במאגר 22 μl EB. Eluate הסופי הוא ~ 21 μl.
- מתאמי רצף לקשור
- הכן מתאמי רצף 28.
- Resuspend כל אוליגו מתאם לריכוז של 100 מיקרומטר ב 10 מ"מ טריס-Cl (pH 7.8), 0.1 מ"מ EDTA (pH 8.0) ו -50 מ"מ NaCl.
- לערבב כמויות שווה של המתאם האוניברסלי 28 ומתאם אינדקס 28.
- לחשל המתאמים בCycler תרמית עם הבאתכנית: 95 מעלות צלזיוס למשך 2 דקות; 140 מחזורים למשך 30 שניות החל מ 95 מעלות צלזיוס, ולהקטין בכ -0.5 מעלות צלזיוס כל מחזור; להחזיק ב 4 מעלות צלזיוס.
- מתאמי Aliquot ולאחסן ב -20 ° C.
- הגדר את תגובה על קרח עם eluate מ3.3.3, 25 μl 2x חיץ קשירה, 1.5 μl 50 מיקרומטר מרותק מתאמי רצף (מופשרים על קרח) מאנזים דנ"א T4 3.4.1.4 ו -2.5 μl. מערבבים היטב עם פיפטה. הימנע קצף ובועות. אם רצף זמנית הוא רצוי, להשתמש במתאם אינדקס שונה עבור כל דגימה.
- לדגור על RT עבור שעה 1.
- לטהר את ה- DNA עם ערכות טיהור PCR או חרוזים SPRI פי הפרוטוקול של היצרן. Elute DNA במאגר 24 μl EB. Eluate הסופי הוא ~ 23 μl.
- הכן מתאמי רצף 28.
- המרת מתאמים בצורת אות V לdsDNA כדי לאפשר קביעה מדויקת של קטע DNA בגדלי 28
- הגדר את תגובה על קרח עם eluate מ3.4.4, H 2 O μl 12.5, 1 μl 25 מיקרומטר פריימר 1 28, 1 μl 25 מיקרומטר פריימר 2 28, 1.5 μl 10mm dNTP, 10 μl 5x PCR הצפת ופולימראז 1 μl DNA (1 U / μl).
- הפעל את ה- PCR כדלקמן: 95 מעלות צלזיוס במשך 3 דקות; 5 מחזורים של 98 מעלות צלזיוס למשך 15 שניות, 63 מעלות צלזיוס למשך 30 שניות, 72 מעלות צלזיוס במשך 30 שניות; 72 מעלות צלזיוס במשך 1 דקות; 4 מעלות צלזיוס בהמתנה.
- לטהר את ה- DNA עם ערכות טיהור PCR או חרוזים SPRI פי הפרוטוקול של היצרן. Elute DNA במאגר 11 μl EB. Eluate הסופי הוא ~ 10 μl.
- גודל בחר הספרייה
- הכן agarose ג'ל 2% עם חיץ 1x טה. להוסיף ethidium ברומיד (זהירות!) לריכוז סופי של 500 ng / ml כאשר פתרון טה-agarose נמס התקרר להימנע משאיפת של ברומיד. ודא שיש מספיק נתיבים לכל הדגימות, סולם DNA ונתיבים ריקים.
- להוסיף צבע טעינת 6x 8 μl לeluate מ3.5.3.
- הכן סולם ה- DNA על ידי ערבוב 1 קילו בתוספת סולם ה- DNA (1.0 מיקרוגרם / μl), צבע טעינת 6x וH 2 O ביחס של 1: 1: 4.
- טען 6 μl סולם ה- DNA, דגימות מ3.6.2 ועוד סולם DNA 6 μl, כל אחד במסלול נפרד ועם השביל ריק לפחות אחד מדגימות / סולמות הסמוכים.
- הפעל את הג'ל על 120 V עבור 60 דקות.
- צפה ג'ל על transilluminator UV (לצמצם את זמן החשיפה לקרינת UV). יש להרכיב משקפיים בטיחות ומגן פנים. ודא לפחות אחד מסולמות DNA לרוץ גם עם מרווח מתאים (מספיק למוציא 3 פרוסות ג'ל) בין 300 נ"ב ו -400 נ"ב להקות. מרווח צר מגביר את הקשיים במוציאים פרוסות ג'ל מרובות, בעוד מרווח רחב מגדיל את נפח פרוסות ג'ל נכרת.
- השתמש באזמל חדש או סכין גילוח לכל מסלול. בלו שלוש פרוסות ג'ל דקות שבין 300 ל -400 נ"ב מנתיב אחד (איור 4) ולמקם אותם כל אחד בצינור microcentrifuge. שמור את עוצמת הקול של כל של ג'לכינים נמוכות כמו (<100 μl) האפשרי.
- מדוד את עוצמת הקול של כל פרוסה ג'ל (ג'ל μl 1 שוקל כ 1 מ"ג), להוסיף כרכי 6x של חיץ QG ולדגור על 50 מעלות צלזיוס.
- מערבולת תערובת QG-ג'ל כל דקות 2-3 עד פרוסת ג'ל נמסה לגמרי. להוסיף כרכי ג'ל 2x של isopropanol ומערבבים.
- מהצעד הזה, לעקוב אחר הפרוטוקול מערכות חילוץ ג'ל. Elute DNA במאגר 51 μl EB. Eluate הסופי הוא ~ 50 μl.
- העשר את שברי DNA מתאם שונה רצף
- לייעל את מספר מחזורי PCR 29.
- הגדר את תגובה על קרח עם eluate 1 μl מ3.6.10, 1 μl 25 מיקרומטר פריימר 1 28, 1 μl 25 מיקרומטר פריימר 2 28, 7 H μl 2 O ו -10 μl SYBR הירוק Supermix. הנפח הכולל הוא 20 μl.
- הפעל qPCR כדלקמן: 95 מעלות צלזיוס במשך 3 דקות; 20 מחזורים של 95 מעלות צלזיוס למשך 30 שניות, 63 מעלות צלזיוס למשך 30 שניות, 72 מעלות צלזיוס למשך 30 שניות, צלחת לקרוא.
- לנתח נתונים באמצעות תוכנת ניתוח qPCR ולקבוע את מחזור כימות (CQ) או מחזור סף (CT) עבור כל דגימה באמצעות הפרוטוקול של היצרן. תמשיך עם הדגימות שיש לי CQ / CT ≤ 14. שימוש המקסימאלי CQ (CQ 0) מינוס 1 (המעוגלים כלפי מעלה למספר השלם הבא גבוה יותר) כמספר מחזור PCR הסופי (N PCR).
- התאם את הכמויות של תבניות DNA, כך שמדגמים שונים יהיו מוגברים לסכומים שווים בערך אחרי שהפעלתי אותו מספר מחזורי PCR. השתמש בתבנית 8 μl למדגם עם CQ הגבוה ביותר (CQ 0), ולחשב את תבנית הנפח של דגימות אחרות עם הנוסחה הבאה:
אני כרך = 8 x 1.8 CQ אני -Cq 0
- הגדרת PCR תגובות על קרח עם כרכי התבנית מחושבים מ3.7.1.4: 1 μl 25 מיקרומטר 1 28, 1 μl 25 מיקרומטר פריימר פריימר 2 28, 1.510 מ"מ dNTP μl, 10 μl 5x חיץ, פולימראז 1 μl DNA (1 U / μl) ולמלא עם H 2 O להיקף כולל של 50 μl.
- הפעל את ה- PCR כדלקמן: 95 מעלות צלזיוס למשך 45 שניות; מחזורי N PCR (שנקבעו ב3.7.1.3) של 98 מעלות צלזיוס למשך 15 שניות, 63 מעלות צלזיוס למשך 30 שניות, 72 מעלות צלזיוס במשך 30 שניות; 72 מעלות צלזיוס במשך 1 דקות; 4 מעלות צלזיוס בהמתנה.
- לנתח 5 מוצרי μl PCR בagarose ג'ל 2% (איור 5 א). להקה "יחידה" ברורה עולה כי שברי דנ"א המוגבר נמצאים בטווח אורך צר ושספריית ה- DNA יכולה להיות כפופה לניתוח נוסף.
- חזור על תגובה אחת לדגימות שנבחרו וברכת ספריות ה- DNA המוגבר מהמדגם זהה.
- לטהר ספריות ה- DNA שנבחרו לריצוף.
- אם הדימרים פריימר / מתאם אינם גלויים ב3.7.4, לטהר ספריות DNA עם ערכות טיהור PCR או חרוזים SPRI פי הפרוטוקול של היצרן. Elute DNA במאגר 21 μl EB.eluate הסופי הוא ~ 20 μl. אם יש מתקן הרצף של המשתמש הוראות ספציפיות במאגר elution, הנפח הסופי, וכו ', להכין דגימות בהתאם.
- אם הדימרים פריימר / מתאם נראה בבירור ב3.7.4, לטהר ספריות כדלקמן.
- DNA עומס מ3.7.5 וסולם DNA ב2% agarose ג'ל טרומי. יכולות להיות מטוהרות דגימות כפי שתוארו ב3.7.6.1 כדי להפחית את עוצמת הקול או ניתן לטעון לתוך בארות מרובות. מניחים את הג'ל agarose במערכת חשמל מתאים. לאפשר ג'ל לרוץ במשך 15 דקות.
- באמצעות transilluminator מתאים, לגזור את הלהקה הרצויה עם אזמל / גילוח נקי עבור כל דגימה ולמקם את פרוסת ג'ל בצינור microcentrifuge 1.5 מיליליטר.
- לבודד DNA באמצעות ערכות חילוץ ג'ל על פי הפרוטוקול של היצרן עם שני שינויים: דגירה תערובת QG-ג'ל חיץ בתרם-מיקסר על 37 מעלות צלזיוס ו1,000 סל"ד במשך 30 דקות, ולהוסיף כרכי ג'ל 2x של isopropanol.
- לייעל את מספר מחזורי PCR 29.
- שלח ספריות ה- DNA מטוהרים למתקן רצף. בצע את כל הנחיות המתקן.
הערה: ניתוח איכות על ידי Bioanalyzer (איור 5) צריכה להתבצע לפני הרצף כדי לקבוע את טווח הגודל המדויק והריכוז של ספריית ה- DNA.
תוצאות
חלבון היתוך דאם-V5-LmnB1 אומת להיות שיתוף מקומי עם Lamin B חלבון אנדוגני על ידי צביעת immunofluorescence (איור 1).
ההגברה PCR המוצלחת של שברי ה- DNA מפוגל אדנין היא צעד מפתח לDamID-seq. דגימות הניסוי צריכים להגביר כתם של 0.2-2 קילו תוך הבקרה השלילית (ללא DpnI, ללא האנזים או בלי תבנית PCR) צריך לגרום לא-ברור או פחות הגברה (איור 2).
שברי DNA מפוגלים הם בטווח 0.2-2 קילו, בעוד שהגודל הכנס הרצוי לספריית NGS הוא 200-300 נקודות בסיס. לכן, זה הכרחי כדי לפצל את מוצרי ה- PCR מתיל לטווח הגודל המתאים. עם זאת, נמצא להיות מעשי לשבור קטעי דנ"א גדולים יותר בו-זמנית עד לגודל מתאיםים ולשמור על הרוב של שברי DNA קטנים שלמים במשך פיצול יחיד. לכן, ניסויים כמובן זמן בוצעו כדי לקבוע את הזמן המינימלי (0.2kb T) זקוק ל- DNA מיקרוגרם בר 1 למריחה מרוכזת ב 200 נ"ב (איור 3). אז 6 משכי זמן במרווחים שווים נבחרו בין 5 דקות ו0.2kb T לפיצול בפועל. הפעילות האנזימטית של גדיל הדנ"א הכפול Fragmentase עשויה להשתנות מתצווי אצווה ועלולה להפחית לאורך זמן, ולכן מומלץ לחזור על שלב זה עבור קבוצה חדשה של Fragmentase או לאחר האחסון לתקופה של זמן.
הגודל הכנס הרצוי הוא בין 200 ל -300 נ"ב מתאים לברי DNA בין 300 ל -400 נ"ב (כולל 121 מתאמי רצף נ"ב) על ג'ל agarose. שלוש פרוסות דקות בטווח זה היו נכרת מכל מדגם ניסיוני כדי לצמצם את טווח הגודל של ספרייה ולהגדיל את האפשרותקבלת ספרייה אחת לפחות מוסמכת רצף (איור 4).
Aliquot של 5 μl של כל ספריית DNA מוגברת נותח על ג'ל agarose כדי לקבוע אילו ספרייה עשוי להעפיל רצף. כפי שניתן לראות באיור 5 א ', להקה אחת ברורה באותו הגודל כמו פרוסת ג'ל נכרת צריכה להיות גלויה על ג'ל agarose (צעד 3.7.4). בשלב הבא, ספריות נבחרו נבדקו על ידי Bioanalyzer (איור 5) כדי לקבוע את טווח הגודל המדויק וריכוזים לפני הרצף. אם תרצה, ספריות ה- DNA מוגברות ניתן לבחון ישירות על ידי Bioanalyzer ללא ניתוח ג'ל. כאשר ספריות מרובות הן באיכות טובה, מומלץ רצף ספריות בגודל דומה נעות לזוג ניסיוני (תאים המבטאים דאם-V5-POI) ושליטה (תאים המבטא V5-דאם) דגימות.
הקצר שנוצר על ידי קוראמערכות רצף מופו ראשון חזרה לגנום המקביל. ייחודי מיושר קורא היו אז עברו לניתוחים שלאחר מכן. צינור לעבד קצר קורא, לבנות מפת אינטראקציה הגנום-NL ולנתח עמותות גן-NL תוארו בפירוט בעבודה הקודמת שלנו 10. נציגי תוצאות מוצגות באיור 6.

איור 1. אימות הגירסות מגוירות subnuclear של חלבוני היתוך על ידי צביעת immunofluorescence. IMR90 תאי פיברובלסטים ריאות האנושיות היו transfected זמני עם פלסמיד להביע דאם-V5-LmnB1 והוכתמו על ידי אנטי-Lamin B (, אדום) ואנטי-V5 (B, יָרוֹק). (ג) מיזוג של תמונות בA ו- B. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של tדמותו.

איור 2. PCR הגברה שברי ה- DNA מפוגל אדנין. Aliquot של 5 μl מכל תגובת PCR נותח על ג'ל agarose 1%. למרוח החל 0.2-2 קילו היה מוגבר מכל מדגם ניסיוני, אבל לא הגברה נצפתה בבקרה שלילית (לא DpnI בשלב 2.1, אין האנזים בשלב 2.2, או לא תבנית PCR). אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של נתון זה.
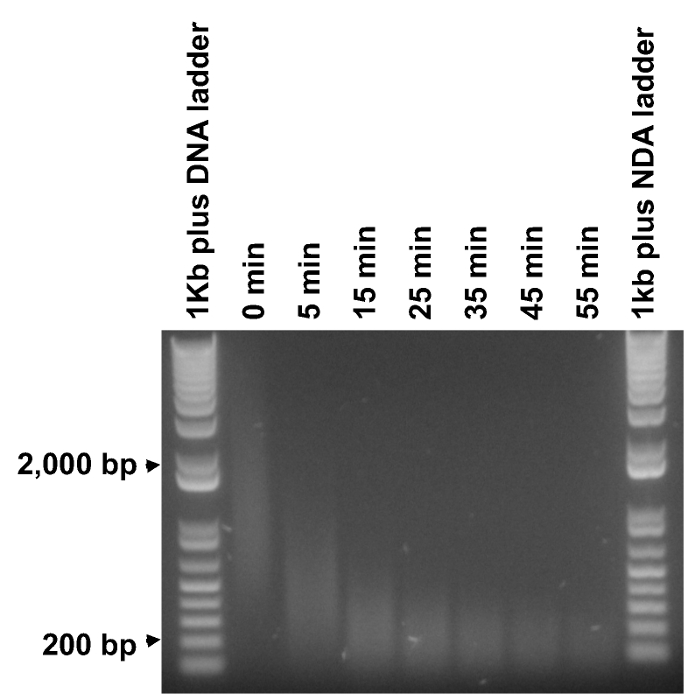
איור 3. קביעת משך פיצול אופטימלי. מוצרי ה- PCR התיל מזוקקים היו כפופים לפיצול לתקופות זמן 5-55 דקות בתוספת של 10 דקות (DNA מעוכל , שכותרתו "מינימום 0"). 1 מיקרוגרם של סולם ה- DNA ושל 0.5 מיקרוגרם של כל דגימת DNA מקוטעת נותחו על ג'ל agarose. הזמן המינימלי לעיכול רוב למרוח DNA לכ -200 נ"ב היה נחוש להיות סביב 35 דקות, לכן שישה משכי זמן מחולק באופן שווה בין 5 ל 35 דקות (5 ו -35 דקות כלולות) נבחרו לבצע את הפיצול בפועל. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.

גודל 4. איור בחירת ספריות ה- DNA. דגימות DNA של הסכר-V5-LmnB1 וV5-דאם מהצעד 3.6 נוהלו על 2% agarose ג'ל, ושלוש פרוסות ג'ל שווה בגודלן (L, M ו- H מתאים לנמוך, בינוני וגבוה בגודל) היו נכרת בין 300 ל -400 נ"ב כפי שמוצג על ידי הקווים הצהובים. אום קבצים / ftp_upload / 53,620 53620fig4large.jpg "target =" / / _ blank "> לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
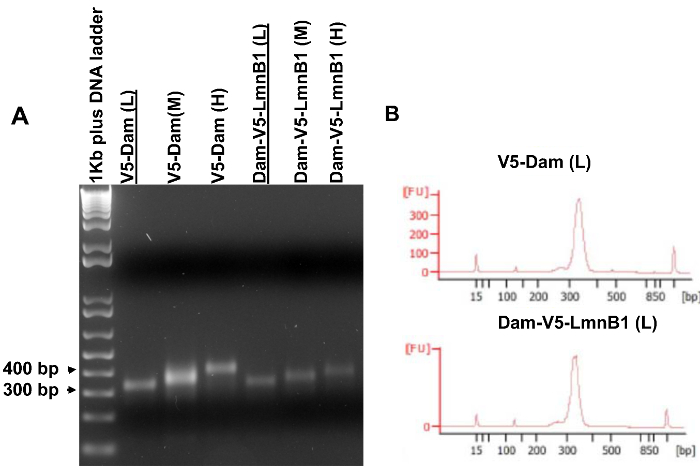
ספריות ה- DNA איור 5. ספריות Amplified DNA עבור סידור תפוקה גבוה. () מוגברת נותחו על ג'ל agarose. תבניות PCR היו מטוהרות מפרוסות ג'ל מוצגות באיור 4. (ב) תוצאות Bioanalyzer של מדגם V5-דאם (L) ומדגם דאם-V5-LmnB1 (L) שהדגישו ב(). היו שתי ספריות אלה טווחי גודל צר דומים ובכך העפיל לרצף תפוקה גבוה. לחצו כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
jpg "/>
6. נתונים NGS איור מוצגים בדפדפן הגנום UCSC. כרומוזום עכבר 1 מוצגים כדוגמא. נתוני רצף הופקו מmyoblasts C2C12 עכבר 10. מסלולים "MB.LmnB1.w2k" ו- "MB.Dam.w2k", מתאים לנתונים מהתאים לבטא דאם-V5-LmnB1 וV5-דאם בהתאמה, צפיפות קריאת עלילה מנורמלות (קוראים לkilobase למיליון ייחודיים ממופים קורא, או RPKM ) בחלונות 2 KB רצופים שאינם חופפים לאורך כרומוזום. עקוב אחר עמותות הגנום-NL חלקות "MB.log2FC.w2k", כלומר log2 RPKM יחסים של LmnB1 מעל הסכר, ברזולוצית 2 KB. עקוב אחר "MB.sLADs" מצייר (sLADs, כלומר אזורים הגנומי שיש צפיפות גבוהה יותר של קריאת LmnB1 מעל הסכר עם מובהקות סטטיסטיות) Lamina Associated תחומים מבוססי רצף שבאינו sLADs השחור, באזורים אפורים ולא נקבעו בלבן. אנא לחץ כאן ל Downloaהיית בקובץ זה.
Discussion
Whether Dam-tagged proteins retain the functions of endogenous proteins should be examined before a DamID-seq experiment. The subcellular localization of Dam-tagged nuclear envelope proteins should always be determined and compared with that of the endogenous proteins. For studying transcription factors, it is suggested to examine whether the Dam-fusion protein can rescue the functions of the endogenous protein in regulating gene expression. This functional test can be performed in organisms in which knockout mutants of endogenous DNA-binding proteins are available. Because advances in genome engineering have potentially allowed knocking out any endogenous gene of interest, functions of Dam-tagged DNA-binding proteins can be examined in cultured mammalian cells.
The critical step in this protocol is to successfully fragment the DpnII-digested DamID PCR products to around 200 bp. This step is designed to render the amplified adenine-methylated fragments to a narrow size range for sequencing and to randomize the starting nucleotides of the DNA fragments in a sequencing library. Inefficient fragmentation will leave the majority of the DNA fragments starting with GATC (the 5'-overhang from the second DpnII digestion), and will result in a much lower performance and yield or even a failure in Illumina sequencing. Other DNA fragmentation methods may be used as an alternative approach.
The resolution of DamID (and DamID-seq described here) is limited by the frequency of GATCs in the genome to be studied. Moreover, even with high throughput sequencing, the genomic localizations of a DNA-binding protein can only be mapped within two consecutive GATCs rather than to the actual DNA-binding sites.
Despite its limitation, the DamID assay has important advantages. Because DamID does not require highly-specific antibodies, it can be used to detect a subset of nuclear proteins that could be difficult to assay by ChIP (such as the nuclear envelope proteins). To study how these proteins regulate genome functions, it is important to integrate and cross-analyze their genome-wide localization data with the current epigenomic mapping data (such as data from the ENCODE and NIH Roadmap Epigenomics Projects 30,31). The DamID-seq approach provides both higher resolution and higher sensitivity than DamID by microarray and enables detecting differential NL-associations within gene structures 10. A combinatorial analysis of DamID-seq data, ChIP-seq data 32 and gene expression data has identified a class of NL-associated genes with distinct epigenetic and transcriptional features (data not published).
Another advantage of DamID is that it only requires a small number of cells. In recent years, there has been an explosion in single cell analysis of gene regulation 33,34. Although genome sequence 35, genome-wide gene expression 36 and chromatin conformation 37 can be assayed in a single cell, there has not been an available approach for detecting protein-DNA interactions genome-wide in a single cell. DamID-seq is a highly promising approach for this goal, and may complement the single cell imaging approach in detecting the dynamics of genome-NL interactions 38. One complication is that because the Dam-fusion protein is expressed at a much lower level than the endogenous protein in the DamID assay, it is possible that the Dam-fusion protein may only occupy a subset of genomic binding sites as compared to the endogenous protein.
DamID assay has mostly been used in cultured animal cells to detect protein-DNA interactions. Notably, developmental biologists have applied this assay in detecting protein-DNA interactions in specific cell types in vivo. For example, Dam-tagged RNA polymerase II was expressed specifically in Drosophila neural stem cells to detect their genome-wide occupancy without cell isolation 39. DamID-seq will be highly useful to study the genome-wide localizations of nuclear envelope proteins, transcription factors and chromatin regulators during development in animal models.
Disclosures
The authors have nothing to disclose.
Acknowledgements
We thank Dr. Bas van Steensel for providing the DamID mammalian expression vectors. We thank Yale Center for Genome Analysis and the Genomics Core in Yale Stem Cell Center for advice on preparing NGS libraries and implementing high throughput DNA sequencing. This work was supported by the startup funding from Yale School of Medicine, a Scientist Development Grant from American Heart Association (12SDG11630031) and a Seed Grant from Connecticut Innovations, Inc. (13-SCA-YALE-15).
Materials
| Name | Company | Catalog Number | Comments |
| ViraPower Lentiviral Expression Systems | Life Technologies | K4950-00, K4960-00, K4970-00, K4975-00, K4980-00, K4985-00, K4990-00, K367-20, K370-20, and K371-20 | |
| Gateway BP Clonase II Enzyme Mix | Life Technologies | 11789-020 | |
| Gateway LR Clonase II Enzyme Mix | Life Technologies | 11791-020 | |
| DNeasy Blood & Tissue Kit (250) | QIAGEN | 69506 or 69504 | |
| Gateway pDONR 201 | Life Technologies | 11798-014 | |
| 293T cells | American Type Culture Collection | CRL-11268 | |
| Trypsin-EDTA (0.05%), phenol red | Life Technologies | 25300-054 | |
| DMEM, high glucose, pyruvate | Life Technologies | 11995-065 | |
| Fetal Bovine Serum | Sigma | F4135 | |
| Tris | brand not critical | ||
| EDTA | brand not critical | ||
| 200 Proof EtOH | brand not critical | ||
| Isopropanol | brand not critical | ||
| Sodium Acetate | brand not critical | ||
| DpnI | New England Biolabs | R0176 | supplied with buffer |
| DamID adaptors "AdRt" and "AdRb" | Integrated DNA Technologies | sequences available in ref. 24; no phosphorylation of the 5' or 3' end to prevent self-ligation. | |
| T4 DNA Ligase | Roche Life Science | 10481220001 | supplied with buffer |
| DpnII | New England Biolabs | R0543 | supplied with buffer |
| DamID PCR primer "AdR_PCR" | Integrated DNA Technologies | sequences available in ref. 24 | |
| Deoxynucleotide (dNTP) Solution Set | New England Biolabs | N0446 | 100 mM each of dATP, dCTP, dGTP and dTTP |
| Advantage 2 Polymerase Mix | Clontech | 639201 | supplied with buffer |
| 1Kb Plus DNA Ladder | Life Technologies | 10787018 | 1.0 µg/µl |
| QIAquick PCR Purification Kit | QIAGEN | 28104 or 28106 | |
| MinElute PCR Purification Kit | QIAGEN | 28004 or 28006 | for an elution volume of less than 30 µl |
| SPRI beads / Agencourt AMPure XP | Beckman Coulter | A63880 | apply extra mixing and more elution time if less than 40 µl elution buffer is used |
| Buffer EB | QIAGEN | 19086 | |
| NEBNext dsDNA Fragmentase | New England Biolabs | M0348 | supplied with buffer |
| T4 DNA Ligase Reaction Buffer | New England Biolabs | B0202 | |
| T4 DNA Polymerase | New England Biolabs | M0203 | |
| DNA Polymerase I, Large (Klenow) Fragment | New England Biolabs | M0210 | |
| T4 Polynuleotide Kinase | New England Biolabs | M0201 | |
| Klenow Fragment (3’ → 5’ exo-) | New England Biolabs | M0212 | supplied with buffer |
| sequencing adaptors | Integrated DNA Technologies | sequences available in ref. 28 | |
| Quick Ligation Kit | New England Biolabs | M2200 | used in 3.4.3; supplied with Quick Ligation Reaction Buffer and Quick T4 DNA Ligase |
| sequencing primer 1 and 2 | Integrated DNA Technologies | sequences available in ref. 28 | |
| KAPA HiFi PCR Kit | Kapa Biosystems | KK2101 or KK2102 | supplied with KAPA HiFi DNA Polymerase, 5x KAPA HiFi Fidelity Buffer and 10 mM dNTP mix |
| agarose | Sigma Aldrich | A4679 | |
| ethidium bromide | Sigma Aldrich | E1510-10ML | 10 mg/ml |
| QIAquick Gel Extraction Kit | QIAGEN | 28704 or 28706 | |
| iTaq Universal SYBR Green Supermix | Bio-Rad Laboratories | 1725121 or 1725122 | |
| Spectrophotometer | brand not critical | ||
| 0.45 μm PVDF Filter | brand not critical | ||
| 25 ml Seringe | brand not critical | ||
| 10 cm Tissue Culture Plates | brand not critical | ||
| 6-well Tissue Culture Plates | brand not critical | ||
| S1000 Thermal Cycler | Bio-Rad Laboratories | ||
| C1000 Touch Thermal Cycler | Bio-Rad Laboratories | for qPCR | |
| Vortex Mixer | brand not critical | ||
| Dry Block Heater or Thermomixer | brand not critical | ||
| Microcentrifuge | brand not critical | ||
| Gel electrophoresis system with power supply | brand not critical | ||
| Magnet stand | for purification of DNA with SPRI beads; should hold 1.5-2 ml tubes; brand not critical | ||
| UV transilluminator | brand not critical | ||
| E-gel electrophoresis system | Life Technologies | G6400, G6500, G6512ST |
References
- van Steensel, B., Delrow, J., Henikoff, S. Chromatin profiling using targeted DNA adenine methyltransferase. Nat Genet. 27, 304-308 (2001).
- van Steensel, B., Henikoff, S. Identification of in vivo DNA targets of chromatin proteins using tethered dam methyltransferase. Nat Biotechnol. 18, 424-428 (2000).
- Fu, A. Q., Adryan, B. Scoring overlapping and adjacent signals from genome-wide ChIP and DamID assays. Mol Biosyst. 5, 1429-1438 (2009).
- Guelen, L. Domain organization of human chromosomes revealed by mapping of nuclear lamina interactions. Nature. 453, 948-951 (2008).
- Kalverda, B., Pickersgill, H., Shloma, V. V., Fornerod, M. Nucleoporins directly stimulate expression of developmental and cell-cycle genes inside the nucleoplasm. Cell. 140, 360-371 (2010).
- Kubben, N. Mapping of lamin A- and progerin-interacting genome regions. Chromosoma. 121, 447-464 (2012).
- Steglich, B., Filion, G. J., van Steensel, B., Ekwall, K. The inner nuclear membrane proteins Man1 and Ima1 link to two different types of chromatin at the nuclear periphery in S. pombe. Nucleus. 3, 77-87 (2012).
- Harr, J. C. Directed targeting of chromatin to the nuclear lamina is mediated by chromatin state and A-type lamins. J Cell Biol. 208, 33-52 (2015).
- Gonzalez-Aguilera, C. Genome-wide analysis links emerin to neuromuscular junction activity in Caenorhabditis elegans. Genome Biol. 15, R21(2014).
- Wu, F., Yao, J. Spatial compartmentalization at the nuclear periphery characterized by genome-wide mapping. BMC Genomics. 14, 591(2013).
- Filion, G. J. Systematic protein location mapping reveals five principal chromatin types in Drosophila cells. Cell. 143, 212-224 (2010).
- Vogel, M. J. Human heterochromatin proteins form large domains containing KRAB-ZNF genes. Genome Res. 16, 1493-1504 (2006).
- Venkatasubrahmanyam, S., Hwang, W. W., Meneghini, M. D., Tong, A. H., Madhani, H. D. Genome-wide, as opposed to local, antisilencing is mediated redundantly by the euchromatic factors Set1 and H2A.Z. Proc Natl Acad Sci U S A. 104, 16609-16614 (2007).
- Shimbo, T. MBD3 localizes at promoters, gene bodies and enhancers of active genes. PLoS Genet. 9, e1004028(2013).
- Orian, A. Genomic binding by the Drosophila Myc, Max, Mad/Mnt transcription factor network. Genes Dev. 17, 1101-1114 (2003).
- Artegiani, B. Tox: a multifunctional transcription factor and novel regulator of mammalian corticogenesis. EMBO J. , (2014).
- Schuster, E. DamID in C. elegans reveals longevity-associated targets of DAF-16/FoxO. Mol Syst Biol. 6, 399(2010).
- Bianchi-Frias, D. Hairy transcriptional repression targets and cofactor recruitment in Drosophila. PLoS Biol. 2, e178(2004).
- Woolcock, K. J., Gaidatzis, D., Punga, T., Buhler, M. Dicer associates with chromatin to repress genome activity in Schizosaccharomyces pombe. Nat Struct Mol Biol. 18, 94-99 (2011).
- Luo, S. D., Shi, G. W., Baker, B. S. Direct targets of the D. melanogaster DSXF protein and the evolution of sexual development. Development. 138, 2761-2771 (2011).
- Germann, S., Gaudin, V. Mapping in vivo protein-DNA interactions in plants by DamID, a DNA adenine methylation-based method. Methods Mol Biol. 754, 307-321 (2011).
- Zhang, X. The Arabidopsis LHP1 protein colocalizes with histone H3 Lys27 trimethylation. Nat Struct Mol Biol. 14, 869-871 (2007).
- Orian, A. Chromatin profiling, DamID and the emerging landscape of gene expression. Curr Opin Genet Dev. 16, 157-164 (2006).
- Vogel, M. J., Peric-Hupkes, D., van Steensel, B. Detection of in vivo protein-DNA interactions using DamID in mammalian cells. Nat Protoc. 2, 1467-1478 (2007).
- Greil, F., Moorman, C., van Steensel, B. DamID: mapping of in vivo protein-genome interactions using tethered DNA adenine methyltransferase. Methods Enzymol. 410, 342-359 (2006).
- de Wit, E., Greil, F., van Steensel, B. Genome-wide HP1 binding in Drosophila: developmental plasticity and genomic targeting signals. Genome Res. 15, 1265-1273 (2005).
- DamID mammalian vectors. , Available from: http://research.nki.nl/vansteensellab/Mammalian_plasmids.htm (2015).
- Illumina TruSeq adaptors & PCR primers. , Available from: https://ethanomics.wordpress.com/chip-seq-library-construction-using-the-illumina-truseq-adapters/ (2015).
- Optimization of PCR cycles for NGS. , Available from: https://ethanomics.wordpress.com/ngs-pcr-cycle-quantitation-protocol/ (2015).
- Bernstein, B. E. The NIH Roadmap Epigenomics Mapping Consortium. Nat Biotechnol. 28, 1045-1048 (2010).
- Encode Project Consortium. A user's guide to the encyclopedia of DNA elements (ENCODE). PLoS Biol. 9, e1001046(2011).
- Asp, P. Genome-wide remodeling of the epigenetic landscape during myogenic differentiation. Proc Natl Acad Sci U S A. 108, E149-E158 (2011).
- Hoppe, P. S., Coutu, D. L., Schroeder, T. Single-cell technologies sharpen up mammalian stem cell research. Nat Cell Biol. 16, 919-927 (2014).
- Avital, G., Hashimshony, T., Yanai, I. Seeing is believing: new methods for in situ single-cell transcriptomics. Genome Biol. 15, 110(2014).
- Navin, N. E. Cancer genomics: one cell at a time. Genome Biol. 15, 452(2014).
- Saliba, A. E., Westermann, A. J., Gorski, S. A., Vogel, J. Single-cell RNA-seq: advances and future challenges. Nucleic Acids Res. 42, 8845-8860 (2014).
- Nagano, T. Single-cell Hi-C reveals cell-to-cell variability in chromosome structure. Nature. 502, 59-64 (2013).
- Kind, J. Single-cell dynamics of genome-nuclear lamina interactions. Cell. 153, 178-192 (2013).
- Southall, T. D. Cell-type-specific profiling of gene expression and chromatin binding without cell isolation: assaying RNA Pol II occupancy in neural stem cells. Dev Cell. 26, 101-112 (2013).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved