Method Article
DamID-след: Генома Отображение белок-ДНК взаимодействий с высокой пропускной секвенирования аденина-метилированных фрагментов ДНК
В этой статье
Резюме
Мы здесь, в описания анализа путем сочетания идентификации ДНК аденин метилтрансферазы (DamID) для высокой пропускной последовательности (DamID-след). Этот усовершенствованный способ обеспечивает более высокое разрешение и широкий динамический диапазон и позволяет анализировать DamID-SEQ данные в сочетании с другими данными секвенирования с высокой пропускной способностью, таких как чип-SEQ, РНК-SEQ и т.д.
Аннотация
The DNA adenine methyltransferase identification (DamID) assay is a powerful method to detect protein-DNA interactions both locally and genome-wide. It is an alternative approach to chromatin immunoprecipitation (ChIP). An expressed fusion protein consisting of the protein of interest and the E. coli DNA adenine methyltransferase can methylate the adenine base in GATC motifs near the sites of protein-DNA interactions. Adenine-methylated DNA fragments can then be specifically amplified and detected. The original DamID assay detects the genomic locations of methylated DNA fragments by hybridization to DNA microarrays, which is limited by the availability of microarrays and the density of predetermined probes. In this paper, we report the detailed protocol of integrating high throughput DNA sequencing into DamID (DamID-seq). The large number of short reads generated from DamID-seq enables detecting and localizing protein-DNA interactions genome-wide with high precision and sensitivity. We have used the DamID-seq assay to study genome-nuclear lamina (NL) interactions in mammalian cells, and have noticed that DamID-seq provides a high resolution and a wide dynamic range in detecting genome-NL interactions. The DamID-seq approach enables probing NL associations within gene structures and allows comparing genome-NL interaction maps with other functional genomic data, such as ChIP-seq and RNA-seq.
Введение
ДНК метилтрансферазы идентификация аденин (DamID) 1,2 является метод обнаружения взаимодействий белок-ДНК в естественных условиях и является альтернативным подходом к иммунопреципитации хроматина (чип) 3. Он использует относительно небольшое количество клеток и не требует химической сшивки белка с ДНК или очень специфических антител. Последнее особенно полезно, когда целевой белок слабо или косвенно связаны с ДНК. DamID успешно используется для отображения сайты связывания различных белков, включая ядерное белков оболочки 4-10, хроматина белков, ассоциированных с 11-13, хроматина изменения ферменты 14, транскрипционных факторов и сопутствующих факторов и РНК-интерференции 15-18 механизмов 19. Метод применим в различных организмов, включая S. Cerevisiae 13, С. pombe 7, С. Элеганс 9,17, Д. MELANOGASTER 5,11,18,20, А. THALIANA 21,22, а также мыши и человека клеточных линий 6,8,10,23,24.
Разработка анализа DamID была основана на специфической детекции аденина-метилированных фрагментов ДНК в эукариотических клетках, которые не имеют эндогенной аденин метилирование 2. Выраженный слитый белок, состоящий из ДНК-связывающего белка, представляющего интерес и Е. палочка ДНК аденин метилтрансферазы (плотины), может метилировать аденин базу в последовательности GATC, которые находятся в пространственной близости (наиболее значительно в течение 1 кб и до примерно 5 кб) сайтов связывания белка в геноме 2. Модифицированные ДНК-фрагменты могут быть специфично амплифицируют и гибридизуют с микрочипов для выявления геномных сайтов связывания белка, представляющего интерес 1,25,26. Это оригинальный способ DamID был ограничен наличием микрочипов и плотности заданных зондов. Поэтому мы интегрировали высокой пропускной последовательности вчтобы DamID 10 и обозначены метод как DamID-SEQ. Большое количество краткосрочных читает полученные от DamID-SEQ позволяет точную локализацию взаимодействий белок-ДНК генома. Мы обнаружили, что DamID-след обеспечил более высокое разрешение и более широкий динамический диапазон, чем DamID по микрочипов для изучения генома ядерной пластинки (NL) ассоциации 10. Это усовершенствованный метод позволяет зондирования NL ассоциации в пределах гена структур 10 и облегчает сравнение с другими данными секвенирования высокой пропускной способностью, таких как чип-SEQ и РНК-SEQ.
DamID-след протокол, описанный здесь был первоначально разработан для отображения генома Л. объединений 10. Мы создали гибридный белок по привязывая мыши или человека Ламин B1 Е. палочка ДНК аденин метилтрансферазы и проходят протокол в 3T3 эмбриональных фибробластов мыши, мыши C2C12 миобластов 10 и IMR90 плода фибробласты легких человека (данные не опубликованы). В этом протоколе, мы начинаем с сonstructing векторы и выражая Дам-привязанных слитые белки от лентивирусного инфекции в клетках млекопитающих 24. Далее, мы опишем подробные протоколы усиления аденин-метилированных фрагментов ДНК и подготовке секвенирования библиотек, которые должны быть применимы в других организмах.
протокол
1. Генерация и экспрессия слитых белков и бесплатно Dam белков
- Клон интерес белок в вектор DamID.
- Amplify кДНК белка интереса (POI) с нужной высокой точностью ДНК-полимеразы и соответствующих праймеров в соответствии с протоколом производителя. Экспериментально определить оптимальные условия амплификации для обеспечения надлежащего усиления вставок.
- Запуск в агарозном геле и очищают амплифицированной кДНК POI с помощью набора для экстракции геля в соответствии с протоколом производителя.
- Клон кДНК POI в вектор pDONR201 использованием BP Clonase II в соответствии с протоколом производителя.
- Клонирование кДНК POI из донорской вектора в вектор назначения pLGW-RFC1-V5-EcoDam или pLGW-EcoDam-V5-RFC1 назначения вектора 27, используя LR Clonase II в соответствии с протоколом производителя, в зависимости от желаемого направления слияния POI к N- или гое С-конец Е. палочка ДНК аденин метилтрансферазы (EcoDam) 27.
- Проверка с помощью секвенирования, что клонированной кДНК имеет правильную последовательность и формы в рамке гибридного EcoDam.
- Создание лентивирусные запасов.
- Создание лентивирусные запасов, выражающие Дам-V5-POI и V5-Дам (от вектора pLGW-V5-EcoDam 27) с помощью лентивирусные системы экспрессии. Используйте вперед процедуру трансфекции в соответствии с протоколом производителя.
- Используйте 293T клетки и липофекцию генерировать лентивирусные запасов в соответствии с протоколом производителя для трансфекции.
- Включают стадию 0,45 мкм PVDF фильтр.
- Создание лентивирусные запасов, выражающие Дам-V5-POI и V5-Дам (от вектора pLGW-V5-EcoDam 27) с помощью лентивирусные системы экспрессии. Используйте вперед процедуру трансфекции в соответствии с протоколом производителя.
- Инфицировать клетки с лентивирус.
- За день до инфекции (день 0), проходят прилипшие клетки культивировали в соответствующей ростовой среде для этого типа клеток в культуре ткани пластины 6-луночного с использованием той же питательной среды без антибиотиков додостижения 50% слияния в день инфекции. Место клеток в 37 ° С инкубатор.
- В день инфицирования (день 1), снимите 2 криопробирки обоих Дам-V5-ТОИ и V5-Дам лентивирусных супернатантах от -80 ° C морозильника и место в 37 ° С водяной бане, чтобы таять.
- Удалить питательной среды от клеток и заменить 0,5 мл свежей питательной среды без антибиотиков.
- Добавить 1 мл талой лентивирус в каждую лунку (2 скважины с V5-Дам и 2 скважины с Дам-V5-POI). Добавить 1 мл питательной среды без антибиотиков на оставшиеся 2 скважин-это будет служить в качестве отрицательного контроля. Осторожно встряхните 6-луночного планшета для смешивания и поместить обратно в 37 ° C инкубатора O / N.
- На следующий день после заражения (день 2), снимите вирусных суспензий из клеток и заменить 2 мл питательной среды без антибиотиков. Место клетки обратно в 37 ° C инкубаторе в течение 48 часов.
- Изолировать GDNA.
- Аспирируйте СМИ из каждой лунки и деTach клетки, используя 250 мкл 0,05% трипсина-EDTA. Инкубируют при 37 ° С в течение 2 мин.
- Вымойте клеток с пластинки с 1 мл питательной среды и пипетки клеток из каждой лунки в 1,5 мл микроцентрифужных трубки. Гранул клетки центрифугированием при 200 х г в течение 5 мин при комнатной температуре.
- Промыть осажденные клетки с 500 мкл PBS и центрифугировать при 200 х г в течение 2 мин при комнатной температуре.
- Ресуспендируют гранулированных клеток в 200 мкл PBS.
- Изолировать GDNA по крови и тканей комплект в соответствии с протоколом производителя. Элюировать гДНК в 200 мкл буфера AE и определить концентрацию путем измерения оптической плотности 260 с помощью спектрофотометра.
Примечание: GDNA из неинфицированных или инфицированных клеток макет может быть выделен в качестве отрицательных контролей. Осадок GDNA более высоких концентраций для длительного хранения.- Добавить 3 объемов 100% этанола и 0,1 объема 3 м ацетата натрия (рН 5,5), и перемешивают переворачиванием трубок 4-6 раз.
- Хранить при -20 ° CO / N.
- Центрифуга на 16000 хг в течение 15 мин при 4 ° С.
- Осторожно удалите супернатант. Промыть гранул с 70% (объем / объем) этанола и центрифугируют при 16000 х г в течение 5 мин при 4 ° С.
- Осторожно снимите этанола и позволяют гранулы высохнуть на воздухе в течение 3 мин при комнатной температуре.
- Растворить в GDNA T 10 E 0,1 (рН 7,5) до примерно 1 мкг / мкл. Бассейн гДНК от каждого экспериментального образца или отрицательного контроля и измерения концентрации. Хранить при -20 ° C.
2. Усиление аденин-метилированных фрагментов ДНК
- Дайджест гДНК с DpnI, что только сокращает аденин-метилированных GATCs.
- Настройка реакции на льду с 2,5 мкг гДНК, 1 мкл 10х буфера, 0,5 мкл DpnI (20 ед / мкл) и залить H 2 O до полного объема 10 мкл. Для каждого образца GDNA, подготовить три реакции - один без DpnI ("нет DpnI" заменить ДПНИ 0,5 мкл H 2 O) и двух жIth DpnI ("с DpnI").
- Дайджест O / N при 37 ° С и инактивируют ДПНИ при 80 ° С в течение 20 мин.
- Перевязывать DamID адаптеров.
- Подготовка DamID адаптеров.
- Ресуспендируют каждый из двух олигонуклеотидов DamID адаптера 24 в H 2 O до конечной концентрации 100 мкМ.
- Комбинат равные объемы двух DamID адаптер олиго, перемешать и поместить в плотно закрытой трубе. Уплотнение трубки парапленкой, сидеть в стойке и места в химический стакан, наполненный водой при 90 ° С. Хранить уровень воды ниже крышки трубки (чтобы избежать попадания воды в трубку), но над поверхностью олиго смеси.
- Дайте воде остыть до комнатной температуры, так что отжиг адаптеры медленно.
- Алиготе Отожженном адаптеры (50 мкм) и хранить при температуре -20 ° C.
- Настройка реакции на льду. В каждую пробирку с 2.1.2, добавить 6,2 мкл H 2 O, 2 мкл 10х буфера для лигирования, 0,8 мкл 50 мкМ DamID объявлениеaptors (размороженные на льду) и 1 мкл ДНК-лигазы Т4 (5 ед / мкл). Общий объем составляет 20 мкл. В одном из двух "с ДПНИ" трубок, замените лигазы с 1 мкл H 2 O ("нет лигазы") Обратите внимание, что каждый образец имеет две гДНК отрицательных контролей -. "Нет ДПНИ» и «нет» лигазы.
- Лигировать O / N при 16 ° С и инактивируют лигазы при 65 ° С в течение 10 мин.
- Подготовка DamID адаптеров.
- Дайджест ДНК с DpnII уничтожить фрагменты, содержащие неметилированные GATCs.
- Настройка реакции на льду. В каждую пробирку с 2.2.3, добавить 24 мкл H 2 O, 5 мкл 10х буфера DpnII и 1 мкл DpnII (10 ед / мкл). Общий объем составляет 50 мкл.
- Дайджест при 37 ° С в течение 2-3 ч и инактивации DpnII при 65 ° С в течение 20 мин.
- Amplify аденин-метилированных фрагментов ДНК.
- Настройка реакции на льду с 5 мкл DpnII переварить из 2.3.2, 5 мкмл 10x ПЦР буфер, 12,5 мкл 5 мкМ DamID ПЦР-праймер 24, 4 мкл 10 мМ дНТФ перемешать, 1 мкл 50x полимеразной смеси и 22,5 мкл H 2 O. Общий объем составляет 50 мкл.
- Запуск ПЦР следующим образом: 68 ° C в течение 10 мин; 94 ° С в течение 1 мин, 65 ° С в течение 5 мин, 68 ° С в течение 15 мин; 4 циклов при 94 ° С в течение 1 мин, 65 ° C в течение 1 мин, 68 ° С в течение 10 мин; 17 циклов при 94 ° С в течение 1 мин, 65 ° С в течение 1 мин, 68 ° С в течение 2 мин.
- Анализ 5 продуктов мкл ПЦР из каждой реакции на 1% агарозном геле. ПЦР-продукты должны появиться в мазке в диапазоне от 0,2 до 2 кб (рис 2). "Нет DpnI" и "нет" лигазы управления должны иметь не менее или явно усиления.
- Если результат от шага 2.4.3 удовлетворительное, повторить шаги 2.4.1-2.4.3 с двумя или тремя реакций экспериментального образца и одной реакции для каждой из двух отрицательных контролей.
- Бассейн и очистки продуктов ПЦР из того жеЭкспериментальный образец с использованием комплектов очистки ПЦР или твердой фазе Реверсивный иммобилизация (SPRI) бусы в соответствии с протоколом производителя. Не очистить "нет ДПНИ" или "Нет" лигазы управления. Элюции ДНК с буферной EB.
- Измерение концентрации очищенной ДНК путем измерения оптической плотности 260, используя спектрофотометр, который должен быть около 0,1 мкг / мкл или выше. Собрать минимум 10 мкг ДНК для каждого образца. При использовании ПЦР наборы для очистки, очистить каждый 50 мкл ПЦР-продуктов с одной колонкой, элюируют в 30 мкл буфера EB и бассейн элюатов.
- Дайджест ДНК с DpnII, чтобы предотвратить DamID ПЦР-праймеров от того секвенировали.
- Настройка реакции на льду с 5 мкг очищенной ДНК из 2.4.6, 5 мкл 10х буфера DpnII, 1 мкл DpnII (10 ед / мкл) и залить H 2 O до полного объема 50 мкл. Приготовьте два или три реакции для каждого образца.
- Дайджест при 37 ° С в течение 2-3 чи инактивируют DpnII при 65 ° С в течение 20 мин.
- Бассейн и очистить дайджестов из того же образца с комплектов очистки ПЦР или бисером SPRI в соответствии с протоколом производителя. Элюции ДНК с буферной EB.
- Измерение концентрации очищенной ДНК, которое должно быть около 0,06 мкг / мкл или выше. Собрать минимум 6 мкг ДНК для каждого образца. При использовании ПЦР наборы для очистки, очистить каждый 50 мкл переварить с одной колонкой, элюируют в 30 мкл буфера EB и бассейн элюатов.
3. Библиотека Подготовка к высокопроизводительного секвенирования
- ДНК-фрагмент
- Экспериментально определить подходящее время пищеварения для каждого нового партии двухцепочечной ДНК Fragmentase. Как активность фермента может уменьшаться с течением времени, повторите тест перед выполнением нового эксперимента. Чтобы сохранить ДНК 2.5.4 для фактического фрагментации, используйте очищенные продукты ПЦР метил из 2.4.6 или дополнительной ДНК из предыдущего experimeНТС в этом шаге.
- Установите мастер-микс с 6 мкг ДНК, 12 мкл 10х буфера Fragmentase и залейте H 2 O до общего объема 114 мкл в.
- Вихрь фондовый флакон Fragmentase в течение 3 сек, 6 мкл добавить в мастер-смеси и вихрь мастер смеси в течение 3 сек. Общий объем составляет 120 мкл.
- Аликвоты 20 мкл мастер смеси в каждую из 5 новых труб. Инкубируйте все 6 пробирки при 37 ° С в течение от 5 до 55 мин при приращения 10 мин. Добавьте 5 мкл 0,5 М ЭДТА для остановки реакции.
- Анализ 12,5 мкл переварить (0,5 мкг ДНК) каждой реакции, а также 0,5 мкг ДНК на непереваренные в агарозном геле (рис 3). Определить минимальное время (Т), необходимое 0.2kb переваривать большинство мазка до примерно 0,2 кб. Выберите 6 Время длительности между 5 мин и Т 0.2kb (в том числе 5 мин и Т 0.2kb) с равными шагом для реальной фрагментации.
- Настройка фактических Фрагменатации, как описано в 3.1.1.1-3.1.1.3. Выдержите реакции при 37 ° С в течение времени продолжительности, определенных в 3.1.1.4.
- Бассейн 6 реакции и очистить дайджестов с комплектов очистки ПЦР или бисером SPRI в соответствии с протоколом производителя. Элюции ДНК в 51 мкл буфера EB. Конечный элюат ~ 50 мкл.
- Экспериментально определить подходящее время пищеварения для каждого нового партии двухцепочечной ДНК Fragmentase. Как активность фермента может уменьшаться с течением времени, повторите тест перед выполнением нового эксперимента. Чтобы сохранить ДНК 2.5.4 для фактического фрагментации, используйте очищенные продукты ПЦР метил из 2.4.6 или дополнительной ДНК из предыдущего experimeНТС в этом шаге.
- Ремонт концы фрагментированной ДНК
- Настройка реакции на льду с элюата из 3.1.3, 25 мкл H 2 O, 10 мкл 10х Т4 ДНК-лигазы буфера с 10 мМ АТФ, 4 мкл 10 мМ дНТФ смеси, 5 мкл ДНК-полимеразы Т4 (3 U / мкл) , 1 мкл Кленова ДНК-полимеразы (5 ед / мкл), и 5 мкл киназы Т4 полинуклеотид. Общий объем составляет 100 мкл. Хорошо перемешать с пипеткой. Избегайте пены и пузырьков.
- Инкубируют при 20 ° С в течение 30 мин.
- Очищают ДНК с комплектов очистки ПЦР или бисером SPRI в соответствии с протоколом производителя. Элюции ДНК в 33 мкл буфера EB. Окончательный элюата ~ 32 & #181; л.
- Добавить "А" свесы
- Настройка реакции на льду с элюата из 3.2.3, 5 мкл 10х буфера Кленова, 10 мкл 1 мМ дНТФ, и 3 мкл Кленова (3 '→ 5' экзо-) (5 ед / мкл). Общий объем составляет 50 мкл. Хорошо перемешать с пипеткой. Избегайте пены и пузырьков.
- Инкубируют при 37 ° С в течение 30 мин.
- Очищают ДНК с комплектов очистки ПЦР или бисером SPRI в соответствии с протоколом производителя. Элюции ДНК в 22 мкл буфера EB. Окончательный элюата ~ 21 мкл.
- Перевязывать секвенирования адаптеры
- Подготовка секвенирования адаптеры 28.
- Ресуспендируют каждый адаптер олиго до концентрации 100 мкМ в 10 мМ Трис-Cl (рН 7,8), 0,1 мМ ЭДТА (рН 8,0) и 50 мМ NaCl.
- Смешайте равные объемы универсального адаптера 28 и индексированной адаптер 28.
- Отжиг адаптеры в термоциклере со следующимиПрограмма: 95 ° С в течение 2 мин; 140 циклов в течение 30 сек, начиная с 95 ° C и снижение на 0,5 ° C в каждом цикле; выдержка при температуре 4 ° С.
- Алиготе адаптеры и хранить при -20 ° C.
- Настройка реакции на льду с элюата из 3.3.3, 25 мкл 2x буфера для лигирования, 1,5 мкл 50 мкМ ренатурированных секвенирования адаптеры (талой на льду) из 3.4.1.4 и 2.5 мкл ДНК-лигазы Т4. Хорошо перемешать с пипеткой. Избегайте пены и пузырьков. Если мультиплекс секвенирования желательно, использовать другой индексированный адаптер для каждого образца.
- Инкубируют при комнатной температуре в течение 1 часа.
- Очищают ДНК с комплектов очистки ПЦР или бисером SPRI в соответствии с протоколом производителя. Элюции ДНК в 24 мкл буфера EB. Окончательный элюата ~ 23 мкл.
- Подготовка секвенирования адаптеры 28.
- Преобразование Y-образный адаптеры двухцепочечной ДНК, чтобы позволить точно определить размеры фрагмент ДНК 28
- Настройка реакции на льду с элюата из 3.4.4, 12.5 мкл H 2 O, 1 мкл 25 мкм грунтовки 1 28, 1 мкл 25 мкм грунтовки 2 28, 1,5 мкл 10 мМ дНТФ, 10 мкл 5х буфера ПЦР и 1 мкл ДНК-полимеразы (1 ед / мкл).
- Запуск ПЦР следующим образом: 95 ° C в течение 3 мин; 5 циклов 98 ° С в течение 15 сек, 63 ° C в течение 30 сек, 72 ° С в течение 30 сек; 72 ° С в течение 1 мин; 4 ° С на удержание.
- Очищают ДНК с комплектов очистки ПЦР или бисером SPRI в соответствии с протоколом производителя. Элюции ДНК в 11 мкл буфера EB. Окончательный элюата ~ 10 мкл.
- Размер выберите библиотеку
- Приготовьте 2% агарозный гель с 1x TAE буфер. Добавить бромистый этидий (Внимание!) До конечной концентрации 500 нг / мл, когда расплавленный агарозном TAE-охлаждения раствора, чтобы избежать вдыхания бромистым этидием. Убедитесь, что есть достаточно полосы для всех образцов ДНК, лестницы и пустых полос.
- Добавить 8 мкл 6x загрузки красителя элюата из 3.5.3.
- Подготовка ДНК лестнице путем смешивания 1 кб плюс ДНК лестнице (1,0 мкг / мкл), 6x загрузки красителя и H 2 O в соотношении 1: 1: 4.
- Нагрузка 6 мкл ДНК лестнице, образцы из 3.6.2 и еще 6 мкл ДНК лестницу, каждый в отдельном ряду и, по крайней мере одной пустой полосе от соседних образцов / лестниц.
- Запустите гель при 120 V в течение 60 мин.
- Просмотр гель на УФ-просвечивания (свести к минимуму время экспозиции УФ). Носите защитные очки и маску. Убедитесь, что по крайней мере один из лестниц ДНК хорошо работать с соответствующим интервалом (достаточно для вырезания 3 ломтика гель) между 300 б.п. и 400 б.п. полос. Узкий интервал увеличивает трудности в вырезая несколько фрагментов гель, в то время как широкий интервал увеличивает объем подакцизных ломтиками гель.
- Используйте новую скальпель или лезвие для каждой полосы. Акцизный три тонкие ломтики гель между 300 и 400 б.п. от каждой полосе (рис 4) и поместите их в каждый микроцентрифужных трубки. Держите объем каждого геля свшей цене возможного (<100 мкл).
- Измерьте объем каждого куска геля (1 мкл гель весит приблизительно 1 мг), добавляют 6x объемы QG буфера и инкубируют при 50 ° С.
- Vortex смеси КГ-гель через 2-3 мин, пока гель не кусочек полностью не растворится. Добавить 2x объемы гель изопропанола и перемешать.
- От этого шага, следовать протоколу от добычи гель комплектов. Элюции ДНК в 51 мкл буфера EB. Конечный элюат ~ 50 мкл.
- Пополните секвенирования адаптер изменены фрагментов ДНК
- Оптимизировать количество циклов ПЦР 29.
- Настройка реакции на льду с 1 мкл элюата из 3.6.10, 1 мкл 25 мкМ праймера 1 28, 1 мкл 25 мкМ праймера 2 28, 7 мкл H 2 O и 10 мкл SYBR Green Supermix. Общий объем составляет 20 мкл.
- Запуск КПЦР следующим образом: 95 ° C в течение 3 мин; 20 циклов 95 ° С в течение 30 секунд, 63 ° C в течение 30 сек, 72 ° С в течение 30 сек, пластины чтения.
- Анализ данных с помощью программного обеспечения КПЦР анализа и определения количественного цикла (CQ) или порогового цикла (CT) для каждого образца с использованием протокола производителя. Продолжить с образцами, которые Cq / КТ ≤ 14. Используйте максимальное Сч (ОВ) 0 минус 1 (с округлением до ближайшего целого числа), как конечное число ПЦР цикла (N ПЦР).
- Регулировка количества ДНК-матриц, так что разные образцы будут усилены приблизительно равных количествах после запуска одинаковое количество циклов ПЦР. Использование шаблона 8 мкл для образца с самой высокой Cq (Cq 0), и рассчитать объем шаблона других образцов по следующей формуле:
Том I = 8 х 1,8 Сч я -cq 0
- Настройка ПЦР реакции на льду с объемами шаблонов, рассчитанных по 3.7.1.4: 1 мкл 25 мкМ праймера 1 28, 1 мкл 25 мкМ праймера 2 28 1.5мкл 10 мМ дНТФ, 10 мкл 5х буфера, 1 мкл ДНК-полимеразы (1 ед / мкл) и залить H 2 O до полного объема 50 мкл.
- Запуск ПЦР следующим образом: 95 ° C в течение 45 сек; N циклов ПЦР (определяется в 3.7.1.3) из 98 ° С в течение 15 сек, 63 ° C в течение 30 сек, 72 ° С в течение 30 сек; 72 ° С в течение 1 мин; 4 ° С на удержание.
- Анализ 5 продуктов ПЦР мкл в 2% агарозном геле (5А). Ясное "сингл" группа указывает, что амплифицированные фрагменты ДНК в пределах узкого диапазона длины и, что библиотека ДНК может быть предметом дальнейшего анализа.
- Повторите одна реакция для выбранных образцов и объединить усиленные библиотеки ДНК из того же образца.
- Очищают выбранных библиотек ДНК для секвенирования.
- Если грунт / адаптер димеры не видны в 3.7.4, очистить библиотеки ДНК с комплектов очистки ПЦР или бисером SPRI в соответствии с протоколом производителя. Элюции ДНК в 21 мкл буфера EB.Окончательный элюата ~ 20 мкл. Если последовательность объект пользователя имеет конкретные инструкции о буфере элюции, в конечном объеме, и т.д., подготовить образцы соответственно.
- Если грунт / адаптер димеры четко видны в 3.7.4, очистить библиотеки следующим образом.
- Нагрузка ДНК из 3.7.5 и лестница ДНК в 2% агарозном геле сборного. Образцы могут быть очищены, как описано в 3.7.6.1, чтобы уменьшить громкость или могут быть загружены в нескольких скважинах. Поместите агарозный гель в соответствующем энергосистемы. Разрешить гель для запуска в течение 15 мин.
- При помощи соответствующего просвечивания, вырезали нужный диапазон с чистой скальпелем / бритва для каждого образца и поместить гель срез в 1,5 мл микроцентрифужных трубки.
- Выделения ДНК с помощью гель-экстракции комплекты в соответствии с протоколом производителя с двух модификаций: инкубируют буфера QG-гель смеси в смесителе термо-при 37 ° С и 1000 оборотов в минуту в течение 30 мин, и добавить 2x объемы гель изопропанола.
- Оптимизировать количество циклов ПЦР 29.
- Отправить очищенные ДНК-библиотек к секвенирования объекта. Выполняйте все рекомендации фонда.
Примечание: Анализ качества по Bioanalyzer (фиг.5В) должны быть выполнены перед секвенированием с целью определения точного диапазон размеров и концентрации ДНК-библиотеки.
Результаты
Гибридный белок Dam-V5-LmnB1 была проверена быть совместно локализованы с эндогенного белка по иммунофлуоресцентного Ламин B (рис 1).
Успешное ПЦР-амплификации из аденина-метилированных фрагментов ДНК является ключевым шагом для DamID-SEQ. Экспериментальные образцы должны усиливать мазок 0,2 - 2 Кб, отрицательных контролей (без DpnI, без лигазы или без ПЦР шаблона) должно привести к не-или-менее ясно усиления (рис 2).
Метилированные фрагменты ДНК находятся в диапазоне от 0,2 до 2 кб, в то время как желаемый размер вставки для библиотеки NGS составляет от 200 до 300 п.н.. Таким образом, важно, чтобы фрагментировать метил продуктов ПЦР в подходящем диапазоне размеров. Тем не менее, было установлено, что непрактично одновременно разрушить большие фрагменты ДНК до подходящего размерас и сохранить большинство более мелких фрагментов ДНК интактных продолжительности одного фрагментации. Таким образом, эксперименты курс время были выполнены, чтобы определить минимальное время (Т 0.2kb), необходимых для фрагмент 1 мкг ДНК в мазке с центром в 200 б.п. (рис 3). Тогда 6 раз продолжительность в равных приращений были отобраны от 5 мин и Т 0.2kb для фактического фрагментации. Ферментативная активность двойной цепи ДНК Fragmentase может варьироваться от партии к партии и может уменьшаться с течением времени, поэтому рекомендуется повторить этот шаг для новой партии Fragmentase или после хранения в течение определенного периода времени.
Желаемый размер вставки между 200 и 300 п.н., соответствующих фрагментов ДНК от 300 пар оснований и 400 (в том числе 121 п.н. секвенирования адаптеров) на агарозном геле. Три тонкие ломтики в этом диапазоне вырезали из каждого экспериментального образца, чтобы сузить диапазон размеров библиотеки и увеличить возможностьполучения по крайней мере одного квалифицированного библиотеку секвенирования (рисунок 4).
Аликвоту из 5 мкл каждого амплифицированной библиотеки ДНК анализировали на агарозном геле, чтобы определить, какие библиотеки могут претендовать на последовательности. Как показано на фиг.5А, четкое одну полосу того же размера, вырезанной куска геля должна быть видна на агарозном геле (этап 3.7.4). Далее, выбранные библиотеки были рассмотрены в Bioanalyzer (рис 5B), чтобы определить точный диапазон размера и концентрации до секвенирования. При желании, амплифицированные ДНК-библиотек могут быть непосредственно осмотрены Bioanalyzer без анализа гель. Когда несколько библиотек имеют хорошее качество, рекомендуется последовательность библиотек аналогичного размера колеблется в течение пары экспериментальных (клеток, экспрессирующих Дам-V5-POI) и контроля (клетки, экспрессирующие V5-Дам) образцы.
Короткий читает порождаетсяупорядоченные системы были впервые отображены обратно к соответствующему генома. Уникально выровнены читает затем были переданы в последующих анализах. Трубопровод для обработки короткого читает, построить генома Л. карту взаимодействия анализа генов и-NL ассоциации были подробно описаны в нашей предыдущей работе 10. Типичные результаты показаны на фиг.6.

Рисунок 1. Проверка субъядерных локализации слитых белков иммунофлуоресцентного. IMR90 человека фибробластов легких клетки трансфицированы плазмидой, выражающей Дам-V5-LmnB1 и окрашивали анти-Ламин B (A, красный) и анти-V5 (B, зеленый). (С) Слияние изображений в А и В. Пожалуйста, нажмите здесь, чтобы посмотреть увеличенную версию Тего фигура.

Рисунок 2. ПЦР амплификации аденин-метилированных фрагментов ДНК. Аликвоту 5 мкл от каждой ПЦР-реакции анализировали на 1% агарозном геле. Мазок от 0,2 до 2 кб усиливается с каждой экспериментальной выборки, но не усиление не наблюдалось отрицательных контролей (не DpnI в шаге 2.1, не лигазы в шаге 2.2, или нет шаблона ПЦР). Пожалуйста, нажмите здесь, чтобы посмотреть большую версию из этой фигуры.
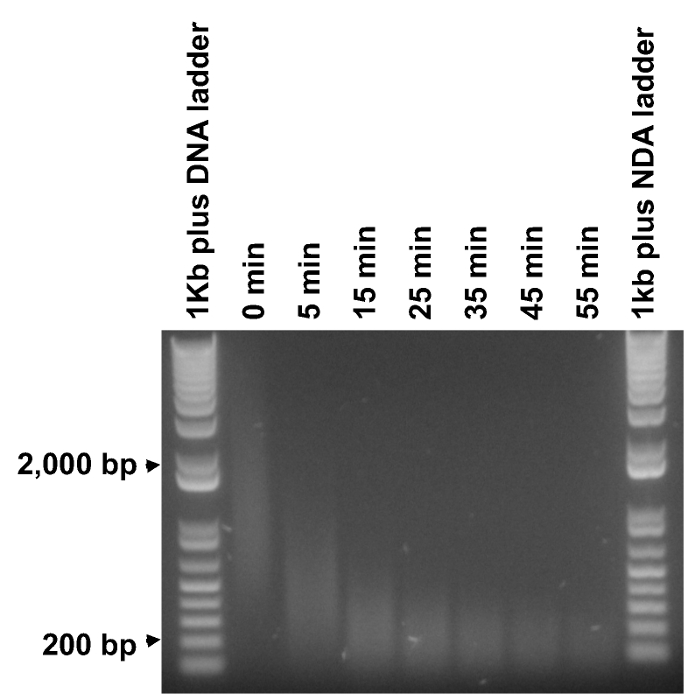
Рисунок 3. Определение оптимальных длительности фрагментации. Очищенные продукты ПЦР метил подлежали дроблению на время длительности от 5 до 55 мин при приращения 10 мин (непереваренной ДНК , Помечены как "0 мин"). 1 мкг ДНК лестнице и 0,5 мкг каждого образца ДНК фрагментированной анализировали на агарозном геле. Минимальное время для переваривания большинство мазка ДНК приблизительно 200 п.н. был определен около 35 мин, поэтому шесть равномерно расположенных времени продолжительности от 5 до 35 мин (5 и 35 мин в комплекте) были выбраны для выполнения фактического фрагментации. Пожалуйста, Нажмите здесь, чтобы посмотреть большую версию этой фигуры.

Рисунок 4. Размер выбора библиотеки ДНК. Образцы ДНК Дам-V5-LmnB1 и V5-Дам с шага 3.6 были работать на 2% агарозном геле, и три одинаковых по размеру кусочки геля (L, M и H, соответствующий низкий, средний и высоко в размере) вырезали между 300 и 400 п.о., как показано желтых линий. ом / файлы / ftp_upload / 53620 / 53620fig4large.jpg "целевых =" _blank "> Нажмите здесь, чтобы посмотреть большую версию этой фигуры.
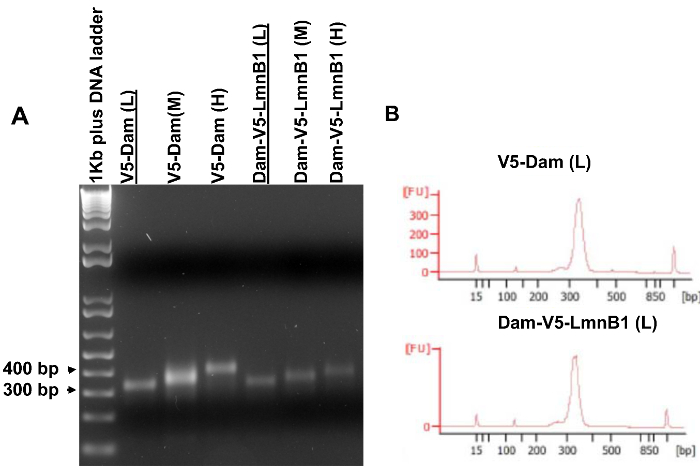
Рисунок 5. Библиотеки амплифицированной ДНК для секвенирования высокой. (А) Усиленный библиотек ДНК анализировали на агарозном геле. Шаблоны ПЦР очищали из геля ломтиками указанных на фиг.4. (Б) Результаты Bioanalyzer о V5-Дам (L) выборки и плотины-V5-LmnB1 (L) выборки, которые были подчеркнуты в (а). Эти две библиотеки были подобные диапазоны узкие размера и, следовательно, право на высокой пропускной последовательности. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
JPG "/>
Рисунок 6. Данные, отображаемые в NGS УСК генома браузер. Mouse хромосомы 1 в качестве примера. Данные последовательности были получены из мыши С2С12 миобластов 10. Треки "MB.LmnB1.w2k" и "MB.Dam.w2k", что соответствует данным клеток, экспрессирующих Дам-V5-LmnB1 и V5-Dam, соответственно, участок нормализуется плотности чтения (чтение за килобаза на миллион однозначно отображается читает, или RPKM ) в непересекающихся подряд окон 2 кб вдоль хромосомы. Трек "MB.log2FC.w2k" участки генома NL ассоциации, то есть log 2 RPKM отношения LmnB1 более плотины, в разрешением 2 кб. Трек "MB.sLADs" рисует секвенирования на основе Lamina Ассошиэйтед Домены (sLADs, т.е. геномных регионов, имеют более высокие плотности читаем LmnB1 над плотины со статистической значимостью) в черной, не sLADs в серых и неопределенных областей в белом. Пожалуйста, нажмите здесь, чтобы downloaD этот файл.
Обсуждение
Whether Dam-tagged proteins retain the functions of endogenous proteins should be examined before a DamID-seq experiment. The subcellular localization of Dam-tagged nuclear envelope proteins should always be determined and compared with that of the endogenous proteins. For studying transcription factors, it is suggested to examine whether the Dam-fusion protein can rescue the functions of the endogenous protein in regulating gene expression. This functional test can be performed in organisms in which knockout mutants of endogenous DNA-binding proteins are available. Because advances in genome engineering have potentially allowed knocking out any endogenous gene of interest, functions of Dam-tagged DNA-binding proteins can be examined in cultured mammalian cells.
The critical step in this protocol is to successfully fragment the DpnII-digested DamID PCR products to around 200 bp. This step is designed to render the amplified adenine-methylated fragments to a narrow size range for sequencing and to randomize the starting nucleotides of the DNA fragments in a sequencing library. Inefficient fragmentation will leave the majority of the DNA fragments starting with GATC (the 5'-overhang from the second DpnII digestion), and will result in a much lower performance and yield or even a failure in Illumina sequencing. Other DNA fragmentation methods may be used as an alternative approach.
The resolution of DamID (and DamID-seq described here) is limited by the frequency of GATCs in the genome to be studied. Moreover, even with high throughput sequencing, the genomic localizations of a DNA-binding protein can only be mapped within two consecutive GATCs rather than to the actual DNA-binding sites.
Despite its limitation, the DamID assay has important advantages. Because DamID does not require highly-specific antibodies, it can be used to detect a subset of nuclear proteins that could be difficult to assay by ChIP (such as the nuclear envelope proteins). To study how these proteins regulate genome functions, it is important to integrate and cross-analyze their genome-wide localization data with the current epigenomic mapping data (such as data from the ENCODE and NIH Roadmap Epigenomics Projects 30,31). The DamID-seq approach provides both higher resolution and higher sensitivity than DamID by microarray and enables detecting differential NL-associations within gene structures 10. A combinatorial analysis of DamID-seq data, ChIP-seq data 32 and gene expression data has identified a class of NL-associated genes with distinct epigenetic and transcriptional features (data not published).
Another advantage of DamID is that it only requires a small number of cells. In recent years, there has been an explosion in single cell analysis of gene regulation 33,34. Although genome sequence 35, genome-wide gene expression 36 and chromatin conformation 37 can be assayed in a single cell, there has not been an available approach for detecting protein-DNA interactions genome-wide in a single cell. DamID-seq is a highly promising approach for this goal, and may complement the single cell imaging approach in detecting the dynamics of genome-NL interactions 38. One complication is that because the Dam-fusion protein is expressed at a much lower level than the endogenous protein in the DamID assay, it is possible that the Dam-fusion protein may only occupy a subset of genomic binding sites as compared to the endogenous protein.
DamID assay has mostly been used in cultured animal cells to detect protein-DNA interactions. Notably, developmental biologists have applied this assay in detecting protein-DNA interactions in specific cell types in vivo. For example, Dam-tagged RNA polymerase II was expressed specifically in Drosophila neural stem cells to detect their genome-wide occupancy without cell isolation 39. DamID-seq will be highly useful to study the genome-wide localizations of nuclear envelope proteins, transcription factors and chromatin regulators during development in animal models.
Раскрытие информации
The authors have nothing to disclose.
Благодарности
We thank Dr. Bas van Steensel for providing the DamID mammalian expression vectors. We thank Yale Center for Genome Analysis and the Genomics Core in Yale Stem Cell Center for advice on preparing NGS libraries and implementing high throughput DNA sequencing. This work was supported by the startup funding from Yale School of Medicine, a Scientist Development Grant from American Heart Association (12SDG11630031) and a Seed Grant from Connecticut Innovations, Inc. (13-SCA-YALE-15).
Материалы
| Name | Company | Catalog Number | Comments |
| ViraPower Lentiviral Expression Systems | Life Technologies | K4950-00, K4960-00, K4970-00, K4975-00, K4980-00, K4985-00, K4990-00, K367-20, K370-20, and K371-20 | |
| Gateway BP Clonase II Enzyme Mix | Life Technologies | 11789-020 | |
| Gateway LR Clonase II Enzyme Mix | Life Technologies | 11791-020 | |
| DNeasy Blood & Tissue Kit (250) | QIAGEN | 69506 or 69504 | |
| Gateway pDONR 201 | Life Technologies | 11798-014 | |
| 293T cells | American Type Culture Collection | CRL-11268 | |
| Trypsin-EDTA (0.05%), phenol red | Life Technologies | 25300-054 | |
| DMEM, high glucose, pyruvate | Life Technologies | 11995-065 | |
| Fetal Bovine Serum | Sigma | F4135 | |
| Tris | brand not critical | ||
| EDTA | brand not critical | ||
| 200 Proof EtOH | brand not critical | ||
| Isopropanol | brand not critical | ||
| Sodium Acetate | brand not critical | ||
| DpnI | New England Biolabs | R0176 | supplied with buffer |
| DamID adaptors "AdRt" and "AdRb" | Integrated DNA Technologies | sequences available in ref. 24; no phosphorylation of the 5' or 3' end to prevent self-ligation. | |
| T4 DNA Ligase | Roche Life Science | 10481220001 | supplied with buffer |
| DpnII | New England Biolabs | R0543 | supplied with buffer |
| DamID PCR primer "AdR_PCR" | Integrated DNA Technologies | sequences available in ref. 24 | |
| Deoxynucleotide (dNTP) Solution Set | New England Biolabs | N0446 | 100 mM each of dATP, dCTP, dGTP and dTTP |
| Advantage 2 Polymerase Mix | Clontech | 639201 | supplied with buffer |
| 1Kb Plus DNA Ladder | Life Technologies | 10787018 | 1.0 µg/µl |
| QIAquick PCR Purification Kit | QIAGEN | 28104 or 28106 | |
| MinElute PCR Purification Kit | QIAGEN | 28004 or 28006 | for an elution volume of less than 30 µl |
| SPRI beads / Agencourt AMPure XP | Beckman Coulter | A63880 | apply extra mixing and more elution time if less than 40 µl elution buffer is used |
| Buffer EB | QIAGEN | 19086 | |
| NEBNext dsDNA Fragmentase | New England Biolabs | M0348 | supplied with buffer |
| T4 DNA Ligase Reaction Buffer | New England Biolabs | B0202 | |
| T4 DNA Polymerase | New England Biolabs | M0203 | |
| DNA Polymerase I, Large (Klenow) Fragment | New England Biolabs | M0210 | |
| T4 Polynuleotide Kinase | New England Biolabs | M0201 | |
| Klenow Fragment (3’ → 5’ exo-) | New England Biolabs | M0212 | supplied with buffer |
| sequencing adaptors | Integrated DNA Technologies | sequences available in ref. 28 | |
| Quick Ligation Kit | New England Biolabs | M2200 | used in 3.4.3; supplied with Quick Ligation Reaction Buffer and Quick T4 DNA Ligase |
| sequencing primer 1 and 2 | Integrated DNA Technologies | sequences available in ref. 28 | |
| KAPA HiFi PCR Kit | Kapa Biosystems | KK2101 or KK2102 | supplied with KAPA HiFi DNA Polymerase, 5x KAPA HiFi Fidelity Buffer and 10 mM dNTP mix |
| agarose | Sigma Aldrich | A4679 | |
| ethidium bromide | Sigma Aldrich | E1510-10ML | 10 mg/ml |
| QIAquick Gel Extraction Kit | QIAGEN | 28704 or 28706 | |
| iTaq Universal SYBR Green Supermix | Bio-Rad Laboratories | 1725121 or 1725122 | |
| Spectrophotometer | brand not critical | ||
| 0.45 μm PVDF Filter | brand not critical | ||
| 25 ml Seringe | brand not critical | ||
| 10 cm Tissue Culture Plates | brand not critical | ||
| 6-well Tissue Culture Plates | brand not critical | ||
| S1000 Thermal Cycler | Bio-Rad Laboratories | ||
| C1000 Touch Thermal Cycler | Bio-Rad Laboratories | for qPCR | |
| Vortex Mixer | brand not critical | ||
| Dry Block Heater or Thermomixer | brand not critical | ||
| Microcentrifuge | brand not critical | ||
| Gel electrophoresis system with power supply | brand not critical | ||
| Magnet stand | for purification of DNA with SPRI beads; should hold 1.5-2 ml tubes; brand not critical | ||
| UV transilluminator | brand not critical | ||
| E-gel electrophoresis system | Life Technologies | G6400, G6500, G6512ST |
Ссылки
- van Steensel, B., Delrow, J., Henikoff, S. Chromatin profiling using targeted DNA adenine methyltransferase. Nat Genet. 27, 304-308 (2001).
- van Steensel, B., Henikoff, S. Identification of in vivo DNA targets of chromatin proteins using tethered dam methyltransferase. Nat Biotechnol. 18, 424-428 (2000).
- Fu, A. Q., Adryan, B. Scoring overlapping and adjacent signals from genome-wide ChIP and DamID assays. Mol Biosyst. 5, 1429-1438 (2009).
- Guelen, L. Domain organization of human chromosomes revealed by mapping of nuclear lamina interactions. Nature. 453, 948-951 (2008).
- Kalverda, B., Pickersgill, H., Shloma, V. V., Fornerod, M. Nucleoporins directly stimulate expression of developmental and cell-cycle genes inside the nucleoplasm. Cell. 140, 360-371 (2010).
- Kubben, N. Mapping of lamin A- and progerin-interacting genome regions. Chromosoma. 121, 447-464 (2012).
- Steglich, B., Filion, G. J., van Steensel, B., Ekwall, K. The inner nuclear membrane proteins Man1 and Ima1 link to two different types of chromatin at the nuclear periphery in S. pombe. Nucleus. 3, 77-87 (2012).
- Harr, J. C. Directed targeting of chromatin to the nuclear lamina is mediated by chromatin state and A-type lamins. J Cell Biol. 208, 33-52 (2015).
- Gonzalez-Aguilera, C. Genome-wide analysis links emerin to neuromuscular junction activity in Caenorhabditis elegans. Genome Biol. 15, R21(2014).
- Wu, F., Yao, J. Spatial compartmentalization at the nuclear periphery characterized by genome-wide mapping. BMC Genomics. 14, 591(2013).
- Filion, G. J. Systematic protein location mapping reveals five principal chromatin types in Drosophila cells. Cell. 143, 212-224 (2010).
- Vogel, M. J. Human heterochromatin proteins form large domains containing KRAB-ZNF genes. Genome Res. 16, 1493-1504 (2006).
- Venkatasubrahmanyam, S., Hwang, W. W., Meneghini, M. D., Tong, A. H., Madhani, H. D. Genome-wide, as opposed to local, antisilencing is mediated redundantly by the euchromatic factors Set1 and H2A.Z. Proc Natl Acad Sci U S A. 104, 16609-16614 (2007).
- Shimbo, T. MBD3 localizes at promoters, gene bodies and enhancers of active genes. PLoS Genet. 9, e1004028(2013).
- Orian, A. Genomic binding by the Drosophila Myc, Max, Mad/Mnt transcription factor network. Genes Dev. 17, 1101-1114 (2003).
- Artegiani, B. Tox: a multifunctional transcription factor and novel regulator of mammalian corticogenesis. EMBO J. , (2014).
- Schuster, E. DamID in C. elegans reveals longevity-associated targets of DAF-16/FoxO. Mol Syst Biol. 6, 399(2010).
- Bianchi-Frias, D. Hairy transcriptional repression targets and cofactor recruitment in Drosophila. PLoS Biol. 2, e178(2004).
- Woolcock, K. J., Gaidatzis, D., Punga, T., Buhler, M. Dicer associates with chromatin to repress genome activity in Schizosaccharomyces pombe. Nat Struct Mol Biol. 18, 94-99 (2011).
- Luo, S. D., Shi, G. W., Baker, B. S. Direct targets of the D. melanogaster DSXF protein and the evolution of sexual development. Development. 138, 2761-2771 (2011).
- Germann, S., Gaudin, V. Mapping in vivo protein-DNA interactions in plants by DamID, a DNA adenine methylation-based method. Methods Mol Biol. 754, 307-321 (2011).
- Zhang, X. The Arabidopsis LHP1 protein colocalizes with histone H3 Lys27 trimethylation. Nat Struct Mol Biol. 14, 869-871 (2007).
- Orian, A. Chromatin profiling, DamID and the emerging landscape of gene expression. Curr Opin Genet Dev. 16, 157-164 (2006).
- Vogel, M. J., Peric-Hupkes, D., van Steensel, B. Detection of in vivo protein-DNA interactions using DamID in mammalian cells. Nat Protoc. 2, 1467-1478 (2007).
- Greil, F., Moorman, C., van Steensel, B. DamID: mapping of in vivo protein-genome interactions using tethered DNA adenine methyltransferase. Methods Enzymol. 410, 342-359 (2006).
- de Wit, E., Greil, F., van Steensel, B. Genome-wide HP1 binding in Drosophila: developmental plasticity and genomic targeting signals. Genome Res. 15, 1265-1273 (2005).
- DamID mammalian vectors. , Available from: http://research.nki.nl/vansteensellab/Mammalian_plasmids.htm (2015).
- Illumina TruSeq adaptors & PCR primers. , Available from: https://ethanomics.wordpress.com/chip-seq-library-construction-using-the-illumina-truseq-adapters/ (2015).
- Optimization of PCR cycles for NGS. , Available from: https://ethanomics.wordpress.com/ngs-pcr-cycle-quantitation-protocol/ (2015).
- Bernstein, B. E. The NIH Roadmap Epigenomics Mapping Consortium. Nat Biotechnol. 28, 1045-1048 (2010).
- Encode Project Consortium. A user's guide to the encyclopedia of DNA elements (ENCODE). PLoS Biol. 9, e1001046(2011).
- Asp, P. Genome-wide remodeling of the epigenetic landscape during myogenic differentiation. Proc Natl Acad Sci U S A. 108, E149-E158 (2011).
- Hoppe, P. S., Coutu, D. L., Schroeder, T. Single-cell technologies sharpen up mammalian stem cell research. Nat Cell Biol. 16, 919-927 (2014).
- Avital, G., Hashimshony, T., Yanai, I. Seeing is believing: new methods for in situ single-cell transcriptomics. Genome Biol. 15, 110(2014).
- Navin, N. E. Cancer genomics: one cell at a time. Genome Biol. 15, 452(2014).
- Saliba, A. E., Westermann, A. J., Gorski, S. A., Vogel, J. Single-cell RNA-seq: advances and future challenges. Nucleic Acids Res. 42, 8845-8860 (2014).
- Nagano, T. Single-cell Hi-C reveals cell-to-cell variability in chromosome structure. Nature. 502, 59-64 (2013).
- Kind, J. Single-cell dynamics of genome-nuclear lamina interactions. Cell. 153, 178-192 (2013).
- Southall, T. D. Cell-type-specific profiling of gene expression and chromatin binding without cell isolation: assaying RNA Pol II occupancy in neural stem cells. Dev Cell. 26, 101-112 (2013).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены