Method Article
3'LIFE Test kullanma Yüksek throughput miRNA Hedefleri tespiti
Bu Makalede
Özet
Luminescent identification of functional elements in 3’ untranslated regions (3’UTRs) (3’LIFE) is a technique to identify functional regulation in 3’UTRs by miRNAs or other regulatory factors. This protocol utilizes high-throughput methodology such as 96-well transfection and luciferase assays to screen hundreds of putative interactions for functional repression.
Özet
3'UTRs fonksiyonel Elementlerin Lüminesans Tanımlama (3'LIFE) sorgulanan 3'UTRs yüzlerce bir dizi içinde spesifik miRNA'ların hedefleri hızlı tanımlanmasını sağlar. Hedef tespiti MiRNA hedefleme işlevsel bir okuma verir translasyon çıkışının ölçülmesi mRNA düzeyinde bağlanmasını saptayan çift lusiferaz deneyi, dayanmaktadır. 3'LIFE genom ekranlar yapma tescilli olmayan tamponlar ve reaktifler ve kamuya açık muhabiri kütüphaneleri kullanır uygulanabilir ve maliyet-etkin. 3'LIFE standart laboratuar ortamında ya yapılmış ya da sıvı taşıma robotlar ve diğer yüksek verimli enstrümantasyon kullanılarak ölçeklendirilebilir. İnsan 96-yuvalı plakalar içinde klonlanmış 3'UTRs ve iki test miRNA'ların bir veri kümesi kullanılarak bir yaklaşımı göstermektedir, salma 7c ve miR-10b. Bu 96 gözlü bir formatta DNA hazırlama, transfeksiyon, hücre kültürü ve lusiferaz tahlilleri yerine ve veri için araçlar temin etmek gösterilmektedirAnaliz. Sonuç olarak 3'LIFE yüksek, tekrarlanabilir hızlı, sistematik ve yüksek güven hedeflerini belirler.
Giriş
Bu yöntemin genel amacı algılamak ve hassas yüksek throughput microRNA (miRNA) hedefleri harita etmektir. MiRNA ~ uzunluğu 22 nükleotid endojen kodlayıcı olmayan RNA'lar bulunmaktadır. Transkripsiyonu ve işleme sonrasında, olgun miRNA'lar bir protein kompleksi içinde dahil edilmiştir kompleksi (RISC) susturulması kaynaklı RNA çağırdı. Her miRNA çeviri baskıya veya mRNA bölünme 1 ya sonuçlanan öncelikle haberci RNA'lar 3 'çevrilmemiş bölgelerine (3'UTRs) (mRNA)' de yer alan unsurları hedef RISC yönlendirir. U baz eşleşmesi sallanmak ve olan dejenere doğada, birden eşleşmeyen baz çifti içeren ve bölgeler şişti: Mirna standart Watson-Crick ve G dayalı hedef siteleri tanır. Birçok miRNA'lar geniş biyolojik rolleri çeşitli bir yelpazede oynamak insanlarda 2,3 için bitkilerden muhafaza edilir. Metazoan'da ise miRNA'lar hücre kaderinin kararları 4, gelişimsel zamanlama 5 dahil olmak üzere birçok biyolojik süreçleri etkileyebilir Ve çoğu zaman dokuya özgü ekspresyonu 6,7 sergiler. MIRNA misexpression aynı zamanda, hedef genlerin işlevine yalnızca temel hücre davranışı üzerinde önemli bir etkiye sahip olabilir olarak anormal gen regülasyonu, neden olabilir. Bu nedenle, miRNA'ların nörodejenerasyon 8,9, diyabet 10 ve kanser de dahil olmak üzere 11, hastalıkların geniş bir bağlantılıdır. Biyoinformatik ve ıslak tezgah yaklaşımları her miRNA yüksek verimli veya genom yaklaşımlar potansiyel etkileşimler bu büyük bir havuz soruşturma için gerekli olduğunu belirten farklı mRNA 12-14 binlerce yüz hedefleme yeteneğine sahip olabileceğini düşündürmektedir.
Hedef genlerin belirlenmesi mekanistik tanımlayan miRNA fonksiyonunun kritik bir bileşenidir, ve bunu yapmak için araştırmacılar büyük çapta hedeflerini ortaya gerekir. Çeşitli yaklaşımlar biyoinformatik tahmin algoritmaları da dahil olmak üzere miRNA hedefler, tanımlamak için geliştirilmiştir, yüksek hacimli dizilemebir mRNA'ları hedeflenen ve raportör deneyleri tabanlı. Bu yaklaşımların her biri doğal güçlü ve zayıf yanları vardır. MiRNA hedefleme sekansı özgüllük tarafından yönlendirilir olduğu göz önüne alındığında, özellikle nükleotid miRNA 2-6, çeşitli algoritmalar birçok organizma genomu boyunca MiRNA hedefleri tahmin etmek için geliştirilmiştir (tohum bölge olarak da adlandırılır). Bu algoritmalar valide miRNA hedeflerinin gözlenen baz eşleşmesi motifleri kullanılarak eğitimli ve sık sık bu tür sıkı tohum eşleştirme, site korunması ve / veya termodinamik stabilite 15 gibi parametreleri kullanan vardır. Bu filtreler sadece yüksek güven hedefleri için yeterli tamamlayıcılık ile olası hedeflerin çok sayıda rafine ederken, onlar son kanıtlar 16-24 yaygın olduğunu düşündürmektedir türe özel ve kanonik olmayan miRNA hedef siteleri, dışarıda olabilir. Ayrıca, bu tahminlerin gibi alternatif poliadenilasyonu olarak miRNA hedef siteleri, dışarıda mRNA işleme hesap mekanizmaları yapmayız25, RNA düzenleme 26, RNA metilasyonu 27 ve kooperatif bağlama. Bu nedenle, yüksek yanlış pozitif ve yanlış negatif oranları birçok algoritmalar 22,24,28 için rapor edilmiştir. Bu algoritmalar sonraki deneysel doğrulama için aday miRNA hedefler belirlemek için yararlı olmakla birlikte, bu yüksek hata oranları sistematik miRNA hedef tespiti için biyoinformatik yaklaşımların etkinliğini sınırlamaktadır.
Sistematik belirli bir miRNA arasındaki etkileşimler ve potansiyel olarak hedeflenmiş 3'UTRs problanması için biz 3'UTRs (3'LIFE) 24 fonksiyon elementleri arasında Lüminesans tanımlanması adı verilen bir yüksek verimli bir deney geliştirilmiştir. Bu tahlil ölçer doğrudan etkileşimleri ve çift lusiferaz haberci sistemini kullanarak bir sorgu miRNA test 3'UTR'sinden translasyon baskı. Bu sistemde, ilgi konusu bir genin 3'UTR ateşböceği lusiferaz (Fluc) raportör okuma çerçevesinin alt klonlanır. muhabir eksilerinitruct HEK293T hücrelerinde bir sorgu miRNA ile kotransfekte edilir. MIRNA hedefleme Test Fluc :: 3'UTR muhabir ve ikinci bir non-spesifik Renilla lusiferaz raportör arasındaki nispi değişimin ölçülmesi ile tespit edilir. Önemli olarak, lusiferaz deneyleri, raportör translasyon çıkış etkileyen fonksiyonel MiRNA / mRNA etkileşimlerini tespit eder. Bu bu 3'UTR tabanlı düzenlemenin bağımsız protein bolca mRNA bozulması ve translasyonel baskıya farklılıkları yanı sıra değişiklikleri atlar, bu tür RT-qPCR ve Western lekeleri olarak miRNA düzenleme, tespit etmek için geleneksel yöntemlere göre önemli bir avantajdır.
Lusiferaz deneyleri, yaygın olarak tüketilebilir reaktifler ile ilişkili yüksek maliyetler ile sınırlıdır, çünkü göreceli basitlik ve hassaslık, ancak, yüksek verimli taramalara kullanımları doğrudan MiRNA hedeflerini doğrulamak için kullanılır, kamu kaynaklarından 3'UTR kütüphanelerinin eksikliği ve yokluğu standart lusiferaz prot arasındaocols, birden veri setleri üzerinde fonksiyonel baskıya karşılaştırılmasında zorluklara yol açmaktadır. 3'LIFE testinin kullanımını kolaylaştırmak için, düzenli olarak güncellenen ve genişletilmiş bir 3'UTR kütüphane oluşturarak, deneysel tasarım, ticari olmayan transfeksiyon 24 ve lusiferaz reaktif 29 kullanımı basitleştirilmesi vurgu ve yoluyla kullanılabilir var kamu plazmid deposu 30.
3'LIFE testinin ölçeklenebilirlik bioinformatically belirlenen genler doğru ekranı eğme olmadan belirli bir miRNA tarafından hedef için büyük bir 3'UTR kütüphanenin tarama sağlar. Kanonik ve tahmin edilen etkileşimleri test etmek için ek olarak, bu sistematik bir yaklaşım, kurallı olmayan ve / veya tür spesifik etkileşimler yoluyla tahrik edilen yeni hedeflerin tanımlanmasına izin verir. Önemli olarak, protein üretimi ile ilgili hedef miRNA etkisi, genel olarak mütevazi bir translasyon baskı 15,31 anlaşılmalıdır/ Sup>, miRNA düzenlemenin bir birincil rolü, protein çıkışı ince ayar gen ifadesinin anormal düzeylerde karşı koruma ve belirli programları 32,33 hücre sağlamlık sağlamak olduğunu düşündürmektedir. 3'LIFE ekranında negatif MiRNA / mRNA etkileşimlerin doğal sayıda kombine lusiferaz deneyi duyarlılığı MiRNA genlerinin bir çok sayıda hedef ince etkilerinin belirlenmesine ve gen ağlarının birden çok bileşenden tanınmasını sağlar Belirli bir miRNA 24 düzenlenir.
Burada 3'LIFE protokol açıklar ve 275 insan 3'UTRs (Şekil 1) 'in bir paneline karşı iyi karakterize edilmiş iki miRNA'lar, miR-10b ve izin-7c taranmasıyla, fizibilite olduğunu göstermektedir.
Protokol
1. Hücre Kültürü (24-48 saat transfeksiyondan önce),
- Transfeksiyon öncesi tohuma 24-48 saat 96 oyuklu plakalar sayısına göre HEK293T hücrelerin yeterli bir miktarı ile transfekte edilir.
NOT: tutarlı transfeksiyonlar için, yeterli bir yoğunlukta plaka hücreleri hızlı bir şekilde bölünme lehine, ancak transfeksiyon zamanı en fazla% 70-90 konfluent olarak vermeye. - Her 96 oyuklu plaka (rezervuar ve çok kanallı pipet kullanımı için hesap oyuk başına 75,000 hücre, plaka başına 120 kuyu) 9 x 10 6 hücre gerektirir. HEK293 hücreleri (tipik olarak ~ 20 saat) iki katına çıkma süresi hesaplayın ve hücreler uygun sayıda transfeksiyon zamanı en az 9 x 10 6 hücre elde etmek üzere tohum. Yeni bir levha reseed kalan hücrelerin ~% 10, ~% 90 konfluansa kadar yetiştirilen bir 145 mm'lik dairesel kültürü plakası, tipik olarak 3 96 oyuklu Transfeksiyonlar için yeterlidir.
Transfeksiyondan 2. Hazırlık Önceki
_content "> Not: bu reaktiflerin hazırlanması zaman alıcı olabilir çünkü aşama 2.0-2.2 tamponların ve plazmid DNA hazırlanması, transfeksiyondan önce günde yapılmalıdır.- 3'LIFE testi ile uyumlu İnsan 3'UTR klonları kamu plazmid deposu 30 edinilebilir. Elle veya sıvı taşıma robotlar ile DNA plazmid arındırın. Bir transfeksiyon dereceli alkali lizis mini hazırlık 96-kuyu kiti kullanın ve üreticinin talimatlarına uyun. Kuyuya ~ 100 ng / ml başına saflaştırılmış vektörler süspanse.
NOT: plazmid konsantrasyonu aşağıda 40 ng / ul düşerse yetersiz lusiferaz sinyali neden olacaktır. - PLIFE-miRNA Şekil 1B 24 boru hattını kullanarak 30 veya klon vektörleri elde edilir. Her miRNA ve boş kontrol plasmidler için 500 ng / ul'lik bir konsantrasyonda vektörleri yeniden süspanse edin.
- Nucleofection tampon koşulları hassasiyeti nedeniyle, sağlamak(hücreler ve plasmidler de dahil olmak üzere), transfekte edilmiş malzemelerin toplam hacmi 96 çukurlu transfeksiyon plakanın her bir toplam sıvı% 10 geçmez. Bunu elde etmek için, en az 500 ng / ul'lik bir konsantrasyonda pLIFE-MiRNA plazmid stok konsantre edilir.
- 10x ateşböceği lusiferaz tampon reaktifleri (Tablo 1) ve 6 aya kadar saklanabilir 1x Renilla lusiferaz tampon reaktifleri (Tablo 2), hazırlayın.
Not: ateşböceği lusiferaz tampon çözeltisi içindeki DTT, tek kullanımlık parçalar halinde -20 ° C'de çözelti içinde saklanabilir olması gerekir.
3. Transfeksiyonu için hemen öncesinde ardından Ürünleri hazırlayın
- PBS ihtiva eden transfeksiyon tamponu,% 1.5 HEPES, pH 7.0 hazırlanır. Bu Transfeksiyon etkinliğindeki fark düşüşler olmadan 4 ° C'de en fazla 1 aya kadar saklanabilir, ancak, bu taze hazırlayın. Tampon ve plazmid DNA hacimleri formüle ederken, su için her 96 oyuklu plaka için 120 reaksiyonlar kabulfficiently pipetle sıvı rezervuarları ve çok kanallı pipet kullanarak kayıp hacim hataları oluşturmaktadır. Kısım kuyu transfeksiyon tamponu başına 18 ul (/ 96-plaka 120 kuyu = plaka başına 2.16 ml) ve bir kenara koyun.
NOT: hücre elektroporasyon cihazı hücrelerini transfekte etmek için kullanılan tampon koşulları son derece duyarlıdır. Hazırlama tamponlar ekipman tutarlı performans sağlayacak Doğruluk. Özellikle bu tampon mikrosantrifüj tüpleri, 96-yuvalı plakalar, rezervuar ve elektrot plakaları içinde ortaya kalan zamanı en aza düşürülmesiyle, tamponlar buharlaşmasını önlemek için tahlil yapmak azami dikkat edilmelidir. - Rezerv Aşağıdaki denetimler için dört kuyu, hiçbir pLIFE-3'UTR (lusiferaz tahlil arka ölçmek için), pLIFE-SV40 3'UTR (negatif hedef kontrolü), miRNA # 1 için pozitif kontrol, miRNA # için pozitif kontrol 2.
NOT: Bu vektörler 30 kamuya mevcuttur. Alternatif olarak, daha önce onaylanmış bir hedefBir pozitif kontrol olarak da kullanılabilir. - Sıcak ortam, 37 ° C tripsin (% 0.25).
- DMEM,% 10 FBS,% 1 Pen / Strep ile takviye edilmiş 96 oyuklu hücre kültürü plakasının her bir oyuğuna, 200 ul ve transfeksiyonu takiben kullanılmak için bir 37 ° C kuluçka makinesi içine yerleştirilmiştir.
- Destek yazılımları tarafından takip tüm cep elektroporasyon aygıtları açın. HEK293T hücreleri ve PBS / HEPES tamponu için Darbe kod FF120 kullanın.
Plazmid DNA ve hücre karışımı 4. hazırlanması
Not: Aşağıdaki protokol, iki miRNA'ların (miRNA- # 1 ve miRNA- # 2) ile birlikte, bir ekran için bir deneyde, üç, 96 gözlü levhalar transfekte varsayar. Her bir plaka pLIFE-3'UTR plasmidlerin, aynı 96-yuvalı plaka karşılık gelir, ve pLIFE-miRNA boş pLIFE-miRNA- 1. veya pLIFE-miRNA- # 2 ile üç kez tedavi edilebilir.
- Her miRNA için pLIFE-miRNA + transfeksiyon tamponu 3 stokları hazırlayın. Bu stok her birinin toplam hacminin% 50'si (10 ul) ayarlanabilmelidiriyi, 120 kuyular ile çarpılır. Böylece, her bir hisse senedi 1.08 ml tampon + 120 ul plazmid DNA (pLIFE-miRNA) içermelidir.
- Medya elüsyon PBS ile hafifçe yıkanır ve 37 ° C'de 5 dakika boyunca ~ 5 mi,% 0.25 tripsin ile muamele etmek suretiyle 145 mm kültür hücreleri plakadan çıkarın. 5 dakika boyunca 300 xg'de eşit medya hacmi ve pelet hücreleri ile tripsin nötralize eder.
- 5-10 ml medya (hücre yoğunluğu ve hücre sayacın doğru aralığına bağlı olarak) ~ tripsini / medya ve tekrar süspansiyon pelet çıkarın.
- Bir hücre sayacını kullanarak hücreleri saymak. % 95 canlı ve makinenin doğru aralığında> hücreler olduğundan emin olun.
NOT: tutarlı hücre sayısı, son derece yüksek hücre konsantrasyonlarında (> 6.0x 10 6 / ml) bir sonucu olabilir. Hücre oranı ve / veya transfeksiyon verimliliği azalan: transfecting çok hücre ölçüde plazmid azaltarak hedef miRNA verimliliğini azaltabilir. - Kısım üç tüp her biri 9 x 10 6 hücre, hücrelere karşılık gelen rBir 96-yuvalı plaka transfeksiyonu için equired. 3 dakika boyunca 300 xg'de hücreleri Spin.
- Ortamı çıkarın. Aşırı medya transfeksiyon etkinliğini etkileyebilir olarak pelet minimal rahatsızlık ile mümkün olduğunca çok ortamı çıkarmak için emin olun.
- 1.2 ml transfeksiyon tamponu / miRNA plazmid karışımı ve bir kenara yeniden süspanse hücreleri.
- Aşağıdaki transfeksiyon tamponunda pLIFE-3'UTR plazmid detay tabanda yineleyin. Bu 96 kuyucuğu oluşur gibi, her zaman plakaları kaplayarak tampon buharlaşmasını önlemek için özen gösterin.
- Bir çok kanallı pipet kullanarak, 96 PCR plakanın her oyuğuna transfeksiyon tamponu ul 32,4 hareket ([transfeksiyon başına] 9 ul * 3: [plakaları] * 1.2 [pipet hata için hesap]).
- Her kuyuya 3.6 ul mini prepped pLIFE-3'UTR plazmid (± 100 ng / | il) ilave edin ve iyice karıştırın.
- Pipet 96 oyuklu transfeksiyon levhasının ve kapağın her oyuğuna Bu karışımın 10 ul.
5. Transfeksiyon
- Rezervuar içine ilk hücre / tampon / pLIFE-miRNA plazmid karışımı 1.2 ml taşıyın. Iyice karıştırın.
- Önce 10 ul transfeksiyon tampon / pLIFE-3'UTR içeren birinci 96 oyuklu transfeksiyon plaka içine bu karışımın 10 ul ekle. Yukarı pipetleme ve birkaç kez aşağı iyice karıştırın.
NOT: tamponunda hücrelerin Eşit süspansiyon bile emin ve küvet ile elektrik akımının geçişi kapsamlı ve transfeksiyon verimliliği maksimize edecek. - Hücre elektroporasyon cihazda 96-iyi transfeksiyon plaka koyun ve transfeksiyon başlatmak.
- Transfeksiyon tamamlandıktan sonra, 96-çukurlu transfeksiyon plakanın her oyuğuna 96 oyuklu bir kültür plakasına gelen önceden ısıtılmış ortam 100 ul ekleyin ve iyice karıştırın. 96-çukurlu kültür plakasına her bir göze 100 ul taşıyın.
- Hücreler w yanlarındaki topaklaşma eğilimi gibi, iyi merkezinde dikey olarak yerleştirilmiş pipetle kültür plakasına hücreleri karıştırınell düzgün karışık sürece.
- Geri kalan iki tabak 5,1-5,5 tekrarlayın.
- Temizleme 96-kuyu transfeksiyon plaka
- 96 oyuklu plakalar, transfeksiyon deneyler arasında, nükleik asitlerin üzerinde hiçbir yürütülmesini sağlamak üzere,% 70 EtOH ile yıkanarak geri dönüştürülebilir. Tamamen elektrot şeritler aşırı EtOH silerek (alt taraf) ve kültürü kaputu tamamen kuru transfeksiyon plakaları izin vererek, ardından her iyi doldurmak için bir sprey şişesi kullanarak iki% 70 EtOH yıkar gerçekleştirin.
NOT: bir plazmid DNA ile% 70 EtOH ile tek bir yıkama ve bir ikinci transfeksiyon, ardından HEK293T hücrelere her biri, plazmid pmaxGFP 2 ug ile 12 kuyu transfekte edilmesiyle carry-over DNA kontaminasyonu için test edilmiştir. Bu, son derece yüksek bir plazmid konsantrasyonu, son derece parlak muhabir ve tek bir yıkama ile, 12 kopya transfeksiyonlar herhangi gözlenebilir bir floresan olmasıdır.
- 96 oyuklu plakalar, transfeksiyon deneyler arasında, nükleik asitlerin üzerinde hiçbir yürütülmesini sağlamak üzere,% 70 EtOH ile yıkanarak geri dönüştürülebilir. Tamamen elektrot şeritler aşırı EtOH silerek (alt taraf) ve kültürü kaputu tamamen kuru transfeksiyon plakaları izin vererek, ardından her iyi doldurmak için bir sprey şişesi kullanarak iki% 70 EtOH yıkar gerçekleştirin.
- Çift lucif ardından 37 ° C'de 48-72 saat süre ile kültür hücreleri,tahlil silebilirsiniz.
6. Hücre Lusiferaz Assay için Lizat hazırlanması
- 4 kısım su, bir rezervuar pasif lizis tamponu 5x 1 parça ile lizis tamponu ile seyreltilir. 26 ul / kuyu, ~ 20 ekstra hacimleri ekleyerek rezervuar kaybı hesaba hesaplayın.
NOT: Tampon -20 ° C'de saklanır ve pipetleme doğruluğu artıracak oda sıcaklığını yaklaşım 5x tampon izin böylece önce, son derece viskoz olabilir. - Floresan mikroskopi kullanılarak transfeksiyon etkinliği için de her bir analiz edin. Verimli transfect vermedi kuyularda tutarsızlıkları (>% 90 transfeksiyon verim) Not veya kalabalık ya da medya bitkinlik göstergesidir RFP düşük seviyelerde ifade edilir. Analizden Bu kuyu çıkarın.
- Tamamen hücreler ayırmak neden olan çok hızlı Zehir için dikkatli olmak, hücrelerden ortamı çıkarın.
NOT: Kalan medya experime genelinde değerleri lizat ve neden dalgalanmaları sulandırmakNTS. - 20-30 dk ~ düşük / orta hızda bir plaka çalkalayıcı / rocker her iyi ve yere lizis tamponu 26 ul ekleyin. Lusiferaz tamponlar hazırlamak lüminometre (ler) yıkayın ve prime ve opak ölçüm plakalarına lizat aktarmak için bu kez kullanın.
7. Çift Lusiferaz Deneyi
NOT: Birden plakaları tek lüminometre üzerinde sırayla ölçülen yapılıyorsa, hemen her plaka kullanmadan önce pH ayarlaması ardından bu reaktif ekleyerek, ATP ve yüzeylerde hariç her şeyi ile tampon ana karışımları oluşturun. ATP ve yüzeyler zamanla düşebilir; Bu reaktifler tampon içinde olan süreyi tutarlılık birden plakalar arasında tutarlılık artıracaktır.
- 1x lusiferaz tamponlar hazırlanması (Tablo 1):
- Yüzeyler ışığa duyarlı olabileceğinden, alüminyum folyo ile ateşböceği ve Renilla tamponlar içeren iki tüpleri (genellikle 15/50 ml santrifüj tüpleri) sarın.
- 1x Firefly lusiferaz tampon hazırlayın. Nihai 1x konsantrasyonu 5 ml H 2 0, EGTA son ekleme, her beş 10x ateşböceği lusiferaz reaktif 1 ml ekleyin.
- Ateşböceği tampon 1x 10 ml, 0.025 gr ATP ilave edin. Birkaç kez baş aşağı çevirerek kanştınn. Her zaman buz üzerinde ATP tutun. ATP düşer, bu nedenle birden fazla plaka sekans ölçülmesi halinde, tampon her bir ek plaka için bu adımda taze başlangıç yapılmalıdır.
- Tampon pH dayalı sarımsı renk değiştirmek gerekir 100 böceği luciferase (alt tabaka) (Tablo 1) 100 ul ekleyin.
- 1x Renilla lusiferaz tamponu sulandırma: 96 oyuklu bir plaka, kısım 1, 10 ml başına "Renilla tampon".
- BSA, 100 ul (44 mg / ml stok) ilave edilir.
- Birden fazla tabak, 10 ml'lik içine ayrı ana karışımı tarama edin.
- Tampon koelenterazin 100 ul ekle (daha önce bölünüp 100x konsantre C'de saklandı.)
- NaOH ve HCI kullanılarak 5.0 1x Renilla tamponu ardından, ateşböceği 8.0 tampon 1x pH ayarlayın.
NOT: Her tampon aktivitesi, ve ateş böceği lusiferaz aktivitesini bastırmak için Renilla tampon kabiliyeti pH değerine oldukça bağlıdır. Tutarlı sonuçlar için bu adımda son derece doğru. - Luminometre astarlanması için yerleştirmek için 10.5 ml (1, 96 oyuklu plakaya tekabül eden) her bir tampon hacmi getirin.
- Opak beyaz plakalara lizat transfer Bu noktada hücreler, 20 dakika için ~ liziz tamponu içinde olmalıdır. Her bir kuyunun çok kanallı pipet kullanarak 25 ul alın, hücre kümeleri break up ve homojenleştirin aşağı iyice yukarı pipetlemeyin emin ve olmayacak.
- Lüminometre hazırlayın. Lüminometre açın ve "iki enjeksiyonları ile DLR" adlı DLR klasöründe protokolü seçin. Diğer formatlar veri analizi boru hattı (aşağıda) ile uyumlu değildir.
- T olmak kuyuları seçinested (bütün kuyular varsayılan).
- (Açıklama için 29 bakınız) 10 sn ölçüm süresi, 5 saniye ayarı 'ölçümden önce Delay' uzatın.
- Kılcal yıkama adımları: Su 3x, EtOH 3x, su 3x, kuru 3x. Arka tampon tüpleri içine bir kez atık haline Başbakan tamponlar ve daha sonra başbakan ikinci kez karıştırma sağlamak. Sağ kılcal Renilla ardından sol kılcal, ilk ve Başbakan ateşböceği tampon enjekte edilir.
- Lusiferaz deneyi başlatın. Her plaka okuma ~ 48 dk almalıdır. Tamamlanmasından sonra ilk dosyayı kaydedin ve sonra yıkama adımları tekrarlayın ve lüminometre kapatın.
NOT: luminometre programı çökmesine nerede Çoklu plakalar okuma ve aynı saklanan veriler excel dosyası olabilir, sorunları birden plaka ile karşılaştı olabilir ancak okur. Ölçümlerin arasındaki tüm verileri kaydetmek ve kaydetme mümkün önce program çökerse ekran görüntüleri almak için emin olun.- Eski tamponlar değiştirinYeni ile yeni plaka başlamadan önce en az iki kez yeni tamponlar ile Başbakan emin olmak.
8. Veri Analizi
- Edinilebilir - ve "multiplate analizi 3'LIFE" - excel tabloları "tek plaka analizi 3'LIFE" yararlanın www.mangonelab.com . Elektronik tablo - "tek plaka analizi 3'LIFE" içine negatif durum, miRNA 1. ve miRNA 2. tekabül yerlere luminometre çıktı dosyasından ateş böceği ve Renilla lusiferaz ölçümleri için ham veri kopyalayın.
- Elektronik tablo otomatik ateşböceği / Renilla oranını hesaplamak ve uygun negatif kontrol her miRNA normalleştirmek ve her plaka üzerinde baskı değerleri normale olacaktır.
NOT: Bu e-tablo otomatik olarak anlamlı bastırılmış kuyu vurgulayın düşük lusiferaz sinyali ile kuyu tespit ve repressi tedbirlerini sağlayacaktüm plaka üzerinde üzerinde. İstatistiksel analiz detaylı açıklama için 24 Bkz.- Test edilen her miRNA için 1. çoğaltmak tekabül eden konumda - "multiplate analizi 3'LIFE" e-tabloda karşılık gelen hücrelere, "Normalize RI Endeksi" kutusundan değerleri kopyalayın. Farklı günlerde yapılan tüm biyolojik çoğaltır için tekrarlayın.
- Arzu edildiği takdirde, sırası ile, sütun B ve D gen isimleri ve hedef tahmini durumunu yerleştirin.
- Elektronik tablo otomatik olarak tüm plaka ortalamasını hesaplamak için izin verin. Bir ısı haritası olarak 96 sıra formatta (Şekil 2) verileri dikkate alınmalıdır. dosyası da liste biçiminde veri (Şekil 2) düzenler.
NOT: baskı endeksi, farazi miRNA hedeflerini tanımlamak için kullanılan bir ölçüdür. Farklı sertlikteki parametreler tek tek tercihlerine bağlı olarak, kullanılabilir. Ortalama olarak biz 0 baskı endeksi altında muhtemel hit düşünün.8 ve t testi (p-değeri <0.05) istatistiksel olarak anlamlı. Şekilde gösterildiği gibi, bu kriterlere göre 3 potansiyel hedefler kırmızı vurgulanır.
- Elektronik tablo otomatik olarak tüm plaka ortalamasını hesaplamak için izin verin. Bir ısı haritası olarak 96 sıra formatta (Şekil 2) verileri dikkate alınmalıdır. dosyası da liste biçiminde veri (Şekil 2) düzenler.
Sonuçlar
luminometre çıktı dosyası hem ateş böceği ve Renilla lusiferaz proteinleri ham ölçümlerini içerir. Ve - Mangone laboratuvar web sitesinden (edinilebilir "3'LIFE multiplate analizi" tabloları - Bu ham biçimi "tek plaka analizi 3'LIFE" ile uyumludur www.mangonelab.com ). Tek plaka analizi tablo otomatik ateşböceği / Renilla oranını hesaplar uygun negatif kontrole her miRNA normalleştirir ve her plaka üzerinde baskı değerlerini normalleştirir. Bu e-tablo otomatik olarak düşük Renilla lusiferaz sinyali ile kuyu tanımlayan negatif kontrol ile karşılaştırıldığında baskıyı sergileyen kuyuları vurgular ve tüm plaka boyunca baskı tedbirleri (Şekil 2) sağlar. İstatistiksel analiz detaylı açıklama için 24 Bkz.
Birden çoğaltır analiz edilebilird "multiplate Analizi" tablo kullanarak. Her tekrarlı yan yana karşılaştırılır ve verilerin istatistiksel ölçümler otomatik olarak (Şekil 3) hesaplanmaktadır. "Normalize Baskı" sütunlu çoğaltır karşılaştıran yanı sıra, kullanıcı "miRNA # 1 / miRNA # 2" sütunları altında iki miRNA'lar arasındaki baskıyı karşılaştırabilirsiniz. Bu önlem, her tekrarında her miRNA için baskı endeksi böler. Bu önlem lusiferaz testte hatalı değerleri gösterebilir (A9 satır, Şekil 3, bkz negatif kontrol ile anormal derecede yüksek veya düşük okumalar, örneğin Rep # 3), ve baskı endeksi önemli baskıyı göstermeyebilir kuyular, ama bunu MiRNA'lar arasında önemli farklılıklar gösterirler. Bu önlem, bir miRNA hedef göstermek için doğrudan kullanılabilir olmayabilir iken, sadece miRNA yeniden yönlendirmek atfedilen olmayan verilerde aykırı, sorunlu kuyuları ya da desenleri tanımlamak için yararlıdırDüzenlemenin.
Baskı Endeksi (RI) daha düşük değerler yüksek bağıl baskıya karşılık gelen, varsayılan bir miRNA hedef çağırmak için kullanılır. varsayılan hedeflere çağırmak için eşik araştırmacı tarafından gerekli sertlik düzeyine bağlıdır, ancak istatistiksel olarak anlamlı p-değerleri (p <0.05) göstermek 3'UTRs ile RI birleştirerek yüksek güven hedefler (Şekil 2 Satırlar B8 bkz gösterir ve B 12).

Şekil 1: 3'LIFE Deneyi (A). 3'LIFE tahlilinde kullanılan ağ uyumlu vektörler. En: Renilla lusiferaz geni (RLuc) kontrol olarak spesifik olmayan SV40 pA 3'UTR kaynaşık ise lusiferaz geni (Fluc), test 3'UTR kaynaştırılır. Alt: RFP- miRNA intron vektör --miRNA öncesi prob artı ~ 400genomik lokus içinde nükleotidleri (endojen MiRNA işleme özetlemek için), DSRed2 florokrom ile ko-ifadesini sağlamak için bir intron içinde klonlanır. Her iki vektör, kamuya açık olan (Seiler ve ark., 2013). (B) 3'LIFE Testi Akış şeması. (C) 3'LIFE Boru: veya MiRNA vektörleri olarak deneme 3'UTR içeren çift lusiferaz vektörü ko-transfekte edilmiş 96 oyuklu plakalar içinde HEK293 hücrelerine bulunmaktadır. miRNA ve iyi niyetli bir 3'UTR hedef arasındaki etkileşim (deneysel plaka turuncu nokta ile örneklenen) belirli kuyularda göreli ışıldama düşürecektir.

Şekil 2: 3'LIFE testi ile üretilen verilerin örnek. MiRNA dört nüsha olarak test edilmiştir Her prob (1-4 çoğaltır). Renkler kırmızı, baskı düzeylerini temsilGüçlü miRNA / 3'UTR etkileşim gösteren renkler. Tüm çoğaltır sarı okla altında özet plakasında gösterilen yüksek kaliteli varsayılan hedeflere üretmek için ortalaması alınır. Beyaz kutu, denetimleri temsil eden düşük transfeksiyon verimlilik ile transfections veya kuyuları başarısız oldu.
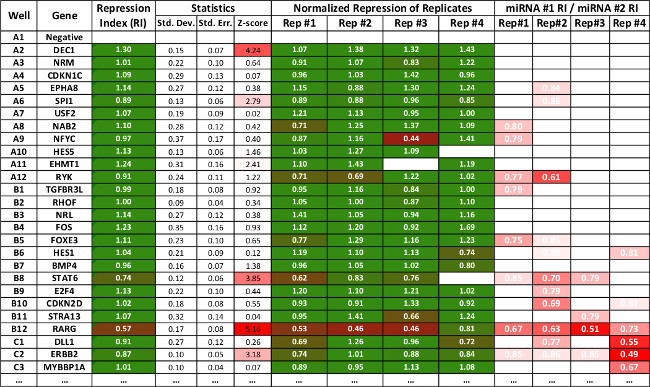
Şekil 3:. 3'LIFE tablo kullanarak üretilen etkileşimlerin bir alt kümesinin özet verileri temsil eden Masa baskı değerleri Şekil 2'de yazılım gibi olan her etkileşim için standart sapma, standart hata ve z-skoru hesaplar. Istatistiksel olarak anlamlı etkileşimler kırmızı olarak işaretlenmiştir. Son dört sıra diğer bir miRNA göreli baskı gösterir ve iki farklı miRNA'ların arasındaki baskı karşılaştırma sekonder göstergesi olarak kullanılır. Elektronik tablo indirilebilir www.mangonelab.com
| Ateş böceği lusiferaz tamponu reaktifler | Nihai konsantrasyon (1x) |
| Glisilglisin | 25 mM |
| K PO 4 x (pH 7.8) | 15 mM |
| MgSO 4 | 15 mM |
| DTT (4º mağaza) | 1 mM |
| EGTA | 4 mM |
| ATP * | 2 mM |
| Beetle luciferin * | 250 uM |
Tablo 1: Hazır ateşböceği lusiferaz reaktifler: glisilglisin 10x stok çözeltileri, K PO 4, MgSO 4 x, DTT ve EGTA ayrı ayrı hazırlanmıştır ve sulandırma tamponunda önceden saklanabilir. 100x Beetle Luciferase (ateşböceği lusiferaz substratı) mağaza olabilir7,134 mi, H 2 0 (25 mM) 'de 50 mg lusiferin çözülmesiyle, d. -80 ºC çözünmüş Beetle tüpler içine luciferase ve mağaza kısım 105 ul / plaka. Promega teknik destek Başına, bu> 6 ay boyunca stabil olmalı, ama ışığa duyarlı olabilir. NOT: EGTA nötr pH çözelti girmeyeceğim. Tamamen eriyene kadar yavaşça EGTA NaOH ekleyin. * Nihai tampona eklenen Reaktifler hemen önce lusiferaz tahlil
| Renilla lusiferaz tamponu reaktifler | Nihai konsantrasyon (1x) |
| NaCl | 1.1 M |
| Na2EDTA | 2.2 mM |
| KH 2 PO 4 | .22 M |
| NaN3 | 1.3 mM |
| BSA * | 0,44 mg / ml |
| Coelenterazine * | 2.5 & #956 M |
Tablo 2: BSA koelenterazin hariç Hazır Renilla lusiferaz tamponu reaktifler tüm reaktifler 1x konsantrasyonda karıştırılır ve oda sıcaklığında saklanabilir. Koelenterazin asitleştirilmiş metanol içinde çözündürüldü ve plaka başına aliquoted olabilir. 5 mM (<3 pH) nihai konsantrasyona HCI eklenerek metanol asitleştirin. 2.36 ml asitleştirilmiş metanol (250 uM) kısım 105 ul / plaka içinde 250 ug coelenterazine eritin. karışım en az 6 ay süreyle stabildir ama hassas ışık olabilir. * Reaktifler lusiferaz tahlil hemen önce tampon eklendi.
Tartışmalar
3'LIFE deneyi yüksek throughput 3'UTRs fonksiyonel MiRNA hedefleri tanımlar. Bu deney, kendi ilgi miRNA için farazi hedef çok sayıda tespit etmek isteyen araştırmacılar için yararlıdır. 3'LIFE tahlil miRNA hedefleme fonksiyonel bir ölçü ve güvenle hitap edebilecek tek bir miRNA karşı 3'UTR'sinden :: tek muhabir ikili test sağlar ki 3'UTR tahrik düzenlenmesi için sorgulamak için güçlü bir yaklaşımdır Bireysel genlerin hedef durumu. Bu yaklaşımı doğrulamak için, 275 3'UTRs oluşan bir panel ekranlı ve iki miRNA'lar karşı, let-7c ve miR-10b ve bu kütüphanede 10 önceden doğrulanmış hedef genleri dahil. Bu on genlerin sekiz baskıyı 24. sergiledi. Biz de unvalidated bioinformatically tahmin hedeflerin belirgin zenginleştirme ve bizim üst isabet arasında kanonik tohum elementleri içeren öngörülemeyen 3'UTRs gözlenen, sugO 3'LIFE gesting iyi niyetli miRNA hedefler belirleme yeteneğine sahiptir.
Yüksek verimli ekranlar duyarlılık önemli bir gösterge yanlış pozitif ve yanlış negatif oranları. Bu testin yalancı pozitiflik oranı hit doğrulamak için ek alternatif yaklaşımlar kullanılarak değerlendirilmesi gereken iken, on pozitif kontrollerin sekiz% 20 yanlış negatif oranı düşündüren, bizim proof-of-prensibi ekran sergilenen baskıya dahil. Bununla birlikte, birçok teknik farklı hücresel bağlamlarda MiRNA hedeflerini tanımlamak için kullanılır, ve trans-etkili faktör tarafından 3'UTR işlenmesi ve ayarı oldukça dokuya özgü olduğu bilinmektedir. Örneğin, 3'UTRs çoğunluğu, olgun mRNA 3'UTR uzunluğunu kontrol birden poliadenilasyon sitesi içerir. Birçok durumda proksimal poliadenilasyon sitelerinin kullanımı doku özgüdür ve miRNA hedef siteleri hariç tutabilir. Ayrıca, kooperatif miRNA hedefleme, rekabet wiinci RNA bağlayıcı proteinler ve mRNA sekonder yapısı, tüm etki 3'LIFE kabiliyeti belirli dokularda MiRNA hedefleri tespit etmek için olabilir. Bu nedenle, 3'LIFE tahlili ile hedeflerin tespiti mutlak hata oranlarının değerlendirilmesi zorlaştıran, tahlil yapıldığı hücresel bağlamda göre değişebilir. Bu protokol, HEK293T hücreleri için optimize edilmiş, ancak bir araştırmacı, belirli bir biyolojik bağlamda tahlili gerçekleştirmek etmek istemesi halinde bir alternatif hücre çizgileri de kullanılabilir. Bununla birlikte, her bir hücre hattı ile transfeksiyon verimliliği ve hücre canlılığının optimizasyonu çok tampon koşulları, darbe kodlarını ve hücre sayısını kullanarak optimize edilmesi gerekir. Optimizasyon şemasının bir örneği, Wolter et al. 24 bulunabilir.
Bu protokol, 96 oyuklu bir formatta optimize edilmiş ve belli bir yüksek verimli enstrümantasyon kullanımını belirtir edilmiştir. Durumunda kurum 96 için gerekli donanıma sahip olmadığınıeh Nucleofection alternatif transfeksiyon reaktifleri sürece transfeksiyon verimi, yüksek kalır, 3'LIFE deneyi gerçekleştirmek için kullanılabilir. Buna ek olarak, lusiferaz deney 3'LIFE tahlilinde en zaman alıcı bir yönüdür. Bu nedenle, birden fazla luminometreyle kullanımı, yüksek verimli taramalar için tavsiye edilir.
Açıklamalar
This work is supported by funds from the College of Liberal Arts and Science and the Biodesign Institute at Arizona State University and NIH Exploratory/Developmental Research Grant 1R21CA179144-01A1.
Teşekkürler
We thank Stephen Blazie, Karen Anderson, Josh LaBaer for advice and discussion. Karen Anderson, John Chaput, and Josh LaBaer for sharing reagents and instrumentation, Michael Gaskin and Andrea Throop for technical advise and protocols. Justin Wolter is a Maher scholar and thanks the Maher family for their generous support.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| glycylglycine | Sigma | G1127-25G | |
| Kx PO4 | Sigma | P2222 | |
| EGTA | Sigma | E3889 | |
| ATP | Sigma | FLAAS | |
| DTT | Sigma | D0632 | |
| MgSO4 | Sigma | M7506 | |
| CoA | Sigma | C4282 | |
| luciferin | Sigma | L9504 | |
| NaCl | Sigma | S7653 | |
| Na2EDTA | Sigma | E0399 | |
| K H2 P O4 | Sigma | 1551139 | |
| BSA | Sigma | A2153 | |
| NaN3 | Sigma | S2002 | |
| Coelenterazine | Sigma | C3230 | |
| PBS/HEPES | Corning | 21-040-CV | |
| DMEM | Sigma | D5546 | |
| FBS | Sigma | F2442 | |
| Pennicilin/Streptomicin | Sigma | P4333 | |
| Trypsin | T2600000 | ||
| Consumables | |||
| MaxiPrep Kit | Promega | A2392 | |
| 96-well miniprep plate | Pall | 8032 | |
| 96-well shuttle plates | Lonza | V4SP-2096 | |
| 5x Lysis Buffer | Promega | E1941 | |
| Instruments | |||
| 96-well GloMax Plate Reader | Promega | E9032 | |
| Biomech FX Liquid Handler Robot | Beckmann | A31842 | |
| 4D-Nucleofector Core Unit | Lonza | AAF-1001 | |
| 96-well Shuttle System | Lonza | AAM-1001 | |
| Cell Counter Countess | Invitrogen | C10227 |
Referanslar
- Bartel, D. P. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell. 116 (2), 281-297 (2004).
- Pasquinelli, A. E., et al. Conservation of the sequence and temporal expression of let-7 heterochronic regulatory RNA. Nature. 408 (6808), 86-89 (2000).
- Reinhart, B. J., Weinstein, E. G., Rhoades, M. W., Bartel, B., Bartel, D. P. MicroRNAs in plants. Genes Dev. 16 (13), 1616-1626 (2002).
- Ivey, K. N., et al. MicroRNA regulation of cell lineages in mouse and human embryonic stem cells. Cell Stem Cell. 2 (3), 219-229 (2008).
- Lee, R. C., Feinbaum, R. L., Ambros, V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 75 (5), 843-854 (1993).
- Wienholds, E., et al. MicroRNA expression in zebrafish embryonic development. Science. 309 (5732), 310-311 (2005).
- Darnell, D. K., et al. MicroRNA expression during chick embryo development. Dev Dyn. 235 (11), 3156-3165 (2006).
- Jin, P., Alisch, R. S., Warren, S. T. RNA and microRNAs in fragile X mental retardation. Nat Cell Biol. 6 (11), 1048-1053 (2004).
- Kim, J., et al. A MicroRNA feedback circuit in midbrain dopamine neurons. Science. 317 (5842), 1220-1224 (2007).
- Poy, M. N., et al. A pancreatic islet-specific microRNA regulates insulin secretion. Nature. 432 (7014), 226-230 (2004).
- Calin, G. A., Croce, C. M. MicroRNA signatures in human cancers. Nature Reviews Cancer. 6 (11), 857-866 (2006).
- Friedman, R. C., Farh, K. K., Burge, C. B., Bartel, D. P. Most mammalian mRNAs are conserved targets of microRNAs. Genome Res. 19 (1), 92-105 (2009).
- Paraskevopoulou, M. D., et al. DIANA-microT web server v5.0: service integration into miRNA functional analysis workflows. Nucleic Acids Res. 41 (Web Server issue), W169-W173 (2013).
- Krek, A., et al. Combinatorial microRNA target predictions. Nat Genet. 37 (5), 495-500 (2005).
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136 (2), 215-233 (2009).
- Vella, M. C., Choi, E. Y., Lin, S. Y., Reinert, K., Slack, F. J. The C. elegans microRNA let-7 binds to imperfect let-7 complementary sites from the lin-41 3'UTR. Genes Dev. 18 (2), 132-137 (2004).
- Lal, A., et al. miR-24 Inhibits cell proliferation by targeting E2F2, MYC, and other cell-cycle genes via binding to 'seedless' 3'UTR microRNA recognition elements. Molecular Cell. 35 (5), 610-625 (2009).
- Cevec, M., Thibaudeau, C., Plavec, J. N. M. R. NMR structure of the let-7 miRNA interacting with the site LCS1 of lin-41 mRNA from Caenorhabditis elegans. Nucleic Acids Res. 38 (21), 7814-7821 (1093).
- Shin, C., et al. Expanding the microRNA targeting code: functional sites with centered pairing. Molecular Cell. 38 (6), 789-802 (2010).
- Azzouzi, I., et al. MicroRNA-96 directly inhibits gamma-globin expression in human erythropoiesis. PLoS One. 6 (7), e22838(2011).
- Chen, J., et al. miR-193b Regulates Mcl-1 in Melanoma. Am J Pathol. 179 (5), 2162-2168 (2011).
- Chi, S. W., Hannon, G. J., Darnell, R. B. An alternative mode of microRNA target recognition. Nat Struct Mol Biol. 19 (3), 321-327 (1038).
- Jiao, L. R., et al. MicroRNAs targeting oncogenes are down-regulated in pancreatic malignant transformation from benign tumors. PLoS One. 7 (2), e32068(2012).
- Wolter, J. M., Kotagama, K., Pierre-Bez, A. C., Firago, M., Mangone, M. 3'LIFE: a functional assay to detect miRNA targets in high-throughput. Nucleic Acids Res. , (2014).
- Mangone, M., et al. The landscape of C. elegans 3'UTRs. Science. 329 (5990), 432-435 (2010).
- Ekdahl, Y., Farahani, H. S., Behm, M., Lagergren, J., Ohman, M. A-to-I editing of microRNAs in the mammalian brain increases during development. Genome Res. 22 (8), 1477-1487 (2012).
- Fu, Y., Dominissini, D., Rechavi, G., He, C. Gene expression regulation mediated through reversible m(6)A RNA methylation. Nat Rev Genet. 15 (5), 293-306 (2014).
- Zhou, P., et al. Large-scale screens of miRNA-mRNA interactions unveiled that the 3'UTR of a gene is targeted by multiple miRNAs. Plos One. 8 (7), e68204(2013).
- Dyer, B. W., Ferrer, F. A., Klinedinst, D. K., Rodriguez, R. A noncommercial dual luciferase enzyme assay system for reporter gene analysis. Anal Biochem. 282 (1), 158-161 (2000).
- Seiler, C. Y., et al. DNASU plasmid and PSI:Biology-Materials repositories: resources to accelerate biological research. Nucleic Acids Res. , (2013).
- Baek, D., et al. The impact of microRNAs on protein output. Nature. 455 (7209), 64-71 (2008).
- Ebert, M. S., Sharp, P. A. Roles for MicroRNAs in Conferring Robustness to Biological Processes. Cell. 149 (3), 515-524 (2012).
- Pelaez, N., Carthew, R. W. Biological robustness and the role of microRNAs: a network perspective. Current Topics in Developmental Biology. 99, 237-255 (2012).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır