Method Article
Nachweis von miRNA Targets in Hochdurchsatz-Verwendung des 3'LIFE Assay
In diesem Artikel
Zusammenfassung
Luminescent identification of functional elements in 3’ untranslated regions (3’UTRs) (3’LIFE) is a technique to identify functional regulation in 3’UTRs by miRNAs or other regulatory factors. This protocol utilizes high-throughput methodology such as 96-well transfection and luciferase assays to screen hundreds of putative interactions for functional repression.
Zusammenfassung
Lumineszierende Bezeichnung von Funktionselementen in 3'UTRs (3'LIFE) erlaubt die schnelle Identifizierung von Zielen bestimmte miRNAs innerhalb einer Anordnung von hunderten abgefragt 3'UTRs. Zielidentifizierung basiert auf dem Dual-Luciferase-Assay, das die Bindung erkennt auf mRNA-Ebene durch Messung translationale Ausgang, so dass ein funktions Auslesen von miRNA gezielt beruht. 3'LIFE nutzt nicht-proprietäre Puffer und Reagenzien, und öffentlich zugängliche Bibliotheken Reporter, dass genomweite Screens durchführbar und kostengünstig. 3'LIFE kann in einem Standard-Testumgebung durchgeführt werden, entweder mit oder up Liquid-Handling-Roboter und andere Hochdurchsatz-Instrumentierung skaliert. Wir veranschaulichen die Vorgehensweise unter Verwendung eines Datensatzes der menschlichen 3'UTRs in 96-Well-Platten kloniert und zwei Test miRNAs, let-7c und miR-10b. Wir zeigen, wie man DNA-Präparation, Transfektion, Zellkultur und Luciferaseassays in 96-Well-Format durchführen, und bieten Werkzeuge zur DatenAnalyse. Abschließend 3'LIFE ist sehr gut reproduzierbar, schnell, systematisch und identifiziert hohes Vertrauen Ziele.
Einleitung
Das übergeordnete Ziel dieser Methode ist, zu erkennen und zu microRNA (miRNA) Ziele präzise Karte im Hochdurchsatz. MiRNAs endogenen nicht-kodierenden RNAs ~ 22 Nukleotide lang. Nach der Transkription und Verarbeitung, sind reife miRNAs in einem Proteinkomplex eingebaut namens RNA induced silencing complex (RISC). Jedes miRNA führt die RISC auf Zielelemente vor allem in den 3'-untranslatierte Regionen (3'UTRs) von Boten-RNAs (mRNAs) gelegen, was entweder Übersetzung Repression oder mRNA-Spaltung 1. MiRNA erkennen, Zielorte basierend auf Standard-Watson-Crick und G: U Wobble-Basenpaarung und entartet in der Natur, mit mehreren fehlgepaarten Basenpaaren und ausgebeult Regionen. Viele miRNAs sind im Großen und Ganzen aus Pflanzen auf den Menschen 2,3, wo sie eine breite Palette von biologischen Rollen spielen konserviert. In Metazoen können miRNAs mehrere biologischen Prozessen einschließlich Zellschicksal Entscheidungen 4, Entwicklungszeit 5 beeinflussen , Und weisen häufig gewebespezifische Expressionsmuster 6,7. MiRNA misexpression kann auch aberrante Genregulation, die wesentlichen Einfluss auf das Zellverhalten ausschließlich auf die Funktion der Zielgene Portals resultieren. Als solche sind miRNAs zu einer Vielzahl von Krankheiten in Verbindung gebracht, einschließlich Neurodegeneration 8,9, Diabetes und Krebs 10 11. Bioinformatic und Nassbank Ansätze vorschlagen, dass jeder miRNA kann in der Lage gezielt Hunderte bis Tausende von verschiedenen mRNAs 12-14 sein, die anzeigt, dass Hochdurchsatz oder genomweite Ansätze sind erforderlich, um diese großen Pool von potentiellen Wechselwirkungen zu untersuchen.
Identifizierung von Zielgenen ist eine kritische Komponente des mechanistisch definieren miRNA-Funktion, und dazu müssen die Forscher in der Lage, Ziele in großem Maßstab zu offenbaren. Verschiedene Ansätze wurden entwickelt, um miRNA Ziele, einschließlich bioinformatische Vorhersage-Algorithmen zu identifizieren, Hochdurchsatz-Sequenzierunggezielte mRNAs und Reporter basierte Assays. Jeder dieser Ansätze hat inhärente Stärken und Schwächen. Da miRNA Targeting durch Sequenzspezifität geführt, insbesondere von Nucleotiden 2-6 der miRNA (der so genannten Seed-Region), mehrere Algorithmen entwickelt, um miRNA gesamten Genom vieler Organismen vorherzusagen. Diese Algorithmen werden mit den beobachteten Basenpaarung Motive validierter miRNA Targets ausgebildet und häufig nutzen, Parameter wie stringent seed Paarung Website Erhaltung und / oder thermodynamische Stabilität 15. Obwohl diese Filter zu verfeinern, die große Anzahl von mutmaßlichen Ziele mit ausreichender Komplementarität zu nur hohen Vertrauens Ziele können sie artspezifisch nichtkanonische miRNA Zielstellen, die den letzten Beweis schlägt sind weit verbreitet 16-24 ausschließen. Darüber hinaus sind diese Prognosen nicht berücksichtigt Mechanismen der mRNA-Prozessierung, die miRNA-Zielstellen, wie zum Beispiel alternative Polyadenylierung ausschließen nehmen25, 26 RNA-Editing, RNA-Methylierung 27 und kooperative Bindung. Als solche haben hohen falsch positiven und falsch negativen Ergebnisse für viele Algorithmen 22,24,28 gemeldet. Während diese Algorithmen sind nützlich, um Kandidaten miRNA Ziele für nachfolgende experimentelle Validierung zu identifizieren, diese hohen Fehlerquoten begrenzen die Wirksamkeit von Bioinformatik-Ansätze für die systematische miRNA Zielerfassung.
Systematisch nach Wechselwirkungen zwischen einem bestimmten miRNA und möglicherweise gezielt 3'UTRs Sonde haben wir ein Hochdurchsatz-Assay genannt Leucht Identifizierung von Funktionselementen in 3'UTRs (3'LIFE) 24 entwickelt. Dieser Assay misst direkte Interaktionen und translationale Repression des Test 3'UTR von einer Abfrage miRNA mit einem Dual-Luciferase-Reportersystems. In diesem System wird die 3'UTR eines Gens von Interesse stromab des Leuchtkäfer-Luciferase (fluc) Reporter-Leserahmen kloniert. Die Reporter construct ist mit einer Abfrage miRNA in HEK293T-Zellen cotransfiziert. MiRNA-Targeting wird durch Messung der relativen Änderung zwischen dem Test Fluc :: 3'UTR-Reporter und eine zweite nicht-spezifische Renilla Luciferase-Reporter bestimmt. Wichtig ist, dass die Luciferaseassays detektieren miRNA / mRNA-Wechselwirkungen, die die Translations Ausgang des Reporter beeinflussen. Dies ist ein entscheidender Vorteil gegenüber herkömmlichen Methoden, um miRNA Regulation, wie RT-qPCR und Western-Blots zu erkennen, daß diese umgeht Unterschiede in mRNA-Abbau und translationale Repression, sowie Änderungen in Proteinmenge unabhängig von 3'UTR basierte Regulierung.
Luciferase-Assays werden häufig genutzt, um direkten miRNA Ziele wegen ihrer relativen Einfachheit und Sensibilität, aber ihre Verwendung in Hochdurchsatz-Screens zu validieren ist von hohen Kosten, die mit verbrauchende Reagenzien Dritter, das Fehlen von 3'UTR Bibliotheken aus öffentlichen Quellen, und das Fehlen standardisierter Luciferase protocols, zu Schwierigkeiten führen beim Vergleich funktionelle Unterdrückung in mehreren Datensätzen. Um die Verwendung des 3'LIFE Assay zu erleichtern, haben wir Wert auf eine Vereinfachung der Versuchsplanung, Nutzung von nicht-kommerziellen Transfektion 24 und Luciferase Reagenzien 29 gestellt haben, die Schaffung einer 3'UTR-Bibliothek, die regelmäßig aktualisiert und erweitert wird, und ist durch die verfügbaren eine öffentliche Plasmid-Repository 30.
Die Skalierbarkeit der 3'LIFE Assay ermöglicht Screening einer großen 3'UTR Bibliothek für die Ausrichtung von einem bestimmten miRNA ohne Vorspannen des Bildschirms in Richtung bioinformatisch identifizierten Gene. Neben der Prüfung kanonischen und vorhergesagten Wechselwirkungen ermöglicht diese systematischen Ansatz die Identifizierung von neuen Targets über nicht-kanonischen und / oder Spezies-spezifische Wechselwirkungen angetrieben. Wichtig ist, dass die Wirkung von miRNA gezielt auf die Proteinproduktion versteht man im allgemeinen in bescheidenen translationale Repression 15,31 / Sup>, was darauf hindeutet, dass eine primäre Rolle der miRNA Regulation ist die Feinabstimmung Protein-Ausgang, Schutz vor abweichenden Genexpressionsniveaus und bieten Robustheit auf bestimmte Programme 32,33 Zelle. Die Empfindlichkeit des Luciferase-Assay in Kombination mit der Natur Vielzahl negativer miRNA / mRNA-Wechselwirkungen im 3'LIFE Bildschirm erlaubt den Nachweis von subtile Effekte der miRNA-Targeting auf einer großen Anzahl von Genen und die Identifizierung von mehreren Komponenten der Gen Netzwerken, werden durch eine miRNA 24 geregelt.
Hier beschreiben wir die 3'LIFE Protokoll, und zeigen es Machbarkeit durch Screening von zwei gut charakterisierten miRNAs, miR-10b und lassen-7c gegen eine Auswahl von 275 menschlichen 3'UTRs (Abbildung 1).
Protokoll
1. Zellkultur (24-48 h vor der Transfektion)
- 24-48 h vor der Transfektion Samen eine ausreichende Menge HEK293T-Zellen basierend auf der Anzahl von Platten mit 96 Vertiefungen transfiziert.
HINWEIS: Für die konsequente Transfektionen Platte Zellen mit einer ausreichenden Dichte schnelle Teilung zu begünstigen, noch nicht mehr als 70-90% Konfluenz werden zum Zeitpunkt der Transfektion. - Jede Platte mit 96 Vertiefungen erfordert 9 x 10 6 Zellen (75.000 Zellen pro Vertiefung, und 120 Vertiefungen pro Platte für die Verwendung von Vorratsbehälter und Mehrkanalpipette Konto). Berechnung der Verdopplungszeit von HEK293-Zellen (~ typischerweise 20 h), und Samen die geeignete Anzahl von Zellen, um mindestens 9 x 10 6 Zellen zur Zeit der Transfektion erhalten. A 145 mm Kreiskulturplatte ist in der Regel ausreichend für 3 96-Loch-Transfektionen, wenn sich zu ~ 90% Konfluenz, mit ~ 10% der Zellen verbleibende, um eine neue Platte reseed.
2. Vorbereitung vor der Transfektion
_content "> HINWEIS: Die Herstellung der Puffer und die Plasmid-DNA in Schritt 2,0-2,2 sollte in den Tagen vor der Transfektion durchgeführt, da die Herstellung dieser Reagenzien können zeitaufwendig sein kann.- Menschen 3'UTR Klone, die mit dem Assay 3'LIFE kompatibel sind, über ein öffentliches Plasmid-Repository 30 zur Verfügung. Reinigen DNA-Plasmid von Hand oder mit Liquid-Handling-Roboter. Verwenden Sie eine Transfektion Grade alkalische Lyse Mini-Prep 96-Loch-Kit und folgen Sie den Anweisungen des Herstellers. Resuspendieren der gereinigten Vektoren auf ~ 100 ng / ul pro Vertiefung.
HINWEIS: Zu wenig Luciferase-Signal wird, wenn die Plasmid-Konzentration unter 40 ng / & mgr; l. - Erhalten die pLIFE-miRNA-Vektoren 30 oder Klon mit der Rohrleitung in 1B 24. Resuspendieren der Vektoren in einer Konzentration von 500 ng / ul für jede miRNA und Blank Steuer Plasmide.
- Aufgrund der Empfindlichkeit der Nukleofektion Pufferbedingungen, sicherzustellen, dassdas Gesamtvolumen der transfizierten Materialien (einschließlich Zellen und Plasmide), 10% der gesamten Flüssigkeit in jede Vertiefung der 96-Loch-Transfektion Platte nicht überschreiten. Um dies zu erreichen, konzentrieren die pLIFE-miRNA Plasmid-Lager in einer Konzentration von mindestens 500 ng / ul.
- Vorbereitung 10x Glühwürmchenluciferase Pufferreagentien (Tabelle 1), und das 1X Renilla Luciferase Pufferreagentien (Tabelle 2), die für bis zu 6 Monate gelagert werden können.
HINWEIS: Der DVB-T in der Glühwürmchen-Luciferase-Puffer muss in Lösung bei -20 ° C in den einmaligen Gebrauch Aliquots gelagert werden.
3. Bereiten Nach Artikel Unmittelbar vor der Transfektion
- Vorbereitung Transfektionspuffer enthaltenden PBS, 1,5% HEPES, pH 7,0. Bereiten Sie diese frischen, auch wenn es für bis zu 1 Monat bei 4 ° C ohne merkliche Abnahme der Transfektionseffizienz gespeichert werden. Bei der Formulierung Puffer und Plasmid-DNA Bände, davon ausgehen, 120 Reaktionen für jede Platte mit 96 Vertiefungen zu sufficiently machen Fehler beim Pipettieren und Lautstärke mit Flüssigkeitsreservoirs und Mehrkanalpipetten verloren. Aliquot 18 ul pro Vertiefung Transfektionspuffer (120 Wells / 96-Well-Platte = 2,16 ml pro Platte), und beiseite stellen.
HINWEIS: Die zell Elektroporationsvorrichtung ist extrem empfindlich auf die Pufferbedingungen verwendet, um Zellen zu transfizieren. Genauigkeit bei der Herstellung von Puffern wird eine konstante Leistung des Gerätes zu gewährleisten. Zusätzliche Vorsicht ist bei der Durchführung des Assays, um Verdunstung von Puffern zu verhindern, und zwar durch Minimieren der Zeit, daß der Puffer gelassen wird in Mikrozentrifugenröhrchen, Platten mit 96 Vertiefungen, Reservoirs und Elektrodenplatten aufgedeckt genommen werden. - Reserve vier Brunnen für die folgenden Kontrollen, keine pLIFE-3'-UTR (zum Hintergrund der Luciferase-Assay zu messen), pLIFE-SV40 3'UTR (negative Zielkontrolle), Positivkontrolle für die miRNA # 1, positive Kontrolle für die miRNA # 2.
HINWEIS: Diese Vektoren sind öffentlich zugänglich 30. Alternativ kann jede zuvor validierten Zielkann als eine Positivkontrolle verwendet werden. - Warmen Medien, Trypsin (0,25%) auf 37 ° C.
- In jede Vertiefung der 96-Loch-Zellkulturplatte, 200 & mgr; l DMEM, ergänzt mit 10% FBS, 1% Pen / Strep und in einem 37 ° C Inkubator für die Verwendung nach der Transfektion platziert.
- Schalten Sie alle Geräte Zelle Elektroporation, gefolgt von der unterstützenden Software. Verwenden Pulse Code FF120 für HEK293T-Zellen und PBS / HEPES-Puffer.
4. Herstellung von Plasmid-DNA und Zellgemisch
HINWEIS: Die folgende Protokoll geht davon Transfektion drei Platten mit 96 Vertiefungen in einem Experiment für einen Bildschirm mit zwei miRNAs (miRNA # 1 und # 2 miRNA). Jede Platte wird auf die gleiche Platte mit 96 Vertiefungen von pLIFE-3'UTR Plasmide entsprechen, und dreimal mit pLIFE-miRNA-blank, pLIFE-miRNA # 1, oder pLIFE-miRNA # 2 behandelt werden.
- Bereiten Sie 3 Bestände pLIFE-miRNA + Transfektionspuffer für jedes miRNA. Dieser Bestand sollte für 50% (10 ul) des Gesamtvolumens der jedes Kontogut, um 120 Brunnen multipliziert. Daher sollte jede Aktien 1,08 ml Puffer + 120 ul Plasmid-DNA (pLIFE-miRNA) enthalten.
- Zellen aus 145 mm-Kulturplatte zu entfernen durch Eluieren Medien, wäscht vorsichtig mit PBS, und Behandeln mit ~ 5 ml 0,25% Trypsin für 5 min bei 37 ° C. Trypsin zu neutralisieren mit einem gleichen Volumen an Medium und Pellet-Zellen bei 300 g für 5 min.
- Entfernen Trypsin / Medien und Pellet in ~ 5-10 ml Medium (je nach Zelldichte und genaue Bereich von Zellzähler).
- Zählen von Zellen mit einem Zellzähler. Stellen Sie sicher, dass Zellen> 95% lebensfähig und in exakte Reihe von Maschine.
HINWEIS: Eine falsche Zellenzahl kann von extrem hohen Zellkonzentrationen (> 6.0x 10 6 / ml) zur Folge haben. Transfektion zu viele Zellen drastisch zu reduzieren Effizienz der miRNA-Targeting durch Reduzierung Plasmid: Zellverhältnis und / oder abnehm Transfektionseffizienz. - Aliquot drei Röhren mit jeweils 9 x 10 6 Zellen, die den Zellen entsprechen rzur Transfektion von einer Platte mit 96 Vertiefungen equired. Spin-Zellen bei 300 × g für 3 min.
- Medien zu entfernen. Achten Sie darauf, so viel wie möglich zu Medien mit minimaler Störung des Pellets zu entfernen, da überschüssige Medien Transfektionseffizienz beeinflussen.
- Die Zellen in 1,2 ml Transfektionspuffer / miRNA Plasmid-Gemisch, und beiseite stellen.
- Die folgenden Schritte detailliert Resuspension pLIFE-3'UTR Plasmid in Transfektionspuffer. Da dies in 96-Well-Platten auf, achten Sie darauf, Verdampfung von Puffer durch Abdeckplatten zu allen Zeiten zu vermeiden.
- Mit Hilfe einer Mehrkanalpipette, bewegen 32,4 ul Transfektionspuffer in jede Vertiefung einer 96-Well-PCR-Platte (9 ul [pro Transfektion] * 3 [Platten] * 1.2 [für Pipettenfehler Konto]).
- In 3,6 & mgr; l (~ 100 ng / ul) von Mini-prepped pLIFE-3'UTR Plasmid in jede Vertiefung geben und gut mischen.
- Je 10 & mgr; l dieser Mischung in jede Vertiefung der 96-Loch-Transfektion Platte und Abdeckung.
5. Transfektion
- Bewegen 1.2 ml der ersten Zelle / Puffer / pLIFE-miRNA Plasmid-Gemisch in das Reservoir. Gut mischen.
- Zugabe von 10 ul dieser Mischung in der ersten 96-Well-Transfektion Platte 10 ul Transfektionspuffer / pLIFE-3'UTR bereits enthält. Gut mischen durch Auf-und Abpipettieren mehrmals.
HINWEIS: Equal Suspension von Zellen in dem Puffer wird auch sichergestellt und gründliche Durchgang des elektrischen Stroms durch die Küvette und maximieren Transfektionseffizienz. - Platzieren Sie 96-Loch-Transfektion Platte auf der Zell Elektroporationsvorrichtung und initiieren Transfektion.
- Nach Transfektion abgeschlossen ist, fügen Sie 100 ul der vorgewärmten Medien von 96-Well-Kulturplatte in jedes Well der 96-Well-Platte Transfektion und gut mischen. Bewegen Sie 100 ul von jeder Vertiefung in die 96-Loch-Kulturplatte.
- Mischen Zellen in Kulturplatten mit Pipette vertikal in der Mitte der Vertiefung angeordnet ist, die als Zellen dazu neigen, an den Seiten des w aggregierenell, wenn richtig gemischt.
- Wiederholen 5,1-5,5 für die verbleibenden zwei Platten.
- Reinigung 96-Well-Platte Transfektion
- Die 96-Well Platten Transfektion kann durch Waschen mit 70% EtOH geführt werden, um keinen Übertrag von Nukleinsäuren zwischen den Experimenten zu gewährleisten. Führen Sie zwei 70% EtOH wäscht mit einer Sprühflasche, um vollständig zu füllen jedes Loch, gefolgt von Abwischen überschüssigen EtOH auf den Elektrodenstreifen (Unterseite) und ermöglicht die Transfektion Platten vollständig trocknen in der Kultur Kapuze.
Anmerkung: Wir haben für Carry-over DNA-Kontamination durch Transfizieren 12 Vertiefungen mit 2 & mgr; pmaxGFP jeweils in HEK293T-Zellen Plasmid getestet, gefolgt von einem einzelnen Waschschritt mit 70% EtOH und eine zweite Transfektion ohne Plasmid-DNA. Mit diesem extrem hohen Plasmidkonzentration, extrem helle Reporter und einem einzigen Wasch, gab es keine beobachtbaren Fluoreszenz in einem der 12 Transfektionen replizieren.
- Die 96-Well Platten Transfektion kann durch Waschen mit 70% EtOH geführt werden, um keinen Übertrag von Nukleinsäuren zwischen den Experimenten zu gewährleisten. Führen Sie zwei 70% EtOH wäscht mit einer Sprühflasche, um vollständig zu füllen jedes Loch, gefolgt von Abwischen überschüssigen EtOH auf den Elektrodenstreifen (Unterseite) und ermöglicht die Transfektion Platten vollständig trocknen in der Kultur Kapuze.
- Kulturzellen für 48-72 h bei 37 ° C, gefolgt von der doppelten luciflöschen Assay.
6. Zelllysat Vorbereitung für Luciferase Assay
- Verdünnen Sie Lysepuffer mit 4 Teilen Wasser, 1 Teil 5x passive Lysepuffer in einem Reservoir. Berechnen Sie 26 ul / Vertiefung, Hinzufügen ~ 20 zusätzliche Mengen, um den Verlust in dem Behälter zu berücksichtigen.
HINWEIS: Der Puffer wird bei -20 ° C gelagert und können äußerst dickflüssig und damit vor so dass der 5-fach-Puffer auf Raumtemperatur zu nähern wird Pipettiergenauigkeit verbessern. - Analysieren Sie jede Vertiefung für Transfektionseffizienz mittels Fluoreszenzmikroskopie. Beachten Sie, etwaige Unstimmigkeiten in Brunnen, die nicht effizient transfizieren haben (> 90% Transfektionseffizienz) oder äußern niedrigen RFP Hinweis auf Überfüllung oder Medien Erschöpfung. Entfernen Sie diese Brunnen aus der Analyse.
- Vollständig zu entfernen Sie das Medium aus den Zellen, man aufpassen, nicht zu schnell zu eluieren, welche dazu führen, Zellen zu lösen wird.
HINWEIS: Verbleibende Medien werden das Lysat und verursachen Schwankungen der Werte über Experime verdünnennts. - In 26 & mgr; l Lysepuffer in jede Vertiefung, und auf einem Plattenschüttler / Wippe bei niedriger / mittlerer Geschwindigkeit für ca. 20-30 min. Nutzen Sie diese Zeit, um die Luciferase-Puffer vorzubereiten, waschen und prime das Luminometer (n), und übertragen Lysat bis opak Messplatten.
7. Doppel Luciferase Assay
Hinweis: Wenn mehrere Platten werden der Reihe nach auf einem Luminometer gemessen, erstellen Puffermastermixen mit alles außer ATP und Substrate, das Hinzufügen dieser Reagenzien, gefolgt von pH-Einstellung unmittelbar vor der Anwendung mit jeder Platte. ATP und Substrate können im Laufe der Zeit verschlechtern; Kohärenz der Zeit sind diese Reagenzien im Puffer Konsistenz über mehrere Platten zu verbessern.
- Bereiten 1x Luciferase-Puffer (Tabelle 1):
- Wickeln Sie zwei Rohre (in der Regel 15/50 ml Zentrifugenröhrchen) mit der Firefly und Renilla-Puffer mit einer Aluminiumfolie, die als Substrate können lichtempfindlich sein.
- Bereiten 1x Firefly Luziferase-Puffer. 1 ml von jeder der fünf 10x Glühwürmchenluciferase Reagenzien Zugabe EGTA letzten, auf 5 ml H 2 0 zu einer endgültigen Konzentration 1x.
- Hinzuzufügen 0,025 g ATP auf 10 ml 1x firefly Puffer. Mischen Sie durch mehrmaliges Wenden. Halten ATP auf Eis zu allen Zeiten. ATP wird abgebaut, so dass, wenn mehr als eine Platte nacheinander messen, müssen Puffer neuen Anfang gemacht werden in diesem Schritt für jede weitere Platte.
- 100 l 100 Käfer-Luciferin (Substrat) (Tabelle 1) Buffer sollte gelbliche Farbe auf Basis von pH ändern.
- 1x Renilla-Luciferase-Puffer Rekonstitution: Per 96-Well-Platte, aliquote 10 ml 1 "Renilla Puffer".
- 100 l BSA (44 mg / ml Stamm).
- Wenn Screening mehr als eine Platte, separate Master-Mix in 10 ml-Teilmengen.
- Fügen Sie 100 ul Coelenterazin zu Puffer (bisher aufgeteilt und bei 100x konz gespeichert.)
- Einstellen des pH auf 8,0 firefly 1x Puffer, gefolgt von dem 1x Renilla-Puffer auf 5,0 unter Verwendung von NaOH und HCl.
HINWEIS: Die Aktivität von jedem Puffer und die Fähigkeit des Renilla Puffer, um die Firefly-Luciferase-Aktivität zu quenchen ist stark abhängig vom pH-Wert. Für gleichbleibende Ergebnisse als äußerst präzise in diesem Schritt. - Bringen Volumen jedes Puffers (entsprechend 1 96-well-Platte) auf 10,5 ml für Luminometer Priming unterzubringen.
- Übertragen Lysat bis opak weißen Platten: An dieser Stelle sollten die Zellen in Lysepuffer für ~ 20 min. 25 & mgr; l aus jede Vertiefung mit einer Mehrkanalpipette, lesen Sie auf- und abpipettieren gründlich zum Aufbrechen der Zellklumpen und homogenisieren Sie das Lysat.
- Bereiten Sie das Luminometer. Schalten Sie das Luminometer und wählen Sie das Protokoll in der DLR-Ordner namens "DLR mit zwei Injektionen". Andere Formate sind nicht mit der Datenanalyse-Pipeline (unten) kompatibel.
- Wählen Sie die Brunnen zu sein tinteressiert (alle Vertiefungen ist die Standardeinstellung).
- Verlängern Sie die 'Verzögerung vor der Messung "Einstellung auf 5 Sekunden, mit einer 10 sec Messzeit (siehe 29 für nähere Information).
- Kapillar-Waschschritte: 3x Wasser, EtOH 3x, Wasser 3x, trocken 3x. Prime Puffer einmal in den Abfall, und dann ein zweites Mal prime zurück in den Adern, um sicherzustellen, Mischen. Spritzen Sie die Glühwürmchen Puffer ersten und prime es in der linken Kapillare, gefolgt von Renilla in der rechten Kapillare.
- Initiieren Sie die Luciferase-Assay. Jede Platte sollte 48 min nehmen ~ zu lesen. Nach Beendigung speichern Sie die Datei zuerst, und dann wiederholen Sie die Waschschritte und schaltete den Luminometer.
HINWEIS: Mehrere Platten können gelesen werden und Daten auf dem gleichen Excel-Datei gespeichert, aber Probleme können mit mehreren Platte auftreten, wo das Luminometer liest Programm abstürzen. Achten Sie darauf, alle Daten zwischen den Messungen speichern und Screenshots, wenn Programm stürzt vor Speichern möglich ist.- Ersetzen Sie alte Puffermit neuen, wobei Sie auf prime mindestens zweimal mit neuen Puffer vor dem Start der neuen Platte.
8. Datenanalyse
- Nutzen Sie Excel-Tabellen "3'LIFE - Einzelplattenanalyse" und "3'LIFE - Mehr Analyse", erhältlich von www.mangonelab.com . Kopieren Sie Rohdaten für Firefly und Renilla-Luciferase-Messungen von Luminometer Ausgabedatei in die entsprechenden Stellen an den negativen Zustand, miRNA # 1, # 2 und miRNA in das "3'LIFE - Einzelplattenanalyse" Tabellenkalkulation.
- Das Spreadsheet berechnet automatisch Firefly / Renilla-Verhältnis und normalisieren jede miRNA auf die entsprechende negative Kontrolle und Repression zu normalisieren Werte in jeder Platte.
HINWEIS: Diese Tabelle wird automatisch Brunnen mit geringer Luciferase-Signal zu identifizieren, markieren Sie deutlich unterdrückte Vertiefungen und bieten Maßnahmen repressiauf über die gesamte Platte. Siehe 24 für eine detaillierte Erläuterung der statistischen Analyse.- Kopieren Sie die Werte aus der "Normalized RI Index" Box, in die entsprechenden Zellen in der "3'LIFE - Mehr Analyse" Tabellenkalkulation an der Position, die für jedes miRNA getestet replizieren # 1. Wiederholen Sie dies für alle biologischen Replikaten an verschiedenen Tagen durchgeführt.
- Falls gewünscht, legen Sie die Gennamen und Zielvorhersagestatus in Spalten B und D auf.
- Lassen Sie die Tabelle, um den Durchschnitt aller Platte automatisch zu berechnen. Beachten Sie die Daten in 96-Well-Format als Wärme Karte (Abbildung 2). Die Datei organisiert auch die Daten in Listenform (Abbildung 2).
HINWEIS: Die Repression Index ist die verwendet werden, um mutmaßlichen miRNA Targets zu identifizieren Maßnahme. Verschiedenen Stringenzbedingungen Parameter verwendet werden, basierend auf individuellen Vorlieben. Im Durchschnitt sehen wir wahrscheinlich Treffern unter Repression Index 0.8 und durch t-Test (p -Wert <0,05) statistisch signifikant. Wie in Abbildung 3 potentielle Ziele auf der Grundlage dieser Kriterien wird in rot hervorgehoben.
- Lassen Sie die Tabelle, um den Durchschnitt aller Platte automatisch zu berechnen. Beachten Sie die Daten in 96-Well-Format als Wärme Karte (Abbildung 2). Die Datei organisiert auch die Daten in Listenform (Abbildung 2).
Ergebnisse
Das Luminometer Ausgabedatei enthält Rohmessungen für beide Firefly und Renilla-Luciferase-Proteine. Das RAW-Format ist mit dem "3'LIFE - Einzelplattenanalyse" kompatibel "3'LIFE - Mehr Analyse" Arbeitsblätter zur Verfügung von der Mangone lab Website ( www.mangonelab.com ). Die einzelne Platte Analyse Tabellenkalkulation berechnet automatisch Firefly / Renilla-Verhältnis, normalisiert jeweils miRNA an die entsprechende Negativkontrolle und normalisiert Repression Werte über jeder Platte. Dieses Arbeitsblatt identifiziert automatisch Brunnen mit geringer Renilla-Luciferase-Signal hebt Brunnen, die Repression zeigen im Vergleich zur Negativkontrolle und bietet Repressionsmaßnahmen über die gesamte Platte (Abbildung 2). Siehe 24 für eine detaillierte Erläuterung der statistischen Analyse.
Mehrere Wiederholungen analysiert werden können,d mit dem "Mehr Analyse" Tabellenkalkulation. Jede Wiederholung wird nebeneinander verglichen und statistische Maße der Daten werden automatisch berechnet (Abbildung 3). Neben einem Abgleich der Wiederholungen mit den Spalten "Normalized Repression", kann der Benutzer Repression zwischen den beiden miRNAs unter "miRNA # 1 / miRNA # 2" Spalten zu vergleichen. Diese Maßnahme teilt die Unterdrückung Index für jedes miRNA für jede Wiederholung. Durch diese Maßnahme kann eine fehlerhafte Werte aus der Luciferase-Assay zeigen (zB hoher oder zu niedriger Messwerte mit der Negativkontrolle, siehe Abbildung 3, Reihe A9, Rep # 3), und Brunnen, wo die Unterdrückung Index kann nicht an erheblichen Repressionen, aber das zu tun zeigen signifikante Unterschiede zwischen den miRNAs. Während diese Maßnahme können nicht direkt an ein miRNA angeben verwendet werden zum Identifizieren von Ausreißern problematisch Vertiefungen oder Muster in den Daten, die nicht allein zugeschrieben werden miRNA re lenken istgulation.
Die Repression Index (RI) wird verwendet, um eine mutmaßliche miRNA Ziel nennen, mit niedrigeren Werten zu höheren relativen Unterdrückung entspricht. Die Schwelle für den Aufruf vermeintlichen Ziele auf der Ebene der Stringenz des Forschers erforderlich basiert, aber die Kombination der RI mit 3'UTRs, die statistisch signifikante p-Werte (p <0.05) Display zeigt hohes Vertrauen Ziele (siehe Abbildung 2 Zeilen und B8 B12).

Abbildung 1: 3'LIFE Assay (A). Gateway-kompatiblen Vektoren im 3'LIFE Assay verwendet. Top: Die Luciferase-Gen (Fluc) auf die Probe 3'UTR fusioniert ist, während die Renilla-Luciferase-Gen (RLuc) an die unspezifischen SV40 pA 3'UTR als Steuer fusioniert. Unten: Die RFP- miRNA-Intron vector - Die Sonde vor, miRNA, zzgl ~ 400Nukleotide innerhalb seiner genomischen Locus (um endogene miRNA Verarbeitungs rekapitulieren) wird innerhalb eines Introns kloniert, um seine Co-Expression mit DsRed2 Fluorochrom ermöglichen. Beide Vektoren sind öffentlich zugänglich (Seiler et al., 2013). (B) Flussdiagramm der 3'LIFE Assay. (C) 3'LIFE Pipeline: Die Dual-Luciferase-Vektor, der die Test 3'UTR mit oder ohne die miRNA Vektoren cotransfiziert in 96-Well-Platten in HEK293-Zellen. Die Wechselwirkung zwischen der miRNA und einem gutgläubigen 3'UTR Ziel wird die relative Lumineszenz in bestimmten Brunnen (von der orangen Fleck in der Versuchsplatte veranschaulicht) zu senken.

Abbildung 2: Beispiel für Daten mit 3'LIFE Assay produziert. Jede Sonde miRNA wird in vierfacher Ausführung getestet (repliziert 1-4). Farben repräsentieren Repression Ebenen, mit rotFarben anzeigt starke miRNA / 3'UTR Interaktion. Alle Replikate gemittelt werden, um qualitativ hochwertige in der Zusammenfassung Platte unter dem gelben Pfeil dargestellt mutmaßlichen Ziele zu erzeugen. White-Box darstellen Kontrollen versagt Transfektionen oder Brunnen mit geringer Transfektionseffizienz.
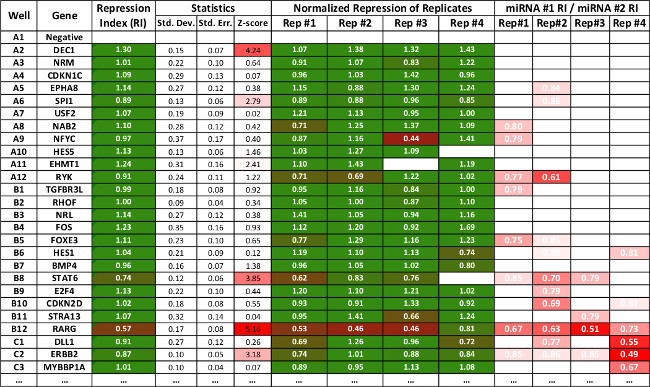
Fig. 3: Tabelle repräsentieren Summendaten einer Untergruppe von Wechselwirkungen mit der 3'LIFE Tabellen erzeugt die Repression Werte sind wie in Figur 2. Die Software berechnet die Standardabweichung, Standardfehler und Z-Score für jede Interaktion. Statistisch signifikante Wechselwirkungen sind rot markiert. Die letzten vier Zeilen zeigen relativen Unterdrückung einer miRNA auf die andere, und wird als Sekundäranzeige verwendet werden, um Repression zwischen zwei unterschiedlichen miRNAs zu vergleichen. Die Tabellenkalkulation kann heruntergeladen werden unter www.mangonelab.com
| Glühwürmchen-Luciferase-Pufferreagentien | Endkonzentration (1x) |
| Glycylglycin | 25 mM |
| K x PO 4 (pH 7,8) | 15 mM |
| MgSO 4 | 15 mM |
| DTT (Speicher bei 4º) | 1 mM |
| EGTA | 4 mM |
| ATP * | 2 mM |
| Beetle Luciferin * | 250 & mgr; M |
Tabelle 1: Auf Glühwürmchenluciferase Reagenzien: 10x Stammlösungen von Glycylglycin, K x PO 4, MgSO 4, DTT und EGTA können getrennt hergestellt und gelagert werden, bevor Rekonstitutionspuffer werden. 100x Beetle Luciferin (Glühwürmchen-Luciferase-Substrat) kann store seind durch Auflösen von 50 mg Luciferin in 7.134 ml H 2 0 (25 mM). Aliquot 105 ul / Platte gelöst Beetle Luciferin in Rohren und bei -80 ºC. Per Promega technischen Support, sollte dies für> 6 Monate stabil sein, sondern kann lichtempfindlich sein. HINWEIS: EGTA nicht in Lösung bei neutralem pH-Wert zu gehen. Gebe langsam NaOH auf EGTA, bis es vollständig auflöst. * Reagenzien zur endgültigen Puffer unmittelbar vor dem Luciferase-Assay
| Renilla-Luciferase Pufferreagentien | Endkonzentration (1x) |
| NaCl | 1.1 M |
| Na 2 EDTA | 2.2 mM |
| KH 2 PO 4 | 0,22 M |
| NaN 3 | 1,3 mM |
| BSA * | 0,44 mg / ml |
| Coelenterazin * | 2,5 & #956; M |
Tabelle 2: Auf Renilla-Luciferase-Puffer Reagenzien Alle Reagenzien außer BSA und Coelenterazin kann zu einem 1x Konzentration gemischt und bei Raumtemperatur gelagert werden. Coelenterazin kann in angesäuertem Methanol gelöst und aliquotiert pro Platte werden. Anzusäuern Methanol durch Zugabe von HCl bis zu einer Endkonzentration von 5 mM (<3 pH). Man löst 250 ug Coelenterazin in 2,36 ml angesäuertem Methanol (250 & mgr; M) aliquoten 105 ul / Platte. Die Mischung ist für mindestens 6 Monate stabil, kann aber lichtempfindlich sein. * Die Reagenzien hinzugefügt, um unmittelbar vor der Luciferase-Assay-Puffer.
Diskussion
Die 3'LIFE Assay identifiziert Funktions miRNA Ziele in 3'UTRs in Hochdurchsatz. Dieser Assay ist nützlich für Forscher, die experimentell Identifizierung einer großen Anzahl von mutmaßlichen Ziele für deren miRNA interessiere möchten. Die 3'LIFE Assay ein leistungsfähiger Ansatz zur 3'UTR angetrieben Regulierung abzufragen, indem der Assay stellt ein funktionelles Maß miRNA Targeting und die binäre Testen eines einzelnen Reporter :: 3'UTR gegen eine einzelne miRNA vertrauens adressieren das Targeting-Status einzelner Gene. Um diesen Ansatz zu validieren, abgeschirmt wir eine Gruppe von 275 3'UTRs und gegen zwei miRNAs, let-7c und miR-10b und 10 enthalten zuvor validierten Zielgene in dieser Bibliothek. Acht dieser zehn Gene zeigten Repression 24. Wir beobachteten auch eine bedeutende Bereicherung unvalidated bioinformatisch vorhergesagt Ziele und unvorhergesehene 3'UTRs die kanonische Samenelemente bei unseren Top-Hits enthalten, sughindeutet, dass 3'LIFE ist in der Lage zu identifizieren bona fide miRNA Ziele.
Ein wichtiger Indikator für die Empfindlichkeit der Hochdurchsatz-Screens ist die falsch positiven und falsch negativen Ergebnisse. Während die falsch positiv Rate von diesem Test muss durch zusätzliche alternative Ansätze zur Treffern zu validieren ausgewertet werden, acht der zehn positive Kontrollen in unseren proof-of-principle-Bildschirm zeigte Repression enthalten, was auf eine falsch negative Rate von 20%. Allerdings sind viele Techniken zur miRNA in verschiedenen zellulären Kontexten zu identifizieren und 3'UTR Verarbeitung und Regelung durch trans-wirkende Faktoren ist bekannt, sehr gewebespezifisch sein. Zum Beispiel kann die Mehrzahl der 3'UTRs enthalten mehrere Polyadenylierungsstellen, die die Länge der 3'-UTR in der reifen mRNA steuern. In vielen Fällen ist der Einsatz von proximal Polyadenylierungsstellen ist gewebespezifisch und können miRNA Zielstellen auszuschließen. Zusätzlich kooperative miRNA Targeting, Wettbewerb with-RNA-bindende Proteine und mRNA-Sekundärstruktur können alle Auswirkungen die Fähigkeit 3'LIFE zu miRNA Ziele in spezifischen Geweben zu detektieren. Aus diesem Grund kann die Erfassung von Zielen von der 3'LIFE Assay vom zellulären Kontext, in dem der Test durchgeführt wird, variieren, erschwert die Auswertung von absoluten Fehlerraten. Dieses Protokoll wird für HEK293T-Zellen optimiert, aber alternative Zelllinien können verwendet werden, wenn sich der Forscher möchte den Assay in einer bestimmten biologischen Kontext auszuführen. Allerdings wird die Optimierung der Transfektionseffizienz und das Überleben der Zellen mit jeder Zelllinie muss mit mehreren Pufferbedingungen Impulscodes und der Anzahl der Zellen optimiert werden. Ein Beispiel einer Optimierungsschema kann Wolter et al. 24 gefunden werden.
Dieses Protokoll wurde in 96-Well-Format optimiert und gibt die Verwendung bestimmter Hochdurch Instrumentierung. Im Falle, dass das Institut nicht die erforderliche Ausrüstung für 96 besitzen-Wanne Nucleofection, alternative Transfektionsreagenzien verwendet werden könnte, um den Assay 3'LIFE solange die Transfektionseffizienz hoch bleibt auszuführen, werden. Zusätzlich ist die Luciferase-Assay der zeitaufwendigste Aspekt der 3'LIFE Assay. Als solche ist die Verwendung von mehreren Luminometer für Hochdurchsatz-empfohlen.
Offenlegungen
This work is supported by funds from the College of Liberal Arts and Science and the Biodesign Institute at Arizona State University and NIH Exploratory/Developmental Research Grant 1R21CA179144-01A1.
Danksagungen
We thank Stephen Blazie, Karen Anderson, Josh LaBaer for advice and discussion. Karen Anderson, John Chaput, and Josh LaBaer for sharing reagents and instrumentation, Michael Gaskin and Andrea Throop for technical advise and protocols. Justin Wolter is a Maher scholar and thanks the Maher family for their generous support.
Materialien
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| glycylglycine | Sigma | G1127-25G | |
| Kx PO4 | Sigma | P2222 | |
| EGTA | Sigma | E3889 | |
| ATP | Sigma | FLAAS | |
| DTT | Sigma | D0632 | |
| MgSO4 | Sigma | M7506 | |
| CoA | Sigma | C4282 | |
| luciferin | Sigma | L9504 | |
| NaCl | Sigma | S7653 | |
| Na2EDTA | Sigma | E0399 | |
| K H2 P O4 | Sigma | 1551139 | |
| BSA | Sigma | A2153 | |
| NaN3 | Sigma | S2002 | |
| Coelenterazine | Sigma | C3230 | |
| PBS/HEPES | Corning | 21-040-CV | |
| DMEM | Sigma | D5546 | |
| FBS | Sigma | F2442 | |
| Pennicilin/Streptomicin | Sigma | P4333 | |
| Trypsin | T2600000 | ||
| Consumables | |||
| MaxiPrep Kit | Promega | A2392 | |
| 96-well miniprep plate | Pall | 8032 | |
| 96-well shuttle plates | Lonza | V4SP-2096 | |
| 5x Lysis Buffer | Promega | E1941 | |
| Instruments | |||
| 96-well GloMax Plate Reader | Promega | E9032 | |
| Biomech FX Liquid Handler Robot | Beckmann | A31842 | |
| 4D-Nucleofector Core Unit | Lonza | AAF-1001 | |
| 96-well Shuttle System | Lonza | AAM-1001 | |
| Cell Counter Countess | Invitrogen | C10227 |
Referenzen
- Bartel, D. P. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell. 116 (2), 281-297 (2004).
- Pasquinelli, A. E., et al. Conservation of the sequence and temporal expression of let-7 heterochronic regulatory RNA. Nature. 408 (6808), 86-89 (2000).
- Reinhart, B. J., Weinstein, E. G., Rhoades, M. W., Bartel, B., Bartel, D. P. MicroRNAs in plants. Genes Dev. 16 (13), 1616-1626 (2002).
- Ivey, K. N., et al. MicroRNA regulation of cell lineages in mouse and human embryonic stem cells. Cell Stem Cell. 2 (3), 219-229 (2008).
- Lee, R. C., Feinbaum, R. L., Ambros, V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 75 (5), 843-854 (1993).
- Wienholds, E., et al. MicroRNA expression in zebrafish embryonic development. Science. 309 (5732), 310-311 (2005).
- Darnell, D. K., et al. MicroRNA expression during chick embryo development. Dev Dyn. 235 (11), 3156-3165 (2006).
- Jin, P., Alisch, R. S., Warren, S. T. RNA and microRNAs in fragile X mental retardation. Nat Cell Biol. 6 (11), 1048-1053 (2004).
- Kim, J., et al. A MicroRNA feedback circuit in midbrain dopamine neurons. Science. 317 (5842), 1220-1224 (2007).
- Poy, M. N., et al. A pancreatic islet-specific microRNA regulates insulin secretion. Nature. 432 (7014), 226-230 (2004).
- Calin, G. A., Croce, C. M. MicroRNA signatures in human cancers. Nature Reviews Cancer. 6 (11), 857-866 (2006).
- Friedman, R. C., Farh, K. K., Burge, C. B., Bartel, D. P. Most mammalian mRNAs are conserved targets of microRNAs. Genome Res. 19 (1), 92-105 (2009).
- Paraskevopoulou, M. D., et al. DIANA-microT web server v5.0: service integration into miRNA functional analysis workflows. Nucleic Acids Res. 41 (Web Server issue), W169-W173 (2013).
- Krek, A., et al. Combinatorial microRNA target predictions. Nat Genet. 37 (5), 495-500 (2005).
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136 (2), 215-233 (2009).
- Vella, M. C., Choi, E. Y., Lin, S. Y., Reinert, K., Slack, F. J. The C. elegans microRNA let-7 binds to imperfect let-7 complementary sites from the lin-41 3'UTR. Genes Dev. 18 (2), 132-137 (2004).
- Lal, A., et al. miR-24 Inhibits cell proliferation by targeting E2F2, MYC, and other cell-cycle genes via binding to 'seedless' 3'UTR microRNA recognition elements. Molecular Cell. 35 (5), 610-625 (2009).
- Cevec, M., Thibaudeau, C., Plavec, J. N. M. R. NMR structure of the let-7 miRNA interacting with the site LCS1 of lin-41 mRNA from Caenorhabditis elegans. Nucleic Acids Res. 38 (21), 7814-7821 (1093).
- Shin, C., et al. Expanding the microRNA targeting code: functional sites with centered pairing. Molecular Cell. 38 (6), 789-802 (2010).
- Azzouzi, I., et al. MicroRNA-96 directly inhibits gamma-globin expression in human erythropoiesis. PLoS One. 6 (7), e22838(2011).
- Chen, J., et al. miR-193b Regulates Mcl-1 in Melanoma. Am J Pathol. 179 (5), 2162-2168 (2011).
- Chi, S. W., Hannon, G. J., Darnell, R. B. An alternative mode of microRNA target recognition. Nat Struct Mol Biol. 19 (3), 321-327 (1038).
- Jiao, L. R., et al. MicroRNAs targeting oncogenes are down-regulated in pancreatic malignant transformation from benign tumors. PLoS One. 7 (2), e32068(2012).
- Wolter, J. M., Kotagama, K., Pierre-Bez, A. C., Firago, M., Mangone, M. 3'LIFE: a functional assay to detect miRNA targets in high-throughput. Nucleic Acids Res. , (2014).
- Mangone, M., et al. The landscape of C. elegans 3'UTRs. Science. 329 (5990), 432-435 (2010).
- Ekdahl, Y., Farahani, H. S., Behm, M., Lagergren, J., Ohman, M. A-to-I editing of microRNAs in the mammalian brain increases during development. Genome Res. 22 (8), 1477-1487 (2012).
- Fu, Y., Dominissini, D., Rechavi, G., He, C. Gene expression regulation mediated through reversible m(6)A RNA methylation. Nat Rev Genet. 15 (5), 293-306 (2014).
- Zhou, P., et al. Large-scale screens of miRNA-mRNA interactions unveiled that the 3'UTR of a gene is targeted by multiple miRNAs. Plos One. 8 (7), e68204(2013).
- Dyer, B. W., Ferrer, F. A., Klinedinst, D. K., Rodriguez, R. A noncommercial dual luciferase enzyme assay system for reporter gene analysis. Anal Biochem. 282 (1), 158-161 (2000).
- Seiler, C. Y., et al. DNASU plasmid and PSI:Biology-Materials repositories: resources to accelerate biological research. Nucleic Acids Res. , (2013).
- Baek, D., et al. The impact of microRNAs on protein output. Nature. 455 (7209), 64-71 (2008).
- Ebert, M. S., Sharp, P. A. Roles for MicroRNAs in Conferring Robustness to Biological Processes. Cell. 149 (3), 515-524 (2012).
- Pelaez, N., Carthew, R. W. Biological robustness and the role of microRNAs: a network perspective. Current Topics in Developmental Biology. 99, 237-255 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten