Method Article
La detección de los genes miARN objetivos en de alto rendimiento utilizando el ensayo 3'LIFE
En este artículo
Resumen
Luminescent identification of functional elements in 3’ untranslated regions (3’UTRs) (3’LIFE) is a technique to identify functional regulation in 3’UTRs by miRNAs or other regulatory factors. This protocol utilizes high-throughput methodology such as 96-well transfection and luciferase assays to screen hundreds of putative interactions for functional repression.
Resumen
Luminiscentes Identificación de elementos funcionales en 3'UTRs (3'LIFE) permite la rápida identificación de los objetivos de miRNAs específicos dentro de una matriz de cientos de 3'UTRs consultados. La identificación del objetivo se basa en el ensayo de luciferasa dual, que detecta la unión a nivel de mRNA mediante la medición de la salida de la traducción, dando una lectura funcional de los genes miARN focalización. 3'LIFE utiliza buffers no propietarios y reactivos, y bibliotecas reportero públicamente disponibles, por lo que las pantallas de todo el genoma viable y rentable. 3'LIFE se puede realizar en un laboratorio estándar o ampliarse el uso de robots de manipulación de líquidos y otros instrumentos de alto rendimiento. Se ilustra el método utilizando un conjunto de datos de 3'UTRs humanos clonados en placas de 96 pocillos, y dos miRNAs prueba, vamos-7c y miR-10b. Demostramos cómo realizar la preparación de ADN, transfección, cultivo celular y ensayos de luciferasa en formato de 96 pocillos, y proporcionar herramientas para datosanálisis. En conclusión 3'LIFE es altamente reproducible, rápido, sistemático, e identifica objetivos de alta confianza.
Introducción
El objetivo general de este método consiste en detectar y mapear precisamente microARN (miARN objetivos) en alto rendimiento. MiRNAs son endógenos ARN no codificantes ~ 22 nucleótidos de longitud. Después de la transcripción y el procesamiento, miRNAs maduros se incorporan en un complejo de proteína llamada el silenciamiento de ARN inducida complejo (RISC). Cada miRNA guía el RISC a elementos situados principalmente en las regiones 3 'no traducida (3'UTRs) de los ARN mensajeros (ARNm) diana, lo que resulta en cualquiera de traducción represión o mRNA división 1. Mirna reconocer sitios diana, según norma Watson-Crick y G: U wobble base de sincronización, y son degenerados en la naturaleza, que contiene varios pares de bases coincidentes y las regiones se hinchó. Muchos miRNAs son ampliamente conservadas de las plantas a los seres humanos 2,3, donde juegan una amplia gama de funciones biológicas. En metazoos miRNAs pueden influir múltiples procesos biológicos incluyendo las decisiones del destino celular 4, el calendario de desarrollo 5 , Y con frecuencia exhiben patrones de expresión específicos de tejido 6,7. MIRNA misexpression también puede resultar en la regulación génica aberrante, que puede tener una influencia sustancial en el comportamiento celular basada únicamente en la función de los genes diana. Como tal, miRNAs están vinculados a una amplia gama de enfermedades, incluyendo neurodegeneración 8,9, diabetes y cáncer 10 11. Bioinformática y enfoques húmedo banco sugieren que cada miARN puede ser capaz de apuntar a cientos de miles de ARNm distintos 12-14, lo que indica que se requieren enfoques sectoriales de alto rendimiento o del genoma para investigar este gran número de interacciones potenciales.
La identificación de genes diana es un componente crítico de la definición de la función mecánica miARN, y hacer lo que los investigadores deben ser capaces de revelar los objetivos a gran escala. Se han desarrollado varios enfoques para identificar los genes miARN objetivos, incluyendo los algoritmos de predicción bioinformáticas, secuenciación de alto rendimientodel objetivo ARNm, y el reportero ensayos basa. Cada uno de estos enfoques tiene fortalezas y debilidades inherentes. Dado que los genes miARN focalización es guiado por la especificidad de secuencia, más notablemente de los nucleótidos 2-6 de la miARN (denominada la región de la semilla), varios algoritmos se han desarrollado para predecir los genes miARN objetivos en todo el genoma de muchos organismos. Estos algoritmos son capacitados utilizando los observados motivos de apareamiento de bases de los genes miARN objetivos validados, y con frecuencia utilizan parámetros como la vinculación estricta de semillas, la conservación de sitios y / o estabilidad termodinámica 15. Si bien estos filtros refinan el gran número de supuestos objetivos con suficiente complementariedad con los objetivos de confianza solamente altas, pueden excluir especies sitios específicos y no canónicos miARN objetivo, que la evidencia reciente sugiere se han generalizado 16-24. Por otra parte, estas predicciones no tienen en cuenta los mecanismos de procesamiento de ARNm que excluyen a los sitios objetivo miARN, como alternativa polyadenylation25, 26 edición de ARN, el ARN metilación 27 y unión cooperativa. Como tal, las altas tasas de falsos negativos y falsos positivos han sido reportados para muchos algoritmos 22,24,28. Mientras que estos algoritmos son útiles para identificar candidatos miARN objetivos para la validación experimental posterior, estas altas tasas de error limitar la eficacia de los enfoques bioinformáticas para la detección sistemática miARN.
Para investigar sistemáticamente la interacción entre un miARN dado y 3'UTRs potencialmente dirigidas hemos desarrollado un ensayo de alto rendimiento llamado luminiscentes Identificación de elementos funcionales en 3'UTRs (3'LIFE) 24. Este ensayo mide las interacciones directas y la represión de traslación de la 3'UTR de prueba por una consulta miARN usando un sistema indicador de luciferasa dual. En este sistema, el 3'UTR de un gen de interés se clona aguas abajo de la luciferasa (Fluc) reportero marco de lectura luciérnaga. Los contras reporterotruct se cotransfecta con una consulta miRNA en células HEK293T. Mirna focalización se determina midiendo la variación relativa entre el Fluc prueba :: reportero 3'UTR y un segundo reportero no específica luciferasa Renilla. Es importante destacar que los ensayos de luciferasa detectan interacciones funcionales miARN / mRNA que influyen en la salida de traslación de la periodista. Esta es una ventaja clave sobre los métodos tradicionales para detectar la regulación de los genes miARN, tales como RT-qPCR y transferencias de Western, en que esta pasa por diferencias en la degradación del ARNm y la represión de traducción, así como los cambios en la abundancia de proteína independiente de la regulación basada 3'UTR.
Los ensayos de luciferasa son ampliamente utilizados para validar blancos directos miARN debido a su sencillez y sensibilidad relativa, sin embargo, su uso en pantallas de alto rendimiento está limitado por los altos costos asociados con reactivos consumibles, la falta de bibliotecas 3'UTR de fuentes públicas, y la ausencia de prot luciferasa normalizadaPROTOCOLOS, dando lugar a dificultades en la comparación de la represión funcional a través de múltiples conjuntos de datos. Para facilitar el uso del ensayo 3'LIFE, hemos puesto énfasis en la simplificación del diseño experimental, la utilización de la transfección 24 y luciferasa reactivos no comerciales 29, la creación de una biblioteca 3'UTR que se actualiza periódicamente y se expandió, y está disponible a través de un repositorio público plásmido 30.
La escalabilidad del ensayo 3'LIFE permite la detección de una gran biblioteca 3'UTR para la orientación por un miARN dado sin sesgar la pantalla hacia genes bioinformatically identificados. Además de probar las interacciones canónicas y previstos, este enfoque sistemático permite la identificación de nuevas dianas impulsadas a través de interacciones no canónicos y / o específicos de las especies. Es importante destacar que el efecto de los genes miARN la orientación en la producción de proteína se entiende generalmente para dar lugar a la represión de traducción modesto 15,31 </ Sup>, lo que sugiere que un papel primordial de la regulación miARN es para poner a punto la producción de proteínas, la protección contra los niveles aberrantes de expresión génica, y proporcionar robustez a la celda programas específicos 32,33. La sensibilidad del ensayo de luciferasa se combina con el inherentemente gran número de interacciones negativas miARN / mRNA en la pantalla 3'LIFE permite la detección de los efectos sutiles de miARN focalización en un gran número de genes, y la identificación de múltiples componentes de las redes de genes que están regulados por un determinado miARN 24.
Aquí se describe el protocolo 3'LIFE, y demostrar que es de viabilidad mediante el cribado de dos miRNAs bien caracterizados, miR-10b y let-7c contra un panel de 275 3'UTRs humanos (Figura 1).
Protocolo
1. Cultivo Celular (24-48 h antes de la transfección)
- 24-48 h antes de la transfección semilla una cantidad suficiente de células HEK293T basado en el número de placas de 96 pocillos se transfectaron.
NOTA: Para las transfecciones consistentes, células de la placa a una densidad suficiente para favorecer la rápida división, sin embargo, no ser en más de 70-90% de confluencia en el momento de la transfección. - Cada placa de 96 pocillos requiere 9 x 10 6 células (75000 células por pocillo, y 120 pocillos por placa para tener en cuenta el uso de depósito y pipeta multicanal). Calcular el tiempo de duplicación de las células HEK293 (típicamente ~ 20 h), y la semilla el número adecuado de células para obtener al menos 9 x 10 6 células en el momento de la transfección. Una placa de cultivo de 145 mm circular es típicamente suficiente para 3 transfecciones 96 pocillos cuando se cultiva a ~ 90% de confluencia, con ~ 10% de las células restantes para sembrar de nuevo una nueva placa.
2. Preparación Antes de la transfección
_content "> NOTA: La preparación de los tampones y ADN plásmido en el paso 2,0-2,2 debe realizarse en los días antes de la transfección desde la preparación de estos reactivos puede llevar mucho tiempo.- 3'UTR clones humanos que son compatibles con el ensayo 3'LIFE están disponibles a través de un repositorio de plásmido público 30. Purificar el ADN del plásmido de forma manual o con los robots de manipulación de líquidos. Utilice un kit de mini-prep de 96 pocillos de grado transfección lisis alcalina y siga las instrucciones del fabricante. Resuspender los vectores purificados a ~ 100 ng / l por pocillo.
NOTA: La señal de luciferasa insuficiente producirá si la concentración de plásmido cae por debajo de 40 ng / l. - Obtenga la pLIFE-miARN vectores 30 o clon usando el gasoducto en la figura 1B 24. Resuspender los vectores a una concentración de 500 ng / l para cada miARN y los plásmidos control en blanco.
- Debido a la sensibilidad de las condiciones de tampón nucleofection, asegurar queel volumen total de materiales (incluyendo las células transfectadas y plásmidos) no supere el 10% del total de líquido en cada pocillo de la placa de la transfección de 96 pocillos. Para lograr esto, concentrar el plásmido de stock pLIFE-miARN a una concentración de por lo menos 500 ng / l.
- Preparar reactivos tampón 10x luciferasa de luciérnaga (Tabla 1), y los reactivos tampón 1x Renilla luciferasa (Tabla 2), que se pueden almacenar durante un máximo de 6 meses.
NOTA: La TDT en el tampón de luciferasa de luciérnaga debe a ser almacenado en solución a -20 ° C en alícuotas de un solo uso.
3. Preparar siguientes artículos Inmediatamente antes de la transfección
- Preparar tampón de transfección que contiene PBS, 1,5% HEPES, pH 7,0. Prepare esta fresca, aunque puede ser almacenado durante un máximo de 1 mes a 4 ° C sin disminuciones notables en la eficiencia de transfección. En la formulación de los volúmenes de tampón y de ADN plásmido, asumir 120 reacciones para cada placa de 96 pocillos para sufficiently cuenta de errores en la pipeta y el volumen perdido el uso de depósitos de líquido y pipetas multicanal. Alícuota 18 l por tampón de transfección bien (120 pozos / 96 pocillos de placas = 2,16 ml por placa) y dejar de lado.
NOTA: El dispositivo celular electroporación es extremadamente sensible a las condiciones de tampón utilizados para transfectar células. Exactitud cuando se preparan tampones garantizar un rendimiento constante del equipo. Un cuidado especial debe ser tomado al realizar el ensayo para evitar la evaporación de los tampones, específicamente al minimizar el tiempo que búfer se deja al descubierto en tubos de microcentrífuga, placas de 96 pocillos, embalses y placas de electrodos. - Reserva cuatro pozos para los siguientes controles, sin pLIFE-3'UTR (para medir el fondo de ensayo de luciferasa), pLIFE-SV40 3'UTR (control de destino negativo), control positivo para miARN # 1, el control positivo para miARN # 2.
NOTA: Estos vectores son a disposición del público 30. Alternativamente, cualquier objetivo previamente validadose puede utilizar como un control positivo. - Medios cálidos, tripsina (0,25%) a 37 ° C.
- A cada pocillo de placa de 96 pocillos de cultivo de células, añadir 200 l de DMEM suplementado con 10% FBS, 1% Pen / Strep, y se coloca en una incubadora a 37 ° para el uso después de la transfección.
- Encienda todos los dispositivos de electroporación de células seguido por el software de soporte. Utilice el código de pulso FF120 para las células HEK293T y tampón PBS / HEPES.
4. Preparación del plásmido de ADN y mezcla de células
NOTA: El siguiente protocolo asume la transfección de tres placas de 96 pocillos en un experimento para una pantalla con dos miRNAs (miRNA- # 1 y # 2 miRNA-). Cada placa se corresponderá con la misma placa de 96 pocillos de plásmidos pLIFE-3'UTR, y ser tratado tres veces con pLIFE-miRNA-blanco, pLIFE-miRNA- # 1, o pLIFE-miRNA- # 2.
- Preparar 3 poblaciones de tampón de transfección pLIFE-miARN + para cada miARN. Esta acción debe representar el 50% (10 l) del volumen total de cadaasí, multiplicado por 120 pozos. Por lo tanto, cada acción debe contener 1,08 ml de tampón + 120 ADN plásmido l (pLIFE-miRNA).
- Retire las células de placa de cultivo de 145 mm eluyendo medios de comunicación, lavar suavemente con PBS, y tratando con 5 ml ~ 0,25% de tripsina durante 5 min a 37 ° C. Neutralizar la tripsina con un volumen igual de medios de comunicación, y las células de pellets a 300 xg durante 5 min.
- Retire tripsina / medios de comunicación, y pellet se resuspende en ~ 5-10 ml de medio (dependiendo de la densidad celular y el rango exacto de contador de células).
- Contar las células utilizando un contador de células. Asegúrese de que las células son> 95% viable y dentro del alcance preciso de la máquina.
NOTA: Un recuento de células incorrecto puede ser resultado de las concentraciones de células extremadamente altos (> 6.0x 10 6 / ml). La transfección de demasiadas células pueden reducir drásticamente la eficiencia de los genes miARN focalización mediante la reducción de plásmido: proporción de células y / o la disminución de la eficiencia de transfección. - Alícuotas tres tubos cada uno conteniendo 9 x 10 6 células, correspondiente a las células rEQUERIDO para la transfección de una placa de 96 pocillos. Girar las células a 300 xg durante 3 min.
- Retire los medios de comunicación. Asegúrese de retirar la mayor cantidad de medios de comunicación como sea posible con el menor trastorno posible de la pastilla como el exceso de medios de comunicación pueden afectar la eficacia de transfección.
- Resuspender las células en 1,2 ml de tampón de transfección / mezcla de plásmido miARN, y dejar de lado.
- Los siguientes pasos detallan resuspensión del plásmido pLIFE-3'UTR en tampón de transfección. Mientras esto ocurre en placas de 96 pocillos, tenga cuidado para evitar la evaporación de tampón cubriendo placas en todo momento.
- Usando una pipeta multicanal, mueva 32,4 l de tampón de transfección en cada pocillo de una placa de PCR 96 (9 l [por transfección] * 3 [placas] * 1,2 [para dar cuenta de error pipeta]).
- Añadir 3,6 l (~ 100 ng / l) de pLIFE-3'UTR plásmido mini-preparada a cada pocillo y mezclar bien.
- Pipeta 10 l de esta mezcla en cada pocillo de la placa de la transfección de 96 pozos y la cubierta.
5. La transfección
- Mueva 1,2 ml de la primera / tampón / mezcla de plásmido pLIFE-miARN celular en el depósito. Mezclar bien.
- Añadir 10 l de esta mezcla en la primera placa de transfección de 96 pocillos ya que contiene 10 l de tampón de transfección / pLIFE-3'UTR. Mezclar bien pipeteando arriba y abajo varias veces.
NOTA: Igual suspensión de células en el tampón se asegurará incluso a fondo y el paso de la corriente eléctrica a través de la cubeta y maximizar la eficiencia de la transfección. - Coloque la placa de 96 pocillos de transfección en el dispositivo de electroporación de células e iniciar la transfección.
- Una vez se haya completado la transfección, añadir 100 l de los medios de comunicación pre-calentado de 96 pocillos placa de cultivo a cada pocillo de la placa de la transfección de 96 pocillos y mezclar bien. Mueva 100 l de cada pocillo en la placa de cultivo de 96 pocillos.
- Mezclar las células en placa de cultivo con la pipeta en posición vertical en el centro del pozo, como las células tienden a agregarse en los lados de la well menos mezclado correctamente.
- Repita 5.1 a 5.5 para las dos placas restantes.
- Limpieza de la placa de la transfección de 96 pocillos
- Las placas de transfección de 96 pocillos pueden ser reciclados por lavado con EtOH al 70% para asegurar que no se arrastren de los ácidos nucleicos entre los experimentos. Realice dos lavados de 70% EtOH utilizando una botella de spray para llenar completamente cada pocillo, seguido por limpiando el exceso de EtOH en las tiras de los electrodos (parte inferior) y permitiendo que las placas de transfección a completamente seca en la campana de cultivo.
NOTA: Hemos probado para traspaso de la contaminación de ADN mediante la transfección de 12 pozos con 2 mg cada pmaxGFP plásmido en células HEK293T, seguido de un solo lavado con EtOH al 70%, y una segunda transfección sin ADN plásmido. Con esta concentración extremadamente alta plásmido, reportero extremadamente brillante, y un solo lavado, no hubo fluorescencia observable en cualquiera de las transfecciones 12 replicados.
- Las placas de transfección de 96 pocillos pueden ser reciclados por lavado con EtOH al 70% para asegurar que no se arrastren de los ácidos nucleicos entre los experimentos. Realice dos lavados de 70% EtOH utilizando una botella de spray para llenar completamente cada pocillo, seguido por limpiando el exceso de EtOH en las tiras de los electrodos (parte inferior) y permitiendo que las placas de transfección a completamente seca en la campana de cultivo.
- Células de cultivo durante 48-72 horas a 37 ° C, seguido por la doble lucifborrar ensayo.
6. Preparación de lisado de la célula para ensayo de luciferasa
- Diluir tampón de lisis con 4 partes de agua, 1 parte de 5x tampón de lisis pasiva en un depósito. Calcula 26 l / pocillo, añadiendo ~ 20 volúmenes adicionales para dar cuenta de la pérdida en el depósito.
NOTA: Buffer se almacena a -20 ºC y puede ser extremadamente viscoso, por lo tanto antes de permitir que el tampón 5x para acercarse a temperatura ambiente mejorará la precisión de pipeteo. - Analizar cada pocillo para la eficiencia de la transfección mediante microscopía de fluorescencia. Nota cualquier inconsistencia en los pozos que no transfectan de manera eficiente (> 90% de eficiencia de transfección), o se expresan bajos niveles de RFP indicativo de hacinamiento o medios agotamiento. Retire estos pozos del análisis.
- Eliminar completamente los medios de comunicación de las células, teniendo cuidado de no eluir demasiado rápido lo que hará que las células que se desprenden.
NOTA: Los medios restantes diluirán las fluctuaciones de lisado y causar en valores a través de EXPERIMEnts. - Añadir 26 l de tampón de lisis a cada pocillo, y colocar en un plato agitador basculante / a velocidad baja / moderada de ~ 20-30 min. Aproveche este tiempo para preparar buffers de luciferasa, lavar y preparar el luminómetro (s), y la transferencia de lisado de placas de medición opacos.
7. Dual Luciferase Assay
NOTA: Si hay varias placas se están midiendo de forma secuencial en un luminómetro, crear mezclas de reacción de amortiguamiento con todo excepto ATP y sustratos, la adición de estos reactivos seguido de ajuste del pH inmediatamente antes de su uso con cada plato. ATP y sustratos pueden degradarse con el tiempo; coherencia en la cantidad de tiempo que estos reactivos están en el buffer mejorará la coherencia en varios platos.
- Preparar buffers de luciferasa 1x (Tabla 1):
- Envuelva dos tubos (típicamente 15/50 ml tubos de centrífuga) que contienen la luciérnaga y Renilla tampones con papel de aluminio, como sustratos pueden ser sensibles a la luz.
- Prepare el buffer de la luciferasa de la luciérnaga 1x. Añadir 1 ml de cada uno de los cinco 10x reactivos de luciferasa de luciérnaga, la adición de EGTA pasado, a 5 ml H 2 0 a una concentración 1x final.
- Añadir 0,025 g ATP a 10 ml 1x tampón de luciérnaga. Mezclar invirtiendo varias veces. Mantenga ATP en hielo en todo momento. ATP se degrada, por lo que si la medición de más de una placa de forma secuencial, tampón debe ser hecho nuevo comienzo en este paso para cada placa adicional.
- Añadir 100 l de 100 luciferina de escarabajo (sustrato) (Tabla 1) Buffer debe cambiar a color amarillento basado en el pH.
- 1x Renilla luciferasa búfer reconstitución: Per placa de 96 pocillos, alícuota de 10 ml de 1 "Renilla tampón".
- Añadir 100 l de BSA (44 mg / ml de acciones).
- Si el cribado más de un plato, mezcla maestra separada en 10 ml alícuotas.
- Añadir 100 l de coelenterazina para amortiguar (previamente en alícuotas y se almacena a 100x conc.)
- Ajustar el pH de 1x tampón de luciérnaga a 8,0, seguido por el tampón 1x Renilla a 5,0 usando NaOH y HCl.
NOTA: La actividad de cada tampón, y la capacidad de la memoria intermedia de Renilla para apagar la actividad de la luciferasa de la luciérnaga es altamente dependiente de pH. Para obtener resultados consistentes ser extremadamente precisa en este paso. - Llevar volumen de cada tampón (correspondiente a la placa 1 de 96 pocillos) a 10,5 ml para acomodar para el cebado luminómetro.
- Transferir el lisado a placas blancas opacas: En este punto las células deben estar en tampón de lisis durante ~ 20 min. Tomar 25 l de cada pocillo usando una pipeta multicanal, asegúrese de pipetear arriba y abajo a fondo para romper los grupos de células y homogeneizar el lisado.
- Preparar el luminómetro. Encienda el luminómetro y seleccione el protocolo en la carpeta DLR, llamado "DLR con dos inyecciones". Otros formatos no son compatibles con la tubería de análisis de datos (a continuación).
- Seleccione los pozos que tsado (todos los pozos es el valor predeterminado).
- Extender el 'Retardo antes de la medición' ajuste a 5 segundos, con un tiempo de medición de 10 segundos (ver 29 para la explicación).
- Capilares pasos de lavado: 3x agua, EtOH 3x, 3x agua, 3x seco. Tampones Prime una vez en los residuos, y el entonces primer por segunda vez de nuevo en los tubos de protección para asegurar la mezcla. Inyectar el buffer luciérnaga primera y primordial en el capilar a la izquierda, seguido de Renilla en el capilar derecha.
- Iniciar el ensayo de luciferasa. Cada placa debe tener ~ 48 min de leer. Después de la terminación guardar el archivo, y luego repetir los pasos de lavado y cierre el luminómetro.
NOTA: placas múltiples se pueden leer los datos almacenados en el mismo archivo de Excel, sin embargo los problemas se pueden encontrar con la placa de varias lecturas donde el programa luminómetro se estrellará. Asegúrese de guardar todos los datos entre las mediciones y realizar capturas de pantalla si programa se bloquea antes de guardar es posible.- Vuelva a colocar tampones viejoscon el nuevo, asegurándose de primer al menos dos veces con nuevos amortiguadores antes de comenzar la nueva placa.
Análisis 8. Datos
- Utilizar tablas excel "3'LIFE - análisis de placa única" y "3'LIFE - análisis multidisco" disponible de www.mangonelab.com . Copie los datos en bruto de luciérnaga y Renilla luciferasa mediciones de archivo de salida luminómetro en lugares correspondientes a la condición negativa, miRNA # 1, # 2 y miRNA en el "3'LIFE - Análisis de placa única" hoja de cálculo.
- La hoja de cálculo calculará automáticamente luciérnaga / relación de Renilla y normalizar cada miARN al control negativo apropiado, y normalizar los valores de la represión a través de cada plato.
NOTA: Esta hoja de cálculo identificará automáticamente pozos con baja señal luciferasa, resalte pozos significativamente reprimidos, y proporcionar medidas de repressien toda la placa. Ver 24 para una explicación detallada de los análisis estadísticos.- Copie los valores en el cuadro de "Normalizado Índice RI", en las células correspondientes en el - de hoja de cálculo "3'LIFE análisis multidisco" en la posición correspondiente para replicar # 1 para cada miARN probado. Repita para todas las réplicas biológicas realizadas en días diferentes.
- Si lo desea, introduzca los nombres de genes y el estado de la predicción de destino en las columnas B y D, respectivamente.
- Deje que la hoja de cálculo para calcular automáticamente el promedio de toda la placa. Observe los datos en formato de 96 así como un mapa de calor (Figura 2). El archivo también organiza los datos en formato de lista (Figura 2).
NOTA: El índice de la represión es la medida utilizada para identificar supuestos objetivos miARN. Diferentes parámetros de rigurosidad se pueden utilizar, en base a las preferencias individuales. En promedio se considera probables éxitos debajo índice represión de 0.8 y estadísticamente significativa por la prueba t (p-valor <0,05). Como se muestra en la Figura 3 posibles objetivos basados en estos criterios se destacan en rojo.
- Deje que la hoja de cálculo para calcular automáticamente el promedio de toda la placa. Observe los datos en formato de 96 así como un mapa de calor (Figura 2). El archivo también organiza los datos en formato de lista (Figura 2).
Resultados
El archivo de salida luminómetro contiene mediciones primas para ambas proteínas luciérnaga y Renilla luciferasa. Este formato RAW es compatible con el "3'LIFE - Análisis de placa única" y "3'LIFE - Análisis multidisco" hojas de cálculo disponible en la página web del laboratorio Mangone ( www.mangonelab.com ). El único análisis placa de hoja de cálculo calcula automáticamente luciérnaga / relación de Renilla, normaliza cada miARN al control negativo apropiado, y normaliza los valores de la represión a través de cada plato. Esta hoja de cálculo identifica automáticamente los pozos con baja señal de luciferasa de Renilla, destaca pozos que muestran la represión en comparación con el control negativo, y proporciona medidas de represión a través de toda la placa (Figura 2). Ver 24 para una explicación detallada de los análisis estadísticos.
Múltiples réplicas se pueden analizard utilizando la hoja de cálculo "análisis multidisco". Cada réplica se compara lado a lado, y las medidas estadísticas de los datos se calculan automáticamente (Figura 3). Además de comparar las réplicas con las columnas "Represión normalizado", el usuario puede comparar la represión entre los dos miRNAs en el marco del "miARN # 1 / # 2 miARN" columnas. Esta medida divide el índice de represión para cada miARN para cada réplica. Esta medida puede indicar valores erróneos desde el ensayo de luciferasa (por ejemplo lecturas anormalmente altas o bajas con el control negativo, véase la figura 3, fila A9, Rep # 3), y pozos donde el índice de represión puede no indicar la represión sustancial, pero que sí lo hacen exhibir diferencias significativas entre los miRNAs. Aunque esta medida no puede ser utilizado directamente para indicar un objetivo miRNA, es útil para la identificación de los valores atípicos, pozos problemáticos, o patrones en los datos que no se asignen exclusivamente para dirigir los genes miARN regulation.
La represión Index (RI) se usa para llamar a un objetivo putativo miARN, con valores más bajos correspondientes a una mayor represión relativa. El umbral para llamar supuestos objetivos se basa en el nivel de rigor requerido por el investigador, pero la combinación de la RI con 3'UTRs que muestran los valores de p estadísticamente significativas (p <0,05) indicará objetivos de alta confianza (ver Figura 2 Filas B8 y B12).

Figura 1: 3'LIFE de ensayo (A). Los vectores compatibles-Puerta de enlace utilizados en el ensayo 3'LIFE. Top: El gen de la luciferasa (Fluc) se fusiona a la 3'UTR de prueba, mientras que el gen de la luciferasa de Renilla (Rluc) se fusiona con el inespecífica SV40 pA 3'UTR como control. Conclusión: El miARN-intrón vector RFP- - La sonda pre-miARN, además de ~ 400nucleótidos dentro de su locus genómico (recapitular procesamiento de miRNA endógeno), se clona dentro de un intrón para permitir su co-expresión con DsRed2 fluorocromo. Ambos vectores son públicamente disponibles (Seiler et al., 2013). (B) Diagrama de flujo del ensayo 3'LIFE. (C) 3'LIFE Pipeline: El vector de doble luciferasa que contiene el 3'UTR de prueba con o sin los vectores de genes miARN son co-transfectaron en células HEK293 en placas de 96 pocillos. La interacción entre el miARN y un objetivo 3'UTR bona fide bajará la luminiscencia relativa en pozos específicos (ejemplificado por la mancha de color naranja en la placa experimental).

Figura 2: Ejemplo de los datos producidos con el ensayo 3'LIFE. Cada sonda miRNA se prueba en cuatro ejemplares (replica 1-4). Los colores representan los niveles de represión, con rojocolores que indican fuerte miARN / interacciones 3'UTR. Todas las réplicas se promedian para producir supuestos objetivos de alta calidad que se muestran en la placa de resumen debajo de la flecha amarilla. Caja blanca representan los controles, no transfecciones o pozos con baja eficiencia de transfección.
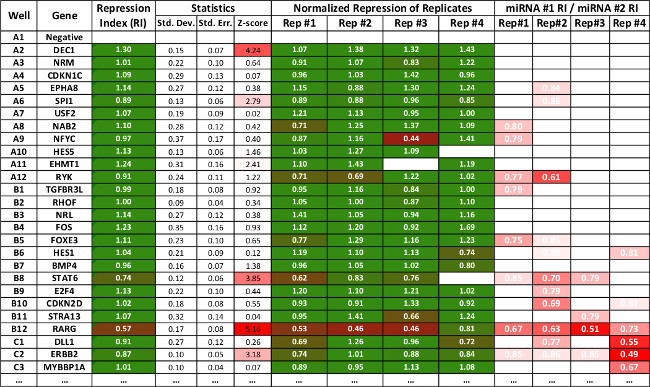
Figura 3:. Tabla que representa los datos de resumen de un subconjunto de las interacciones producidas utilizando la hoja de cálculo 3'LIFE Los valores de represión son como en la figura 2. El software calcula la desviación estándar, error estándar y el z-score para cada interacción. Estadísticamente interacciones significativas están marcados en rojo. Los últimos cuatro filas muestran la represión relativa de uno de los genes miARN a la otra, y se utiliza como indicador secundario para comparar la represión entre dos miRNAs diferentes. La hoja de cálculo se puede descargar desde www.mangonelab.com
| Firefly reactivos tampón luciferasa | La concentración final (1x) |
| Glicilglicina | 25 mM |
| K x PO 4 (pH 7,8) | 15 mM |
| MgSO 4 | 15 mM |
| TDT (almacenar a 4º) | 1 mM |
| EGTA | 4 mM |
| ATP * | 2 mM |
| Escarabajo luciferina * | 250 M |
Tabla 1: Stock reactivos de luciferasa de luciérnaga: 10x soluciones madre de Glicilglicina, K x PO 4, MgSO4, TDT y EGTA se pueden preparar por separado y se almacenan antes de amortiguar la reconstitución. 100x Escarabajo luciferina (sustrato de luciferasa de luciérnaga) puede ser tiendad disolviendo 50 mg luciferina en 7.134 ml H 2 0 (25 mM). Alícuota 105 l / plato de disuelto luciferina Escarabajo en tubos y almacenar a -80 ºC. Por el apoyo técnico Promega, esto debe ser estable durante> 6 meses, pero puede ser sensible a la luz. NOTA: EGTA no voy a entrar en solución a pH neutro. Se añade lentamente NaOH a EGTA hasta que se disuelva completamente. * Reactivos añadidos a tampón final inmediatamente antes de el ensayo de luciferasa
| Renilla luciferasa reactivos tampón | La concentración final (1x) |
| NaCl | 1,1 M |
| Na 2 EDTA | 2,2 mM |
| KH 2 PO 4 | 0,22 M |
| NaN 3 | 1,3 mM |
| BSA * | 0,44 mg / ml |
| Coelenterazina * | 2.5 & #956; M |
Tabla 2: Stock Renilla luciferasa reactivos tampón Todos los reactivos excepto BSA y coelenterazina se pueden mezclar a una concentración de 1x y se almacenaron a temperatura ambiente. Coelenterazina se puede disolver en metanol acidificado y se dividió en alícuotas por placa. Acidificar metanol mediante la adición de HCl a una concentración final de 5 mM (<3 pH). Disolver 250 g coelenterazina en 2,36 ml de metanol acidificado (250 M) alícuotas de 105 ul / placa. La mezcla es estable durante al menos 6 meses, pero puede ser sensible a la luz. * Reactivos añadió al tampón inmediatamente antes del ensayo de luciferasa.
Discusión
El ensayo 3'LIFE identifica los genes miARN objetivos funcionales en 3'UTRs en alto rendimiento. Este ensayo es útil para los investigadores que desean identificar experimentalmente un gran número de supuestos objetivos para su miARN de interés. El ensayo 3'LIFE es un enfoque poderoso para consultar para la regulación impulsado 3'UTR, en que el ensayo proporciona una medida funcional de los genes miARN focalización, y la prueba binario de un solo reportero :: 3'UTR contra un solo miARN puede abordar con confianza el estado de la orientación de los genes individuales. Para validar este enfoque, que exhibió un panel de 275 3'UTRs y en contra de dos miRNAs, vamos-7c y miR-10b, y se incluyeron 10 genes diana previamente validados en esta biblioteca. Ocho de estos diez genes mostraron represión 24. También se observó un significativo enriquecimiento de objetivos bioinformatically predichos no validados y 3'UTRs imprevistos que contienen elementos de semillas canónicas entre nuestros grandes éxitos, suggesting que 3'LIFE es capaz de identificar los genes miARN objetivos de buena fe.
Un indicador clave de la sensibilidad de las pantallas de alto rendimiento es la tasa de falsos negativos y falsos positivos. Mientras que la tasa de falsos positivos de este ensayo debe ser evaluado utilizando enfoques alternativos adicionales para validar hits, ocho de los diez controles positivos incluidos en nuestra pantalla de la represión exhibido prueba de principio, lo que sugiere una tasa de falsos negativos de 20%. Sin embargo, muchas de las técnicas se utilizan para identificar los genes miARN objetivos en diferentes contextos celulares, y el procesamiento 3'UTR y regulación por factores que actúan en trans es conocido por ser altamente tejido específico. Por ejemplo, la mayoría de 3'UTRs contiene múltiples sitios de poliadenilación, que controlan la longitud de la 3'UTR en el ARNm maduro. En muchos casos el uso de sitios de poliadenilación proximales es específica de tejido, y puede excluir sitios diana miARN. Además, la orientación miARN cooperativa, wi competenciaproteínas de unión de ARN-th y estructura secundaria de ARNm pueden todo el impacto de la capacidad de 3'LIFE para detectar los genes miARN objetivos en tejidos específicos. Debido a esto, la detección de blancos por el ensayo 3'LIFE puede variar en función del contexto celular en el que se realiza el ensayo, lo que complica la evaluación de las tasas de error absoluto. Este protocolo se ha optimizado para las células HEK293T, pero las líneas celulares alternativas se puede utilizar si el investigador desea realizar el ensayo en un contexto biológico específico. Sin embargo, la optimización de la eficiencia de transfección y la supervivencia celular con cada línea celular tendrá que ser optimizado utilizando múltiples condiciones de tampón, códigos de pulso, y el número de células. Un ejemplo de un esquema de optimización se puede encontrar en Wolter et. Al 24.
Este protocolo se ha optimizado en formato de 96 pocillos y se especifica el uso de cierta instrumentación de alto rendimiento. En el caso de que la institución no posee el equipo necesario para 96-bien Nucleofection, reactivos de transfección alternativa podría ser utilizado para realizar el ensayo 3'LIFE, siempre y cuando la eficacia de transfección sigue siendo alta. Además, el ensayo de luciferasa es el tiempo que consume aspecto más del ensayo 3'LIFE. Como tal, se recomienda el uso de múltiples luminómetros para pantallas de alto rendimiento.
Divulgaciones
This work is supported by funds from the College of Liberal Arts and Science and the Biodesign Institute at Arizona State University and NIH Exploratory/Developmental Research Grant 1R21CA179144-01A1.
Agradecimientos
We thank Stephen Blazie, Karen Anderson, Josh LaBaer for advice and discussion. Karen Anderson, John Chaput, and Josh LaBaer for sharing reagents and instrumentation, Michael Gaskin and Andrea Throop for technical advise and protocols. Justin Wolter is a Maher scholar and thanks the Maher family for their generous support.
Materiales
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| glycylglycine | Sigma | G1127-25G | |
| Kx PO4 | Sigma | P2222 | |
| EGTA | Sigma | E3889 | |
| ATP | Sigma | FLAAS | |
| DTT | Sigma | D0632 | |
| MgSO4 | Sigma | M7506 | |
| CoA | Sigma | C4282 | |
| luciferin | Sigma | L9504 | |
| NaCl | Sigma | S7653 | |
| Na2EDTA | Sigma | E0399 | |
| K H2 P O4 | Sigma | 1551139 | |
| BSA | Sigma | A2153 | |
| NaN3 | Sigma | S2002 | |
| Coelenterazine | Sigma | C3230 | |
| PBS/HEPES | Corning | 21-040-CV | |
| DMEM | Sigma | D5546 | |
| FBS | Sigma | F2442 | |
| Pennicilin/Streptomicin | Sigma | P4333 | |
| Trypsin | T2600000 | ||
| Consumables | |||
| MaxiPrep Kit | Promega | A2392 | |
| 96-well miniprep plate | Pall | 8032 | |
| 96-well shuttle plates | Lonza | V4SP-2096 | |
| 5x Lysis Buffer | Promega | E1941 | |
| Instruments | |||
| 96-well GloMax Plate Reader | Promega | E9032 | |
| Biomech FX Liquid Handler Robot | Beckmann | A31842 | |
| 4D-Nucleofector Core Unit | Lonza | AAF-1001 | |
| 96-well Shuttle System | Lonza | AAM-1001 | |
| Cell Counter Countess | Invitrogen | C10227 |
Referencias
- Bartel, D. P. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell. 116 (2), 281-297 (2004).
- Pasquinelli, A. E., et al. Conservation of the sequence and temporal expression of let-7 heterochronic regulatory RNA. Nature. 408 (6808), 86-89 (2000).
- Reinhart, B. J., Weinstein, E. G., Rhoades, M. W., Bartel, B., Bartel, D. P. MicroRNAs in plants. Genes Dev. 16 (13), 1616-1626 (2002).
- Ivey, K. N., et al. MicroRNA regulation of cell lineages in mouse and human embryonic stem cells. Cell Stem Cell. 2 (3), 219-229 (2008).
- Lee, R. C., Feinbaum, R. L., Ambros, V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 75 (5), 843-854 (1993).
- Wienholds, E., et al. MicroRNA expression in zebrafish embryonic development. Science. 309 (5732), 310-311 (2005).
- Darnell, D. K., et al. MicroRNA expression during chick embryo development. Dev Dyn. 235 (11), 3156-3165 (2006).
- Jin, P., Alisch, R. S., Warren, S. T. RNA and microRNAs in fragile X mental retardation. Nat Cell Biol. 6 (11), 1048-1053 (2004).
- Kim, J., et al. A MicroRNA feedback circuit in midbrain dopamine neurons. Science. 317 (5842), 1220-1224 (2007).
- Poy, M. N., et al. A pancreatic islet-specific microRNA regulates insulin secretion. Nature. 432 (7014), 226-230 (2004).
- Calin, G. A., Croce, C. M. MicroRNA signatures in human cancers. Nature Reviews Cancer. 6 (11), 857-866 (2006).
- Friedman, R. C., Farh, K. K., Burge, C. B., Bartel, D. P. Most mammalian mRNAs are conserved targets of microRNAs. Genome Res. 19 (1), 92-105 (2009).
- Paraskevopoulou, M. D., et al. DIANA-microT web server v5.0: service integration into miRNA functional analysis workflows. Nucleic Acids Res. 41 (Web Server issue), W169-W173 (2013).
- Krek, A., et al. Combinatorial microRNA target predictions. Nat Genet. 37 (5), 495-500 (2005).
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136 (2), 215-233 (2009).
- Vella, M. C., Choi, E. Y., Lin, S. Y., Reinert, K., Slack, F. J. The C. elegans microRNA let-7 binds to imperfect let-7 complementary sites from the lin-41 3'UTR. Genes Dev. 18 (2), 132-137 (2004).
- Lal, A., et al. miR-24 Inhibits cell proliferation by targeting E2F2, MYC, and other cell-cycle genes via binding to 'seedless' 3'UTR microRNA recognition elements. Molecular Cell. 35 (5), 610-625 (2009).
- Cevec, M., Thibaudeau, C., Plavec, J. N. M. R. NMR structure of the let-7 miRNA interacting with the site LCS1 of lin-41 mRNA from Caenorhabditis elegans. Nucleic Acids Res. 38 (21), 7814-7821 (1093).
- Shin, C., et al. Expanding the microRNA targeting code: functional sites with centered pairing. Molecular Cell. 38 (6), 789-802 (2010).
- Azzouzi, I., et al. MicroRNA-96 directly inhibits gamma-globin expression in human erythropoiesis. PLoS One. 6 (7), e22838(2011).
- Chen, J., et al. miR-193b Regulates Mcl-1 in Melanoma. Am J Pathol. 179 (5), 2162-2168 (2011).
- Chi, S. W., Hannon, G. J., Darnell, R. B. An alternative mode of microRNA target recognition. Nat Struct Mol Biol. 19 (3), 321-327 (1038).
- Jiao, L. R., et al. MicroRNAs targeting oncogenes are down-regulated in pancreatic malignant transformation from benign tumors. PLoS One. 7 (2), e32068(2012).
- Wolter, J. M., Kotagama, K., Pierre-Bez, A. C., Firago, M., Mangone, M. 3'LIFE: a functional assay to detect miRNA targets in high-throughput. Nucleic Acids Res. , (2014).
- Mangone, M., et al. The landscape of C. elegans 3'UTRs. Science. 329 (5990), 432-435 (2010).
- Ekdahl, Y., Farahani, H. S., Behm, M., Lagergren, J., Ohman, M. A-to-I editing of microRNAs in the mammalian brain increases during development. Genome Res. 22 (8), 1477-1487 (2012).
- Fu, Y., Dominissini, D., Rechavi, G., He, C. Gene expression regulation mediated through reversible m(6)A RNA methylation. Nat Rev Genet. 15 (5), 293-306 (2014).
- Zhou, P., et al. Large-scale screens of miRNA-mRNA interactions unveiled that the 3'UTR of a gene is targeted by multiple miRNAs. Plos One. 8 (7), e68204(2013).
- Dyer, B. W., Ferrer, F. A., Klinedinst, D. K., Rodriguez, R. A noncommercial dual luciferase enzyme assay system for reporter gene analysis. Anal Biochem. 282 (1), 158-161 (2000).
- Seiler, C. Y., et al. DNASU plasmid and PSI:Biology-Materials repositories: resources to accelerate biological research. Nucleic Acids Res. , (2013).
- Baek, D., et al. The impact of microRNAs on protein output. Nature. 455 (7209), 64-71 (2008).
- Ebert, M. S., Sharp, P. A. Roles for MicroRNAs in Conferring Robustness to Biological Processes. Cell. 149 (3), 515-524 (2012).
- Pelaez, N., Carthew, R. W. Biological robustness and the role of microRNAs: a network perspective. Current Topics in Developmental Biology. 99, 237-255 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados