Method Article
Обнаружение микроРНК цели в высокой пропускной Использование 3'LIFE Пробирной
В этой статье
Резюме
Luminescent identification of functional elements in 3’ untranslated regions (3’UTRs) (3’LIFE) is a technique to identify functional regulation in 3’UTRs by miRNAs or other regulatory factors. This protocol utilizes high-throughput methodology such as 96-well transfection and luciferase assays to screen hundreds of putative interactions for functional repression.
Аннотация
Люминесцентные Идентификация функциональных элементов в 3'UTRs (3'LIFE) позволяет быструю идентификацию целей конкретных микроРНК в массиве сотен запрашиваемых 3'UTRs. Целевой идентификации основан на двойной люциферазы, который обнаруживает связывания на уровне мРНК путем измерения поступательное выход, давая функциональный считывание микроРНК ориентации. 3'LIFE использует неимущественные буферы и реагенты, и публично доступные репортер библиотеки, делая генома экраны целесообразным и экономически эффективным. 3'LIFE может быть выполнена либо в стандартном лабораторных или расширены с использованием жидких роботов обработки и другой высокой пропускной приборы. Проиллюстрируем подход с использованием набора данных человеческих 3'UTRs клонированных в 96-луночных планшетах, и двух испытаний микроРНК, пусть-7c, и микроРНК-10b. Мы показываем, как выполнять препарат ДНК, трансфекцию, культуры клеток и люциферазы анализов в формате 96-луночного, и предоставляют инструменты для передачи данныхАнализ. В заключение 3'LIFE хорошо воспроизводим, быстрый, систематический и определяет высокие цели доверительные.
Введение
Общая цель этого метода заключается в выявлении и точно карту микроРНК (микроРНК) цели в высокой пропускной. МикроРНК эндогенные некодирующие РНК ~ 22 нуклеотидов в длину. После транскрипции и обработки, зрелые микроРНК включены в белковый комплекс называется РНК индуцированного глушителей комплекса (RISC). Каждый микроРНК направляет RISC целевых элементов, расположенных в основном в регионах 3'untranslated (3'UTRs) в матричных РНК (мРНК), в результате чего либо репрессий перевода или мРНК расщепления 1. Мирна признать целевые сайты, основанные на стандартной Уотсона-Крика и G: U раскачиваться спаривание оснований, и вырождаются в природе, содержащий несколько ошибочно спаренных пар оснований и выпучил регионы. Многие микроРНК широко сохраняется от растений до человека 2,3, где они играют широкий спектр биологических функций. В многоклеточных микроРНК могут влиять несколько биологических процессов, включая судьбы клеток решениями 4, сроков развития 5 И часто демонстрируют тканеспецифические паттерны экспрессии 6,7. Мирна неправильная экспрессия может также привести к аномальным регуляции генов, которые могут иметь существенное влияние на поведение клеток, основанной исключительно на функции генов-мишеней. Таким образом, микроРНК связаны с широким спектром заболеваний, в том числе нейродегенеративных 8,9, диабета и рака 10 11. Биоинформационный и мокрого стендовые подходы позволяют предположить, что каждая микроРНК могут быть способны ориентации от сотен до тысяч различных мРНК 12-14, что указывает на высокую пропускную способность или геномные широкие подходы для зонда этот большой круг потенциальных взаимодействий.
Определение целевых генов критическим компонентом механистически определяющей функции микроРНК, и сделать так, исследователи должны быть в состоянии выявить цели на больших масштабах. Несколько подходов были разработаны для выявления микроРНК целей, в том числе по биоинформатике алгоритмов прогнозирования, высокой пропускной последовательностицелевых мРНК, и журналистом анализов. Каждый из этих подходов имеет присущие сильные и слабые стороны. Учитывая, что микроРНК нацеливание ориентируется на специфичность последовательности, в первую очередь из нуклеотидов 2-6 миРНК (называемый запальной зоне), несколько алгоритмов были разработаны, чтобы предсказать целевые микроРНК во всем геноме многих организмов. Эти алгоритмы обучаются с помощью наблюдаемых спаривание оснований мотивы проверенных целей микроРНК, и часто используют такие параметры, как строгий семян спаривания, сохранения сайте, и / или термодинамической стабильности 15. В то время как эти фильтры результаты большого количества предполагаемых целей с достаточной комплементарности к только высоких целей доверия, они могут исключить видовой и неканонические микроРНК целевых сайтов, которые последние данные свидетельствуют о широко распространены 16-24. Кроме того, эти прогнозы не учитывают механизмы процессинга мРНК, которые исключают микроРНК целевых сайтов, таких как альтернативного полиаденилирования25, 26 редактирование РНК, РНК метилирование 27 и кооперативное связывание. Таким образом, высокие ложные положительные и ложные отрицательные темпы были зарегистрированы в течение многих алгоритмов 22,24,28. В то время как эти алгоритмы полезны для выявления возможных микроРНК целей для последующей экспериментальной проверки, эти высокие коэффициенты ошибок ограничить эффективность биоинформатики подходов к систематической микроРНК обнаружения цели.
Систематически исследовать взаимодействие между данной микроРНК и потенциально целевых 3'UTRs мы разработали высокой пропускной анализа под названием Люминесцентные Идентификация функциональных элементов в 3'UTRs (3'LIFE) 24. Этот анализ мер прямого взаимодействия и репрессии трансляции испытательного 3'UTR запросом микроРНК с использованием двойной системы люциферазы репортер. В этой системе, 3'-UTR из интересующего гена клонируют на выходе из люциферазы светлячка (fluc) репортера рамке считывания. Репортер минусыtruct котрансфицирована с запросом микроРНК в клетках HEK293T. МикроРНК нацеливание определяется путем измерения относительного изменения между испытательной fluc :: 3'UTR репортера и второй неспецифической репортера люциферазы Renilla. Важно отметить, что люциферазы анализы обнаружить функциональные взаимодействия микроРНК / мРНК, которые влияют на поступательное выход репортера. Это главное преимущество по сравнению с традиционными методами для обнаружения регулирования микроРНК, например, RT-КПЦР и западных пятна, что это обходит различия в деградации мРНК и репрессии трансляции, а также изменения в белковых изобилии независимого регулирования, основанного 3'UTR.
Люциферазы анализы широко используются для проверки прямых целей микроРНК из-за их относительной простоты и чувствительности, но их использование в экранах с высокой пропускной способностью ограничено высоких затрат, связанных с расходными реагентами, отсутствие 3'UTR библиотек из общедоступных источников, и отсутствие стандартизированных люциферазы протocols, что приводит к трудностям в сравнении функциональную репрессии по нескольким наборам данных. Для облегчения использования 3'LIFE анализа, мы делали акцент на упрощение экспериментального проектирования, использования некоммерческих трансфекции 24 и люциферазных реагентов 29, создавая 3'UTR библиотеку, которая регулярно обновляется и расширяется, и доступна через хранилище общественной плазмиды 30.
Масштабируемость 3'LIFE анализа позволяет скрининг большого 3'UTR библиотеки для нацеливания на заданное миРНК без смещения в направлении экрана bioinformatically идентифицированных генов. В дополнение к тестированию канонические и предсказанные взаимодействия, это системный подход позволяет выявить новых целей приводом с помощью неканонических и / или видов-специфических взаимодействий. Важно отметить, что эффект микроРНК ориентации на производство белка, как правило, понимается привести к скромной репрессии трансляции 15,31 </ SUP>, предполагая, что основная роль регулирования микроРНК является точной настройки выходного белка, защиты от аберрантных уровней экспрессии генов, а также обеспечить надежность на мобильные конкретные программы 32,33. Чувствительность анализа люциферазы в сочетании с изначально большим числом отрицательных взаимодействий микроРНК / мРНК в экране 3'LIFE позволяет обнаруживать тонких эффектов микроРНК ориентации на большом количестве генов, и идентификацию множества компонентов генных сетей, которые регулируются данной микроРНК 24.
Здесь мы опишем протокол 3'LIFE, и продемонстрировать это технико-экономическое путем скрининга два хорошо охарактеризованные микроРНК Мир-10b, и пусть-7c против панели 275 человека 3'UTRs (рисунок 1).
протокол
1. Культура клеток (24-48 ч до трансфекции)
- 24-48 ч до трансфекции семени достаточное количество HEK293T элементов на основе числа 96-луночных планшетах быть трансфицированы.
Примечание: В соответствии трансфекции, клетки пластины в достаточной плотности, чтобы способствовать быстрому деление, пока не будет более чем в 70-90% слияния во время трансфекции. - Каждый 96-луночный планшет требуется 9 х 10 6 клеток (75000 клеток на лунку, и 120 скважин на чашку для учета использования водоема и многоканальной пипетки). Вычислить время удвоения клеток НЕК293 (обычно ~ 20 час), и семян соответствующее количество клеток для получения по меньшей мере, 9 × 10 6 клеток на момент трансфекции. 145 мм круглая пластина культура, как правило, достаточно для 3 трансфекций 96-а, когда вырос до ~ 90% слияния, с ~ 10% клеток, оставшихся повторно заполнить новую пластинку.
2. Подготовка перед трансфекцией
_content "> Примечание: подготовка буферов и плазмидной ДНК на стадии 2,0-2,2 должна быть выполнена за несколько дней до трансфекции с подготовкой этих реагентов может занять много времени.- Клоны человека 3'UTR, которые совместимы с 3'LIFE анализа доступны через репозиторий общественного плазмиды 30. Очищают плазмиду ДНК вручную или с жидкими роботов обработки. Использование комплекта мини-Prep 96-а класса трансфекции щелочного лизиса и следуйте инструкциям изготовителя. Ресуспендируют очищенные векторы ~ 100 нг / мкл на лунку.
ПРИМЕЧАНИЕ: сигнал на вашем люциферазы приведет, если концентрация плазмиды падает ниже 40 нг / мкл. - Получить pLIFE-микроРНК векторы 30 или клон с помощью трубопровода на рисунке 1В 24. Ресуспендируют векторы в концентрации 500 нг / мкл каждого миРНК и пустой плазмиды управления.
- Благодаря чувствительности условиях nucleofection буферных убедитесь, чтоОбщий объем трансфицированных материалов (в том числе клеток и плазмиды), не превышает 10% от полного количества жидкости, в каждую лунку трансфекции 96-луночного планшета. Для достижения этой цели, концентрировать плазмиды запас pLIFE-микроРНК в концентрации менее 500 нг / мкл.
- Подготовка 10х буфера люциферазы светляков реагентов (таблица 1), и 1x Renilla люциферазы буферные реагенты (таблица 2), которые могут быть сохранены на срок до 6 месяцев.
Примечание: ДТТ в буфере светлячка люциферазы должны быть сохранены в растворе при -20 ° С в отдельных аликвот использования.
3. Подготовить следующие предметы непосредственно перед Трансфекцию
- Подготовка трансфекции буфер, содержащий PBS, 1,5% HEPES, рН 7,0. Подготовка эта свежая, хотя это может храниться до 1 месяца при 4 ° С без заметных уменьшается в эффективности трансфекции. При формулировании буфера и плазмидной ДНК объемы, предположим, 120 реакций для каждого 96-луночного планшета в Суfficiently зарегистрированы ошибки в пипетки и объема потерянной помощью хранилища жидких и многоканальные пипетки. Алиготе 18 мкл на лунку буфера трансфекции (120 скважин / 96-луночного планшета = 2,16 мл на чашку), и отложите в сторону.
Примечание: Сотовый электропорации устройства чрезвычайно чувствительны к условиям буфер, используемый для трансфекции клеток. Точность при подготовке буферы обеспечит стабильную работу оборудования. Особую осторожность следует проявлять при выполнении анализа для предотвращения испарения буферов, в частности, за счет минимизации времени, что буфер влево обнаружены в микропробирок, 96-луночные планшеты, водоемов, и электродных пластин. - Резервный четыре скважины для следующих элементов управления, нет pLIFE-3'UTR (для измерения фона люциферазы), pLIFE-SV40 3'UTR (отрицательный контроль целевого), положительный контроль для микроРНК # 1, положительный контроль для микроРНК # 2.
ПРИМЕЧАНИЕ: Эти векторы публично доступны 30. Кроме того, все ранее утверждена целеваяможет быть использован в качестве положительного контроля. - Теплые информации, трипсина (0,25%) до 37 ° С.
- В каждую лунку 96-луночного культурального планшета клеток, добавить 200 мкл DMEM, дополненной 10% FBS, 1% Pen / Strep, и помещают в 37 ° С инкубатор для использования после трансфекции.
- Включите все клетки электропорации устройств с последующим соответствующим программным обеспечением. Используйте импульсно-кодовой FF120 для HEK293T клеток и PBS / HEPES буфера.
4. Подготовка плазмидной ДНК и клеточной смеси
ПРИМЕЧАНИЕ: Следующий протокол предполагает трансфекции три 96-луночных в одном эксперименте для экрана с двумя микроРНК (miRNA- # 1 и # 2 miRNA-). Каждая пластина будет соответствовать той же 96-луночного планшета в pLIFE-3'UTR плазмид, и лечиться три раза pLIFE-микроРНК-пустой, pLIFE-miRNA- # 1, или pLIFE-miRNA- # 2.
- Подготовьте 3 запасы pLIFE-микроРНК + буфер трансфекции для каждого микроРНК. Эта акция должна составлять 50% (10 мкл) от общего объема каждогохорошо, умноженное на 120 скважинах. Таким образом, каждая акция должна содержать 1,08 мл буфера + 120 мкл ДНК плазмиды (pLIFE-микроРНК).
- Удалить клетки от 145 мм планшет для культивирования при элюировании носитель, промывки PBS осторожно и обработки ~ 5 мл 0,25% трипсина в течение 5 мин при 37 ° С. Нейтрализовать трипсина с равным объемом информации, и гранул клеток в 300 мкг в течение 5 мин.
- Удалить трипсин / СМИ и ресуспендируют осадок в ~ 5-10 мл средства массовой информации (в зависимости от плотности клеток и точного диапазона счетчика клеток).
- Граф клеток с использованием клеток счетчика. Убедитесь, что клетки> 95% жизнеспособным и в точной диапазоне машины.
Примечание: неточно количество клеток может быть результатом очень высокой концентрации клеток (> 6.0x 10 6 / мл). Трансфекции клетки слишком много может резко снизить эффективность миРНК ориентации за счет снижения плазмиды: соотношение клеток и / или снижению эффективности трансфекции. - Кратные три пробирки, содержащие каждый 9 х 10 6 клеток, соответствующих клетках Required для трансфекции одного 96-луночного планшета. Спин клетки при 300 мкг в течение 3 мин.
- Удалить СМИ. Будьте уверены, чтобы удалить как можно больше средств массовой информации, как это возможно с минимальным нарушением таблетки, как избыток информации может повлиять эффективности трансфекции.
- Ресуспендируют клеток в 1,2 мл буфера трансфекции / микроРНК плазмиды смеси, и отставьте в сторону.
- Ниже подробно ресуспендирование pLIFE-3'UTR плазмиды трансфекции буфера. Как это происходит в 96-луночных, заботиться, чтобы избежать испарения буфера, покрывая пластины во все времена.
- Используя многоканальную пипетку, переместить 32,4 мкл буфера трансфекции в каждую лунку 96-луночного ПЦР пластины (9 мкл [за трансфекции] * [3 пластины] * 1.2 [для учета погрешности пипетки]).
- Добавить 3,6 мкл (~ 100 нг / мкл) мини-нацелен pLIFE-3'UTR плазмиды в каждую лунку и тщательно перемешать.
- Пипетка 10 мкл этой смеси в каждую лунку трансфекции пластины и крышки 96-луночного.
5. Трансфекция
- Перемещение 1,2 мл первой ячейки буфера / / pLIFE-микроРНК плазмиды смеси в резервуаре. Хорошо перемешать.
- Добавить 10 мкл этой смеси в первой трансфекции 96-луночного планшета, уже содержащего 10 мкл буфера трансфекции / pLIFE-3'UTR. Все хорошо перемешать с помощью пипетки вверх и вниз несколько раз.
Примечание: равное суспензию клеток в буфере будет гарантировать равномерное и тщательное прохождение электрического тока через кювету и максимизировать эффективность трансфекции. - Поместите трансфекции 96-луночного планшета на сотовый электропорации устройства и инициировать трансфекции.
- После трансфекции завершена, добавить 100 мкл подогретого СМИ из культуральной 96-луночного планшета в каждую лунку 96-луночного трансфекции пластины и хорошо перемешать. Перемещение по 100 мкл из каждой лунки в культуральную 96-луночного планшета.
- Смешайте клетки в культуральной пластины с пипетки, расположенной вертикально в центре скважины, а клетки будут иметь тенденцию к агрегации на сторонах WELL, если не смешивать правильно.
- Повторите 5,1-5,5 для остальных двух пластин.
- Очистка трансфекции 96-луночный планшет
- Трансфекции 96-луночные могут быть переработаны путем промывки 70% этанола, чтобы убедиться в отсутствии перенести нуклеиновых кислот между экспериментами. Выполнение двух 70% этанола моет помощью пульверизатора, чтобы полностью заполнить каждую лунку, после чего протирки избыток EtOH на электродных полос (нижняя сторона) и позволяет трансфекции пластины полностью высохнуть в капот культуры.
Примечание: Мы протестировали для уноса загрязнений ДНК путем трансфекции 12 скважин с 2 мкг плазмидной pmaxGFP друг в клетках HEK293T, после чего одной промывки 70% этанолом, и второй трансфекции не плазмидной ДНК. С этой очень высокой концентрации плазмиды, очень яркий репортера, и одной стирки, не было наблюдаемой флуоресценции в любом из 12 повторных трансфекции.
- Трансфекции 96-луночные могут быть переработаны путем промывки 70% этанола, чтобы убедиться в отсутствии перенести нуклеиновых кислот между экспериментами. Выполнение двух 70% этанола моет помощью пульверизатора, чтобы полностью заполнить каждую лунку, после чего протирки избыток EtOH на электродных полос (нижняя сторона) и позволяет трансфекции пластины полностью высохнуть в капот культуры.
- Культуры клеток в течение 48-72 ч при 37 ° С, а затем двойной lucifстереть анализа.
6. клеточного лизата Подготовка к люциферазы
- Развести буфера для лизиса с 4 частями воды, 1 часть 5-кратным пассивного буфера для лизиса в резервуар. Рассчитать 26 мкл / лунку, добавляя ~ 20 дополнительных томов к ответственности за потери в водохранилище.
ПРИМЕЧАНИЕ: Буфер хранили при -20 ° С и может быть чрезвычайно вязким, таким образом, до позволяя 5x буфер подойти комнатной температуры будет повысить точность пипетки. - Анализ каждую лунку для повышения эффективности трансфекции с использованием флуоресцентной микроскопии. Обратите внимание на любые несоответствия в скважинах, которые не трансфецировать эффективно (> 90% эффективности трансфекции), или выражая низкие уровни ППП индикативного переполненности или медиа истощения. Удалить эти скважины из анализа.
- Полностью извлеките носитель из клеток, стараясь не слишком быстро вымывается, которые будут вызывать клетки, чтобы отсоединить.
ПРИМЕЧАНИЕ: Остальные СМИ будут разбавлять лизат и вызывают колебания значений по experimeНТС. - Добавить 26 мкл буфера для лизиса в каждую лунку, и место на тарелке шейкера / рокера при низкой / средней скорости для ~ 20-30 мин. Используйте это время, чтобы подготовиться люциферазы буферы, вымыть и премьер люминометра (ы), и передать лизата непрозрачных пластин измерений.
7. Двойной анализа люциферазы
ПРИМЕЧАНИЕ: Если несколько пластин измеряется последовательно на одном люминометра, создать буферные смеси мастер со всем, кроме АТФ и субстратов, добавив эти реагенты с последующим доведением рН непосредственно перед использованием с каждой пластины. АТФ и субстраты могут со временем ухудшаться; последовательность в количестве времени, эти реагенты в буфере улучшит согласованность между несколькими пластинами.
- Приготовьте 1х буферов люциферазы (Таблица 1):
- Оберните две трубы (обычно 15/50 мл центрифужные пробирки), содержащие светлячка и Renilla буферов с алюминиевой фольгой, в качестве подложек могут быть чувствительны к свету.
- Приготовьте 1х люциферазы светляков буфер. Добавить 1 мл каждого из пяти 10x люциферазы светлячка реагентов, добавлением EGTA наконец, 5 мл Н 2 0 до конечной концентрации 1х.
- Добавить 0,025 г АТФ до 10 мл 1x светлячка буфера. Смешайте путем обращения в несколько раз. Держите АТФ на льду во все времена. АТФ будет деградировать, так что если измерения более одной пластины последовательно, буфер должен быть свежим начало на этом этапе для каждого дополнительного пластины.
- Добавить 100 мкл 100 жука люциферина (подложки) (таблица 1) Буфер должен измениться на желтоватого цвета на основе рН.
- 1x Renilla люциферазы буфера восстановление: Пер 96-луночного планшета, аликвоты 10 мл 1 "Renilla буфер".
- Добавить 100 мкл BSA (44 мг / мл стоковый).
- Если скрининг более одной пластины, отдельный мастер смеси в 10 мл аликвотах.
- Добавить 100 мкл буфера для коэлентеразином (ранее аликвоты и хранили при 100x конц.)
- Регулировка рН 1x светлячка буфер до 8,0, после чего буфер 1x Renilla до 5,0 с помощью NaOH и HCl.
Примечание: активность каждого буфера, а также возможность буфера Renilla, чтобы подавить активность люциферазы Firefly сильно зависит от рН. Для получения стабильных результатов, чрезвычайно точны в этом шаге. - Доведите объем каждого буфера (соответствующий 1 96-луночный планшет) в 10,5 мл, чтобы приспособить для фотометра грунтовки.
- Перевести лизата непрозрачных белых пластин: В этот момент клетки должны быть в буфере для лизиса для ~ 20 мин. Возьмите 25 мкл из каждой лунки, используя многоканальную пипетку, обязательно пипеткой вверх и вниз тщательно, чтобы разбить скопления клеток и гомогенизации лизат.
- Подготовка люминометра. Включите фотометр и выберите протокол в папку DLR, под названием "DLR с двумя инъекциями". Другие форматы не совместимы с анализа данных трубопровода (ниже).
- Выберите скважин быть тЗаинтересованное (все скважины является по умолчанию).
- Продлить 'задержки перед измерением' в значение 5 секунд, со временем измерения 10 сек (см 29 для объяснения).
- Капиллярные шаги мыть: вода 3x, 3x этанола, воды 3x 3x, сухие. Премьер буферы раз в отходах, а затем премьер-второй раз назад в буфер труб для обеспечения смешивания. Вводите светлячка буфер первый и премьер его в левом капилляра, а затем Renilla в правильном капилляра.
- Инициировать люциферазы. Каждая пластина должна принять ~ 48 минут, чтобы читать. После завершения сохранения файла, а затем повторите шаги мыть и выключить люминометра.
ПРИМЕЧАНИЕ: Несколько пластин можно прочитать и данные, хранящиеся на файл преуспеть же, однако проблемы могут встречаться с несколькими плите, гласит, где программа люминометра рухнет. Будьте уверены, чтобы сохранить все данные между измерениями и делать скриншоты, если программа закроется, прежде чем сохранения возможно.- Замените старые буферыс новым, будучи уверенным, что премьер-крайней мере, в два раза с новых буферов перед началом новой пластинкой.
Анализ 8. Данные
- Использование таблиц Excel "3'LIFE - одноместное анализа тарелка" и "3'LIFE - анализ многодисковое" доступны www.mangonelab.com . Скопируйте исходные данные для светлячков и Renilla люциферазных измерений из выходного файла люминометра в местах, соответствующих негативном состоянии, микроРНК # 1, и микроРНК # 2 в "3'LIFE - одноместное анализа пластина" таблицы.
- Таблица будет автоматически рассчитывать светлячка / соотношение Renilla и нормализовать каждый Мирна к соответствующему отрицательного контроля, и нормализовать значения репрессий по каждой пластины.
ПРИМЕЧАНИЕ: Эта таблица будет автоматически определять скважин с низким уровнем сигнала люциферазы, выделите значительно репрессированных скважин, а также обеспечить меры repressiна по всей пластине. См 24 для детального объяснения статистического анализа.- Копирование значения из коробки "стандартизированный индекс РИ", в соответствующих клетках в "3'LIFE - анализ многодисковой" таблицы в положении, соответствующем повторить # 1 для каждого тестируемого миРНК. Повторите для всех биологических повторяет выполненных в разные дни.
- При желании, вставить имена генов и предсказания состояния цель в колонках В и D, соответственно.
- Разрешить таблицы автоматически вычислить среднее всех пластине. Соблюдайте данные в 96-луночного формата, как тепловой карте (рис 2). Файл также организует данные в виде списка (рисунок 2).
ПРИМЕЧАНИЕ: индекс репрессии мера используется для идентификации предполагаемых целей микроРНК. Различные параметры жесткости могут быть использованы, в зависимости от индивидуальных предпочтений. В среднем мы считаем вероятные хиты ниже репрессий индексом 0.8 и статистически значимым по Стьюдента (р <0,05 -Value). Как показано на рисунке 3 потенциальных задачи на основе этих критериев будут выделены красным цветом.
- Разрешить таблицы автоматически вычислить среднее всех пластине. Соблюдайте данные в 96-луночного формата, как тепловой карте (рис 2). Файл также организует данные в виде списка (рисунок 2).
Результаты
Выход люминометра файл содержит сырые измерения для обоих светлячков и Renilla люциферазных белков. Это сырье формат совместим с "3'LIFE - одноместное анализа пластины" и "3'LIFE - многодисковое анализа" электронных таблиц доступной из лаборатории сайт Mangone ( www.mangonelab.com ). Одну таблицу анализ пластина автоматически вычисляет светлячка / соотношение Renilla, нормализует каждый Мирна к соответствующему отрицательного контроля, нормализует значения репрессий по каждой пластины. Эта таблица автоматически определяет скважин с низким Renilla люциферазы сигнала, подчеркивает скважин, которые обладают репрессии по сравнению с отрицательным контролем, и обеспечивает репрессии по всей пластине (рисунок 2). См 24 для детального объяснения статистического анализа.
Несколько повторов можно анализироватьд с помощью "анализа многодисковое" таблицу. Каждый повторной сравнивается бок о бок, и статистические показатели данных рассчитываются автоматически (рисунок 3). В дополнение к сравнению повторов с «нормализованной репрессий" колонки, пользователь может сравнить репрессии между двумя микроРНК под "микроРНК # 1 / # 2 микроРНК" столбцов. Эта мера делит индекс репрессии для каждого микроРНК для каждой повторности. Эта мера может указывать ошибочных значений из люциферазы (например аномально высокие или низкие показания с отрицательным контролем, рисунок 3, строка A9, Rep # 3), и лунки, где индекс репрессии не могут указывать значительную репрессии, но, что делать демонстрируют существенные различия между микроРНК. В то время как эта мера не может быть использован непосредственно для указания цели микроРНК, это полезно для выявления выбросов, проблемные скважины, или закономерности в данных, которые не только отнести направить микроРНК Regulation.
Репрессии индекс (RI), используется для вызова предполагаемый микроРНК цель, с более низкими значениями, соответствующими высокой относительной репрессий. Порог для вызова предполагаемых целей на основе уровня жесткости, требуемого исследователя, но комбинируя RI с 3'UTRs, которые отображают статистически значимые р-значения (р <0,05) укажет высокое доверие цели (рис 2 Строки В8 и В12).

Рисунок 1: 3'LIFE анализ (А). Шлюз-совместимые векторы, используемые в анализе 3'LIFE. Вверху: ген люциферазы (FLuc) слит с 3'-UTR тестовой, в то время как ген люциферазы Renilla (RLuc) слит с неспецифической SV40 рА 3'-UTR в качестве контроля. Внизу: RFP- микроРНК-интрон вектор - зонд предварительно микроРНК, плюс ~ 400нуклеотидов в пределах своей геномной локуса (резюмировать эндогенного обработку микроРНК), клонируют в интрона, чтобы его сотрудничество с выражением DsRed2 флуорохромом. Оба вектора публично доступны (Зайлер и др., 2013). (Б) Блок-схема на 3'LIFE анализе. (С) 3'LIFE трубопроводов: двойной люциферазы вектор, содержащий тестовый 3'UTR с или без векторов микроРНК ко-трансфицировали в клетки HEK293 в 96-луночных планшетах. Взаимодействие между микроРНК и добросовестного 3'UTR цели снизит относительную свечение в отдельных скважинах (на примере оранжевым пятном в экспериментальной пластины).

Рисунок 2: Пример данных, полученных с 3'LIFE анализа. Каждый зонд микроРНК тестируется в четырех (повторяет 1-4). Цвета представляют уровни репрессий, с краснымцвета, указывающие сильный микроРНК / 3'UTR взаимодействия. Все повторности усредняются для получения высокого качества предполагаемых целей, показанные в итоговом пластины ниже желтой стрелкой. Белая коробка представляет управления, не удалось трансфекций или скважин с низкой эффективностью трансфекции.
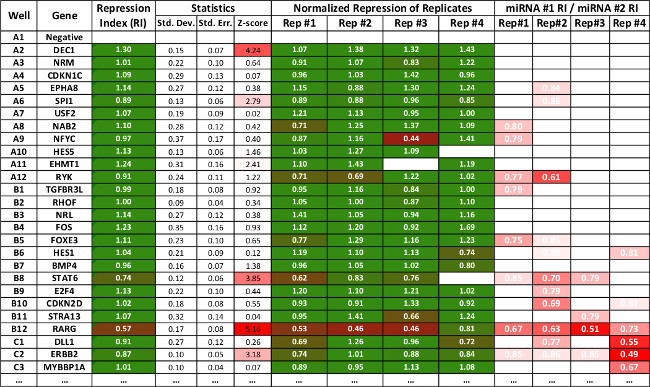
Рисунок 3:. Таблица, представляющая сводные данные подмножества взаимодействий, произведенных с использованием 3'LIFE таблицу Значения репрессии являются как показано на рисунке 2. Программа вычисляет стандартное отклонение, стандартная ошибка и Z-счет для каждого взаимодействия. Статистически значимых взаимодействий отмечены красным. Последние четыре строки показывают относительную репрессии одной микроРНК в друга, и используется в качестве вторичного показателя для сравнения репрессии между двумя различными микроРНК. Таблицы могут быть загружены с www.mangonelab.com
Таблица 1: Фото люциферазы светляков реактивы: 10x Исходные растворы глицилглицин, К х PO 4, MgSO 4, DVB-T и ЭГТА могут быть получены отдельно и хранятся до буфера восстановления. 100x Жук Люциферин (люциферазы светляков субстрат) может быть магазинd путем растворения 50 мг люциферин в 7,134 мл H 2 0 (25 мм). Алиготе 105 мкл / чашку растворенного Beetle люциферина в пробирки и хранят при -80 ° С. За технической поддержкой Промега, это должно быть стабильным в течение> 6 месяцев, но может быть чувствительна к свету. ПРИМЕЧАНИЕ: ЭГТА не переходят в раствор при нейтральном рН. Медленно добавляют NaOH до EGTA, пока он не полностью растворяется. * Реагенты добавл ли до конечных буфере непосредственно перед люциферазы
| Renilla люциферазы буферные реагенты | Конечная концентрация (1x) |
| NaCl | 1.1 М |
| На 2 ЭДТА | 2,2 мм |
| KH 2 PO 4 | .22 М |
| NaN 3 | 1,3 мм |
| БСА * | 0,44 мг / мл |
| Коэлентеразин * | 2.5 & #956; М |
Таблица 2: сток Renilla люциферазы буферные реагенты Все реагенты, кроме BSA и коэлентеразином могут быть смешаны в концентрации 1х и хранили при комнатной температуре. Коэлентеразином можно растворить в подкисленным метанолом и аликвоты каждой чашке. Подкислите метанол добавлением HCl до конечной концентрации 5 мМ (<3 рН). Растворите 250 мкг коэлентеразин в 2,36 мл метанола подкисляют (250 мкМ) аликвотные 105 мкл / пластина. Смесь стабильна в течение по крайней мере 6 месяцев, но может быть чувствительна к свету. * Реагенты добавляют в буфер непосредственно перед люциферазы.
Обсуждение
3'LIFE анализ выявляет функциональные цели микроРНК в 3'UTRs в высокой пропускной. Этот анализ является полезным для исследователей, которые хотят, чтобы экспериментально определить большое количество предполагаемых целей их микроРНК интерес. 3'LIFE анализ является мощным подход для запроса 3'UTR приводом регулирования, в том анализ обеспечивает функциональную меру микроРНК адресности и двоичный тестирование одного репортера :: 3'UTR против одного микроРНК можно с уверенностью решения таргетинг состояние отдельных генов. Для проверки этого подхода, мы провели скрининг панель 275 3'UTRs и против двух микроРНК, пусть-7с и Мир-10b, и включены 10 ранее утвержденные целевые гены в этой библиотеке. Восемь из этих десяти генов выставлены репрессии 24. Мы также наблюдали значительное обогащение необоснованных bioinformatically прогнозируемых целей, и непредвиденных 3'UTRs, которые содержат канонические элементы семян среди наших топ-хитов, SuGgesting что 3'LIFE способна определять добросовестных микроРНК целей.
Ключевым показателем чувствительности экранов высокой пропускной это ложные положительные и ложные отрицательные темпы. В то время как процент ложных срабатываний этого анализа необходимо оценить с помощью дополнительных альтернативных подходов для проверки хиты, восемь из десяти положительных контролей включены в наш доказательством правильности принципа экран выставленный репрессий, предлагая ложные отрицательные темпы 20%. Тем не менее, многие методы используются для идентификации микроРНК цели в различных клеточных контекстах, и обработка 3'-UTR и регулирование транс-действующих факторов, как известно, высоко специфический ткани. Например, большинство 3'UTRs содержать несколько сайты полиаденилирования, которые контролируют длину 3'-UTR в зрелой мРНК. Во многих случаях использование проксимальных участках полиаденилирования специфичен ткани, и может исключить микроРНК целевых сайтов. Кроме того, кооператив таргетинг микроРНК, конкуренция Wiй РНК-связывающие белки, и мРНК вторичная структура может все это влияет способность 3'LIFE обнаруживать цели микроРНК в определенных тканях. Из-за этого, обнаружение мишеней в анализе 3'LIFE может изменяться в зависимости от клеточного контекста, в котором осуществляется анализ, усложняя оценку абсолютных коэффициентов ошибок. Этот протокол оптимизирован для HEK293T клеток, но альтернативные клеточные линии могут быть использованы, если исследователь хочет выполнить анализ в специфической биологической контексте. Тем не менее, оптимизации эффективности трансфекции и выживание клеток с каждой клеточной линии должны быть оптимизированы с использованием нескольких буферных условий, импульсные коды, и количество клеток. Пример схемы оптимизации можно найти на Wolter соавт. 24.
Этот протокол был оптимизирован в формате 96-луночного и определяет использование определенной высокой пропускной приборов. В случае, учреждение не имеет оборудование, необходимое для 96Ну Nucleofection, альтернативные реагенты трансфекции может быть использован для выполнения анализа 3'LIFE, при условии, что эффективность трансфекции остается высокой. Кроме того, анализ люциферазы является наиболее трудоемким аспектом 3'LIFE анализа. Таким образом, использование нескольких люминометров рекомендуется для экранов высокой пропускной.
Раскрытие информации
This work is supported by funds from the College of Liberal Arts and Science and the Biodesign Institute at Arizona State University and NIH Exploratory/Developmental Research Grant 1R21CA179144-01A1.
Благодарности
We thank Stephen Blazie, Karen Anderson, Josh LaBaer for advice and discussion. Karen Anderson, John Chaput, and Josh LaBaer for sharing reagents and instrumentation, Michael Gaskin and Andrea Throop for technical advise and protocols. Justin Wolter is a Maher scholar and thanks the Maher family for their generous support.
Материалы
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| glycylglycine | Sigma | G1127-25G | |
| Kx PO4 | Sigma | P2222 | |
| EGTA | Sigma | E3889 | |
| ATP | Sigma | FLAAS | |
| DTT | Sigma | D0632 | |
| MgSO4 | Sigma | M7506 | |
| CoA | Sigma | C4282 | |
| luciferin | Sigma | L9504 | |
| NaCl | Sigma | S7653 | |
| Na2EDTA | Sigma | E0399 | |
| K H2 P O4 | Sigma | 1551139 | |
| BSA | Sigma | A2153 | |
| NaN3 | Sigma | S2002 | |
| Coelenterazine | Sigma | C3230 | |
| PBS/HEPES | Corning | 21-040-CV | |
| DMEM | Sigma | D5546 | |
| FBS | Sigma | F2442 | |
| Pennicilin/Streptomicin | Sigma | P4333 | |
| Trypsin | T2600000 | ||
| Consumables | |||
| MaxiPrep Kit | Promega | A2392 | |
| 96-well miniprep plate | Pall | 8032 | |
| 96-well shuttle plates | Lonza | V4SP-2096 | |
| 5x Lysis Buffer | Promega | E1941 | |
| Instruments | |||
| 96-well GloMax Plate Reader | Promega | E9032 | |
| Biomech FX Liquid Handler Robot | Beckmann | A31842 | |
| 4D-Nucleofector Core Unit | Lonza | AAF-1001 | |
| 96-well Shuttle System | Lonza | AAM-1001 | |
| Cell Counter Countess | Invitrogen | C10227 |
Ссылки
- Bartel, D. P. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell. 116 (2), 281-297 (2004).
- Pasquinelli, A. E., et al. Conservation of the sequence and temporal expression of let-7 heterochronic regulatory RNA. Nature. 408 (6808), 86-89 (2000).
- Reinhart, B. J., Weinstein, E. G., Rhoades, M. W., Bartel, B., Bartel, D. P. MicroRNAs in plants. Genes Dev. 16 (13), 1616-1626 (2002).
- Ivey, K. N., et al. MicroRNA regulation of cell lineages in mouse and human embryonic stem cells. Cell Stem Cell. 2 (3), 219-229 (2008).
- Lee, R. C., Feinbaum, R. L., Ambros, V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 75 (5), 843-854 (1993).
- Wienholds, E., et al. MicroRNA expression in zebrafish embryonic development. Science. 309 (5732), 310-311 (2005).
- Darnell, D. K., et al. MicroRNA expression during chick embryo development. Dev Dyn. 235 (11), 3156-3165 (2006).
- Jin, P., Alisch, R. S., Warren, S. T. RNA and microRNAs in fragile X mental retardation. Nat Cell Biol. 6 (11), 1048-1053 (2004).
- Kim, J., et al. A MicroRNA feedback circuit in midbrain dopamine neurons. Science. 317 (5842), 1220-1224 (2007).
- Poy, M. N., et al. A pancreatic islet-specific microRNA regulates insulin secretion. Nature. 432 (7014), 226-230 (2004).
- Calin, G. A., Croce, C. M. MicroRNA signatures in human cancers. Nature Reviews Cancer. 6 (11), 857-866 (2006).
- Friedman, R. C., Farh, K. K., Burge, C. B., Bartel, D. P. Most mammalian mRNAs are conserved targets of microRNAs. Genome Res. 19 (1), 92-105 (2009).
- Paraskevopoulou, M. D., et al. DIANA-microT web server v5.0: service integration into miRNA functional analysis workflows. Nucleic Acids Res. 41 (Web Server issue), W169-W173 (2013).
- Krek, A., et al. Combinatorial microRNA target predictions. Nat Genet. 37 (5), 495-500 (2005).
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136 (2), 215-233 (2009).
- Vella, M. C., Choi, E. Y., Lin, S. Y., Reinert, K., Slack, F. J. The C. elegans microRNA let-7 binds to imperfect let-7 complementary sites from the lin-41 3'UTR. Genes Dev. 18 (2), 132-137 (2004).
- Lal, A., et al. miR-24 Inhibits cell proliferation by targeting E2F2, MYC, and other cell-cycle genes via binding to 'seedless' 3'UTR microRNA recognition elements. Molecular Cell. 35 (5), 610-625 (2009).
- Cevec, M., Thibaudeau, C., Plavec, J. N. M. R. NMR structure of the let-7 miRNA interacting with the site LCS1 of lin-41 mRNA from Caenorhabditis elegans. Nucleic Acids Res. 38 (21), 7814-7821 (1093).
- Shin, C., et al. Expanding the microRNA targeting code: functional sites with centered pairing. Molecular Cell. 38 (6), 789-802 (2010).
- Azzouzi, I., et al. MicroRNA-96 directly inhibits gamma-globin expression in human erythropoiesis. PLoS One. 6 (7), e22838(2011).
- Chen, J., et al. miR-193b Regulates Mcl-1 in Melanoma. Am J Pathol. 179 (5), 2162-2168 (2011).
- Chi, S. W., Hannon, G. J., Darnell, R. B. An alternative mode of microRNA target recognition. Nat Struct Mol Biol. 19 (3), 321-327 (1038).
- Jiao, L. R., et al. MicroRNAs targeting oncogenes are down-regulated in pancreatic malignant transformation from benign tumors. PLoS One. 7 (2), e32068(2012).
- Wolter, J. M., Kotagama, K., Pierre-Bez, A. C., Firago, M., Mangone, M. 3'LIFE: a functional assay to detect miRNA targets in high-throughput. Nucleic Acids Res. , (2014).
- Mangone, M., et al. The landscape of C. elegans 3'UTRs. Science. 329 (5990), 432-435 (2010).
- Ekdahl, Y., Farahani, H. S., Behm, M., Lagergren, J., Ohman, M. A-to-I editing of microRNAs in the mammalian brain increases during development. Genome Res. 22 (8), 1477-1487 (2012).
- Fu, Y., Dominissini, D., Rechavi, G., He, C. Gene expression regulation mediated through reversible m(6)A RNA methylation. Nat Rev Genet. 15 (5), 293-306 (2014).
- Zhou, P., et al. Large-scale screens of miRNA-mRNA interactions unveiled that the 3'UTR of a gene is targeted by multiple miRNAs. Plos One. 8 (7), e68204(2013).
- Dyer, B. W., Ferrer, F. A., Klinedinst, D. K., Rodriguez, R. A noncommercial dual luciferase enzyme assay system for reporter gene analysis. Anal Biochem. 282 (1), 158-161 (2000).
- Seiler, C. Y., et al. DNASU plasmid and PSI:Biology-Materials repositories: resources to accelerate biological research. Nucleic Acids Res. , (2013).
- Baek, D., et al. The impact of microRNAs on protein output. Nature. 455 (7209), 64-71 (2008).
- Ebert, M. S., Sharp, P. A. Roles for MicroRNAs in Conferring Robustness to Biological Processes. Cell. 149 (3), 515-524 (2012).
- Pelaez, N., Carthew, R. W. Biological robustness and the role of microRNAs: a network perspective. Current Topics in Developmental Biology. 99, 237-255 (2012).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены