Method Article
Detecção de alvos miRNA em métodos de alta capacidade utilizando o ensaio 3'LIFE
Neste Artigo
Resumo
Luminescent identification of functional elements in 3’ untranslated regions (3’UTRs) (3’LIFE) is a technique to identify functional regulation in 3’UTRs by miRNAs or other regulatory factors. This protocol utilizes high-throughput methodology such as 96-well transfection and luciferase assays to screen hundreds of putative interactions for functional repression.
Resumo
Luminescente Identificação de elementos funcionais em 3'UTRs (3'LIFE) permite a rápida identificação de alvos de miRNAs específicos dentro de uma matriz de centenas de 3'UTRs consultados. Identificação do alvo baseia-se no ensaio de luciferase dupla, que detecta a ligação ao nível do ARNm por medição da produção de translação, dando uma leitura funcional de miARN alvo. 3'LIFE usa buffers não-proprietários e reagentes, e bibliotecas repórter publicamente disponíveis, tornando as telas do genoma viável e rentável. 3'LIFE pode ser realizada quer num ambiente de laboratório padrão ou ampliados usando robôs de manipulação de líquidos e outros instrumentos de alto rendimento. Nós ilustrar a abordagem que utiliza um conjunto de dados de 3'UTRs humanos clonados em placas de 96 alvéolos, e duas miARNs de teste, deixa-7c e miR-10b. Nós demonstramos como realizar a preparação de DNA, transfecção, cultura de células e ensaios de luciferase em formato de 96 poços, e fornecer ferramentas para dadosanálise. Em conclusão 3'LIFE é altamente reprodutível, rápida, sistemática e identifica alvos elevados de confiança.
Introdução
O objetivo geral deste método é detectar e mapear precisamente microRNA (miRNA) em alvos de alto rendimento. MiRNAs são ARNs endógeno não-codificantes ~ 22 nucleótidos de comprimento. Após a transcrição e processamento, miARNs maduros são incorporadas num complexo proteína chamada o ARN de silenciamento induzido complexo (RISC). Cada miRNA orienta o RISC de elementos localizados principalmente nas regiões 3'untranslated (3'UTRs) de RNA mensageiro (mRNA) alvo, resultando em repressão ou tradução ou clivagem mRNA 1. Mirna reconhecer sites de destino com base no padrão de Watson-Crick e G: U oscilar base emparelhamento, e são degenerados na natureza, que contém vários pares de bases incompatíveis e regiões incharam. Muitos miARNs são amplamente conservada de plantas para os seres humanos 2,3, em que desempenham uma grande variedade de funções biológicas. Em metazoans miRNAs pode influenciar vários processos biológicos incluindo as decisões do destino da célula 4, o calendário de desenvolvimento 5 E, freqüentemente, apresentam padrões de expressão específicos de tecidos 6,7. MiARN misexpression também pode resultar na regulação do gene aberrante, que pode ter uma influência significativa no comportamento das células com base unicamente na função de genes alvo. Como tal, miARNs estão ligadas a uma ampla gama de doenças, incluindo a neurodegeneração 8,9, diabetes 10 e 11 do cancro. Bioinformática e abordagens molhado de bancada sugerem que cada miARN pode ser capaz de se direccionar centenas a milhares de ARNm distintas 12-14, o que indica que de alto rendimento ou do genoma abordagens de largura são necessários para investigar este grande grupo de potenciais interacções.
Identificar genes-alvo é um componente crítico de mechanistically definindo função miRNA, e para isso os pesquisadores devem ser capazes de revelar alvos em grande escala. Várias abordagens foram desenvolvidas para identificar alvos de miARN, incluindo algoritmos de previsão de bioinformática, sequenciação de alto rendimentodo ARNm alvo, e ensaios baseados repórter. Cada uma dessas abordagens tem pontos fortes e fraquezas inerentes. Dado que miARN direccionamento é guiado pela especificidade da sequência, especialmente de 2-6 nucleótidos de miARN (denominada região da semente), vários algoritmos foram desenvolvidos para prever alvos miARN ao longo do genoma de muitos organismos. Esses algoritmos são treinados usando os motivos de emparelhamento de bases observados de alvos miRNA validados, e freqüentemente utilizam parâmetros como emparelhamento rigorosas de sementes, conservação de sítios, e / ou estabilidade termodinâmica 15. Embora esses filtros refinar o grande número de possíveis alvos com complementaridade suficiente para destinos somente altos de confiança, eles podem excluir espécies locais específicos e não-canônicos alvo de miRNA, evidências recentes sugerem que são difundidos 16-24. Além disso, estas previsões não têm em conta os mecanismos de processamento de mRNA que excluem locais alvo de miRNA, tais como poliadenilação alternativa25, edição de 26 de RNA, RNA metilação 27, e ligação cooperativa. Como tal, as altas taxas positivos e falsos negativos falsos têm sido relatados por muitos algoritmos 22,24,28. Enquanto estes algoritmos são úteis para identificar candidatos alvos miRNA para validação experimental posterior, estas taxas de erro elevadas limitar a eficácia de abordagens de bioinformática para miRNA detecção do alvo sistemático.
Para sondar sistematicamente as interacções entre uma dada miARN e 3'UTRs potencialmente segmentados temos desenvolvido um ensaio de elevado débito chamados luminescentes Identificação de elementos funcionais na 3'UTRs (3'LIFE 24). Este ensaio mede interacções directas e repressão de translação da 3'UTR de teste por uma consulta miARN utilizando um sistema repórter de luciferase duplo. Neste sistema, o 3 'UTR de um gene de interesse é clonado a jusante da luciferase (FLUC) repórter grelha de leitura do pirilampo. Os contras repórtertruct é co-transfectado com uma consulta de miARN em células HEK293T. MiARN direccionamento é determinada pela medição da alteração relativa entre o FLUC teste :: repórter 3'UTR e um segundo não específica repórter de luciferase de Renilla. Importante, ensaios de luciferase detectar interacções funcionais miARN / ARNm que influenciam a saída de translação do repórter. Esta é uma das principais vantagens sobre os métodos tradicionais para detectar regulamento miRNA, como RT-qPCR e Western blot, em que este ignora diferenças na degradação do mRNA e repressão translacional, bem como mudanças na abundância de proteína independente de regulação baseada 3'UTR.
Os ensaios de luciferase são amplamente utilizados para validar alvos diretos de miRNA por causa de sua relativa simplicidade e sensibilidade, mas seu uso em telas de alto rendimento é limitado por elevados custos associados com reagentes de consumo, a falta de bibliotecas 3'UTR de fontes públicas, ea ausência de prot luciferase padronizadaocols, levando a dificuldades na comparação de repressão funcional em vários conjuntos de dados. Para facilitar a utilização do ensaio 3'LIFE, nós colocamos ênfase na simplificação do design experimental, a utilização de transfecção 24 e luciferase reagentes não-comerciais 29, criando uma biblioteca 3'UTR que é regularmente atualizada e expandida, e está disponível através um repositório plasmídeo público 30.
A escalabilidade do ensaio 3'LIFE permite o rastreio de uma grande biblioteca 3'UTR para segmentação por um determinado miRNA sem polarização da tela em direção genes bioinformatically identificados. Além de testar interacções canónicos e preditos, esta abordagem permite a identificação sistemática de novos alvos impulsionados através de interacções não canónicos e / ou específica da espécie. Importantemente, o efeito da miARN alvo na produção de proteína é geralmente entendida para resultar em modesta repressão translacional 15,31 </ Sup>, sugerindo que um papel principal de regulação miRNA é para afinar saída proteína, proteger contra os níveis de expressão de genes aberrantes, e proporcionam robustez a célula programas específicos 32,33. A sensibilidade do ensaio de luciferase combinado com o inerentemente grande número de interacções miARN / ARNm negativos na tela de 3'LIFE permite a detecção de efeitos subtis da miARN alvo em um grande número de genes, e a identificação de múltiplos componentes de redes de genes que são regulados por um determinado miRNA 24.
Aqui nós descrevemos o protocolo 3'LIFE, e demonstrar que é viabilidade pela triagem dois miRNAs bem caracterizados, miR-10b e deixe-7c contra um painel de 275 3'UTRs humanos (Figura 1).
Protocolo
1. Cultura celular (24-48 h antes da transfecção)
- 24-48 h antes da transfecção as sementes uma quantidade suficiente de células HEK293T com base no número de placas de 96 poços a ser transfectada.
NOTA: Para transfecções consistentes, as células em placas a uma densidade suficiente para favorecer a divisão rápida, ainda não estar em mais do que 70-90% de confluência no momento da transfecção. - Cada placa de 96 poços requer 9 x 10 6 células (75.000 células por poço, e 120 poços por placa para ter em conta o uso de reservatório e pipeta multicanal). Calcular o tempo de duplicação de células HEK293 (tipicamente ~ 20 h), e semear o número adequado de células para se obter, pelo menos 9 x 10 6 células no momento da transfecção. Uma placa de cultura de 145 milímetros circular é tipicamente suficiente para três transfecções de 96 poços em que cresceram para ~ 90% de confluência, com ~ 10% de células restantes para propagar novamente uma nova placa.
2. Preparação antes da transfecção
_content "> NOTA: A preparação de tampões e de DNA plasmídeo em 2,0-2,2 passo deve ser realizado no dia antes da transfecção, uma vez a preparação destes reagentes pode ser demorado.- Clones 3'UTR humanos que são compatíveis com o ensaio 3'LIFE estão disponíveis através de um repositório plasmídeo público 30. Purifica-se o plasmídeo de ADN manualmente ou com robôs de manipulação de líquidos. Usar um grau de lise alcalina mini-prep de transfecção de 96 poços kit e seguir as instruções do fabricante. Ressuspender os vectores purificadas a ~ 100 ng / mL por poço.
NOTA: O sinal da luciferase insuficiente resultará se a concentração de plasmídeo cai abaixo de 40 ng / ul. - Obter o pLIFE-miRNA vetores 30 ou clone usando o pipeline na Figura 1B 24. Ressuspender os vectores a uma concentração de 500 ng / mL para cada miARN e plasmídeos de controlo em branco.
- Devido à sensibilidade das condições de tampão nucleofection, assegurar queo volume total de materiais transfectadas (incluindo células e plasmídeos) não excede 10% do total de líquido em cada poço da placa de transfecção de 96 poços. Para conseguir isso, concentra-se o plasmídeo da pLIFE-miARN a uma concentração de pelo menos 500 ng / ul.
- Prepare 10x tampão reagentes luciferase de pirilampo (Tabela 1), e os reagentes tampão 1x Renilla luciferase (Tabela 2), que podem ser armazenadas por até 6 meses.
NOTA: O DTT no tampão de luciferase de pirilampo deve ser armazenada em solução a -20 ° C em alíquotas de uso único.
3. Prepare Seguindo itens imediatamente antes da transfecção
- Preparar tampão de transfecção contendo PBS, 1,5% de HEPES, pH 7,0. Preparar esta fresco, embora possa ser armazenado durante até um mês a 4 ° C sem diminuições visíveis na eficiência de transfecção. Na formulação de volumes de tampão e de DNA plasmídeo, assumir 120 reacções para cada placa de 96 poços para sufficiently responsáveis por erros de pipetagem e volume perdido usando reservatórios líquidos e pipetas multicanal. Alíquota de 18 ul por tampão de transfecção bem (120 poços / placa de 96 poços = 2,16 ml por placa), e reserve.
NOTA: O dispositivo de célula de electroporação é extremamente sensível às condições de tampão utilizadas para transfectar as células. Precisão quando preparando buffers irá garantir um desempenho consistente do equipamento. Extra cuidado deve ser tomado quando da realização do ensaio para evitar a evaporação de tampões, especificamente através da minimização do tempo que o tampão é deixada descoberta em tubos de microcentrífuga, placas de 96 poços, reservatórios, e placas de eléctrodos. - Reserve quatro poços para os seguintes controles, não pLIFE-3'UTR (para medir fundo de ensaio de luciferase), pLIFE-SV40 3'UTR (controle de destino negativo), controlo positivo para miRNA # 1, o controlo positivo para miRNA # 2.
NOTA: Estes vectores estão disponíveis publicamente 30. Alternativamente, qualquer alvo previamente validadopode ser usado como um controlo positivo. - Meios quentes, tripsina (0,25%) a 37 ° C.
- A cada poço da placa de 96 poços de cultura de células, adicionar 200 ul de DMEM suplementado com 10% FBS, 1% de Pen / Strep, e colocados num banho a 37 ° C incubadora para utilização após a transfecção.
- Ligue todos os dispositivos eletroporação célula seguidos pelo software de suporte. Use código de pulso FF120 para células HEK293T e tampão PBS / HEPES.
4. Preparação do plasmídeo de DNA e da mistura de células
NOTA: O protocolo a seguir pressupõe transfecção de três placas de 96 poços em um experimento para uma tela com dois miRNAs (miRNA- # 1 e # 2 miRNA-). Cada placa irá corresponder à mesma placa de 96 poços de plasmídeos pLIFE-3'UTR, e ser tratado três vezes com pLIFE-miARN-branco, pLIFE-miRNA- # 1, ou pLIFE-miRNA- # 2.
- Prepare três estoques de tampão de transfecção pLIFE-miRNA + para cada miRNA. Este estoque devem responder por 50% (10 mL) de volume total de cadaassim, multiplicado por 120 poços. Assim, cada unidade deve conter 1,08 ml de tampão + 120 l DNA plasmídeo (pLIFE-miRNA).
- Remover as células a partir de 145 mm Placa de cultura por meios eluindo, lavando suavemente com PBS, e o tratamento com ~ 5 ml de tripsina a 0,25% durante 5 min a 37 ° C. Neutralizar a tripsina com um volume igual de meio, e as células de pelotas a 300 xg durante 5 min.
- Remover tripsina / mídia e ressuspender sedimento em ~ 5-10 ml de mídia (dependendo da densidade celular e alcance exato de contador de células).
- Contagem de células utilizando um contador de células. Certifique-se de que as células são> 95% viáveis e dentro do alcance exato da máquina.
NOTA: Uma contagem de células imprecisa pode resultar de concentrações extremamente elevadas de células (> 6.0 x 10 6 / ml). Transfecção de muitas células pode reduzir drasticamente a eficiência da miARN alvo, reduzindo plasmídeo: índice de célula e / ou a diminuir a eficiência da transfecção. - Alíquotas três tubos cada um contendo 9 x 10 6 células, o que corresponde às células de required para a transfecção de uma placa de 96 poços. Rotação células a 300 xg durante 3 min.
- Remova a mídia. Certifique-se de remover o máximo de mídia como possível com o mínimo de perturbação do sedimento como excesso de mídia pode afetar a eficiência de transfecção.
- Ressuspender as células em tampão 1,2 ml de transfecção / mistura plasmídeo miRNA, e reserve.
- Os seguintes passos detalham ressuspensão de pLIFE-3'UTR plasmídeo em tampão de transfecção. Enquanto isto ocorre em placas de 96 poços, ter o cuidado de evitar a evaporação do tampão por placas de cobertura em todos os momentos.
- Usando uma pipeta de canais múltiplos, mover 32,4 ul de tampão de transfecção para cada poço de uma placa de 96 poços de PCR (9 ul [por transfecção] * [3 placas] * 1.2 [para contabilizar erro pipeta]).
- Adicionar 3,6 mL (~ 100 ng / uL) preparada de mini-pLIFE-3'UTR plasmídeo a cada poço e homogeneizar.
- Pipetar 10 ul desta mistura em cada poço da placa de 96 poços de transfecção e da tampa.
5. Transfecção
- Mova 1,2 ml do primeiro / buffer / mistura plasmídeo pLIFE-miRNA celular em reservatório. Misture bem.
- Adicionar 10 ul desta mistura na primeira placa de 96 poços de transfecção já contendo 10 ul de tampão de transfecção / pLIFE-3'UTR. Misture bem, pipetando cima e para baixo várias vezes.
NOTA: Igual suspensão de células no mesmo tampão assegurará completa e passagem de corrente eléctrica através da cuvete e maximizar a eficiência da transfecção. - Coloque placa de 96 poços de transfecção no dispositivo de electroporação de células e iniciar a transfecção.
- Uma vez que a transfecção é completa, adicionar 100 uL de meio de pré-aquecido a partir de placa de 96 poços de cultura de cada poço da placa de transfecção de 96 poços e misture bem. Mova 100 uL de cada poço na placa de cultura de 96 poços.
- Misturar as células em placa de cultura com uma pipeta posicionado verticalmente no centro do poço, enquanto as células tendem a agregar nos lados do well a menos misturado adequadamente.
- Repita 5,1-5,5 para os restantes duas placas.
- Limpeza placa de transfecção de 96 poços
- As placas de transfecção de 96 poços podem ser reciclados por lavagem com etanol 70% para garantir que não haja transição de ácidos nucleicos entre experimentos. Execute duas lavagens de 70% EtOH utilizando um frasco de spray para encher completamente cada poço, seguido por limpando o excesso de EtOH nas tiras de eletrodos (lado inferior) e permitindo que as placas de transfecção para completamente seca na capa de cultura.
NOTA: Testámos a transitar contaminação de ADN por transfecção de 12 poços com 2 ug pmaxGFP cada plasmídeo em células HEK293T, seguido por uma única lavagem com EtOH a 70%, e uma segunda transfecção com plasmídeo sem DNA. Com esta concentração extremamente elevada de plasmídeo, extremamente brilhante repórter, e uma única lavagem, não havia nenhuma fluorescência observável em qualquer um dos 12 transfecções em duplicado.
- As placas de transfecção de 96 poços podem ser reciclados por lavagem com etanol 70% para garantir que não haja transição de ácidos nucleicos entre experimentos. Execute duas lavagens de 70% EtOH utilizando um frasco de spray para encher completamente cada poço, seguido por limpando o excesso de EtOH nas tiras de eletrodos (lado inferior) e permitindo que as placas de transfecção para completamente seca na capa de cultura.
- Células de cultura durante 48-72 horas a 37 ° C, seguido pela dupla Lucifapagar ensaio.
6. Lisado celular Preparação de Ensaio de Luciferase
- Diluir tampão de lise com água 4 partes, uma parte de 5x tampão de lise passivo em um reservatório. Calcular 26 ul / poço, acrescentando ~ 20 volumes adicionais para contribuir para uma perda no reservatório.
NOTA: Tampão é armazenado a -20 ° C e pode ser extremamente viscoso, ou seja, antes de permitir que o tampão 5x para aproximar temperatura ambiente irá melhorar a precisão de pipetagem. - Analisar cada poço para a eficiência de transfecção utilizando a microscopia de fluorescência. Nota quaisquer inconsistências em poços que não transfectar de forma eficiente (> 90% de eficiência de transfecção), ou estão expressando baixos níveis de RFP indicativo de superlotação ou mídia exaustão. Remova os poços da análise.
- Completamente remover a mídia a partir das células, tomando cuidado para não eluir muito rapidamente que fará com que as células de separar.
NOTA: mídia restante irá diluir o ligado e causar flutuações de valores em todo experiments. - Adicionar 26 mL de tampão de lise a cada poço, e coloque em um prato shaker / balancim em velocidade baixa / moderada para ~ 20-30 min. Use esse tempo para preparar tampões de luciferase, lavar e preparar o luminómetro (s), e transferir ligado às placas de medição opacos.
7. Ensaio de Luciferase Duplo
NOTA: Se várias placas estão sendo medidos sequencialmente em um luminómetro, criar misturas principais tampão com tudo, exceto ATP e substratos, acrescentando estes reagentes seguido por ajuste de pH imediatamente antes da utilização, com cada placa. ATP e substratos podem degradar ao longo do tempo; consistência na quantidade de tempo que estes reagentes estão no buffer irá melhorar a consistência entre vários pratos.
- Prepare tampões luciferase 1x (Tabela 1):
- Enrole dois tubos (tipicamente 15/50 ml tubos de centrífuga) contendo o vaga-lume e buffers Renilla com folha de alumínio, como substratos pode ser sensível à luz.
- Prepare tampão luciferase 1x Firefly. Adicionar 1 ml de cada um dos cinco 10x reagentes luciferase de pirilampo, a adição de EGTA a última, a 5 ml de H 2 0 a uma concentração final de 1x.
- Adicionar 0,025 g ATP para 10 ml de 1x tampão de pirilampo. Misture invertendo várias vezes. Manter em gelo ATP em todos os momentos. ATP irá degradar, por isso, se medindo mais de uma placa sequencialmente, buffer deve ser feita no início desta etapa para cada placa adicional.
- Adicionar 100 ul de luciferina 100 besouro (substrato) (Tabela 1) Tampão devem mudar a cor amarelada com base no pH.
- 1x tampão de luciferase de Renilla reconstituição: Por placa de 96 poços, uma alíquota de 10 ml de um "tampão de Renilla".
- Adiciona-se 100 ul de BSA (44 mg / ml de stock).
- Se o rastreio mais de uma placa, mix principal separado em 10 ml alíquotas.
- Adicionar 100 ul de tampão de coelenterazina (previamente divididos em alíquotas e armazenado a 100x conc.)
- Ajustar o pH de 1x tampão de pirilampo para 8,0, seguido de tampão 1x a Renilla para 5,0 usando NaOH e HCl.
NOTA: A actividade de cada tampão, e a capacidade da memória intermédia de Renilla para extinguir a actividade da luciferase do pirilampo é altamente dependente do pH. Para obter resultados consistentes ser extremamente precisos nesta etapa. - Levar o volume de cada tampão (correspondente a uma placa de 96 poços) a 10,5 ml de acomodar para luminómetro escorva.
- Transferir o lisado para placas brancas opacas: Neste ponto, as células devem ser em tampão de lise durante ~ 20 min. Pegue 25 ul de cada poço utilizando uma pipeta multicanal, certifique-se de pipeta cima e para baixo cuidadosamente para quebrar os aglomerados de células e homogeneizar o ligado.
- Prepara-se o luminómetro. Ligue o luminómetro e selecione o protocolo na pasta DLR, chamado de "DLR com duas injeções". Outros formatos não são compatíveis com o pipeline de análise de dados (abaixo).
- Selecione os poços para ser tresse (todos os poços é o padrão).
- Estender o "Atraso antes da medição 'configuração para 5 segundos, com um tempo de medição de 10 segundos (ver 29 para explicação).
- Capilares passos de lavagem: 3x água, EtOH 3x 3x, água, 3x seco. Buffers Prime uma vez para o lixo, e então primeiro pela segunda vez de volta para os tubos de tampão para assegurar a mistura. Injectar o buffer vaga-lume primeiro e principal-lo no capilar esquerda, seguido de Renilla no capilar direita.
- Iniciar o ensaio de luciferase. Cada placa deve ter ~ 48 min de ler. Após a conclusão salvar o arquivo primeiro e, em seguida, repita os passos de lavagem e desligar o luminómetro.
NOTA: Vários pratos podem ser lidos e os dados armazenados no mesmo arquivo excel, no entanto questões podem ser encontradas com placa de várias leituras onde o programa lumin�etro irá falhar. Certifique-se de salvar todos os dados entre as medições e tirar screenshots se programa trava antes de save é possível.- Substitua buffers velhoscom o novo, certificando-se de primordial pelo menos duas vezes com novos amortecedores antes de iniciar a nova placa.
Análise 8. Dados
- Utilize tabelas do Excel "3'LIFE - análise de placa única" e "3'LIFE - análise multiplate" disponível a partir www.mangonelab.com . Copie dados brutos para vaga-lume e Renilla luciferase medições de arquivo de saída luminómetro em locais correspondentes à condição negativa, miRNA # 1, # 2 e miRNA no "3'LIFE - análise placa single" planilha.
- A planilha calculará automaticamente vaga-lume rácio / Renilla e normalizar cada miRNA para o controlo negativo adequado, e normalizar os valores de repressão através de cada prato.
NOTA: Esta planilha irá identificar automaticamente poços com sinal de baixa luciferase, realce poços significativamente reprimidas, e fornecer medidas de repressina em toda a placa. Veja 24 para explicação detalhada de análise estatística.- Copiar os valores de caixa "Normalized Índice RI", em células correspondentes no "3'LIFE - análise multipanel" folha de cálculo na posição correspondente para replicar # 1 para cada miARN testado. Repetir para todos os replicados biológicos realizados em dias diferentes.
- Se desejado, inserir os nomes de genes alvo e estado de predição nas colunas B e D, respectivamente.
- Permitir que a planilha para calcular automaticamente a média de todas as placas. Observar os dados em formato de 96 cavidades como um mapa de calor (Figura 2). O arquivo também organiza os dados em formato de lista (Figura 2).
NOTA: O índice de repressão é a medida utilizada para identificar alvos de miARN putativos. Restringência parâmetros diferentes pode ser utilizado, com base nas preferências individuais. Em média, nós consideramos prováveis sucessos abaixo índice de repressão de 0.8 e estatisticamente significativa pelo teste t (valor p <0,05). Como mostrado na Figura 3 alvos potenciais com base nesses critérios serão destacadas em vermelho.
- Permitir que a planilha para calcular automaticamente a média de todas as placas. Observar os dados em formato de 96 cavidades como um mapa de calor (Figura 2). O arquivo também organiza os dados em formato de lista (Figura 2).
Resultados
O arquivo de saída lumin�etro contém medidas brutos para ambas as proteínas vaga-lume e Renilla luciferase. Este formato bruto é compatível com o "3'LIFE - análise de placa única" e "3'LIFE - análise multiplate" planilhas disponíveis no site do laboratório Mangone ( www.mangonelab.com ). A única planilha análise placa calcula automaticamente vaga-lume rácio / Renilla, normaliza cada miRNA para o controlo negativo adequado, e normaliza valores repressão através de cada prato. Esta planilha identifica automaticamente poços com sinal luciferase Renilla baixo, destaca poços que apresentam repressão em relação ao controle negativo, e fornece medidas de repressão em toda a placa (Figura 2). Veja 24 para explicação detalhada de análise estatística.
Várias repetições pode ser analisard utilizando a "análise multiplate" planilha. Cada repetição é comparados lado a lado, e as medidas estatísticas dos dados são calculados automaticamente (Figura 3). Além de comparar repetições, com as colunas "repressão normalizado", o usuário pode comparar repressão entre os dois miRNAs no âmbito do "miRNA # 1 / miRNA # 2" colunas. Esta medida divide o índice de repressão para cada miRNA para cada repetição. Esta medida pode indicar valores erróneos a partir do ensaio de luciferase (por exemplo leituras anormalmente altas ou baixas com o controlo negativo, ver Figura 3, linha A9, Rep # 3), e os poços em que o índice de repressão não pode indicar repressão substancial, mas que fazer apresentam diferenças significativas entre os miRNAs. Enquanto esta medida não pode ser utilizado directamente para indicar um miARN alvo, é útil para a identificação de pontos extremos, poços problemáticos, ou padrões nos dados que não são atribuídos exclusivamente para dirigir miARN regulamento.
A repressão Index (RI) é usado para chamar um alvo putativo miRNA, com menores valores correspondentes a maior repressão relativa. O limiar para chamar alvos putativos é baseado no nível de rigor exigido pelo pesquisador, mas combinando o RI com 3'UTRs que exibem valores de p estatisticamente significativa (p <0,05) vai indicar alvos de alto confiança (ver Figura 2 linhas e B8 B12).

Figura 1: 3'LIFE Ensaio (A). Vectores Gateway compatível utilizado no ensaio 3'LIFE. Topo: O gene da luciferase (Fluc) está fundido com o 3 'UTR de teste, enquanto que o gene da luciferase de Renilla (RLuc) está fundido com o inespecífica SV40 pA 3'UTR como controlo. Resumindo: O RFP- vector miRNA-intron - A sonda pré-miRNA, além de ~ 400nucleotídeos dentro de seu locus genómico (recapitular processamento de miRNA endógeno), é clonado dentro de um intrão para permitir a sua co-expressão com DsRed2 fluorocromo. Ambos os vectores estão disponíveis publicamente (Seiler et al., 2013). (B) Fluxograma do 3'LIFE Ensaio. (C) 3'LIFE Pipeline: O vector de luciferase dupla contendo a 3'UTR de teste com ou sem os vectores de miARN são co-transfectados em células HEK293 em placas de 96 poços. A interacção entre a miARN alvo e uma UTR 3? Bona fide irá diminuir a luminescência relativa em poços específicos (exemplificado pela mancha cor de laranja na placa experimental).

Figura 2: Exemplo de dados produzidos com ensaio 3'LIFE. Cada sonda miRNA é testada em quadruplicado (replica 1-4). Cores representam os níveis de repressão, com vermelhocores que indicam forte miRNA / interação 3'UTR. Todas as réplicas são calculados para produzir alvos putativos de alta qualidade mostrados na placa de resumo que se segue a seta amarela. Caixa branca representam os controles, não transfections ou poços com baixa eficiência de transfecção.
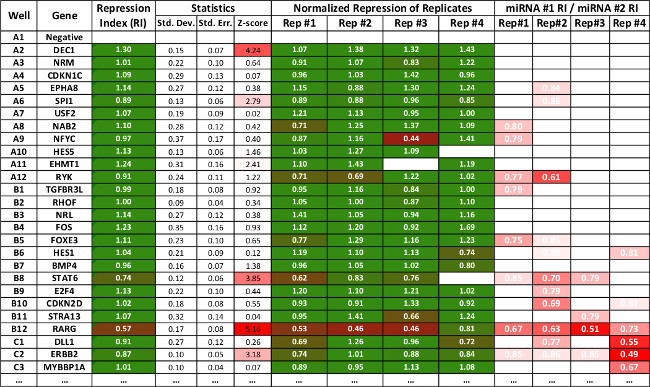
Figura 3:. Tabela representando dados de resumo de um subconjunto de interacções produzidos utilizando a folha de cálculo 3'LIFE Os valores de repressão são como na Figura 2. O software calcula o desvio padrão, e o erro padrão Z-pontuação para cada interacção. Estatisticamente interações significativas estão marcados em vermelho. Os últimos quatro linhas mostram repressão relativa de um miARN para o outro, e é usado como indicador secundário para comparar repressão entre dois miARNs diferentes. A planilha pode ser descarregado a partir www.mangonelab.com
| Firefly reagentes tampão luciferase | A concentração final (1x) |
| Glicilglicina | 25 mM |
| K x PO 4 (pH 7,8) | 15 mM |
| MgSO4 | 15 mM |
| DTT (loja no 4º) | 1 mM |
| EGTA | 4 mM |
| ATP * | 2 mM |
| Beetle luciferina * | 250 uM |
Tabela 1: os reagentes da luciferase do pirilampo: As soluções-mãe 10x de glicilglicina, K x PO 4, MgSO 4, DTT e EGTA podem ser preparados separadamente e armazenados antes da reconstituição tampão. 100x Beetle Luciferin (substrato de luciferase de vaga-lume) pode ser lojad por dissolução de 50 mg de luciferina em 7,134 ml de H 2 0 (25 mM). Alíquota de 105 mL / placa de luciferina Beetle dissolvido em tubos e armazenar a -80 ºC. Por suporte técnico Promega, este deve ser estável por> 6 meses, mas pode ser sensível à luz. NOTA: EGTA não vou entrar em solução a pH neutro. Lentamente adicionar NaOH para EGTA até que se dissolva completamente. * Os reagentes adicionados ao tampão final, imediatamente antes do ensaio de luciferase
| Renilla reagentes tampão luciferase | A concentração final (1x) |
| NaCl | 1,1 M |
| Na2EDTA | 2,2 mM |
| KH 2 PO 4 | 0,22 M |
| NaN3 | 1,3 mM |
| BSA * | 0,44 mg / mL |
| Coelenterazina * | 2.5 & #956; M |
Tabela 2: da Renilla luciferase reagentes tampão Todos os reagentes excepto BSA e coelenterazina pode ser misturado a uma concentração 1x e armazenado à temperatura ambiente. Coelenterazina pode ser dissolvido em metanol e acidificou-se aliquotas por placa. Acidifica-se por adição de HCl metanol até à concentração final de 5 mM (<3 pH). Dissolver 250 g coelenterazina em 2,36 ml de metanol acidificado (250 M) alíquotas 105 ul / placa. A mistura é estável durante pelo menos 6 meses, mas pode ser sensível à luz. * Os reagentes adicionados para tamponar imediatamente antes do ensaio de luciferase.
Discussão
O ensaio 3'LIFE identifica alvos miRNA funcionais em 3'UTRs em high-throughput. Este ensaio é útil para investigadores que desejam identificar experimentalmente um grande número de possíveis alvos para a sua miARN de interesse. O ensaio 3'LIFE é uma abordagem poderosa para consultar 3'UTR regulação accionado, em que o ensaio fornece uma medida funcional de miARN direccionamento, e o teste de binário de um único repórter :: 3'UTR contra um único miARN pode confiantemente tratar o estado de direccionamento de genes individuais. Para validar esta abordagem, nós analisamos um painel de 275 3'UTRs e contra dois miRNAs, deixe-7c e miR-10b, e incluiu 10 genes alvo previamente validadas nesta biblioteca. Oito desses dez genes exibiu repressão 24. Observou-se também um enriquecimento significativo de unvalidated alvos bioinformatically previstos, e 3'UTRs imprevisíveis que contêm elementos canônicos de sementes entre os nossos maiores sucessos, sugrindo que 3'LIFE é capaz de identificar alvos miRNA de boa-fé.
Um dos principais indicadores da sensibilidade de telas de alto rendimento é as taxas de positivos e falsos negativos falsos. Enquanto a taxa de falsos positivos deste ensaio tem de ser avaliada utilizando abordagens alternativas adicionais para validar hits, oito dos dez controlos positivos incluído em nossa tela de repressão de prova de princípio exibiu, sugerindo uma taxa de falso negativo de 20%. No entanto, muitas técnicas são utilizadas para identificar alvos de miARN em diferentes contextos celulares, e de processamento 3 'UTR e regulação dos factores que actuam em trans é conhecido por ser altamente específico de tecido. Por exemplo, a maioria dos 3'UTRs conter vários locais de poliadenilação, que controlam o comprimento da 3'UTR do ARNm maduro. Em muitos casos, a utilização de locais de poliadenilação proximais é específico de tecido, e pode excluir sites miARN alvo. Além disso, cooperativa segmentação miRNA, wi competiçãoproteínas de ligação a ARN-po, e a estrutura secundária do ARNm podem todos impacto a capacidade de 3'LIFE para detectar alvos de miARN em tecidos específicos. Devido a isso, a detecção de alvos de ensaio 3'LIFE pode variar com base no contexto celular, em que o ensaio é realizado, o que complica a avaliação de taxas de erro absoluto. Este protocolo foi optimizado para as células HEK293T, mas linhas celulares alternativos pode ser usado se o investigador pretende realizar o ensaio num contexto biológico específico. No entanto, a optimização da eficiência da transfecção e a sobrevivência das células com cada linha de células de terá de ser optimizada utilizando várias condições de tampão, códigos de impulsos, o número de células. Um exemplo de um esquema de optimização podem ser encontradas em Wolter et ai. 24.
Este protocolo foi optimizado em formato de 96 poços e especifica a utilização de certa instrumentação de alto rendimento. No caso em que a instituição não possuir o equipamento necessário para 96-bem Nucleofection, reagentes de transfecção alternativos podem ser usadas para realizar o ensaio 3'LIFE, contanto que a eficiência de transfecção continua a ser elevada. Além disso, o ensaio de luciferase é o mais demorado aspecto do ensaio 3'LIFE. Como tal, recomenda-se o uso de múltiplos luminómetros para telas de alto rendimento.
Divulgações
This work is supported by funds from the College of Liberal Arts and Science and the Biodesign Institute at Arizona State University and NIH Exploratory/Developmental Research Grant 1R21CA179144-01A1.
Agradecimentos
We thank Stephen Blazie, Karen Anderson, Josh LaBaer for advice and discussion. Karen Anderson, John Chaput, and Josh LaBaer for sharing reagents and instrumentation, Michael Gaskin and Andrea Throop for technical advise and protocols. Justin Wolter is a Maher scholar and thanks the Maher family for their generous support.
Materiais
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| glycylglycine | Sigma | G1127-25G | |
| Kx PO4 | Sigma | P2222 | |
| EGTA | Sigma | E3889 | |
| ATP | Sigma | FLAAS | |
| DTT | Sigma | D0632 | |
| MgSO4 | Sigma | M7506 | |
| CoA | Sigma | C4282 | |
| luciferin | Sigma | L9504 | |
| NaCl | Sigma | S7653 | |
| Na2EDTA | Sigma | E0399 | |
| K H2 P O4 | Sigma | 1551139 | |
| BSA | Sigma | A2153 | |
| NaN3 | Sigma | S2002 | |
| Coelenterazine | Sigma | C3230 | |
| PBS/HEPES | Corning | 21-040-CV | |
| DMEM | Sigma | D5546 | |
| FBS | Sigma | F2442 | |
| Pennicilin/Streptomicin | Sigma | P4333 | |
| Trypsin | T2600000 | ||
| Consumables | |||
| MaxiPrep Kit | Promega | A2392 | |
| 96-well miniprep plate | Pall | 8032 | |
| 96-well shuttle plates | Lonza | V4SP-2096 | |
| 5x Lysis Buffer | Promega | E1941 | |
| Instruments | |||
| 96-well GloMax Plate Reader | Promega | E9032 | |
| Biomech FX Liquid Handler Robot | Beckmann | A31842 | |
| 4D-Nucleofector Core Unit | Lonza | AAF-1001 | |
| 96-well Shuttle System | Lonza | AAM-1001 | |
| Cell Counter Countess | Invitrogen | C10227 |
Referências
- Bartel, D. P. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell. 116 (2), 281-297 (2004).
- Pasquinelli, A. E., et al. Conservation of the sequence and temporal expression of let-7 heterochronic regulatory RNA. Nature. 408 (6808), 86-89 (2000).
- Reinhart, B. J., Weinstein, E. G., Rhoades, M. W., Bartel, B., Bartel, D. P. MicroRNAs in plants. Genes Dev. 16 (13), 1616-1626 (2002).
- Ivey, K. N., et al. MicroRNA regulation of cell lineages in mouse and human embryonic stem cells. Cell Stem Cell. 2 (3), 219-229 (2008).
- Lee, R. C., Feinbaum, R. L., Ambros, V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 75 (5), 843-854 (1993).
- Wienholds, E., et al. MicroRNA expression in zebrafish embryonic development. Science. 309 (5732), 310-311 (2005).
- Darnell, D. K., et al. MicroRNA expression during chick embryo development. Dev Dyn. 235 (11), 3156-3165 (2006).
- Jin, P., Alisch, R. S., Warren, S. T. RNA and microRNAs in fragile X mental retardation. Nat Cell Biol. 6 (11), 1048-1053 (2004).
- Kim, J., et al. A MicroRNA feedback circuit in midbrain dopamine neurons. Science. 317 (5842), 1220-1224 (2007).
- Poy, M. N., et al. A pancreatic islet-specific microRNA regulates insulin secretion. Nature. 432 (7014), 226-230 (2004).
- Calin, G. A., Croce, C. M. MicroRNA signatures in human cancers. Nature Reviews Cancer. 6 (11), 857-866 (2006).
- Friedman, R. C., Farh, K. K., Burge, C. B., Bartel, D. P. Most mammalian mRNAs are conserved targets of microRNAs. Genome Res. 19 (1), 92-105 (2009).
- Paraskevopoulou, M. D., et al. DIANA-microT web server v5.0: service integration into miRNA functional analysis workflows. Nucleic Acids Res. 41 (Web Server issue), W169-W173 (2013).
- Krek, A., et al. Combinatorial microRNA target predictions. Nat Genet. 37 (5), 495-500 (2005).
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136 (2), 215-233 (2009).
- Vella, M. C., Choi, E. Y., Lin, S. Y., Reinert, K., Slack, F. J. The C. elegans microRNA let-7 binds to imperfect let-7 complementary sites from the lin-41 3'UTR. Genes Dev. 18 (2), 132-137 (2004).
- Lal, A., et al. miR-24 Inhibits cell proliferation by targeting E2F2, MYC, and other cell-cycle genes via binding to 'seedless' 3'UTR microRNA recognition elements. Molecular Cell. 35 (5), 610-625 (2009).
- Cevec, M., Thibaudeau, C., Plavec, J. N. M. R. NMR structure of the let-7 miRNA interacting with the site LCS1 of lin-41 mRNA from Caenorhabditis elegans. Nucleic Acids Res. 38 (21), 7814-7821 (1093).
- Shin, C., et al. Expanding the microRNA targeting code: functional sites with centered pairing. Molecular Cell. 38 (6), 789-802 (2010).
- Azzouzi, I., et al. MicroRNA-96 directly inhibits gamma-globin expression in human erythropoiesis. PLoS One. 6 (7), e22838(2011).
- Chen, J., et al. miR-193b Regulates Mcl-1 in Melanoma. Am J Pathol. 179 (5), 2162-2168 (2011).
- Chi, S. W., Hannon, G. J., Darnell, R. B. An alternative mode of microRNA target recognition. Nat Struct Mol Biol. 19 (3), 321-327 (1038).
- Jiao, L. R., et al. MicroRNAs targeting oncogenes are down-regulated in pancreatic malignant transformation from benign tumors. PLoS One. 7 (2), e32068(2012).
- Wolter, J. M., Kotagama, K., Pierre-Bez, A. C., Firago, M., Mangone, M. 3'LIFE: a functional assay to detect miRNA targets in high-throughput. Nucleic Acids Res. , (2014).
- Mangone, M., et al. The landscape of C. elegans 3'UTRs. Science. 329 (5990), 432-435 (2010).
- Ekdahl, Y., Farahani, H. S., Behm, M., Lagergren, J., Ohman, M. A-to-I editing of microRNAs in the mammalian brain increases during development. Genome Res. 22 (8), 1477-1487 (2012).
- Fu, Y., Dominissini, D., Rechavi, G., He, C. Gene expression regulation mediated through reversible m(6)A RNA methylation. Nat Rev Genet. 15 (5), 293-306 (2014).
- Zhou, P., et al. Large-scale screens of miRNA-mRNA interactions unveiled that the 3'UTR of a gene is targeted by multiple miRNAs. Plos One. 8 (7), e68204(2013).
- Dyer, B. W., Ferrer, F. A., Klinedinst, D. K., Rodriguez, R. A noncommercial dual luciferase enzyme assay system for reporter gene analysis. Anal Biochem. 282 (1), 158-161 (2000).
- Seiler, C. Y., et al. DNASU plasmid and PSI:Biology-Materials repositories: resources to accelerate biological research. Nucleic Acids Res. , (2013).
- Baek, D., et al. The impact of microRNAs on protein output. Nature. 455 (7209), 64-71 (2008).
- Ebert, M. S., Sharp, P. A. Roles for MicroRNAs in Conferring Robustness to Biological Processes. Cell. 149 (3), 515-524 (2012).
- Pelaez, N., Carthew, R. W. Biological robustness and the role of microRNAs: a network perspective. Current Topics in Developmental Biology. 99, 237-255 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados