Method Article
Floresan Bazlı Astar Uzatma Tekniği Transkripsiyonal Başlangıç Noktalarını ve RNases yarılması Siteleri belirleme
Bu Makalede
Özet
We here describe a fluorescence based primer extension method to determine transcriptional starting points from bacterial transcripts and RNA processing in vivo using an automated gel sequencer.
Özet
Flüoresan göre primer uzatma (FPE) transkripsiyonel başlangıç noktalarının ya da RNA moleküllerinin işlenmesi bölgelerini belirlemek için moleküler bir yöntemdir. Bu özel floresanla işaretlenmiş primerler ve poliakrilamid jel elektroforezi ile denaturasyon ile elde edilen cDNA parçaları daha sonra analizi kullanılarak, ilgi konusu RNA'nın ters transkripsiyonu ile elde edilir. Aynı zamanda, geleneksel bir Sanger dizileme Reaksiyon bunların tam karşılık gelen tabanlarına cDNA fragmanlarının uçları eşlemek için jel üzerinde çalıştırılır. Ürün klonlanmış olması ve çok sayıda aday dizilenmiştir 5'-RACE (cDNA uçlarının hızlı büyütülmesi), aksine, primer uzama yolu ile üretilen bir cDNA fragmanlarının toplu eşzamanlı olarak bir jel vadede tespit edilebilir. Buna ek olarak, (sonuçların son analizde, ters transkripsiyon ile ilgili) bütün işlem bir iş günü içinde tamamlanabilir. Floresanla işaretlenmiş primerler kullanılarak, tehlikeli radyoaktif izotop kullanımı reaktifler etiketliönlenebilir ve ürünler elektroforez işlemi sırasında tespit edilebilir işlem süreleri azaltılır.
Aşağıdaki protokol, güvenilir ve hızlı S. (örneğin, toksin antitoksin sistem bileşenleri ile) bir başlangıç noktası ve RNA işleme the transkripsiyonel anlamak için RNA'ların 5 'uçlarını tespit etmek için, bir in vivo floresan primer uzatma yöntemi tarif S. aureus, E. E. coli ve diğer bakteriler.
Giriş
Primer uzatma 1 bir taban çözünürlüğe kadar özel bir RNA moleküllerinin 5 'ucunun saptanması için moleküler bir yöntemdir. 5'-RACE (cDNA uçlarının hızlı yükseltilmesi) gibi diğer yöntemlerin bir avantajı, hızlı bir geri dönüş süresi ve kolay bir RNA moleküllerinin farklı uzunluklarının bir karışımı analiz yeteneğidir.
Bu yöntem, RNA molekülleri, belirli uzunluktaki cDNA fragmanlarının üretilmesi, özel bir flüoresan primerler kullanılarak transkripsiyon reaksiyonlarını ters tabi tutarak çalışır. Bu cDNA molekülleri denatüre edici poliakrilamid jelleri üzerinde geleneksel Sanger dizileme reaksiyonlarının 2 yanına uygulanır ve bağlı floresan etiketli primerlerin kullanımı ile fluoresansları ile tespit edilebilir. CDNA fragmanlarının uzunlukları 5 'RNA uçlarının eşleşmesini sağlayan bir sıralama merdiven karşılaştırılarak değerlendirilir.
Geleneksel olarak, primer uzatma reaksiyonları bağlantılı olarak kullanılanradyoaktif izotoplar X-ray filmlerde cDNA molekülleri tespit etmek. Onların duyarlılığı biraz daha düşük de olsa dolayı sağlık tehlikeleri, atık bertaraf sorunları ve kullanım kolaylığı, yeni protokoller, otomatik dizicilerle ile primer uzatma tespiti için floresan kullanmaktadır. Floresan primerler (bizim elimizde bir yıldan daha fazla), uzun bir süre için kararlı olduğu gibi, floresanla işaretlenmiş primerler kullanılarak, tekrar eden radyo-etiketleme prosedürü, ihmal edilebilir.
Burada açıklandığı gibi bir yöntem otomatik bir jel sekanslayıcı kullanır, ancak küçük değişiklikler yapılarak, kapillerli da cDNA ayrılması ve keşfi 3 için kullanılabilir. Jel analizi paralel yapısı mümkün RNA bölünme ve işlem daha az miktarda tespit edilebilir. Diğer bir avantajı tespit edilebilir terminali bölünme ya da bir bazın işleme gibi, bu yöntemin yüksek çözünürlüktür.
RNA bölünme ya da işlem, t tespiti ile ilgili olarakprimer uzantıları ypically iki farklı tür ayırt edilir. Diğer bir durumda, işlem, in vivo olarak yapılması ve sonuçta elde edilen RNA saflaştırılmıştır ise Bir durumda, olan enzimatik işlem, saflaştırılmış RNA ve arıtılmış enzim kullanılarak in vitro olarak yapılır. Bir primer uzatma in vitro olarak, her iki durumda da RNA RNA kaynağına bağlı olarak, ancak, tabi tutulur, bu yöntem, bir in vitro veya in vivo olarak, primer genişlemesini, adlandırılır ya. Burada mevcut protokol, bunların kullanım kolaylığı (herhangi bir saflaştırılmış protein için) ve aynı zamanda kopyalama başlangıç noktaları ve belirlemek için olasılığı, sadece, in vivo bir primer uzatma odaklanır. Bununla birlikte in vitro primer uzantıları ile aynı şekilde ayarlanır, prensip olarak ve bu protokol, bir başlangıç noktası olarak görev yapabilir.
Burada gösterilen yöntem sürece yüksek giriş oranlı gibi pek çok bakteri türleri uygulanabilirNükleik asitlerin saflığı ve yüksek verimli hazırlanması.
Laboratuarımızda araştırma toksin antitoksin (TA-) sistemler 4,5, primer uzatma yöntemi yaygın olarak kullanıldığı bir alanın düzenleme kapsamında odaklanmaktadır. TA-sistemleri istikrarlı ve endojen aktif toksik protein ve toksisite 6,7 zıt bir çoğunlukla kararsız protein ya da RNA antitoksin oluşmaktadır prokaryotik genomları mevcut küçük genetik unsurlardır. Toksin aktivitesi bazen RNaz etkinliğinin 8,9 En sık replikasyonu, hücre çeperi sentezi veya başka mekanizmalar inhibisyonu tarafından uygulanan, ancak. Tipik haliyle, RNaz özgüllük primer uzatma yöntemdir biri farklı testler, iletken ile belirlenir. Parçalanır ve tam uzunlukta parçalarının bir karışımı, aynı zamanda, 5 'ucunun saptanması için analiz edilebilir Primer uzatma reaksiyonları, bu uygulama için uygundur. In vitro ve in vivo primeri uzantılarında bir karışımını kullanarak,spesifik toksin RNaz ayrılma, örneğin, sekans özgüllük 10-13 belirlenebilir.
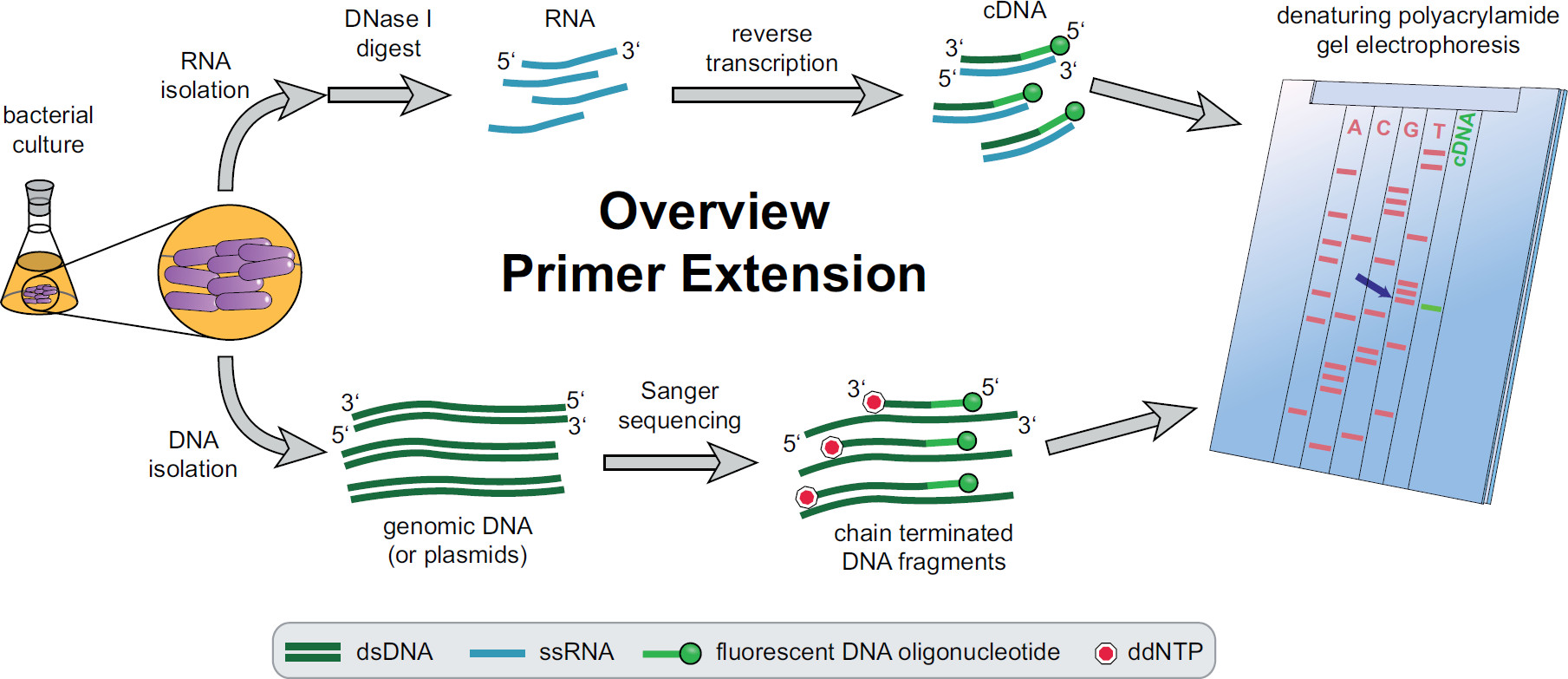
. Primer uzatma prosedürünün 1. Genel Bakış Şekil Bakteriyel kültürler deneysel ihtiyaçlarına göre kuluçkaya ve tedavi edilir. Toplam RNA, hücre cDNA elde edildi hedefe özgü floresan DNA primerler kullanılarak bir ters transkripsiyon reaksiyonu, DNA izlerini ortadan kaldırmak için DNase I ile işleme tabi tutuldu ve tabi elde edilir. Genomik DNA ya da plazmidler ekstre edilmiş ve daha sonra cDNA fragmanları ile boyut karşılaştırma için floresan Sanger dizileme reaksiyonları için kullanılır. Primer uzatma ürünleri denatüre üre poliakrilamid jel üzerinde Sanger dizileme ürünlerin yanında çalıştırmak ve otomatik lazer ve mikroskop ile analiz edilir. La cDNA bandı ile çizgiler kadar olan sıralama bazSt olarak 5 'cDNA ucu (mavi ok) tabanı. Fekete daha fazla bilgi, vd. 3 Bu rakamın büyük bir versiyonunu görmek için lütfen buraya tıklayınız.
Bütün primer uzatma işlemi ile ilgili bilgi, Şekil 1 'de bulunabilir. Kısaca, bakteri hücreleri, hasat edilir, kültürlenir, hücre topağı eritilir ve RNA ekstre edildi. Arındırılmış RNA daha sonra ters transkriptaz için şablon olarak hareket edebilir DNA moleküllerinin izlerini ortadan kaldırmak için DNase I ile işleme tabi tutulur. Özel flüoresan primerler ilgi bölgeye hibridize ve daha sonra tek sarmallı Tamamlayıcı DNA (cDNA) 'de elde edilen, ters transkribe edilmiş RNA ilave edilir. Bir sıralama merdiven floresan primer kullanılarak, geleneksel Sanger dizileme ile oluşturulan ve primer uzatma cDNA parçaları ile birlikte bir denatüre edici poliakrilamid jeli üzerinde ayrılır. Elde edilenJel ilgi 5 'uçlarının tanımlanmasına izin veren, floresan bantları karşılaştırılarak analiz edilir. Transkripsiyonal başlangıç noktaları ve işleme siteleri daha sonra sırası karşılaştırmaları tek tek değerlendirilir.
Protokol
1. Yüksek Verim RNA Hazırlık
- RNA'nın izole edilmesi,
Not: toplam RNA yüksek konsantrasyonları primer uzatma reaksiyonuna sokulması için gereklidir. Spin kolon takımları, genellikle gerekli RNA miktarını tespit edilmemektedir (~ 5-16 ug 5 ul hacim içinde). Bu nedenle asit guanidinyum thiosiyanat-fenol-kloroform ekstraksiyon yöntemi kullanılarak yapılan saflaştırma işlemi, aşağıda belirtileceği üzere, tavsiye edilir.
NOT: Fenol, kanserojen toksik ve tahriş edicidir. Malzeme güvenlik bilgi formlarını okuyun ve uygun koruma ile bir davlumbaz altında kullanın!- İstenilen ve hasat 4,600 x g'de 10 dakika santrifüj edildi ve 4 ° C ile olduğu gibi bakteriyel hücreler (bu örnekte S. aureus veya E.coli) büyütün ya da tedavi. Not: Tipik olarak 20 600 toplam OD hasat - 70 Hücre peletleri -20 ° C'de birkaç hafta muhafaza edilebilir.
- Asit guanidinyum thiosiyanat-fenol-kloroform çözeltisi 1 ml hücre pelletini ve transfer2 ml 0.1 mm cam zirkonyum / silika boncuk 0.5 ml içeren fincan vida.
- Lyse hücreleri, her çalışmadan sonra 5 dakika boyunca buz üzerinde örnekleri soğutma üç tur için 30 saniye için 6.5 m / sn hızla hazırlık / boncuk çırpıcı üç kez. Not: Homojenize Örnek birkaç hafta -80 ° C'de saklanabilir.
- Oda sıcaklığında 5 dakika boyunca lisat inkübe ve daha sonra kloroform içinde 200 ul ilave edin.
- Çalkalayın veya RNA çıkarmak için 30 saniye için örnek vorteks.
- 15,000 x g ve 4 ° C - daha sonra 13,000 C'de 15 dakika süreyle santrifüj 3 dakika boyunca oda sıcaklığında inkübe edin. Not: Solventler (beyaz DNA içerir), (pembe, proteinleri içerir) daha düşük bir organik faza bir interfaz ayrıldı ve bir üst sulu faz (net, RNA içerir) vardır.
NOT: Bu adımdan kullanımı sadece RNazsız miyar ve plastik eşya! - Uygun bir şekilde, taze RNase içermeyen 1.5 ml'lik tepkime tüpleri, etiket hazırlayın ve yaklaşık olarak aynı vo kullanımı (% 100 RNazsız izopropanol her biri 500 ul eklelume) bir önceki boru içindeki su fazı olarak.
- Bir açıyla boru tutun ve RNazsız uçları kullanılarak hazırlanan tüplerine sulu faz (yaklaşık 500 ul) aktarın. Interfazda rahatsız etmeyin.
- Birkaç kez baş aşağı ve oda sıcaklığında 10 dakika boyunca inkübe edilerek RNA hızlandırabilir.
- 15.000 x g ve 4 ° C ve pipetleme veya aspirasyon (su püskürtme pompası ve besleme şişesi) ile süpernatan - 13,000 C'de 15 dakika örnekleri santrifüjleyin. Altındaki beyaz şeffaf RNA pelet rahatsız etmeyin.
- Yıkamak için% 70-80 RNazsız etanol (do not girdabın) 1 ml ekleyin. Not: etanol RNA, -20 ° C'de birkaç hafta muhafaza edilebilir.
- 7,500 x g'de 5 dakika 4 ° C'de santrifüj ve pipetleme veya tercihen aspirasyon ile supernatant atın.
- Davlumbaz altında 30 dakika - 15 hava kuru RNA pelet. Overdry etmeyin, aksi takdirde pelet çözülür için zor olabilir.
- 5 pelletiniRNazsız GKD 2 O veya RNA depolama tampon 0 ul.
- Bir microvolume UV-Vis spektrofotometre veya kuvars küvetine (ve konvansiyonel fotometre) ile RNA konsantrasyonu ölçmek ve Dnaz ben sindirim geçin.
- RNA DNase I sindirim, DNA izlerini ortadan kaldırmak için
Not: DNA, ters transkripsiyon (primer uzatma) reaksiyonu sahte bir şablon olarak hareket edebildiği için, bu örnek çıkarılmalıdır. RNA, çözeltilerden DNA'nın uzaklaştırılması için çeşitli yöntemler, genellikle, DNase sindirim dayananlar bulunmaktadır. DNA çıkarılması için basit ama etkili ve maliyet etkin bir yöntem aşağıda belirtilmiştir.- 37 ° C'ye kadar ısıtın su banyosu.
- Bir 1.5 ml'lik tepkime tüpü içinde, Tablo 1 de gösterilen bileşikleri karıştırın.
- Bir su banyosu içinde 37 ° C de 1 saat süreyle inkübe karışımı, ve daha sonra fenol / kloroform ile ekstre etmeden doğrudan devam edin. Not: DNase I'inin ısı inaktivasyonu tavsiye edilmez, bu RNA düşürebilir gibi.
- DNase I sindirim sonra RNA fenol / kloroform ile ekstraksiyon
Not: RNA, DNase I sindirim ari nükleotidler, DNA fragmanlarının ve tampon bileşenleri çıkarmak için arındırılmalıdır. Fenol / kloroform ile ekstre RNA numunesinin yüksek geri kazanım ve konsantrasyon sağlar ve bu nedenle de aşağıda özetlenmiştir. Bunlar, bu gereksinimleri karşılaması durumunda, RNA saflaştırma için diğer yöntemler de kullanılabilir.- I sindirimi, 500 ul, DNase 2 ml'lik tepkime tüpleri iki 250 ul numuneler halinde karıştırın Böl.
- Asidik P / C / I çözeltisi 1 hacim (250 ul) (: 24: 1 - 5, pH 4.5 su doymuş fenol, kloroform ve izopentanol içinde, 25 oran) ekleyin.
NOT: P / C / I çözümü, kanserojen toksik ve tahriş edicidir. Malzeme güvenlik bilgi formlarını okuyun ve uygun koruma ile bir davlumbaz altında kullanın! - 3 dakika - 1 için vorteksleme platformda şiddetle girdap veya yer.
- 15.000 x g ve 4 ° C - 13.000 ° C'de 30 dakika boyunca santrifüje.
- Üst (sulu) faz toplamak ve tüpe (250 ul) transfer edilir.
- 3 M sodyum asetat pH 5.2 ve 1/9 hacim (28 ul) eklenir.
- Saf etanol (700 ul) 2.5-3 hacimlerini ekleyin.
- 3 saat - 30 dakika süreyle ya da 2, -20 ° C de, -80 ° C'de kısa bir süre ve yeri vorteks ile karıştırın. Gerekirse, -20 ° C'de RNA O / N saklayın.
- 15.000 x g ve 4 ° C - 13,000 arasında 60 dakika - 30 santrifüjleyin.
- Pipetle veya aspirasyon ile süpernatantı.
- Pelet üzerine,% 70 etanol, 1 ml ekleyerek Yıkama pelet. Vorteks örnek yok.
- 15,000 x g ve 4 ° C - 13,000 C'de 5 dakika boyunca santrifüje numunesi.
- Pipetle veya aspirasyon ile süpernatantı.
- Davlumbaz altında hava kuru pelet. Gerekli -20 ° CO / N ise pelet saklayın.
- 2 dakika boyunca karıştırın H 2 O tedavi edilen 30 ul DEPC pelet eritin ve pe çözmek için bu çözümü kullanmakörnek başına karşılık gelen ikinci tübün LLET (bir ekstre çifti başına 30 ul çözelti).
- RNA konsantrasyonu ölçümü, ve ortalama olarak ifade mRNA'ların primer uzatma kullanım için 1 ug / ul aşmasını sağlamak.
- Gerekirse, birkaç aya kadar bir kaç hafta boyunca -20 ° C'de RNA saklayın.
2. Primer Uzatma reaksiyonu
- Primer tasarımı
NOT: Bir astar uzatma deney için primerler tasarlanırken, (manuel bu yazıda daha çok bilgi ve tartışma bölümüne otomatik jel sequencer eşlik bakınız) PCR primer dizaynı genel kurallarına uyun.- Spesifik olarak, primerler, (i) bazların çalışır içermeyen sağlamak, (ii) 3 'ucunda, G ya da C, sahip, (iii), dengelenmiş bir GC vardır: bir oranda, (iv) yaklaşık bir tavlama sıcaklığı 55 - ilgi bölgenin aşağı 60 ° C ve (v) bağlama, en az 50 bp, daha iyi 100 bp net görüntü almak için.
- Primer uzatma reaksiyonuna sokulması
Not: primer uzatma reaksiyonu (cDNA sentez) şablonu RNA, yüksek miktarda gerektirir. Kullanılan RNA miktarları düşük bir şekilde seçilir, sinyal tespit etmek için çok düşük olabilir! Yukarıda tarif edildiği gibi biz bu nedenle RNA saflaştırma önerilir.
NOT: DİKKAT: kullan RNazsız miyar ve plastik eşya !!!- 95 ° C'lik bir sıcaklığa değin ısı-cycler önceden ısıtmak ve kullanım ve çoğaltılabilirlik kolaylığı için bir termo-döngü cihazı, diğer tüm inkübasyon adımları gerçekleştirmek.
- Her RNA numunesinin bir PCR tüpü Tablo 2'den elde edilen bileşikleri karıştırın.
- 95 ° C'de 1 dakika boyunca numune denatüre.
- RNA ve primerler hibridize etmek için 5 dakika boyunca buz ve soğuk tüpler yerleştirin.
- 47 ° C'ye kadar PCR ayarlamak.
- Tablo 3'te tarif edildiği gibi arada bir ters transkripsiyon ana karışımı hazırlayın.
- Her hibritlenmiş RNA ters transkripsiyon ana karışımı 4 ul ekleyinörnek.
- 47 ° C de 1 saat süreyle inkübe edin. Not: AMV RT için optimum sıcaklık 42 ° C'dir, ancak daha yüksek sıcaklıklarda, RNA moleküllerinin ikincil yapılar örterler.
- 2 dakika boyunca 95 ° C'ye kadar numunelerin ısıtılmasıyla reaksiyonu durdurun.
NOT: Formamid, korozif, toksik ve karnındaki çocuğa zararlı olabilir. , Malzeme güvenlik bilgi formlarını okuyun dikkatle ele almak ve uygun bir koruma giymek Lütfen! - Karanlıkta -20 ° C'de O / N ve mağaza iki hafta (a / h) bromofenol mavisi% 0.05 w (% 95 (h / h), deiyonize formamid, 10 mM EDTA), formamid yükleme boyası 6 ul ekleyin.
Sıralama Merdiven 3. hazırlanması
NOT: sıralama merdiven reaksiyon plazmid ya da genomik DNA'nın yüksek miktarda orta miktarda ya gerektirir. Mümkün durumlarda sekans reaksiyonda plasmidlerin kullanımı nedeniyle izolasyonu ve yüksek SIG kolaylığı önerilirnal yoğunluğu. Diğer durumlarda, rutin E. genomik DNA hazırlamak için Marmur'un 5,14 benimsenen bir yöntem kullanabilirsiniz E. coli ve S. gerek kalmadan aureus hücreleri fenol kullanımı. Temel olarak, yüksek miktarda ve genomik DNA saflığı veren herhangi bir yöntem kullanılabilir.
- Genomik DNA izolasyonu
- E. 10 ml büyümek E. coli veya S. S. aureus hücreleri O / LB, BM 5 veya TSB ortamı içinde K.
- 15 ml Falcon tüpüne 4.600 x g'de 10 dakika boyunca santrifüjleme ile hasat hücreleri.
- Bazı mini hazırlık kitleri bulunan olarak 2 ml tampon P1 içinde yeniden süspansiyona alınmış pelet (50 mM Tris-HCI, pH 8.0, 10 mM EDTA, 100 ug / ml Rnaz A).
- 40 ul lysostaphin (-20 ° C'de, 0.5 mg / ml, depolama) - 20, 60 dakika - 45 Hücreleri. Not: E. İçin E. coli hücreleri, enzimatik ön arıtma ihmal edilebilir ya da lisozim kullanılır.
- Süspansiyon ve incubat üzere (% 45 etanol içinde), doymuş, SDS-çözeltisinin 100 ul ekle37 ° C'de 5 dakika boyunca, e.
- 650 ul 5 M NaClO 4 ve kısaca vorteks hücreleri ekleyin.
NOT: Kloroform potansiyel bir kanserojendir. Malzeme güvenlik bilgi formlarını okuyun ve uygun koruma ile bir davlumbaz altında kullanın lütfen !!! - Karışıma: (1 oranında 24) ve en az 60 saniye boyunca çalkalanır kloroform / izopentanol 3 ml ilave edilir. Not: Sıvı homojen bir beyaz emülsiyon haline dönmelidir.
- Fazlar ayrıldı C'de 10 dakika 4600 x g ve RT için santrifüj numunesi.
- Dikkatli bir şekilde yeni bir tüp açık üst (sulu) faz transfer. Çözelti bulanık olduğu takdirde, kloroform / izopentanol çıkarma tekrarlayın. DNA çözeltisinin hacmini ölçün ve etanol (% 100) 2 hacim ile yeni bir tüp hazırlanır.
- Yavaş süzün veya tüp içeren etanol içine DNA çözüm pipetle. Not: DNA alt veya tamamen susuz bir yüzen beyaz küme olarak şeffaf, yoğun bobinler çöktürmek gerekir.
- D AlUA cam Pasteur pipet (Şekil 2) elde edilen kancalar ile ve 1 ml% 70 etanol içinde bir ayrı tüp içine daldırma ile iki kez, her örnek yıkayın.
- Rafa dik kanca yerleştirin ve hava 60 dakika pelet kurulayın. Gerekirse, oda sıcaklığında birkaç gün boyunca kurutuldu DNA saklayın.
- 500 ul GKD 2 O - DNA kaplı cam kanca kopan ve 100 içeren 2.0 ml reaksiyon tüpüne koyarak DNA çözülür 1500 ng / ul - 1,000 nihai DNA konsantrasyonu ses seviyesini ayarlayın. Genomik DNA, 18 ug - tek bir dizileme reaksiyonu için, 10 kullanımı.

Şekil DNA olta nasıl oluşturulacağı 2. Talimat. Bunsen brülör alev içine bir cam Pasteur pipet ucu tutun. Bu t küçük bir kanca oluştururken, birkaç saniye sonra erimeye başlaması camını neden olurdiye biter. Çabuk alev kaldırmak ve 1 dakika boyunca soğumaya bırakın.
- Plasmid İzolasyon
- Standart mini hazırlık kiti kullanılarak plazmidler hazırlanması ve elüsyon tamponu (10 mM Tris-Cl, pH 8.5) içinde çözülür. Bir sıralama merdiven için plazmidin 500 ng - plazmid boyutuna bağlı olarak, 100 kullanımı.
- Sanger dizileme reaksiyonu
Not: astar uzantılarının amaç için çalışır 7-deaza-dGTP bir floresan etiketli primer dizileme kiti kullanarak basit bir protokol altında bak. Ayrıntılı bilgi için dizileme kiti kılavuzuna başvurun. Dizileme reaksiyonu, aynı uzunlukta ürünleri oluşturmak için primer uzatma reaksiyonuna sokulması ile aynı primer kullanılması gerektiğine dikkat ediniz.- 1 ul DMSO, 1 ul floresan etiketli primer (/ ul 2 pmol) - ile (15 ug ~ 10) genomik DNA, 12 ul karıştırın.
- Dört dizi Reaksiyon karışımları (A, C, G veya T) her birinden 1 ul için, DNA / DMSO / primer karışımı için 3 ul ekleyin.
- PCR makinenin içine numuneler ve aşağıdaki PCR programı çalıştırmak: 2 dakika boyunca 95 ° C; 20 saniye, 20 saniye boyunca 54 ° C, 30 sn için 70 ° C sıcaklıkta 95 ° C'lik 35 döngü; 4 ° C'de sonsuza dek tutmak.
- Run sonra, makineden örneklerini kaldırmak hafta birkaç gün boyunca buz (kısa vadeli) veya -20 ° C'de üzerinde yükleme boyası ve mağaza 6 ul ekleyin.
4. Jel Kurulum ve Aparatı Run
Not: dizileme jeli üzerine Cihaz monte edildiğinde ilgili ayrıntılı bilgi, jel hazırlanır ve nasıl jel çalıştırılan üretici protokolünde bulunabilir.
- Hazırlıklar
- Tablo 4'te gösterildiği gibi 10x TBE hazırlayın.
- Jel çalışma gününde, aşırı saf GKD 2 O ile 1 x TBE tampon maddesi 1 L 'nin Hazırlanması
- % 10 (ağ / hac) APS hazırlayın. Not: birkaç ay boyunca -20 ° C'de, 200 ul alikolar içinde saklanabilir, fakat aktivitesi zamanla azalabilir <./ Li>
- Jel döküm odasının Meclisi
- Cam plakalar arasında toz ve tüy bırakmayan kaçının. Islak mendil kullanarak nedenle iyice temiz çalışma yüzeyleri.
- Cam plakaların iç tarafı için izopropanol daha sonra her iki tarafta tek kullanımlık kağıt havlu ve damıtılmış su kullanılarak 25 cm 'lik cam bir çift levha temizleyin ve.
- (Şekil 3), arka cam plaka üzerinde 0.25 mm pulları yerlerine yerleştirin ve üstüne çentikli cam plakasını indirin.
- Çentikli ucu ile cam plakalar ve yukarı bakacak ray giriş pilotların her iki taraf için jel rayları takın ve hafifçe düğmeleri sıkın.

Şekil 3. jel elektroforez cam plakalar açılmış görünüşüdür. Cam levhalar yönlü olarak kullanılmalıdır. Içeri cam plakaların iç tarafı ve dış yüzü için özendışa doğru yan.

Şekil 4 monte edilmiş bir jel tertibatına görünümü. Jel çözeltisi enjekte edildikten sonra, cep boşluk cam levhalar arasındaki çözeltisi yerleştirilir. Döküm plaka daha sonra ön cam plaka ve jel raylar arasına kaydırdı ve demiryolu topuzlar sıkarak sabitlenir.
- Jel Döküm
NOT: sigara polimerize akrilamid nörotoksiktir! Malzeme güvenlik bilgi formlarını okuyun ve uygun koruma ile kullanın lütfen !!!- Bir beher içine, Tablo 5'de listelenen bileşikler ilave edin ve bir karıştırma çubuğu ve manyetik bir karıştırıcı kullanılarak karıştırılır.
- Hemen APS ve TEMED ekledikten sonra, 50 ml şırınga içinde jel çözümü sürebilir ve ucunda bir 0.45 nm filtre yerleştirin.
- Ya tek elle cam plakanın üst kenarını tutun veya panel duvarları yerleştirin20 ° - bir jel döküm h 10 açılı bir eğim oluşturmak için stand.
- Yavaş yavaş sürekli olarak, diğer bir taraftan, şırınga ucu hareket ederken cam plakalar arasında jel çözelti tevzi ve jel çözeltisi alt ucu yerine getirdiği durdurun.
- Tarafına hareket ettirin ya da tamamen kabarcık kanca kullanarak herhangi kurdu kabarcıklarını çıkarın.
- Çentikli sonunda cam plakalar arasındaki jel cep parçasını (0.25 mm) kaydırın, jel çözeltisi içine daldırın ve döküm plaka takarak sabitleyin.
- Sabitleyin üst ray vidaları hafifçe (tamamen monte aparatı için Bakınız Şekil 4).
- 2 saat - 1 jel seti edelim.
- Döküm plaka ve cep ayırıcı kaldırmak ve tuz ve jel kalıntılarından cebi temizleyin.
- GKD 2 O ile durulayın ve doku kağıtları ile fazla çözüm silin.
- Koşu ve jel görüntülenmesi
Not: sekans oluşturma jellerinde doğrudan ise, jel görüntüleyici içinde elektroforez işlemine tabi tutulurFloresan aynı anda bir lazer mikroskobu ile tespit edilir. Jel, ilk çalışma ve daha sonra lekeli ve görselleştirildi geleneksel jel elektroforezi, aksine, algılama birimi sabitlenir ve lazer geçerken gerçek zamanlı olarak bantları tarar. Daha yeni sürümleri için kabul edilebilir özetlenen OS / 2 ImagIR Veri toplama yazılımı için bir prosedür aşağıda. Daha fazla bilgi için kullanım kılavuzuna bakın.- Ön cam plakalar üzerinde jel rayların içine tampon tankı yuvasını ve düğmeleri sıkın.
- Isıtma plakası karşı otomatik jel görüntüleyici alt jel tankına jel yerleştirin ve cihaz parantez içine ray giriş pilotu kaydırarak düzeltmek.
- Güç kablosunu kullanarak iktidara üst tampon odasını alt tampon odasına yakın ve bağlamak, alt ve üst jel tampon odalarına 1x TBE tamponu doldurun.
- Eğer varsa, tekrar tekrar cebine tampon pipetle tuz-kalıntılarından jel cebi temizleyin.
- Üst tampon kapağı kullanılarak üst tampon tankı odasına kapatın.
- Makine kapılarını kapatın ve görüntüleyici ve bilgisayar açmak ve Taban ImagIR Veri toplama yazılımını başlatın.
- Yeni bir proje dosyası (Dosya-> Yeni ...) oluşturmak, proje dosya adı girin uygun lazer aralıkları (700 veya 800 nm) seçin ve OK ile onaylayın.
- Seçenekleri-> Otomatik kazanç ... üstündeki görüntü menüden seçin, otomatik kazanç ölçümünü başlatmak ve OK tıklayarak ayarları kabul etmek için Otomatik seçeneğini tıklatın.
- Odak ... tarayıcı kontrol menüsünden, Otomatik düğmesini tıklayarak ve OK tıklayarak ayarları kabul> Options seçerek lazer odaklanın.
- Yeni odaklı bölgeye ayarlamak için otomatik kazanç işlemi tekrarlayın.
- Kur bu ayarlara göre tarayıcı kontrolü: 2000 V, 35 mA, 45 W, 45 ° C, Tarama filtresi: 3, Tarama hızı: 3.
- 20 dakika boyunca boş jel (seçme gerilimi AÇIN ve basın İşlem öncesi ).
- Bu arada, daha sonra, 90 ° C, 2 dakika için bir PCR makinesi içinde sıralama merdiveni ve primer uzatma ürünleri ısıtmak buz üzerinde soğutulur.
- , Elektroforez durdurmak otomatik jel sequencer açın ve üst tampon tankı kapağını çıkarın.
- Cam levha arasına köpekbalığı diş tarak yerleştirin ve hafif köpekbalığı dişleri ile jel delmek (bakınız Şekil 5).
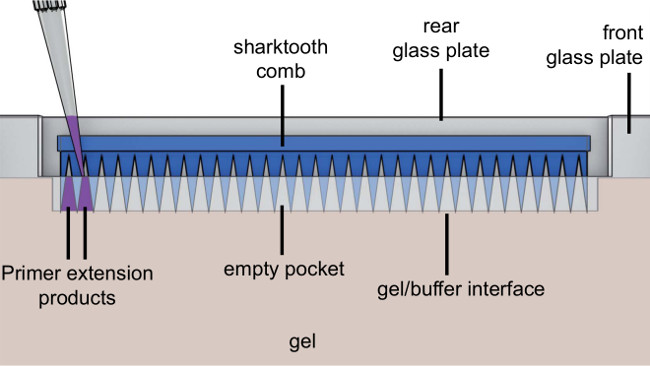
Şekil 5. Close-up köpekbalığı dişli tarak ile jel görünümü. Örnek (mor) köpekbalığı dişleri arasında uygulanır.
- Pipet ya 1 - (köpekbalığı dişleri tarafından oluşturulan) her jel cebine uzatma ürünleri veya sıralama merdiven reaksiyonları primer 2 ul.
- Tüm cepler ihtiyaç varsa, önlemeye yükleme boyası ile boş ceplerini doldurmaktutarlı çalışma davranışı.
- Jel sequencer tampon tankı ve kapağını kapatın.
- Elektroforez başlatın ve lazer (Seç Gerilim ON ve Lazer AÇIK ve açmak için tuşuna basın ) .
- Ilgi bölgesi lazer geçtikten sonra elektroforez durdurun.
Sonuçlar
Şekil 6'da gösterildiği gibi, bir primer uzatma reaksiyonu ilgi transkript transkripsiyonel başlangıç noktalarını belirlemek için kullanılabilir ve (tipik olarak -10 ila -35 elemanlar tarafından tanımlanan) hızlandırıcı bölgeler anlamak için yardımcı olabilir. En üstteki (uzun) cDNA parçası, mRNA 5 'ucunu temsil eder ve bu sıralama merdiven göre kolayca eşlenebilir.

Bir primer uzatma reaksiyonuna sokulması Şekil 6. Örnek sonucu. Sol tarafta E. tam bir in vivo olarak bir primer uzatma jelinde Çeşitli plasmidler ile E. coli gösterilmiştir. Ilgi Bireysel alanları genişlemiştir. En iyi (A) parçası olarak, ompA transkripsiyonel başlangıç noktasının belirlenmesi gösterilmektedir. Ters transkripsiyon mRNA'nın 5 'ucunda durur ve bu ind (tam uzunluktaki RNA bir bant oluşturur) bir ok ile gili. Ekteki sekans gösterildiği gibi sıralama merdiveni cDNA bant hizalayarak, mRNA'nın 5 'ucu belirlenebilir. Alt parçanın (B) 'de, YoeB-seq2 RNaz ompA transkriptin ayrılması gösterilmiştir. Geçitler 1 - yokluğunda ve cDNA ürünlerinin varlığı ile örtüşen, 5 aktif RNase ile numunelerini - 3 4 şeritli ise toksin YoeB-seq2 eksik veya inaktif olduğu örnekleri, temsil eder. Oklarla gösterildiği üzere, iki ana parçalanma ürünleri oluşturulur. Her kuşaktan ve sıralama merdivenin karşılık gelen RNA, baz kullanılır ddNTP'yi edilir. Transkript parçalar eflatun yazı kodon mavi yazı, promotör elemanların ve Ağustos başından ile gösterilir. Daha fazla bilgi için; Takipsizlik ark gelen orijinal araştırma makalesine bakın., Mikrobiyoloji 159, 1575-1585 (2013). için tıklayınızBu rakamın büyük bir versiyonunu görmek.
Şekil 6'da gösterilen örnekte, ompA mRNA TSP (jel aşağıdaki sıraya göre, bir ok ile işaretlenmiştir) G taban olarak belirlendi. Bu 15 önce yayınlanmış ompA TSP ile tutarlıdır. Motif sadece ilgili konsensüs dizilerinin (sırasıyla TTGACA ve TATAAT) için iki baz her bir farklı olarak promotörünün -35 ile -10 elemanları TTGTAA ve TAGACT 16 olacak şekilde çıkarılabilir.
Örneğin 5 'RACE gibi diğer yöntemlerin aksine, primer uzatma reaksiyonları hassas olarak belirlenmesi ve RNA moleküllerinin bölünmesini ölçmek için kullanılabilir. RNA moleküllerinin bölünmesi aynı jel cDNA bantlar olarak tespit edilebilir serbest 5 'uçları oluşturur. Birkaç PCR ürünü, parçalanma ürünlerinin elde edilmesi için dizilenmiştir zorunda olması yüzünden tek bir mRNA bazı parçalanma ürünlerinin belirlenmesi, 5 'RACE deneyleri, daha zordursadece toplam (işlenmemiş) RNA toplu küçük bir bölümünü temsil etmektedir.
Şekil 6'nın alt kısmında, bir RNaz bölünme RNA haritalama tasvir edilmiştir. Önce 5, RNaz YoeB-seq2, S., TA-sisteminin bir parçasını tarif edildiği gibi 17 equorum, başlangıç kodonu yakın cleaves mRNAlar. Bu ayrışma, aynı kökenli antitoksin YefM-seq2 ile inhibe edilebilir. RNaz YoeB (kulvarlar 1-3) mevcut veya etkin olmayan olduğu zaman, primer uzatma ürünleri yakın başlangıç kodonu için oluşturulmuştur. RNaz etkinliği kısa bir süre aşağı kodon görünen başlangıç mevcut (kuşak 4 ve 5), iki güçlü bant olduğunda. Bu yaklaşımı kullanarak, klivaj paternleri kolayca tespit edilebilir.
Şekil 7, bir başarısız primer uzatma deneyi gösterir. Ters transkripsiyon reaksiyonunda fazla RNA, cDNA üretilen miktarının bu türden bir güçlü bir sinyal oluşturur nedeniyle, bu, tek bir cDNA bants ayırt edilemez. Bu 5 'RNA sonu belirlenmesi imkansız hale getirir.
(Şekil 7'de gösterildiği gibi) bu tür bir ribozomal RNA gibi primer uzatma reaksiyonunda yüksek derecede ifade edilen RNA, kullanılırken aşırı maruz kalan alanlar riski artar. Bu şekilde, toplam şablon RNA miktarının söz konusu RNA çokluğuna göre ayarlanması gerekir. Ilgi transkript sadece toplam RNA küçük bir kısmını oluşturur Bunun tersine, ters transkripsiyon reaksiyonunda miktarının da sinyaller çok soluk olacaktır arttırılmalıdır (gösterilmemiştir). Bu aynı zamanda çok daha güçlü sinyallerine neden (örneğin rRNA genleri veya plazmidler üzerinde kodlanmış genler gibi) hücre başına multikopya şablon olarak, Sanger dizileme reaksiyonu için geçerlidir.
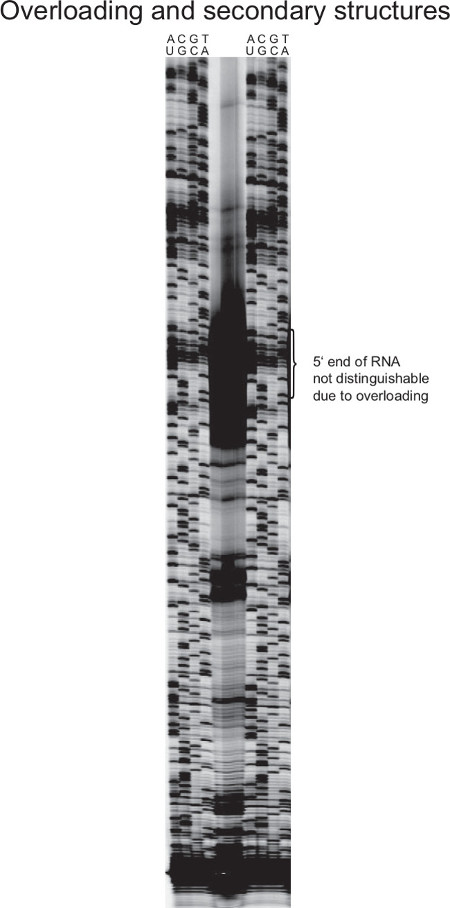
Şekil 7. başarısız bir primer uzatma Temsilcisi sonucu ng> S. aureus. 16S RNA bakteri hücrelerinde oldukça boldur. Toplam RNA'nın orta miktarlarda kullanıldığında, bu mutlaka ters transkripsiyon sinyalleri yol açar. Burada tanımlanan durumda, bu kuvvetli cDNA bantları tam bir 5 'ucuna ve bu nedenle 5 önlemek' maske eşleme. Buna ek olarak, ribozomal RNA'lar derece yapılandırılmış ve bu nedenle tam uzunlukta parçaları temsil etmemektedir kısa cDNA ürünleri üreten, zamanından önce ters transkripsiyonu kesebilirler. Isıya dayanıklı bir ters transkriptaz ile birlikte, ters transkripsiyon artan sıcaklık Bu durumda, daha kolay geçen ikincil yapılar sentezlenmesi için yapabilir. Nedeniyle genomik DNA dizisinin çoklu kopya, Sanger dizileme merdiveni tek kopyalı gen için daha güçlüdür. Jel resmi iyileştirmek için, RNA ve DNA, ters transkripsiyon için miktarları ve Sanger reaksiyonu, azaltılmalı ve / veya daha az ürün jele tatbik._upload / 52134 / 52134fig7large.jpg "target =" _ blank "> Bu rakamın büyük bir versiyonunu görmek için lütfen buraya tıklayınız.
Tablo 1: RNA, DNase I sindirimi.
| Madde | Miktar |
| Yukarıdaki aşama RNA, | 70-100 ug |
| 10x DNase I tamponu (100 mM Tris, pH 7.5, 25 mM MgCl2, 2 5 mM CaC) | 50 ul |
| DNase I, RNaz içermeyen (2 U / ml) | 10.0 ul (RNA kadar 10 ug DNase 1 ul için) |
| GKD 2 O ile 500 ul hacim getirmek (DEPC tedavi) | |
DNAaz'ın reaksiyon hacmi ve miktarı ben miktarı o bağlı büyütülüyor ya da aşağı olabilirf RNA kullanılmıştır.
Tablo 2: RNA Astar melezleme.
| Bileşik | Bir reaksiyon için Miktarı |
| RNA | 5-15 ug |
| Floresan etiketli astar | 2 pmol |
| GKD ile 6 ul hacmi getirin 2 O (DEPC tedavi) | |
RNA örnek ve primer başına bir reaksiyonu kurmak.
Tablo 3: Primer uzatma master karışımı.
| Bileşik | Bir reaksiyon için Miktarı |
| GKD 2 O (DEPC işlenmiş) | 1.3 ul |
| AMV RT Buffer (10x) | 1.0ul |
| dNTP (10 mM, RNazsız) | 1.0 ul |
| RNaz inhibitör | 0.2 ul |
| AMV Ters Transkriptaz (RT) (20-25 U / ul) | 0.5 ul |
Gerekli reaksiyonların sayısı büyütmek.
Tablo 4: 10x TBE tarifi.
| Madde | Miktar |
| Tris baz | 107.8 g |
| Borik Asit | 55.0 gr |
| EDTA | 7.4 gr |
| Tozu ve kiri için ultra saf GKD 2 O ve filtre ile 1,000 ml hacim getirmek | |
4 ° toz ve tüy bırakmayan ve deposunu kaldırmak için filtreC.
Tablo 5: Dizi jel tarifi.
| Bileşik | Miktar |
| Üre | 10.5 gr |
| GKD 2 O (MilliQ) | 13.0 mi |
| 10x TBE | 2.5 mi |
| XL, hızlı jel çözeltisi, | 5.0 mi |
| TEMED (N, N, N ', N'-Tetramethylethane-1,2-diamin) | 25 ul |
| APS (amonyum persülfat,% 10) | 175 ul |
Hızlı TEMED ve APS ilave edildikten sonra işlem jel çözeltisi.
Tartışmalar
Floresan primer uzatma TSP- veya ikincil bir RNA işleme tanımlanması için iki, RNA'ların 5 'uçlarını belirlemek için basit ve hızlı bir yöntemdir. Dolayı floresan primerlerin kullanımı ile reaksiyonlar, ayarlanabilir ve (radyoaktif olarak etiketlenmiş primerler aksine olarak) ilave güvenlik önlemleri olmadan çalışabilir. Numuneler floresan ile tespit edilir elektroforez X-ışını filmleri yaygın olarak kullanılan radyoaktif yöntemlerle karşılaştırıldığında hızlı bir analizine izin verir işlemi sırasında, bunlar görüntülenebilir.
Genel olarak, primer uzatma reaksiyonuna sokulması kalite derecesi ile primerin bağlama özelliklerine sıkı bir şekilde bağlıdır. Bağlama sahası ilgi alanına çok yakın seçilirse, astar smear sinyali maskeleyebilir, bir bağlanma ise çok uzakta (> 300 bp), 5 'ucundan bir zayıf sinyal ile sonuçlanabilir.
Floresan boyasının kovalent olarak bağlanmış olması gerekirSentez ve oligonükleotidin sırasında özel bir DNA oligonükleotidin 5 'ucu kalıntı tuzları, ters transkripsiyon reaksiyonu engel olmamak için HPLC ile arındırılmalıdır. Uygun boya modifikasyonu (uyumlu boyalar hakkında daha fazla bilgi için reaktifler liste tabloya bakınız) ile Oligonükleotidler oligonükleotid sentezi şirketlerden sipariş edilebilir ve karanlıkta saklanmalıdır. Radyo-etiketli nükleotidler ile mümkün olduğu gibi Ne yazık ki, daha önce mevcut oligonükleotitler için boya bağlamak için bir enzimatik tekniklerin farkında değildir.
Burada anlatılan dizileme sisteminde, iki farklı floresan boyalar, uyarılma gibi, aynı anda iki numune tespit etmek için (yaklaşık 700 ve 800 nm) kullanılır ve emisyon spektrumlarına farklı girmektedir. Yanı sıra orijinal üretici boyalar gibi reaktifler listede belirtildiği gibi diğer boyalar mükemmel sonuçlar elde etmek için kullanılabilir.
Pr için diğer önemli faktörimer yapılan uzatma reaksiyonları, RNA kalitesi ve miktarı. Bakım böyle AMV RT olarak ters transkriptazlar şablon 18 olarak DNA gibi kullanabilirsiniz, DNA kirleri çıkarmak için önlemler alınmalıdır.
Şekil 7'de gösterildiği gibi, jel vadede bantların bulgulanan sinyal gücü, ters transkripsiyon reaksiyonunda kullanılan RNA miktarına bağlıdır. Bu numunede ilgi mevcut olan RNA'nın nispi miktarına bağlı olarak, toplam RNA miktarını ayarlamak için elzemdir. Düşük ifade RNA'lar tespit etmek zor olabilir, bu yöntemin düşük duyarlılık, aynı zamanda dezavantajlarından biridir. Herhangi bir yayın her tespit edilebilir ise, toplam RNA miktarı arttırılabilir veya ilgi duyulan RNA yapay bir plazmidden fazla ifade edilebilir.
Primer uzatma bazlı floresan için jel algılama hassasiyeti primer uzatma 19 tabanlı 32 P veya 33 P radyoizotop daha düşük faktörü on hakkında. Ancak, bu dezavantaj, jel ya da ters bir transkripsiyon reaksiyonunda kullanılan RNA şablonunun miktarının arttırılmasıyla yüklü cDNA miktarını ayarlayarak telafi edilebilir. Çoğu durumda, hassasiyet tatmin edici sonuçlar 4,5 'için yeteri kadar yüksektir. Kısa pozlama süreleri avantajı ile, bir kılcal sequencer 3 ile birlikte floresan primerler kullanarak radyoaktif primer uzantıları benzer duyarlılıkları bildirilmiştir.
Böyle uygun makinelerde, astar, merdiven reaksiyonlar, kullanılabilirlik ve radyoaktif maddeler, radyoaktif atıklar, eğitim ve bireysel sağlık riskleri bertaraf çalışması için bir laboratuvar bakımı gibi çeşitli faktörlere bağlıdır astar uzantıları bazlı floresan ve radyoaktivite ile karşılaştırmalar, zor Maliyetleri . Floresan primerler etiketli olmayan standart primerler (20 bp), yaklaşık 5-10 kat daha pahalıdır. Bununla birlikte, bu primerler, en az kullanılabiliryılda çok daha kısa bir yarılanma ömrüne sahip 32P etiketli primerlerin ile karşılaştırıldığında (muhtemel birkaç yıl). Yeniden etiketleme primerler nedeniyle sık aralıklarla yeni radyoaktif malzeme için ihtiyacı, zaman alıcı ve maliyetlidir. Primerler, aynı resim, uzun bir süre boyunca kullanıldığı takdirde, floresan primer normal, radyoaktif olarak etiketlenmiş primerler daha ucuzdur. Ana maliyet nokta ancak floresan sequencer veya görüntüleyiciyi ve maliyet-etkili olmayabilir primer uzantıları amacıyla sadece bu cihazı satın olacaktır. Burada açıklanan yöntem sahip olan veya bu tür bir makineye erişimi gruplar için oldukça ilginçtir.
Otomatik bir jel dizi mevcut değilse, flüoresan tespiti için diğer yöntemler de kullanılabilir. Bu durumda, jel gerekirse, kurutuldu ve daha sonra bir floresan görüntüleyici aktarılır, standart bir elektroforez aygıtı içinde çalıştırılabilir (modelleri fl benzer olarak temin edilebiliratbed tarayıcılar). Çalışma sırasında jel görselleştirme avantajı kaybı olmasına rağmen, radyoaktif izotoplar kullanımı deneyi için bu şekilde, önemli bir avantaj önlenebilir. Buna ek olarak, eğer varsa, bir kılcal dizi duyarlılığını artırmak için yardımcı olabilir ayırma ve gerçek zamanlı saptama için kullanılabilir.
Çeşitli yöntemler, ancak bu yöntemlerin sık sık 19 (kirleri ve çıkarmak) örnek konsantre bir cDNA çökeltme adımı gerektiren, otomatik jel sıralama makineleri veya kılcal sequencer ile floresan astar uzantısı nasıl kullanılacağı yayınlanmıştır. Burada yer alan yöntemde, DNA, çöktürme ve böylece işlem süresini azaltmak gerekli değildir. Çökeltme işleminin giderilecek gibi yarı kantitatif numunede RNA moleküllerinin miktarının belirlenmesi için Ancak daha da önemlisi, bu adımı atlayarak mümkün kılar.
Bu, kaydetti, ancak o edilmelidirRNA moleküllerinin 5 'uçları, primer uzatma reaksiyonları eşleştirilir işlenmiş ve birinci uçlarının (transkripsiyonel başlangıç noktalarının) kolayca ayırt edilemez olabilir. Bu kısıtlamaları aşmak için, uygun kontroller ile deneylerin titiz bir planlama esastır. Apaçık promotör dizileri transkripsiyon başlangıç noktasının varlığını gösterebilir ve mutasyona veya silme promotör elemanları, daha sonra cDNA bantları ortadan kaldırmak gerekir. Bu tür sekanslar, eksik ya da spesifik diziler mevcut olması halinde, diğer yandan, bantlar, RNA işleme neden olabilir. Işlem enzimi bilinir ve saflaştırılabilir durumunda, in vitro olarak astar uzantıları transkripsiyon gibi, açıklama getirebilir ve işleme ayrılabilir. Buna ek olarak, (işlenmemiş RNA moleküllerinin enzimatik zenginleştirilmesi de dahil olmak üzere) için diğer yöntemler, örneğin, 5 'RACE RNA işleme ile ilgili transkripsiyon başlangıç the ayırt primeri uzantıları tamamlayabilir.
The primer uzatma yöntemi genellikle 5 'RACE ve S1 nükleaz koruma deneyleri ve dolayısıyla yararlılığının bazen sorgulandığı gibi diğer yöntemlerle karşılaştırıldığında. Örneğin yeni nesil dizileme ile birlikte RNAseq teknikleri ancak gerekli mali yük ve Biyoinformatik çalışma ilgi tek RNA'lar için oldukça ekonomik olmayan yapmak TSPs ve paralel RNA'ların birçok işleme siteleri belirlemek için yardımcı olabilir. Öte yandan 5 'RACE ucuzdur ve sonuçlar analiz etmek daha kolaydır, ancak RNA birden çok şekilde ya da daha fazla transkripsiyonel başlangıç noktalarının içinde mevcut olan işleme durumunda, ürün klonlanmış olması ve bir aday büyük miktarda için dizilenmiştir gerekir ilgilenilen bir RNA'lar temsil eden bir bakış elde edilir.
Yeni yöntemler inadına yılda ortaya çıkmış olan bu nedenle, bugün bile primer uzantıları nedeniyle özellikle doğrudur kullanımı, düşük maliyet ve kısa gerçekleştirme süresi kolaylığı, onların varoluş nedeni varBurada yer floresans bazlı yöntem.
Açıklamalar
The authors have nothing to disclose that would present a conflict of interest.
Teşekkürler
We thank Anne Wochele for her assistance in the laboratory and Vera Augsburger for help with the automated gel sequencer. We thank the Deutsche Forschungsgemeinschaft for funding by grants BE4038/2 and BE4038/5 within the “priority programmes” SPP1316 and SPP1617.
Malzemeler
| Name | Company | Catalog Number | Comments |
| AMV Reverse Transcriptase (20-25 U/µl) | NEB / Roche | NEB: M0277-T / Roche: 10109118001 | |
| DNase I (RNase free) | Ambion (life technologies) | AM2222 | |
| FastPrep-24 Instrument | MPBio | 116004500 | |
| Fluorescently labeled primers | Biomers | n/a | 5’ DY-681 modification of “ordinary” DNA oligonucleotides. Compatible dyes such as the LICOR IRDye 700/800 are also available from other suppliers such as IDTdna. |
| Li-Cor 4200 Sequencer incl. ImagIR Data collection software | Li-Cor | Product discontinued | |
| NanoDrop 2000 | Thermo Scientific | ||
| Nuclease free water | Ambion (life technologies) | AM9915G | |
| Plasmid mini preparation kit | QIAGEN | 12125 | |
| RapidGel-XL-40% Concentrate | USB | US75863 | |
| RNA STORAGE BUFFER | Ambion (life technologies) | AM7000 | |
| Roti-Aqua-P/C/I | Carl Roth | X985.3 | Alternative: “Acid-Phenol:Chloroform, pH 4.5 (with IAA, 125:24:1)” from Ambion (AM9720) |
| SUPERase•In RNase Inhibitor | Ambion (life technologies) | AM2696 | |
| Thermo Sequenase fluorescently labelled primer cycle sequencing kit with 7-deaza-dGTP | GE Healthcare | RPN2538 | |
| TRIzol reagent | life technologies | 15596-026 | |
| Zirconia/Silica Beads 0.1 mm | BioSpec | 11079101z |
Referanslar
- Simpson, C. G., Brown, J. W. Primer extension assay. Methods Mol. Biol. 49, 249-256 (1995).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proc. Natl. Acad. Sci. U. S. A. 74, 5463-5467 (1977).
- Fekete, R. A., Miller, M. J., Chattoraj, D. K. Fluorescently labeled oligonucleotide extension: a rapid and quantitative protocol for primer extension. Biotechniques. 35, 90-94 (2003).
- Schuster, C. F., et al. Characterization of a mazEF Toxin-Antitoxin Homologue from Staphylococcus equorum. J. Bacteriol. 195, 115-125 (2013).
- Nolle, N., Schuster, C. F., Bertram, R. Two paralogous yefM-yoeB loci from Staphylococcus equorum encode functional toxin-antitoxin systems. Microbiology. 159, 1575-1585 (2013).
- Yamaguchi, Y., Park, J. H., Inouye, M. Toxin-antitoxin systems in bacteria and archaea. Annu. Rev. Genet. 45, 61-79 (2011).
- Schuster, C. F., Bertram, R. Toxin-antitoxin systems are ubiquitous and versatile modulators of prokaryotic cell fate. FEMS Microbiol. Lett. 340, 73-85 (2013).
- Goeders, N., Van Melderen, L. Toxin-antitoxin systems as multilevel interaction systems. Toxins. 6, 304-324 (2014).
- Yamaguchi, Y., Inouye, M. mRNA interferases, sequence-specific endoribonucleases from the toxin-antitoxin systems. Progress in molecular biology and translational science. 85, 467-500 (2009).
- Park, J. H., Yamaguchi, Y., Inouye, M. Bacillus subtilis MazF-bs (EndoA) is a UACAU-specific mRNA interferase. FEBS Lett. 585, 2526-2532 (2011).
- Zhu, L., et al. et al.Staphylococcus aureus MazF specifically cleaves a pentad sequence, UACAU, which is unusually abundant in the mRNA for pathogenic adhesive factor SraP. J. Bacteriol. 191, 3248-3255 (2009).
- Zhu, L., et al. The mRNA interferases, MazF-mt3 and MazF-mt7 from Mycobacterium tuberculosis target unique pentad sequences in single-stranded RNA. Mol. Microbiol. 69, 559-569 (2008).
- Fu, Z., Donegan, N. P., Memmi, G., Cheung, A. L. Characterization of MazFSa, an endoribonuclease from Staphylococcus aureus. J. Bacteriol. 189, 8871-8879 (2007).
- Marmur, J. A procedure for the isolation of deoxyribonucleic acid from micro-organisms. J. Mol. Biol. 3, 208-218 (1961).
- Emory, S. A., Belasco, J. G. The ompA 5' untranslated RNA segment functions in Escherichia coli as a growth-rate-regulated mRNA stabilizer whose activity is unrelated to translational efficiency. J. Bacteriol. 172, 4472-4481 (1990).
- Cole, S. T., Bremer, E., Hindennach, I., Henning, U. Characterisation of the promoters for the ompA gene which encodes a major outer membrane protein of Escherichia coli. Mol. Gen. Genet. 188, 472-479 (1982).
- Schleifer, K. H., Kilpper-Bälz, R., Devriese, L. Staphylococcus arlettae sp. nov., S. equorum sp. nov. and S. kloosii sp. nov.: Three New Coagulase-Negative, Novobiocin-Resistant Species from Animals. Syst. Appl. Microbiol. 5, 501-509 Forthcoming.
- Yu, H., Goodman, M. F. Comparison of HIV-1 and avian myeloblastosis virus reverse transcriptase fidelity on RNA and DNA templates. J. Biol. Chem. 267, 10888-10896 (1992).
- Ying, B. W., Fourmy, D., Yoshizawa, S. Substitution of the use of radioactivity by fluorescence for biochemical studies of RNA. RNA. 13, 2042-2050 (2007).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır