Method Article
Fluorescencia Basado Técnica de extensión del cebador para determinar la transcripción puntos de partida y los sitios de escisión de RNasas
En este artículo
Resumen
We here describe a fluorescence based primer extension method to determine transcriptional starting points from bacterial transcripts and RNA processing in vivo using an automated gel sequencer.
Resumen
Fluorescencia de extensión del cebador basado (FPE) es un método molecular para determinar los puntos de partida de la transcripción o sitios de procesamiento de moléculas de ARN. Esto se consigue mediante la transcripción inversa del ARN de interés usando cebadores marcados con fluorescencia específicos y el análisis posterior de los fragmentos de ADNc resultantes mediante electroforesis en gel desnaturalizante de poliacrilamida. Simultáneamente, una reacción tradicional de secuenciación de Sanger se ejecuta en el gel para mapear los extremos de los fragmentos de ADNc a sus bases correspondientes exactas. En contraste con 5'-RACE (amplificación rápida de extremos de ADNc), donde el producto debe ser clonado y secuenciado múltiples candidatos, la mayor parte de fragmentos de ADNc generados por la extensión del cebador se puede detectar simultáneamente en una carrera gel. Además, todo el procedimiento (de la transcripción inversa para el análisis final de los resultados) se puede completar en un día laborable. Mediante el uso de cebadores marcados con fluorescencia, el uso de isótopos radiactivos peligrosos reactivos marcadosse pueden evitar y los tiempos de procesamiento se reducen como productos pueden ser detectados durante el procedimiento de electroforesis.
En el siguiente protocolo, se describe un método de extensión de cebador fluorescente in vivo para detectar de forma fiable y rápida los extremos 5 'de los ARN para deducir puntos de partida de la transcripción y sitios de procesamiento del ARN (por ejemplo, por componentes del sistema toxina-antitoxina) en S. aureus, E. coli y otras bacterias.
Introducción
La extensión del cebador 1 es un método molecular para determinar los extremos 5 'de moléculas de ARN específicas hasta una resolución una base. La ventaja de otros métodos tales como 5'-RACE (amplificación rápida de extremos de ADNc) es el tiempo de respuesta rápido y la capacidad de analizar fácilmente una mezcla de diferentes longitudes de moléculas de ARN.
Este método funciona sometiendo moléculas de ARN para revertir reacciones de transcripción utilizando cebadores fluorescentes específicas, generando fragmentos de ADNc de ciertas longitudes. Estas moléculas de ADNc se ejecutan junto tradicionales reacciones de secuenciación Sanger 2 en geles de poliacrilamida desnaturalizantes y pueden ser detectados por su fluorescencia debido a la utilización de cebadores marcados con fluorescencia. Las longitudes de los fragmentos de ADNc se evalúan por comparación con la escalera de secuenciación, lo que permite el mapeo de los extremos 5 'de ARN.
Tradicionalmente, las reacciones de extensión de cebadores se utilizan en combinacióncon isótopos radioactivos para detectar moléculas de ADNc de películas de rayos X. Debido a los peligros de salud, problemas de eliminación de residuos y la facilidad de manejo, nuevos protocolos utilizan la fluorescencia para la detección de la extensión del cebador con secuenciadores automatizados, aunque su sensibilidad es ligeramente inferior. El uso de cebadores marcados con fluorescencia, el procedimiento marcado radiactivo recurrentes se puede omitir, como cebadores fluorescentes son estables durante un largo tiempo (más de un año en nuestras manos).
El método se describe aquí utiliza un secuenciador automatizado de gel, pero con ligeras modificaciones, secuenciadores capilares puede también ser utilizado para la separación y detección de cDNA 3. La naturaleza paralela de análisis en gel hace que sea posible detectar incluso una pequeña cantidad de escisión de ARN o de procesamiento. Otra ventaja es la alta resolución de este método, como terminal de división o transformación de una sola base puede ser detectado.
En lo que se refiere a la detección de la escisión de ARN o el procesamiento, typically dos diferentes tipos de extensiones de imprimación se distinguen. En un caso, el tratamiento enzimático se realiza in vitro usando ARN purificado y enzima purificada, mientras que en el otro caso, el procesamiento se realiza in vivo y el ARN resultante se purifica. En ambos casos, el ARN se somete a una extensión del cebador lleva a cabo in vitro, sin embargo, dependiendo de la fuente de ARN, ya sea el método se llama una extensión del cebador vivo in vitro o in. En el protocolo que presentamos aquí, nos centramos únicamente en la extensión del cebador en vivo, debido a la facilidad de uso (no hay proteínas purificadas necesario) y la posibilidad de determinar los puntos de partida de la transcripción y procesamiento a la vez. Sin embargo, in vitro extensiones de cebadores son, en principio, establecer la misma manera y este protocolo pueden servir como punto de partida.
El método ilustrado aquí se puede aplicar a muchas especies bacterianas mientras que son susceptibles de altopreparación pureza y alto rendimiento de los ácidos nucleicos.
La investigación en nuestro laboratorio se centra en el alcance de la regulación de la toxina-antitoxina (TA) sistemas de 4,5, un campo en el que se utiliza ampliamente el método de extensión del cebador. TA-sistemas son pequeños elementos genéticos presentes en los genomas de procariotas que constan de una proteína tóxica estable y endógena activa y una proteína o ARN antitoxina en su mayoría inestables que contrarresta la toxicidad 6,7. Actividad de la toxina a veces se ejerce mediante la inhibición de la replicación, síntesis de la pared celular u otros mecanismos, pero más a menudo por la actividad de RNasa 8,9. Típicamente, la especificidad de RNasa se determina mediante la realización de diferentes pruebas, uno de los cuales es el método de extensión del cebador. Reacciones de extensión de cebador son muy adecuados para esta aplicación, como una mezcla de fragmentos de longitud escindidos y completo se puede analizar de forma simultánea para determinar sus extremos 5 '. Usando una mezcla de in vitro e in vivo extensiones de cebadores, latoxina RNasa escisión específica, por ejemplo, la especificidad de secuencia se puede determinar 10-13.
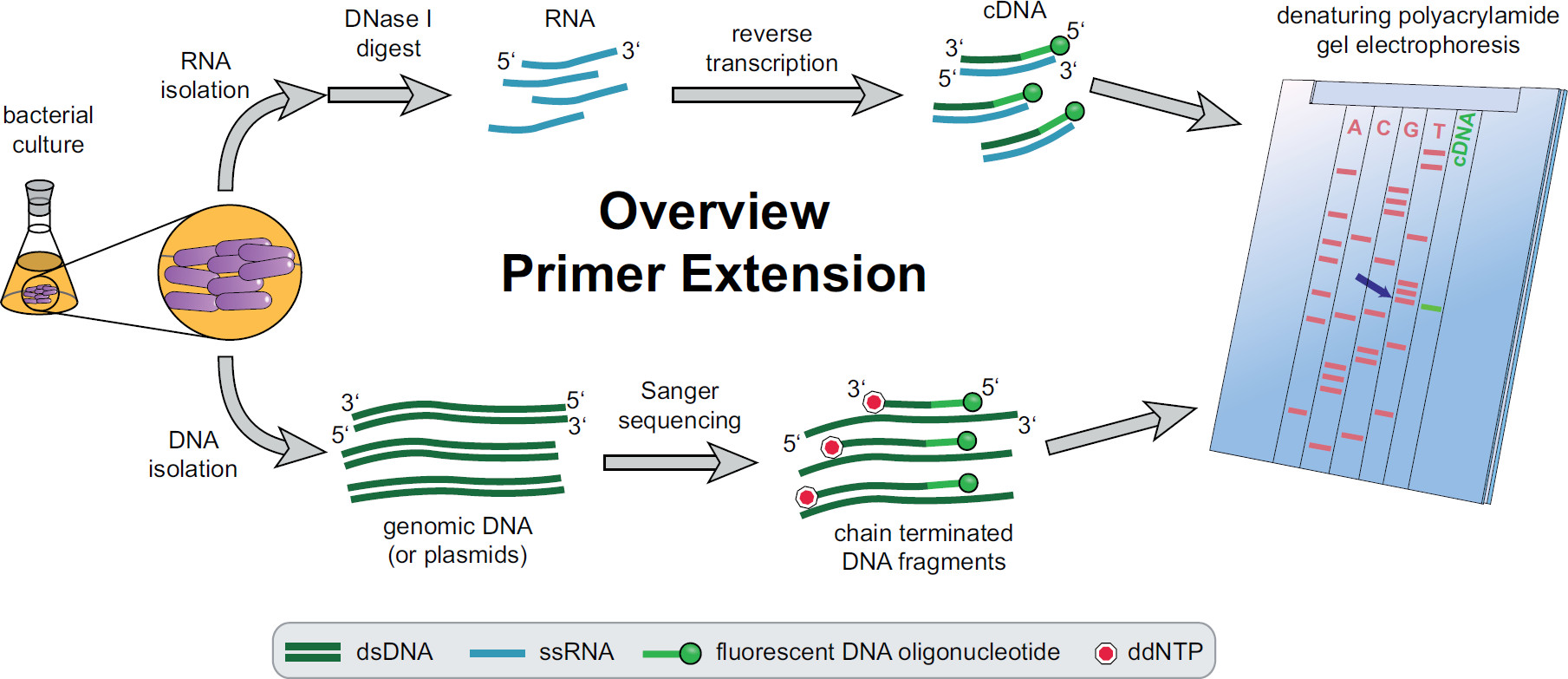
Figura 1. Descripción general del procedimiento de extensión del cebador. Los cultivos bacterianos se incubaron y tratados de acuerdo con las necesidades experimentales. El ARN total se extrajo de las células, se trató con DNasa I para eliminar las trazas de ADN y se sometieron a una reacción de transcripción inversa usando cebadores fluorescentes específicos diana de ADN rendimiento de ADNc. El ADN genómico o plásmidos se extrajeron y posteriormente utilizarse para reacciones de secuenciación Sanger fluorescentes para la comparación de tamaño con los fragmentos de ADNc. Productos de extensión del cebador se ejecutan junto a los productos de secuenciación de Sanger en un gel de poliacrilamida desnaturalizante de urea y se analizaron con un láser automatizado y el microscopio. La base de la secuencia que se alinea con la banda de ADNc es la last base del extremo 5 'de ADNc (flecha azul). Más información en Fekete, et al. 3 Haga clic aquí para ver una versión más grande de esta cifra.
Una visión general de todo el procedimiento de extensión del cebador se puede encontrar en la Figura 1. Brevemente, las células bacterianas se cultivan, cosechado, el sedimento de células se lisaron y el ARN extraído. ARN purificado se trata a continuación con DNasa I para eliminar trazas de moléculas de ADN que podrían actuar como plantillas para la transcriptasa inversa. Cebadores fluorescentes específicos se añaden a la ARN, hibridado a la región de interés y posteriormente a transcripción inversa, lo que resulta en ADN complementario monocatenario (ADNc). Una escalera de secuenciación se crea mediante secuenciación Sanger tradicional empleando cebadores fluorescentes y se separó en un gel de poliacrilamida desnaturalizante junto de los fragmentos de ADNc de extensión del cebador. La resultantegel se analizó mediante la comparación de las bandas fluorescentes, lo que permite la identificación de los extremos 5 'de interés. Puntos de partida de la transcripción y sitios de procesamiento son luego evaluados individualmente por comparación de secuencias.
Protocolo
1. Alto Rendimiento Preparación de ARN
- Aislamiento de ARN
NOTA: Las altas concentraciones de ARN total se necesitan para la reacción de extensión del cebador. Kits columna de centrifugación por lo general no producen la cantidad de ARN necesaria (~ 5-16 g en 5 l de volumen). Por lo tanto se recomienda la purificación utilizando el método de extracción de tiocianato-fenol-cloroformo de guanidinio ácido, se describe a continuación.
NOTA: El fenol es cancerígeno, tóxico y corrosivo. Por favor, lea las hojas de datos de seguridad de los materiales y el uso bajo una campana de humos con la protección adecuada!- Crecer o tratar las células bacterianas (S. aureus o E. coli en este ejemplo) como se desee y la cosecha por 10 min de centrifugación a 4600 × g y 4 ° C. Nota: Por lo general se cosecha un total de 600 OD de 20 - 70. Los sedimentos celulares se pueden almacenar durante varias semanas a -20 ° C.
- Resuspender el sedimento celular en 1 ml de solución de tiocianato-fenol-cloroformo de guanidinio ácido y transferir a una2 ml de tornillo taza contiene 0,5 ml de 0,1 mm perlas de cristal de zirconio / sílice.
- Lisar las células tres veces en un batidor de preparación rápida / perlas de 6,5 m / s durante 30 s durante tres rondas, el enfriamiento de las muestras en hielo durante 5 minutos después de cada carrera. Nota: muestra homogeneizada se puede almacenar a -80 ° C durante varias semanas.
- Incubar lisado durante 5 min a RT y luego añadir 200 l de cloroformo.
- Agite vigorosamente o vórtice de la muestra durante 30 segundos para extraer ARN.
- Incubar a temperatura ambiente durante 3 min centrifugar durante 15 min a 13.000 - 15.000 × g y 4 ° C. Nota: Los disolventes se separan en una fase orgánica inferior (rosa, contiene proteínas), una interfase (blanco, contiene ADN) y una fase acuosa superior (claro, contiene ARN).
NOTA: A partir de este paso en el uso sólo RNasa reactivos libres y artículos de plástico! - Preparar fresco 1,5 ml tubos de reacción, etiqueta RNasa libre de manera adecuada y añadir unos 500 l de isopropanol 100% de RNasa libre de cada uno (utilizar aproximadamente la misma voLume como la fase acuosa en el tubo anterior).
- Sostenga el tubo en un ángulo y transferir la fase acuosa (aproximadamente 500 l) para los tubos preparados usando RNasa consejos gratis. No moleste a la interfase.
- Precipitar el RNA invirtiendo varias veces e incubando durante 10 min a TA.
- Centrifugar las muestras durante 15 min a 13.000 - 15.000 × g y 4 ° C y retirar el sobrenadante mediante pipeteado o aspiración (bomba de chorro de agua y la botella de alimentación). No perturbar el sedimento de ARN blanco transparente en la parte inferior.
- Añadir 1 ml de etanol 70-80% de RNasa libre (no hacer vórtice) para lavar. Nota: ARN en etanol se puede almacenar durante varias semanas a -20 ° C.
- Centrifugar durante 5 min a 7500 × g y 4 ° C y desechar el sobrenadante con la pipeta o preferiblemente la aspiración.
- Aire seco sedimento de ARN por 15 - 30 minutos bajo la campana de humos. No secar en exceso, de lo contrario gránulos pueden ser difíciles de disolver.
- Resuspender el precipitado en 50 l de RNasa libre de ddH 2 O o tampón de almacenamiento de ARN.
- Medir la concentración de ARN con una microvolumen UV-Vis o una cubeta de cuarzo (y fotómetro convencional) y proceder a la digestión DNasa I.
- DNasa I digestión de ARN para eliminar las trazas de DNA
NOTA: Dado que el ADN puede actuar como una plantilla espuria en la reacción de transcripción inversa (extensión del cebador), debe ser retirado de la muestra. Varios métodos para la eliminación de ADN a partir de soluciones de ARN están disponibles, que generalmente se basan en la digestión DNasa. Un método eficiente simple pero efectivo y el costo para la extracción de ADN se describe a continuación.- Precaliente el baño de agua a 37 ° C.
- Mezclar los compuestos enumerados en la Tabla 1 en un tubo de 1,5 ml de reacción.
- Incubar la mezcla durante 1 hora a 37 ° C en un baño de agua, y luego proceder directamente a la extracción con fenol / cloroformo. Nota: No se recomienda la inactivación por calor de la DNasa I, ya que esto podría degradar el ARN.
- Extracción con fenol / cloroformo de ARN después de DNasa I digestión
NOTA: El ARN debe ser purificada para eliminar los nucleótidos libres, fragmentos de ADN y los componentes del tampón de la DNasa I digestión. Extracción con fenol / cloroformo permite una alta recuperación y concentración de la muestra de ARN y por lo tanto se describe a continuación. Otros métodos para la purificación de ARN también pueden ser utilizados, si cumplen con estos requisitos.- Dividir la DNasa I digestión 500 l mezclar en dos muestras de 250 mu l en 2 ml tubos de reacción.
- Añadir 1 volumen (250 l) de solución ácida P / C / I (en agua saturada de fenol, cloroformo y isopentanol, relación de 25: 24: 1, pH 4,5-5).
NOTA: P solución / C / I es cancerígeno, tóxico y corrosivo. Por favor, lea las hojas de datos de seguridad de los materiales y el uso bajo una campana de humos con la protección adecuada! - Vigorosamente vórtice o lugar en una plataforma de agitación en vórtex durante 1 - 3 minutos.
- Centrifugar durante 30 min a 13.000 - 15.000 × g y 4 ° C.
- Recoger la fase superior (acuosa) y transferir a un tubo nuevo (250 l).
- Añadir 1/9 de volumen (28 l) de acetato de sodio 3 M pH 5,2.
- Añadir 2,5-3 volúmenes de etanol puro (700 l).
- Mezclar mediante agitación breve y lugar a -80 ° C durante 30 min o a -20 ° C por 2 - 3 horas. Si es necesario, guarde el ARN O / N a -20 ° C.
- Centrifugar durante 30 - 60 min a 13.000 - 15.000 × g y 4 ° C.
- Aspirar el sobrenadante con la pipeta o la aspiración.
- Lavar la pella mediante la adición de 1 ml de etanol al 70% en el sedimento. No te muestra vórtice.
- Centrifugar la muestra durante 5 min a 13.000 - 15.000 × g y 4 ° C.
- Aspirar el sobrenadante con la pipeta o la aspiración.
- Aire pellet seco bajo la campana de humos. Guarde el sedimento a -20 ° CO / N si es necesario.
- Disolver el precipitado en 30 l DEPC tratada con H 2 O por agitación durante 2 min y utilizar esta solución para disolver el PEllet del segundo tubo correspondiente por muestra (30 l solución por un par de extracción).
- Medir la concentración de ARN, y asegúrese de que sea superior a 1 mg / l para el uso en la extensión del cebador de ARNm expresadas medianamente.
- Si es necesario, almacenar el ARN a -20 ° C durante varias semanas hasta algunos meses.
2. cebador de extensión Reacción
- Primer diseño
NOTA: En el diseño de cebadores para un experimento de extensión del cebador, obedecer las directrices generales de diseño de cebadores de PCR (ver el manual que acompaña al secuenciador gel automatizado para obtener más información y discusión sección de este documento).- Específicamente, asegúrese de que los cebadores (i) no contienen tramos de bases, (ii) poseer una G o C en el extremo 3 ', (iii) tener un GC equilibrada: AT ratio, (iv) tienen una temperatura de recocido de alrededor de 55 - 60 ° C y (v) se unen al menos 50 pb, 100 pb mejor aguas abajo de la región de interés para recibir imágenes claras.
- Reacción de extensión del cebador
NOTA: La reacción de extensión del cebador (síntesis de ADNc) requiere grandes cantidades de ARN de plantilla. Si se eligen las cantidades de ARN que se utilizan para bajo, la señal puede ser demasiado baja para detectar! Por lo tanto recomendamos la purificación del ARN como se describe anteriormente.
NOTA: PRECAUCIÓN: El uso de RNasa reactivos libres y utensilios de plástico !!!- Precalentar el termociclador a una temperatura de 95 ° C y llevar a cabo todos los pasos adicionales de incubación en un termociclador para la facilidad de uso y reproducibilidad.
- Mezclar los compuestos de la Tabla 2 en un tubo de PCR para cada muestra de ARN.
- Desnaturalizar las muestras durante 1 min a 95 ° C.
- Colocar los tubos en el hielo y el frío durante 5 min para hibridar ARN y cebadores.
- Ajuste la máquina de PCR a 47 ° C.
- Mientras tanto preparar la mezcla maestra de transcripción inversa como se describe en la Tabla 3.
- Añadir 4 l de mezcla maestra de transcripción inversa para cada ARN hibridadomuestra.
- Incubar los tubos durante 1 hora a 47 ° C. Nota: La temperatura óptima para AMV RT es 42 ° C, las temperaturas más altas sin embargo ayudan a superar las estructuras secundarias de las moléculas de ARN.
- Detener la reacción por calentamiento de las muestras a 95 ° C durante 2 min.
NOTA: La formamida es corrosivo, tóxico y puede ser perjudicial para el feto. Por favor, lea las hojas de datos de seguridad de los materiales, manejar con cuidado y use protección adecuada! - Añadir 6 l de colorante de carga de formamida (95% (v / v) de formamida desionizada, EDTA 10 mM, 0,05% (w / v) de azul de bromofenol) y tienda para O / N dos semanas a -20 ° C en la oscuridad.
3. Preparación de la escalera de secuenciación
NOTA: La reacción escalera secuenciación requiere ya sea una cantidad moderada de plásmidos o grandes cantidades de ADN genómico. Siempre que sea posible, se recomienda el uso de plásmidos en la reacción de secuencia debido a la facilidad de aislamiento y de alta sigintensidad nal. En otros casos, se utiliza rutinariamente un método adoptado de Marmur 5,14 para preparar ADN genómico de E. coli y S. aureus células sin la necesidad de utilizar fenol. En principio cualquier método que produce grandes cantidades y pureza del ADN genómico se puede utilizar.
- Aislamiento de ADN genómico
- Crecer 10 ml de E. coli o S. células aureus O / N en LB, BM 5 o medio TSB.
- Cosecha células por centrifugación durante 10 min a 4600 × g en un tubo Falcon de 15 ml.
- Pellet se resuspendió en 2 ml de tampón P1 como se encuentra en algunos kits de preparación de los mini (Tris-HCl 50 mM pH 8,0, EDTA 10 mM, 100 mg / ml de RNasa A).
- Lisar las células durante 45 - 60 min con 20 - 40 l lisostafina (0,5 mg / ml, el almacenamiento a -20 ° C). Nota: Para E. células de E. coli el pretratamiento enzimático puede o bien omitirse o lisozima utilizado.
- Añadir 100 l de SDS-solución saturada (en 45% de etanol) a la suspensión y incubate durante 5 min a 37 ° C.
- Añadir 650 l 5 M NaClO 4 y las células de vórtice brevemente.
NOTA: El cloroformo es un carcinógeno potencial. Por favor, lea las hojas de datos de seguridad de los materiales y el uso bajo una campana de humos con la protección adecuada !!! - Añadir 3 ml de cloroformo / isopentanol (24: 1) a la mezcla y se agita durante al menos 60 segundos. Nota: El líquido debe convertirse en una emulsión blanca homogénea.
- Muestra Centrifugar durante 10 min a 4600 × g y RT para separar las fases.
- Transferir cuidadosamente la fase superior transparente (acuosa) a un tubo nuevo. Si la solución está turbia, repetir la extracción con cloroformo / isopentanol. Medir el volumen de la solución de ADN y prepare un tubo nuevo con 2 volúmenes de etanol (100%).
- Lentamente decantar o una pipeta la solución de ADN en el etanol que contiene el tubo. Nota: ADN debería precipitar como transparentes, bobinas densas en la parte inferior o cuando está completamente deshidratado como un racimo blanco flotante.
- Recuperar la DNA usando ganchos hechos de pipetas Pasteur de vidrio (Figura 2) y se lava dos veces cada muestra mediante inmersión en un tubo individual de 1 ml de etanol de 70%.
- Coloque los ganchos en posición vertical en un bastidor y secar al aire el sedimento durante 60 min. Si es necesario, almacenar el ADN se secó durante varios días a temperatura ambiente.
- Disolver el ADN por la ruptura de los ganchos de ADN cubierta de vidrio y colocar en un tubo de reacción de 2,0 ml que contiene 100 - 500 l ddH 2 O. Ajuste el volumen a una concentración final de ADN de 1.000 - 1.500 ng / l. Para una reacción de secuenciación, utilizar 10 - 18 g de ADN genómico.

Figura 2. Instrucción sobre cómo crear una barra de pesca de ADN. Sostenga la punta de una pipeta Pasteur de vidrio en la llama de un mechero Bunsen. Esto hace que el vidrio para iniciar la fusión después de varios segundos, creando un pequeño gancho en tél termina. Quite rápidamente del fuego y dejar enfriar durante 1 min.
- Aislamiento del plásmido
- Preparar plásmidos usando kits Mini Preparación estándar y disolver en tampón de elución (10 mM Tris-Cl, pH 8,5). Dependiendo del tamaño del plásmido, utilice 100 - 500 ng de plásmido para una escalera de secuenciación.
- Reacción de secuenciación de Sanger
NOTA: Encuentra debajo de un protocolo simple que utiliza un kit de secuenciación del cebador marcado con fluorescencia con 7-deaza-dGTP que funciona bien para el propósito de las extensiones de los cebadores. Consulte el manual del kit de secuenciación para obtener información detallada. Tenga en cuenta que la reacción de secuenciación debe utilizar el mismo cebador como la reacción de extensión del cebador para crear productos de la misma longitud.- Mezclar 12 l de ADN genómico (~ 10 - 15 g) con 1 l de DMSO y 1 l de cebador marcado con fluorescencia (2 pmol / l).
- A cada 1 l de las cuatro mezclas de reacción de secuenciación (A, C, G o T), añadir 3 l de la ADN / DMSO / mezcla de cebadores.
- Coloque las muestras en una máquina de PCR, y ejecute el siguiente programa de PCR: 95 ° C durante 2 min; 35 ciclos de 95 ° C durante 20 seg, 54 ° C durante 20 s, 70 ° C durante 30 s; mantener a 4 ° C para siempre.
- Después de la carrera, retirar las muestras de la máquina, añadir 6 l de colorante de carga y almacenar en hielo (a corto plazo) oa -20 ° C durante varios días o semanas.
4. Configuración del gel y Aparato Run
NOTA: La información detallada sobre cómo se monta el aparato de gel de secuenciación, el gel se prepara el gel y cómo se ejecuta se puede encontrar en el protocolo del fabricante.
- Preparativos
- Preparar 10x TBE como se indica en la Tabla 4.
- En el día de la carrera gel preparar 1 L de tampón 1x TBE con ultrapura ddH 2 O.
- Preparar 10% (w / v) APS. Nota: Puede ser almacenado en 200 ml de alícuotas a -20 ° C durante varios meses, pero la actividad puede disminuir con el tiempo <./ Li>
- Asamblea de cámara de colada de gel
- Evite el polvo y la pelusa entre las placas de vidrio. Superficies de trabajo tanto limpiar a fondo utilizando toallitas húmedas.
- Limpiar un par de placas de vidrio de 25 cm utilizando toallas de papel desechables y agua destilada en ambos lados y luego isopropanol para el lado interior de las placas de vidrio.
- Coloque 0.25 mm espaciadores en la placa de vidrio trasero y baje la placa de vidrio con muescas en la parte superior (Figura 3).
- Coloque los rieles de gel a ambos lados de las placas de vidrio con el extremo con muescas y los pilotos de entrada ferrocarril hacia arriba y apriete las perillas a la ligera.

Figura 3. Vista de las placas de vidrio de electroforesis en gel de despiece. Las placas de vidrio se deben utilizar direccionalmente. Tenga cuidado de hacer frente a la cara interior de las placas de vidrio hacia el interior y el exteriorlado hacia el exterior.

Figura 4. Vista de un aparato de gel montado. Después de inyectar la solución de gel, el bolsillo espaciador se coloca en la solución de entre las placas de vidrio. La placa de fundición se desliza entonces entre la placa de cristal frontal y los carriles de gel y se fija apretando las perillas de ferrocarril.
- Lanzar el gel
NOTA: acrilamida no polimerizada es neurotóxico! Por favor, lea las hojas de datos de seguridad de los materiales y su uso con la protección adecuada !!!- Añadir los compuestos enumerados en la Tabla 5 en un vaso de precipitados y mezclar usando una barra de agitación y un agitador magnético.
- Inmediatamente después de la adición de APS y TEMED, tomar la solución de gel en una jeringa de 50 ml y colocar un filtro de 0,45 nm en la punta.
- Cualquiera de sujetar el borde superior de la placa de vidrio con una mano o colocar la sandwich en una colada de gel de pie para crear una pendiente en ángulo 10-20 °.
- Lentamente dispensar la solución de gel entre las placas de vidrio mientras que se mueve continuamente la punta de la jeringa de un lado al otro y detener una vez que la solución de gel se encuentra con el extremo inferior.
- Mover a un lado o eliminar por completo las burbujas de forma usando un gancho de burbujas.
- Deslice el espaciador bolsillo gel (0,25 mm) entre las placas de vidrio en el extremo con muescas, se sumergen en la solución de gel y fijar por fijación de la placa de colada.
- Tornillos de los rieles superior Sujete ligeramente (Ver Figura 4 para el aparato completamente montado).
- Deje conjunto gel para 1 - 2 horas.
- Retire la placa y el bolsillo de fundición espaciador y limpiar el bolsillo de los residuos de sal y gel.
- Enjuague con ddH2O y limpie el exceso de solución con papeles de seda.
- Correr y visualizar el gel
NOTA: Los geles de secuenciación se sometieron a electroforesis directamente en el generador de imágenes de gel, mientrasla fluorescencia se detecta simultáneamente por un microscopio láser. En contraste a electroforesis en gel convencional, en el que el gel se ejecuta primero y luego teñidas y se visualizó, la unidad de detección se fija y escanea las bandas en tiempo real a medida que pasan el láser. A continuación un procedimiento para el software de recogida de datos ImagIR en OS / 2 se esboza, que puede ser adoptada para las versiones más recientes. Para obtener más información, consulte el manual de usuario.- Deslice el soporte de depósito de inercia en los carriles de gel sobre las placas de vidrio del frente y apriete las perillas.
- Coloque gel en el depósito de gel inferior del generador de imágenes de gel automatizado contra la placa de calentamiento y fijar deslizando el piloto de entrada ferroviaria en los soportes de aparatos.
- Rellene tampón TBE 1x en las cámaras de amortiguación de gel inferior y superior, cierre la cámara intermedia inferior y conecte la cámara de amortiguación superior a la corriente mediante el cable de alimentación.
- Si está presente, limpiar la bolsa de gel de salinas residuos pipeteando repetidamente búfer en el bolsillo.
- Cierre la cámara de la parte superior del tanque buffer usando la tapa de amortiguación superior.
- Cierre la puerta de la máquina y encienda el reproductor de imágenes y la computadora e iniciar el software de colección de datos Base ImagIR.
- Crear un nuevo archivo de proyecto (Archivo-> Nuevo ...), introduzca un nombre de archivo del proyecto, seleccione los rangos de láser apropiados (700 o 800 nm) y confirme con OK.
- Seleccione Opciones-> Auto ganancia ... en el menú de la imagen en la parte superior, haga clic en Auto para iniciar la medición de ganancia automática y aceptar la configuración haciendo clic en Aceptar.
- Enfoque el láser seleccionando Opciones-> Enfoque ... en el menú de control del escáner, haga clic en el botón Auto y aceptar la configuración haciendo clic en Aceptar.
- Repita el procedimiento automático de ganancia para ajustar a la región recién centrado.
- Configuración del control del escáner de acuerdo con estos valores: 2000 V, 35 mA, 45 W, 45 ° C, el filtro de exploración: 3, velocidad de escaneo: 3.
- Ejecución previa de la gel de vacío durante 20 minutos (SELECT en tensión y pulse ).
- Mientras tanto, calentar la escalera de secuenciación y los productos de extensión de cebadores en una máquina de PCR durante 2 min a 90 ° C, a continuación, enfriar en hielo.
- Detener la electroforesis, abrir el secuenciador automático de gel y retirar la tapa del tanque de amortiguación superior.
- Insertar el tiburón de dientes de peine entre las placas de vidrio y Pierce ligeramente el gel con los dientes de tiburón (véase la Figura 5).
Figura 5. Vista de primer plano de gel con peine de dientes de tiburón. Muestra (púrpura) se aplica entre los dientes de tiburón.
- Pipetear ya sea 1 - 2 l de los productos de extensión de escalera o reacciones de secuenciación del cebador en cada bolsillo de gel (formado por los dientes de tiburón).
- Si se necesitan no todos los bolsillos, llenar los bolsillos vacíos con colorante de carga para evitar que encomportamiento de marcha constante.
- Cerrar el depósito y la puerta del secuenciador gel buffer.
- Iniciar electroforesis y encienda láser (Seleccione Voltaje ON y Laser ON y pulse ).
- Deje de electroforesis vez región de interés ha pasado el láser.
Resultados
Como se representa en la figura 6, una reacción de extensión del cebador se puede utilizar para determinar los puntos de partida de la transcripción de las transcripciones de interés y puede ayudar a deducir regiones promotoras (normalmente identificado por -10 y -35 elementos). El fragmento de ADNc más alta (más larga) representa el extremo 5 'del ARNm y por lo tanto se puede asignar fácilmente cuando se compara con la escalera de secuenciación.

Figura 6. resultado representativo de una reacción de extensión del cebador. En el lado izquierdo de una imprimación vivo lleno de gel en la extensión de E. coli con diversos plásmidos se muestra. Áreas individuales de interés están agrandados. En la parte superior (A), la determinación del punto de partida transcripcional ompA se representa. La transcripción inversa se detiene en el extremo 5 'del ARNm y por lo tanto crea una banda del ARN de longitud completa (indicated por una flecha). Mediante la alineación de la banda de ADNc con la escalera de secuenciación, el extremo 5 'del ARNm se puede determinar como se muestra en la secuencia adjunta. En la parte inferior (B), se muestra la escisión de la transcripción ompA por la RNasa YoeB-seq2. Carriles 1 - 3 representan muestras en las que la toxina YoeB-seq2 falte o inactivos, mientras que los carriles 4 - 5 representan muestras con una RNasa activa, coincidiendo con la ausencia y presencia de productos de ADNc. Dos productos principales de escisión se crean como se indica por las flechas. El ddNTP utilizado en cada carril y la base RNA correspondiente de la escalera de secuenciación está marcado. Transcripción partes se indican con fuente azul, elementos promotores y comienzo codón AUG por fuente magenta. Para obtener más información; ver el artículo de investigación original a partir de Nolle et al., Microbiología 159, 1575-1585 (2013). Por favor, haga clic aquí paraver una versión más grande de esta cifra.
En el ejemplo mostrado en la Figura 6, el TSP de la ompA mRNA se determinó que era una base de G (marcada con una flecha en la secuencia por debajo de la gel). Esto es consistente con el ompA TSP publicada antes de 15. Los elementos -35 y -10 del promotor se pueden deducir a ser TTGTAA y TAGACT 16 como los motivos sólo se diferencian dos bases de cada una de sus respectivas secuencias de consenso (TTGACA y TATAAT respectivamente).
En contraste con otros métodos tales como 5 'RACE, reacciones de extensión de cebadores se pueden usar para determinar y cuantificar la escisión de moléculas de ARN con precisión. La escisión de moléculas de ARN libres crea extremos 5 ', que pueden ser detectados simultáneamente como bandas de ADNc en el gel. La identificación de varios productos de escisión de un mRNA es más difícil en 5 'RACE experimentos, ya que varios productos de PCR deben ser secuenciados para obtener productos de escisiónque sólo representan una pequeña fracción del total (sin procesar) RNA a granel.
En la parte inferior de la Figura 6, la asignación de ARN de la escisión por una RNasa se representa. Como se describió anteriormente 5, la RNasa YoeB-seq2, parte de un sistema de TA de S. equorum 17, mRNAs escinde cerca del codón de inicio. Esta escisión puede ser inhibida por el anticuerpo anti-toxina YefM-seq2 afín. Cuando la RNasa YoeB no está presente o inactivo (carriles 1 - 3), no hay productos de extensión del cebador se forman cerca del codón de inicio. Siempre que la actividad de RNasa está presente (carriles 4 y 5), dos bandas fuertes poco corriente abajo del codón de inicio aparecen. Usando este enfoque, los patrones de escisión pueden ser fácilmente identificados.
La Figura 7 muestra un experimento de extensión del cebador fallado. Debido al exceso de ARN en la reacción de transcripción inversa, la cantidad generada de cDNA crea una señal tan fuerte, que la banda de ADNc individuos no se pueden distinguir. Esto hace que la determinación final 5 'del ARN imposible.
Cuando se utiliza ARN altamente expresado en la reacción de extensión del cebador, tal como un ARN ribosomal (como se representa en la figura 7), el riesgo de aumentos de áreas sobre-expuestas. Por lo tanto, la cantidad de RNA total plantilla debe ser ajustado a la abundancia del ARN de interés. En contraste, cuando las transcripciones de interés sólo constituyen una pequeña cantidad de RNA total, la cantidad en la reacción de transcripción inversa se debe aumentar, de lo contrario las señales serán demasiado débil (no mostrado). Esto también se aplica a la reacción de secuenciación de Sanger, como plantillas de múltiples copias por célula (tales como genes de ARNr o genes codificados en plásmidos) resultar en señales mucho más fuertes.
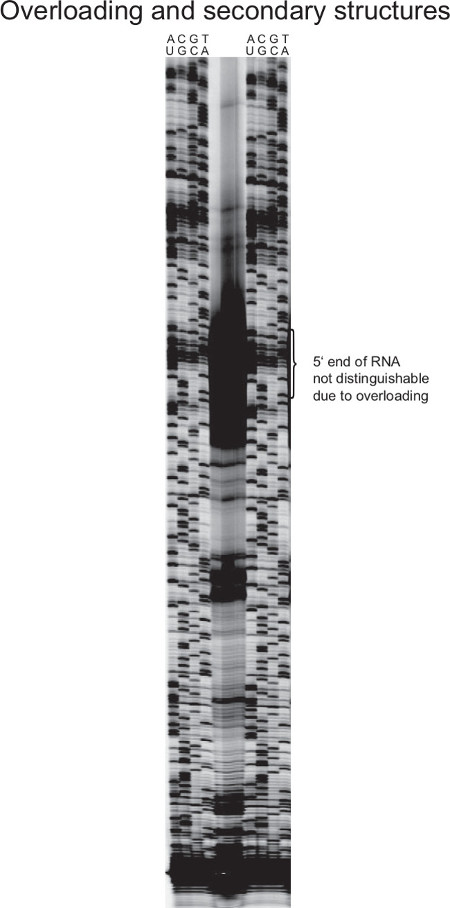
Figura 7. Representante resultado de una extensión del cebador fracasado desde ng> S. aureus. 16S RNA es muy abundante en las células bacterianas. Esto lleva a fuertes señales de transcripción inversa cuando se utilizan cantidades moderadas de ARN total. En el caso representado aquí, las fuertes bandas de ADNc enmascaran el 'final y por lo tanto evitan 5' exacto 5 cartografía. Además, ARN ribosomal son altamente estructurado y, por tanto, pueden terminar la transcripción inversa antes de tiempo, la producción de productos de ADNc cortos que no representan fragmentos de longitud completa. En este caso el aumento de temperatura de la transcripción inversa, junto con una transcriptasa inversa resistente al calor puede hacer que sea más fácil de sintetizar estructuras secundarias últimos. Debido a las múltiples copias de la secuencia en el ADN genómico, la escalera de secuenciación de Sanger es más fuerte que para los genes de copia única. Para mejorar la imagen de gel, ARN y ADN asciende para la transcripción inversa y reacción en Sanger deben reducirse y / o menos producto aplicado al gel._upload / 52134 / 52134fig7large.jpg "target =" _ blank "> Haga clic aquí para ver una versión más grande de esta cifra.
Tabla 1: DNasa I digestión de ARN.
| Sustancia | Cantidad |
| ARN de paso por encima | 70 a 100 g |
| 10x tampón de ADNasa I (100 mM Tris, pH 7,5, 25 mM de MgCl 2, 5 mM CaCl 2) | 50 l |
| DNasa I, RNasa libre (2 U / l) | 10,0 l (hasta 10 g de ARN por 1 l de DNasa I) |
| Traiga volumen de hasta 500 l con ddH2O (DEPC tratada) | |
Volumen de reacción y la cantidad de DNasa I se pueden escalar hacia arriba o hacia abajo dependiendo de la cantidad of RNA utilizado.
Tabla 2: ARN-Primer hibridación.
| Compuesto | Importe para una reacción |
| ARN | 5-15 g |
| Cebador marcado con fluorescencia | 2 pmol |
| Traiga volumen de 6 l con ddH2O (DEPC tratada) | |
Configurar una reacción por cada muestra de ARN y de imprimación.
Tabla 3: mezcla maestra La extensión del cebador.
| Compuesto | Importe para una reacción |
| ddH2O (DEPC tratada) | 1.3 l |
| AMV RT Buffer (10x) | 1.0l |
| dNTPs (10 mM, RNasa libre) | 1.0 l |
| Inhibidor de RNasa | 0.2 l |
| AMV transcriptasa inversa (RT) (20 - 25 U / l) | 0.5 l |
Escalar hasta el número de reacciones necesarias.
Tabla 4: 10x TBE receta.
| Sustancia | Cantidad |
| Tris Base | 107,8 g |
| Ácido bórico | 55,0 g |
| EDTA | 7,4 g |
| Traiga volumen hasta 1000 ml con ddH 2 O ultrapura y se filtra para eliminar el polvo y la pelusa | |
Se filtra para eliminar el polvo y la pelusa y se almacena a 4 °C.
Tabla 5: Receta gel de secuenciación.
| Compuesto | Cantidad |
| Urea | 10,5 g |
| ddH2O (MilliQ) | 13,0 ml |
| 10x TBE | 2,5 ml |
| Solución de gel XL Rápida | 5,0 ml |
| TEMED (N, N, N ', N'-Tetramethylethane-1,2-diamina) | 25 l |
| APS (persulfato de amonio, 10%) | 175 l |
Proceso de solución de gel rápidamente después de la adición de TEMED y APS.
Discusión
Extensión del cebador fluorescente es un método sencillo y rápido para determinar los extremos 5 'de los ARN, ya sea para TSP- o identificación procesamiento del ARN secundaria. Debido a la utilización de cebadores fluorescentes, las reacciones se pueden configurar y ejecutar sin las precauciones de seguridad adicionales (a diferencia de en el caso de cebadores marcados radiactivamente). Como las muestras son detectados por fluorescencia, que pueden obtenerse imágenes, mientras que la electroforesis está en progreso que permite un análisis rápido en comparación con los métodos radiactivos que se utilizan comúnmente películas de rayos X.
En general, la calidad de la reacción de extensión del cebador depende en gran medida del grado y capacidades de unión del cebador. Si el sitio de unión se elige demasiado cerca de la zona de interés, los frotis de cebadores pueden enmascarar la señal, mientras que un sitio de unión demasiado lejos (> 300 pb) desde el extremo 5 'puede resultar en una señal pobre.
El colorante fluorescente debe ser unido covalentemente a laExtremo 5 'del oligonucleótido de ADN durante la síntesis de encargo y el oligonucleótido debe ser purificado por HPLC para evitar la interferencia con la reacción de transcripción inversa por las sales residuales. Los oligonucleótidos con modificaciones tinte adecuado (vea la tabla de lista de reactivos para obtener más información sobre los colorantes compatibles) pueden solicitarse a la mayoría de las compañías de síntesis de oligonucleótidos y se deben almacenar en la oscuridad. Por desgracia, no nos damos cuenta de las técnicas enzimáticas para fijar el tinte de oligonucleótidos previamente existentes, como es posible con nucleótidos radiomarcados.
En el sistema de secuenciación se describe aquí, dos colorantes fluorescentes diferentes pueden ser usados para detectar simultáneamente dos muestras, ya que su excitación (alrededor de 700 y 800 nm) y los espectros de emisión son distintos. Aparte de los tintes originales del fabricante, otros colorantes, como se indica en la lista de los reactivos se pueden utilizar para producir excelentes resultados.
Otro factor importante para la prreacciones de extensión Imer es la calidad y la cantidad de ARN. Se debe tener cuidado para eliminar los contaminantes de ADN, como transcriptasas inversas como la AMV RT pueden usar el ADN como una plantilla 18.
Como se muestra en la Figura 7, la intensidad de la señal detectada de bandas en el gel de ejecución es dependiente de la cantidad de ARN utilizado en la reacción de transcripción inversa. Por tanto, es esencial para ajustar la cantidad de RNA total, dependiendo de la cantidad proporcional de la RNA de interés presente en la muestra. La baja sensibilidad de este método es también una de sus desventajas, como los ARN expresados bajas pueden ser difíciles de detectar. Si no hay señales pueden ser detectados en absoluto, la cantidad de RNA total se puede aumentar o el ARN de interés puede ser sobreexpresado artificialmente a partir de un plásmido.
Gel sensibilidad de detección de fluorescencia basada en la extensión del cebador es de aproximadamente factor de diez menor que 32 o 33 P P radioisótopo basado extensión del cebador 19. Sin embargo, esta desventaja se puede compensar mediante el ajuste de la cantidad de cDNA cargada en el gel o mediante el aumento de la cantidad de molde de ARN utilizado en la reacción de transcripción inversa. En la mayoría de los casos, la sensibilidad es lo suficientemente alta como para obtener resultados satisfactorios 4,5. Sensibilidades similares a las extensiones de cebadores radioactivos han sido reportados cuando utilizando cebadores fluorescentes en combinación con un secuenciador capilar 3, con la ventaja de tiempos de exposición cortos.
Costos de las comparaciones entre la fluorescencia y la radioactividad basados extensiones de cebadores son difíciles, ya que dependen de varios factores, tales como las máquinas disponibles, los cebos, las reacciones de la escalera, disponibilidad y mantenimiento de un laboratorio para el trabajo con sustancias radiactivas, la eliminación de desechos radiactivos, la capacitación y los riesgos de salud individuales . Cebadores fluorescentes son alrededor de cinco a diez veces más caros que los cebadores estándar no etiquetados (20 pb). Sin embargo, estos cebadores pueden usarse para, al menos,un año (más probablemente varios años) en comparación con cebadores marcados 32 P, que tienen una vida media mucho más corta. Re-etiquetado de los cebadores es lento y costoso, debido a la necesidad de que el nuevo material radiactivo en intervalos frecuentes. Si se utiliza el mismo conjunto de cebadores a lo largo de un período de tiempo largo, cebadores fluorescentes son más baratos que los cebadores regulares, marcados radiactivamente. Sin embargo, el punto principal costo será el secuenciador fluorescente o de imágenes y la adquisición de este aparato únicamente con el propósito de extensiones de primers podría no ser rentable. El método descrito aquí es bastante interesante para los grupos que están en posesión de, o tener acceso a una máquina de este tipo.
Si un secuenciador automatizado de gel no está disponible, otros métodos para la detección de la fluorescencia se pueden utilizar también. En este caso, el gel se puede ejecutar en un aparato de electroforesis estándar, se seca si es necesario y después se transfirió a un generador de imágenes de fluorescencia (modelos están disponibles, que son similares a un flescáneres atbed). Aunque se pierde la ventaja de visualizar el gel durante la carrera, el uso de isótopos radiactivos se puede evitar de esta manera, una ventaja importante para el experimentador. Además, si está disponible, un secuenciador capilar puede ser utilizado para la separación y detección en tiempo real, que puede ayudar a aumentar la sensibilidad.
Varios métodos han sido publicados sobre el uso de la extensión del cebador fluorescente con máquinas de secuenciación automatizada de gel o secuenciadores capilares, sin embargo, estos métodos a menudo requieren una etapa de precipitación de cDNA para concentrar la muestra (y eliminar las impurezas) 19. En el método presentado aquí, la precipitación de ADN no es necesario reduciendo así el tiempo de preparación. Más importante, sin embargo, la omisión de este paso hace que sea posible determinar semi-cuantitativamente la cantidad de moléculas de ARN en la muestra como las inconsistencias del procedimiento de precipitación se pueden eliminar.
Cabe señalar, que aunquelos extremos 5 'de las moléculas de ARN se pueden asignar en las reacciones de extensión de cebador, procesados y extremos primarios (puntos de partida de la transcripción) no pueden distinguirse fácilmente. Para sortear estas restricciones, la planificación minuciosa de los experimentos con los controles adecuados es esencial. Obvias secuencias promotoras pueden indicar la presencia de un punto de partida transcripcional y mutando o eliminación de elementos promotores entonces deben abolir bandas de ADNc. Por otro lado, si tales secuencias no están presentes o secuencias de consenso específicas están presentes, las bandas pueden ser causados por el procesamiento del ARN. Si la enzima de procesamiento es conocido y puede ser purificado, en vitro extensiones de cebadores pueden aportar aclaraciones, como la transcripción y procesamiento pueden ser separados. Además, otros métodos tales como 5 'RACE (incluyendo el enriquecimiento enzimática de moléculas de ARN no procesados) puede complementar extensiones de cebadores para distinguir los sitios de inicio transcripcional de procesamiento del ARN.
The método de extensión de cebador es a menudo comparado con otros métodos, tales como 5 'RACE y ensayos de protección de nucleasa S1 y por lo tanto su utilidad veces se cuestiona. Técnicas RNAseq en combinación con la secuenciación de próxima generación, por ejemplo, pueden ayudar a determinar el PAT y sitios de procesamiento de muchos de los ARN en paralelo, sin embargo, la carga financiera y el trabajo requerido bioinformatical hacen bastante poco rentable para los ARNs individuales de interés. 5'-RACE, por otro lado es más barato y los resultados son más fáciles de analizar, pero si RNAs se procesan en múltiples formas o varios puntos de partida de la transcripción están presentes, el producto debe ser clonado y una gran cantidad de candidatos deben ser secuenciadas a obtener una visión representativa de los ARN de interés.
Por lo tanto, a pesar de los nuevos métodos de haber surgido en los últimos años, incluso hoy en día imprimación extensiones tienen su razón de ser debido a la facilidad de uso, bajo costo y el tiempo de respuesta corto, lo cual es especialmente ciertopara el método de fluorescencia basado presentado aquí.
Divulgaciones
The authors have nothing to disclose that would present a conflict of interest.
Agradecimientos
We thank Anne Wochele for her assistance in the laboratory and Vera Augsburger for help with the automated gel sequencer. We thank the Deutsche Forschungsgemeinschaft for funding by grants BE4038/2 and BE4038/5 within the “priority programmes” SPP1316 and SPP1617.
Materiales
| Name | Company | Catalog Number | Comments |
| AMV Reverse Transcriptase (20-25 U/µl) | NEB / Roche | NEB: M0277-T / Roche: 10109118001 | |
| DNase I (RNase free) | Ambion (life technologies) | AM2222 | |
| FastPrep-24 Instrument | MPBio | 116004500 | |
| Fluorescently labeled primers | Biomers | n/a | 5’ DY-681 modification of “ordinary” DNA oligonucleotides. Compatible dyes such as the LICOR IRDye 700/800 are also available from other suppliers such as IDTdna. |
| Li-Cor 4200 Sequencer incl. ImagIR Data collection software | Li-Cor | Product discontinued | |
| NanoDrop 2000 | Thermo Scientific | ||
| Nuclease free water | Ambion (life technologies) | AM9915G | |
| Plasmid mini preparation kit | QIAGEN | 12125 | |
| RapidGel-XL-40% Concentrate | USB | US75863 | |
| RNA STORAGE BUFFER | Ambion (life technologies) | AM7000 | |
| Roti-Aqua-P/C/I | Carl Roth | X985.3 | Alternative: “Acid-Phenol:Chloroform, pH 4.5 (with IAA, 125:24:1)” from Ambion (AM9720) |
| SUPERase•In RNase Inhibitor | Ambion (life technologies) | AM2696 | |
| Thermo Sequenase fluorescently labelled primer cycle sequencing kit with 7-deaza-dGTP | GE Healthcare | RPN2538 | |
| TRIzol reagent | life technologies | 15596-026 | |
| Zirconia/Silica Beads 0.1 mm | BioSpec | 11079101z |
Referencias
- Simpson, C. G., Brown, J. W. Primer extension assay. Methods Mol. Biol. 49, 249-256 (1995).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proc. Natl. Acad. Sci. U. S. A. 74, 5463-5467 (1977).
- Fekete, R. A., Miller, M. J., Chattoraj, D. K. Fluorescently labeled oligonucleotide extension: a rapid and quantitative protocol for primer extension. Biotechniques. 35, 90-94 (2003).
- Schuster, C. F., et al. Characterization of a mazEF Toxin-Antitoxin Homologue from Staphylococcus equorum. J. Bacteriol. 195, 115-125 (2013).
- Nolle, N., Schuster, C. F., Bertram, R. Two paralogous yefM-yoeB loci from Staphylococcus equorum encode functional toxin-antitoxin systems. Microbiology. 159, 1575-1585 (2013).
- Yamaguchi, Y., Park, J. H., Inouye, M. Toxin-antitoxin systems in bacteria and archaea. Annu. Rev. Genet. 45, 61-79 (2011).
- Schuster, C. F., Bertram, R. Toxin-antitoxin systems are ubiquitous and versatile modulators of prokaryotic cell fate. FEMS Microbiol. Lett. 340, 73-85 (2013).
- Goeders, N., Van Melderen, L. Toxin-antitoxin systems as multilevel interaction systems. Toxins. 6, 304-324 (2014).
- Yamaguchi, Y., Inouye, M. mRNA interferases, sequence-specific endoribonucleases from the toxin-antitoxin systems. Progress in molecular biology and translational science. 85, 467-500 (2009).
- Park, J. H., Yamaguchi, Y., Inouye, M. Bacillus subtilis MazF-bs (EndoA) is a UACAU-specific mRNA interferase. FEBS Lett. 585, 2526-2532 (2011).
- Zhu, L., et al. et al.Staphylococcus aureus MazF specifically cleaves a pentad sequence, UACAU, which is unusually abundant in the mRNA for pathogenic adhesive factor SraP. J. Bacteriol. 191, 3248-3255 (2009).
- Zhu, L., et al. The mRNA interferases, MazF-mt3 and MazF-mt7 from Mycobacterium tuberculosis target unique pentad sequences in single-stranded RNA. Mol. Microbiol. 69, 559-569 (2008).
- Fu, Z., Donegan, N. P., Memmi, G., Cheung, A. L. Characterization of MazFSa, an endoribonuclease from Staphylococcus aureus. J. Bacteriol. 189, 8871-8879 (2007).
- Marmur, J. A procedure for the isolation of deoxyribonucleic acid from micro-organisms. J. Mol. Biol. 3, 208-218 (1961).
- Emory, S. A., Belasco, J. G. The ompA 5' untranslated RNA segment functions in Escherichia coli as a growth-rate-regulated mRNA stabilizer whose activity is unrelated to translational efficiency. J. Bacteriol. 172, 4472-4481 (1990).
- Cole, S. T., Bremer, E., Hindennach, I., Henning, U. Characterisation of the promoters for the ompA gene which encodes a major outer membrane protein of Escherichia coli. Mol. Gen. Genet. 188, 472-479 (1982).
- Schleifer, K. H., Kilpper-Bälz, R., Devriese, L. Staphylococcus arlettae sp. nov., S. equorum sp. nov. and S. kloosii sp. nov.: Three New Coagulase-Negative, Novobiocin-Resistant Species from Animals. Syst. Appl. Microbiol. 5, 501-509 Forthcoming.
- Yu, H., Goodman, M. F. Comparison of HIV-1 and avian myeloblastosis virus reverse transcriptase fidelity on RNA and DNA templates. J. Biol. Chem. 267, 10888-10896 (1992).
- Ying, B. W., Fourmy, D., Yoshizawa, S. Substitution of the use of radioactivity by fluorescence for biochemical studies of RNA. RNA. 13, 2042-2050 (2007).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados