Method Article
基于荧光引物延伸法确定转录起点和核糖核酸酶的切割位点
摘要
We here describe a fluorescence based primer extension method to determine transcriptional starting points from bacterial transcripts and RNA processing in vivo using an automated gel sequencer.
摘要
基于荧光的引物延伸(FPE)是一个分子的方法来确定转录起始点或RNA分子的加工位点。这是通过使用特定的荧光标记的引物,将所得cDNA片段通过变性聚丙烯酰胺凝胶电泳随后分析感兴趣的RNA的反转录来实现的。同时,传统的Sanger测序反应是在凝胶上,以该cDNA片段的末端映射到它们的确切相应的碱。而相比之下,5'-RACE(cDNA末端的快速扩增),其中,所述产品必须被克隆和测序的多个候选,堆积由引物延伸所产生的cDNA片段可以同时在一个凝胶运行检测。此外,整个过程(从反转录到的结果的最终分析)可以在一个工作日内完成。通过使用荧光标记的引物,使用危险的放射性同位素标记的试剂可避免与处理时间缩短为可以在电泳过程中,被检测的产品。
在下面的协议中,我们描述了一种在体内的荧光引物延伸的方法来可靠地和快速地检测RNA的5'末端推断的转录起始点和RNA加工位点( 例如,通过毒素-抗毒素系统组件)的S.金黄色葡萄球菌,大肠杆菌大肠杆菌和其它细菌。
引言
引物延伸1是一个分子的方法来确定特定RNA分子的5'末端到一基座分辨率。其它方法,如5'-RACE(cDNA的末端快速扩增)的优点是快速的周转时间,并很容易地分析的RNA分子的长度不同的混合物的能力。
这种方法的工作原理是使RNA分子利用特异性荧光引物反转录反应,生成一定长度的cDNA片段。这些cDNA分子是在变性聚丙烯酰胺凝胶上运行除了传统Sanger测序反应2,并且可以通过它们的荧光由于使用了荧光标记的引物进行检测。的cDNA片段的长度,然后通过比较来测序梯评估,允许5'核糖核酸端的映射。
传统上,引物延伸反应中结合使用用放射性同位素检测到对X射线胶片的cDNA分子。由于对健康的危害,废物处理问题和易于处理,新的协议利用荧光进行引物延伸用自动测序仪的检测中,尽管其灵敏度稍低。使用荧光标记的引物,由重复的无线标签的过程可以省略,如荧光引物是稳定很长一段时间(超过一年中的手)。
我们在这里描述的方法利用了一种自动凝胶序列,但有轻微的修改,毛细管测序仪还可以用于cDNA的分离和检测3。凝胶分析的并行特性使得它能够检测到少量的RNA切割或加工。另一个优点是该高分辨率这种方法,当终端裂解或者甚至一种碱处理可被检测到。
对于RNA裂解或加工,吨的检测ypically两种不同类型的引物延伸的是有区别的。在一种情况下,酶处理完成后在体外用纯化的RNA和纯化的酶,而在其他情况下,该处理完成的体内 ,将所得的RNA进行纯化。在这两种情况下的RNA,进行引物延伸在体外进行,但视RNA的源上,该方法无论是所谓的体外或体内引物延伸。在我们这里介绍的协议中,我们侧重因为易用性(无纯化的蛋白质需要的话),并有可能在同一时间,以确定转录起始点和处理仅仅对体内引物延伸。然而, 体外引物延伸在原理建立相同的方式,该协议可以作为一个起点。
这里说明的方法可以,只要它们适于高被应用于许多种细菌纯度和高收益的准备核酸。
该研究在我们的实验室着重于毒素抗毒素(TA-)系统4,5,其中,所述引物延伸的方法被广泛使用的字段的调节范围。 TA-系统都存在于原核细胞的基因组,它由一个稳定的和内源性活性的毒性蛋白和大多不稳定的蛋白质或RNA的抗毒素是抵消毒性6,7的小遗传元件。毒素活性有时被抑制复制,细胞壁合成或其他机制通过RNase活性8,9-作用,但是最常见的。典型地,RNA酶的特异性是通过进行不同的测试,其中之一是在引物延伸方法测定的。引物延伸反应是非常适合于该应用程序,如裂解和全长片段的混合物可以同时分析,以确定它们的5'末端。使用体外和体内引物延伸的混合,该具体的毒素RNA酶裂解, 例如,序列特异性,可确定10-13。
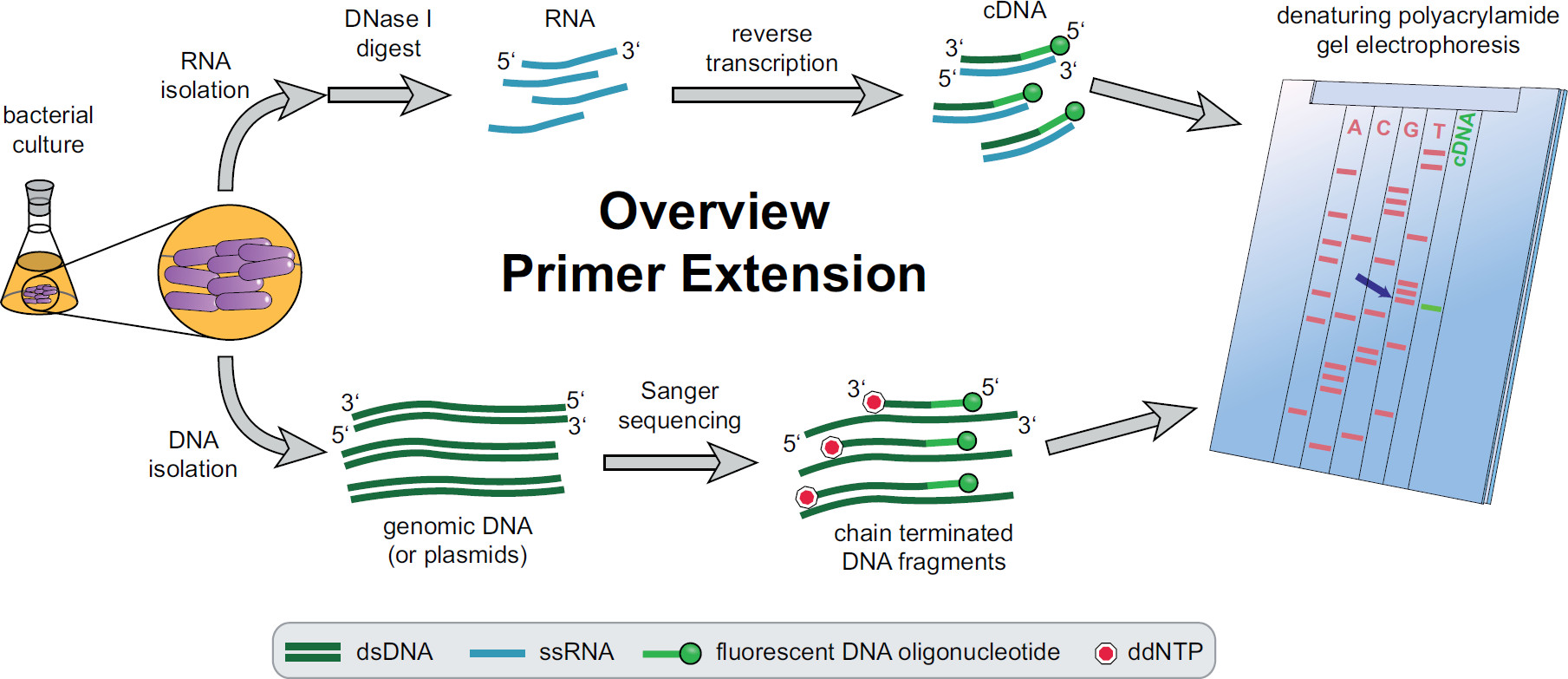
图的引物延伸步骤(1)概述。细菌培养物,根据实验需要孵育和治疗。总RNA从细胞中,用DNA酶I处理以除去痕量的DNA,并进行使用靶特异性荧光的DNA引物产生的cDNA逆转录反应萃取。基因组DNA或质粒提取并随后用于荧光桑格测序反应中与cDNA片段的大小进行比较。引物延伸产物在变性尿素聚丙烯酰胺凝胶上同时运行桑格测序产物和用自动激光和显微镜分析。测序基础,线与基因带是洛杉矶ST 5'cDNA末端(蓝色箭头)的基地。在菲克特更多信息, 等 。3 ,请点击这里查看该图的放大版本。
整个引物延伸过程的概述可以在图1中找到。简言之,将细菌细胞进行培养,收获,将细胞沉淀溶解和RNA的提取。纯化的RNA,然后用DNA酶I处理以除去的DNA分子,其可作为用于反转录酶的模板的痕迹。特定的荧光引物被加入到RNA的杂交于感兴趣的区域,并随后反向转录,从而产生单链互补DNA(cDNA)。测序梯子是由传统的Sanger测序创建使用荧光引物和分离,旁边的引物延伸cDNA片段的变性聚丙烯酰胺凝胶。由此产生的凝胶是通过比较荧光条带,使所关注的5'端的识别分析。转录的出发点和加工点,然后通过序列比单独评估。
研究方案
1.高产RNA制备
- RNA分离
注意:高浓度的总RNA,都需要引物延伸反应。离心柱试剂盒通常不产生所需要的RNA的量(〜5 - 16微克在5微升体积)。因此,建议使用酸性硫氰酸胍 - 苯酚 - 氯仿提取法纯化,概述如下。
注:苯酚是致癌物质,有毒和腐蚀性。请阅读材料安全数据表,并在通风橱中使用适当的保护!- 生长或治疗细菌细胞(S。在本实施例金黄色葡萄球菌或大肠杆菌 ),为期望的和收获的10分钟离心分离,在4600×g下4℃。注意:通常我们收获总的OD 600为20 - 70.细胞沉淀可以在-20℃保存数周。
- 重悬细胞沉淀在1ml的酸性硫氰酸胍 - 苯酚 - 氯仿溶液并转移至2毫升螺丝含0.5毫升0.1毫米的玻璃锆/硅珠杯。
- 裂解的细胞在快速准备/珠打浆机三次在6.5米/秒,30秒为三轮,在冰上冷却样品每次运行后5分钟。注意:匀浆样品可以储存在-80℃下为几个星期。
- 孵育裂解物5分钟,在室温,然后加入200μl的氯仿中。
- 用力摇动或旋涡振荡样品30秒,以提取的RNA。
- 在室温下孵育3分钟,然后离心15分钟,在13000 - 15000×g下4℃。注:溶剂被分离为下层有机相(粉红色,含有蛋白质),其相间(白色,含有DNA)和上部的水相(清晰,包含RNA)。
注:从这一步骤上只使用无RNA酶的试剂和塑料制品! - 适当地准备新鲜的无RNA酶的1.5毫升反应管,标签,加入约500微升的100%无RNA酶的异丙醇(每使用大致相同的VOlume明如前面的管内的水相)。
- 持所述管以一定的角度,并使用无RNA酶的尖端的水相(约500微升)转移到制备管中。请勿打扰相间。
- 沉淀出的RNA通过反转几次并孵育在室温10分钟。
- 离心样品15分钟,在13000 - 15000×g下4℃,并通过移液,或抽吸(水喷射泵和奶瓶)除去上清液。请勿打扰的白色透明RNA沉淀在底部。
- 加入1毫升70%-80%无RNA酶的乙醇(不涡)洗。注意:在乙醇中的RNA可在-20℃保存数周。
- 离心5分钟,在7500×g下4℃,并通过移液,或优选吸液弃去上清液。
- 空气干燥RNA沉淀为15 - 通风柜下30分钟。不干燥过度,否则粒料可能难以再溶解。
- 悬浮在5颗粒0微升无RNA酶的DDH 2 O的或RNA的存储缓冲区。
- 衡量一个微量紫外可见分光光度计或石英比色皿(和常规光度计)RNA的浓度,并继续抗酶消化。
- RNA的DNA酶I消化去除DNA的痕迹
注意:由于DNA可作为在逆转录(引物延伸)反应的乱真模板,应该从样品中除去。可用通常依赖于DNA酶消化的各种方法用于从RNA的溶液中除去DNA。用于DNA去除一个简单但有效的和成本有效的方法概述如下。- 预热水浴至37℃。
- 混合表1中列出的化合物在1.5ml反应管中。
- 为1小时,在37℃的水浴中孵育该混合物中,然后直接进行苯酚/氯仿萃取。注:抗酶热失活,不推荐,因为这可能会降低RNA。
- 酚/氯仿抽提RNA的DNA酶I消化后,
注:将RNA必须进行纯化,从DNA酶I消化除去游离核苷酸,DNA片段和缓冲液组分。苯酚/氯仿萃取允许高回收率的RNA样品的浓度,并与下面因此概括。其它方法用于RNA纯化也可使用,如果它们满足这些要求。- 拆分500微升的DNA酶I消化混合成两个250微升的样品在2毫升反应管。
- 加入1倍体积(250微升)的酸性的P / C / I的溶液(在水饱和的苯酚,氯仿和异戊醇,在25比24:1,pH为4.5 - 5)。
注:P / C / I的解决方案是致癌物质,有毒和腐蚀性。请阅读材料安全数据表,并在通风橱中使用适当的保护! - 大力旋涡,或发生在涡旋平台,为1 - 3分钟。
- 离心30分钟,在13000 - 15000×g下4℃。
- 收集上层(水相),并转移到新管(250微升)。
- 加的3M醋酸钠pH值为5.2 1/9体积(28微升)。
- 加入2.5-3倍体积的纯乙醇(700微升)。
- 3小时 - 由不久和地方涡旋在-80℃下30分钟或在-20℃下搅拌2混合。如果需要的话,存储的RNA O / N在-20℃。
- 离心机30 - 15000×g下4℃ - 13,000 60分钟。
- 用移液吸出或取出上清液。
- 洗涤沉淀加入1ml的70%乙醇到沉淀。不要旋涡样本。
- 离心样品5分钟,在13000 - 15000×g下4℃。
- 用移液吸出或取出上清液。
- 通风柜在空气干燥的颗粒。储存在-20°CO / N,如果需要沉淀。
- 通过涡旋2分钟,溶解在处理过的H 2ö30微升DEPC处理的沉淀,并使用该溶液,以溶解的pe每个样本对应的第二管的llet(每1提取对30微升的溶液)。
- 测量RNA浓度,并确保它超过1微克/微升在的平均表达的mRNA的引物延伸用。
- 如果需要的话,存储的RNA于-20℃下几个星期到数月。
2.引物延伸反应的
- 引物设计
注:当为引物延伸实验设计引物,服从的PCR引物设计的一般原则(请参阅本文附带的手册自动化凝胶音序器的详细信息和讨论部分)。- 具体地说,确保引物(ⅰ)不含有碱的运行,(ⅱ)具有一个G或C,在3'末端,(ⅲ)有一个平衡的GC:AT比;(4)具有约的退火温度55 - 60℃及(v)结合的至少50碱基对,更好地为100bp的感兴趣区域的下游以接收清晰的图像。
- 引物延伸反应
注:该引物延伸反应(cDNA合成),需要大量的RNA模板。如果RNA的使用量被选择为低,该信号可能太低,以检测!因此,我们建议RNA的纯化,如上所述。
注意:注意:使用无RNA酶的试剂和塑料制品!- 预热的热循环仪中以95℃的温度下,进行了热循环仪的所有进一步的孵育步骤为便于使用和可再现性。
- 混合表2中的化合物在PCR管中的每个RNA样品。
- 变性的样品1分钟,在95℃下。
- 放置在冰上,并寒意的管5分钟,以杂交的RNA和引物。
- PCR仪设定为47℃。
- 在此期间,如表3中所述制备的反转录主混合物。
- 加入4微升反转录预混每个杂交的RNA样本。
- 孵育管1小时在47℃。注意:为AMV RT的最适温度是42℃,但较高的温度有利于克服在RNA分子的二级结构。
- 停止反应,将样品加热至95℃持续2分钟。
注意:甲酰胺是有腐蚀性,有毒,会损害胎儿。请阅读材料安全数据表,轻拿轻放,并穿戴适当的防护! - 加入6微升甲酰胺上样染料(95%(体积/体积)的去离子甲酰胺,10毫摩尔EDTA,0.05%(重量/体积)溴酚蓝),并存储为O / N到两周,在-20℃下在黑暗中。
3.制备测序天梯
注:排序阶梯反应需要两种适量的质粒或大量基因组DNA中。只要有可能,在测序反应中使用的质粒是因为很容易的分离和高的签名的推荐最终强度。在其他情况下,我们经常使用的Marmur 5,14采用的方法制备的大肠杆菌基因组DNA 大肠杆菌和S.金黄色葡萄球菌细胞,而无需使用酚。在主要能产生高量的和基因组DNA的纯度的任何方法都可以使用。
- 基因组DNA提取
- 成长10毫升E的大肠杆菌或S.金黄色葡萄球菌细胞O / N在LB,BM 5或TSB培养基。
- 收获细胞,离心,在4600×g下在15ml falcon管10分钟。
- 在2ml缓冲液P1的重新悬浮沉淀,以一些微型制备试剂盒中找到(50毫摩尔的Tris-HCl pH值为8.0,10mM的EDTA,100μg/ ml的RNA酶A)。
- 裂解细胞45 - 60分钟,用20 - 40微升溶葡萄球菌素(0.5毫克/毫升,保存于-20℃)。注意:对于E.大肠杆菌细胞中的酶的前处理可以被省略或溶菌酶使用。
- 添加饱和的SDS-溶液100微升(在45%乙醇中)的悬浮液和incubatE代表5分钟,在37℃。
- 加入650微升5米的NaClO 4,简要涡细胞。
注:氯仿是潜在的致癌物质。请阅读材料安全数据表,并在通风橱中使用适当的保护! - 添加3毫升氯仿/异戊醇(24:1的比例)的混合物,振摇至少60秒。注意:该液体应该变成均匀的白色乳状液。
- 离心样品10分钟,在4600×g下和室温,分离相。
- 清澈的上层(水相)小心地转移到新的管中。如果溶液混浊,重复氯仿/异戊醇抽提。测量DNA溶液的体积,并准备新的管中与2倍体积的乙醇(100%)。
- 慢慢倒出或吸液管将DNA溶液为含有管的乙醇。注意:DNA应沉淀为透明的,致密的线圈上的底部或当完全脱水的浮白集群。
- 找回对DNA使用从玻璃巴斯德移液管( 图2)制成的钩子和通过浸渍在1ml 70%乙醇中的一个单独的管冲洗各样品的两倍。
- 将钩直立在一个机架和空气干燥沉淀60分钟。如果需要的话,储存在室温几天的干燥的DNA。
- 500微升双蒸2 O -通过破坏掉DNA玻璃盖钩和放置在含有100 2.0毫升的反应管中溶解DNA调节音量到最终1000 DNA浓度 - 1500纳克/微升。一个测序反应中,使用10 - 18微克基因组DNA。

图2说明了如何创建一个DNA钓竿。保持巴斯德玻璃吸管的尖端插入本生灯火焰。这会导致玻璃开始几秒钟后融化,创造了小钩在t他结束。从火焰迅速捞出晾凉,持续1分钟。
- 质粒隔离
- 使用标准的微型制备试剂盒制备的质粒,并溶解于洗脱缓冲液(10mM三Cl,pH值8.5)。根据质粒大小,使用100 - 500毫微克质粒的一个排序阶梯。
- 桑格测序反应
注:请看下面使用荧光标记的引物测序试剂盒与7脱氮-dGTP行之有效的引物延伸的目的,一个简单的协议。请参阅测序试剂盒说明书的详细信息。请注意,该测序反应,必须使用相同的引物作为引物延伸反应,以创建具有相同长度的产品。- 混合12微升基因组DNA(〜10 - 15微克)用1微升的DMSO和1μl的荧光标记的引物(2皮摩尔/微升)。
- 每个1微升4测序反应混合物(A,C,G或T),加入3微升的DNA / DMSO /引物混合物。
- 将样本分为PCR仪,并运行下列PCR程序:95℃2分钟; 35个循环的95℃20秒,54℃20秒,70℃,30秒;保持在4℃,直到永远。
- 运行后,从机器中取出的样品,几天增加6微升样染料和存储的冰(短期)或-20°C至数周。
4.凝胶设置和设备上运行
注:详细信息在测序凝胶装置是如何组装的,该凝胶的制备,以及如何将凝胶中运行所用的制造商协议中找到。
- 准备工作
- 制备10×TBE,如表4所示。
- 在凝胶上跑的一天做好准备1升1X TBE缓冲液用超纯水双蒸2 O.
- 制备10%(重量/体积)的APS。注:可存储在200μl等分试样在-20℃下数月,但活性可能随时间降低<。/ LI>
- 凝胶浇铸室的装配
- 避免玻璃板之间的灰尘和绒毛。用湿纸巾因此彻底清洁工作表面。
- 清洁一对使用一次性纸巾和蒸馏水两侧25厘米玻璃板,再用异丙醇进行玻璃板的内侧面。
- 放置在后玻璃板0.25毫米隔板和降低切口玻璃板上顶端( 图3)。
- 附加凝胶轨到玻璃板与切口端和朝上导轨条目飞行员的两侧并拧紧旋钮轻轻。

图3分解图的凝胶电泳玻璃板。玻璃板应定向使用。照顾到面对玻璃板的内侧向内和外一面朝外。

图4显示的组装凝胶装置。注入的凝胶溶液后,将袋垫片被放置在玻璃板之间的溶液。铸塑板然后在前面玻璃板和凝胶导轨之间滑动,并通过紧固导轨旋钮固定。
- 铸造凝胶
注:非聚合丙烯酰胺具有神经毒性!请阅读材料安全数据表,并与适当的保护使用!- 添加在表5中列出的化合物引入到烧杯中,并混合使用搅拌棒和磁力搅拌器。
- 立即加入APS和TEMED后,占用的50ml注射器,凝胶溶液,并放置一0.45纳米的过滤器上的小费。
- 要么保持玻璃板的顶边,用一只手或放置sandwich的凝胶浇注站创建一个斜坡倾斜的10 - 20°。
- 慢慢地分配所述玻璃板之间的凝胶溶液中,同时连续移动的注射器尖端从一侧到另一和停止一次的凝胶溶液达到底端。
- 移动到一侧或删除完全用泡沫挂钩的任何形成的气泡。
- 滑动玻璃板之间的凝胶袋垫片(0.25mm)的在切口端,浸入到凝胶溶液并通过将浇铸板固定。
- 法尔胜上轨螺丝轻轻(参见图4完全组装设备)。
- 让1硅胶套 - 2小时。
- 取出铸板和口袋间隔和盐凝胶残留清洁袋。
- 冲洗DDH 2 O的擦了纸巾多余的溶液。
- 运行和可视化的凝胶
注:该测序凝胶直接进行电泳在凝胶成像仪,而该荧光通过激光显微镜同时检测。与传统的凝胶电泳,其中所述凝胶中运行,然后再染色和可视化,所述检测单元是固定的,并且扫描带的实时,因为他们通过激光。下面的步骤用于OS / 2上的ImagIR数据收集软件概述,这可以通过向更近的版本。欲了解更多信息,请参见用户手册。- 缓冲罐支架滑入前玻璃板的凝胶导轨并拧紧旋钮。
- 放置凝胶成对着加热板的自动凝胶成像仪的下凝胶罐,并通过滑动导轨条目导频插入装置支架固定。
- 补1×TBE缓冲液中进下和上凝胶缓冲室,关闭下部缓冲室,并连接上缓冲液槽使用的电源线的电源。
- 如果存在,通过反复吹打缓冲区纳入囊中,从清洗盐残留凝胶的口袋里。
- 使用顶部缓冲盖关闭顶部缓冲箱室。
- 关闭机门并打开相机和电脑,并启动相应的ImagIR数据采集软件。
- 创建一个新的项目文件( 文件- >新建...),输入项目文件名 ,选择合适的激光范围(700或800纳米),按OK确认。
- 选择选项 - >自动增益...从顶部图像菜单中,单击自动启动自动增益测量并接受设置,单击OK(确定)。
- 聚焦激光通过选择选项 - >焦点......从扫描仪的控制菜单中,点击自动按钮,然后单击OK(确定)接受设置。
- 重复该自动增益过程,以适应新的重点区域。
- 安装程序会根据这些设定扫描仪控制:2,000 V 35毫安,45 W,45°C,扫描过滤器:3,扫描速度:3。
- 预运行20分钟的空胶(选择电压,然后按<回车> )。
- 在此期间,加热该测序梯和引物延伸产物在PCR机器为2分钟至90℃,然后冷却,在冰上。
- 停止电泳,打开自动凝胶音序器,取下上部缓冲罐的盖子。
- 将鲨鱼齿形梳在玻璃板之间,并微微刺进与鲨鱼齿凝胶(参见图5)。
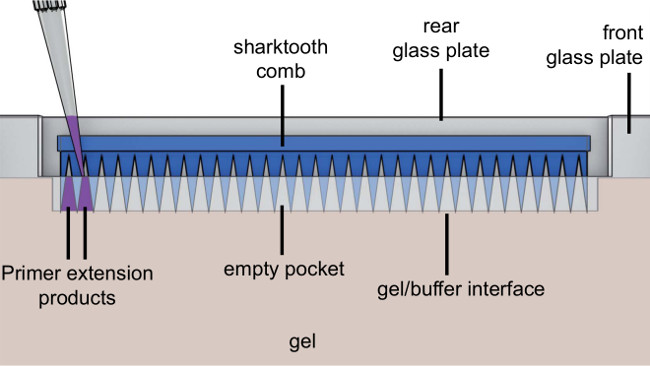
图5.特写视图凝胶鲨鱼齿梳。样品(紫色)是鲨鱼的牙齿之间。
- 移液管是1 - 2μl的引物延伸产物或测序梯反应到每个凝胶袋(由鲨齿形成)。
- 如果需要不是所有的口袋,填补空口袋样染料,以防止一致的运转性能。
- 关闭缓冲罐凝胶音序器和门。
- 开始电泳并打开激光(选择电压和激光ON,然后按 <回车>)。
- 停止电泳一旦感兴趣区域已通过激光器。
结果
如示于图6中 ,引物延伸反应可以用来确定感兴趣转录物的转录起点,并可以帮助推断启动子区域(典型地由在-10和-35元件识别)。最上面的(最长)的cDNA片段代表了mRNA的5'端,因此可相比的测序梯容易地进行映射。

引物延伸反应的图6。代表性结果 。左边一个完整的活体引物延伸,从E.凝胶显示大肠杆菌各种质粒。感兴趣的各个领域被放大。在顶端部分(A),所述的ompA转录起始点的确定是描绘。逆转录停止在mRNA的5'末端,从而产生全长的RNA的条带(的ind由箭头icated)。通过对准的cDNA条带的测序梯,在mRNA的5'末端可被确定为显示在所附的序列。在所述下部(B)中 ,示出了的ompA转录由的YoeB-SEQ2核糖核酸酶的切割。泳道1 - 3表示样品中的毒素的YoeB-SEQ2缺失或不活动的,而泳道4 - 5表示样品有活性RNA酶,与缺少和的cDNA产物的存在相一致。创建两个主要裂解产物中的箭头所指示的。在每个泳道中并测序梯的相应的RNA基所用的ddNTP的标记。谈话部分由蓝色字体,子元素和AUG起始用红色字体标明密码子。欲了解更多信息;看到中止等人的原创性研究文章。,159微生物学 ,1575年至1585年(2013年)。 请点击这里查看该图的放大版本。
在图6所示的例子中, 的ompA mRNA的TSP被确定为一个G为基础的(标记为凝胶下面的序列中的箭头)。这与15年前出版的的ompA TSP一致。启动子的-35和-10元件可以推导为TTGTAA和TAGACT 16的基序的区别仅在于每两个碱基从它们各自的共有序列(TTGACA和TATAAT分别)。
相对于其他方法,如5'RACE,引物延伸反应可以用来精确地确定和定量RNA分子的切割。 RNA分子的裂解产生游离5'端,可以同时检测作为在凝胶中的cDNA条带。标识若干裂解产物1的mRNA在5'RACE实验比较困难,因为几个PCR产物必须被测序,以获得裂解产物仅代表总(未处理)的RNA本体的一小部分。
在图6的下部,所述裂解的RNA酶的RNA作图描述。如前面所描述的图5中,RNA酶的YoeB-SEQ2,的TA系统从S的部分equorum 17,切割mRNA的靠近起始密码子。该裂解可通过同源抗毒素YefM-SEQ2被抑制。当RNA酶的YoeB不存在或不活动的(泳道1 - 3),无引物延伸产物形成靠近起始密码子。每当RNase活性存在(泳道4&5),两强带起始密码子出现短期内下游。使用这种方法,切割图案可以容易地识别。
图7示出一个失败的引物延伸实验。由于在逆转录反应过量的RNA的cDNA的生成量产生如此强烈的信号,即个体的cDNA带S不能区分。这使得5'RNA一端的决心是不可能的。
当使用高表达的RNA的引物延伸反应,如核糖体RNA(如在图7中示出),过度曝光区域的危险性增大。因此,总的模板RNA的量必须被调整到感兴趣的RNA的丰度。与此相反,当所关心的转录只占少量的总RNA,在逆转录反应的量必须增加,否则信号将太微弱(未示出)。这也适用于Sanger测序反应中,每个细胞的多拷贝模板(如rRNA基因或基因编码在质粒上),导致更强的信号。
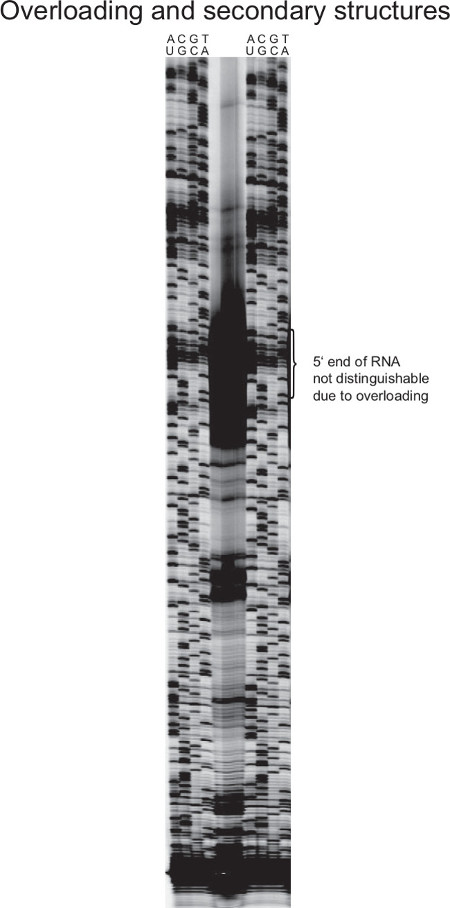
图7.引物延伸,从失败的代表性结果 NG> S.金黄色葡萄球菌 。16S RNA是在细菌细胞中高丰度。这会导致强烈的反转录信号时使用适量的总RNA。在这里所示的情况下,强的cDNA条带掩模的精确5'末端,从而防止5'映射。此外,核糖体RNA是高度结构化的,因此,可以终止逆转录过早,产生不表示全长片段短cDNA产物。在这种情况下,与耐热性逆转录增加反转录温度下,一起可以更容易地合成以往的二级结构。由于在基因组DNA序列的多个拷贝,桑格测序梯比单拷贝基因更强。以改善凝胶图像,RNA和DNA量为逆转录和桑格反应应减少和/或更低的产品加到凝胶。_upload / 52134 / 52134fig7large.jpg"TARGET ="_空白">请点击这里查看该图的放大版本。
表1:RNA的DNA酶I消化。
| 物质 | 量 |
| 从以上步骤RNA | 70 - 100微克 |
| 10倍的DNA酶I缓冲液(100毫摩尔Tris,pH为7.5,25mM的MgCl 2的,5mM的氯化钙2) | 50微升 |
| DNA酶I,RNA酶(2 U /μL) | 10.0微升(最多10个核糖核酸微克每1μl的DNA酶I)的 |
| 把音量开到500微升与DDH 2 O的(DEPC处理) | |
反应体积的DNA酶和量我可以放大或缩小根据定量O˚FRNA使用。
表2:RNA的引物杂交。
| 复合 | 量为1反应 |
| RNA | 5 - 15微克 |
| 荧光标记的引物 | 2皮摩尔 |
| 带量至6微升与DDH 2 O的(DEPC处理) | |
设置各RNA样品和引物一个反应。
表3:引物延伸母液。
| 复合 | 量为1反应 |
| DDH 2 O的(DEPC处理) | 1.3微升 |
| AMV RT缓冲液(10X) | 1.0微升 |
| 的dNTP(10毫米,无RNA酶) | 1.0微升 |
| 核糖核酸酶抑制剂 | 0.2微升 |
| AMV逆转录酶(RT)(20 - 25 U /μL) | 0.5微升 |
向上扩展到所需要的反应的数量。
表4:10X TBE配方。
| 物质 | 量 |
| 三基地 | 107.8克 |
| 硼酸 | 55.0克 |
| EDTA | 7.4克 |
| 带量高达1000毫升超纯DDH 2 O的和过滤器清除灰尘和毛絮 | |
过滤器清除灰尘和毛絮和储存在4℃C.
表5:测序凝胶配方。
| 复合 | 量 |
| 尿素 | 10.5克 |
| DDH 2 O的(MilliQ中) | 13.0毫升 |
| 10X TBE | 2.5毫升 |
| XL快速凝胶溶液 | 5.0毫升 |
| TEMED(N,N,N',N'- Tetramethylethane -1,2-二胺) | 25微升 |
| 的APS(过硫酸铵,10%) | 175微升 |
后迅速加入TEMED和APS的过程中凝胶溶液。
讨论
荧光引物延伸是用于确定RNA的5'末端的简单和快速的方法,无论是对TSP-或仲RNA加工鉴定。由于利用荧光引物,所述反应可以建立和无需额外的安全预防措施,运行(不像在壳体的放射性标记的引物)。作为样品,通过荧光检测到,它们可以被成像,而在电泳过程中,它允许快速的分析相比,放射性的方法,其中X线片是常用的。
在一般情况下,引物延伸反应的质量在很大程度上取决于等级和引物的结合能力。如果该结合位点被选择太靠近感兴趣区域,引物涂片可能掩盖的信号,而一个结合位点太远(> 300 bp)的从5'末端,可能会导致差的信号。
荧光染料必须被共价连接到定制的DNA寡核苷酸的5'端合成与寡核苷酸中应通过HPLC进行纯化,以防止由残留的盐逆转录反应的干扰。用适当的染料修饰(见试剂名单表上兼容染料的详细信息)的寡核苷酸可以从最寡核苷酸合成的公司订购并应贮存在黑暗中。不幸的是,我们不知道任何酶技术,以高度的染料先前存在的寡核苷酸,是可能用放射性标记的核苷酸。
在这里所描述的测序系统中,两种不同的荧光染料可用于同时检测两个样品,因为它们的激发(约700和800纳米)和发射光谱是不同的。除了原有的制造染料,其它染料,例如在试剂列表中指示可用于产生极好的效果。
对于公关的另一重要因素IMER延伸反应是RNA的质量和数量。应当小心以除去DNA污染物,如逆转录酶,如AMV逆转录可以使用DNA作为模板18。
如图7所示,在凝胶的运行频带的检测到的信号强度依赖于在逆转录反应中使用的RNA的量。因此,必须调整总RNA的量,取决于样品中存在的感兴趣的RNA的比例量。该方法的灵敏度低也是其缺点之一,因为低表达的RNA可以是难以察觉。如果没有信号可以在所有被检测到,总RNA的量可以增加或感兴趣的RNA可从质粒被人为地过度表达。
基于引物延伸的荧光凝胶检测灵敏度大约是基于引物延伸19因子10比32 P或33 P放射性同位素低。但是,该缺点可以通过调整的cDNA的量上样到凝胶或通过增加RNA模板的逆转录反应中使用的量进行补偿。在大多数情况下,灵敏度是令人满意的结果4,5-不够高。在组合使用时,荧光引物,用毛细管测序3,具有短曝光时间的优点敏感性相似,放射性引物延伸的报道。
收费是基于引物延伸的荧光和放射性之间的比较是困难的,因为它们依赖于几个因素,如可用的机器,引物,梯反应,可用性和维护一个实验室用于与放射性物质,处置放射性废物,训练和个人的健康风险的工作。荧光引物是大约五至十倍比非标记的标准引物(20 bp)的更昂贵。然而,这些引物可用于至少一年(更可能几年)相比,32 P标记的引物,其具有更短的半衰期。重新标号的引物是耗时和昂贵的,由于需要在频繁的时间间隔的新的放射性物质。如果一组相同的引物被用于在整个长的时间周期,荧光引物是不是规则的,放射性标记的引物便宜。然而,主要的成本点将会是荧光测序或成像仪和单独购买本设备用于引物延伸的目的可能不符合成本效益。这里介绍的方法是相当有趣的谁是持有还是有机会获得这样的计算机组。
如果一个自动化凝胶序器不可用时,可以使用其它方法进行荧光检测。在这种情况下,该凝胶可以在一个标准电泳装置中运行,如果需要干燥,然后转移至荧光成像仪(型号均有其类似于一个佛罗里达州atbed扫描仪)。虽然可视化在运行过程中的凝胶的优点丧失,使用放射性同位素,可避免这种方式,一个重要的优点为实验者。另外,如果有的话,一个毛细管测序可以用于分离和实时检测,这可以帮助提高灵敏度。
各种方法已经公布关于如何使用荧光引物延伸与自动化凝胶测序仪或毛细管测序仪,然而这些方法通常需要的cDNA沉淀步骤以浓缩样品(和去除杂质)19。在这里提出的方法中,DNA的沉淀是没有必要的因此降低了制备时间。然而,更重要,省略此步骤使得可以半定量地测定RNA分子的量与样品中的沉淀过程中的不一致之处可以被消除。
应当指出的是,尽管RNA分子的5'末端可被映射在引物延伸反应,处理和初级端(转录起始点)不能很容易地分辨。为了规避这些限制,适当控制实验的精心策划是必不可少的。明显的启动子序列可以表示一个转录起始点的存在下,然后突变或缺失的启动子元件应取消的cDNA条带。在另一方面,如果这样的序列缺失或特定的共有序列都存在,该带可通过RNA的加工造成的。如果处理的酶是已知的并且可被纯化, 在体外引物延伸可以使澄清,如转录和处理可被分离。此外,其他方法,如5'RACE(包括未加工的RNA分子的酶富集)可以互补的引物延伸来区分的RNA加工的转录起始位点。
日Ë引物延伸方法是经常与其它方法相比,如5'RACE和S1核酸酶保护测定,因此其有用有时质疑。与新一代测序例如组合RNAseq技术可以帮助确定的TSP和许多并行的RNA的加工点,但所需的财政负担,生物信息学的工作,使其不经济的,而单RNA的兴趣。另一方面5'-RACE的是更便宜的,其结果是更容易分析,但如果RNA的多种方法或几个转录起点被处理都存在,该产品必须被克隆,并需要大量的候选的待测序以得到感兴趣的RNA的一个代表性视图。
因此,在出现多年来尽管新方法具有,即使在今天,引物延伸有其存在的理由,由于易于使用,低成本和短的周转时间,这是特别真实对于这里提出的基于荧光的方法。
披露声明
The authors have nothing to disclose that would present a conflict of interest.
致谢
We thank Anne Wochele for her assistance in the laboratory and Vera Augsburger for help with the automated gel sequencer. We thank the Deutsche Forschungsgemeinschaft for funding by grants BE4038/2 and BE4038/5 within the “priority programmes” SPP1316 and SPP1617.
材料
| Name | Company | Catalog Number | Comments |
| AMV Reverse Transcriptase (20-25 U/µl) | NEB / Roche | NEB: M0277-T / Roche: 10109118001 | |
| DNase I (RNase free) | Ambion (life technologies) | AM2222 | |
| FastPrep-24 Instrument | MPBio | 116004500 | |
| Fluorescently labeled primers | Biomers | n/a | 5’ DY-681 modification of “ordinary” DNA oligonucleotides. Compatible dyes such as the LICOR IRDye 700/800 are also available from other suppliers such as IDTdna. |
| Li-Cor 4200 Sequencer incl. ImagIR Data collection software | Li-Cor | Product discontinued | |
| NanoDrop 2000 | Thermo Scientific | ||
| Nuclease free water | Ambion (life technologies) | AM9915G | |
| Plasmid mini preparation kit | QIAGEN | 12125 | |
| RapidGel-XL-40% Concentrate | USB | US75863 | |
| RNA STORAGE BUFFER | Ambion (life technologies) | AM7000 | |
| Roti-Aqua-P/C/I | Carl Roth | X985.3 | Alternative: “Acid-Phenol:Chloroform, pH 4.5 (with IAA, 125:24:1)” from Ambion (AM9720) |
| SUPERase•In RNase Inhibitor | Ambion (life technologies) | AM2696 | |
| Thermo Sequenase fluorescently labelled primer cycle sequencing kit with 7-deaza-dGTP | GE Healthcare | RPN2538 | |
| TRIzol reagent | life technologies | 15596-026 | |
| Zirconia/Silica Beads 0.1 mm | BioSpec | 11079101z |
参考文献
- Simpson, C. G., Brown, J. W. Primer extension assay. Methods Mol. Biol. 49, 249-256 (1995).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proc. Natl. Acad. Sci. U. S. A. 74, 5463-5467 (1977).
- Fekete, R. A., Miller, M. J., Chattoraj, D. K. Fluorescently labeled oligonucleotide extension: a rapid and quantitative protocol for primer extension. Biotechniques. 35, 90-94 (2003).
- Schuster, C. F., et al. Characterization of a mazEF Toxin-Antitoxin Homologue from Staphylococcus equorum. J. Bacteriol. 195, 115-125 (2013).
- Nolle, N., Schuster, C. F., Bertram, R. Two paralogous yefM-yoeB loci from Staphylococcus equorum encode functional toxin-antitoxin systems. Microbiology. 159, 1575-1585 (2013).
- Yamaguchi, Y., Park, J. H., Inouye, M. Toxin-antitoxin systems in bacteria and archaea. Annu. Rev. Genet. 45, 61-79 (2011).
- Schuster, C. F., Bertram, R. Toxin-antitoxin systems are ubiquitous and versatile modulators of prokaryotic cell fate. FEMS Microbiol. Lett. 340, 73-85 (2013).
- Goeders, N., Van Melderen, L. Toxin-antitoxin systems as multilevel interaction systems. Toxins. 6, 304-324 (2014).
- Yamaguchi, Y., Inouye, M. mRNA interferases, sequence-specific endoribonucleases from the toxin-antitoxin systems. Progress in molecular biology and translational science. 85, 467-500 (2009).
- Park, J. H., Yamaguchi, Y., Inouye, M. Bacillus subtilis MazF-bs (EndoA) is a UACAU-specific mRNA interferase. FEBS Lett. 585, 2526-2532 (2011).
- Zhu, L., et al. et al.Staphylococcus aureus MazF specifically cleaves a pentad sequence, UACAU, which is unusually abundant in the mRNA for pathogenic adhesive factor SraP. J. Bacteriol. 191, 3248-3255 (2009).
- Zhu, L., et al. The mRNA interferases, MazF-mt3 and MazF-mt7 from Mycobacterium tuberculosis target unique pentad sequences in single-stranded RNA. Mol. Microbiol. 69, 559-569 (2008).
- Fu, Z., Donegan, N. P., Memmi, G., Cheung, A. L. Characterization of MazFSa, an endoribonuclease from Staphylococcus aureus. J. Bacteriol. 189, 8871-8879 (2007).
- Marmur, J. A procedure for the isolation of deoxyribonucleic acid from micro-organisms. J. Mol. Biol. 3, 208-218 (1961).
- Emory, S. A., Belasco, J. G. The ompA 5' untranslated RNA segment functions in Escherichia coli as a growth-rate-regulated mRNA stabilizer whose activity is unrelated to translational efficiency. J. Bacteriol. 172, 4472-4481 (1990).
- Cole, S. T., Bremer, E., Hindennach, I., Henning, U. Characterisation of the promoters for the ompA gene which encodes a major outer membrane protein of Escherichia coli. Mol. Gen. Genet. 188, 472-479 (1982).
- Schleifer, K. H., Kilpper-Bälz, R., Devriese, L. Staphylococcus arlettae sp. nov., S. equorum sp. nov. and S. kloosii sp. nov.: Three New Coagulase-Negative, Novobiocin-Resistant Species from Animals. Syst. Appl. Microbiol. 5, 501-509 Forthcoming.
- Yu, H., Goodman, M. F. Comparison of HIV-1 and avian myeloblastosis virus reverse transcriptase fidelity on RNA and DNA templates. J. Biol. Chem. 267, 10888-10896 (1992).
- Ying, B. W., Fourmy, D., Yoshizawa, S. Substitution of the use of radioactivity by fluorescence for biochemical studies of RNA. RNA. 13, 2042-2050 (2007).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。