Method Article
Fluorescence Basé Technique d'extension d'amorce pour déterminer transcription Points de départ et des sites de clivage de RNases
Dans cet article
Résumé
We here describe a fluorescence based primer extension method to determine transcriptional starting points from bacterial transcripts and RNA processing in vivo using an automated gel sequencer.
Résumé
L'extension d'amorce à base de fluorescence (FPE) est une méthode moléculaire pour déterminer les points de départ de la transcription ou des sites de traitement de molécules d'ARN. Ce résultat est obtenu par transcription inverse de l'ARN d'intérêt en utilisant des amorces marquées par fluorescence spécifiques et l'analyse ultérieure des fragments d'ADNc obtenus par électrophorèse dénaturante sur gel de polyacrylamide. En même temps, une réaction classique de séquençage de Sanger est exécuté sur le gel vers la carte les extrémités des fragments d'ADNc à leurs bases correspondantes exactes. Contrairement à 5'-RACE (Rapid Amplification of cDNA Ends), où le produit doit être cloné et séquencé plusieurs candidats, la majeure partie des fragments d'ADNc générés par extension d'amorce peut être détecté simultanément en une seule opération de gel. En outre, l'ensemble de la procédure (à partir de la transcription inverse de l'analyse finale des résultats) peut être complété en une journée de travail. En utilisant des amorces marquées par fluorescence, l'utilisation de réactifs dangereux isotope radioactif étiquetépeut être évitée et les délais de traitement sont réduites comme produits peuvent être détectés au cours de la procédure d'électrophorèse.
Dans le protocole suivant, nous décrivons une méthode d'extension d'amorce fluorescente in vivo pour détecter de manière fiable et rapide des extrémités 5 'des ARN de déduire la transcription des points de départ et les sites de traitement de l'ARN (par exemple, par les composants du système toxine-antitoxine) en S. aureus, E. coli et d'autres bactéries.
Introduction
Une extension d'amorce est une méthode moléculaire pour déterminer les extrémités 5 'des molécules d'ARN spécifiques à une résolution d'une base. L'avantage à d'autres méthodes telles que 5'-RACE (amplification rapide des extrémités d'ADNc) est le délai d'exécution rapide et la capacité d'analyser facilement un mélange de différentes longueurs de molécules d'ARN.
Cette méthode fonctionne en soumettant des molécules d'ARN à des réactions de transcription inverse en utilisant des amorces fluorescentes spécifiques, générant des fragments d'ADNc de certaines longueurs. Ces molécules d'ADNc sont longent les réactions de séquençage de Sanger traditionnels 2 sur des gels dénaturants de polyacrylamide et peuvent être détectés par leur fluorescence en raison de l'utilisation d'amorces marquées par fluorescence. Les longueurs des fragments d'ADNc sont ensuite évaluées par comparaison de l'échelle de séquençage, ce qui permet la mise en correspondance de l'extrémité 5 'de l'ARN.
Traditionnellement, les réactions d'extension d'amorces sont utilisées en conjonctionavec des isotopes radioactifs pour détecter des molécules d'ADNc sur des films radiographiques. En raison de risques pour la santé, les problèmes d'élimination des déchets et la facilité de manipulation, des protocoles plus récents utilisent la fluorescence pour la détection de l'extension d'amorce avec des séquenceurs automatiques, mais leur sensibilité est légèrement inférieure. En utilisant des amorces marquées par fluorescence, la procédure récurrente de radio-étiquetage peut être omis, comme des amorces fluorescentes sont stables pendant une longue période (plus d'un an dans nos mains).
La méthode que nous décrivons ici utilise un séquenceur automatisé de gel, mais avec de légères modifications, séquenceurs capillaires peut être également utilisée pour la séparation et la détection de l'ADNc 3. La nature de l'analyse parallèle de gel permet de détecter même une petite quantité de clivage de l'ARN ou de traitement. Un autre avantage est la haute résolution de cette méthode, comme clivage ou le traitement d'une seule base de la borne peut être détecté.
En ce qui concerne la détection de clivage d'ARN ou de transformation, tabituellement deux différents types d'extensions d'amorces sont distingués. Dans un cas, le traitement enzymatique est effectué in vitro en utilisant l'ARN purifié et enzyme purifiée, alors que dans l'autre cas, le traitement est réalisé in vivo et l'ARN obtenu est purifié. Dans les deux cas, l'ARN est soumis à une extension d'amorce effectuée in vitro, cependant, en fonction de la source d'ARN, le procédé est appelé soit une extension d'amorce in vitro ou in vivo. Dans le protocole que nous présentons ici, nous nous concentrons uniquement sur l'extension vivo en primaire, en raison de la facilité d'utilisation (pas de protéines purifiées nécessaire) et la possibilité de déterminer les points de départ de la transcription et de traitement dans le même temps. Cependant, in vitro extensions d'amorces sont en principe mis en place de la même manière et ce protocole peuvent servir de point de départ.
Le procédé illustré ici peut être appliquée à de nombreuses espèces bactériennes tant qu'ils se prêtent à une grandepureté et à haut rendement de préparation d'acides nucléiques.
La recherche dans notre laboratoire se concentre sur la portée réglementaire de la toxine-antitoxine (TA-systèmes) 4,5, un domaine dans lequel la méthode d'extension d'amorce est largement utilisé. AT-systèmes sont de petits éléments génétiques présents dans les génomes procaryotes qui sont constitués d'une protéine toxique et stable et actif de manière endogène une protéine ou un ARN essentiellement instable antitoxine qui neutralise la toxicité 6,7. l'activité de la toxine est parfois exercée par inhibition de la réplication, la synthèse de la paroi cellulaire ou d'autres mécanismes, mais le plus souvent par l'activité RNase 8,9. En général, la spécificité de la RNase est déterminée en effectuant des tests différents, dont l'un est le procédé d'extension d'amorce. Réactions d'extension d'amorce sont bien adaptés pour cette application, comme un mélange de fragments de longueur clivés et complètes peuvent être analysés simultanément pour déterminer leurs extrémités 5 '. Utilisation d'un mélange de in vitro et in vivo des extensions d'amorce, latoxine spécifique clivage RNase, par exemple, une spécificité de séquence peut être déterminée 10 à 13.
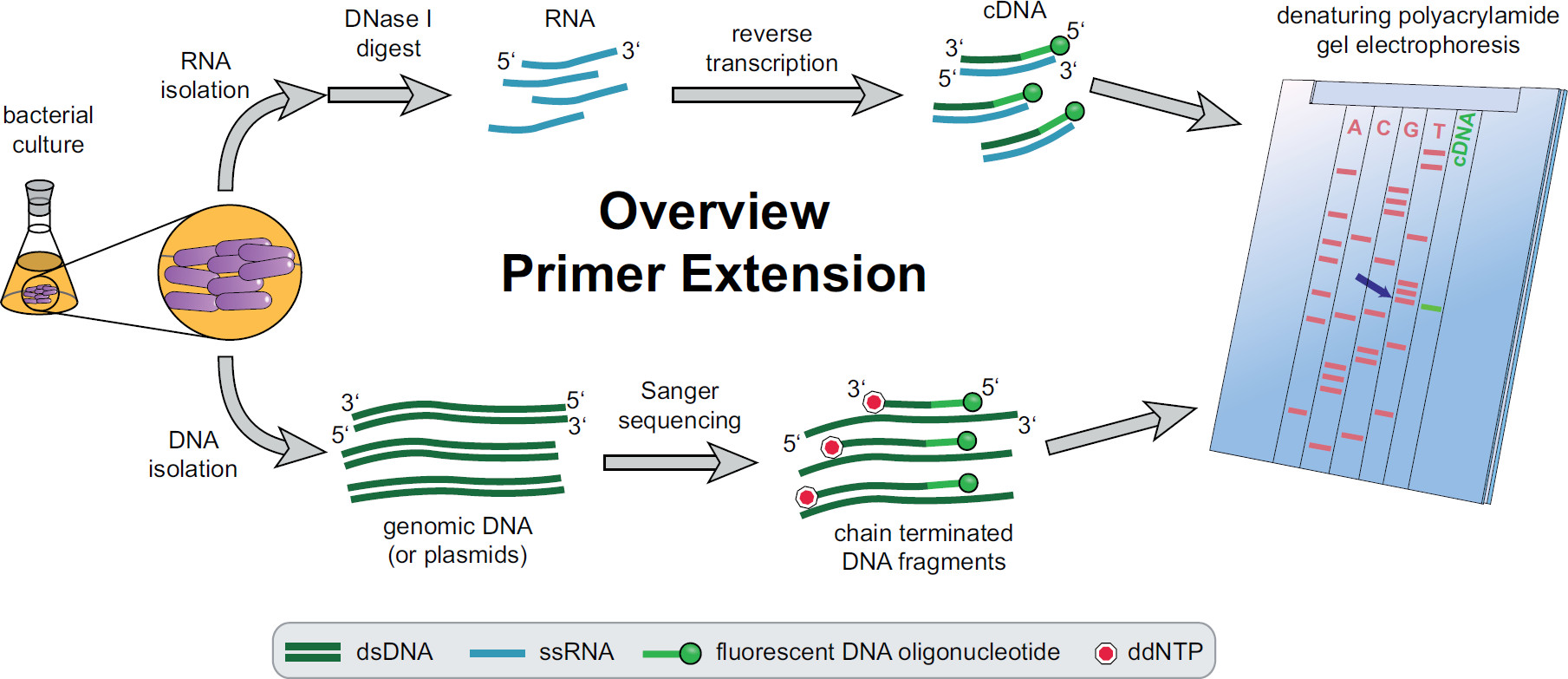
Figure 1. Vue d'ensemble de la procédure d'extension d'amorce. Les cultures bactériennes sont incubées et traitées selon les besoins expérimentaux. L'ARN total est extrait des cellules, traités à la DNase I pour éliminer les traces d'ADN et soumis à une réaction de transcription inverse en utilisant des amorces fluorescentes spécifiques des cibles d'ADN donnant des ADNc. L'ADN génomique ou de plasmides sont extraites et utilisées par la suite pour des réactions de séquençage de Sanger fluorescentes pour la comparaison de la taille des fragments d'ADNc. Produits d'extension d'amorce sont longent produits de séquençage Sanger sur un gel de polyacrylamide dénaturant urée et analysés avec un laser automatisé et microscope. La base de séquençage qui aligne avec la bande d'ADNc est laer but de l'extrémité 5 'de l'ADNc (flèche bleue). Plus d'informations dans Fekete, et al. 3 Se il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Une vue d'ensemble de l'ensemble de la procédure d'extension d'amorce se trouve dans la figure 1. En bref, les cellules bactériennes sont cultivées, récoltées, le culot de cellules lysées et l'ARN extrait. L'ARN purifié est ensuite traité avec de la DNase I pour éliminer les traces de molécules d'ADN qui pourraient agir en tant que modèles pour la transcriptase inverse. Des amorces fluorescentes spécifiques sont ajoutés à l'ARN, hybride à la région d'intérêt et inverser ensuite transcrit, résultant en un ADN complémentaire simple brin (ADNc). Une échelle de séquençage est réalisé par séquençage de Sanger traditionnel utilisant des amorces fluorescentes et séparé sur un gel de polyacrylamide dénaturant à côté de l'extension des fragments d'ADNc d'amorces. La résultantegel est analysé par comparaison des bandes fluorescentes, permettant l'identification des extrémités 5 'd'intérêt. Points de départ de la transcription et des sites de traitement sont ensuite évaluées individuellement par des comparaisons de séquences.
Protocole
1. ARN à haut rendement Préparation
- Isolement de l'ARN
REMARQUE: Les fortes concentrations d'ARN total sont nécessaires pour la réaction d'extension d'amorce. Kits de colonne de centrifugation ne donnent généralement pas la quantité d'ARN nécessaire (~ 5 à 16 ug dans un volume de 5 ul). Par conséquent, la purification en utilisant la méthode d'extraction thiocyanate-phénol-chloroforme guanidinium acide est recommandé, décrit ci-dessous.
REMARQUE: Le phénol est cancérigène, toxique et corrosif. Se il vous plaît lire les fiches de données de sécurité des matériaux et utiliser sous une hotte avec une protection appropriée!- Développer ou traiter les cellules bactériennes (S. aureus ou E. coli dans cet exemple) comme vous le souhaitez et la récolte par centrifugation 10 min à 4600 x g et 4 ° C. Remarque: En général, nous récoltons un OD totale de 20 600 - 70. Les culots cellulaires peuvent être stockés pendant plusieurs semaines à -20 ° C.
- Remettre en suspension le culot cellulaire dans 1 ml de solution de thiocyanate-phénol-chloroforme guanidinium acide et transférer à un2 ml vis tasse contenant 0,5 ml de 0,1 mm perles de verre zirconium / silice.
- Lyse des cellules à trois reprises un batteur préparation rapide / perle à 6,5 m / s pendant 30 secondes pour trois tours de refroidissement, les échantillons sur la glace pendant 5 min après chaque course. Remarque: échantillon homogénéisé peut être conservé à -80 ° C pendant plusieurs semaines.
- Incuber lysat pendant 5 min à TA, puis ajouter 200 ul de chloroforme.
- Secouez vigoureusement ou vortex de l'échantillon pendant 30 secondes pour extraire l'ARN.
- Incuber à température ambiante pendant 3 minutes, puis centrifuger pendant 15 min à 13 000 - 15 000 x g et 4 ° C. Remarque: Les solvants sont séparés en une phase organique inférieure (rose, contient des protéines), une interphase (blanc, contient de l'ADN) et une phase aqueuse supérieure (clair, contient de l'ARN).
NOTE: A partir de cette étape sur l'utilisation que RNase réactifs libres et les articles en plastique! - Préparer une solution fraîche sans RNase-tubes de 1,5 ml de réaction, l'étiquette appropriée et ajouter environ 500 pi de 100% RNase isopropanol chaque (utiliser environ la même volume comme phase aqueuse dans le tube précédent).
- Tenir le tube à un angle et transférer la phase aqueuse (environ 500 pi) de tubes préparés à la RNase conseils gratuits. Ne pas déranger l'interphase.
- Précipiter l'ARN en retournant plusieurs fois et en incubant pendant 10 min à température ambiante.
- Centrifuger les échantillons pendant 15 min à 13 000 - 15 000 × g et 4 ° C et éliminer le surnageant par pipetage ou aspiration (pompe à jet d'eau et biberon). Ne pas perturber le culot d'ARN blanc transparent en bas.
- Ajouter 1 ml de 70-80% RNase éthanol libre (ne pas vortex) à laver. Remarque: l'ARN dans de l'éthanol peut être stocké pendant plusieurs semaines à -20 ° C.
- Centrifugeuse pendant 5 min à 7500 x g et 4 ° C et éliminer le surnageant par pipetage ou de préférence aspiration.
- Air culot d'ARN sécher pendant 15 - 30 min sous la hotte. Ne pas trop sécher, sinon pellets peuvent être difficiles à dissoudre.
- Reprendre le culot dans 50 ul de RNase ddH 2 O ou tampon de stockage de l'ARN.
- Mesurer la concentration de l'ARN avec un micro-volume spectrophotomètre UV-Vis ou une cuve en quartz (photomètre et classique) et procéder à la DNase I digestion.
- DNase I digestion de l'ARN pour éliminer les traces d'ADN
NOTE: L'ADN peut agir en tant que matrice dans la réaction parasite de transcription inverse (extension d'amorce), elle doit être retirée de l'échantillon. Diverses méthodes pour enlever l'ADN de solutions d'ARN sont disponibles qui reposent généralement sur digestion par la DNase. Une méthode simple et efficace, mais efficace et le coût pour l'enlèvement d'ADN est décrite ci-dessous.- Bain d'eau préchauffé à 37 ° C.
- Mélanger les composés énumérés dans le tableau 1 dans un tube de 1,5 ml de réaction.
- Incuber le mélange pendant 1 heure à 37 ° C dans un bain d'eau, puis procéder directement à l'extraction au phénol / chloroforme. Remarque: inactivation par la chaleur de la DNase I est pas recommandé, car cela pourrait dégrader l'ARN.
- Extraction phénol / chloroforme de l'ARN après DNase I Digestion
NOTE: L'ARN doit être purifié pour éliminer les nucléotides libres, des fragments d'ADN et les composants du tampon de digestion de la DNase I. Extraction au phénol / chloroforme permet une récupération élevée et la concentration de l'échantillon d'ARN et est par conséquent décrit ci-dessous. D'autres procédés de purification de l'ARN pourraient également être utilisées, si elles satisfont à ces exigences.- Diviser la DNase I 500 ul digestion mélanger en deux échantillons de 250 pi dans 2 ml de tubes de réaction.
- Ajouter 1 volume (250 pi) d'une solution d'acide P / C / I (dans de l'eau saturée de phénol, chloroforme et l'isopentanol, le rapport de 25: 24: 1, à pH 4,5 à 5).
REMARQUE: solution de P / C / I est cancérigène, toxique et corrosif. Se il vous plaît lire les fiches de données de sécurité des matériaux et utiliser sous une hotte avec une protection appropriée! - Vigoureusement vortex ou dans une plate-forme de vortex pendant 1 - 3 min.
- Centrifuger pendant 30 min à 13 000 - 15 000 x g et 4 ° C.
- Recueillir le (aqueuse) phase supérieure et transférer à nouveau tube (250 pi).
- Ajouter 1/9 volume (28 pi) de 3 M d'acétate de sodium pH 5,2.
- Ajouter 2,5-3 volumes d'éthanol pur (700 pi).
- Mélanger au vortex peu et lieu à -80 ° C pendant 30 min ou à -20 ° C pour les 2 - 3 heures. Si nécessaire, stocker l'ARN O / N à -20 ° C.
- Centrifuger pendant 30 - 60 min à 13 000 - 15 000 x g et 4 ° C.
- Retirer le surnageant par pipetage ou aspiration.
- Laver la pastille en ajoutant 1 ml d'éthanol à 70% sur la pastille. Ne échantillon de vortex.
- échantillon de centrifugeuse pendant 5 min à 13 000 - 15 000 × g et 4 ° C.
- Retirer le surnageant par pipetage ou aspiration.
- Air culot sec sous la hotte. Conservez le culot à -20 ° CO / N si nécessaire.
- Dissoudre le culot dans 30 ul traitée DEPC H 2 O par vortex pendant 2 min et en utilisant cette solution de dissoudre la pellet du second tube correspondant par l'échantillon (30 ul de solution par une paire d'extraction).
- Mesurer la concentration d'ARN, et veiller à ce qu'elle dépasse 1 pg / pl pour une utilisation dans l'extension d'amorce d'ARNm moyennement exprimées.
- Si nécessaire, stocker l'ARN à -20 ° C pendant plusieurs semaines à quelques mois.
Réaction 2. Primer Extension
- Conception Primer
REMARQUE: Lors de la conception d'amorces pour une expérience d'extension d'amorce, obéir directives générales de conception d'amorce PCR (voir le manuel accompagnant le séquenceur automatisé gel pour plus de section information et de discussion dans cet article).- En particulier, faire en sorte que les amorces (i) ne contiennent pas de pistes de bases, (ii) posséder un G ou C à l'extrémité 3 ', (iii) avoir une GC équilibrée: AT rapport, (iv) avoir une température de recuit de l'ordre de 55 - 60 ° C et (c) se lient au moins 50 pb, 100 pb en aval mieux de la région d'intérêt à recevoir des images claires.
- Réaction d'extension d'amorce
REMARQUE: La réaction d'extension d'amorce (de synthèse d'ADNc) nécessite de grandes quantités d'ARN matrice. Si les quantités d'ARN utilisés sont choisis à bas, le signal peut être trop faible pour détecter! Nous vous recommandons donc la purification de l'ARN comme décrit ci-dessus.
NOTE: ATTENTION: L'utilisation RNase réactifs libres et les articles en plastique !!!- Préchauffer le thermo-cycleur à une température de 95 ° C et procéder à toutes les autres étapes d'incubation dans un thermo-cycleur pour la facilité d'utilisation et de reproductibilité.
- Mélanger les composés du Tableau 2 dans un tube de PCR pour chaque échantillon d'ARN.
- Dénaturer les échantillons pendant 1 min à 95 ° C.
- Placer les tubes sur la glace et le froid pendant 5 min à hybrider les ARN et des amorces.
- Régler la machine PCR à 47 ° C.
- Dans le même temps de préparer le mélange maître de transcription inverse comme décrit dans le tableau 3.
- Ajouter 4 pi de transcription inverse mélange maître à chaque ARN hybrideéchantillon.
- Incuber les tubes pendant 1 heure à 47 ° C. Note: La température optimale pour AMV RT est de 42 ° C, des températures plus élevées mais aident à surmonter les structures secondaires des molécules d'ARN.
- Arrêter la réaction en chauffant les échantillons à 95 ° C pendant 2 min.
REMARQUE: Le formamide est corrosif, toxique et peut être nocif pour l'enfant à naître. Se il vous plaît lire les fiches de données de sécurité des matériaux, manipuler avec soin et porter une protection appropriée! - Ajouter 6 pi de colorant de charge formamide (95% (v / v) formamide désionisée, EDTA 10 mM, 0,05% (p / v) bleu de bromophénol) et magasin pour O / N à deux semaines à -20 ° C dans l'obscurité.
3. Préparation de l'échelle de séquençage
NOTE: La réaction de séquençage de l'échelle nécessite soit des quantités modérées de plasmides ou de grandes quantités d'ADN génomique. Chaque fois que possible, l'utilisation de plasmides dans la séquence de réaction est recommandé en raison de la facilité d'isolement et de haute sigintensité nal. Dans d'autres cas, nous utilisons systématiquement une méthode adoptée de Marmur 5,14 pour préparer l'ADN génomique de E. coli et S. aureus cellules sans la nécessité d'utiliser du phénol. En principe, n'importe quel procédé qui produit des quantités élevées de pureté et de l'ADN génomique peut être utilisé.
- Isolement de l'ADN génomique
- Croître de 10 ml de E. coli ou S. cellules aureus O / N en LB, BM 5 ou milieu du BST.
- cellules de récolte par centrifugation pendant 10 min à 4600 x g dans un tube Falcon de 15 ml.
- Culot remis en suspension dans 2 ml de tampon P1 que l'on trouve dans certains kits de préparation de mini (Tris-HCl 50 mM pH 8,0, EDTA 10 mM, 100 ug / ml de RNase A).
- Lyse des cellules pendant 45 - 60 min à 20 - 40 pi de la lysostaphine (0,5 mg / ml, le stockage à -20 ° C). Remarque: Pour E. cellules de E. coli le prétraitement enzymatique peut être soit omises ou lysozyme utilisé.
- Ajouter 100 ul de solution saturée de SDS-(dans 45% d'éthanol) à la suspension et Incubate pendant 5 min à 37 ° C.
- Ajouter 650 ul 5 M NaClO 4 et les cellules de vortex brièvement.
REMARQUE: Le chloroforme est un cancérogène potentiel. Se il vous plaît lire les fiches de données de sécurité des matériaux et utiliser sous une hotte avec une protection appropriée !!! - Ajouter 3 ml de chloroforme / isopentanol (rapport 24: 1) au mélange et agiter pendant au moins 60 secondes. Remarque: Le liquide doit se transformer en une émulsion blanche homogène.
- échantillon de centrifugeuse pendant 10 min à 4600 x g et RT pour séparer les phases.
- Soigneusement transférer la phase claire supérieure (aqueuse) dans un nouveau tube. Si la solution est trouble, répéter l'extraction chloroforme / isopentanol. Mesurer le volume de la solution d'ADN et préparer un nouveau tube avec 2 volumes d'éthanol (100%).
- Lentement décanter ou d'une pipette la solution d'ADN dans l'éthanol contenant du tube. Remarque: l'ADN devrait précipiter, bobines denses comme transparentes sur le fond ou quand il est complètement déshydraté comme un cluster blanc flottant.
- Récupérer le DNA utilisant des crochets fabriqués à partir de pipettes de Pasteur en verre (figure 2) et laver chaque échantillon deux fois par immersion dans un tube individuel de 1 ml d'éthanol à 70%.
- Placez les crochets en position verticale dans un rack et sécher à l'air le culot pendant 60 min. Si nécessaire, l'ADN ranger séché pendant plusieurs jours à température ambiante.
- Dissoudre l'ADN par la rupture des ADN verre recouverte de crochets et en plaçant dans un tube de réaction de 2,0 ml contenant 100 - 500 ul trou DDH 2 O. Ajuster le volume à une concentration finale d'ADN de 1 000 - 1 500 ng / pl. Pour une réaction de séquençage, utiliser 10 à 18 ug d'ADN génomique.

Figure 2. Instruction sur la manière de créer une canne à pêche de l'ADN. Maintenez la pointe d'une pipette Pasteur en verre dans la flamme d'un brûleur Bunsen. Cela provoque le verre pour commencer la fusion après quelques secondes, la création d'un petit crochet à til se termine. Retirer rapidement du feu et laisser refroidir pendant 1 min.
- Plasmide Isolation
- Préparation des plasmides en utilisant des kits de préparation de mini standard et dissoudre dans du tampon d'élution (10 mM Tris-Cl, pH 8,5). En fonction de la taille du plasmide, en utilisant de 100 à 500 ng de plasmide pour une échelle de séquençage.
- Réaction de séquençage Sanger
REMARQUE: Nous mettons un protocole simple qui utilise un kit d'amorce de séquençage marquée par fluorescence avec 7-déaza-dGTP qui fonctionne bien dans le but d'extensions d'amorces. Reportez-vous au manuel du kit de séquençage pour des informations détaillées. Se il vous plaît noter que la réaction de séquençage doit utiliser la même amorce que la réaction d'extension d'amorce pour créer des produits de la même longueur.- Mélanger 12 pl de l'ADN génomique (~ 10 à 15 ug) avec 1 pi de DMSO et 1 ul d'amorce marqué par fluorescence (2 pmol / pl).
- Pour chaque 1 pl des quatre mélanges réactionnels de séquençage (A, C, G ou T), ajouter 3 pl de l'ADN / DMSO / mélange d'amorces.
- Placer les échantillons dans un appareil de PCR, et exécuter le programme PCR suivant: 95 ° C pendant 2 min; 35 cycles de 95 ° C pendant 20 sec, 54 ° C pendant 20 sec, 70 ° C pendant 30 sec; conserve à 4 ° C pour toujours.
- Après la course, retirer les échantillons de la machine, ajouter 6 pi de colorant de charge et conserver dans la glace (à court terme) ou à -20 ° C pendant plusieurs jours à plusieurs semaines.
4. Gel installation et appareil Run
REMARQUE: Des informations détaillées sur la façon dont le dispositif de gel de séquençage est assemblé, le gel est préparé le gel et la manière dont est exécuté se trouve dans le protocole du fabricant.
- Préparations
- Préparer TBE 10x comme indiqué dans le tableau 4.
- Le jour de la course de gel préparer 1 L de tampon TBE 1x avec ultrapure ddH 2 O.
- Préparer 10% (p / v) APS. Remarque: peut être stocké dans 200 aliquotes à -20 ° C pendant plusieurs mois, mais l'activité peut diminuer au fil du temps <./ Li>
- Assemblée de la chambre de coulée de gel
- Éviter la poussière et les peluches entre les plaques de verre. Surfaces de travail a donc bien nettoyer à l'aide de lingettes humides.
- Nettoyer une paire de plaques de verre de 25 cm à l'aide de serviettes en papier jetables et de l'eau distillée des deux côtés de l'isopropanol et ensuite de la face interne des plaques de verre.
- Placez 0,25 mm entretoises sur la plaque de verre arrière et abaisser la plaque de verre crantée sur le dessus (Figure 3).
- Fixez les rails de gel pour les deux côtés des plaques de verre avec la fin crantée et les pilotes d'entrée de rail vers le haut et serrer les boutons légèrement.

Figure 3. Vue des plaques de verre d'électrophorèse sur gel éclatée. Les plaques de verre doivent être utilisées de manière directionnelle. Prenez soin de faire face à la face interne des plaques de verre vers l'intérieur et l'extérieurl'autre vers l'extérieur.

Figure 4. Vue d'un dispositif de gel assemblé. Après injection de la solution de gel, la poche d'espacement est placé dans la solution entre les plaques de verre. La plaque de coulée est ensuite glissée entre la plaque de verre avant et les rails de gel et fixé par les boutons de fixation de rail.
- Couler le gel
REMARQUE: acrylamide non polymérisé est neurotoxique! Se il vous plaît lire les fiches de données de sécurité des matériaux et utiliser une protection appropriée !!!- Ajouter les composés énumérés dans le tableau 5 dans un bécher et mélanger à l'aide d'un agitateur et d'un agitateur magnétique.
- Immédiatement après l'ajout d'APS et TEMED, prendre la solution de gel dans une seringue de 50 ml et placer un filtre de 0,45 nm sur la pointe.
- Soit maintenir le bord supérieur de la plaque de verre avec une main ou placer le, savoureuxh dans une coulée de gel support pour créer une pente inclinée 10 - 20 °.
- Passer lentement la solution de gel entre les plaques de verre tout en déplaçant en continu la pointe de la seringue d'un côté à l'autre et d'arrêt une fois que la solution de gel répond à l'extrémité inférieure.
- Déplacer vers le côté ou supprimer complètement les bulles formées à l'aide d'un crochet de bulle.
- Faites glisser l'entretoise de poche de gel (0,25 mm) entre les plaques de verre à l'extrémité entaillée, plonger dans la solution de gel et fixer par la fixation de la plaque de coulée.
- Tirefonds supérieure Fixer légèrement (voir la figure 4 pour appareil entièrement assemblé).
- Laissez ensemble de gel pour 1 - 2 h.
- Retirer la plaque et poche de coulée d'écartement et nettoyer la poche de résidus de sel et gel.
- Rincer avec le trou DDH 2 O et essuyez l'excédent de solution avec de papiers tissu.
- Courir et visualiser le gel
REMARQUE: Les gels de séquençage sont directement soumis à une électrophorèse en gel de l'imageur, tout enla fluorescence est détectée simultanément par un microscope laser. Contrairement à l'électrophorèse sur gel classique, où le gel est exécutée en premier et ensuite coloré et visualisée, l'unité de détection est fixe et balaie les bandes en temps réel lors de leur passage au laser. Ci-dessous une procédure pour le logiciel de collecte de données sur ImagIR OS / 2 est décrite, qui peut être adoptée pour les versions plus récentes. Pour plus d'informations, voir le manuel d'utilisation.- Faites glisser le support de réservoir tampon dans les rails de gel sur les plaques de verre avant et serrez les boutons.
- Placer le gel dans la cuve de gel inférieure de l'appareil d'imagerie automatisée gel contre la plaque de chauffage et de fixer par glissement de l'entrée pilote du rail dans les supports de l'appareil.
- Remplissez tampon TBE 1x dans les chambres de tampons gel inférieures et supérieures, fermer la chambre de tampon inférieure et relier la chambre de tampon supérieure à la puissance à l'aide du cordon d'alimentation.
- Le cas échéant, nettoyer la poche de gel de sel résidus par pipetage à plusieurs reprises tampon dans la poche.
- Fermez la chambre de réservoir tampon supérieure en utilisant le couvercle du tampon haut.
- Fermez la porte de la machine et de passer sur l'imageur et l'ordinateur et lancer le logiciel de collecte de données de base ImagIR.
- Créer un nouveau fichier de projet (Fichier> Nouveau ...), entrez un nom de fichier de projet, sélectionner les gammes de laser appropriés (700 ou 800 nm) et validez avec OK.
- Sélectionnez Options-> gain automatique ... dans le menu d'image en haut, cliquez sur Auto pour démarrer la mesure de gain automatique et accepter les paramètres en cliquant sur OK.
- Concentrer le laser en sélectionnant Options-> Zoom ... dans le menu de commande du scanner, en cliquant sur le bouton Auto et accepter les paramètres en cliquant sur OK.
- Répétez la procédure de gain automatique pour ajuster à la nouvelle région ciblée.
- Configurer le contrôle du scanner en fonction de ces paramètres: 2000 V, 35 mA, 45 W, 45 ° C, filtre de numérisation: 3, Vitesse de numérisation: 3.
- PreRun le gel vide pendant 20 min (sélectionnez tension ON et appuyez sur ).
- Dans le même temps, la chaleur de l'échelle de séquençage et les produits d'extension d'amorce dans une machine de PCR pendant 2 min à 90 ° C, puis refroidir sur de la glace.
- Arrêtez le électrophorèse, ouvrez le séquenceur automatisé gel et enlever le couvercle du réservoir tampon supérieure.
- Insérez la dent de requin-peigne entre les plaques de verre et légèrement percer le gel avec les dents de requin (voir Figure 5).
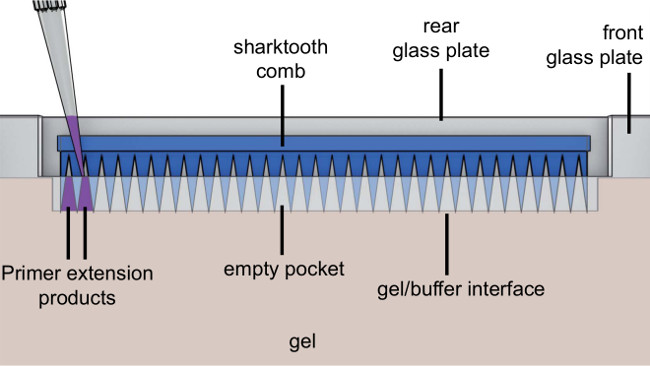
Figure 5. Vue rapprochée de gel avec un peigne dent de requin. Exemple (violet) est appliquée entre les dents de requins.
- Pipette soit 1 - 2 pl de l'amorce produits d'extension ou de réactions de séquençage de l'échelle dans chaque poche de gel (formé par les dents de requin).
- Si toutes les poches sont nécessaires, remplir les poches vides avec colorant de charge pour éviter danscomportement de marche constante.
- Fermer le réservoir tampon et la porte du séquenceur de gel.
- Lancer électrophorèse et allumer laser (Select tension sur et Laser ON et appuyez sur ).
- Arrêtez une fois électrophorèse région d'intérêt a passé le laser.
Résultats
Comme le montre la figure 6, une réaction d'extension d'amorce peut être utilisée pour déterminer les points de départ de la transcription de transcrits d'intérêt et peut aider à déduire régions promotrices (typiquement identifié par -10 et -35 éléments). La plus haute (la plus longue) représente un fragment d'ADNc de l'extrémité 5 de l'ARNm et peut donc être facilement mis en correspondance par rapport à l'échelle de séquençage.

Figure 6. Représentant résultat d'une réaction d'extension d'amorce. Sur le côté gauche une vivo de gel de l'extension complète en amorce de E. coli avec différents plasmides est représentée. Les différents domaines d'intérêt sont agrandies. Dans la partie supérieure (A), la détermination du point de départ de la transcription ompA est représenté. La transcription inverse arrête à l'extrémité 5 'de l'ARNm et crée une bande de l'ARN de pleine longueur (donc indicated par une flèche). En alignant la bande d'ADNc avec l'échelle de séquençage, l'extrémité 5 'de l'ARNm peut être déterminée comme indiqué dans la séquence d'accompagnement. Dans la partie inférieure (B), le clivage de la transcription par la RNase ompA-YoeB seq2 est représenté. Les pistes 1-3 représentent des échantillons dans lesquels la toxine YoeB-seq2 est manquant ou inactifs, alors que les voies 4-5 représentent des échantillons avec une RNase actif, coïncidant avec l'absence et en présence de produits d'ADNc. Les deux principaux produits de dissociation sont créés comme indiqué par les flèches. Les ddNTP utilisés dans chaque voie et la base de l'ARN correspondant de l'échelle de séquençage est marquée. parties de transcription sont indiqués par une police bleue, les éléments promoteurs et début codon AUG par la police de magenta. Pour plus d'informations; voir l'article de recherche original de Nolle et al., microbiologie 159, 1575-1585 (2013). Se il vous plaît cliquez ici pourvoir une version plus grande de cette figure.
Dans l'exemple représenté sur la figure 6, le PTS de la ompA ARNm a été déterminée comme étant une base de G (indiqué par une flèche dans la séquence ci-dessous le gel). Ceci est cohérent avec le TSP ompA publiée avant 15. Les éléments -35 et -10 du promoteur peuvent être déduites à être TTGTAA TAGACT 16 et que les motifs ne diffèrent que deux bases chacune de leurs séquences consensus respectifs (TATAAT et TTGACA respectivement).
Contrairement à d'autres procédés tels que la 5 'RACE, des réactions d'extension d'amorces peuvent être utilisés pour déterminer et quantifier le clivage de molécules d'ARN avec précision. Le clivage des molécules d'ARN libre crée des extrémités 5 ', qui peuvent être détectés en même temps que des bandes d'ADNc dans le gel. L'identification de plusieurs produits de clivage de l'ARNm de l'un est plus difficile dans les expériences 5 'RACE, étant donné que plusieurs produits de PCR doivent être séquences pour obtenir des produits de clivagequi ne représentent qu'une petite fraction du total (non traité) ARN vrac.
Dans la partie inférieure de la figure 6, l'application de l'ARN de la coupure par une RNase est représenté. 5 Comme décrit précédemment, la RNase-YoeB seq2, qui fait partie d'un système de TA S. equorum 17, clive les ARNm à proximité du codon de départ. Ce clivage peut être inhibée par l'anti-toxine YefM-seq2 apparenté. Lorsque la RNase YoeB est absente ou inactive (voies 1 - 3), pas de produits d'extension d'amorce sont formés à proximité du codon d'initiation. Chaque fois que l'activité RNase est présent (pistes 4 et 5), de deux bandes intenses, peu en aval du codon d'initiation apparaissent. En utilisant cette approche, les modèles de clivage peuvent être facilement identifiés.
La figure 7 montre une expérience d'extension d'amorce a échoué. En raison de l'ARN en excès dans la réaction de transcription inverse, la quantité d'ADNc produite crée un signal fort, cette bande d'ADNc individuels ne peut pas être distingués. Cela rend l'ARN détermination d'extrémité 5 'impossible.
Lors de l'utilisation des ARN hautement exprimés dans la réaction d'extension d'amorce, par exemple un ARN ribosomique (comme représenté sur la figure 7), le risque de zones surexposées augmente. Par conséquent, la quantité de matrice d'ARN total doit être ajusté à l'abondance de l'ARN d'intérêt. En revanche, lorsque les transcriptions d'intérêt constituent seulement une petite quantité de l'ARN total, le montant de la réaction de transcription inverse doit être augmenté, sinon les signaux seront trop faibles (non représenté). Cela vaut également pour la réaction de séquençage de Sanger, en tant que modèles à copies multiples par cellule (tels que les gènes d'ARNr ou des gènes codés sur des plasmides) conduisent à des signaux beaucoup plus forts.
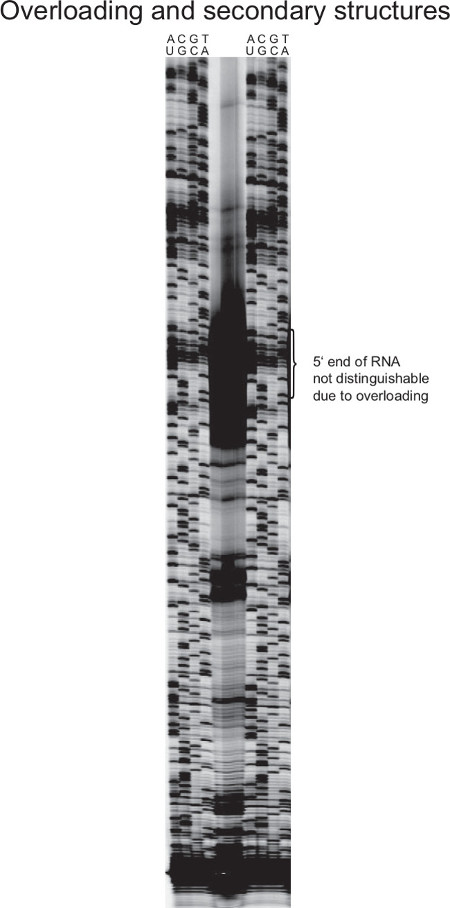
Figure 7. résultat Représentant d'une extension d'amorce manqué de ng> S. aureus. ARN 16S est très abondant dans les cellules bactériennes. Cela conduit à des signaux forts de transcription inverse quand des quantités modérées d'ARN total sont utilisés. Dans le cas représenté ici, les bandes d'ADNc fortes masquent l'extrémité 5 'et par conséquent empêchent 5' exacte correspondance. En outre, les ARN ribosomique sont très structurés et peuvent donc résilier prématurément la transcription inverse, la production de courts produits d'ADNc qui ne représentent pas des fragments de pleine longueur. Dans ce cas, l'augmentation de température de transcription inverse, avec une transcriptase inverse résistant à la chaleur peut rendre plus facile à synthétiser des structures secondaires dernières. En raison de multiples copies de la séquence dans l'ADN génomique, le séquençage de Sanger échelle est plus forte que pour des gènes à copie unique. Pour améliorer l'image de gel, l'ARN et l'ADN des montants pour la transcription inverse et réaction Sanger devraient être réduites et / ou moins de produit appliqué sur le gel._upload / 52134 / 52134fig7large.jpg "target =" _ blank "> Se il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Tableau 1: DNase I digestion de l'ARN.
| Substance | Montant |
| L'ARN provenant de l'étape ci-dessus | 70-100 pg |
| DNase I Buffer 10x (100 mM de Tris, pH 7,5, 25 mM de MgCl2, 5 mM de CaCl2) | 50 ul |
| DNase I, RNase (2 U / pl) | 10,0 pi (jusqu'à 10 pg d'ARN par une pi de DNase I) |
| Amener le volume à 500 pi avec le trou DDH 2 O (traitée au DEPC) | |
volume de réaction et la quantité de DNase I peuvent être agrandies ou réduites en fonction de la quantité of ARN utilisé.
Tableau 2: ARN-hybridation de l'amorce.
| Composé | Montant pour une réaction |
| ARN | 5-15 pg |
| Amorce marquée par fluorescence | 2 pmol |
| Amener le volume de 6 pi avec le trou DDH 2 O (traitée au DEPC) | |
Mettre en place une réaction par échantillon d'ARN et de l'amorce.
Tableau 3: extension d'amorce maître mix.
| Composé | Montant pour une réaction |
| ddH 2 O (DEPC traitée) | 1,3 pl |
| RT Buffer AMV (de 10x) | 1.0pl |
| dNTP (10 mM, RNase) | 1,0 ul |
| RNase Inhibitor | 0,2 pl |
| AMV transcriptase inverse (RT) (20-25 U / pl) | 0,5 ul |
Élargir le nombre de réactions nécessaires.
Tableau 4: 10x TBE recette.
| Substance | Montant |
| Base Tris | 107,8 g |
| L'acide borique | 55,0 g |
| EDTA | 7,4 g |
| Amener le volume à 1000 ml avec ultrapure ddH 2 O et le filtre pour enlever la poussière et les peluches | |
Filtrer pour retirer la poussière et les peluches et conserver à 4 °C.
Tableau 5: gel de séquençage recette.
| Composé | Montant |
| Urée | 10,5 g |
| ddH 2 O (MilliQ) | 13,0 ml |
| 10x TBE | 2,5 ml |
| Solution de gel XL rapide | 5,0 ml |
| TEMED (N, N, N ', N'-Tetramethylethane-1,2-diamine) | 25 ul |
| APS (persulfate d'ammonium, 10%) | 175 pi |
solution de gel de traiter rapidement après l'addition de TEMED et APS.
Discussion
Extension d'amorce fluorescente est une méthode simple et rapide pour déterminer les extrémités 5 'des ARN, soit pour TSP ou l'identification de la transformation secondaire de l'ARN. Grâce à l'utilisation d'amorces fluorescentes, les réactions peuvent être mises en place et fonctionnent sans précautions de sécurité supplémentaires (contrairement au cas des amorces marquées de manière radioactive). Étant donné que les échantillons sont détectés par fluorescence, ils peuvent être visualisés pendant l'électrophorèse est en cours qui permet une analyse rapide en comparaison avec les méthodes radioactives, où des films radiographiques sont généralement utilisés.
En général, la qualité de la réaction d'extension d'amorce est fortement dépendante de la capacité de liaison de l'amorce et de qualité. Si le site de liaison est choisi trop près de la zone d'intérêt, les frottis de primaire peuvent masquer le signal, alors que le site de liaison trop loin (> 300 pb) de l'extrémité 5 'peut entraîner un mauvais signal.
Le colorant fluorescent doit être liée de manière covalente à laL'extrémité 5 'de l'oligonucléotide d'ADN de synthèse et personnalisée au cours de l'oligonucléotide doit être purifié par HPLC pour éviter les interférences avec la réaction de transcription inverse par les sels résiduels. Les oligonucléotides avec modification de colorant approprié (voir tableau de la liste des réactifs pour plus d'informations sur les colorants compatibles) peuvent être commandées à partir de la plupart des entreprises de la synthèse d'oligonucléotides et doivent être stockés dans l'obscurité. Malheureusement, nous ne sommes pas conscients de toutes les techniques enzymatiques pour fixer le colorant à oligonucléotides déjà existants, comme cela est possible avec des nucléotides radiomarqués.
Dans le système de séquençage décrit ici, deux colorants fluorescents différents peuvent être utilisés pour détecter simultanément deux échantillons, en tant que leur excitation (environ 700 et 800 nm) et les spectres d'émission sont distinctes. Outre les colorants du fabricant d'origine, d'autres colorants tels qu'indiqués dans la liste des réactifs peuvent être utilisés pour produire d'excellents résultats.
Un autre facteur important pour prdes réactions d'extension imer est la qualité et de la quantité d'ARN. Des précautions doivent être prises pour éliminer les contaminants ADN, comme transcriptases inverses telles que l'AMV RT peuvent utiliser l'ADN comme un modèle 18.
Comme le montre la Figure 7, la force du signal détecté de bandes de la piste de gel dépend de la quantité d'ARN utilisée dans la réaction de transcription inverse. Il est donc essentiel de régler la quantité d'ARN total, en fonction de la quantité proportionnelle de l'ARN d'intérêt présente dans l'échantillon. La faible sensibilité de cette méthode est aussi l'un de ses inconvénients, comme les ARN faibles exprimées peuvent être difficiles à détecter. Si aucun signal ne peut être détecté du tout, la quantité de l'ARN total peut être augmentée ou l'ARN d'intérêt peut être surexprimé artificiellement à partir d'un plasmide.
sensibilité de détection de gel pour la fluorescence en fonction extension d'amorce est d'environ dix facteur inférieur à 32 P ou 33 P radio-isotope basé extension d'amorce 19. Cependant, cet inconvénient peut être compensé par ajustement de la quantité d'ADNc chargée sur le gel, soit en augmentant la quantité de matrice d'ARN utilisé dans la réaction de transcription inverse. Dans la plupart des cas, la sensibilité est suffisamment élevée pour résultats satisfaisants 4,5. Les sensibilités similaires aux extensions d'amorces radioactives ont été rapportées pour l'utilisation des amorces fluorescentes, en combinaison avec un séquenceur capillaire 3, avec l'avantage de temps d'exposition courts.
Coûts des comparaisons entre la fluorescence et de la radioactivité sur la base des extensions d'amorces sont difficiles, car ils dépendent de plusieurs facteurs tels que les machines disponibles, les amorces, les réactions de l'échelle, de la disponibilité et de l'entretien d'un laboratoire pour le travail avec des substances radioactives, l'élimination des déchets radioactifs, la formation et les risques sanitaires individuels . Amorces fluorescentes sont cinq à dix fois plus cher que les amorces non standard marqué (20 pb). Cependant, ces amorces peuvent être utilisées pour au moinsun an plus probables (plusieurs années) par rapport à P 32 amorces marquées, qui ont une demi-vie beaucoup plus courte. Re-étiquetage des amorces est long et coûteux, en raison de la nécessité d'une nouvelle matière radioactive dans des intervalles fréquents. Si le même jeu d'amorces est utilisée tout au long d'une longue période de temps, des amorces fluorescentes sont moins chers que les amorces réguliers, marquées radioactivement. Le point de coût principal sera toutefois le séquenceur fluorescent ou imageur et l'achat de cet appareil uniquement dans le but d'extensions d'amorces pourrait ne pas être rentable. La méthode décrite ici est plutôt intéressant pour les groupes qui sont en la possession ou avoir accès à une telle machine.
Si un séquenceur automatisé de gel ne sont pas disponibles, d'autres procédés de détection par fluorescence peuvent être utilisés aussi bien. Dans ce cas, le gel peut être exécuté dans un appareil d'électrophorèse standard, séché si nécessaire, et ensuite transféré à un système d'imagerie fluorescente (modèles sont disponibles, qui sont semblables à un flscanners atbed). Bien que l'avantage de la visualisation du gel au cours de la course est perdu, l'utilisation d'isotopes radioactifs peut être évitée de cette façon, un avantage important pour l'expérimentateur. En outre, le cas échéant, un séquenceur capillaire peut être utilisée pour la séparation et la détection en temps réel, ce qui peut aider à augmenter la sensibilité.
Diverses méthodes ont été publiés sur la façon d'utiliser l'extension d'amorce fluorescente avec des machines de séquençage automatisé de gel ou séquenceurs capillaires, cependant, ces méthodes nécessitent souvent une étape ADNc de précipitation pour concentrer l'échantillon (et éliminer les impuretés) 19. Dans la méthode présentée ici, la précipitation de l'ADN ne sont pas nécessaires ce qui réduit le temps de préparation. Plus important cependant, omettre cette étape, il est possible de déterminer semi-quantitativement la quantité de molécules d'ARN dans l'échantillon que les incohérences de la procédure de précipitation peuvent être éliminés.
Il convient de noter que, bien queles extrémités 5 'des molécules d'ARN peuvent être mappés à des réactions d'extension d'amorce, traitées et extrémités primaires (points de départ de la transcription) ne peuvent pas être facilement distingués. Pour contourner ces restrictions, la planification minutieuse des expériences avec des contrôles appropriés est essentiel. Séquences promotrices évidents peuvent indiquer la présence d'un point de départ de la transcription et de mutation ou de supprimer des éléments promoteurs doivent alors abolir bandes d'ADNc. D'autre part, si de telles séquences sont manquantes ou des séquences consensus spécifiques sont présentes, les bandes peuvent être provoqués par traitement de l'ARN. Si l'enzyme de traitement est connu et peut être purifié, in vitro extensions d'amorces peuvent apporter des précisions, comme la transcription et de traitement peuvent être séparés. En outre, d'autres méthodes telles que RACE 5 '(y compris l'enrichissement enzymatique de molécules d'ARN non transformés) peut compléter extensions d'amorces pour distinguer les sites d'initiation de la transcription de traitement de l'ARN.
The méthode d'extension d'amorce est souvent comparé à d'autres méthodes telles que RACE 5 'et S1 essais de protection de nucléase et donc son utilité est parfois contestée. techniques de RNAseq en combinaison avec le séquençage de prochaine génération par exemple, peuvent aider à déterminer FST et les sites de traitement d'un grand nombre d'ARN en parallèle, mais le fardeau financier et bioinformatiques travail requis, il plutôt rentable pour ARN simple d'intérêt. 5'-RACE en revanche est moins cher et les résultats sont plus faciles à analyser, mais si les ARN sont traitées de plusieurs façons ou plusieurs points de départ de la transcription sont présents, le produit doit être cloné et une grande quantité de candidats doivent être séquencé à obtenir une vue représentative des ARN d'intérêt.
Par conséquent, en dépit de nouvelles méthodes ayant surgi au fil des ans, même aujourd'hui amorce extensions ont leur raison d'être en raison de la facilité d'utilisation, faible coût et peu de temps d'exécution, ce qui est particulièrement vraipour la méthode de fluorescence à base présentée ici.
Déclarations de divulgation
The authors have nothing to disclose that would present a conflict of interest.
Remerciements
We thank Anne Wochele for her assistance in the laboratory and Vera Augsburger for help with the automated gel sequencer. We thank the Deutsche Forschungsgemeinschaft for funding by grants BE4038/2 and BE4038/5 within the “priority programmes” SPP1316 and SPP1617.
matériels
| Name | Company | Catalog Number | Comments |
| AMV Reverse Transcriptase (20-25 U/µl) | NEB / Roche | NEB: M0277-T / Roche: 10109118001 | |
| DNase I (RNase free) | Ambion (life technologies) | AM2222 | |
| FastPrep-24 Instrument | MPBio | 116004500 | |
| Fluorescently labeled primers | Biomers | n/a | 5’ DY-681 modification of “ordinary” DNA oligonucleotides. Compatible dyes such as the LICOR IRDye 700/800 are also available from other suppliers such as IDTdna. |
| Li-Cor 4200 Sequencer incl. ImagIR Data collection software | Li-Cor | Product discontinued | |
| NanoDrop 2000 | Thermo Scientific | ||
| Nuclease free water | Ambion (life technologies) | AM9915G | |
| Plasmid mini preparation kit | QIAGEN | 12125 | |
| RapidGel-XL-40% Concentrate | USB | US75863 | |
| RNA STORAGE BUFFER | Ambion (life technologies) | AM7000 | |
| Roti-Aqua-P/C/I | Carl Roth | X985.3 | Alternative: “Acid-Phenol:Chloroform, pH 4.5 (with IAA, 125:24:1)” from Ambion (AM9720) |
| SUPERase•In RNase Inhibitor | Ambion (life technologies) | AM2696 | |
| Thermo Sequenase fluorescently labelled primer cycle sequencing kit with 7-deaza-dGTP | GE Healthcare | RPN2538 | |
| TRIzol reagent | life technologies | 15596-026 | |
| Zirconia/Silica Beads 0.1 mm | BioSpec | 11079101z |
Références
- Simpson, C. G., Brown, J. W. Primer extension assay. Methods Mol. Biol. 49, 249-256 (1995).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proc. Natl. Acad. Sci. U. S. A. 74, 5463-5467 (1977).
- Fekete, R. A., Miller, M. J., Chattoraj, D. K. Fluorescently labeled oligonucleotide extension: a rapid and quantitative protocol for primer extension. Biotechniques. 35, 90-94 (2003).
- Schuster, C. F., et al. Characterization of a mazEF Toxin-Antitoxin Homologue from Staphylococcus equorum. J. Bacteriol. 195, 115-125 (2013).
- Nolle, N., Schuster, C. F., Bertram, R. Two paralogous yefM-yoeB loci from Staphylococcus equorum encode functional toxin-antitoxin systems. Microbiology. 159, 1575-1585 (2013).
- Yamaguchi, Y., Park, J. H., Inouye, M. Toxin-antitoxin systems in bacteria and archaea. Annu. Rev. Genet. 45, 61-79 (2011).
- Schuster, C. F., Bertram, R. Toxin-antitoxin systems are ubiquitous and versatile modulators of prokaryotic cell fate. FEMS Microbiol. Lett. 340, 73-85 (2013).
- Goeders, N., Van Melderen, L. Toxin-antitoxin systems as multilevel interaction systems. Toxins. 6, 304-324 (2014).
- Yamaguchi, Y., Inouye, M. mRNA interferases, sequence-specific endoribonucleases from the toxin-antitoxin systems. Progress in molecular biology and translational science. 85, 467-500 (2009).
- Park, J. H., Yamaguchi, Y., Inouye, M. Bacillus subtilis MazF-bs (EndoA) is a UACAU-specific mRNA interferase. FEBS Lett. 585, 2526-2532 (2011).
- Zhu, L., et al. et al.Staphylococcus aureus MazF specifically cleaves a pentad sequence, UACAU, which is unusually abundant in the mRNA for pathogenic adhesive factor SraP. J. Bacteriol. 191, 3248-3255 (2009).
- Zhu, L., et al. The mRNA interferases, MazF-mt3 and MazF-mt7 from Mycobacterium tuberculosis target unique pentad sequences in single-stranded RNA. Mol. Microbiol. 69, 559-569 (2008).
- Fu, Z., Donegan, N. P., Memmi, G., Cheung, A. L. Characterization of MazFSa, an endoribonuclease from Staphylococcus aureus. J. Bacteriol. 189, 8871-8879 (2007).
- Marmur, J. A procedure for the isolation of deoxyribonucleic acid from micro-organisms. J. Mol. Biol. 3, 208-218 (1961).
- Emory, S. A., Belasco, J. G. The ompA 5' untranslated RNA segment functions in Escherichia coli as a growth-rate-regulated mRNA stabilizer whose activity is unrelated to translational efficiency. J. Bacteriol. 172, 4472-4481 (1990).
- Cole, S. T., Bremer, E., Hindennach, I., Henning, U. Characterisation of the promoters for the ompA gene which encodes a major outer membrane protein of Escherichia coli. Mol. Gen. Genet. 188, 472-479 (1982).
- Schleifer, K. H., Kilpper-Bälz, R., Devriese, L. Staphylococcus arlettae sp. nov., S. equorum sp. nov. and S. kloosii sp. nov.: Three New Coagulase-Negative, Novobiocin-Resistant Species from Animals. Syst. Appl. Microbiol. 5, 501-509 Forthcoming.
- Yu, H., Goodman, M. F. Comparison of HIV-1 and avian myeloblastosis virus reverse transcriptase fidelity on RNA and DNA templates. J. Biol. Chem. 267, 10888-10896 (1992).
- Ying, B. W., Fourmy, D., Yoshizawa, S. Substitution of the use of radioactivity by fluorescence for biochemical studies of RNA. RNA. 13, 2042-2050 (2007).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon