Method Article
Скрининг сперматозоидов для быстрого выделения изменений зародышевой линии у рыбок данио-рерио
В этой статье
Резюме
Технологии CRISPR-Cas произвели революцию в области редактирования генома. Тем не менее, поиск и изоляция желаемого редактирования зародышевой линии остается основным узким местом. Таким образом, в этом протоколе описывается надежный метод быстрого скрининга сперматозоидов рыбок данио, вводимых с помощью F0 CRISPR, на предмет редактирования зародышевой линии с использованием стандартных методов ПЦР, рестрикционного дайджеста и гель-электрофореза.
Аннотация
Появление целевых нуклеазных технологий CRISPR-Cas произвело революцию в способности выполнять точное редактирование генома как в устоявшихся, так и в новых модельных системах. Системы редактирования генома CRISPR-Cas используют синтетическую направляющую РНК (sgRNA) для нацеливания CRISPR-ассоциированной (Cas) эндонуклеазы на конкретные локусы геномной ДНК, где эндонуклеаза Cas генерирует двухцепочечный разрыв. Восстановление двухцепочечных разрывов внутренними механизмами, подверженными ошибкам, приводит к вставкам и/или делециям, нарушая локус. В качестве альтернативы, включение двухцепочечных доноров ДНК или одноцепочечных олигонуклеотидов ДНК в этот процесс может вызвать включение точных изменений генома, начиная от однонуклеотидных полиморфизмов и заканчивая небольшими иммунологическими метками или даже большими флуоресцентными белковыми конструкциями. Однако основным узким местом в этой процедуре может быть поиск и изоляция нужного редактирования в зародышевой линии. В этом протоколе изложен надежный метод скрининга и выделения мутаций зародышевой линии в определенных локусах у Danio rerio (рыбки данио); Тем не менее, эти принципы могут быть адаптированы в любой модели, где возможен сбор спермы in vivo .
Введение
Система CRISPR (Clustered Regular Interspaced Short Palindromic Repeats)/Cas является мощным инструментом для выполнения локус-специфического мутагенеза и точного редактирования генома в модельной системе Danio rerio (рыбки данио) 1,2,3,4. Cas-рибонуклеопротеин (RNP) состоит из двух основных компонентов: эндонуклеазы Cas (обычно Cas9 или Cas12a) и локус-специфической синтетической направляющей РНК (sgRNA)5. Вместе Cas-RNP генерирует двухцепочечный разрыв (DSB) в желаемом локусе, который может быть восстановлен одним из двух внутренних механизмов репарации. Механизм репарации негомологичного соединения концов (NHEJ) подвержен ошибкам и часто приводит к различным вставкам или делециям (инделам) вокруг DSB. Эти инделы могут быть вредными, если они вносят мутацию со сдвигом рамки или преждевременную остановку в результирующей белковой последовательности. В качестве альтернативы, механизм гомологической репарации (HDR) использует донорский шаблон с областями гомологии, окружающими сайт DSB, для восстановления повреждения. Исследователи могут воспользоваться преимуществами системы HDR для создания точных геномных правок. В частности, они могут совместно вводить двухцепочечную донорскую конструкцию ДНК, которая содержит желаемые изменения, а также области гомологии, фланкирующие сайт DSB в геноме. Возросшая экономия за счет масштаба этих коммерчески производимых компонентов CRISPR значительно снизила барьеры для скрининга нескольких локусов и создания более масштабных усилий для точного редактирования генома. Однако в моделях животных, размножающихся половым путем, основным узким местом является идентификация и изоляция устойчивых к зародышевой линии мутантных животных.
Модельная система рыбок данио-рерио демонстрирует несколько ключевых качеств, которые расширяют ее использование в обратных генетических исследованиях. Их легко выращивать в больших количествах с помощью основного оборудования для содержания в воде, а самки демонстрируют высокую плодовитость круглый год6. Более того, их внешняя яйцекладка и оплодотворение делают их поддающимися микроинъекциям компонентов CRISPR/Cas. Cas-RNP обычно вводят эмбрионам рыбок данио-рерио на одной клеточной стадии для генерации DSB / репарации, которая теоретически наследуется всеми дочерними клетками. Однако диплоидные геномы требуют двух событий DSB/репарации для мутагенизации обеих гомологичных хромосом. Кроме того, несмотря на то, что Cas-RNP вводится на стадии одной клетки, DSB / репарация может не произойти до более поздних точек развития. Вместе эти факторы способствуют мозаичной природе рыбы, введенной в F0. Распространенной практикой является скрещивание рыбы, введенной в F0, и проверка потомства F1 на наличие инделов / специфических правок. Однако, поскольку не все рыбы, введенные в F0, обладают мутациями зародышевой линии, эта практика приводит к множеству непродуктивных скрещиваний, которые не приводят к желаемому редактированию. Скрининг зародышевой линии F0, а не соматической ткани F1 увеличивает вероятность выделения желаемого редактирования зародышевой линии и уменьшает количество животных, необходимых в этом процессе.
Сперма может быть легко собрана у рыбок данио-рерио с инъекцией F0 без необходимости эвтаназии. Эта функция позволяет криоконсервировать и извлекать замороженные запасы спермы7, но также может быть использована для быстрого скрининга, идентификации и изоляции носителей зародышевой линии желаемых геномных мутаций 8,9. Brocal et al. (2016) ранее описали основанный на секвенировании метод скрининга изменений зародышевой линии у самцов рыбокданио-рерио 10, введенных в F0. Несмотря на то, что этот подход полезен для идентификации мутировавших аллелей, присутствующих в зародышевой линии, он может стать дорогостоящим при высокой пропускной способности и может быть доступен не для всех лабораторий. В отличие от этого, текущий протокол предлагает доступную и экономически эффективную стратегию на основе электрофореза для выявления правок зародышевой линии. В частности, в этом протоколе описывается надежный метод скрининга и выделения мутаций зародышевой линии в определенных локусах с использованием электрофореза в агарозном геле высокого разрешения. Кроме того, в этом протоколе описывается аналогичная стратегия выявления успешной интеграции донорской конструкции, содержащей конкретные изменения. Как всегда, если требуются конкретные изменения, стратегии на основе секвенирования могут быть выполнены в тандеме с протоколом, описанным ниже. Хотя этот протокол специфичен для модельной системы рыбок данио, эти принципы должны быть адаптированы к любой модели, в которой сбор спермы является рутинной процедурой. Вместе эти стратегии позволят идентифицировать мужчин, введенных в F0, с зародышевыми инделами/правками, которые могут быть разрешены на геле после стандартной полимеразной цепной реакции (ПЦР) и/или рестрикционного дайджеста.
протокол
Это исследование было проведено в соответствии с руководящими принципами, изложенными в Руководстве по уходу за лабораторными животными и их использованию Национальных институтов здравоохранения. Протокол был одобрен Комитетом по уходу за животными и их использованию Техасского университета в Остине (AUP-2021-00254).
1. Проектирование сгРНК для мутагенеза CRISPR
- Получите последовательность экзонов, содержащую целевые локусы.
- Разработка синтетической направляющей РНК (сгРНК) с соседним сайтом протоспейсерного мотива (PAM), специфичным для эндонуклеазы Cas.
- Если вы вводите донорскую конструкцию, спроектируйте сгРНК так, чтобы прогнозируемый участок разреза был как можно ближе к желаемым изменениям.
ПРИМЕЧАНИЕ: APE — это бесплатное программное обеспечение с инструментом для поиска сайтов сгРНКCas9 11. В качестве альтернативы можно использовать такие инструменты, как CHOPCHOP, который предлагает онлайн-интерфейс для проектирования сгРНК, который учитывает различные сайты PAM Cas9 и других эндонуклеаз Cas12.
- Если вы вводите донорскую конструкцию, спроектируйте сгРНК так, чтобы прогнозируемый участок разреза был как можно ближе к желаемым изменениям.
- Разработайте прямые и обратные праймеры, чтобы усилить область, окружающую спроектированный участок разреза CRISPR/Cas. Продукт ПЦР должен иметь длину примерно 200-400 пар оснований (.н.), чтобы иметь возможность разрешать небольшие инделы на геле позже в протоколе.
ПРИМЕЧАНИЕ: CHOPCHOP11 автоматически разрабатывает праймеры для каждой выводимой сгРНК. - Необязательно: Разработайте конструкцию донора ДНК с желаемыми изменениями.
- Получают геномную последовательность экзона, содержащую локусы нужной мутации.
ПРИМЕЧАНИЕ: Интронные последовательности могут варьироваться у разных людей, поэтому лучше избегать включения интронных последовательностей в донорскую конструкцию, так как это может помешать гомологически зависимому пути репарации. - Если требуется конкретное изменение аминокислоты, измените соответствующий кодон.
ПРИМЕЧАНИЕ: При внесении этих изменений помните о частоте кодонов. Если возможно, выбирайте кодон с интенсивным использованием. - Чтобы предотвратить переваривание CRISPR/Cas донорской конструкции, сделайте дополнительные синонимичные мутации, которые (i) нарушают PAM и/или (ii) нарушают последовательность sgRNA, при этом предпочтение отдается внесению несинонимичных изменений ближе к сайту PAM.
- Используйте средство поиска сайтов рестрикции (APE11 также имеет эту функцию), чтобы найти уникальные сайты рестрикции (те, которые встречаются только один раз) в предсказанном продукте ПЦР последовательности дикого типа.
- Повторите этот процесс для прогнозируемого продукта ПЦР, который отражает успешную интеграцию донорской конструкции.
- Сравните список сайтов рестрикции между последовательностями дикого типа и последовательностями, интегрированными с донором, чтобы найти сайт рестрикции, который присутствует только в отредактированной последовательности.
- Если уникальный сайт рестрикции не найден, продолжайте вносить синонимичные изменения в донорской конструкции до тех пор, пока это не будет достигнуто.
ПРИМЕЧАНИЕ: Если возможно, продолжайте делать синонимичные мутации, которые нарушат связывание sgРНК с донорской конструкцией.
- Обрежьте донорскую последовательность до приемлемого размера (50-100.н.) с гомологией не менее 20.н., окружающей прогнозируемый сайт разреза CRISPR/Cas, и отредактируйте, если это возможно.
- Получают геномную последовательность экзона, содержащую локусы нужной мутации.
- Получить разработанные олигонуклеотиды сгРНК и/или донора ДНК, а также любые праймеры, необходимые для генотипирования.
ПРИМЕЧАНИЕ: Чтобы предотвратить расщепление олигонуклеотидов донора ДНК нуклеазами, рекомендуется модифицировать первые три фосфатные связи на 5'- и 3'-концах олигонуклеотидов ДНК фосфоротиоатом. - Введите 1 нл инъекционной смеси эмбрионам на одноклеточной стадии. Как правило, стандартная инъекционная смесь содержит следующее:
1 мкл 25 мкМ локус-специфической сгРНК
1 мкл 25 мкМ эндонуклеазы Cas9
Дополнительно: 1 мкл донорской конструкции ДНК 3 мкМ- Доведите общий объем до 5 мкл с 0,1 М РНКазы KCl.
- Позвольте индивидуумам, получившим инъекцию F0, развиться в половозрелых взрослых. Оставшийся протокол фокусируется на скрининге мужчин, которым вводили F0, на предмет редактирования зародышевой линии.
2. Установка резервуаров для разведения
- Подготовьте самцов F0 к спариванию, установив резервуары для разведения с разделителями между самцами и самками F0.
- Чтобы свести к минимуму количество необходимых резервуаров для размножения, установите трех самцов напротив двух самок в каждом аквариуме.
- Настройте как можно больше мужчин F0, которые могут быть эффективно проверены и размещены как отдельные лица после сбора спермы. Генотип самок не важен, так как их гаметы не будут собраны во время этой процедуры.
- Установите один аквариум для размножения самцов дикого типа напротив самок. Сперматозоиды дикого типа будут служить контролем для интерпретации результатов позже в процедуре.
- Чтобы свести к минимуму количество необходимых резервуаров для размножения, установите трех самцов напротив двух самок в каждом аквариуме.
- Подготовленные емкости для разведения инкубируйте в течение ночи.
3. Подготовка материалов для процедуры забора спермы
- Приготовьте достаточное количество обезболивающего раствора (0,016% трикаина-HCl в рыбьей воде), чтобы заполнить небольшую стеклянную миску.
- Распределите 40 мкл 50 мМ NaOH в пробирки для ПЦР объемом 0,2 мл. Подготовьте одну пробирку для каждого образца спермы, который будет собран. Хранить на льду.
- Подготовьте индивидуальные резервуары объемом 0,8 л для каждой рыбы, которая будет отобрана во время процедуры. В этих резервуарах будут размещаться отобранные образцы рыбы до тех пор, пока генотипирование не будет завершено.
ПРИМЕЧАНИЕ: Это основной фактор, который ограничивает количество рыб, которые могут быть эффективно проверены в этом протоколе. Обеспечьте достаточное пространство для размещения отобранных особей до завершения генотипирования. - Смочите губку размером 1 x 1 дюйм (разрежьте неглубоким выступом овальной формы, чтобы удерживать самца рыбки данио-рерио брюшной стороной вверх во время процедуры) в рыбьей воде и отожмите лишнюю жидкость.
- Расположите губку в поле зрения стереомикроскопа при малом увеличении (рис. 1А).
- Расположите наклонное верхнее освещение для оптимального просмотра.
4. Обезболивание самца рыбы и сбор спермы
- Подготовьте чистую капиллярную трубку.
- Осторожно встряхните контейнер с трубкой, чтобы освободить одну капиллярную трубку.
- Поместите капиллярную трубку в прилагаемую колбу и отложите ее в сторону.
- Переведите самца рыбы в приготовленный обезболивающий раствор (0,016% трикаина-HCl в рыбьей воде).
- Аккуратно размешайте раствор шумовкой, чтобы ускорить процесс обезболивания.
- Как только движение оперкулума (жабр) замедлится (примерно на 1-2 мин), приступайте к сбору спермы.
- Шумовкой переложите рыбу на чистую стопку бумажных полотенец.
- Шумовкой аккуратно скатайте рыбу, чтобы удалить лишнюю воду.
- Аккуратно промокните рыбу чистой сложенной папиросной бумагой, чтобы удалить остатки воды.
- Шумовкой переложите рыбу на подготовленную губку (рис. 1Б).
- Расположите рыбу брюшной стороной вверх так, чтобы ее голова была ближе всего к доминирующей руке человека, выполняющего процедуру.
- Аккуратно промокните область вокруг анальных плавников чистой сложенной папиросной бумагой.
- Соберите сперму.
- Используя конец капиллярной трубки, осторожно переместите брюшные плавники вбок от средней линии, чтобы обнажить клоаку (рис. 1C).
- Расположите конец капиллярной трубки возле клоаки.
- С помощью щипцов осторожно сожмите бока рыбы, начиная чуть ниже жабр и заканчивая клоакой (рис. 1C').
- Соберите сперму в капиллярную трубку капиллярным действием. Для этой процедуры достаточно примерно 1 мкл (рис. 1D).
ПРИМЕЧАНИЕ: Некоторые лаборатории сообщают об использовании наконечников пипеток p10 вместо капиллярных трубок для сбора спермы. - Если сперматозоиды не высвобождаются при мягком надавливании, верните рыбок в системную воду.
- Следите за восстановлением после анестезии.
- Не сжимайте рыбу сильно, чтобы изгнать сперму, так как это может травмировать рыбу.
- Дайте рыбе отдохнуть в течение 2 недель, прежде чем снова пытаться собрать сперму.
- После сбора поместите рыбок в чистый изолирующий резервуар с системной водой для индивидуального содержания.
- Следите за рыбой на предмет восстановления после анестезии.
- Дайте рыбе отдохнуть в течение 2 недель, прежде чем снова пытаться собрать сперму.
- Поместите капиллярную трубку с собранной спермой в подготовленную пробирку для ПЦР (с 40 мкл 50 мМ NaOH).
- Сожмите резиновую грушу, чтобы удалить собранный образец.
- Утилизируйте использованную капиллярную трубку в утвержденный стеклянный контейнер для отходов.
- Инкубируйте образцы на льду до тех пор, пока все самцы не будут выжаты.
- Повторите шаги 4.1-4.5 для каждого отдельного мужчины.
- Пометьте резервуары для изоляции и образцы спермы таким образом, чтобы окончательные результаты генотипирования сперматозоидов можно было проследить до соответствующего человека с предполагаемыми мутациями.
- Следите за всеми особями и убедитесь, что они находятся в вертикальном положении и исследуют свои резервуары, прежде чем снова включать резервуары в систему.
- Поместите самок, используемых для заправки, обратно в соответствующие резервуары в системе.
- Если какая-либо рыба не оправится от анестезии через 10 минут, следуйте процедуре эвтаназии, одобренной учреждением, связанным с лабораторией.
- Пример: Быстрое охлаждение человека на ледяной бане с рыбьей водой с температурой 2-4 °C.
- Утилизируйте усыпленную рыбу в соответствующий контейнер для отходов туш животных в соответствии с процедурой, утвержденной учреждением, связанным с лабораторией.
5. Извлечение ДНК из образцов спермы
- Ненадолго опустите все образцы спермы в миницентрифугу и поместите ПЦР-пробирки в термоциклер.
- Закройте крышку термоциклера.
- Выполните следующие настройки в термоциклере:
- Нагревайте образцы в течение 40 мин при температуре 95 °C.
- Охладите образцы до 25 °C.
- Извлеките образцы из термоциклера.
- С помощью чистого наконечника пипетки для каждого образца нейтрализуйте pH, добавив 10 мкл 1 М Tris-HCl (буферизованный до pH 8).
- Хорошо перемешайте, пипеткой вверх и вниз.
- Кратковременно раскрутите образцы в миницентрифуге.
- Образцы геномной ДНК могут храниться в течение нескольких дней при 4 ° C или помещаться при -20 ° C на срок до 6 месяцев.
6. ПЦР-амплификация (и/или рестрикционный дайджест) нужного локуса
- Амплифицируйте нужный локус с помощью стандартного протокола ПЦР.
- Предварительно нагрейте термоциклер до начальной температуры денатурации в реакции ПЦР, приведенной ниже.
- Приготовьте реакционную смесь по 25 мкл для каждого образца спермы в пробирках для ПЦР:
12,5 мкл 2x Taq Polymerase Master Mix
1,5 мкл прямого праймера 10 мкМ
1,5 мкл обратного праймера 10 мкМ
4,5 мкл безнуклеазной воды
5 мкл нейтрализованного образца ДНК сперматозоидов - Хорошо перемешайте, пипеткой вверх и вниз.
- Кратковременно раскрутите образцы в миницентрифуге.
- Поместите образцы в предварительно нагретый термоциклер со следующими настройками:
Начальная денатурация: 95 °C в течение 3 мин
35 циклов денатурации (95 °C в течение 30 с), отжига (55 °C в течение 30 с) и удлинения (72 °C в течение 30 с)
Окончательное продление: 72 °C в течение 5 минут
ПРИМЕЧАНИЕ: Может потребоваться регулировка температуры и/или времени расширения в зависимости от конкретной Taq-полимеразы и ожидаемой длины продукта амплификации ПЦР. - Извлеките образцы ПЦР из термоциклера.
- Дополнительно: Проведите специфический для донора рестрикционный дайджест на небольшом объеме продукта ПЦР, оставив не менее 5-10 мкл непереваренного продукта ПЦР для гель-электрофореза.
- Приготовьте реакцию 30 мкл в пробирках для ПЦР объемом 0,2 мл:
10 мкл неочищенного ПЦР-продукта
15 мкл безнуклеазной воды
3 мкл 10-кратного буфера рестрикционного фермента
2 мкл фермента рестрикции (BstNI используется в репрезентативных результатах для анализа аллеля ДНК10 )
ПРИМЕЧАНИЕ: Многие рестрикционные ферменты все еще активны в типичном реакционном буфере ПЦР; Тем не менее, добавление буфера дайджеста ограничений может помочь улучшить результаты. При возникновении каких-либо проблем обратитесь к конкретным инструкциям производителя для устранения неполадок. - Хорошо перемешайте, пипеткой вверх и вниз.
- Кратковременно раскрутите образцы в миницентрифуге.
- Поместите образцы в термоциклер со следующими настройками:
- Разрежьте ампликон ПЦР при 60 °C в течение 1 ч.
ПРИМЕЧАНИЕ: Температура среза и время инкубации, возможно, должны быть скорректированы для конкретного используемого фермента рестрикции. - Инактивируйте рестрикционный фермент необходимой температурой и временем инкубации, если это указано производителем.
ПРИМЕЧАНИЕ: Фермент BstNI не требует тепловой инактивации.
- Разрежьте ампликон ПЦР при 60 °C в течение 1 ч.
- Приготовьте реакцию 30 мкл в пробирках для ПЦР объемом 0,2 мл:
7. Проведение гель-электрофореза для разделения ПЦР-ампликонов разного размера
- Приготовьте 500 мл 0,5-кратного буфера КЭ: добавьте 50 мл 10-кратного буферного концентрата КЭ к 450 мл деионизированной воды.
- Приготовьте 100 мл 4% агарозного геля в 0,5-кратном рабочем буфере КЭ: добавьте 4 г агарозы с 10 мкл 10 000-кратного гелевого красителя к 100 мл 0,5-кратного рабочего буфера КЭ. Разогрейте в микроволновой печи до полного растворения порошка агарозы.
- Налейте гелевый раствор в рамку для литья геля соответствующего размера и вставьте расческу с одной стороны геля.
ПРИМЕЧАНИЕ: Больший размер геля (рекомендуется: 15 см x 15 см) выгоден для этой процедуры, так как он дает ампликонам больше места для разрешения на геле.- Если необходимо проанализировать рестрикционный дайджест-продукт, лучше всего запустить его на том же геле, что и продукт ПЦР, чтобы можно было сравнить результаты. Отрегулируйте размер лунки гребня по мере необходимости, чтобы все образцы поместились на геле.
- Оставьте гель при комнатной температуре до застывания. Аккуратно снимите расческу. Аккуратно извлеките гель из отливочной рамы.
- Налейте гелевый раствор в рамку для литья геля соответствующего размера и вставьте расческу с одной стороны геля.
- Установите установку для гель-электрофореза.
- Поместите гель в установку для электрофореза так, чтобы лунки были ближе всего к отрицательному электроду. Залейте 0,5x проточный буфер TBE в установку для электрофореза до тех пор, пока скважины не будут полностью погружены в воду.
- Загрузите гель.
- Загрузите 5 мкл ДНК-лестницы в первую лунку (выберите ДНК-лестницу, содержащую фрагменты ДНК, аналогичные по размеру ампликону ПЦР).
- Используя новый наконечник пипетки для каждого образца (включая контрольный образец дикого типа), загрузите 5-10 мкл продукта ПЦР в оставшиеся лунки.
- Дополнительно: Если необходимо проанализировать рестрикционный продукт, загрузите эти образцы также в гель.
ПРИМЕЧАНИЕ: Лучше всего загружать равное количество ДНК между ПЦР и продуктом рестрикционного дайджеста. Например, в рестрикционную реакционную смесь 30 мкл добавляли 10 мкл продукта ПЦР. Таким образом, концентрация ДНК в 15 мкл продукта переваривания была сопоставима с 5 мкл неочищенного продукта ПЦР.
- Запустите гель.
- Подключите электроды к установке для гель-электрофореза и подайте напряжение 150 В в течение не менее 1 ч, чтобы обеспечить адекватное разделение ампликонов.
- Изображение геля для просмотра полос ДНК.
ПРИМЕЧАНИЕ: Сильно мутагенизированные локусы, нацеленные на CRISPR, будут работать в виде нескольких размеров полос, представляющих комбинацию аллелей вставки/делеции с ампликонами дикого типа.- Если гелевые полосы не рассасываются через 1 ч, нанесите на гель 150 В с интервалом 15 мин, пока полосы на геле не будут достаточно разделены.
- Необязательно: При скрининге на вставку точного редактирования, такого как SNP, эпитопная метка или флуоресцентная метка, проведите скрининг ДНК сперматозоида с помощью методов ПЦР, специфичных для желаемого редактирования. Например, при идентификации донорского специфического сайта рестрикции в продукте рестрикционного дайджеста будет присутствовать дополнительная полоса по сравнению с продуктом ПЦР образца спермы.
8. Выделение стабильных аллелей зародышевой линии
- После 2 недель отдыха после процедуры выдавливания сперматозоидов пересеките людей F0, чья геномная ДНК сперматозоидов содержала желаемые изменения.
ПРИМЕЧАНИЕ: Генерация сперматозоидов из отдельных клонов сперматогонии может быть циклической у рыбок данио; Таким образом, желаемый аллель может не производить сперматозоиды во время скрещивания. По этой причине, возможно, придется выполнить дополнительное скрещивание более одного раза, чтобы выделить аллель, вовлеченный в первоначальный скрининг. - После того, как скрещенные рыбы F1 достигнут половозрелого возраста, приступайте к стандартным методам генотипирования (например, ДНК с плавниковым зажимом) с использованием того же протокола ПЦР и гель-электрофореза, который выполняется во время скрининга геномной ДНК сперматозоидов.
- Сэнгер секвенирует продукты ПЦР, чтобы идентифицировать людей F1 с таким же желаемым редактированием.
- Необязательно: если требуемое изменение вводит или нарушает работу сайта ограничений, отобразите соответствующий протокол дайджеста ограничений.
- Скрещивающие гетерозиготы F1 с одинаковой желаемой правкой.
- Проверьте потомство F2 на предмет ожидаемого фенотипа и/или геномных изменений.
Результаты
Экспериментальные подходы, описанные в этом протоколе, позволяют более быстро идентифицировать изменения генома или предполагаемые вредные аллели, сосредоточив внимание на анализе тысяч геномов, полученных из коллекции F0-инъекционных мужских сперматозоидов. На рисунке 2 показано, как интерпретировать результаты, полученные с помощью этого протокола.
Для генерации мутаций в локусе p2ry12 эмбрионам рыбок данио-рерио на одноклеточной стадии вводили эндонуклеазу Cas9 и p2ry12-специфическую сгРНК (рис. 2B, серый свет). Рыбы, введенные в F0, были помещены в систему до половой зрелости, а сперма была собрана у шести отдельных самцов. ДНК была извлечена из образцов сперматозоидов с использованием высокой температуры и основных условий (экстракция ДНК HotSHOT) и нейтрализована перед ПЦР-амплификацией локуса p2ry12. Продукты ПЦР разделяли на высокопроцентном (4%) агарозном геле в течение 1,5 ч при высоком напряжении (150 В). В этом примере ампликон дикого типа представлял собой одну яркую полосу длиной около 250 пар оснований (рис. 2C; WT). Напротив, инъецированные F0 мужские ампликоны, содержащие инделы, работали на геле в виде нескольких полос (рис. 2C; полосы #1-2, #5-8). В качестве альтернативы, мутировавшие F0-инъецированные мужские ампликоны могут работать как одна полоса с измененной подвижностью геля (не показано в примере p2ry12, но очевидно в продукте ПЦР ДНК10 F0-male #2; Рисунок 2Е). На примере геля p2ry12 было менее ясно, содержат ли образцы #3 и #4 инделы по сравнению с группой дикого типа, поэтому эти люди могут быть не лучшими кандидатами-основателями. Выделение аллелей от самцов F0 с более характерными изменениями ампликонов лучше всего подходит для размножения стабильных носителей зародышевой линии, поскольку они легко забиваются на 4% агарозном геле. Например, образец сперматозоидов F0-самца #6, по-видимому, содержал большую делецию в одном из аллелей (рис. 2C; полоса 6; яркая полоса с повышенной подвижностью геля). Если бы этот большой аллель делеции был выбран в поколении F1, его было бы легко отличить от аллеля WT на 4% агарозном геле. В качестве альтернативы, если желателен набор различных аллелей, F0-male #7 может быть эффективным кандидатом-основателем, поскольку его продукт ПЦР, по-видимому, содержит несколько дискретных аллелей различной подвижности. После того, как мужчина-основатель выбран, желаемый аллель может быть выделен путем скрещивания человека обратно с исходным штаммом дикого типа, используемым для инъекций.
Чтобы создать специфическую мутацию в гене DNAH10, эмбрионам рыбок данио-рерио на одноклеточной стадии вводили эндонуклеазу Cas9, сгРНК, специфическую для ДНК10 сгРНК (рис. 2D; выделение серым цветом) и олигонуклеотид донора ДНК, содержащий желаемое редактирование (рис. 2D; красный) и донор-специфический сайт рестрикции BstNI (рис. 2D , в скобках). Такая конструкция позволяет легко идентифицировать интеграцию донора с рестрикционным дайджестом после ПЦР. Кроме того, изменяя пары оснований в сайте распознавания сгРНК (рис. 2D; серый свет), эта конструкция предотвращает расщепление Cas9 донорской последовательности. После того, как рыбы, введенные в F0, достигли половой зрелости, сперма была собрана, и ДНК была извлечена с использованием метода горячего выстрела. Из этих образцов локус ДНК10 амплифицировали с помощью ПЦР, а продукты разделяли на высокопроцентном (4%) агарозном геле в течение 1 ч при высоком напряжении (150 В). В этом примере ампликон дикого типа представлял собой одну яркую полосу длиной около 400 пар оснований (рис. 2E; верхняя панель: WT). Напротив, инъецированные F0 мужские ампликоны, содержащие инделы, работали как несколько полос (рис. 2E; верхняя панель: полосы #1, #4 и #5) или как одна полоса с уменьшенной подвижностью геля (рис. 2E; верхняя панель: полоса #2). Чтобы определить, содержит ли какой-либо из образцов сперматозоидов интегрированные с донорами аллели, продукты ПЦР переваривали ферментом рестрикции BstNI в течение 1 ч, и продукт запускали на 4% агарозном геле в течение 1 ч при высоком напряжении (150 В). Сравнение непереваренного продукта ПЦР (рис. 2D; верхняя панель) с переваренным продуктом (рис. 2D; нижние панели) позволяет выявить образцы с вероятной интеграцией донорской конструкции. В этом примере мужчина #1, которому вводили F0, имел дополнительную полосу в переваренном продукте, которая отсутствовала в продукте ПЦР (рис. 2D; нижняя панель: полоса #1, обведена). Таким образом, этот самец представляет собой лучшего кандидата-основателя для создания мутантной линии с желаемым нокаутом.
После того, как предполагаемый носитель F0 самец скрещивается с аллелем, аллель должен быть проверен последовательностью в потомстве F1. Предлагается непосредственно секвенировать ампликоны, полученные из гетерозиготных рыб F1, с последующим выполнением методов анализа гетерозиготных аллелей, таких как Poly Peak Parser13, или с использованием различных методов, основанных на секвенировании следующего поколения, таких как секвенирование MiSeq 14 или Hi-Tom15. Это необходимый и взаимодополняющий подход, гарантирующий, что точное редактирование или вредная мутация индела действительно идет по зародышевой линии, а не является просто артефактом анализа гель-электрофореза. Действительно, использование подходов к секвенированию следующего поколения для секвенирования ДНК сперматозоидов из носителей F0 может быть легко использовано вместо анализа гель-электрофореза, описанного в этом протоколе. Тем не менее, наличие легко обжигаемого метода генотипирования в агарозном геле является более экономичным и эгалитарным подходом для мирового сообщества исследователей рыбок данио.

Рисунок 1: Настройка процедуры выжимания сперматозоидов . (A) Губка расположена под стереомикроскопом при малом увеличении с верхним освещением. (B) Смоченная губка размером 1 х 1 дюйм, нарезанная овальной дивотой (пунктирной линией), используется для удержания анестезированного самца рыбы брюшной стороной вверх во время процедуры. (C) Анатомия брюшной стороны анестезированного самца рыбы с изображением анальных плавников (af, розовый), брюшных плавников (pf, синий) и клоаки (стрелка), куда сперматозоиды будут выбрасываться во время процедуры. (С') Фильтрующие щипцы используются для осторожного выдавливания анестезированного самца рыбы из жабр в клоаку, в то время как изгнанная сперма втягивается в стеклянную пипетку капиллярным действием (белая стрелка). (D) Капиллярная трубка, содержащая достаточное количество вытесненных сперматозоидов (непрозрачная жидкость; черная скобка) для последующей экстракции и анализа ДНК. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
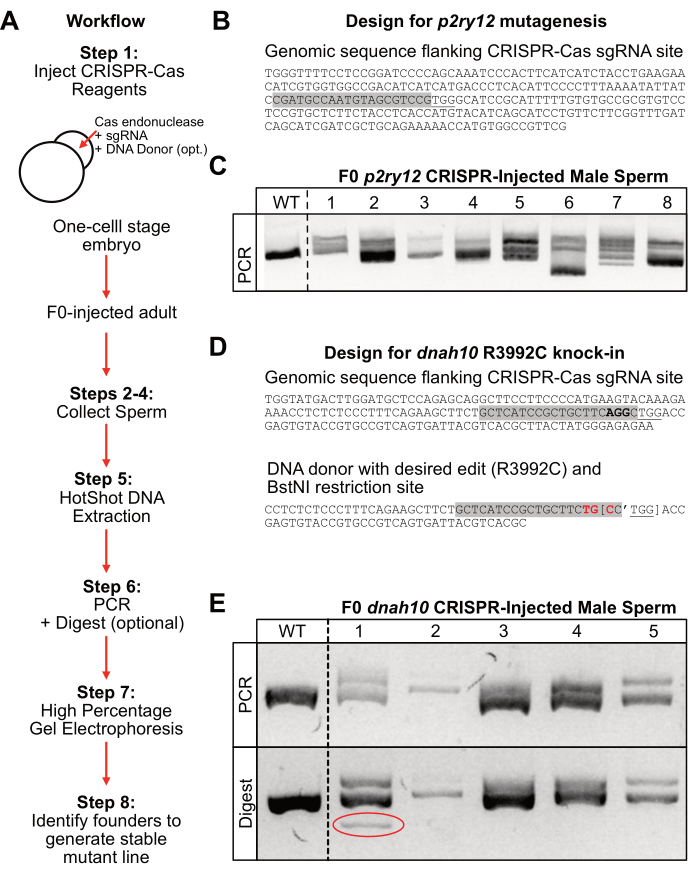
Рисунок 2: Рабочий процесс и репрезентативные результаты . А) Общий рабочий процесс протокола. (B) Репрезентативный дизайн сайта sgRNA p2ry12 (серый свет) с Cas9-специфическим сайтом PAM (подчеркнут). (C) Репрезентативные результаты 4% гель-электрофореза через 1,5 ч при 150 В. Контроль дикого типа (WT) и образцы мужской спермы F0-p2ry12 (1-8) после амплификации ПЦР. (D) Репрезентативный дизайн сайта sgRNA DNAH10 (серый свет) с Cas9-специфическим сайтом PAM (подчеркнут) рядом с целевым кодоном (выделено жирным шрифтом). Донорская последовательность ДНК с нужным редактированием кодона (красным жирным шрифтом) и рестрикционным сайтом BstNI (скобки с апострофом, обозначающим место разреза). (E) Репрезентативные результаты 4% гель-электрофореза через 1 ч при 150 В. Вверху: продукт ПЦР образцов мужской спермы дикого типа (WT) и F0 dnah10 , введенных CRISPR (1-5). Внизу: продукт рестрикционного дайджеста BstNI из приведенных выше образцов. Пример 1 демонстрирует успешную интеграцию донорской конструкции на основе дополнительной полосы после разложения (красный кружок). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Обсуждение
Этот протокол описывает метод быстрой характеристики предполагаемых изменений генома или целевых мутаций с использованием технологии CRISPR-Cas путем целенаправленного анализа мужских геномов сперматозоидов F0. Этот протокол должен быть совместим с другими моделями животных, где сперма легко доступна для отбора проб без эвтаназии. Этот метод увеличит пропускную способность скрининга на наличие желаемых правок и особенно полезен для выявления редких событий, опосредованных HDR. Этот подход также служит для сокращения числа экспериментальных животных, используемых для поиска стабильного редактирования зародышевой линии, облегчая быстрый скрининг потенциально тысяч геномов в одном образце спермы от предполагаемого носителя F0, что контрастирует с более традиционными подходами, которые могут потребовать скрининга сотен эмбрионов, полученных от скрещивания предполагаемых носителей F0.
Этот протокол основан на установленных протоколах сбора спермы у рыбок данио-рерио 7,10,14,16,17,18 путем включения воспроизводимого метода идентификации изменений зародышевой линии с использованием гель-электрофореза высокого разрешения. Этот подход может быть легко включен в любой стандартный рабочий процесс CRISPR/Cas для увеличения пропускной способности для скрининга и выделения целевых правок генома. Кроме того, этот протокол подходит для лабораторий, укомплектованных персоналом с различной подготовкой и опытом, а также для учебных лабораторий. Однако наш метод сбора спермы недостаточен для криоконсервации, которая была экспертно описана в предыдущих публикациях10,17.
В этом протоколе используется электрофорез в агарозном геле высокого разрешения для идентификации предполагаемых мужчин-носителей желаемых индельных и точных геномных мутаций. Тем не менее, геномная ДНК сперматозоидов поддается множеству других подходов, включая флуоресцентный анализ фрагментов или секвенированиештрих-кода 14, анализ расплава с высоким разрешением18 или простое обнаружение ампликонов флуоресцентных меток или эпитопов с использованием стандартных подходов гель-электрофореза. Последующая валидация всех аллелей с использованием таких подходов, как секвенирование на основе Сэнгера или секвенирование следующего поколения, должна быть выполнена до начала экспериментальной работы над предполагаемыми аллелями. Действительно, существующие методы скрининга мутаций у эмбрионов с использованием подходасеквенирования следующего поколения 14 могут обойти необходимость анализа на агарозных гелях, который описан в этом протоколе. Тем не менее, использование метода генотипирования с помощью геля является более экономичным и эгалитарным подходом, учитывая, что не все лаборатории имеют легкий доступ к дешевым методам секвенирования следующего поколения во время изолированных и экспериментальных этапов работы с каждым мутантным штаммом.
Таким образом, этот протокол предоставляет пошаговые инструкции по воспроизводимому скринингу геномов сперматозоидов мужчин, отредактированных CRISPR/Cas, так что для скрининга желаемых изменений требуется меньше скрещиваний и меньше ПЦР-скрининга эмбрионов. Применение этого метода уменьшит количество рыб, которые необходимо создать и проанализировать для успешной идентификации отредактированных интересующих аллелей, что также сократит время и затраты персонала на создание стабильных линий.
Раскрытие информации
Никакой.
Благодарности
Мы хотели бы поблагодарить Анну Хайндес из Медицинской школы Вашингтонского университета за ее первоначальные усилия по получению геномной ДНК сперматозоидов хорошего качества с использованием метода горячего выстрела. Эта работа была профинансирована Национальным институтом артрита, скелетно-мышечных и кожных заболеваний Национального института здравоохранения в рамках премии (R01AR072009 для R.S.G.).
Материалы
| Name | Company | Catalog Number | Comments |
| Agarose powder | Fisher BioReagents | BP1356-100 | |
| Breeding tanks | Carolina Biological | 161937 | |
| BstNI Restriction Enzyme | NEB | R0168S | |
| Cas9 Endonuclease | IDT | 1081060 | |
| DNA Ladder, 100 bp | Thermo Scientific | FERSM0241 | |
| dnah10 donor construct | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry. Phosphorothioate bond on the donor at the first three phosphate bonds on both the 5’ and 3’ ends (5'-CCTCTCTCCCTTTCAGAAGCTTC TGCTCATCCGCTGCTTCTGCCT GGACCGAGTGTACCGTGCCGTC AGTGATTACGTCACGC-3') | |
| dnah10 forward primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CATGGAACTCTTTCCTAATGAGT TTGGC-3') | |
| dnah10 reverse primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry ('5-AGTAGAGATCACACATCAACAGA ATACAGC-3') | |
| dnah10 synthetic sgRNA | Synthego | Synthetic sgRNA, target sequence: 5'-GCTCATCCGCTGCTTCAGGC-3' | |
| Electrophoresis power supply | Thermo Scientific | 105ECA-115 | |
| Filter forceps | Millipore | XX6200006P | |
| Fish (system) water | Generic | n/a | |
| Gel electrophoresis system (including casting frame, comb, and electrophoresis chamber) | Thermo Scientific | B2 | |
| Gel imaging light box | Azure Biosystems | AZI200-01 | |
| Gel stain, 10000X | Invitrogen | S33102 | |
| Glass bowl, 250 mL | Generic | n/a | |
| Isolation tanks, 0.8 L | Aquaneering | ZT080 | |
| Microcap capillary tube with bulb, 20 µL | Drummond | 1-000-0020/CA | |
| Minicentrifuge | Bio-Rad | 12011919EDU | |
| Micropipettes, various with appropriate tips | Generic | n/a | |
| Microwave | Generic | n/a | |
| Nuclease free water | Promega | P119-C | |
| Paper towels | Generic | n/a | |
| PCR tubes, 0.2 mL | Bioexpress | T-3196-1 | |
| Plastic spoon, with drilled holes/slots | Generic | n/a | |
| KCl solution, 0.2 M RNAse Free | Sigma-Aldrich | P9333 | |
| p2ry12 forward primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CCCAAATGTAATCCTGACCAGT -3') | |
| p2ry12 reverse primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CCAGGAACACATTAACCTGGAT -3') | |
| p2ry12 synthetic sgRNA | Synthego | Synthetic sgRNA, target sequence: 5'-GGCCGCACGAGGTCTCCGCG-3' | |
| Restriction Enzyme 10X Buffer | NEB | B6003SVIAL | |
| NaOH solution, 50 mM | Thermo Scientific | S318; 424330010 | |
| Sponge, 1-inch x 1-inch cut with small oval divot | Generic | n/a | |
| Stereomicroscope | Zeiss | Stemi 508 | |
| Taq polymerase master mix, 2X | Promega | M7122 | |
| TBE Buffer Concentrate, 10X | VWR | E442 | |
| Thermal Cycler | Bio-Rad | 1861096 | |
| Tissue paper | Fisher Scientific | 06-666 | |
| Tricaine-methanesulfonate solution (Syncaine, MS-222), 0.016% in fish water (pH 7.0±0.2) | Syndel | 200-266 | |
| Tris Base, 1M (Buffered with HCl to ph 8.0) | Promega | H5131 |
Ссылки
- Auer, T. O., Duroure, K., De Cian, A., Concordet, J. P., Del Bene, F. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair. Genome Research. 24 (1), 142-153 (2014).
- Hwang, W. Y., et al. Efficient genome editing in zebrafish using a CRISPR-Cas system. Nature Biotechnology. 31 (3), 227-229 (2013).
- Jao, L. E., Wente, S. R., Chen, W. Efficient multiplex biallelic zebrafish genome editing using a CRISPR nuclease system. Proceedings of the National Academy of Sciences of the United States of America. 110 (34), 13904-13909 (2013).
- Troutwine, B. R., et al. The Reissner fiber is highly dynamic in vivo and controls morphogenesis of the spine. Current Biology. 30 (12), 2353-2362 (2020).
- Xu, Y., Li, Z. CRISPR-Cas systems: Overview, innovations and applications in human disease research and gene therapy. Computational and Structural Biotechnology Journal. 18, 2401-2415 (2020).
- Creaser, C. W. The technic of handling the zebrafish (Brachydanio rerio) for the production of eggs which are favorable for embryological research and are available at any specified time throughout the year. Copeia. 1930 (4), 159-161 (1934).
- Carmichael, C., Westerfiel, M., Varga, Z. M. Cryopreservatin and in vitro fertilization at the zebrafish international resource center. Methods in Molecular Biology. 546, 45-65 (2009).
- Wang, Y., Troutwine, B. R., Zhang, H., Gray, R. S. The axonemal dynein heavy chain 10 gene is essential for monocilia motility and spine alignment in zebrafish. Developmental Biology. 482, 82-90 (2021).
- Gray, R. S., et al. Postembryonic screen for mutations affecting spine development in zebrafish. Developmental Biology. 471, 18-33 (2021).
- Brocal, I., et al. Efficient identification of CRISPR/Cas9-induced insertions/deletions by direct germline screening in zebrafish. BMC Genomics. 17, 259 (2016).
- Davis, M. W., Jorgensen, E. M. ApE, A Plasmid Editor: A freely available DNA manipulation and visualization program. Frontiers in Bioinformatics. 2, 816619 (2022).
- Labun, K., et al. CHOPCHOP v3: Expanding the CRISPR web toolbox beyond genome editing. Nucleic Acids Research. 47, 171-174 (2019).
- Hill, J. T., et al. Poly Peak Parser: Method and software for identification of unknown indels using sanger sequencing of polymerase chain reaction products. Developmental Dynamics. 243 (12), 1632-1636 (2014).
- Varshney, G. K., et al. High-throughput gene targeting and phenotyping in zebrafish using CRISPR/Cas9. Genome Research. 25 (7), 1030-1042 (2015).
- Liu, Q., et al. Hi-TOM: A platform for high-throughput tracking of mutations induced by CRISPR/Cas systems. Science China. Life Sciences. 62 (1), 1-7 (2019).
- Sorlien, E. L., Witucki, M. A., Ogas, J. Efficient production and identification of CRISPR/Cas9-generated gene knockouts in the model system Danio rerio. Journal of Visualized Experiments. (138), e56969 (2018).
- Draper, B. W., Moens, C. B. A high-throughput method for zebrafish sperm cryopreservation and in vitro fertilization. Journal of Visualized Experiments. (29), e1395 (2009).
- Parant, J. M., George, S. A., Pryor, R., Wittwer, C. T., Yost, H. J. A rapid and efficient method of genotyping zebrafish mutants. Developmental Dynamics. 238 (12), 3168-3174 (2009).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены