Method Article
筛选精子以快速分离斑马鱼的种系编辑
摘要
CRISPR-Cas技术彻底改变了基因组编辑领域。然而,寻找和分离所需的种系编辑仍然是一个主要瓶颈。因此,该协议描述了一种使用标准PCR,限制性酶切和凝胶电泳技术快速筛选F0 CRISPR注射斑马鱼精子以进行种系编辑的稳健方法。
摘要
靶向CRISPR-Cas核酸酶技术的出现彻底改变了在已建立和新兴模型系统中进行精确基因组编辑的能力。CRISPR-Cas基因组编辑系统使用合成向导RNA(sgRNA)将CRISPR相关(Cas)核酸内切酶靶向特定的基因组DNA位点,其中Cas核酸内切酶产生双链断裂。通过固有的容易出错机制修复双链断裂会导致插入和/或删除,从而破坏位点。或者,在此过程中包含双链DNA供体或单链DNA寡核苷酸可以引发精确的基因组编辑,范围从单核苷酸多态性到小免疫标签甚至大荧光蛋白构建体。然而,这个过程的一个主要瓶颈可能是在种系中发现和分离所需的编辑。该协议概述了一种在 Danio rerio (斑马鱼)的特定位点筛选和分离种系突变的可靠方法;然而,这些原则可能适用于任何可以 体内精子收集 的模型。
引言
CRISPR(簇状规则间隔短回文重复序列)/Cas系统是在Danio rerio(斑马鱼)模型系统1,2,3,4中进行位点特异性诱变和精确基因组编辑的强大工具。Cas-核糖核蛋白 (RNP) 由两种主要成分组成:Cas核酸内切酶(通常为 Cas9 或 Cas12a)和位点特异性合成向导 RNA (sgRNA)5。Cas-RNP一起在所需的位点中产生双链断裂(DSB),可以通过两种内在修复机制之一进行修复。非同源端接 (NHEJ) 修复机制容易出错,并且通常会导致 DSB 周围的各种插入或缺失 (插入缺失)。如果这些插入缺失在所得蛋白质序列中引入移码突变或过早停止,则可能是有害的。或者,同源定向修复 (HDR) 机制使用供体模板,在 DSB 位点周围具有同源区域来修复损伤。研究人员可以利用HDR系统生成精确的基因组编辑。具体来说,他们可以共同注入双链DNA供体构建体,其中包含所需的编辑以及基因组中DSB位点两侧的同源区域。这些商业生产的CRISPR组件的规模经济的提高大大减少了筛选多个位点和为精确基因组编辑建立更大规模努力的障碍。然而,在有性繁殖动物模型中,一个主要瓶颈是种系稳定的突变动物的鉴定和分离。
斑马鱼模型系统表现出几个关键品质,可以增强其在反向遗传研究中的使用。它们很容易通过基本的水生住房设备大量饲养,雌性全年表现出很高的繁殖力6。此外,它们的外部产卵和受精使它们适合显微注射CRISPR / Cas成分。Cas-RNP通常被注射到单细胞阶段斑马鱼胚胎中以产生DSB/修复,理论上,DSBs由所有子细胞遗传。然而,二倍体基因组需要两个DSB/修复事件来诱变两个同源染色体。此外,尽管Cas-RNP是在单细胞阶段注射的,但DSB/修复可能要到发育的后期才会发生。这些因素共同促成了注入F0的鱼的马赛克性质。一种常见的做法是杂交F0注入的鱼,并筛选F1后代的插入/特定编辑。然而,由于并非所有注射F0的鱼都具有种系突变,这种做法会导致许多非生产性杂交,无法产生所需的编辑。筛选F0种系而不是F1体细胞组织增加了分离所需种系编辑的可能性,并减少了该过程所需的动物数量。
精子可以很容易地从注射F0的斑马鱼中收集,而无需安乐死。此功能允许冷冻保存和再衍生冷冻精子原种7 ,但也可用于快速筛选,鉴定和分离所需基因组突变的种系携带者8,9。Brocal等人(2016)之前描述了一种基于测序的方法,用于筛选注射F0的雄性斑马鱼10的种系编辑。虽然这种方法可用于鉴定种系中存在的突变等位基因,但在高通量下可能会变得昂贵,并且可能不适用于所有实验室。相比之下,目前的协议提供了一种平易近人且具有成本效益的基于电泳的策略来鉴定种系编辑。具体来说,该协议概述了使用高分辨率琼脂糖凝胶电泳筛选和分离特定位点种系突变的稳健方法。此外,该协议描述了一种类似的策略,用于识别包含特定编辑的供体构建体的成功整合。与往常一样,如果需要特定的编辑,可以与下面描述的协议一起执行基于测序的策略。尽管该协议特定于斑马鱼模型系统,但这些原则应适用于任何将精子收集作为常规程序的模型。总之,这些策略将允许鉴定具有种系插入缺失/编辑的F0注射雄性,这些插入缺失/编辑可以在标准聚合酶链反应(PCR)和/或限制性酶切后在凝胶上解决。
研究方案
这项研究是根据美国国立卫生研究院实验动物护理和使用指南中的指南进行的。该协议已获得德克萨斯大学奥斯汀分校动物护理和使用委员会 (AUP-2021-00254) 的批准。
1. 设计用于CRISPR诱变的sgRNA
- 获取包含目标位点的外显子序列。
- 设计具有原间隔邻基序 (PAM) 位点的合成向导 RNA(sgRNA),该位点特定于 Cas 核酸内切酶。
- 如果共注射供体构建体,请将sgRNA设计为具有尽可能接近所需编辑的预测切割位点。
注意:APE是一款免费软件,带有查找Cas9 sgRNA位点的工具11。或者,可以使用诸如CHOPCHOP之类的工具,它为设计sgRNA提供了一个在线界面,该界面考虑了Cas9和其他Cas 内切酶12的不同PAM位点。
- 如果共注射供体构建体,请将sgRNA设计为具有尽可能接近所需编辑的预测切割位点。
- 设计正向和反向引物,以扩增设计的CRISPR/Cas切割位点周围的区域。PCR产物的长度应约为200-400个碱基对(bp),以便能够在方案后期分离凝胶上的小插入缺失。
注意:CHOPCHOP11 自动为其输出的每个 sgRNA 设计引物。 - 可选:设计具有所需编辑的DNA供体构建体。
- 获得包含所需突变位点的外显子的基因组序列。
注意:内含子序列可能因个体而异,因此最好避免在供体构建体中包含内含子序列,因为这可能会干扰同源依赖性修复途径。 - 如果需要特定的氨基酸变化,请相应地改变特定的密码子。
注意:进行这些更改时,请注意密码子频率。如果可能,请选择使用率高的密码子。 - 为了防止CRISPR / Cas消化供体构建体,进行额外的同义突变,(i)破坏PAM和/或(ii)破坏sgRNA序列,更倾向于使非同 义 变化更接近PAM位点。
- 使用限制性内切位点查找器(APE11 也具有此功能)在野生型序列的预测 PCR 产物中查找独特的限制性位点(仅出现一次的位点)。
- 对预测的PCR产物重复此过程,以反映供体构建体的成功整合。
- 比较野生型序列和供体整合序列之间的限制性位点列表,以找到 仅 存在于编辑序列中的限制性位点。
- 如果没有找到唯一的限制性位点,请继续在供体构建体中进行同义更改,直到实现为止。
注意:如果可能,继续进行同义突变,这些突变会破坏sgRNA与供体构建体的结合。
- 如果可能的话,将供体序列修剪到可管理的大小(50-100 bp),在预测的CRISPR / Cas切割位点周围至少有20 bp的同源性并进行编辑。
- 获得包含所需突变位点的外显子的基因组序列。
- 获取设计的 sgRNA 和/或 DNA 供体寡核苷酸,以及基因分型所需的任何引物。
注意:为了防止核酸酶消化DNA供体寡核苷酸,建议用硫代磷酸酯修饰DNA寡核苷酸5'和3'端的前三个磷酸键。 - 将1nL注射混合物注射到单细胞阶段胚胎中。通常,标准注射混合物包含以下内容:
1 μL 25 μM 位点特异性 sgRNA
1 μL 25 μM Cas9 核酸内切酶
可选:1 μL 3 μM DNA 供体构建体- 使用不含 0.1 M RNase 的 KCl 使总体积达到 5 μL。
- 让注射F0的个体发育成性成熟的成年人。其余方案侧重于筛选F0注射的雄性以进行种系编辑。
2. 设置育种池
- 通过在F0雄性和雌性之间设置带有分隔器的繁殖池来主要F0雄配。
- 为了尽量减少所需的繁殖池数量,在每个水箱中设置三只雄性,对面有两只雌性。
- 在精子采集后,尽可能多地设置可以有效筛选和安置的F0男性。雌性的基因型并不重要,因为在此过程中不会收集它们的配子。
- 在雌性对面设置一个野生型雄性的繁殖池。野生型精子将作为手术后期解释结果的对照。
- 为了尽量减少所需的繁殖池数量,在每个水箱中设置三只雄性,对面有两只雌性。
- 将准备好的繁殖池孵育过夜。
3. 准备精子采集程序的材料
- 准备足够的麻醉溶液(0.016%三卡因-HCl鱼水溶液)以装满一个小玻璃碗。
- 将 40 μL 的 50 mM NaOH 分配到 0.2 mL PCR 管中。为将要收集的每个精子样本准备一根管。存放在冰上。
- 为将在手术过程中取样的每条鱼准备单独的 0.8 升水箱。这些水箱将容纳采样的鱼,直到基因分型完成。
注意:这是限制本协议中可以有效筛选的鱼类数量的主要因素。确保有足够的空间容纳采样的个体,直到基因分型完成。 - 在鱼水中弄湿 1 英寸 x 1 英寸的海绵(用浅椭圆形凹槽切割,以在手术过程中将雄性斑马鱼腹侧朝上)并挤出多余的液体。
- 将海绵以低放大倍率放置在体视显微镜的视野中(图1A)。
- 放置倾斜的头顶照明以获得最佳观看效果。
4.麻醉雄鱼并收集精子
- 准备一个干净的毛细管。
- 轻轻摇动管容器以释放单个毛细管。
- 将毛细管放入提供的灯泡中,并将其放在一边。
- 将雄鱼转移到制备的麻醉溶液中(鱼水中0.016%三卡因-HCl)。
- 用漏勺轻轻搅拌溶液以加快麻醉过程。
- 一旦盖膜(鳃)运动减慢(约1-2分钟),继续进行精子收集。
- 使用漏勺将鱼转移到一叠干净的纸巾上。
- 用漏勺轻轻滚动鱼以去除多余的水分。
- 用干净的折叠薄纸轻轻吸干鱼,以去除任何残留的水。
- 使用漏勺将鱼转移到准备好的海绵上(图1B)。
- 将鱼的腹侧朝上,头部最靠近执行手术的人的惯用手。
- 用干净的折叠薄纸轻轻擦拭臀鳍周围的区域。
- 收集精子。
- 使用毛细管的末端,轻轻地将骨盆鳍横向移离中线以露出泄殖腔(图1C)。
- 将毛细管的末端靠近泄殖腔。
- 使用镊子轻轻挤压鱼的侧面,从鳃下方开始,到泄殖腔结束(图1C')。
- 通过毛细管作用将精子收集到毛细管中。大约 1 μL 足以完成此过程(图 1D)。
注意:一些实验室报告使用p10移液管吸头而不是毛细管进行精子收集。 - 如果精子没有以温和的压力释放,请将鱼放回系统水中。
- 监测麻醉恢复情况。
- 不要用力挤压鱼以排出精子,因为这会伤害鱼。
- 让鱼休息 2 周,然后再尝试再次收集精子。
- 收集后,将鱼放入带有系统水的干净隔离池中,用于个人住房。
- 监测鱼从麻醉中恢复的情况。
- 让鱼休息 2 周,然后再尝试再次收集精子。
- 将带有收集的精子的毛细管放入制备的PCR管(40μL的50mM NaOH)中。
- 挤压橡胶球以排出收集的样品。
- 将用过的毛细管丢弃到批准的玻璃废物容器中。
- 将样品在冰上孵育,直到所有雄性都被挤压。
- 对每个雄性重复步骤4.1-4.5。
- 标记分离罐和精子样本,以便最终的精子基因分型结果可以追溯到具有假定突变的相应个体。
- 监控所有人员,并确保他们直立并探索他们的坦克,然后再将坦克放回系统上。
- 将用于启动的雌性放回系统上各自的水箱中。
- 如果任何鱼在 10 分钟后没有从麻醉中恢复,请按照与实验室相关的机构批准的安乐死程序进行操作。
- 示例:在冰浴中用2-4°C的鱼水快速冷却个体。
- 根据与实验室相关的机构批准的程序,将安乐死的鱼丢弃在适当的动物尸体废物容器中。
5. 从精子样本中提取DNA
- 在微型离心机中短暂旋转所有精子样品,并将PCR管放入热循环仪中。
- 合上热循环仪的盖子。
- 在热循环仪中运行以下设置:
- 将样品在95°C下加热40分钟。
- 将样品冷却至25°C。
- 从热循环仪中取出样品。
- 使用干净的移液器吸头对每个样品进行中和,加入 10 μL 的 1 M Tris-HCl(缓冲至 pH 8)来中和 pH。
- 通过上下移液充分混合。
- 在小型离心机中短暂旋转样品。
- 基因组DNA样品可以在4°C下储存数天或在-20°C下放置长达6个月。
6. 所需基因座的PCR扩增(和/或限制性酶切)
- 使用标准PCR方案扩增所需的位点。
- 在下面的PCR反应中将热循环仪预热至初始变性温度。
- 为PCR管中的每个精子样品制备25μL反应混合物:
12.5 μL 2x Taq 聚合酶预混液
1.5 μL 10 μM 正向引物
1.5 μL 10 μM 反向引物
4.5 μL 无核酸酶水
5 μL 中和精子 DNA 样品 - 通过上下移液充分混合。
- 在小型离心机中短暂旋转样品。
- 使用以下设置将样品放入预热的热循环仪中:
初始变性:95°C3分钟
35个变性循环(95°C持续30秒),退火(55°C持续30秒)和延伸(72°C持续30秒)
最终延伸:72°C5分钟
注意:可能需要根据特定的Taq聚合酶和PCR扩增产物的预期长度调整延伸温度和/或时间。 - 从热循环仪中取出PCR样品。
- 可选:对少量PCR产物进行供体特异性限制性酶切,留下至少5-10 μL未消化的PCR产物用于凝胶电泳。
- 在 0.2 mL PCR 管中制备 30 μL 反应:
10 μL 未纯化的 PCR 产物
15 μL 无核酸酶水
3 μL 10x 限制性内切酶缓冲液
2 μL 限制性内切酶(BstNI 用于分析 dnah10 敲入等位基因的代表性结果)
注意:许多限制性内切酶在典型的PCR反应缓冲液中仍然有效;然而,添加限制性酶切缓冲液有助于改善结果。如果出现任何问题,请参阅特定制造商的说明以进行故障排除。 - 通过上下移液充分混合。
- 在小型离心机中短暂旋转样品。
- 将样品放入具有以下设置的热循环仪中:
- 在60°C下切割PCR扩增子1小时。
注意:可能需要根据所使用的特定限制性内切酶调整切割温度和孵育时间。 - 如果制造商指示,则以所需的温度和孵育时间灭活限制性内切酶。
注意:BstNI酶不需要热灭活。
- 在60°C下切割PCR扩增子1小时。
- 在 0.2 mL PCR 管中制备 30 μL 反应:
7. 进行凝胶电泳以分离不同大小的PCR扩增子
- 准备 500 mL 的 0.5x TBE 电泳缓冲液:将 50 mL 的 10x TBE 缓冲液浓缩物加入 450 mL 去离子水中。
- 在 0.5x TBE 电泳缓冲液中制备 100 mL 4% 琼脂糖凝胶:将 4 g 琼脂糖和 10 μL 10,000x 凝胶染色剂加入 100 mL 0.5x TBE 电泳缓冲液中。微波直至琼脂糖粉末完全溶解。
- 将凝胶溶液倒入适当大小的凝胶灌制框架中,然后将梳子插入凝胶的一侧。
注意:较大的凝胶尺寸(推荐:15 cm x 15 cm)对于此过程是有利的,因为它为扩增子提供了更多的空间来分离凝胶。- 如果要分析限制性酶切产物,最好将其与PCR产物在同一凝胶上运行,以便比较结果。根据需要调整梳孔尺寸,以将所有样品放在凝胶上。
- 将凝胶保持在室温下直至凝固。小心地取下梳子。小心地从浇注架上取出凝胶。
- 将凝胶溶液倒入适当大小的凝胶灌制框架中,然后将梳子插入凝胶的一侧。
- 设置凝胶电泳台。
- 将凝胶放置在电泳台中,使孔最靠近负极。将0.5x TBE电泳缓冲液倒入电泳台中,直到孔完全浸没。
- 加载凝胶。
- 将 5 μL DNA 分子量标准品加载到第一个孔中(选择包含与 PCR 扩增子大小相似的 DNA 片段的 DNA 分子量标准)。
- 对每个样品(包括野生型对照样品)使用新的移液器吸头,将 5-10 μL PCR 产物加载到剩余孔中。
- 可选:如果要分析限制性酶切产物,请将这些样品也加载到凝胶上。
注意:最好在PCR和限制性酶切产物之间加载等量的DNA。例如,在 30 μL 限制性酶切反应混合物中,加入 10 μL PCR 产物。因此,15 μL 消化产物的 DNA 浓度与 5 μL 未纯化的 PCR 产物相当。
- 运行凝胶。
- 将电极连接到凝胶电泳台,并施加150 V至少1小时以确保扩增子的充分分离。
- 对凝胶进行成像以查看DNA条带。
注意:强诱变的CRISPR靶向位点将以多个条带大小运行,代表插入/缺失等位基因与野生型扩增子的组合。- 如果凝胶条带在1小时后未分离,则每隔150分钟向凝胶施加150 V,直到条带在凝胶上充分分离。
- 可选:如果筛选精确编辑(如 SNP、表位标签或荧光标签)的插入,请使用特定于所需编辑的 PCR 方法筛选精子 DNA。例如,在鉴定供体特异性限制性位点时,与精子样本的PCR产物相比,限制性酶切产物中将存在额外的条带。
8. 分离种系稳定等位基因
- 从精子挤压过程中休息 2 周后,超越精子基因组 DNA 包含所需编辑的 F0 个体。
注意:斑马鱼中单个精原体克隆的精子生成可能是周期性的;因此,所需的等位基因在异交时可能不会产生精子。出于这个原因,可能必须进行多次额外的杂交,以分离与初始筛选有关的等位基因。 - 一旦F1杂交鱼性成熟,使用在精子基因组DNA筛选期间执行的相同PCR和凝胶电泳方案进行标准基因分型方法(例如,鳍夹DNA)。
- Sanger对PCR产物进行测序,以鉴定具有相同所需编辑的F1个体。
- 可选:如果所需的编辑引入或中断限制性站点,请使用适当的限制摘要协议进行筛选。
- 具有相同所需编辑的杂交 F1 杂合子。
- 筛选 F2 后代的预期表型和/或基因组编辑。
结果
该协议中描述的实验方法允许通过专注于分析来自F0注射的男性精子收集的数千个基因组来更快速地识别基因组编辑或假定的有害等位基因。 图 2 重点介绍了如何解释使用此协议获得的结果。
为了在p2ry12位点中产生突变,向单细胞阶段斑马鱼胚胎注射Cas9核酸内切酶和p2ry12特异性sgRNA(图2B,灰色突出显示)。将注射F0的鱼放入系统中直到性成熟,并从六个雄性个体中收集精子。使用高温和碱性条件(HotSHOT DNA提取)从精子样品中提取DNA,并在p2ry12位点的PCR扩增之前中和。将PCR产物在高电压(150V)下在高百分比(4%)琼脂糖凝胶上分离1.5小时。在本例中,野生型扩增子作为长度约为250个碱基对的单个亮带运行(图2C;WT)。相反,含有插入缺失的F0注射雄性扩增子在凝胶上以多条带的形式运行(图2C;泳道#1-2,#5-8)。或者,突变的F0注射雄性扩增子可以作为单条带运行,凝胶迁移率改变(p2ry12示例中未显示,但在dnah10 F0-male #2的PCR产物中很明显;图2E)。在p2ry12示例凝胶上,与野生型带相比,样品#3和样品#4是否含有插入缺失不太清楚,因此这些人可能不是最佳的创始人候选人。从具有更独特扩增子改变的F0雄性中分离等位基因最适合繁殖稳定的种系携带者,因为它们很容易在4%琼脂糖凝胶上评分。例如,F0-male #6精子样本似乎在其中一个等位基因中含有大量缺失(图2C;泳道6;凝胶流动性增加的亮带)。如果在F1代中选择了这个大的缺失等位基因,则很容易将其与4%琼脂糖凝胶上的WT等位基因区分开来。或者,如果需要不同等位基因的集合,F0-male #7可能是一个有效的创始人候选者,因为它的PCR产物似乎包含几个不同迁移率的离散等位基因。一旦选择了创始雄性,就可以通过将个体杂交回用于注射的原始野生型菌株来分离所需的等位基因。
为了在dnah10基因中产生特异性敲入突变,在单细胞阶段斑马鱼胚胎中注射了Cas9核酸内切酶,dnah10特异性sgRNA(图2D;灰色突出显示)和含有所需编辑的DNA供体寡核苷酸(图2D;红色)和供体特异性BstNI限制性位点(图2D,括号)。这种设计允许在PCR后通过限制性酶切轻松识别供体整合。此外,通过改变sgRNA识别位点内的碱基对(图2D;灰色突出显示),这种设计可防止供体序列的Cas9消化。一旦注射F0的鱼达到性成熟,收集精子,并使用热射法提取DNA。从这些样品中,使用PCR扩增dnah10位点,并将产物在高百分比(4%)琼脂糖凝胶上在高压(150V)下分离1小时。在本例中,野生型扩增子作为长度约为400个碱基对的单个亮带运行(图2E;上图:WT)。相比之下,含有插入缺失的F0注射雄性扩增子以多个条带的形式运行(图2E;上图:泳道#1、#4和#5)或凝胶迁移率降低的单个条带(图2E;上图:泳道#2)。为了确定是否有任何精子样品含有供体整合的等位基因,将PCR产物用BstNI限制性内切酶消化1小时,并将产品在4%琼脂糖凝胶上在高压(150V)下运行1小时。将未消化的PCR产物(图2D;上图)与消化产物(图2D;底图)进行比较,可以发现样品可能整合了供体构建体。在本例中,F0注射的男性#1在消化产物中具有PCR产物中不存在的额外条带(图2D;底图:泳道#1,圈出)。因此,这只雄性代表了建立具有所需敲入的突变系的最佳创始人候选人。
一旦推定的F0携带者雄性杂交,必须在F1后代中对等位基因进行序列验证。建议直接对源自F1杂合子鱼的扩增子进行Sanger测序,然后进行杂合等位基因分析方法,如Poly Peak Parser13 或使用各种基于下一代测序的方法,如MiSeq14 或Hi-Tom15 测序。这是一种必要且互补的方法,可确保精确的编辑或有害的插入缺失突变实际上是种系的,而不仅仅是凝胶电泳分析的伪影。事实上,使用下一代测序方法对来自F0载体的精子DNA进行测序可以很容易地代替本协议中描述的凝胶电泳分析。然而,对于全球斑马鱼研究人员社区来说,拥有易于烧焦的琼脂糖凝胶基因分型方法是一种更经济、更平等的方法。

图1:精子挤压程序的设置 。 (A)将海绵放置在具有头顶照明的低放大倍率的体视显微镜下方。(B)湿润的1英寸x 1海绵,用椭圆形凹陷(虚线)切割,用于在手术过程中保持麻醉的雄鱼腹侧朝上。(C)麻醉雄鱼腹侧的解剖图,描绘了臀鳍(AF,粉红色),盆鳍(pf,蓝色)和泄殖腔(箭头),精子将在手术过程中排出。(C')过滤钳用于轻轻地将麻醉的雄性鱼从鳃挤压到泄殖腔,同时通过毛细管作用将排出的精子吸入玻璃移液管(白色箭头)。(D)含有足够排出的精子(不透明液体;黑色支架)的毛细管,用于下游DNA提取和分析。 请点击此处查看此图的大图。
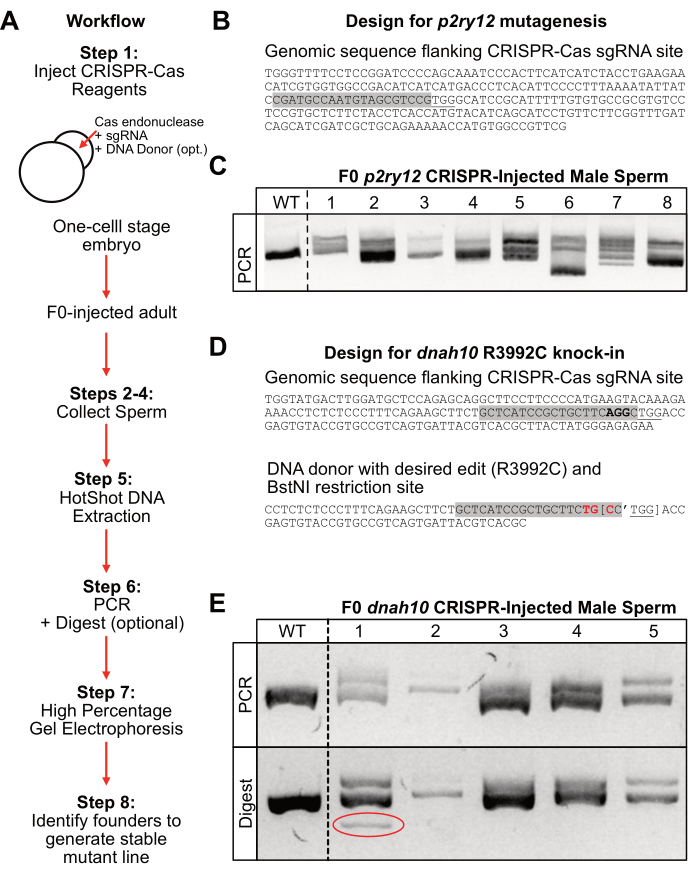
图 2:工作流程和代表性结果 。 (A) 协议的一般工作流程。(B) p2ry12 sgRNA位点的代表性设计(灰色突出显示)与Cas9特异性PAM位点(下划线)。(C)在150 V.野生型(WT)对照和F0-p2ry12 CRISPR注射的男性精子样品(1-8)后1.5小时后4%凝胶电泳的代表性结果。 (D) dnah10 sgRNA位点的代表性设计(灰色突出显示),靶向密码子附近有一个Cas9特异性PAM位点(下划线)(粗体)。具有所需密码子编辑(红色粗体)和BstNI限制性位点(带有撇号标记切割位点的括号)的DNA供体序列。(E)在150V下1小时后4%凝胶电泳的代表性结果, 顶部:野生型(WT)和F0 dnah10 CRISPR注射的男性精子样品的PCR产物(1-5)。 下图:上述样品的BstNI限制性酶切产物。样品1展示了基于消化后附加条带(红色圆圈)的供体构建体的成功整合。 请点击此处查看此图的大图。
讨论
该协议描述了一种通过聚焦分析F0男性精子基因组,使用CRISPR-Cas技术快速表征假定基因组编辑或靶向突变的方法。该协议应适用于其他动物模型,其中精子很容易采样而无需安乐死。这种方法将提高筛选所需编辑的吞吐量,对于识别罕见的 HDR 介导的敲入事件特别有用。这种方法还有助于减少用于寻找稳定种系编辑的实验动物的数量,通过促进从推定的F0载体中快速筛选一个精子样本中潜在的数千个基因组,这与可能需要筛选数百个来自假定F0携带者杂交的胚胎的更传统方法形成鲜明对比。
该协议建立在斑马鱼7,10,14,16,17,18 精子收集的既定协议之上,包括使用高分辨率凝胶电泳鉴定种系编辑的可重复技术。这种方法可以很容易地整合到任何标准的CRISPR/Cas工作流程中,以提高靶基因组编辑筛选和分离的通量。此外,该协议适用于配备具有一系列培训和经验的人员的实验室以及教学实验室。然而,我们的精子收集方法不足以冷冻保存,这在以前的出版物10,17中已经进行了专业描述。
该协议使用高分辨率琼脂糖凝胶电泳来鉴定所需插入缺失和精确基因组突变的假定男性携带者。然而,精子基因组DNA适用于无数其他方法,包括荧光片段分析或条形码测序14,高分辨率熔解分析18,或使用标准凝胶电泳方法对荧光标签或表位进行简单的扩增子检测。在开始对假定等位基因的实验工作之前,必须使用基于Sanger或下一代测序等方法对所有等位基因进行下游验证。事实上,使用下一代测序方法14 筛选胚胎突变的现有方法可以避免对琼脂糖凝胶进行分析的需要,这在本协议中描述。然而,鉴于并非所有实验室在处理每种突变菌株的分离和实验阶段都可以轻松获得廉价的下一代测序方法,因此拥有凝胶可灼热的基因分型方法是一种更具成本效益和平等主义的方法。
总之,该协议为可重复地筛选来自CRISPR / Cas编辑的男性的精子基因组提供了分步指导,因此需要更少的杂交和更少的胚胎PCR筛选来筛选所需的编辑。该方法的应用将减少需要创建和分析以成功识别编辑的感兴趣等位基因的鱼的数量,这也减少了生成稳定品系的人员时间和成本。
披露声明
没有。
致谢
我们要感谢华盛顿大学医学院的Anna Hindes,感谢她使用热射法获得高质量精子基因组DNA的初步努力。这项工作由美国国立卫生研究院国家关节炎,肌肉骨骼和皮肤病研究所资助(R01AR072009至RSG)。
材料
| Name | Company | Catalog Number | Comments |
| Agarose powder | Fisher BioReagents | BP1356-100 | |
| Breeding tanks | Carolina Biological | 161937 | |
| BstNI Restriction Enzyme | NEB | R0168S | |
| Cas9 Endonuclease | IDT | 1081060 | |
| DNA Ladder, 100 bp | Thermo Scientific | FERSM0241 | |
| dnah10 donor construct | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry. Phosphorothioate bond on the donor at the first three phosphate bonds on both the 5’ and 3’ ends (5'-CCTCTCTCCCTTTCAGAAGCTTC TGCTCATCCGCTGCTTCTGCCT GGACCGAGTGTACCGTGCCGTC AGTGATTACGTCACGC-3') | |
| dnah10 forward primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CATGGAACTCTTTCCTAATGAGT TTGGC-3') | |
| dnah10 reverse primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry ('5-AGTAGAGATCACACATCAACAGA ATACAGC-3') | |
| dnah10 synthetic sgRNA | Synthego | Synthetic sgRNA, target sequence: 5'-GCTCATCCGCTGCTTCAGGC-3' | |
| Electrophoresis power supply | Thermo Scientific | 105ECA-115 | |
| Filter forceps | Millipore | XX6200006P | |
| Fish (system) water | Generic | n/a | |
| Gel electrophoresis system (including casting frame, comb, and electrophoresis chamber) | Thermo Scientific | B2 | |
| Gel imaging light box | Azure Biosystems | AZI200-01 | |
| Gel stain, 10000X | Invitrogen | S33102 | |
| Glass bowl, 250 mL | Generic | n/a | |
| Isolation tanks, 0.8 L | Aquaneering | ZT080 | |
| Microcap capillary tube with bulb, 20 µL | Drummond | 1-000-0020/CA | |
| Minicentrifuge | Bio-Rad | 12011919EDU | |
| Micropipettes, various with appropriate tips | Generic | n/a | |
| Microwave | Generic | n/a | |
| Nuclease free water | Promega | P119-C | |
| Paper towels | Generic | n/a | |
| PCR tubes, 0.2 mL | Bioexpress | T-3196-1 | |
| Plastic spoon, with drilled holes/slots | Generic | n/a | |
| KCl solution, 0.2 M RNAse Free | Sigma-Aldrich | P9333 | |
| p2ry12 forward primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CCCAAATGTAATCCTGACCAGT -3') | |
| p2ry12 reverse primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CCAGGAACACATTAACCTGGAT -3') | |
| p2ry12 synthetic sgRNA | Synthego | Synthetic sgRNA, target sequence: 5'-GGCCGCACGAGGTCTCCGCG-3' | |
| Restriction Enzyme 10X Buffer | NEB | B6003SVIAL | |
| NaOH solution, 50 mM | Thermo Scientific | S318; 424330010 | |
| Sponge, 1-inch x 1-inch cut with small oval divot | Generic | n/a | |
| Stereomicroscope | Zeiss | Stemi 508 | |
| Taq polymerase master mix, 2X | Promega | M7122 | |
| TBE Buffer Concentrate, 10X | VWR | E442 | |
| Thermal Cycler | Bio-Rad | 1861096 | |
| Tissue paper | Fisher Scientific | 06-666 | |
| Tricaine-methanesulfonate solution (Syncaine, MS-222), 0.016% in fish water (pH 7.0±0.2) | Syndel | 200-266 | |
| Tris Base, 1M (Buffered with HCl to ph 8.0) | Promega | H5131 |
参考文献
- Auer, T. O., Duroure, K., De Cian, A., Concordet, J. P., Del Bene, F. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair. Genome Research. 24 (1), 142-153 (2014).
- Hwang, W. Y., et al. Efficient genome editing in zebrafish using a CRISPR-Cas system. Nature Biotechnology. 31 (3), 227-229 (2013).
- Jao, L. E., Wente, S. R., Chen, W. Efficient multiplex biallelic zebrafish genome editing using a CRISPR nuclease system. Proceedings of the National Academy of Sciences of the United States of America. 110 (34), 13904-13909 (2013).
- Troutwine, B. R., et al. The Reissner fiber is highly dynamic in vivo and controls morphogenesis of the spine. Current Biology. 30 (12), 2353-2362 (2020).
- Xu, Y., Li, Z. CRISPR-Cas systems: Overview, innovations and applications in human disease research and gene therapy. Computational and Structural Biotechnology Journal. 18, 2401-2415 (2020).
- Creaser, C. W. The technic of handling the zebrafish (Brachydanio rerio) for the production of eggs which are favorable for embryological research and are available at any specified time throughout the year. Copeia. 1930 (4), 159-161 (1934).
- Carmichael, C., Westerfiel, M., Varga, Z. M. Cryopreservatin and in vitro fertilization at the zebrafish international resource center. Methods in Molecular Biology. 546, 45-65 (2009).
- Wang, Y., Troutwine, B. R., Zhang, H., Gray, R. S. The axonemal dynein heavy chain 10 gene is essential for monocilia motility and spine alignment in zebrafish. Developmental Biology. 482, 82-90 (2021).
- Gray, R. S., et al. Postembryonic screen for mutations affecting spine development in zebrafish. Developmental Biology. 471, 18-33 (2021).
- Brocal, I., et al. Efficient identification of CRISPR/Cas9-induced insertions/deletions by direct germline screening in zebrafish. BMC Genomics. 17, 259 (2016).
- Davis, M. W., Jorgensen, E. M. ApE, A Plasmid Editor: A freely available DNA manipulation and visualization program. Frontiers in Bioinformatics. 2, 816619 (2022).
- Labun, K., et al. CHOPCHOP v3: Expanding the CRISPR web toolbox beyond genome editing. Nucleic Acids Research. 47, 171-174 (2019).
- Hill, J. T., et al. Poly Peak Parser: Method and software for identification of unknown indels using sanger sequencing of polymerase chain reaction products. Developmental Dynamics. 243 (12), 1632-1636 (2014).
- Varshney, G. K., et al. High-throughput gene targeting and phenotyping in zebrafish using CRISPR/Cas9. Genome Research. 25 (7), 1030-1042 (2015).
- Liu, Q., et al. Hi-TOM: A platform for high-throughput tracking of mutations induced by CRISPR/Cas systems. Science China. Life Sciences. 62 (1), 1-7 (2019).
- Sorlien, E. L., Witucki, M. A., Ogas, J. Efficient production and identification of CRISPR/Cas9-generated gene knockouts in the model system Danio rerio. Journal of Visualized Experiments. (138), e56969 (2018).
- Draper, B. W., Moens, C. B. A high-throughput method for zebrafish sperm cryopreservation and in vitro fertilization. Journal of Visualized Experiments. (29), e1395 (2009).
- Parant, J. M., George, S. A., Pryor, R., Wittwer, C. T., Yost, H. J. A rapid and efficient method of genotyping zebrafish mutants. Developmental Dynamics. 238 (12), 3168-3174 (2009).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。