Method Article
제브라피쉬에서 생식계열 편집의 신속한 분리를 위한 정자 스크리닝
요약
CRISPR-Cas 기술은 게놈 편집 분야에 혁명을 일으켰습니다. 그러나 원하는 생식계열 편집을 찾고 분리하는 것은 여전히 주요 병목 현상으로 남아 있습니다. 따라서 이 프로토콜은 표준 PCR, 제한 분해 및 겔 전기영동 기술을 사용하여 생식계열 편집을 위해 F0 CRISPR 주입 제브라피쉬 정자를 신속하게 스크리닝하는 강력한 방법을 설명합니다.
초록
표적 CRISPR-Cas 뉴클레아제 기술의 출현은 기존 모델 시스템과 새로운 모델 시스템 모두에서 정밀한 게놈 편집을 수행하는 능력에 혁명을 일으켰습니다. CRISPR-Cas 게놈 편집 시스템은 합성 가이드 RNA(sgRNA)를 사용하여 CRISPR 관련(Cas) 엔도뉴클레아제를 특정 게놈 DNA 유전자좌에 표적으로 삼으며, 여기서 Cas 엔도뉴클레아제는 이중 가닥 절단을 생성합니다. 본질적으로 오류가 발생하기 쉬운 메커니즘에 의한 이중 가닥 파손의 복구는 삽입 및/또는 삭제로 이어져 궤적을 방해합니다. 대안적으로, 이 공정에 이중 가닥 DNA 공여체 또는 단일 가닥 DNA 올리고뉴클레오티드를 포함시키면 단일 뉴클레오티드 다형성에서 작은 면역학적 태그 또는 큰 형광 단백질 구조에 이르기까지 정확한 게놈 편집을 유도할 수 있습니다. 그러나 이 절차의 주요 병목 현상은 생식계열에서 원하는 편집을 찾아 분리하는 것일 수 있습니다. 이 프로토콜은 Danio rerio (제브라피쉬)의 특정 유전자좌에서 생식계열 돌연변이를 스크리닝하고 분리하기 위한 강력한 방법을 설명합니다. 그러나 이러한 원칙은 생체 내 정자 수집이 가능한 모든 모델에 적용할 수 있습니다.
서문
CRISPR(Clustered Regularly Interspaced Short Palindromic Repeats)/Cas 시스템은 Danio rerio(제브라피쉬) 모델 시스템 1,2,3,4에서 유전자좌 특이적 돌연변이 유발 및 정확한 게놈 편집을 수행하는 강력한 도구입니다. Cas-리보핵단백질(RNP)은 Cas 엔도뉴클레아제(일반적으로 Cas9 또는 Cas12a)와 유전자좌 특이적 합성 가이드 RNA(sgRNA)의 두 가지 주요 구성 요소로 구성됩니다.5. Cas-RNP는 함께 원하는 유전자좌에 이중 가닥 파손(DSB)을 생성하며, 이 절단은 두 가지 고유 복구 메커니즘 중 하나로 복구할 수 있습니다. NHEJ(Non-homologous End Joining) 복구 메커니즘은 오류가 발생하기 쉬우며 종종 DSB 주위에 다양한 삽입 또는 삭제(indels)가 발생합니다. 이러한 indel은 결과 단백질 서열에 프레임시프트 돌연변이 또는 조기 정지를 도입하는 경우 해로울 수 있습니다. 대안적으로, 상동성 지향 복구(HDR) 메커니즘은 손상을 복구하기 위해 DSB 부위를 둘러싸는 상동성 영역을 갖는 기증자 템플릿을 사용한다. 연구원은 HDR 시스템을 활용하여 정확한 게놈 편집을 생성할 수 있습니다. 특히, 그들은 게놈의 DSB 부위 옆에 있는 상동성 영역뿐만 아니라 원하는 편집을 포함하는 이중 가닥 DNA 기증자 구조를 공동 주입할 수 있습니다. 상업적으로 생산된 이러한 CRISPR 구성 요소의 규모 경제가 증가함에 따라 여러 유전자좌를 스크리닝하고 정확한 게놈 편집을 위한 대규모 노력을 설정하는 데 대한 장벽이 크게 감소했습니다. 그러나 유성 생식 동물 모델에서 주요 병목 현상은 생식계열 안정성 돌연변이 동물의 식별 및 분리입니다.
제브라피쉬 모델 시스템은 역 유전 연구에서 사용을 향상시키는 몇 가지 주요 특성을 보여줍니다. 기본적인 수생 주거 장비로 대량으로 쉽게 키울 수 있으며 암컷은 일년 내내 높은 번식력을 보입니다6. 또한, 외부 알을 낳고 수정하면 CRISPR/Cas 구성 요소의 미세 주입이 가능합니다. Cas-RNP는 일반적으로 이론적으로 모든 딸 세포에 의해 유전되는 DSB/복구를 생성하기 위해 1세포 단계 제브라피쉬 배아에 주입됩니다. 그러나 이배체 게놈은 두 상동 염색체를 돌연변이화하기 위해 두 개의 DSB/복구 사건이 필요합니다. 또한, Cas-RNP는 one-cell 단계에서 주입되지만, DSB/복구는 개발 후반 시점까지 발생하지 않을 수 있습니다. 함께, 이러한 요인들은 F0 주입 물고기의 모자이크 특성에 기여합니다. 일반적인 관행은 F0 주입 물고기를 능가하고 F1 자손을 선별하여 인델/특정 편집을 하는 것입니다. 그러나 모든 F0 주입 물고기가 생식계열 돌연변이를 가지고 있는 것은 아니기 때문에 이 관행은 원하는 편집을 생성하지 않는 많은 비생산적인 교배를 초래합니다. F1 체세포가 아닌 F0 생식계열을 스크리닝하면 원하는 생식계열 편집을 분리할 확률이 증가하고 이 과정에 필요한 동물의 수가 줄어듭니다.
안락사 없이 F0 주사 제브라피쉬에서 정자를 쉽게 채취할 수 있습니다. 이 기능은 냉동 정자 스톡의 냉동 보존 및 재유도를 가능하게 하지만7 원하는 게놈 돌연변이8,9의 생식계열 운반체를 신속하게 스크리닝, 식별 및 분리하는 데에도 활용될수 있다. Brocal et al. (2016)은 이전에 F0 주입 수컷 제브라피쉬10에서 생식계열 편집을 스크리닝하기 위한 시퀀싱 기반 방법을 설명했습니다. 생식계열에 존재하는 돌연변이 대립유전자를 식별하는 데 유용하지만 이 접근 방식은 처리량이 높을수록 비용이 많이 들 수 있으며 모든 실험실에서 액세스할 수 있는 것은 아닙니다. 대조적으로, 현재 프로토콜은 생식계열 편집을 식별하기 위한 접근 가능하고 비용 효율적인 전기영동 기반 전략을 제공합니다. 특히, 이 프로토콜은 고분해능 아가로스 겔 전기영동을 사용하여 특정 유전자좌에서 생식계열 돌연변이를 스크리닝하고 분리하는 강력한 방법을 설명합니다. 또한 이 프로토콜은 특정 편집을 포함하는 기증자 구성의 성공적인 통합을 식별하기 위한 유사한 전략을 설명합니다. 항상 그렇듯이 특정 편집이 필요한 경우 아래에 설명된 프로토콜과 함께 시퀀싱 기반 전략을 수행할 수 있습니다. 이 프로토콜은 제브라피쉬 모델 시스템에만 해당되지만 이러한 원칙은 정자 수집이 일상적인 절차인 모든 모델에 적용할 수 있어야 합니다. 함께, 이러한 전략을 통해 표준 중합효소 연쇄 반응(PCR) 및/또는 제한 소화 후 겔에서 해결할 수 있는 생식계열 인델/편집이 있는 F0 주입 수컷을 식별할 수 있습니다.
프로토콜
이 연구는 국립 보건원(National Institutes of Health)의 실험실 동물 관리 및 사용 가이드(Guide for the Care and Use of Laboratory Animals)의 지침에 따라 수행되었습니다. 이 프로토콜은 텍사스 대학교 오스틴 동물 관리 및 사용 위원회(AUP-2021-00254)의 승인을 받았습니다.
1. CRISPR 돌연변이 유발을 위한 sgRNA 설계
- 표적 유전자좌를 포함하는 엑손 서열을 얻습니다.
- Cas 엔도뉴클레아제에 특이적인 PAM(protospacer adjacent motif) 부위를 갖는 합성 가이드 RNA(sgRNA)를 설계합니다.
- 공여자 구조체를 공동 주입하는 경우, 가능한 한 원하는 편집에 가까운 예측 절단 부위를 갖도록 sgRNA를 설계합니다.
참고: APE는 Cas9 sgRNA 부위를 찾을 수 있는 도구가 포함된 무료 소프트웨어입니다11. 대안적으로, Cas9 및 다른 Cas 엔도뉴클레아제12의 상이한 PAM 부위를 고려하는 sgRNA를 설계하기 위한 온라인 인터페이스를 제공하는 CHOPCHOP과 같은 도구를 사용할 수 있다.
- 공여자 구조체를 공동 주입하는 경우, 가능한 한 원하는 편집에 가까운 예측 절단 부위를 갖도록 sgRNA를 설계합니다.
- 설계된 CRISPR/Cas 절단 부위 주변 영역을 증폭하기 위해 정방향 및 역방향 프라이머를 설계합니다. PCR 산물은 프로토콜의 후반부에 겔의 작은 인델을 분해할 수 있도록 길이가 약 200-400 염기쌍(bp)이어야 합니다.
참고: CHOPCHOP11 은 출력하는 모든 sgRNA에 대한 프라이머를 자동으로 설계합니다. - 선택 사항: 원하는 편집으로 DNA 기증자 구조를 설계합니다.
- 원하는 돌연변이의 유전자좌를 포함하는 엑손의 게놈 서열을 얻습니다.
참고: Intronic 서열은 개인마다 다를 수 있으므로 기증자 구성에 intronic 서열을 포함하면 상동성 의존적 복구 경로를 방해할 수 있으므로 피하는 것이 가장 좋습니다. - 특정 아미노산 변경이 필요한 경우 그에 따라 특정 코돈을 변경합니다.
참고: 이러한 변경을 수행할 때 코돈 주파수에 유의하십시오. 가능하면 사용량이 많은 코돈을 선택하십시오. - 공여자 구성물의 CRISPR/Cas 소화를 방지하기 위해 (i) PAM을 방해하거나 (ii) sgRNA 서열을 방해하는 추가 동의어 돌연변이를 만들고, PAM 부위에 더 가까운 비동의 어 변경을 수행하는 것이 더 선호됩니다.
- 제한 부위 찾기(APE11 에도 이 기능이 있음)를 사용하여 야생형 서열의 예측된 PCR 산물에서 고유한 제한 부위(한 번만 발생하는 부위)를 찾습니다.
- 공여체 구축물의 성공적인 통합을 반영하는 예측된 PCR 산물에 대해 이 과정을 반복합니다.
- 야생형 염기서열과 기증자 통합 염기서열 사이의 제한 부위 목록을 비교하여 편집된 염기서열 에만 존재하는 제한 부위를 찾습니다.
- 고유한 제한 부위가 발견되지 않으면 이것이 달성될 때까지 기증자 구성 내에서 동의어 변경을 계속합니다.
참고: 가능하면 기증자 구조에 대한 sgRNA 결합을 방해하는 동의어 돌연변이를 계속 만드십시오.
- 가능한 경우 예측된 CRISPR/Cas 절단 부위 및 편집을 둘러싼 최소 20bp의 상동성을 가진 관리 가능한 크기(50-100bp)로 기증자 서열을 다듬습니다.
- 원하는 돌연변이의 유전자좌를 포함하는 엑손의 게놈 서열을 얻습니다.
- 설계된 sgRNA 및/또는 DNA 기증자 올리고뉴클레오티드와 유전형 분석에 필요한 프라이머를 얻을 수 있습니다.
참고: 뉴클레아제에 의한 DNA 공여체 올리고뉴클레오티드의 소화를 방지하기 위해 DNA 올리고뉴클레오티드의 5' 및 3' 말단에 있는 처음 3개의 인산염 결합을 포스포로티오에이트로 수정하는 것이 좋습니다. - 주입 혼합물 1nL을 1세포 단계 배아에 주입합니다. 일반적으로 표준 주입 혼합물에는 다음이 포함됩니다.
25μM 유전자좌 특이적 sgRNA 1μL
1 μL의 25 μM Cas9 엔도뉴클레아제
선택 사항: 1μL의 3μM DNA 기증자 구조- 0.1 M RNase-free KCl로 총 부피를 5 μL로 가져옵니다.
- F0 주사를 맞은 개인이 성적으로 성숙한 성인으로 발전하도록 합니다. 나머지 프로토콜은 생식계열 편집을 위해 F0 주입된 수컷을 스크리닝하는 데 중점을 둡니다.
2. 번식 탱크 설치
- F0 수컷과 암컷 사이에 칸막이가 있는 번식 탱크를 설치하여 짝짓기를 위한 F0 수컷을 프라임합니다.
- 필요한 번식 탱크의 수를 최소화하려면 각 탱크에 두 마리의 암컷 맞은 편에 세 마리의 수컷을 설치하십시오.
- 정자 채취 후 개인으로 효과적으로 선별하고 수용할 수 있는 한 많은 F0 남성을 설정합니다. 암컷의 유전자형은 이 과정에서 배우자가 수집되지 않기 때문에 중요하지 않습니다.
- 암컷 맞은편에 야생형 수컷의 번식 탱크를 하나 설치하십시오. 야생형 정자는 절차 후반부에 결과를 해석하기 위한 제어 역할을 합니다.
- 필요한 번식 탱크의 수를 최소화하려면 각 탱크에 두 마리의 암컷 맞은 편에 세 마리의 수컷을 설치하십시오.
- 준비된 번식 탱크를 밤새 배양하십시오.
3. 정자 채취 절차를 위한 재료 준비
- 작은 유리 그릇을 채우기에 충분한 마취 용액 (생선 물에 0.016 % tricaine-HCl)을 준비하십시오.
- 50mM NaOH 40μL를 0.2mL PCR 튜브에 분주합니다. 수집 할 각 정자 샘플에 대해 하나의 튜브를 준비하십시오. 얼음 위에 보관하십시오.
- 절차 중에 샘플링 할 각 물고기에 대해 개별 0.8 L 탱크를 준비하십시오. 이 탱크는 유전형 분석이 완료될 때까지 샘플링된 물고기를 수용합니다.
참고: 이것은 이 프로토콜에서 효과적으로 선별할 수 있는 물고기의 수를 제한하는 주요 요소입니다. 유전형 분석이 완료될 때까지 샘플링된 개체를 수용할 수 있는 적절한 공간을 확보합니다. - 1인치 x 1인치 스폰지(절차 중에 수컷 제브라피쉬 복부 면이 위로 향하도록 얕은 타원형 디봇으로 자른 것)를 생선 물에 적셔 과도한 액체를 짜냅니다.
- 스폰지를 저배율로 실체 현미경의 시야에 배치합니다(그림 1A).
- 최적의 viewed를 위해 비스듬한 머리 위 조명.
4. 수컷 물고기를 마취시키고 정자를 채취합니다.
- 깨끗한 모세관을 준비하십시오.
- 튜브 용기를 부드럽게 흔들어 단일 모세관을 분리합니다.
- 모세관을 제공된 전구에 넣고 따로 보관합니다.
- 수컷 물고기를 준비된 마취 용액 (물고기 물 중 0.016 % tricaine-HCl)에 옮깁니다.
- 마취 과정을 가속화하기 위해 슬롯 형 스푼으로 용액을 부드럽게 저어줍니다.
- operculum (아가미) 움직임이 느려지면(약 1-2분) 정자 채취를 진행합니다.
- 홈이 있는 스푼을 사용하여 생선을 깨끗한 종이 타월 더미에 옮깁니다.
- 홈이 있는 스푼을 사용하여 생선을 부드럽게 굴려 과도한 물을 제거합니다.
- 깨끗하고 접힌 티슈 페이퍼로 생선을 부드럽게 닦아 남아 있는 물기를 제거합니다.
- 홈이 있는 스푼을 사용하여 생선을 준비된 스폰지로 옮깁니다(그림 1B).
- 절차를 수행하는 사람의 지배적 인 손에 가장 가까운 머리가 있도록 물고기 복부면을 위로 향하게합니다.
- 깨끗하고 접힌 티슈 페이퍼로 항문 지느러미 주변을 부드럽게 닦아냅니다.
- 정자를 모으십시오.
- 모세관의 끝을 사용하여 골반 지느러미를 정중선에서 옆으로 부드럽게 움직여 배설물을 노출시킵니다(그림 1C).
- 모세관의 끝을 배설물 근처에 놓습니다.
- 집게를 사용하여 아가미 바로 아래에서 시작하여 배설물에서 끝나는 물고기의 측면을 부드럽게 짜냅니다(그림 1C').
- 모세관 작용에 의해 모세관으로 정자를 수집합니다. 이 절차에는 약 1μL이면 충분합니다(그림 1D).
참고: 일부 실험실에서는 정자 수집을 위해 모세관 대신 p10 피펫 팁을 사용한다고 보고합니다. - 부드러운 압력으로 정자가 방출되지 않으면 물고기를 시스템 물로 되돌립니다.
- 마취 회복을 모니터링하십시오.
- 물고기를 다치게 할 수 있으므로 정자를 배출하기 위해 물고기를 세게 짜지 마십시오.
- 정자를 다시 채취하기 전에 2주 동안 물고기를 쉬게 하십시오.
- 수집 후, 개별 하우징을위한 시스템 용수가있는 깨끗한 격리 탱크에 물고기를 넣으십시오.
- 마취에서 회복하기 위해 물고기를 모니터하십시오.
- 정자를 다시 채취하기 전에 2주 동안 물고기를 쉬게 하십시오.
- 수집된 정자가 있는 모세관을 준비된 PCR 튜브(50mM NaOH 40μL 포함)에 넣습니다.
- 고무 전구를 꽉 쥐어 수집 된 샘플을 배출하십시오.
- 사용한 모세관은 승인된 유리 폐기물 용기에 폐기하십시오.
- 모든 수컷이 압착 될 때까지 얼음 위에서 샘플을 배양하십시오.
- 각 남성에 대해 4.1-4.5 단계를 반복하십시오.
- 최종 정자 유전형 분석 결과가 추정 돌연변이가 있는 해당 개인으로 추적할 수 있도록 분리 탱크와 정자 샘플에 라벨을 붙입니다.
- 모든 개인을 모니터링하고 탱크를 시스템에 다시 장착하기 전에 똑바로 서서 탱크를 탐색하는지 확인하십시오.
- 프라이밍에 사용되는 암을 시스템의 해당 탱크에 다시 넣습니다.
- 10분 후에도 마취에서 회복되지 않는 물고기가 있으면 실험실과 관련된 기관에서 승인한 안락사 절차를 따르십시오.
- 예: 2-4 °C의 물고기 물이 담긴 얼음 욕조에서 개인을 급속 냉각시킵니다.
- 안락사된 물고기는 실험실과 관련된 기관에서 승인한 절차에 따라 적절한 동물 사체 폐기물 용기에 폐기하십시오.
5. 정자 샘플에서 DNA 추출
- 미니 원심분리기에서 모든 정자 샘플을 간단히 회전시키고 PCR 튜브를 열 순환기에 넣습니다.
- 열 순환기의 뚜껑을 닫습니다.
- 열 순환기에서 다음 설정을 실행합니다.
- 샘플을 95°C에서 40분 동안 가열합니다.
- 샘플을 25°C로 냉각합니다.
- 열 순환기에서 샘플을 제거합니다.
- 각 샘플에 대해 깨끗한 피펫 팁을 사용하여 10μL의 1M Tris-HCl(pH 8로 완충됨)을 추가하여 pH를 중화합니다.
- 위아래로 피펫팅하여 잘 섞습니다.
- 미니 원심분리기에서 샘플을 간단히 회전시킵니다.
- 게놈 DNA 샘플은 4°C에서 며칠 동안 보관하거나 -20°C에서 최대 6개월 동안 보관할 수 있습니다.
6. 원하는 유전자좌의 PCR 증폭(및/또는 제한 분해)
- 표준 PCR 프로토콜을 사용하여 원하는 유전자좌를 증폭합니다.
- 열순환기를 아래의 PCR 반응에서 초기 변성 온도로 예열한다.
- PCR 튜브에서 각 정자 샘플에 대해 25 μL 반응 혼합물을 준비합니다.
12.5 μL의 2x Taq 중합효소 마스터 믹스
1.5μL의 10μM 정방향 프라이머
1.5μL의 10μM 역방향 프라이머
뉴클레아제가 없는 물 4.5μL
중화된 정자 DNA 샘플 5μL - 위아래로 피펫팅하여 잘 섞습니다.
- 미니 원심분리기에서 샘플을 간단히 회전시킵니다.
- s를 배치amp다음 설정으로 예열된 열 순환기에 s:
초기 변성: 95°C에서 3분 동안
변성(30초 동안 95°C), 어닐링(30초 동안 55°C) 및 연장(30초 동안 72°C)의 35 사이클
최종 연장: 72°C에서 5분
참고: 특정 Taq 중합효소 및 PCR 증폭 산물의 예상 길이에 따라 확장 온도 및/또는 시간을 조정해야 할 수도 있습니다. - 열 순환기에서 PCR 샘플을 제거합니다.
- 선택 사항: 겔 전기영동을 위해 소량의 PCR 산물에 대해 기증자별 제한 분해를 수행하고 소화되지 않은 PCR 산물을 최소 5-10μL 남깁니다.
- 0.2mL PCR 튜브에서 30μL 반응을 준비합니다.
정제되지 않은 PCR 산물 10μL
뉴클레아제가 없는 물 15μL
3 μL의 10x 제한 효소 완충액
2 μL의 제한효소 (BstNI는 dnah10 knock-in 대립유전자를 분석하기 위한 대표적인 결과에 사용됨)
참고: 많은 제한 효소는 일반적인 PCR 반응 완충액에서 여전히 활성입니다. 그러나 제한 다이제스트 버퍼를 추가하면 결과를 개선하는 데 도움이 될 수 있습니다. 문제가 발생할 경우 문제 해결을 위해 특정 제조업체 지침을 참조하십시오. - 위아래로 피펫팅하여 잘 섞습니다.
- 미니 원심분리기에서 샘플을 간단히 회전시킵니다.
- 다음 설정을 사용하여 열 순환기에 샘플을 배치합니다.
- PCR 앰플리콘을 60°C에서 1시간 동안 절단합니다.
알림: 절단 온도와 배양 시간은 사용 중인 특정 제한 효소에 맞게 조정해야 할 수 있습니다. - 제조업체가 지시한 경우 필요한 온도와 배양 시간으로 제한 효소를 비활성화합니다.
참고: BstNI 효소는 열 비활성화가 필요하지 않습니다.
- PCR 앰플리콘을 60°C에서 1시간 동안 절단합니다.
- 0.2mL PCR 튜브에서 30μL 반응을 준비합니다.
7. 겔 전기영동을 수행하여 다양한 크기의 PCR 앰플리콘을 분리합니다.
- 500mL의 0.5x TBE 실행 완충액 준비: 50mL의 10x TBE 완충액 농축액을 450mL의 탈이온수에 추가합니다.
- 0.5x TBE 실행 완충액에 4% 아가로스 겔 100mL 준비: 10μL의 10,000x 겔 염색과 함께 4g의 아가로스를 100mL의 0.5x TBE 실행 완충액에 추가합니다. 아가로스 분말이 완전히 용해될 때까지 전자레인지에 돌립니다.
- 젤 용액을 적절한 크기의 젤 캐스팅 프레임에 붓고 젤의 한쪽 면에 빗을 삽입합니다.
알림: 더 큰 젤 크기(권장: 15cm x 15cm)는 amplicons에 젤에서 해결할 수 있는 더 많은 공간이 있습니다.- 제한효소 분해 산물을 분석해야 하는 경우 결과를 비교할 수 있도록 PCR 산물과 동일한 겔에서 실행하는 것이 가장 좋습니다. 모든 샘플에 맞게 필요에 따라 빗 웰 크기를 조정합니다.amp젤에.
- 굳을 때까지 젤을 실온에 두십시오. 빗을 조심스럽게 제거하십시오. 주조 프레임에서 젤을 조심스럽게 제거합니다.
- 젤 용액을 적절한 크기의 젤 캐스팅 프레임에 붓고 젤의 한쪽 면에 빗을 삽입합니다.
- 겔 전기영동 장비를 설정합니다.
- 웰이 음극에 가장 가깝도록 전기 영동 장비에 겔을 배치합니다. 우물이 완전히 잠길 때까지 0.5x TBE 실행 버퍼를 전기 영동 장비에 붓습니다.
- 젤을 넣습니다.
- 5 μL의 DNA ladder를 첫 번째 웰에 로드합니다(PCR 앰플리콘과 크기가 유사한 DNA 단편이 포함된 DNA ladder 선택).
- 각 샘플(야생형 대조군 샘플 포함)에 대해 새 피펫 팁을 사용하여 5-10 μL의 PCR 산물을 나머지 웰에 로드합니다.
- 선택 사항: 제한 분해 제품을 분석해야 하는 경우 이러한 샘플도 겔에 로드합니다.
참고: PCR과 제한 소화 제품 사이에 동일한 양의 DNA를 로드하는 것이 가장 좋습니다. 예를 들어, 30 μL 제한 분해 반응 혼합물에, 10 μL의 PCR 생성물을 첨가하였다. 따라서 소화 산물 15μL의 DNA 농도는 정제되지 않은 PCR 산물 5μL와 비슷했습니다.
- 젤을 실행하십시오.
- 전극을 겔 전기영동 장비에 연결하고 최소 1시간 동안 150V를 적용하여 앰플리콘이 적절하게 분리되도록 합니다.
- 젤을 이미지화하여 DNA 밴드를 봅니다.
참고: 강력하게 돌연변이된 CRISPR 표적 유전자좌는 삽입/삭제 대립유전자와 야생형 앰플리콘의 조합을 나타내는 여러 밴드 크기로 실행됩니다.- 1시간 후에도 겔 밴드가 분해되지 않으면 밴드가 겔에서 충분히 분리될 때까지 15분 간격으로 겔에 150V를 적용합니다.
- 선택 사항: SNP, 에피토프 태그 또는 형광 태그와 같은 정확한 편집의 삽입을 위해 스크리닝하는 경우 원하는 편집에 특정한 PCR 방법으로 정자 DNA를 스크리닝합니다. 예를 들어, 기증자 특이적 제한 부위를 식별할 때, 정자 샘플의 PCR 산물과 비교하여 제한 소화 산물에 추가 밴드가 존재할 것입니다.
8. 생식계열 안정 대립유전자 분리
- 정자 압착 절차에서 2주간의 휴식 후, 정자 게놈 DNA에 원하는 편집이 포함된 F0 개체를 교차시킵니다.
참고: 개별 정자 클론의 정자 생성은 제브라피쉬에서 주기적일 수 있습니다. 따라서 원하는 대립 유전자는 교배 당시 정자를 생산하지 않을 수 있습니다. 이러한 이유로 초기 스크리닝과 관련된 대립 유전자를 분리하기 위해 추가 교차를 두 번 이상 수행해야 할 수 있습니다. - F1 교배된 어류가 성적으로 성숙하면 정자 게놈 DNA를 스크리닝하는 동안 수행된 것과 동일한 PCR 및 겔 전기영동 프로토콜을 사용하여 표준 유전자형 분석 방법(예: 지느러미 클립 DNA)을 진행합니다.
- Sanger는 동일한 원하는 편집을 가진 F1 개체를 식별하기 위해 PCR 산물을 시퀀싱합니다.
- 선택 사항: 원하는 편집으로 인해 제한 사이트가 도입되거나 중단되는 경우 적절한 제한 다이제스트 프로토콜을 사용하여 화면을 표시합니다.
- 동일한 원하는 편집을 가진 교차 F1 이형 접합체.
- 예상되는 표현형 및/또는 게놈 편집에 대해 F2 자손을 스크리닝합니다.
결과
이 프로토콜에 설명된 실험적 접근 방식은 F0 주입된 남성 정자 수집에서 파생된 수천 개의 게놈 분석에 초점을 맞춰 게놈 편집 또는 추정되는 유해한 대립 유전자를 보다 신속하게 식별할 수 있도록 합니다. 그림 2 는 이 프로토콜을 사용하여 얻은 결과를 해석하는 방법을 강조합니다.
p2ry12 유전자좌에서 돌연변이를 생성하기 위해 1세포 단계 제브라피쉬 배아에 Cas9 엔도뉴클레아제와 p2ry12 특이적 sgRNA를 주입했습니다(그림 2B, 회색 강조 표시). F0 주사 된 물고기는 성적으로 성숙 할 때까지 시스템에 넣었고 6 명의 개별 수컷으로부터 정자를 채취했습니다. DNA는 고열 및 염기성 조건(HotSHOT DNA 추출)을 사용하여 정자 샘플에서 추출하고 p2ry12 유전자좌의 PCR 증폭 전에 중화시켰다. PCR 산물을 고전압(150V)에서 1.5시간 동안 고-백분율(4%) 아가로스 겔 상에서 분리하였다. 이 예에서, 야생형 앰플리콘은 길이가 약 250 염기쌍인 단일 밝은 띠로 실행되었습니다(그림 2C; WT)입니다. 대조적으로, 인델을 포함하는 F0 주입된 수컷 앰플리콘은 겔에서 여러 밴드로 실행되었습니다(그림 2C; 레인 #1-2, #5-8). 대안적으로, 돌연변이된 F0-주입된 수컷 앰플리콘은 변경된 겔 이동성을 갖는 단일 밴드로서 실행될 수 있다(p2ry12 예에는 표시되지 않지만, dnah10 F0-male #2의 PCR 산물에서 분명함; 그림 2E). p2ry12 예제 젤에서 샘플 #3 및 샘플 #4가 야생형 밴드와 비교하여 인델을 포함하는지 여부가 덜 명확했기 때문에 이러한 개인이 최고의 창시자 후보가 아닐 수 있습니다. 더 독특한 앰플리콘 변형이 있는 F0 수컷으로부터 대립유전자를 분리하는 것은 4% 아가로스 겔에서 쉽게 점수를 매기기 때문에 안정적인 생식계열 운반체를 전파하는 데 가장 좋습니다. 예를 들어, F0-수컷 #6 정자 샘플은 대립유전자 중 하나에서 큰 결실을 포함하는 것으로 나타났습니다(그림 2C, 레인 6, 증가된 겔 이동성을 갖는 밝은 밴드). 이 큰 결실 대립유전자가 F1 세대에서 선택되었다면, 4% 아가로스 겔 상의 WT 대립유전자와 쉽게 구별될 수 있을 것이다. 대안적으로, 상이한 대립유전자의 집합체가 필요한 경우, F0-수컷 #7은 PCR 산물이 다양한 이동성의 여러 개별 대립유전자를 포함하는 것으로 나타났기 때문에 효과적인 창시자 후보가 될 수 있다. 창시자 수컷이 선택되면, 원하는 대립 유전자는 주사에 사용 된 원래의 야생형 균주로 개인을 다시 교배시킴으로써 분리 될 수 있습니다.
dnah10 유전자에서 특이적 knock-in 돌연변이를 생성하기 위해 1세포 단계 제브라피쉬 배아에 Cas9 엔도뉴클레아제, dnah10 특이적 sgRNA(그림 2D, 회색 강조 표시) 및 원하는 편집(그림 2D; 빨간색) 및 기증자 특이적 BstNI 제한 부위(그림 2D)를 포함하는 DNA 공여체 올리고뉴클레오티드를 주입했습니다 , 대괄호). 이 설계를 통해 PCR 후 제한 다이제스트와 기증자 통합을 쉽게 식별할 수 있습니다. 또한, sgRNA 인식 부위 내의 염기쌍을 변경함으로써(그림 2D; 회색 강조 표시) 이 설계는 공여자 서열의 Cas9 분해를 방지합니다. F0 주사 된 물고기가 성적으로 성숙하면 정자를 채취하고 핫 샷 방법을 사용하여 DNA를 추출했습니다. 이들 샘플로부터, dnah10 유전자좌는 PCR을 사용하여 증폭되었고, 생성물은 고전압(150 V)에서 1시간 동안 높은 비율(4%) 아가로스 겔 상에서 분리되었다. 이 예에서 야생형 앰플리콘은 길이가 약 400 염기쌍인 단일 밝은 띠로 실행되었습니다(그림 2E, 상단 패널: WT). 대조적으로, 인델을 포함하는 F0 주입된 수컷 앰플리콘은 여러 밴드(그림 2E, 상단 패널: 레인 #1, #4 및 #5) 또는 겔 이동성이 감소된 단일 밴드(그림 2E, 상단 패널: 레인 #2)로 실행되었습니다. 정자 샘플에 기증자 통합 대립 유전자가 포함되어 있는지 확인하기 위해 PCR 산물을 BstNI 제한 효소로 1시간 동안 분해하고 산물을 고전압(150V)에서 1시간 동안 4% 아가로스 겔에서 실행했습니다. 소화되지 않은 PCR 산물(그림 2D; 상단 패널)과 소화된 산물(그림 2D; 하단 패널)을 비교하면 공여체 구조가 통합될 가능성이 있는 샘플이 나타납니다. 이 예에서, F0-주입된 수컷 #1은 PCR 산물에 존재하지 않았던 소화된 산물에 추가적인 밴드를 가졌다(도 2D; 하단 패널: 레인 #1, 동그라미로 표시됨). 따라서 이 수컷은 원하는 녹인(knock-in)을 가진 돌연변이 계통을 확립하기 위한 최고의 창시자 후보를 나타냅니다.
추정되는 F0 운반체 수컷이 교배되면 F1 자손에서 대립유전자를 서열로 확인해야 합니다. F1 이형접합 어류에서 유래한 앰플리콘을 직접 Sanger하여 염기서열을 분석한 후 Poly Peak Parser13 과 같은 이형접합 대립유전자 분석 방법을 수행하거나 MiSeq14 또는 Hi-Tom15 시퀀싱과 같은 다양한 차세대 시퀀싱 기반 방법을 사용하는 것이 좋습니다. 이것은 정확한 편집 또는 유해한 indel 돌연변이가 실제로 생식계열로 진행되고 겔 전기영동 분석의 인공물이 아닌지 확인하기 위해 필요하고 보완적인 접근 방식입니다. 실제로, F0 운반체로부터 정자 DNA를 시퀀싱하기 위한 차세대 시퀀싱 접근법의 사용은 이 프로토콜에 설명된 겔 전기영동 분석 대신 쉽게 사용할 수 있습니다. 그러나 쉽게 점수를 매길 수 있는 아가로스 겔 유전형 분석법을 사용하는 것은 제브라피쉬 연구자들의 글로벌 커뮤니티를 위한 보다 경제적이고 평등한 접근 방식입니다.

그림 1: 정자 짜내기 절차를 위한 설정 . (A) 스펀지는 오버헤드 조명이 있는 저배율로 실체현미경 아래에 위치합니다. (B) 타원형 디봇(점선)으로 자른 적신 1 in x 1 in 스폰지는 시술 중에 마취된 수컷 물고기의 복부 면을 위로 향하게 하는 데 사용됩니다. (C) 항문 지느러미(af, 분홍색), 골반 지느러미(pf, 파란색) 및 배설물(화살표)을 묘사한 마취된 수컷 물고기의 복부 쪽의 해부학으로, 시술 중에 정자가 배출됩니다. (씨') 필터 집게는 마취 된 수컷 물고기를 아가미에서 배설물로 부드럽게 짜내는 데 사용되며, 배출 된 정자는 모세관 작용 (흰색 화살표)에 의해 유리 피펫으로 끌어 들입니다. (D) 다운스트림 DNA 추출 및 분석을 위해 충분히 배출된 정자(불투명한 액체, 검은색 브래킷)를 포함하는 모세관. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
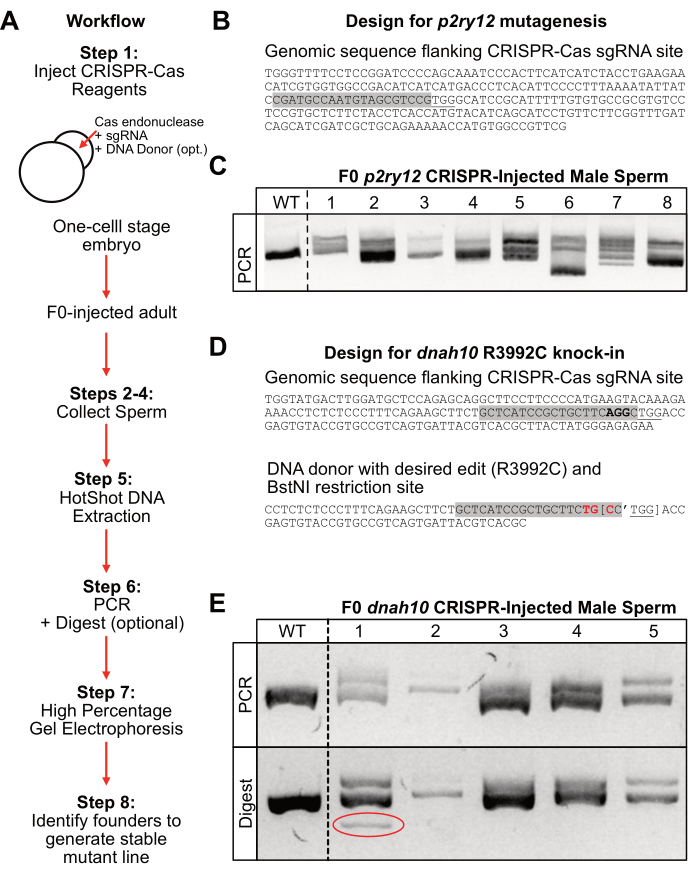
그림 2: 워크플로 및 대표 결과. (A) 프로토콜의 일반적인 워크플로. (B) Cas9 특이적 PAM 부위(밑줄 그은)가 있는 p2ry12 sgRNA 부위(회색 강조 표시)의 대표 디자인. (C) 150V에서 1.5시간 후 4% 겔 전기영동의 대표적인 결과. 야생형(WT) 대조군 및 F0-p2ry12 CRISPR 주입 남성 정자 샘플(1-8) PCR 증폭 후. (D) 표적 코돈 근처에 Cas9 특이적 PAM 부위(밑줄이 그어진 것)가 있는 dnah10 sgRNA 부위(회색 강조 표시)의 대표적인 디자인(굵은 글씨). 원하는 코돈 편집(빨간색 굵은 글씨) 및 BstNI 제한 부위(절단 부위를 표시하는 아포스트로피가 있는 괄호)가 있는 DNA 기증자 서열. (E) 150 V에서 1시간 후 4% 겔 전기영동의 대표적인 결과. 위: 야생형(WT) 및 F0 dnah10 CRISPR 주입 남성 정자 샘플의 PCR 산물(1-5). 하단: 위 샘플의 BstNI 제한 다이제스트 제품. 샘플 1은 소화 후 추가 밴드(빨간색 원)를 기반으로 하는 공여자 구축물의 성공적인 통합을 보여줍니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
이 프로토콜은 F0 남성 정자 게놈에 대한 집중 분석을 통해 CRISPR-Cas 기술을 사용하여 추정 게놈 편집 또는 표적 돌연변이를 신속하게 특성화하는 방법을 설명합니다. 이 프로토콜은 안락사 없이 정자를 샘플링할 수 있는 다른 동물 모델에 적용할 수 있어야 합니다. 이 방법은 원하는 편집에 대한 스크리닝 처리량을 증가시키며 드문 HDR 매개 knock-in 이벤트를 식별하는 데 특히 유용합니다. 이 접근법은 또한 추정 F0 보균자로부터 하나의 정자 샘플에서 잠재적으로 수천 개의 게놈을 신속하게 스크리닝하는 것을 용이하게 함으로써 안정적인 생식계열 편집을 찾는 데 사용되는 실험 동물의 수를 줄이는 역할을 하며, 이는 추정 F0 보균자의 교차에서 파생된 수백 개의 배아를 스크리닝해야 할 수 있는 보다 전통적인 접근 방식과 대조됩니다.
이 프로토콜은 제브라피쉬 7,10,14,16,17,18의 정자 수집을 위해 확립된 프로토콜을 기반으로 하며, 고해상도 겔 전기영동을 사용하여 생식계열 편집을 식별하는 재현 가능한 기술을 포함합니다. 이 접근법은 모든 표준 CRISPR/Cas 워크플로우에 쉽게 통합되어 표적 게놈 편집의 스크리닝 및 분리를 위한 처리량을 높일 수 있습니다. 또한 이 프로토콜은 다양한 교육과 경험을 갖춘 직원이 있는 실험실과 교육 실험실에 적합합니다. 그러나 우리의 정자 수집 방법은 이전 간행물10,17에서 전문적으로 설명된 냉동 보존에 충분하지 않습니다.
이 프로토콜은 고분해능 아가로스 겔 전기영동을 사용하여 원하는 인델 및 정확한 게놈 돌연변이의 추정 수컷 운반체를 식별합니다. 그러나, 정자 게놈 DNA는 형광 단편 분석 또는 바코드 시퀀싱(barcoded sequencing)14, 고분해능 용융 분석(high-resolution melt analysis)18, 또는 표준 겔 전기영동 접근법을 이용한 형광 태그 또는 에피토프의 간단한 앰플리콘 검출(amplicon detection of fluorescent tag or epitopes)을 포함한 무수히 많은 다른 접근법을 사용할 수 있다. Sanger 기반 또는 차세대 시퀀싱과 같은 접근 방식을 사용하는 모든 대립 유전자의 다운스트림 검증은 추정 대립 유전자에 대한 실험 작업을 시작하기 전에 수행해야 합니다. 실제로, 차세대 염기서열 분석 접근법14 을 사용하여 배아에서 돌연변이를 스크리닝하기 위한 기존의 방법은 이 프로토콜에 기술되어 있는 아가로스 겔에 대한 분석의 필요성을 회피할 수 있다. 그러나 모든 실험실이 각 돌연변이 균주에 대한 분리 및 실험 단계에서 저렴한 차세대 염기서열 분석 방법에 쉽게 접근할 수 있는 것은 아니라는 점을 감안할 때 겔 스코어링 가능한 유전형 분석법을 사용하는 것이 더 비용 효율적이고 평등한 접근 방식입니다.
요약하면, 이 프로토콜은 CRISPR/Cas 편집 남성의 정자 게놈을 재현 가능하게 스크리닝하기 위한 단계별 지침을 제공하여 원하는 편집을 스크리닝하는 데 더 적은 수의 교차와 배아의 PCR 스크리닝이 필요하지 않습니다. 이 방법을 적용하면 편집된 관심 대립 유전자를 성공적으로 식별하기 위해 생성 및 분석해야 하는 물고기의 수가 줄어들고 안정적인 라인을 생성하는 인력의 시간과 비용도 줄어듭니다.
공개
없음.
감사의 말
핫샷 방법을 사용하여 양질의 정자 게놈 DNA를 얻기 위한 초기 노력에 대해 Washington University School of Medicine의 Anna Hindes에게 감사드립니다. 이 연구는 국립 보건원 (National Institutes of Health)의 국립 관절염 및 근골격계 및 피부 질환 연구소 (National Institute of Arthritis and Musculoskeletal and Skin Diseases)가 상을 수상했습니다 (R01AR072009에서 RSG).
자료
| Name | Company | Catalog Number | Comments |
| Agarose powder | Fisher BioReagents | BP1356-100 | |
| Breeding tanks | Carolina Biological | 161937 | |
| BstNI Restriction Enzyme | NEB | R0168S | |
| Cas9 Endonuclease | IDT | 1081060 | |
| DNA Ladder, 100 bp | Thermo Scientific | FERSM0241 | |
| dnah10 donor construct | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry. Phosphorothioate bond on the donor at the first three phosphate bonds on both the 5’ and 3’ ends (5'-CCTCTCTCCCTTTCAGAAGCTTC TGCTCATCCGCTGCTTCTGCCT GGACCGAGTGTACCGTGCCGTC AGTGATTACGTCACGC-3') | |
| dnah10 forward primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CATGGAACTCTTTCCTAATGAGT TTGGC-3') | |
| dnah10 reverse primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry ('5-AGTAGAGATCACACATCAACAGA ATACAGC-3') | |
| dnah10 synthetic sgRNA | Synthego | Synthetic sgRNA, target sequence: 5'-GCTCATCCGCTGCTTCAGGC-3' | |
| Electrophoresis power supply | Thermo Scientific | 105ECA-115 | |
| Filter forceps | Millipore | XX6200006P | |
| Fish (system) water | Generic | n/a | |
| Gel electrophoresis system (including casting frame, comb, and electrophoresis chamber) | Thermo Scientific | B2 | |
| Gel imaging light box | Azure Biosystems | AZI200-01 | |
| Gel stain, 10000X | Invitrogen | S33102 | |
| Glass bowl, 250 mL | Generic | n/a | |
| Isolation tanks, 0.8 L | Aquaneering | ZT080 | |
| Microcap capillary tube with bulb, 20 µL | Drummond | 1-000-0020/CA | |
| Minicentrifuge | Bio-Rad | 12011919EDU | |
| Micropipettes, various with appropriate tips | Generic | n/a | |
| Microwave | Generic | n/a | |
| Nuclease free water | Promega | P119-C | |
| Paper towels | Generic | n/a | |
| PCR tubes, 0.2 mL | Bioexpress | T-3196-1 | |
| Plastic spoon, with drilled holes/slots | Generic | n/a | |
| KCl solution, 0.2 M RNAse Free | Sigma-Aldrich | P9333 | |
| p2ry12 forward primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CCCAAATGTAATCCTGACCAGT -3') | |
| p2ry12 reverse primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CCAGGAACACATTAACCTGGAT -3') | |
| p2ry12 synthetic sgRNA | Synthego | Synthetic sgRNA, target sequence: 5'-GGCCGCACGAGGTCTCCGCG-3' | |
| Restriction Enzyme 10X Buffer | NEB | B6003SVIAL | |
| NaOH solution, 50 mM | Thermo Scientific | S318; 424330010 | |
| Sponge, 1-inch x 1-inch cut with small oval divot | Generic | n/a | |
| Stereomicroscope | Zeiss | Stemi 508 | |
| Taq polymerase master mix, 2X | Promega | M7122 | |
| TBE Buffer Concentrate, 10X | VWR | E442 | |
| Thermal Cycler | Bio-Rad | 1861096 | |
| Tissue paper | Fisher Scientific | 06-666 | |
| Tricaine-methanesulfonate solution (Syncaine, MS-222), 0.016% in fish water (pH 7.0±0.2) | Syndel | 200-266 | |
| Tris Base, 1M (Buffered with HCl to ph 8.0) | Promega | H5131 |
참고문헌
- Auer, T. O., Duroure, K., De Cian, A., Concordet, J. P., Del Bene, F. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair. Genome Research. 24 (1), 142-153 (2014).
- Hwang, W. Y., et al. Efficient genome editing in zebrafish using a CRISPR-Cas system. Nature Biotechnology. 31 (3), 227-229 (2013).
- Jao, L. E., Wente, S. R., Chen, W. Efficient multiplex biallelic zebrafish genome editing using a CRISPR nuclease system. Proceedings of the National Academy of Sciences of the United States of America. 110 (34), 13904-13909 (2013).
- Troutwine, B. R., et al. The Reissner fiber is highly dynamic in vivo and controls morphogenesis of the spine. Current Biology. 30 (12), 2353-2362 (2020).
- Xu, Y., Li, Z. CRISPR-Cas systems: Overview, innovations and applications in human disease research and gene therapy. Computational and Structural Biotechnology Journal. 18, 2401-2415 (2020).
- Creaser, C. W. The technic of handling the zebrafish (Brachydanio rerio) for the production of eggs which are favorable for embryological research and are available at any specified time throughout the year. Copeia. 1930 (4), 159-161 (1934).
- Carmichael, C., Westerfiel, M., Varga, Z. M. Cryopreservatin and in vitro fertilization at the zebrafish international resource center. Methods in Molecular Biology. 546, 45-65 (2009).
- Wang, Y., Troutwine, B. R., Zhang, H., Gray, R. S. The axonemal dynein heavy chain 10 gene is essential for monocilia motility and spine alignment in zebrafish. Developmental Biology. 482, 82-90 (2021).
- Gray, R. S., et al. Postembryonic screen for mutations affecting spine development in zebrafish. Developmental Biology. 471, 18-33 (2021).
- Brocal, I., et al. Efficient identification of CRISPR/Cas9-induced insertions/deletions by direct germline screening in zebrafish. BMC Genomics. 17, 259 (2016).
- Davis, M. W., Jorgensen, E. M. ApE, A Plasmid Editor: A freely available DNA manipulation and visualization program. Frontiers in Bioinformatics. 2, 816619 (2022).
- Labun, K., et al. CHOPCHOP v3: Expanding the CRISPR web toolbox beyond genome editing. Nucleic Acids Research. 47, 171-174 (2019).
- Hill, J. T., et al. Poly Peak Parser: Method and software for identification of unknown indels using sanger sequencing of polymerase chain reaction products. Developmental Dynamics. 243 (12), 1632-1636 (2014).
- Varshney, G. K., et al. High-throughput gene targeting and phenotyping in zebrafish using CRISPR/Cas9. Genome Research. 25 (7), 1030-1042 (2015).
- Liu, Q., et al. Hi-TOM: A platform for high-throughput tracking of mutations induced by CRISPR/Cas systems. Science China. Life Sciences. 62 (1), 1-7 (2019).
- Sorlien, E. L., Witucki, M. A., Ogas, J. Efficient production and identification of CRISPR/Cas9-generated gene knockouts in the model system Danio rerio. Journal of Visualized Experiments. (138), e56969 (2018).
- Draper, B. W., Moens, C. B. A high-throughput method for zebrafish sperm cryopreservation and in vitro fertilization. Journal of Visualized Experiments. (29), e1395 (2009).
- Parant, J. M., George, S. A., Pryor, R., Wittwer, C. T., Yost, H. J. A rapid and efficient method of genotyping zebrafish mutants. Developmental Dynamics. 238 (12), 3168-3174 (2009).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유