Method Article
فحص الحيوانات المنوية للعزل السريع لتعديلات الخط الجرثومي في الزرد
In This Article
Summary
أحدثت تقنيات كريسبر-كاس ثورة في مجال تحرير الجينوم. ومع ذلك ، فإن العثور على تعديل الخط الجرثومي المطلوب وعزله لا يزال يمثل عنق الزجاجة الرئيسي. لذلك ، يصف هذا البروتوكول طريقة قوية لفحص الحيوانات المنوية لأسماك الزرد المحقونة ب F0 CRISPR بسرعة لإجراء تعديلات على الخط الجرثومي باستخدام تقنيات تفاعل البوليميراز المتسلسل القياسية وهضم التقييد والرحلان الكهربائي الهلامي.
Abstract
أحدث ظهور تقنيات CRISPR-Cas nuclease المستهدفة ثورة في القدرة على إجراء تحرير دقيق للجينوم في كل من أنظمة النماذج الراسخة والناشئة. تستخدم أنظمة تحرير الجينوم CRISPR-Cas الحمض النووي الريبي الإرشادي الاصطناعي (sgRNA) لاستهداف نوكلياز داخلي مرتبط ب CRISPR (Cas) إلى مواقع الحمض النووي الجينومية المحددة ، حيث يولد Cas endonuclease كسرا مزدوجا. يؤدي إصلاح فواصل الخيوط المزدوجة بواسطة آليات معرضة للخطأ الجوهري إلى عمليات الإدراج و / أو الحذف ، مما يؤدي إلى تعطيل الموضع. بدلا من ذلك ، يمكن أن يؤدي إدراج متبرعين بالحمض النووي المزدوج أو قليل النيوكليوتيدات أحادية الشريط من الحمض النووي في هذه العملية إلى إدراج تعديلات دقيقة للجينوم تتراوح من تعدد أشكال النوكليوتيدات المفردة إلى العلامات المناعية الصغيرة أو حتى تركيبات البروتين الفلوري الكبيرة. ومع ذلك ، يمكن أن يكون عنق الزجاجة الرئيسي في هذا الإجراء هو العثور على التعديل المطلوب في الخط الجرثومي وعزله. يحدد هذا البروتوكول طريقة قوية لفحص وعزل طفرات الخط الجرثومي في مواقع محددة في Danio rerio (الزرد). ومع ذلك ، قد تكون هذه المبادئ قابلة للتكيف في أي نموذج حيث يمكن جمع الحيوانات المنوية في الجسم الحي .
Introduction
يعد نظام CRISPR (التكرارات العنقودية القصيرة المتباعدة بانتظام) / Cas أداة قوية لإجراء طفرات خاصة بالموقع وتحرير الجينوم الدقيق في نظام نموذج Danio rerio (الزرد)1،2،3،4. يتكون Cas-ribonucleoprotein (RNP) من مكونين رئيسيين: Cas endonuclease (عادة Cas9 أو Cas12a) و RNA دليل اصطناعي خاص بالموقع (sgRNA) 5. معا ، يولد Cas-RNP استراحة مزدوجة تقطعت بهم السبل (DSB) في الموضع المطلوب الذي يمكن إصلاحه بواسطة إحدى آليتي الإصلاح الجوهريتين. آلية إصلاح الانضمام النهائي غير المتماثل (NHEJ) عرضة للخطأ وغالبا ما تؤدي إلى مجموعة متنوعة من عمليات الإدراج أو الحذف (indels) حول DSB. يمكن أن تكون هذه الإندل ضارة إذا أدخلت طفرة في إزاحة الإطار أو توقفا سابقا لأوانه في تسلسل البروتين الناتج. بدلا من ذلك ، تستخدم آلية الإصلاح الموجه بالتماثل (HDR) نموذجا مانحا مع مناطق التماثل المحيطة بموقع DSB لإصلاح الضرر. يمكن للباحثين الاستفادة من نظام HDR لتوليد تعديلات جينومية دقيقة. على وجه التحديد ، يمكنهم المشاركة في حقن بنية مانحة مزدوجة الحمض النووي التي تقطعت بها السبل والتي تحتوي على التعديلات المطلوبة بالإضافة إلى مناطق التماثل التي تحيط بموقع DSB في الجينوم. وقد أدى الاقتصاد المتزايد في الحجم لمكونات كريسبر المنتجة تجاريا إلى تقليل الحواجز التي تحول دون فحص مواقع متعددة وإقامة جهود واسعة النطاق لتحرير الجينوم بدقة. ومع ذلك ، في النماذج الحيوانية التي تتكاثر جنسيا ، يتمثل عنق الزجاجة الرئيسي في تحديد وعزل الحيوانات الطافرة المستقرة للخط الجرثومي.
يظهر نظام نموذج الزرد العديد من الصفات الرئيسية التي تعزز استخدامه في الدراسات الجينية العكسية. من السهل تربيتها بأعداد كبيرة مع معدات الإسكان المائية الأساسية ، وتظهر الإناث خصوبة عالية على مدار السنة6. علاوة على ذلك ، فإن وضع البيض الخارجي والتسميد يجعلها قابلة للحقن الدقيق لمكونات CRISPR / Cas. عادة ما يتم حقن Cas-RNP في أجنة الزرد ذات المرحلة الواحدة لتوليد DSBs / إصلاح ، من الناحية النظرية ، موروثة من قبل جميع الخلايا الوليدة. ومع ذلك ، تتطلب الجينومات ثنائية الصيغة الصبغية حدثين DSB / إصلاح لإحداث طفرة في كلا الكروموسومين المتماثلين. علاوة على ذلك ، على الرغم من حقن Cas-RNP في مرحلة الخلية الواحدة ، فقد لا يحدث DSB / الإصلاح حتى نقاط لاحقة في التطوير. تساهم هذه العوامل معا في الطبيعة الفسيفسائية للأسماك المحقونة ب F0. من الممارسات الشائعة تهجين الأسماك المحقونة ب F0 وفحص ذرية F1 بحثا عن تعديلات indels / محددة. ومع ذلك ، نظرا لعدم امتلاك جميع الأسماك المحقونة ب F0 طفرات في الخط الجرثومي ، فإن هذه الممارسة تؤدي إلى العديد من التهجينات غير المنتجة التي لا تولد التعديل المطلوب. يزيد فحص الخط الجرثومي F0 بدلا من الأنسجة الجسدية F1 من احتمالية عزل تحرير الخط الجرثومي المطلوب ويقلل من عدد الحيوانات المطلوبة في هذه العملية.
يمكن بسهولة جمع الحيوانات المنوية من الزرد المحقون ب F0 دون الحاجة إلى القتل الرحيم. تسمح هذه الميزة بالحفظ بالتبريد وإعادة اشتقاق مخزون الحيوانات المنوية المجمدة7 ولكن يمكن أيضا استغلالها لفحص وتحديد وعزل ناقلات الخط الجرثومي للطفرات الجينومية المرغوبة بسرعة 8,9. وصف Brocal et al. (2016) سابقا طريقة قائمة على التسلسل لفحص تعديلات الخط الجرثومي في ذكور الزرد 10 المحقونة بF0. على الرغم من أن هذا النهج مفيد لتحديد الأليلات الطافرة الموجودة في الخط الجرثومي ، إلا أنه يمكن أن يصبح مكلفا في الإنتاجية العالية وقد لا يكون متاحا لجميع المختبرات. في المقابل ، يقدم البروتوكول الحالي استراتيجية سهلة الاستخدام وفعالة من حيث التكلفة قائمة على الرحلان الكهربائي لتحديد تعديلات الخط الجرثومي. على وجه التحديد ، يحدد هذا البروتوكول طريقة قوية لفحص وعزل طفرات الخط الجرثومي في مواقع محددة باستخدام الرحلان الكهربائي لهلام الأغاروز عالي الدقة. بالإضافة إلى ذلك ، يصف هذا البروتوكول استراتيجية مماثلة لتحديد التكامل الناجح لبنية مانحة تحتوي على تعديلات محددة. كما هو الحال دائما ، إذا كانت هناك حاجة إلى تعديلات محددة ، فيمكن تنفيذ الاستراتيجيات القائمة على التسلسل جنبا إلى جنب مع البروتوكول الموضح أدناه. على الرغم من أن هذا البروتوكول خاص بنظام نموذج الزرد ، إلا أن هذه المبادئ يجب أن تكون قابلة للتكيف مع أي نموذج يكون فيه جمع الحيوانات المنوية إجراء روتينيا. معا ، ستسمح هذه الاستراتيجيات بتحديد الذكور المحقونين ب F0 مع indels / edits الخط الجرثومي التي يمكن حلها على هلام بعد تفاعل البوليميراز المتسلسل القياسي (PCR) و / أو هضم التقييد.
Protocol
أجريت هذه الدراسة بما يتماشى مع المبادئ التوجيهية الواردة في دليل رعاية واستخدام المختبر للمعاهد الوطنية للصحة. تمت الموافقة على البروتوكول من قبل جامعة تكساس في لجنة أوستن لرعاية واستخدام الحيوان (AUP-2021-00254).
1. تصميم sgRNA لطفرات كريسبر
- الحصول على تسلسل إكسون الذي يحتوي على المواقع المستهدفة.
- صمم دليلا اصطناعيا للحمض النووي الريبي (sgRNA) مع موقع شكل مجاور أولي (PAM) خاص ب Cas endonuclease.
- في حالة المشاركة في حقن بنية مانحة ، صمم sgRNA بحيث يكون موقع القطع المتوقع أقرب ما يمكن إلى التعديلات المطلوبة.
ملاحظة: APE هو برنامج مجاني مع أداة للعثور على مواقع Cas9 sgRNA11. بدلا من ذلك ، يمكن استخدام أدوات مثل CHOPCHOP ، والتي توفر واجهة عبر الإنترنت لتصميم sgRNAs التي تأخذ في الاعتبار مواقع PAM المختلفة ل Cas9 وغيرها من نوكلياز Cas12.
- في حالة المشاركة في حقن بنية مانحة ، صمم sgRNA بحيث يكون موقع القطع المتوقع أقرب ما يمكن إلى التعديلات المطلوبة.
- صمم مواد أولية للأمام وللخلف لتضخيم المنطقة المحيطة بموقع القطع CRISPR / Cas المصمم. يجب أن يكون طول منتج تفاعل البوليميراز المتسلسل حوالي 200-400 زوج أساسي (bp) ليتمكن من حل indels الصغيرة على هلام لاحقا في البروتوكول.
ملاحظة: يقوم CHOPCHOP11 تلقائيا بتصميم مواد أولية لكل sgRNA ينتجها. - اختياري: تصميم بنية مانحة للحمض النووي مع التعديل (التعديلات) المطلوبة.
- احصل على التسلسل الجينومي للإكسون الذي يحتوي على مواضع الطفرة المطلوبة.
ملاحظة: يمكن أن تختلف التسلسلات الداخلية بين الأفراد ، لذلك من الأفضل تجنب تضمين التسلسلات الإنترونية في بنية المتبرع لأن هذا قد يتداخل مع مسار الإصلاح المعتمد على التماثل. - إذا كنت ترغب في تغيير معين في الأحماض الأمينية ، فقم بتغيير الكودون المحدد وفقا لذلك.
ملاحظة: عند إجراء هذه التغييرات، كن على دراية بتردد الكودون. إذا أمكن ، اختر كودون عالي الاستخدام. - لمنع هضم كريسبر / كاس للبنية المانحة ، قم بعمل طفرات مرادفة إضافية (i) تعطل PAM و / أو (ii) تعطل تسلسل sgRNA ، مع تفضيل أعلى لإجراء تغييرات غير مرادفة أقرب إلى موقع PAM.
- استخدم مكتشف موقع التقييد (يحتوي APE11 أيضا على هذه الميزة) للعثور على مواقع تقييد فريدة (تلك التي تحدث مرة واحدة فقط) في منتج PCR المتوقع لتسلسل النوع البري.
- كرر هذه العملية لمنتج تفاعل البوليميراز المتسلسل المتوقع الذي يعكس التكامل الناجح للبنية المانحة.
- قارن قائمة مواقع التقييد بين التسلسلات من النوع البري والتسلسلات المتكاملة للمانحين للعثور على موقع تقييد موجود فقط في التسلسل المحرر.
- إذا لم يتم العثور على موقع تقييد فريد ، فاستمر في إجراء تغييرات مرادفة داخل بنية الجهة المانحة حتى يتم تحقيق ذلك.
ملاحظة: إذا أمكن ، استمر في عمل طفرات مرادفة من شأنها تعطيل ارتباط sgRNA ببناء المتبرع.
- قم بقص تسلسل المانحين إلى حجم يمكن التحكم فيه (50-100 نقطة أساس) مع ما لا يقل عن 20 نقطة أساس من التماثل المحيط بموقع قطع CRISPR / Cas المتوقع وتعديلاته ، إن أمكن.
- احصل على التسلسل الجينومي للإكسون الذي يحتوي على مواضع الطفرة المطلوبة.
- احصل على قليل النوكليوتيدات المصمم sgRNA و / أو DNA المانحة ، بالإضافة إلى أي مواد أولية مطلوبة للتنميط الجيني.
ملاحظة: لمنع هضم قليل النيوكليوتيدات المانحة للحمض النووي بواسطة النيوكليازات ، يوصى بتعديل روابط الفوسفات الثلاثة الأولى على نهايات 5 'و 3' من oligonucleotides الحمض النووي مع الفوسفورثيوات. - حقن 1 نانولتر من مزيج الحقن في أجنة مرحلة خلية واحدة. عادة ، يحتوي مزيج الحقن القياسي على ما يلي:
1 ميكرولتر من 25 ميكرومتر sgRNA الخاص بالموقع
1 ميكرولتر من 25 ميكرومتر كاس9 نوكلياز
اختياري: 1 ميكرولتر من 3 ميكرومتر بنية مانح الحمض النووي- ارفع الحجم الإجمالي إلى 5 ميكرولتر مع KCl الخالي من RNase 0.1 M.
- اسمح للأفراد الذين تم حقنهم ب F0 بالتطور إلى بالغين ناضجين جنسيا. يركز البروتوكول المتبقي على فحص الذكور المحقونة ب F0 لتعديلات الخط الجرثومي.
2. إعداد خزانات التربية
- ذكور F0 الرئيسية للتزاوج عن طريق إنشاء خزانات تربية مع فواصل بين ذكور وإناث F0.
- لتقليل عدد خزانات التربية المطلوبة ، قم بإعداد ثلاثة ذكور مقابل إناث في كل حوض.
- قم بإعداد أكبر عدد ممكن من الذكور F0 الذين يمكن فحصهم وإيواؤهم بشكل فعال كأفراد بعد جمع الحيوانات المنوية. النمط الوراثي للإناث ليس مهما لأنه لن يتم جمع الأمشاج أثناء هذا الإجراء.
- قم بإعداد خزان تربية واحد من الذكور من النوع البري مقابل الإناث. ستعمل الحيوانات المنوية من النوع البري كعنصر تحكم لتفسير النتائج لاحقا في الإجراء.
- لتقليل عدد خزانات التربية المطلوبة ، قم بإعداد ثلاثة ذكور مقابل إناث في كل حوض.
- احتضان خزانات التربية المعدة بين عشية وضحاها.
3. تحضير المواد اللازمة لإجراء جمع الحيوانات المنوية
- قم بإعداد محلول تخدير كاف (0.016٪ تريكايين-حمض الهيدروكلوريك في ماء السمك) لملء وعاء زجاجي صغير.
- قم بتوزيع 40 ميكرولتر من 50 mM NaOH في أنابيب PCR سعة 0.2 مل. قم بإعداد أنبوب واحد لكل عينة من الحيوانات المنوية التي سيتم جمعها. تخزينها على الجليد.
- قم بإعداد خزانات فردية سعة 0.8 لتر لكل سمكة سيتم أخذ عينات منها أثناء الإجراء. ستؤوي هذه الخزانات الأسماك التي تم أخذ عينات منها حتى يكتمل التنميط الجيني.
ملاحظة: هذا هو العامل الأساسي الذي يحد من عدد الأسماك التي يمكن فحصها بشكل فعال في هذا البروتوكول. تأكد من وجود مساحة كافية لإيواء الأفراد الذين تم أخذ عينات منهم حتى اكتمال التنميط الجيني. - بلل إسفنجة مقاس 1 بوصة × 1 بوصة (مقطوعة بأنبوب ضحل بيضاوي الشكل لتثبيت الجانب البطني لذكر الزرد أثناء العملية) في ماء السمك والضغط على السائل الزائد.
- ضع الإسفنجة في مجال رؤية المجهر المجسم عند التكبير المنخفض (الشكل 1 أ).
- ضع الإضاءة العلوية المائلة للحصول على عرض مثالي.
4. تخدير ذكور الأسماك وجمع الحيوانات المنوية
- تحضير أنبوب شعري نظيف.
- هز وعاء الأنبوب برفق لتحرير أنبوب شعري واحد.
- ضع الأنبوب الشعري في المصباح المقدم، وضعه جانبا.
- نقل سمكة الذكور إلى محلول التخدير المحضر (0.016٪ تريكايين-حمض الهيدروكلوريك في ماء السمك).
- حرك المحلول برفق بملعقة ذات فترة زمنية محددة لتسريع عملية التخدير.
- بمجرد أن تتباطأ حركة الخياشيم (الخيشومية) (حوالي 1-2 دقيقة) ، تابع جمع الحيوانات المنوية.
- استخدم الملعقة ذات فترة زمنية محددة لنقل السمك إلى كومة نظيفة من المناشف الورقية.
- استخدم الملعقة ذات فترة زمنية محددة للف السمك برفق لإزالة الماء الزائد.
- امسح السمك برفق بمناديل ورقية نظيفة ومطوية لإزالة أي ماء متبقي.
- استخدم الملعقة ذات فترة زمنية محددة لنقل السمك إلى الإسفنجة المعدة (الشكل 1 ب).
- ضع الجانب البطني للسمكة لأعلى مع وضع رأسها بالقرب من اليد المهيمنة للشخص الذي يقوم بإجراء العملية.
- امسح المنطقة المحيطة بالزعانف الشرجية برفق بمناديل ورقية نظيفة ومطوية.
- جمع الحيوانات المنوية.
- باستخدام نهاية الأنبوب الشعري ، حرك زعانف الحوض برفق جانبيا بعيدا عن خط الوسط لكشف العباءة (الشكل 1C).
- ضع نهاية الأنبوب الشعري بالقرب من العباءة.
- باستخدام الملقط ، اضغط برفق على جوانب السمكة بدءا من أسفل الخياشيم مباشرة وتنتهي عند العباءة (الشكل 1 ج).
- جمع الحيوانات المنوية في الأنبوب الشعري عن طريق العمل الشعري. يكفي حوالي 1 ميكرولتر لهذا الإجراء (الشكل 1 د).
ملاحظة: أبلغت بعض المختبرات عن استخدام أطراف ماصة p10 بدلا من الأنابيب الشعرية لجمع الحيوانات المنوية. - إذا لم يتم إطلاق الحيوانات المنوية بضغط لطيف ، فأعد الأسماك إلى ماء النظام.
- مراقبة للتعافي من التخدير.
- لا تضغط على الأسماك بقوة لطرد الحيوانات المنوية ، لأن هذا يمكن أن يصيب الأسماك.
- راحة الأسماك لمدة 2 أسابيع قبل محاولة جمع الحيوانات المنوية مرة أخرى.
- بعد التجميع ، ضع الأسماك في خزان عزل نظيف مع مياه النظام للسكن الفردي.
- مراقبة الأسماك للتعافي من التخدير.
- راحة الأسماك لمدة 2 أسابيع قبل محاولة جمع الحيوانات المنوية مرة أخرى.
- ضع الأنبوب الشعري مع الحيوانات المنوية المجمعة في أنبوب PCR المحضر (مع 40 ميكرولتر من 50 mM NaOH).
- اضغط على المصباح المطاطي لطرد العينة التي تم جمعها.
- تخلص من الأنبوب الشعري المستخدم في حاوية نفايات زجاجية معتمدة.
- احتضان العينات على الجليد حتى يتم ضغط جميع الذكور.
- كرر الخطوات 4.1-4.5 لكل ذكر على حدة.
- قم بتسمية خزانات العزل وعينات الحيوانات المنوية بحيث يمكن تتبع نتائج التنميط الجيني النهائي للحيوانات المنوية إلى الفرد المقابل مع الطفرات المفترضة.
- راقب جميع الأفراد ، وتأكد من أنهم منتصبون ويستكشفون دباباتهم قبل إعادة الدبابات إلى النظام.
- ضع الإناث المستخدمة في التحضير مرة أخرى في خزاناتها على النظام.
- إذا لم تتعافى أي سمكة من التخدير بعد 10 دقائق ، فاتبع إجراء القتل الرحيم المعتمد من قبل المؤسسة المرتبطة بالمختبر.
- مثال: قم بتبريد الفرد بسرعة في حمام جليدي بماء السمك الذي يتراوح بين 2-4 درجة مئوية.
- التخلص من الأسماك التي تم قتلها رحيما في حاوية نفايات ذبيحة الحيوانات المناسبة وفقا للإجراء المعتمد من قبل المؤسسة المرتبطة بالمختبر.
5. استخراج الحمض النووي من عينات الحيوانات المنوية
- قم بتدوير جميع عينات الحيوانات المنوية لفترة وجيزة في جهاز طرد مركزي صغير ، وضع أنابيب PCR في جهاز تدوير حراري.
- أغلق غطاء جهاز الدورة الحرارية.
- قم بتشغيل الإعدادات التالية في جهاز تدوير الحرارة:
- سخني العينات لمدة 40 دقيقة عند 95 درجة مئوية.
- تبريد العينات إلى 25 درجة مئوية.
- قم بإزالة العينات من جهاز التدوير الحراري.
- باستخدام طرف ماصة نظيف لكل عينة ، قم بتحييد الرقم الهيدروجيني بإضافة 10 ميكرولتر من 1 M Tris-HCl (مخزن مؤقتا إلى الرقم الهيدروجيني 8).
- تخلط جيدا عن طريق سحب الماصة لأعلى ولأسفل.
- تدور لفترة وجيزة أسفل العينات في جهاز طرد مركزي صغير.
- يمكن تخزين عينات الحمض النووي الجينومي لعدة أيام عند 4 درجات مئوية أو وضعها عند -20 درجة مئوية لمدة تصل إلى 6 أشهر.
6. تضخيم تفاعل البوليميراز المتسلسل (و / أو هضم التقييد) للموضع المطلوب
- تضخيم الموضع المطلوب باستخدام بروتوكول PCR قياسي.
- سخن جهاز التدوير الحراري إلى درجة حرارة التمسخ الأولية في تفاعل تفاعل تفاعل البوليميراز المتسلسل أدناه.
- تحضير خليط تفاعل 25 ميكرولتر لكل عينة من الحيوانات المنوية في أنابيب تفاعل البوليميراز المتسلسل:
12.5 ميكرولتر من 2x تاك بوليميراز ماستر ميكس
1.5 ميكرولتر من 10 ميكرومتر التمهيدي الأمامي
1.5 ميكرولتر من التمهيدي العكسي 10 ميكرومتر
4.5 ميكرولتر من الماء الخالي من النيوكلياز
5 ميكرولتر من عينة الحمض النووي للحيوانات المنوية المحايدة - تخلط جيدا عن طريق سحب الماصة لأعلى ولأسفل.
- تدور لفترة وجيزة أسفل العينات في جهاز طرد مركزي صغير.
- ضع العينات في جهاز تدوير حراري مسخن مسبقا بالإعدادات التالية:
تمسخ أولي: 95 درجة مئوية لمدة 3 دقائق
35 دورة من التمسخ (95 درجة مئوية لمدة 30 ثانية) ، والتلدين (55 درجة مئوية لمدة 30 ثانية) ، والتمديد (72 درجة مئوية لمدة 30 ثانية)
التمديد النهائي: 72 درجة مئوية لمدة 5 دقائق
ملاحظة: قد يكون من الضروري ضبط درجة حرارة التمديد و / أو الوقت اعتمادا على Taq polymerase المحدد والطول المتوقع لمنتج تضخيم PCR. - قم بإزالة عينات تفاعل البوليميراز المتسلسل من جهاز التدوير الحراري.
- اختياري: قم بإجراء هضم التقييد الخاص بالمتبرع على حجم صغير من منتج تفاعل البوليميراز المتسلسل ، مع ترك ما لا يقل عن 5-10 ميكرولتر من منتج تفاعل البوليميراز المتسلسل غير المهضوم للرحلان الكهربائي الهلامي.
- تحضير تفاعل 30 ميكرولتر في أنابيب PCR سعة 0.2 مل:
10 ميكرولتر من منتج تفاعل البوليميراز المتسلسل غير المنقى
15 ميكرولتر من الماء الخالي من النيوكلياز
3 ميكرولتر من 10x تقييد إنزيم العازلة
2 ميكرولتر من إنزيم التقييد (يستخدم BstNI في النتائج التمثيلية لتحليل أليل dnah10 knock-in)
ملاحظة: لا تزال العديد من إنزيمات التقييد نشطة في مخزن مؤقت نموذجي لتفاعل تفاعل البوليميراز المتسلسل. ومع ذلك ، فإن إضافة المخزن المؤقت لهضم التقييد يمكن أن يساعد في تحسين النتائج. راجع إرشادات الشركة المصنعة المحددة لاستكشاف الأخطاء وإصلاحها في حالة حدوث أي مشاكل. - تخلط جيدا عن طريق سحب الماصة لأعلى ولأسفل.
- تدور لفترة وجيزة أسفل العينات في جهاز طرد مركزي صغير.
- ضع العينات في جهاز تدوير حراري بالإعدادات التالية:
- قطع amplicon PCR عند 60 درجة مئوية لمدة 1 ساعة.
ملاحظة: قد تحتاج درجة حرارة القطع ووقت الحضانة إلى تعديل إنزيم التقييد المحدد المستخدم. - قم بتعطيل إنزيم التقييد بدرجة الحرارة المطلوبة ووقت الحضانة ، إذا أشارت الشركة المصنعة إلى ذلك.
ملاحظة: لا يتطلب إنزيم BstNI تعطيلا للحرارة.
- قطع amplicon PCR عند 60 درجة مئوية لمدة 1 ساعة.
- تحضير تفاعل 30 ميكرولتر في أنابيب PCR سعة 0.2 مل:
7. إجراء رحلان كهربائي هلامي لفصل مضخمات PCR بأحجام مختلفة
- تحضير 500 مل من 0.5x TBE تشغيل المخزن المؤقت: أضف 50 مل من 10x TBE العازلة المركزة إلى 450 مل من الماء منزوع الأيونات.
- قم بإعداد 100 مل من جل الأغاروز 4٪ في مخزن مؤقت للتشغيل 0.5x TBE: أضف 4 جم من الأغاروز مع 10 ميكرولتر من صبغة جل 10000x إلى 100 مل من 0.5x TBE تشغيل المخزن المؤقت. الميكروويف حتى يذوب مسحوق الأغاروز بالكامل.
- صب محلول الجل في إطار صب جل بحجم مناسب ، وأدخل المشط على جانب واحد من الجل.
ملاحظة: حجم الجل الأكبر (موصى به: 15 سم × 15 سم) مفيد لهذا الإجراء ، لأنه يوفر للمضخمات مساحة أكبر للتحليل على الجل.- إذا كان سيتم تحليل منتج هضم التقييد ، فمن الأفضل تشغيله على نفس الجل مثل منتج PCR بحيث يمكن مقارنة النتائج. اضبط حجم بئر المشط حسب الضرورة لتناسب جميع العينات الموجودة على الجل.
- اترك الجل في درجة حرارة الغرفة حتى يصلب. إزالة بعناية المشط. قم بإزالة الجل بعناية من إطار الصب.
- صب محلول الجل في إطار صب جل بحجم مناسب ، وأدخل المشط على جانب واحد من الجل.
- قم بإعداد جهاز الرحلان الكهربائي الهلامي.
- ضع الجل في جهاز الرحلان الكهربائي بحيث تكون الآبار أقرب إلى القطب السالب. صب 0.5x TBE تشغيل العازلة في جهاز الرحلان الكهربائي حتى يتم غمر الآبار بالكامل.
- تحميل الجل.
- قم بتحميل 5 ميكرولتر من سلم الحمض النووي في البئر الأول (اختر سلم الحمض النووي الذي يحتوي على شظايا الحمض النووي التي تشبه في الحجم مضخم PCR).
- باستخدام طرف ماصة جديد لكل عينة (بما في ذلك عينة التحكم من النوع البري) ، قم بتحميل 5-10 ميكرولتر من منتج تفاعل البوليميراز المتسلسل في الآبار المتبقية.
- اختياري: إذا كان سيتم تحليل منتج هضم التقييد ، فقم بتحميل هذه العينات على الجل أيضا.
ملاحظة: من الأفضل تحميل كميات متساوية من الحمض النووي بين تفاعل البوليميراز المتسلسل ومنتج هضم التقييد. على سبيل المثال ، في خليط تفاعل هضم تقييد 30 ميكرولتر ، تمت إضافة 10 ميكرولتر من منتج PCR. لذلك ، كان تركيز الحمض النووي ل 15 ميكرولتر من المنتج المهضوم مشابها ل 5 ميكرولتر من منتج تفاعل البوليميراز المتسلسل غير المنقى.
- تشغيل الجل.
- قم بتوصيل الأقطاب الكهربائية بجهاز الرحلان الكهربائي الهلامي ، وقم بتطبيق 150 فولت لمدة 1 ساعة على الأقل لضمان الفصل الكافي للمضخمات.
- تخيل الجل لعرض أشرطة الحمض النووي.
ملاحظة: سيتم تشغيل المواقع المستهدفة بتقنية كريسبر شديدة التغير كأحجام نطاقات متعددة تمثل مزيجا من أليلات الإدخال/الحذف مع مضخمات من النوع البري.- إذا لم يتم حل أشرطة الهلام بعد 1 ساعة ، ضع 150 فولت على الجل لمدة 15 دقيقة حتى يتم فصل العصابات بشكل كاف على الجل.
- اختياري: في حالة الفحص لإدخال تعديل دقيق مثل SNP أو علامة الخاتمة أو علامة الفلورسنت ، قم بفحص الحمض النووي للحيوانات المنوية باستخدام طرق تفاعل البوليميراز المتسلسل الخاصة بالتعديل المطلوب. على سبيل المثال ، عند تحديد موقع تقييد خاص بالمتبرع ، سيكون هناك شريط إضافي في منتج هضم التقييد مقارنة بمنتج تفاعل البوليميراز المتسلسل لعينة الحيوانات المنوية.
8. عزل الأليلات المستقرة للخط الجرثومي
- بعد 2 أسابيع من الراحة من إجراء ضغط الحيوانات المنوية ، تجاوز الأفراد F0 الذين يحتوي الحمض النووي الجيني للحيوانات المنوية على التعديلات المطلوبة.
ملاحظة: قد يكون توليد الحيوانات المنوية من الحيوانات المنوية الفردية المستنسخة دورية في الزرد. ومن ثم، قد لا ينتج الأليل المطلوب الحيوانات المنوية وقت التهجين. لهذا السبب ، قد يتعين إجراء عبور إضافي أكثر من مرة لعزل الأليل المتورط في الفحص الأولي. - بمجرد أن تنضج الأسماك المهجنة F1 جنسيا ، تابع طرق التنميط الجيني القياسية (على سبيل المثال ، الحمض النووي لمقطع الزعانف) باستخدام نفس بروتوكول PCR و gel الكهربائي الذي يتم إجراؤه أثناء فحص الحمض النووي الجيني للحيوانات المنوية.
- يقوم سانجر بتسلسل منتجات تفاعل البوليميراز المتسلسل لتحديد أفراد F1 بنفس التعديل المطلوب.
- اختياري: إذا أدى التعديل المطلوب إلى إدخال موقع تقييد أو تعطيله، يمكنك إجراء فحص باستخدام بروتوكول مناسب لملخص القيود.
- في تقاطع F1 متغاير الزيجوت مع نفس التعديل المطلوب.
- فحص ذرية F2 للنمط الظاهري المتوقع و / أو التعديلات الجينومية.
النتائج
تسمح الأساليب التجريبية الموصوفة في هذا البروتوكول بتحديد أسرع لتعديلات الجينوم أو الأليلات الضارة المفترضة من خلال التركيز على تحليل آلاف الجينومات المشتقة من مجموعة الحيوانات المنوية الذكرية المحقونة ب F0. يوضح الشكل 2 كيفية تفسير النتائج التي تم الحصول عليها باستخدام هذا البروتوكول.
لتوليد طفرات في موضع p2ry12 ، تم حقن أجنة الزرد ذات المرحلة الواحدة ب Cas9 endonuclease و sgRNA الخاص ب p2ry12 (الشكل 2B ، تمييز رمادي). تم وضع الأسماك المحقونة ب F0 في النظام حتى النضج الجنسي ، وتم جمع الحيوانات المنوية من ستة ذكور فردية. تم استخراج الحمض النووي من عينات الحيوانات المنوية باستخدام حرارة عالية وظروف أساسية (استخراج الحمض النووي HotSHOT) وتحييده قبل تضخيم تفاعل البوليميراز المتسلسل لموضع p2ry12. تم فصل منتجات PCR على هلام أغاروز بنسبة عالية (4٪) لمدة 1.5 ساعة بجهد عالي (150 فولت). في هذا المثال ، ركض amplicon من النوع البري كشريط ساطع واحد يبلغ طوله حوالي 250 زوجا من القواعد (الشكل 2C ؛ الشكل 2C). بالوزن). في المقابل ، ركضت الأمبليكونات الذكرية المحقونة ب F0 والتي تحتوي على indels على الجل كنطاقات متعددة (الشكل 2C ؛ الممرات # 1-2 ، # 5-8). بدلا من ذلك ، يمكن تشغيل مضخمات الذكور المحقونة ب F0 المتحورة كشريط واحد مع حركة هلام متغيرة (غير مذكورة في مثال p2ry12 ، ولكنها واضحة في منتج تفاعل البوليميراز المتسلسل ل dnah10 F0-male # 2; الشكل 2E). كان أقل وضوحا في مثال هلام p2ry12 ما إذا كانت العينة #3 والعينة #4 تحتوي على indels مقارنة بالفرقة من النوع البري ، لذلك قد لا يكون هؤلاء الأفراد أفضل المرشحين المؤسسين. إن عزل الأليلات من ذكور F0 مع تعديلات أمبليكون أكثر تميزا هو الأفضل لنشر ناقلات الخط الجرثومي المستقرة حيث يتم تسجيلها بسهولة على 4٪ هلام أغاروز. على سبيل المثال ، يبدو أن عينة الحيوانات المنوية F0-male # 6 تحتوي على حذف كبير في أحد الأليلات (الشكل 2C ؛ الحارة 6 ؛ شريط ساطع مع زيادة حركة الهلام). إذا تم اختيار أليل الحذف الكبير هذا في جيل F1 ، فسيكون من السهل تمييزه عن أليل WT على هلام أغاروز 4٪. بدلا من ذلك ، إذا كانت هناك رغبة في الحصول على مجموعة من الأليلات المختلفة ، يمكن أن يكون F0-male # 7 مرشحا مؤسسا فعالا نظرا لأن منتج تفاعل البوليميراز المتسلسل الخاص به يبدو أنه يحتوي على العديد من الأليلات المنفصلة لمختلف التنقلات. بمجرد اختيار ذكر مؤسس ، يمكن عزل الأليل المطلوب عن طريق تهجين الفرد مرة أخرى إلى السلالة البرية الأصلية المستخدمة في الحقن.
لتوليد طفرة محددة في جين dnah10 ، تم حقن أجنة الزرد ذات المرحلة الواحدة ب Cas9 endonuclease ، وهو sgRNA خاص ب dnah10 (الشكل 2D ؛ تمييز رمادي) ، وقليل النوكليوتيد المتبرع بالحمض النووي يحتوي على التعديل المطلوب (الشكل 2D ؛ أحمر) وموقع تقييد BstNI خاص بالجهة المانحة (الشكل 2D ، بين قوسين). يسمح هذا التصميم بسهولة تحديد تكامل المانحين مع ملخص التقييد بعد تفاعل البوليميراز المتسلسل. علاوة على ذلك ، من خلال تغيير أزواج القواعد داخل موقع التعرف على sgRNA (الشكل 2D ؛ تمييز رمادي) ، يمنع هذا التصميم هضم Cas9 لتسلسل المتبرع. بمجرد وصول الأسماك المحقونة ب F0 إلى مرحلة النضج الجنسي ، تم جمع الحيوانات المنوية ، وتم استخراج الحمض النووي باستخدام طريقة الطلقة الساخنة. من هذه العينات ، تم تضخيم موضع dnah10 باستخدام تفاعل البوليميراز المتسلسل ، وتم فصل المنتجات على هلام أغاروز بنسبة عالية (4٪) لمدة 1 ساعة عند جهد عالي (150 فولت). في هذا المثال ، تم تشغيل amplicon من النوع البري كشريط ساطع واحد يبلغ طوله حوالي 400 زوج أساسي (الشكل 2E ؛ اللوحة العلوية: WT). في المقابل ، تم تشغيل مضخمات الذكور المحقونة ب F0 والتي تحتوي على indels كنطاقات متعددة (الشكل 2E ؛ اللوحة العلوية: الممرات # 1 و # 4 و # 5) أو كشريط واحد مع انخفاض حركة الهلام (الشكل 2E ؛ اللوحة العلوية: حارة # 2). لتحديد ما إذا كانت أي من عينات الحيوانات المنوية تحتوي على أليلات متكاملة مع المتبرعين ، تم هضم منتجات تفاعل البوليميراز المتسلسل مع إنزيم تقييد BstNI لمدة 1 ساعة ، وتم تشغيل المنتج على هلام أغاروز 4٪ لمدة 1 ساعة عند الجهد العالي (150 فولت). تكشف مقارنة منتج تفاعل البوليميراز المتسلسل غير المهضوم (الشكل 2 د ؛ اللوحة العلوية) بالمنتج المهضوم (الشكل 2 د ؛ الألواح السفلية) عن عينات ذات تكامل محتمل للبنية المانحة. في هذا المثال ، كان للذكر #1 المحقون ب F0 شريط إضافي في المنتج المهضوم لم يكن موجودا في منتج تفاعل البوليميراز المتسلسل (الشكل 2D ؛ اللوحة السفلية: حارة # 1 ، محاطة بدائرة). لذلك ، يمثل هذا الذكر أفضل مرشح مؤسس لإنشاء خط متحور مع الضربة القاضية المطلوبة.
بمجرد تجاوز الذكر الحامل المفترض F0 ، يجب التحقق من تسلسل الأليل في ذرية F1. يقترح تسلسل سانجر مباشرة للمضخمات المشتقة من الأسماك غير المتجانسة F1 متبوعة بأداء طرق تحليل الأليل غير المتجانس مثل Poly Peak Parser13 أو باستخدام مجموعة متنوعة من الأساليب القائمة على التسلسل من الجيل التالي مثل تسلسل MiSeq 14 أو Hi-Tom15. هذا نهج ضروري ومكمل للتأكد من أن التعديل الدقيق أو طفرة indel الضارة تسير بالفعل في الخط الجرثومي وليست مجرد قطعة أثرية لتحليل الرحلان الكهربائي الهلامي. في الواقع ، يمكن بسهولة استخدام نهج تسلسل الجيل التالي لتسلسل الحمض النووي للحيوانات المنوية من ناقلات F0 بدلا من تحليل الرحلان الكهربائي الهلامي الموصوف في هذا البروتوكول. ومع ذلك ، فإن وجود طريقة التنميط الجيني لهلام الأغاروز سهلة الاحتراق هو نهج أكثر اقتصادا ومساواة للمجتمع العالمي للباحثين في أسماك الزرد.

الشكل 1: الإعداد لإجراء ضغط الحيوانات المنوية . (أ) توضع الإسفنجة أسفل مجهر مجسم عند تكبير منخفض مع إضاءة علوية. (ب) يتم استخدام الإسفنج المبلل 1 × 1 ، المقطوع بخط بيضاوي (خط متقطع) ، لتثبيت الجانب البطني لذكر السمك المخدر لأعلى أثناء العملية. ج: تشريح الجانب البطني من ذكور الأسماك المخدرة التي تصور الزعانف الشرجية (AF ، الوردي) ، زعانف الحوض (pf ، الأزرق) ، والعباءة (السهم) ، حيث سيتم طرد الحيوانات المنوية أثناء العملية. (ج') تستخدم ملقط المرشح للضغط بلطف على ذكور الأسماك المخدرة من الخياشيم إلى العباءة ، بينما يتم سحب الحيوانات المنوية المطرودة إلى ماصة زجاجية عن طريق العمل الشعري (السهم الأبيض). د: أنبوب شعري يحتوي على عدد كاف من الحيوانات المنوية المطرودة (سائل معتم؛ قوس أسود) لاستخلاص الحمض النووي (DNA) وتحليله. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
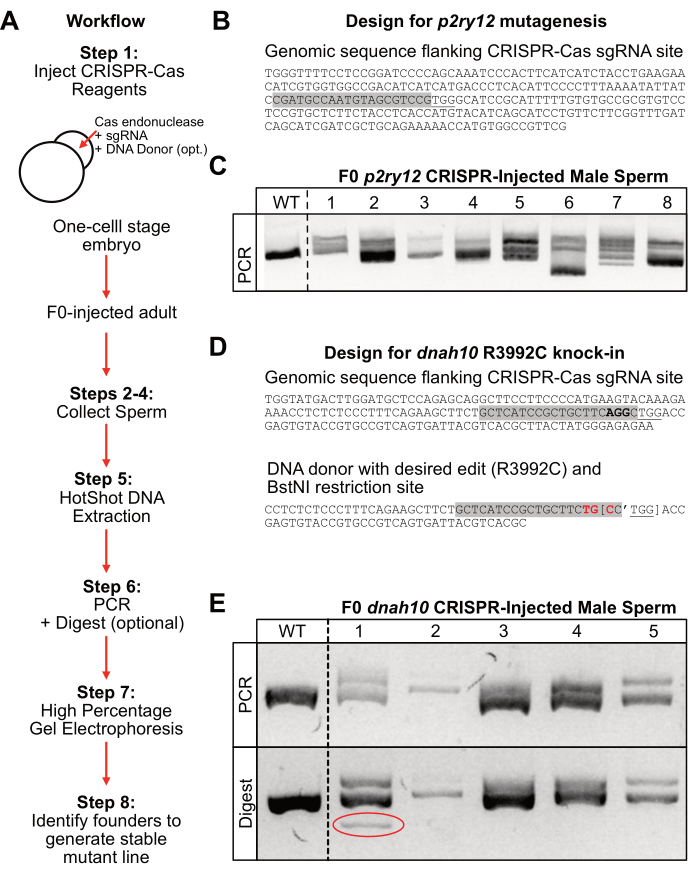
الشكل 2: سير العمل والنتائج التمثيلية. (أ) سير العمل العام للبروتوكول. (ب) تصميم تمثيلي لموقع p2ry12 sgRNA (تمييز رمادي) مع موقع PAM خاص ب Cas9 (تحته خط). (C) النتائج التمثيلية للرحلان الكهربائي الهلامي بنسبة 4٪ بعد 1.5 ساعة عند 150 فولت. التحكم في النوع البري (WT) وعينات الحيوانات المنوية الذكرية المحقونة ب F0-p2ry12 CRISPR (1-8) بعد تضخيم تفاعل البوليميراز المتسلسل. (د) تصميم تمثيلي لموقع dnah10 sgRNA (تمييز رمادي) مع موقع PAM خاص ب Cas9 (تحته خط) بالقرب من الكودون المستهدف (غامق). تسلسل مانح الحمض النووي مع تحرير الكودون المطلوب (أحمر غامق) وموقع تقييد BstNI (أقواس مع الفاصلة العليا التي تشير إلى موقع القطع). (ه) النتائج التمثيلية للرحلان الكهربائي الهلامي بنسبة 4٪ بعد 1 ساعة عند 150 فولت. أعلى: منتج تفاعل البوليميراز المتسلسل من النوع البري (WT) وعينات الحيوانات المنوية الذكرية المحقونة ب F0 dnah10 CRISPR (1-5). أسفل: BstNI تقييد هضم المنتج من العينات المذكورة أعلاه. توضح العينة 1 التكامل الناجح للبنية المانحة بناء على الشريط الإضافي بعد الهضم (الدائرة الحمراء). يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
Discussion
يصف هذا البروتوكول طريقة للتوصيف السريع لتعديلات الجينوم المفترضة أو الطفرات المستهدفة باستخدام تقنية CRISPR-Cas من خلال التحليل المركز على جينومات الحيوانات المنوية الذكرية F0. يجب أن يكون هذا البروتوكول قابلا للنماذج الحيوانية الأخرى حيث تكون الحيوانات المنوية متاحة بسهولة لأخذ العينات دون القتل الرحيم. ستزيد هذه الطريقة من إنتاجية الفحص للتعديلات المرغوبة وهي مفيدة بشكل خاص لتحديد الأحداث النادرة بوساطة HDR. يعمل هذا النهج أيضا على تقليل عدد التجارب المستخدمة للعثور على تعديل مستقر للخط الجرثومي من خلال تسهيل الفحص السريع لآلاف الجينومات المحتملة في عينة واحدة من الحيوانات المنوية من حامل F0 مفترض ، وهو ما يتناقض مع الأساليب التقليدية التي قد تتطلب فحص مئات الأجنة المشتقة من تهجين ناقلات F0 المفترضة.
يعتمد هذا البروتوكول على البروتوكولات المعمول بها لجمع الحيوانات المنوية في الزرد7،10،14،16،17،18 من خلال تضمين تقنية قابلة للتكرار لتحديد تعديلات الخط الجرثومي باستخدام الرحلان الكهربائي للهلام عالي الدقة. يمكن دمج هذا النهج بسهولة في أي سير عمل قياسي ل CRISPR / Cas لزيادة الإنتاجية لفحص وعزل تعديلات الجينوم المستهدفة. بالإضافة إلى ذلك ، هذا البروتوكول مناسب للمختبرات المزودة بموظفين لديهم مجموعة من التدريب والخبرة ، بالإضافة إلى مختبرات التدريس. ومع ذلك ، فإن طريقتنا في جمع الحيوانات المنوية ليست كافية للحفظ بالتبريد ، والتي تم وصفها بخبرة في المنشورات السابقة10،17.
يستخدم هذا البروتوكول رحلان كهربائي عالي الدقة لهلام الأغاروز لتحديد الناقلات الذكور المفترضة للطفرات الجينومية المرغوبة والدقيقة. ومع ذلك ، فإن الحمض النووي الجيني للحيوانات المنوية قابل لعدد لا يحصى من الأساليب الأخرى ، بما في ذلك تحليل شظايا الفلورسنت أو التسلسل المشفر14 ، أو تحليل الذوبان عالي الدقة18 ، أو الكشف البسيط عن amplicon لعلامات الفلورسنت أو الحواتم باستخدام نهج الرحلان الكهربائي الهلامي القياسي. يجب أن يتم التحقق من صحة جميع الأليلات باستخدام مناهج مثل التسلسل القائم على سانجر أو الجيل التالي قبل بدء العمل التجريبي على الأليلات المفترضة. في الواقع ، يمكن للطرق الحالية لفحص الطفرات في الأجنة باستخدام نهج تسلسل الجيل التالي14 أن تتحايل على الحاجة إلى التحليل على هلام الأغاروز ، الموصوف في هذا البروتوكول. ومع ذلك ، فإن وجود طريقة التنميط الجيني القابلة للحرق بالهلام هو نهج أكثر فعالية من حيث التكلفة والمساواة يجب مراعاته نظرا لأنه ليس من السهل على جميع المختبرات الوصول إلى طرق تسلسل الجيل التالي الرخيصة أثناء مراحل العزل والتجارب للعمل مع كل سلالة متحولة.
باختصار ، يوفر هذا البروتوكول توجيهات خطوة بخطوة لفحص جينومات الحيوانات المنوية بشكل متكرر من الذكور المحررين بتقنية كريسبر / كاس بحيث تكون هناك حاجة إلى عدد أقل من التهجينات وتقليل فحص تفاعل البوليميراز المتسلسل للأجنة لفحص التعديلات المطلوبة. سيؤدي تطبيق هذه الطريقة إلى تقليل عدد الأسماك التي يجب إنشاؤها وتحليلها لتحديد الأليلات المحررة ذات الأهمية بنجاح ، مما يقلل أيضا من وقت وتكلفة الموظفين لتوليد خطوط مستقرة.
Disclosures
اي.
Acknowledgements
نود أن نشكر آنا هيندز في كلية الطب بجامعة واشنطن على جهودها الأولية في الحصول على الحمض النووي الجيني للحيوانات المنوية عالي الجودة باستخدام طريقة الطلقة الساخنة. تم تمويل هذا العمل من قبل المعهد الوطني لالتهاب المفاصل وأمراض الجهاز العضلي الهيكلي والجلد التابع للمعاهد الوطنية للصحة بموجب جائزة (R01AR072009 إلى R.S.G.).
Materials
| Name | Company | Catalog Number | Comments |
| Agarose powder | Fisher BioReagents | BP1356-100 | |
| Breeding tanks | Carolina Biological | 161937 | |
| BstNI Restriction Enzyme | NEB | R0168S | |
| Cas9 Endonuclease | IDT | 1081060 | |
| DNA Ladder, 100 bp | Thermo Scientific | FERSM0241 | |
| dnah10 donor construct | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry. Phosphorothioate bond on the donor at the first three phosphate bonds on both the 5’ and 3’ ends (5'-CCTCTCTCCCTTTCAGAAGCTTC TGCTCATCCGCTGCTTCTGCCT GGACCGAGTGTACCGTGCCGTC AGTGATTACGTCACGC-3') | |
| dnah10 forward primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CATGGAACTCTTTCCTAATGAGT TTGGC-3') | |
| dnah10 reverse primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry ('5-AGTAGAGATCACACATCAACAGA ATACAGC-3') | |
| dnah10 synthetic sgRNA | Synthego | Synthetic sgRNA, target sequence: 5'-GCTCATCCGCTGCTTCAGGC-3' | |
| Electrophoresis power supply | Thermo Scientific | 105ECA-115 | |
| Filter forceps | Millipore | XX6200006P | |
| Fish (system) water | Generic | n/a | |
| Gel electrophoresis system (including casting frame, comb, and electrophoresis chamber) | Thermo Scientific | B2 | |
| Gel imaging light box | Azure Biosystems | AZI200-01 | |
| Gel stain, 10000X | Invitrogen | S33102 | |
| Glass bowl, 250 mL | Generic | n/a | |
| Isolation tanks, 0.8 L | Aquaneering | ZT080 | |
| Microcap capillary tube with bulb, 20 µL | Drummond | 1-000-0020/CA | |
| Minicentrifuge | Bio-Rad | 12011919EDU | |
| Micropipettes, various with appropriate tips | Generic | n/a | |
| Microwave | Generic | n/a | |
| Nuclease free water | Promega | P119-C | |
| Paper towels | Generic | n/a | |
| PCR tubes, 0.2 mL | Bioexpress | T-3196-1 | |
| Plastic spoon, with drilled holes/slots | Generic | n/a | |
| KCl solution, 0.2 M RNAse Free | Sigma-Aldrich | P9333 | |
| p2ry12 forward primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CCCAAATGTAATCCTGACCAGT -3') | |
| p2ry12 reverse primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CCAGGAACACATTAACCTGGAT -3') | |
| p2ry12 synthetic sgRNA | Synthego | Synthetic sgRNA, target sequence: 5'-GGCCGCACGAGGTCTCCGCG-3' | |
| Restriction Enzyme 10X Buffer | NEB | B6003SVIAL | |
| NaOH solution, 50 mM | Thermo Scientific | S318; 424330010 | |
| Sponge, 1-inch x 1-inch cut with small oval divot | Generic | n/a | |
| Stereomicroscope | Zeiss | Stemi 508 | |
| Taq polymerase master mix, 2X | Promega | M7122 | |
| TBE Buffer Concentrate, 10X | VWR | E442 | |
| Thermal Cycler | Bio-Rad | 1861096 | |
| Tissue paper | Fisher Scientific | 06-666 | |
| Tricaine-methanesulfonate solution (Syncaine, MS-222), 0.016% in fish water (pH 7.0±0.2) | Syndel | 200-266 | |
| Tris Base, 1M (Buffered with HCl to ph 8.0) | Promega | H5131 |
References
- Auer, T. O., Duroure, K., De Cian, A., Concordet, J. P., Del Bene, F. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair. Genome Research. 24 (1), 142-153 (2014).
- Hwang, W. Y., et al. Efficient genome editing in zebrafish using a CRISPR-Cas system. Nature Biotechnology. 31 (3), 227-229 (2013).
- Jao, L. E., Wente, S. R., Chen, W. Efficient multiplex biallelic zebrafish genome editing using a CRISPR nuclease system. Proceedings of the National Academy of Sciences of the United States of America. 110 (34), 13904-13909 (2013).
- Troutwine, B. R., et al. The Reissner fiber is highly dynamic in vivo and controls morphogenesis of the spine. Current Biology. 30 (12), 2353-2362 (2020).
- Xu, Y., Li, Z. CRISPR-Cas systems: Overview, innovations and applications in human disease research and gene therapy. Computational and Structural Biotechnology Journal. 18, 2401-2415 (2020).
- Creaser, C. W. The technic of handling the zebrafish (Brachydanio rerio) for the production of eggs which are favorable for embryological research and are available at any specified time throughout the year. Copeia. 1930 (4), 159-161 (1934).
- Carmichael, C., Westerfiel, M., Varga, Z. M. Cryopreservatin and in vitro fertilization at the zebrafish international resource center. Methods in Molecular Biology. 546, 45-65 (2009).
- Wang, Y., Troutwine, B. R., Zhang, H., Gray, R. S. The axonemal dynein heavy chain 10 gene is essential for monocilia motility and spine alignment in zebrafish. Developmental Biology. 482, 82-90 (2021).
- Gray, R. S., et al. Postembryonic screen for mutations affecting spine development in zebrafish. Developmental Biology. 471, 18-33 (2021).
- Brocal, I., et al. Efficient identification of CRISPR/Cas9-induced insertions/deletions by direct germline screening in zebrafish. BMC Genomics. 17, 259 (2016).
- Davis, M. W., Jorgensen, E. M. ApE, A Plasmid Editor: A freely available DNA manipulation and visualization program. Frontiers in Bioinformatics. 2, 816619 (2022).
- Labun, K., et al. CHOPCHOP v3: Expanding the CRISPR web toolbox beyond genome editing. Nucleic Acids Research. 47, 171-174 (2019).
- Hill, J. T., et al. Poly Peak Parser: Method and software for identification of unknown indels using sanger sequencing of polymerase chain reaction products. Developmental Dynamics. 243 (12), 1632-1636 (2014).
- Varshney, G. K., et al. High-throughput gene targeting and phenotyping in zebrafish using CRISPR/Cas9. Genome Research. 25 (7), 1030-1042 (2015).
- Liu, Q., et al. Hi-TOM: A platform for high-throughput tracking of mutations induced by CRISPR/Cas systems. Science China. Life Sciences. 62 (1), 1-7 (2019).
- Sorlien, E. L., Witucki, M. A., Ogas, J. Efficient production and identification of CRISPR/Cas9-generated gene knockouts in the model system Danio rerio. Journal of Visualized Experiments. (138), e56969 (2018).
- Draper, B. W., Moens, C. B. A high-throughput method for zebrafish sperm cryopreservation and in vitro fertilization. Journal of Visualized Experiments. (29), e1395 (2009).
- Parant, J. M., George, S. A., Pryor, R., Wittwer, C. T., Yost, H. J. A rapid and efficient method of genotyping zebrafish mutants. Developmental Dynamics. 238 (12), 3168-3174 (2009).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved