Method Article
Screening von Spermien zur schnellen Isolierung von Keimbahnveränderungen im Zebrafisch
In diesem Artikel
Zusammenfassung
Die CRISPR-Cas-Technologien haben die Genom-Editierung revolutioniert. Das Finden und Isolieren der gewünschten Keimbahnreduktion bleibt jedoch ein großer Engpass. Daher beschreibt dieses Protokoll eine robuste Methode zum schnellen Screening von F0 CRISPR-injizierten Zebrafischspermien auf Keimbahn-Editierungen unter Verwendung von Standard-PCR-, Restriktionsverdauungs- und Gelelektrophoresetechniken.
Zusammenfassung
Das Aufkommen zielgerichteter CRISPR-Cas-Nuklease-Technologien hat die Fähigkeit revolutioniert, sowohl in etablierten als auch in neu entstehenden Modellsystemen eine präzise Genom-Editierung durchzuführen. CRISPR-Cas-Genomeditierungssysteme verwenden eine synthetische Guide-RNA (sgRNA), um eine CRISPR-assoziierte (Cas) Endonuklease auf bestimmte genomische DNA-Loci auszurichten, wobei die Cas-Endonuklease einen Doppelstrangbruch erzeugt. Die Reparatur von Doppelstrangbrüchen durch intrinsische fehleranfällige Mechanismen führt zu Insertionen und/oder Deletionen, die den Locus stören. Alternativ kann der Einschluss von doppelsträngigen DNA-Donoren oder einzelsträngigen DNA-Oligonukleotiden in diesen Prozess die Einbeziehung präziser Genom-Editierungen hervorrufen, die von Einzelnukleotid-Polymorphismen über kleine immunologische Markierungen bis hin zu großen fluoreszierenden Proteinkonstrukten reichen. Ein großer Engpass bei diesem Verfahren kann jedoch darin bestehen, die gewünschte Bearbeitung in der Keimbahn zu finden und zu isolieren. Dieses Protokoll beschreibt eine robuste Methode zum Screening und zur Isolierung von Keimbahnmutationen an bestimmten Loci in Danio rerio (Zebrafisch); Diese Prinzipien können jedoch in jedem Modell adaptiert werden, in dem eine In-vivo-Spermienentnahme möglich ist.
Einleitung
Das CRISPR-System (Clustered Regularly Interspaced Short Palindromic Repeats)/Cas ist ein leistungsfähiges Werkzeug zur Durchführung von Loci-spezifischer Mutagenese und präziser Genom-Editierung im Danio rerio (Zebrafisch) Modellsystem 1,2,3,4. Das Cas-Ribonukleoprotein (RNP) besteht aus zwei Hauptkomponenten: einer Cas-Endonuklease (üblicherweise Cas9 oder Cas12a) und einer locusspezifischen synthetischen Guide-RNA (sgRNA)5. Zusammen erzeugen die Cas-RNP einen doppelsträngigen Bruch (DSB) im gewünschten Locus, der durch einen von zwei intrinsischen Reparaturmechanismen repariert werden kann. Der Reparaturmechanismus der nicht-homologen Endverknüpfung (NHEJ) ist fehleranfällig und führt häufig zu einer Vielzahl von Insertionen oder Deletionen (Indels) um das DSB herum. Diese Indels können schädlich sein, wenn sie eine Frameshift-Mutation oder einen vorzeitigen Stopp in der resultierenden Proteinsequenz einführen. Alternativ verwendet der Homologie-gerichtete Reparaturmechanismus (HDR) eine Spenderschablone mit Homologieregionen, die die DSB-Stelle umgeben, um den Schaden zu reparieren. Forscher können das HDR-System nutzen, um präzise genomische Bearbeitungen zu generieren. Konkret können sie ein doppelsträngiges DNA-Spenderkonstrukt koinjizieren, das die gewünschten Bearbeitungen sowie Homologieregionen enthält, die die DSB-Stelle im Genom flankieren. Die erhöhten Skaleneffekte für diese kommerziell hergestellten CRISPR-Komponenten haben die Hürden für das Screening mehrerer Loci und für die Einrichtung größerer Anstrengungen zur präzisen Genom-Editierung erheblich verringert. Ein großer Engpass bei der sexuell reproduzierenden Tiervermehrung ist jedoch die Identifizierung und Isolierung keimbahnstabiler mutierter Tiere.
Das Zebrafisch-Modellsystem weist mehrere Schlüsseleigenschaften auf, die seine Verwendung in reversen genetischen Studien verbessern. Sie lassen sich leicht in großer Zahl aufziehen, und die Weibchen weisen das ganze Jahr über eine hohe Fruchtbarkeit auf6. Darüber hinaus sind sie durch ihre äußere Eiablage und Befruchtung für die Mikroinjektion von CRISPR/Cas-Komponenten zugänglich. Das Cas-RNP wird üblicherweise in Zebrafischembryonen im Ein-Zell-Stadium injiziert, um DSBs/Reparaturen zu erzeugen, die theoretisch von allen Tochterzellen vererbt werden. Diploide Genome benötigen jedoch zwei DSB/Reparaturereignisse, um beide homologen Chromosomen zu mutagenisieren. Obwohl Cas-RNP im Ein-Zell-Stadium injiziert wird, kann es sein, dass die DSB/Reparatur erst zu einem späteren Zeitpunkt in der Entwicklung erfolgt. Zusammen tragen diese Faktoren zur Mosaiknatur von F0-injizierten Fischen bei. Eine gängige Praxis ist es, F0-injizierte Fische zu kreuzen und die F1-Nachkommen auf Indels/spezifische Bearbeitungen zu untersuchen. Da jedoch nicht alle F0-injizierten Fische Keimbahnmutationen aufweisen, führt diese Praxis zu vielen unproduktiven Kreuzungen, die nicht den gewünschten Edit erzeugen. Das Screening der F0-Keimbahn anstelle des F1-Samengewebes erhöht die Wahrscheinlichkeit, die gewünschte Keimbahnreduktion zu isolieren, und reduziert die Anzahl der für diesen Prozess erforderlichen Tiere.
Spermien können leicht aus F0-injizierten Zebrafischen entnommen werden, ohne dass eine Euthanasie erforderlich ist. Diese Funktion ermöglicht die Kryokonservierung und Rederivation von gefrorenen Spermienbeständen7, kann aber auch genutzt werden, um die Keimbahnträger der gewünschten genomischen Mutationen 8,9 schnell zu screenen, zu identifizieren und zu isolieren. Brocal et al. (2016) beschrieben zuvor eine sequenzierungsbasierte Methode zum Screening von Keimbahn-Edits bei F0-injizierten männlichen Zebrafischen10. Obwohl dieser Ansatz für die Identifizierung der in der Keimbahn vorhandenen mutierten Allele nützlich ist, kann er im Hochdurchsatz kostspielig werden und ist möglicherweise nicht für alle Labore zugänglich. Im Gegensatz dazu bietet das aktuelle Protokoll eine zugängliche und kostengünstige elektrophoresebasierte Strategie zur Identifizierung von Keimbahn-Editierungen. Konkret beschreibt dieses Protokoll eine robuste Methode zum Screening und zur Isolierung von Keimbahnmutationen an bestimmten Loci mittels hochauflösender Agarose-Gelelektrophorese. Darüber hinaus beschreibt dieses Protokoll eine ähnliche Strategie zur Identifizierung der erfolgreichen Integration eines Spenderkonstrukts, das spezifische Bearbeitungen enthält. Wie immer, wenn bestimmte Bearbeitungen gewünscht werden, können sequenzierungsbasierte Strategien zusammen mit dem unten beschriebenen Protokoll durchgeführt werden. Obwohl dieses Protokoll spezifisch für das Zebrafisch-Modellsystem ist, sollten diese Prinzipien auf jedes Modell übertragbar sein, in dem die Entnahme von Spermien ein Routineverfahren ist. Zusammen ermöglichen diese Strategien die Identifizierung von F0-injizierten Männchen mit Keimbahn-Indels/Editits, die nach Standard-Polymerase-Kettenreaktion (PCR) und/oder Restriktionsverdauung auf einem Gel aufgelöst werden können.
Protokoll
Diese Studie wurde in Übereinstimmung mit den Richtlinien des Leitfadens für die Pflege und Verwendung von Versuchstieren der National Institutes of Health durchgeführt. Das Protokoll wurde vom Animal Care and Use Committee der University of Texas at Austin genehmigt (AUP-2021-00254).
1. Design der sgRNA für die CRISPR-Mutagenese
- Ermitteln Sie die Exon-Sequenz, die die Zielorte enthält.
- Entwerfen Sie eine synthetische Guide-RNA (sgRNA) mit einer Protospacer-benachbarten Motivstelle (PAM), die spezifisch für die Cas-Endonuklease ist.
- Wenn Sie ein Spenderkonstrukt koinjizieren, entwerfen Sie die sgRNA so, dass sie eine vorhergesagte Schnittstelle aufweist, die so nah wie möglich an den gewünschten Bearbeitungen liegt.
HINWEIS: APE ist eine freie Software mit einem Werkzeug zum Auffinden von Cas9-sgRNA-Stellen11. Alternativ können Werkzeuge wie CHOPCHOP verwendet werden, das eine Online-Schnittstelle für das Design von sgRNAs bietet, die die verschiedenen PAM-Stellen von Cas9 und anderen Cas-Endonukleasenberücksichtigt 12.
- Wenn Sie ein Spenderkonstrukt koinjizieren, entwerfen Sie die sgRNA so, dass sie eine vorhergesagte Schnittstelle aufweist, die so nah wie möglich an den gewünschten Bearbeitungen liegt.
- Entwerfen Sie Vorwärts- und Rückwärtsprimer, um den Bereich um die entworfene CRISPR/Cas-Schnittstelle herum zu verstärken. Das PCR-Produkt sollte etwa 200-400 Basenpaare (bp) lang sein, um später im Protokoll kleine Indels auf einem Gel auflösen zu können.
HINWEIS: CHOPCHOP11 entwirft automatisch Primer für jede ausgegebene sgRNA. - Optional: Entwerfen Sie ein DNA-Spenderkonstrukt mit den gewünschten Änderungen.
- Erhalten Sie die genomische Sequenz des Exons, die die Loci der gewünschten Mutation enthält.
HINWEIS: Intronische Sequenzen können von Person zu Person variieren, daher ist es am besten, die Aufnahme von intronischen Sequenzen in das Donorkonstrukt zu vermeiden, da dies den homologieabhängigen Reparaturweg stören könnte. - Wenn eine bestimmte Aminosäureänderung gewünscht ist, ändern Sie das spezifische Codon entsprechend.
HINWEIS: Achten Sie bei diesen Änderungen auf die Codon-Frequenz. Wenn möglich, wählen Sie ein Codon mit hoher Nutzung. - Um eine CRISPR/Cas-Verdauung des Donorkonstrukts zu verhindern, müssen zusätzliche synonyme Mutationen vorgenommen werden, die (i) das PAM und/oder (ii) die sgRNA-Sequenz stören, wobei nicht-synonyme Veränderungen näher an der PAM-Stelle bevorzugt werden.
- Verwenden Sie einen Restriktionsstellenfinder (APE11 verfügt ebenfalls über diese Funktion), um eindeutige Restriktionsstellen (solche, die nur einmal auftreten) im vorhergesagten PCR-Produkt der Wildtyp-Sequenz zu finden.
- Wiederholen Sie diesen Vorgang für das vorhergesagte PCR-Produkt, das die erfolgreiche Integration des Spenderkonstrukts widerspiegelt.
- Vergleichen Sie die Liste der Restriktionsstellen zwischen der Wildtyp- und der Donor-integrierten Sequenz, um eine Restriktionsstelle zu finden, die nur in der bearbeiteten Sequenz vorhanden ist.
- Wenn keine eindeutige Restriktionsstelle gefunden wird, nehmen Sie weitere synonyme Änderungen innerhalb des Spenderkonstrukts vor, bis dies erreicht ist.
HINWEIS: Wenn möglich, machen Sie weiterhin synonyme Mutationen, die die sgRNA-Bindung an das Spenderkonstrukt stören würden.
- Kürzen Sie die Spendersequenz auf eine überschaubare Größe (50-100 bp) mit mindestens 20 bp Homologie, die die vorhergesagte CRISPR/Cas-Schnittstelle umgibt, und bearbeiten Sie sie, wenn möglich.
- Erhalten Sie die genomische Sequenz des Exons, die die Loci der gewünschten Mutation enthält.
- Besorgen Sie sich die entworfenen sgRNA- und/oder DNA-Donor-Oligonukleotide sowie alle für die Genotypisierung erforderlichen Primer.
Anmerkung: Um den Aufschluss der DNA-Donor-Oligonukleotide durch Nukleasen zu verhindern, wird empfohlen, die ersten drei Phosphatbindungen an den 5'- und 3'-Enden der DNA-Oligonukleotide mit Phosphorothioat zu modifizieren. - Injizieren Sie 1 nL der Injektionsmischung in einzellige Embryonen. In der Regel enthält eine Standard-Injektionsmischung Folgendes:
1 μL 25 μM locus-spezifische sgRNA
1 μL 25 μM Cas9-Endonuklease
Optional: 1 μL 3 μM DNA-Spenderkonstrukt- Bringen Sie das Gesamtvolumen mit 0,1 M RNase-freiem KCl auf 5 μL.
- Erlauben Sie den mit F0 injizierten Personen, sich zu geschlechtsreifen Erwachsenen zu entwickeln. Das verbleibende Protokoll konzentriert sich auf das Screening der F0-injizierten Männchen auf Keimbahn-Editierungen.
2. Aufstellen der Aufzuchtbecken
- Bereiten Sie F0-Männchen auf die Paarung vor, indem Sie Zuchtbecken mit Trennwänden zwischen den F0-Männchen und -Weibchen aufstellen.
- Um die Anzahl der benötigten Zuchtbecken zu minimieren, stellen Sie drei Männchen gegenüber zwei Weibchen in jedem Aquarium auf.
- Stellen Sie so viele F0-Männchen wie möglich auf und bringen Sie sie nach der Spermienentnahme als Individuen unter. Der Genotyp der Weibchen ist nicht von Bedeutung, da ihre Gameten bei diesem Eingriff nicht entnommen werden.
- Stellen Sie ein Zuchtbecken mit Wildtyp-Männchen gegenüber den Weibchen auf. Wildtyp-Spermien dienen als Kontrolle für die spätere Interpretation der Ergebnisse.
- Um die Anzahl der benötigten Zuchtbecken zu minimieren, stellen Sie drei Männchen gegenüber zwei Weibchen in jedem Aquarium auf.
- Bebrüten Sie die vorbereiteten Aufzuchtbecken über Nacht.
3. Vorbereitung der Materialien für die Samenentnahme
- Bereiten Sie genügend Anästhesielösung (0,016% Tricain-HCl in Fischwasser) vor, um eine kleine Glasschale zu füllen.
- Geben Sie 40 μl 50 mM NaOH in 0,2 mL PCR-Röhrchen. Bereiten Sie für jede Samenprobe, die entnommen wird, ein Röhrchen vor. Auf Eis lagern.
- Bereiten Sie für jeden Fisch, der während des Verfahrens beprobt wird, einzelne 0,8-Liter-Tanks vor. In diesen Becken werden die beprobten Fische untergebracht, bis die Genotypisierung abgeschlossen ist.
HINWEIS: Dies ist der Hauptfaktor, der die Anzahl der Fische begrenzt, die in diesem Protokoll effektiv untersucht werden können. Sorgen Sie für ausreichend Platz für die Unterbringung der beprobten Individuen, bis die Genotypisierung abgeschlossen ist. - Befeuchten Sie einen 1 Zoll x 1 Zoll großen Schwamm (geschnitten mit einem flachen, ovalen Divot, um einen männlichen Zebrafisch während des Vorgangs mit der Bauchseite nach oben zu halten) in Fischwasser und drücken Sie überschüssige Flüssigkeit aus.
- Positionieren Sie den Schwamm bei geringer Vergrößerung im Sichtfeld eines Stereomikroskops (Abbildung 1A).
- Positionieren Sie die schräge Deckenbeleuchtung für eine optimale Sicht.
4. Betäubung des männlichen Fisches und Entnahme der Spermien
- Bereiten Sie ein sauberes Kapillarröhrchen vor.
- Schütteln Sie den Röhrchenbehälter vorsichtig, um ein einzelnes Kapillarröhrchen freizugeben.
- Setzen Sie das Kapillarrohr in die mitgelieferte Glühbirne ein und legen Sie es beiseite.
- Übertragen Sie einen männlichen Fisch in die vorbereitete Betäubungslösung (0,016% Tricain-HCl in Fischwasser).
- Rühren Sie die Lösung vorsichtig mit einem Schaumlöffel um, um den Anästhesievorgang zu beschleunigen.
- Sobald sich die Bewegung des Operculums (Kiemen) verlangsamt hat (ca. 1-2 Minuten), fahren Sie mit der Spermienentnahme fort.
- Verwenden Sie den geschlitzten Löffel, um den Fisch auf einen sauberen Stapel Papiertücher zu geben.
- Verwenden Sie den geschlitzten Löffel, um den Fisch vorsichtig zu rollen, um überschüssiges Wasser zu entfernen.
- Tupfen Sie den Fisch vorsichtig mit sauberem, gefaltetem Seidenpapier ab, um das restliche Wasser zu entfernen.
- Verwenden Sie den Schaumlöffel, um den Fisch auf den vorbereiteten Biskuit zu geben (Abbildung 1B).
- Positionieren Sie den Fisch mit der Bauchseite nach oben, wobei der Kopf der dominanten Hand der Person, die den Eingriff durchführt, am nächsten kommt.
- Tupfen Sie den Bereich um die Afterflossen vorsichtig mit sauberem, gefaltetem Seidenpapier ab.
- Sammle das Sperma.
- Bewegen Sie die Beckenflossen mit dem Ende des Kapillarrohrs vorsichtig seitlich von der Mittellinie weg, um die Kloake freizulegen (Abbildung 1C).
- Positionieren Sie das Ende des Kapillarrohrs in der Nähe der Kloake.
- Drücken Sie mit der Pinzette vorsichtig die Seiten des Fisches zusammen, beginnend knapp unter den Kiemen und endend an der Kloake (Abbildung 1C').
- Sammeln Sie die Spermien durch Kapillarwirkung in der Kapillarröhre. Für dieses Verfahren ist ca. 1 μL ausreichend (Abbildung 1D).
HINWEIS: Einige Labore berichten, dass sie p10-Pipettenspitzen anstelle von Kapillarröhrchen für die Spermienentnahme verwenden. - Wenn das Sperma nicht mit sanftem Druck freigesetzt wird, geben Sie den Fisch wieder in das Systemwasser zurück.
- Überwachen Sie, ob Sie sich von der Narkose erholen.
- Drücken Sie den Fisch nicht fest zusammen, um Sperma auszustoßen, da dies den Fisch verletzen kann.
- Ruhen Sie den Fisch 2 Wochen lang aus, bevor Sie erneut versuchen, Spermien zu sammeln.
- Legen Sie die Fische nach der Entnahme in ein sauberes Isolationsbecken mit Systemwasser für die individuelle Unterbringung.
- Überwachen Sie die Fische, um sich von der Narkose zu erholen.
- Ruhen Sie den Fisch 2 Wochen lang aus, bevor Sie erneut versuchen, Spermien zu sammeln.
- Das Kapillarröhrchen mit den entnommenen Spermien wird in das vorbereitete PCR-Röhrchen (mit 40 μL 50 mM NaOH) eingesetzt.
- Drücken Sie den Gummikolben zusammen, um die gesammelte Probe auszustoßen.
- Entsorgen Sie das gebrauchte Kapillarrohr in einem zugelassenen Glasabfallbehälter.
- Inkubieren Sie die Proben auf Eis, bis alle Männchen zusammengedrückt sind.
- Wiederholen Sie die Schritte 4.1-4.5 für jedes einzelne Männchen.
- Beschriften Sie die Isolationstanks und Samenproben so, dass die endgültigen Ergebnisse der Spermiengenotypisierung auf das entsprechende Individuum mit mutmaßlichen Mutationen zurückgeführt werden können.
- Überwachen Sie alle Personen und stellen Sie sicher, dass sie aufrecht stehen und ihre Tanks erkunden, bevor Sie die Tanks wieder auf das System setzen.
- Setzen Sie die Weibchen, die zum Ansaugen verwendet wurden, wieder in ihre jeweiligen Becken auf dem System.
- Wenn sich ein Fisch nach 10 Minuten nicht von der Narkose erholt, befolgen Sie das Euthanasieverfahren, das von der mit dem Labor verbundenen Einrichtung genehmigt wurde.
- Beispiel: Kühlen Sie die Person in einem Eisbad mit 2-4 °C heißem Fischwasser schnell ab.
- Entsorgen Sie die eingeschläferten Fische in einem geeigneten Tierkörperabfallbehälter gemäß dem von der dem Labor angeschlossenen Institution genehmigten Verfahren.
5. Extraktion von DNA aus den Spermaproben
- Schleudern Sie alle Spermaproben kurz in einer Minizentrifuge herunter und legen Sie die PCR-Röhrchen in einen Thermocycler.
- Schließen Sie den Deckel des Thermocyclers.
- Führen Sie die folgenden Einstellungen im Thermocycler aus:
- Die Proben werden 40 min bei 95 °C erhitzt.
- Kühlen Sie die Proben auf 25 °C ab.
- Nehmen Sie die Proben aus dem Thermocycler.
- Mit einer sauberen Pipettenspitze für jede Probe neutralisieren Sie den pH-Wert, indem Sie 10 μl 1 M Tris-HCl (gepuffert auf pH 8) hinzufügen.
- Gut mischen, indem Sie auf und ab pipettieren.
- Schleudern Sie die Proben kurz in einer Minizentrifuge herunter.
- Genomische DNA-Proben können mehrere Tage bei 4 °C gelagert oder bis zu 6 Monate bei −20 °C gelagert werden.
6. PCR-Amplifikation (und/oder Restriktionsverdauung) des gewünschten Locus
- Amplifizieren Sie den gewünschten Locus mit einem Standard-PCR-Protokoll.
- Heizen Sie den Thermocycler auf die anfängliche Denaturierungstemperatur in der folgenden PCR-Reaktion vor.
- Bereiten Sie für jede Spermienprobe eine 25-μl-Reaktionsmischung in PCR-Röhrchen vor:
12,5 μL 2x Taq-Polymerase-Mastermix
1,5 μL 10 μM Vorwärtsprimer
1,5 μL 10 μM Reverse Primer
4,5 μl nukleasefreies Wasser
5 μl neutralisierte Spermien-DNA-Probe - Gut mischen, indem Sie auf und ab pipettieren.
- Schleudern Sie die Proben kurz in einer Minizentrifuge herunter.
- Legen Sie die Proben mit den folgenden Einstellungen in den vorgeheizten Thermocycler:
Initiale Denaturierung: 95 °C für 3 min
35 Denaturierungszyklen (95 °C für 30 s), Glühen (55 °C für 30 s) und Verlängern (72 °C für 30 s)
Endstrecke: 72 °C für 5 min
HINWEIS: Es kann erforderlich sein, die Verlängerungstemperatur und/oder -zeit in Abhängigkeit von der spezifischen Taq-Polymerase und der erwarteten Länge des PCR-Amplifikationsprodukts anzupassen. - Nehmen Sie die PCR-Proben aus dem Thermocycler.
- Optional: Führen Sie den spenderspezifischen Restriktionsaufschluss an einem kleinen Volumen des PCR-Produkts durch, wobei mindestens 5-10 μl unverdautes PCR-Produkt für die Gelelektrophorese übrig bleiben.
- Bereiten Sie eine 30-μl-Reaktion in 0,2-ml-PCR-Röhrchen vor:
10 μl ungereinigtes PCR-Produkt
15 μl nukleasefreies Wasser
3 μL 10x Restriktionsenzympuffer
2 μL Restriktionsenzym (BstNI wird in den repräsentativen Ergebnissen für die Analyse des dnah10 knock-in-Allels verwendet)
HINWEIS: Viele Restriktionsenzyme sind in einem typischen PCR-Reaktionspuffer noch aktiv; Das Hinzufügen von Restriktion Digest Buffer kann jedoch dazu beitragen, die Ergebnisse zu verbessern. Beziehen Sie sich auf die spezifischen Anweisungen des Herstellers, um Probleme zu beheben, wenn Probleme auftreten. - Gut mischen, indem Sie auf und ab pipettieren.
- Schleudern Sie die Proben kurz in einer Minizentrifuge herunter.
- Legen Sie die Proben in einen Thermocycler mit den folgenden Einstellungen:
- Schneiden Sie das PCR-Amplikon bei 60 °C für 1 h.
Anmerkungen: Die Schnitttemperatur und die Inkubationszeit müssen möglicherweise für das verwendete Restriktionsenzym angepasst werden. - Inaktivieren Sie das Restriktionsenzym mit der erforderlichen Temperatur und Inkubationszeit, falls vom Hersteller angegeben.
HINWEIS: Das BstNI-Enzym erfordert keine Hitzeinaktivierung.
- Schneiden Sie das PCR-Amplikon bei 60 °C für 1 h.
- Bereiten Sie eine 30-μl-Reaktion in 0,2-ml-PCR-Röhrchen vor:
7. Durchführung einer Gelelektrophorese zur Trennung von PCR-Amplikons unterschiedlicher Größe
- Bereiten Sie 500 ml 0,5x TBE-Laufpuffer vor: 50 ml 10x TBE-Pufferkonzentrat zu 450 mL deionisiertem Wasser hinzufügen.
- Bereiten Sie 100 ml 4%iges Agarose-Gel in 0,5-fachem FSME-Laufpuffer vor: 4 g Agarose mit 10 μl 10.000-facher Gelfärbung zu 100 ml 0,5-fachem TBE-Laufpuffer hinzufügen. Mikrowelle stellen, bis sich das Agarosepulver vollständig aufgelöst hat.
- Gießen Sie die Gellösung in einen entsprechend großen Gelgussrahmen und führen Sie den Kamm auf einer Seite des Gels ein.
HINWEIS: Eine größere Gelgröße (empfohlen: 15 cm x 15 cm) ist für dieses Verfahren von Vorteil, da sie den Amplikons mehr Platz bietet, um sich auf dem Gel aufzulösen.- Wenn ein Restriktionsverdauungsprodukt analysiert werden soll, ist es am besten, es auf demselben Gel wie das PCR-Produkt laufen zu lassen, damit die Ergebnisse verglichen werden können. Passen Sie die Größe der Kammvertiefung nach Bedarf an, damit alle Proben auf das Gel passen.
- Lassen Sie das Gel bei Raumtemperatur, bis es fest ist. Entfernen Sie vorsichtig den Kamm. Entfernen Sie das Gel vorsichtig aus dem Gussrahmen.
- Gießen Sie die Gellösung in einen entsprechend großen Gelgussrahmen und führen Sie den Kamm auf einer Seite des Gels ein.
- Richten Sie das Gelelektrophorese-Rig ein.
- Positionieren Sie das Gel so im Elektrophorese-Rig, dass die Vertiefungen der negativen Elektrode am nächsten sind. Gießen Sie 0,5x TBE-Laufpuffer in den Elektrophorese-Rig, bis die Wells vollständig eingetaucht sind.
- Füllen Sie das Gel.
- Laden Sie 5 μl DNA-Leiter in die erste Vertiefung (wählen Sie eine DNA-Leiter, die DNA-Fragmente enthält, die in ihrer Größe dem PCR-Amplikon ähneln).
- Laden Sie mit einer neuen Pipettenspitze für jede Probe (einschließlich der Wildtyp-Kontrollprobe) 5-10 μl des PCR-Produkts in die verbleibenden Vertiefungen.
- Optional: Wenn ein Restriktionsverdauungsprodukt analysiert werden soll, laden Sie diese Proben ebenfalls auf das Gel.
HINWEIS: Es ist am besten, gleiche Mengen an DNA zwischen die PCR und das Restriktionsverdauungsprodukt zu laden. Beispielsweise wurden in der 30-μl-Restriktionsverdauungsreaktionsmischung 10 μl PCR-Produkt zugegeben. Daher war die DNA-Konzentration der 15 μl des Aufschlussprodukts vergleichbar mit den 5 μl des ungereinigten PCR-Produkts.
- Lassen Sie das Gel laufen.
- Schließen Sie die Elektroden an das Gelelektrophorese-Rig an und legen Sie mindestens 1 Stunde lang 150 V an, um eine ausreichende Trennung der Amplikone zu gewährleisten.
- Bilde das Gel ab, um die DNA-Banden zu sehen.
HINWEIS: Stark mutagenisierte CRISPR-gerichtete Loci werden in mehreren Bandgrößen ausgeführt, die eine Kombination von Insertions-/Deletionsallelen mit den Wildtyp-Amplikons darstellen.- Wenn sich die Gelbänder nach 1 h nicht aufgelöst haben, legen Sie 150 V in 15-minütigen Intervallen auf das Gel auf, bis die Banden auf dem Gel ausreichend getrennt sind.
- Optional: Wenn Sie nach der Insertion eines präzisen Edits, wie z. B. eines SNP, Epitop-Tags oder Fluoreszenz-Tags, suchen, screenen Sie die Spermien-DNA mit PCR-Methoden, die für den gewünschten Edit spezifisch sind. Wenn Sie beispielsweise eine spenderspezifische Restriktionsstelle identifizieren, ist im Vergleich zum PCR-Produkt der Samenprobe eine zusätzliche Bande im Restriktionsverdauungsprodukt vorhanden.
8. Isolierung keimbahnstabiler Allele
- Nach 2 Wochen Pause von der Spermienquetschprozedur kreuzen Sie die F0-Individuen, deren genomische DNA der Spermien die gewünschten Bearbeitungen enthielt.
HINWEIS: Die Spermienerzeugung aus einzelnen Spermatogonien-Klonen kann bei Zebrafischen zyklisch sein. Daher kann es sein, dass das gewünschte Allel zum Zeitpunkt der Kreuzung keine Spermien produziert. Aus diesem Grund kann es sein, dass eine zusätzliche Kreuzung mehr als einmal durchgeführt werden muss, um das Allel zu isolieren, das durch das anfängliche Screening impliziert wurde. - Sobald die ausgekreuzten F1-Fische geschlechtsreif sind, fahren Sie mit Standard-Genotypisierungsmethoden (z. B. Flossenclip-DNA) fort, wobei dasselbe PCR- und Gelelektrophoreseprotokoll verwendet wird, das während des Screenings der genomischen DNA der Spermien durchgeführt wird.
- Sanger sequenziert die PCR-Produkte, um die F1-Individuen mit der gleichen gewünschten Bearbeitung zu identifizieren.
- Optional: Wenn durch die gewünschte Bearbeitung eine Einschränkungswebsite eingeführt oder unterbrochen wird, überprüfen Sie mit einem geeigneten Einschränkungs-Digestprotokoll.
- In-Cross-F1-Heterozygoten mit der gleichen gewünschten Bearbeitung.
- Screenen Sie die F2-Nachkommen auf den erwarteten Phänotyp und/oder genomische Veränderungen.
Ergebnisse
Die in diesem Protokoll beschriebenen experimentellen Ansätze ermöglichen eine schnellere Identifizierung von Genom-Edits oder vermeintlich schädlichen Allelen, indem sie sich auf die Analyse von Tausenden von Genomen konzentrieren, die aus der Sammlung von F0-injizierten männlichen Spermien stammen. Abbildung 2 zeigt, wie die mit diesem Protokoll erzielten Ergebnisse zu interpretieren sind.
Um Mutationen im p2ry12-Locus zu erzeugen, wurden Zebrafischembryonen im Einzelzellstadium mit Cas9-Endonuklease und einer p2ry12-spezifischen sgRNA injiziert (Abbildung 2B, graue Hervorhebung). F0-injizierte Fische wurden bis zur Geschlechtsreife in das System gegeben, und von sechs einzelnen Männchen wurden Spermien gesammelt. Die DNA wurde unter Verwendung hoher Hitze und basischer Bedingungen (HotSHOT-DNA-Extraktion) aus den Spermienproben extrahiert und vor der PCR-Amplifikation des p2ry12-Locus neutralisiert. Die PCR-Produkte wurden an einem hochprozentigen (4%) Agarosegel für 1,5 h bei hoher Spannung (150 V) getrennt. In diesem Beispiel verlief das Wildtyp-Amplikon als einzelnes helles Band mit einer Länge von etwa 250 Basenpaaren (Abbildung 2C; WT). Im Gegensatz dazu liefen die F0-injizierten männlichen Amplikons, die Indels enthielten, als mehrere Banden auf dem Gel (Abbildung 2C; Bahnen #1-2, #5-8). Alternativ könnten die mutierten F0-injizierten männlichen Amplikons als Einzelband mit veränderter Gelbeweglichkeit verlaufen (nicht im p2ry12-Beispiel gezeigt, aber im PCR-Produkt des dnah10 F0-Männchens #2; Abbildung 2E). Auf dem p2ry12-Beispielgel war weniger klar, ob Probe #3 und Probe #4 im Vergleich zur Wildtyp-Bande Indels enthielten, so dass diese Individuen möglicherweise nicht die besten Gründerkandidaten sind. Die Isolierung von Allelen von F0-Männchen mit ausgeprägteren Amplikonveränderungen eignet sich am besten für die Vermehrung stabiler Keimbahnträger, da sie auf 4% Agarosegel leicht zu bewerten sind. Zum Beispiel schien die Spermienprobe des F0-Männchens #6 eine große Deletion in einem der Allele zu enthalten (Abbildung 2C; Spur 6; helles Band mit erhöhter Gelbeweglichkeit). Wenn dieses große Deletionsallel in der F1-Generation selektiert würde, wäre es leicht vom WT-Allel auf einem 4%igen Agarose-Gel zu unterscheiden. Wenn eine Sammlung verschiedener Allele gewünscht wird, könnte alternativ das F0-Männchen #7 ein effektiver Gründerkandidat sein, da sein PCR-Produkt mehrere diskrete Allele mit unterschiedlichen Mobilitäten zu enthalten schien. Sobald ein männlicher Gründerstamm ausgewählt wurde, kann das gewünschte Allel isoliert werden, indem das Individuum auf den ursprünglichen Wildtyp-Stamm zurückgekreuzt wird, der für die Injektionen verwendet wurde.
Um eine spezifische Knock-in-Mutation im dnah10-Gen zu erzeugen, wurden Zebrafischembryonen im Einzellstadium mit Cas9-Endonuklease, einer dnah10-spezifischen sgRNA (Abbildung 2D; graue Hervorhebung) und einem DNA-Donor-Oligonukleotid, das den gewünschten Edit (Abbildung 2D; rot) und eine spenderspezifische BstNI-Restriktionsstelle enthielt (Abbildung 2D) injiziert , Klammern). Dieses Design ermöglicht die einfache Identifizierung der Spenderintegration mit einem Restriktionsdigest nach PCR. Darüber hinaus verhindert dieses Design durch die Veränderung von Basenpaaren innerhalb der sgRNA-Erkennungsstelle (Abbildung 2D; graue Hervorhebung) die Cas9-Verdauung der Donorsequenz. Sobald die mit F0 injizierten Fische die Geschlechtsreife erreicht hatten, wurden Spermien gesammelt und die DNA mit der Hot-Shot-Methode extrahiert. Aus diesen Proben wurde der dnah10-Locus mittels PCR amplifiziert und die Produkte an einem hochprozentigen (4%) Agarosegel für 1 h bei hoher Spannung (150 V) getrennt. In diesem Beispiel verlief das Wildtyp-Amplikon als einzelnes helles Band mit einer Länge von etwa 400 Basenpaaren (Abbildung 2E; oberes Bild: WT). Im Gegensatz dazu liefen die F0-injizierten männlichen Amplikons, die Indels enthielten, als mehrere Bänder (Abbildung 2E; oberes Bild: Bahnen #1, #4 und #5) oder als einzelnes Band mit verminderter Gelbeweglichkeit (Abbildung 2E; oberes Bild: Spur #2). Um festzustellen, ob eine der Samenproben Spender-integrierte Allele enthielt, wurden die PCR-Produkte 1 h lang mit dem BstNI-Restriktionsenzym verdaut und das Produkt 1 h lang mit einem 4%igen Agarose-Gel bei Hochspannung (150 V) betrieben. Vergleicht man das unverdaute PCR-Produkt (Abbildung 2D; oberes Bild) mit dem aufgeschlossenen Produkt (Abbildung 2D; untere Tafeln), so ergeben sich Proben mit wahrscheinlicher Integration des Spenderkonstrukts. In diesem Beispiel hatte das F0-injizierte Männchen #1 eine zusätzliche Bande im verdauten Produkt, die im PCR-Produkt nicht vorhanden war (Abbildung 2D; unten: Bahn #1, eingekreist). Daher stellt dieses Männchen den besten Gründerkandidaten für die Etablierung einer Mutantenlinie mit dem gewünschten Knock-In dar.
Sobald das vermeintliche F0-Trägermännchen ausgekreuzt ist, muss das Allel in den F1-Nachkommen sequenzverifiziert werden. Es wird empfohlen, die aus dem F1-heterozygoten Fisch gewonnenen Amplikons direkt zu sequenzieren und anschließend heterozygote Allelanalysemethoden wie Poly Peak Parser 13 durchzuführen oder eine Vielzahl von Next-Generation-Sequencing-basierten Methoden wie MiSeq14 oder Hi-Tom15-Sequenzierung zu verwenden. Dies ist ein notwendiger und komplementärer Ansatz, um sicherzustellen, dass die präzise Bearbeitung oder schädliche Indel-Mutation tatsächlich in die Keimbahn gelangt und nicht nur ein Artefakt der Gelelektrophorese-Analyse ist. In der Tat kann die Verwendung von Next-Generation-Sequencing-Ansätzen zur Sequenzierung von Spermien-DNA von F0-Trägern problemlos anstelle der in diesem Protokoll beschriebenen Gelelektrophorese-Analyse verwendet werden. Eine leicht zu bewertende Methode zur Genotypisierung von Agarose-Gelen ist jedoch ein wirtschaftlicherer und egalitärerer Ansatz für die globale Gemeinschaft von Zebrafischforschern.

Abbildung 1: Aufbau für das Spermienquetschverfahren . (A) Der Schwamm wird bei geringer Vergrößerung mit Deckenbeleuchtung unter einem Stereomikroskop positioniert. (B) Der angefeuchtete 1 Zoll x 1 Zoll Schwamm, der mit einem ovalen Divot (gestrichelte Linie) geschnitten wird, wird verwendet, um den betäubten männlichen Fisch während des Eingriffs mit der ventralen Seite nach oben zu halten. (C) Anatomie der ventralen Seite des betäubten männlichen Fisches mit Darstellung der Afterflossen (af, rosa), der Beckenflossen (pf, blau) und der Kloake (Pfeil), wo die Spermien während des Eingriffs ausgestoßen werden. (C') Mit einer Filterzange wird der betäubte männliche Fisch sanft von den Kiemen bis zur Kloake gedrückt, während das ausgestoßene Sperma durch Kapillarwirkung in eine Glaspipette gezogen wird (weißer Pfeil). (D) Ein Kapillarröhrchen, das ausreichend ausgestoßene Spermien enthält (undurchsichtige Flüssigkeit; schwarze Klammer) für die nachgeschaltete DNA-Extraktion und -Analyse. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
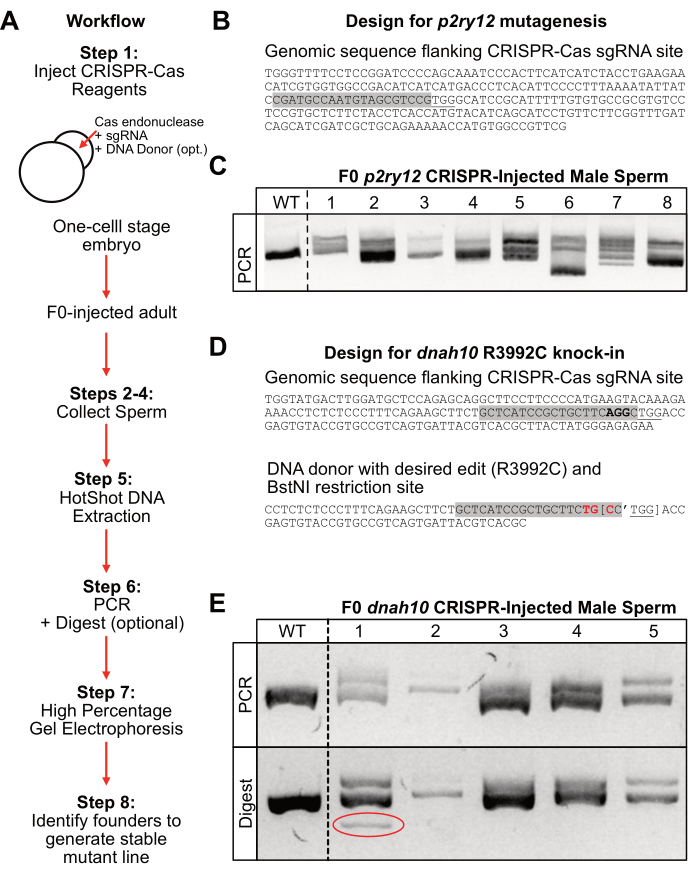
Abbildung 2: Arbeitsablauf und repräsentative Ergebnisse. (A) Allgemeiner Arbeitsablauf des Protokolls. (B) Repräsentatives Design der p2ry12 sgRNA-Stelle (graue Hervorhebung) mit einer Cas9-spezifischen PAM-Stelle (unterstrichen). (C) Repräsentative Ergebnisse der 4%igen Gelelektrophorese nach 1,5 h bei 150 V. Wildtyp (WT)-Kontrolle und F0-p2ry12 CRISPR-injizierte männliche Spermienproben (1-8) nach PCR-Amplifikation. (D) Repräsentatives Design der dnah10 sgRNA-Stelle (graue Hervorhebung) mit einer Cas9-spezifischen PAM-Stelle (unterstrichen) in der Nähe des Zielcodons (fett). DNA-Spendersequenz mit dem gewünschten Codon-Edit (rot fett) und BstNI-Restriktionsstelle (Klammern mit Apostroph, die die Schnittstelle markieren). (E) Repräsentative Ergebnisse der 4%igen Gelelektrophorese nach 1 h bei 150 V. Oben: PCR-Produkt des Wildtyps (WT) und F0 dnah10 CRISPR-injizierte männliche Spermienproben (1-5). Unten: BstNI-Restriktionsaufschlussprodukt der oben genannten Proben. Probe 1 zeigt die erfolgreiche Integration des Spenderkonstrukts anhand der zusätzlichen Bande nach der Verdauung (roter Kreis). Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Diskussion
Dieses Protokoll beschreibt eine Methode zur schnellen Charakterisierung von mutmaßlichen Genomänderungen oder gezielten Mutationen mit Hilfe der CRISPR-Cas-Technologie durch gezielte Analyse von F0-männlichen Spermiengenomen. Dieses Protokoll sollte für andere Tiermodelle zugänglich sein, bei denen Spermien ohne Euthanasie leicht für die Probenahme zur Verfügung stehen. Diese Methode erhöht den Durchsatz beim Screening auf gewünschte Bearbeitungen und ist besonders nützlich für die Identifizierung seltener HDR-vermittelter Knock-In-Ereignisse. Dieser Ansatz dient auch dazu, die Anzahl der Versuchstiere zu reduzieren, die verwendet werden, um einen stabilen Keimbahnschnitt zu finden, indem er das schnelle Screening von potenziell Tausenden von Genomen in einer Spermienprobe eines mutmaßlichen F0-Trägers erleichtert, was im Gegensatz zu traditionelleren Ansätzen steht, die das Screening von Hunderten von Embryonen erfordern können, die aus Kreuzungen von mutmaßlichen F0-Trägern stammen.
Dieses Protokoll baut auf etablierten Protokollen für die Spermiengewinnung im Zebrafisch 7,10,14,16,17,18 auf, indem es eine reproduzierbare Technik zur Identifizierung von Keimbahnveränderungen mittels hochauflösender Gelelektrophorese enthält. Dieser Ansatz kann leicht in jeden Standard-CRISPR/Cas-Workflow integriert werden, um den Durchsatz für das Screening und die Isolierung von Zielgenom-Editierungen zu erhöhen. Darüber hinaus eignet sich dieses Protokoll für Labore, die mit Personal mit einer Reihe von Schulungen und Erfahrungen besetzt sind, sowie für Lehrlabore. Unsere Methode der Samengewinnung ist jedoch nicht ausreichend für die Kryokonservierung, die in früheren Veröffentlichungen fachmännisch beschrieben wurde10,17.
Dieses Protokoll verwendet eine hochauflösende Agarose-Gelelektrophorese, um mögliche männliche Träger der gewünschten Indel- und präzisen genomischen Mutationen zu identifizieren. Die genomische DNA von Spermien ist jedoch für eine Vielzahl anderer Ansätze zugänglich, darunter die Analyse von Fluoreszenzfragmenten oder die Barcode-Sequenzierung14, die hochauflösende Schmelzanalyse18 oder die einfache Amplikon-Detektion von fluoreszierenden Markierungen oder Epitopen unter Verwendung von Standard-Gelelektrophorese-Ansätzen. Die nachgelagerte Validierung aller Allele mit Ansätzen wie Sanger-basierter oder Next-Generation-Sequenzierung muss vor Beginn der experimentellen Arbeit an putativen Allelen erfolgen. Bestehende Methoden für das Screening auf Mutationen in Embryonen, die den Next-Generation-Sequencing-Ansatz14 verwenden, könnten die in diesem Protokoll beschriebene Notwendigkeit einer Analyse an Agarosegelen umgehen. Eine Gel-Scorable-Genotypisierungsmethode ist jedoch ein kostengünstigerer und egalitärerer Ansatz, da nicht alle Labore während der Isolations- und Versuchsphasen der Arbeit mit jedem mutierten Stamm einfachen Zugang zu billigen Next-Generation-Sequenzierungsmethoden haben.
Zusammenfassend lässt sich sagen, dass dieses Protokoll Schritt-für-Schritt-Anleitungen für das reproduzierbare Screening von Spermiengenomen von CRISPR/Cas-editierten Männern bietet, so dass weniger Kreuzungen und weniger PCR-Screenings von Embryonen erforderlich sind, um nach gewünschten Bearbeitungen zu suchen. Die Anwendung dieser Methode verringert die Anzahl der Fische, die erzeugt und analysiert werden müssen, um die editierten Allele von Interesse erfolgreich zu identifizieren, was auch den Zeit- und Kostenaufwand für die Generierung stabiler Linien reduziert.
Offenlegungen
Nichts.
Danksagungen
Wir danken Anna Hindes von der Washington University School of Medicine für ihre anfänglichen Bemühungen, mit der Hot-Shot-Methode genomische DNA von Spermien in guter Qualität zu erhalten. Diese Arbeit wurde vom National Institute of Arthritis and Muskuloskeletal and Skin Diseases der National Institutes of Health unter Auszeichnung (R01AR072009 an R.S.G.) finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Agarose powder | Fisher BioReagents | BP1356-100 | |
| Breeding tanks | Carolina Biological | 161937 | |
| BstNI Restriction Enzyme | NEB | R0168S | |
| Cas9 Endonuclease | IDT | 1081060 | |
| DNA Ladder, 100 bp | Thermo Scientific | FERSM0241 | |
| dnah10 donor construct | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry. Phosphorothioate bond on the donor at the first three phosphate bonds on both the 5’ and 3’ ends (5'-CCTCTCTCCCTTTCAGAAGCTTC TGCTCATCCGCTGCTTCTGCCT GGACCGAGTGTACCGTGCCGTC AGTGATTACGTCACGC-3') | |
| dnah10 forward primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CATGGAACTCTTTCCTAATGAGT TTGGC-3') | |
| dnah10 reverse primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry ('5-AGTAGAGATCACACATCAACAGA ATACAGC-3') | |
| dnah10 synthetic sgRNA | Synthego | Synthetic sgRNA, target sequence: 5'-GCTCATCCGCTGCTTCAGGC-3' | |
| Electrophoresis power supply | Thermo Scientific | 105ECA-115 | |
| Filter forceps | Millipore | XX6200006P | |
| Fish (system) water | Generic | n/a | |
| Gel electrophoresis system (including casting frame, comb, and electrophoresis chamber) | Thermo Scientific | B2 | |
| Gel imaging light box | Azure Biosystems | AZI200-01 | |
| Gel stain, 10000X | Invitrogen | S33102 | |
| Glass bowl, 250 mL | Generic | n/a | |
| Isolation tanks, 0.8 L | Aquaneering | ZT080 | |
| Microcap capillary tube with bulb, 20 µL | Drummond | 1-000-0020/CA | |
| Minicentrifuge | Bio-Rad | 12011919EDU | |
| Micropipettes, various with appropriate tips | Generic | n/a | |
| Microwave | Generic | n/a | |
| Nuclease free water | Promega | P119-C | |
| Paper towels | Generic | n/a | |
| PCR tubes, 0.2 mL | Bioexpress | T-3196-1 | |
| Plastic spoon, with drilled holes/slots | Generic | n/a | |
| KCl solution, 0.2 M RNAse Free | Sigma-Aldrich | P9333 | |
| p2ry12 forward primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CCCAAATGTAATCCTGACCAGT -3') | |
| p2ry12 reverse primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CCAGGAACACATTAACCTGGAT -3') | |
| p2ry12 synthetic sgRNA | Synthego | Synthetic sgRNA, target sequence: 5'-GGCCGCACGAGGTCTCCGCG-3' | |
| Restriction Enzyme 10X Buffer | NEB | B6003SVIAL | |
| NaOH solution, 50 mM | Thermo Scientific | S318; 424330010 | |
| Sponge, 1-inch x 1-inch cut with small oval divot | Generic | n/a | |
| Stereomicroscope | Zeiss | Stemi 508 | |
| Taq polymerase master mix, 2X | Promega | M7122 | |
| TBE Buffer Concentrate, 10X | VWR | E442 | |
| Thermal Cycler | Bio-Rad | 1861096 | |
| Tissue paper | Fisher Scientific | 06-666 | |
| Tricaine-methanesulfonate solution (Syncaine, MS-222), 0.016% in fish water (pH 7.0±0.2) | Syndel | 200-266 | |
| Tris Base, 1M (Buffered with HCl to ph 8.0) | Promega | H5131 |
Referenzen
- Auer, T. O., Duroure, K., De Cian, A., Concordet, J. P., Del Bene, F. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair. Genome Research. 24 (1), 142-153 (2014).
- Hwang, W. Y., et al. Efficient genome editing in zebrafish using a CRISPR-Cas system. Nature Biotechnology. 31 (3), 227-229 (2013).
- Jao, L. E., Wente, S. R., Chen, W. Efficient multiplex biallelic zebrafish genome editing using a CRISPR nuclease system. Proceedings of the National Academy of Sciences of the United States of America. 110 (34), 13904-13909 (2013).
- Troutwine, B. R., et al. The Reissner fiber is highly dynamic in vivo and controls morphogenesis of the spine. Current Biology. 30 (12), 2353-2362 (2020).
- Xu, Y., Li, Z. CRISPR-Cas systems: Overview, innovations and applications in human disease research and gene therapy. Computational and Structural Biotechnology Journal. 18, 2401-2415 (2020).
- Creaser, C. W. The technic of handling the zebrafish (Brachydanio rerio) for the production of eggs which are favorable for embryological research and are available at any specified time throughout the year. Copeia. 1930 (4), 159-161 (1934).
- Carmichael, C., Westerfiel, M., Varga, Z. M. Cryopreservatin and in vitro fertilization at the zebrafish international resource center. Methods in Molecular Biology. 546, 45-65 (2009).
- Wang, Y., Troutwine, B. R., Zhang, H., Gray, R. S. The axonemal dynein heavy chain 10 gene is essential for monocilia motility and spine alignment in zebrafish. Developmental Biology. 482, 82-90 (2021).
- Gray, R. S., et al. Postembryonic screen for mutations affecting spine development in zebrafish. Developmental Biology. 471, 18-33 (2021).
- Brocal, I., et al. Efficient identification of CRISPR/Cas9-induced insertions/deletions by direct germline screening in zebrafish. BMC Genomics. 17, 259 (2016).
- Davis, M. W., Jorgensen, E. M. ApE, A Plasmid Editor: A freely available DNA manipulation and visualization program. Frontiers in Bioinformatics. 2, 816619 (2022).
- Labun, K., et al. CHOPCHOP v3: Expanding the CRISPR web toolbox beyond genome editing. Nucleic Acids Research. 47, 171-174 (2019).
- Hill, J. T., et al. Poly Peak Parser: Method and software for identification of unknown indels using sanger sequencing of polymerase chain reaction products. Developmental Dynamics. 243 (12), 1632-1636 (2014).
- Varshney, G. K., et al. High-throughput gene targeting and phenotyping in zebrafish using CRISPR/Cas9. Genome Research. 25 (7), 1030-1042 (2015).
- Liu, Q., et al. Hi-TOM: A platform for high-throughput tracking of mutations induced by CRISPR/Cas systems. Science China. Life Sciences. 62 (1), 1-7 (2019).
- Sorlien, E. L., Witucki, M. A., Ogas, J. Efficient production and identification of CRISPR/Cas9-generated gene knockouts in the model system Danio rerio. Journal of Visualized Experiments. (138), e56969 (2018).
- Draper, B. W., Moens, C. B. A high-throughput method for zebrafish sperm cryopreservation and in vitro fertilization. Journal of Visualized Experiments. (29), e1395 (2009).
- Parant, J. M., George, S. A., Pryor, R., Wittwer, C. T., Yost, H. J. A rapid and efficient method of genotyping zebrafish mutants. Developmental Dynamics. 238 (12), 3168-3174 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten