Method Article
Screening dello sperma per il rapido isolamento delle modifiche germinali nel pesce zebra
In questo articolo
Riepilogo
Le tecnologie CRISPR-Cas hanno rivoluzionato il campo dell'editing del genoma. Tuttavia, trovare e isolare la modifica della linea germinale desiderata rimane un importante collo di bottiglia. Pertanto, questo protocollo descrive un metodo robusto per lo screening rapido dello sperma di zebrafish iniettato con F0 CRISPR per le modifiche della linea germinale utilizzando PCR standard, digestione di restrizione e tecniche di elettroforesi su gel.
Abstract
L'avvento delle tecnologie mirate di nucleasi CRISPR-Cas ha rivoluzionato la capacità di eseguire un accurato editing del genoma in sistemi modello sia consolidati che emergenti. I sistemi di editing del genoma CRISPR-Cas utilizzano un RNA guida sintetico (sgRNA) per indirizzare un'endonucleasi associata a CRISPR (Cas) a specifici loci del DNA genomico, dove l'endonucleasi Cas genera una rottura a doppio filamento. La riparazione delle rotture del doppio filamento mediante meccanismi intrinseci soggetti a errori porta a inserimenti e / o delezioni, interrompendo il locus. In alternativa, l'inclusione di donatori di DNA a doppio filamento o oligonucleotidi di DNA a singolo filamento in questo processo può suscitare l'inclusione di precise modifiche del genoma che vanno dai polimorfismi a singolo nucleotide a piccoli tag immunologici o persino grandi costrutti proteici fluorescenti. Tuttavia, un importante collo di bottiglia in questa procedura può essere trovare e isolare la modifica desiderata nella linea germinale. Questo protocollo delinea un metodo robusto per lo screening e l'isolamento delle mutazioni germinali in loci specifici in Danio rerio (zebrafish); Tuttavia, questi principi possono essere adattabili in qualsiasi modello in cui è possibile la raccolta di spermatozoi in vivo .
Introduzione
Il sistema CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats)/Cas è un potente strumento per eseguire mutagenesi loci-specifica e editing preciso del genoma nel sistema modello Danio rerio (zebrafish) 1,2,3,4. La Cas-ribonucleoproteina (RNP) è composta da due componenti principali: un'endonucleasi Cas (comunemente Cas9 o Cas12a) e un RNA guida sintetico locus-specifico (sgRNA)5. Insieme, il Cas-RNP genera una rottura a doppio filamento (DSB) nel locus desiderato che può essere riparata da uno dei due meccanismi di riparazione intrinseca. Il meccanismo di riparazione della giunzione finale non omologa (NHEJ) è soggetto a errori e spesso si traduce in una varietà di inserzioni o delezioni (indel) attorno al DSB. Questi indels possono essere deleteri se introducono una mutazione frameshift o un arresto prematuro nella sequenza proteica risultante. In alternativa, il meccanismo di riparazione diretta dall'omologia (HDR) utilizza un modello di donatore con regioni di omologia che circondano il sito DSB per riparare il danno. I ricercatori possono sfruttare il sistema HDR per generare modifiche genomiche precise. In particolare, possono co-iniettare un costrutto di donatore di DNA a doppio filamento che contiene le modifiche desiderate e le regioni di omologia che fiancheggiano il sito DSB nel genoma. La maggiore economia di scala per questi componenti CRISPR prodotti commercialmente ha notevolmente ridotto le barriere allo screening di più loci e alla creazione di sforzi su larga scala per l'editing preciso del genoma. Tuttavia, nei modelli animali che si riproducono sessualmente, un importante collo di bottiglia è l'identificazione e l'isolamento di animali mutanti stabili alla linea germinale.
Il sistema modello zebrafish presenta diverse qualità chiave che ne migliorano l'uso negli studi genetici inversi. Sono facili da allevare in gran numero con attrezzature di base per l'alloggiamento acquatico e le femmine mostrano un'elevata fecondità tutto l'anno6. Inoltre, la loro deposizione esterna delle uova e la fertilizzazione li rendono suscettibili alla microiniezione di componenti CRISPR / Cas. Il Cas-RNP viene comunemente iniettato in embrioni di zebrafish a una cellula per generare DSB / riparazione che è, in teoria, ereditato da tutte le cellule figlie. Tuttavia, i genomi diploidi richiedono due eventi DSB/riparazione per mutagenizzare entrambi i cromosomi omologhi. Inoltre, sebbene Cas-RNP venga iniettato allo stadio di una cellula, il DSB / riparazione potrebbe non verificarsi fino a punti successivi nello sviluppo. Insieme, questi fattori contribuiscono alla natura a mosaico del pesce iniettato con F0. Una pratica comune è quella di incrociare i pesci iniettati con F0 e controllare la progenie F1 per indel/modifiche specifiche. Tuttavia, poiché non tutti i pesci iniettati con F0 possiedono mutazioni germinali, questa pratica si traduce in molti incroci improduttivi che non generano la modifica desiderata. Lo screening della linea germinale F0 piuttosto che del tessuto somatico F1 aumenta la probabilità di isolare la modifica germinale desiderata e riduce il numero di animali necessari in questo processo.
Lo sperma può essere facilmente raccolto dal pesce zebra iniettato con F0 senza bisogno di eutanasia. Questa caratteristica consente la crioconservazione e la riderivazione di scorte di spermatozoi congelati7 ma può anche essere sfruttata per vagliare, identificare e isolare rapidamente i portatori della linea germinale delle mutazioni genomiche desiderate 8,9. Brocal et al. (2016) hanno precedentemente descritto un metodo basato sul sequenziamento per lo screening delle modifiche della linea germinale nel pesce zebra maschio iniettato con F010. Sebbene utile per identificare gli alleli mutati presenti nella linea germinale, questo approccio può diventare costoso in termini di produttività elevata e potrebbe non essere accessibile a tutti i laboratori. Al contrario, il protocollo attuale offre una strategia basata sull'elettroforesi accessibile ed economica per identificare le modifiche della linea germinale. In particolare, questo protocollo delinea un metodo robusto per lo screening e l'isolamento delle mutazioni germinali in loci specifici utilizzando l'elettroforesi su gel di agarosio ad alta risoluzione. Inoltre, questo protocollo descrive una strategia simile per identificare l'integrazione riuscita di un costrutto donatore contenente modifiche specifiche. Come sempre, se si desiderano modifiche specifiche, le strategie basate sul sequenziamento possono essere eseguite in tandem con il protocollo descritto di seguito. Sebbene questo protocollo sia specifico per il sistema modello del pesce zebra, questi principi dovrebbero essere adattabili a qualsiasi modello in cui la raccolta di spermatozoi è una procedura di routine. Insieme, queste strategie consentiranno l'identificazione di maschi iniettati in F0 con indels/edits germinali che possono essere risolti su un gel dopo reazione a catena della polimerasi standard (PCR) e/o digestione di restrizione.
Protocollo
Questo studio è stato condotto in linea con le linee guida della Guida per la cura e l'uso degli animali da laboratorio del National Institutes of Health. Il protocollo è stato approvato dall'Università del Texas presso il Comitato per la cura e l'uso degli animali di Austin (AUP-2021-00254).
1. Progettazione dello sgRNA per la mutagenesi di CRISPR
- Ottenere la sequenza di esoni contenente i loci mirati.
- Progettare un RNA guida sintetico (sgRNA) con un sito di motivo adiacente protospaziatore (PAM) specifico per l'endonucleasi Cas.
- Se si co-inietta un costrutto donatore, progettare l'sgRNA in modo da avere un sito di taglio previsto il più vicino possibile alle modifiche desiderate.
NOTA: APE è un software gratuito con uno strumento per trovare siti sgRNA Cas911. In alternativa, è possibile utilizzare strumenti come CHOPCHOP, che offre un'interfaccia online per la progettazione di sgRNA che considera i diversi siti PAM di Cas9 e altre endonucleasi Cas12.
- Se si co-inietta un costrutto donatore, progettare l'sgRNA in modo da avere un sito di taglio previsto il più vicino possibile alle modifiche desiderate.
- Progetta primer avanti e indietro per amplificare la regione che circonda il sito di taglio CRISPR / Cas progettato. Il prodotto PCR dovrebbe essere lungo circa 200-400 paia di basi (bp) per essere in grado di risolvere piccoli indels su un gel più avanti nel protocollo.
NOTA: CHOPCHOP11 progetta automaticamente primer per ogni sgRNA che emette. - Facoltativo: progettare un costrutto di donatore di DNA con le modifiche desiderate.
- Ottenere la sequenza genomica dell'esone contenente i loci della mutazione desiderata.
NOTA: le sequenze introniche possono variare tra gli individui, quindi è meglio evitare l'inclusione di sequenze introniche nel costrutto del donatore in quanto ciò potrebbe interferire con il percorso di riparazione dipendente dall'omologia. - Se si desidera un cambiamento specifico dell'amminoacido, modificare di conseguenza il codone specifico.
NOTA: quando si apportano queste modifiche, prestare attenzione alla frequenza del codone. Se possibile, scegli un codone con un uso elevato. - Per prevenire la digestione CRISPR / Cas del costrutto donatore, effettuare ulteriori mutazioni sinonimi che (i) interrompono il PAM e / o (ii) interrompono la sequenza di sgRNA, con una maggiore preferenza per apportare modifiche non sinonimi più vicine al sito PAM.
- Utilizzare un cercatore di siti di restrizione (APE11 ha anche questa caratteristica) per trovare siti di restrizione unici (quelli che si verificano solo una volta) nel prodotto PCR previsto della sequenza wild-type.
- Ripetere questo processo per il prodotto PCR previsto che riflette la corretta integrazione del costrutto del donatore.
- Confrontare l'elenco dei siti di restrizione tra le sequenze wild-type e quelle integrate nel donatore per trovare un sito di restrizione presente solo nella sequenza modificata.
- Se non viene trovato alcun sito di restrizione univoco, continuare ad apportare modifiche sinonimi all'interno del costrutto del donatore fino a quando questo non viene raggiunto.
NOTA: Se possibile, continuare a fare mutazioni sinonimi che interromperebbero il legame dell'sgRNA al costrutto del donatore.
- Tagliare la sequenza del donatore a una dimensione gestibile (50-100 bp) con almeno 20 bp di omologia che circonda il sito di taglio CRISPR / Cas previsto e le modifiche, se possibile.
- Ottenere la sequenza genomica dell'esone contenente i loci della mutazione desiderata.
- Ottenere gli oligonucleotidi sgRNA e/o donatori di DNA progettati, nonché tutti i primer necessari per la genotipizzazione.
NOTA: Per impedire la digestione degli oligonucleotidi del donatore di DNA da parte delle nucleasi, si raccomanda di modificare i primi tre legami fosfato sulle estremità 5' e 3' degli oligonucleotidi del DNA con fosforotioato. - Iniettare 1 nL della miscela iniettabile in embrioni monocellulari. Tipicamente, una miscela di iniezione standard contiene quanto segue:
1 μL di sgRNA locus-specifico da 25 μM
1 μL di endonucleasi Cas9 25 μM
Opzionale: 1 μL di costrutto di donatore di DNA da 3 μM- Portare il volume totale a 5 μL con KCl privo di RNasi 0,1 M.
- Consentire agli individui iniettati F0 di svilupparsi in adulti sessualmente maturi. Il protocollo rimanente si concentra sullo screening dei maschi iniettati con F0 per le modifiche della linea germinale.
2. Allestimento delle vasche di allevamento
- Primi maschi F0 per l'accoppiamento allestendo vasche di riproduzione con divisori tra i maschi e le femmine F0.
- Per ridurre al minimo il numero di vasche di riproduzione necessarie, allestire tre maschi di fronte a due femmine in ogni vasca.
- Impostare il maggior numero di maschi F0 che possono essere efficacemente sottoposti a screening e ospitati come individui dopo la raccolta dello sperma. Il genotipo delle femmine non è importante poiché i loro gameti non saranno raccolti durante questa procedura.
- Istituire una vasca di riproduzione di maschi selvatici di fronte alle femmine. Lo sperma wild-type servirà come controllo per l'interpretazione dei risultati più avanti nella procedura.
- Per ridurre al minimo il numero di vasche di riproduzione necessarie, allestire tre maschi di fronte a due femmine in ogni vasca.
- Incubare le vasche di riproduzione preparate durante la notte.
3. Preparazione dei materiali per la procedura di raccolta dello sperma
- Preparare una soluzione anestetizzante sufficiente (0,016% tricaina-HCl in acqua di pesce) per riempire una piccola ciotola di vetro.
- Erogare 40 μL di 50 mM NaOH in provette PCR da 0,2 mL. Preparare una provetta per ogni campione di sperma che verrà raccolto. Conservare in ghiaccio.
- Preparare le singole vasche da 0,8 L per ogni pesce che verrà campionato durante la procedura. Queste vasche ospiteranno i pesci campionati fino al completamento della genotipizzazione.
NOTA: Questo è il fattore principale che limita il numero di pesci che possono essere efficacemente sottoposti a screening in questo protocollo. Garantire uno spazio adeguato per ospitare gli individui campionati fino al completamento della genotipizzazione. - Inumidire una spugna da 1 pollice x 1 pollice (tagliata con un divot poco profondo di forma ovale per tenere un maschio di zebrafish lato ventrale verso l'alto durante la procedura) in acqua di pesce e spremere il liquido in eccesso.
- Posizionare la spugna nel campo visivo di uno stereomicroscopio a basso ingrandimento (Figura 1A).
- Posizionare l'illuminazione obliqua dall'alto per una visione ottimale.
4. Anestetizzare il pesce maschio e raccogliere lo sperma
- Preparare un tubo capillare pulito.
- Agitare delicatamente il contenitore del tubo per rilasciare un singolo tubo capillare.
- Posizionare il tubo capillare nel bulbo fornito e metterlo da parte.
- Trasferire un pesce maschio nella soluzione anestetizzante preparata (0,016% tricaina-HCl in acqua di pesce).
- Mescolare delicatamente la soluzione con un mestolo forato per accelerare il processo di anestesia.
- Una volta che il movimento dell'opercolo (branchia) rallenta (circa 1-2 minuti), procedere con la raccolta degli spermatozoi.
- Usa il mestolo forato per trasferire il pesce su una pila pulita di carta assorbente.
- Utilizzare il mestolo forato per arrotolare delicatamente il pesce per rimuovere l'acqua in eccesso.
- Tamponare delicatamente il pesce con carta velina pulita e piegata per rimuovere l'acqua residua.
- Utilizzare il mestolo forato per trasferire il pesce nella spugna preparata (Figura 1B).
- Posizionare il pesce con il lato ventrale verso l'alto con la testa più vicina alla mano dominante della persona che esegue la procedura.
- Asciugare delicatamente l'area intorno alle pinne anali con carta velina pulita e piegata.
- Raccogli lo sperma.
- Utilizzando l'estremità del tubo capillare, spostare delicatamente le pinne pelviche lateralmente lontano dalla linea mediana per esporre la cloaca (Figura 1C).
- Posizionare l'estremità del tubo capillare vicino alla cloaca.
- Usando la pinza, stringere delicatamente i lati del pesce iniziando appena sotto le branchie e terminando alla cloaca (Figura 1C').
- Raccogliere lo sperma nel tubo capillare per azione capillare. Circa 1 μL è sufficiente per questa procedura (Figura 1D).
NOTA: Alcuni laboratori riferiscono di utilizzare punte di pipette p10 invece di tubi capillari per la raccolta dello sperma. - Se lo sperma non viene rilasciato con una leggera pressione, riportare il pesce nell'acqua del sistema.
- Monitor per il recupero dall'anestesia.
- Non spremere duramente il pesce per espellere lo sperma, poiché ciò può ferire il pesce.
- Riposare il pesce per 2 settimane prima di tentare di raccogliere nuovamente lo sperma.
- Dopo la raccolta, posizionare il pesce in una vasca di isolamento pulita con acqua di sistema per l'alloggiamento individuale.
- Monitorare il pesce per il recupero dall'anestesia.
- Riposare il pesce per 2 settimane prima di tentare di raccogliere nuovamente lo sperma.
- Posizionare il tubo capillare con lo sperma raccolto nel tubo PCR preparato (con 40 μL di 50 mM NaOH).
- Spremere il bulbo di gomma per espellere il campione raccolto.
- Smaltire il tubo capillare usato in un contenitore di rifiuti di vetro approvato.
- Incubare i campioni sul ghiaccio fino a quando tutti i maschi sono stati spremuti.
- Ripetere i passaggi 4.1-4.5 per ogni singolo maschio.
- Etichettare i serbatoi di isolamento e i campioni di sperma in modo tale che i risultati finali della genotipizzazione degli spermatozoi possano essere ricondotti all'individuo corrispondente con mutazioni putative.
- Monitora tutti gli individui e assicurati che siano in posizione verticale ed esplorino i loro serbatoi prima di rimettere i serbatoi sul sistema.
- Metti le femmine utilizzate per l'innesco nelle rispettive vasche sul sistema.
- Se un pesce non si riprende dall'anestesia dopo 10 minuti, seguire la procedura di eutanasia approvata dall'istituzione associata al laboratorio.
- Esempio: raffreddare rapidamente l'individuo in un bagno di ghiaccio con acqua di pesce a 2-4 °C.
- Smaltire il pesce eutanasia in un apposito contenitore per i rifiuti delle carcasse animali secondo la procedura approvata dall'istituzione associata al laboratorio.
5. Estrazione del DNA dai campioni di sperma
- Girare brevemente tutti i campioni di sperma in una minicentrifuga e posizionare i tubi PCR in un termociclatore.
- Chiudere il coperchio del termociclatore.
- Eseguire le seguenti impostazioni nel termociclatore:
- Riscaldare i campioni per 40 minuti a 95 °C.
- Raffreddare i campioni a 25 °C.
- Rimuovere i campioni dal termociclatore.
- Con una punta di pipetta pulita per ciascun campione, neutralizzare il pH aggiungendo 10 μL di 1 M Tris-HCl (tamponato a pH 8).
- Mescolare bene pipettando su e giù.
- Girare brevemente i campioni in una minicentrifuga.
- I campioni di DNA genomico possono essere conservati per diversi giorni a 4 °C o posti a -20 °C per un massimo di 6 mesi.
6. Amplificazione PCR (e/o digestione di restrizione) del locus desiderato
- Amplifica il locus desiderato utilizzando un protocollo PCR standard.
- Preriscaldare il termociclatore alla temperatura di denaturazione iniziale nella reazione PCR sottostante.
- Preparare una miscela di reazione da 25 μL per ogni campione di sperma in provette per PCR:
12,5 μL di 2x Taq Polymerase Master Mix
1,5 μL di primer forward da 10 μM
1,5 μL di primer inverso da 10 μM
4,5 μL di acqua esente da nucleasi
5 μL di campione di DNA spermatico neutralizzato - Mescolare bene pipettando su e giù.
- Girare brevemente i campioni in una minicentrifuga.
- Posizionare i campioni nel termociclatore preriscaldato con le seguenti impostazioni:
Denaturazione iniziale: 95 °C per 3 min
35 cicli di denaturazione (95 °C per 30 s), ricottura (55 °C per 30 s) ed estensione (72 °C per 30 s)
Estensione finale: 72 °C per 5 min
NOTA: Potrebbe essere necessario regolare la temperatura e/o il tempo di estensione in base alla Taq polimerasi specifica e alla lunghezza prevista del prodotto di amplificazione PCR. - Rimuovere i campioni di PCR dal termociclatore.
- Facoltativo: eseguire il digest di restrizione specifico del donatore su un piccolo volume del prodotto PCR, lasciando almeno 5-10 μL di prodotto PCR non digerito per l'elettroforesi su gel.
- Preparare una reazione da 30 μL in provette da 0,2 mL PCR:
10 μL di prodotto PCR non purificato
15 μL di acqua senza nucleasi
3 μL di tampone enzimatico di restrizione 10x
2 μL di enzima di restrizione (BstNI è usato nei risultati rappresentativi per l'analisi dell'allele knock-in dnah10 )
NOTA: Molti enzimi di restrizione sono ancora attivi in un tipico tampone di reazione PCR; Tuttavia, l'aggiunta di Restriction Digest Buffer può contribuire a migliorare i risultati. Fare riferimento alle istruzioni specifiche del produttore per la risoluzione dei problemi in caso di problemi. - Mescolare bene pipettando su e giù.
- Girare brevemente i campioni in una minicentrifuga.
- Posizionare i campioni in un termociclatore con le seguenti impostazioni:
- Tagliare l'amplicone PCR a 60 °C per 1 ora.
NOTA: Potrebbe essere necessario regolare la temperatura di taglio e il tempo di incubazione per l'enzima di restrizione specifico utilizzato. - Inattivare l'enzima di restrizione con la temperatura e il tempo di incubazione richiesti, se indicato dal produttore.
NOTA: L'enzima BstNI non richiede inattivazione termica.
- Tagliare l'amplicone PCR a 60 °C per 1 ora.
- Preparare una reazione da 30 μL in provette da 0,2 mL PCR:
7. Esecuzione dell'elettroforesi su gel per separare ampliconi PCR di varie dimensioni
- Preparare 500 mL di tampone corrente 0,5x TBE: aggiungere 50 mL di concentrato tampone 10x TBE a 450 mL di acqua deionizzata.
- Preparare 100 ml di gel di agarosio al 4% in tampone corrente TBE 0,5x: aggiungere 4 g di agarosio con 10 μL di colorante gel 10.000x a 100 ml di tampone corrente 0,5x TBE. Microonde fino a quando la polvere di agarosio è completamente sciolta.
- Versare la soluzione di gel in un telaio di fusione in gel di dimensioni appropriate e inserire il pettine su un lato del gel.
NOTA: Una dimensione del gel più grande (consigliata: 15 cm x 15 cm) è vantaggiosa per questa procedura, in quanto fornisce agli ampliconi più spazio per risolvere sul gel.- Se un prodotto digestivo di restrizione deve essere analizzato, è meglio eseguirlo sullo stesso gel del prodotto PCR in modo che i risultati possano essere confrontati. Regolare le dimensioni del pettine secondo necessità per adattare tutti i campioni sul gel.
- Lasciare il gel a temperatura ambiente fino a solidificarsi. Rimuovere con cura il pettine. Rimuovere con attenzione il gel dal telaio di fusione.
- Versare la soluzione di gel in un telaio di fusione in gel di dimensioni appropriate e inserire il pettine su un lato del gel.
- Impostare il rig di elettroforesi su gel.
- Posizionare il gel nel rig di elettroforesi in modo che i pozzetti siano più vicini all'elettrodo negativo. Versare un buffer di funzionamento TBE 0,5x nel carro di elettroforesi fino a quando i pozzetti non sono completamente sommersi.
- Caricare il gel.
- Caricare 5 μL di scala del DNA nel primo pozzetto (scegliere una scala di DNA che contenga frammenti di DNA di dimensioni simili all'amplicone PCR).
- Utilizzando una nuova punta per pipetta per ciascun campione (incluso il campione di controllo wild-type), caricare 5-10 μL del prodotto PCR nei pozzetti rimanenti.
- Facoltativo: se è necessario analizzare un prodotto digestivo di restrizione, caricare anche questi campioni sul gel.
NOTA: è meglio caricare quantità uguali di DNA tra la PCR e il prodotto digestivo di restrizione. Ad esempio, nella miscela di reazione digestiva a restrizione da 30 μL, sono stati aggiunti 10 μL di prodotto PCR. Pertanto, la concentrazione di DNA dei 15 μL del prodotto digesto era paragonabile ai 5 μL del prodotto PCR non purificato.
- Esegui il gel.
- Collegare gli elettrodi al rig di elettroforesi su gel e applicare 150 V per almeno 1 ora per garantire un'adeguata separazione degli ampliconi.
- Immagina il gel per visualizzare le bande di DNA.
NOTA: I loci fortemente mutagenizzati mirati a CRISPR verranno eseguiti come dimensioni di banda multiple che rappresentano una combinazione di alleli di inserzione/delezione con gli ampliconi wild-type.- Se le bande di gel non si sciolgono dopo 1 ora, applicare 150 V sul gel per intervalli di 15 minuti fino a quando le bande sono sufficientemente separate sul gel.
- Facoltativo: se lo screening per l'inserimento di una modifica precisa come un SNP, un tag epitopo o un tag fluorescente, eseguire lo screening del DNA spermatico con metodi PCR specifici per la modifica desiderata. Ad esempio, quando si identifica un sito di restrizione specifico del donatore, una banda aggiuntiva sarà presente nel prodotto digestivo di restrizione rispetto al prodotto PCR del campione di sperma.
8. Isolamento degli alleli stabili della linea germinale
- Dopo 2 settimane di riposo dalla procedura di spremitura dello sperma, superare gli individui F0 il cui DNA genomico dello sperma conteneva le modifiche desiderate.
NOTA: La generazione di spermatozoi da singoli cloni di spermatogoni può essere ciclica nel pesce zebra; Pertanto, l'allele desiderato potrebbe non produrre spermatozoi al momento dell'incrocio. Per questo motivo, potrebbe essere necessario eseguire più di una volta ulteriori attraversamenti per isolare l'allele implicato dallo screening iniziale. - Una volta che i pesci incrociati F1 sono sessualmente maturi, procedere con metodi di genotipizzazione standard (ad esempio, DNA pinna-clip) utilizzando lo stesso protocollo di PCR e elettroforesi su gel eseguito durante lo screening del DNA genomico degli spermatozoi.
- Sanger sequenzia i prodotti PCR per identificare gli individui F1 con la stessa modifica desiderata.
- Facoltativo: se la modifica desiderata introduce o interrompe un sito con restrizioni, eseguire lo screening con un protocollo digest di restrizione appropriato.
- Eterozigoti F1 incrociati con la stessa modifica desiderata.
- Screening della progenie F2 per il fenotipo atteso e/o le modifiche genomiche.
Risultati
Gli approcci sperimentali descritti in questo protocollo consentono la più rapida identificazione di modifiche del genoma o presunti alleli deleteri concentrandosi sull'analisi di migliaia di genomi derivati dalla raccolta di spermatozoi maschili iniettati in F0. La Figura 2 illustra come interpretare i risultati ottenuti utilizzando questo protocollo.
Per generare mutazioni nel locus p2ry12, sono stati iniettati embrioni di zebrafish a una cellula con endonucleasi Cas9 e un sgRNA specifico per p2ry12 (Figura 2B, evidenziazione grigia). I pesci iniettati con F0 sono stati inseriti nel sistema fino alla maturità sessuale e lo sperma è stato raccolto da sei singoli maschi. Il DNA è stato estratto dai campioni di sperma utilizzando calore elevato e condizioni di base (estrazione del DNA HotSHOT) e neutralizzato prima dell'amplificazione PCR del locus p2ry12. I prodotti PCR sono stati separati su un gel di agarosio ad alta percentuale (4%) per 1,5 ore ad alta tensione (150 V). In questo esempio, l'amplicone wild-type funzionava come una singola banda luminosa di circa 250 coppie di basi di lunghezza (Figura 2C; WT). Al contrario, gli ampliconi maschili iniettati con F0 contenenti indels correvano sul gel come bande multiple (Figura 2C; corsie #1-2, #5-8). In alternativa, gli ampliconi maschili iniettati in F0 mutati potrebbero funzionare come una singola banda con mobilità del gel alterata (non mostrata nell'esempio p2ry12, ma evidente nel prodotto PCR del dnah10 F0-male #2; Figura 2E). Era meno chiaro sul gel di esempio p2ry12 se il campione #3 e il campione #4 contenessero indels rispetto alla banda wild-type, quindi questi individui potrebbero non essere i migliori candidati fondatori. L'isolamento degli alleli dai maschi F0 con alterazioni ampliconiche più distintive è la soluzione migliore per la propagazione di portatori stabili della linea germinale in quanto sono facilmente segnati su gel di agarosio al 4%. Ad esempio, il campione di sperma F0-maschio # 6 sembrava contenere una grande delezione in uno degli alleli (Figura 2C; corsia 6; banda luminosa con maggiore mobilità del gel). Se questo grande allele di delezione fosse selezionato nella generazione F1, sarebbe facilmente distinguibile dall'allele WT su un gel di agarosio al 4%. In alternativa, se si desidera una collezione di alleli diversi, il maschio F0 #7 potrebbe essere un candidato fondatore efficace poiché il suo prodotto PCR sembrava contenere diversi alleli discreti di varie mobilità. Una volta selezionato un maschio fondatore, l'allele desiderato può essere isolato incrociando l'individuo con il ceppo wild-type originale utilizzato per le iniezioni.
Per generare una specifica mutazione knock-in nel gene dnah10, sono stati iniettati embrioni di zebrafish allo stadio unicellulare con endonucleasi Cas9, uno sgRNA specifico per dnah10 (Figura 2D; evidenziazione grigia) e un oligonucleotide donatore di DNA contenente la modifica desiderata (Figura 2D; rosso) e un sito di restrizione BstNI specifico del donatore (Figura 2D , parentesi). Questo design consente una facile identificazione dell'integrazione del donatore con un digest di restrizione dopo PCR. Inoltre, alterando le coppie di basi all'interno del sito di riconoscimento dell'sgRNA (Figura 2D; evidenziazione grigia), questo disegno impedisce la digestione Cas9 della sequenza donatrice. Una volta che il pesce iniettato con F0 ha raggiunto la maturità sessuale, lo sperma è stato raccolto e il DNA è stato estratto utilizzando il metodo hot shot. Da questi campioni, il locus dnah10 è stato amplificato mediante PCR e i prodotti sono stati separati su un gel di agarosio ad alta percentuale (4%) per 1 ora ad alta tensione (150 V). In questo esempio, l'amplicone wild-type funzionava come una singola banda luminosa di circa 400 coppie di basi di lunghezza (Figura 2E; pannello superiore: WT). Al contrario, gli ampliconi maschili iniettati con F0 contenenti indels correvano come bande multiple (Figura 2E; pannello superiore: corsie #1, #4 e #5) o come una singola banda con ridotta mobilità del gel (Figura 2E; pannello superiore: corsia #2). Per determinare se uno qualsiasi dei campioni di sperma conteneva alleli integrati nel donatore, i prodotti PCR sono stati digeriti con l'enzima di restrizione BstNI per 1 ora e il prodotto è stato eseguito su un gel di agarosio al 4% per 1 ora ad alta tensione (150 V). Il confronto tra il prodotto PCR non digerito (Figura 2D; pannello superiore) con il prodotto digerito (Figura 2D; pannelli inferiori) rivela campioni con probabile integrazione del costrutto del donatore. In questo esempio, il maschio iniettato F0 #1 aveva una banda aggiuntiva nel prodotto digerito che non era presente nel prodotto PCR (Figura 2D; pannello inferiore: corsia #1, cerchiata). Pertanto, questo maschio rappresenta il miglior candidato fondatore per stabilire una linea mutante con il knock-in desiderato.
Una volta che il presunto maschio portatore di F0 viene incrociato, l'allele deve essere verificato in sequenza nella progenie F1. Si suggerisce di sequenziare direttamente Sanger gli ampliconi derivati dal pesce eterozigote F1 seguito eseguendo metodi di analisi allelica eterozigote come Poly Peak Parser13 o utilizzando una varietà di metodi basati sul sequenziamento di nuova generazione come il sequenziamento MiSeq 14 o Hi-Tom15. Questo è un approccio necessario e complementare per garantire che l'edit preciso o la deleteria mutazione indel stia effettivamente diventando germinale e non sia solo un artefatto dell'analisi dell'elettroforesi su gel. In effetti, l'uso di approcci di sequenziamento di nuova generazione per sequenziare il DNA spermatico da portatori di F0 può essere facilmente utilizzato al posto dell'analisi di elettroforesi su gel descritta in questo protocollo. Tuttavia, avere un metodo di genotipizzazione del gel di agarosio facilmente valutabile è un approccio più economico ed egualitario per la comunità globale dei ricercatori di zebrafish.

Figura 1: Impostazione per la procedura di spremitura dello sperma . (A) La spugna è posizionata sotto uno stereomicroscopio a basso ingrandimento con illuminazione dall'alto. (B) La spugna inumidita da 1 in x 1, tagliata con un divot ovale (linea tratteggiata), viene utilizzata per tenere il pesce maschio anestetizzato con il lato ventrale verso l'alto durante la procedura. (C) Anatomia del lato ventrale del pesce maschio anestetizzato raffigurante le pinne anali (af, rosa), le pinne pelviche (pf, blu) e la cloaca (freccia), dove lo sperma verrà espulso durante la procedura. (C') Le pinze filtranti vengono utilizzate per spremere delicatamente il pesce maschio anestetizzato dalle branchie alla cloaca, mentre lo sperma espulso viene aspirato in una pipetta di vetro per azione capillare (freccia bianca). (D) Un tubo capillare contenente spermatozoi espulsi sufficienti (liquido opaco; parentesi nera) per l'estrazione e l'analisi del DNA a valle. Fare clic qui per visualizzare una versione ingrandita di questa figura.
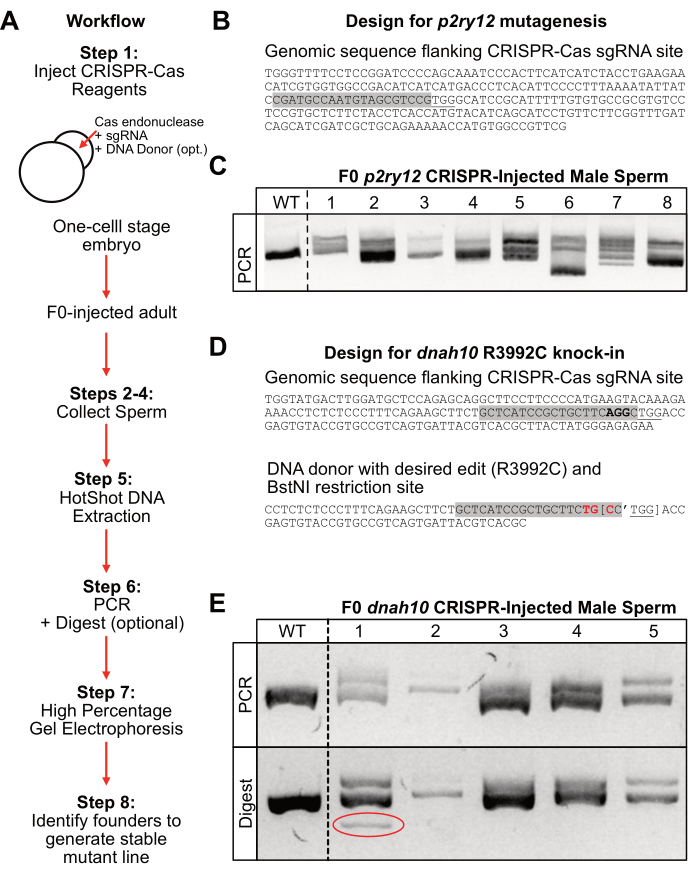
Figura 2: Flusso di lavoro e risultati rappresentativi . (A) Flusso di lavoro generale del protocollo. (B) Disegno rappresentativo del sito sgRNA p2ry12 (evidenziazione grigia) con un sito PAM specifico per Cas9 (sottolineato). (C) Risultati rappresentativi dell'elettroforesi su gel al 4% dopo 1,5 ore a 150 V. Controllo wild-type (WT) e campioni di spermatozoi maschili iniettati con F0-p2ry12 CRISPR (1-8) dopo amplificazione PCR. (D) Disegno rappresentativo del sito sgRNA dnah10 (evidenziazione grigia) con un sito PAM specifico per Cas9 (sottolineato) vicino al codone bersaglio (grassetto). Sequenza del donatore di DNA con la modifica del codone desiderata (grassetto rosso) e il sito di restrizione BstNI (parentesi con apostrofo che contrassegna il sito tagliato). (E) Risultati rappresentativi dell'elettroforesi su gel al 4% dopo 1 ora a 150 V. In alto: prodotto PCR dei campioni di spermatozoi maschili iniettati in wild-type (WT) e F0 dnah10 CRISPR (1-5). In basso: prodotto digest di restrizione BstNI dei campioni di cui sopra. Il campione 1 dimostra la riuscita integrazione del costrutto donatore basato sulla banda aggiuntiva dopo la digestione (cerchio rosso). Fare clic qui per visualizzare una versione ingrandita di questa figura.
Discussione
Questo protocollo descrive un metodo per caratterizzare rapidamente le modifiche putative del genoma o le mutazioni mirate utilizzando la tecnologia CRISPR-Cas mediante analisi focalizzata sui genomi degli spermatozoi maschili F0. Questo protocollo dovrebbe essere suscettibile di altri modelli animali in cui lo sperma è prontamente disponibile per il campionamento senza eutanasia. Questo metodo aumenterà il throughput dello screening per le modifiche desiderate ed è particolarmente utile per identificare rari eventi knock-in mediati da HDR. Questo approccio serve anche a ridurre il numero di animali da esperimento utilizzati per trovare una modifica stabile della linea germinale facilitando lo screening rapido di potenzialmente migliaia di genomi in un campione di sperma da un presunto portatore di F0, che è in contrasto con approcci più tradizionali che possono richiedere lo screening di centinaia di embrioni derivati da incroci di portatori putativi di F0.
Questo protocollo si basa su protocolli stabiliti per la raccolta dello sperma nel pesce zebra 7,10,14,16,17,18 includendo una tecnica riproducibile per identificare le modifiche della linea germinale utilizzando l'elettroforesi su gel ad alta risoluzione. Questo approccio può essere facilmente incorporato in qualsiasi flusso di lavoro CRISPR / Cas standard per aumentare il throughput per lo screening e l'isolamento delle modifiche del genoma target. Inoltre, questo protocollo è appropriato per i laboratori con personale con una vasta gamma di formazione ed esperienza, nonché per i laboratori didattici. Tuttavia, il nostro metodo di raccolta dello sperma non è sufficiente per la crioconservazione, che è stato sapientemente descritto nelle precedenti pubblicazioni10,17.
Questo protocollo utilizza un'elettroforesi su gel di agarosio ad alta risoluzione per identificare presunti portatori maschi di mutazioni genomiche indel e precise desiderate. Tuttavia, il DNA genomico degli spermatozoi è suscettibile di una miriade di altri approcci, tra cui l'analisi dei frammenti fluorescenti o il sequenziamento con codice a barre14, l'analisi della fusione ad alta risoluzione18 o il semplice rilevamento di ampliconi di tag fluorescenti o epitopi utilizzando approcci standard di elettroforesi su gel. La convalida a valle di tutti gli alleli utilizzando approcci come il sequenziamento basato su Sanger o di nuova generazione deve essere effettuata prima di iniziare il lavoro sperimentale sugli alleli putativi. In effetti, i metodi esistenti per lo screening delle mutazioni negli embrioni utilizzando l'approccio di sequenziamento di nuova generazione14 potrebbero aggirare la necessità di analisi su gel di agarosio, che è descritto in questo protocollo. Tuttavia, avere un metodo di genotipizzazione con punteggio in gel è un approccio più economico ed egualitario da considerare, dato che non tutti i laboratori hanno facile accesso a metodi di sequenziamento economici di nuova generazione durante le fasi di isolamento e sperimentali di lavoro con ciascun ceppo mutante.
In sintesi, questo protocollo fornisce istruzioni passo-passo per lo screening riproducibile dei genomi degli spermatozoi da maschi modificati da CRISPR / Cas in modo tale che siano necessari meno incroci e meno screening PCR degli embrioni per lo screening delle modifiche desiderate. L'applicazione di questo metodo ridurrà il numero di pesci che devono essere creati e analizzati per identificare con successo gli alleli di interesse modificati, il che riduce anche i tempi e i costi del personale per generare linee stabili.
Divulgazioni
Nessuno.
Riconoscimenti
Vorremmo ringraziare Anna Hindes della Washington University School of Medicine per i suoi sforzi iniziali nell'ottenere DNA genomico spermatico di buona qualità utilizzando il metodo hot shot. Questo lavoro è stato finanziato dal National Institute of Arthritis and Musculoskeletal and Skin Diseases del National Institutes of Health under Award (R01AR072009 to R.S.G.).
Materiali
| Name | Company | Catalog Number | Comments |
| Agarose powder | Fisher BioReagents | BP1356-100 | |
| Breeding tanks | Carolina Biological | 161937 | |
| BstNI Restriction Enzyme | NEB | R0168S | |
| Cas9 Endonuclease | IDT | 1081060 | |
| DNA Ladder, 100 bp | Thermo Scientific | FERSM0241 | |
| dnah10 donor construct | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry. Phosphorothioate bond on the donor at the first three phosphate bonds on both the 5’ and 3’ ends (5'-CCTCTCTCCCTTTCAGAAGCTTC TGCTCATCCGCTGCTTCTGCCT GGACCGAGTGTACCGTGCCGTC AGTGATTACGTCACGC-3') | |
| dnah10 forward primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CATGGAACTCTTTCCTAATGAGT TTGGC-3') | |
| dnah10 reverse primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry ('5-AGTAGAGATCACACATCAACAGA ATACAGC-3') | |
| dnah10 synthetic sgRNA | Synthego | Synthetic sgRNA, target sequence: 5'-GCTCATCCGCTGCTTCAGGC-3' | |
| Electrophoresis power supply | Thermo Scientific | 105ECA-115 | |
| Filter forceps | Millipore | XX6200006P | |
| Fish (system) water | Generic | n/a | |
| Gel electrophoresis system (including casting frame, comb, and electrophoresis chamber) | Thermo Scientific | B2 | |
| Gel imaging light box | Azure Biosystems | AZI200-01 | |
| Gel stain, 10000X | Invitrogen | S33102 | |
| Glass bowl, 250 mL | Generic | n/a | |
| Isolation tanks, 0.8 L | Aquaneering | ZT080 | |
| Microcap capillary tube with bulb, 20 µL | Drummond | 1-000-0020/CA | |
| Minicentrifuge | Bio-Rad | 12011919EDU | |
| Micropipettes, various with appropriate tips | Generic | n/a | |
| Microwave | Generic | n/a | |
| Nuclease free water | Promega | P119-C | |
| Paper towels | Generic | n/a | |
| PCR tubes, 0.2 mL | Bioexpress | T-3196-1 | |
| Plastic spoon, with drilled holes/slots | Generic | n/a | |
| KCl solution, 0.2 M RNAse Free | Sigma-Aldrich | P9333 | |
| p2ry12 forward primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CCCAAATGTAATCCTGACCAGT -3') | |
| p2ry12 reverse primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CCAGGAACACATTAACCTGGAT -3') | |
| p2ry12 synthetic sgRNA | Synthego | Synthetic sgRNA, target sequence: 5'-GGCCGCACGAGGTCTCCGCG-3' | |
| Restriction Enzyme 10X Buffer | NEB | B6003SVIAL | |
| NaOH solution, 50 mM | Thermo Scientific | S318; 424330010 | |
| Sponge, 1-inch x 1-inch cut with small oval divot | Generic | n/a | |
| Stereomicroscope | Zeiss | Stemi 508 | |
| Taq polymerase master mix, 2X | Promega | M7122 | |
| TBE Buffer Concentrate, 10X | VWR | E442 | |
| Thermal Cycler | Bio-Rad | 1861096 | |
| Tissue paper | Fisher Scientific | 06-666 | |
| Tricaine-methanesulfonate solution (Syncaine, MS-222), 0.016% in fish water (pH 7.0±0.2) | Syndel | 200-266 | |
| Tris Base, 1M (Buffered with HCl to ph 8.0) | Promega | H5131 |
Riferimenti
- Auer, T. O., Duroure, K., De Cian, A., Concordet, J. P., Del Bene, F. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair. Genome Research. 24 (1), 142-153 (2014).
- Hwang, W. Y., et al. Efficient genome editing in zebrafish using a CRISPR-Cas system. Nature Biotechnology. 31 (3), 227-229 (2013).
- Jao, L. E., Wente, S. R., Chen, W. Efficient multiplex biallelic zebrafish genome editing using a CRISPR nuclease system. Proceedings of the National Academy of Sciences of the United States of America. 110 (34), 13904-13909 (2013).
- Troutwine, B. R., et al. The Reissner fiber is highly dynamic in vivo and controls morphogenesis of the spine. Current Biology. 30 (12), 2353-2362 (2020).
- Xu, Y., Li, Z. CRISPR-Cas systems: Overview, innovations and applications in human disease research and gene therapy. Computational and Structural Biotechnology Journal. 18, 2401-2415 (2020).
- Creaser, C. W. The technic of handling the zebrafish (Brachydanio rerio) for the production of eggs which are favorable for embryological research and are available at any specified time throughout the year. Copeia. 1930 (4), 159-161 (1934).
- Carmichael, C., Westerfiel, M., Varga, Z. M. Cryopreservatin and in vitro fertilization at the zebrafish international resource center. Methods in Molecular Biology. 546, 45-65 (2009).
- Wang, Y., Troutwine, B. R., Zhang, H., Gray, R. S. The axonemal dynein heavy chain 10 gene is essential for monocilia motility and spine alignment in zebrafish. Developmental Biology. 482, 82-90 (2021).
- Gray, R. S., et al. Postembryonic screen for mutations affecting spine development in zebrafish. Developmental Biology. 471, 18-33 (2021).
- Brocal, I., et al. Efficient identification of CRISPR/Cas9-induced insertions/deletions by direct germline screening in zebrafish. BMC Genomics. 17, 259 (2016).
- Davis, M. W., Jorgensen, E. M. ApE, A Plasmid Editor: A freely available DNA manipulation and visualization program. Frontiers in Bioinformatics. 2, 816619 (2022).
- Labun, K., et al. CHOPCHOP v3: Expanding the CRISPR web toolbox beyond genome editing. Nucleic Acids Research. 47, 171-174 (2019).
- Hill, J. T., et al. Poly Peak Parser: Method and software for identification of unknown indels using sanger sequencing of polymerase chain reaction products. Developmental Dynamics. 243 (12), 1632-1636 (2014).
- Varshney, G. K., et al. High-throughput gene targeting and phenotyping in zebrafish using CRISPR/Cas9. Genome Research. 25 (7), 1030-1042 (2015).
- Liu, Q., et al. Hi-TOM: A platform for high-throughput tracking of mutations induced by CRISPR/Cas systems. Science China. Life Sciences. 62 (1), 1-7 (2019).
- Sorlien, E. L., Witucki, M. A., Ogas, J. Efficient production and identification of CRISPR/Cas9-generated gene knockouts in the model system Danio rerio. Journal of Visualized Experiments. (138), e56969 (2018).
- Draper, B. W., Moens, C. B. A high-throughput method for zebrafish sperm cryopreservation and in vitro fertilization. Journal of Visualized Experiments. (29), e1395 (2009).
- Parant, J. M., George, S. A., Pryor, R., Wittwer, C. T., Yost, H. J. A rapid and efficient method of genotyping zebrafish mutants. Developmental Dynamics. 238 (12), 3168-3174 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon