Method Article
בדיקת זרע לבידוד מהיר של עריכות נבט בדגי זברה
In This Article
Summary
טכנולוגיות CRISPR-Cas חוללו מהפכה בתחום עריכת הגנום. עם זאת, מציאת ובידוד עריכת קו הנבט הרצויה נותרה צוואר בקבוק משמעותי. לכן, פרוטוקול זה מתאר שיטה חזקה לסינון מהיר של זרע דג זברה מוזרק F0 CRISPR לעריכות קו הנבט באמצעות טכניקות PCR סטנדרטיות, תקציר הגבלה ואלקטרופורזה בג'ל.
Abstract
הופעתן של טכנולוגיות נוקלאז CRISPR-Cas ממוקדות חוללה מהפכה ביכולת לבצע עריכת גנום מדויקת במערכות מודל מבוססות ומתפתחות כאחד. מערכות עריכת הגנום של CRISPR-Cas משתמשות ב-RNA מנחה סינתטי (sgRNA) כדי לכוון אנדונוקלאז הקשור לקריספר (Cas) ל-DNA loci גנומי ספציפי, שבו אנדונוקלאז Cas יוצר שבר דו-גדילי. תיקון שברים דו-גדיליים על ידי מנגנונים המועדים לטעויות מהותיות מוביל להחדרות ו/או מחיקות, המשבשות את הלוקוס. לחלופין, הכללתם של תורמי DNA דו-גדיליים או אוליגונוקלאוטידים חד-גדיליים של DNA בתהליך זה יכולה לעורר הכללת עריכות גנום מדויקות החל מפולימורפיזמים של נוקלאוטידים בודדים ועד תגים אימונולוגיים קטנים או אפילו מבנים חלבוניים פלואורסצנטיים גדולים. עם זאת, צוואר בקבוק משמעותי בהליך זה יכול להיות מציאת ובידוד העריכה הרצויה בקו הנבט. פרוטוקול זה מתאר שיטה חזקה לסינון ובידוד מוטציות של תאי נבט במוקדים ספציפיים בדגי זברה ( Danio rerio ); עם זאת, עקרונות אלה עשויים להיות ניתנים להתאמה בכל מודל שבו איסוף זרע in vivo אפשרי.
Introduction
מערכת CRISPR (Clustered Regular Interspaced Short Palindromic Repeats)/Cas היא כלי רב עוצמה לביצוע מוטגנזה ספציפית ללוקי ועריכת גנום מדויקת במערכת מודל Danio rerio (דג זברה) 1,2,3,4. Cas-ribonucleoprotein (RNP) מורכב משני מרכיבים עיקריים: Cas endonuclease (בדרך כלל Cas9 או Cas12a) ו- RNA מדריך סינתטי ספציפי ללוקוס (sgRNA)5. יחד, Cas-RNP יוצר שבר דו-גדילי (DSB) במיקום הרצוי שניתן לתקן על ידי אחד משני מנגנוני תיקון פנימיים. מנגנון התיקון הלא הומולוגי של הצטרפות הקצה (NHEJ) מועד לשגיאות ולעתים קרובות גורם למגוון של הוספות או מחיקות (indels) סביב DSB. אינדלים אלה יכולים להיות מזיקים אם הם מציגים מוטציה frameshift או עצירה מוקדמת ברצף החלבונים המתקבל. לחלופין, מנגנון התיקון מונחה הומולוגיה (HDR) משתמש בתבנית תורם עם אזורי הומולוגיה המקיפים את אתר DSB כדי לתקן את הנזק. חוקרים יכולים לנצל את מערכת HDR כדי ליצור עריכות גנומיות מדויקות. באופן ספציפי, הם יכולים להזריק במשותף מבנה תורם DNA דו-גדילי המכיל את העריכות הרצויות, כמו גם אזורים של הומולוגיה הצמודים לאתר DSB בגנום. הגידול בכלכלת הגודל של רכיבי CRISPR המיוצרים באופן מסחרי צמצם מאוד את המחסומים לסינון אתרים מרובים ולהקמת מאמצים בקנה מידה גדול יותר לעריכה מדויקת של הגנום. עם זאת, במודלים של בעלי חיים המתרבים מינית, צוואר בקבוק עיקרי הוא זיהוי ובידוד של בעלי חיים מוטנטיים יציבי נבט.
מערכת מודל דגי הזברה מציגה מספר תכונות מפתח המשפרות את השימוש בה במחקרים גנטיים הפוכים. קל לגדל אותם במספרים גדולים עם ציוד דיור ימי בסיסי, והנקבות מפגינות פריון גבוה כל השנה6. יתר על כן, הטלת הביצים וההפריה החיצונית שלהם הופכות אותם למקובלים למיקרו-הזרקה של רכיבי CRISPR/Cas. ה- Cas-RNP מוזרק בדרך כלל לעוברים של דגי זברה בשלב חד תאי כדי ליצור DSBs/תיקון שהוא, בתיאוריה, בירושה על ידי כל תאי הבת. עם זאת, גנומים דיפלואידים דורשים שני אירועי DSB/תיקון כדי לבצע מוטגניזציה של שני הכרומוזומים ההומולוגיים. יתר על כן, למרות ש- Cas-RNP מוזרק בשלב התא האחד, ה- DSB/תיקון עשוי להתרחש רק בנקודות מאוחרות יותר בהתפתחות. יחד, גורמים אלה תורמים לאופי הפסיפס של דגים המוזרקים F0. נוהג נפוץ הוא לחצות דגים המוזרקים F0 ולסנן את צאצאי F1 עבור indels / עריכות ספציפיות. עם זאת, מכיוון שלא לכל הדגים המוזרקים F0 יש מוטציות בקו הנבט, נוהג זה גורם להצלבות לא פרודוקטיביות רבות שאינן מייצרות את העריכה הרצויה. סינון הרקמה הסומטית F0 במקום F1 מגדיל את ההסתברות לבידוד מערך הנבט הרצוי ומקטין את מספר בעלי החיים הדרושים בתהליך זה.
ניתן לאסוף זרע בקלות מדגי זברה המוזרקים F0 ללא צורך בהמתת חסד. תכונה זו מאפשרת שימור בהקפאה וגזירה מחדש של מלאי זרע קפוא7 אך ניתן גם לנצל אותה כדי לסנן, לזהות ולבודד במהירות את נשאי הנבט של מוטציות גנומיות רצויות 8,9. Brocal et al. (2016) תיארו בעבר שיטה מבוססת ריצוף לסינון עריכות קו הנבט בדגי זברה זכרים בהזרקת F010. למרות שהיא שימושית לזיהוי האללים המוטנטים הנמצאים בקו הנבט, גישה זו עלולה להיות יקרה בתפוקה גבוהה וייתכן שלא תהיה נגישה לכל המעבדות. לעומת זאת, הפרוטוקול הנוכחי מציע אסטרטגיה נגישה וחסכונית מבוססת אלקטרופורזה לזיהוי עריכות קו הנבט. באופן ספציפי, פרוטוקול זה מתאר שיטה חזקה לסינון ובידוד מוטציות של קו הנבט באתרים ספציפיים באמצעות אלקטרופורזה של ג'ל אגרוז ברזולוציה גבוהה. בנוסף, פרוטוקול זה מתאר אסטרטגיה דומה לזיהוי שילוב מוצלח של מבנה תורם המכיל עריכות ספציפיות. כמו תמיד, אם יש צורך בעריכות ספציפיות, ניתן לבצע אסטרטגיות מבוססות רצף במקביל לפרוטוקול המתואר להלן. למרות שפרוטוקול זה הוא ספציפי למערכת מודל דגי הזברה, עקרונות אלה צריכים להיות ניתנים להתאמה לכל מודל שבו איסוף הזרע הוא הליך שגרתי. יחד, אסטרטגיות אלה יאפשרו זיהוי של זכרים עם הזרקת F0 עם indels/edits germline שניתן לפתור על ג'ל לאחר תגובת שרשרת פולימראז סטנדרטית (PCR) ו / או הגבלת digest.
Protocol
מחקר זה בוצע בהתאם להנחיות במדריך לטיפול ושימוש בחיות מעבדה של המכונים הלאומיים לבריאות. הפרוטוקול אושר על ידי אוניברסיטת טקסס בוועדה לטיפול ושימוש בבעלי חיים באוסטין (AUP-2021-00254).
1. תכנון sgRNA עבור מוטגנזה של קריספר
- השג את רצף האקסון המכיל את loci היעד.
- תכנן רנ"א מנחה סינתטי (sgRNA) עם אתר מוטיב סמוך פרוטוספייסר (PAM) הספציפי לאנדונוקלאז Cas.
- אם אתם מזריקים במשותף מבנה תורם, תכננו את ה-sgRNA כך שיהיה לו אתר חיתוך חזוי קרוב ככל האפשר לעריכות הרצויות.
הערה: APE היא תוכנה חופשית עם כלי למציאת אתרי Cas9 sgRNA11. לחלופין, ניתן להשתמש בכלים כגון CHOPCHOP, המציע ממשק מקוון לתכנון sgRNA המתחשב באתרי PAM השונים של Cas9 ואנדונוקלאזות Cas אחרות12.
- אם אתם מזריקים במשותף מבנה תורם, תכננו את ה-sgRNA כך שיהיה לו אתר חיתוך חזוי קרוב ככל האפשר לעריכות הרצויות.
- עצבו פריימרים קדימה ואחורה כדי להגביר את האזור שמסביב לאתר חיתוך CRISPR/Cas מעוצב. מוצר ה-PCR צריך להיות באורך של כ-200-400 זוגות בסיסים (bp) כדי להיות מסוגל לפתור אינדל קטן על ג'ל בהמשך הפרוטוקול.
הערה: CHOPCHOP11 מעצב פריימרים באופן אוטומטי עבור כל sgRNA שהוא מפיק. - אופציונלי: תכנון מבנה תורם DNA עם העריכות הרצויות.
- קבל את הרצף הגנומי של האקסון המכיל את המוקדים של המוטציה הרצויה.
הערה: רצפים אינטרוניים יכולים להשתנות בין אנשים, ולכן עדיף להימנע מהכללת רצפים אינטרוניים במבנה התורם מכיוון שהדבר עלול להפריע למסלול התיקון התלוי בהומולוגיה. - אם רוצים שינוי מסוים בחומצת האמינו, שנו את הקודון הספציפי בהתאם.
הערה: בעת ביצוע שינויים אלה, שים לב לתדירות הקודון. במידת האפשר, בחר קודון עם שימוש גבוה. - כדי למנוע עיכול CRISPR/Cas של מבנה התורם, צור מוטציות נרדפות נוספות אשר (i) משבשות את ה- PAM ו / או (ii) משבשות את רצף sgRNA, עם עדיפות גבוהה יותר לביצוע שינויים לא נרדפים קרוב יותר לאתר ה- PAM.
- השתמש במאתר אתרי הגבלה (APE11 כולל גם תכונה זו) כדי למצוא אתרי הגבלה ייחודיים (אלה שמתרחשים רק פעם אחת) במכפלת ה- PCR החזויה של הרצף מסוג wild.
- חזור על תהליך זה עבור מוצר ה- PCR החזוי המשקף את השילוב המוצלח של מבנה התורם.
- השווה את רשימת אתרי ההגבלה בין הרצפים המשולבים בטבע לבין הרצפים המשולבים על ידי תורמים כדי למצוא אתר הגבלה שנמצא רק ברצף הערוך.
- אם לא נמצא אתר הגבלה ייחודי, המשך לבצע שינויים נרדפים במבנה התורם עד להשגת מטרה זו.
הערה: במידת האפשר, המשך ליצור מוטציות נרדפות שישבשו את קשירת sgRNA למבנה התורם.
- חתוך את רצף התורמים לגודל הניתן לניהול (50-100 bp) עם לפחות 20 bp של הומולוגיה סביב אתר חיתוך CRISPR/Cas החזוי ועריכות, במידת האפשר.
- קבל את הרצף הגנומי של האקסון המכיל את המוקדים של המוטציה הרצויה.
- השג את האוליגונוקלאוטידים המתוכננים של sgRNA ו/או תורם DNA, כמו גם כל פריימר הדרוש לגנוטיפ.
הערה: כדי למנוע עיכול של אוליגונוקלאוטידים מתורם DNA על ידי נוקלאזות, מומלץ לשנות את שלושת קשרי הפוספט הראשונים בקצוות 5' ו-3' של אוליגונוקלאוטידים של DNA עם זרחן. - יש להזריק 1 nL מתערובת ההזרקה לעוברים בשלב חד-תאי. בדרך כלל, תערובת הזרקה סטנדרטית מכילה את הדברים הבאים:
1 μL של 25 μM sgRNA ספציפי ללוקוס
1 μL של 25 מיקרומטר Cas9 אנדונוקלאז
אופציונלי: 1 μL של 3 מיקרומטר DNA מבנה תורם- הביאו את הנפח הכולל ל-5 מיקרוליטר עם KCl נטול RNase בנפח 0.1 M.
- לאפשר לאנשים המוזרקים F0 להתפתח למבוגרים בוגרים מינית. שאר הפרוטוקול מתמקד בסינון הזכרים שהזריקו F0 לעריכות קו הנבט.
2. הקמת מיכלי הגידול
- זכרי F0 ראשוניים להזדווגות על ידי הקמת מיכלי רבייה עם מחיצות בין זכרי F0 לנקבות.
- כדי למזער את מספר מכלי הרבייה הדרושים, הציבו שלושה זכרים מול שתי נקבות בכל טנק.
- הגדר כמה שיותר זכרי F0 שניתן לבדוק ביעילות ולשכן כפרטים לאחר איסוף זרע. הגנוטיפ של הנקבות אינו חשוב מכיוון שהגמטות שלהן לא ייאספו במהלך הליך זה.
- הקימו מיכל רבייה אחד של זכרים פראיים מול הנקבות. זרע מסוג פרא ישמש כבקרה לפענוח התוצאות בהמשך ההליך.
- כדי למזער את מספר מכלי הרבייה הדרושים, הציבו שלושה זכרים מול שתי נקבות בכל טנק.
- לדגור על מיכלי הרבייה המוכנים למשך הלילה.
3. הכנת החומרים להליך איסוף הזרע
- הכינו מספיק תמיסת הרדמה (0.016% טריקאין-HCl במי דגים) כדי למלא קערת זכוכית קטנה.
- יש להוציא 40 μL של 50 mM NaOH לתוך צינורות PCR של 0.2 מ"ל. הכינו צינורית אחת לכל דגימת זרע שתיאסף. יש לאחסן על קרח.
- הכינו מיכלי 0.8 ליטר נפרדים לכל דג שיידגמו במהלך ההליך. מכלים אלה יאחסנו את הדגים שנדגמו עד להשלמת הגנוטיפ.
הערה: זהו הגורם העיקרי המגביל את מספר הדגים שניתן לסנן ביעילות בפרוטוקול זה. להבטיח מקום מספיק לשיכון הפרטים שנדגמו עד להשלמת הגנוטיפ. - יש להרטיב ספוג בגודל 1 אינץ' על 1 אינץ' (לחתוך עם דיווט רדוד בצורת אליפסה כדי להחזיק את הצד הגחוני של דג זברה זכר למעלה במהלך ההליך) במי דגים ולסחוט את עודפי הנוזלים.
- מקמו את הספוג בשדה הראייה של סטריאומיקרוסקופ בהגדלה נמוכה (איור 1A).
- מקם תאורה עילית אלכסונית לצפייה אופטימלית.
4. הרדמת הדג הזכר ואיסוף הזרע
- הכינו צינור נימי נקי.
- נערו בעדינות את מיכל הצינור כדי לשחרר צינור נימי יחיד.
- מניחים את צינור הנימים לתוך הנורה שסופקה, ומניחים אותו בצד.
- מעבירים דג זכר לתמיסת הרדמה מוכנה (0.016% טריקאין-HCl במי דגים).
- ערבבו בעדינות את התמיסה עם כף מחורצת כדי לזרז את תהליך ההרדמה.
- לאחר שתנועת האופרקולום (זמיר) מאטה (כ-1-2 דקות), המשיכו באיסוף הזרע.
- השתמשו בכף המחורצת כדי להעביר את הדג לערימה נקייה של מגבות נייר.
- השתמשו בכף המחורצת כדי לגלגל בעדינות את הדג כדי להסיר עודפי מים.
- הכתימו בעדינות את הדג בנייר טישו נקי ומקופל כדי להסיר את שאריות המים.
- השתמשו בכף המחוררת כדי להעביר את הדג לספוג המוכן (איור 1B).
- מקם את הצד הגחוני של הדג כלפי מעלה כשראשו קרוב ליד הדומיננטית של האדם המבצע את ההליך.
- כתם בעדינות את האזור סביב סנפירי פי הטבעת עם נייר טישו נקי ומקופל.
- לאסוף את הזרע.
- בעזרת קצה צינור הנימים, הזיזו בעדינות את סנפירי האגן לרוחב מקו האמצע כדי לחשוף את הקלואקה (איור 1C).
- מקם את קצה צינור הנימים ליד cloaca.
- בעזרת המלקחיים, סחטו בעדינות את דפנות הדג שמתחיל ממש מתחת לזימים ומסתיים בקלואקה (איור 1C).
- לאסוף זרע לתוך צינור נימי על ידי פעולה נימית. בערך 1 μL מספיק עבור הליך זה (איור 1D).
הערה: מעבדות מסוימות מדווחות על שימוש בקצוות פיפט p10 במקום צינורות נימים לאיסוף זרע. - אם הזרע אינו משתחרר בלחץ עדין, יש להחזיר את הדג למי המערכת.
- יש לעקוב אחר ההתאוששות מההרדמה.
- אין לסחוט את הדג קשה כדי לגרש זרע, כמו זה יכול לפצוע את הדג.
- הניחו את הדג במשך שבועיים לפני שתנסו לאסוף זרע שוב.
- לאחר האיסוף, הכניסו את הדגים למיכל בידוד נקי עם מי מערכת לדיור אישי.
- עקוב אחר הדג להתאוששות מההרדמה.
- הניחו את הדג במשך שבועיים לפני שתנסו לאסוף זרע שוב.
- מניחים את הצינור הנימי עם הזרע שנאסף לתוך צינור PCR מוכן (עם 40 μL של 50 mM NaOH).
- סחטו את נורת הגומי כדי לגרש את הדגימה שנאספת.
- יש להשליך את צינור הנימים המשומש למיכל פסולת זכוכית מאושר.
- דוגרים על הדגימות על קרח עד שכל הזכרים נסחטים.
- חזור על שלבים 4.1-4.5 עבור כל זכר בודד.
- תייגו את מיכלי הבידוד ואת דגימות הזרע כך שניתן יהיה לייחס את תוצאות הגנוטיפ הסופיות של הזרע לאדם המתאים עם מוטציות משוערות.
- עקוב אחר כל האנשים, וודא שהם זקופים ובוחנים את הטנקים שלהם לפני שתחזיר את הטנקים למערכת.
- החזירו את הנקבות המשמשות להכנה חזרה למכלים שלהן במערכת.
- אם דג כלשהו אינו מתאושש מההרדמה לאחר 10 דקות, בצע את הליך המתת חסד שאושר על ידי המוסד המשויך למעבדה.
- דוגמה: קירור מהיר של האדם באמבט קרח עם מי דגים שהם 2-4 מעלות צלזיוס.
- יש להשליך את הדגים המורדמים במיכל פסולת מתאים של פגרי בעלי חיים בהתאם לנוהל שאושר על ידי המוסד הקשור למעבדה.
5. מיצוי DNA מדגימות הזרע
- סובבו בקצרה את כל דגימות הזרע במיניצנטריפוגה, והכניסו צינורות PCR למחזור תרמי.
- סגור את המכסה של מחזור תרמי.
- הפעל את ההגדרות הבאות במחזור התרמי:
- חממו את הדגימות במשך 40 דקות בטמפרטורה של 95°C.
- מצננים את הדגימות ל-25°C.
- הסר את הדגימות מהמחזור התרמי.
- עם קצה פיפטה נקי לכל דגימה, נטרלו את ה- pH על ידי הוספת 10 μL של 1 M Tris-HCl (חוצץ ל- pH 8).
- מערבבים היטב על ידי פיפטינג למעלה ולמטה.
- סובבו בקצרה את הדגימות במיניצנטריפוגה.
- דגימות DNA גנומיות ניתן לאחסן במשך מספר ימים ב 4 ° C או להציב ב -20 ° C עד 6 חודשים.
6. הגברה PCR (ו/או תקציר הגבלה) של הלוקוס הרצוי
- הגבירו את הלוקוס הרצוי באמצעות פרוטוקול PCR סטנדרטי.
- חממו מראש את המחזור התרמי לטמפרטורת הדנטורציה הראשונית בתגובת ה-PCR למטה.
- הכינו תערובת תגובה של 25 μL לכל דגימת זרע בצינורות PCR:
12.5 μL של 2x Taq Polymerase Master Mix
1.5 μL של פריימר קדמי 10 μM
1.5 μL של פריימר הפוך 10 מיקרומטר
4.5 מיקרוליטר מים ללא נוקלאז
5 μL של דגימת DNA זרע מנוטרלת - מערבבים היטב על ידי פיפטינג למעלה ולמטה.
- סובבו בקצרה את הדגימות במיניצנטריפוגה.
- מקם את הדגימות במחזור תרמי שחומם מראש עם ההגדרות הבאות:
דנטורציה ראשונית: 95 °C למשך 3 דקות
35 מחזורים של דנטורציה (95 ° C עבור 30 שניות), חישול (55 ° C עבור 30 שניות), והרחבה (72 ° C עבור 30 שניות)
הארכה סופית: 72°C למשך 5 דקות
הערה: ייתכן שיהיה צורך להתאים את טמפרטורת ההרחבה ו / או הזמן בהתאם לפולימראז Taq הספציפי והאורך הצפוי של מוצר הגברה PCR. - הסר את דגימות ה- PCR מהמחזור התרמי.
- אופציונלי: בצע את תקציר ההגבלה הספציפי לתורם על נפח קטן של מוצר ה- PCR, תוך השארת לפחות 5-10 מיקרוליטר של מוצר PCR לא מעוכל לאלקטרופורזה בג'ל.
- הכן תגובה של 30 μL בצינורות PCR של 0.2 מ"ל:
10 μL של מוצר PCR לא מטוהר
15 מיקרוליטר מים ללא נוקלאז
3 μL של מאגר אנזים הגבלה פי 10
2 μL של אנזים הגבלה (BstNI משמש בתוצאות המייצגות לניתוח אלל הנוק-אין DNAH10 )
הערה: אנזימי הגבלה רבים עדיין פעילים במאגר תגובת PCR טיפוסי; עם זאת, הוספת מאגר Restriction Digest יכולה לעזור לשפר את התוצאות. עיין בהוראות יצרן ספציפיות לפתרון בעיות במקרה של בעיות. - מערבבים היטב על ידי פיפטינג למעלה ולמטה.
- סובבו בקצרה את הדגימות במיניצנטריפוגה.
- מקם את הדגימות במחזור תרמי עם ההגדרות הבאות:
- חתכו את אמפליקון ה-PCR ב-60°C למשך שעה אחת.
הערה: ייתכן שיהיה צורך להתאים את טמפרטורת החיתוך ואת זמן הדגירה לאנזים ההגבלה הספציפי הנמצא בשימוש. - השבת את אנזים ההגבלה עם הטמפרטורה וזמן הדגירה הנדרשים, אם צוין על ידי היצרן.
הערה: האנזים BstNI אינו דורש השבתת חום.
- חתכו את אמפליקון ה-PCR ב-60°C למשך שעה אחת.
- הכן תגובה של 30 μL בצינורות PCR של 0.2 מ"ל:
7. ביצוע אלקטרופורזה בג'ל להפרדת אמפליקונים PCR בגדלים שונים
- הכן 500 מ"ל של חיץ ריצה TBE 0.5x: הוסף 50 מ"ל של תרכיז חיץ TBE 10x ל- 450 מ"ל מים נטולי יונים.
- הכינו 100 מ"ל של 4% ג'ל אגרוז במאגר ריצה של 0.5x TBE: הוסיפו 4 גרם אגרוז עם 10 מיקרוליטר של כתם ג'ל 10,000x ל-100 מ"ל של מאגר ריצה TBE 0.5x. יש לחמם במיקרוגל עד להמסה מלאה של אבקת האגרוז.
- יוצקים את תמיסת הג'ל למסגרת יציקת ג'ל בגודל מתאים, ומכניסים את המסרק בצד אחד של הג'ל.
הערה: גודל ג'ל גדול יותר (מומלץ: 15 ס"מ x 15 ס"מ) הוא יתרון עבור הליך זה, כפי שהוא מספק את amplicons עם יותר מקום לפתור על הג'ל.- אם יש לנתח מוצר תקציר הגבלה, עדיף להפעיל אותו על אותו ג'ל כמו מוצר PCR, כך שניתן יהיה להשוות את התוצאות. התאימו את גודל המסרק לפי הצורך כך שיתאים לכל הדגימות שעל הג'ל.
- השאירו את הג'ל בטמפרטורת החדר עד להתייצבות. הסר בזהירות את המסרק. בזהירות להסיר את הג'ל ממסגרת הליהוק.
- יוצקים את תמיסת הג'ל למסגרת יציקת ג'ל בגודל מתאים, ומכניסים את המסרק בצד אחד של הג'ל.
- הגדר את מתקן אלקטרופורזה ג'ל.
- מקמו את הג'ל במתקן האלקטרופורזה כך שהבארות יהיו הקרובות ביותר לאלקטרודה השלילית. יוצקים חיץ ריצה של 0.5x TBE לתוך אסדת האלקטרופורזה עד שהבארות שקועות במלואן.
- טוענים את הג'ל.
- טען 5 μL של סולם DNA לתוך הבאר הראשונה (בחר סולם DNA המכיל מקטעי DNA הדומים בגודלם למגבר PCR).
- באמצעות קצה פיפטה חדש עבור כל דגימה (כולל דגימת הבקרה מסוג Wild), לטעון 5-10 μL של מוצר PCR לתוך הבארות הנותרות.
- אופציונלי: אם רוצים לנתח מוצר לעיכול הגבלה, יש להעמיס את הדגימות גם על הג'ל.
הערה: עדיף לטעון כמויות שוות של DNA בין PCR לבין מוצר העיכול הגבלה. לדוגמה, בתערובת תגובת ההגבלה של 30 μL, נוסף 10 μL של מוצר PCR. לכן, ריכוז הדנ"א של 15 μL של מוצר העיכול היה דומה ל 5 μL של מוצר PCR לא מטוהר.
- הפעל את הג'ל.
- חבר את האלקטרודות למתקן אלקטרופורזה ג'ל, והפעל 150 V למשך שעה לפחות כדי להבטיח הפרדה נאותה של האמליקונים.
- דמיינו את הג'ל כדי להציג את רצועות הדנ"א.
הערה: אתרים ממוקדי קריספר בעלי מוטגן חזק יפעלו כגדלים מרובים של רצועות המייצגות שילוב של אללי החדרה/מחיקה עם האמפליקונים מסוג Wild.- אם רצועות הג'ל אינן נפתרות לאחר שעה אחת, יש למרוח 150 וולט על הג'ל למשך 15 דקות עד שהרצועות מופרדות מספיק על הג'ל.
- אופציונלי: אם בודקים להחדרת עריכה מדויקת כגון SNP, תג אפיטופ או תג פלואורסצנטי, סנן את DNA הזרע בשיטות PCR ספציפיות לעריכה הרצויה. לדוגמה, בעת זיהוי אתר הגבלה ספציפי לתורם, רצועה נוספת תהיה נוכחת במוצר תקציר ההגבלה בהשוואה לתוצר ה- PCR של דגימת הזרע.
8. בידוד אללים יציבים של קו הנבט
- לאחר שבועיים של מנוחה מהליך סחיטת הזרע, חצו את הפרטים F0 שהדנ"א הגנומי של הזרע שלהם הכיל את העריכות הרצויות.
הערה: ייצור זרע משיבוטים בודדים של זרעונים עשוי להיות מחזורי בדגי זברה; לכן, ייתכן שהאלל הרצוי לא מייצר זרע בזמן ההצלבה. מסיבה זו, ייתכן שיהיה צורך לבצע חצייה נוספת יותר מפעם אחת כדי לבודד את האלל המעורב בסינון הראשוני. - לאחר שדגי F1 מוצלבים מגיעים לבגרות מינית, המשיכו בשיטות גנוטיפ סטנדרטיות (לדוגמה, DNA קליפ סנפיר) תוך שימוש באותו פרוטוקול אלקטרופורזה של PCR וג'ל המבוצע במהלך בדיקת הדנ"א הגנומי של הזרע.
- Sanger רצף את מוצרי PCR כדי לזהות את האנשים F1 עם אותה עריכה רצויה.
- אופציונלי: אם העריכה הרצויה מציגה או משבשת אתר הגבלה, סנן עם פרוטוקול תקציר הגבלות מתאים.
- הטרוזיגוטים F1 בהצלבה עם אותה עריכה רצויה.
- סנן את צאצאי F2 עבור הפנוטיפ ו/או העריכות הגנומיות הצפויות.
תוצאות
הגישות הניסיוניות המתוארות בפרוטוקול זה מאפשרות זיהוי מהיר יותר של עריכות גנום או אללים מזיקים לכאורה על ידי התמקדות בניתוח אלפי גנומים הנגזרים מאוסף הזרע הגברי המוזרק F0. איור 2 מדגיש כיצד לפרש את התוצאות המתקבלות באמצעות פרוטוקול זה.
כדי ליצור מוטציות בלוקוס p2ry12, הוזרקו לעוברים של דגי זברה בשלב חד-תאי Cas9 אנדונוקלאז ו-sgRNA ספציפי ל-p2ry12 (איור 2B, אור אפור). דגים בהזרקת F0 הוכנסו למערכת עד לבגרות מינית, וזרע נאסף משישה זכרים בודדים. DNA הופק מדגימות הזרע בחום גבוה ובתנאים בסיסיים (מיצוי DNA HotSHOT) ונוטרל לפני הגברת ה-PCR של מוקד p2ry12. מוצרי ה-PCR הופרדו על אחוז גבוה (4%) של ג'ל אגרוז למשך שעה וחצי במתח גבוה (150 וולט). בדוגמה זו, האמפליקון מסוג פרא רץ כרצועה בהירה אחת באורך של כ-250 זוגות בסיסים (איור 2C; WT). לעומת זאת, האמפליקונים הזכריים שהוזרקו ב-F0 והכילו אינדלים רצו על הג'ל כרצועות מרובות (איור 2C; נתיבים #1-2, #5-8). לחלופין, האמפליקונים הזכריים שעברו מוטציה בהזרקת F0 יכולים לרוץ כרצועה אחת עם תנועתיות ג'ל שונה (לא מוצג בדוגמה p2ry12, אך ניכר במכפלת PCR של DNAH10 F0-male #2; איור 2E). היה פחות ברור בג'ל לדוגמה p2ry12 אם מדגם #3 ומדגם #4 מכילים אינדל בהשוואה ללהקה מסוג פרא, כך שאנשים אלה עשויים שלא להיות המועמדים הטובים ביותר למייסד. הבידוד של אללים מזכרי F0 עם שינויים ייחודיים יותר באמפליקון הוא הטוב ביותר להפצת נשאי נבט יציבים מכיוון שהם מקבלים ניקוד בקלות על 4% ג'ל אגרוז. לדוגמה, דגימת הזרע #6 של זכר F0 הכילה מחיקה גדולה באחד האללים (איור 2C; נתיב 6; פס בהיר עם ניידות ג'ל מוגברת). אם אלל מחיקה גדול זה היה נבחר בדור F1, ניתן היה להבחין בו בקלות מאלל WT על ג'ל אגרוז 4%. לחלופין, אם רוצים אוסף של אללים שונים, F0-male #7 יכול להיות מועמד יעיל למייסד מכיוון שנראה כי מוצר ה-PCR שלו מכיל מספר אללים בדידים בעלי ניידות שונות. לאחר בחירת זכר מייסד, ניתן לבודד את האלל הרצוי על ידי הצלבת הפרט בחזרה לזן המקורי מסוג פרא ששימש לזריקות.
כדי ליצור מוטציה ספציפית בגן dnah10, הוזרקו לעוברים של דגי זברה בשלב חד-תאי Cas9 endonuclease, sgRNA ספציפי ל-dnah10 (איור 2D; אור אפור), אוליגונוקלאוטיד של תורם DNA שהכיל את העריכה הרצויה (איור 2D; אדום) ואתר הגבלה ספציפי של BstNI (איור 2D סוגריים מרובעים)., עיצוב זה מאפשר זיהוי קל של שילוב התורם עם תקציר הגבלה לאחר PCR. יתר על כן, על-ידי שינוי זוגות בסיסים בתוך אתר זיהוי sgRNA (איור 2D; אור אפור), עיצוב זה מונע עיכול Cas9 של רצף התורמים. ברגע שהדג שמוזרק F0 הגיע לבגרות מינית, נאסף זרע, והדנ"א הופק בשיטת הזריקה החמה. מדגימות אלה, מוקד ה- DNAH10 הוגבר באמצעות PCR, והמוצרים הופרדו על אחוז גבוה (4%) ג'ל אגרוז למשך שעה אחת במתח גבוה (150 V). בדוגמה זו, האמפליקון מסוג פרא רץ כרצועה בהירה יחידה באורך של כ-400 זוגות בסיסים (איור 2E; לוח עליון: WT). לעומת זאת, האמפליקונים הזכריים שהוזרקו ב-F0 והכילו אינדלים רצו כרצועות מרובות (איור 2E; פאנל עליון: נתיבים #1, #4 ו-#5) או כרצועה אחת עם ניידות ג'ל מופחתת (איור 2E; פאנל עליון: נתיב #2). כדי לקבוע אם אחת מדגימות הזרע הכילה אללים משולבים בתורם, מוצרי ה-PCR עוכלו עם אנזים הגבלת BstNI למשך שעה אחת, והמוצר הופעל על ג'ל אגרוז 4% למשך שעה אחת במתח גבוה (150 V). השוואת מוצר ה-PCR הלא מעוכל (איור 2D; פאנל עליון) למוצר המעוכל (איור 2D; לוחות תחתונים) מגלה דגימות עם אינטגרציה אפשרית של מבנה התורם. בדוגמה זו, לזכר #1 שמוזרק F0 הייתה רצועה נוספת במוצר המעוכל שלא הייתה קיימת במוצר ה-PCR (איור 2D; לוח תחתון: נתיב #1, בעיגול). לכן, זכר זה מייצג את המועמד המייסד הטוב ביותר להקמת קו מוטנטי עם הנוק-אין הרצוי.
ברגע שהזכר נושא ה-F0 מוציא את הזכר, האלל חייב להיות מאומת ברצף בצאצאי F1. מוצע לרצף ישירות את האמפליקונים שמקורם בדג ההטרוזיגוטי F1, ולאחר מכן לבצע שיטות ניתוח אללים הטרוזיגוטיות כגון Poly Peak Parser13 או להשתמש במגוון שיטות מבוססות ריצוף מהדור הבא, כגון ריצוף MiSeq 14 או Hi-Tom15. זוהי גישה הכרחית ומשלימה כדי להבטיח שהעריכה המדויקת או מוטציית האינדל המזיקה אכן הולכת לנבט ואינה רק תוצר של ניתוח אלקטרופורזה בג'ל. ואכן, השימוש בגישות ריצוף של הדור הבא לריצוף DNA זרע מנשאי F0 יכול לשמש בקלות במקום ניתוח אלקטרופורזה בג'ל המתואר בפרוטוקול זה. עם זאת, שיטת גנוטיפ ג'ל אגרוז הניתנת לניקוד בקלות היא גישה חסכונית ושוויונית יותר עבור הקהילה העולמית של חוקרי דגי זברה.

איור 1: הכנה להליך סחיטת הזרע . (A) הספוג ממוקם מתחת לסטריאומיקרוסקופ בהגדלה נמוכה עם תאורה עילית. (B) הספוג הלח 1 ל-x 1 בספוג, חתוך עם דיוות אליפסה (קו מקווקו), משמש להחזקת הצד הגחוני של הדג הזכר המורדם כלפי מעלה במהלך ההליך. (C) אנטומיה של הצד הגחוני של הדג הזכר המורדם המתארת את הסנפירים האנאליים (af, pink), סנפירי האגן (pf, כחול) וקלואקה (חץ), שם יסולק זרע במהלך ההליך. (ג') מלקחיים פילטרים משמשים לסחיטה עדינה של הדג הזכר המורדם מהזימים אל הקלואקה, בעוד שהזרע המגורש נשאב לתוך פיפטת זכוכית על ידי פעולה נימית (חץ לבן). (D) צינור נימי המכיל כמות מספקת של זרע מגורש (נוזל אטום; סוגר שחור) לצורך מיצוי וניתוח DNA במורד הזרם. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
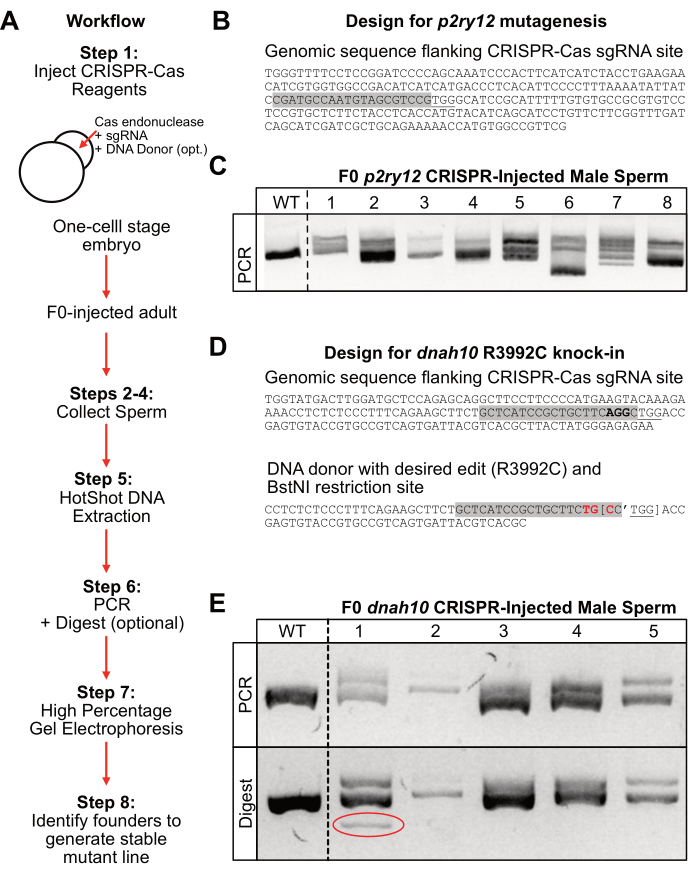
איור 2: זרימת עבודה ותוצאות מייצגות. (A) זרימת עבודה כללית של הפרוטוקול. (B) עיצוב מייצג של אתר p2ry12 sgRNA (סימון אפור) עם אתר PAM ספציפי ל-Cas9 (מסומן בקו תחתון). (C) תוצאות מייצגות של אלקטרופורזה בג'ל 4% לאחר 1.5 שעות בבקרת 150 וולט מסוג Wild (WT) ודגימות זרע זכריות בהזרקת קריספר F0-p2ry12 (1-8) לאחר הגברה של PCR. (D) עיצוב מייצג של אתר dnah10 sgRNA (הדגשה אפורה) עם אתר PAM ספציפי ל-Cas9 (מסומן בקו תחתון) ליד קודון המטרה (מודגש). רצף תורם DNA עם עריכת הקודון הרצויה (אדום מודגש) ואתר הגבלת BstNI (סוגריים עם גרש המסמן את אתר החיתוך). (E) תוצאות מייצגות של אלקטרופורזה בג'ל 4% לאחר שעה אחת ב- 150 V. למעלה: תוצר PCR של דגימות זרע זכריות מסוג פרא (WT) ו- F0 dnah10 בהזרקת קריספר (1-5). למטה: תוצר עיכול הגבלה BstNI של הדגימות הנ"ל. דוגמה 1 מדגימה את השילוב המוצלח של מבנה התורם בהתבסס על הרצועה הנוספת לאחר העיכול (עיגול אדום). אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
Discussion
פרוטוקול זה מתאר שיטה לאפיון מהיר של עריכות גנום משוערות או מוטציות ממוקדות באמצעות טכנולוגיית CRISPR-Cas על ידי ניתוח ממוקד של גנומי זרע זכריים F0. פרוטוקול זה צריך להיות מקובל על מודלים אחרים של בעלי חיים שבהם הזרע זמין לדגימה ללא המתת חסד. שיטה זו תגדיל את תפוקת הסינון לעריכות רצויות והיא שימושית במיוחד לזיהוי אירועי דפיקה נדירים בתיווך HDR. גישה זו משמשת גם להפחתת מספר חיות הניסוי המשמשות למציאת עריכה יציבה של קו הנבט על ידי הקלה על סינון מהיר של אלפי גנומים פוטנציאליים בדגימת זרע אחת מנשא F0 משוער, בניגוד לגישות מסורתיות יותר שעשויות לדרוש סינון של מאות עוברים שמקורם בהצלבות של נשאי F0 משוערים.
פרוטוקול זה מתבסס על פרוטוקולים מבוססים לאיסוף זרע בדגי זברה 7,10,14,16,17,18 על ידי הכללת טכניקה הניתנת לשחזור לזיהוי עריכות קו הנבט באמצעות אלקטרופורזה בג'ל ברזולוציה גבוהה. ניתן לשלב גישה זו בקלות בכל זרימת עבודה סטנדרטית של CRISPR/Cas כדי להגדיל את התפוקה לסינון ובידוד של עריכות גנום מטרה. בנוסף, פרוטוקול זה מתאים למעבדות המאוישות בכוח אדם בעל הכשרה וניסיון מגוון, כמו גם למעבדות הוראה. עם זאת, שיטת איסוף הזרע שלנו אינה מספיקה לשימור בהקפאה, אשר תואר במומחיות בפרסומים קודמים10,17.
פרוטוקול זה משתמש באלקטרופורזה של ג'ל אגרוז ברזולוציה גבוהה כדי לזהות נשאים זכריים משוערים של מוטציות גנומיות רצויות ומדויקות. עם זאת, DNA גנומי של זרע מקובל על מספר עצום של גישות אחרות, כולל ניתוח מקטע פלואורסצנטי או ריצוף ברקודי14, ניתוח התכה ברזולוציה גבוהה18, או זיהוי אמפליקון פשוט של תגים פלואורסצנטיים או אפיטופים באמצעות גישות אלקטרופורזה סטנדרטיות בג'ל. האימות במורד הזרם של כל האללים באמצעות גישות כגון ריצוף מבוסס סנגר או הדור הבא חייב להיעשות לפני תחילת העבודה הניסיונית על אללים משוערים. ואכן, השיטות הקיימות לבדיקת מוטציות בעוברים באמצעות גישת ריצוף הדור הבא14 יכולות לעקוף את הצורך בניתוח על ג'ל אגרוז, המתואר בפרוטוקול זה. עם זאת, שיטת גנוטיפ הניתנת לניקוד בג'ל היא גישה חסכונית ושוויונית יותר שיש לקחת בחשבון, בהתחשב בכך שלא לכל המעבדות יש גישה קלה לשיטות ריצוף זולות של הדור הבא במהלך שלבי הבידוד והניסוי של העבודה עם כל זן מוטנטי.
לסיכום, פרוטוקול זה מספק הוראות שלב אחר שלב לסינון גנום זרע מזכרים ערוכים בקריספר/Cas, כך שיהיה צורך בפחות הצלבות ופחות בדיקת PCR של עוברים כדי לסנן עריכות רצויות. היישום של שיטה זו יקטין את מספר הדגים שיש ליצור ולנתח כדי לזהות בהצלחה את האללים הערוכים של עניין, אשר גם מקטין את הזמן ואת העלות של כוח אדם ליצירת קווים יציבים.
Disclosures
ללא.
Acknowledgements
ברצוננו להודות לאנה הינדס מבית הספר לרפואה של אוניברסיטת וושינגטון על מאמציה הראשוניים להשיג DNA גנומי זרע באיכות טובה בשיטת הזריקה החמה. עבודה זו מומנה על ידי המכון הלאומי לדלקת פרקים ומחלות שרירים ושלד ועור של המכונים הלאומיים לבריאות תחת פרס (R01AR072009 ל- R.S.G).
Materials
| Name | Company | Catalog Number | Comments |
| Agarose powder | Fisher BioReagents | BP1356-100 | |
| Breeding tanks | Carolina Biological | 161937 | |
| BstNI Restriction Enzyme | NEB | R0168S | |
| Cas9 Endonuclease | IDT | 1081060 | |
| DNA Ladder, 100 bp | Thermo Scientific | FERSM0241 | |
| dnah10 donor construct | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry. Phosphorothioate bond on the donor at the first three phosphate bonds on both the 5’ and 3’ ends (5'-CCTCTCTCCCTTTCAGAAGCTTC TGCTCATCCGCTGCTTCTGCCT GGACCGAGTGTACCGTGCCGTC AGTGATTACGTCACGC-3') | |
| dnah10 forward primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CATGGAACTCTTTCCTAATGAGT TTGGC-3') | |
| dnah10 reverse primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry ('5-AGTAGAGATCACACATCAACAGA ATACAGC-3') | |
| dnah10 synthetic sgRNA | Synthego | Synthetic sgRNA, target sequence: 5'-GCTCATCCGCTGCTTCAGGC-3' | |
| Electrophoresis power supply | Thermo Scientific | 105ECA-115 | |
| Filter forceps | Millipore | XX6200006P | |
| Fish (system) water | Generic | n/a | |
| Gel electrophoresis system (including casting frame, comb, and electrophoresis chamber) | Thermo Scientific | B2 | |
| Gel imaging light box | Azure Biosystems | AZI200-01 | |
| Gel stain, 10000X | Invitrogen | S33102 | |
| Glass bowl, 250 mL | Generic | n/a | |
| Isolation tanks, 0.8 L | Aquaneering | ZT080 | |
| Microcap capillary tube with bulb, 20 µL | Drummond | 1-000-0020/CA | |
| Minicentrifuge | Bio-Rad | 12011919EDU | |
| Micropipettes, various with appropriate tips | Generic | n/a | |
| Microwave | Generic | n/a | |
| Nuclease free water | Promega | P119-C | |
| Paper towels | Generic | n/a | |
| PCR tubes, 0.2 mL | Bioexpress | T-3196-1 | |
| Plastic spoon, with drilled holes/slots | Generic | n/a | |
| KCl solution, 0.2 M RNAse Free | Sigma-Aldrich | P9333 | |
| p2ry12 forward primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CCCAAATGTAATCCTGACCAGT -3') | |
| p2ry12 reverse primer | Sigma-Aldrich | DNA Oligo in Tube; 0.025 nM, standard desalt purification, dry (5'-CCAGGAACACATTAACCTGGAT -3') | |
| p2ry12 synthetic sgRNA | Synthego | Synthetic sgRNA, target sequence: 5'-GGCCGCACGAGGTCTCCGCG-3' | |
| Restriction Enzyme 10X Buffer | NEB | B6003SVIAL | |
| NaOH solution, 50 mM | Thermo Scientific | S318; 424330010 | |
| Sponge, 1-inch x 1-inch cut with small oval divot | Generic | n/a | |
| Stereomicroscope | Zeiss | Stemi 508 | |
| Taq polymerase master mix, 2X | Promega | M7122 | |
| TBE Buffer Concentrate, 10X | VWR | E442 | |
| Thermal Cycler | Bio-Rad | 1861096 | |
| Tissue paper | Fisher Scientific | 06-666 | |
| Tricaine-methanesulfonate solution (Syncaine, MS-222), 0.016% in fish water (pH 7.0±0.2) | Syndel | 200-266 | |
| Tris Base, 1M (Buffered with HCl to ph 8.0) | Promega | H5131 |
References
- Auer, T. O., Duroure, K., De Cian, A., Concordet, J. P., Del Bene, F. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair. Genome Research. 24 (1), 142-153 (2014).
- Hwang, W. Y., et al. Efficient genome editing in zebrafish using a CRISPR-Cas system. Nature Biotechnology. 31 (3), 227-229 (2013).
- Jao, L. E., Wente, S. R., Chen, W. Efficient multiplex biallelic zebrafish genome editing using a CRISPR nuclease system. Proceedings of the National Academy of Sciences of the United States of America. 110 (34), 13904-13909 (2013).
- Troutwine, B. R., et al. The Reissner fiber is highly dynamic in vivo and controls morphogenesis of the spine. Current Biology. 30 (12), 2353-2362 (2020).
- Xu, Y., Li, Z. CRISPR-Cas systems: Overview, innovations and applications in human disease research and gene therapy. Computational and Structural Biotechnology Journal. 18, 2401-2415 (2020).
- Creaser, C. W. The technic of handling the zebrafish (Brachydanio rerio) for the production of eggs which are favorable for embryological research and are available at any specified time throughout the year. Copeia. 1930 (4), 159-161 (1934).
- Carmichael, C., Westerfiel, M., Varga, Z. M. Cryopreservatin and in vitro fertilization at the zebrafish international resource center. Methods in Molecular Biology. 546, 45-65 (2009).
- Wang, Y., Troutwine, B. R., Zhang, H., Gray, R. S. The axonemal dynein heavy chain 10 gene is essential for monocilia motility and spine alignment in zebrafish. Developmental Biology. 482, 82-90 (2021).
- Gray, R. S., et al. Postembryonic screen for mutations affecting spine development in zebrafish. Developmental Biology. 471, 18-33 (2021).
- Brocal, I., et al. Efficient identification of CRISPR/Cas9-induced insertions/deletions by direct germline screening in zebrafish. BMC Genomics. 17, 259 (2016).
- Davis, M. W., Jorgensen, E. M. ApE, A Plasmid Editor: A freely available DNA manipulation and visualization program. Frontiers in Bioinformatics. 2, 816619 (2022).
- Labun, K., et al. CHOPCHOP v3: Expanding the CRISPR web toolbox beyond genome editing. Nucleic Acids Research. 47, 171-174 (2019).
- Hill, J. T., et al. Poly Peak Parser: Method and software for identification of unknown indels using sanger sequencing of polymerase chain reaction products. Developmental Dynamics. 243 (12), 1632-1636 (2014).
- Varshney, G. K., et al. High-throughput gene targeting and phenotyping in zebrafish using CRISPR/Cas9. Genome Research. 25 (7), 1030-1042 (2015).
- Liu, Q., et al. Hi-TOM: A platform for high-throughput tracking of mutations induced by CRISPR/Cas systems. Science China. Life Sciences. 62 (1), 1-7 (2019).
- Sorlien, E. L., Witucki, M. A., Ogas, J. Efficient production and identification of CRISPR/Cas9-generated gene knockouts in the model system Danio rerio. Journal of Visualized Experiments. (138), e56969 (2018).
- Draper, B. W., Moens, C. B. A high-throughput method for zebrafish sperm cryopreservation and in vitro fertilization. Journal of Visualized Experiments. (29), e1395 (2009).
- Parant, J. M., George, S. A., Pryor, R., Wittwer, C. T., Yost, H. J. A rapid and efficient method of genotyping zebrafish mutants. Developmental Dynamics. 238 (12), 3168-3174 (2009).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved