Method Article
Лазерная микродиссекция для видогностических однотканных применений
В этой статье
Резюме
Описан протокол, который использует лазерную микродиссекцию для выделения отдельных тканей нематоды для секвенирования РНК. Протокол не требует видоспецифических генетических инструментов, что позволяет сравнивать профили экспрессии генов между различными видами на уровне образцов одной ткани.
Аннотация
Одноклеточные методологии произвели революцию в анализе транскриптомов конкретных типов клеток. Тем не менее, они часто требуют видоспецифических генетических «инструментов», таких как промоторы, управляющие тканеспецифической экспрессией флуоресцентных белков. Кроме того, протоколы, которые разрушают ткани для изоляции отдельных клеток, удаляют клетки из их родной среды (например, передача сигналов от соседей) и могут привести к стрессовым реакциям или другим отличиям от состояний экспрессии нативных генов. В настоящем протоколе лазерная микродиссекция (LMD) оптимизирована для выделения отдельных кончиков хвоста нематоды для изучения экспрессии генов во время морфогенеза кончика хвоста самца.
LMD позволяет изолировать часть животного без необходимости клеточного нарушения или видоспецифических инструментариев и, таким образом, применим к любому виду. Впоследствии одноклеточные протоколы подготовки библиотеки РНК-seq, такие как CEL-Seq2, могут быть применены к изолированным LMD одиночным тканям и проанализированы с использованием стандартных конвейеров, учитывая, что для вида доступен хорошо аннотированный геном или транскриптом. Такие данные могут быть использованы для установления того, насколько сохранены или различны транскриптомы, лежащие в основе развития этой ткани у разных видов.
Ограничения включают в себя возможность вырезать интересующую ткань и размер выборки. Анализ мощности показывает, что для 80% мощности требуется всего 70 кончиков хвоста на условие. Тесная синхронизация развития необходима для получения этого количества животных на одной стадии развития. Таким образом, описан способ синхронизации животных с интервалом 1 ч.
Введение
Нематоды, особенно рабдитидные нематоды, связанные с модельной системой Caenorhabditis elegans, являются замечательной группой животных для эволюционной биологии развития (EDB) по многим причинам 1,2. Преимущества включают в себя небольшое количество клеток, определенные и последовательные клеточные линии, прозрачность и простоту культивирования и ведения. Есть также много доступных ресурсов, в том числе высококачественные геномы для нескольких видов и для C. elegans, обширные молекулярно-генетические инструменты и знания о развитии, генетике, анатомии и физиологии 3,4,5,6.
Как и у многих других организмов, способность характеризовать динамику транскриптома в отдельных тканях или одиночных клетках произвела революцию в анализе развития у C. elegans 7,8,9,10. Возможность сравнивать одноклеточные транскриптомы у нематод аналогичным образом преобразует EDB с помощью этих организмов. Например, такие сравнения дадут представление о том, как развивались генные регуляторные сети для персонажей (признаков), которые были сохранены, для персонажей, которые разошлись, или для персонажей, которые развивались независимо.
Тем не менее, изоляция определенных тканей или клеток от нематод является одной из больших проблем. Для многих организмов одиночные клетки могут быть диссоциированы от тканей и собраны непредвзятым образом или могут быть помечены тканеспецифической экспрессией флуоресцентного белка и отсортированы с помощью флуоресцентно-активированной сортировки клеток (FACS)11. У C. elegans высокопроизводительная (HTP) изоляция клеток была ограничена в основном эмбрионами, потому что жесткая внешняя кутикула (и гидростатический скелет) препятствовала изоляции клеток от личинок и взрослых особей. Чтобы обойти эту проблему, некоторые методы использовали генетические инструменты для целых червей C. elegans, таких как тканеспецифическая мРНК-метка12, и дифференциальные сравнения экспрессии между диким типом и мутантами, влияющими на клетку типа13. Более поздние методы преодолели проблему, растворив кутикулу, чтобы выделить ядра14 или целые клетки 8,9,15. Однако клеточная изоляция и клеточная культура имеют очевидные недостатки, заключающиеся в том, что клетки удаляются из их естественного контекста развития или анатомии , например, вдали от клеточной сигнализации и контакта с внеклеточным матриксом, что, как ожидается, повлияет на профиль экспрессии генов15. Кроме того, генетические инструменты и тканеспецифические маркеры являются видоспецифичными (т. е. они могут быть использованы только у C. elegans).
LMD обеспечивает альтернативный метод выделения тканей без нарушения естественного контекста клеток. Что важно для EDB, LMD также позволяет сравнивать транскриптомы из гомологичных тканей разных видов без необходимости в видоспецифических генетических инструментариях, если доступны геномные или целые последовательности транскриптомов этих видов. LMD включает в себя нацеливание на ткани путем прямого микроскопического наблюдения и использования лазерного микролуча, интегрированного в оптику микроскопа, для вырезания и сбора (захвата) интересующей ткани16. Ограничения LMD заключаются в том, что он не способствует очень HTP-подходам (хотя профили транскрипции для хвостовых кончиков, как описано в этом протоколе, были надежными с ~ 70 образцами), некоторые образцы может быть трудно препарировать, а разрезы ограничены точностью лазера и тем, что может быть визуализировано в микроскопе.
Целью настоящего протокола является описание того, как LMD, за которым следует однотканевая РНК-Seq, может быть использована для получения специфических для стадии и тканей данных транскриптома от нематод. В частности, он демонстрирует LMD для изоляции кончиков хвоста от личинок четвертой стадии (L4) C. elegans. Однако этот метод может быть адаптирован к другим тканям и, конечно же, к разным видам.
У C. elegans есть 4 клетки, которые составляют кончик хвоста как у самцов, так и у гермафродитов. Во время стадии L4 у самцов, но не у гермафродитов, клетки хвостового кончика меняют свою форму и мигрируют кпереди и внутрь. Этот процесс также происходит у некоторых, но не у всех других видов рабдитидных нематод. Поэтому кончик хвоста является хорошей моделью для эволюции полового диморфного морфогенеза. Из-за своего положения хвостовой кончик также легко изолируется с помощью LMD.
Для получения профилей транскриптома из кончиков хвоста настоящий протокол использует CEL-Seq2, метод РНК-seq, разработанный для одиночных клеток17,18. Этот метод имеет ряд преимуществ для тканей, полученных из LMD. CEL-Seq2 очень чувствителен и эффективен, используя уникальные молекулярные идентификаторы (UMI), чтобы обеспечить простую количественную оценку считывания мРНК, транскрипцию in vitro для обеспечения линейной амплификации и штрих-кодирование, которое позволяет мультиплексировать отдельные образцы тканей. Единственным ограничением CEL-Seq2 является то, что восстановленные показания смещены к 3'-концу мРНК, и большинство изоформ, таким образом, не могут быть различимы.
протокол
1. Синхронизация червей
ПРИМЕЧАНИЕ: Ниже описаны два метода синхронизации развития C. elegans и других видов rhabditid.
- Синхронизация при остановке первой личиночной стадии (L1) после обработки щелочным гипохлоритом (отбеливателем).
ПРИМЕЧАНИЕ: Этот метод был подробно описан ранее19. Этот метод опирается на две особенности C. elegans , которые также верны для нескольких других видов рабдитидов: (1) яичная скорлупа устойчива к отбеливанию, тогда как кутикула, окружающая взрослых и личиночных червей, нет. (2) Личинки первой стадии останавливают развитие при содержании без пищи20.- Обработайте гравидных гермафродитов (или самок) разбавленным раствором отбеливателя, чтобы разрушить их кутикулу и освободить эмбрионы.
- Извлеките эмбрионы из отбеливателя и держите их без пищи, пока не вылупится весь L1.
- Поместите арестованный L1 на еду, где все возобновят развитие примерно в одно и то же время.
ПРИМЕЧАНИЕ: Выход из ареста L1 может произойти в течение одного часа.
- Синхронизация с методом «hatch-off» (используется здесь; Рисунок 1 вверху):
ПРИМЕЧАНИЕ: Метод вылупления обеспечивает плотную синхронизацию без нарушения разработки (остановка L1 влияет на развитие даже на более поздних стадиях21). Протокол адаптирован из Pepper et al.22. Целью этого метода является сбор L1, которые вылупились в течение определенного периода из пластины, содержащей только эмбрионы.- Выберите матерей: Вечером перед выполнением вылупления соберите ~ 30 гравид гермафродитов на тарелку, засеянную кишечной палочкой OP50.
- Инкубировать при температуре 25 °C для яйцекладки в течение ночи.
ПРИМЕЧАНИЕ: Выберите тарелку без трещин или пузырьков, где черви могут застрять. Избегайте тарелок с очень толстым бактериальным газоном, так как позже будет трудно удалить всех червей. При работе с чувствительным к температуре штаммом отрегулируйте время откладывания яиц, чтобы учесть более длительный эмбриогенез. Выбирайте матерей на максимальной стадии яйцекладки. - Удалить матерей и личинок: на следующее утро под рассекающим микроскопом (20-кратное увеличение) аккуратно пипетку 1-2 мл буфера М9 прижимают к стенке пластины без брызг; закрутите пластину, чтобы выбить червей.
- Удалите и выбросьте всю жидкость и червей, приложив наконечник пипетки к стенке пластины на краю агара, чтобы избежать протыкания отверстий. Убедитесь, что на пластине не осталось червей (и только яиц/эмбрионов), особенно L1; в противном случае повторите стирку.
- Поместите пластину при температуре 25 °C в течение 1 часа и подождите, пока вылупятся некоторые L1.
- Соберите только что вылупившиеся L1s: осторожно опустите 1 мл буфера M9 на агар. Закрутите пластину, чтобы выбить L1, но не эмбрионы. Аккуратно поместите пипетку в буфер и червей в центрифужную трубку объемом 1 мл.
- Центрифугируйте трубку в течение 1 мин при ~18 000 × г. Удалите супернатант.
- Пипетка L1 прямо на бактериальный газон семенной пластины. Проверьте под микроскопом вскрытия, что взрослые черви или эмбрионы не присутствуют.
- Держите червей при 25 °C до тех пор, пока они не разовьются до нужной стадии.
ПРИМЕЧАНИЕ: Если условия являются оптимальными, с той же пластины можно собрать еще две партии L1. Осмотрите начальную пластину, чтобы убедиться, что L1 отсутствует. При необходимости снова вымойте. Повторите шаги 1.2.5-1.2.9. -
Проверьте сроки разработки. Прежде чем перейти к последующему применению, осмотрите некоторых червей под составным микроскопом при 400-кратном увеличении, чтобы подтвердить, что они достигли желаемой стадии развития, здесь L3.
ПРИМЕЧАНИЕ: Миграционное расстояние дистальных кончиков клеток или линкерных клеток может быть использовано в качестве ориентира, в дополнение к развитию вульвы. Для развития вульвы Mock et al.23 представляют собой полезное руководство, хотя время в этом исследовании было определено при 20 °C. При 25 °C дикие C. elegans будут подвергаться линьке L3-L4 через 24 часа после вылупления.
2. Сбор самцов L4 и гермафродитов и фиксация
- Перед фиксацией готовят без РНКаза, холодный (-20 °C), 70% метанол.
- Под рассеченным микроскопом при 30-50-кратном увеличении начните отбирать самцов и гермафродитов из синхронных пластин на отдельные несеянные пластины, как только полы можно будет различить (~ 21 ч после вылупления, рисунок 2), и продолжайте сбор в течение 1-2 ч или до тех пор, пока не будет собрано 200 животных.
- Держите червей при температуре 25 °C до тех пор, пока они не достигнут желаемой стадии для эксперимента.
- Смойте червей с тарелки с 1-2 мл буфера M9 с помощью наконечника пипетки, предварительно промытого буфером M9, содержащим 0,01% моющего средства (чтобы предотвратить прилипание червей к наконечнику).
- Перенесите червей в трубку центрифуги объемом 1 мл.
- Вращайте в течение 1 мин при 21 000 × г , чтобы гранулировать червей. Удалите супернатант.
- Добавьте 1 мл буфера M9 и перемешайте, чтобы разбить гранулу.
- Открутите в течение 1 мин при 21 000 × г , чтобы гранулировать червей. Удалите супернатант.
- Повторите стирку.
- Добавьте 1 мл ледяного 70% метанола и хорошо перемешайте.
- Вращайте в течение 1 мин при 21 000 × г , чтобы гранулировать червей. Удалите супернатант.
- Повторите шаги 2.10 и 2.11.
- Добавьте 500 мкл 70% метанола, перемешайте и храните при 4 °C в течение 1 ч до ночи.
3. Лазерная микродиссекция
ПРИМЕЧАНИЕ: С этого момента используйте реагенты и расходные материалы, не содержащие РНКазы; используйте наконечники фильтров.
- Если для обработки образцов используется метод CEL-Seq2, подготовьте мастер-микс для каждой грунтовки CEL-Seq2 (Дополнительная таблица S1): пипетку 2 мкл грунтовки CEL-Seq2, 1 мкл 10 мМ dNTP и 9 мкл 1% β-меркаптоэтанола (в воде без РНКазы) в маркированную трубку объемом 200 мкл.
- Монтаж на слайде
- Под рассеченным микроскопом пипетку 20 мкл фиксированных червей (20-40 червей из стадии 2.13) на матовую сторону полиэтиленнафталата (PEN) - мембранного стеклянного предметного стекла (где находится мембрана).
- Подождите, пока метанол испарится. Используйте скользящий подогреватель, чтобы ускорить испарение.
ПРИМЕЧАНИЕ: Можно нанести дополнительные капли метанола и наконечник пипетки, используемый для распространения червей, если они начинают слипаться по мере высыхания. Когда черви находятся в скоплениях, их может быть трудно рассечь.
- Настройка микроскопа
ПРИМЕЧАНИЕ: Следующий протокол относится к инструменту, указанному в Таблице материалов. Его необходимо отрегулировать, если используется другой LMD-микроскоп.- Поместите настольный увлажнитель за сценой на сторону микроскопа LMD. Убедитесь, что пар дует прямо на сцену.
ПРИМЕЧАНИЕ: Увлажнитель полезен для уменьшения статического электричества, которое в противном случае может предотвратить падение небольшой секции мембраны в крышку трубки. - Поверните ключ для лазерной энергии.
- Включите питание управления сценой.
- Включите блок управления микроскопом.
- Открытое программное обеспечение для лазерной микродиссекции.
- Снимите пластиковый экран над сценой.
- Нажмите кнопку выгрузки со стрелкой вверх для загрузки мембранных слайдов.
- Убедитесь, что горка полностью сухая, переверните так, чтобы мембрана была обращена вниз.
- Вставьте слайд и нажмите кнопку Продолжить в окне изменения образца .
- Замените пластиковый щит.
- В нижней части экрана выберите, какой держатель слайда содержит слайд.
- Чтобы загрузить трубки, нажмите кнопку выгрузки со стрелкой вниз.
- Вытащите лоток и извлеките блок трубки.
ПРИМЕЧАНИЕ: Блок трубок, используемый для этого эксперимента, предназначен для 500 мкл ПЦР-пробирок. - Вставьте в держатель колпачки трубки ПЦР объемом 500 мкл и сложите трубку под нее.
- Верните блок в лоток и сдвиньте лоток обратно в стадию микроскопа.
- Во всплывающем окне смены коллекторного устройства выберите трубки ПЦР и нажмите кнопку ОК.
- Нажмите на пустое место трубки в левом нижнем углу экрана под крышками трубок коллекторного устройства.
- На панели управления «Микроскоп» выберите TL-BF для пропускаемого света яркого поля.
- Поместите настольный увлажнитель за сценой на сторону микроскопа LMD. Убедитесь, что пар дует прямо на сцену.
- Режущий
ПРИМЕЧАНИЕ: Данный протокол относится к инструменту, указанному в Таблице материалов.- Используя объектив 2,5x, отрегулируйте фокус до тех пор, пока черви и структура мембраны не станут видимыми.
- Переключитесь на объектив 20x.
- Переместите сцену в регион без червей. Отрегулируйте фокус таким образом, чтобы пузырькообразные структуры в мембране имели желтоватый цвет (рисунок 3A, B), чтобы сфокусировать лазер на правильной фокальной плоскости.
- Установка параметров лазера; для кончиков хвоста начните с Power 45, диафрагмы 30 и скорости 20.
- На панели управления лазером выберите Калибровка. Следуйте инструкциям.
ПРИМЕЧАНИЕ: Инструмент выполнит этот шаг автоматически. Это гарантирует, что фигура, нарисованная мышью на экране, идентична форме, вырезанной лазером. - В нижней части экрана у коллекторного устройства колпачки трубки нажмите на позицию А.
- В правой части экрана выберите одну фигуру | Нарисовать + Вырезать. В левой части экрана выберите PtoP.
- Нарисуйте линию.
- Нажмите кнопку Начать резать , чтобы лазер прорезал мембрану.
ПРИМЕЧАНИЕ: Он также может вытравить линию в стакане. - Если этот тестовый срез выглядит хорошо (мембрана разрезана, края разреза выглядят гладкими), переходите к следующему шагу. В противном случае отрегулируйте фокус и вырежьте еще одну линию.
- Найдите червя. Переключитесь на Move + Cut и используйте мышь, чтобы разрезать хвост.
ПРИМЕЧАНИЕ: Если лазер не разрезает хвост, отрегулируйте фокус и увеличьте мощность лазера. Для более толстых тканей мощность лазера, возможно, придется установить на 60. - Сохраните параметры: вкладка «Файл» | Сохранить конфигурацию приложения; для последующего извлечения восстановите конфигурацию приложения.
- Чтобы собрать образец, переключитесь на настройку Draw + Cut с функцией PtoP и нарисуйте фигуру, чтобы завершить разрез секции мембраны (рисунок 3C).
ПРИМЕЧАНИЕ: Более крупные мембранные секции и секции в форме прямоугольников или треугольников, а не кругов или овалов, легче найти в крышке коллекторной трубки. - Выберите следующую трубку в разделе Collector Device Tube Cap в нижней части экрана и вырежьте следующий кончик хвоста.
- После того, как четыре хвоста будут разрезаны, разгрузите стойку трубки (нажмите Выгрузить стрелкой вниз) и найдите участки мембраны под рассекающим микроскопом (рисунок 3D).
ПРИМЕЧАНИЕ: Секции могут быть расположены в середине крышки трубки или приклеены к боковой части колпачка. - Продолжите работу с подчиненным приложением. Для CEL-Seq2 пипетка 1,2 мкл мастер-микса для грунтовки CEL-Seq2 (из шага 3.1) непосредственно поверх образца.
- Закройте трубку, пометьте номер грунтовки и немедленно поместите крышку трубки непосредственно на кусок сухого льда, чтобы заморозить образец и предотвратить деградацию РНК.
- Загрузите больше трубок, верните блок труб на сцену и вырежьте больше образцов. Добавьте различную смесь грунтовки CEL-Seq2 к каждому кончику хвоста.
- Хранить все тубы при температуре -70 °C.
4. Секвенирование однохвостой РНК с помощью CEL-Seq2
ПРИМЕЧАНИЕ: Для получения полной информации о протоколе CEL-Seq2 см. Yanai и Hashimshony18.
- Очистите область лабораторного стенда раствором для обеззараживания РНКазы, чтобы предотвратить деградацию РНК.
- Готовьте мастер-миксы и держите их на льду.
- Приготовьте мастер-микс с обратной транскрипцией: 0,4 мкл буфера первой нити, 0,1 мкл 0,1 М DTT, 0,1 мкл ингибитора РНКазы и 0,1 мкл обратной транскриптазы на образец.
- Готовят вторую цепь реакционной мастер-смеси: 7 мкл воды, 2,31 мкл буфера второй цепи, 0,23 мкл dNTP, 0,08 мкл E. coli лигазы, 0,3 мкл ДНК-полимеразы E. coli , 0,08 мкл RNaseH на образец.
- Вскрытие ячеек и отжиг грунтовками (полный список грунтовок см. в Дополнительной таблице S1 ):
- Запрограммируйте термоциклер и его крышку на 65 °C.
- Извлеките образцы при температуре -70 °C и инкубируйте их в термоциклере в течение 2,5 мин.
- Отжим при 21 000 × г в течение 30-40 с.
- Инкубировать при 65 °C в течение 2,5 мин.
- Немедленно переместите их на лед.
- Вращайтесь при 21 000 × г в течение 30-40 с и возвращайте их в лед.
- Преобразование РНК в кДНК:
- Добавьте 0,8 мкл смеси обратной транскрипции к каждому кончику хвоста.
- Инкубировать при 42 °C в течение 1 ч.
- Нагревать-инактивировать при 70 °C в течение 10 мин.
- Сразу же переместите его на лед.
- Добавьте 10 мкл смеси второй нити к каждому кончику хвоста.
- Проведите пальцем по образцам.
- Отжим при 21 000 × г в течение 30-40 с.
- Инкубировать при 16 °C в течение 2 ч.
- Очистка кДНК:
- Предварительно прогрейте БУСИНЫ ДЛЯ ОЧИСТКИ ДНК до комнатной температуры.
- Соберите до 40 образцов в центрифужной трубке объемом 1,5 мл (до 480 мкл).
- Перемешайте шарики до тех пор, пока они не станут хорошо диспергированными, и добавьте 20 мкл бусин и 100 мкл буфера связывания бисера на каждые 100 мкл объединенного образца (на 480 мкл образца добавьте 480 мкл бусинового буфера и 96 мкл бусин к конечному объему до 1056 мкл). Хорошо перемешать путем пипетки.
- Инкубировать при комнатной температуре в течение 15 мин.
- Поместите на магнитную подставку не менее 5 минут, пока жидкость не станет прозрачной.
- Удалите и выбросьте все, кроме 20 мкл супернатанта.
- Добавьте 200 мкл свежеприготовленного 80% этанола.
- Инкубируйте не менее 30 с, удалите супернатант, пипетируя его, не нарушая бусины. Выбросьте супернатант.
- Повторите шаги 4.5.7 и 4.5.8 один раз.
- Высушите бусины на воздухе в течение 15 минут или до полного высыхания.
- Повторно суспендируйте бусины (~6,4 мкл) 6,4 мкл воды. Тщательно перемешайте, пипетируя весь объем вверх и вниз десять раз.
- Инкубировать при комнатной температуре в течение 2 мин.
- Переходите прямо к транскрипции in vitro (IVT).
- Транскрипция и фрагментация in vitro:
- К пробирке, содержащей 6,4 мкл образца и шариков, добавляют следующую смесь (всего 9,6 мкл): 1,6 мкл 10x T7 Buffer, 1,6 мкл АТФ, 1,6 мкл UTP, 1,6 мкл CTP и 1,6 мкл GTP (каждый dNTP при концентрации 75 мМ) 1,6 мкЛоф фермента T7.
- Инкубировать в течение 13 ч при 37 °C с трюмом 4 °C.
- Добавьте 6 мкл раствора экзонуклеазы (конечный объем должен быть 22 мкл).
- Инкубировать в течение 15 мин при 37 °C.
- Поместите трубку обратно на лед и добавьте 5,5 мкл буфера фрагментации (0,25 × реакционного объема).
- Инкубировать в течение 3 мин при 94 °C.
- Немедленно переместите трубку в лед и добавьте 2,75 мкл буфера остановки фрагментации (0,5 × добавлен объем буфера фрагментации).
- Снимите шарики, поместив трубку на магнитную подставку не менее чем на 5 минут, пока жидкость не станет прозрачной.
- Перенесите супернатант в новую трубку.
- Очистка амплифицированной РНК (аРНК):
- Предварительно прогрейте шарики РНК до комнатной температуры.
- Перемешайте бусины до тех пор, пока они не станут хорошо диспергированными.
- Пипетка 55 мкл шариков (1,8 × реакционного объема).
- Инкубировать их при комнатной температуре в течение 10 мин.
- Поместите трубку на магнитную подставку не менее чем на 5 минут, пока жидкость не станет прозрачной.
- Удалите и выбросьте 80 мкл надосадочного вещества.
- Добавить 200 мкл свежеприготовленного 70% этанола.
- Насиживайте не менее 30 с, удаляйте супернатант путем пипетки, не нарушая бусины. Выбросьте супернатант.
- Повторите промывку этанолом еще два раза.
- Высушите бусины на воздухе в течение 15 минут или до полного высыхания.
- Повторно суспендируйте бусины 7 мкл воды. Пипетка всей громкости вверх и вниз 10 раз для тщательного перемешивания.
- Инкубировать при комнатной температуре в течение 2 мин.
- Поместите трубку с шариками на магнитную подставку на 5 минут, пока жидкость не станет прозрачной.
- Перенесите супернатант в новую трубку.
ПРИМЕЧАНИЕ: Точка остановки: образцы могут храниться при температуре -70 °C.
- Необязательно: Проверьте количество и качество аРНК с помощью автоматизированной системы электрофореза в соответствии с протоколом производителя.
- Подготовка библиотеки:
- К 5 мкл РНК добавляют 1 мкл 100 мкМ случайного гексамерного праймера RT (см. Таблицу материалов) и 0,5 мкл 10 мМ dNTP.
- Инкубировать при 65 °C в течение 5 мин.
- Добавьте 4 мкл следующей смеси при комнатной температуре: 2 мкл буфера первой нити, 1 мкл 0,1 МДДТ, 0,5 мкл ингибитора РНКАЗЫ, 0,5 мкл обратной транскриптазы.
- Инкубировать при 25 °C в течение 10 мин.
- Инкубировать при 42 °C в течение 1 ч (в гибридизационной печи или предварительно нагретом термоциклере с крышкой, установленной на 50 °C).
- Инкубировать при 70 °C в течение 10 мин.
- Переложить 5 мкл в новую пробирку (оставить остальную часть реакции при -20 °C). Добавьте 5,5 мкл сверхчистой воды, 1 мкл РНК ПЦР Праймер (RP1), 1 мкл индексированной РНК ПЦР Праймер (RPIX) и 12,5 мкл смеси ПЦР.
- Используйте следующую программу на термоциклере: 30 с при 98 °C, 11 циклов: (10 с при 98 °C, 30 с при 60 °C, 30 с при 72 °C), 10 мин при 72 °C, держите при 4 °C.
ПРИМЕЧАНИЕ: Температура остановки: образцы могут храниться при температуре -20 °C.
- Очистка библиотеки:
- Предварительно прогрейте БУСИНЫ ДЛЯ ОЧИСТКИ ДНК до комнатной температуры.
- Перемешайте бусины до тех пор, пока они не станут хорошо диспергированными.
- Добавьте 25 мкл шариков в реакцию ПЦР. Хорошо перемешать путем пипетки.
- Инкубировать при комнатной температуре в течение 15 мин.
- Поместите трубку на магнитную подставку не менее чем на 5 минут, пока жидкость не станет прозрачной.
- Удалите и выбросьте 45 мкл супернатанта.
- Добавьте 200 мкл свежеприготовленного 80% этанола.
- Насиживайте не менее 30 с, удаляйте и выбрасывайте супернатант, не нарушая бусины.
- Повторите промывку этанолом один раз.
- Бусины высушиваются на воздухе в течение 15 мин или до полного высыхания.
- Повторно суспендируйте их 25 мкл воды. Хорошо перемешать путем пипетки.
- Инкубировать при комнатной температуре в течение 2 мин.
- Поместите трубку на магнитную подставку на 5 минут, пока жидкость не станет прозрачной.
- Перенесите 25 мкл супернатанта в новую трубку.
- Повторите шаги 4.10.2-4.10.10 один раз.
- Повторное суспендирование с 10,5 мкл воды. Хорошо перемешать путем пипетки.
- Инкубировать при комнатной температуре в течение 2 мин.
- Поместите трубку на магнитную подставку на 5 минут, пока жидкость не станет прозрачной.
- Переложите 10 мкл супернатанта в новую трубку и храните при -20 °C.
- Оцените качество и количество библиотеки в соответствии с требованиями средства секвенирования.
Результаты
После лазерного захвата микродиссекции отдельные кончики хвоста самцов и гермафродитов в 4 временных точках (L3 через 22 ч после вылупления; L4 24, 26 и 28 ч после вылупления) были подготовлены к секвенированию РНК с использованием протокола CEL-Seq2. Грунтовки CEL-Seq2 содержат уникальные штрих-коды, которые позволяют биоинформатически идентифицировать показания секвенирования из конкретного образца (в данном случае отдельного кончика хвоста). Данные секвенирования были сгенерированы с помощью этого метода в общей сложности для 557 кончиков хвостов (266 гермафродитов и 291 самец в 4 временных точках развития, 59-78 хвостов на пол и точку времени). Штрих-коды CEL-Seq2 были восстановлены для 97% (т.е. 543) этих хвостовых кончиков (Дополнительная таблица S2). Для большинства библиотек скорость восстановления составляла 99-100%; тем не менее, это было 88% для одного мужского периода времени. Стоит отметить, что около половины мужских кончиков хвоста с 22, 24 и 28 часов хранились при -80 ° C в течение ~ 4 месяцев из-за задержек, связанных с COVID-19. Это показывает, что, хотя идеально подготовить библиотеки виртуализации вскоре после выборки, можно хранить препарированные образцы в течение более длительного времени до подготовки библиотеки.
Праймеры CEL-Seq2 также добавляют UMI к каждому транскрипту мРНК. Это позволяет удалять дубликаты ПЦР и точно определять экспрессию генов в образце. Число UMI резко варьировалось в зависимости от кончиков хвоста (рисунок 4; мужское среднее = 92 560; мужское мин. = 155; мужское макс. = 1 183 998; гермафродиты среднее = 67 597; гермафродиты мин. = 132; гермафродиты макс. = 630 427). Сведения о количестве UMI на кончик хвоста см. в дополнительной таблице S3. Из-за низкого количества входной РНК для подготовки одноклеточной библиотеки данные секвенирования одной клетки, как известно, имеют большое количество технического шума. Следовательно, рекомендуется фильтровать образцы, которые имеют очень низкое или очень высокое количество UMI перед анализом24.
Пакет R powsimR25 использовался для оценки требований к статистической мощности и размеру выборки для надежного обнаружения дифференциально экспрессированных (DE) генов в одноклеточных или объемных экспериментах RNA-seq. Параметры моделирования были основаны на наборе данных секвенирования из 70 отдельных мужских кончиков хвоста (в точке времени 24 часа), полученных с помощью метода, описанного здесь. Ожидаемые изменения логарифмических складок были основаны на результатах отдельного эксперимента RNA-seq, который объединил 80-100 кончиков хвоста. Моделирование определило, что данные с одним хвостом обладают достаточной мощностью (True Positive Rate = TPR) для обнаружения генов DE, за исключением генов, которые имеют очень низкое среднее значение экспрессии ( верхняя часть рисунка 5; пунктирная линия представляет 80% TPR). Добавление большего количества смоделированных кончиков хвоста в каждый момент времени несколько увеличило мощность для низко экспрессированных генов. Аналогичная картина наблюдается для коэффициента ложного обнаружения (FDR). FDR высок (>0,10) для низко экспрессированных генов; однако для более высоко экспрессированных генов он падает на уровне или ниже номинального отсечения 0,10 (пунктирная линия для FDR в нижней части рисунка 5). Таким образом, увеличение количества хвостов, отобранных за точку времени выше 70, мало что сделает для снижения FDR или увеличения мощности. Тем не менее, 70 хвостовых кончиков обеспечивают гораздо меньшую FDR и более сильную мощность, чем 30 кончиков хвоста.

Рисунок 1: Обзор процедуры синхронизации Caenorhabditis elegans с методом вылупления и лазерной микродиссекцией кончиков хвоста. Сокращения: L1-L4 = личиночные стадии с 1 по 4; PEN = полиэтиленнафталат; LMD = лазерная микродиссекция. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
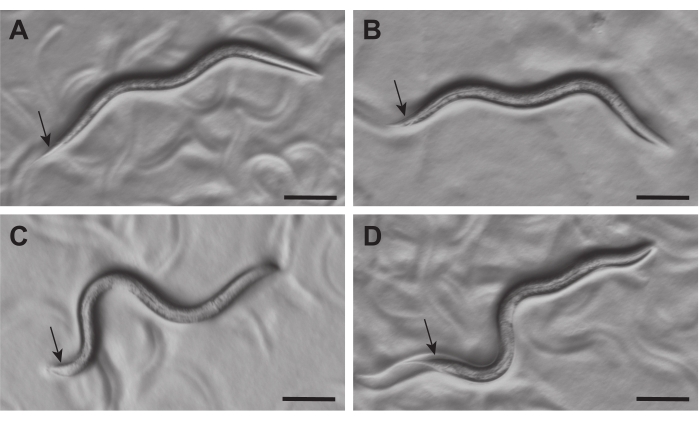
Рисунок 2: Появление гермафродитов C. elegans L3 и самцов под рассеченным микроскопом. Гермафродитов (A, B) и самцов (C, D) через 21-23 ч после вылупления можно отличить под рассеченным микроскопом (~50-кратное увеличение) по морфологии их хвостов (стрелок). Хвост гермафродитов узкий, в то время как хвост самцов опухший и кажется прозрачным. Шкала = 0,1 мм. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.

Рисунок 3: Внешний вид скользящей структуры мембраны PEN и хвоста червя. Фокусировка корректна для рассечения ткани, просматриваемой с помощью линзы 20x (A) и 40x (B) под микроскопом. (C) Рассечен хвост и частично вырезана пен-мембрана. После закрытия зазора в разрезе, кусок мембраны упадет в крышку трубки под затвором. (D) Колпачок трубки с мембранной секцией PEN, содержащей рассеченный хвостовой наконечник. Шкала стержней = 0,1 мм (A-C), 1 мм (D). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
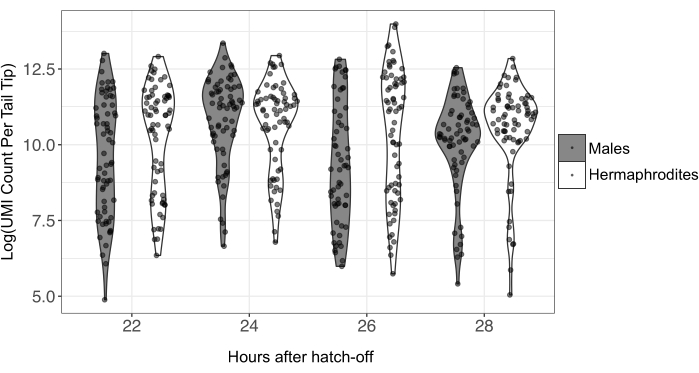
Рисунок 4: Естественный логарифмический трансформированный UMI на отдельный кончик хвоста для разных временных точек и полов. РНК из отдельных хвостов готовили к секвенированию методом CEL-Seq2; В общей сложности было секвенировано 557 хвостов, с 59-78 хвостами на пол и точку времени. Чрезвычайно низкие и высокие выбросы UMI будут удалены из данных перед анализом. Аббревиатура: UMI = уникальный молекулярный идентификатор. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.

Рисунок 5: Результаты анализа апостериорной мощности с использованием моделирования с помощью powsimR. Программное обеспечение powsimR определяет количество независимых образцов, необходимых для обнаружения генов DE на различных уровнях экспрессии. Гены бинируются средней экспрессией, преобразованной как естественный журнал подсчетов UMI. (A) Мощность (TPR) для обнаружения генов DE между двумя состояниями (здесь мужской и гермафродит) для четырех различных симуляций (разных цветных графиков), включающих различные размеры выборки (количество отдельных хвостовых кончиков) для каждого условия. Пунктирная линия указывает на 80% TPR. (B) FDR в тех же четырех симуляциях, что и в (A), пунктирная линия, указывающая на 10% FDR. Графики показывают, что размер выборки в 70 кончиков хвоста (зеленый) на условие достаточен для обнаружения генов DE, за исключением генов с очень низким уровнем экспрессии. То есть, мощность и частота ложных обнаружений для таких генов не могут быть значительно улучшены путем увеличения размера выборки более 70. Сокращения: DE = дифференциально выраженные; UMI = уникальный молекулярный идентификатор; TPR = истинный положительный показатель; FDR = коэффициент ложного обнаружения. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Дополнительная таблица S1: Последовательности праймеров, используемых в протоколе CEL-Seq2. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительная таблица S2: Выборка в сравнении с восстановленными отдельными кончиками хвоста. Грунтовки CEL-Seq2 содержат уникальные штрих-коды, которые позволяют идентифицировать показания секвенирования из каждого образца для биоинформатической идентификации. Показания были извлечены из 97% образцов хвостового кончика, подготовленных для секвенирования. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительная таблица S3: UMI подсчитывает все образцы с восстановленными штрих-кодами, как указано в дополнительной таблице S2. Аббревиатура: UMI = уникальный молекулярный идентификатор. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Обсуждение
Критические этапы метода
При правильном выполнении способ, описанный здесь, позволит получить надежные профили РНК с относительно небольшим количеством образцов, рассеченных лазером (70 хвостовых кончиков в этом примере). Однако для образцов от развивающихся животных жесткая синхронизация имеет решающее значение для уменьшения изменчивости между образцами. По этой причине протокол рекомендует метод вылупления для синхронизации червей. Здесь исследователь может определить и точно контролировать разницу в возрасте между людьми (1 ч в настоящем протоколе). Кроме того, метод вылупления применим к любым видам, даже если эмбрионы чувствительны к отбеливанию, L1 не останавливается, или восстановление после остановки L1 является переменным. Для успешной синхронизации путем вылупления решающее значение имеют этапы промывки: все взрослые особи и личинки должны быть удалены в начале периода вылупления, и никакие эмбрионы не должны смываться вместе с только что вылупившимся L1 в конце периода вылупления. Это удается только в том случае, если агаровая поверхность пластины не повреждена трещинами, отверстиями или пузырьками, бактериальный газон на тарелке свежий и не слишком густой, а жидкость добавляется и перемешивается только очень осторожно.
Если данные должны быть получены отдельно по мужчинам и гермафродитам/самкам, то также важна надежная идентификация полов. Различение личинок L3 по полу (см. Рисунок 2) требует опыта. Рекомендуется практиковать отбор самцов L3 и гермафродитов / самок и проверять вероятность успеха после того, как животные превратились во взрослых особей и полы легко различимы. После одноткановой РНК-Seq выбросы также могут быть идентифицированы методом анализа основных компонентов и удалены, при необходимости.
Для успешного восстановления образцов лазерной резки важно максимально снизить статическое электричество. Заряженные части PEN-мембраны часто не опускаются в крышку трубки, а прилипают к затвору или любой другой части микроскопа. Одним из средств является повышение влажности в комнате и, в частности, вокруг микроскопа, путем размещения небольшого увлажнителя рядом со сценой. Кроме того, мембранные слайды могут быть обработаны ультрафиолетовым светом. Для этого инкубируют слайды в поперечной камере UV-C (254 нм) и подают не менее 1 джоуля энергии или подвергают слайды воздействию ультрафиолетового света в ламинарной скамье воздушного потока в течение 30 мин.
Поскольку целью протокола является RNA-Seq, поддержание рабочей среды, свободной от РНКазы, имеет решающее значение. Начиная с фиксирующего раствора, реагенты, контейнеры и расходные материалы должны быть без РНКазы, рабочая поверхность должна быть обеззаражена, а исследователи должны носить чистые перчатки. Рассеченные образцы должны быть заморожены как можно скорее и сохранены при температуре -70 °C до дальнейшей обработки. Также рекомендуется использовать трубки с низким уровнем удержания и наконечники для части протокола CEL-Seq2.
В настоящей статье представлен лишь базовый обзор протокола CEL-Seq2, который ранее был опубликован его разработчиками с полезными примечаниями и советами 17,18. Рекомендуется ознакомиться с этими публикациями перед использованием метода CEL-Seq2.
Данные LMD-RNA-Seq могут быть подтверждены флуоресцентной гибридизацией in-situ (smRNA FISH)26,27,28. smRNA FISH широко используется у C. elegans и поддается другим видам нематод, в отличие от иммуноокрашения существующими антителами (которые могут не перекрестно реагировать) или введения транскрипционных репортеров через трансгенез. Последний хорошо работает у C. elegans и некоторых родственных видов Caenorhabditis 29, но трансгенез может быть более сложным у других видов нематод30,31.
Ограничения метода
Описанный здесь метод очень хорошо работает для сбора кончиков хвоста, тонкой ткани на конце червя. Рассечение тканей в более толстой середине старых личинок или взрослых особей является более сложной задачей. Программное обеспечение инструмента, используемого здесь, включает в себя настройку для множественных разрезов, размещенных на последующих более глубоких уровнях в ткани. Эта настройка может быть использована для резки более толстых участков животного. Поскольку черви должны быть зафиксированы перед рассечением, структурные детали трудно увидеть, что препятствует точному рассечению конкретных небольших структур. Как упоминалось выше, LMD-RNA-Seq не является методом HTP. Тем не менее, 50-70 образцов могут быть вскрыты за один день.
Значение метода по отношению к существующим/альтернативным методам
LMD-RNA-Seq может использоваться у любых видов, даже если нет трансгенных инструментов. Другие методы основаны на сортировке FACS флуоресцентно меченых клеток 8,9,32 или выделении меченых ядер33,34 и, таким образом, требуют трансгенных животных. Методы, которые диссоциируют и изолируют клетки у постэмбриональных C. elegans, как правило, пропускают ткани на двух концах червя (Дилан Рахе, личное общение). Эти предостережения преодолеваются путем объединения одноклеточной РНК-Seq с криосекцией целых червей (РНК-томография)35. Этот метод был использован для сравнения пространственной экспрессии генов между C. elegans и другой рабдитидной нематодой, Pristionchus pacificus36. В качестве альтернативы можно экспериментировать с формалин-фиксированными парафин-внедренными (FFPE) червями. Такой материал был успешно использован для RNA-Seq после LMD образцов тканей млекопитающих37. Однако РНК-томография и ЛМД червей FFPE ограничены анализом лишь горстки животных. Поэтому они не так хорошо подходят для изучения динамической экспрессии генов в развивающихся тканях, как LMD-RNA-Seq.
Раскрытие информации
Все авторы заявляют, что у них нет конфликта интересов.
Благодарности
Эта работа финансировалась грантами NIH (R01GM141395) и NSF (1656736) для DF и стипендией NIH (F32GM136170) для AW. Рисунок 1 был создан с помощью BioRender.com.
Материалы
| Name | Company | Catalog Number | Comments |
| 0.5 µM PEN membrane glass slides RNase free | Leica | 11600288 | for LMD |
| 500 µL PCR tubes (nuclease-free) | Axygen | 732-0675 | to cut the tail tips into |
| Compound microscope with 40x objective and DIC | any | to check age of worms | |
| Desktop humidifier | any | ||
| Dissection microscope with transmitted light base | any | for all worm work | |
| glass pasteur pipets | any | handle of worm pick | |
| glass slides and coverslips | any | to check age of worms | |
| LMD6 microdissection system | Leica | multiple | to cut tail tips |
| LoBind tubes 0.5 mL | Eppendorf | 22431005 | |
| M9 Buffer | Recipe in WormBook | ||
| Methanol 99.8% | Sigma | 322415 | to fix worms |
| NGM growth medium | US Biological | N1000 | Buffers and salts need to be added: Recipe in WormBook |
| P10 pipette variablle volume | e.g. Gilson | ||
| P1000 pipette variable volume | e.g. Gilson | ||
| P2 pipette variable volume | e.g. Gilson | ||
| Pipette tips 1,000 µL | any | ||
| Pipette tips 1-10 µL filtered | any | ||
| platinum iridium wire | Tritech | PT-9010 | to make worm pick |
| sterile and nuclease-free 1 mL centrfuge tubes | any | ||
| Tween 20 | Sigma | P9416 | Add a very small amount to M9 buffer to prevent worms from sticking to the pipet tips |
| vented 6 mm plastic Petri dishes | any | ||
| For CEL-Seq2 | |||
| 4200 TapeStation System with reagents for high-sensitivity RNA and DNA detection | Aligent | automated electrophoresis system | |
| AMPure XP beads | Beckman Coulter | A63880 | DNA cleanup beads |
| Bead binding buffer 20% PEG8000, 2.5 M NaCl | |||
| CEL-Seq2 primers (see Table S1) | Sigma Genosys Mastercycler Nexus GX2 Eppendorf | 6335000020 | Thermal cycler with programmable lid and block for 200 µl tubes. |
| DNA Polymerase I (E. coli) | Invitrogen | 18052-025 | |
| dNTP mix 10 mM | any | ||
| E. coli DNA ligase | Invitrogen | 18052-019 | |
| Ethanol | |||
| ExoSAP-IT For PCR Product Clean-Up | Affymetrix | 78200 | exonuclease solution |
| MEGAscript T7 Transcription Kit | Ambion | AM1334 | For step 4.6.1 |
| Nuclease-free water | any | ||
| Phusion High-Fidelity PCR Master Mix with HF Buffer | NEB | M0531 | PCR mix step 4.9.7 |
| random hexamer RT primer GCCTTGGCACCCGAGAATTCCA NNNNNN | IDT | a primer with 6 nucleotides that are random | |
| RNA Fragmentation buffer | NEB | E6150S | |
| RNA Fragmentation stop buffer | NEB | E6150S | |
| RNA PCR Index Primers (RPI1–RPI48) | Illumina, NEB, or IDT | RPIX in protocol step 4.9.7, sequences available from Illumina | |
| RNAClean XP beads | Beckman Coulter | A63987 | |
| RNase AWAY Surface Decontaminant | Thermo Scientific | 7000TS1 | or any other similar product |
| RNaseH (E. coli) | Invitrogen | 18021-071 | |
| RNaseOUT Recombinant Ribonuclease Inhibitor | Invitrogen | 10777-019 | |
| Second strand buffer | Invitrogen | 10812-014 | |
| Superscripit II | Invitrogen | 18064-014 | reverse transcriptase |
Ссылки
- Haag, E. S., Fitch, D. H. A., Delattre, M. From "the worm" to "the worms" and back again: the evolutionary developmental biology of nematodes. Genetics. 210 (2), 397-433 (2018).
- Sommer, R. J., Bumbarger, D. J. Nematode model systems in evolution and development. Wiley Interdisciplinary Reviews: Developmental Biology. 1 (3), 389-400 (2012).
- . WormBase Available from: https://wormbase.org/#012-3-6 (2022)
- . WormAtlas Available from: https://wormatlas.org (2022)
- . WormBook in Genetics Available from: https://academic-oup-com.remotexs.ntu.edu.sg/genetics/pages/wormbook (2022)
- Cao, J., et al. Comprehensive single-cell transcriptional profiling of a multicellular organism. Science. 357 (6352), 661-667 (2017).
- Kaletsky, R., Murphy, C. T. Transcriptional profiling of C. elegans adult cells and tissues with age. Methods in Molecular Biology. 2144, 177-186 (2020).
- Kaletsky, R., et al. Transcriptome analysis of adult Caenorhabditis elegans cells reveals tissue-specific gene and isoform expression. PLoS Genetics. 14 (8), 1007559 (2018).
- Packer, J. S., et al. A lineage-resolved molecular atlas of C. elegans embryogenesis at single-cell resolution. Science. 365 (6459), 1971 (2019).
- Kulkarni, A., Anderson, A. G., Merullo, D. P., Konopka, G. Beyond bulk: a review of single cell transcriptomics methodologies and applications. Current Opinion in Biotechnology. 58, 129-136 (2019).
- Von Stetina, S. E., et al. Cell-specific microarray profiling experiments reveal a comprehensive picture of gene expression in the C. elegans nervous system. Genome Biology. 8 (7), 135 (2007).
- Baugh, L. R., et al. The homeodomain protein PAL-1 specifies a lineage-specific regulatory network in the C. elegans embryo. Development. 132 (8), 1843-1854 (2005).
- Haenni, S., et al. Analysis of C. elegans intestinal gene expression and polyadenylation by fluorescence-activated nuclei sorting and 3'-end-seq. Nucleic Acids Research. 40 (13), 6304-6318 (2012).
- Zhang, S., Banerjee, D., Kuhn, J. R. Isolation and culture of larval cells from C. elegans. PLoS One. 6 (4), 19505 (2011).
- Emmert-Buck, M. R., et al. Laser capture microdissection. Science. 274 (5289), 998-1001 (1996).
- Hashimshony, T., et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biology. 17, 77 (2016).
- Yanai, I., Hashimshony, T. CEL-Seq2-single-cell RNA sequencing by multiplexed linear amplification. Methods in Molecular Biology. 1979, 45-56 (2019).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Ceron, J. Basic Caenorhabditis elegans methods: synchronization and observation. Journal of Visualized Experiments: JoVE. (64), e4019 (2012).
- Baugh, L. R. To grow or not to grow: nutritional control of development during Caenorhabditis elegans L1 arrest. Genetics. 194 (3), 539-555 (2013).
- Baugh, L. R., Hu, P. J. Starvation responses throughout the Caenorhabditis elegans life cycle. Genetics. 216 (4), 837-878 (2020).
- Pepper, A. S., Killian, D. J., Hubbard, E. J. Genetic analysis of Caenorhabditis elegans glp-1 mutants suggests receptor interaction or competition. Genetics. 163 (1), 115-132 (2003).
- Mok, D. Z., Sternberg, P. W., Inoue, T. Morphologically defined sub-stages of C. elegans vulval development in the fourth larval stage. BMC Developmental Biology. 15, 26 (2015).
- Lytal, N., Ran, D., An, L. Normalization methods on single-cell RNA-seq data: an empirical survey. Frontiers in Genetics. 11, 41 (2020).
- Vieth, B., Ziegenhain, C., Parekh, S., Enard, W., Hellmann, I. powsimR: power analysis for bulk and single cell RNA-seq experiments. Bioinformatics. 33 (21), 3486-3488 (2017).
- Bolkova, J., Lanctot, C. Quantitative gene expression analysis in Caenorhabditis elegans using single molecule RNA FISH. Methods. 98, 42-49 (2016).
- Ji, N., van Oudenaarden, A. Single molecule fluorescent in situ hybridization (smFISH) of C. elegans worms and embryos. WormBook. , 1-16 (2012).
- Lee, C., et al. Single-molecule RNA fluorescence in situ hybridization (smFISH) in Caenorhabditis elegans. Bio-Protocol. 7 (12), 2357 (2017).
- Baird, S. E., Chamberlin, H. M. Caenorhabditis briggsae methods. WormBook. , 1-9 (2006).
- Felix, M. A. Oscheius tipulae. WormBook. , 1-8 (2006).
- Han, Z., et al. Improving transgenesis efficiency and CRISPR-associated tools through codon optimization and native intron addition in Pristionchus nematodes. Genetics. 216 (4), 947-956 (2020).
- Fernandes Povoa, E. E., Ebbing, A. L. P., Betist, M. C., vander Veen, C., Korswagen, H. C. An optimized dissociation protocol for FACS-based isolation of rare cell types from Caenorhabditis elegans L1 larvae. MethodsX. 7, 100922 (2020).
- Han, M., Wei, G., McManus, C. E., Hillier, L. W., Reinke, V. Isolated C. elegans germ nuclei exhibit distinct genomic profiles of histone modification and gene expression. BMC Genomics. 20 (1), 500 (2019).
- Steiner, F. A., Henikoff, S. Cell type-specific affinity purification of nuclei for chromatin profiling in whole animals. Methods in Molecular Biology. 1228, 3-14 (2015).
- Ebbing, A. Spatial transcriptomics of C. elegans males and hermaphrodites identifies sex-specific differences in gene expression patterns. Developmental Cell. 47 (6), 801-813 (2018).
- Rödelsperger, C., et al. Spatial transcriptomics of nematodes identifies sperm cells as a source of genomic novelty and rapid evolution. Molecular Biology and Evolution. 38 (1), 229-243 (2021).
- Amini, P., et al. An optimised protocol for isolation of RNA from small sections of laser-capture microdissected FFPE tissue amenable for next-generation sequencing. BMC Molecular Biology. 18 (1), 22 (2017).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены