Method Article
種にとらわれない単一組織アプリケーションのためのレーザーマイクロダイセクション
要約
レーザーマイクロダイセクションを使用してRNAシーケンシングのために個々の線虫組織を単離するプロトコルが記載されている。このプロトコルは、種固有の遺伝子ツールキットを必要とせず、単一組織サンプルのレベルで異なる種間で遺伝子発現プロファイルを比較することを可能にする。
要約
単一細胞方法論は、特定の細胞型のトランスクリプトームの解析に革命をもたらしました。しかし、それらはしばしば、蛍光タンパク質の組織特異的発現を駆動するプロモーターなどの種特異的な遺伝的「ツールキット」を必要とする。さらに、個々の細胞を単離するために組織を破壊するプロトコルは、細胞をそれらの天然環境から除去し(例えば、隣人からのシグナル伝達)、ストレス応答または他の天然遺伝子発現状態からの他の相違をもたらし得る。本プロトコールにおいて、レーザーマイクロダイセクション(LMD)は、雄尾先端形態形成中の遺伝子発現の研究のために個々の線虫尾先端を単離するように最適化されている。
LMDは、細胞破壊または種固有のツールキットを必要とせずに動物の一部を単離することを可能にし、したがってあらゆる種に適用可能である。その後、CEL-Seq2などの単一細胞RNA-seqライブラリー調製プロトコルをLMD単離単一組織に適用し、適切に注釈付きのゲノムまたはトランスクリプトームが種に利用可能であることを考えると、標準的なパイプラインを使用して分析することができます。このようなデータは、異なる種におけるその組織の発達の根底にあるトランスクリプトームがどれほど保存されているか、または異なっているかを確立するために使用することができる。
制限には、目的の組織を切り出す能力とサンプルサイズが含まれます。検出力解析では、80%の検出力に必要なテールチップは条件ごとにわずか70個であることが示されています。同じ発達段階でこの数の動物を得るためには、発達の緊密な同期が必要です。したがって、1時間間隔で動物を同期させる方法も記載されている。
概要
線虫、特にモデル系Caenorhabditis elegansに関連する横紋線虫は、多くの理由で進化発生生物学(EDB)のための動物の素晴らしいグループです1,2。利点には、少数の細胞、定義済みで一貫した細胞系譜、透明性、および培養および飼育の容易さが含まれる。また、複数の種のための高品質のゲノム、およびC. elegansのための、広範な分子遺伝学的ツールおよび発生、遺伝学、解剖学、および生理学に関する知識を含む多くの利用可能なリソースがあります3,4,5,6。
他の多くの生物と同様に、単一組織または単一細胞におけるトランスクリプトームダイナミクスを特徴付ける能力は、C. elegans 7,8,9,10における発生の分析に革命をもたらしました。線虫間で単一細胞トランスクリプトームを比較できれば、これらの生物を用いてEDBを同様に変換するであろう。例えば、このような比較は、保存された文字(形質)、発散した文字、または独立して進化した文字について、遺伝子調節ネットワークがどのように進化したかについての洞察を提供するであろう。
しかし、線虫から特定の組織または細胞を単離することは、大きな課題の1つです。多くの生物にとって、単一細胞を組織から解離させて公平な方法で採取するか、蛍光タンパク質の組織特異的発現で標識し、蛍光活性化細胞選別(FACS)によって選別することができる11。C. elegansでは、丈夫な外側のキューティクル(および静水圧骨格)が幼虫および成虫からの細胞分離を妨げているため、細胞のハイスループット(HTP)単離は主に胚に限定されてきた。この課題を回避するために、いくつかの方法は、組織特異的mRNAタグ付け12、および細胞型13に影響を及ぼす野生型と変異体との間の差次的発現比較など、C. elegansワーム全体における遺伝的ツールを採用している。より最近の方法は、キューティクルを溶解して核14または細胞全体を単離することによって課題を克服している8、9、15。しかし、細胞単離および細胞培養には明らかな欠点があり、細胞が自然な発生的または解剖学的文脈から(例えば、細胞間シグナル伝達および細胞外マトリックスとの接触から離れるなど)除去され、遺伝子発現プロファイルに影響を与えることが予想される15。さらに、遺伝的ツールおよび組織特異的マーカーは種特異的である(すなわち、それらはC. elegansにおいてのみ使用することができる)。
LMDは、細胞の自然な文脈を破壊することなく組織を単離するための代替方法を提供する。EDBにとって有意に、LMDはまた、これらの種のゲノムまたはトランスクリプトーム配列全体が利用可能な場合、種固有の遺伝的ツールキットを必要とせずに、異なる種の相同組織からのトランスクリプトームを比較することを可能にする。LMDは、直接顕微鏡観察によって組織を標的化し、顕微鏡の光学系に統合されたレーザーマイクロビームを使用して、関心組織を切り出して採取(捕捉)することを含む16。LMDの限界は、それが非常にHTPアプローチに役立たないということです (ただし、テールチップの転写プロファイル, このプロトコルに記載されているように, 〜で堅牢でした 70 サンプル), 特定のサンプルを解剖するのが難しいかもしれません, そして、カットは、レーザーの精度と顕微鏡で視覚化することができるものに制限されています.
本プロトコールの目的は、LMD、それに続く単一組織RNA-Seqが、線虫から段階的および組織特異的トランスクリプトームデータを取得するためにどのように使用できるかを記述することである。具体的には、 C. elegansの第4段階幼虫(L4)から尾先を単離するためのLMDを実証する。しかしながら、この方法は、他の組織およびもちろん、異なる種に適合させることができる。
C. elegansでは、男性と雌雄同体の両方で尾の先端を作る4つの細胞があります。男性のL4段階では、雌雄同体ではそうではないが、尾先細胞は形を変え、前方および内側に移動する。このプロセスは、他の一部のラブディチッド線虫種でも起こるが、すべてではない。したがって、尾の先端は性的二型形態形成の進化のための良いモデルである。その位置のために、尾先端もLMDによって分離することが容易である。
テールチップからトランスクリプトームプロファイルを得るために、本プロトコルは、単一細胞のために開発されたRNA-seq法であるCEL-Seq2を使用する17、18。この方法は、LMD由来組織に対していくつかの利点を有する。CEL-Seq2は、mRNA読み取りの簡単な定量を可能にする一意の分子識別子(UMI)、線形増幅を保証する ためのインビトロ 転写、および個々の組織サンプルの多重化を可能にするバーコードを使用して、非常に高感度で効率的です。CEL-Seq2の唯一の制限は、回収された読み取りがmRNAの3'末端に偏っており、したがってほとんどのアイソフォームが区別できないことです。
プロトコル
1. ワームの同期
注: C. elegans と他のラブディチッド種の発達を同期させるために、2つの方法を以下に説明します。
- アルカリ性次亜塩素酸塩(漂白剤)処理後の最初の幼虫期(L1)停止によって同期する。
注: このメソッドは、前に詳細19 で説明しました。この方法は、他のいくつかの横紋岩種にも当てはまるC. elegansの2つの特徴に依存している:(1)卵殻は漂白剤に耐性があるが、成虫および幼虫のワームを囲むキューティクルはそうではない。- 重質雌雄同体(または雌)を希釈漂白剤溶液で処理して、キューティクルを分解して胚を放出する。
- 漂白剤から胚を取り出し、すべてのL1が孵化するまで食物なしでそれらを保管する。
- 逮捕されたL1を食物の上に置き、そこで全員がほぼ同時に発達を再開する。
メモ: L1 停止からの出口は 1 時間以内に発生することがあります。
- 「ハッチオフ」方式(ここで使用; 図1上 ):
注: ハッチオフ方式では、開発を中断することなく緊密な同期が可能になります (L1 停止は、後のステージ21 の発達にも影響します)。このプロトコルは、Pepperら22から適応されている。この方法の目的は、胚のみを含むプレートから特定の期間にわたって孵化したL1を収集することである。- 母親を選ぶ: ハッチオフを行う前の夕方に、 大腸菌 OP50を播種したプレート上に〜30個の重力雌雄同体をピックします。
- 一晩産卵のために25°Cで孵化させる。
メモ:ワームが立ち往生する可能性のある亀裂や泡のないプレートを選択してください。後ですべてのワームを取り除くのは難しいので、非常に厚い細菌芝生のプレートは避けてください。温度感受性株で作業する場合は、より長い胚発生を説明するために産卵時間を調整してください。最大の産卵段階で母親を選ぶ。 - 母親と幼虫を取り除く: 翌朝、解剖顕微鏡(倍率20倍)の下で、噴出せずにプレートの壁に対してM9バッファー1〜2mLを穏やかにピペットでピペットする。プレートを渦巻いてワームを取り除きます。
- 穴を突かないように、ピペットチップを寒天の端にあるプレートの壁に押し付けて、すべての液体とワームを取り除いて捨てます。プレート上にワーム(および卵/胚のみ)が残っていないこと、特にL1が残っていないことを確認してください。それ以外の場合は、洗浄を繰り返します。
- プレートを25°Cで1時間置き、L1がハッチングするのを待ちます。
- 新しく孵化したL1を集める:1mLのM9バッファーを寒天に慎重に落とします。プレートを渦巻いてL1を取り除きますが、胚は取り除きません。緩衝液およびワームを1mLの遠沈管に穏やかにピペットで入れる。
- チューブを〜18,000 × gで1分間遠心分離 する。上清を除去する。
- ピペットL1を播種したプレートの細菌芝生に直接塗布する。解剖顕微鏡で成虫や胚が存在しないことを確認します。
- ワームは、目的の段階まで発達するまで25°Cに保ちます。
注:条件が最適であれば、同じプレートからさらに2つのバッチのL1を収集できます。初期プレートを調べて、L1が存在しないことを確認します。必要に応じて、もう一度洗ってください。手順 1.2.5 ~ 1.2.9 を繰り返します。 -
発達のタイミングを確認してください。 下流のアプリケーションに進む前に、400倍の倍率で化合物顕微鏡下でいくつかのワームを検査し、それらが所望の発生段階(ここではL3)に達したことを確認する。
注:外陰部の発生に加えて、遠位先端細胞またはリンカー細胞の遊走距離をガイドとして使用することができる。外陰部の発生については、Mockら23が有用なガイドを提供しているが、その研究のタイミングは20 °Cで決定された。 25°Cでは、野生型 C.エレガンスは 孵化後24時間でL3-L4脱皮を受ける。
2. L4オスと雌雄同体と固着の収集
- 固定前にRNAseフリー、コールド(-20°C)、70%メタノールを調製する。
- 30〜50倍の倍率の解剖顕微鏡下で、性別が区別できるようになったらすぐに(孵化後〜21時間、 図2)、1〜2時間または200匹の動物が収集されるまで、同期プレートから別々の播種されていないプレートに雄雄および雌雄同体をピッキングし始める。
- ワームを実験の所望の段階に達するまで25°Cに保ちます。
- 0.01%洗剤を含むM9バッファーで予備洗浄したピペットチップを使用して、1〜2 mLのM9バッファーでプレートからワームを洗い流します(ワームがチップに付着するのを防ぐため)。
- ワームを1mLの遠沈管に移す。
- 21,000 × g で1分間回転させてワームをペレット化します。上清を除去する。
- 1 mLのM9バッファーを加え、混合してペレットを分解します。
- 21,000 × g で1分間回転させてワームをペレット化します。上清を除去する。
- 洗浄を繰り返します。
- 氷冷70%メタノール1mLを加え、よく混ぜる。
- 21,000 × g で1分間回転させてワームをペレット化します。上清を除去する。
- 手順 2.10 と 2.11 を繰り返します。
- 70%メタノール500 μLを加え、混合し、4°Cで1時間~一晩保存する。
3. レーザーマイクロダイセクション
注:ここからは、RNaseフリーの試薬と消耗品を使用してください。フィルターのヒントを使用します。
- サンプルの処理にCEL-Seq2法を使用する場合は、各CEL-Seq2プライマー(補足表S1)のマスターミックスを調製します:ピペット2 μLのCEL-Seq2プライマー、10 mM dNTPの1 μL、および9 μLの1%β-メルカプトエタノール(RNaseフリー水中)を標識200 μLチューブに入れます。
- スライドへの取り付け
- 解剖顕微鏡下で、固定されたワーム(ステップ2.13から20〜40個のワーム)をポリエチレンナフタレート(PEN)膜スライドガラスのマット側(膜がある場所)に20μLのピペットで送ります。
- メタノールが蒸発するのを待ちます。スライドウォーマーを使用して蒸発をスピードアップします。
注:追加のメタノール滴を適用することができ、乾燥時にワームが凝集し始めた場合にワームを広げるために使用されるピペットチップ。ワームが塊になっているとき、それらは解剖するのが難しいかもしれません。
- 顕微鏡のセットアップ
メモ:以下のプロトコルは、 材料表に記載されている機器に固有のものです。別のLMD顕微鏡を使用する場合は、調整する必要があります。- LMD顕微鏡の側面にあるステージの後ろにデスクトップ加湿器を置きます。蒸気がステージに直接吹き込まれていることを確認します。
注:加湿器は静電気を減らすのに役立ち、そうでなければ小さな膜部分がチューブキャップに落ちるのを防ぐことができます。 - レーザー出力のキーを回します。
- ステージ制御の電源をオンにします。
- 顕微鏡コントロールボックスをオンにします。
- レーザーマイクロダイセクションソフトウェアを開きます。
- ステージ上のプラスチックシールドを取り外します。
- 上向き矢印の付いたアンロードボタンをクリックして、メンブレンスライドを ロード します。
- スライドが完全に乾いていることを確認し、メンブレンが下を向くように反転させます。
- スライドを挿入し、試料の変更ウィンドウで「続行」をクリックします。
- プラスチックシールドを交換してください。
- 画面の下部で、スライドを含むスライドホルダーを選択します。
- チューブをロードするには、下向き矢印の付いた アンロード ボタンをクリックします。
- トレイを引き出し、チューブブロックを取り外します。
注: この実験に使用したチューブブロックは、500 μL PCR チューブ用です。 - 500 μL PCR チューブのチューブキャップをホルダーに挿入し、チューブを下に折ります。
- ブロックをトレイに戻し、トレイを顕微鏡ステージに戻します。
- 変更コレクターデバイスのポップアップウィンドウで、PCR チューブを選択し、「OK」をクリックします。
- 画面左下の コレクターデバイスチューブキャップの下にある空のチューブ位置をクリックします。
- 顕微鏡コントロールパネルで、透過光明視野照明にTL-BFを選択します。
- LMD顕微鏡の側面にあるステージの後ろにデスクトップ加湿器を置きます。蒸気がステージに直接吹き込まれていることを確認します。
- 切断
メモ:このプロトコルは、材料表に記載されている機器に 固有のものです。- 2.5xレンズを使用して、ワームと膜の構造が見えるまで焦点を調整します。
- 20xレンズに切り替えます。
- ステージをワームのない領域に移動します。膜内の気泡のような構造が黄色がかった色(図3A、B)になるように焦点を調整して、レーザーを正しい焦点面に焦点を合わせます。
- レーザーパラメータを設定します。テールチップの場合は、 パワー 45、 絞り 30、 速度 20 から始めます。
- レーザーコントロールパネルで、キャリブレーションを選択します。指示に従ってください。
メモ:計測器はこの手順を自動的に実行します。これにより、画面上にマウスで描画された形状が、レーザーによって切り取られた形状と同一であることが保証されます。 - コレクターデバイスチューブキャップの画面下部で、位置Aをクリックします。
- 画面の右側で、 単一の図形|描画+カット。画面の左側で、[ PtoP] を選択します。
- 線を引きます。
- 「カット開始」をクリックして、レーザーが膜を切断します。
注:また、ガラスに線を刻むこともあります。 - このテストカットが良好に見える場合(膜が切断され、カットのエッジが滑らかに見える場合)、次のステップに進みます。それ以外の場合は、フォーカスを調整して別の線を切り取ります。
- ワームを見つけます。 [移動 + カット] に切り替え、マウスを使用して尾を切り取ります。
メモ:レーザーが尾部を貫通しない場合は、焦点を調整してレーザー出力を上げます。より厚い組織の場合、レーザー出力を60に設定する必要があるかもしれません。 - パラメータの保存: [ファイル] タブの|アプリケーション構成を保存します。後で取得するには、「 アプリケーション構成を復元」します。
- サンプルを収集するには、PtoP機能でドロー+カット設定に切り替え、形状を描画して膜断面のカットを完了します(図3C)。
メモ:大きな膜セクションと、円や楕円形ではなく長方形や三角形のような形をしたセクションは、コレクターチューブキャップ内で見つけやすいです。 - 画面下部の コレクタデバイスチューブキャップ で次のチューブを選択し、次のテールチップをカットします。
- 4つのテールが切断されたら、チューブラックをアンロードし(下向き矢印で アンロード をクリック)、解剖顕微鏡で膜セクションを見つけます(図3D)。
メモ:セクションは、チューブキャップの中央に配置するか、キャップの側面に貼り付けることができます。 - ダウンストリーム・アプリケーションを続行します。CEL-Seq2の場合、1.2 μLのCEL-Seq2プライマーマスターミックス(ステップ3.1から)をサンプルの上に直接ピペットで送ります。
- チューブを閉じ、プライマー番号でラベルを付け、すぐにチューブ キャップをドライアイスの上に直接置き、サンプルをフラッシュフリーズさせ、RNA の分解を防ぎます。
- より多くのチューブをロードし、チューブブロックをステージに戻し、より多くのサンプルを切断します。異なるCEL-Seq2プライマーミックスを各テールチップに加える。
- すべてのチューブを-70°Cで保管してください。
4. CEL-Seq2によるシングルテールRNAシーケンシング
注: CEL-Seq2 プロトコルの詳細については、Yanai および Hashimshony18 を参照してください。
- ラボベンチエリアをRNase除染液で清掃し、RNAの分解を防ぎます。
- マスターミックスを準備し、氷の上に保管してください。
- 逆転写マスターミックスを調製する:サンプルあたり、0.4 μLの第1鎖バッファー、0.1M DTT0.1 μL、RNase阻害剤0.1 μL、および逆転写酵素0.1 μL。
- 第2の鎖反応マスターミックスを調製する:7 μLの水、2.31 μLの第2の鎖緩衝液、0.23 μLのdNTP、0.08 μLの大腸菌リガーゼ、0.3 μLの大腸菌DNAポリメラーゼ、0.08 μLのRNaseH/サンプル。
- 連続気泡の破壊およびプライマーによるアニーリング(プライマーの完全なリストについては 、補足表S1 を参照):
- サーモサイクラーとその蓋を65°Cにプログラムします。
- -70°Cからサンプルを取り出し、サーモサイクラーで2.5分間インキュベートします。
- 21,000 × g で30〜40秒間回転します。
- 65°Cで2.5分間インキュベートする。
- すぐに氷に移してください。
- 21,000 × g で30〜40秒間回転させ、氷に戻します。
- RNAをcDNAに変換する:
- 0.8 μL の逆転写ミックスを各テールチップに加えます。
- 42°Cで1時間インキュベートする。
- 70°Cで10分間加熱失活する。
- すぐに氷に移してください。
- 第2のストランドミックスを10μLずつ各尾先端に加える。
- サンプルをフリックします。
- 21,000 × g で 30 ~ 40 秒間回転します。
- 16°Cで2時間インキュベートする。
- cDNAクリーンアップ:
- DNAクリーンアップビーズを室温に予温する。
- 最大 40 サンプルを 1.5 mL 遠沈管にプールします(最大 480 μL)。
- ビーズがよく分散するまで混合し、プールしたサンプル100 μLごとに20 μLのビーズと100 μLのビーズ結合バッファーを追加します(480 μLのサンプルの場合は、ビーズバッファー480 μLとビーズ96 μLを最終容量まで1,056 μLまで加えます)。ピペッティングでよく混ぜる。
- 室温で15分間インキュベートする。
- 液体が透明に見えるまで、磁気スタンドに少なくとも5分間置きます。
- 上清の20 μLを除くすべての上清を取り出して廃棄します。
- 新たに調製した80%エタノールを200μL加える。
- 少なくとも30秒間インキュベートし、ビーズを乱すことなくピペッティングして上清を除去する。上清を捨てる。
- 手順 4.5.7 と 4.5.8 を 1 回繰り返します。
- ビーズを15分間、または完全に乾くまで風乾します。
- ビーズ(約6.4 μL)を6.4 μLの水で再懸濁します。ボリューム全体を10回上下にピペッティングして徹底的に混ぜます。
- 室温で2分間インキュベートする。
- インビトロ転写(IVT)に直行します。
- インビトロ 転写および断片化:
- 6.4 μL のサンプルとビーズを含むチューブに、1.6 μL の 10x T7 バッファー、1.6 μL の ATP、1.6 μL の UTP、1.6 μL の CTP、および 1.6 μL の GTP (75 mM 濃度で各 dNTP) 1.6 μL の T7 酵素のミックス (合計 9.6 μL) を追加します。
- 4°Cのホールドで37°Cで13時間インキュベートする。
- 6 μLのエキソヌクレアーゼ溶液を加える(最終容量は22 μLでなければならない)。
- 37°Cで15分間インキュベートする。
- チューブを氷の上に置き、5.5 μL のフラグメンテーションバッファー (反応容量 0.25 ×) を加えます。
- 94°Cで3分間インキュベートする。
- 直ちにチューブを氷上に移動し、2.75 μLのフラグメンテーション停止バッファー(0.5 ×量のフラグメンテーションバッファーを追加)を加えます。
- 液体が透明になるまで、チューブを磁気スタンドに少なくとも5分間置いてビーズを取り外します。
- 上清を新しいチューブに移す。
- 増幅されたRNA(aRNA)クリーンアップ:
- RNAクリーンアップビーズを室温に予温する。
- ビーズがよく分散するまで混ぜる。
- 55 μLのビーズ(反応容量1.8×)をピペットで流します。
- 室温で10分間インキュベートする。
- 液体が透明になるまで、チューブを磁気スタンドに少なくとも5分間置きます。
- 80 μLの上清を取り出して廃棄する。
- 新たに調製した70%エタノールを200μL加える。
- 少なくとも30秒間インキュベートし、ビーズを乱すことなくピペッティングによって上清を除去する。上清を捨てる。
- エタノール洗浄をさらに2回繰り返す。
- ビーズを15分間、または完全に乾くまで風乾します。
- ビーズを7μLの水で再懸濁する。全容量を10回上下にピペットで完全に混合する。
- 室温で2分間インキュベートする。
- ビーズの入ったチューブを磁気スタンドに5分間置き、液体が透明に見えるまで置きます。
- 上清を新しいチューブに移す。
メモ:停止点:サンプルは-70°Cに保つことができます。
- オプション:メーカーのプロトコルに従って自動電気泳動システムでaRNAの量と品質を確認します。
- ライブラリの準備:
- 5 μL の RNA に、1 μL の 100 μM ランダムヘキサマー RT プライマー ( 材料表を参照) および 0.5 μL の 10 mM dNTP を追加します。
- 65°Cで5分間インキュベートする。
- 室温で次のミックスを4 μL加えます:ファーストストランドバッファー2 μL、0.1 M DDT1 μL、RNase阻害剤0.5 μL、逆転写酵素0.5 μL。
- 25°Cで10分間インキュベートする。
- 42°Cで1時間インキュベートする(ハイブリダイゼーションオーブンまたは蓋を50°Cに設定した予熱サーマルサイクラー中)。
- 70°Cで10分間インキュベートする。
- 5 μLを新しいチューブに移します(残りの反応を-20°Cに保ちます)。5.5 μL の超純水、1 μL の RNA PCR プライマー (RP1)、1 μL のインデックス付き RNA PCR プライマー (RPIX)、および 12.5 μL の PCR ミックスを追加します。
- サーモサイクラーで次のプログラムを使用します:98°Cで30秒、11サイクル:(98°Cで10秒、60°Cで30秒、72°Cで30秒)、72°Cで10分、4°Cで保持します。
メモ:停止点:サンプルは-20°Cに保つことができます。
- ライブラリのクリーンアップ:
- DNAクリーンアップビーズを室温に予温する。
- ビーズがよく分散するまで混ぜる。
- 25 μL のビーズを PCR 反応に加えます。ピペッティングでよく混ぜる。
- 室温で15分間インキュベートする。
- 液体が透明になるまで、チューブを磁気スタンドに少なくとも5分間置きます。
- 45 μLの上清を取り出して廃棄する。
- 新たに調製した80%エタノールを200μL加える。
- 少なくとも30秒間インキュベートし、ビーズを乱すことなく上清を除去し、廃棄する。
- エタノール洗浄を1回繰り返す。
- ビーズを15分間、または完全に乾燥するまで空気乾燥させます。
- それらを25μLの水で再懸濁する。ピペッティングでよく混ぜる。
- 室温で2分間インキュベートする。
- 液体が透明になるまで、チューブを磁気スタンドに5分間置きます。
- 25 μLの上清を新しいチューブに移します。
- 手順 4.10.2-4.10.10 を 1 回繰り返します。
- 10.5μLの水で再懸濁する。ピペッティングでよく混ぜる。
- 室温で2分間インキュベートする。
- 液体が透明になるまで、チューブを磁気スタンドに5分間置きます。
- 上清10 μLを新しいチューブに移し、-20°Cで保存します。
- シーケンシング施設の要件に従ってライブラリの品質と量を評価します。
結果
レーザー捕獲微小解剖に続いて、4つの時点における男性および雌雄同体(L3 22時間後;L4 24、26、および28時間後)を、CEL-Seq2プロトコルを用いたRNAシーケンシングのために調製した。CEL-Seq2プライマーには、特定のサンプル(この場合は個々のテールチップ)からのシーケンシング読み取りをバイオインフォマティカルに識別できる独自のバーコードが含まれています。この方法でシーケンシングデータは、合計557本の尾先(4つの発達時点にわたる266頭の雌雄同体と291人の男性、性別および時間点ごとに59〜78人の尾)について生成された。CEL-Seq2バーコードは、これらのテールチップの97%(すなわち、543)について回収された(補足表S2)。ほとんどの図書館では、回復率は99-100%でした。しかし、男性の1つの時点については88%でした。COVID-19関連の遅延のために、22時間、24時間、および28時間の時点からのオスの尾の先端の約半分が、〜4ヶ月間-80°Cで保存されたことは注目に値します。これは、サンプリングの直後にシーケンシングライブラリを調製することが理想的である一方で、解剖されたサンプルをライブラリ調製前により長い時間保存することが可能であることを示しています。
CEL-Seq2プライマーはまた、各mRNA転写物にUMIを付加する。これにより、PCR重複除去とサンプル中の遺伝子発現の正確な定量が可能になります。UMIの数は尾の先端で劇的に変化した(図4;男性平均= 92,560;男性最小= 155;男性最大= 1,183,998;雌雄同体平均= 67,597;雌雄同体最小= 132;雌雄同体最大= 630,427)。テールチップあたりのUMI数については、 補足表S3を参照してください。単一細胞ライブラリー調製のための入力RNAの量が少ないため、単一細胞シーケンシングデータは大量の技術的ノイズを有することが知られている。したがって、分析24 の前に、UMI カウントが非常に低い、または非常に高いサンプルをフィルタリングすることをお勧めします。
RパッケージpowsimR25 を使用して、単一細胞またはバルクRNA-seq実験で発現差(DE)遺伝子を確実に検出するための統計的検出力およびサンプルサイズ要件を評価しました。シミュレーションのパラメータは、ここで説明する方法で得られた70個のオスの尾端(24時間の時点で)のシーケンシングデータセットに基づいていました。予想される対数倍の変化は、80〜100個のテールチップをプールした別のRNA-seq実験の結果に基づいていました。シミュレーションの結果、シングルテールチップデータは、平均発現値が非常に低い遺伝子を除き、DE遺伝子を検出するのに十分な検出力(真陽性率 = TPR)を有することが決定されました( 図5の上部、破線は80%TPRを表します)。タイムポイントごとにシミュレートされたテールチップを追加すると、低発現遺伝子のパワーが幾分増加しました。同様のパターンが誤検出率(FDR)でも見られます。FDRは、低発現遺伝子に対して高い(>0.10)。しかし、より高発現の遺伝子の場合、公称0.10カットオフ( 図5の下部にあるFDRの破線)以下に該当します。要約すると、タイムポイントごとにサンプリングされるテールの数を70以上に増やしても、FDRを下げたり、パワーを上げたりすることはほとんどありません。しかし、70個のテールチップは、30個のテールチップよりもはるかに低いFDRと強力なパワーを提供します。

図1: Caenorhabditis elegans とハッチオフ法および尾部先端のレーザー微小解剖との同期のための手順の概要。 略語:L1-L4=幼虫期1〜4;PEN=ポリエチレンナフタレート;LMD = レーザーマイクロダイセクション。 この図の拡大版を表示するには、ここをクリックしてください。
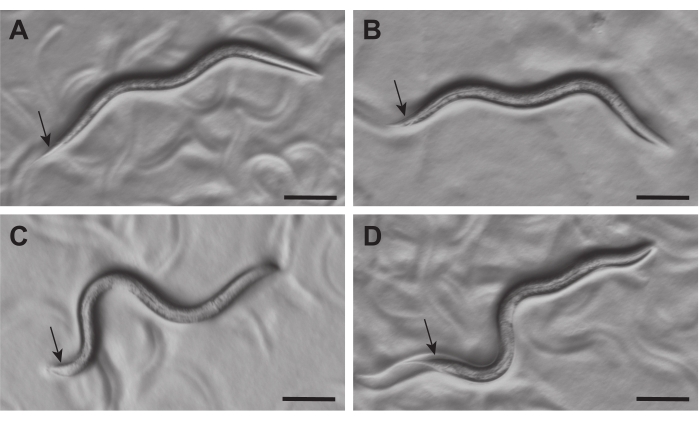
図2:解剖顕微鏡下での C. elegans L3雌雄同体および雄の出現。 孵化後21〜23時間における雌雄同体(A、B)および雄(C、D)は、解剖顕微鏡(〜50倍の倍率)下で、それらの尾(矢印)の形態によって区別することができる。雌雄同体の尾は狭く、男性の尾は腫れてはっきりと見えます。スケール バー = 0.1 mm 。この図の拡大版を表示するには、ここをクリックしてください。

図3:PEN膜スライド構造とワームテールの外観 焦点は、顕微鏡で20倍(A)および40倍(B)レンズで見た組織の解剖に正しい。(c)尾部を解剖し、PEN膜を部分的に切り出した。カットの隙間を閉じた後、膜ピースはスライドの下のチューブキャップに落ちます。(D)解剖された尾先端を含むPEN膜セクションを備えたチューブキャップ。スケール バー = 0.1 mm (A-C)、1 mm (D)。 この図の拡大版を表示するには、ここをクリックしてください。
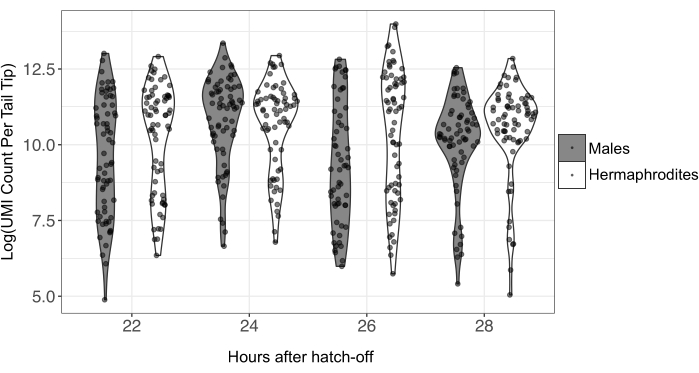
図4:異なる時点および性別について、個々のテールチップあたりの自然対数変換UMIカウント。 個々の尾部からのRNAを、CEL-Seq2法を用いたシークエンシングのために調製した。合計で557尾が配列決定され、性別および時点ごとに59〜78尾が配列された。極端に低いUMI外れ値と高いUMI外れ値は、分析前にデータから削除されます。略語: UMI = 一意の分子識別子。 この図の拡大版を表示するには、ここをクリックしてください。

図5:powsimRによるシミュレーションを使用した 事後 検出力解析の結果。 powsimRソフトウェアは、さまざまな発現レベルでDE遺伝子を検出するために必要な独立したサンプルの数を決定します。遺伝子は、UMIカウントの自然対数として変換された平均発現によってビン化される。(A)条件ごとに異なるサンプルサイズ(個々のテールチップの数)を組み込んだ4つの異なるシミュレーション(異なる色グラフ)のために、2つの条件(ここでは雄対雌雄同体)間のDE遺伝子を検出するパワー(TPR)。破線は 80% TPR を示します。(B)(A)と同じ4つのシミュレーションにおけるFDR、破線は10%FDRを示す。グラフは、発現レベルが非常に低い遺伝子を除き、1条件あたり70テールチップ(緑色)のサンプルサイズでDE遺伝子を検出するのに十分であることを示しています。つまり、このような遺伝子の検出力と誤発見率は、サンプルサイズを70を超えて増やしても大幅に改善することはできません。略語: DE = 差分的に表現;UMI = 一意の分子識別子;TPR = 真陽性率;FDR = 誤検出率。 この図の拡大版を表示するには、ここをクリックしてください。
補足表S1:CEL-Seq2プロトコルで用いたプライマーの配列。このファイルをダウンロードするには、ここをクリックしてください。
補足表S2:サンプリングされた個々のテールチップと回収された個々のテールチップ。 CEL-Seq2プライマーには、各サンプルからのシーケンシング読み取りの識別を可能にする固有のバーコードが含まれており、バイオインフォマティクスで識別できます。シーケンシング用に調製したテールチップサンプルの97%からリードを回収した。 このファイルをダウンロードするには、ここをクリックしてください。
補足表 S3:補足表S2に示されているように、回収されたバーコードを持つすべてのサンプルのUMIカウント。略語: UMI = 一意の分子識別子。このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
メソッドの重要な手順
正しく実行すれば、ここで説明する方法は、比較的少数のレーザー解剖サンプル(この例では70テールチップ)で堅牢なRNAプロファイルを取得します。しかし、発生中の動物からのサンプルの場合、サンプル間のばらつきを減らすためには、緊密な同期が不可欠です。このため、プロトコルではワーム同期のハッチオフ方式を推奨しています。ここで、研究者は、個人間の年齢差(本プロトコールでは1時間)を決定し、正確に制御することができる。さらに、ハッチオフ法は、胚が漂白剤に敏感であっても、L1が停止しない、またはL1停止からの回復が可変であっても、任意の種に適用可能である。ハッチオフによる同期を成功させるためには、洗浄ステップが重要です:すべての成虫と幼虫は孵化期間の初めに除去されなければならず、孵化期間の終わりに新しく孵化したL1と一緒に胚を洗い流すべきではありません。これは、プレートの寒天表面が亀裂、穴、または気泡によって損傷を受けておらず、プレート上の細菌芝生が新鮮で厚すぎず、液体が非常に穏やかに添加および攪拌される場合にのみ成功する。
男性と女性について別々にデータを取得する場合、性別の信頼できる識別も重要です。L3幼虫を性別で区別するには( 図2参照)、経験が必要です。L3の雄と雌雄同体/雌を選ぶ練習をし、動物が大人に発達し、性別が容易に区別された後の成功率をチェックすることをお勧めします。単一組織RNA-Seqの後、外れ値は主成分分析によって同定され、必要に応じて除去することもできる。
レーザーカットサンプルの回収を成功させるには、静電気をできるだけ低減することが重要です。帯電したPEN膜片は、多くの場合、チューブキャップに落ちるのではなく、スライドまたは顕微鏡の他の部分に付着します。1つの救済策は、ステージの隣に小さな加湿器を置くことによって、部屋の湿度、特に顕微鏡の周りの湿度を上げることです。さらに、メンブレンスライドはUV光で処理することができます。これを行うには、UV-C(254nm)架橋チャンバ内でスライドをインキュベートし、少なくとも1ジュールのエネルギーを送達するか、層流ベンチ内のUV光にスライドを30分間さらす。
プロトコルの目標はRNA-Seqであるため、RNaseフリーの作業環境を維持することが重要です。固定溶液から始めて、試薬、容器、消耗品はRNaseフリーでなければならず、作業面は除染され、研究者は清潔な手袋を着用する必要があります。解剖されたサンプルはできるだけ早く凍結し、さらなる処理まで-70°Cに保持する必要があります。また、プロトコルのCEL-Seq2部分には低保持チューブとチップを使用することも推奨されます。
この記事では、開発者によって以前に公開されたCEL-Seq2プロトコルの基本的な概要と、役立つメモとヒント17,18のみを提供します。CEL-Seq2 メソッドを使用する前に、これらのパブリケーションを参照することをお勧めします。
LMD-RNA-Seqデータは、一分子RNA蛍光in-situハイブリダイゼーション(smRNA FISH)26、27、28によって検証することができる。smRNA FISHはC. elegansで広く使用されており、既存の抗体(交差反応しない可能性がある)による免疫染色やトランスジェネシスによる転写レポーターの導入とは異なり、他の線虫種に順応しています。後者はC. elegansおよびいくつかの関連するCaenorhabditis種29ではうまく機能するが、トランスジェネシスは他の線虫種30,31においてより困難であり得る。
メソッドの制限
ここで説明する方法は、ワームの端にある薄い組織である尾の先端を集めるのに非常に効果的です。古い幼虫または成虫のより厚い真ん中で組織を解剖することはより困難です。ここで使用される器具のソフトウェアには、組織内のその後より深いレベルに配置された複数の切開のための設定が含まれています。この設定は、動物のより厚い領域を切断するために使用することができます。ワームは解剖前に固定する必要があるため、構造の詳細が見えにくく、特定の小さな構造物の正確な解剖が妨げられます。前述のように、LMD-RNA-SeqはHTP法ではありません。しかし、50-70サンプルは、午後に解剖することができます。
既存/代替方法に対する本方法の意義
LMD-RNA-Seqは、トランスジェニックツールが利用できない場合でも、どの種でも使用できます。他の方法は、蛍光標識細胞8、9、32のFACS選別または標識核33、34の単離に依存しており、したがってトランスジェニック動物を必要とする。胚後C.エレガンスの細胞を解離および単離する方法は、ワームの両端の組織を見逃す傾向がある(Dylan Rahe、個人的なコミュニケーション)。これらの警告は、単一細胞RNA-Seqとワーム全体の凍結切断(RNA断層撮影)を組み合わせることによって克服されます35。この方法は、C. elegansと別の横紋線虫、Pristionchus pacificus36との間の空間的遺伝子発現を比較するために使用された。あるいは、ホルマリン固定パラフィン埋め込み(FFPE)ワームを試すこともできます。このような物質は、哺乳動物組織試料37のRNA-Seq以下のLMDに首尾よく使用されている。しかし、FFPEワームのRNA断層撮影法およびLMDは、ほんの一握りの動物の分析に限定されている。したがって、それらはLMD-RNA-Seqほど発達中の組織における動的遺伝子発現の研究には適していない。
開示事項
すべての著者は、利益相反がないと宣言します。
謝辞
この研究は、DFへのNIH(R01GM141395)およびNSF(1656736)助成金とAWへのNIHフェローシップ(F32GM136170)によって資金提供されました。 図 1 は、 BioRender.com の助けを借りて作成されました。
資料
| Name | Company | Catalog Number | Comments |
| 0.5 µM PEN membrane glass slides RNase free | Leica | 11600288 | for LMD |
| 500 µL PCR tubes (nuclease-free) | Axygen | 732-0675 | to cut the tail tips into |
| Compound microscope with 40x objective and DIC | any | to check age of worms | |
| Desktop humidifier | any | ||
| Dissection microscope with transmitted light base | any | for all worm work | |
| glass pasteur pipets | any | handle of worm pick | |
| glass slides and coverslips | any | to check age of worms | |
| LMD6 microdissection system | Leica | multiple | to cut tail tips |
| LoBind tubes 0.5 mL | Eppendorf | 22431005 | |
| M9 Buffer | Recipe in WormBook | ||
| Methanol 99.8% | Sigma | 322415 | to fix worms |
| NGM growth medium | US Biological | N1000 | Buffers and salts need to be added: Recipe in WormBook |
| P10 pipette variablle volume | e.g. Gilson | ||
| P1000 pipette variable volume | e.g. Gilson | ||
| P2 pipette variable volume | e.g. Gilson | ||
| Pipette tips 1,000 µL | any | ||
| Pipette tips 1-10 µL filtered | any | ||
| platinum iridium wire | Tritech | PT-9010 | to make worm pick |
| sterile and nuclease-free 1 mL centrfuge tubes | any | ||
| Tween 20 | Sigma | P9416 | Add a very small amount to M9 buffer to prevent worms from sticking to the pipet tips |
| vented 6 mm plastic Petri dishes | any | ||
| For CEL-Seq2 | |||
| 4200 TapeStation System with reagents for high-sensitivity RNA and DNA detection | Aligent | automated electrophoresis system | |
| AMPure XP beads | Beckman Coulter | A63880 | DNA cleanup beads |
| Bead binding buffer 20% PEG8000, 2.5 M NaCl | |||
| CEL-Seq2 primers (see Table S1) | Sigma Genosys Mastercycler Nexus GX2 Eppendorf | 6335000020 | Thermal cycler with programmable lid and block for 200 µl tubes. |
| DNA Polymerase I (E. coli) | Invitrogen | 18052-025 | |
| dNTP mix 10 mM | any | ||
| E. coli DNA ligase | Invitrogen | 18052-019 | |
| Ethanol | |||
| ExoSAP-IT For PCR Product Clean-Up | Affymetrix | 78200 | exonuclease solution |
| MEGAscript T7 Transcription Kit | Ambion | AM1334 | For step 4.6.1 |
| Nuclease-free water | any | ||
| Phusion High-Fidelity PCR Master Mix with HF Buffer | NEB | M0531 | PCR mix step 4.9.7 |
| random hexamer RT primer GCCTTGGCACCCGAGAATTCCA NNNNNN | IDT | a primer with 6 nucleotides that are random | |
| RNA Fragmentation buffer | NEB | E6150S | |
| RNA Fragmentation stop buffer | NEB | E6150S | |
| RNA PCR Index Primers (RPI1–RPI48) | Illumina, NEB, or IDT | RPIX in protocol step 4.9.7, sequences available from Illumina | |
| RNAClean XP beads | Beckman Coulter | A63987 | |
| RNase AWAY Surface Decontaminant | Thermo Scientific | 7000TS1 | or any other similar product |
| RNaseH (E. coli) | Invitrogen | 18021-071 | |
| RNaseOUT Recombinant Ribonuclease Inhibitor | Invitrogen | 10777-019 | |
| Second strand buffer | Invitrogen | 10812-014 | |
| Superscripit II | Invitrogen | 18064-014 | reverse transcriptase |
参考文献
- Haag, E. S., Fitch, D. H. A., Delattre, M. From "the worm" to "the worms" and back again: the evolutionary developmental biology of nematodes. Genetics. 210 (2), 397-433 (2018).
- Sommer, R. J., Bumbarger, D. J. Nematode model systems in evolution and development. Wiley Interdisciplinary Reviews: Developmental Biology. 1 (3), 389-400 (2012).
- WormBase. , Available from: https://wormbase.org/#012-3-6 (2022).
- WormAtlas. , Available from: https://wormatlas.org (2022).
- WormBook. , Available from: http://wormbook.org (2022).
- WormBook in Genetics. , Available from: https://academic-oup-com.remotexs.ntu.edu.sg/genetics/pages/wormbook (2022).
- Cao, J., et al. Comprehensive single-cell transcriptional profiling of a multicellular organism. Science. 357 (6352), 661-667 (2017).
- Kaletsky, R., Murphy, C. T. Transcriptional profiling of C. elegans adult cells and tissues with age. Methods in Molecular Biology. 2144, 177-186 (2020).
- Kaletsky, R., et al. Transcriptome analysis of adult Caenorhabditis elegans cells reveals tissue-specific gene and isoform expression. PLoS Genetics. 14 (8), 1007559(2018).
- Packer, J. S., et al. A lineage-resolved molecular atlas of C. elegans embryogenesis at single-cell resolution. Science. 365 (6459), 1971(2019).
- Kulkarni, A., Anderson, A. G., Merullo, D. P., Konopka, G. Beyond bulk: a review of single cell transcriptomics methodologies and applications. Current Opinion in Biotechnology. 58, 129-136 (2019).
- Von Stetina, S. E., et al. Cell-specific microarray profiling experiments reveal a comprehensive picture of gene expression in the C. elegans nervous system. Genome Biology. 8 (7), 135(2007).
- Baugh, L. R., et al. The homeodomain protein PAL-1 specifies a lineage-specific regulatory network in the C. elegans embryo. Development. 132 (8), 1843-1854 (2005).
- Haenni, S., et al. Analysis of C. elegans intestinal gene expression and polyadenylation by fluorescence-activated nuclei sorting and 3'-end-seq. Nucleic Acids Research. 40 (13), 6304-6318 (2012).
- Zhang, S., Banerjee, D., Kuhn, J. R. Isolation and culture of larval cells from C. elegans. PLoS One. 6 (4), 19505(2011).
- Emmert-Buck, M. R., et al. Laser capture microdissection. Science. 274 (5289), 998-1001 (1996).
- Hashimshony, T., et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biology. 17, 77(2016).
- Yanai, I., Hashimshony, T. CEL-Seq2-single-cell RNA sequencing by multiplexed linear amplification. Methods in Molecular Biology. 1979, 45-56 (2019).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Ceron, J. Basic Caenorhabditis elegans methods: synchronization and observation. Journal of Visualized Experiments: JoVE. (64), e4019(2012).
- Baugh, L. R. To grow or not to grow: nutritional control of development during Caenorhabditis elegans L1 arrest. Genetics. 194 (3), 539-555 (2013).
- Baugh, L. R., Hu, P. J. Starvation responses throughout the Caenorhabditis elegans life cycle. Genetics. 216 (4), 837-878 (2020).
- Pepper, A. S., Killian, D. J., Hubbard, E. J. Genetic analysis of Caenorhabditis elegans glp-1 mutants suggests receptor interaction or competition. Genetics. 163 (1), 115-132 (2003).
- Mok, D. Z., Sternberg, P. W., Inoue, T. Morphologically defined sub-stages of C. elegans vulval development in the fourth larval stage. BMC Developmental Biology. 15, 26(2015).
- Lytal, N., Ran, D., An, L. Normalization methods on single-cell RNA-seq data: an empirical survey. Frontiers in Genetics. 11, 41(2020).
- Vieth, B., Ziegenhain, C., Parekh, S., Enard, W., Hellmann, I. powsimR: power analysis for bulk and single cell RNA-seq experiments. Bioinformatics. 33 (21), 3486-3488 (2017).
- Bolkova, J., Lanctot, C. Quantitative gene expression analysis in Caenorhabditis elegans using single molecule RNA FISH. Methods. 98, 42-49 (2016).
- Ji, N., van Oudenaarden, A. Single molecule fluorescent in situ hybridization (smFISH) of C. elegans worms and embryos. WormBook. , 1-16 (2012).
- Lee, C., et al. Single-molecule RNA fluorescence in situ hybridization (smFISH) in Caenorhabditis elegans. Bio-Protocol. 7 (12), 2357(2017).
- Baird, S. E., Chamberlin, H. M. Caenorhabditis briggsae methods. WormBook. , 1-9 (2006).
- Felix, M. A. Oscheius tipulae. WormBook. , 1-8 (2006).
- Han, Z., et al. Improving transgenesis efficiency and CRISPR-associated tools through codon optimization and native intron addition in Pristionchus nematodes. Genetics. 216 (4), 947-956 (2020).
- Fernandes Povoa, E. E., Ebbing, A. L. P., Betist, M. C., vander Veen, C., Korswagen, H. C. An optimized dissociation protocol for FACS-based isolation of rare cell types from Caenorhabditis elegans L1 larvae. MethodsX. 7, 100922(2020).
- Han, M., Wei, G., McManus, C. E., Hillier, L. W., Reinke, V. Isolated C. elegans germ nuclei exhibit distinct genomic profiles of histone modification and gene expression. BMC Genomics. 20 (1), 500(2019).
- Steiner, F. A., Henikoff, S. Cell type-specific affinity purification of nuclei for chromatin profiling in whole animals. Methods in Molecular Biology. 1228, 3-14 (2015).
- Ebbing, A. Spatial transcriptomics of C. elegans males and hermaphrodites identifies sex-specific differences in gene expression patterns. Developmental Cell. 47 (6), 801-813 (2018).
- Rödelsperger, C., et al. Spatial transcriptomics of nematodes identifies sperm cells as a source of genomic novelty and rapid evolution. Molecular Biology and Evolution. 38 (1), 229-243 (2021).
- Amini, P., et al. An optimised protocol for isolation of RNA from small sections of laser-capture microdissected FFPE tissue amenable for next-generation sequencing. BMC Molecular Biology. 18 (1), 22(2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved