Method Article
التشريح المجهري بالليزر لتطبيقات الأنسجة الأحادية التي لا تعرف الأنواع
In This Article
Summary
يتم وصف بروتوكول يستخدم التشريح المجهري بالليزر لعزل أنسجة الديدان الخيطية الفردية لتسلسل الحمض النووي الريبي. لا يتطلب البروتوكول مجموعات أدوات وراثية خاصة بالأنواع ، مما يسمح بمقارنة ملامح التعبير الجيني بين الأنواع المختلفة على مستوى عينات الأنسجة المفردة.
Abstract
أحدثت منهجيات الخلية الواحدة ثورة في تحليل النسخ لأنواع معينة من الخلايا. ومع ذلك ، فإنها غالبا ما تتطلب "مجموعات أدوات" وراثية خاصة بالأنواع ، مثل المروجين الذين يقودون التعبير الخاص بالأنسجة عن البروتينات الفلورية. علاوة على ذلك ، فإن البروتوكولات التي تعطل الأنسجة لعزل الخلايا الفردية تزيل الخلايا من بيئتها الأصلية (على سبيل المثال ، الإشارات من الجيران) وقد تؤدي إلى استجابات الإجهاد أو الاختلافات الأخرى عن حالات التعبير الجيني الأصلي. في هذا البروتوكول ، تم تحسين التشريح المجهري بالليزر (LMD) لعزل أطراف ذيل الديدان الخيطية الفردية لدراسة التعبير الجيني أثناء تشكل طرف ذيل الذكور.
يسمح LMD بعزل جزء من الحيوان دون الحاجة إلى تعطيل خلوي أو مجموعات أدوات خاصة بالأنواع ، وبالتالي فهو قابل للتطبيق على أي نوع. بعد ذلك ، يمكن تطبيق بروتوكولات إعداد مكتبة RNA-seq أحادية الخلية مثل CEL-Seq2 على الأنسجة المفردة المعزولة بواسطة LMD وتحليلها باستخدام خطوط أنابيب قياسية ، بالنظر إلى أن الجينوم أو النسخ المشروح جيدا متاح للأنواع. يمكن استخدام هذه البيانات لتحديد مدى حفظ أو اختلاف النسخ التي تكمن وراء تطور هذا النسيج في الأنواع المختلفة.
تشمل القيود القدرة على قطع الأنسجة ذات الأهمية وحجم العينة. يظهر تحليل الطاقة أن هناك حاجة إلى ما لا يقل عن 70 طرف ذيل لكل حالة للحصول على 80٪ من الطاقة. هناك حاجة إلى تزامن ضيق للتنمية للحصول على هذا العدد من الحيوانات في نفس مرحلة النمو. وبالتالي ، يتم وصف طريقة لمزامنة الحيوانات على فترات 1 ساعة.
Introduction
الديدان الخيطية - وخاصة الديدان الخيطية الرابديتيد المرتبطة بالنظام النموذجي Caenorhabditis elegans - هي مجموعة رائعة من الحيوانات لبيولوجيا التطور التطوري (EDB) لأسباب عديدة 1,2. وتشمل المزايا العدد الصغير من الخلايا ، وسلالات الخلايا المحددة والمتسقة ، والشفافية ، وسهولة الزراعة والتربية. هناك أيضا العديد من الموارد المتاحة ، بما في ذلك الجينوم عالي الجودة لأنواع متعددة ، وبالنسبة ل C. elegans ، الأدوات الوراثية الجزيئية الواسعة والمعرفة حول التنمية وعلم الوراثة والتشريح وعلم وظائف الأعضاء3،4،5،6.
كما هو الحال مع العديد من الكائنات الحية الأخرى ، فإن القدرة على توصيف ديناميكيات النسخ في الأنسجة المفردة أو الخلايا المفردة قد أحدثت ثورة في تحليل التطور في C. elegans 7,8,9,10. إن القدرة على مقارنة النسخ أحادية الخلية عبر الديدان الخيطية من شأنها أن تحول EDB بالمثل باستخدام هذه الكائنات الحية. على سبيل المثال، من شأن مثل هذه المقارنات أن توفر نظرة ثاقبة حول كيفية تطور الشبكات التنظيمية الجينية للشخصيات (السمات) التي تم الحفاظ عليها، أو للشخصيات التي تباين، أو للشخصيات التي تطورت بشكل مستقل.
ومع ذلك ، فإن عزل أنسجة أو خلايا معينة عن الديدان الخيطية هو أحد التحديات الكبيرة. بالنسبة للعديد من الكائنات الحية، يمكن فصل الخلايا المفردة عن الأنسجة وحصادها بطريقة غير متحيزة أو يمكن تصنيفها بتعبير خاص بالأنسجة لبروتين فلورسنت وفرزها بواسطة فرز الخلايا المنشط بالفلور (FACS)11. في C. elegans ، اقتصر عزل الخلايا عالي الإنتاجية (HTP) في الغالب على الأجنة لأن البشرة الخارجية الصلبة (والهيكل العظمي الهيدروستاتيكي) أعاقت عزل الخلايا عن اليرقات والبالغين. للتغلب على هذا التحدي ، استخدمت بعض الطرق أدوات وراثية في ديدان C. elegans بأكملها ، مثل وضع علامة mRNA الخاصة بالأنسجة12 ، ومقارنات التعبير التفاضلي بين النوع البري والمسوخ التي تؤثر على نوع الخلية13. وقد تغلبت الطرق الحديثة على التحدي عن طريق إذابة البشرة لعزل النوى14 أو خلايا كاملة8،9،15. ومع ذلك ، فإن عزل الخلايا وزراعة الخلايا لهما عيوب واضحة تتمثل في إزالة الخلايا من سياقها التنموي أو التشريحي الطبيعي - على سبيل المثال ، بعيدا عن إشارات الخلايا الخلوية والاتصال بالمصفوفة خارج الخلية - والتي من المتوقع أن تؤثر على ملف تعريف التعبير الجيني15. علاوة على ذلك ، فإن الأدوات الوراثية والعلامات الخاصة بالأنسجة خاصة بالأنواع (أي أنه لا يمكن استخدامها إلا في C. elegans).
يوفر LMD طريقة بديلة لعزل الأنسجة دون تعطيل السياق الطبيعي للخلايا. ويسمح LMD أيضا بمقارنة النسخ من الأنسجة المتجانسة من الأنواع المختلفة دون الحاجة إلى مجموعات أدوات وراثية خاصة بالأنواع إذا كان الجينوم أو تسلسل النسخ الكامل لهذه الأنواع متاحا. يتضمن LMD استهداف الأنسجة عن طريق المراقبة المجهرية المباشرة واستخدام شعاع ليزر مجهري - مدمج في بصريات المجهر - لقطع وحصاد (التقاط) الأنسجة ذات الأهمية16. تتمثل قيود LMD في أنها لا تفضي إلى نهج HTP للغاية (على الرغم من أن ملفات تعريف النسخ لنصائح الذيل ، كما هو موضح في هذا البروتوكول ، كانت قوية مع حوالي 70 عينة) ، وقد يكون من الصعب تشريح بعض العينات ، وتقتصر التخفيضات على دقة الليزر وما يمكن تصوره في المجهر.
الغرض من هذا البروتوكول هو وصف كيفية استخدام LMD ، متبوعا بالحمض النووي الريبي أحادي النسيج ، للحصول على بيانات النسخ الخاصة بالمرحلة والأنسجة من الديدان الخيطية. على وجه التحديد ، فإنه يوضح LMD لعزل أطراف الذيل من يرقات المرحلة الرابعة (L4) من C. elegans. ومع ذلك ، يمكن تكييف هذه الطريقة مع الأنسجة الأخرى ، وبالطبع الأنواع المختلفة.
في C. elegans ، هناك 4 خلايا تجعل طرف الذيل في كل من الذكور والخنثى. خلال مرحلة L4 في الذكور - ولكن ليس في الخنثى - تغير خلايا طرف الذيل شكلها وتهاجر أماميا وداخليا. تحدث هذه العملية أيضا في بعض أنواع الديدان الخيطية الرابديتيد الأخرى وليس كلها. لذلك ، فإن طرف الذيل هو نموذج جيد لتطور التشكل الجنسي ثنائي الشكل. بسبب موقعه ، من السهل أيضا عزل طرف الذيل بواسطة LMD.
للحصول على ملفات تعريف transcriptome من نصائح الذيل ، يستخدم البروتوكول الحالي CEL-Seq2 ، وهي طريقة RNA-seq تم تطويرها للخلايا المفردة17,18. هذه الطريقة لها العديد من المزايا للأنسجة المشتقة من LMD. يتميز CEL-Seq2 بحساسية وكفاءة عالية، حيث يستخدم معرفات جزيئية فريدة (UMIs) للسماح بالقياس الكمي المباشر لقراءات الحمض النووي الريبوزي المرسال، والنسخ في المختبر لضمان التضخيم الخطي، والترميز الشريطي الذي يسمح بتعدد الإرسال لعينات الأنسجة الفردية. القيد الوحيد ل CEL-Seq2 هو أن القراءات المستردة متحيزة إلى نهاية 3' من mRNAs ، وبالتالي لا يمكن تمييز معظم الأشكال المتساوية.
Protocol
1. مزامنة الدودة
ملاحظة: يتم وصف طريقتين أدناه لمزامنة تطور C. elegans والأنواع الأخرى من الرابديتيد.
- المزامنة عن طريق القبض على المرحلة اليرقية الأولى (L1) بعد معالجة هيبوكلوريت قلوية (تبييض).
ملاحظة: تم وصف هذه الطريقة سابقا بالتفصيل19. تعتمد هذه الطريقة على سمتين من C. elegans تنطبق أيضا على العديد من الأنواع الرابديتيد الأخرى: (1) قشر البيض مقاوم للتبييض ، في حين أن البشرة المحيطة بالديدان البالغة واليرقية ليست كذلك. (2) توقف يرقات المرحلة الأولى التطور عند الاحتفاظ بها بدون طعام20.- عالج الخنثى الودثومي (أو الإناث) بمحلول مبيض مخفف لتفتيت بشرتها وإطلاق الأجنة.
- قم بإزالة الأجنة من المبيض واحتفظ بها بدون طعام حتى تفقس جميع L1.
- ضع L1 المعتقل على الطعام ، حيث يستأنف الجميع التطوير في نفس الوقت تقريبا.
ملاحظة: يمكن أن يحدث الخروج من اعتقال L1 في غضون ساعة واحدة.
- المزامنة مع طريقة "الفتحة" (المستخدمة هنا ؛ الشكل 1 أعلاه):
ملاحظة: تسمح طريقة الفقس بالمزامنة الضيقة دون تعطيل التطوير (يؤثر اعتقال L1 على التطور حتى في المراحل اللاحقة21). البروتوكول مقتبس من Pepper et al.22. الهدف من هذه الطريقة هو جمع L1 التي فقست خلال فترة محددة من لوحة تحتوي فقط على الأجنة.- اختيار الأمهات: في المساء قبل إجراء الفقس ، اختر ~ 30 خنثى محبب على طبق مزروع بالإشريكية القولونية OP50.
- احتضان في 25 درجة مئوية لوضع البيض بين عشية وضحاها.
ملاحظة: اختر صفيحة بدون شقوق أو فقاعات حيث يمكن أن تتعثر الديدان. تجنب الألواح ذات العشب البكتيري السميك جدا لأنه سيكون من الصعب إزالة جميع الديدان لاحقا. إذا كنت تعمل مع سلالة حساسة لدرجة الحرارة، فاضبط وقت وضع البيض لحساب التكوين الجنيني الأطول. اختر الأمهات في مرحلة وضع البيض القصوى. - إزالة الأمهات واليرقات: في صباح اليوم التالي ، تحت المجهر تشريح (تكبير 20x) ، ماصة بلطف 1-2 مل من المخزن المؤقت M9 على جدار اللوحة دون رش ؛ دوامة اللوحة لإزاحة الديدان.
- قم بإزالة جميع السوائل والديدان والتخلص منها عن طريق وضع طرف الماصة على جدار اللوحة على حافة الأجار لتجنب ثقب الثقوب. تأكد من عدم ترك أي ديدان (والبيض / الأجنة فقط) على اللوحة ، خاصة L1s ؛ خلاف ذلك ، كرر الغسيل.
- ضع اللوحة عند 25 درجة مئوية لمدة 1 ساعة وانتظر حتى تفقس بعض L1s.
- جمع L1s الفقس حديثا: إسقاط بعناية 1 مل M9 العازلة على أجار. قم بتدوير اللوحة لإزاحة L1 ولكن ليس الأجنة. ماصة بلطف العازلة والديدان في أنبوب الطرد المركزي 1 مل.
- جهاز الطرد المركزي للأنبوب لمدة 1 دقيقة في ~ 18000 × غرام. إزالة supernatant.
- ماصة L1 مباشرة على العشب البكتيري للوحة البذور. تحقق تحت مجهر التشريح من عدم وجود ديدان أو أجنة بالغة.
- حافظ على الديدان عند 25 درجة مئوية حتى تتطور إلى المرحلة المطلوبة.
ملاحظة: إذا كانت الظروف مثالية، يمكن جمع دفعتين أخريين من L1 من نفس اللوحة. افحص اللوحة الأولية للتأكد من عدم وجود L1. إذا لزم الأمر ، اغسل مرة أخرى. كرر الخطوات 1.2.5-1.2.9. -
تحقق من توقيت التطوير. قبل الشروع في تطبيق المصب ، افحص بعض الديدان تحت المجهر المركب عند تكبير 400x للتأكد من أنها وصلت إلى مرحلة النمو المطلوبة ، هنا L3.
ملاحظة: يمكن استخدام مسافة ترحيل خلايا الطرف البعيدة أو خلايا الرابط كدليل ، بالإضافة إلى تطور الفرج. بالنسبة لتطور الفرج ، يوفر Mock et al.23 دليلا مفيدا ، على الرغم من أن التوقيت في تلك الدراسة تم تحديده عند 20 درجة مئوية. عند 25 درجة مئوية ، ستخضع C. elegans من النوع البري لذوبان L3-L4 بعد 24 ساعة من الفقس.
2. جمع الذكور L4 والخنثى والتثبيت
- تحضير خالية من الحمض النووي الريبي ، باردة (-20 درجة مئوية) ، 70 ٪ الميثانول قبل التثبيت.
- تحت مجهر التشريح عند تكبير 30-50x ، ابدأ في اختيار الذكور والخنثى من لوحات التزامن على ألواح منفصلة غير مصنفة بمجرد التمييز بين الجنسين (~ 21 ساعة بعد الفقس ، الشكل 2) ، واستمر في الانتقاء لمدة 1-2 ساعة أو حتى يتم جمع 200.
- حافظ على الديدان عند 25 درجة مئوية حتى تصل إلى المرحلة المطلوبة للتجربة.
- اغسل الديدان من اللوحة باستخدام 1-2 مل من المخزن المؤقت M9 باستخدام طرف ماصة مغسول مسبقا بمخزن مؤقت M9 يحتوي على منظف بنسبة 0.01٪ (لمنع الديدان من الالتصاق بالطرف).
- نقل الديدان إلى أنبوب طرد مركزي 1 مل.
- تدور لمدة 1 دقيقة في 21000 × غرام لتكوير الديدان. إزالة supernatant.
- أضف 1 مل من المخزن المؤقت M9 واخلطه لتفتيت الكريات.
- تدور لمدة 1 دقيقة في 21000 × غرام لتكوير الديدان. إزالة supernatant.
- كرر الغسيل.
- أضف 1 مل من الميثانول المثلج بنسبة 70٪ واخلطه جيدا.
- تدور لمدة 1 دقيقة في 21000 × غرام لتكوير الديدان. إزالة supernatant.
- كرر الخطوتين 2.10 و2.11.
- أضف 500 ميكرولتر من 70٪ ميثانول ، واخلطها ، واحفظها في 4 درجات مئوية لمدة 1 ساعة بين عشية وضحاها.
3. التشريح المجهري بالليزر
ملاحظة: من هنا فصاعدا ، استخدم الكواشف والمواد الاستهلاكية الخالية من RNase ؛ استخدام نصائح التصفية.
- إذا تم استخدام طريقة CEL-Seq2 لمعالجة العينات، فقم بإعداد مزيج رئيسي لكل تمهيدي CEL-Seq2 (الجدول التكميلي S1): ماصة 2 ميكرولتر من التمهيدي CEL-Seq2، و 1 ميكرولتر من 10 mM dNTP، و 9 ميكرولتر من 1٪ β-Mercaptoethanol (في المياه الخالية من RNase) في أنبوب 200 ميكرولتر موسوم.
- التركيب على الشريحة
- تحت مجهر التشريح ، ماصة 20 ميكرولتر من الديدان الثابتة (20-40 دودة من الخطوة 2.13) على الجانب غير اللامع من زجاج غشاء البولي إيثيلين النفثالات (PEN) (حيث يوجد الغشاء).
- انتظر حتى يتبخر الميثانول. استخدم جهاز تدفئة الشرائح لتسريع التبخر.
ملاحظة: يمكن تطبيق قطرات إضافية من الميثانول ، ويستخدم طرف ماصة لنشر الديدان إذا بدأت في التكتل أثناء جفافها. عندما تكون الديدان في كتل ، قد يكون من الصعب تشريحها.
- إعداد المجهر
ملاحظة: البروتوكول التالي خاص بالصك المدرج في جدول المواد. يجب تعديله إذا تم استخدام مجهر LMD مختلف.- ضع مرطب سطح المكتب خلف المسرح على جانب مجهر LMD. تأكد من أن البخار يهب مباشرة على المسرح.
ملاحظة: المرطب مفيد لتقليل الكهرباء الساكنة ، والتي يمكن أن تمنع قسم الغشاء الصغير من السقوط في غطاء الأنبوب. - أدر المفتاح للحصول على طاقة الليزر.
- قم بتشغيل طاقة التحكم في المرحلة.
- قم بتشغيل صندوق التحكم في المجهر.
- افتح برنامج التشريح المجهري بالليزر .
- قم بإزالة الدرع البلاستيكي فوق المسرح.
- انقر فوق زر إلغاء التحميل باستخدام السهم لأعلى لتحميل شرائح الغشاء.
- تأكد من أن الشريحة جافة تماما ، واقلبها بحيث يكون الغشاء متجها لأسفل.
- أدخل الشريحة وانقر فوق متابعة في نافذة نموذج التغيير .
- استبدل الدرع البلاستيكي.
- في أسفل الشاشة، اختر حامل الشريحة الذي يحتوي على الشريحة.
- لتحميل الأنابيب، انقر فوق زر إلغاء التحميل مع السهم لأسفل.
- اسحب الدرج للخارج وأزل كتلة الأنبوب.
ملاحظة: كتلة الأنبوب المستخدمة في هذه التجربة هي لأنابيب PCR سعة 500 ميكرولتر. - أدخل أغطية الأنبوب المكونة من 500 ميكرولتر من أنابيب PCR في الحامل وقم بطي الأنبوب تحتها.
- أعد الكتلة إلى الدرج وحرك الدرج مرة أخرى إلى مرحلة المجهر.
- في النافذة المنبثقة لجهاز تجميع التغييرات ، حدد أنابيب تفاعل البوليميراز المتسلسل (PCR ) وانقر فوق موافق.
- انقر على موقع الأنبوب الفارغ في أسفل يسار الشاشة أسفل أغطية أنبوب جهاز التجميع.
- في لوحة تحكم المجهر ، حدد TL-BF لإضاءة الضوء الساطع المرسلة.
- ضع مرطب سطح المكتب خلف المسرح على جانب مجهر LMD. تأكد من أن البخار يهب مباشرة على المسرح.
- قطع
ملاحظة: هذا البروتوكول خاص بالصك المدرج في جدول المواد.- باستخدام عدسة 2.5x ، اضبط التركيز البؤري حتى تظهر الديدان وبنية الغشاء.
- قم بالتبديل إلى عدسة 20x.
- انقل المرحلة إلى منطقة خالية من الديدان. اضبط التركيز بحيث يكون للهياكل الشبيهة بالفقاعات في الغشاء لون مصفر (الشكل 3A ، B) لتركيز الليزر على المستوى البؤري الصحيح.
- تعيين معلمات الليزر. للحصول على نصائح الذيل، ابدأ باستخدام Power 45 وفتحة العدسة 30 والسرعة 20.
- في لوحة التحكم بالليزر ، حدد المعايرة. اتبع التعليمات.
ملاحظة: ستقوم الأداة بتنفيذ هذه الخطوة تلقائيا. سيضمن أن الشكل المرسوم باستخدام الماوس على الشاشة مطابق للشكل المقطوع بواسطة الليزر. - في الجزء السفلي من الشاشة عند أغطية أنبوب جهاز التجميع ، انقر فوق الموضع A.
- على الجانب الأيسر من الشاشة، حدد شكلا واحدا | رسم + قص. على الجانب الأيمن من الشاشة، حدد PtoP.
- ارسم خطا.
- انقر فوق بدء القطع بحيث يخترق الليزر الغشاء.
ملاحظة: قد يقوم أيضا بحفر خط في الزجاج. - إذا كان هذا الاختبار يبدو جيدا (يتم قطع الغشاء ، تبدو حواف القطع سلسة) ، فتابع الخطوة التالية. خلاف ذلك ، اضبط التركيز البؤري وقطع خط آخر.
- العثور على دودة. قم بالتبديل إلى Move + Cut واستخدم الماوس لقطع الذيل.
ملاحظة: إذا لم يقطع الليزر الذيل ، فاضبط التركيز وزيادة طاقة الليزر. بالنسبة للأنسجة الأكثر سمكا ، قد يلزم ضبط طاقة الليزر على 60. - حفظ المعلمات: علامة التبويب ملف | حفظ تكوين التطبيق; للاسترجاع لاحقا، استعادة تكوين التطبيق.
- لجمع العينة، قم بالتبديل إلى إعداد Draw + Cut باستخدام الدالة PtoP وارسم شكلا لإكمال قطع مقطع غشاء (الشكل 3C).
ملاحظة: من الأسهل تحديد موقع أقسام الغشاء الأكبر حجما وأقسامه على شكل مستطيلات أو مثلثات بدلا من الدوائر أو الأشكال البيضاوية في غطاء أنبوب التجميع. - حدد الأنبوب التالي في غطاء أنبوب جهاز المجمع في أسفل الشاشة واقطع طرف الذيل التالي.
- بمجرد قطع أربعة ذيول ، قم بتفريغ رف الأنبوب (انقر فوق إلغاء التحميل بسهم لأسفل) وابحث عن أقسام الغشاء تحت مجهر تشريح (الشكل 3D).
ملاحظة: قد تكون الأقسام موجودة في منتصف غطاء الأنبوب أو عالقة على جانب الغطاء. - تابع تطبيق المصب. بالنسبة ل CEL-Seq2 ، ماصة 1.2 ميكرولتر من مزيج رئيسي تمهيدي CEL-Seq2 (من الخطوة 3.1) مباشرة أعلى العينة.
- أغلق الأنبوب، وقم بلصقه برقم التمهيدي، وضع غطاء الأنبوب على الفور مباشرة على قطعة من الثلج الجاف لتجميد العينة ومنع تدهور الحمض النووي الريبي.
- قم بتحميل المزيد من الأنابيب ، وأعد كتلة الأنبوب إلى المرحلة ، وقطع المزيد من العينات. أضف مزيجا مختلفا من التمهيدي CEL-Seq2 إلى كل طرف ذيل.
- تخزين جميع الأنابيب في -70 درجة مئوية.
4. تسلسل الحمض النووي الريبي أحادي الذيل مع CEL-Seq2
ملاحظة: للحصول على التفاصيل الكاملة حول بروتوكول CEL-Seq2، راجع Yanai و Hashimshony18.
- نظف منطقة مقعد المختبر باستخدام محلول إزالة التلوث RNase لمنع تدهور الحمض النووي الريبي.
- تحضير الخلطات الرئيسية والاحتفاظ بها على الجليد.
- قم بإعداد المزيج الرئيسي للنسخ العكسي: 0.4 ميكرولتر من المخزن المؤقت الأول للحبلا ، و 0.1 ميكرولتر من 0.1M DTT ، و 0.1 ميكرولتر من مثبط RNase ، و 0.1 ميكرولتر من النسخ العكسي لكل عينة.
- تحضير المزيج الرئيسي لتفاعل الخيط الثاني: 7 ميكرولتر من الماء ، 2.31 ميكرولتر من المخزن المؤقت الثاني للحبلا ، 0.23 ميكرولتر من dNTP ، 0.08 ميكرولتر من E. coli ligase ، 0.3 ميكرولتر من بوليميراز الحمض النووي E. coli ، 0.08 ميكرولتر من RNaseH لكل عينة.
- كسر الخلايا المفتوحة والتلدين باستخدام الاشعال (انظر الجدول التكميلي S1 للحصول على القائمة الكاملة للاشعال :
- قم ببرمجة جهاز تدوير الحرارة وغطائه إلى 65 درجة مئوية.
- استرجع العينات من -70 درجة مئوية واحتضنها في جهاز الدورة الحرارية لمدة 2.5 دقيقة.
- تدور في 21000 × غرام لمدة 30-40 ثانية.
- تحضن عند 65 درجة مئوية لمدة 2.5 دقيقة.
- انقلها على الفور إلى الجليد.
- تدور في 21000 × غرام لمدة 30-40 ثانية وإعادتها إلى الجليد.
- تحويل الحمض النووي الريبي إلى cDNA:
- أضف 0.8 ميكرولتر من مزيج النسخ العكسي إلى كل طرف ذيل.
- تحضن عند 42 درجة مئوية لمدة 1 ساعة.
- الحرارة تعطيلها عند 70 درجة مئوية لمدة 10 دقائق.
- حركه على الفور إلى الجليد.
- أضف 10 ميكرولتر من مزيج الخيط الثاني إلى كل طرف ذيل.
- قم بالنقر فوق العينات.
- تدور في 21000 × غرام لمدة 30-40 ثانية.
- حضانة في 16 درجة مئوية لمدة 2 ساعة.
- تنظيف الحمض النووي الريبوزي منقوص الأكسجين:
- قم بتسخين حبات تنظيف الحمض النووي مسبقا إلى درجة حرارة الغرفة.
- تجميع ما يصل إلى 40 عينة في أنبوب طرد مركزي 1.5 مل (حتى 480 ميكرولتر).
- امزج الخرز حتى يتم تشتيته جيدا وأضف 20 ميكرولتر من الخرز و 100 ميكرولتر من المخزن المؤقت لربط الخرز لكل 100 ميكرولتر من العينة المجمعة (بالنسبة ل 480 ميكرولتر من العينة ، أضف 480 ميكرولتر من المخزن المؤقت للخرز و 96 ميكرولتر من الخرز إلى حجم نهائي يصل إلى 1056 ميكرولتر). تخلط جيدا عن طريق السحب.
- تحضن في درجة حرارة الغرفة لمدة 15 دقيقة.
- ضعه على حامل مغناطيسي لمدة 5 دقائق على الأقل حتى يظهر السائل صافيا.
- قم بإزالة وتجاهل جميع ما عدا 20 ميكرولتر من supernatant.
- أضف 200 ميكرولتر من الإيثانول الطازج بنسبة 80٪.
- احتضن لمدة 30 ثانية على الأقل ، وقم بإزالة supernatant عن طريق سحبه دون إزعاج الخرز. تخلص من السوبرناتانت.
- كرر الخطوتين 4.5.7 و 4.5.8 مرة واحدة.
- جفف الخرز في الهواء لمدة 15 دقيقة أو حتى يجف تماما.
- أعد تعليق الخرز (~ 6.4 ميكرولتر) مع 6.4 ميكرولتر من الماء. اخلطي جيدا عن طريق سحب مستوى الصوت بالكامل لأعلى ولأسفل عشر مرات.
- حضانة في درجة حرارة الغرفة لمدة 2 دقيقة.
- انتقل مباشرة إلى النسخ في المختبر (IVT).
- النسخ والتجزؤ في المختبر:
- إلى الأنبوب الذي يحتوي على 6.4 ميكرولتر من العينة والخرز ، أضف المزيج التالي (إجمالي 9.6 ميكرولتر): 1.6 ميكرولتر من 10x T7 Buffer ، و 1.6 ميكرولتر من ATP ، و 1.6 ميكرولتر من UTP ، و 1.6 ميكرولتر من CTP ، و 1.6 ميكرولتر من GTP (كل dNTP بتركيز 75 mM) 1.6 μLof T7 إنزيم.
- احتضان لمدة 13 ساعة عند 37 درجة مئوية مع عقد 4 درجة مئوية.
- أضف 6 ميكرولتر من محلول exonuclease (يجب أن يكون الحجم النهائي 22 ميكرولتر).
- احتضان لمدة 15 دقيقة عند 37 درجة مئوية.
- ضع الأنبوب مرة أخرى على الجليد وأضف 5.5 ميكرولتر من المخزن المؤقت للتجزئة (0.25 × حجم التفاعل).
- احتضن لمدة 3 دقائق عند 94 درجة مئوية.
- حرك الأنبوب على الفور إلى الجليد وأضف 2.75 ميكرولتر من المخزن المؤقت لإيقاف التجزئة (0.5 × حجم المخزن المؤقت للتجزئة المضاف).
- قم بإزالة الخرز عن طريق وضع الأنبوب على الحامل المغناطيسي لمدة 5 دقائق على الأقل حتى يبدو السائل واضحا.
- انقل السوبرناتانت إلى أنبوب جديد.
- تنظيف الحمض النووي الريبي المضخم (aRNA):
- قم بتسخين حبات تنظيف الحمض النووي الريبي مسبقا إلى درجة حرارة الغرفة.
- اخلطي الخرز حتى يتم تشتيته جيدا.
- ماصة 55 ميكرولتر من الخرز (1.8 × حجم التفاعل).
- احتضانها في درجة حرارة الغرفة لمدة 10 دقائق.
- ضع الأنبوب على الحامل المغناطيسي لمدة 5 دقائق على الأقل حتى يظهر السائل صافيا.
- إزالة وتجاهل 80 ميكرولتر من supernatant.
- أضف 200 ميكرولتر من الإيثانول الطازج بنسبة 70٪.
- احتضان لمدة 30 ثانية على الأقل ، وإزالة supernatant عن طريق سحب دون إزعاج الخرز. تخلص من السوبرناتانت.
- كرر غسل الإيثانول مرتين أخريين.
- جفف الخرز في الهواء لمدة 15 دقيقة أو حتى يجف تماما.
- أعد تعليق الخرز ب 7 ميكرولتر من الماء. ماصة الحجم بأكمله لأعلى ولأسفل 10 مرات لتخلط جيدا.
- حضانة في درجة حرارة الغرفة لمدة 2 دقيقة.
- ضع الأنبوب مع الخرز على الحامل المغناطيسي لمدة 5 دقائق حتى يظهر السائل صافيا.
- انقل السوبرناتانت إلى أنبوب جديد.
ملاحظة: نقطة التوقف: يمكن الاحتفاظ بالعينات عند -70 درجة مئوية.
- اختياري: تحقق من كمية الحمض النووي الريبي المرسال وجودته باستخدام نظام الرحلان الكهربائي الآلي باتباع بروتوكول الشركة المصنعة.
- إعداد المكتبة:
- إلى 5 ميكرولتر من الحمض النووي الريبي، أضف 1 ميكرولتر من 100 ميكرومتر عشوائي hexamer RT primer (انظر جدول المواد) و 0.5 ميكرولتر من 10 mM dNTP.
- تحضن عند 65 درجة مئوية لمدة 5 دقائق.
- أضف 4 ميكرولتر من المزيج التالي في درجة حرارة الغرفة: 2 ميكرولتر من المخزن المؤقت First Strand ، 1 ميكرولتر من 0.1 M DDT ، 0.5 ميكرولتر من مثبط RNase ، 0.5 ميكرولتر من النسخ العكسي.
- تحضن عند 25 درجة مئوية لمدة 10 دقائق.
- احتضان في 42 درجة مئوية لمدة 1 ساعة (في فرن التهجين أو دورة حرارية مسخنة مسبقا مع ضبط الغطاء على 50 درجة مئوية).
- تحضن في 70 درجة مئوية لمدة 10 دقائق.
- انقل 5 ميكرولتر إلى أنبوب جديد (حافظ على بقية التفاعل عند -20 درجة مئوية). أضف 5.5 ميكرولتر من الماء فائق النقاء ، و 1 ميكرولتر من الحمض النووي الريبي PCR Primer (RP1) ، و 1 ميكرولتر من الحمض النووي الريبي المفهرس PCR Primer (RPIX) ، و 12.5 ميكرولتر من مزيج PCR.
- استخدم البرنامج التالي على جهاز التدوير الحراري: 30 ثانية عند 98 درجة مئوية ، 11 دورة من: (10 ثانية عند 98 درجة مئوية ، 30 ثانية عند 60 درجة مئوية ، 30 ثانية عند 72 درجة مئوية) ، 10 دقائق عند 72 درجة مئوية ، عقد عند 4 درجات مئوية.
ملاحظة: نقطة التوقف: يمكن الاحتفاظ بالعينات عند -20 درجة مئوية.
- تنظيف المكتبة:
- قم بتسخين حبات تنظيف الحمض النووي مسبقا إلى درجة حرارة الغرفة.
- اخلطي الخرز حتى يتم تشتيته جيدا.
- أضف 25 ميكرولتر من الخرز إلى تفاعل PCR. تخلط جيدا عن طريق السحب.
- تحضن في درجة حرارة الغرفة لمدة 15 دقيقة.
- ضع الأنبوب على الحامل المغناطيسي لمدة 5 دقائق على الأقل حتى يظهر السائل صافيا.
- إزالة وتجاهل 45 ميكرولتر من supernatant.
- أضف 200 ميكرولتر من الإيثانول الطازج بنسبة 80٪.
- احتضان لمدة 30 ثانية على الأقل ، وإزالة ، والتخلص من supernatant دون إزعاج الخرز.
- كرر غسل الإيثانول مرة واحدة.
- الخرز الجاف في الهواء لمدة 15 دقيقة أو حتى يجف تماما.
- أعد تعليقها ب 25 ميكرولتر من الماء. تخلط جيدا عن طريق السحب.
- حضانة في درجة حرارة الغرفة لمدة 2 دقيقة.
- ضع الأنبوب على الحامل المغناطيسي لمدة 5 دقائق حتى يظهر السائل صافيا.
- نقل 25 ميكرولتر من supernatant إلى أنبوب جديد.
- كرر الخطوات 4.10.2-4.10.10 مرة واحدة.
- أعد التعليق مع 10.5 ميكرولتر من الماء. تخلط جيدا عن طريق السحب.
- حضانة في درجة حرارة الغرفة لمدة 2 دقيقة.
- ضع الأنبوب على الحامل المغناطيسي لمدة 5 دقائق حتى يظهر السائل صافيا.
- انقل 10 ميكرولتر من supernatant إلى أنبوب جديد وخزنه عند -20 درجة مئوية.
- تقييم جودة المكتبة وكميتها وفقا لمتطلبات مرفق التسلسل.
النتائج
بعد التشريح المجهري لالتقاط الليزر ، نصائح الذيل الفردية للذكور والخنثى في 4 نقاط زمنية (L3 22 h بعد الفقس; L4 24 و 26 و 28 ساعة بعد الفتح) تم إعدادها لتسلسل الحمض النووي الريبي باستخدام بروتوكول CEL-Seq2. تحتوي الاشعال CEL-Seq2 على رموز شريطية فريدة تمكن من تحديد قراءات التسلسل من عينة معينة (في هذه الحالة طرف ذيل فردي) بالمعلوماتية الحيوية. تم إنشاء بيانات التسلسل باستخدام هذه الطريقة لما مجموعه 557 نصيحة ذيل (266 خنثى و 291 ذكرا عبر 4 نقاط زمنية للتطور ، 59-78 ذيلا لكل جنس ونقطة زمنية). تم استرداد الباركود CEL-Seq2 ل 97٪ (أي 543) من نصائح الذيل هذه (الجدول التكميلي S2). وبالنسبة لمعظم المكتبات، كان معدل الاسترداد 99-100 في المائة؛ ومع ذلك ، كان 88 ٪ لنقطة زمنية واحدة للذكور. تجدر الإشارة إلى أن حوالي نصف أطراف ذيل الذكور من النقاط الزمنية 22 و 24 و 28 ساعة تم تخزينها عند -80 درجة مئوية لمدة 4 أشهر تقريبا بسبب التأخير المرتبط ب COVID-19. وهذا يدل على أنه في حين أنه من المثالي إعداد مكتبات التسلسل بعد فترة وجيزة من أخذ العينات، فمن الممكن تخزين العينات المشوهة لفترة أطول قبل إعداد المكتبة.
تضيف الاشعال CEL-Seq2 أيضا UMI إلى كل نسخة mRNA. وهذا يمكن من إزالة تكرار تفاعل البوليميراز المتسلسل وتحديد كمي دقيق للتعبير الجيني في العينة. تباين عدد UMIs بشكل كبير عبر أطراف الذيل (الشكل 4 ؛ متوسط الذكور = 92,560 ؛ الحد الأدنى للذكور = 155 ؛ الحد الأقصى للذكور = 1,183,998 ؛ متوسط الخنثى = 67,597 ؛ خنثى الحد الأدنى = 132 ؛ الخنثى كحد أقصى. = 630,427). لمعرفة عدد UMI لكل طرف ذيل، راجع الجدول التكميلي S3. نظرا لانخفاض كمية الحمض النووي الريبي المدخلة لإعداد مكتبة أحادية الخلية ، من المعروف أن بيانات تسلسل الخلية الواحدة تحتوي على كمية كبيرة من الضوضاء التقنية. وبالتالي ، يوصى بتصفية العينات التي تحتوي على عدد UMI منخفض جدا أو مرتفع جدا قبل التحليل24.
تم استخدام حزمة R powsimR25 لتقييم متطلبات القوة الإحصائية وحجم العينة للكشف بشكل موثوق عن الجينات المعبر عنها بشكل تفاضلي (DE) في تجارب الحمض النووي الريبي أحادي الخلية أو السائبة. استندت معلمات عمليات المحاكاة إلى مجموعة بيانات تسلسل مكونة من 70 نصيحة ذيل ذكور فردية (في النقطة الزمنية 24 ساعة) تم الحصول عليها بالطريقة الموضحة هنا. استندت التغييرات المتوقعة في طي السجل إلى نتائج تجربة منفصلة ل RNA-seq جمعت 80-100 طرف ذيل. حددت عمليات المحاكاة أن بيانات الطرف الواحد الذيل لديها طاقة كافية (المعدل الإيجابي الحقيقي = TPR) للكشف عن جينات DE ، باستثناء الجينات التي لها قيمة تعبير متوسطة منخفضة للغاية (أعلى الشكل 5 ؛ يمثل الخط المتقطع 80٪ TPR). إضافة المزيد من أطراف الذيل المحاكية لكل نقطة زمنية زادت من الطاقة إلى حد ما للجينات المعبر عنها بشكل متواضع. وينظر إلى نمط مماثل لمعدل الاكتشاف الخاطئ (FDR). FDR مرتفع (>0.10) للجينات المعبر عنها بشكل منخفض. ومع ذلك ، بالنسبة للجينات الأكثر تعبيرا ، فإنه يقع عند أو أقل من الحد الاسمي 0.10 (خط متقطع ل FDR في أسفل الشكل 5). وباختصار، فإن زيادة عدد الذيول التي أخذت عينات منها في كل نقطة زمنية فوق 70 لن تفعل شيئا يذكر لخفض FDR أو زيادة الطاقة. ومع ذلك ، فإن 70 نصيحة ذيل توفر FDR أقل بكثير وقوة أقوى من 30 نصيحة ذيل.

الشكل 1: نظرة عامة على الإجراء لمزامنة Caenorhabditis elegans مع طريقة الفقس والتشريح المجهري بالليزر لأطراف الذيل. الاختصارات: L1-L4 = مراحل اليرقات من 1 إلى 4 ؛ القلم = البولي إيثيلين النفثالات; LMD = التشريح المجهري بالليزر. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
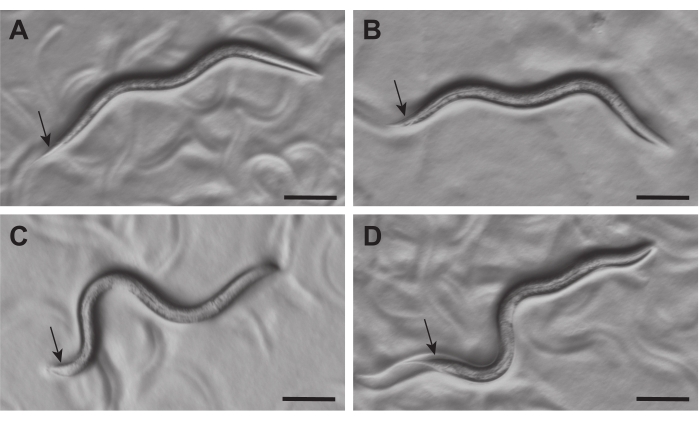
الشكل 2: ظهور خنثى C. elegans L3 والذكور تحت مجهر التشريح. يمكن تمييز الخنثى (A ، B) والذكور (C ، D) في 21-23 ساعة بعد الفتحة تحت مجهر تشريح (~ 50x تكبير) من خلال مورفولوجيا ذيولهم (السهام). ذيل الخنثى ضيق ، في حين أن ذيل الذكور متورم ويبدو واضحا. أشرطة المقياس = 0.1 مم. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 3: ظهور هيكل شريحة غشاء القلم وذيل الدودة. التركيز صحيح لتشريح الأنسجة التي يتم عرضها باستخدام عدسة 20x (A) و 40x (B) في المجهر. (ج) تشريح الذيل وقطع غشاء القلم جزئيا. بعد إغلاق الفجوة في القطع ، ستسقط قطعة الغشاء في غطاء الأنبوب أسفل الشريحة. (د) غطاء أنبوب مع قسم غشاء القلم يحتوي على طرف ذيل تشريحي. أشرطة الميزان = 0.1 مم (A-C)، 1 مم (D). يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
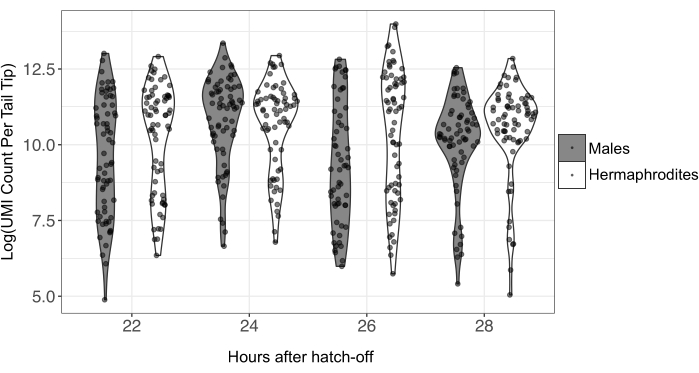
الشكل 4: حساب UMI الطبيعي المحول بالسجل لكل طرف ذيل فردي لنقاط زمنية وجنسين مختلفين. تم إعداد الحمض النووي الريبي من الذيول الفردية للتسلسل باستخدام طريقة CEL-Seq2 ؛ تم تسلسل 557 ذيلا في المجموع ، مع 59-78 ذيلا لكل جنس ونقطة زمنية. ستتم إزالة القيم المتطرفة UMI المنخفضة والعالية للغاية من البيانات قبل التحليل. اختصار: UMI = معرف جزيئي فريد. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 5: نتائج تحليل الطاقة الخلفي باستخدام المحاكاة باستخدام powsimR. يحدد برنامج powsimR عدد العينات المستقلة المطلوبة للكشف عن جينات DE على مستويات التعبير المختلفة. يتم ربط الجينات بواسطة التعبير المتوسط الذي يتم تحويله كسجل طبيعي لعدد UMI. (أ) القدرة (TPR) للكشف عن جينات DE بين شرطين (هنا ، الذكور مقابل الخنثى) لأربع عمليات محاكاة مختلفة (رسوم بيانية ملونة مختلفة) تتضمن أحجام عينات مختلفة (أعداد أطراف الذيل الفردية) لكل حالة. يشير الخط المتقطع إلى 80٪ TPR. (ب) FDR في نفس عمليات المحاكاة الأربعة كما في (A) ، خط متقطع يشير إلى 10٪ FDR. تظهر الرسوم البيانية أن حجم العينة البالغ 70 طرف ذيل (أخضر) لكل حالة يكفي للكشف عن جينات DE ، باستثناء الجينات ذات مستويات التعبير المنخفضة للغاية. وهذا يعني أنه لا يمكن تحسين معدل القوة والاكتشاف الخاطئ لمثل هذه الجينات بشكل كبير عن طريق زيادة حجم العينة إلى ما بعد 70. الاختصارات: DE = يتم التعبير عنها بشكل تفاضلي ؛ UMI = معرف جزيئي فريد ؛ TPR = معدل إيجابي حقيقي ؛ FDR = معدل الاكتشاف الخاطئ. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
الجدول التكميلي S1: تسلسل الاشعال المستخدمة في بروتوكول CEL-Seq2. يرجى النقر هنا لتنزيل هذا الملف.
الجدول التكميلي S2: أخذ عينات مقابل نصائح الذيل الفردية المستردة. تحتوي الاشعال CEL-Seq2 على رموز شريطية فريدة تمكن من تحديد قراءات التسلسل من كل عينة ليتم تحديدها بالمعلوماتية الحيوية. تم استرداد القراءات من 97٪ من عينات طرف الذيل المعدة للتسلسل. يرجى النقر هنا لتنزيل هذا الملف.
الجدول التكميلي دال-3: تحسب UMI جميع العينات ذات الرموز الشريطية المستردة ، كما هو مذكور في الجدول التكميلي S2. اختصار: UMI = معرف جزيئي فريد. يرجى النقر هنا لتنزيل هذا الملف.
Discussion
الخطوات الحاسمة للطريقة
إذا تم تنفيذها بشكل صحيح ، فستحصل الطريقة الموضحة هنا على ملفات تعريف RNA قوية مع عدد صغير نسبيا من العينات التي تم تشريحها بالليزر (70 نصيحة ذيل في هذا المثال). ومع ذلك ، بالنسبة للعينات من الحيوانات النامية ، فإن التزامن الضيق أمر بالغ الأهمية لتقليل التباين بين العينات. لهذا السبب ، يوصي البروتوكول بطريقة الفقس لمزامنة الدودة. هنا ، يمكن للباحث تحديد والتحكم بدقة في فرق العمر بين الأفراد (1 ساعة في هذا البروتوكول). بالإضافة إلى ذلك ، فإن طريقة الفقس قابلة للتطبيق على أي نوع ، حتى لو كانت الأجنة حساسة للتبييض ، أو L1 لا توقف ، أو الانتعاش من اعتقال L1 متغير. من أجل تزامن ناجح عن طريق الفقس ، تعد خطوات الغسيل حاسمة: يجب إزالة جميع البالغين واليرقات في بداية فترة الفقس ، ويجب عدم غسل أي أجنة مع L1 الذي تم فقسه حديثا في نهاية فترة الفقس. ينجح هذا فقط إذا كان سطح الأجار في اللوحة غير تالف بسبب الشقوق أو الثقوب أو الفقاعات ، والعشب البكتيري على اللوحة طازج وليس سميكا جدا ، ويتم إضافة السائل وتحريكه بلطف شديد فقط.
وإذا أريد الحصول على بيانات منفصلة عن الذكور والخنثى/الإناث، فمن المهم أيضا تحديد هوية الجنسين على نحو يعول عليه. يتطلب التمييز بين يرقات L3 حسب الجنس (انظر الشكل 2) خبرة. يوصى بممارسة اختيار الذكور L3 والخنثى / الإناث والتحقق من معدل النجاح بعد تطور الحيوانات إلى بالغين وتمييز الجنسين بسهولة. بعد الحمض النووي الريبي أحادي النسيج ، يمكن أيضا تحديد القيم المتطرفة عن طريق تحليل المكون الرئيسي وإزالتها ، إذا لزم الأمر.
من أجل الاسترداد الناجح للعينات المقطوعة بالليزر ، من المهم تقليل الكهرباء الساكنة قدر الإمكان. غالبا ما لا تسقط قطع غشاء القلم المشحونة في غطاء الأنبوب ولكنها تلتصق بالشريحة أو أي جزء آخر من المجهر. أحد العلاجات هو رفع الرطوبة في الغرفة وتحديدا حول المجهر عن طريق وضع مرطب صغير بجوار المرحلة. بالإضافة إلى ذلك ، يمكن معالجة شرائح الغشاء بالأشعة فوق البنفسجية. للقيام بذلك ، احتضن الشرائح في غرفة متقاطعة UV-C (254 نانومتر) وقم بتوصيل 1 جول على الأقل من الطاقة ، أو قم بتعريض الشرائح لضوء الأشعة فوق البنفسجية في مقعد تدفق الهواء الرقائقي لمدة 30 دقيقة.
نظرا لأن الهدف من البروتوكول هو RNA-Seq ، فإن الحفاظ على بيئة عمل خالية من RNASE أمر بالغ الأهمية. بدءا من محلول التثبيت ، يجب أن تكون الكواشف والحاويات والمواد الاستهلاكية خالية من RNase ، ويجب تطهير سطح العمل ، ويجب على الباحثين ارتداء قفازات نظيفة. يجب تجميد العينات التي تم تشريحها في أقرب وقت ممكن والاحتفاظ بها عند -70 درجة مئوية حتى تتم معالجتها مرة أخرى. يوصى أيضا باستخدام أنابيب ونصائح منخفضة الاحتفاظ لجزء CEL-Seq2 من البروتوكول.
تقدم هذه المقالة فقط مخططا أساسيا لبروتوكول CEL-Seq2 ، الذي نشره مطوروه سابقا مع ملاحظات ونصائح مفيدة17,18. يوصى بالرجوع إلى هذه المنشورات قبل استخدام طريقة CEL-Seq2.
يمكن التحقق من صحة بيانات LMD-RNA-Seq عن طريق التهجين الفلوري في الموقع أحادي الجزيء - الحمض النووي الريبي (smRNA FISH)26،27،28. تم استخدام smRNA FISH على نطاق واسع في C. elegans وهو قابل لأنواع الديدان الخيطية الأخرى ، يختلف عن التلطيخ المناعي بالأجسام المضادة الموجودة (التي قد لا تتفاعل معها) أو إدخال مراسلين نسخيين من خلال التكاثر. هذا الأخير يعمل بشكل جيد في C. elegans وبعض أنواع Caenorhabditis ذات الصلة29 ، ولكن يمكن أن يكون التحول أكثر تحديا في أنواع الديدان الخيطية الأخرى30,31.
قيود الطريقة
الطريقة الموضحة هنا تعمل بشكل جيد للغاية لجمع نصائح الذيل ، وهي نسيج رقيق في نهاية الدودة. تشريح الأنسجة في الوسط الأكثر سمكا من اليرقات الأكبر سنا أو البالغين هو أكثر تحديا. يتضمن برنامج الأداة المستخدمة هنا إعدادا للجروح المتعددة الموضوعة على مستويات أعمق لاحقا في الأنسجة. يمكن استخدام هذا الإعداد لقطع المناطق السميكة من الحيوان. نظرا لأن الديدان تحتاج إلى إصلاح قبل التشريح ، يصعب رؤية التفاصيل الهيكلية ، مما يمنع التشريح الدقيق لهياكل صغيرة محددة. كما ذكر أعلاه ، LMD-RNA-Seq ليست طريقة HTP. ومع ذلك ، يمكن تشريح 50-70 عينة في فترة ما بعد الظهر الواحدة.
أهمية الطريقة فيما يتعلق بالطرق الموجودة/البديلة
يمكن استخدام LMD-RNA-Seq في أي نوع حتى لو لم تكن هناك أدوات معدلة وراثيا متاحة. تعتمد الطرق الأخرى على فرز FACS للخلايا المصنفة بالفلورسنت 8,9,32 أو عزل النوى المصنفة 33,34 وبالتالي تتطلب معدلة وراثيا. تميل الطرق التي تفكك وتعزل الخلايا في C. elegans بعد الجنين إلى تفويت الأنسجة الموجودة في طرفي الدودة (ديلان راهي ، التواصل الشخصي). يتم التغلب على هذه المحاذير من خلال الجمع بين الحمض النووي الريبي أحادي الخلية و التشريح المبرد للديدان بأكملها (التصوير المقطعي للحمض النووي الريبي)35. تم استخدام هذه الطريقة لمقارنة التعبير الجيني المكاني بين C. elegans والديدان الخيطية الرابديتية الأخرى ، Pristionchus pacificus36. بدلا من ذلك ، يمكن للمرء تجربة الديدان المدمجة في البارافين الثابت بالفورمالين (FFPE). وقد استخدمت هذه المواد بنجاح في الحمض النووي الريبي بعد LMD من عينات أنسجة الثدييات37. ومع ذلك ، يقتصر التصوير المقطعي للحمض النووي الريبي و LMD من ديدان FFPE على تحليل حفنة من الحيوانات فقط. وبالتالي ، فهي ليست مناسبة تماما لدراسة التعبير الجيني الديناميكي في الأنسجة النامية مثل LMD-RNA-Seq.
Disclosures
يعلن جميع المؤلفين أنه ليس لديهم تضارب في المصالح.
Acknowledgements
تم تمويل هذا العمل من قبل منح المعاهد الوطنية للصحة (R01GM141395) و NSF (1656736) إلى DF وزمالة المعاهد الوطنية للصحة (F32GM136170) إلى AW. تم إنشاء الشكل 1 بمساعدة BioRender.com.
Materials
| Name | Company | Catalog Number | Comments |
| 0.5 µM PEN membrane glass slides RNase free | Leica | 11600288 | for LMD |
| 500 µL PCR tubes (nuclease-free) | Axygen | 732-0675 | to cut the tail tips into |
| Compound microscope with 40x objective and DIC | any | to check age of worms | |
| Desktop humidifier | any | ||
| Dissection microscope with transmitted light base | any | for all worm work | |
| glass pasteur pipets | any | handle of worm pick | |
| glass slides and coverslips | any | to check age of worms | |
| LMD6 microdissection system | Leica | multiple | to cut tail tips |
| LoBind tubes 0.5 mL | Eppendorf | 22431005 | |
| M9 Buffer | Recipe in WormBook | ||
| Methanol 99.8% | Sigma | 322415 | to fix worms |
| NGM growth medium | US Biological | N1000 | Buffers and salts need to be added: Recipe in WormBook |
| P10 pipette variablle volume | e.g. Gilson | ||
| P1000 pipette variable volume | e.g. Gilson | ||
| P2 pipette variable volume | e.g. Gilson | ||
| Pipette tips 1,000 µL | any | ||
| Pipette tips 1-10 µL filtered | any | ||
| platinum iridium wire | Tritech | PT-9010 | to make worm pick |
| sterile and nuclease-free 1 mL centrfuge tubes | any | ||
| Tween 20 | Sigma | P9416 | Add a very small amount to M9 buffer to prevent worms from sticking to the pipet tips |
| vented 6 mm plastic Petri dishes | any | ||
| For CEL-Seq2 | |||
| 4200 TapeStation System with reagents for high-sensitivity RNA and DNA detection | Aligent | automated electrophoresis system | |
| AMPure XP beads | Beckman Coulter | A63880 | DNA cleanup beads |
| Bead binding buffer 20% PEG8000, 2.5 M NaCl | |||
| CEL-Seq2 primers (see Table S1) | Sigma Genosys Mastercycler Nexus GX2 Eppendorf | 6335000020 | Thermal cycler with programmable lid and block for 200 µl tubes. |
| DNA Polymerase I (E. coli) | Invitrogen | 18052-025 | |
| dNTP mix 10 mM | any | ||
| E. coli DNA ligase | Invitrogen | 18052-019 | |
| Ethanol | |||
| ExoSAP-IT For PCR Product Clean-Up | Affymetrix | 78200 | exonuclease solution |
| MEGAscript T7 Transcription Kit | Ambion | AM1334 | For step 4.6.1 |
| Nuclease-free water | any | ||
| Phusion High-Fidelity PCR Master Mix with HF Buffer | NEB | M0531 | PCR mix step 4.9.7 |
| random hexamer RT primer GCCTTGGCACCCGAGAATTCCA NNNNNN | IDT | a primer with 6 nucleotides that are random | |
| RNA Fragmentation buffer | NEB | E6150S | |
| RNA Fragmentation stop buffer | NEB | E6150S | |
| RNA PCR Index Primers (RPI1–RPI48) | Illumina, NEB, or IDT | RPIX in protocol step 4.9.7, sequences available from Illumina | |
| RNAClean XP beads | Beckman Coulter | A63987 | |
| RNase AWAY Surface Decontaminant | Thermo Scientific | 7000TS1 | or any other similar product |
| RNaseH (E. coli) | Invitrogen | 18021-071 | |
| RNaseOUT Recombinant Ribonuclease Inhibitor | Invitrogen | 10777-019 | |
| Second strand buffer | Invitrogen | 10812-014 | |
| Superscripit II | Invitrogen | 18064-014 | reverse transcriptase |
References
- Haag, E. S., Fitch, D. H. A., Delattre, M. From "the worm" to "the worms" and back again: the evolutionary developmental biology of nematodes. Genetics. 210 (2), 397-433 (2018).
- Sommer, R. J., Bumbarger, D. J. Nematode model systems in evolution and development. Wiley Interdisciplinary Reviews: Developmental Biology. 1 (3), 389-400 (2012).
- WormBase. , Available from: https://wormbase.org/#012-3-6 (2022).
- WormAtlas. , Available from: https://wormatlas.org (2022).
- WormBook. , Available from: http://wormbook.org (2022).
- WormBook in Genetics. , Available from: https://academic-oup-com.remotexs.ntu.edu.sg/genetics/pages/wormbook (2022).
- Cao, J., et al. Comprehensive single-cell transcriptional profiling of a multicellular organism. Science. 357 (6352), 661-667 (2017).
- Kaletsky, R., Murphy, C. T. Transcriptional profiling of C. elegans adult cells and tissues with age. Methods in Molecular Biology. 2144, 177-186 (2020).
- Kaletsky, R., et al. Transcriptome analysis of adult Caenorhabditis elegans cells reveals tissue-specific gene and isoform expression. PLoS Genetics. 14 (8), 1007559(2018).
- Packer, J. S., et al. A lineage-resolved molecular atlas of C. elegans embryogenesis at single-cell resolution. Science. 365 (6459), 1971(2019).
- Kulkarni, A., Anderson, A. G., Merullo, D. P., Konopka, G. Beyond bulk: a review of single cell transcriptomics methodologies and applications. Current Opinion in Biotechnology. 58, 129-136 (2019).
- Von Stetina, S. E., et al. Cell-specific microarray profiling experiments reveal a comprehensive picture of gene expression in the C. elegans nervous system. Genome Biology. 8 (7), 135(2007).
- Baugh, L. R., et al. The homeodomain protein PAL-1 specifies a lineage-specific regulatory network in the C. elegans embryo. Development. 132 (8), 1843-1854 (2005).
- Haenni, S., et al. Analysis of C. elegans intestinal gene expression and polyadenylation by fluorescence-activated nuclei sorting and 3'-end-seq. Nucleic Acids Research. 40 (13), 6304-6318 (2012).
- Zhang, S., Banerjee, D., Kuhn, J. R. Isolation and culture of larval cells from C. elegans. PLoS One. 6 (4), 19505(2011).
- Emmert-Buck, M. R., et al. Laser capture microdissection. Science. 274 (5289), 998-1001 (1996).
- Hashimshony, T., et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biology. 17, 77(2016).
- Yanai, I., Hashimshony, T. CEL-Seq2-single-cell RNA sequencing by multiplexed linear amplification. Methods in Molecular Biology. 1979, 45-56 (2019).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Ceron, J. Basic Caenorhabditis elegans methods: synchronization and observation. Journal of Visualized Experiments: JoVE. (64), e4019(2012).
- Baugh, L. R. To grow or not to grow: nutritional control of development during Caenorhabditis elegans L1 arrest. Genetics. 194 (3), 539-555 (2013).
- Baugh, L. R., Hu, P. J. Starvation responses throughout the Caenorhabditis elegans life cycle. Genetics. 216 (4), 837-878 (2020).
- Pepper, A. S., Killian, D. J., Hubbard, E. J. Genetic analysis of Caenorhabditis elegans glp-1 mutants suggests receptor interaction or competition. Genetics. 163 (1), 115-132 (2003).
- Mok, D. Z., Sternberg, P. W., Inoue, T. Morphologically defined sub-stages of C. elegans vulval development in the fourth larval stage. BMC Developmental Biology. 15, 26(2015).
- Lytal, N., Ran, D., An, L. Normalization methods on single-cell RNA-seq data: an empirical survey. Frontiers in Genetics. 11, 41(2020).
- Vieth, B., Ziegenhain, C., Parekh, S., Enard, W., Hellmann, I. powsimR: power analysis for bulk and single cell RNA-seq experiments. Bioinformatics. 33 (21), 3486-3488 (2017).
- Bolkova, J., Lanctot, C. Quantitative gene expression analysis in Caenorhabditis elegans using single molecule RNA FISH. Methods. 98, 42-49 (2016).
- Ji, N., van Oudenaarden, A. Single molecule fluorescent in situ hybridization (smFISH) of C. elegans worms and embryos. WormBook. , 1-16 (2012).
- Lee, C., et al. Single-molecule RNA fluorescence in situ hybridization (smFISH) in Caenorhabditis elegans. Bio-Protocol. 7 (12), 2357(2017).
- Baird, S. E., Chamberlin, H. M. Caenorhabditis briggsae methods. WormBook. , 1-9 (2006).
- Felix, M. A. Oscheius tipulae. WormBook. , 1-8 (2006).
- Han, Z., et al. Improving transgenesis efficiency and CRISPR-associated tools through codon optimization and native intron addition in Pristionchus nematodes. Genetics. 216 (4), 947-956 (2020).
- Fernandes Povoa, E. E., Ebbing, A. L. P., Betist, M. C., vander Veen, C., Korswagen, H. C. An optimized dissociation protocol for FACS-based isolation of rare cell types from Caenorhabditis elegans L1 larvae. MethodsX. 7, 100922(2020).
- Han, M., Wei, G., McManus, C. E., Hillier, L. W., Reinke, V. Isolated C. elegans germ nuclei exhibit distinct genomic profiles of histone modification and gene expression. BMC Genomics. 20 (1), 500(2019).
- Steiner, F. A., Henikoff, S. Cell type-specific affinity purification of nuclei for chromatin profiling in whole animals. Methods in Molecular Biology. 1228, 3-14 (2015).
- Ebbing, A. Spatial transcriptomics of C. elegans males and hermaphrodites identifies sex-specific differences in gene expression patterns. Developmental Cell. 47 (6), 801-813 (2018).
- Rödelsperger, C., et al. Spatial transcriptomics of nematodes identifies sperm cells as a source of genomic novelty and rapid evolution. Molecular Biology and Evolution. 38 (1), 229-243 (2021).
- Amini, P., et al. An optimised protocol for isolation of RNA from small sections of laser-capture microdissected FFPE tissue amenable for next-generation sequencing. BMC Molecular Biology. 18 (1), 22(2017).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved