Method Article
Microdissezione laser per applicazioni a tessuto singolo indipendenti dalla specie
In questo articolo
Riepilogo
Viene descritto un protocollo che utilizza la microdissezione laser per isolare i singoli tessuti nematodi per il sequenziamento dell'RNA. Il protocollo non richiede toolkit genetici specie-specifici, consentendo di confrontare i profili di espressione genica tra diverse specie a livello di campioni di tessuto singolo.
Abstract
Le metodologie a singola cellula hanno rivoluzionato l'analisi dei trascrittomi di specifici tipi cellulari. Tuttavia, spesso richiedono "toolkit" genetici specie-specifici, come i promotori che guidano l'espressione tessuto-specifica di proteine fluorescenti. Inoltre, i protocolli che interrompono i tessuti per isolare le singole cellule rimuovono le cellule dal loro ambiente nativo (ad esempio, la segnalazione dai vicini) e possono causare risposte allo stress o altre differenze dagli stati di espressione genica nativa. Nel presente protocollo, la microdissezione laser (LMD) è ottimizzata per isolare le singole punte della coda dei nematodi per lo studio dell'espressione genica durante la morfogenesi della punta della coda maschile.
LMD consente l'isolamento di una parte dell'animale senza la necessità di interruzioni cellulari o toolkit specifici per specie ed è quindi applicabile a qualsiasi specie. Successivamente, i protocolli di preparazione della libreria RNA-seq a singola cellula come CEL-Seq2 possono essere applicati a singoli tessuti isolati da LMD e analizzati utilizzando pipeline standard, dato che per la specie è disponibile un genoma o trascrittoma ben annotato. Tali dati possono essere utilizzati per stabilire quanto siano conservati o diversi i trascrittomi che sono alla base dello sviluppo di quel tessuto in specie diverse.
Le limitazioni includono la possibilità di tagliare il tessuto di interesse e la dimensione del campione. Un'analisi di potenza mostra che sono necessarie solo 70 punte di coda per condizione per l'80% di potenza. È necessaria una stretta sincronizzazione dello sviluppo per ottenere questo numero di animali nella stessa fase di sviluppo. Pertanto, viene descritto anche un metodo per sincronizzare gli animali a intervalli di 1 ora.
Introduzione
I nematodi, in particolare i nematodi rabditidi correlati al sistema modello Caenorhabditis elegans, sono un meraviglioso gruppo di animali per la biologia evolutiva dello sviluppo (EDB) per molte ragioni 1,2. I vantaggi includono il loro piccolo numero di cellule, lignaggi cellulari definiti e coerenti, la trasparenza e la facilità di coltura e allevamento. Ci sono anche molte risorse disponibili, tra cui genomi di alta qualità per più specie e per C. elegans, ampi strumenti genetici molecolari e conoscenze sullo sviluppo, la genetica, l'anatomia e la fisiologia 3,4,5,6.
Come per molti altri organismi, la capacità di caratterizzare la dinamica del trascrittoma in singoli tessuti o singole cellule ha rivoluzionato l'analisi dello sviluppo in C. elegans 7,8,9,10. Essere in grado di confrontare i trascrittomi unicellulari tra i nematodi trasformerebbe allo stesso modo l'EDB usando questi organismi. Ad esempio, tali confronti fornirebbero informazioni su come le reti di regolazione genica si sono evolute per i caratteri (tratti) che sono stati conservati, per i caratteri che si sono divergenti o per i caratteri che si sono evoluti in modo indipendente.
Tuttavia, isolare particolari tessuti o cellule dai nematodi è una delle grandi sfide. Per molti organismi, le singole cellule possono essere dissociate dai tessuti e raccolte in modo imparziale o possono essere etichettate con l'espressione tessuto-specifica di una proteina fluorescente e ordinate mediante selezione cellulare attivata dalla fluorescenza (FACS)11. In C. elegans, l'isolamento ad alto rendimento (HTP) delle cellule è stato limitato principalmente agli embrioni perché la cuticola esterna dura (e lo scheletro idrostatico) ha ostacolato l'isolamento cellulare da larve e adulti. Per aggirare questa sfida, alcuni metodi hanno impiegato strumenti genetici in interi vermi C. elegans, come l'mRNA12 specifico del tessuto e i confronti di espressione differenziale tra wild-type e mutanti che colpiscono un tipo di cellula13. Metodi più recenti hanno superato la sfida sciogliendo la cuticola per isolare nuclei14 o intere cellule 8,9,15. L'isolamento cellulare e la coltura cellulare hanno gli ovvi svantaggi, tuttavia, che le cellule vengono rimosse dal loro naturale contesto di sviluppo o anatomico , ad esempio lontano dalla segnalazione cellula-cellula e dal contatto con la matrice extracellulare - che dovrebbero avere un impatto sul profilo di espressione genica15. Inoltre, gli strumenti genetici e i marcatori tessuto-specifici sono specie-specifici (cioè, possono essere utilizzati solo in C. elegans).
LMD fornisce un metodo alternativo per isolare i tessuti senza interrompere il contesto naturale delle cellule. Significativamente per l'EDB, LMD consente anche di confrontare i trascrittomi di tessuti omologhi di diverse specie senza la necessità di toolkit genetici specie-specifici se sono disponibili sequenze di genoma o intere sequenze di trascrittomi di queste specie. LMD prevede il targeting dei tessuti mediante osservazione microscopica diretta e l'utilizzo di un microfascio laser, integrato nell'ottica del microscopio, per ritagliare e raccogliere (catturare) il tessuto di interesse16. I limiti di LMD sono che non è favorevole ad approcci molto HTP (anche se i profili di trascrizione per le punte di coda, come descritto in questo protocollo, erano robusti con ~ 70 campioni), alcuni campioni potrebbero essere difficili da sezionare e i tagli sono limitati alla precisione del laser e a ciò che può essere visualizzato al microscopio.
Lo scopo del presente protocollo è quello di descrivere come LMD, seguito da RNA-Seq a tessuto singolo, può essere utilizzato per ottenere dati sul trascrittoma specifico dello stadio e del tessuto dai nematodi. In particolare, dimostra LMD per isolare le punte della coda dalle larve del quarto stadio (L4) di C. elegans. Tuttavia, questo metodo può essere adattato ad altri tessuti e, naturalmente, a specie diverse.
In C. elegans, ci sono 4 cellule che fanno la punta della coda sia nei maschi che negli ermafroditi. Durante lo stadio L4 nei maschi, ma non negli ermafroditi, le cellule della punta della coda cambiano forma e migrano anteriormente e verso l'interno. Questo processo si verifica anche in alcune ma non in tutte le altre specie di nematodi rabditidi. Pertanto, la punta della coda è un buon modello per l'evoluzione della morfogenesi dimorfica sessuale. A causa della sua posizione, la punta della coda è anche facile da isolare da LMD.
Per ottenere profili di trascrittoma dalle punte della coda, il presente protocollo utilizza CEL-Seq2, un metodo RNA-seq sviluppato per singole cellule17,18. Questo metodo ha diversi vantaggi per i tessuti derivati da LMD. CEL-Seq2 è altamente sensibile ed efficiente, utilizzando identificatori molecolari univoci (UMI) per consentire una quantificazione diretta delle letture dell'mRNA, la trascrizione in vitro per garantire l'amplificazione lineare e il codice a barre che consente il multiplexing di singoli campioni di tessuto. L'unica limitazione di CEL-Seq2 è che le letture recuperate sono distorte verso l'estremità 3' degli mRNA, e la maggior parte delle isoforme quindi non può essere distinta.
Protocollo
1. Sincronizzazione worm
NOTA: Di seguito sono descritti due metodi per sincronizzare lo sviluppo di C. elegans e di altre specie di rabditidi.
- Sincronizzazione mediante arresto del primo stadio larvale (L1) dopo trattamento con ipoclorito alcalino (candeggina).
NOTA: questo metodo è stato descritto in precedenza in dettaglio19. Questo metodo si basa su due caratteristiche di C. elegans che sono vere anche per molte altre specie di rabditidi: (1) Il guscio d'uovo è resistente alla candeggina, mentre la cuticola che circonda i vermi adulti e larvali non lo è. (2) Le larve di primo stadio arrestano lo sviluppo se tenute senza cibo20.- Trattare gli ermafroditi gravidi (o femmine) con una soluzione di candeggina diluita per rompere la cuticola e rilasciare embrioni.
- Rimuovere gli embrioni dalla candeggina e tenerli senza cibo fino a quando tutti gli L1 non si sono schiusi.
- Posiziona l'arrestato L1 sul cibo, dove tutti riprendono lo sviluppo all'incirca nello stesso momento.
NOTA: l'uscita dall'arresto L1 può avvenire entro un'ora.
- Sincronizzazione con il metodo "hatch-off" (usato qui; Figura 1 in alto):
NOTA: il metodo hatch-off consente una stretta sincronizzazione senza interruzioni dello sviluppo (l'arresto L1 influisce sullo sviluppo anche delle fasi successive21). Il protocollo è adattato da Pepper et al.22. L'obiettivo di questo metodo è quello di raccogliere L1 che si sono schiuse in un periodo specifico da una piastra che contiene solo embrioni.- Scegli le madri: La sera prima di eseguire il hatch-off, raccogliere ~ 30 ermafroditi gravidi su un piatto seminato con E. coli OP50.
- Incubare a 25 °C per la deposizione delle uova durante la notte.
NOTA: Scegli una piastra senza crepe o bolle in cui i vermi potrebbero rimanere bloccati. Evitare piatti con un prato batterico molto spesso in quanto sarà difficile rimuovere tutti i vermi in seguito. Se si lavora con un ceppo sensibile alla temperatura, regolare il tempo di deposizione delle uova per tenere conto di un'embriogenesi più lunga. Scegli le madri nella fase massima di deposizione delle uova. - Rimuovere madri e larve: la mattina successiva, sotto il microscopio di dissezione (ingrandimento 20x), pipettare delicatamente 1-2 ml di tampone M9 contro la parete della piastra senza schizzi; ruotare la piastra per rimuovere i vermi.
- Rimuovere e scartare tutto il liquido e i vermi posizionando la punta della pipetta contro la parete della piastra sul bordo dell'agar per evitare di bucare. Controllare che non rimangano vermi (e solo uova/embrioni) sul piatto, specialmente non L1; altrimenti, ripetere il lavaggio.
- Posizionare la piastra a 25 °C per 1 ora e attendere che alcuni L1 si schiudano.
- Raccogliere gli L1 appena schiusi: far cadere con cura 1 mL di tampone M9 sull'agar. Ruotare la piastra per rimuovere L1 ma non gli embrioni. Tampone a pipetta delicata e vermi in un tubo centrifuga da 1 mL.
- Centrifugare il tubo per 1 minuto a ~ 18.000 × g. Rimuovere il surnatante.
- Pipetta L1 direttamente sul prato batterico di un piatto seminato. Verificare al microscopio di dissezione che non siano presenti vermi o embrioni adulti.
- Mantenere i vermi a 25 °C fino a quando non si sono sviluppati allo stadio desiderato.
NOTA: se le condizioni sono ottimali, è possibile prelevare altri due lotti di L1 dalla stessa piastra. Ispezionare la piastra iniziale per assicurarsi che non siano presenti L1. Se necessario, lavare di nuovo. Ripetere i passaggi 1.2.5-1.2.9. -
Controllare i tempi di sviluppo. Prima di procedere all'applicazione a valle, ispezionare alcuni vermi al microscopio composto con ingrandimento 400x per confermare che hanno raggiunto lo stadio di sviluppo desiderato, qui L3.
NOTA: La distanza di migrazione delle cellule della punta distale o delle cellule linker può essere utilizzata come guida, oltre allo sviluppo della vulva. Per lo sviluppo della vulva, Mock et al.23 forniscono una guida utile, anche se i tempi in quello studio sono stati determinati a 20 °C. A 25 °C, i C. elegans selvatici subiranno la muta L3-L4 24 ore dopo la schiusa.
2. Raccolta di maschi L4 ed ermafroditi e fissazione
- Preparare il metanolo al 70% senza RNAsi, freddo (-20 °C) prima della fissazione.
- Sotto un microscopio di dissezione con ingrandimento 30-50x, iniziare a raccogliere maschi ed ermafroditi dalle piastre di sincronizzazione su piastre separate senza semi non appena i sessi possono essere distinti (~ 21 ore dopo la schiusa, Figura 2) e continuare a raccogliere per 1-2 ore o fino a quando non vengono raccolti 200 animali.
- Mantenere i vermi a 25 °C fino a quando non raggiungono lo stadio desiderato per l'esperimento.
- Lavare i vermi dalla piastra con 1-2 ml di tampone M9 utilizzando una punta della pipetta prelavata con tampone M9 contenente lo 0,01% di detergente (per evitare che i vermi si attacchino alla punta).
- Trasferire i vermi in un tubo centrifuga da 1 mL.
- Girare per 1 minuto a 21.000 × g per pellettare i vermi. Rimuovere il surnatante.
- Aggiungere 1 mL di tampone M9 e mescolare per rompere il pellet.
- Girare per 1 minuto a 21.000 × g per pellettare i vermi. Rimuovere il surnatante.
- Ripetere il lavaggio.
- Aggiungere 1 mL di metanolo ghiacciato al 70% e mescolare bene.
- Girare per 1 minuto a 21.000 × g per pellettare i vermi. Rimuovere il surnatante.
- Ripetere i passaggi 2.10 e 2.11.
- Aggiungere 500 μL di metanolo al 70%, mescolare e conservare a 4 °C per 1 ora fino a notte.
3. Microdissezione laser
NOTA: Da qui in poi, utilizzare reagenti e materiali di consumo privi di RNasi; utilizzare suggerimenti per i filtri.
- Se per elaborare i campioni viene utilizzato il metodo CEL-Seq2, preparare una miscela master per ciascun primer CEL-Seq2 (Tabella supplementare S1): pipettare 2 μL di primer CEL-Seq2, 1 μL di 10 mM dNTP e 9 μL di 1% di β-Mercaptoetanolo (in acqua priva di RNasi) in un tubo etichettato da 200 μL.
- Montaggio su slitta
- Al microscopio di dissezione, pipettare 20 μL dei vermi fissi (20-40 vermi dal punto 2.13) sul lato opaco di un vetrino a membrana di polietilene naftalato (PEN) (dove si trova la membrana).
- Attendere che il metanolo evapori. Utilizzare uno scaldafilo per accelerare l'evaporazione.
NOTA: è possibile applicare ulteriori gocce di metanolo e utilizzare una punta di pipetta per stendere i vermi se iniziano a raggrupparsi mentre si asciugano. Quando i vermi sono in ciuffi, possono essere difficili da sezionare.
- Impostazione del microscopio
NOTA: Il seguente protocollo è specifico per lo strumento elencato nella Tabella dei materiali. Deve essere regolato se viene utilizzato un microscopio LMD diverso.- Posizionare un umidificatore desktop dietro il palco sul lato del microscopio LMD. Assicurarsi che il vapore soffi direttamente sul palco.
NOTA: L'umidificatore è utile per ridurre l'elettricità statica, che altrimenti può impedire alla piccola sezione della membrana di cadere nel cappuccio del tubo. - Gira la chiave per l'alimentazione laser.
- Accendi il controllo del palco.
- Accendere la scatola di controllo del microscopio.
- Aprire il software di microdissezione laser .
- Rimuovere lo scudo di plastica sul palco.
- Fare clic sul pulsante di scarico con la freccia verso l'alto per caricare le diapositive a membrana.
- Assicurarsi che la diapositiva sia completamente asciutta, capovolgere in modo che la membrana sia rivolta verso il basso.
- Inserite la diapositiva e fate clic su Continua (Continue ) nella finestra cambia campione .,
- Sostituire lo scudo di plastica.
- Nella parte inferiore dello schermo, scegli quale supporto per diapositive contiene la diapositiva.
- Per caricare i tubi, fare clic sul pulsante di scarico con la freccia verso il basso.
- Estrarre il vassoio e rimuovere il blocco del tubo.
NOTA: il blocco di tubi utilizzato per questo esperimento è per tubi PCR da 500 μL. - Inserire i tappi dei tubi PCR da 500 μL nel supporto e piegare il tubo sotto.
- Riportare il blocco sul vassoio e riportarlo nella fase del microscopio.
- Nella finestra popup del dispositivo di raccolta delle modifiche , selezionare i tubi PCR e fare clic su OK.
- Fare clic sulla posizione del tubo vuoto in basso a sinistra dello schermo sotto i tappi del tubo del dispositivo di raccolta.
- Nel pannello di controllo del microscopio , selezionare TL-BF per l'illuminazione a campo luminoso della luce trasmessa.
- Posizionare un umidificatore desktop dietro il palco sul lato del microscopio LMD. Assicurarsi che il vapore soffi direttamente sul palco.
- Taglio
NOTA: Questo protocollo è specifico per lo strumento elencato nella Tabella dei materiali.- Utilizzando l'obiettivo 2,5x, regolare la messa a fuoco fino a quando i vermi e la struttura della membrana sono visibili.
- Passare all'obiettivo 20x.
- Spostare lo stage in una regione senza worm. Regolare la messa a fuoco in modo tale che le strutture a forma di bolla nella membrana abbiano un colore giallastro (Figura 3A, B) per focalizzare il laser sul piano focale corretto.
- Impostare i parametri laser; per le punte di coda, inizia con Power 45, apertura 30 e velocità 20.
- Nel pannello Controllo laser , selezionate Calibra. Seguire le istruzioni.
NOTA: lo strumento eseguirà automaticamente questo passaggio. Garantirà che una forma disegnata con il mouse sullo schermo sia identica alla forma ritagliata dal laser. - Nella parte inferiore dello schermo in corrispondenza dei tappi del tubo del dispositivo di raccolta, fare clic sulla posizione A.
- Sul lato destro dello schermo, seleziona forma singola | Disegna + Taglia. Sul lato sinistro dello schermo, seleziona PtoP.
- Disegna una linea.
- Fare clic su Avvia taglio in modo che il laser tagli attraverso la membrana.
NOTA: Può anche incidere una linea nel vetro. - Se questo taglio di prova sembra buono (la membrana è tagliata, i bordi del taglio sembrano lisci), continua con il passaggio successivo. Altrimenti, regola la messa a fuoco e taglia un'altra linea.
- Trova un verme. Passare a Sposta + Taglia e utilizzare il mouse per tagliare la coda.
NOTA: se il laser non taglia la coda, regolare la messa a fuoco e aumentare la potenza del laser. Per i tessuti più spessi, potrebbe essere necessario impostare la potenza del laser su 60. - Salva i parametri: scheda File | Salva configurazione applicazione; per un successivo recupero, ripristinare la configurazione dell'applicazione.
- Per raccogliere il campione, passare all'impostazione Disegna + Taglia con la funzione PtoP e disegnare una forma per completare il taglio di una sezione di membrana (Figura 3C).
NOTA: sezioni di membrana più grandi e sezioni a forma di rettangoli o triangoli piuttosto che cerchi o ovali sono più facili da individuare nel cappuccio del tubo del collettore. - Selezionare il tubo successivo in Collector Device Tube Cap nella parte inferiore dello schermo e tagliare la punta della coda successiva.
- Una volta tagliate quattro code, scaricare il rack del tubo (fare clic su Scarica con freccia verso il basso) e trovare le sezioni della membrana sotto un microscopio di dissezione (Figura 3D).
NOTA: le sezioni possono essere posizionate al centro del tappo del tubo o attaccate al lato del cappuccio. - Continuare con l'applicazione a valle. Per CEL-Seq2, pipetta 1,2 μL di una miscela master di primer CEL-Seq2 (dal punto 3.1) direttamente sopra il campione.
- Chiudere il tubo, etichettare con il numero di primer e posizionare immediatamente il cappuccio del tubo direttamente su un pezzo di ghiaccio secco per congelare il campione e prevenire la degradazione dell'RNA.
- Caricare più tubi, riportare il blocco del tubo sul palco e tagliare più campioni. Aggiungere un diverso mix di primer CEL-Seq2 a ciascuna punta della coda.
- Conservare tutti i tubi a -70 °C.
4. Sequenziamento dell'RNA a coda singola con CEL-Seq2
NOTA: per i dettagli completi sul protocollo CEL-Seq2, vedere Yanai e Hashimshony18.
- Pulire l'area del banco di laboratorio con la soluzione di decontaminazione della RNasi per prevenire la degradazione dell'RNA.
- Preparare i mix master e tenerli sul ghiaccio.
- Preparare il mix master di trascrizione inversa: 0,4 μL di tampone del primo filamento, 0,1 μL di 0,1 M DTT, 0,1 μL di inibitore della RNasi e 0,1 μL di trascrittasi inversa per campione.
- Preparare la miscela principale di reazione del secondo filamento: 7 μL di acqua, 2,31 μL di tampone del secondo filamento, 0,23 μL di dNTP, 0,08 μL di E. coli ligasi, 0,3 μL di E. coli DNA polimerasi, 0,08 μL di RNaseH per campione.
- Rottura delle cellule aperte e ricottura con primer (vedere tabella supplementare S1 per l'elenco completo dei primer):
- Programmare il termociclatore e il suo coperchio a 65 °C.
- Prelevare i campioni da -70 °C e incubarli nel termociclatore per 2,5 minuti.
- Ruotare a 21.000 × g per 30-40 s.
- Incubare a 65 °C per 2,5 min.
- Spostali immediatamente sul ghiaccio.
- Ruotare a 21.000 × g per 30-40 s e riportarli sul ghiaccio.
- Conversione dell'RNA in cDNA:
- Aggiungere 0,8 μL della miscela di trascrizione inversa a ciascuna punta della coda.
- Incubare a 42 °C per 1 ora.
- Inattivazione termica a 70 °C Per 10 min.
- Spostalo immediatamente sul ghiaccio.
- Aggiungere 10 μL della miscela del secondo filo a ciascuna punta della coda.
- Scorrere i campioni.
- Ruotare a 21.000 × g per 30-40 s.
- Incubare a 16 °C per 2 ore.
- Pulizia del cDNA:
- Prima del riscaldamento le perle di pulizia del DNA a temperatura ambiente.
- Raggruppare fino a 40 campioni in un tubo centrifugo da 1,5 mL (fino a 480 μL).
- Mescolare le perline fino a quando non sono ben disperse e aggiungere 20 μL di perline e 100 μL di tampone legante perline per ogni 100 μL del campione aggregato (per 480 μL di campione aggiungere 480 μL di tampone perline e 96 μL di perline a un volume finale fino a 1.056 μL). Mescolare bene con il pipettaggio.
- Incubare a temperatura ambiente per 15 min.
- Posizionare su un supporto magnetico per almeno 5 minuti fino a quando il liquido appare limpido.
- Rimuovere ed eliminare tutti tranne 20 μL del surnatante.
- Aggiungere 200 μL di etanolo all'80% appena preparato.
- Incubare per almeno 30 s, rimuovere il surnatante pipettandolo senza disturbare le perline. Scartare il surnatante.
- Ripetere una volta i passaggi 4.5.7 e 4.5.8.
- Asciugare all'aria le perline per 15 minuti o fino a quando non sono completamente asciutte.
- Sospendere le perline (~6,4 μL) con 6,4 μL di acqua. Mescolare accuratamente pipettando l'intero volume su e giù dieci volte.
- Incubare a temperatura ambiente per 2 min.
- Vai direttamente alla trascrizione in vitro (IVT).
- Trascrizione e frammentazione in vitro:
- Alla provetta contenente 6,4 μL di campione e le perline, aggiungere la seguente miscela (9,6 μL totali): 1,6 μL di 10x T7 Buffer, 1,6 μL di ATP, 1,6 μL di UTP, 1,6 μL di CTP e 1,6 μL di GTP (ogni dNTP a concentrazione di 75 mM) 1,6 μLof T7 enzima.
- Incubare per 13 ore a 37 °C con una tenuta di 4 °C.
- Aggiungere 6 μL di soluzione di esonucleasi (il volume finale deve essere di 22 μL).
- Incubare per 15 min a 37 °C.
- Riposizionare il tubo sul ghiaccio e aggiungere 5,5 μL di tampone di frammentazione (0,25 × volume di reazione).
- Incubare per 3 minuti a 94 °C.
- Spostare immediatamente il tubo sul ghiaccio e aggiungere 2,75 μL di buffer di arresto della frammentazione (0,5 × volume di buffer di frammentazione aggiunto).
- Rimuovere le perline posizionando il tubo sul supporto magnetico per almeno 5 minuti fino a quando il liquido appare limpido.
- Trasferire il surnatante in un nuovo tubo.
- Pulizia dell'RNA amplificato (aRNA):
- Preriscaldare le perle di pulizia dell'RNA a temperatura ambiente.
- Mescolare le perline fino a quando non sono ben disperse.
- Pipetta 55 μL di perline (1,8 × volume di reazione).
- Incubarli a temperatura ambiente per 10 min.
- Posizionare il tubo sul supporto magnetico per almeno 5 minuti fino a quando il liquido appare limpido.
- Rimuovere ed eliminare 80 μL del surnatante.
- Aggiungere 200 μL di etanolo al 70% appena preparato.
- Incubare per almeno 30 s, rimuovere il surnatante mediante pipettaggio senza disturbare le perline. Scartare il surnatante.
- Ripetere il lavaggio con etanolo altre due volte.
- Asciugare all'aria le perline per 15 minuti o fino a quando non sono completamente asciutte.
- Sospendere le perline con 7 μL di acqua. Pipettare l'intero volume su e giù 10 volte per mescolare accuratamente.
- Incubare a temperatura ambiente per 2 min.
- Posizionare il tubo con le perline sul supporto magnetico per 5 minuti fino a quando il liquido appare limpido.
- Trasferire il surnatante in un nuovo tubo.
NOTA: Punto di arresto: i campioni possono essere conservati a -70 °C.
- Opzionale: controllare la quantità e la qualità dell'aRNA con un sistema di elettroforesi automatizzato seguendo il protocollo del produttore.
- Preparazione della biblioteca:
- A 5 μL di RNA, aggiungere 1 μL di primer RT ad esamero casuale da 100 μM (vedere la Tabella dei materiali) e 0,5 μL di 10 mM dNTP.
- Incubare a 65 °C per 5 min.
- Aggiungere 4 μL della seguente miscela a temperatura ambiente: 2 μL di tampone First Strand, 1 μL di 0,1 M DDT, 0,5 μL di inibitore della RNasi, 0,5 μL di trascrittasi inversa.
- Incubare a 25 °C per 10 min.
- Incubare a 42 °C per 1 ora (in un forno di ibridazione o in un termociclatore preriscaldato con il coperchio impostato a 50 °C).
- Incubare a 70 °C per 10 min.
- Trasferire 5 μL in un nuovo tubo (mantenere il resto della reazione a -20 °C). Aggiungere 5,5 μL di acqua ultrapura, 1 μL di RNA PCR Primer (RP1), 1 μL di RNA PCR Primer indicizzato (RPIX) e 12,5 μL di miscela PCR.
- Utilizzare il seguente programma sul termociclatore: 30 s a 98 °C, 11 cicli di: (10 s a 98 °C, 30 s a 60 °C, 30 s a 72 °C), 10 min a 72 °C, tenere a 4 °C.
NOTA: Punto di arresto: i campioni possono essere conservati a -20 °C.
- Pulizia della libreria:
- Prima del riscaldamento le perle di pulizia del DNA a temperatura ambiente.
- Mescolare le perline fino a quando non sono ben disperse.
- Aggiungere 25 μL di perline alla reazione PCR. Mescolare bene con il pipettaggio.
- Incubare a temperatura ambiente per 15 min.
- Posizionare il tubo sul supporto magnetico per almeno 5 minuti fino a quando il liquido appare limpido.
- Rimuovere ed eliminare 45 μL del surnatante.
- Aggiungere 200 μL di etanolo all'80% appena preparato.
- Incubare per almeno 30 s, rimuovere e scartare il surnatante senza disturbare le perline.
- Ripetere il lavaggio con etanolo una volta.
- Perline asciutte all'aria per 15 minuti o fino a quando non sono completamente asciutte.
- Risospenderli con 25 μL di acqua. Mescolare bene con il pipettaggio.
- Incubare a temperatura ambiente per 2 min.
- Posizionare il tubo sul supporto magnetico per 5 minuti fino a quando il liquido appare limpido.
- Trasferire 25 μL di surnatante in un nuovo tubo.
- Ripetere una volta i passaggi 4.10.2-4.10.10.
- Sospendere con 10,5 μL di acqua. Mescolare bene con il pipettaggio.
- Incubare a temperatura ambiente per 2 min.
- Posizionare il tubo sul supporto magnetico per 5 minuti fino a quando il liquido appare limpido.
- Trasferire 10 μL del surnatante in un nuovo tubo e conservare a -20 °C.
- Valutare la qualità e la quantità della libreria in base alle esigenze della struttura di sequenziamento.
Risultati
Dopo la microdissezione di cattura laser, le singole punte della coda di maschi ed ermafroditi a 4 punti temporali (L3 22 h dopo la schiusa; L4 24, 26 e 28 h dopo la schiusa) sono stati preparati per il sequenziamento dell'RNA utilizzando il protocollo CEL-Seq2. I primer CEL-Seq2 contengono codici a barre univoci che consentono di identificare bioinformaticamente le letture di sequenziamento da un particolare campione (in questo caso una singola punta di coda). I dati di sequenziamento sono stati generati con questo metodo per un totale di 557 punte della coda (266 ermafroditi e 291 maschi in 4 punti temporali di sviluppo, 59-78 code per sesso e punto temporale). I codici a barre CEL-Seq2 sono stati recuperati per il 97% (cioè 543) di queste punte di coda (Tabella supplementare S2). Per la maggior parte delle biblioteche, il tasso di recupero è stato del 99-100%; tuttavia, era dell'88% per un punto temporale maschile. Vale la pena notare che circa la metà delle punte della coda maschile dei punti temporali di 22, 24 e 28 ore sono state conservate a -80 ° C per ~ 4 mesi a causa di ritardi legati a COVID-19. Ciò dimostra che, mentre è ideale preparare librerie di sequenziamento poco dopo il campionamento, è possibile conservare i campioni sezionati per un tempo più lungo prima della preparazione della libreria.
I primer CEL-Seq2 aggiungono anche un UMI a ciascuna trascrizione di mRNA. Ciò consente la rimozione dei duplicati della PCR e la quantificazione precisa dell'espressione genica nel campione. Il numero di UMI variava notevolmente tra le punte della coda (Figura 4; media maschile = 92.560; min. maschile = 155; maschio max. = 1.183.998; media ermafroditi = 67.597; ermafroditi min. = 132; ermafroditi max. = 630.427). Per i conteggi UMI per punta di coda, vedere Tabella supplementare S3. A causa della bassa quantità di RNA in ingresso per la preparazione della libreria a singola cellula, i dati di sequenziamento a singola cellula sono noti per avere una grande quantità di rumore tecnico. Pertanto, si consiglia di filtrare campioni che hanno conteggi UMI molto bassi o molto alti prima dell'analisi24.
Il pacchetto R powsimR25 è stato utilizzato per valutare la potenza statistica e i requisiti di dimensione del campione per rilevare in modo affidabile i geni differenzialmente espressi (DE) in esperimenti RNA-seq a singola cellula o di massa. I parametri per le simulazioni erano basati su un set di dati di sequenziamento di 70 singole punte della coda maschile (al punto temporale di 24 ore) ottenuto con il metodo qui descritto. I cambiamenti attesi di log-fold erano basati sui risultati di un esperimento separato di RNA-seq che ha raggruppato 80-100 punte della coda. Le simulazioni hanno determinato che i dati a punta singola hanno una potenza sufficiente (True Positive Rate = TPR) per rilevare i geni DE, ad eccezione dei geni che hanno un valore di espressione medio molto basso (in alto nella Figura 5; la linea tratteggiata rappresenta l'80% di TPR). L'aggiunta di più punte di coda simulate per punto temporale ha aumentato in qualche modo la potenza per i geni espressi in modo modesto. Un modello simile è visto per il False Discovery Rate (FDR). FDR è alto (>0,10) per i geni scarsamente espressi; tuttavia, per i geni più altamente espressi, scende al di sotto o al di sotto del limite nominale di 0,10 (linea tratteggiata per FDR nella parte inferiore della Figura 5). In sintesi, aumentare il numero di code campionate per punto temporale al di sopra di 70 farebbe poco per abbassare l'FDR o aumentare la potenza. Tuttavia, 70 punte di coda forniscono un FDR molto più basso e una potenza più forte di 30 punte di coda.

Figura 1: Panoramica della procedura per la sincronizzazione di Caenorhabditis elegans con il metodo hatch-off e la microdissezione laser delle punte della coda. Abbreviazioni: L1-L4 = stadi larvali da 1 a 4; PEN = polietilene naftalato; LMD = microdissezione laser. Fare clic qui per visualizzare una versione più grande di questa figura.
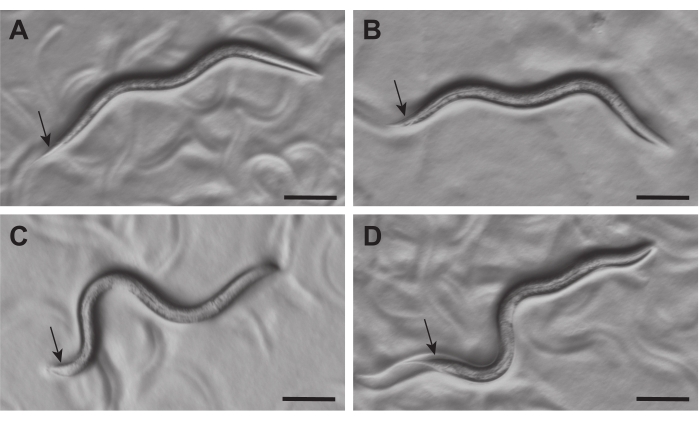
Figura 2: Comparsa di Ermafroditi e maschi di C. elegans L3 al microscopio di dissezione. Gli ermafroditi (A, B) e i maschi (C, D) a 21-23 ore dopo la schiusa possono essere distinti al microscopio di dissezione (ingrandimento ~ 50x) dalla morfologia delle loro code (frecce). La coda degli ermafroditi è stretta, mentre quella dei maschi è gonfia e appare chiara. Barre della scala = 0,1 mm. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Aspetto della struttura del vetrino della membrana PEN e della coda a vite senza fine. La messa a fuoco è corretta per la dissezione del tessuto visualizzato con la lente 20x (A) e 40x (B) al microscopio. (C) Coda sezionata e membrana PEN parzialmente tagliata. Dopo aver chiuso lo spazio nel taglio, il pezzo di membrana cadrà nel cappuccio del tubo sotto la diapositiva. (D) Cappuccio del tubo con una sezione di membrana PEN contenente una punta della coda sezionata. Barre di scala = 0,1 mm (A-C), 1 mm (D). Fare clic qui per visualizzare una versione più grande di questa figura.
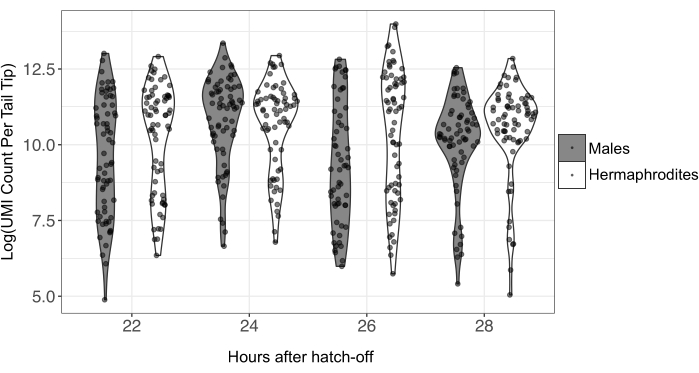
Figura 4: Conteggi UMI naturali trasformati in log per singola punta di coda per diversi punti temporali e sessi. L'RNA delle singole code è stato preparato per il sequenziamento utilizzando il metodo CEL-Seq2; 557 code sono state sequenziate in totale, con 59-78 code per sesso e punto temporale. I valori anomali UMI estremamente bassi e alti verrebbero rimossi dai dati prima dell'analisi. Abbreviazione: UMI = identificatore molecolare univoco. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 5: Risultati di un'analisi di potenza a posteriori utilizzando simulazioni con powsimR. Il software powsimR determina il numero di campioni indipendenti necessari per rilevare i geni DE a vari livelli di espressione. I geni sono legati dall'espressione media trasformata come il registro naturale dei conteggi UMI. (A) Potenza (TPR) per rilevare i geni DE tra due condizioni (qui, maschio vs ermafrodita) per quattro diverse simulazioni (diversi grafici colorati) che incorporano diverse dimensioni del campione (numero di singole punte della coda) per condizione. La linea tratteggiata indica l'80% di TPR. (B) FDR nelle stesse quattro simulazioni di cui alla (A), linea tratteggiata che indica il 10% di FDR. I grafici mostrano che una dimensione del campione di 70 punte della coda (verde) per condizione è sufficiente per rilevare i geni DE, ad eccezione dei geni con livelli di espressione molto bassi. Cioè, la potenza e il tasso di falsa scoperta per tali geni non possono essere notevolmente migliorati aumentando la dimensione del campione oltre 70. Abbreviazioni: DE = espresso in modo differenziale; UMI = identificatore molecolare univoco; TPR = tasso di positività reale; FDR = tasso di falsa scoperta. Fare clic qui per visualizzare una versione più grande di questa figura.
Tabella supplementare S1: Sequenze di primer utilizzati nel protocollo CEL-Seq2. Fare clic qui per scaricare questo file.
Tabella supplementare S2: Punte della coda individuali campionate rispetto a quelle recuperate. I primer CEL-Seq2 contengono codici a barre univoci che consentono di identificare bioinformaticamente le letture di sequenziamento da ciascun campione. Le letture sono state recuperate dal 97% dei campioni di punta della coda preparati per il sequenziamento. Fare clic qui per scaricare questo file.
Tabella supplementare S3: UMI conta tutti i campioni con codici a barre recuperati, come indicato nella Tabella supplementare S2. Abbreviazione: UMI = identificatore molecolare univoco. Fare clic qui per scaricare questo file.
Discussione
Passaggi critici del metodo
Se eseguito correttamente, il metodo qui descritto otterrà profili di RNA robusti con un numero relativamente piccolo di campioni sezionati al laser (70 punte di coda in questo esempio). Tuttavia, per i campioni provenienti da animali in via di sviluppo, una stretta sincronizzazione è fondamentale per ridurre la variabilità tra i campioni. Per questo motivo, il protocollo consiglia il metodo hatch-off per la sincronizzazione worm. Qui, il ricercatore può determinare e controllare con precisione la differenza di età tra gli individui (1 ora nel presente protocollo). Inoltre, il metodo hatch-off è applicabile a qualsiasi specie, anche se gli embrioni sono sensibili alla candeggina, L1 non arrestano o il recupero dall'arresto L1 è variabile. Per una sincronizzazione di successo mediante schiusa, le fasi di lavaggio sono cruciali: tutti gli adulti e le larve devono essere rimossi all'inizio del periodo di schiusa e nessun embrione deve essere lavato via insieme alla L1 appena schiusa alla fine del periodo di schiusa. Questo ha successo solo se la superficie dell'agar della piastra non è danneggiata da crepe, fori o bolle, il prato batterico sul piatto è fresco e non troppo spesso e il liquido viene aggiunto e agitato solo molto delicatamente.
Se i dati devono essere ottenuti separatamente per maschi ed ermafroditi/femmine, è importante anche un'identificazione affidabile dei sessi. Distinguere le larve L3 per sesso (vedi Figura 2) richiede esperienza. Si raccomanda di esercitarsi a scegliere maschi l3 ed ermafroditi / femmine e controllare il tasso di successo dopo che gli animali si sono sviluppati in adulti e i sessi sono facilmente distinguibili. Dopo l'RNA-Seq a tessuto singolo, i valori anomali possono anche essere identificati mediante analisi dei componenti principali e rimossi, se necessario.
Per il successo del recupero dei campioni tagliati al laser, è importante ridurre il più possibile l'elettricità statica. I pezzi caricati della membrana PEN spesso non cadono nel cappuccio del tubo ma si attaccano al vetrino o a qualsiasi altra parte del microscopio. Un rimedio è aumentare l'umidità nella stanza e in particolare intorno al microscopio posizionando un piccolo umidificatore accanto al palco. Inoltre, i vetrini a membrana possono essere trattati con luce UV. Per fare ciò, incubare i vetrini in una camera di reticolazione UV-C (254 nm) e fornire almeno 1 joule di energia, oppure esporre i vetrini alla luce UV in un banco di flusso d'aria laminare per 30 minuti.
Poiché l'obiettivo del protocollo è RNA-Seq, mantenere un ambiente di lavoro privo di RNasi è fondamentale. A partire dalla soluzione di fissaggio, i reagenti, i contenitori e i materiali di consumo dovrebbero essere privi di RNasi, la superficie di lavoro dovrebbe essere decontaminata e i ricercatori dovrebbero indossare guanti puliti. I campioni sezionati devono essere congelati il prima possibile e conservati a -70 °C fino a un'ulteriore trasformazione. Si raccomanda inoltre di utilizzare tubi e punte a bassa ritenzione per la parte CEL-Seq2 del protocollo.
Il presente articolo fornisce solo uno schema di base del protocollo CEL-Seq2, che è stato precedentemente pubblicato dai suoi sviluppatori con note utili e suggerimenti17,18. Si raccomanda di consultare queste pubblicazioni prima di utilizzare il metodo CEL-Seq2.
I dati LMD-RNA-Seq possono essere convalidati mediante ibridazione fluorescente in situ a singola molecola-RNA (smRNA FISH)26,27,28. smRNA FISH è stato ampiamente utilizzato in C. elegans ed è suscettibile di altre specie di nematodi, diverso dall'immunocolorazione con anticorpi esistenti (che non possono reagire in modo incrociato) o dall'introduzione di reporter trascrizionali attraverso la transgenesi. Quest'ultimo funziona bene in C. elegans e in alcune specie di Caenorhabditis 29 correlate, ma la transgenesi può essere più impegnativa in altre specie di nematodi30,31.
Limitazioni del metodo
Il metodo qui descritto funziona molto bene per la raccolta delle punte della coda, un tessuto sottile alla fine di un verme. Sezionare i tessuti nel mezzo più spesso di larve più vecchie o adulti è più impegnativo. Il software dello strumento qui utilizzato include un'impostazione per tagli multipli posti a livelli successivamente più profondi nel tessuto. Questa impostazione può essere utilizzata per tagliare aree più spesse dell'animale. Poiché i vermi devono essere riparati prima della dissezione, i dettagli strutturali sono difficili da vedere, il che impedisce la dissezione precisa di specifiche piccole strutture. Come accennato in precedenza, LMD-RNA-Seq non è un metodo HTP. Tuttavia, 50-70 campioni possono essere sezionati in un pomeriggio.
Importanza del metodo rispetto ai metodi esistenti/alternativi
LMD-RNA-Seq può essere utilizzato in qualsiasi specie anche se non sono disponibili strumenti transgenici. Altri metodi si basano sullo smistamento FACS di cellule marcate fluorescentemente 8,9,32 o sull'isolamento di nuclei marcati33,34 e quindi richiedono animali transgenici. I metodi che dissociano e isolano le cellule in C. elegans postembrionale tendono a perdere i tessuti alle due estremità del verme (Dylan Rahe, comunicazione personale). Questi avvertimenti vengono superati combinando l'RNA-Seq a singola cellula con la criosezione di interi vermi (tomografia a RNA)35. Questo metodo è stato utilizzato per confrontare l'espressione genica spaziale tra C. elegans e un altro nematode rabditide, Pristionchus pacificus36. In alternativa, si può sperimentare con vermi incorporati in paraffina fissata in formalina (FFPE). Tale materiale è stato utilizzato con successo per l'RNA-Seq in seguito a LMD di campioni di tessuto di mammifero37. Tuttavia, la tomografia a RNA e l'LMD dei vermi FFPE sono limitate all'analisi di solo una manciata di animali. Non sono, quindi, così adatti per lo studio dell'espressione genica dinamica nei tessuti in via di sviluppo come LMD-RNA-Seq.
Divulgazioni
Tutti gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
Questo lavoro è stato finanziato da NIH (R01GM141395) e NSF (1656736) sovvenzioni a DF e NIH fellowship (F32GM136170) ad AW. La Figura 1 è stata creata con l'aiuto di BioRender.com.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.5 µM PEN membrane glass slides RNase free | Leica | 11600288 | for LMD |
| 500 µL PCR tubes (nuclease-free) | Axygen | 732-0675 | to cut the tail tips into |
| Compound microscope with 40x objective and DIC | any | to check age of worms | |
| Desktop humidifier | any | ||
| Dissection microscope with transmitted light base | any | for all worm work | |
| glass pasteur pipets | any | handle of worm pick | |
| glass slides and coverslips | any | to check age of worms | |
| LMD6 microdissection system | Leica | multiple | to cut tail tips |
| LoBind tubes 0.5 mL | Eppendorf | 22431005 | |
| M9 Buffer | Recipe in WormBook | ||
| Methanol 99.8% | Sigma | 322415 | to fix worms |
| NGM growth medium | US Biological | N1000 | Buffers and salts need to be added: Recipe in WormBook |
| P10 pipette variablle volume | e.g. Gilson | ||
| P1000 pipette variable volume | e.g. Gilson | ||
| P2 pipette variable volume | e.g. Gilson | ||
| Pipette tips 1,000 µL | any | ||
| Pipette tips 1-10 µL filtered | any | ||
| platinum iridium wire | Tritech | PT-9010 | to make worm pick |
| sterile and nuclease-free 1 mL centrfuge tubes | any | ||
| Tween 20 | Sigma | P9416 | Add a very small amount to M9 buffer to prevent worms from sticking to the pipet tips |
| vented 6 mm plastic Petri dishes | any | ||
| For CEL-Seq2 | |||
| 4200 TapeStation System with reagents for high-sensitivity RNA and DNA detection | Aligent | automated electrophoresis system | |
| AMPure XP beads | Beckman Coulter | A63880 | DNA cleanup beads |
| Bead binding buffer 20% PEG8000, 2.5 M NaCl | |||
| CEL-Seq2 primers (see Table S1) | Sigma Genosys Mastercycler Nexus GX2 Eppendorf | 6335000020 | Thermal cycler with programmable lid and block for 200 µl tubes. |
| DNA Polymerase I (E. coli) | Invitrogen | 18052-025 | |
| dNTP mix 10 mM | any | ||
| E. coli DNA ligase | Invitrogen | 18052-019 | |
| Ethanol | |||
| ExoSAP-IT For PCR Product Clean-Up | Affymetrix | 78200 | exonuclease solution |
| MEGAscript T7 Transcription Kit | Ambion | AM1334 | For step 4.6.1 |
| Nuclease-free water | any | ||
| Phusion High-Fidelity PCR Master Mix with HF Buffer | NEB | M0531 | PCR mix step 4.9.7 |
| random hexamer RT primer GCCTTGGCACCCGAGAATTCCA NNNNNN | IDT | a primer with 6 nucleotides that are random | |
| RNA Fragmentation buffer | NEB | E6150S | |
| RNA Fragmentation stop buffer | NEB | E6150S | |
| RNA PCR Index Primers (RPI1–RPI48) | Illumina, NEB, or IDT | RPIX in protocol step 4.9.7, sequences available from Illumina | |
| RNAClean XP beads | Beckman Coulter | A63987 | |
| RNase AWAY Surface Decontaminant | Thermo Scientific | 7000TS1 | or any other similar product |
| RNaseH (E. coli) | Invitrogen | 18021-071 | |
| RNaseOUT Recombinant Ribonuclease Inhibitor | Invitrogen | 10777-019 | |
| Second strand buffer | Invitrogen | 10812-014 | |
| Superscripit II | Invitrogen | 18064-014 | reverse transcriptase |
Riferimenti
- Haag, E. S., Fitch, D. H. A., Delattre, M. From "the worm" to "the worms" and back again: the evolutionary developmental biology of nematodes. Genetics. 210 (2), 397-433 (2018).
- Sommer, R. J., Bumbarger, D. J. Nematode model systems in evolution and development. Wiley Interdisciplinary Reviews: Developmental Biology. 1 (3), 389-400 (2012).
- WormBase. , Available from: https://wormbase.org/#012-3-6 (2022).
- WormAtlas. , Available from: https://wormatlas.org (2022).
- WormBook. , Available from: http://wormbook.org (2022).
- WormBook in Genetics. , Available from: https://academic-oup-com.remotexs.ntu.edu.sg/genetics/pages/wormbook (2022).
- Cao, J., et al. Comprehensive single-cell transcriptional profiling of a multicellular organism. Science. 357 (6352), 661-667 (2017).
- Kaletsky, R., Murphy, C. T. Transcriptional profiling of C. elegans adult cells and tissues with age. Methods in Molecular Biology. 2144, 177-186 (2020).
- Kaletsky, R., et al. Transcriptome analysis of adult Caenorhabditis elegans cells reveals tissue-specific gene and isoform expression. PLoS Genetics. 14 (8), 1007559(2018).
- Packer, J. S., et al. A lineage-resolved molecular atlas of C. elegans embryogenesis at single-cell resolution. Science. 365 (6459), 1971(2019).
- Kulkarni, A., Anderson, A. G., Merullo, D. P., Konopka, G. Beyond bulk: a review of single cell transcriptomics methodologies and applications. Current Opinion in Biotechnology. 58, 129-136 (2019).
- Von Stetina, S. E., et al. Cell-specific microarray profiling experiments reveal a comprehensive picture of gene expression in the C. elegans nervous system. Genome Biology. 8 (7), 135(2007).
- Baugh, L. R., et al. The homeodomain protein PAL-1 specifies a lineage-specific regulatory network in the C. elegans embryo. Development. 132 (8), 1843-1854 (2005).
- Haenni, S., et al. Analysis of C. elegans intestinal gene expression and polyadenylation by fluorescence-activated nuclei sorting and 3'-end-seq. Nucleic Acids Research. 40 (13), 6304-6318 (2012).
- Zhang, S., Banerjee, D., Kuhn, J. R. Isolation and culture of larval cells from C. elegans. PLoS One. 6 (4), 19505(2011).
- Emmert-Buck, M. R., et al. Laser capture microdissection. Science. 274 (5289), 998-1001 (1996).
- Hashimshony, T., et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biology. 17, 77(2016).
- Yanai, I., Hashimshony, T. CEL-Seq2-single-cell RNA sequencing by multiplexed linear amplification. Methods in Molecular Biology. 1979, 45-56 (2019).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Ceron, J. Basic Caenorhabditis elegans methods: synchronization and observation. Journal of Visualized Experiments: JoVE. (64), e4019(2012).
- Baugh, L. R. To grow or not to grow: nutritional control of development during Caenorhabditis elegans L1 arrest. Genetics. 194 (3), 539-555 (2013).
- Baugh, L. R., Hu, P. J. Starvation responses throughout the Caenorhabditis elegans life cycle. Genetics. 216 (4), 837-878 (2020).
- Pepper, A. S., Killian, D. J., Hubbard, E. J. Genetic analysis of Caenorhabditis elegans glp-1 mutants suggests receptor interaction or competition. Genetics. 163 (1), 115-132 (2003).
- Mok, D. Z., Sternberg, P. W., Inoue, T. Morphologically defined sub-stages of C. elegans vulval development in the fourth larval stage. BMC Developmental Biology. 15, 26(2015).
- Lytal, N., Ran, D., An, L. Normalization methods on single-cell RNA-seq data: an empirical survey. Frontiers in Genetics. 11, 41(2020).
- Vieth, B., Ziegenhain, C., Parekh, S., Enard, W., Hellmann, I. powsimR: power analysis for bulk and single cell RNA-seq experiments. Bioinformatics. 33 (21), 3486-3488 (2017).
- Bolkova, J., Lanctot, C. Quantitative gene expression analysis in Caenorhabditis elegans using single molecule RNA FISH. Methods. 98, 42-49 (2016).
- Ji, N., van Oudenaarden, A. Single molecule fluorescent in situ hybridization (smFISH) of C. elegans worms and embryos. WormBook. , 1-16 (2012).
- Lee, C., et al. Single-molecule RNA fluorescence in situ hybridization (smFISH) in Caenorhabditis elegans. Bio-Protocol. 7 (12), 2357(2017).
- Baird, S. E., Chamberlin, H. M. Caenorhabditis briggsae methods. WormBook. , 1-9 (2006).
- Felix, M. A. Oscheius tipulae. WormBook. , 1-8 (2006).
- Han, Z., et al. Improving transgenesis efficiency and CRISPR-associated tools through codon optimization and native intron addition in Pristionchus nematodes. Genetics. 216 (4), 947-956 (2020).
- Fernandes Povoa, E. E., Ebbing, A. L. P., Betist, M. C., vander Veen, C., Korswagen, H. C. An optimized dissociation protocol for FACS-based isolation of rare cell types from Caenorhabditis elegans L1 larvae. MethodsX. 7, 100922(2020).
- Han, M., Wei, G., McManus, C. E., Hillier, L. W., Reinke, V. Isolated C. elegans germ nuclei exhibit distinct genomic profiles of histone modification and gene expression. BMC Genomics. 20 (1), 500(2019).
- Steiner, F. A., Henikoff, S. Cell type-specific affinity purification of nuclei for chromatin profiling in whole animals. Methods in Molecular Biology. 1228, 3-14 (2015).
- Ebbing, A. Spatial transcriptomics of C. elegans males and hermaphrodites identifies sex-specific differences in gene expression patterns. Developmental Cell. 47 (6), 801-813 (2018).
- Rödelsperger, C., et al. Spatial transcriptomics of nematodes identifies sperm cells as a source of genomic novelty and rapid evolution. Molecular Biology and Evolution. 38 (1), 229-243 (2021).
- Amini, P., et al. An optimised protocol for isolation of RNA from small sections of laser-capture microdissected FFPE tissue amenable for next-generation sequencing. BMC Molecular Biology. 18 (1), 22(2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon