Method Article
Laser-Mikrodissektion für speziesunabhängige Einzelgewebeanwendungen
In diesem Artikel
Zusammenfassung
Es wird ein Protokoll beschrieben, das Laser-Mikrodissektion verwendet, um einzelne Nematodengewebe für die RNA-Sequenzierung zu isolieren. Das Protokoll erfordert keine artspezifischen genetischen Toolkits, so dass Genexpressionsprofile zwischen verschiedenen Spezies auf der Ebene von Einzelgewebeproben verglichen werden können.
Zusammenfassung
Einzelzellmethoden haben die Analyse der Transkriptome bestimmter Zelltypen revolutioniert. Sie erfordern jedoch oft speziesspezifische genetische "Toolkits", wie z.B. Promotoren, die die gewebespezifische Expression fluoreszierender Proteine vorantreiben. Darüber hinaus entfernen Protokolle, die Gewebe stören, um einzelne Zellen zu isolieren, Zellen aus ihrer nativen Umgebung (z. B. Signalisierung von Nachbarn) und können zu Stressreaktionen oder anderen Unterschieden von nativen Genexpressionszuständen führen. Im vorliegenden Protokoll ist die Lasermikrodissektion (LMD) optimiert, um einzelne Nematodenschwanzspitzen für die Untersuchung der Genexpression während der Morphogenese der männlichen Schwanzspitze zu isolieren.
LMD ermöglicht die Isolierung eines Teils des Tieres ohne zelluläre Störung oder artspezifische Toolkits und ist somit auf jede Art anwendbar. Anschließend können einzellige RNA-seq-Bibliotheksvorbereitungsprotokolle wie CEL-Seq2 auf LMD-isolierte Einzelgewebe angewendet und mithilfe von Standardpipelines analysiert werden, vorausgesetzt, dass ein gut annotiertes Genom oder Transkriptom für die Spezies verfügbar ist. Solche Daten können verwendet werden, um festzustellen, wie konserviert oder unterschiedlich die Transkriptome sind, die der Entwicklung dieses Gewebes bei verschiedenen Arten zugrunde liegen.
Zu den Einschränkungen gehört die Möglichkeit, das interessierende Gewebe und die Stichprobengröße auszuschneiden. Eine Leistungsanalyse zeigt, dass nur 70 Heckspitzen pro Bedingung für 80% Leistung benötigt werden. Eine enge Synchronisation der Entwicklung ist erforderlich, um diese Anzahl von Tieren im gleichen Entwicklungsstadium zu erhalten. Somit wird auch eine Methode beschrieben, Tiere in 1-Stunden-Intervallen zu synchronisieren.
Einleitung
Nematoden – insbesondere die Rhabditiden-Nematoden, die mit dem Modellsystem Caenorhabditis elegans verwandt sind – sind aus vielen Gründen eine wunderbare Gruppe von Tieren für die evolutionäre Entwicklungsbiologie (EDB) 1,2. Zu den Vorteilen gehören die geringe Anzahl von Zellen, definierte und konsistente Zelllinien, Transparenz und einfache Kultur und Haltung. Es gibt auch viele Ressourcen, darunter hochwertige Genome für mehrere Arten und für C. elegans, umfangreiche molekulargenetische Werkzeuge und Wissen über Entwicklung, Genetik, Anatomie und Physiologie 3,4,5,6.
Wie bei vielen anderen Organismen hat die Fähigkeit, die Transkriptomdynamik in einzelnen Geweben oder einzelnen Zellen zu charakterisieren, die Analyse der Entwicklung in C. elegans 7,8,9,10 revolutioniert. In der Lage zu sein, einzellige Transkriptome über Nematoden hinweg zu vergleichen, würde EDB in ähnlicher Weise mit diesen Organismen transformieren. Zum Beispiel würden solche Vergleiche Aufschluss darüber geben, wie sich genregulatorische Netzwerke für Charaktere (Merkmale) entwickelt haben, die konserviert wurden, für Charaktere, die divergiert sind, oder für Charaktere, die sich unabhängig voneinander entwickelt haben.
Die Isolierung bestimmter Gewebe oder Zellen von Nematoden ist jedoch eine der großen Herausforderungen. Für viele Organismen können einzelne Zellen aus Geweben dissoziiert und unvoreingenommen geerntet oder mit gewebespezifischer Expression eines fluoreszierenden Proteins markiert und durch fluoreszenzaktivierte Zellsortierung (FACS)11 sortiert werden. Bei C. elegans war die Hochdurchsatzisolierung (HTP) von Zellen hauptsächlich auf Embryonen beschränkt, da die zähe äußere Kutikula (und das hydrostatische Skelett) die Zellisolierung von Larven und Erwachsenen behindert hat. Um diese Herausforderung zu umgehen, haben einige Methoden genetische Werkzeuge in ganzen C. elegans-Würmern eingesetzt, wie z.B. gewebespezifisches mRNA-Tagging12 und differentielle Expressionsvergleiche zwischen Wildtyp und Mutanten, die einen Zelltyp13 beeinflussen. Neuere Methoden haben die Herausforderung überwunden, indem sie die Kutikula aufgelöst haben, um Kerne 14 oder ganze Zellen 8,9,15 zu isolieren. Zellisolierung und Zellkultur haben jedoch die offensichtlichen Nachteile, dass Zellen aus ihrem natürlichen Entwicklungs- oder anatomischen Kontext entfernt werden - z. B. weg von der Zell-Zell-Signalisierung und dem Kontakt mit der extrazellulären Matrix -, von denen erwartet wird, dass sie das Genexpressionsprofilbeeinflussen 15. Darüber hinaus sind die genetischen Werkzeuge und gewebespezifischen Marker artspezifisch (d.h. sie können nur in C. elegans verwendet werden).
LMD bietet eine alternative Methode zur Isolierung von Geweben, ohne den natürlichen Kontext von Zellen zu stören. Bezeichnend für EDB ist, dass LMD es auch ermöglicht, Transkriptome aus homologen Geweben verschiedener Spezies zu vergleichen, ohne dass speziesspezifische genetische Toolkits erforderlich sind, wenn Genom- oder ganze Transkriptomsequenzen dieser Spezies verfügbar sind. LMD beinhaltet das Targeting von Geweben durch direkte mikroskopische Beobachtung und die Verwendung eines Laser-Mikrostrahls - integriert in die Optik des Mikroskops -, um das Gewebe von Interesse auszuschneiden und zu ernten (einzufangen) 16. Einschränkungen von LMD sind, dass es für sehr HTP-Ansätze nicht förderlich ist (obwohl die Transkriptionsprofile für Schwanzspitzen, wie in diesem Protokoll beschrieben, mit ~ 70 Proben robust waren), bestimmte Proben könnten schwierig zu sezieren sein, und Schnitte sind auf die Präzision des Lasers beschränkt und was im Mikroskop visualisiert werden kann.
Der Zweck des vorliegenden Protokolls ist es, zu beschreiben, wie LMD, gefolgt von RNA-Seq aus Einzelgewebe, verwendet werden kann, um stadien- und gewebespezifische Transkriptomdaten von Nematoden zu erhalten. Insbesondere zeigt es LMD zur Isolierung von Schwanzspitzen aus Larven der vierten Stufe (L4) von C. elegans. Diese Methode kann jedoch an andere Gewebe und natürlich an verschiedene Arten angepasst werden.
Bei C. elegans gibt es 4 Zellen, die sowohl bei Männchen als auch bei Hermaphroditen die Schwanzspitze bilden. Während des L4-Stadiums bei Männern - aber nicht bei Hermaphroditen - verändern die Schwanzspitzenzellen ihre Form und wandern anterior und innerlich. Dieser Prozess tritt auch bei einigen, aber nicht allen anderen Rhabditiden-Nematodenarten auf. Daher ist die Schwanzspitze ein gutes Modell für die Evolution der sexuell-dimorphen Morphogenese. Aufgrund ihrer Position ist die Heckspitze auch leicht mit LMD zu isolieren.
Um Transkriptomprofile von Schwanzspitzen zu erhalten, verwendet das vorliegende Protokoll CEL-Seq2, eine RNA-Seq-Methode, die für einzelne Zellen17,18 entwickelt wurde. Diese Methode hat mehrere Vorteile für LMD-abgeleitete Gewebe. CEL-Seq2 ist hochempfindlich und effizient und verwendet eindeutige molekulare Identifikatoren (UMIs), um eine einfache Quantifizierung von mRNA-Lesevorgängen zu ermöglichen, In-vitro-Transkription, um eine lineare Amplifikation zu gewährleisten, und Barcodes, die das Multiplexen einzelner Gewebeproben ermöglichen. Die einzige Einschränkung von CEL-Seq2 besteht darin, dass wiederhergestellte Lesevorgänge auf das 3'-Ende von mRNAs verzerrt sind und die meisten Isoformen daher nicht unterschieden werden können.
Protokoll
1. Wurmsynchronisation
HINWEIS: Im Folgenden werden zwei Methoden beschrieben, um die Entwicklung von C. elegans und anderen rhabditidischen Arten zu synchronisieren.
- Synchronisieren Sie durch den ersten Larvenstadium (L1) nach der alkalischen Hypochloritbehandlung (Bleichmittel).
HINWEIS: Diese Methode wurde zuvor ausführlichbeschrieben 19. Diese Methode beruht auf zwei Merkmalen von C. elegans , die auch für mehrere andere Rhabditid-Arten gelten: (1) Die Eierschale ist resistent gegen Bleichmittel, während die Kutikula, die erwachsene und Larvenwürmer umgibt, dies nicht tut. (2) Larven im ersten Stadium halten die Entwicklung fest, wenn sie ohne Nahrung gehaltenwerden 20.- Behandeln Sie gravide Hermaphroditen (oder Frauen) mit einer verdünnten Bleichlösung, um ihre Nagelhaut aufzubrechen und Embryonen freizusetzen.
- Entfernen Sie die Embryonen aus dem Bleichmittel und bewahren Sie sie ohne Nahrung auf, bis alle L1 geschlüpft sind.
- Legen Sie die verhafteten L1 auf Lebensmittel, wo alle die Entwicklung ungefähr zur gleichen Zeit wieder aufnehmen.
HINWEIS: Der Ausstieg aus der L1-Verhaftung kann innerhalb einer Stunde erfolgen.
- Synchronisation mit der "Hatch-off"-Methode (hier verwendet; Abbildung 1 oben):
HINWEIS: Die Hatch-Off-Methode ermöglicht eine enge Synchronisation ohne Unterbrechung der Entwicklung (L1-Arrest wirkt sich auch auf die Entwicklung späterer Stadien21 aus). Das Protokoll ist von Pepper et al.22 adaptiert. Das Ziel dieser Methode ist es, L1, die über einen bestimmten Zeitraum geschlüpft sind, von einer Platte zu sammeln, die nur Embryonen enthält.- Wähle Mütter: Am Abend vor dem Hatch-Off pflücken Sie ~ 30 gravide Hermaphroditen auf eine Platte, die mit E. coli OP50 gesät ist.
- Bei 25 °C für die Eiablage über Nacht inkubieren.
HINWEIS: Wählen Sie eine Platte ohne Risse oder Blasen, auf der Würmer stecken bleiben könnten. Vermeiden Sie Platten mit einem sehr dicken Bakterienrasen, da es schwierig sein wird, alle Würmer später zu entfernen. Wenn Sie mit einem temperaturempfindlichen Stamm arbeiten, passen Sie die Eiablagezeit an, um eine längere Embryogenese zu berücksichtigen. Wählen Sie Mütter in der maximalen Eiablagephase. - Entfernen Sie Mütter und Larven: Am nächsten Morgen unter dem Seziermikroskop (20-fache Vergrößerung) pipettieren Sie vorsichtig 1-2 ml M9-Puffer gegen die Wand der Platte, ohne zu spritzen; Wirbeln Sie die Platte, um die Würmer zu entfernen.
- Entfernen und entsorgen Sie alle Flüssigkeiten und Würmer, indem Sie die Pipettenspitze an die Wand der Platte am Rand des Agars legen, um zu vermeiden, dass Löcher stechen. Stellen Sie sicher, dass keine Würmer (und nur Eier / Embryonen) auf dem Teller verbleiben, insbesondere keine L1s; Andernfalls wiederholen Sie die Wäsche.
- Legen Sie die Platte für 1 h auf 25 °C und warten Sie, bis einige L1 schlüpfen.
- Sammeln Sie frisch geschlüpfte L1s: Lassen Sie vorsichtig 1 ml M9-Puffer auf den Agar fallen. Schwenken Sie die Platte, um L1, aber nicht Embryonen zu entfernen. Pipettieren Sie vorsichtig Puffer und Würmer in ein 1 ml Zentrifugenröhrchen.
- Zentrifen Sie das Röhrchen für 1 min bei ~18.000 × g. Entfernen Sie den Überstand.
- Pipette L1 direkt auf den Bakterienrasen einer ausgesäten Platte. Überprüfen Sie unter dem Seziermikroskop, dass keine erwachsenen Würmer oder Embryonen vorhanden sind.
- Halten Sie Würmer bei 25 °C, bis sie sich auf das gewünschte Stadium entwickelt haben.
HINWEIS: Wenn die Bedingungen optimal sind, können zwei weitere Chargen L1 von derselben Platte gesammelt werden. Überprüfen Sie die ursprüngliche Platte, um sicherzustellen, dass keine L1 vorhanden sind. Bei Bedarf erneut waschen. Wiederholen Sie die Schritte 1.2.5-1.2.9. -
Überprüfen Sie das Entwicklungstiming. Bevor Sie mit der nachgeschalteten Anwendung fortfahren, untersuchen Sie einige Würmer unter einem zusammengesetzten Mikroskop bei 400-facher Vergrößerung, um zu bestätigen, dass sie das gewünschte Entwicklungsstadium erreicht haben, hier L3.
HINWEIS: Die Migrationsentfernung von distalen Spitzenzellen oder Linkerzellen kann zusätzlich zur Vulvaentwicklung als Leitfaden verwendet werden. Für die Vulva-Entwicklung bieten Mock et al.23 einen nützlichen Leitfaden, obwohl das Timing in dieser Studie bei 20 ° C bestimmt wurde. Bei 25 °C werden Wildtyp-C. elegans nach dem Schlüpfen 24 h der L3-L4-Häutung unterzogen.
2. Sammeln von L4-Männchen und Hermaphroditen und Fixierung
- Vor der Fixierung RNAsefreies, kaltes (-20 °C), 70% Methanol herstellen.
- Beginnen Sie unter einem Seziermikroskop bei 30-50-facher Vergrößerung mit der Ernte von Männchen und Hermaphroditen von den Synchronisationsplatten auf separaten ungesäten Platten, sobald die Geschlechter unterschieden werden können (~ 21 h nach dem Schlüpfen, Abbildung 2), und pflücken Sie für 1-2 h oder bis 200 Tiere gesammelt werden.
- Halten Sie die Würmer bei 25 ° C, bis sie das gewünschte Stadium für das Experiment erreicht haben.
- Waschen Sie die Würmer mit 1-2 ml M9-Puffer von der Platte mit einer Pipettenspitze, die mit M9-Puffer vorgewaschen wurde, der 0,01% Reinigungsmittel enthält (um zu verhindern, dass Würmer an der Spitze haften bleiben).
- Überführen Sie die Würmer in ein 1 ml Zentrifugenröhrchen.
- Drehen Sie für 1 min bei 21.000 × g , um die Würmer zu pelletieren. Entfernen Sie den Überstand.
- Fügen Sie 1 ml M9-Puffer hinzu und mischen Sie, um das Pellet aufzubrechen.
- Drehen Sie für 1 min bei 21.000 × g , um die Würmer zu pelletieren. Entfernen Sie den Überstand.
- Wiederholen Sie die Wäsche.
- Fügen Sie 1 ml eiskaltes 70% Methanol hinzu und mischen Sie gut.
- Drehen Sie für 1 min bei 21.000 × g , um die Würmer zu pelletieren. Entfernen Sie den Überstand.
- Wiederholen Sie die Schritte 2.10 und 2.11.
- Fügen Sie 500 μL 70% Methanol hinzu, mischen Sie und lagern Sie es bei 4 °C für 1 h bis über Nacht.
3. Laser-Mikrodissektion
HINWEIS: Verwenden Sie von nun an RNase-freie Reagenzien und Verbrauchsmaterialien. Verwenden Sie Filtertipps.
- Wenn die CEL-Seq2-Methode zur Verarbeitung der Proben verwendet wird, bereiten Sie eine Mastermischung für jeden CEL-Seq2-Primer vor (Zusatztabelle S1): Pipetten Sie 2 μL CEL-Seq2-Primer, 1 μL 10 mM dNTP und 9 μL 1% β-Mercaptoethanol (in RNase-freiem Wasser) in ein markiertes 200-μL-Röhrchen.
- Montage auf Rutsche
- Unter einem Seziermikroskop werden 20 μL der fixierten Würmer (20-40 Würmer aus Schritt 2.13) auf die matte Seite eines Polyethylennaphthalat (PEN)-Membranglasobjektträgers pipettiert (wo sich die Membran befindet).
- Warten Sie, bis das Methanol verdampft ist. Verwenden Sie einen Schieberwärmer, um die Verdunstung zu beschleunigen.
HINWEIS: Zusätzliche Tropfen Methanol können aufgetragen werden, und eine Pipettenspitze wird verwendet, um die Würmer auszubreiten, wenn sie beim Trocknen zu verklumpen beginnen. Wenn sich Würmer in Klumpen befinden, können sie schwer zu sezieren sein.
- Einrichten des Mikroskops
HINWEIS: Das folgende Protokoll ist spezifisch für das in der Materialtabelle aufgeführte Instrument. Es muss angepasst werden, wenn ein anderes LMD-Mikroskop verwendet wird.- Stellen Sie einen Desktop-Luftbefeuchter hinter der Bühne an der Seite des LMD-Mikroskops auf. Stellen Sie sicher, dass der Dampf direkt auf die Bühne bläst.
HINWEIS: Der Luftbefeuchter ist hilfreich, um statische Elektrizität zu reduzieren, die sonst verhindern kann, dass der kleine Membranabschnitt in die Rohrkappe fällt. - Drehen Sie den Schlüssel für Laserleistung.
- Schalten Sie die Bühnensteuerung ein.
- Schalten Sie die Mikroskop-Steuerbox ein.
- Öffnen Sie die Laser-Mikrodissektionssoftware .
- Entfernen Sie die Kunststoffabschirmung über der Bühne.
- Klicken Sie auf die Schaltfläche Entladen mit dem Aufwärtspfeil, um die Membranschienen zu laden.
- Stellen Sie sicher, dass der Objektträger vollständig trocken ist, drehen Sie ihn um, so dass die Membran nach unten zeigt.
- Fügen Sie die Folie ein, und klicken Sie im Fenster "Muster ändern" auf "Weiter".
- Ersetzen Sie die Kunststoffabschirmung (plastic shield).
- Wählen Sie unten auf dem Bildschirm aus, welcher Folienhalter die Folie enthält.
- Um die Rohre zu laden, klicken Sie auf die Schaltfläche Entladen mit dem Abwärtspfeil.
- Ziehen Sie das Fach heraus und entfernen Sie den Rohrblock.
HINWEIS: Der für dieses Experiment verwendete Röhrchenblock ist für 500 μL PCR-Röhren. - Stecken Sie die Röhrchenkappen von 500 μL PCR-Röhrchen in den Halter und falten Sie das Röhrchen darunter.
- Bringen Sie den Block in das Fach zurück und schieben Sie das Fach zurück in den Mikroskoptisch.
- Wählen Sie im Popup-Fenster des Change Collector Device die Option PCR-Röhren aus und klicken Sie auf OK.
- Klicken Sie auf die leere Röhrenposition unten links auf dem Bildschirm unter den Röhrenkappen des Kollektorgeräts.
- Wählen Sie im Bedienfeld des Mikroskops TL-BF für die Hellfeldbeleuchtung des Durchlichts aus.
- Stellen Sie einen Desktop-Luftbefeuchter hinter der Bühne an der Seite des LMD-Mikroskops auf. Stellen Sie sicher, dass der Dampf direkt auf die Bühne bläst.
- Schneiden
HINWEIS: Dieses Protokoll ist spezifisch für das in der Materialtabelle aufgeführte Instrument.- Stellen Sie mit dem 2,5-fachen Objektiv den Fokus ein, bis die Würmer und die Struktur der Membran sichtbar sind.
- Wechseln Sie zum 20-fachen Objektiv.
- Verschieben der Bühne in einen Bereich ohne Würmer Stellen Sie den Fokus so ein, dass die blasenartigen Strukturen in der Membran eine gelbliche Farbe haben (Abbildung 3A,B), um den Laser auf die richtige Fokusebene zu fokussieren.
- Stellen Sie die Laserparameter ein; Für Heckspitzen beginnen Sie mit Power 45, Aperture 30 und Speed 20.
- Wählen Sie im Bedienfeld "Lasersteuerung" die Option "Kalibrieren". Folgen Sie den Anweisungen.
HINWEIS: Das Instrument führt diesen Schritt automatisch aus. Dadurch wird sichergestellt, dass eine mit der Maus auf dem Bildschirm gezeichnete Form mit der vom Laser ausgeschnittenen Form identisch ist. - Klicken Sie unten auf dem Bildschirm an den Röhrenkappen des Kollektorgeräts auf Position A.
- Wählen Sie auf der rechten Seite des Bildschirms eine einzelne Form | Zeichnen + Schneiden. Wählen Sie auf der linken Seite des Bildschirms PtoP aus.
- Zeichnen Sie eine Linie.
- Klicken Sie auf Start Cut , damit der Laser die Membran durchschneidet.
HINWEIS: Es kann auch eine Linie in das Glas ätzen. - Wenn dieser Testschnitt gut aussieht (die Membran ist geschnitten, die Schnittkanten sehen glatt aus), fahren Sie mit dem nächsten Schritt fort. Andernfalls passen Sie den Fokus an und schneiden Sie eine andere Linie aus.
- Finde einen Wurm. Wechseln Sie zu Move + Cut und verwenden Sie die Maus, um den Schwanz zu durchschneiden.
HINWEIS: Wenn der Laser den Schwanz nicht durchschneidet, passen Sie den Fokus an und erhöhen Sie die Laserleistung. Für dickeres Gewebe muss die Laserleistung möglicherweise auf 60 eingestellt werden. - Speichern Sie die Parameter: Registerkarte Datei | Anwendungskonfiguration speichern; für den späteren Abruf die Anwendungskonfiguration wiederherstellen.
- Um die Probe zu erfassen, wechseln Sie mit der Funktion "PtoP" zur Einstellung "Zeichnen + Schneiden" und zeichnen Sie eine Form, um den Schnitt eines Membranabschnitts abzuschließen (Abbildung 3C).
HINWEIS: Größere Membranabschnitte und Abschnitte, die wie Rechtecke oder Dreiecke anstelle von Kreisen oder Ovalen geformt sind, sind in der Kollektorrohrkappe leichter zu finden. - Wählen Sie die nächste Röhre unter Collector Device Tube Cap am unteren Bildschirmrand aus und schneiden Sie die nächste Heckspitze ab.
- Sobald vier Schwänze geschnitten sind, entladen Sie das Rohrgestell (klicken Sie auf Entladen mit Pfeil nach unten) und suchen Sie die Membranabschnitte unter einem Seziermikroskop (Abbildung 3D).
HINWEIS: Die Abschnitte können sich in der Mitte der Rohrkappe befinden oder an der Seite der Kappe haften. - Fahren Sie mit der nachgelagerten Anwendung fort. Für CEL-Seq2 pipettieren Sie 1,2 μL eines CEL-Seq2-Primer-Mastermixes (aus Schritt 3.1) direkt auf die Probe.
- Schließen Sie das Röhrchen, beschriften Sie es mit der Primernummer und legen Sie die Tubenkappe sofort direkt auf ein Stück Trockeneis, um die Probe schockgefrieren zu lassen und den RNA-Abbau zu verhindern.
- Laden Sie weitere Röhrchen, bringen Sie den Röhrenblock auf die Bühne zurück und schneiden Sie weitere Proben. Fügen Sie jeder Heckspitze eine andere CEL-Seq2-Primermischung hinzu.
- Lagern Sie alle Rohre bei -70 °C.
4. Single-Tail-RNA-Sequenzierung mit CEL-Seq2
HINWEIS: Ausführliche Informationen zum CEL-Seq2-Protokoll finden Sie unter Yanai und Hashimshony18.
- Reinigen Sie den Labortisch mit RNase-Dekontaminationslösung, um den RNA-Abbau zu verhindern.
- Bereiten Sie Master-Mixe vor und bewahren Sie sie auf Eis auf.
- Bereiten Sie den Reverse-Transkriptions-Mastermix vor: 0,4 μL Puffer des ersten Strangs, 0,1 μL 0,1 M DTT, 0,1 μL RNase-Inhibitor und 0,1 μL Reverse-Transkriptase pro Probe.
- Es wird die Mastermischung der zweiten Strangreaktion hergestellt: 7 μL Wasser, 2,31 μL zweiter Strangpuffer, 0,23 μL dNTP, 0,08 μL E. coli-Ligase, 0,3 μL E. coli-DNA-Polymerase, 0,08 μL RNaseH pro Probe.
- Aufbrechen von Zellen und Glühen mit Primern (siehe Zusatztabelle S1 für die vollständige Liste der Primer):
- Programmieren Sie den Thermocycler und seinen Deckel auf 65 °C.
- Entnehmen Sie die Proben bei -70 °C und inkubieren Sie sie im Thermocycler für 2,5 min.
- Drehen Sie mit 21.000 × g für 30-40 s.
- 2,5 min bei 65 °C inkubieren.
- Bewegen Sie sie sofort auf Eis.
- Drehen Sie sich bei 21.000 × g für 30-40 s und bringen Sie sie auf Eis zurück.
- Umwandlung von RNA in cDNA:
- Geben Sie 0,8 μL der umgekehrten Transkriptionsmischung zu jeder Schwanzspitze.
- Bei 42 °C für 1 h inkubieren.
- Hitzeinaktivierung bei 70 °C Für 10 min.
- Bewegen Sie es sofort auf Eis.
- Fügen Sie 10 μL der zweiten Strangmischung zu jeder Schwanzspitze hinzu.
- Streichen Sie durch die Beispiele.
- Drehen Sie bei 21.000 × g für 30-40 s.
- 2 h bei 16 °C inkubieren.
- cDNA-Bereinigung:
- Erwärmen Sie die DNA-Reinigungsperlen auf Raumtemperatur.
- Sammeln Sie bis zu 40 Proben in einem 1,5 ml Zentrifugenröhrchen (bis zu 480 μL).
- Mischen Sie die Perlen, bis sie gut verteilt sind, und fügen Sie 20 μL Perlen und 100 μL Perlenbindungspuffer für jeweils 100 μL der gepoolten Probe hinzu (für 480 μL Probe 480 μL Perlenpuffer und 96 μL Perlen zu einem Endvolumen von bis zu 1.056 μL hinzufügen). Durch Pipettieren gut mischen.
- Bei Raumtemperatur 15 min inkubieren.
- Mindestens 5 Minuten auf einen Magnetständer stellen, bis die Flüssigkeit klar erscheint.
- Entfernen und verwerfen Sie alle bis auf 20 μL des Überstandes.
- 200 μL frisch zubereitetes 80% Ethanol hinzufügen.
- Inkubieren Sie für mindestens 30 s, entfernen Sie den Überstand, indem Sie ihn abpipettieren, ohne die Perlen zu stören. Verwerfen Sie den Überstand.
- Wiederholen Sie die Schritte 4.5.7 und 4.5.8 einmal.
- Trocknen Sie die Perlen für 15 Minuten an der Luft oder bis sie vollständig trocken sind.
- Resuspendieren Sie die Perlen (~ 6,4 μL) mit 6,4 μL Wasser. Mischen Sie gründlich, indem Sie die gesamte Lautstärke zehnmal nach oben und unten pipettieren.
- Bei Raumtemperatur 2 min inkubieren.
- Gehen Sie direkt zur In-vitro-Transkription (IVT).
- In-vitro-Transkription und - Fragmentierung:
- Zu dem Röhrchen, das 6,4 μL Probe und die Kügelchen enthält, wird die folgende Mischung (insgesamt 9,6 μL) zugegeben: 1,6 μL 10x T7-Puffer, 1,6 μL ATP, 1,6 μL UTP, 1,6 μL CTP und 1,6 μL GTP (jedes dNTP bei 75 mM-Konzentration) 1,6 μL T7-Enzym.
- Inkubieren Sie für 13 h bei 37 °C mit einem 4 °C Halt.
- Fügen Sie 6 μL Exonuklease-Lösung hinzu (das Endvolumen sollte 22 μL betragen).
- 15 min bei 37 °C inkubieren.
- Legen Sie das Röhrchen wieder auf Eis und fügen Sie 5,5 μL Fragmentierungspuffer (0,25 × Reaktionsvolumen) hinzu.
- 3 min bei 94 °C inkubieren.
- Bewegen Sie die Röhre sofort auf Eis und fügen Sie 2,75 μL Fragmentierungsstopppuffer hinzu (0,5 × Volumen des Fragmentierungspuffers hinzugefügt).
- Entfernen Sie die Perlen, indem Sie das Rohr mindestens 5 Minuten lang auf den Magnetständer legen, bis die Flüssigkeit klar erscheint.
- Übertragen Sie den Überstand auf eine neue Röhre.
- Amplifizierte RNA (aRNA) Bereinigung:
- Erwärmen Sie die RNA-Reinigungsperlen auf Raumtemperatur.
- Mischen Sie die Perlen, bis sie gut verteilt sind.
- Pipette 55 μL Kügelchen (1,8 × Reaktionsvolumen).
- Inkubieren Sie sie bei Raumtemperatur für 10 min.
- Legen Sie das Rohr mindestens 5 Minuten lang auf den Magnetständer, bis die Flüssigkeit klar erscheint.
- Entfernen und verwerfen Sie 80 μL des Überstandes.
- 200 μL frisch zubereitetes 70% Ethanol hinzufügen.
- Mindestens 30 s inkubieren, den Überstand durch Pipettieren entfernen, ohne die Perlen zu stören. Verwerfen Sie den Überstand.
- Wiederholen Sie die Ethanolwäsche noch zwei weitere Male.
- Trocknen Sie die Perlen für 15 Minuten an der Luft oder bis sie vollständig trocken sind.
- Suspendieren Sie die Perlen mit 7 μL Wasser. Pippen Sie die gesamte Lautstärke 10 Mal auf und ab, um sie gründlich zu mischen.
- Bei Raumtemperatur 2 min inkubieren.
- Legen Sie das Rohr mit den Perlen für 5 Minuten auf den Magnetständer, bis die Flüssigkeit klar erscheint.
- Übertragen Sie den Überstand auf eine neue Röhre.
HINWEIS: Haltepunkt: Proben können bei -70 °C aufbewahrt werden.
- Optional: Überprüfen Sie die aRNA-Menge und -Qualität mit einem automatisierten Elektrophoresesystem gemäß dem Protokoll des Herstellers.
- Bibliotheksvorbereitung:
- Zu 5 μL RNA fügen Sie 1 μL 100 μM zufälligen Hexamer RT-Primer (siehe Materialtabelle) und 0,5 μL 10 mM dNTP hinzu.
- 5 min bei 65 °C inkubieren.
- Fügen Sie 4 μL der folgenden Mischung bei Raumtemperatur hinzu: 2 μL First Strand Puffer, 1 μL 0,1 M DDT, 0,5 μL RNase-Inhibitor, 0,5 μL Reverse Transkriptase.
- Bei 25 °C 10 min inkubieren.
- Inkubieren bei 42 °C für 1 h (in einem Hybridisierungsofen oder einem vorgeheizten Thermocycler mit einem auf 50 °C eingestellten Deckel).
- Bei 70 °C 10 min inkubieren.
- Übertragen Sie 5 μL auf ein neues Röhrchen (halten Sie den Rest der Reaktion bei -20 ° C). Fügen Sie 5,5 μL Reinstwasser, 1 μL RNA PCR Primer (RP1), 1 μL indizierten RNA PCR Primer (RPIX) und 12,5 μL PCR-Mix hinzu.
- Verwenden Sie das folgende Programm auf dem Thermocycler: 30 s bei 98 °C, 11 Zyklen von: (10 s bei 98 °C, 30 s bei 60 °C, 30 s bei 72 °C), 10 min bei 72 °C, halten bei 4 °C.
HINWEIS: Haltepunkt: Proben können bei -20 °C aufbewahrt werden.
- Bibliotheksbereinigung:
- Erwärmen Sie die DNA-Reinigungsperlen auf Raumtemperatur.
- Mischen Sie die Perlen, bis sie gut verteilt sind.
- Geben Sie 25 μL der Perlen zur PCR-Reaktion. Durch Pipettieren gut mischen.
- Bei Raumtemperatur 15 min inkubieren.
- Legen Sie das Rohr mindestens 5 Minuten lang auf den Magnetständer, bis die Flüssigkeit klar erscheint.
- Entfernen und verwerfen Sie 45 μL des Überstandes.
- 200 μL frisch zubereitetes 80% Ethanol hinzufügen.
- Inkubieren Sie für mindestens 30 s, entfernen und entsorgen Sie den Überstand, ohne die Perlen zu stören.
- Wiederholen Sie die Ethanolwäsche einmal.
- Lufttrockene Perlen für 15 min oder bis sie vollständig trocken sind.
- Resuspendieren Sie mit 25 μL Wasser. Durch Pipettieren gut mischen.
- Bei Raumtemperatur 2 min inkubieren.
- Legen Sie das Rohr für 5 Minuten auf den Magnetständer, bis die Flüssigkeit klar erscheint.
- Übertragen Sie 25 μL Überstand auf eine neue Röhre.
- Wiederholen Sie die Schritte 4.10.2-4.10.10 einmal.
- Resuspendieren Sie mit 10,5 μL Wasser. Durch Pipettieren gut mischen.
- Bei Raumtemperatur 2 min inkubieren.
- Legen Sie das Rohr für 5 Minuten auf den Magnetständer, bis die Flüssigkeit klar erscheint.
- 10 μL des Überstands auf ein neues Röhrchen übertragen und bei -20 °C lagern.
- Bewerten Sie die Qualität und Quantität der Bibliothek entsprechend den Anforderungen der Sequenzierungseinrichtung.
Ergebnisse
Nach der Laser-Capture-Mikrodissektion einzelne Schwanzspitzen von Männchen und Hermaphroditen an 4 Zeitpunkten (L3 22 h nach dem Schlüpfen; L4 24, 26 und 28 h nach dem Schlüpfen) wurden für die RNA-Sequenzierung unter Verwendung des CEL-Seq2-Protokolls hergestellt. CEL-Seq2-Primer enthalten eindeutige Barcodes, mit denen Sequenzlesevorgänge aus einer bestimmten Probe (in diesem Fall einer einzelnen Schwanzspitze) bioinformatisch identifiziert werden können. Sequenzierungsdaten wurden mit dieser Methode für insgesamt 557 Schwanzspitzen (266 Hermaphroditen und 291 Männchen über 4 Entwicklungszeitpunkte, 59-78 Schwänze pro Geschlecht und Zeitpunkt) generiert. CEL-Seq2-Barcodes wurden für 97% (d. h. 543) dieser Heckspitzen wiederhergestellt (Supplemental Table S2). Für die meisten Bibliotheken lag die Wiederherstellungsrate bei 99-100%; Es waren jedoch 88% für einen männlichen Zeitpunkt. Es ist erwähnenswert, dass etwa die Hälfte der männlichen Schwanzspitzen aus den 22-, 24- und 28-Stunden-Zeitpunkten aufgrund von COVID-19-bedingten Verzögerungen für ~ 4 Monate bei -80 ° C gelagert wurden. Dies zeigt, dass es zwar ideal ist, Sequenzierungsbibliotheken kurz nach der Probenahme vorzubereiten, es jedoch möglich ist, sezierte Proben vor der Bibliotheksvorbereitung länger zu lagern.
Die CEL-Seq2-Primer fügen jedem mRNA-Transkript auch eine UMI hinzu. Dies ermöglicht die Entfernung von PCR-Duplikaten und die präzise Quantifizierung der Genexpression in der Probe. Die Anzahl der UMIs variierte dramatisch über die Schwanzspitzen hinweg (Abbildung 4; männlicher Mittelwert = 92.560; männlich min. = 155; männlich max. = 1.183.998; Hermaphroditen Mittelwert = 67.597; Hermaphrodites min. = 132; Hermaphrodites max. = 630.427). Informationen zu UMI-Zählungen pro Schwanzspitze finden Sie unter Ergänzende Tabelle S3. Aufgrund der geringen Menge an Eingangs-RNA für die Einzelzellbibliotheksvorbereitung ist bekannt, dass Einzelzellsequenzierungsdaten eine große Menge an technischem Rauschen aufweisen. Daher wird empfohlen, Proben mit sehr niedrigen oder sehr hohen UMI-Werten vor der Analysezu filtern 24.
Das R-Paket powsimR25 wurde verwendet, um die statistische Aussagekraft und die Anforderungen an die Stichprobengröße für den zuverlässigen Nachweis differentiell exprimierter (DE) Gene in Einzelzell- oder Massen-RNA-seq-Experimenten zu bewerten. Die Parameter für die Simulationen basierten auf einem Sequenzdatensatz von 70 einzelnen männlichen Schwanzspitzen (zum 24-Stunden-Zeitpunkt), der mit der hier beschriebenen Methode erhalten wurde. Die erwarteten Log-Fold-Änderungen basierten auf den Ergebnissen eines separaten RNA-Seq-Experiments, bei dem 80-100 Schwanzspitzen gepoolt wurden. Die Simulationen ergaben, dass die Single-Tail-Tip-Daten eine ausreichende Aussagekraft (True Positive Rate = TPR) haben, um DE-Gene nachzuweisen, mit Ausnahme von Genen, die einen sehr niedrigen mittleren Expressionswert haben (oben in Abbildung 5; gestrichelte Linie entspricht 80% TPR). Das Hinzufügen von mehr simulierten Schwanzspitzen pro Zeitpunkt erhöhte die Leistung für niedrig exprimierte Gene etwas. Ein ähnliches Muster wird für die False Discovery Rate (FDR) beobachtet. FDR ist hoch (>0,10) für die niedrig exprimierten Gene; Bei stärker exprimierten Genen liegt es jedoch bei oder unter dem nominalen Cutoff von 0,10 (gestrichelte Linie für FDR am unteren Rand von Abbildung 5). Zusammenfassend lässt sich sagen, dass eine Erhöhung der Anzahl der pro Zeitpunkt beprobten Schwänze über 70 wenig dazu beitragen würde, die FDR zu senken oder die Leistung zu erhöhen. 70 Heckspitzen bieten jedoch einen viel niedrigeren FDR und eine stärkere Leistung als 30 Heckspitzen.

Abbildung 1: Verfahrensübersicht zur Synchronisation von Caenorhabditis elegans mit der Schlupfmethode und Lasermikrodissektion von Schwanzspitzen. Abkürzungen: L1-L4 = Larvenstadien 1 bis 4; PEN = Polyethylennaphthalat; LMD = Laser-Mikrodissektion. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
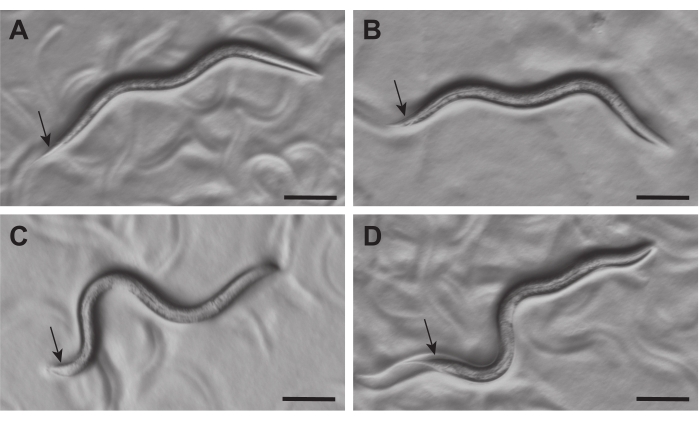
Abbildung 2: Auftreten von C. elegans L3 Hermaphroditen und Männchen unter einem Dissektionsmikroskop. Hermaphroditen (A, B) und Männchen (C, D) bei 21-23 h nach dem Schlüpfen können unter einem Seziermikroskop (~50-fache Vergrößerung) durch die Morphologie ihrer Schwänze (Pfeile) unterschieden werden. Der Schwanz von Hermaphroditen ist schmal, während der von Männchen geschwollen ist und klar erscheint. Maßstabsbalken = 0,1 mm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Aussehen der PEN-Membranschlittenstruktur und des Schneckenschwanzes. Der Fokus ist richtig für die Dissektion des Gewebes, das mit der 20x (A) und 40x (B) Linse am Mikroskop betrachtet wird. (C) Zerlegter Schwanz und teilweise ausgeschnittene PEN-Membran. Nach dem Schließen der Lücke im Schnitt fällt das Membranstück in die Rohrkappe unterhalb des Objektträgers. (D) Rohrkappe mit einem PEN-Membranabschnitt, der eine sezierte Schwanzspitze enthält. Maßstabsstäbe = 0,1 mm (A-C), 1 mm (D). Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
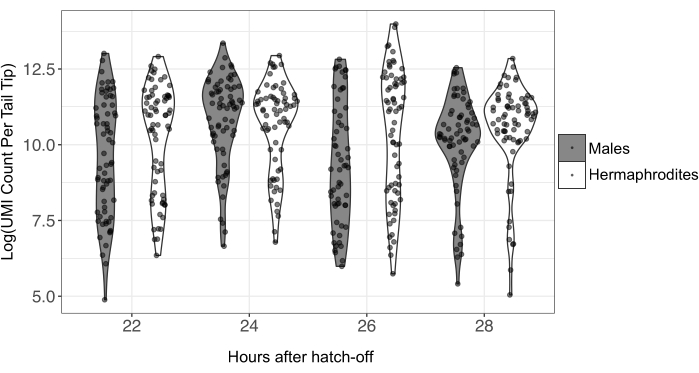
Abbildung 4: Natürliche log-transformierte UMI-Zählungen pro individueller Schwanzspitze für verschiedene Zeitpunkte und Geschlechter. RNA aus einzelnen Schwänzen wurde für die Sequenzierung mit der CEL-Seq2-Methode hergestellt; Insgesamt wurden 557 Schwänze sequenziert, mit 59-78 Schwänzen pro Geschlecht und Zeitpunkt. Extrem niedrige und hohe UMI-Ausreißer würden vor der Analyse aus den Daten entfernt. Abkürzung: UMI = Unique Molecular Identifier. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 5: Ergebnisse einer a posteriori Leistungsanalyse mittels Simulationen mit powsimR. Die powsimR-Software bestimmt die Anzahl der unabhängigen Proben, die zum Nachweis von DE-Genen auf verschiedenen Expressionsebenen erforderlich sind. Gene werden durch den Mittelausdruck zusammengefasst, der als natürliches Protokoll der UMI-Zählungen transformiert wird. (A) Power (TPR) zum Nachweis von DE-Genen zwischen zwei Bedingungen (hier männlich vs. Hermaphrodit) für vier verschiedene Simulationen (verschiedenfarbige Grafiken) mit unterschiedlichen Probengrößen (Anzahl der einzelnen Schwanzspitzen) pro Zustand. Die gestrichelte Linie zeigt 80% TPR an. (B) FDR in den gleichen vier Simulationen wie in (A), gestrichelte Linie, die 10% FDR anzeigt. Die Grafiken zeigen, dass eine Stichprobengröße von 70 Schwanzspitzen (grün) pro Bedingung ausreicht, um DE-Gene nachzuweisen, mit Ausnahme von Genen mit sehr niedrigen Expressionsniveaus. Das heißt, die Leistungs- und Falschentdeckungsrate für solche Gene kann nicht wesentlich verbessert werden, indem die Stichprobengröße über 70 hinaus erhöht wird. Abkürzungen: DE = differentiell ausgedrückt; UMI = eindeutiger molekularer Identifikator; TPR = True Positive Rate; FDR = False-Discovery-Rate. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Ergänzende Tabelle S1: Sequenzen von Primern, die im CEL-Seq2-Protokoll verwendet werden. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzungstabelle S2: Beprobte versus wiedergewonnene einzelne Schwanzspitzen. CEL-Seq2-Primer enthalten eindeutige Barcodes, mit denen Sequenzierungswerte aus jeder Probe bioinformatisch identifiziert werden können. Die Messwerte wurden aus 97% der für die Sequenzierung vorbereiteten Schwanzspitzenproben gewonnen. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Tabelle S3: UMI-Zählungen aller Proben mit wiedergefundenen Barcodes, wie in der Ergänzungstabelle S2 angegeben. Abkürzung: UMI = Unique Molecular Identifier. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Kritische Schritte der Methode
Bei korrekter Durchführung erhält die hier beschriebene Methode robuste RNA-Profile mit einer relativ kleinen Anzahl laserdissektierter Proben (70 Schwanzspitzen in diesem Beispiel). Für Proben von sich entwickelnden Tieren ist jedoch eine enge Synchronisation entscheidend, um die Variabilität zwischen den Proben zu reduzieren. Aus diesem Grund empfiehlt das Protokoll die Hatch-Off-Methode für die Wurmsynchronisation. Hier kann der Forscher den Altersunterschied zwischen Individuen (1 h im vorliegenden Protokoll) bestimmen und genau kontrollieren. Darüber hinaus ist die Hatch-Off-Methode auf jede Spezies anwendbar, auch wenn die Embryonen empfindlich auf Bleichmittel reagieren, L1 nicht festhalten oder die Erholung von L1-Arrest variabel ist. Für eine erfolgreiche Synchronisation durch Schlüpfen sind die Waschschritte entscheidend: Alle Erwachsenen und Larven müssen zu Beginn der Schlupfzeit entfernt werden, und am Ende der Schlupfzeit sollten keine Embryonen zusammen mit dem frisch geschlüpften L1 abgewaschen werden. Dies gelingt nur, wenn die Agaroberfläche der Platte durch Risse, Löcher oder Blasen unbeschädigt ist, der Bakterienrasen auf der Platte frisch und nicht zu dick ist und die Flüssigkeit nur sehr schonend zugegeben und gerührt wird.
Sollen Daten für Männchen und Hermaphroditen/Weibchen getrennt gewonnen werden, ist auch eine zuverlässige Identifizierung der Geschlechter wichtig. Die Unterscheidung von L3-Larven nach Geschlecht (siehe Abbildung 2) erfordert Erfahrung. Es wird empfohlen, L3-Männchen und Hermaphroditen / Weibchen zu pflücken und die Erfolgsrate zu überprüfen, nachdem sich die Tiere zu Erwachsenen entwickelt haben und die Geschlechter leicht zu unterscheiden sind. Nach Einzelgewebe-RNA-Seq können die Ausreißer auch durch Hauptkomponentenanalyse identifiziert und gegebenenfalls entfernt werden.
Für eine erfolgreiche Rückgewinnung von lasergeschnittenen Proben ist es wichtig, statische Elektrizität so weit wie möglich zu reduzieren. Geladene PEN-Membranstücke fallen oft nicht in die Röhrenkappe, sondern haften am Objektträger oder einem anderen Teil des Mikroskops. Ein Mittel ist die Erhöhung der Luftfeuchtigkeit im Raum und speziell um das Mikroskop herum, indem ein kleiner Luftbefeuchter neben der Bühne platziert wird. Zusätzlich können die Membranobjektträger mit UV-Licht behandelt werden. Inkubieren Sie dazu Objektträger in einer UV-C (254 nm) Vernetzungskammer und liefern Sie mindestens 1 Joule Energie oder setzen Sie die Objektträger 30 min lang dem UV-Licht in einer laminaren Luftstrombank aus.
Da das Ziel des Protokolls RNA-Seq ist, ist die Aufrechterhaltung einer RNase-freien Arbeitsumgebung von entscheidender Bedeutung. Beginnend mit der Fixierlösung sollten Reagenzien, Behälter und Verbrauchsmaterialien RNase-frei sein, die Arbeitsfläche sollte dekontaminiert werden und die Forscher sollten saubere Handschuhe tragen. Die sezierten Proben sollten so schnell wie möglich eingefroren und bis zur Weiterverarbeitung bei -70 °C aufbewahrt werden. Es wird auch empfohlen, Röhren und Spitzen mit geringer Retention für den CEL-Seq2-Teil des Protokolls zu verwenden.
Der vorliegende Artikel bietet nur einen grundlegenden Überblick über das CEL-Seq2-Protokoll, das zuvor von seinen Entwicklern mit hilfreichen Hinweisen und Tipps17,18 veröffentlicht wurde. Es wird empfohlen, diese Veröffentlichungen vor der Anwendung der CEL-Seq2-Methode zu konsultieren.
Die LMD-RNA-Seq-Daten können durch Einzelmolekül-RNA-Fluoreszenz-in-situ-Hybridisierung (smRNA FISH)26,27,28 validiert werden. smRNA FISH wurde in großem Umfang in C. elegans verwendet und ist für andere Nematodenarten zugänglich, anders als die Immunfärbung mit bestehenden Antikörpern (die möglicherweise nicht kreuzreagieren) oder die Einführung von transkriptionellen Reportern durch Transgenese. Letzteres funktioniert gut bei C. elegans und einigen verwandten Caenorhabditis-Arten 29, aber die Transgenese kann bei anderen Nematodenarten schwieriger sein30,31.
Einschränkungen der Methode
Die hier beschriebene Methode funktioniert sehr gut zum Sammeln von Schwanzspitzen, einem dünnen Gewebe am Ende eines Wurms. Das Sezieren von Geweben in der dickeren Mitte älterer Larven oder Erwachsener ist eine größere Herausforderung. Die Software des hier verwendeten Instruments enthält eine Einstellung für mehrere Schnitte, die auf später tieferen Ebenen im Gewebe platziert werden. Diese Einstellung kann zum Schneiden dickerer Bereiche des Tieres verwendet werden. Da die Würmer vor der Dissektion fixiert werden müssen, sind strukturelle Details schwer zu erkennen, was die präzise Dissektion bestimmter kleiner Strukturen verhindert. Wie oben erwähnt, ist LMD-RNA-Seq keine HTP-Methode. An einem Nachmittag können jedoch 50-70 Proben seziert werden.
Bedeutung der Methode im Vergleich zu bestehenden/alternativen Methoden
LMD-RNA-Seq kann in jeder Spezies eingesetzt werden, auch wenn keine transgenen Werkzeuge zur Verfügung stehen. Andere Methoden beruhen auf der FACS-Sortierung fluoreszierend markierter Zellen 8,9,32 oder der Isolierung markierter Kerne33,34 und erfordern daher transgene Tiere. Methoden, die Zellen im postembryonalen C. elegans dissoziieren und isolieren, neigen dazu, das Gewebe an den beiden Enden des Wurms zu übersehen (Dylan Rahe, persönliche Kommunikation). Diese Vorbehalte werden durch die Kombination von Einzelzell-RNA-Seq mit der Kryosektion ganzer Würmer (RNA-Tomographie) überwunden35. Diese Methode wurde verwendet, um die räumliche Genexpression zwischen C. elegans und einem anderen Rhabditid-Nematoden, Pristionchus pacificus36, zu vergleichen. Alternativ kann man mit formalinfixierten paraffineingebetteten (FFPE) Würmern experimentieren. Solches Material wurde erfolgreich für RNA-Seq nach LMD von Säugetiergewebeproben37 verwendet. RNA-Tomographie und LMD von FFPE-Würmern beschränken sich jedoch auf die Analyse von nur einer Handvoll Tieren. Sie eignen sich daher nicht so gut für die Untersuchung der dynamischen Genexpression in sich entwickelnden Geweben wie LMD-RNA-Seq.
Offenlegungen
Alle Autoren erklären, dass sie keine Interessenkonflikte haben.
Danksagungen
Diese Arbeit wurde durch NIH (R01GM141395) und NSF (1656736) Zuschüsse an DF und NIH Fellowship (F32GM136170) an AW finanziert. Abbildung 1 wurde mit Hilfe von BioRender.com erstellt.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.5 µM PEN membrane glass slides RNase free | Leica | 11600288 | for LMD |
| 500 µL PCR tubes (nuclease-free) | Axygen | 732-0675 | to cut the tail tips into |
| Compound microscope with 40x objective and DIC | any | to check age of worms | |
| Desktop humidifier | any | ||
| Dissection microscope with transmitted light base | any | for all worm work | |
| glass pasteur pipets | any | handle of worm pick | |
| glass slides and coverslips | any | to check age of worms | |
| LMD6 microdissection system | Leica | multiple | to cut tail tips |
| LoBind tubes 0.5 mL | Eppendorf | 22431005 | |
| M9 Buffer | Recipe in WormBook | ||
| Methanol 99.8% | Sigma | 322415 | to fix worms |
| NGM growth medium | US Biological | N1000 | Buffers and salts need to be added: Recipe in WormBook |
| P10 pipette variablle volume | e.g. Gilson | ||
| P1000 pipette variable volume | e.g. Gilson | ||
| P2 pipette variable volume | e.g. Gilson | ||
| Pipette tips 1,000 µL | any | ||
| Pipette tips 1-10 µL filtered | any | ||
| platinum iridium wire | Tritech | PT-9010 | to make worm pick |
| sterile and nuclease-free 1 mL centrfuge tubes | any | ||
| Tween 20 | Sigma | P9416 | Add a very small amount to M9 buffer to prevent worms from sticking to the pipet tips |
| vented 6 mm plastic Petri dishes | any | ||
| For CEL-Seq2 | |||
| 4200 TapeStation System with reagents for high-sensitivity RNA and DNA detection | Aligent | automated electrophoresis system | |
| AMPure XP beads | Beckman Coulter | A63880 | DNA cleanup beads |
| Bead binding buffer 20% PEG8000, 2.5 M NaCl | |||
| CEL-Seq2 primers (see Table S1) | Sigma Genosys Mastercycler Nexus GX2 Eppendorf | 6335000020 | Thermal cycler with programmable lid and block for 200 µl tubes. |
| DNA Polymerase I (E. coli) | Invitrogen | 18052-025 | |
| dNTP mix 10 mM | any | ||
| E. coli DNA ligase | Invitrogen | 18052-019 | |
| Ethanol | |||
| ExoSAP-IT For PCR Product Clean-Up | Affymetrix | 78200 | exonuclease solution |
| MEGAscript T7 Transcription Kit | Ambion | AM1334 | For step 4.6.1 |
| Nuclease-free water | any | ||
| Phusion High-Fidelity PCR Master Mix with HF Buffer | NEB | M0531 | PCR mix step 4.9.7 |
| random hexamer RT primer GCCTTGGCACCCGAGAATTCCA NNNNNN | IDT | a primer with 6 nucleotides that are random | |
| RNA Fragmentation buffer | NEB | E6150S | |
| RNA Fragmentation stop buffer | NEB | E6150S | |
| RNA PCR Index Primers (RPI1–RPI48) | Illumina, NEB, or IDT | RPIX in protocol step 4.9.7, sequences available from Illumina | |
| RNAClean XP beads | Beckman Coulter | A63987 | |
| RNase AWAY Surface Decontaminant | Thermo Scientific | 7000TS1 | or any other similar product |
| RNaseH (E. coli) | Invitrogen | 18021-071 | |
| RNaseOUT Recombinant Ribonuclease Inhibitor | Invitrogen | 10777-019 | |
| Second strand buffer | Invitrogen | 10812-014 | |
| Superscripit II | Invitrogen | 18064-014 | reverse transcriptase |
Referenzen
- Haag, E. S., Fitch, D. H. A., Delattre, M. From "the worm" to "the worms" and back again: the evolutionary developmental biology of nematodes. Genetics. 210 (2), 397-433 (2018).
- Sommer, R. J., Bumbarger, D. J. Nematode model systems in evolution and development. Wiley Interdisciplinary Reviews: Developmental Biology. 1 (3), 389-400 (2012).
- . WormBase Available from: https://wormbase.org/#012-3-6 (2022)
- . WormAtlas Available from: https://wormatlas.org (2022)
- . WormBook in Genetics Available from: https://academic-oup-com.remotexs.ntu.edu.sg/genetics/pages/wormbook (2022)
- Cao, J., et al. Comprehensive single-cell transcriptional profiling of a multicellular organism. Science. 357 (6352), 661-667 (2017).
- Kaletsky, R., Murphy, C. T. Transcriptional profiling of C. elegans adult cells and tissues with age. Methods in Molecular Biology. 2144, 177-186 (2020).
- Kaletsky, R., et al. Transcriptome analysis of adult Caenorhabditis elegans cells reveals tissue-specific gene and isoform expression. PLoS Genetics. 14 (8), 1007559 (2018).
- Packer, J. S., et al. A lineage-resolved molecular atlas of C. elegans embryogenesis at single-cell resolution. Science. 365 (6459), 1971 (2019).
- Kulkarni, A., Anderson, A. G., Merullo, D. P., Konopka, G. Beyond bulk: a review of single cell transcriptomics methodologies and applications. Current Opinion in Biotechnology. 58, 129-136 (2019).
- Von Stetina, S. E., et al. Cell-specific microarray profiling experiments reveal a comprehensive picture of gene expression in the C. elegans nervous system. Genome Biology. 8 (7), 135 (2007).
- Baugh, L. R., et al. The homeodomain protein PAL-1 specifies a lineage-specific regulatory network in the C. elegans embryo. Development. 132 (8), 1843-1854 (2005).
- Haenni, S., et al. Analysis of C. elegans intestinal gene expression and polyadenylation by fluorescence-activated nuclei sorting and 3'-end-seq. Nucleic Acids Research. 40 (13), 6304-6318 (2012).
- Zhang, S., Banerjee, D., Kuhn, J. R. Isolation and culture of larval cells from C. elegans. PLoS One. 6 (4), 19505 (2011).
- Emmert-Buck, M. R., et al. Laser capture microdissection. Science. 274 (5289), 998-1001 (1996).
- Hashimshony, T., et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biology. 17, 77 (2016).
- Yanai, I., Hashimshony, T. CEL-Seq2-single-cell RNA sequencing by multiplexed linear amplification. Methods in Molecular Biology. 1979, 45-56 (2019).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Ceron, J. Basic Caenorhabditis elegans methods: synchronization and observation. Journal of Visualized Experiments: JoVE. (64), e4019 (2012).
- Baugh, L. R. To grow or not to grow: nutritional control of development during Caenorhabditis elegans L1 arrest. Genetics. 194 (3), 539-555 (2013).
- Baugh, L. R., Hu, P. J. Starvation responses throughout the Caenorhabditis elegans life cycle. Genetics. 216 (4), 837-878 (2020).
- Pepper, A. S., Killian, D. J., Hubbard, E. J. Genetic analysis of Caenorhabditis elegans glp-1 mutants suggests receptor interaction or competition. Genetics. 163 (1), 115-132 (2003).
- Mok, D. Z., Sternberg, P. W., Inoue, T. Morphologically defined sub-stages of C. elegans vulval development in the fourth larval stage. BMC Developmental Biology. 15, 26 (2015).
- Lytal, N., Ran, D., An, L. Normalization methods on single-cell RNA-seq data: an empirical survey. Frontiers in Genetics. 11, 41 (2020).
- Vieth, B., Ziegenhain, C., Parekh, S., Enard, W., Hellmann, I. powsimR: power analysis for bulk and single cell RNA-seq experiments. Bioinformatics. 33 (21), 3486-3488 (2017).
- Bolkova, J., Lanctot, C. Quantitative gene expression analysis in Caenorhabditis elegans using single molecule RNA FISH. Methods. 98, 42-49 (2016).
- Ji, N., van Oudenaarden, A. Single molecule fluorescent in situ hybridization (smFISH) of C. elegans worms and embryos. WormBook. , 1-16 (2012).
- Lee, C., et al. Single-molecule RNA fluorescence in situ hybridization (smFISH) in Caenorhabditis elegans. Bio-Protocol. 7 (12), 2357 (2017).
- Baird, S. E., Chamberlin, H. M. Caenorhabditis briggsae methods. WormBook. , 1-9 (2006).
- Felix, M. A. Oscheius tipulae. WormBook. , 1-8 (2006).
- Han, Z., et al. Improving transgenesis efficiency and CRISPR-associated tools through codon optimization and native intron addition in Pristionchus nematodes. Genetics. 216 (4), 947-956 (2020).
- Fernandes Povoa, E. E., Ebbing, A. L. P., Betist, M. C., vander Veen, C., Korswagen, H. C. An optimized dissociation protocol for FACS-based isolation of rare cell types from Caenorhabditis elegans L1 larvae. MethodsX. 7, 100922 (2020).
- Han, M., Wei, G., McManus, C. E., Hillier, L. W., Reinke, V. Isolated C. elegans germ nuclei exhibit distinct genomic profiles of histone modification and gene expression. BMC Genomics. 20 (1), 500 (2019).
- Steiner, F. A., Henikoff, S. Cell type-specific affinity purification of nuclei for chromatin profiling in whole animals. Methods in Molecular Biology. 1228, 3-14 (2015).
- Ebbing, A. Spatial transcriptomics of C. elegans males and hermaphrodites identifies sex-specific differences in gene expression patterns. Developmental Cell. 47 (6), 801-813 (2018).
- Rödelsperger, C., et al. Spatial transcriptomics of nematodes identifies sperm cells as a source of genomic novelty and rapid evolution. Molecular Biology and Evolution. 38 (1), 229-243 (2021).
- Amini, P., et al. An optimised protocol for isolation of RNA from small sections of laser-capture microdissected FFPE tissue amenable for next-generation sequencing. BMC Molecular Biology. 18 (1), 22 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten