Method Article
Microdisección láser para aplicaciones de un solo tejido independientes de la especie
En este artículo
Resumen
Se describe un protocolo que utiliza la microdisección láser para aislar tejidos de nematodos individuales para la secuenciación de ARN. El protocolo no requiere kits de herramientas genéticas específicas de la especie, lo que permite comparar los perfiles de expresión génica entre diferentes especies a nivel de muestras de un solo tejido.
Resumen
Las metodologías unicelulares han revolucionado el análisis de los transcriptomas de tipos celulares específicos. Sin embargo, a menudo requieren "kits de herramientas" genéticos específicos de la especie, como promotores que impulsan la expresión específica de proteínas fluorescentes en los tejidos. Además, los protocolos que interrumpen los tejidos para aislar células individuales eliminan las células de su entorno nativo (por ejemplo, la señalización de los vecinos) y pueden provocar respuestas al estrés u otras diferencias con los estados de expresión génica nativa. En el presente protocolo, la microdisección láser (LMD) está optimizada para aislar las puntas individuales de la cola de los nematodos para el estudio de la expresión génica durante la morfogénesis de la punta de la cola masculina.
LMD permite el aislamiento de una porción del animal sin la necesidad de interrupción celular o kits de herramientas específicos de la especie y, por lo tanto, es aplicable a cualquier especie. Posteriormente, los protocolos de preparación de la biblioteca de ARN-seq de una sola célula, como CEL-Seq2, se pueden aplicar a tejidos individuales aislados de LMD y analizarse utilizando tuberías estándar, dado que se dispone de un genoma o transcriptoma bien anotado para la especie. Dichos datos se pueden utilizar para establecer qué tan conservados o diferentes son los transcriptomas que subyacen al desarrollo de ese tejido en diferentes especies.
Las limitaciones incluyen la capacidad de cortar el tejido de interés y el tamaño de la muestra. Un análisis de potencia muestra que se requieren tan solo 70 puntas de cola por condición para obtener un 80% de potencia. Se necesita una estrecha sincronización del desarrollo para obtener este número de animales en la misma etapa de desarrollo. Por lo tanto, también se describe un método para sincronizar animales a intervalos de 1 h.
Introducción
Los nematodos, particularmente los nematodos rabdíidos relacionados con el sistema modelo Caenorhabditis elegans, son un maravilloso grupo de animales para la biología evolutiva del desarrollo (EDB) por muchas razones 1,2. Las ventajas incluyen su pequeño número de células, linajes celulares definidos y consistentes, transparencia y facilidad de cultivo y cría. También hay muchos recursos disponibles, incluidos genomas de alta calidad para múltiples especies, y para C. elegans, amplias herramientas genéticas moleculares y conocimientos sobre desarrollo, genética, anatomía y fisiología 3,4,5,6.
Al igual que con muchos otros organismos, la capacidad de caracterizar la dinámica del transcriptoma en tejidos individuales o células individuales ha revolucionado el análisis del desarrollo en C. elegans 7,8,9,10. Ser capaz de comparar transcriptomas unicelulares entre nematodos transformaría de manera similar EDB utilizando estos organismos. Por ejemplo, tales comparaciones proporcionarían información sobre cómo las redes reguladoras de genes han evolucionado para los caracteres (rasgos) que se han conservado, para los caracteres que han divergido o para los caracteres que evolucionaron de forma independiente.
Sin embargo, aislar tejidos o células particulares de los nematodos es uno de los grandes desafíos. Para muchos organismos, las células individuales pueden disociarse de los tejidos y cosecharse de manera imparcial o pueden etiquetarse con la expresión específica del tejido de una proteína fluorescente y clasificarse mediante clasificación celular activada por fluorescencia (FACS)11. En C. elegans, el aislamiento de células de alto rendimiento (HTP) se ha limitado principalmente a embriones porque la cutícula externa dura (y el esqueleto hidrostático) ha obstaculizado el aislamiento celular de larvas y adultos. Para sortear este desafío, algunos métodos han empleado herramientas genéticas en gusanos C. elegans completos, como el etiquetado de ARNm específico del tejido12 y las comparaciones de expresión diferencial entre el tipo salvaje y los mutantes que afectan a un tipo celular13. Métodos más recientes han superado el desafío disolviendo la cutícula para aislar núcleos14 o células enteras 8,9,15. Sin embargo, el aislamiento celular y el cultivo celular tienen las desventajas obvias de que las células se eliminan de su contexto anatómico o de desarrollo natural, por ejemplo, lejos de la señalización célula-célula y el contacto con la matriz extracelular, que se espera que afecten el perfil de expresión génica15. Además, las herramientas genéticas y los marcadores específicos de tejidos son específicos de la especie (es decir, solo se pueden usar en C. elegans).
LMD proporciona un método alternativo para aislar tejidos sin interrumpir el contexto natural de las células. Significativamente para EDB, LMD también permite comparar transcriptomas de tejidos homólogos de diferentes especies sin la necesidad de kits de herramientas genéticas específicas de la especie si se dispone de secuencias del genoma o del transcriptoma completo de estas especies. LmD consiste en dirigirse a los tejidos mediante la observación microscópica directa y el uso de una microhaz láser, integrada en la óptica del microscopio, para cortar y cosechar (capturar) el tejido de interés16. Las limitaciones de LMD son que no es propicio para enfoques muy HTP (aunque los perfiles de transcripción para las puntas de la cola, como se describe en este protocolo, eran robustos con ~ 70 muestras), ciertas muestras pueden ser difíciles de diseccionar, y los cortes se limitan a la precisión del láser y lo que se puede visualizar en el microscopio.
El propósito del presente protocolo es describir cómo lmD, seguido de ARN-Seq de un solo tejido, se puede utilizar para obtener datos de transcriptoma específicos de estadios y tejidos de nematodos. Específicamente, demuestra LMD para aislar las puntas de la cola de larvas de cuarta etapa (L4) de C. elegans. Sin embargo, este método se puede adaptar a otros tejidos y, por supuesto, a diferentes especies.
En C. elegans, hay 4 células que hacen la punta de la cola tanto en machos como en hermafroditas. Durante la etapa L4 en los machos, pero no en los hermafroditas, las células de la punta de la cola cambian su forma y migran hacia arriba y hacia adentro. Este proceso también ocurre en algunas pero no en todas las demás especies de nematodos rabdítidos. Por lo tanto, la punta de la cola es un buen modelo para la evolución de la morfogénesis dimórfica sexual. Debido a su posición, la punta de la cola también es fácil de aislar por LMD.
Para obtener perfiles de transcriptoma a partir de puntas de cola, el presente protocolo utiliza CEL-Seq2, un método de ARN-seq desarrollado para células individuales17,18. Este método tiene varias ventajas para los tejidos derivados de LMD. CEL-Seq2 es altamente sensible y eficiente, utilizando identificadores moleculares únicos (UMI) para permitir la cuantificación directa de las lecturas de ARNm, la transcripción in vitro para garantizar la amplificación lineal y el código de barras que permite la multiplexación de muestras de tejido individuales. La única limitación de CEL-Seq2 es que las lecturas recuperadas están sesgadas al extremo 3' de los ARNm, y la mayoría de las isoformas no se pueden distinguir.
Protocolo
1. Sincronización de gusanos
NOTA: A continuación se describen dos métodos para sincronizar el desarrollo de C. elegans y otras especies rabdítidas.
- Sincronizar por primera parada en estadio larvario (L1) después del tratamiento con hipoclorito alcalino (lejía).
NOTA: Este método fue descrito anteriormente en detalle19. Este método se basa en dos características de C. elegans que también son ciertas para varias otras especies de rabditides: (1) La cáscara del huevo es resistente a la lejía, mientras que la cutícula que rodea a los gusanos adultos y larvales no lo es. (2) Las larvas de primera etapa detienen el desarrollo cuando se mantienen sin alimento20.- Trate a los hermafroditas grávidos (o hembras) con una solución de lejía diluida para romper su cutícula y liberar embriones.
- Retire los embriones de la lejía y manténgalos sin alimentos hasta que todo el L1 haya eclosionado.
- Coloque el L1 arrestado en la comida, donde todos reanudan el desarrollo aproximadamente al mismo tiempo.
NOTA: La salida del arresto L1 puede ocurrir dentro de una hora.
- Sincronización con el método "hatch-off" (utilizado aquí; Figura 1 arriba):
NOTA: El método de eclosión permite una sincronización estrecha sin interrupción del desarrollo (la detención L1 afecta el desarrollo incluso de etapas posteriores21). El protocolo está adaptado de Pepper et al.22. El objetivo de este método es recolectar L1 que han eclosionado durante un período específico de una placa que solo contiene embriones.- Elige madres: En la noche antes de realizar la eclosión, escoja ~ 30 hermafroditas grávidos en una placa sembrada con E. coli OP50.
- Incubar a 25 °C para la puesta de huevos durante la noche.
NOTA: Elija un plato sin grietas o burbujas donde los gusanos puedan atascarse. Evite las placas con un césped bacteriano muy grueso, ya que será difícil eliminar todos los gusanos más adelante. Si trabaja con una cepa sensible a la temperatura, ajuste el tiempo de puesta de huevos para tener en cuenta una embriogénesis más larga. Elija a las madres en la etapa máxima de puesta de huevos. - Retire las madres y las larvas: a la mañana siguiente, bajo el microscopio de disección (aumento de 20x), pipetee suavemente 1-2 ml de tampón M9 contra la pared de la placa sin chorrear; girar la placa para desalojar los gusanos.
- Retire y deseche todo el líquido y los gusanos colocando la punta de la pipeta contra la pared de la placa en el borde del agar para evitar agujeros. Verifique que no queden gusanos (y solo huevos / embriones) en el plato, especialmente no L1; de lo contrario, repita el lavado.
- Coloque la placa a 25 °C durante 1 h y espere a que eclosionen algunas L1.
- Recoja los L1 recién eclosionados: deje caer cuidadosamente el búfer M9 de 1 ml sobre el agar. Girar la placa para desalojar L1 pero no embriones. Pipete suavemente el tampón y los gusanos en un tubo de centrífuga de 1 ml.
- Centrifugar el tubo durante 1 min a ~18,000 × g. Retire el sobrenadante.
- Pipeta L1 directamente sobre el césped bacteriano de una placa sembrada. Verifique bajo el microscopio de disección que no hay gusanos o embriones adultos presentes.
- Mantenga los gusanos a 25 °C hasta que se hayan desarrollado a la etapa deseada.
NOTA: Si las condiciones son óptimas, se pueden recolectar dos lotes más de L1 de la misma placa. Inspeccione la placa inicial para asegurarse de que no haya L1 presente. Si es necesario, lávese de nuevo. Repita los pasos 1.2.5-1.2.9. -
Verifique el momento del desarrollo. Antes de proceder a la aplicación aguas abajo, inspeccione algunos gusanos bajo un microscopio compuesto a un aumento de 400x para confirmar que alcanzaron la etapa de desarrollo deseada, aquí L3.
NOTA: La distancia de migración de las células de la punta distal o las células enlazadoras se puede utilizar como guía, además del desarrollo de la vulva. Para el desarrollo de la vulva, Mock et al.23 proporcionan una guía útil, aunque el tiempo en ese estudio se determinó a 20 °C. A 25 °C, los C. elegans de tipo salvaje se someterán a la muda L3-L4 24 h después de la eclosión.
2. Recolección de machos L4 y hermafroditas y fijación
- Preparar sin RNAse, frío (-20 °C), 70% de metanol antes de la fijación.
- Bajo un microscopio de disección a un aumento de 30-50x, comience a recoger machos y hermafroditas de las placas de sincronización en placas separadas sin sembrar tan pronto como se puedan distinguir los sexos (~ 21 h después de la eclosión, Figura 2), y continúe recogiendo durante 1-2 h o hasta que se recolecten 200 animales.
- Mantenga los gusanos a 25 °C hasta que alcancen la etapa deseada para el experimento.
- Lave los gusanos de la placa con 1-2 ml de tampón M9 usando una punta de pipeta prelavada con tampón M9 que contenga detergente al 0.01% (para evitar que los gusanos se peguen a la punta).
- Transfiera los gusanos a un tubo centrífugo de 1 ml.
- Girar durante 1 min a 21.000 × g para peletizar los gusanos. Retire el sobrenadante.
- Agregue 1 ml de tampón M9 y mezcle para romper el pellet.
- Girar durante 1 min a 21.000 × g para peletizar los gusanos. Retire el sobrenadante.
- Repita el lavado.
- Añadir 1 ml de metanol helado al 70% y mezclar bien.
- Girar durante 1 min a 21.000 × g para peletizar los gusanos. Retire el sobrenadante.
- Repita los pasos 2.10 y 2.11.
- Agregue 500 μL de metanol al 70%, mezcle y almacene a 4 °C durante 1 h hasta la noche.
3. Microdisección láser
NOTA: De aquí en adelante, use reactivos y consumibles libres de RNasa; use puntas de filtro.
- Si se utiliza el método CEL-Seq2 para procesar las muestras, prepare una mezcla maestra para cada imprimación CEL-Seq2 (Tabla suplementaria S1): pipeta 2 μL de imprimación CEL-Seq2, 1 μL de 10 mM dNTP y 9 μL de 1% β-Mercaptoetanol (en agua libre de RNasa) en un tubo etiquetado de 200 μL.
- Montaje en diapositiva
- Bajo un microscopio de disección, pipetee 20 μL de los gusanos fijos (20-40 gusanos del paso 2.13) en el lado mate de un portaobjetos de vidrio de membrana de naftalato de polietileno (PEN) (donde está la membrana).
- Espere a que el metanol se evapore. Use un calentador deslizante para acelerar la evaporación.
NOTA: Se pueden aplicar gotas adicionales de metanol y se usa una punta de pipeta para extender los gusanos si comienzan a agruparse a medida que se secan. Cuando los gusanos están en grupos, pueden ser difíciles de diseccionar.
- Configuración del microscopio
NOTA: El siguiente protocolo es específico para el instrumento enumerado en la Tabla de Materiales. Debe ajustarse si se utiliza un microscopio LMD diferente.- Coloque un humidificador de escritorio detrás del escenario en el lado del microscopio LMD. Asegúrese de que el vapor sople directamente sobre el escenario.
NOTA: El humidificador es útil para reducir la electricidad estática, que de lo contrario puede evitar que la pequeña sección de la membrana caiga en la tapa del tubo. - Gire la llave para la potencia del láser.
- Encienda el control del escenario.
- Encienda la caja de control del microscopio.
- Abra el software de microdisección láser.
- Retire el escudo de plástico sobre el escenario.
- Haga clic en el botón de descarga con la flecha hacia arriba para cargar las diapositivas de membrana.
- Asegúrese de que la diapositiva esté completamente seca, voltee para que la membrana esté hacia abajo.
- Inserte la diapositiva y haga clic en continuar en la ventana de cambio de muestra .
- Reemplace el escudo de plástico.
- En la parte inferior de la pantalla, elija qué soporte deslizante contiene la diapositiva.
- Para cargar los tubos, haga clic en el botón de descarga con la flecha hacia abajo.
- Saque la bandeja y retire el bloque de tubo.
NOTA: El bloque de tubo utilizado para este experimento es para tubos de PCR de 500 μL. - Inserte las tapas de los tubos de PCR de 500 μL en el soporte y doble el tubo debajo.
- Devuelva el bloque a la bandeja y deslice la bandeja de nuevo en la etapa del microscopio.
- En la ventana emergente del dispositivo colector de cambios , seleccione Tubos pcr y haga clic en Aceptar.
- Haga clic en la ubicación vacía del tubo en la parte inferior izquierda de la pantalla debajo de las tapas del tubo del dispositivo colector.
- En el panel de control microscopio , seleccione TL-BF para la iluminación de campo brillante de luz transmitida.
- Coloque un humidificador de escritorio detrás del escenario en el lado del microscopio LMD. Asegúrese de que el vapor sople directamente sobre el escenario.
- Cortante
NOTA: Este protocolo es específico para el instrumento enumerado en la Tabla de Materiales.- Usando la lente 2.5x, ajuste el enfoque hasta que los gusanos y la estructura de la membrana sean visibles.
- Cambie a la lente 20x.
- Mueva el escenario a una región sin gusanos. Ajuste el enfoque de tal manera que las estructuras en forma de burbuja en la membrana tengan un color amarillento (Figura 3A, B) para enfocar el láser en el plano focal correcto.
- Ajuste los parámetros del láser; para las puntas de la cola, comience con Power 45, apertura 30 y velocidad 20.
- En el panel Control láser , seleccione calibrar. Siga las instrucciones.
NOTA: El instrumento realizará este paso automáticamente. Se asegurará de que una forma dibujada con el mouse en la pantalla sea idéntica a la forma recortada por el láser. - En la parte inferior de la pantalla, en las tapas del tubo del dispositivo colector, haga clic en la posición A.
- En el lado derecho de la pantalla, seleccione una sola forma | Dibujar + Cortar. En el lado izquierdo de la pantalla, seleccione PtoP.
- Dibuja una línea.
- Haga clic en Iniciar corte para que el láser corte a través de la membrana.
NOTA: También puede grabar una línea en el vidrio. - Si este corte de prueba se ve bien (la membrana está cortada, los bordes del corte se ven lisos), continúe con el siguiente paso. De lo contrario, ajuste el enfoque y corte otra línea.
- Encuentra un gusano. Cambie a Mover + Cortar y use el mouse para cortar la cola.
NOTA: Si el láser no corta la cola, ajuste el enfoque y aumente la potencia del láser. Para tejidos más gruesos, la potencia del láser puede tener que ajustarse a 60. - Guardar los parámetros: ficha Archivo | Guardar la configuración de la aplicación; para su posterior recuperación, Restaurar configuración de la aplicación.
- Para recoger la muestra, cambie a la configuración Dibujar + Cortar con la función PtoP y dibuje una forma para completar el corte de una sección de membrana (Figura 3C).
NOTA: Las secciones de membrana más grandes y las secciones con forma de rectángulos o triángulos en lugar de círculos u óvalos son más fáciles de ubicar en la tapa del tubo colector. - Seleccione el siguiente tubo en Collector Device Tube Cap en la parte inferior de la pantalla y corte la siguiente punta de la cola.
- Una vez cortadas cuatro colas, descargue el bastidor del tubo (haga clic en Descargar con la flecha hacia abajo) y busque las secciones de membrana bajo un microscopio de disección (Figura 3D).
NOTA: Las secciones pueden estar ubicadas en el centro de la tapa del tubo o pegadas al costado de la tapa. - Continúe con la aplicación descendente. Para CEL-Seq2, pipetee 1,2 μL de una mezcla maestra de imprimación CEL-Seq2 (del paso 3.1) directamente encima de la muestra.
- Cierre el tubo, etiquete con el número de imprimación e inmediatamente coloque la tapa del tubo directamente sobre un trozo de hielo seco para congelar la muestra y evitar la degradación del ARN.
- Cargue más tubos, devuelva el bloque de tubos al escenario y corte más muestras. Agregue una mezcla de imprimación CEL-Seq2 diferente a cada punta de la cola.
- Conservar todos los tubos a -70 °C.
4. Secuenciación de ARN de cola única con CEL-Seq2
NOTA: Para obtener detalles completos sobre el protocolo CEL-Seq2, consulte Yanai y Hashimshony18.
- Limpie el área del banco de laboratorio con una solución de descontaminación de RNasa para evitar la degradación del ARN.
- Prepare mezclas maestras y manténgalas en hielo.
- Preparar la mezcla maestra de transcripción inversa: 0,4 μL de tampón de primera hebra, 0,1 μL de 0,1 M de TDT, 0,1 μL de inhibidor de la RNasa y 0,1 μL de transcriptasa inversa por muestra.
- Preparar la mezcla maestra de reacción de la segunda hebra: 7 μL de agua, 2,31 μL de tampón de segunda hebra, 0,23 μL de dNTP, 0,08 μL de E. coli ligasa, 0,3 μL de E. coli ADN polimerasa, 0,08 μL de RNasaH por muestra.
- Romper las celdas abiertas y recocir con cebadores (consulte la Tabla suplementaria S1 para obtener la lista completa de cebadores):
- Programe el termociclador y su tapa a 65 °C.
- Recuperar las muestras a partir de -70 °C e incubarlas en el termociclador durante 2,5 min.
- Girar a 21.000 × g durante 30-40 s.
- Incubar a 65 °C durante 2,5 min.
- Muévelos inmediatamente al hielo.
- Girar a 21.000 × g durante 30-40 s y devolverlos al hielo.
- Conversión de ARN a ADNc:
- Agregue 0.8 μL de la mezcla de transcripción inversa a cada punta de la cola.
- Incubar a 42 °C durante 1 h.
- Inactivar térmicamente a 70 °C durante 10 min.
- Muévelo inmediatamente al hielo.
- Agregue 10 μL de la segunda mezcla de hebras a cada punta de la cola.
- Desplácese por las muestras.
- Girar a 21.000 × g durante 30-40 s.
- Incubar a 16 °C durante 2 h.
- Limpieza de ADNc:
- Precalentar las cuentas de limpieza de ADN a temperatura ambiente.
- Acumule hasta 40 muestras en un tubo centrífugo de 1,5 ml (hasta 480 μL).
- Mezcle las perlas hasta que estén bien dispersas y agregue 20 μL de perlas y 100 μL de tampón de unión de perlas por cada 100 μL de la muestra agrupada (para 480 μL de muestra agregue 480 μL de tampón de cuentas y 96 μL de perlas a un volumen final de hasta 1.056 μL). Mezclar bien mediante pipeteo.
- Incubar a temperatura ambiente durante 15 min.
- Coloque en un soporte magnético durante al menos 5 minutos hasta que el líquido parezca claro.
- Retire y deseche todos menos 20 μL del sobrenadante.
- Añadir 200 μL de etanol 80% recién preparado.
- Incubar durante al menos 30 s, retirar el sobrenadante pipeteándolo sin molestar las perlas. Deseche el sobrenadante.
- Repita los pasos 4.5.7 y 4.5.8 una vez.
- Seque al aire las perlas durante 15 minutos o hasta que estén completamente secas.
- Resuspend las perlas (~6.4 μL) con 6.4 μL de agua. Mezclar bien canalizando todo el volumen hacia arriba y hacia abajo diez veces.
- Incubar a temperatura ambiente durante 2 min.
- Vaya directamente a la transcripción in vitro (IVT).
- Transcripción y fragmentación in vitro :
- Al tubo que contiene 6,4 μL de muestra y las perlas, añadir la siguiente mezcla (9,6 μL en total): 1,6 μL de 10x T7 Buffer, 1,6 μL de ATP, 1,6 μL de UTP, 1,6 μL de CTP y 1,6 μL de GTP (cada dNTP a 75 mM concentración) 1,6 μLof T7 enzima.
- Incubar durante 13 h a 37 °C con una retención de 4 °C.
- Añadir 6 μL de solución de exonucleasa (el volumen final debe ser de 22 μL).
- Incubar durante 15 min a 37 °C.
- Vuelva a colocar el tubo sobre hielo y agregue 5,5 μL de tampón de fragmentación (0,25 × volumen de reacción).
- Incubar durante 3 min a 94 °C.
- Mueva inmediatamente el tubo al hielo y agregue 2,75 μL de búfer de parada de fragmentación (0,5 × volumen de búfer de fragmentación añadido).
- Retire las perlas colocando el tubo en el soporte magnético durante al menos 5 minutos hasta que el líquido parezca claro.
- Transfiera el sobrenadante a un tubo nuevo.
- Limpieza de ARN amplificado (aRNA):
- Precalentar las perlas de limpieza de ARN a temperatura ambiente.
- Mezcle las cuentas hasta que estén bien dispersas.
- Pipeta 55 μL de perlas (1,8 × volumen de reacción).
- Incubarlos a temperatura ambiente durante 10 min.
- Coloque el tubo en el soporte magnético durante al menos 5 minutos hasta que el líquido parezca claro.
- Retire y deseche 80 μL del sobrenadante.
- Añadir 200 μL de etanol 70% recién preparado.
- Incubar durante al menos 30 s, retirar el sobrenadante pipeteando sin molestar las perlas. Deseche el sobrenadante.
- Repita el lavado con etanol dos veces más.
- Seque al aire las perlas durante 15 minutos o hasta que estén completamente secas.
- Resuspendir las perlas con 7 μL de agua. Pipetear todo el volumen hacia arriba y hacia abajo 10 veces para mezclar bien.
- Incubar a temperatura ambiente durante 2 min.
- Coloque el tubo con las perlas en el soporte magnético durante 5 minutos hasta que el líquido parezca claro.
- Transfiera el sobrenadante a un tubo nuevo.
NOTA: Punto de parada: las muestras se pueden mantener a -70 °C.
- Opcional: Compruebe la cantidad y calidad de aRNA con un sistema de electroforesis automatizado siguiendo el protocolo del fabricante.
- Preparación de la biblioteca:
- A 5 μL de ARN, añadir 1 μL de cebador RT de hexámero aleatorio de 100 μM (véase la Tabla de Materiales) y 0,5 μL de 10 mM dNTP.
- Incubar a 65 °C durante 5 min.
- Añadir 4 μL de la siguiente mezcla a temperatura ambiente: 2 μL de tampón First Strand, 1 μL de 0,1 M DDT, 0,5 μL de inhibidor de la RNasa, 0,5 μL de transcriptasa inversa.
- Incubar a 25 °C durante 10 min.
- Incubar a 42 °C durante 1 h (en un horno de hibridación o en un termociclador precalentado con la tapa ajustada a 50 °C).
- Incubar a 70 °C durante 10 min.
- Transfiera 5 μL a un tubo nuevo (mantenga el resto de la reacción a -20 °C). Agregue 5,5 μL de agua ultrapura, 1 μL de ARN PCR Primer (RP1), 1 μL de RNA PCR Indexado (RPIX) y 12,5 μL de mezcla de PCR.
- Utilice el siguiente programa en el termociclador: 30 s a 98 °C, 11 ciclos de: (10 s a 98 °C, 30 s a 60 °C, 30 s a 72 °C), 10 min a 72 °C, mantener a 4 °C.
NOTA: Punto de parada: las muestras se pueden mantener a -20 °C.
- Limpieza de bibliotecas:
- Precalentar las cuentas de limpieza de ADN a temperatura ambiente.
- Mezcle las cuentas hasta que estén bien dispersas.
- Añadir 25 μL de las perlas a la reacción de PCR. Mezclar bien mediante pipeteo.
- Incubar a temperatura ambiente durante 15 min.
- Coloque el tubo en el soporte magnético durante al menos 5 minutos hasta que el líquido parezca claro.
- Retire y deseche 45 μL del sobrenadante.
- Añadir 200 μL de etanol 80% recién preparado.
- Incubar durante al menos 30 s, retirar y desechar el sobrenadante sin molestar las perlas.
- Repita el lavado con etanol una vez.
- Seque las perlas al aire durante 15 minutos o hasta que estén completamente secas.
- Resuspenderlos con 25 μL de agua. Mezclar bien mediante pipeteo.
- Incubar a temperatura ambiente durante 2 min.
- Coloque el tubo en el soporte magnético durante 5 minutos hasta que el líquido aparezca claro.
- Transfiera 25 μL de sobrenadante a un tubo nuevo.
- Repita los pasos 4.10.2-4.10.10 una vez.
- Resuspend con 10,5 μL de agua. Mezclar bien mediante pipeteo.
- Incubar a temperatura ambiente durante 2 min.
- Coloque el tubo en el soporte magnético durante 5 minutos hasta que el líquido aparezca claro.
- Transfiera 10 μL del sobrenadante a un nuevo tubo y guárdelo a -20 °C.
- Evaluar la calidad y cantidad de la biblioteca de acuerdo con los requisitos de la instalación de secuenciación.
Resultados
Después de la microdisección de captura láser, puntas de cola individuales de machos y hermafroditas en 4 puntos de tiempo (L3 22 h después de la eclosión; L4 24, 26 y 28 h después de la eclosión) se prepararon para la secuenciación de ARN utilizando el protocolo CEL-Seq2. Los cebadores CEL-Seq2 contienen códigos de barras únicos que permiten identificar bioinformáticamente las lecturas de secuenciación de una muestra en particular (en este caso, una punta de cola individual). Los datos de secuenciación se generaron con este método para un total de 557 puntas de cola (266 hermafroditas y 291 machos en 4 puntos de tiempo de desarrollo, 59-78 colas por sexo y punto de tiempo). Los códigos de barras CEL-Seq2 se recuperaron para el 97% (es decir, 543) de estas puntas de cola (Tabla suplementaria S2). Para la mayoría de las bibliotecas, la tasa de recuperación fue del 99-100%; sin embargo, fue del 88% para un punto de tiempo masculino. Vale la pena señalar que aproximadamente la mitad de las puntas de la cola masculina de los puntos de tiempo de 22, 24 y 28 h se almacenaron a -80 ° C durante ~ 4 meses debido a retrasos relacionados con COVID-19. Esto demuestra que, si bien es ideal preparar bibliotecas de secuenciación poco después del muestreo, es posible almacenar muestras disecadas durante más tiempo antes de la preparación de la biblioteca.
Los cebadores CEL-Seq2 también agregan una UMI a cada transcripción de ARNm. Esto permite la eliminación de duplicados de PCR y la cuantificación precisa de la expresión génica en la muestra. El número de UMI varió dramáticamente entre las puntas de la cola (Figura 4; media masculina = 92,560; macho min. = 155; macho máx. = 1,183,998; hermafroditas media = 67,597; hermafroditas min. = 132; hermafroditas máx. = 630,427). Para conocer los recuentos de UMI por punta de cola, consulte la Tabla suplementaria S3. Debido a la baja cantidad de ARN de entrada para la preparación de bibliotecas de una sola célula, se sabe que los datos de secuenciación de una sola célula tienen una gran cantidad de ruido técnico. Por lo tanto, se recomienda filtrar las muestras que tienen recuentos de UMI muy bajos o muy altos antes del análisis24.
El paquete R powsimR25 se utilizó para evaluar el poder estadístico y los requisitos de tamaño de la muestra para detectar de manera confiable genes expresados diferencialmente (DE) en experimentos de ARN-seq unicelulares o a granel. Los parámetros para las simulaciones se basaron en un conjunto de datos de secuenciación de 70 puntas de cola masculinas individuales (en el punto de tiempo de 24 h) obtenidas con el método descrito aquí. Los cambios esperados en el pliegue logarítmico se basaron en los resultados de un experimento separado de ARN-seq que agrupó 80-100 puntas de cola. Las simulaciones determinaron que los datos de punta de cola única tienen suficiente potencia (tasa positiva verdadera = TPR) para detectar genes DE, excepto los genes que tienen un valor de expresión medio muy bajo (parte superior de la Figura 5; la línea discontinua representa el 80% de TPR). Agregar más puntas de cola simuladas por punto de tiempo aumentó un poco la potencia para los genes poco expresados. Un patrón similar se observa para la tasa de descubrimiento falso (FDR). La FDR es alta (>0,10) para los genes poco expresados; sin embargo, para genes más altamente expresados, cae en o por debajo del punto de corte nominal de 0.10 (línea discontinua para FDR en la parte inferior de la Figura 5). En resumen, aumentar el número de colas muestreadas por punto de tiempo por encima de 70 haría poco para reducir el FDR o aumentar la potencia. Sin embargo, 70 puntas de cola proporcionan un FDR mucho más bajo y una potencia más fuerte que 30 puntas de cola.

Figura 1: Descripción general del procedimiento para la sincronización de Caenorhabditis elegans con el método de eclosión y microdisección láser de las puntas de la cola. Abreviaturas: L1-L4 = estadios larvales 1 a 4; PEN = naftalato de polietileno; LMD = microdisección láser. Haga clic aquí para ver una versión más grande de esta figura.
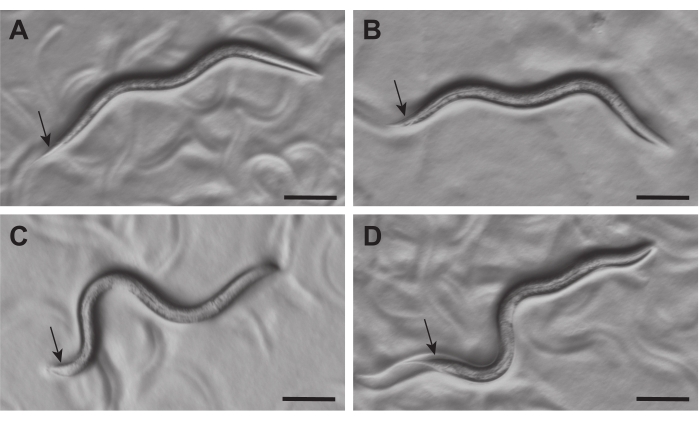
Figura 2: Aparición de hermafroditas y machos de C. elegans L3 bajo un microscopio de disección. Hermafroditas (A, B) y machos (C, D) a las 21-23 h después de la eclosión se pueden distinguir bajo un microscopio de disección (~ 50x aumento) por la morfología de sus colas (flechas). La cola de los hermafroditas es estrecha, mientras que la de los machos está hinchada y parece clara. Barras de escala = 0.1 mm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Apariencia de la estructura de deslizamiento de la membrana PEN y la cola de gusano. El enfoque es correcto para la disección del tejido visto con la lente 20x (A) y 40x (B) en el microscopio. (C) Cola diseccionada y membrana PEN parcialmente cortada. Después de cerrar el espacio en el corte, la pieza de membrana caerá en la tapa del tubo debajo de la diapositiva. (D) Tapa de tubo con una sección de membrana PEN que contiene una punta de cola diseccionada. Barras de escala = 0,1 mm (A-C), 1 mm (D). Haga clic aquí para ver una versión más grande de esta figura.
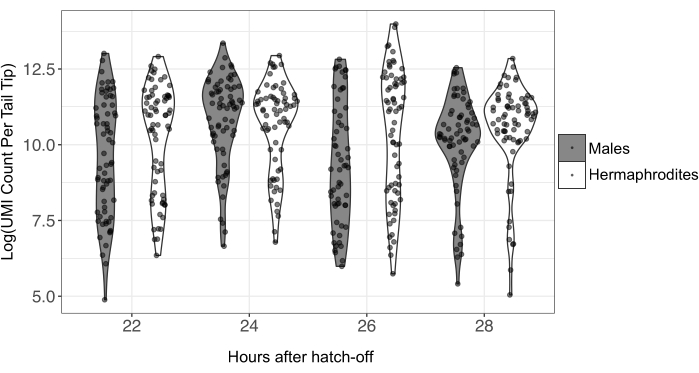
Figura 4: Recuentos naturales de UMI transformados en logarítmica por punta de cola individual para diferentes puntos de tiempo y sexos. El ARN de las colas individuales se preparó para la secuenciación utilizando el método CEL-Seq2; Se secuenciaron 557 colas en total, con 59-78 colas por sexo y punto de tiempo. Los valores atípicos de UMI extremadamente bajos y altos se eliminarían de los datos antes del análisis. Abreviatura: UMI = identificador molecular único. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Resultados de un análisis de potencia a posteriori mediante simulaciones con powsimR. El software powsimR determina el número de muestras independientes necesarias para detectar genes DE en varios niveles de expresión. Los genes se agrupan por expresión media transformada como el registro natural de los recuentos de UMI. (A) Potencia (TPR) para detectar genes DE entre dos condiciones (aquí, macho vs hermafrodita) para cuatro simulaciones diferentes (gráficos de diferentes colores) que incorporan diferentes tamaños de muestra (números de puntas de cola individuales) por condición. La línea discontinua indica un 80% de TPR. (B) FDR en las mismas cuatro simulaciones que en (A), línea discontinua que indica 10% FDR. Los gráficos muestran que un tamaño de muestra de 70 puntas de cola (verde) por condición es suficiente para detectar genes DE, excepto para genes con niveles de expresión muy bajos. Es decir, el poder y la tasa de descubrimiento falso de tales genes no se pueden mejorar en gran medida aumentando el tamaño de la muestra más allá de 70. Abreviaturas: DE = expresado diferencialmente; UMI = identificador molecular único; TPR = tasa positiva verdadera; FDR = tasa de descubrimiento falso. Haga clic aquí para ver una versión más grande de esta figura.
Tabla suplementaria S1: Secuencias de cebadores utilizados en el protocolo CEL-Seq2. Haga clic aquí para descargar este archivo.
Tabla suplementaria S2: Puntas de cola individuales muestreadas versus recuperadas. Los cebadores CEL-Seq2 contienen códigos de barras únicos que permiten identificar bioinformáticamente las lecturas de secuenciación de cada muestra. Las lecturas se recuperaron del 97% de las muestras de la punta de la cola preparadas para la secuenciación. Haga clic aquí para descargar este archivo.
Tabla suplementaria S3: Recuentos UMI de todas las muestras con códigos de barras recuperados, como se indica en la Tabla suplementaria S2. Abreviatura: UMI = identificador molecular único. Haga clic aquí para descargar este archivo.
Discusión
Pasos críticos del método
Si se realiza correctamente, el método descrito aquí obtendrá perfiles de ARN robustos con un número relativamente pequeño de muestras diseccionadas con láser (70 puntas de cola en este ejemplo). Sin embargo, para las muestras de animales en desarrollo, la sincronización estrecha es fundamental para reducir la variabilidad entre las muestras. Por esta razón, el protocolo recomienda el método de sombreado para la sincronización de gusanos. Aquí, el investigador puede determinar y controlar con precisión la diferencia de edad entre individuos (1 h en el presente protocolo). Además, el método de eclosión es aplicable a cualquier especie, incluso si los embriones son sensibles a la lejía, L1 no se detiene o la recuperación de la detención L1 es variable. Para una sincronización exitosa por eclosión, los pasos de lavado son cruciales: todos los adultos y larvas deben eliminarse al comienzo del período de eclosión, y no se deben lavar los embriones junto con el L1 recién eclosionado al final del período de eclosión. Esto solo tiene éxito si la superficie de agar de la placa no está dañada por grietas, agujeros o burbujas, el césped bacteriano en el plato es fresco y no demasiado espeso, y el líquido se agrega y agita solo muy suavemente.
Si se van a obtener datos por separado para los machos y los hermafroditas/hembras, también es importante una identificación fiable de los sexos. Distinguir las larvas L3 por sexo (ver Figura 2) requiere experiencia. Se recomienda practicar la selección de machos L3 y hermafroditas / hembras y verificar la tasa de éxito después de que los animales se hayan convertido en adultos y los sexos se distingan fácilmente. Después del ARN-Seq de un solo tejido, los valores atípicos también se pueden identificar mediante el análisis del componente principal y eliminarse, si es necesario.
Para una recuperación exitosa de muestras cortadas con láser, es importante reducir la electricidad estática tanto como sea posible. Las piezas cargadas de la membrana PEN a menudo no caen en la tapa del tubo, sino que se adhieren a la diapositiva o a cualquier otra parte del microscopio. Un remedio es elevar la humedad en la habitación y específicamente alrededor del microscopio colocando un pequeño humidificador al lado del escenario. Además, los portaobjetos de membrana se pueden tratar con luz UV. Para hacer esto, incube las diapositivas en una cámara de reticulación UV-C (254 nm) y entregue al menos 1 julio de energía, o exponga las diapositivas a la luz UV en un banco de flujo de aire laminar durante 30 minutos.
Dado que el objetivo del protocolo es RNA-Seq, mantener un entorno de trabajo libre de RNasa es fundamental. Comenzando con la solución de fijación, los reactivos, contenedores y consumibles deben estar libres de RNasa, la superficie de trabajo debe estar descontaminada y los investigadores deben usar guantes limpios. Las muestras disecadas deben congelarse lo antes posible y mantenerse a -70 °C hasta su posterior procesamiento. También se recomienda utilizar tubos y puntas de baja retención para la parte CEL-Seq2 del protocolo.
El presente artículo proporciona solo un esquema básico del protocolo CEL-Seq2, que fue publicado previamente por sus desarrolladores con notas y consejos útiles17,18. Se recomienda consultar estas publicaciones antes de utilizar el método CEL-Seq2.
Los datos LMD-RNA-Seq pueden ser validados por hibridación fluorescente in situ de una sola molécula-ARN (smRNA FISH)26,27,28. smRNA FISH se ha utilizado ampliamente en C. elegans y es susceptible a otras especies de nematodos, diferente de la inmunotinción con anticuerpos existentes (que pueden no reaccionar de forma cruzada) o la introducción de reporteros transcripcionales a través de la transgénesis. Este último funciona bien en C. elegans y algunas especies relacionadas de Caenorhabditis 29, pero la transgénesis puede ser más desafiante en otras especies de nematodos30,31.
Limitaciones del método
El método descrito aquí funciona muy bien para recolectar las puntas de la cola, un tejido delgado al final de un gusano. La disección de tejidos en el medio más grueso de larvas mayores o adultos es más desafiante. El software del instrumento utilizado aquí incluye un ajuste para múltiples cortes colocados posteriormente en niveles más profundos en el tejido. Este ajuste se puede utilizar para cortar áreas más gruesas del animal. Debido a que los gusanos deben fijarse antes de la disección, los detalles estructurales son difíciles de ver, lo que impide la disección precisa de estructuras pequeñas específicas. Como se mencionó anteriormente, LMD-RNA-Seq no es un método HTP. Sin embargo, 50-70 muestras se pueden diseccionar en una tarde.
Importancia del método con respecto a los métodos existentes/alternativos
LMD-RNA-Seq se puede utilizar en cualquier especie, incluso si no hay herramientas transgénicas disponibles. Otros métodos se basan en la clasificación FACS de células marcadas fluorescentemente 8,9,32 o el aislamiento de núcleos marcados33,34 y, por lo tanto, requieren animales transgénicos. Los métodos que disocian y aíslan las células en C. elegans postembrionario tienden a perder los tejidos en los dos extremos del gusano (Dylan Rahe, comunicación personal). Estas advertencias se superan combinando RNA-Seq unicelular con crioseccionamiento de gusanos enteros (tomografía de ARN)35. Este método se utilizó para comparar la expresión génica espacial entre C. elegans y otro nematodo rabdítido, Pristionchus pacificus36. Alternativamente, se puede experimentar con gusanos incrustados en parafina fijada en formalina (FFPE). Dicho material se ha utilizado con éxito para RNA-Seq después de LMD de muestras de tejido de mamíferos37. Sin embargo, la tomografía de ARN y la LMD de gusanos FFPE se limitan al análisis de solo un puñado de animales. Por lo tanto, no son tan adecuados para el estudio de la expresión génica dinámica en tejidos en desarrollo como LMD-RNA-Seq.
Divulgaciones
Todos los autores declaran que no tienen conflictos de intereses.
Agradecimientos
Este trabajo fue financiado por las subvenciones NIH (R01GM141395) y NSF (1656736) a DF y nih fellowship (F32GM136170) a AW. La Figura 1 fue creada con la ayuda de BioRender.com.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.5 µM PEN membrane glass slides RNase free | Leica | 11600288 | for LMD |
| 500 µL PCR tubes (nuclease-free) | Axygen | 732-0675 | to cut the tail tips into |
| Compound microscope with 40x objective and DIC | any | to check age of worms | |
| Desktop humidifier | any | ||
| Dissection microscope with transmitted light base | any | for all worm work | |
| glass pasteur pipets | any | handle of worm pick | |
| glass slides and coverslips | any | to check age of worms | |
| LMD6 microdissection system | Leica | multiple | to cut tail tips |
| LoBind tubes 0.5 mL | Eppendorf | 22431005 | |
| M9 Buffer | Recipe in WormBook | ||
| Methanol 99.8% | Sigma | 322415 | to fix worms |
| NGM growth medium | US Biological | N1000 | Buffers and salts need to be added: Recipe in WormBook |
| P10 pipette variablle volume | e.g. Gilson | ||
| P1000 pipette variable volume | e.g. Gilson | ||
| P2 pipette variable volume | e.g. Gilson | ||
| Pipette tips 1,000 µL | any | ||
| Pipette tips 1-10 µL filtered | any | ||
| platinum iridium wire | Tritech | PT-9010 | to make worm pick |
| sterile and nuclease-free 1 mL centrfuge tubes | any | ||
| Tween 20 | Sigma | P9416 | Add a very small amount to M9 buffer to prevent worms from sticking to the pipet tips |
| vented 6 mm plastic Petri dishes | any | ||
| For CEL-Seq2 | |||
| 4200 TapeStation System with reagents for high-sensitivity RNA and DNA detection | Aligent | automated electrophoresis system | |
| AMPure XP beads | Beckman Coulter | A63880 | DNA cleanup beads |
| Bead binding buffer 20% PEG8000, 2.5 M NaCl | |||
| CEL-Seq2 primers (see Table S1) | Sigma Genosys Mastercycler Nexus GX2 Eppendorf | 6335000020 | Thermal cycler with programmable lid and block for 200 µl tubes. |
| DNA Polymerase I (E. coli) | Invitrogen | 18052-025 | |
| dNTP mix 10 mM | any | ||
| E. coli DNA ligase | Invitrogen | 18052-019 | |
| Ethanol | |||
| ExoSAP-IT For PCR Product Clean-Up | Affymetrix | 78200 | exonuclease solution |
| MEGAscript T7 Transcription Kit | Ambion | AM1334 | For step 4.6.1 |
| Nuclease-free water | any | ||
| Phusion High-Fidelity PCR Master Mix with HF Buffer | NEB | M0531 | PCR mix step 4.9.7 |
| random hexamer RT primer GCCTTGGCACCCGAGAATTCCA NNNNNN | IDT | a primer with 6 nucleotides that are random | |
| RNA Fragmentation buffer | NEB | E6150S | |
| RNA Fragmentation stop buffer | NEB | E6150S | |
| RNA PCR Index Primers (RPI1–RPI48) | Illumina, NEB, or IDT | RPIX in protocol step 4.9.7, sequences available from Illumina | |
| RNAClean XP beads | Beckman Coulter | A63987 | |
| RNase AWAY Surface Decontaminant | Thermo Scientific | 7000TS1 | or any other similar product |
| RNaseH (E. coli) | Invitrogen | 18021-071 | |
| RNaseOUT Recombinant Ribonuclease Inhibitor | Invitrogen | 10777-019 | |
| Second strand buffer | Invitrogen | 10812-014 | |
| Superscripit II | Invitrogen | 18064-014 | reverse transcriptase |
Referencias
- Haag, E. S., Fitch, D. H. A., Delattre, M. From "the worm" to "the worms" and back again: the evolutionary developmental biology of nematodes. Genetics. 210 (2), 397-433 (2018).
- Sommer, R. J., Bumbarger, D. J. Nematode model systems in evolution and development. Wiley Interdisciplinary Reviews: Developmental Biology. 1 (3), 389-400 (2012).
- . WormBase Available from: https://wormbase.org/#012-3-6 (2022)
- . WormAtlas Available from: https://wormatlas.org (2022)
- . WormBook in Genetics Available from: https://academic-oup-com.remotexs.ntu.edu.sg/genetics/pages/wormbook (2022)
- Cao, J., et al. Comprehensive single-cell transcriptional profiling of a multicellular organism. Science. 357 (6352), 661-667 (2017).
- Kaletsky, R., Murphy, C. T. Transcriptional profiling of C. elegans adult cells and tissues with age. Methods in Molecular Biology. 2144, 177-186 (2020).
- Kaletsky, R., et al. Transcriptome analysis of adult Caenorhabditis elegans cells reveals tissue-specific gene and isoform expression. PLoS Genetics. 14 (8), 1007559 (2018).
- Packer, J. S., et al. A lineage-resolved molecular atlas of C. elegans embryogenesis at single-cell resolution. Science. 365 (6459), 1971 (2019).
- Kulkarni, A., Anderson, A. G., Merullo, D. P., Konopka, G. Beyond bulk: a review of single cell transcriptomics methodologies and applications. Current Opinion in Biotechnology. 58, 129-136 (2019).
- Von Stetina, S. E., et al. Cell-specific microarray profiling experiments reveal a comprehensive picture of gene expression in the C. elegans nervous system. Genome Biology. 8 (7), 135 (2007).
- Baugh, L. R., et al. The homeodomain protein PAL-1 specifies a lineage-specific regulatory network in the C. elegans embryo. Development. 132 (8), 1843-1854 (2005).
- Haenni, S., et al. Analysis of C. elegans intestinal gene expression and polyadenylation by fluorescence-activated nuclei sorting and 3'-end-seq. Nucleic Acids Research. 40 (13), 6304-6318 (2012).
- Zhang, S., Banerjee, D., Kuhn, J. R. Isolation and culture of larval cells from C. elegans. PLoS One. 6 (4), 19505 (2011).
- Emmert-Buck, M. R., et al. Laser capture microdissection. Science. 274 (5289), 998-1001 (1996).
- Hashimshony, T., et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biology. 17, 77 (2016).
- Yanai, I., Hashimshony, T. CEL-Seq2-single-cell RNA sequencing by multiplexed linear amplification. Methods in Molecular Biology. 1979, 45-56 (2019).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Ceron, J. Basic Caenorhabditis elegans methods: synchronization and observation. Journal of Visualized Experiments: JoVE. (64), e4019 (2012).
- Baugh, L. R. To grow or not to grow: nutritional control of development during Caenorhabditis elegans L1 arrest. Genetics. 194 (3), 539-555 (2013).
- Baugh, L. R., Hu, P. J. Starvation responses throughout the Caenorhabditis elegans life cycle. Genetics. 216 (4), 837-878 (2020).
- Pepper, A. S., Killian, D. J., Hubbard, E. J. Genetic analysis of Caenorhabditis elegans glp-1 mutants suggests receptor interaction or competition. Genetics. 163 (1), 115-132 (2003).
- Mok, D. Z., Sternberg, P. W., Inoue, T. Morphologically defined sub-stages of C. elegans vulval development in the fourth larval stage. BMC Developmental Biology. 15, 26 (2015).
- Lytal, N., Ran, D., An, L. Normalization methods on single-cell RNA-seq data: an empirical survey. Frontiers in Genetics. 11, 41 (2020).
- Vieth, B., Ziegenhain, C., Parekh, S., Enard, W., Hellmann, I. powsimR: power analysis for bulk and single cell RNA-seq experiments. Bioinformatics. 33 (21), 3486-3488 (2017).
- Bolkova, J., Lanctot, C. Quantitative gene expression analysis in Caenorhabditis elegans using single molecule RNA FISH. Methods. 98, 42-49 (2016).
- Ji, N., van Oudenaarden, A. Single molecule fluorescent in situ hybridization (smFISH) of C. elegans worms and embryos. WormBook. , 1-16 (2012).
- Lee, C., et al. Single-molecule RNA fluorescence in situ hybridization (smFISH) in Caenorhabditis elegans. Bio-Protocol. 7 (12), 2357 (2017).
- Baird, S. E., Chamberlin, H. M. Caenorhabditis briggsae methods. WormBook. , 1-9 (2006).
- Felix, M. A. Oscheius tipulae. WormBook. , 1-8 (2006).
- Han, Z., et al. Improving transgenesis efficiency and CRISPR-associated tools through codon optimization and native intron addition in Pristionchus nematodes. Genetics. 216 (4), 947-956 (2020).
- Fernandes Povoa, E. E., Ebbing, A. L. P., Betist, M. C., vander Veen, C., Korswagen, H. C. An optimized dissociation protocol for FACS-based isolation of rare cell types from Caenorhabditis elegans L1 larvae. MethodsX. 7, 100922 (2020).
- Han, M., Wei, G., McManus, C. E., Hillier, L. W., Reinke, V. Isolated C. elegans germ nuclei exhibit distinct genomic profiles of histone modification and gene expression. BMC Genomics. 20 (1), 500 (2019).
- Steiner, F. A., Henikoff, S. Cell type-specific affinity purification of nuclei for chromatin profiling in whole animals. Methods in Molecular Biology. 1228, 3-14 (2015).
- Ebbing, A. Spatial transcriptomics of C. elegans males and hermaphrodites identifies sex-specific differences in gene expression patterns. Developmental Cell. 47 (6), 801-813 (2018).
- Rödelsperger, C., et al. Spatial transcriptomics of nematodes identifies sperm cells as a source of genomic novelty and rapid evolution. Molecular Biology and Evolution. 38 (1), 229-243 (2021).
- Amini, P., et al. An optimised protocol for isolation of RNA from small sections of laser-capture microdissected FFPE tissue amenable for next-generation sequencing. BMC Molecular Biology. 18 (1), 22 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados