Method Article
Microdisseção a laser para aplicações de tecido único agnóstico de espécies
Neste Artigo
Resumo
Um protocolo é descrito que usa microdissecção a laser para isolar tecidos nematoides individuais para sequenciamento de RNA. O protocolo não exige kits de ferramentas genéticas específicos para espécies, permitindo que perfis de expressão genética sejam comparados entre diferentes espécies ao nível de amostras de tecido único.
Resumo
Metodologias unicelulares revolucionaram a análise dos transcritos de tipos celulares específicos. No entanto, eles geralmente requerem "kits de ferramentas" genéticos específicos das espécies, como promotores que conduzem a expressão específica do tecido de proteínas fluorescentes. Além disso, protocolos que interrompem tecidos para isolar células individuais removem células de seu ambiente nativo (por exemplo, sinalizando de vizinhos) e podem resultar em respostas ao estresse ou outras diferenças em relação aos estados de expressão genética nativa. No presente protocolo, a microdisseção a laser (LMD) é otimizada para isolar pontas individuais de cauda de nematoide para o estudo da expressão genética durante a morfogênese da ponta da cauda masculina.
O LMD permite o isolamento de uma porção do animal sem a necessidade de interrupção celular ou kits de ferramentas específicos para espécies e, portanto, é aplicável a qualquer espécie. Posteriormente, protocolos de preparação de bibliotecas RNA-seq de células únicas, como o CEL-Seq2, podem ser aplicados a tecidos únicos isolados de LMD e analisados usando dutos padrão, dado que um genoma ou transcriptome bem anotado está disponível para a espécie. Esses dados podem ser usados para estabelecer o quão conservados ou diferentes são os transcriptomes que estão por trás do desenvolvimento desse tecido em diferentes espécies.
As limitações incluem a capacidade de cortar o tecido de interesse e o tamanho da amostra. Uma análise de energia mostra que apenas 70 pontas traseiras por condição são necessárias para 80% de potência. Uma sincronização rigorosa do desenvolvimento é necessária para obter esse número de animais no mesmo estágio de desenvolvimento. Assim, também é descrito um método para sincronizar animais em intervalos de 1h.
Introdução
Nematoides - particularmente os nematoides rabditidos relacionados ao sistema modelo Caenorhabditis elegans - são um grupo maravilhoso de animais para biologia evolutiva do desenvolvimento (EDB) por muitas razões 1,2. As vantagens incluem seu pequeno número de células, linhagens celulares definidas e consistentes, transparência e facilidade de cultura e criação. Há também muitos recursos disponíveis, incluindo genomas de alta qualidade para múltiplas espécies, e para C. elegans, extensas ferramentas genéticas moleculares e conhecimento sobre desenvolvimento, genética, anatomia e fisiologia 3,4,5,6.
Como em muitos outros organismos, a capacidade de caracterizar a dinâmica do transcriptome em tecidos únicos ou células únicas revolucionou a análise do desenvolvimento em C. elegans 7,8,9,10. Ser capaz de comparar transcritos unicelulares entre nematoides transformaria igualmente o EDB usando esses organismos. Por exemplo, tais comparações forneceriam uma visão de como as redes reguladoras genéticas evoluíram para caracteres (traços) que foram conservados, para caracteres que divergiram, ou para caracteres que evoluíram independentemente.
No entanto, isolar tecidos ou células particulares de nematoides é um dos grandes desafios. Para muitos organismos, as células únicas podem ser dissociadas a partir de tecidos e colhidas de forma imparcial ou podem ser rotuladas com expressão específica do tecido de uma proteína fluorescente e classificadas por triagem celular ativada por fluorescência (FACS)11. Em C. elegans, o isolamento de células de alta produtividade (HTP) tem sido limitado principalmente a embriões porque a cutícula externa dura (e esqueleto hidrostático) tem dificultado o isolamento celular de larvas e adultos. Para contornar esse desafio, alguns métodos têm utilizado ferramentas genéticas em vermes c. elegans inteiros, como mRNA-tagging12 específico de tecido, e comparações de expressão diferencial entre tipo selvagem e mutantes que afetam uma célula tipo13. Métodos mais recentes superaram o desafio dissolvendo a cutícula para isolar núcleos14 ou células inteiras 8,9,15. O isolamento celular e a cultura celular têm as óbvias desvantagens, no entanto, de que as células são removidas de seu contexto natural de desenvolvimento ou anatômico — por exemplo, longe da sinalização celular e contato com a matriz extracelular — que devem impactar o perfil de expressão genética15. Além disso, as ferramentas genéticas e marcadores específicos do tecido são específicos das espécies (ou seja, elas só podem ser usadas em C. elegans).
A LMD fornece um método alternativo para isolar tecidos sem interromper o contexto natural das células. Significativamente para o EDB, a LMD também permite que transcritos de tecidos homólogos de diferentes espécies sejam comparados sem a necessidade de kits de ferramentas genéticas específicos para espécies se o genoma ou sequências de transcriptome inteiros dessas espécies estiverem disponíveis. A LMD envolve direcionar tecidos por observação microscópica direta e usar um micróbio laser — integrado à óptica do microscópio — para cortar e colher (capturar) o tecido de interesse16. As limitações do LMD são que não é propícia a abordagens muito HTP (embora os perfis de transcrição para pontas traseiras, como descrito neste protocolo, fossem robustos com ~70 amostras), certas amostras podem ser difíceis de dissecar, e os cortes são limitados à precisão do laser e ao que pode ser visualizado no microscópio.
O objetivo do presente protocolo é descrever como a LMD, seguida pelo RNA-Seq de tecido único, pode ser usada para obter dados de transcriptome específicos de estágio e tecido de nematoides. Especificamente, ele demonstra LMD para isolar pontas traseiras de larvas de quarto estágio (L4) de C. elegans. No entanto, este método pode ser adaptado a outros tecidos e, claro, espécies diferentes.
Em C. elegans, há 4 células que fazem a ponta da cauda em ambos os machos e hermafroditas. Durante a fase L4 em machos — mas não em hermafroditas — as células da ponta traseira mudam de forma e migram anteriormente e interiormente. Esse processo também ocorre em algumas, mas não em todas as outras espécies de nematode rabditid. Portanto, a ponta da cauda é um bom modelo para a evolução da morfogênese dimórfica sexual. Por causa de sua posição, a ponta traseira também é fácil de isolar por LMD.
Para obter perfis de transcriptome a partir de pontas traseiras, o presente protocolo utiliza o CEL-Seq2, um método RNA-seq desenvolvido para células únicas17,18. Este método tem várias vantagens para tecidos derivados de LMD. O CEL-Seq2 é altamente sensível e eficiente, usando identificadores moleculares únicos (UMIs) para permitir quantificação direta de leituras de mRNA, transcrição in vitro para garantir amplificação linear e codificação de barras que permite multiplexação de amostras de tecido individuais. A única limitação do CEL-Seq2 é que as leituras recuperadas são tendenciosas para o final dos mRNAs de 3' e, portanto, a maioria dos isoformes, portanto, não pode ser distinguida.
Protocolo
1. Sincronização de vermes
NOTA: Dois métodos são descritos abaixo para sincronizar o desenvolvimento de C. elegans e outras espécies rabiscadas.
- Sincronizar pelo primeiro estágio larval (L1) detenção após o tratamento alcalino hipoclorito (alvejante).
NOTA: Este método foi descrito anteriormente no detalhe19. Este método se baseia em duas características de C. elegans que também são verdadeiras para várias outras espécies de rabiscos: (1) A casca de ovo é resistente ao alvejante, enquanto a cutícula em torno de vermes adultos e larvas não é. (2) Desenvolvimento de apreensão de larvas em primeiro estágio quando mantida sem alimento20.- Trate hermafroditas gravid (ou fêmeas) com uma solução alvejante diluída para quebrar sua cutícula e liberar embriões.
- Remova os embriões do alvejante e mantenha-os sem comida até que todos os L1 tenham eclodido.
- Coloque o L1 preso em alimentos, onde todos retomam o desenvolvimento ao mesmo tempo.
NOTA: A saída da L1 pode ocorrer dentro de uma hora.
- Sincronização com o método "hatch-off" (usado aqui; Figura 1 superior:
NOTA: O método hatch-off permite uma sincronização apertada sem interrupção do desenvolvimento (a prisão L1 afeta o desenvolvimento mesmo dos estágiosposteriores 21). O protocolo é adaptado de Pepper et al.22. O objetivo deste método é coletar L1 que chocaram durante um período específico a partir de uma placa que contém apenas embriões.- Escolha mães: Na noite anterior à realização da escotilha, pegue ~30 hermafroditas gravid em uma placa semeada com E. coli OP50.
- Incubar a 25 °C para a colocação de ovos durante a noite.
NOTA: Escolha um prato sem rachaduras ou bolhas onde os vermes possam ficar presos. Evite placas com um gramado bacteriano muito espesso, pois será difícil remover todos os vermes mais tarde. Se trabalhar com uma cepa sensível à temperatura, ajuste o tempo de colocação de ovos para explicar a embrigênese mais longa. Escolha as mães na fase máxima de colocação de ovos. - Remova as mães e as larvas: na manhã seguinte, sob o microscópio dissecando (ampliação de 20x), pipeta suavemente 1-2 mL de tampão M9 contra a parede da placa sem esguicharar; redemoinho a placa para desalojar os vermes.
- Remova e descarte todos os líquidos e vermes colocando a ponta da pipeta contra a parede da placa na borda do ágar para evitar furos. Verifique se não restam vermes (e apenas ovos/embriões) na placa, especialmente L1s; caso contrário, repita a lavagem.
- Coloque a placa a 25 °C por 1h e espere alguns L1s para chocar.
- Colete L1s recém-eclodidos: solte cuidadosamente 1 mL M9 tampão no ágar. Gire a placa para desalojar L1, mas não embriões. Tampão de pipeta suavemente e worms em um tubo centrífuga de 1 mL.
- Centrifugar o tubo por 1 min a ~18.000 × g. Remova o supernatante.
- Pipeta L1 diretamente no gramado bacteriano de uma placa semeada. Verifique sob o microscópio de dissecção que não há vermes adultos ou embriões.
- Mantenha os vermes a 25 °C até que eles tenham se desenvolvido para o estágio desejado.
NOTA: Se as condições forem ótimas, mais dois lotes de L1 podem ser coletados da mesma placa. Inspecione a placa inicial para ter certeza de que não há L1. Se necessário, lave novamente. Repetição de passos 1.2.5-1.2.9. -
Verifique o tempo de desenvolvimento. Antes de prosseguir para a aplicação a jusante, inspecione alguns worms sob um microscópio composto a 400x de ampliação para confirmar que chegaram ao estágio de desenvolvimento desejado, aqui L3.
NOTA: A distância de migração de células de ponta distal ou células de linker pode ser usada como guia, além do desenvolvimento da vulva. Para o desenvolvimento da vulva, Mock et al.23 fornecem um guia útil, embora o tempo nesse estudo tenha sido determinado a 20 °C. A 25 °C, os elegans do tipo selvagem passarão pelo molt L3-L4 24 h após a eclosão.
2. Coleta de L4 machos e hermafroditas e fixação
- Prepare o rnAse, frio (-20 °C), 70% de metanol antes da fixação.
- Sob um microscópio de dissecção a 30-50x de ampliação, comece a colher machos e hermafroditas das placas de sincronização em placas separadas não semeadas assim que os sexos puderem ser distinguidos (~21 h após a eclosão, Figura 2), e continue escolhendo por 1-2 h ou até que 200 animais sejam coletados.
- Mantenha os vermes a 25 °C até chegarem ao estágio desejado para o experimento.
- Lave os vermes da placa com 1-2 mL de tampão M9 usando uma ponta de pipeta pré-lavada com tampão M9 contendo detergente de 0,01% (para evitar que os vermes grudem na ponta).
- Transfira os vermes para um tubo de centrífuga de 1 mL.
- Gire por 1 min a 21.000 × g para pelotar os vermes. Remova o supernatante.
- Adicione 1 mL de tampão M9 e misture para quebrar a pelota.
- Gire por 1 min a 21.000 × g para pelotar os vermes. Remova o supernatante.
- Repita a lavagem.
- Adicione 1 mL de gelo frio 70% de metanol e misture bem.
- Gire por 1 min a 21.000 × g para pelotar os vermes. Remova o supernatante.
- Repita as etapas 2.10 e 2.11.
- Adicione 500 μL de 70% de metanol, misture e armazene a 4 °C por 1h a noite.
3. Microdisseção a laser
NOTA: A partir de agora, use reagentes e consumíveis livres de RNase; usar dicas de filtro.
- Se o método CEL-Seq2 for usado para processar as amostras, prepare uma mistura mestra para cada primer CEL-Seq2 (Tabela Suplementar S1): pipeta 2 μL de primer CEL-Seq2, 1 μL de 10 mM dNTP e 9 μL de 1% β-Mercaptoetanol (em água livre de RNase) em um tubo de 200 ΜL etiquetado.
- Montagem no slide
- Sob um microscópio de dissecção, pipeta 20 μL dos vermes fixos (20-40 vermes da etapa 2.13) para o lado fosco de um naphtalato de polietileno (PEN)-membrana deslizamento de vidro (onde a membrana está).
- Espere o metanol evaporar. Use um aquecedor de slides para acelerar a evaporação.
NOTA: Podem ser aplicadas gotas adicionais de metanol, e uma ponta de pipeta usada para espalhar os vermes se eles começarem a se agrupar à medida que secam. Quando os vermes estão em aglomerados, eles podem ser difíceis de dissecar.
- Configuração do microscópio
NOTA: O protocolo a seguir é específico do instrumento listado na Tabela de Materiais. Ele precisa ser ajustado se um microscópio LMD diferente for usado.- Coloque um umidificador de desktop atrás do palco na lateral do microscópio LMD. Certifique-se de que o vapor está soprando diretamente para o palco.
NOTA: O umidificador é útil para reduzir a eletricidade estática, o que de outra forma pode evitar que a pequena seção de membrana caia na tampa do tubo. - Gire a chave para a potência laser.
- Ligue a potência do controle do palco.
- Ligue a caixa de controle do microscópio.
- Software de microdisseção a laser aberto.
- Remova o escudo plástico sobre o palco.
- Clique no botão de descarga com a seta ascendente para carregar os slides da membrana.
- Certifique-se de que o slide está completamente seco, gire para que a membrana esteja virada para baixo.
- Insira o slide e clique em continuar na janela do espécime de alteração .
- Substitua o escudo plástico.
- Na parte inferior da tela, escolha qual porta-slides contém o slide.
- Para carregar os tubos, clique no botão de descarga com a seta para baixo.
- Puxe a bandeja para fora e remova o bloco do tubo.
NOTA: O bloco de tubos utilizado para este experimento é para tubos PCR de 500 μL. - Insira as tampas do tubo de tubos de 500 μL PCR no suporte e dobre o tubo sob.
- Volte o bloco para a bandeja e deslize a bandeja de volta para o estágio do microscópio.
- Na janela popup do dispositivo de coletor de alterações , selecione tubos PCR e clique em ok.
- Clique no local vazio do tubo no canto inferior esquerdo da tela sob tampas de tubo do dispositivo coletor.
- No painel de controle do microscópio , selecione TL-BF para iluminar a luz transmitida.
- Coloque um umidificador de desktop atrás do palco na lateral do microscópio LMD. Certifique-se de que o vapor está soprando diretamente para o palco.
- Corte
NOTA: Este protocolo é específico para o instrumento listado na Tabela de Materiais.- Usando a lente 2,5x, ajuste o foco até que os vermes e a estrutura da membrana sejam visíveis.
- Mude para a lente 20x.
- Mova o palco para uma região sem vermes. Ajuste o foco de modo que as estruturas semelhantes a bolhas na membrana tenham uma cor amarelada (Figura 3A,B) para focar o laser no plano focal correto.
- Definir os parâmetros do laser; para pontas traseiras, comece com Power 45, abertura 30 e velocidade 20.
- No painel controle a laser , selecione calibrar. Siga as instruções.
NOTA: O instrumento executará esta etapa automaticamente. Ele garantirá que uma forma desenhada com o mouse na tela seja idêntica à forma cortada pelo laser. - Na parte inferior da tela em tampas de tubo do dispositivo coletor, clique na posição A.
- No lado direito da tela, selecione a forma única | Desenhar + Corte. No lado esquerdo da tela, selecione PtoP.
- Desenhe uma linha.
- Clique em Iniciar Corte para que o laser corte através da membrana.
NOTA: Também pode gravar uma linha no vidro. - Se este corte de teste parecer bom (a membrana é cortada, as bordas do corte parecem lisas), continue com o próximo passo. Caso contrário, ajuste o foco e corte outra linha.
- Encontre um verme. Mude para Mover + Corte e use o mouse para cortar a cauda.
NOTA: Se o laser não cortar a cauda, ajuste o foco e aumente a potência do laser. Para tecidos mais grossos, a potência do laser pode ter que ser definida como 60. - Salve os parâmetros: | da guia arquivo Salvar configuração do aplicativo; para recuperação posterior, restaurar configuração do aplicativo.
- Para coletar a amostra, mude para a configuração Desenhar + Corte com a função PtoP e desenhe uma forma para completar o corte de uma seção de membrana (Figura 3C).
NOTA: Seções de membrana maiores e seções em forma de retângulos ou triângulos em vez de círculos ou ovais são mais fáceis de localizar na tampa do tubo coletor. - Selecione o próximo tubo na Tampa do Tubo do Dispositivo coletor na parte inferior da tela e corte a próxima ponta da cauda.
- Uma vez que quatro caudas são cortadas, descarregue o rack do tubo (clique em Descarregar com seta para baixo) e encontre as seções de membrana sob um microscópio dissecando (Figura 3D).
NOTA: As seções podem estar localizadas no meio da tampa do tubo ou presas ao lado da tampa. - Continue com a aplicação a jusante. Para CEL-Seq2, pipeta 1,2 μL de uma mistura master de primer CEL-Seq2 (a partir da etapa 3.1) diretamente em cima da amostra.
- Feche o tubo, rotule com o número do primer e coloque imediatamente a tampa do tubo diretamente em um pedaço de gelo seco para congelar a amostra e evitar a degradação do RNA.
- Carregue mais tubos, devolva o bloco do tubo ao palco e corte mais amostras. Adicione uma mistura diferente de primer CEL-Seq2 a cada ponta traseira.
- Armazene todos os tubos a -70 °C.
4. Sequenciamento de RNA de cauda única com CEL-Seq2
NOTA: Para obter detalhes completos sobre o protocolo CEL-Seq2, consulte Yanai e Hashimshony18.
- Limpe a área do banco de laboratório com solução de descontaminação RNase para evitar a degradação do RNA.
- Prepare misturas mestras e mantenha-as no gelo.
- Prepare a mistura mestra de transcrição reversa: 0,4 μL do primeiro buffer de fio, 0,1 μL de 0,1M DTT, 0,1 μL do inibidor de RNase e 0,1 μL de transcriptase reversa por amostra.
- Prepare a segunda mistura mestre de reação do fio: 7 μL de água, 2,31 μL de tampão de segunda linha, 0,23 μL de dNTP, 0,08 μL de E. coli ligase, 0,3 μL de polimerase de DNA E. coli , 0,08 μL de RNaseH por amostra.
- Quebrando células abertas e ressarcendo com primers (consulte Tabela Suplementar S1 para a lista completa de primers):
- Programe o termociclador e sua tampa a 65 °C.
- Recupere as amostras a partir de -70 °C e incuba-as no termociclador por 2,5 min.
- Gire a 21.000 × g para 30-40 s.
- Incubar a 65 °C por 2,5 min.
- Mova-os imediatamente para o gelo.
- Gire a 21.000 × g para 30-40 s e devolva-os ao gelo.
- Conversão do RNA para cDNA:
- Adicione 0,8 μL da mistura de transcrição reversa a cada ponta traseira.
- Incubar a 42 °C por 1 h.
- Inativação a calor a 70 °C Por 10 min.
- Mova-o imediatamente para o gelo.
- Adicione 10 μL da segunda mistura de fios a cada ponta da cauda.
- Passe as amostras.
- Gire a 21.000 × g para 30-40 s.
- Incubar a 16 °C por 2 h.
- limpeza cDNA:
- Pré-aquecimento as contas de limpeza de DNA à temperatura ambiente.
- Acumule até 40 amostras em um tubo centrífuga de 1,5 mL (até 480 μL).
- Misture as contas até que elas estejam bem dispersas e adicione 20 μL de contas e 100 μL de tampão de ligação de contas para cada 100 μL da amostra agrupada (para 480 μL de amostra adicione 480 μL de tampão de contas e 96 μL de contas a um volume final até 1.056 μL). Misture bem por pipetação.
- Incubar em temperatura ambiente por 15 minutos.
- Coloque em uma posição magnética por pelo menos 5 minutos até que o líquido pareça limpo.
- Remova e descarte todos, exceto 20 μL do supernaspeso.
- Adicione 200 μL de etanol recém-preparado.
- Incubar por pelo menos 30 s, remova o supernatante pipetando-o sem perturbar as contas. Descarte o supernatante.
- Repetir as etapas 4.5.7 e 4.5.8 uma vez.
- Seque as contas por 15 minutos ou até ficarem completamente secas.
- Resuspende as contas (~6,4 μL) com 6,4 μL de água. Misture bem, encanar todo o volume dez vezes.
- Incubar em temperatura ambiente por 2 minutos.
- Vá direto para transcrição in vitro (IVT).
- Transcrição in vitro e fragmentação:
- Para o tubo contendo 6,4 μL de amostra e as contas, adicionar o mix seguinte (9,6 μL total): 1,6 μL de 10x T7 Buffer, 1,6 μL de ATP, 1,6 μL de UTP, 1,6 μL de CTP e 1,6 μL de GTP (cada dNTP a 75 mM de concentração) 1,6 μL de T7 enme.
- Incubar por 13 h a 37 °C com uma espera de 4 °C.
- Adicione 6 μL de solução exonuclease (o volume final deve ser de 22 μL).
- Incubar por 15 min a 37 °C.
- Coloque o tubo de volta no gelo e adicione 5,5 μL de tampão de fragmentação (0,25 × volume de reação).
- Incubar por 3 min a 94 °C.
- Mova imediatamente o tubo para o gelo e adicione 2,75 μL de buffer de parada de fragmentação (0,5 × volume de buffer de fragmentação adicionado).
- Remova as contas colocando o tubo no suporte magnético por pelo menos 5 minutos até que o líquido pareça limpo.
- Transfira o supernatante para um novo tubo.
- Limpeza de RNA amplificado (aRNA):
- Pré-aquecimento as contas de limpeza do RNA à temperatura ambiente.
- Misture as contas até que elas estejam bem dispersas.
- Pipeta 55 μL de contas (1,8 × volume de reação).
- Incuba-os à temperatura ambiente por 10 minutos.
- Coloque o tubo no suporte magnético por pelo menos 5 minutos até que o líquido pareça limpo.
- Remova e descarte 80 μL do supernante.
- Adicione 200 μL de etanol recém-preparado.
- Incubar por pelo menos 30 s, remova o supernatante por pipetação sem perturbar as contas. Descarte o supernatante.
- Repita a lavagem do etanol mais duas vezes.
- Seque as contas por 15 minutos ou até ficarem completamente secas.
- Resuspenda as contas com 7 μL de água. Pipete todo o volume para cima e para baixo 10 vezes para misturar bem.
- Incubar em temperatura ambiente por 2 minutos.
- Coloque o tubo com as contas no suporte magnético por 5 minutos até que o líquido pareça limpo.
- Transfira o supernatante para um novo tubo.
NOTA: Ponto de parada: as amostras podem ser mantidas a -70 °C.
- Opcional: Verifique a quantidade e a qualidade do ARNA com um sistema automatizado de eletroforese seguindo o protocolo do fabricante.
- Preparação da biblioteca:
- A 5 μL de RNA, adicione 1 μL de primer RT de hexamer aleatório de 100 μM (ver a tabela de materiais) e 0,5 μL de 10 mM dNTP.
- Incubar a 65 °C por 5 min.
- Adicione 4 μL da seguinte mistura à temperatura ambiente: 2 μL de primeiro tampão de fio, 1 μL de 0,1 M DDT, 0,5 μL de inibidor de RNase, 0,5 μL de transcriptase reversa.
- Incubar a 25 °C por 10 min.
- Incubar a 42 °C por 1h (em forno de hibridização ou um cicloviário térmico pré-aquecido com a tampa definida a 50 °C).
- Incubar a 70 °C por 10 min.
- Transfira 5 μL para um novo tubo (mantenha o resto da reação a -20 °C). Adicione 5,5 μL de água ultrauso, 1 μL de RNA PCR Primer (RP1), 1 μL de RNA PCR Primer indexado (RPIX) e 12,5 μL de mix PCR.
- Use o seguinte programa no termociclador: 30 s a 98 °C, 11 ciclos de: (10 s a 98 °C, 30 s a 60 °C, 30 s a 72 °C), 10 min a 72 °C, mantém em 4 °C.
NOTA: Ponto de parada: as amostras podem ser mantidas a -20 °C.
- Limpeza da biblioteca:
- Pré-aquecimento as contas de limpeza de DNA à temperatura ambiente.
- Misture as contas até que elas estejam bem dispersas.
- Adicione 25 μL das contas à reação do PCR. Misture bem por pipetação.
- Incubar em temperatura ambiente por 15 minutos.
- Coloque o tubo no suporte magnético por pelo menos 5 minutos até que o líquido pareça limpo.
- Remova e descarte 45 μL do supernaspeso.
- Adicione 200 μL de etanol recém-preparado.
- Incubar por pelo menos 30 s, remover e descartar o supernatante sem perturbar as contas.
- Repita a lavagem do etanol uma vez.
- Contas secas por 15 minutos ou até ficarem completamente secas.
- Resuspensá-los com 25 μL de água. Misture bem por pipetação.
- Incubar em temperatura ambiente por 2 minutos.
- Coloque o tubo no suporte magnético por 5 minutos até que o líquido pareça limpo.
- Transfira 25 μL de supernante para um novo tubo.
- Repetir as etapas 4.10.2-4.10.10 uma vez.
- Resuspend com 10,5 μL de água. Misture bem por pipetação.
- Incubar em temperatura ambiente por 2 minutos.
- Coloque o tubo no suporte magnético por 5 minutos até que o líquido pareça limpo.
- Transfira 10 μL do supernante para um novo tubo e armazene a -20 °C.
- Avalie a qualidade e quantidade da biblioteca de acordo com a exigência da instalação de sequenciamento.
Resultados
Após microdisseção de captura a laser, pontas traseiras individuais de machos e hermafroditas em 4 pontos de tempo (L3 22 h após a escotilha; L4 24, 26 e 28 h após o hatch) foram preparados para sequenciamento de RNA utilizando o protocolo CEL-Seq2. Os primers CEL-Seq2 contêm códigos de barras exclusivos que permitem que leituras de sequenciamento de uma determinada amostra (neste caso uma ponta de cauda individual) sejam identificadas bioinformáticamente. Os dados de sequenciamento foram gerados com este método para um total de 557 pontas traseiras (266 hermafroditas e 291 machos em 4 pontos de tempo de desenvolvimento, 59-78 caudas por sexo e ponto de tempo). Os códigos de barras CEL-Seq2 foram recuperados por 97% (ou seja, 543) dessas pontas traseiras (Tabela Suplementar S2). Para a maioria das bibliotecas, a taxa de recuperação foi de 99-100%; no entanto, foi de 88% para um ponto de tempo masculino. Vale ressaltar que cerca de metade das pontas de cauda masculinas dos pontos de tempo de 22, 24 e 28 h foram armazenadas a -80 °C por ~4 meses devido a atrasos relacionados ao COVID-19. Isso demonstra que, embora seja ideal preparar bibliotecas de sequenciamento logo após a amostragem, é possível armazenar amostras dissecadas por mais tempo antes da preparação da biblioteca.
Os primers CEL-Seq2 também adicionam um UMI a cada transcrição mRNA. Isso permite a remoção duplicada do PCR e quantificação precisa da expressão genética na amostra. O número de UMIs variou drasticamente entre as pontas da cauda (Figura 4; média masculina = 92.560; min. masculino = 155; máximo masculino. = 1.183.998; hermafroditas média = 67.597; hermafroditas min. = 132; hermafroditas max. = 630.427). Para contagem de UMI por ponta traseira, consulte Tabela Suplementar S3. Devido à baixa quantidade de RNA de entrada para preparação de biblioteca de células únicas, os dados de sequenciamento de células únicas são conhecidos por ter uma grande quantidade de ruído técnico. Assim, recomenda-se filtrar amostras que tenham contagens muito baixas ou muito altas de UMI antes da análise24.
O pacote R powsimR25 foi utilizado para avaliar os requisitos estatísticos de potência e tamanho da amostra para detectar genes de forma confiável (DE) em experimentos RNA-seq unicelular ou a granel. Os parâmetros para as simulações basearam-se em um conjunto de dados de sequenciamento de 70 pontas individuais de cauda masculina (no ponto de tempo de 24h) obtido com o método descrito aqui. As alterações esperadas do registro foram baseadas nos resultados de um experimento RNA-seq separado que reuniu 80-100 pontas traseiras. As simulações determinaram que os dados de ponta única têm potência suficiente (Taxa Real Positiva = TPR) para detectar genes DE, exceto para genes que têm um valor de expressão média muito baixo (topo da Figura 5; linha tracejada representa 80% de TPR). Adicionar mais pontas de cauda simuladas por ponto de tempo aumentou um pouco a potência para genes mal expressos. Um padrão semelhante é visto para a Taxa de Descoberta Falsa (FDR). FDR é alto (>0,10) para os genes humildemente expressos; no entanto, para genes mais altamente expressos, ele cai a ou abaixo do corte nominal de 0,10 (linha tracejada para FDR na parte inferior da Figura 5). Em resumo, aumentar o número de caudas amostradas por ponto de tempo acima de 70 faria pouco para baixar o FDR ou aumentar a potência. No entanto, 70 pontas traseiras fornecem um FDR muito mais baixo e potência mais forte do que 30 pontas traseiras.

Figura 1: Visão geral do procedimento para sincronização de elegans caenorhabditis com o método hatch-off e microdisseção a laser de pontas traseiras. Abreviaturas: L1-L4 = estágios larvais 1 a 4; PEN = naftato de polietileno; LMD = microdisseção a laser. Clique aqui para ver uma versão maior desta figura.
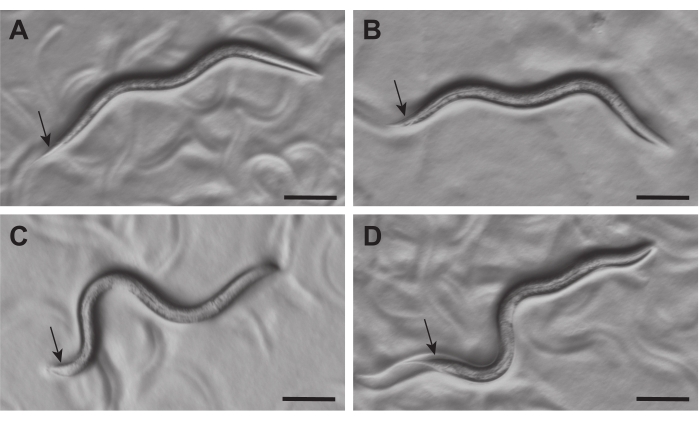
Figura 2: Aparecimento de C. elegans L3 hermafroditas e machos sob um microscópio de dissecção. Hermafroditas (A, B) e machos (C, D) a 21-23 h após a escotilha podem ser distinguidos sob um microscópio de dissecção (~50x de ampliação) pela morfologia de suas caudas (setas). A cauda das hermafroditas é estreita, enquanto a dos machos está inchada e parece clara. Barras de escala = 0,1 mm. Clique aqui para ver uma versão maior desta figura.

Figura 3: Aparecimento da estrutura de lâmina da membrana PEN e cauda de verme. O foco é correto para dissecção do tecido visualizado com a lente 20x (A) e 40x (B) no microscópio. (C) Cauda dissecada e membrana PEN parcialmente cortada. Depois de fechar a abertura do corte, a peça de membrana cairá na tampa do tubo abaixo do slide. (D) Tampa do tubo com uma seção de membrana PEN contendo uma ponta traseira dissecada. Barras de escala = 0,1 mm (A-C), 1 mm (D). Clique aqui para ver uma versão maior desta figura.
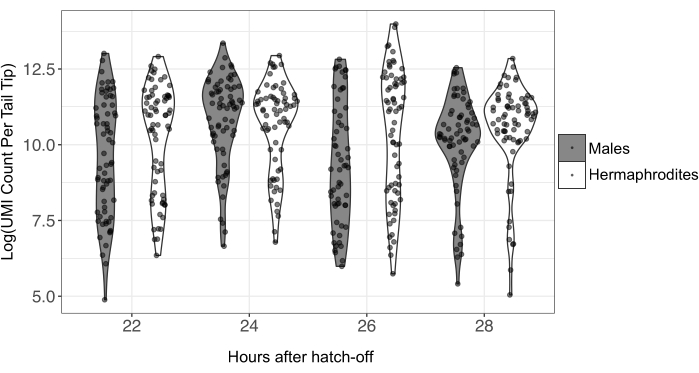
Figura 4: Contagem natural de UMI transformada em troncos por ponta de cauda individual para diferentes pontos de tempo e sexos. O RNA de caudas individuais foi preparado para sequenciamento utilizando o método CEL-Seq2; 557 caudas foram sequenciadas no total, com 59-78 caudas por sexo e ponto de tempo. Outliers umi extremamente baixos e altos seriam removidos dos dados antes da análise. Abreviação: UMI = identificador molecular único. Clique aqui para ver uma versão maior desta figura.

Figura 5: Resultados de uma análise de energia posteriori usando simulações com powsimR. O software powsimR determina o número de amostras independentes necessárias para detectar genes DE em vários níveis de expressão. Genes são binados pela expressão média transformada como o registro natural da UMI conta. (A) Poder (TPR) para detectar genes DE entre duas condições (aqui, masculino vs hermafrodita) para quatro simulações diferentes (diferentes gráficos coloridos) incorporando diferentes tamanhos de amostra (números de pontas traseiras individuais) por condição. A linha tracejada indica 80% TPR. (B) FDR nas mesmas quatro simulações que em (A), linha tracejada indicando 10% FDR. Os gráficos mostram que um tamanho amostral de 70 pontas de cauda (verde) por condição é suficiente para detectar genes DE, exceto para genes com níveis de expressão muito baixos. Ou seja, o poder e a falsa taxa de descoberta para tais genes não podem ser muito melhorados aumentando o tamanho da amostra para além de 70. Abreviaturas: DE = expresso diferencialmente; UMI = identificador molecular único; TPR = taxa positiva verdadeira; FDR = taxa de descoberta falsa. Clique aqui para ver uma versão maior desta figura.
Tabela Suplementar S1: Sequências de primers utilizados no protocolo CEL-Seq2. Clique aqui para baixar este Arquivo.
Tabela Suplementar S2: Amostradas versus pontas de cauda individuais recuperadas. Os primers CEL-Seq2 contêm códigos de barras exclusivos que permitem identificar leituras de sequenciamento de cada amostra a serem identificadas bioinformáticamente. As leituras foram recuperadas de 97% das amostras de ponta de cauda preparadas para sequenciamento. Clique aqui para baixar este Arquivo.
Tabela Suplementar S3: Umi conta todas as amostras com códigos de barras recuperados, conforme observado na Tabela Suplementar S2. Abreviação: UMI = identificador molecular único. Clique aqui para baixar este Arquivo.
Discussão
Passos críticos do método
Se executado corretamente, o método descrito aqui obterá perfis robustos de RNA com um número relativamente pequeno de amostras dissecadas a laser (70 pontas de cauda neste exemplo). No entanto, para amostras de animais em desenvolvimento, a sincronização apertada é fundamental para reduzir a variabilidade entre as amostras. Por essa razão, o protocolo recomenda o método hatch-off para sincronização de vermes. Aqui, o pesquisador pode determinar e controlar precisamente a diferença de idade entre os indivíduos (1h no presente protocolo). Além disso, o método hatch-off é aplicável a qualquer espécie, mesmo que os embriões sejam sensíveis ao alvejante, L1 não prendem ou a recuperação da prisão de L1 é variável. Para uma sincronização bem sucedida por hatch-off, as etapas de lavagem são cruciais: todos os adultos e larvas devem ser removidos no início do período de eclosão, e nenhum embrião deve ser lavado junto com o recém-eclodido L1 no final do período de eclosão. Isso só é bem sucedido se a superfície do ágar da placa não for danificada por rachaduras, buracos ou bolhas, o gramado bacteriano na placa é fresco e não muito grosso, e o líquido é adicionado e agitado apenas muito suavemente.
Se os dados forem obtidos separadamente para machos e hermafroditas/fêmeas, a identificação confiável dos sexos também é importante. Distinguir larvas L3 por sexo (ver Figura 2) requer experiência. Recomenda-se praticar a colheita de machos L3 e hermafroditas/fêmeas e verificar a taxa de sucesso depois que os animais se desenvolveram em adultos e os sexos são facilmente distinguidos. Após o RNA-Seq de tecido único, os outliers também podem ser identificados pela análise dos componentes principais e removidos, se necessário.
Para uma recuperação bem-sucedida de amostras cortadas a laser, é importante reduzir a eletricidade estática tanto quanto possível. As peças de membrana PEN carregada muitas vezes não caem na tampa do tubo, mas grudam no slide ou em qualquer outra parte do microscópio. Um remédio é aumentar a umidade na sala e especificamente ao redor do microscópio, colocando um pequeno umidificador ao lado do palco. Além disso, as lâminas de membrana podem ser tratadas com luz UV. Para isso, incubar slides em uma câmara de crosslink UV-C (254 nm) e entregar pelo menos 1 joule de energia, ou expor os slides à luz UV em um banco de fluxo de ar laminar por 30 minutos.
Como o objetivo do protocolo é o RNA-Seq, manter um ambiente de trabalho livre de RNase é fundamental. A partir da solução de fixação, reagentes, recipientes e consumíveis devem ser livres de RNase, a superfície de trabalho deve ser descontaminada, e os pesquisadores devem usar luvas limpas. As amostras dissecadas devem ser congeladas o mais rápido possível e mantidas a -70 °C até um novo processamento. Também é recomendado o uso de tubos de baixa retenção e dicas para a parte CEL-Seq2 do protocolo.
O presente artigo fornece apenas um esboço básico do protocolo CEL-Seq2, que foi publicado anteriormente por seus desenvolvedores com notas úteis e dicas17,18. Recomenda-se que essas publicações sejam consultadas antes do uso do método CEL-Seq2.
Os dados LMD-RNA-Seq podem ser validados pela hibridização fluorescente in-situ de molécula única (smRNA FISH)26,27,28. smRNA FISH tem sido amplamente utilizado em C. elegans e é favorável a outras espécies de nematoides, diferente de imunosmunagem com anticorpos existentes (que não podem cruzar) ou a introdução de repórteres transcricionais através da transgênese. Este último funciona bem em C. elegans e algumas espécies relacionadas de Caenorhabditis 29, mas a transgênese pode ser mais desafiadora em outras espécies de nematoides30,31.
Limitações do método
O método descrito aqui funciona muito bem para coletar pontas de cauda, um tecido fino na extremidade de um verme. Dissecar tecidos no meio mais espesso de larvas mais velhas ou adultos é mais desafiador. O software do instrumento utilizado aqui inclui uma configuração para múltiplos cortes colocados em níveis posteriormente mais profundos no tecido. Esta configuração pode ser usada para cortar áreas mais espessas do animal. Como os vermes precisam ser corrigidos antes da dissecação, os detalhes estruturais são difíceis de ver, o que impede a dissecção precisa de pequenas estruturas específicas. Como mencionado acima, lMD-RNA-Seq não é um método HTP. No entanto, 50-70 amostras podem ser dissecadas em uma tarde.
Significância do método em relação aos métodos existentes/alternativos
LMD-RNA-Seq pode ser usado em qualquer espécie, mesmo que não haja ferramentas transgênicas disponíveis. Outros métodos dependem da triagem FACS de células fluorescentes rotuladas 8,9,32 ou isolamento de núcleos rotulados33,34 e, portanto, requerem animais transgênicos. Métodos que dissociam e isolam células em pós-mbryônico C. elegans tendem a perder os tecidos nas duas extremidades do verme (Dylan Rahe, comunicação pessoal). Essas ressalvas são superadas pela combinação de RNA-Seq unicelular com criosectioning de vermes inteiros (tomografia de RNA)35. Este método foi utilizado para comparar a expressão genética espacial entre C. elegans e outro nematode rabditid, Pristionchus pacificus36. Alternativamente, pode-se experimentar com vermes parafina-fixados formalin (FFPE). Esse material foi usado com sucesso para RNA-Seq após LMD de amostras de tecidomamínico 37. No entanto, a tomografia de RNA e LMD de vermes FFPE estão limitadas à análise de apenas um punhado de animais. Eles, portanto, não são tão adequados para o estudo da expressão genética dinâmica no desenvolvimento de tecidos como LMD-RNA-Seq.
Divulgações
Todos os autores declaram que não têm conflitos de interesse.
Agradecimentos
Este trabalho foi financiado por subvenções do NIH (R01GM141395) e NSF (1656736) para bolsa DF e NIH (F32GM136170) para a AW. A Figura 1 foi criada com a ajuda de BioRender.com.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.5 µM PEN membrane glass slides RNase free | Leica | 11600288 | for LMD |
| 500 µL PCR tubes (nuclease-free) | Axygen | 732-0675 | to cut the tail tips into |
| Compound microscope with 40x objective and DIC | any | to check age of worms | |
| Desktop humidifier | any | ||
| Dissection microscope with transmitted light base | any | for all worm work | |
| glass pasteur pipets | any | handle of worm pick | |
| glass slides and coverslips | any | to check age of worms | |
| LMD6 microdissection system | Leica | multiple | to cut tail tips |
| LoBind tubes 0.5 mL | Eppendorf | 22431005 | |
| M9 Buffer | Recipe in WormBook | ||
| Methanol 99.8% | Sigma | 322415 | to fix worms |
| NGM growth medium | US Biological | N1000 | Buffers and salts need to be added: Recipe in WormBook |
| P10 pipette variablle volume | e.g. Gilson | ||
| P1000 pipette variable volume | e.g. Gilson | ||
| P2 pipette variable volume | e.g. Gilson | ||
| Pipette tips 1,000 µL | any | ||
| Pipette tips 1-10 µL filtered | any | ||
| platinum iridium wire | Tritech | PT-9010 | to make worm pick |
| sterile and nuclease-free 1 mL centrfuge tubes | any | ||
| Tween 20 | Sigma | P9416 | Add a very small amount to M9 buffer to prevent worms from sticking to the pipet tips |
| vented 6 mm plastic Petri dishes | any | ||
| For CEL-Seq2 | |||
| 4200 TapeStation System with reagents for high-sensitivity RNA and DNA detection | Aligent | automated electrophoresis system | |
| AMPure XP beads | Beckman Coulter | A63880 | DNA cleanup beads |
| Bead binding buffer 20% PEG8000, 2.5 M NaCl | |||
| CEL-Seq2 primers (see Table S1) | Sigma Genosys Mastercycler Nexus GX2 Eppendorf | 6335000020 | Thermal cycler with programmable lid and block for 200 µl tubes. |
| DNA Polymerase I (E. coli) | Invitrogen | 18052-025 | |
| dNTP mix 10 mM | any | ||
| E. coli DNA ligase | Invitrogen | 18052-019 | |
| Ethanol | |||
| ExoSAP-IT For PCR Product Clean-Up | Affymetrix | 78200 | exonuclease solution |
| MEGAscript T7 Transcription Kit | Ambion | AM1334 | For step 4.6.1 |
| Nuclease-free water | any | ||
| Phusion High-Fidelity PCR Master Mix with HF Buffer | NEB | M0531 | PCR mix step 4.9.7 |
| random hexamer RT primer GCCTTGGCACCCGAGAATTCCA NNNNNN | IDT | a primer with 6 nucleotides that are random | |
| RNA Fragmentation buffer | NEB | E6150S | |
| RNA Fragmentation stop buffer | NEB | E6150S | |
| RNA PCR Index Primers (RPI1–RPI48) | Illumina, NEB, or IDT | RPIX in protocol step 4.9.7, sequences available from Illumina | |
| RNAClean XP beads | Beckman Coulter | A63987 | |
| RNase AWAY Surface Decontaminant | Thermo Scientific | 7000TS1 | or any other similar product |
| RNaseH (E. coli) | Invitrogen | 18021-071 | |
| RNaseOUT Recombinant Ribonuclease Inhibitor | Invitrogen | 10777-019 | |
| Second strand buffer | Invitrogen | 10812-014 | |
| Superscripit II | Invitrogen | 18064-014 | reverse transcriptase |
Referências
- Haag, E. S., Fitch, D. H. A., Delattre, M. From "the worm" to "the worms" and back again: the evolutionary developmental biology of nematodes. Genetics. 210 (2), 397-433 (2018).
- Sommer, R. J., Bumbarger, D. J. Nematode model systems in evolution and development. Wiley Interdisciplinary Reviews: Developmental Biology. 1 (3), 389-400 (2012).
- WormBase. , Available from: https://wormbase.org/#012-3-6 (2022).
- WormAtlas. , Available from: https://wormatlas.org (2022).
- WormBook. , Available from: http://wormbook.org (2022).
- WormBook in Genetics. , Available from: https://academic-oup-com.remotexs.ntu.edu.sg/genetics/pages/wormbook (2022).
- Cao, J., et al. Comprehensive single-cell transcriptional profiling of a multicellular organism. Science. 357 (6352), 661-667 (2017).
- Kaletsky, R., Murphy, C. T. Transcriptional profiling of C. elegans adult cells and tissues with age. Methods in Molecular Biology. 2144, 177-186 (2020).
- Kaletsky, R., et al. Transcriptome analysis of adult Caenorhabditis elegans cells reveals tissue-specific gene and isoform expression. PLoS Genetics. 14 (8), 1007559(2018).
- Packer, J. S., et al. A lineage-resolved molecular atlas of C. elegans embryogenesis at single-cell resolution. Science. 365 (6459), 1971(2019).
- Kulkarni, A., Anderson, A. G., Merullo, D. P., Konopka, G. Beyond bulk: a review of single cell transcriptomics methodologies and applications. Current Opinion in Biotechnology. 58, 129-136 (2019).
- Von Stetina, S. E., et al. Cell-specific microarray profiling experiments reveal a comprehensive picture of gene expression in the C. elegans nervous system. Genome Biology. 8 (7), 135(2007).
- Baugh, L. R., et al. The homeodomain protein PAL-1 specifies a lineage-specific regulatory network in the C. elegans embryo. Development. 132 (8), 1843-1854 (2005).
- Haenni, S., et al. Analysis of C. elegans intestinal gene expression and polyadenylation by fluorescence-activated nuclei sorting and 3'-end-seq. Nucleic Acids Research. 40 (13), 6304-6318 (2012).
- Zhang, S., Banerjee, D., Kuhn, J. R. Isolation and culture of larval cells from C. elegans. PLoS One. 6 (4), 19505(2011).
- Emmert-Buck, M. R., et al. Laser capture microdissection. Science. 274 (5289), 998-1001 (1996).
- Hashimshony, T., et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biology. 17, 77(2016).
- Yanai, I., Hashimshony, T. CEL-Seq2-single-cell RNA sequencing by multiplexed linear amplification. Methods in Molecular Biology. 1979, 45-56 (2019).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Ceron, J. Basic Caenorhabditis elegans methods: synchronization and observation. Journal of Visualized Experiments: JoVE. (64), e4019(2012).
- Baugh, L. R. To grow or not to grow: nutritional control of development during Caenorhabditis elegans L1 arrest. Genetics. 194 (3), 539-555 (2013).
- Baugh, L. R., Hu, P. J. Starvation responses throughout the Caenorhabditis elegans life cycle. Genetics. 216 (4), 837-878 (2020).
- Pepper, A. S., Killian, D. J., Hubbard, E. J. Genetic analysis of Caenorhabditis elegans glp-1 mutants suggests receptor interaction or competition. Genetics. 163 (1), 115-132 (2003).
- Mok, D. Z., Sternberg, P. W., Inoue, T. Morphologically defined sub-stages of C. elegans vulval development in the fourth larval stage. BMC Developmental Biology. 15, 26(2015).
- Lytal, N., Ran, D., An, L. Normalization methods on single-cell RNA-seq data: an empirical survey. Frontiers in Genetics. 11, 41(2020).
- Vieth, B., Ziegenhain, C., Parekh, S., Enard, W., Hellmann, I. powsimR: power analysis for bulk and single cell RNA-seq experiments. Bioinformatics. 33 (21), 3486-3488 (2017).
- Bolkova, J., Lanctot, C. Quantitative gene expression analysis in Caenorhabditis elegans using single molecule RNA FISH. Methods. 98, 42-49 (2016).
- Ji, N., van Oudenaarden, A. Single molecule fluorescent in situ hybridization (smFISH) of C. elegans worms and embryos. WormBook. , 1-16 (2012).
- Lee, C., et al. Single-molecule RNA fluorescence in situ hybridization (smFISH) in Caenorhabditis elegans. Bio-Protocol. 7 (12), 2357(2017).
- Baird, S. E., Chamberlin, H. M. Caenorhabditis briggsae methods. WormBook. , 1-9 (2006).
- Felix, M. A. Oscheius tipulae. WormBook. , 1-8 (2006).
- Han, Z., et al. Improving transgenesis efficiency and CRISPR-associated tools through codon optimization and native intron addition in Pristionchus nematodes. Genetics. 216 (4), 947-956 (2020).
- Fernandes Povoa, E. E., Ebbing, A. L. P., Betist, M. C., vander Veen, C., Korswagen, H. C. An optimized dissociation protocol for FACS-based isolation of rare cell types from Caenorhabditis elegans L1 larvae. MethodsX. 7, 100922(2020).
- Han, M., Wei, G., McManus, C. E., Hillier, L. W., Reinke, V. Isolated C. elegans germ nuclei exhibit distinct genomic profiles of histone modification and gene expression. BMC Genomics. 20 (1), 500(2019).
- Steiner, F. A., Henikoff, S. Cell type-specific affinity purification of nuclei for chromatin profiling in whole animals. Methods in Molecular Biology. 1228, 3-14 (2015).
- Ebbing, A. Spatial transcriptomics of C. elegans males and hermaphrodites identifies sex-specific differences in gene expression patterns. Developmental Cell. 47 (6), 801-813 (2018).
- Rödelsperger, C., et al. Spatial transcriptomics of nematodes identifies sperm cells as a source of genomic novelty and rapid evolution. Molecular Biology and Evolution. 38 (1), 229-243 (2021).
- Amini, P., et al. An optimised protocol for isolation of RNA from small sections of laser-capture microdissected FFPE tissue amenable for next-generation sequencing. BMC Molecular Biology. 18 (1), 22(2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados