Method Article
Protocolos de Triagem Experimental, Imunocitoquímica e Técnicas de Imagem Baseadas em Microscopia para Penium margaritaceum
Neste Artigo
Resumo
Este relatório descreve os métodos fundamentais usados para cultivar e manipular experimentalmente a alga estreptófita unicelular, Penium margaritaceum. Ele também fornece protocolos fundamentais de imagens baseadas em microscopia, incluindo marcação de células vivas com anticorpos monoclonais e outras sondas fluorescentes e microscopia eletrônica de varredura.
Resumo
A parede celular é o primeiro componente da recepção/transdução de sinal para uma célula vegetal durante o desenvolvimento e ao responder a estressores ambientais abióticos e bióticos. A célula monitora constantemente a integridade de sua parede celular e a modula em resposta ao estresse. Elucidar as modulações estruturais e bioquímicas específicas que ocorrem na parede celular é uma tarefa difícil, especialmente quando se emprega plantas multicelulares e seus órgãos/tecidos. Isso se deve aos limites do que pode ser resolvido em uma célula individual que faz parte de uma rede multicelular complexa. A alga estreptófita unicelular, Penium margaritaceum, tem sido recentemente usada em investigações da dinâmica da pectina, plasticidade fenotípica baseada na parede celular e múltiplos aspectos da biologia celular de algas. Seu fenótipo simples, parede celular distinta que possui muitos componentes notavelmente semelhantes às paredes celulares de plantas terrestres e facilidade em estudos imunocitoquímicos e experimentais o tornam um poderoso organismo modelo na biologia da parede celular vegetal. O objetivo deste estudo é fornecer as técnicas básicas para cultivo, manipulação experimental e triagem de estressores aplicados. Protocolos de triagem para imunocitoquímica, imagens de microscopia de varredura a laser confocal e imagens de microscopia eletrônica de varredura da estrutura da parede celular. Da mesma forma, muitas das técnicas descritas podem ser modificadas para uma ampla gama de outros estudos celulares e moleculares.
Introdução
A parede celular de uma planta é uma rede polimérica complexa que tem múltiplos papéis na vida de uma célula vegetal1. A integridade da parede celular é constantemente monitorada pela célula durante o desenvolvimento e em resposta ao estresse ambiental e modula em química e estrutura de acordo. Penium margaritaceum é uma alga verde unicelular que tem sido recentemente utilizada em estudos de algas estreptófitas (Streptophyta, o grupo de algas verdes mais intimamente relacionadas e ancestrais das plantas terrestres2).
Nas últimas duas décadas, P. margaritaceum tem sido um organismo importante nas investigações da dinâmica da parede celular e da matriz extracelular, atividades do sistema endomembranar, manifestação da forma celular e evolução das plantas 3,4,5,6,7,8,9,10,11 . O objetivo deste trabalho é fornecer aos pesquisadores da parede celular vegetal os métodos fundamentais de cultivo de P. margaritaceum, manipulando-o experimentalmente usando técnicas baseadas em microplacas e monitorando a estrutura de sua parede celular usando marcação imunocitoquímica de células vivas e imagens com técnicas de microscopia de luz e eletrônica. P. margaritaceum tem muitas semelhanças na bioquímica da parede celular com as paredes celulares primárias das plantas terrestres. Desenvolvemos vários protocolos que aproveitam o fenótipo unicelular exclusivo dessa alga e fornecem um meio rápido de estudar a dinâmica da parede celular que muitas vezes é difícil de monitorar em plantas multicelulares. Essas técnicas serão úteis para os biólogos da parede celular de plantas que desejam elucidar a dinâmica detalhada da parede celular, especialmente aquelas que lidam com pectina, e servirão como ponto de partida para estudos que lidam com biologia celular de plantas e algas estreptófitas.
Protocolo
NOTA: Penium margaritaceum é obtido na Sammlung von Algenkulturen der Universität Göttingen - Coleção de Cultura de Algas da Universidade de Göttingen, SAG; Cepa # 2640.
1. Manutenção de culturas
- Mantenha a alga em meio líquido Woods Hole (WH12) que também pode ser suplementado com extrato de água do solo (ou seja, 40 mL por L de meio). Manter as culturas a 20-25 °C com um ciclo claro: escuro de 16 h:8 h com 3,5 klux (74 μmol fótons/m2/s) de luz fluorescente branca fria.
- Prepare subculturas semanalmente e use culturas de células quando tiverem 10-14 dias de idade. As culturas de penium serão viáveis por 6 meses e podem ser usadas para iniciar subculturas durante esse período. A alga também crescerá em 1% -2% de ágar WHM solidificado.
- Para sincronização rotineira do ciclo celular, coloque as células de culturas de 10 a 14 dias no escuro por 2 a 6 semanas a 20 a 25 ° C. Após este tempo, lave as células com WHM fresco (veja abaixo) e cultura conforme descrito acima.
2. Marcação da parede celular com anticorpos monoclonais
NOTA: P. margaritaceum é coberto por uma parede celular primária que possui muitos dos mesmos constituintes encontrados nas paredes celulares primárias de plantas terrestres11. Muitos dos anticorpos monoclonais (mAbs) criados contra epítopos da parede celular de plantas terrestres reconhecem componentes da parede celular de P. margaritaceum . As fontes desses anticorpos incluem o Centro de Pesquisa de Carboidratos Complexos da Universidade da Geórgia (ccrc@uga.edu) ou Kerafast (kerafast.com). Após a marcação da parede celular de células vivas com mAbs primários específicos para epítopos da parede celular e anticorpos secundários conjugados com fluoróforo (por exemplo, FITC, TRITC), as células podem ser colocadas de volta em cultura sem afetar a saúde da célula ou a deposição da parede celular. A marcação fluorescente da parede celular permanece indefinidamente, e a parede celular recém-secretada se apresenta como zonas escuras (ou seja, não marcadas) que podem ser medidas para determinar as taxas de expansão celular e / ou marcadas novamente com mAbs ou outras sondas.
- Remova 5 mL de cultura de células líquidas em crescimento ativo (10-14 dias de idade) e coloque-o em um tubo de centrífuga de plástico de 15 mL. Centrifugue em uma centrífuga de mesa a 1.000 x g por 1 min.
- Despeje e descarte o sobrenadante. Ressuspenda o pellet em 5 ml de WHM fresco. Coloque a tampa firmemente e agite vigorosamente o tubo por 10 s para ressuspender o pellet e remover qualquer substância polimérica extracelular ou EPS da superfície da parede celular.
- Centrifugue a 1.000 x g por 1 min. Repita a etapa 2.2 e centrifugue. Ressuspenda o pellet em 1 mL de WHM fresco e transfira alíquotas de 200 μL da suspensão celular para tubos de microcentrífuga de 1,5 mL.
- Tampe os tubos e centrifugue em uma microcentrífuga a 1.000 x g. Remover o sobrenadante e ressuspender o sedimento em 400 μl de WHM fresco.
- À suspensão, adicione 20 μL de mAb (por exemplo, JIM5, um mAb de rato com especificidade para homogalacturonano esterificado com baixo teor de metila; a diluição final é de 1/20 com WHM). Vortex o tubo, envolva-o em papel alumínio e coloque-o em um rotador de laboratório por 90 min. Para obter melhores resultados, faça um vórtice no tubo 2x durante a etapa de incubação de 90 minutos.
- Centrifugue a suspensão da célula a 1.000 x g por 1 min. Remover o sobrenadante e ressuspender o sedimento em 500 μL de WHM fresco. Tampe o tubo e vortex a suspensão da célula por 10 s.
- Repita a etapa 2.6 2x. Após centrifugação, ressuspender o pellet em 400 μL de WHM e 8 μL de TRITC ou FITC anti-rato caprino (diluído 1/75 com WHM). Vortex, enrole o tubo em papel alumínio e coloque-o em um rotador de laboratório por 90 min.
- Repita as etapas 2.6 e 2.7. Ressuspenda o pellet em 100 μL de meio de crescimento, tampe o tubo e envolva-o em papel alumínio até que esteja pronto para a imagem.
NOTA: A incubação das células em WHM contendo um agente bloqueador como leite instantâneo sem gordura (1%) ou albumina de soro bovino (1%) antes da incubação nos mAbs também pode ser usada, mas em nossa experiência não faz nada para rotular a qualidade ou intensidade. O penium não expande sua parede celular nem produz grandes quantidades de EPS no escuro. O anticorpo marcado permanece intacto por pelo menos 3-4 dias. Portanto, a imagem não precisa ser realizada imediatamente. Outros mAbs podem ser aplicados aqui, mas as concentrações precisarão ser testadas. Para estudos de triagem química e física, pode-se rotular vários mL de suspensão celular, mantida no escuro por vários dias e usada em estudos de microplacas. As células também podem ser marcadas com o anticorpo primário apenas antes do tratamento, seguido pelo anticorpo secundário posteriormente.
3. Medição da parede celular e das taxas de expansão celular ao longo do tempo
- Pegue 1 mL de células marcadas com JIM5-TRITC e coloque 1 mL de WHM em um tubo de microcentrífuga de 1,5 mL. Centrifugue a 1.000 x g por 1 min e descarte o sobrenadante. Ressuspenda o pellet em 250 μL de WHM e vórtice para misturar as células.
- Para cada poço de uma placa de Petri de 12 poços, adicione 1 mL de WHM. Neste momento, inibidores específicos e reguladores de crescimento podem ser adicionados a cada poço. Neste artigo, demonstramos os efeitos do aumento dos níveis de cálcio durante a incubação.
- Pegue 30 μL de suspensão celular marcada (etapa 1) e adicione a cada poço da microplaca. Gire suavemente o prato para misturar. Selo com filme transparente.
- Cultura como acima (etapa 1.1) por 24 h, 48 h ou 72 h. Em horários especificados, remova 250 mL de suspensão celular de cada poço e coloque-o em um tubo de microcentrífuga de 1,5 μL.
- Centrifugue a 1.000 x g por 1 min. Remova o sobrenadante. Ressuspenda o pellet em 50 μL de meio de crescimento e vórtice.
- Coloque uma gota de 15 μL da suspensão da célula em uma lâmina de vidro e cubra com uma lamínula de 22 x 22 (espessura de 1.5). Observe as células com um microscópio de fluorescência usando um filtro TRITC em 10x -20x.
- Capture imagens de pelo menos 50 a 100 células. Para cada célula, meça e registre o comprimento total da célula e o comprimento da zona escura (ou seja, a nova parede produzida durante a incubação após a rotulagem).
- Determine a % média de novas paredes celulares na cultura. Repita para células coletadas após 48 h e 72 h para obter informações sobre a expansão da parede celular ao longo do tempo.
NOTA: As etapas 3.5-3.8 produzem uma suspensão de células densas que permite a aquisição de imagens de muitas células em um campo de visão. Isso torna a medição manual muito mais conveniente. Muitos programas de software de câmera fornecem medição conveniente e/ou automática das dimensões das células que podem ser usadas com esta alga. Também é possível coletar as células e rotulá-las como acima com JIM5, mas substituir o anticorpo anti-rato FITC. Usando um microscópio confocal de varredura a laser (CLSM), pode-se obter imagens de células com os filtros FITC e TRITC. Os sinais fluorescentes podem então ser atribuídos a diferentes pseudocores. Isso também permite distinguir a estrutura da nova parede celular produzida durante vários tratamentos (marcada com JIM5-FITC) em comparação com as paredes celulares pré-marcadas (marcadas com JIM5-TRITC).
4. Imagem em timelapse da expansão da parede celular
- Rotule a parede celular com JIM5-TRITC conforme descrito acima (etapas 2.1-2.8). Dilua as células 10x em WHM e adicione uma gota de 50 μL de células em uma lamínula ou placa de Petri com fundo de vidro.
- Deixe as células aderirem ao vidro por 2 minutos no escuro. Enxágue suavemente as células que não grudaram no vidro com 1 mL de WHM. Use uma micropipeta e adicione cuidadosamente WHM gota a gota ao copo.
- Adicione 30 μL de WHM em cima das células presas ao vidro. Adicione 30 μL de agarose/WHM quente a 4% em cima da gota de WHM/células. Deixe a agarose esfriar até a temperatura ambiente e solidificar.
- Para amostras em uma placa de Petri, adicione WHM suficiente para cobrir completamente as células embebidas em agarose. Para células em uma lamínula, inverta a lamínula e coloque-a suavemente sobre uma lâmina de depressão cheia de WHM.
- Monte as células em um microscópio fluorescente. A iluminação externa (lâmpada) pode ser configurada, ou a luz do próprio microscópio pode ser usada para fornecer energia para o crescimento e movimento celular. Em um microscópio Olympus Ix83 ou Ix63, uma potência de 5-6 V para a lâmpada trans funciona bem. Isso pode exigir tentativa e erro para o seu sistema.
- Imagem a cada 10-30 minutos usando o conjunto de filtros TRITC para rastreamento da expansão da parede celular. Adicione produtos químicos/enzimas ao WHM usado na etapa 3.8, se houver.
5. Observando a produção de substâncias poliméricas extracelulares (EPS)
- Preparação das células: Obtenha 5 mL de células de culturas de 10 a 14 dias e lave-as como nas etapas 1 a 4 acima.
- Preparação de esferas fluorescentes: Em um tubo de microcentrífuga de 1,5 mL, adicione 1 gota (aproximadamente 100 μL) de esferas fluorescentes de 0,75 μm (Polisfera). Adicione 1 mL de WHM ao tubo e agite vigorosamente para ressuspender os grânulos. Centrifugue a 10.000 x g por 3 min. Remova o sobrenadante. Adicione 1 mL de WHM ao pellet e agite. Centrifugue a 10.000 x g por 3 min. Ressuspenda o pellet em 500 μL de WHM.
- Preparação de poços: Adicione 1 mL de meio WHM aos poços de uma placa de 12 poços. Adicione inibidores ou reguladores de crescimento à concentração desejada e agite suavemente. Adicione 10 μL da solução de esferas e gire suavemente a placa para misturar as contas. Adicione 10 μL de células lavadas a cada poço e gire a placa suavemente.
- Cultivar a placa de células como acima (1.1). Após 24 h, coloque suavemente a placa (cuidado para não misturar) em um microscópio de fluorescência invertida equipado com um filtro FITC. As contas aderem ao EPS e revelam os padrões de liberação do EPS. Fotografe as células em 4x, 10x ou 20x.
6. Imagem em timelapse da formação de rastros de EPS
- Realize imagens de lapso de tempo em uma placa de 12 poços (etapa 3.2), conforme preparado acima, ou em uma placa de Petri individual. Siga as etapas 1 a 4. Em vez de cultivar as células sob iluminação fluorescente, as células podem ser cultivadas enquanto montadas em um microscópio fluorescente invertido.
- Células de imagem a cada 5-10 minutos para melhor capturar o movimento da célula. As esferas fluorescentes são visíveis usando um conjunto de filtros FITC, mas para contas concentradas use o canal de campo claro. A iluminação externa (lâmpada) pode ser configurada, ou a luz do próprio microscópio pode ser usada para fornecer energia para o crescimento e movimento celular. Em um microscópio Olympus Ix83, uma potência de 5-6 V para a lâmpada trans funciona bem. Isso pode exigir tentativa e erro para o seu sistema.
7. Análise estrutural correlativa da parede celular com microscopia eletrônica de varredura (MEV)
NOTA: Características alteradas da parede celular observadas em células vivas marcadas com anticorpos específicos da célula podem ser visualizadas para características detalhadas usando SEM. Essa abordagem correlativa permite a obtenção de dados ultraestruturais que podem ser comparados com os dados de fluorescência.
- Obtenha 1 mL de uma suspensão celular (controle ou tratada experimentalmente) em um tubo de microcentrífuga de 1,5 mL. Centrifugue a 4.000 x g por 1 min.
- Descarte o sobrenadante. Mergulhe o tubo que contém o pellet em nitrogênio líquido ou, se não estiver disponível, coloque-o em um freezer a -80 °C. As células congeladas podem ser armazenadas a -80 °C por vários meses.
- No momento do processamento da parede celular, retire o tubo do freezer e deixe-o descongelar por 15 min. Ressuspenda o pellet em 20 μL de WHM e coloque uma gota de suspensão de célula densa em uma lamínula de 45 mm x 50 mm.
- Coloque uma segunda lamínula em cima da gota para criar um sanduíche. Coloque em uma mesa de laboratório e pressione continuamente o sanduíche para romper as células (por exemplo, 30 s).
- Separe cuidadosamente as lamínulas de vidro e lave as células rompidas em um tubo de centrífuga de 15 mL. Centrifugue a 500 x g por 1 min.
- Despeje o sobrenadante. O pellet deve ser branco ou ligeiramente verde. Se a imagem subsequente mostrar que um número suficiente de células não foi rompido, repita a etapa 3 com o pellet aqui.
- Ressuspenda o pellet que contém paredes celulares em DH2O e repita as etapas 7.4 e 7.5. Ressuspenda o pellet em 100 μL de DH2O e coloque-o em um tubo de centrífuga de 1,5 mL.
- Obtenha um stub de Cambridge (raio de 6 mm ou 8 mm) e cole fita de carbono (EMS) em sua superfície. Em seguida, coloque gotas de 5 μL da suspensão da parede celular ressuspensa (etapa 7) na fita de carbono. Observe o número de paredes celulares usando um microscópio de dissecação. Se a suspensão for muito densa, dilua-a com D-H2O.
- Coloque o toco em um recipiente coberto e deixe secar (2 h durante a noite). Cubra o toco por 50 s usando um alvo de paládio (outros alvos também podem ser empregados). Observe as células a 5 kV, tamanho do ponto, a 10 cm do detector de elétrons secundário.
NOTA: Diluir a suspensão da parede celular antes de depositar gotas na fita de carbono limitará o depósito das paredes celulares umas nas outras.
Resultados
A marcação da parede celular de P. margaritaceum com mAbs anti-pectina (por exemplo, JIM5) revela uma rede de fibras e projeções complexadas com cálcio que formam um padrão regular ou rede (Figura 1). A pectina é depositada no centro celular ou istmo, onde desloca a pectina mais antiga em direção aos pólos (Figura 2). A marcação com um JIM7 semelhante a um anticorpo de pectina diferente destaca a secreção inicial de pectina altamente metilesterificada em uma faixa estreita no istmo ( Figura 3A, B ). Outros polímeros localizados na parede celular, incluindo a proteína arabinogalactana que é marcada com o mAb, JIM13 (Figura 4). Grandes quantidades de EPS são secretadas externamente à parede celular (Figura 5), permitindo o deslizamento e a formação de agregados celulares. Várias dessas técnicas de marcação podem ser integradas em estudos de desenvolvimento, incluindo exame quantitativo da parede celular e expansão celular (Figura 6). Da mesma forma, estudos estruturais correlativos da pectina usando imagens de MEV fornecem informações ultraestruturais detalhadas (Figura 7) que podem ser comparadas com a microscopia baseada em fluorescência de marcação de células vivas.
Os métodos fornecidos fornecem um guia fundamental para o emprego de uma alga unicelular como P. margaritaceum para vários tipos de análises. A capacidade de realizar a imunomarcação de células vivas da parede celular e acompanhar eventos de desenvolvimento subsequentes é uma vantagem particular, muitas vezes não permitida em sistemas multicelulares. P. margaritaceum também oferece uma parede celular onde a pectina está na superfície externa da parede celular (ou seja, diretamente acessível para microscopia). A aquisição de imagens e experimentos com células em poços de placas de micropoços oferece um meio conveniente para coletar dados rapidamente. Finalmente, os meios para correlacionar dados CLSM com dados SEM fornecem um canal valioso para comparar eventos de células vivas com informações ultraestruturais.

Figura 1: Marcação da rede de pectina com o anticorpo monoclonal. A especificidade do anticorpo JIM5 é homogalacturonano de baixa esterificação. Observe as projeções distintas (setas brancas) na superfície da parede celular. A zona de expansão (seta preta) é onde novos materiais da parede celular são adicionados. Clique aqui para ver uma versão maior desta figura.

Figura 2: Marcação de células JIM5 durante o desenvolvimento. A célula foi inicialmente marcada com JIM5-TRITC (laranja e *) e colocada de volta em cultura. Após 24 h, as células foram coletadas e marcadas com JIM5-FITC (verde e seta). Esta zona representa um novo crescimento. Clique aqui para ver uma versão maior desta figura.

Figura 3: Marcação JIM7 de pectina altamente esterificada. (A) Marcação de pectina altamente esterificada com o anticorpo monoclonal, JIM7 (especificidade: homogalacturonano altamente esterificado) na zona de expansão. Observe a camada estreita (seta branca) na superfície da parede celular. (B) Co-marcação de pectina esterificada alta e baixa da parede celular durante a expansão celular. A célula foi inicialmente marcada com JIM5-TRITC (laranja e *) e colocada de volta em cultura. Às 24 h depois, as células foram coletadas e marcadas com JIM7-FITC (verde e seta). As zonas escuras entre as zonas rotuladas representam pectina recém-adicionada (setas pretas). Clique aqui para ver uma versão maior desta figura.

Figura 4: Proteínas arabinolactanas da parede celular. Marcação das proteínas arabinogalactanas da superfície da parede celular (AGPs) com o anticorpo monoclonal, JIM13. Observe a associação frouxa dos AGPs com a parede celular (setas). Clique aqui para ver uma versão maior desta figura.
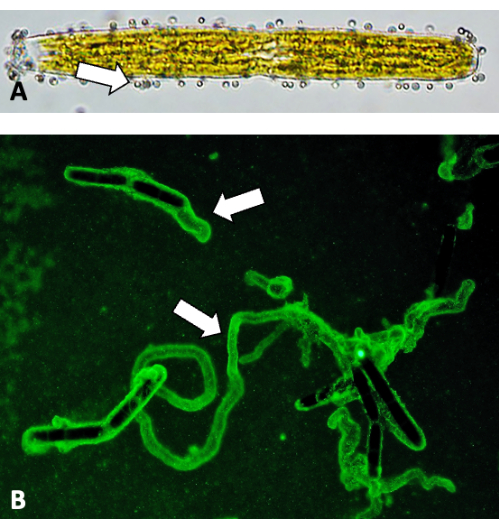
Figura 5: Imagens de trilhas planadoras com esferas fluorescentes. (A) Contas fluorescentes de 0,75 μm presas à superfície da parede celular (setas). (B) Encadernação de esferas fluorescentes de trilhas de EPS. Clique aqui para ver uma versão maior desta figura.
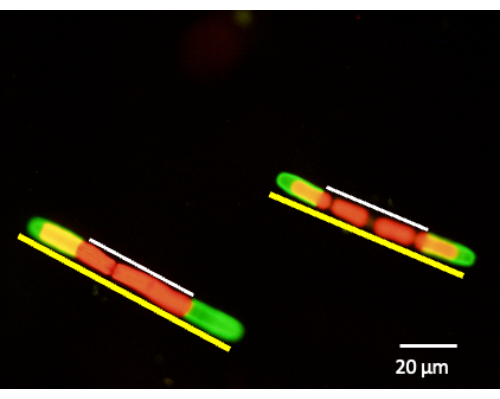
Figura 6: A medição da expansão da parede celular pode ser realizada após a marcação JIM5-TRITC. As células são colocadas de volta em cultura e deixadas crescer por 24 h, 48 h e 72 h. Alíquotas de células podem ser removidas em um período de tempo específico e visualizadas com um microscópio confocal de varredura a laser de fluorescência. A porcentagem de novo crescimento é determinada medindo a parede celular não marcada (linha branca) versus todo o comprimento da célula (linha amarela). A fluorescência vermelha é a autofluorescência da clorofila. Clique aqui para ver uma versão maior desta figura.

Figura 7: Estudos correlativos. (A) A rede típica é feita de uma malha de fibras que muitas vezes terminam externamente em projeções distintas (seta). (B) Quando tratada com grandes quantidades de cálcio, a rede se transforma em depósitos irregulares (setas). Célula marcada com JIM5. (C) Quando as paredes celulares são tratadas como em (B) e examinadas com MEV, a pectina desorganizada (seta) é observada em detalhes. Clique aqui para ver uma versão maior desta figura.
Discussão
P. margaritaceum é um organismo eficaz para elucidar a dinâmica do desenvolvimento da parede celular e secreção de MEC em plantas e algas estreptófitas. Os principais atributos incluem um hábito unicelular e facilidade na manutenção da cultura e manipulação experimental, uma parede celular primária com uma rede de pectina externa distinta e outros polímeros, facilidade na marcação de células vivas com mAbs direcionados à parede celular que podem ser seguidos a tempo para estudos subsequentes de desenvolvimento e/ou experimentais e a produção de grandes quantidades de EPS que podem ser monitoradas em tempo real usando esferas fluorescentes1, 2,3,6. Além disso, a maioria das técnicas de rotulagem oferece meios simples para medições quantitativas.
P. maragaritaceum é um organismo robusto que pode acomodar algumas mudanças modestas nos protocolos descritos aqui. No entanto, é importante ressaltar que a) para imunocitoquímica e manipulação experimental, utilizar células de culturas com menos de 21 dias de idade; b) cultura em temperaturas não superiores a 28 °C; c) manter as diluições de anticorpos conforme prescrito. A marcação fluorescente JIM5-TRITC durará vários dias se as células forem mantidas no escuro. As células marcadas com JIM7 devem ser visualizadas dentro de 24 h. Para a marcação de esferas fluorescentes, a imagem das células é melhor em 24 horas. Para experimentos específicos, especialmente aqueles que usam ensaios de microplacas, várias concentrações de agentes devem primeiro ser testadas para encontrar o mais ativo. Também é fundamental que experimentos de recuperação sejam realizados para ter certeza de que um agente não simplesmente alterou a viabilidade da cultura de células.
Modificações simples nos protocolos descritos podem ser facilmente feitas para se adequar aos objetivos do pesquisador. Muitos outros anticorpos estão disponíveis para estudar a parede celular que ainda não foi testada com P. margaritaceum. Para experimentos específicos, especialmente aqueles que usam o ensaio de microplaca, várias concentrações de agentes devem primeiro ser testadas para encontrar o mais ativo. A imagem em microplacas de células marcadas com os anticorpos fluorescentes pode ser avaliada rapidamente usando a triagem baseada em microscopia de poços individuais. Também é fundamental que experimentos de recuperação sejam realizados para garantir que um agente não altere a viabilidade da cultura de células.
Divulgações
Nenhum conflito de interesses é relatado.
Agradecimentos
Este trabalho foi apoiado pela National Science Foundation (NSF) (número de concessão MCB 2129443 para DD).
Materiais
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcent. Tubes | Fisher Scientific | 01-549-740 | |
| 12 welled microplate | Fisher Scientific | 50-233-6077 | |
| 22 x 22 mm coverslips | Fisher Scientific | 12-541-016 | |
| 45x 50 cm coverslips | Brain Research | 4550-1.5D | |
| Agar | Sigma Aldrich | A9414 | |
| anti-rat FITC | Sigma Aldrich | F6258 | |
| anti-rat TRITC | Sigma Aldrich | T4280 | |
| calcium chloride | Sigma Aldrich | C4981 | |
| Cambbridge stubs | EMS | 75183-65 | |
| Fluoview CLSM | Evident | Fluoview 1200 | |
| JIM5 | Kerafast | ELD004 | |
| JIM7 | Kerafast | ELD005 | |
| Microcentrifuge | Fisher Scientific | 13-100-675 | |
| Micropipetors | BioRad | 1660499EDU | |
| Penium margaritaceum | Sammlung von Algenkulturen der Universität Göttingen - Culture Collection of Algae at Göttingen University | 2640 | |
| Polysphere kit | Polysciences | 18336 | |
| SEM | ThermoFisher | Quattro SEM | |
| sputter coater | EMS | Q150V | |
| Vortex mixer | Fisher Scientific | 02-215-414 |
Referências
- Delmer, D., Dixon, R. A., Keegstra, K., Mohnen, D. The plant cell wall-dynamic, strong, and adaptable-is a natural shapeshifter. Plant Cell. 36 (5), 1257-1311 (2024).
- Bierenbroodspot, M. J., et al. Phylogeny and evolution of streptophyte algae. Ann Bot. 134 (3), 385-400 (2024).
- Domozych, D. S., et al. Endomembrane architecture and dynamics during secretion of the extracellular matrix of the unicellular charophyte, Penium margaritaceum. J Exp Bot. 71 (11), 3323-3339 (2020).
- Feng, X., et al. Genomes of multicellular algal sisters to land plants illuminate signaling network evolution. Nat Genet. 56 (5), 1018-1031 (2024).
- Jiao, C., et al. The Penium margaritaceum genome: Hallmarks of the origins of land plants. Cell. 181, 1097-1111 (2020).
- LoRicco, J. G., et al. The multifunctional roles of the extracellular matrix in the sessile life of the zygnematophyte Penium margaritaceum: stick, glide and cluster. Physiologie Plantarum. 176 (5), e14520 (2024).
- LoRicco, J. G., et al. Aberrant growth and expansion in Penium margaritaceum triggered by disruption of microtubules and the cell wall. J Exp Bot. erae387, (2024).
- LoRicco, J. G., et al. Chemically induced phenotype plasticity in the unicellular zygnematophyte, Penium margaritaceum. Protoplasma. 261 (6), 1233-1249 (2024).
- Davis, D. J., et al. Callose deposition is essential for the completion of cytokinesis in the unicellular alga, Penium margaritaceum. J Cell Sci. 133 (19), jcs.249599 (2020).
- Rydahl, M. G., et al. Penium margaritaceum as a model organism for cell wall analysis of expanding plant cells. Methods Mol Biol. 1242, 1-22 (2015).
- Carrillo-Carrasco, V. P., Hernández-García, J., Weijers, D. Electroporation-based delivery of proteins in Penium margaritaceum and other zygnematophycean algae. Physiol Plant. 175 (6), e14121 (2023).
- Nichols, H. W. Growth media-freshwater. Handbook Phycol Methods. , 16-17 (1973).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados