Method Article
Protocolli di Screening Sperimentale, Tecniche di Immunocitochimica e Imaging Microscopico per Penium margaritaceum
In questo articolo
Riepilogo
Questo rapporto descrive i metodi fondamentali utilizzati per coltivare e manipolare sperimentalmente l'alga streptofita unicellulare, Penium margaritaceum. Fornisce inoltre protocolli fondamentali di imaging basato sulla microscopia, tra cui la marcatura di cellule vive con anticorpi monoclonali e altre sonde fluorescenti e la microscopia elettronica a scansione.
Abstract
La parete cellulare è il primo componente della ricezione/trasduzione del segnale per una cellula vegetale durante lo sviluppo e quando risponde a fattori di stress abiotici e biotici ambientali. La cellula monitora costantemente l'integrità della sua parete cellulare e la modula in risposta allo stress. Chiarire le specifiche modulazioni strutturali e biochimiche che si verificano nella parete cellulare è un compito difficile, soprattutto quando si impiegano piante multicellulari e i loro organi/tessuti. Ciò è dovuto ai limiti su ciò che può essere risolto in una singola cellula che fa parte di una complessa rete multicellulare. L'alga streptofita unicellulare, Penium margaritaceum, è stata recentemente utilizzata in studi sulla dinamica delle pectine, sulla plasticità fenotipica basata sulla parete cellulare e su molteplici aspetti della biologia cellulare algale. Il suo fenotipo semplice, la parete cellulare distinta che ha molti componenti notevolmente simili alle pareti cellulari delle piante terrestri e la facilità negli studi immunocitochimici e sperimentali lo rendono un potente organismo modello nella biologia della parete cellulare vegetale. L'obiettivo di questo studio è quello di fornire le tecniche di base per la coltura, la manipolazione sperimentale e lo screening dei fattori di stress applicati. Protocolli di screening per immunocitochimica, imaging al microscopio a scansione laser confocale e imaging al microscopio elettronico a scansione della struttura della parete cellulare. Allo stesso modo, molte delle tecniche descritte possono essere modificate per una vasta gamma di altri studi cellulari e molecolari.
Introduzione
La parete cellulare di una pianta è una complessa rete polimerica che ha molteplici ruoli nella vita di una cellula vegetale1. L'integrità della parete cellulare è costantemente monitorata dalla cellula durante lo sviluppo e in risposta allo stress ambientale e, modula di conseguenza in chimica e struttura. Penium margaritaceum è un'alga verde unicellulare che è stata recentemente utilizzata negli studi sulle alghe streptofite (Streptophyta, il gruppo di alghe verdi più strettamente imparentato e ancestrale alle piante terrestri2).
Negli ultimi due decenni, P. margaritaceum è stato un organismo importante nelle indagini sulla parete cellulare e sulla dinamica della matrice extracellulare, sulle attività del sistema endomembrana, sulla manifestazione della forma cellulare e sull'evoluzione delle piante 3,4,5,6,7,8,9,10,11 . L'obiettivo di questo lavoro è quello di fornire ai ricercatori della parete cellulare vegetale i metodi fondamentali per coltivare P. margaritaceum, manipolarlo sperimentalmente utilizzando tecniche basate su micropiastre e monitorare la struttura della sua parete cellulare utilizzando la marcatura immunocitochimica su cellule vive e l'imaging con tecniche di microscopia ottica ed elettronica. P. margaritaceum ha molte somiglianze nella biochimica delle pareti cellulari con le pareti cellulari primarie delle piante terrestri. Abbiamo ideato diversi protocolli che sfruttano il fenotipo unicellulare unico di quest'alga e forniscono un mezzo rapido per studiare le dinamiche della parete cellulare che sono spesso difficili da monitorare nelle piante multicellulari. Queste tecniche saranno di aiuto ai biologi della parete cellulare delle piante che desiderano chiarire le dinamiche dettagliate della parete cellulare, in particolare quelle che si occupano della pectina, e serviranno come punto di partenza per gli studi che si occupano della biologia cellulare delle alghe delle piante e degli streptofiti.
Protocollo
NOTA: Il Penium margaritaceum è ottenuto presso la Sammlung von Algenkulturen der Universität Göttingen - Collezione di colture di alghe presso l'Università di Gottinga, SAG; Ceppo #2640.
1. Mantenimento delle culture
- Mantenere l'alga in un terreno liquido Woods Hole (WH12) che può anche essere integrato con estratto di acqua del suolo (cioè 40 ml per litro di terreno). Mantenere le colture a 20-25 °C con un ciclo luce: buio di 16 h:8 h con 3,5 klux (74 μmol di fotoni/m2/s) di luce fluorescente bianca fredda.
- Prepara le sottocolture settimanalmente e usa le colture cellulari quando hanno 10-14 giorni. Le colture di penium saranno vitali per 6 mesi e possono essere utilizzate per avviare le sottocolture durante questo periodo. L'alga crescerà anche con l'1%-2% di WHM solidificato con agar.
- Per la sincronizzazione di routine del ciclo cellulare, posizionare al buio le cellule provenienti da colture di 10-14 giorni per 2-6 settimane a 20-25 °C. Trascorso questo tempo, lavare le cellule con WHM fresco (vedi sotto) e coltura come descritto sopra.
2. Marcatura della parete cellulare con anticorpi monoclonali
NOTA: P. margaritaceum è ricoperto da una parete cellulare primaria che ha molti degli stessi costituenti che si trovano nelle pareti cellulari primarie delle piante terrestri11. Molti degli anticorpi monoclonali (mAb) sollevati contro gli epitopi della parete cellulare delle piante terrestri riconoscono i componenti della parete cellulare di P. margaritaceum . Le fonti di questi anticorpi includono il Complex Carbohydrate Research Center dell'Università della Georgia (ccrc@uga.edu) o Kerafast (kerafast.com). Dopo la marcatura della parete cellulare di cellule vive con mAb primari specifici per epitopi della parete cellulare e anticorpi secondari coniugati con fluorofori (ad es. FITC, TRITC), le cellule possono essere rimesse in coltura senza influire sulla salute della cellula o sulla deposizione della parete cellulare. La marcatura fluorescente della parete cellulare rimane a tempo indeterminato e la parete cellulare appena secreta si presenta come zone scure (cioè non marcate) che possono essere misurate per determinare i tassi di espansione cellulare e/o marcate nuovamente con mAb o altre sonde.
- Rimuovere 5 mL di coltura cellulare liquida in crescita attiva (10-14 giorni) e metterli in una provetta da centrifuga di plastica da 15 mL. Centrifugare su una centrifuga da tavolo a 1.000 x g per 1 min.
- Versare e gettare il surnatante. Risospendere il pellet in 5 mL di WHM fresco. Posizionare il tappo saldamente e agitare energicamente la provetta per 10 s per risospendere il pellet e rimuovere qualsiasi sostanza polimerica extracellulare o EPS dalla superficie della parete cellulare.
- Centrifugare a 1.000 x g per 1 min. Ripetere il passaggio 2.2 e centrifugare. Risospendere il pellet in 1 mL di WHM fresco e trasferire 200 μL di aliquote della sospensione cellulare in provette per microcentrifuga da 1,5 mL.
- Tappare le provette e centrifugare in una microcentrifuga a 1.000 x g. Rimuovere il surnatante e risospendere il pellet in 400 μL di WHM fresco.
- Alla sospensione, aggiungere 20 μL di mAb (ad es. JIM5, un mAb di ratto con specificità per l'omogalatturone esterificato a basso metile; la diluizione finale è 1/20 con WHM). Agitare la provetta, avvolgerla in un foglio di alluminio e posizionarla su un rotatore da laboratorio per 90 minuti. Per ottenere i migliori risultati, agitare la provetta 2 volte durante la fase di incubazione di 90 minuti.
- Centrifugare la sospensione cellulare a 1.000 x g per 1 minuto. Rimuovere il surnatante e risospendere il pellet in 500 μL di WHM fresco. Tappare il tubo e agitare la sospensione cellulare per 10 s.
- Ripetere il passaggio 2.6 2 volte. Dopo la centrifugazione, risospendere il pellet in 400 μL di WHM e 8 μL di capra anti-ratto TRITC o FITC (diluito 1/75 con WHM). Vortex, avvolgere la provetta in un foglio di alluminio e posizionarla su un rotatore da laboratorio per 90 minuti.
- Ripetere i passaggi 2.6 e 2.7. Risospendere il pellet in 100 μL di terreno di coltura, tappare il tubo e avvolgerlo in un foglio di alluminio fino al momento dell'immagine.
NOTA: L'incubazione delle cellule in WHM contenenti un agente bloccante come il latte istantaneo di garofano senza grassi (1%) o l'albumina sierica bovina (1%) prima dell'incubazione negli anticorpi monoclonali può anche essere utilizzata, ma nella nostra esperienza non fa nulla per etichettare la qualità o l'intensità. Il penio non espande la sua parete cellulare né produce grandi quantità di EPS al buio. L'anticorpo marcato rimane intatto per almeno 3-4 giorni. Pertanto, l'imaging non deve essere eseguito immediatamente. In questo caso è possibile applicare altri anticorpi monoclonali, ma è necessario testare le concentrazioni. Per gli studi di screening chimico e fisico, è possibile marcare diversi mL di sospensione cellulare, tenuti al buio per diversi giorni e utilizzati negli studi su micropiastre. Le cellule possono anche essere marcate con l'anticorpo primario solo prima del trattamento, seguito dall'anticorpo secondario in seguito.
3. Misurazione della parete cellulare e dei tassi di espansione delle celle nel tempo
- Prelevare 1 mL di cellule marcate con JIM5-TRITC e inserire 1 mL di WHM in una provetta da microcentrifuga da 1,5 mL. Centrifugare a 1.000 x g per 1 minuto ed eliminare il surnatante. Risospendere il pellet in 250 μL di WHM e vorticare per mescolare le cellule.
- A ciascun pozzetto di una capsula di Petri a 12 pozzetti aggiungere 1 mL di WHM. A questo punto, è possibile aggiungere inibitori e regolatori di crescita specifici a ciascun pozzetto. In questo articolo, dimostriamo gli effetti dell'aumento dei livelli di calcio durante l'incubazione.
- Prelevare 30 μl di sospensione cellulare marcata (fase 1) e aggiungerle a ciascun pozzetto della micropiastra. Agitare delicatamente il piatto per mescolare. Sigillare con pellicola trasparente.
- Coltura come sopra (passaggio 1.1) per 24 ore, 48 ore o 72 ore. A orari specificati, rimuovere 250 mL di sospensione cellulare da ciascun pozzetto e metterlo in una provetta da microcentrifuga da 1,5 μL.
- Centrifugare a 1.000 x g per 1 min. Rimuovere il surnatante. Risospendere il pellet in 50 μL di terreno di crescita e vortice.
- Posizionare una goccia di 15 μl della sospensione cellulare su un vetrino e coprire con un vetrino coprioggetti 22 x 22 (spessore 1,5). Osservare le cellule con un microscopio a fluorescenza utilizzando un filtro TRITC a 10x -20x.
- Cattura immagini di almeno 50-100 cellule. Per ogni cellula, misurare e registrare la lunghezza totale della cellula e la lunghezza della zona scura (cioè la nuova parete prodotta durante l'incubazione dopo la marcatura).
- Determinare la percentuale media di nuove pareti cellulari nella coltura. Ripetere per le cellule raccolte dopo 48 ore e 72 ore per ottenere informazioni sull'espansione della parete cellulare nel tempo.
NOTA: I passaggi 3.5-3.8 producono una sospensione cellulare densa che consente l'imaging di molte cellule in un unico campo visivo. Questo rende la misurazione manuale molto più comoda. Molti programmi software per fotocamere forniscono una misurazione comoda e/o automatica delle dimensioni delle celle che possono essere utilizzate con questa alga. Si possono anche raccogliere le cellule ed etichettarle come sopra con JIM5 ma sostituire FITC anti-ratto come anticorpo secondario. Utilizzando un microscopio a scansione laser confocale (CLSM), è possibile visualizzare le cellule con entrambi i filtri FITC e TRITC. I segnali fluorescenti possono quindi essere assegnati a diversi pseudocolori. Ciò consente anche di distinguere la struttura della nuova parete cellulare prodotta durante vari trattamenti (marcata con JIM5-FITC) rispetto alle pareti cellulari pre-marcate (marcate con JIM5-TRITC).
4. Imaging timelapse dell'espansione della parete cellulare
- Etichettare la parete cellulare con JIM5-TRITC come descritto sopra (passaggi 2.1-2.8). Diluire le cellule 10 volte in WHM e aggiungere una goccia di 50 μl di cellule su un vetrino coprioggetti o su una piastra di Petri con fondo di vetro.
- Lasciare che le celle aderiscano al vetro per 2 minuti al buio. Sciacquare delicatamente le cellule che non si sono attaccate al bicchiere con 1 mL di WHM. Utilizzare una micropipetta e aggiungere con cura WHM goccia a goccia al bicchiere.
- Aggiungere 30 μl di WHM sopra le celle attaccate al vetro. Aggiungere 30 μl di agarosio caldo al 4%/WHM sopra la gocciolina di WHM/cellule. Lasciare raffreddare l'agarosio a temperatura ambiente e solidificare.
- Per i campioni in una capsula di Petri, aggiungere una quantità di WHM sufficiente a coprire completamente le cellule incorporate nell'agarosio. Per le cellule su un vetrino coprioggetti, capovolgere il vetrino coprioggetti e posizionarlo delicatamente su un vetrino a depressione riempito di WHM.
- Montare le cellule su un microscopio a fluorescenza. È possibile impostare l'illuminazione esterna (lampada) o la luce del microscopio stesso per fornire energia per la crescita e il movimento cellulare. Su un microscopio Olympus Ix83 o Ix63, una potenza di 5-6 V per la lampada trans funziona bene. Ciò potrebbe richiedere tentativi ed errori per il sistema.
- Immagine ogni 10-30 minuti utilizzando il set di filtri TRITC per il tracciamento dell'espansione della parete cellulare. Aggiungere sostanze chimiche/enzimi al WHM utilizzato nel passaggio 3.8, se presenti.
5. Osservazione della produzione di sostanze polimeriche extracellulari (EPS)
- Preparazione delle cellule: Prelevare 5 mL di cellule da colture di 10-14 giorni e lavarle come nei passaggi 1-4 precedenti.
- Preparazione delle microsfere fluorescenti: A una provetta da microcentrifuga da 1,5 mL, aggiungere 1 goccia (circa 100 μL) di microsfere fluorescenti da 0,75 μm (Polisfera). Aggiungere 1 mL di WHM alla provetta e agitare energicamente per risospendere le perline. Centrifugare a 10.000 x g per 3 min. Rimuovere il surnatante. Aggiungere 1 ml di WHM al pellet e agitare. Centrifugare a 10.000 x g per 3 min. Risospendere il pellet in 500 μL di WHM.
- Preparazione dei pozzetti: Aggiungere 1 mL di terreno WHM ai pozzetti di una piastra a 12 pozzetti. Aggiungere inibitori o regolatori di crescita alla concentrazione desiderata e agitare delicatamente. Aggiungere 10 μl della soluzione di perline e agitare delicatamente la piastra per mescolare le microsfere. Aggiungere 10 μl di cellule lavate a ciascun pozzetto e agitare delicatamente la piastra.
- Coltivare la piastra di cellule come sopra (1.1). Dopo 24 h, posizionare delicatamente la piastra (facendo attenzione a non mescolare) su un microscopio a fluorescenza invertita dotato di filtro FITC. Le perline aderiscono all'EPS e rivelano i modelli di rilascio dell'EPS. Fotografa le celle a 4x, 10x o 20x.
6. Imaging timelapse della formazione di scie EPS
- Eseguire l'imaging time-lapse in una piastra a 12 pozzetti (passaggio 3.2), come preparato sopra, o in una singola piastra di Petri. Segui i passaggi 1-4. Piuttosto che coltivare le cellule sotto l'illuminazione fluorescente, le cellule possono essere coltivate mentre sono montate su un microscopio a fluorescenza invertito.
- Immagini di celle ogni 5-10 minuti per catturare al meglio il movimento delle celle. Le perle fluorescenti sono visibili utilizzando un set di filtri FITC, ma per le perle concentrate utilizzare il canale del campo chiaro. È possibile impostare l'illuminazione esterna (lampada) o la luce del microscopio stesso per fornire energia per la crescita e il movimento cellulare. Su un microscopio Olympus Ix83, una potenza di 5-6 V per la lampada trans funziona bene. Ciò potrebbe richiedere tentativi ed errori per il sistema.
7. Analisi strutturale correlativa della parete cellulare con microscopia elettronica a scansione (SEM)
NOTA: Le caratteristiche alterate della parete cellulare osservate nelle cellule vive marcate con anticorpi specifici della cellula possono essere visualizzate per caratteristiche dettagliate utilizzando il SEM. Questo approccio correlativo consente di ottenere dati ultrastrutturali che possono essere confrontati con i dati di fluorescenza.
- Ottenere 1 mL di una sospensione cellulare (di controllo o trattata sperimentalmente) in una provetta da microcentrifuga da 1,5 mL. Centrifugare a 4.000 x g per 1 min.
- Scartare il surnatante. Immergere il tubo contenente il pellet nell'azoto liquido o, se non disponibile, metterlo in un congelatore a -80 °C. Le celle congelate possono essere conservate a -80 °C per diversi mesi.
- Al momento dell'elaborazione della parete cellulare, rimuovere il tubo dal congelatore e lasciarlo scongelare per 15 minuti. Risospendere il pellet in 20 μL di WHM e posizionare una goccia di sospensione cellulare densa su un vetrino coprioggetti da 45 mm x 50 mm.
- Posiziona un secondo vetrino coprioggetti sopra la goccia per creare un panino. Posizionare su un tavolo da laboratorio e premere continuamente sul sandwich per rompere le cellule (ad es. 30 s).
- Separare accuratamente i vetrini coprioggetti e lavare le cellule rotte in una provetta da centrifuga da 15 mL. Centrifugare a 500 x g per 1 min.
- Versare il surnatante. Il pellet deve essere bianco o leggermente verde. Se l'imaging successivo mostra che un numero sufficiente di cellule non è stato rotto, ripetere qui il passaggio 3 con il pellet.
- Risospendere il pellet che contiene le pareti cellulari in D-H2O e ripetere i passaggi 7.4 e 7.5. Risospendere il pellet in 100 μL di D-H2O e metterlo in una provetta da centrifuga da 1,5 mL.
- Procurarsi un tronchetto Cambridge (raggio di 6 mm o 8 mm) e far aderire il nastro di carbonio (EMS) alla sua superficie. Quindi, posizionare 5 μL di gocce della sospensione della parete cellulare risoluta (passaggio 7) sul nastro di carbonio. Osservare il numero di pareti cellulari utilizzando un microscopio da dissezione. Se la sospensione è troppo densa, diluirla con D-H2O.
- Metti il mozzicone in un contenitore coperto e lascialo asciugare (da 2 ore a tutta la notte). Rivestire lo stub per 50 s utilizzando un bersaglio di palladio (possono essere utilizzati anche altri bersagli). Osservare le celle a 5 kV, dimensione del punto, a 10 cm dal rivelatore di elettroni secondario.
NOTA: Diluire la sospensione della parete cellulare prima di depositare le gocce sul nastro di carbonio limiterà il deposito delle pareti cellulari l'una sull'altra.
Risultati
L'etichettatura della parete cellulare di P. margaritaceum con anticorpi monoclonali anti-pectina (ad esempio, JIM5) rivela una rete di fibre e proiezioni complessate con calcio che formano un modello regolare o reticolo (Figura 1). La pectina si deposita nel centro della cellula o istmo, dove sposta la pectina più vecchia verso i poli (Figura 2). La marcatura con un diverso anticorpo pectina-simile a JIM7 evidenzia la secrezione iniziale di pectina altamente metil-esterificata in una banda stretta all'istmo (Figura 3A, B). Altri polimeri situati nella parete cellulare, tra cui la proteina arabinogalattano marcata con l'anticorpo monoclonale, JIM13 (Figura 4). Grandi quantità di EPS vengono secrete esternamente alla parete cellulare (Figura 5), consentendo lo scivolamento e la formazione di aggregati cellulari. Molte di queste tecniche di marcatura possono essere integrate negli studi sullo sviluppo, tra cui l'esame quantitativo della parete cellulare e dell'espansione cellulare (Figura 6). Allo stesso modo, gli studi strutturali correlati della pectina utilizzando l'imaging SEM forniscono informazioni ultrastrutturali dettagliate (Figura 7) che possono essere confrontate con la microscopia basata sulla fluorescenza per la marcatura di cellule vive.
I metodi forniti forniscono una guida fondamentale per l'impiego di un'alga unicellulare come la P. margaritaceum per vari tipi di analisi. La capacità di eseguire l'immunomarcatura della parete cellulare con cellule vive e di seguire i successivi eventi di sviluppo è un vantaggio particolare spesso non consentito con i sistemi multicellulari. P. margaritaceum offre anche una parete cellulare in cui la pectina si trova sulla superficie della parete cellulare esterna (cioè direttamente accessibile per la microscopia). L'imaging e la sperimentazione con le cellule nei pozzetti delle piastre a micropozzetti offrono un mezzo conveniente per raccogliere rapidamente i dati. Infine, i mezzi per correlare i dati CLSM con i dati SEM forniscono un prezioso canale per confrontare gli eventi di cellule vive con le informazioni ultrastrutturali.

Figura 1: Marcatura del reticolo della pectina con l'anticorpo monoclonale. La specificità dell'anticorpo JIM5 è l'omogalatturonano poco esterificato. Si notino le sporgenze distinte (frecce bianche) sulla superficie della parete cellulare. La zona di espansione (freccia nera) è quella in cui vengono aggiunti nuovi materiali per le pareti cellulari. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Marcatura JIM5 delle cellule durante lo sviluppo. La cellula è stata inizialmente marcata con JIM5-TRITC (arancione e *) e riposta in coltura. Dopo 24 ore, le cellule sono state raccolte e marcate con JIM5-FITC (verde e freccia). Questa zona rappresenta una nuova crescita. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Marcatura JIM7 della pectina altamente esterificata. (A) Marcatura della pectina altamente esterificata con l'anticorpo monoclonale JIM7 (specificità: omogalatturonano altamente esterificato) nella zona di espansione. Si noti lo strato stretto (freccia bianca) sulla superficie della parete cellulare. (B) Co-marcatura di pectina alta e bassa esterificata della parete cellulare durante l'espansione cellulare. La cellula è stata inizialmente marcata con JIM5-TRITC (arancione e *) e riposta in coltura. A 24 ore dopo, le cellule sono state raccolte e marcate con JIM7-FITC (verde e freccia). Le zone scure tra le zone etichettate rappresentano la pectina appena aggiunta (frecce nere). Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: Proteine arabinoglactan della parete cellulare. Marcatura delle proteine arabinogalattano (AGP) della superficie della parete cellulare con l'anticorpo monoclonale JIM13. Si noti l'associazione libera degli AGP con la parete cellulare (frecce). Clicca qui per visualizzare una versione più grande di questa figura.
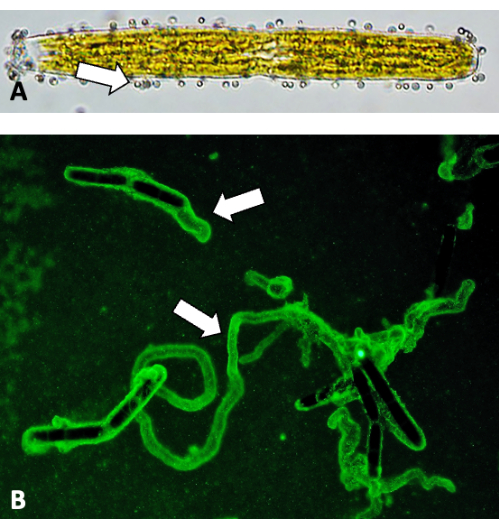
Figura 5: Imaging delle scie di scorrimento con perline fluorescenti. (A) Sfere fluorescenti da 0,75 μm attaccate alla superficie della parete cellulare (frecce). (B) Legatura a perline fluorescenti di scie in EPS. Clicca qui per visualizzare una versione più grande di questa figura.
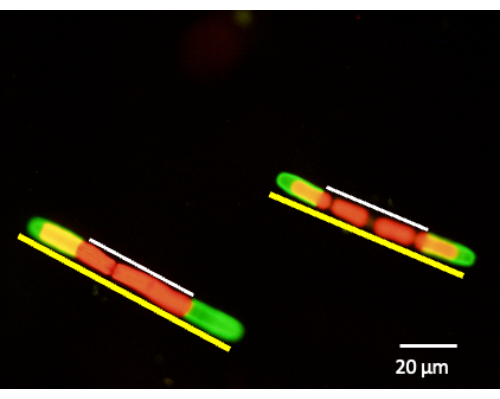
Figura 6: La misurazione dell'espansione della parete cellulare può essere eseguita dopo la marcatura JIM5-TRITC. Le cellule vengono rimesse in coltura e lasciate crescere per 24 ore, 48 ore e 72 ore. Le aliquote di cellule possono essere rimosse in un periodo di tempo specifico e visualizzate con un microscopio a scansione laser confocale a fluorescenza. La percentuale di nuova crescita è determinata misurando la parete cellulare non marcata (linea bianca) rispetto all'intera lunghezza della cellula (linea gialla). La fluorescenza rossa è l'autofluorescenza della clorofilla. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 7: Studi sui corelativi. (A) Il reticolo tipico è costituito da una rete di fibre che spesso terminano esternamente in proiezioni distinte (freccia). (B) Se trattato con grandi quantità di calcio, il reticolo si trasforma in depositi irregolari (frecce). Cella etichettata JIM5. (C) Quando le pareti cellulari vengono trattate come in (B) ed esaminate con SEM, la pectina disorganizzata (freccia) viene osservata in dettaglio. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
P. margaritaceum è un organismo efficace per chiarire le dinamiche dello sviluppo della parete cellulare e della secrezione di ECM nelle piante e nelle alghe streptofite. Le caratteristiche principali includono un'abitudine unicellulare e facilità nel mantenimento della coltura e nella manipolazione sperimentale, una parete cellulare primaria con un reticolo esterno di pectina distinto e altri polimeri, facilità nella marcatura di cellule vive con anticorpi monoclonali diretti alla parete cellulare che possono essere seguiti in tempo per successivi studi di sviluppo e/o sperimentali e la produzione di grandi quantità di EPS che possono essere monitorate in tempo reale utilizzando perle fluorescenti1, 2,3,6. Inoltre, la maggior parte delle tecniche di etichettatura offre mezzi semplici per le misurazioni quantitative.
P. maragaritaceum è un organismo robusto che può adattarsi ad alcune modeste modifiche ai protocolli qui descritti. Tuttavia, è importante notare che a) per l'immunocitochimica e la manipolazione sperimentale, utilizzare cellule provenienti da colture di età inferiore a 21 giorni; b) coltura a temperature non superiori a 28 °C; c) mantenere le diluizioni anticorpali come prescritto. L'etichettatura fluorescente JIM5-TRITC durerà per diversi giorni se le cellule vengono tenute al buio. Le cellule marcate con JIM7 devono essere visualizzate entro 24 ore. Per l'etichettatura con microsfere fluorescenti, l'imaging delle cellule è migliore entro 24 ore. Per esperimenti specifici, in particolare quelli che utilizzano saggi su micropiastra, è necessario testare prima varie concentrazioni di agenti per trovare quello più attivo. È anche fondamentale che vengano effettuati esperimenti di recupero per essere sicuri che un agente non abbia semplicemente modificato la vitalità della coltura cellulare.
Semplici modifiche ai protocolli descritti possono essere facilmente apportate per adattarle agli obiettivi del ricercatore. Molti altri anticorpi sono disponibili per studiare la parete cellulare che deve ancora essere provata con P. margaritaceum. Per esperimenti specifici, in particolare quelli che utilizzano il test su micropiastra, è necessario testare prima varie concentrazioni di agenti per trovare quello più attivo. L'imaging su micropiastra di cellule marcate con gli anticorpi fluorescenti può quindi essere valutato rapidamente utilizzando lo screening basato sulla microscopia dei singoli pozzetti. È inoltre fondamentale che vengano effettuati esperimenti di recupero per essere sicuri che un agente non abbia modificato la vitalità della coltura cellulare.
Divulgazioni
Non viene segnalato alcun conflitto di interessi.
Riconoscimenti
Questo lavoro è stato sostenuto dalla National Science Foundation (NSF) (numero di sovvenzione MCB 2129443 a DD).
Materiali
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcent. Tubes | Fisher Scientific | 01-549-740 | |
| 12 welled microplate | Fisher Scientific | 50-233-6077 | |
| 22 x 22 mm coverslips | Fisher Scientific | 12-541-016 | |
| 45x 50 cm coverslips | Brain Research | 4550-1.5D | |
| Agar | Sigma Aldrich | A9414 | |
| anti-rat FITC | Sigma Aldrich | F6258 | |
| anti-rat TRITC | Sigma Aldrich | T4280 | |
| calcium chloride | Sigma Aldrich | C4981 | |
| Cambbridge stubs | EMS | 75183-65 | |
| Fluoview CLSM | Evident | Fluoview 1200 | |
| JIM5 | Kerafast | ELD004 | |
| JIM7 | Kerafast | ELD005 | |
| Microcentrifuge | Fisher Scientific | 13-100-675 | |
| Micropipetors | BioRad | 1660499EDU | |
| Penium margaritaceum | Sammlung von Algenkulturen der Universität Göttingen - Culture Collection of Algae at Göttingen University | 2640 | |
| Polysphere kit | Polysciences | 18336 | |
| SEM | ThermoFisher | Quattro SEM | |
| sputter coater | EMS | Q150V | |
| Vortex mixer | Fisher Scientific | 02-215-414 |
Riferimenti
- Delmer, D., Dixon, R. A., Keegstra, K., Mohnen, D. The plant cell wall-dynamic, strong, and adaptable-is a natural shapeshifter. Plant Cell. 36 (5), 1257-1311 (2024).
- Bierenbroodspot, M. J., et al. Phylogeny and evolution of streptophyte algae. Ann Bot. 134 (3), 385-400 (2024).
- Domozych, D. S., et al. Endomembrane architecture and dynamics during secretion of the extracellular matrix of the unicellular charophyte, Penium margaritaceum. J Exp Bot. 71 (11), 3323-3339 (2020).
- Feng, X., et al. Genomes of multicellular algal sisters to land plants illuminate signaling network evolution. Nat Genet. 56 (5), 1018-1031 (2024).
- Jiao, C., et al. The Penium margaritaceum genome: Hallmarks of the origins of land plants. Cell. 181, 1097-1111 (2020).
- LoRicco, J. G., et al. The multifunctional roles of the extracellular matrix in the sessile life of the zygnematophyte Penium margaritaceum: stick, glide and cluster. Physiologie Plantarum. 176 (5), e14520 (2024).
- LoRicco, J. G., et al. Aberrant growth and expansion in Penium margaritaceum triggered by disruption of microtubules and the cell wall. J Exp Bot. erae387, (2024).
- LoRicco, J. G., et al. Chemically induced phenotype plasticity in the unicellular zygnematophyte, Penium margaritaceum. Protoplasma. 261 (6), 1233-1249 (2024).
- Davis, D. J., et al. Callose deposition is essential for the completion of cytokinesis in the unicellular alga, Penium margaritaceum. J Cell Sci. 133 (19), jcs.249599 (2020).
- Rydahl, M. G., et al. Penium margaritaceum as a model organism for cell wall analysis of expanding plant cells. Methods Mol Biol. 1242, 1-22 (2015).
- Carrillo-Carrasco, V. P., Hernández-García, J., Weijers, D. Electroporation-based delivery of proteins in Penium margaritaceum and other zygnematophycean algae. Physiol Plant. 175 (6), e14121 (2023).
- Nichols, H. W. Growth media-freshwater. Handbook Phycol Methods. , 16-17 (1973).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon