Method Article
Edição do genoma no mosquito da febre amarela Aedes aegypti usando CRISPR-Cas9
Neste Artigo
Resumo
Aqui, descrevemos um protocolo detalhado para edição do genoma por meio de microinjeção embrionária no mosquito A. aegypti usando a tecnologia CRISPR-Cas9.
Resumo
O surgimento da tecnologia de repetições palindrômicas curtas agrupadas, regularmente intercaladas (CRISPR)-Cas9 revolucionou o campo da engenharia genética e abriu as portas para a edição precisa do genoma em várias espécies, incluindo organismos não modelo. No mosquito Aedes aegypti, mutações de perda de função e inserções de DNA foram realizadas com essa tecnologia. Aqui, descrevemos um protocolo detalhado para edição de genoma por meio de microinjeção embrionária no mosquito A. aegypti usando a tecnologia CRISPR-Cas9, com foco na geração de linhas de nocaute e knockin de genes. Neste protocolo, as agulhas de quartzo são preenchidas com uma mistura de RNA guia, Cas9 recombinante e um plasmídeo contendo um de DNA que codifica um gene para um marcador fluorescente, se o gene knockin for desejado. Os embriões no estágio de pré-blastoderma são alinhados em uma tira de fita adesiva dupla face colocada em uma lamínula, que é posteriormente montada em uma lâmina de vidro. Com a ajuda de um microinjetor, as agulhas são inseridas suavemente na extremidade posterior dos embriões e um pequeno volume da mistura CRISPR é dispensado. Quando os embriões são eclodidos, as larvas são verificadas sob o escopo fluorescente e as pupas são classificadas por sexo e separadas em gaiolas diferentes. Uma vez que os adultos emergem, eles são cruzados reciprocamente com indivíduos do tipo selvagem, alimentados com sangue e colocados para postura de ovos. Uma vez que esses ovos são eclodidos, as larvas fluorescentes coletadas representam indivíduos com inserção estável do de DNA em seu genoma. Essas larvas são então cultivadas até o estágio adulto, cruzadas para indivíduos do tipo selvagem e, em seguida, avaliadas por meio de técnicas moleculares para confirmar que a sequência exata do de DNA está presente no local desejado do genoma do mosquito. Linhas homozigóticas também podem ser obtidas seguindo o pipeline fornecido de esquema de cruzamento e triagem molecular das mutações.
Introdução
A edição precisa do genoma tornou-se indiscutivelmente mais fácil, mas possível, com o estabelecimento das tecnologias CRISPR-Cas de tesouras moleculares1. Essas tecnologias aproveitam um mecanismo que o sistema imunológico procariótico usa para combater infecções por fagos2. Entre esses sistemas, repetições palindrômicas curtas agrupadas, regularmente intercaladas (CRISPR), juntamente com a nuclease Cas9, geralmente dependem de 20 RNAs de pares de bases, os RNAs guia (gRNAs), com sequências homólogas ao DNA alvo, que são seguidas por uma sequência de motivo adjacente ao protoespaçador NGG (PAM)3. Os gRNAs carregados no Cas9 guiam essas nucleases precisamente para locais-alvo específicos no genoma, desencadeando quebras de DNA de fita dupla3.
As quebras de fita dupla de DNA induzem os mecanismos de reparo a remendar a dupla hélice4. Embora se espere que qualquer reparo de DNA seja preciso, existem mecanismos de reparo de DNA menos precisos que podem deixar cicatrizes de sequência e, por sua vez, mutações de perda de função4. Entre os mecanismos de reparo de DNA propensos a erros, o Non-Homousus End-Joining (NHEJ) pode causar mutações de mudança de quadro, incluindo pequenas deleções, inserções e alterações de nucleotídeos (SNPs), que podem resultar em mutação de perda de função. O mecanismo de Reparo Dirigido por Homologia (HDR), por outro lado, depende do cromossomo homólogo como modelo para copiar a sequência exata do alelo não danificado e fazer um reparo perfeito da sequência de DNA alvo4.
Com base nesse conhecimento, a tecnologia CRISPR-Cas9 foi desenvolvida para editar genomas com precisão, sem dúvida em qualquer sequência contendo um sítio PAM3. Em mosquitos, a tecnologia CRISPR-Cas9 tem sido usada para nocautear uma variedade de genes, por meio da microinjeção embrionária de uma mistura de Cas9 e gRNAs, aproveitando o mecanismo de reparo do NHEJ 5,6. Mutagênese germinativa semelhante é obtida com a injeção da mistura gRNA + Cas9 na hemolinfa de mosquitos fêmeas adultas7. Essa tecnologia foi denominada ReMOT control e se baseia em uma versão modificada de um Cas9 marcado com um peptídeo que é absorvido pelos ovários por meio de endocitose durante o processo de desenvolvimento do óvulo (vitelogênese)7. A inserção de de genes específicos em um genoma só é possível por meio da microinjeção embrionária de uma mistura de gRNA e Cas9 (ou um plasmídeo expressando essas moléculas) junto com um plasmídeo que codifica um de DNA desejado8. Aproveitando o mecanismo HDR, o plasmídeo contendo o de DNA de interesse flanqueado por sequências homólogas (500-1.000 pb) 9 , 10 a montante e a jusante do local alvo é usado como modelo para reescrever a quebra de fita dupla, copiando também o de DNA para a sequência alvo9.
A tecnologia CRISPR-Cas9 tem sido usada para eliminar vários genes envolvidos principalmente com os sistemas sensoriais do mosquito Aedes aegypti11, mas também genes associados à fertilidade masculina e viabilidade feminina (PgSIT) para controle populacional12. A eliminação de genes-alvo também foi realizada pela inserção de genes que codificam marcadores fluorescentes nas sequências codificantes de genes específicos13,14. Essa estratégia tem a vantagem de não apenas induzir mutações frame-shift, mas também permitir o uso de luz fluorescente para classificar os indivíduos da nova linha de nocaute 13,14. O genoma do A. aegypti também foi editado com sequências de sistemas de expressão binária, como o sistema Q (QF-QUAS)11. A introdução do gene que codifica o transativador QF a jusante de um promotor de um gene específico garante a expressão espaço-temporal definida do transativador 15,16. Uma vez que uma linha de mosquito que expressa QF é cruzada com outra linha de mosquito contendo os locais de ligação (QUAS) para QF, este último se liga a ela e desencadeia a expressão de genes a jusante da sequência QUAS15,16. Esse sistema, em geral, permite a expressão específica do tecido e do tempo de tais genes efetores, que podem ser marcadores fluorescentes usados para localização celular ou detecção de atividade neuronal, e até mesmo nucleases Cas9 para interromper genes em tecidos específicos (ou seja, nocaute somático)11.
Dadas todas as informações disponíveis para a transformação genética do A. aegypti , fornecemos aqui um protocolo detalhado com instruções passo a passo para realizar a edição do genoma com o sistema CRISPR-Cas9 por meio de microinjeção embrionária. Estratégias para gerar knockout, por meio de mutações e deleções de mudança de quadro mediadas por NHEJ, e linhas knockin, por inserções de de genes mediadas por HDR, são discutidas.
Protocolo
Os detalhes relacionados aos equipamentos e reagentes utilizados neste protocolo estão listados na Tabela de Materiais. Todos os animais foram manipulados seguindo o Guia para o Cuidado e Uso de Animais de Laboratório, conforme recomendado pelo National Institutes of Health. Os procedimentos foram aprovados pelo Comitê Institucional de Cuidados e Uso de Animais da UCSD (IACUC, Protocolo de Uso de Animais #S17187) e pela Autorização de Uso Biológico da UCSD (BUA #R2401).
1. gRNAs e design de plasmídeo de doadores
- Para fazer mutantes knockout, projete dois gRNAs espaçados por ~ 20-100 bp ( Figura 1A ).
- Projete gRNAs de 20 pb para CRISPR-Cas9, excluindo a sequência PAM (NGG) usando uma ferramenta online (Figura 1A), como CHOPCHOP (https://chopchop.cbu.uib.no/)17ou Benchling (benchling.com) ou CRISPOR (http://crispor.gi.ucsc.edu/)18. Selecione os gRNAs mais específicos e livres de alvos para experimentos, conforme sugerido pela ferramenta online.
- Certifique-se de que um local de corte de enzima de restrição eficiente seja incluído na sequência entre os locais de corte de gRNA (Figura 1A). Inclua um local de enzima de restrição entre os locais de corte do gRNA para permitir a confirmação visual rápida de edições bem-sucedidas.
NOTA: Quando ocorre a deleção, o local de restrição é removido, de modo que a enzima não cortará a sequência, produzindo uma única banda não cortada em um gel para confirmar a exclusão.
- Para inserções de de genes mediadas por HDR, projete vários gRNAs usando as ferramentas mencionadas para fazer mutantes knockout e selecione o mais eficaz após avaliação posterior.
- Projete plasmídeos doadores para reparo dirigido por homologia (HDR) contendo braços de homologia do local alvo, a sequência de DNA de carga, um marcador fluorescente, a origem da replicação e um gene de resistência a antibióticos (Figura 1B).
- Escolha os braços de homologia das regiões a montante e a jusante do local alvo, cada um abrangendo 500-1.000 pb10 (Figura 1B).
- Selecione a sequência de carga, que pode incluir um marcador fluorescente, um gene de interesse ou um elemento regulador (por exemplo, QF2).
- Selecione um marcador fluorescente. Marcadores fluorescentes comuns usados para A. aegypti estão incluídos em um de DNA contendo um promotor, um gene codificador de marcador fluorescente e uma sequência 3' UTR. Veja a discussão para mais detalhes.
2. Preparação da mistura de injeção
- Diluir a proteína Cas9 com o tampão de diluição Cas9 até 1 μg/μl.
NOTA: Não descongele e congele Cas9 mais de 2x. É aconselhável fazer alíquotas da proteína Cas9. - Compre os gRNAs ou produza-os internamente.
NOTA: Para produção interna, use práticas padrão de descontaminação de RNA e materiais livres de RNAse.- Projete 4-6 gRNAs e escolha os dois melhores gRNAs para injeção (veja o ensaio de clivagem in vitro abaixo).
- Projete um primer direto que inclua a sequência do promotor T7 a montante da sequência de gRNA. Use o primer reverso universal de gRNA para as reações de PCR não modelo, conforme descrito abaixo. A base da sequência sobreposta emparelha-se com a sequência correspondente no primer reverso universal (negrito e sublinhado), criando um modelo para a DNA polimerase amplificar suas sequências.
NOTA: Para essas reações de PCR, o primer direto deve conter o promotor T7 (negrito), seguido por duas guaninas (importante para o início da transcrição via RNA polimerase T7), a sequência de 20 nucleotídeos do gRNA (N20; sem a sequência PAM) e a sequência de sobreposição do primer (sublinhado).
Primer para a frente: 5'- GAAATTAATACGACTCACTATAGGN20 GTTTTAGAGCTAGAAATAGC- 3'
Primer reverso: 5 '' - AAAAGCACCGACTCGGTGCCACTTTTT CAAGTTGATAACGGACTAGC
CTTATT TTAACTT GCTATTTCTAGCTCTAAAAC - 3' - Sintetize o molde de DNA por PCR sem molde. Configure várias reações (cada uma contendo 12,5 μL da mistura principal 2x, 1,25 μL da solução de 10 μM de primer direto, 1,25 μL da solução de 10 μM de primer reverso e 10 μL de água ultrapura) para que produto de PCR suficiente (300 ng) esteja disponível para a reação de transcrição in vitro . Utilizar as seguintes condições de PCR: desnaturação inicial durante 30 s a 98 °C; 35 ciclos de amplificação durante 10 s a 98 °C, 10 s a 62 °C e 10 s a 72 °C; extensão final a 72 °C por 2 min; e armazenamento a 4 °C.
- Confirme a amplificação de um único fragmento de DNA (122 pares de bases) em um gel de agarose (2%).
- Limpe o modelo de PCR usando um kit de purificação de PCR, seguindo as recomendações do fabricante.
- Realize uma reação de transcrição in vitro com um kit de transcrição T7. Misture 2 μL de tampão de reação 10x, 2 μL de nucleotídeos (ATP, CTP, UTP e GTP), 2 μL de RNA polimerase T7, 3 μL do DNA molde (100 ng/μL) e 5 μL de água ultrapura. Incubar a mistura a 37 °C durante pelo menos 2 h (não mais de 16 h) a uma noite (12 h).
NOTA: Nesta reação, a RNA polimerase T7 se liga ao promotor T7 incluído na sequência direta do primer, o que leva à transcrição do gRNA. - Trate a reação de transcrição com DNase adicionando 1 μL de DNase (misture bem) e incubando a 37 ° C por 15 min.
- Purifique os sgRNAs sintetizados com um kit de limpeza de transcrição seguindo as recomendações do fabricante.
- Realize um ensaio de clivagem in vitro para avaliar a eficiência de corte dos gRNAs selecionados (Figura 1C).
- Projete primers para amplificar um fragmento de DNA (500-1.000 pb) flanqueando o local de corte do gRNA.
- Configure as reações de PCR da seguinte forma: 12,5 μL da mistura principal 2x, 1,25 μL da solução de 10 μM de primer direto, 1,25 μL da solução de 10 μM de primer reverso, 9 μL de água ultrapura e 1 μL de DNA molde de 5 ng/μL.
- Utilizar as seguintes condições de PCR: condições: desnaturação inicial durante 30 s a 98 °C; 35 ciclos de amplificação por 10 s a 98 °C, 15-30 s a X °C (temperatura ideal de recozimento para primers) e 35 s a 72 °C; extensão final a 72 °C por 2 min; e armazenamento final a longo prazo a 4 °C.
- Consulte as diretrizes do fabricante para obter as melhores temperaturas de amplificação e intervalo de tempo. Agrupe pelo menos cinco reações ou aumente os volumes dos reagentes para que produto de PCR suficiente (1,5-2 μg) seja obtido após a limpeza da PCR (banda única) ou purificação da banda de PCR em gel de agarose.
- Configure as reações de clivagem de Cas9 com Cas9 recombinante incubando a seguinte mistura a 37 ° C por 1 h: 1 μL de tampão de reação 10x Cas9, 0,35 μL de Cas9 recombinante (1 μg / μL), 1 μL de gRNA (100 ng / μL), 6,65 μL de água ultrapura e 1 μL de 300 ng / μL de DNA molde.
- Configure uma reação de controle negativo sem nenhum gRNA.
- Verifique a eficiência da clivagem executando reações em um gel de agarose (1,5-2,0%; Figura 1C).
3. Montagem do plasmídeo doador
- PCR-amplifica os braços de homologia do DNA genômico de A. aegypti.
- Para o design do primer, consulte as diretrizes do fabricante do kit de clonagem. Use uma ferramenta online (por exemplo, Benchling), que fornece suporte para design e montagem de plasmídeos usando diferentes estratégias de clonagem (por exemplo, Gibson, Golden Gate e clonagem baseada em enzimas de restrição).
- Amplifique a sequência de carga do DNA genômico de A. aegypti ou solicite um fragmento de gene sintetizado comercialmente.
- Amplificar marcadores fluorescentes de plasmídeos internamente.
- Ligue fragmentos de DNA das etapas 3.1.1 a 3.1.3 na espinha dorsal de um plasmídeo existente pela montagem de Gibson. Incubar uma mistura de 10 μL de 2x master mix, X μL de todos os fragmentos de DNA, 10-X μL de água ultrapura a 50 °C por 1 h.
NOTA: Consulte as diretrizes do fabricante para calcular a proporção ideal de insertos para a estrutura do plasmídeo.- Calcule as concentrações de cada fragmento de clonagem em pico mols: pmols = (peso em ng) × 1.000/(pares de bases × 650 daltons).
- Use 50-100 ng de esqueleto de plasmídeo e excesso molar de 2-3 vezes de cada inserto.
- Se estiver montando 2-3 fragmentos, adicione 0,02-0,5 pmols de cada fragmento na reação de Gibson. Se estiver montando 4-6 fragmentos, adicione 0,2-1,0 pmols de cada um.
- Use 3-5 μL da reação de montagem de Gibson para transformar células competentes para E. coli JM109.
NOTA: O JM109 foi escolhido para a montagem de Gibson devido ao seu genótipo recA- , que reduz eventos indesejáveis de recombinação e evita o transporte de nuclease durante a colheita celular, garantindo a integridade dos fragmentos de DNA montados
Para plasmídeos maiores que 10 kb, recomendamos a transformação de células competentes para DH5α usando o protocolo estendido, seguindo as recomendações do fabricante. - Expanda as bactérias transformadas e purifique o plasmídeo usando um kit de minipreparação.
- Para confirmação da montagem correta do plasmídeo, execute uma digestão diagnóstica da enzima de restrição com o DNA do plasmídeo purificado e visualize por eletroforese em gel de agarose ( Figura 1D , E ).
NOTA: Sugerimos o uso de uma ferramenta online para a seleção de algumas enzimas de restrição que podem cortar o plasmídeo em um único local. Softwares como o Benchling possuem uma ferramenta integrada que realiza a digestão virtual de sequências de plasmídeos com as enzimas de restrição selecionadas, exibindo o padrão de banda de DNA esperado em um gel de eletroforese virtual (Figura 1D).- Efectuar a digestão da enzima de restrição do plasmídeo. Incubar uma mistura de 1 μL de tampão de digestão de restrição 10x, 0,5 μL de enzima de restrição 1, 0,5 μL de enzima de restrição 2, 1 μL de DNA de plasmídeo (300 ng/mL) e 7 μL de água ultrapura a 37 °C por 2 h.
- Execute as reações de digestão de restrição em um gel de agarose (1,5%).
NOTA: O padrão de banda de DNA (Figura 1E) deve se assemelhar ao padrão de banda de resumo virtual (Figura 1D). Os plasmídeos também podem ser enviados para sequenciamento de plasmídeo completo se o serviço estiver disponível.
- Cultive o clone do plasmídeo em 150 mL de meio LB.
- Realize o plasmídeo maxiprep, seguindo as orientações do fabricante.
- Suspender o plasmídeo em água ultrapura.
4. Misturando a construção de injeção
NOTA: As faixas de concentração final sugeridas para cada construto são fornecidas na Tabela 1. Comece com uma proporção de 2:1:2 (ng Cas9: ng sgRNA: ng plasmídeo doador) e ajuste conforme necessário para otimizar a eficiência do HDR. O monitoramento dos resultados ajudará a identificar a combinação mais eficaz.
- Para fazer mutantes nocaute,
- Diluir a alíquota da proteína Cas9 até à concentração desejada utilizando o tampão de diluição Cas9 e diluir a alíquota do RNAg com água ultrapura.
- Pré-misture a proteína Cas9 com cada gRNA para formar um complexo de ribonucleoproteína (RNP). Combine as soluções pré-misturadas.
- Para inserções de de genes mediadas por HDR,
- Dilua e misture a proteína Cas9 e os gRNAs conforme sugerido para fazer mutantes nocaute.
- Diluir o plasmídeo dador com água ultrapura.
- Combine todas as construções.
5. Puxando e carregando agulhas de microinjeção
- Use filamento de quartzo para puxar as agulhas de injeção (Figura 2). Certifique-se de que o filamento tenha 10 cm de comprimento, com um diâmetro externo de 1 mm e um diâmetro interno de 0,7 mm.
- Puxe as agulhas usando um extrator de micropipeta a laser com um dos dois programas a seguir:
Programa 1: Calor 805, Filamento 4, Velocidade 50, Atraso 145, Tração 145
Programa 2: Calor 650, Filamento 4, Velocidade 40, Atraso 150, Tração 156
NOTA: O programa 1 resulta em uma ponta de agulha mais fina, porém mais longa, tornando-o adequado para injeções com construções de concentração mais baixa, onde uma ponta mais fina é benéfica para a precisão. O programa 2 produz uma ponta de agulha mais curta, porém mais grossa, ideal para injeções de maior concentração, pois a construção é mais espessa e requer uma agulha mais resistente.
6. Colheita de embriões
- Prepare um aspirador elétrico ou manual para coletar mosquitos e use frascos de plástico para coleta e coleta de embriões.
- Umedeça os papéis de filtro de círculo branco e coloque-os na parede interna ou em cima do algodão úmido no coletor.
- Coloque de 5 a 10 mosquitos fêmeas que foram alimentadas com sangue há 5 a 10 dias no coletor. Coloque o coletor no escuro por 45 min.
- Retire os papéis de filtro para a colheita de embriões.
7. Linha de embriões
- Use uma cepa selvagem (Liverpool) de A. aegypti para injeção de embriões.
- Selecione embriões em estágio de pré-blastoderma, particularmente aqueles de cor cinza claro, do papel de colheita (Figura 3).
- Transfira alguns embriões com um pincel umedecido para a fita adesiva dupla face colocada em cima de uma lamínula. Alinhe os embriões em paralelo enquanto seus arredores ainda estão molhados, garantindo que estejam lado a lado, com todas as extremidades posteriores voltadas para a frente.
- Uma vez posicionado corretamente, deixe o ambiente secar um pouco para prender os embriões no lugar.
- Adicione óleo de halocarbono 700 aos embriões durante o alinhamento para evitar a dessecação.
NOTA: Certifique-se de que os embriões não estejam cercados por água ao adicionar o óleo, pois isso faria com que os embriões flutuassem no óleo.
8. Microinjeção de embriões
NOTA: A injeção é realizada à temperatura ambiente ou a 18 °C. A temperatura de 18°C é recomendada por razões práticas, pois atrasa o desenvolvimento do embrião.
- Configure um microinjetor com os seguintes parâmetros: pressão de compensação (Pc) 300 hPa, pressão de injeção (Pi) 500 hPa. Ajuste essas condições sempre que necessário.
- Carregue 3 μL da construção mista em uma agulha usando um microcarregador.
- Coloque a lamínula com embriões alinhados em cima de uma lâmina de microscópio (Figura 3). Posicione a lamínula e deslize sob o microscópio para injeção. Certifique-se de que a extremidade posterior do embrião esteja posicionada em direção à agulha.
- Coloque uma agulha em um porta-agulha junto com um micromanipulador e posicione a agulha em um ângulo de 10° em direção à extremidade posterior dos embriões alinhados, mantendo-a estacionária (Figura 4A). Abra suavemente a agulha sob um microscópio e toque-a levemente com a ponta de uma lamínula.
NOTA: Outra opção é criar uma pequena abertura na agulha batendo nela com um embrião. No entanto, recomenda-se uma abertura de agulha um pouco mais larga, pois a viscosidade da proteína Cas9 pode entupir a agulha. - Mova o vidro deslizante em direção à agulha para injeção (Figura 5).
9. Cuidados posteriores ao embrião injetado
- Use lenços descartáveis e sem fiapos para remover o óleo ao redor dos embriões.
- Adicione água deionizada para enxaguar os embriões e mova os embriões para um papel de filtro úmido. Coloque o papel de filtro úmido em um lenço úmido dentro de copos Karat de 9 onças (Figura 4B e Figura 6A,B).
- Coloque o algodão úmido no fundo do copo para manter a umidade (Figura 6B).
- Conservar os embriões injectados em papel de filtro húmido.
10. Eclosão de embriões e triagem de larvas G0
- Pelo menos 4 dias após a injeção, transfira o papel de filtro com embriões para aproximadamente 3 L de água deionizada em panelas Sterilite de 6 litros para incubação.
NOTA: Os ovos normalmente eclodem com mais eficiência dentro de 2 semanas após a injeção. Certifique-se de que os ovos permaneçam úmidos durante este período. O papel de filtro não deve estar muito úmido, pois a umidade excessiva pode levar à eclosão precoce dos embriões. Embora a preservação mais longa possa melhorar as taxas de eclosão, não exceda um mês para obter os melhores resultados. O uso de água desoxigenada também pode ajudar na eclosão. - Assim que as larvas G0 eclodirem, adicione comida de peixe misturada com água à panela como alimento.
- Rastreie as larvas G0 para o marcador fluorescente no estágio larval de 3º a 4º ínstar (Figura 7).
- Mantenha as larvas separadamente com base em seu status fluorescente, com larvas fluorescentes positivas e fluorescentes negativas mantidas em panelas separadas.
11. Pupas de classificação sexual e cruzamento com o tipo selvagem
- Separe os mosquitos injetados por sexo quando eles se transformam em pupa, usando estruturas específicas de tamanho e sexo no lobo genital para identificação:
- Machos: Tamanho menor, lobo genital mais proeminente e pontiagudo e pás mais largas (estruturas na extremidade da cauda das pupas; Figura 8A 1,A2).
- Fêmeas: Tamanho maior, lobo genital menor e menos pronunciado e pás mais estreitas (Figura 8B1, B2).
- Agrupar mosquitos fluorescentes positivos ou negativos fluorescentes de cada sexo.
- Cruze os mosquitos agrupados com mosquitos do sexo oposto da cepa Liverpool. Use uma proporção de 3:1 a 5:1 do tipo selvagem para o mosquito G0.
- Atravesse os mosquitos por 4 dias.
- Forneça às fêmeas uma refeição de sangue após a travessia.
12. Triagem G1
- Três dias após a alimentação com sangue, forneça copos de ovos colocando uma toalha de papel dentro da parede dos copos Karat de 9 onças e adicionando cerca de 3 onças de água deionizada.
- Após 3-4 dias, colha e ecloda os embriões G1. Rastreie as larvas G1 para o marcador fluorescente nos estágios de 3º a 4º ínstar (Figura 7).
- Colete as larvas fluorescentes, sugerindo uma inserção de HDR. Quando os indivíduos fluorescentes G1 atingirem o estágio de pupa, separe-os por sexo e coloque cada sexo em gaiolas separadas.
- Cruze os mosquitos fluorescentes G1 com indivíduos do sexo oposto da cepa Liverpool.
13. Confirmação do local de inserção G1
- Depois de fornecer a refeição de sangue, prepare as fêmeas G1 para postura de ovos de fêmeas únicas, colocando um pequeno pedaço de papel toalha em frascos individuais de Drosophila e adicionando 3 mL de água deionizada.
- Para mutantes knockout:
- Após a postura dos ovos, a água evapora e a toalha de papel seca, choca os ovos das fêmeas que exibem claramente mutações nocaute e obtém a geração G2.
- Colete o corpo das mães G1 que eclodiram com sucesso a prole G2 e extraia o DNA.
- Use o DNA como modelo para PCR para amplificar a sequência que cobre o local alvo. Certifique-se de que os primers amplificam um fragmento de DNA de ~ 200 pb. Configure as reações (cada uma contendo 12,5 μL da mistura principal 2x, 1,25 μL da solução de 10 μM de primer direto, 1,25 μL da solução de 10 μM de primer reverso, 9 μL de água ultrapura e 1 μL de DNA molde [5 ng/μL]). Utilizar as seguintes condições de PCR: desnaturação inicial durante 30 s a 98 °C; 35 ciclos de amplificação por 10 s a 98 °C, 15-30 s a X °C (temperatura ideal de recozimento dos primers) e 10 s a 72 °C; extensão final a 72 °C por 2 min; e armazenamento final a longo prazo a 4 °C.
- Purifique os fragmentos de PCR e realize a digestão enzimática de restrição dos fragmentos de PCR incubando uma mistura de 1 μL de tampão de digestão de restrição 10x, 0,5 μL de enzima de restrição 1, 0,5 μL de enzima de restrição 2, 1 μL de fragmento de PCR (200 ng/mL) e 7 μL de água ultrapura a 37 °C por 30 min.
NOTA: Certas enzimas de restrição podem ser usadas na mistura de PCR sem purificação prévia. - Visualize a digestão do fragmento de PCR por eletroforese em gel para confirmar se ocorreu clivagem.
- Sequencie o fragmento de PCR não digerido para identificar as mutações nocaute.
- Para inserções mediadas por HDR, após a postura:
- Colete o corpo das mães G1 e extraia o DNA.
- Use o DNA como modelo para PCR para amplificar a sequência que cobre o local alvo.
- Realize o sequenciamento do fragmento de DNA amplificado para confirmar a integridade do de DNA inserido.
- Chocar o ovo e obter a geração G2.
14. Expandindo as novas linhas CRISPR
- Para linhagens mutantes nocaute:
- Configure fêmeas G2 para postura de ovos, com 20 fêmeas por G1. Cada G1 representa uma linha.
- Após a postura dos ovos, confirme as mutações nocaute da mesma maneira que para G1.
- Prossiga com cinco linhagens G1 que exibem mutações claramente identificadas em G1 e G2. Chocar os ovos coletados do G2 de cada linhagem, dando origem à geração G3.
- Das cinco linhas cruzadas, selecione duas linhas com mutações claramente identificadas e alta aptidão ou o fenótipo desejado.
- Cruze fêmeas G3 de cada linhagem selecionada com machos da linhagem Liverpool.
- Configure 50 fêmeas solteiras para postura de ovos de cada linhagem. Confirme as mutações nocaute como feito anteriormente para G1.
NOTA: Aumente o número de mulheres na configuração para manter a diversidade. - Repita os procedimentos por mais duas gerações com as duas linhas (Figura 4J).
- Para novas linhas de inserções mediadas por HDR:
- Verificar a presença de indivíduos fluorescentes na geração G2.
- Cruze os mosquitos G2, de cada indivíduo G1, com a cepa Liverpool. Cada indivíduo G1 representa uma linha.
- Prossiga com mais três gerações de cruzamentos. Verifique continuamente o marcador fluorescente e observe a adequação19 de cada linha (Figura 4x).
- Selecione uma linha com inserções corretas e alta aptidão e o fenótipo desejado.
15. Faça linhas homozigóticas
- Para linhagens mutantes nocaute:
- Geração G6:
- Chocar os ovos produzidos pelo G5 e obter a geração G6.
- Intercruze adultos G6 dentro de suas respectivas linhas (Figura 4K e Figura 9A).
- Configure 50 fêmeas G6 solteiras para postura de ovos por linha e choque os ovos conforme descrito anteriormente (Figura 9B).
- Colete as fêmeas G6 que eclodiram com sucesso a prole G7, extraia o DNA e realize PCR e digestão da enzima de restrição como feito para G1 (Figura 9C).
NOTA: Um tampão de compressão simples e barato (SB) pode ser usado nesta etapa para extração de DNA.- Usando um moedor portátil, macere um único mosquito em 100 μL de 1x SB (10 mM Tris-HCl pH 8, 1 mM EDTA e 25 mM NaCl). Adicione a proteinase K (200 μg/mL) e incube a 37 °C por 30 min, seguido de 95 °C por 2 min. Gire para baixo a 10.000 × g por 5 min e colete o sobrenadante20.
- Confirme a mutação visualizando os resultados em um gel.
- Descarte os ovos produzidos por fêmeas sem mutações (Figura 9D).
- Mantenha larvas de fêmeas com mutações confirmadas para estabelecer várias linhagens G7 e descarte ninhadas sem mutações (Figura 9D).
- Geração G7:
- Intercruze fêmeas e machos dentro de cada linhagem G7 (Figura 9E).
- Configure 10 fêmeas G7 únicas para postura de ovos por linha e choque os ovos conforme descrito anteriormente (Figura 9F).
- Colete as fêmeas G7 que eclodiram com sucesso a prole G7, extraia DNA e realize PCR e digestão de enzimas de restrição (Figura 9G).
- Detecte mutações nocaute em ambos os alelos ou em um único alelo visualizando os resultados em um gel.
- Manter larvas de fêmeas G7 confirmadas com nocautes em ambos os alelos, produzindo a geração G8 (Figura 9H). Descarte as ninhadas sem mutações (Figura 9H).
NOTA: Esta etapa garante que as fêmeas sejam homozigóticas. No G8, testar a homozigosidade da progênie confirma se os pais também são homozigotos.
- Geração G8:
- Cruze adultos G8 para cada linhagem feminina G7 (Figura 9I).
- Escolha aleatoriamente cinco machos G8, extraia DNA e realize PCR e digestão de enzimas de restrição (Figura 9J).
- Forneça uma refeição de sangue e choque os ovos de fêmeas G8 que foram cruzadas com machos G8 confirmados como nocautes em ambos os alelos.
- Continue com as linhagens G7 onde todos os machos detectados têm nocautes em ambos os alelos, indicando que essas linhagens são homozigotos.
- Geração G6:
- Para linhas de inserções mediadas por HDR:
- Chocar os ovos produzidos pelo G5 e obter a geração G6. Filtre as larvas no estágio G6 para marcadores fluorescentes.
- Descarte as larvas sem marcadores fluorescentes (Figura 4xi).
- Mosquitos cruzados que exibem marcadores fluorescentes.
- Continue este procedimento para cada geração subsequente até que nenhuma larva não fluorescente seja observada. Neste ponto, a linha do mosquito será homozigótica.
Resultados
Desenho e validação de direcionamento de genes mediados por gRNA para recombinação de homologia HDR
Para garantir que o gene desejado seja direcionado com precisão, recomendamos selecionar alguns gRNAs e posicionar os braços de homologia 5 'e 3' próximos ao local de corte para recombinação homóloga mediada por HDR ( Figura 1A ). Por exemplo, projetamos dois gRNAs para atingir ambos os lados do códon inicial do gene de interesse e usamos o QF2-Hsp70-OpIE2-ECFP-SV40 como marcador, inserindo-o no gene alvo por meio de braços de homologia de 1 kb por meio do mecanismo HDR (Figura 1B). As quebras de fita dupla levam a uma deleção de um pequeno fragmento de DNA entre os locais de corte dos gRNAs (Figura 1A).
A eficiência de clivagem dos gRNAs e sua capacidade de cortar o gene alvo foram testadas por ensaio de clivagem Cas9 in vitro . Os fragmentos de PCR do gene alvo foram clivados em dois fragmentos por Cas9, carregados com os gRNAs (Figura 1C). Além disso, as enzimas de restrição podem ser usadas para verificar a integridade do plasmídeo doador antes de passar para as etapas posteriores do protocolo. Os resultados da digestão do plasmídeo devem exibir uma ou duas bandas no gel, com a presença de uma pequena banda indicando digestão bem-sucedida em um fragmento curto (Figura 1D, E).
Preparação de agulhas de microinjeção
Antes de iniciar o processo de injeção, é importante preparar agulhas de microinjeção novas. As agulhas devem ter uma forma protuberante com uma ligeira curva. Cerca de 2/3 da agulha deve ser estreita na parte superior, enquanto o 1/3 restante é mais largo. A extremidade frontal da ponta da agulha não deve exceder 0,5 cm de comprimento da parte estreita para a larga (Figura 2A). Depois de usar a borda da lamínula para abrir a agulha, uma agulha com ponta chanfrada é preferível para microinjeção, pois é mais fácil de usar depois de quebrar a agulha tocando suavemente a superfície do embrião de mosquito preto (Figura 2B). Por outro lado, uma agulha de ponta romba pode dificultar a perfuração de embriões cinza claro durante a microinjeção (Figura 2C). As agulhas não devem ser muito macias nem muito duras para penetrar nos embriões do mosquito de forma eficaz para evitar quebrar a agulha.
Injeção de embriões
Para garantir a injeção bem-sucedida, comece colhendo embriões de mosquito cinza claro e alinhando-os de forma que o pólo posterior fique voltado para a mesma direção (Figura 3A). Esse alinhamento direcional ajuda a injetar o plasmídeo próximo às células germinativas (pólo posterior), o que é crucial para a produção de mutantes que podem ser herdados pela próxima geração. Selecione embriões frescos, uniformes e cinza claro de fêmeas que foram alimentadas com sangue 5 a 10 dias antes (Figura 3A1). Evite usar embriões que sejam desigualmente cinza ou marrom (Figura 3A2), pois eles podem não se desenvolver adequadamente.
Alinhe os embriões de mosquito um a um na fita dupla-face para facilitar a injeção precisa e eficiente. Aplique óleo de halocarbono antes que os ovos sequem e escureçam para uma cor cinza média ou escura (Figura 3B).
Ao injetar, concentre-se na superfície do pólo posterior de cada embrião. O lado anterior do embrião (lado direito na Figura 5), por outro lado, apresenta uma superfície plana e dura (micrópila). Use suavemente uma agulha chanfrada contendo a mistura de injeção para perfurar o embrião (Figura 5). A superfície do embrião se deprime e ricocheteia, indicando que a agulha penetrou no embrião. Após a injeção bem-sucedida, a cor do embrião clareia em poucos segundos (curva amarela na Figura 5, canto superior direito).
Após a injeção, use cuidadosamente a escova para pegar e transferir os embriões para outro papel de filtro úmido usando um lenço de laboratório para remover o excesso de óleo (Figura 6A). Mantenha os embriões em uma toalha de papel úmida por vários dias em temperatura de criação (Figura 6B). O excesso de óleo pode reduzir a taxa de eclosão de embriões de mosquitos. Use bolas de algodão com água ultrapura para manter a hidratação. É improvável que os embriões que permanecem cinza ou marrom (seta amarela) após 2 h após a injeção eclodam ( Figura 6C ).
Triagem e cruzamento
Assim que os embriões amadurecerem, adicione água ultrapura ao copo do ovo e coloque-o em uma incubadora a 27 °C para incubação. Normalmente, leva de 5 a 7 dias para as larvas se desenvolverem até o estágio de 3º ínstar. Inicialmente, pré-selecionamos as larvas G0 que exibiam fluorescência, embora fossem mais fracas ou parciais em comparação com outras linhagens mutantes. Isso garantiu que o marcador fosse expresso corretamente e que os plasmídeos fossem injetados com sucesso nos embriões.
Essas larvas foram então cruzadas com mosquitos do tipo selvagem (WT). Durante as triagens subsequentes, selecionamos e retivemos as larvas G1 que exibiram expressão de fluorescência mais forte e completa para criação, expandindo o estoque de linhagens e conduzindo novos experimentos. As larvas fluorescentes G1 podem ser identificadas com base no padrão de expressão da proteína fluorescente fornecido pela combinação do promotor repórter e da proteína fluorescente: o promotor Hr5Ie1-eGFP (Figura 7A) impulsiona a expressão da proteína fluorescente verde em todo o corpo; Hr5Ie1-DsRed (Figura 7B) leva à expressão de fluorescência vermelha no corpo; OpIE2-CFP (Figura 7C) induz a expressão da proteína de fluorescência ciano (azul) apenas em uma parte específica do corpo. Quando a combinação 3xP3-TdTomato é usada, a proteína fluorescente vermelha é especificamente expressa nos olhos (Figura 7D). Esta linha específica de mosquito transgênico também contém o promotor OpIE2 que conduz a expressão de CFP em todo o corpo (Figura 7D).
Processo especial de estabelecimento homozigoto para mutantes knockout
Para simplificar a manutenção da linha e garantir que todos os mosquitos sejam mutantes, implementamos um pipeline para gerar mosquitos mutantes homozigotos não fluorescentes. Conforme descrito na etapa 15 do protocolo, após o cruzamento com mosquitos WT, colocamos os mosquitos mutantes G6 na mesma gaiola para cruzamento (Figura 9A). Após o cruzamento, as fêmeas foram coletadas em frascos individuais para postura de ovos (Figura 9B). Esta etapa permitiu a separação de fêmeas com diferentes genótipos.
Após a coleta de ovos para obtenção do G7, o DNA genômico foi extraído das fêmeas do G6, seguido de PCR e eletroforese em gel dos fragmentos de PCR, tratados ou não com enzima de restrição (Figura 9C). Linhas mostrando apenas fragmentos de PCR digeridos, indicando que não há mutações, foram descartadas. Enquanto aquelas linhas que mostram bandas de PCR duplas, indicando a presença de mutações e, por sua vez, fêmeas heterozigóticas, foram mantidas e eclodidas (Figura 9D). Os mosquitos G7 foram então deixados amadurecer e todos os adultos foram colocados na mesma gaiola para outra rodada de cruzamento para aumentar a probabilidade de estabelecer uma linha homozigótica (Figura 9E). Fêmeas G7 alimentadas com sangue foram novamente coletadas individualmente para coleta de ovos G8, representando três genótipos potenciais (Figura 9F). O DNA genômico foi extraído das fêmeas do G7 e a PCR seguida de eletroforese em gel foi realizada para determinar quais eram homozigotas (Figura 9G). Ovos de fêmeas não mutantes ou heterozigotas foram descartados, e apenas aqueles com resultados homozigotos foram mantidos (Figura 9H). Os ovos G8 da etapa H foram eclodidos e os adultos resultantes foram cruzados em uma gaiola separada (Figura 9I). De cada gaiola, 10 machos adultos foram selecionados aleatoriamente para eletroforese em gel e sequenciamento (Figura 9J). Esta etapa deve produzir indivíduos homozigotos, que foram então mantidos como estoque (Figura 9K).
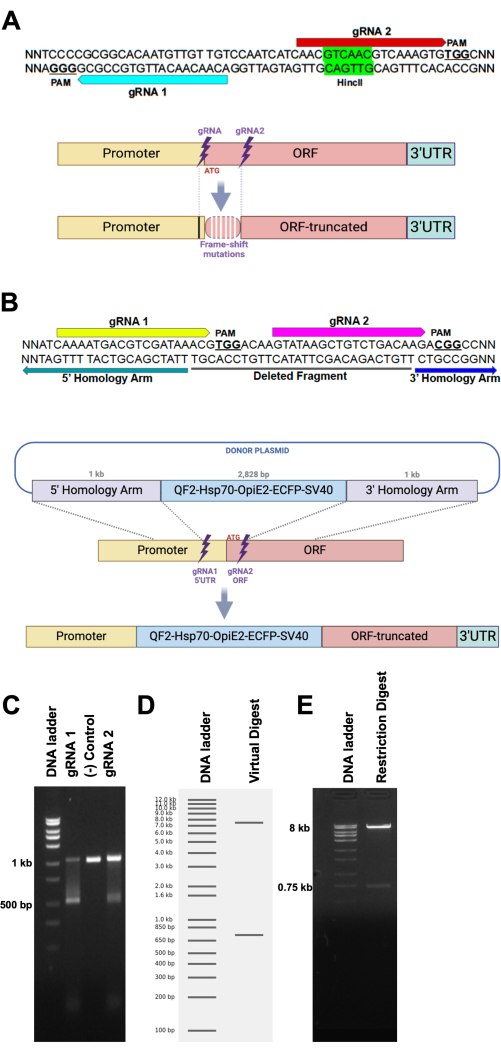
Figura 1: Esquema do projeto do plasmídeo doador de gRNA e HDR. (A, topo) Sequência alvo de sequência de DNA hipotética para dois gRNAs (setas azuis e vermelhas e seus locais PAM associados, juntamente com um local alvo de enzima de restrição (HincII). (A, embaixo) Representação esquemática de um local alvo submetido a NHEJ, levando ao surgimento de mutações de mudança de quadro (barras verticais brancas) devido a uma pequena deleção ou inserção de sequência. As mutações podem se estender além do local de clivagem dos gRNAs (barra vertical preta). (B, topo) Sequência de DNA hipotética representando os locais-alvo para gRNA 1 (seta amarela) e gRNA 2 (seta magenta), seguidos por locais PAM (NGG, negrito e sublinhado). A sequência de DNA potencialmente excluída entre os dois gRNAs é sublinhada por uma barra preta (fragmento excluído). As setas verde e azul destacam a sequência mais 3' do braço de homologia 5' e a sequência mais 5' do braço de homologia 3', respectivamente. (B, embaixo) Representação esquemática de uma recombinação HDR de um de DNA de um plasmídeo para um local homólogo no genoma. (C) Ensaio de clivagem in vitro . A incubação de um fragmento de DNA de 1 kb é digerida por Cas9 carregado com gRNA 1 (faixa esquerda) ou gRNA 2 (faixa direita), resultando em um fragmento de 500 pb. A faixa central mostra a reação de controle negativo, que foi desprovida de qualquer gRNA. (D) Digestão virtual de um plasmídeo (8,75 kb) com duas enzimas de restrição de corte único, resultando em praticamente duas bandas (8 kb e 750 bp) em um gel hipotético. (E) Um gel de agarose carregado com a reação de digestão de restrição de um plasmídeo, representando os resultados esperados fornecidos pelo resumo virtual. Clique aqui para ver uma versão maior desta figura.

Figura 2: Formato da agulha de microinjeção. (A) Imagem representativa da ponta de uma agulha. (B) Formato ideal da ponta da agulha para microinjeção de ovos de mosquito. (C) Formato da ponta da agulha que deve ser evitado. Clique aqui para ver uma versão maior desta figura.

Figura 3: Colheita de embriões. (A) Coleta de embriões cinza claro e (A1) homogêneos, mas não (A2) irregulares para injeções; Barra de escala = 500 μm. (B) Imagem para os embriões alinhados; Barra de escala = 500 μm. Clique aqui para ver uma versão maior desta figura.
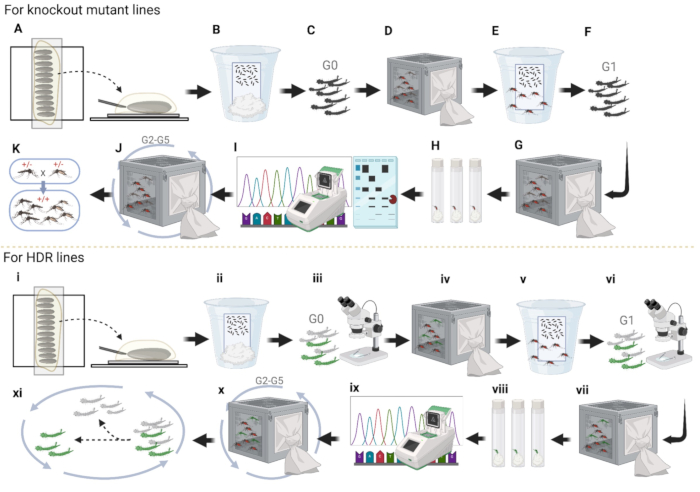
Figura 4: Pipeline de microinjeção de embriões de mosquito. (A-K) Injeções de linha de nocaute. (A) Alinhe os embriões de mosquito e injete o plasmídeo na extremidade posterior. (B) Armazenamento de embriões injetados no copo com papel úmido; (C) Larvas G0 eclodindo e separando-se para ambos os sexos. (D) Ambos os sexos de adultos que cruzam para WT em gaiolas separadas; (E) coletar os ovos das fêmeas no copo de ovos. (F) Larvas G1 eclodindo e separando-se para ambos os sexos. (G) Ambos os sexos de adultos cruzando para WT em gaiolas separadas. (H) Colete os ovos fêmeas G1 individualmente em frascos de plástico e choque os ovos. (I) PCR, execute o gel com fragmentos tratados com enzimas e sequenciamento para fêmeas solteiras G1 que eclodiram ovos com sucesso. (J) Cruzar G2 com WT por mais três gerações e sequenciar os mosquitos da mesma maneira que foi feito para G1. (K) Estabelecer homozigoto por intercruzamento. (i-xi) Injeções de linha HDR. (i) Alinhamento de embriões. (ii) Armazenamento de embriões injetados no copo com papel úmido. (iii) Pré-triagem de larvas G0 para fluorescência. (iv) Transforme larvas fluorescentes em adultos e cruze com WT em uma gaiola. (v) Coleta de ovos G1 no copo de ovos. (vi) Triagem e seleção de larvas fluorescentes G1. (vii) Adultos fluorescentes G1 cruzados com WT. (viii) Fêmeas adultas fluorescentes G1 alimentadas com sangue que põem ovos individualmente em frascos plásticos. (ix) Coleta e sequenciamento de adultos fluorescentes G1 individualmente. (x) Cruzamento com o tipo selvagem por mais três gerações adicionais e (xi) descartar larvas sem fluorescência para estabelecer homozigotos. Clique aqui para ver uma versão maior desta figura.

Figura 5: Injetando embriões. O momento da injeção de plasmídeo em embriões de mosquitos. Inserção: mistura de injeção visualizada como um líquido turvo dentro do embrião (linha pontilhada amarela); Barra de escala = 200 μm. Clique aqui para ver uma versão maior desta figura.
Figura 6: Coleta de embriões. (A) Colheita de embriões após a injeção e transferência para um papel de filtro úmido. Barra de escala = 7,5 mm. (B) Manutenção de embriões injetados em um copo. Barra de escala = 25 mm. (C) Embriões escuros após a injeção; Barra de escala = 500 μm. Clique aqui para ver uma versão maior desta figura.
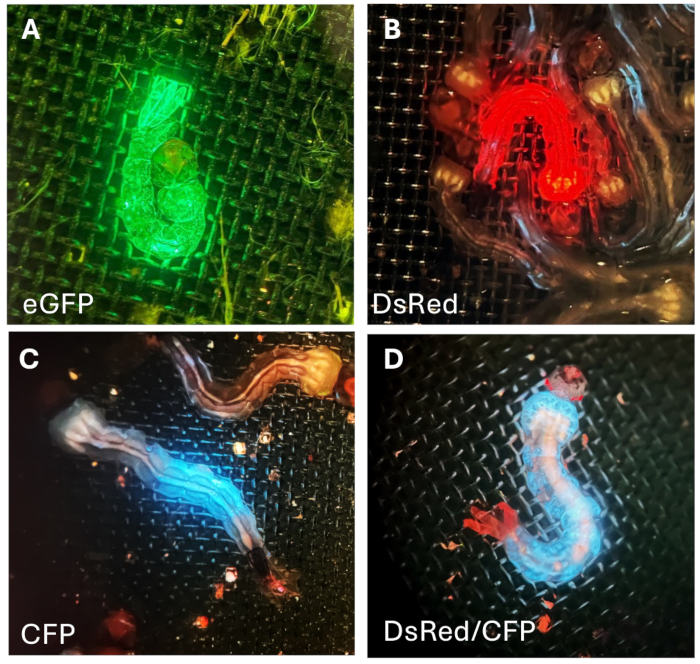
Figura 7: Fluorescência em larvas G1. (A) Verde, (B) vermelho e (C)proteínas fluorescentes azuis no corpo da larva do mosquito. (D) Larva exibindo fluorescência vermelha específica do olho e fluorescência azul específica do corpo. Clique aqui para ver uma versão maior desta figura.

Figura 8: Dimorfismo sexual na morfologia da pupa. (A1,A2) As pupas masculinas são caracterizadas por um tamanho corporal menor, um lobo genital proeminente e pontiagudo e pás mais largas na extremidade da cauda. (B1, B2) As pupas femininas exibem um tamanho corporal maior, um lobo genital menor e menos pronunciado e pás mais estreitas na extremidade da cauda. Clique aqui para ver uma versão maior desta figura.
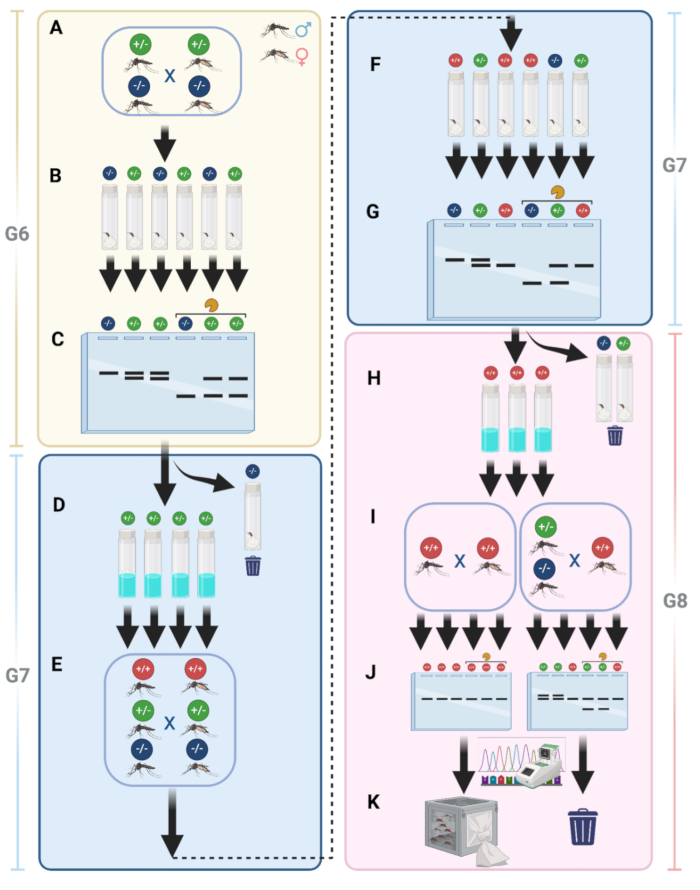
Figura 9: Detalhes do estabelecimento homozigoto para linhagens mutantes nocaute. (A) Os mosquitos mutantes são cruzados na mesma gaiola após o cruzamento com WT. (B) Após o cruzamento, as fêmeas individuais são coletadas em frascos para postura de ovos. (C) O DNA genômico é extraído de fêmeas G6, seguido de PCR e eletroforese em gel com fragmentos tratados e não tratados com enzimas. (D) Ovos G7 com resultados positivos de PCR (heterozigotos) são chocados. (E) Adultos do G7 são cruzados novamente para aumentar as chances de obter indivíduos homozigotos. (F) Fêmeas G7 alimentadas com sangue são coletadas individualmente para coleta de ovos G8, representando três genótipos. (G) Extração de DNA genômico, PCR e eletroforese em gel são realizados em fêmeas G7 para identificar indivíduos homozigotos. (H) Ovos de fêmeas não mutantes ou heterozigóticas são descartados; ovos homozigotos são mantidos. (I) Os ovos G8 são chocados e cruzados em uma gaiola separada. (J) Dez machos adultos são selecionados aleatoriamente de cada gaiola para eletroforese em gel e sequenciamento. (K) Os mosquitos homozigotos são identificados e mantidos como estoque. Clique aqui para ver uma versão maior desta figura.
| Construção de injeção | Concentração final |
| Proteína Cas9 | 100-300 ng/μL |
| gRNA | 50-100 ng/μL |
| plasmídeo doador | 100-500 ng/μL |
Tabela 1: As faixas de concentração final sugeridas para cada construção de injeção.
Discussão
A tecnologia CRISPR-Cas mudou o cenário da edição do genoma, promovendo mudanças específicas do alvo nos cromossomos1. Embora os elementos transponíveis tenham sido essenciais para a geração dos primeiros mosquitos transgênicos, seus locais de inserção são um tanto aleatórios, e a expressão da construção de carga (promotor + gene) pode não corresponder ao perfil de expressão do gene real devido a um efeito posicional do genoma (ou seja, local de inserção), que geralmente leva à expressão ectópica21. Antecedendo o surgimento da tesoura molecular CRISPR-Cas9, foram desenvolvidas tecnologias que promovem quebras de fita dupla de DNA em locais-alvo específicos e se basearam nas propriedades de ligação ao DNA de domínios proteicos específicos marcados com a enzima de restrição FokI, como TALEN22 e Zinc Finger23. Embora a última tecnologia tenha sido utilizada com sucesso para a engenharia genética de mosquitos24,25, o alto custo de engenharia de tais domínios e o potencial de clivagem fora do alvo levaram à descontinuação de tais tecnologias. Desde então, a tecnologia CRISPR-Cas9 tornou-se a tesoura molecular de escolha11.
A engenharia genética de mosquitos tem sido, em sua maior parte, realizada por injeção da mistura de transformação em embriões 26,27. A criação de microinjeção embrionária de mosquito requer foco, paciência e atenção aos detalhes. Durante as 2 h após a postura dos ovos, esses embriões devem ser alinhados, injetados e lavados com o máximo de cuidado possível para não atrapalhar seu desenvolvimento. O foco é uma habilidade essencial para reduzir a possibilidade de os óvulos serem danificados ou os embriões serem dessecados durante o alinhamento. Diante de incidentes inesperados, como entupimento da agulha, quebra da ponta e vazamento da mistura de injeção, que acontecem com bastante frequência quando a proteína Cas9 está na mistura, a paciência é outra habilidade essencial para gerenciar essas situações. Pocking embriões quando a ponta da agulha é muito grande ou muita mistura de injeção está vazando só pode danificar o embrião. A melhor solução é substituir a agulha e continuar com as injeções. Não menos importante é a atenção aos detalhes. Os mosquitos fêmeas mais jovens (6 a 10 dias de idade) põem mais ovos do que os mais velhos, o que é útil para colher embriões saudáveis. Escolha água deionizada ou ultrapura e toalhas de papel marrom (não branqueadas) para armazenar os embriões após a microinjeção, pois qualquer contato com água contaminada ou superfícies branqueadas pode aumentar significativamente a mortalidade embrionária. Os embriões cinza claro (<1 h de idade) são mais macios, o que facilita o procedimento de injeção, aumentando as chances de obter a linhagem mutante. Além disso, os embriões injetados devem repousar em papel de filtro umidificado por pelo menos 5 dias antes de serem chocados para permitir a recuperação e cicatrização completas.
A qualidade e pureza da mistura injetável são fundamentais para a sobrevivência do embrião. Nem tampões, sais ou álcoois de kits de extração de plasmídeo ou kits de síntese de gRNA devem ser transferidos para as misturas de injeção. Embora cara, a opção de solicitar os gRNAs pode aumentar a probabilidade de dobramento adequado do RNA. Também recomendamos injetar a proteína Cas9 recombinante sobre um plasmídeo que codifica Cas9, pois acreditamos que a versão recombinante já está carregada e pronta para realizar quebras de fita dupla em comparação com uma fonte de plasmídeo que precisa ser transcrita e traduzida e, em seguida, carregada com gRNAs para começar a agir sobre o DNA.
Embora tenhamos descrito o protocolo que funciona melhor em nossas mãos no que diz respeito ao uso da tecnologia CRISPR-Cas9 para edição do genoma do mosquito A. aegypti, reagentes alternativos foram descritos na literatura que também podem ser usados como substitutos, se necessário. Por exemplo, suspendemos todos os reagentes e diluímos as misturas de injeção com água ultrapura. Alternativamente, as misturas injetáveis podem ser diluídas em um tampão injetável feito de 5 mM KCl e 0,1 M NaPO4 (pH = 6,8)26,28. Além de usar óleo de halocarbono 700 para proteger os embriões da dessecação após alinhá-los, o óleo de halocarbono 2729, bem como uma mistura de 27 e 70030, fornecem proteção semelhante. A versão concentrada 10x do tampão de injeção descrita acima tem sido usada alternativamente para prevenir a dessecação do embrião26. Como mencionado acima, os plasmídeos que codificam Cas9 estão disponíveis comercialmente (addgene) e têm sido usados para edição de genoma mediada por CRISPR-Cas9 em mosquitos31.
Entre as estratégias para obter uma linhagem transgênica de mosquitos, estão disponíveis abordagens de knockout mediadas por NHEJ e knockin mediadas por HDR. Enquanto a taxa de sucesso do primeiro é muito maior do que a do último32,33, o esquema de cruzamento e o processo de triagem são muito mais demorados para obter um nocaute do que uma linha de knockin, conforme descrito na Figura 4. Como uma abordagem alternativa para obter uma linha de nocaute sem passar por esquemas de cruzamento complicados, o knockin mediado por HDR de um de genes que codifica um marcador fluorescente oferece a possibilidade de interromper a sequência de codificação de um gene e selecionar a linha transgênica de mosquito usando o marcador fluorescente14,34.
Embora este protocolo se concentre apenas nas etapas básicas para a transformação genética de mosquitos, projetar plasmídeos doadores para HDR é uma etapa essencial que precisa ser considerada. É importante usar promotores como 3xP3, OpIE2, Hr5-IE1 e Pub 6,35,36,37,38. 3xP3 é um promotor sintético comumente usado para impulsionar a expressão no sistema nervoso e nos olhos de insetos. OpIE2 e Hr5-IE1 são promotores derivados de baculovírus que são usados para impulsionar uma forte expressão em células de insetos. O promotor de pub impulsiona a expressão em todo o corpo. O promotor U6 é usado para a expressão de gRNAs. Marcadores fluorescentes como dsRed (e TdTomato), eGFP, CFP e YFP também são usados para plasmídeos doadores para HDR. Estas são proteínas fluorescentes que emitem fluorescência vermelha, verde, ciano e amarela, respectivamente. Para UTRs 3 ', p1039 e SV4040 são frequentemente usados para aumentar a estabilidade do mRNA e a eficiência da tradução em transgenes.
Como resumo do procedimento de microinjeção, para as linhagens mutantes knockout, primeiro alinhe os embriões de mosquito em fita dupla face lado a lado para injeção (Figura 4A). Em segundo lugar, remova a maior parte do óleo de halocarbono para facilitar a taxa de sobrevivência dos embriões e transfira os embriões da fita dupla-face para um papel de filtro úmido limpo. Mantenha-os em um copo com água ultrapura para manter a umidade alta por alguns dias, esperando que os embriões se desenvolvam completamente (Figura 4B). Terceiro, ecloda os embriões injetados, separe as pupas em ambos os sexos e coloque-as em gaiolas diferentes (Figura 4C). Quarto, coloque todos os machos fluorescentes em uma gaiola para cruzar com fêmeas WT de 5 a 7 dias de idade e vice-versa (Figura 4D). Quinto, colete ovos G1 das fêmeas WT cruzadas para expandir o número de mosquitos no início (Figura 4E). Sexto, incubação de ovos G1 e separação das pupas em ambos os sexos (Figura 4F). Sétimo, adultos do G1 cruzam novamente com o WT (Figura 4G). Oitavo, coletar fêmeas adultas G1 em frascos para coleta individual de ovos G2 e manter água ultrapura no fundo dos frascos usando bolas de algodão ou papel toalha por alguns dias (Figura 4H). Nono, coletar fêmeas G1 após a postura dos ovos e extrair o DNA genômico das fêmeas G1 para PCR e sequenciamento (Figura 4I). Décimo, chocar ovos G2 corretos para manutenção de linha e cruzamentos com WT por mais três gerações para remover a maioria das mutações de fundo não vinculadas (Figura 4J). Décimo primeiro, estabelecer uma linhagem de mosquito homozigoto usando PCR, ensaio de enzima de restrição, eletroforese em gel e sequenciamento para rastrear mutações (Figura 4K).
Para o estabelecimento de linhas HDR, alinhe os embriões de mosquito em fita dupla face lado a lado para microinjeção de embriões (Figura 4i). Em segundo lugar, após as injeções, remova a maior parte do óleo de halocarbono e armazene os embriões em um copo com água ultrapura para manter a umidade por alguns dias e aguarde o amadurecimento dos embriões (Figura 4ii). Terceiro, eclodir embriões injetados e rastrear larvas G0 para classificar aqueles com ou sem fluorescência (Figura 4iii); larvas com fluorescência significa que o plasmídeo foi injetado em embriões de mosquitos com sucesso. Quarto, permitir que as larvas fluorescentes amadureçam e se tornem adultas e colocar todos os machos fluorescentes em uma gaiola para cruzar com as fêmeas WT, e vice-versa, para expandir o número de mosquitos (Figura 4iv). Quinto, coletar ovos das fêmeas WT cruzadas (Figura 4v). Sexto, eclodir, peneirar e classificar as larvas de fluorescência G1 e descartar as larvas sem fluorescência (Figura 4vi). Sétimo, cruze adultos de fluorescência G1 com WT (Figura 4vii). Oitavo, coletar ovos G2 em frascos plásticos individualmente para separar os diferentes locais de inserção do plasmídeo nos mosquitos (Figura 4viii). Nono, chocar os ovos, verificar a aptidão do mosquito e realizar o sequenciamento (Figura 4ix). Décimo, selecione duas linhagens com fenótipo mais forte e cruze com WT por mais três gerações (Figura 4x). Décimo primeiro, selecionando larvas com fluorescência para se cruzarem por mais duas gerações para estabelecer uma linha homozigótica para novos experimentos (Figura 4xi).
Aqui, fornecemos o protocolo mais abrangente para edição do genoma de A. aegypti com CRISPR-Cas9 que usamos para gerar várias linhagens transgênicas. Sugerimos que este protocolo seja usado como ponto de partida para outros investigadores, que devem fazer alterações com base na experiência obtida com seus ensaios de microinjeção. Diferentes reagentes e condições devem sempre ser testados. A taxa de sucesso da edição de genes em mosquitos ainda não foi totalmente otimizada e muito poucos laboratórios foram capazes de produzir mosquitos transgênicos via CRISPR-Cas9.
Divulgações
A O.S.A. é fundadora da Agragene, Inc. e da Synvect, Inc. com participação acionária. Os termos deste acordo foram revisados e aprovados pela Universidade da Califórnia, San Diego, de acordo com suas políticas de conflito de interesses. Os demais autores declaram não haver interesses conflitantes.
Agradecimentos
Os autores agradecem a Judy Ishikawa e Ava Stevenson por ajudarem na criação de mosquitos. Este trabalho foi apoiado por financiamento de prêmios do NIH (R01AI151004, RO1AI148300, RO1AI175152) concedidos à OSA e K22AI166268 ao NHR As figuras foram criadas usando o BioRender.
Materiais
| Name | Company | Catalog Number | Comments |
| 10x Cas9 reaction buffer | PNA Bio | CB01 | |
| Benchling software | Benchling | N/A | www.benchling.com |
| Cas9 dilution buffer | PNA Bio | CB03 | |
| Cas9 protein | PNA Bio | CP01-50 | |
| DH5α E. coli Competent Cells | New England Biolabs | C2987 | |
| Double-sided sticky tape | Scotch Permanent | 3136 | |
| Drosophila vials | Genesee Scientific | 32-109 | |
| Filter papers | GE Healthcare Life Science | 1450-042 | |
| Fish food | Tetra | B00025Z6YI | goldfish flakes |
| Flugs | Genesee Scientific | AS273 | |
| Fluorescent microscope | Leica Microsystems | M165 FC | |
| Gene fragment | Integrated DNA Technologies | N/A | |
| gRNA | Synthego | N/A | |
| Halocarbon oil 700 | Sigma-Aldrich | H8898 | |
| Injection microscope | Leica Microsystems | DM2000 | |
| JM109 E. coli Competent Cells | Zymo Research | T3005 | |
| Microinjector | Eppendorf | FemtoJet 4x | |
| Microloader Tips for Filling Femtotips | Eppendorf | E5242956003 | |
| Micromanipulator | Eppendorf | TransferMan 4r | |
| Micropipette Pullers | Sutter Instrument | P-2000 | |
| Microscope Cover Glass | Fisherbrand | 12-542-B | |
| Microscope slide | Eisco | 12-550-A3 | |
| Mouse blood (live mice used for feeding) | University of California | IACUC, Animal Use Protocol #S17187 | Used for mosquito blood feeding; details comply with animal ethics protocols |
| NEB Q5 High-Fidelity DNA polymerase | New England Biolabs | M0491S | |
| PCR Purification Kit | Qiagen | 28004 | |
| Plasmid Miniprep Kit | Zymo Research | D4036 | |
| Quartz filament | Sutter Instruments | QF100-70-10 | |
| Transcription Clean-Up Kit | Fisher Scientific | AM1908 | |
| Ultra-pure water | Life Technologies | 10977-023 |
Referências
- Anzalone, A. V., Koblan, L. W., Liu, D. R. Genome editing with CRISPR-Cas nucleases, base editors, transposases and prime editors. Nat Biotechnol. 38 (7), 824-844 (2020).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Shen, H., Li, Z. DNA double-strand break repairs and their application in plant DNA integration. Genes (Basel). 13 (2), 322 (2022).
- Vinauger, C., et al. Modulation of host learning in Aedes aegypti mosquitoes. Curr Biol. 28 (3), 333-344.e8 (2018).
- Li, M., Bui, M., Yang, T., Bowman, C. S., White, B. J., Akbari, O. S. Germline Cas9 expression yields highly efficient genome engineering in a major worldwide disease vector. Proc Natl Acad Sci U S A. 114 (49), E10540-E10549 (2017).
- Chaverra-Rodriguez, D., et al. Targeted delivery of CRISPR-Cas9 ribonucleoprotein into arthropod ovaries for heritable germline gene editing. Nat Commun. 9 (1), 3008 (2018).
- Rouyar, A., et al. Transgenic line for characterizing GABA-receptor expression to study the neural basis of olfaction in the yellow-fever mosquito. Front Physiol. 15, 1381164 (2024).
- Ang, J. X. D., et al. Considerations for homology-based DNA repair in mosquitoes: Impact of sequence heterology and donor template source. PLoS Genet. 18 (2), e1010060 (2022).
- Zhang, J. -. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biol. 18 (1), 35 (2017).
- Coutinho-Abreu, I. V., Akbari, O. S. Technological advances in mosquito olfaction neurogenetics. Trends Genet. 39 (2), 154-166 (2023).
- Li, M., et al. Targeting sex determination to suppress mosquito populations. eLife. 12, RP90199 (2024).
- Zhan, Y., Alonso San Alberto, D., Rusch, C., Riffell, J. A., Montell, C. Elimination of vision-guided target attraction in Aedes aegypti using CRISPR. Current Biol. 31 (18), 4180-4187.e6 (2021).
- Greppi, C., et al. Mosquito heat seeking is driven by an ancestral cooling receptor. Science. 367 (6478), 681-684 (2020).
- Potter, C. J., Tasic, B., Russler, E. V., Liang, L., Luo, L. The Q system: a repressible binary system for transgene expression, lineage tracing, and mosaic analysis. Cell. 141 (3), 536-548 (2010).
- Riabinina, O., et al. Improved and expanded Q-system reagents for genetic manipulations. Nat Methods. 12 (3), 219-222 (2015).
- Labun, K., et al. CHOPCHOP v3: expanding the CRISPR web toolbox beyond genome editing. Nucleic Acids Res. 47 (W1), W171-W174 (2019).
- Concordet, J. -. P., Haeussler, M. CRISPOR: intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Res. 46 (W1), W242-W245 (2018).
- Williams, A. E., et al. Quantifying fitness costs in transgenic Aedes aegypti mosquitoes. J Vis Exp. , e65136 (2023).
- Bassett, A. R., Tibbit, C., Ponting, C. P., Liu, J. -. L. Highly efficient targeted mutagenesis of Drosophila with the CRISPR/Cas9 system. Cell Rep. 4 (1), 220-228 (2013).
- Coutinho-Abreu, I. V., Zhu, K. Y., Ramalho-Ortigao, M. Transgenesis and paratransgenesis to control insect-borne diseases: current status and future challenges. Parasitol Int. 59 (1), 1-8 (2010).
- Christian, M., et al. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics. 186 (2), 757-761 (2010).
- Kim, Y. G., Cha, J., Chandrasegaran, S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain. Proc Natl Acad Sci U S A. 93 (3), 1156-1160 (1996).
- DeGennaro, M., et al. orco mutant mosquitoes lose strong preference for humans and are not repelled by volatile DEET. Nature. 498 (7455), 487-491 (2013).
- McMeniman, C. J., Corfas, R. A., Matthews, B. J., Ritchie, S. A., Vosshall, L. B. Multimodal integration of carbon dioxide and other sensory cues drives mosquito attraction to humans. Cell. 156 (5), 1060-1071 (2014).
- Lobo, N. F., Clayton, J. R., Fraser, M. J., Kafatos, F. C., Collins, F. H. High efficiency germ-line transformation of mosquitoes. Nat Protoc. 1 (3), 1312-1317 (2006).
- Kistler, K. E., Vosshall, L. B., Matthews, B. J. Genome engineering with CRISPR-Cas9 in the mosquito Aedes aegypti. Cell Rep. 11 (1), 51-60 (2015).
- Handler, A. M., Harrell, R. A. 2nd Transformation of the Caribbean fruit fly, Anastrephasuspensa, with a piggyBac vector marked with polyubiquitin-regulated GFP. Insect Biochem Mol Biol. 31 (2), 199-205 (2001).
- Harrell, R. A. . 2nd Mosquito embryo microinjection under halocarbon oil or in aqueous solution. 2024 (7), (2024).
- Sun, R., Raban, R., Akbari, O. S. Generating mutant strains with transgenic Cas9. Cold Spring Harb Protoc. 2023 (9), 671-678 (2023).
- Giraldo, D., et al. An expanded neurogenetic toolkit to decode olfaction in the African malaria mosquito Anopheles gambiae. Cell Rep Methods. 4 (2), 100714 (2024).
- Liu, G., Lin, Q., Jin, S., Gao, C. The CRISPR-Cas toolbox and gene editing technologies. Mol Cell. 82 (2), 333-347 (2022).
- Chu, V. T., et al. Increasing the efficiency of homology-directed repair for CRISPR-Cas9-induced precise gene editing in mammalian cells. Nat Biotechnol. 33 (5), 543-548 (2015).
- Laursen, W. J., et al. Humidity sensors that alert mosquitoes to nearby hosts and egg-laying sites. Neuron. 111 (6), 874-887.e8 (2023).
- Weng, S. -. C., Antoshechkin, I., Marois, E., Akbari, O. S. Efficient sex separation by exploiting differential alternative splicing of a dominant marker in Aedes aegypti. PLoS Genet. 19 (11), e1011065 (2023).
- Li, M., et al. Development of a confinable gene drive system in the human disease vector. eLife. 9, e51701 (2020).
- Dalla Benetta, E., et al. Engineered Antiviral Sensor Targets Infected Mosquitoes. The CRISPR journal. 6 (6), 543-556 (2023).
- Li, H. -. H., et al. C-Type lectins link immunological and reproductive processes in Aedes aegypti. iScience. 23 (9), 101486 (2020).
- van Oers, M. M., Vlak, J. M., Voorma, H. O., Thomas, A. A. M. Role of the 3' untranslated region of baculovirus p10 mRNA in high-level expression of foreign genes. J Gen Virol. 80 (Pt 8), 2253-2262 (1999).
- Salem, T. Z., et al. The influence of SV40 polyA on gene expression of baculovirus expression vector systems. PloS One. 10 (12), e0145019 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados