Method Article
CRISPR-Cas9を用いた黄熱病ネ ッタイシマカ のゲノム編集
要約
ここでは、CRISPR-Cas9技術を用いた蚊A . aegypti への胚マイクロインジェクションによるゲノム編集の詳細なプロトコールについて説明します。
要約
クラスター化され、規則的に散在する短い回文反復(CRISPR)-Cas9技術の出現は、遺伝子工学分野に革命をもたらし、非モデル生物を含む複数の種における正確なゲノム編集の扉を開きました。ネ ッタイシマカでは、この技術により機能喪失型突然変異とDNA挿入が達成されています。ここでは、CRISPR-Cas9技術を用いた蚊A . aegypti への胚マイクロインジェクションによるゲノム編集の詳細なプロトコールについて、遺伝子ノックアウトラインとノックインラインの両方に焦点を当てて説明します。このプロトコルでは、石英針は、ガイドRNA、組換えCas9、および遺伝子ノックインが所望される場合には、蛍光マーカー用の遺伝子をコードするDNAカセットを含むプラスミドの混合物で満たされる。胚盤葉前胚盤葉期の胚は、カバーガラス上に置かれた両面粘着テープのストリップに並べられ、続いてスライドガラスに取り付けられます。マイクロインジェクターの助けを借りて、針を胚の後端に穏やかに挿入し、少量のCRISPR混合物を分注します。胚が孵化すると、幼虫は蛍光スコープでチェックされ、蛹は性別に分類され、異なるケージに分離されます。成虫が出てくると、これらは野生型の個体と相互に交配され、血液を与えられ、産卵のために配置されます。これらの卵が孵化すると、収集された蛍光幼虫は、DNAカセットがゲノムに安定して挿入された個体を表しています。次に、これらの幼虫を成虫期まで成長させ、野生型の個体と交配した後、分子技術によってさらに評価を行い、DNAカセットの正確な配列が蚊のゲノムの所望の部位に存在することを確認します。ホモ接合型ラインは、提供されたクロッシングスキーマおよび突然変異の分子スクリーニングのパイプラインに従うことによっても得ることができる。
概要
正確なゲノム編集は、分子はさみのCRISPR-Cas技術の確立により、間違いなく容易になりましたが、可能になりました1。これらの技術は、原核生物の免疫系がファージ感染と戦うために使用するメカニズムを利用しています2。これらの系の中で、Cas9ヌクレアーゼとともにクラスター化された規則的に散在する短い回文反復(CRISPR)は、通常、標的DNAと相同な配列を持つ20の塩基対RNA、ガイドRNA(gRNA)に依存し、その後にNGGプロトスペーサー隣接モチーフ(PAM)配列3が続きます。Cas9にロードされたgRNAは、これらのヌクレアーゼをゲノムの特定の標的部位に正確に導き、二本鎖DNA切断3を引き起こします。
DNA二本鎖切断は、二重らせんにパッチを当てる修復機構を誘導する4。DNA修復には正確であることが期待されますが、精度の低いDNA修復メカニズムが存在し、その結果、配列の傷跡が残り、ひいては機能喪失型突然変異が生じる可能性があります4。エラーを起こしやすいDNA修復メカニズムの中で、非相同末端結合(NHEJ)は、小さな欠失、挿入、ヌクレオチド変化(SNP)などのフレームシフト変異を引き起こし、機能喪失型変異を引き起こす可能性があります。一方、HDR(Homology Directed Repair)メカニズムは、損傷を受けていない対立遺伝子の正確な配列をコピーし、標的DNA配列4を完全に修復するためのテンプレートとして相同染色体に依存しています。
この知識に基づいて、CRISPR-Cas9技術は、おそらくPAM部位3を含む任意の配列でゲノムを正確に編集するために開発されました。蚊では、NHEJの修復機構5,6を利用して、Cas9とgRNAの混合物を胚マイクロインジェクションすることで、CRISPR-Cas9技術を用いてさまざまな遺伝子をノックアウトしています。同様の生殖細胞系突然変異誘発は、成虫の雌の蚊の血リンパにgRNA+Cas9ミックスを注入することで得られる7。この技術はReMOTコントロールと呼ばれ、卵子の発育(ビテロジェネシス)の過程で卵巣に取り込まれるペプチドでタグ付けされたCas9の改変バージョンに依存しています7。特定の遺伝子カセットをゲノムにノックインするには、gRNAとCas9(またはそれらの分子を発現するプラスミド)の混合物と、目的のDNAカセット8をコードするプラスミドを胚マイクロインジェクションすることによってのみ可能です。HDRメカニズムを利用して、標的部位の上流および下流に相同配列(500-1,000bp)9,10が隣接する目的のDNAカセットを含むプラスミドをテンプレートとして使用して、二本鎖切断を書き換え、DNAカセットも標的配列9にコピーする。
CRISPR-Cas9技術は、ネッタイシマカ11の感覚系に主に関与する複数の遺伝子をノックアウトするために使用されてきたが、人口制御のための雄の生殖能力と雌の生存能力(PgSIT)に関連する遺伝子もノックアウトするために使用されてきた12。標的遺伝子のノックアウトは、蛍光マーカーをコードする遺伝子を特定の遺伝子のコード配列にノックインすることによっても達成されている13,14。この戦略は、フレームシフト突然変異を誘導するだけでなく、蛍光光を使用して新しいノックアウトライン13,14の個体を選別することを可能にするという利点を有する。A. aegyptiのゲノムは、Qシステム(QF-QUAS)11などのバイナリ発現系の配列でも編集されています。特定の遺伝子のプロモーターに下流のQFトランスアクチベーターをコードする遺伝子をノックインすると、トランスアクチベーター15,16の明確な時空間的発現が保証されます。QFを発現する蚊の系統が、QFの結合部位(QUAS)を含む別の蚊の系統に渡ると、後者はそれに結合し、QUAS配列15,16の下流の遺伝子の発現を誘発する。このシステムは、全体として、細胞の局在化またはニューロン活動の検出に使用される蛍光マーカーであり、さらには特定の組織の遺伝子を破壊する(すなわち、体細胞ノックアウト)ためのCas9ヌクレアーゼである可能性のある、そのようなエフェクター遺伝子の組織特異的および時間特異的な発現を可能にする11。
A. aegyptiの遺伝子形質転換について利用可能なすべての情報を考慮して、ここでは、胚マイクロインジェクションを通じてCRISPR-Cas9システムを使用してゲノム編集を実行するための段階的な指示を含む詳細なプロトコルを提供します。NHEJによって媒介されるフレームシフト変異と欠失によるノックアウトと、HDRを介した遺伝子カセット挿入によるノックインラインの両方を生成するための戦略について説明します。
プロトコル
このプロトコールで使用される機器および試薬に関連する詳細は、 資料表に記載されています。すべての動物は、国立衛生研究所が推奨する実験動物の世話と使用のためのガイドに従って取り扱われました。この手順は、UCSDの施設内動物管理および使用委員会(IACUC、動物使用プロトコル #S17187)およびUCSD生物学的使用許可(BUA #R2401)によって承認されました。
1. gRNAとドナープラスミドの設計
- ノックアウト変異体を作製するには、20~100 bpの間隔で2つのgRNAを設計します(図1A)。
- CHOPCHOP(https://chopchop.cbu.uib.no/)17、Benchling(benchling.com)、CRISPOR(http://crispor.gi.ucsc.edu/)18などのオンラインツール(図1A)を使用して、PAM配列(NGG)を除くCRISPR-Cas9用の20 bp gRNAを設計します。オンラインツールが提案するように、実験に最も特異的でオフターゲットフリーのgRNAを選択します。
- 効率的な制限酵素切断部位がgRNA切断部位間の配列に含まれていることを確認します(図1A)。gRNAカット部位の間に制限酵素部位を含めることで、編集の成功を視覚的に素早く確認することができます。
注:欠失が起こると、制限部位が除去されるため、酵素は配列を切断せず、ゲル上に単一の未切断バンドを生成して欠失を確認します。
- HDRを介した遺伝子カセット挿入の場合、ノックアウト変異体の作成について言及したツールを使用して複数のgRNAを設計し、後で評価した後に最も効果的なものを選択します。
- 標的部位の相同性アーム、カーゴDNA配列、蛍光マーカー、複製の起源、および抗生物質耐性遺伝子を含むHomology-Directed Repair(HDR)のドナープラスミドを設計します(図1B)。
- ターゲットサイトの上流領域と下流領域からホモロジーアームを選択し、それぞれが500〜1,000 bp10 に及びます(図1B)。
- カーゴ配列を選択し、これには蛍光マーカー、目的の遺伝子、または調節要素(QF2など)が含まれ得る。
- 蛍光マーカーを選択します。 A. aegypti に使用される一般的な蛍光マーカーは、プロモーター、蛍光マーカーをコードする遺伝子、および3' UTR配列を含むDNAカセットに含まれています。詳細については、ディスカッションを参照してください。
2.インジェクションミックスの準備
- Cas9タンパク質をCas9希釈バッファーで1 μg/μLに希釈します。
注意: Cas9を2倍以上解凍して凍結しないでください。Cas9タンパク質のアリコートを作ることをお勧めします。 - gRNAを購入するか、社内で作製します。
注:自社生産の場合は、標準的なRNA除染方法とRNAseフリーの材料を使用してください。- 4-6 gRNAを設計し、注射に最適な2つのgRNAを選択します(以下の in vitro 切断アッセイを参照)。
- gRNA配列の上流にT7プロモーター配列を含むフォワードプライマーを設計します。以下で説明するように、非テンプレートPCR反応にはユニバーサルgRNAリバースプライマーを使用してください。オーバーラップする配列の塩基は、ユニバーサルリバースプライマー(太字と下線)の対応する配列とペアになり、DNAポリメラーゼがそれらの配列を増幅するためのテンプレートを作成します。
注:これらのPCR反応では、フォワードプライマーにはT7プロモーター(太字)、2つのグアニン(T7 RNAポリメラーゼを介した転写開始に重要)、gRNAの20ヌクレオチド配列(N20、PAM配列なし)、およびプライマーオーバーラップ配列(下線部)が含まれている必要があります。
プライマーフォワード:5'- GAAATTAATACGACTCACTATAGGN20 GTTTTAGAGCTAGAAATAGC- 3'
プライマーリバース:5''-AAAAGCACCGACTCGTGCCACTTTTT CAAGTTGATAACGGACTAGC
CTTATT TTAACTT GCTATTTCTAGCTCTAAAAC - 3' - 非テンプレートPCRによりDNAテンプレートを合成します。 in vitro 転写反応に十分なPCR産物(300 ng)が得られるように、複数の反応(それぞれに2x Master Mixが12.5 μL、フォワードプライマーの10 μM溶液が1.25 μL、リバースプライマーの10 μM溶液が1.25 μL、超純水が10 μL)をセットします。以下のPCR条件を使用してください:98°Cで30秒間の初期変性;98 °C で 10 秒、62 °C で 10 秒、72 °C で 10 秒の 35 サイクルの増幅。72°Cで2分間の最終延長。4°Cで保存します。
- アガロースゲル上での単一DNA断片(122塩基対)の増幅を確認します(2%)。
- PCR精製キットを使用して、メーカーの推奨に従ってPCRテンプレートをクリーンアップします。
- T7 Transcription Kitを用いて in vitro 転写反応を行います。10x反応バッファー2μL、ヌクレオチド(ATP、CTP、UTP、GTP)各2μL、T7 RNAポリメラーゼ2μL、テンプレートDNA(100 ng/μL)3μL、超純水5μLを混合します。混合物を37°Cで少なくとも2時間(16時間以内)から一晩(12時間)インキュベートします。
注:この反応では、T7 RNAポリメラーゼはプライマーフォワード配列に含まれるT7プロモーターに結合し、gRNAの転写につながります。 - DNaseで転写反応を処理するには、1 μLのDNaseを添加し(よく混合)、37°Cで15分間インキュベートします。
- 合成されたsgRNAは、メーカーの推奨に従って転写クリーンアップキットで精製します。
- in vitro切断アッセイを実施して、選択したgRNAの切断効率を評価します(図1C)。
- gRNA切断部位に隣接するDNAフラグメント(500-1,000 bp)を増幅するプライマーを設計します。
- PCR反応液は、2xマスターミックス12.5 μL、フォワードプライマー10 μM溶液1.25 μL、リバースプライマー10 μM溶液1.25 μL、超純水9 μL、5 ng/μLテンプレートDNA1 μLで設定します。
- 以下のPCR条件を使用してください:条件:98°Cで30秒間の初期変性;98°Cで10秒、X°Cで15〜30秒(プライマーの理想的なアニーリング温度)、および72°Cで35秒間の35サイクルの増幅。72°Cで2分間の最終延長。最終的な長期保存は4°Cで行います。
- 最適な増幅温度と時間寿命については、メーカーのガイドラインを参照してください。PCRクリーンアップ(シングルバンド)またはアガロースゲルPCRバンド精製後に十分なPCR産物(1.5-2 μg)が得られるように、少なくとも5つの反応をプールするか、試薬の量を増やします。
- 10x Cas9反応バッファー1 μL、組換えCas9 0.35 μL (1 μg/μL)、gRNA 1 μL (100 ng/μL)、6.65 μL の超純水、300 ng/μL のテンプレート DNA 1 μL の混合物を 37 °C で 1 時間インキュベートして、組換え Cas9 との Cas9 切断反応を設定します。
- gRNAを含まないネガティブコントロール反応を設定します。
- アガロースゲル(1.5-2.0%)で反応を実行して、切断効率を確認します。 図1C)。
3. ドナープラスミドの組み立て
- A. aegyptiのゲノムDNAから相同性アームをPCR増幅します。
- プライマーのデザインについては、クローニングキットメーカーのガイドラインを参照してください。さまざまなクローニング戦略(Gibson、Golden Gate、制限酵素ベースのクローニングなど)を使用したプラスミドの設計とアセンブリをサポートするオンラインツール(Benchlingなど)を使用してください。
- A. aegyptiゲノムDNAからカーゴ配列を増幅するか、商業的に合成された遺伝子断片を注文します。
- プラスミドからの蛍光マーカーを社内で増幅します。
- ステップ3.1.1から3.1.3までのDNA断片を、ギブソンアセンブリにより既存のプラスミドの骨格にライゲーションします。10 μL の 2x マスターミックス、X μL の全 DNA フラグメント、10-X μL の超純水の混合物を 50 °C で 1 時間インキュベートします。
注:プラスミドバックボーンに対するインサートの理想的な比率を計算するためのメーカーのガイドラインを参照してください。- 各クローニングフラグメントの濃度をピコモルで計算します:pmols = (重量 (ng) × 1,000/(塩基対 × 650 ダルトン)。
- 50〜100 ngのプラスミド骨格と各インサートの2〜3倍のモル過剰を使用します。
- 2〜3個のフラグメントを組み立てる場合は、各フラグメントの0.02〜0.5 pmolsをGibson反応に加えます。4〜6個のフラグメントを組み立てる場合は、各フラグメントに0.2〜1.0 pmolsを追加します。
- 3-5 μLのギブソンアセンブリ反応を使用して、 大腸菌 コンピテントセルJM109を形質転換します。
注:JM109は、その recA- 遺伝子型により、望ましくない組換えイベントを減らし、細胞収穫中のヌクレアーゼキャリーオーバーを防ぎ、組み立てられたDNA断片の完全性を確保するため、ギブソンアセンブリに選択されました
10 kbを超えるプラスミドの場合、メーカーの推奨に従って、拡張プロトコルを使用したDH5αコンピテントセルの形質転換を推奨します。 - 形質転換細菌を増殖させ、miniprepキットを使用してプラスミドを精製します。
- 正しいプラスミドアセンブリを確認するために、精製したプラスミドDNAで診断用制限酵素消化物を実行し、アガロースゲル電気泳動で可視化します(図1D、E)。
注:プラスミドを1つの部位で切断できるいくつかの制限酵素を選択するためのオンラインツールの使用をお勧めします。Benchlingのようなソフトウェアには、選択した制限酵素によるプラスミド配列の仮想消化を行うツールが組み込まれており、仮想電気泳動ゲル上に予想されるDNAバンドパターンを表示します(図1D)。- プラスミド制限酵素消化を行います。1 μL の 10x 制限酵素消化バッファー、0.5 μL の制限酵素 1、0.5 μL の制限酵素 2、1 μL のプラスミド DNA (300 ng/mL)、および7 μL の超純水の混合物を 37 °C で 2 時間インキュベートします。
- 制限酵素消化反応をアガロースゲル(1.5%)で実行します。
注:DNAバンドパターン(図1E)は、仮想消化バンドパターン(図1D)に似ている必要があります。プラスミドは、サービスが利用可能であれば、全プラスミドシーケンシングのために送ることもできます。
- プラスミドクローンを150 mLのLB培地に培養します。
- プラスミドmaxiprepは、メーカーのガイドラインに従って実施してください。
- プラスミドを超純水に懸濁します。
4. インジェクションコンストラクトの混合
注:各コンストラクトの推奨最終濃度範囲を 表1に示します。2:1:2(ng Cas9:ng sgRNA:ng donor plusid)の比率から始めて、必要に応じて調整してHDR効率を最適化します。結果をモニタリングすることで、最も効果的な組み合わせを特定することができます。
- ノックアウトミュータントを作るために、
- Cas9希釈バッファーを使用してCas9タンパク質のアリコートを所望の濃度に希釈し、gRNAのアリコートを超純水で希釈します。
- Cas9タンパク質を各gRNAとあらかじめ混合して、リボ核タンパク質(RNP)複合体を形成します。予混合溶液を組み合わせます。
- HDRを介した遺伝子カセット挿入の場合、
- Cas9タンパク質とgRNAを希釈して混合し、ノックアウト変異体を作製します。
- ドナープラスミドを超純水で希釈します。
- すべてのコンストラクトを結合します。
5. マイクロインジェクション針の引っ張りと装填
- クォーツフィラメントを使用して注射針を引っ張ります(図2)。フィラメントの長さが10cm、外径が1mm、内径が0.7mmであることを確認してください。
- レーザーマイクロピペットプーラーと次の2つのプログラムのいずれかを使用して針を引っ張ります。
プログラム1:ヒート805、フィラメント4、速度50、遅延145、プル145
プログラム2:ヒート650、フィラメント4、ベロシティ40、ディレイ150、プル156
注:プログラム1は、針先が細く、長くなるため、より細い先端が精度に有益である低濃度のコンストラクトでの注射に適しています。プログラム2は、より短く、より太い針先をもたらし、構造がより厚く、より頑丈な針を必要とするため、高濃度の注射に最適です。
6.胚採取
- 蚊を捕集するための電気または手動の吸引器を準備し、収集と胚の収穫にプラスチック製のバイアルを使用します。
- 白い丸の濾紙を湿らせ、コレクターの内壁または湿った綿の上に置きます。
- 5〜10日前に血液を与えられた5〜10匹のメスの蚊をコレクターに入れます。コレクターを暗闇に45分間置きます。
- 胚採取用の濾紙を取り出します。
7. 胚のラインナップ
- 胚注入には A. aegypti の野生型株(Liverpool)を使用してください。
- 採取紙から胚盤葉前胚葉期の胚、特に色が薄い胚を選択します(図3)。
- 濡れたブラシでいくつかの胚をカバースリップの上に置かれた両面粘着テープに移します。胚の周囲がまだ濡れている間に胚を平行に整列させ、すべての後端が正面を向くように、胚が並んでいることを確認します。
- 正しく配置されたら、環境を少し乾かして胚を所定の位置に固定します。
- アライメント中にハロカーボンオイル700を胚に添加して、乾燥を防ぎます。
注:油を加えるときは、胚が油に浮くため、胚が水に囲まれていないことを確認してください。
8. 胚マイクロインジェクション
注:注入は室温または18°Cで行われます。 18°Cの温度は、胚の発生を遅らせるため、実用的な理由から推奨されます。
- 次のパラメータでマイクロインジェクターをセットアップします: 補償圧力(Pc)300 hPa、注入圧力(Pi)500 hPa。必要に応じて、これらの条件を調整してください。
- 混合したコンストラクトの3μLをマイクロローダーを使用して針にロードします。
- 胚を整列させたカバーガラスを顕微鏡スライドの上に置きます(図3)。カバーガラスをセットし、顕微鏡下にスライドさせて注射します。胚の後端が針の方に配置されていることを確認してください。
- 1本の針をマイクロマニピュレーターと一緒にニードルホルダーに入れ、整列した胚の後端に向かって針を10°の角度で配置し、静止させます(図4A)。顕微鏡で針をそっと開き、カバースリップの端で軽く触れます。
注:別のオプションは、胚で針を軽くたたいて針に小さな開口部を作ることです。ただし、Cas9タンパク質の粘着性が針を詰まらせる可能性があるため、針の開口部を少し広げることをお勧めします。 - スライドガラスを注射針の方に動かします(図5)。
9.注入された胚のアフターケア
- 糸くずの出ない使い捨てワイプを使用して、胚の周りの油を取り除きます。
- 脱イオン水を加えて胚を洗い流し、湿った濾紙の上に胚を移します。濡れた濾紙をカラット9オンスのカップ(図4B および 図6A、B)内の濡れたティッシュの上に置きます。
- 湿気を保つために、濡れた綿をカップの底に置きます(図6B)。
- 注入した胚を濡れた濾紙の上に保存します。
10. 胚の孵化とG0幼虫のスクリーニング
- 注射後少なくとも4日後、胚を含む濾紙をSterilite 6クォートパンで約3 Lの脱イオン水に移し、孵化させます。.
注:卵は通常、注射後2週間以内により効率的に孵化します。この期間中、卵が湿ったままであることを確認してください。濾紙は、過度の湿気が胚の早期孵化につながる可能性があるため、濡れすぎないようにする必要があります。保存期間が長いと孵化率が向上する可能性がありますが、最適な結果を得るには1か月を超えないようにしてください。脱酸素水を使用すると、孵化にも役立つ場合があります。 - G0の幼虫が孵化したら、水と混ぜた魚の餌を餌として鍋に加えます。
- G0幼虫の3番目から4番目の幼虫期の蛍光マーカーをスクリーニングします(図7)。
- 幼虫は蛍光状態に基づいて別々に維持し、蛍光陽性の幼虫と蛍光陰性の幼虫は別々の鍋に保管します。
11.性選別蛹と野生型への交配
- 蛹になるときに注射された蚊を性別で分離し、生殖葉のサイズと性別固有の構造を使用して識別します。
- 男性:サイズが小さく、性器葉がより目立ち、尖っており、パドルが広くなっています(蛹の尾端の構造。 図8A、1、A2)。
- 女性:サイズが大きく、性器葉が小さく目立たず、パドルが狭い(図8B1、B2)。
- 各性別の蛍光陽性または蛍光陰性の蚊をプールします。
- プールされた蚊とリバプール株の異性の蚊を交配します。野生型とG0蚊の比率は3:1から5:1です。
- 蚊を4日間渡ります。
- 渡った後、女性に血の食事を提供します。
12. G1スクリーニング
- 採血の3日後、カラット9オンスカップの壁の内側にペーパータオルを置き、約3オンスの脱イオン水を追加して、卵カップを提供します。
- 3〜4日後、G1胚を収穫して孵化させます。G1幼虫の蛍光マーカーを3番目から4番目のインスターステージでスクリーニングします(図7)。
- 蛍光幼虫を収集し、HDR挿入を提案します。蛍光G1個体が蛹期に到達したら、性別ごとに分け、各性別を別々のケージに入れます。
- 蛍光G1蚊をリバプール株の異性の個体に交配します。
13. G1挿入部位の確認
- 血粉を提供した後、個々の ショウジョウバエ のバイアルに小さなペーパータオルを置き、3 mLの脱イオン水を加えることにより、G1メスを1匹のメスの産卵用にセットアップします。
- ノックアウト変異体の場合:
- 産卵後、水分が蒸発し、ペーパータオルが乾くと、ノックアウト変異がはっきりと現れる雌の卵を孵化させ、G2世代を得ます。
- G2の子孫を無事に孵化させたG1の母親の体を採取し、DNAを抽出します。
- DNAをPCRのテンプレートとして使用して、標的部位を覆う配列を増幅します。プライマーが~200 bpのDNA断片を増幅することを確認してください。反応液をセットアップします(それぞれに2倍マスターミックス12.5 μL、フォワードプライマー10 μM溶液1.25 μL、リバースプライマー10 μM溶液1.25 μL、超純水9 μL、テンプレートDNA [5 ng/μL]1 μLを含有)。以下のPCR条件を使用してください:98°Cで30秒間の初期変性;98 °C で 10 秒、X °C (プライマーの理想的なアニーリング温度) で 15-30 秒、72 °C で 10 秒の増幅を 35 サイクル行いました。72°Cで2分間の最終延長。最終的な長期保存は4°Cで行います。
- 1 μL の 10x 制限酵素消化バッファー、0.5 μL の制限酵素 1、0.5 μL の制限酵素 2、1 μL の PCR フラグメント (200 ng/mL)、および7 μL の超純水の混合物を 37 °C で 30 分間インキュベートすることにより、PCR フラグメントを精製し、PCR フラグメントの制限酵素消化を行います。
注:特定の制限酵素は、事前の精製なしでPCRミックスに使用することができます。 - ゲル電気泳動によるPCRフラグメントの消化を可視化し、切断が発生したかどうかを確認します。
- 未消化のPCRフラグメントのシーケンシングを行い、ノックアウト変異を同定します。
- HDRを介した挿入の場合、産卵後:
- G1の母親の体を採取し、DNAを抽出。
- DNAをPCRのテンプレートとして使用して、標的部位をカバーする配列を増幅します。
- 増幅したDNA断片のシーケンシングを行い、挿入したDNAカセットの完全性を確認します。
- 卵を孵化させ、G2世代を入手します。
14. 新しいCRISPRラインの拡大
- ノックアウト変異株の場合:
- G2の雌を産卵用にセットアップし、G1ごとに20匹の雌を設定します。各 G1 は 1 つの線を表します。
- 産卵後、G1と同様にノックアウト変異を確認します。
- G1とG2の両方で明確に識別された変異を示す5つのG1系統に進みます。各系統のG2から集めた卵を孵化させ、G3世代を誕生させます。
- 5つの交差系統から、明確に同定された変異と高い適応度、または目的の表現型を持つ2つの系統を選択します。
- 選択した各系統のG3メスをリバプール系統のオスとアウトクロスします。
- 各ラインから産卵用の50匹の独身メスを設定します。G1に対して以前に行ったようにノックアウト変異を確認します。
注:多様性を維持するために、セットアップ内の女性の数を増やします。 - 2 本の線を使用して、さらに 2 つの世代に対して手順を繰り返します (図 4J)。
- 新しい HDR を介した挿入行の場合:
- G2世代における蛍光個体の存在を確認します。
- 各G1個体からのG2蚊をリバプール株と交配します。各 G1 個体は 1 本の線を表します。
- さらに3世代のアウトクロスを進めます。蛍光マーカーを連続的にチェックし、各ラインの適応度19 を観察します(図4x)。
- 正しい挿入と高い適応度、および目的の表現型の1つのラインを選択します。
15.ホモ接合線を作る
- ノックアウト変異株の場合:
- ジェネレーションG6:
- G5で産んだ卵を孵化させ、G6世代を獲得する。
- それぞれのライン内でG6成虫をインタークロスします(図4K および 図9A)。
- 1行あたり50匹のG6メスを産卵用に設定し、前述のように卵を孵化させます(図9B)。
- G7の子孫を成功裏に孵化させたG6の雌を採取し、DNAを抽出し、G1の場合と同様にPCRおよび制限酵素消化を行います(図9C)。
注:このステップでは、シンプルで安価なスクイッシングバッファー(SB)をDNA抽出に使用できます。- ハンドヘルドグラインダーを使用して、1x SB(10 mM Tris-HCl pH 8、1 mM EDTA、および25 mM NaCl)の100 μLで1匹の蚊を浸軟させます。プロテイナーゼK(200 μg/mL)を添加し、37°Cで30分間インキュベートし、続いて95°Cで2分間インキュベートします。10,000 × g で5分間スピンダウンし、上清20を回収します。
- ゲル上で結果を視覚化することにより、突然変異を確認します。
- 突然変異のない女性によって作られた卵子を捨てます(図9D)。
- 変異が確認された雌の幼虫を維持して、いくつかのG7系統を確立し、突然変異のない雛を捨てます(図9D)。
- ジェネレーションG7:
- 各G7系統内の雌と雄を交配します(図9E)。
- 1行あたり10匹のG7メスを産卵用に設定し、前述のように卵を孵化させます(図9F)。
- G7の子孫をうまく孵化させたG7の雌を採取し、DNAを抽出し、PCRおよび制限酵素消化を行います(図9G)。
- ゲル上で結果を視覚化することにより、両方の対立遺伝子または単一の対立遺伝子のノックアウト変異を検出します。
- 両方の対立遺伝子にノックアウトがあることが確認されたG7雌の幼虫を維持し、G8世代を生成します(図9H)。突然変異のない雛を捨てます(図9H)。
注:この手順により、雌がホモ接合体であることが保証されます。G8では、子孫のホモ接合性を検査することで、父親もホモ接合体であるかどうかが確認されます。
- ジェネレーションG8:
- G7の女性系統ごとにG8の成体を交配します(図9I)。
- 5匹のG8オスをランダムに選択し、DNAを抽出し、PCRおよび制限酵素消化を実行します(図9J)。
- 血の食事を提供し、両方の対立遺伝子にノックアウトがあることが確認されたG8男性と交配したG8女性からの卵を孵化させます。
- 検出されたすべての雄が両方の対立遺伝子にノックアウトを持っているG7系統を続けて、これらの系統がホモ接合体であることを示しています。
- ジェネレーションG6:
- HDR を介した挿入行の場合:
- G5で産んだ卵を孵化させ、G6世代を獲得する。G6段階で幼虫を蛍光マーカーのスクリーニングします。
- 蛍光マーカーのない幼虫を捨てます(図4xi)。
- 蛍光マーカーを示すインタークロス蚊。
- 非蛍光性の幼虫が観察されなくなるまで、後続の各世代でこの手順を続けます。この時点で、蚊の系統はホモ接合体になります。
結果
HDR相同組換えのためのgRNA媒介遺伝子ターゲティングの設計と検証
目的の遺伝子を正確に標的にするために、いくつかのgRNAを選択し、5'および3'相同性アームを切断部位の近くに配置して、HDRを介した相同組換えを行うことをお勧めします(図1A)。例えば、目的遺伝子の開始コドンの両側を標的とする2つのgRNAを設計し、QF2-Hsp70-OpIE2-ECFP-SV40カセットをマーカーとして、HDR機構を介して1kbの相同性アームを介して標的遺伝子に挿入しました(図1B)。二本鎖切断は、gRNAの切断部位間の短いDNA断片の欠失につながります(図1A)。
gRNAの切断効率と標的遺伝子を切断する能力を、 in vitro Cas9切断アッセイによって試験しました。標的遺伝子由来のPCR断片をCas9によって2つの断片に切断し、gRNAをロードしました(図1C)。さらに、制限酵素を使用して、プロトコルの下流ステップに進む前に、ドナープラスミドの完全性を確認することができます。プラスミド消化の結果は、ゲル上に1つまたは2つのバンドを表示し、小さなバンドの存在は短いフラグメントへの消化が成功したことを示します(図1D、E)。
マイクロインジェクション針の調製
注射プロセスを開始する前に、新しいマイクロインジェクション針を準備することが重要です。針は、わずかに湾曲した突起状の形状をしている必要があります。針の約2/3は上部が狭く、残りの1/3は幅が広くする必要があります。針先の先端の前端は、細い部分から幅の広い部分までの長さが0.5cmを超えてはなりません(図2A)。カバースリップの端を使用して針を開いた後、黒い蚊の胚の表面にそっと触れて針を折った後に使用するのが簡単なため、先端が斜めの針がマイクロインジェクションに適しています(図2B)。一方、先端が鈍い針は、マイクロインジェクション中に薄い灰色の胚を突き刺すのを困難にする可能性があります(図2C)。針が折れるのを避けるために、針は蚊の胚を効果的に貫通するために柔らかすぎたり硬すぎたりしてはいけません。
胚の注入
注入を成功させるには、まず薄い灰色の蚊の胚を採取し、後極が同じ方向を向くように位置合わせします(図3A)。この方向性の整列は、プラスミドを生殖細胞(後極)の近くに注入するのに役立ち、これは次世代に受け継がれる可能性のある変異体を生成するために重要です。5〜10日前に血液で育てられた女性から新鮮で均一な薄い灰色の胚を選択します(図3A1)。不均一な灰色または茶色の胚(図3A2)は、適切に発生しない可能性があるため、使用しないでください。
蚊の胚を両面テープに1つずつ位置合わせして、正確で効率的な注入を容易にします。卵が乾燥して中程度または濃い灰色に暗くなる前に、ハロカーボンオイルを塗布します(図3B)。
注入するときは、各胚の後極の表面に焦点を合わせます。一方、胚の前面( 図5の右側)は、平らで硬い表面(マイクロパイル)を持っています。注入混合物を含む斜めの針をそっと使用して、胚を突き刺します(図5)。胚の表面が陥凹して跳ね返り、針が胚を貫通したことを示します。注入が成功すると、胚の色は数秒以内に明るくなります( 図5の右上の黄色の曲線)。
注入後、ブラシを慎重に使用して胚を拾い上げ、実験室のワイプを使用して別の湿った濾紙に移し、余分な油分を取り除きます(図6A)。胚を濡れたペーパータオルの上に数日間、飼育温度で保管します(図6B)。過剰な油は、蚊の胚の孵化率を低下させる可能性があります。超純水を入れたコットンボールを使用して水分補給を維持します。注入後2時間後に灰色または茶色(黄色の矢印)のままの胚は、孵化する可能性は低いです(図6C)。
スクリーニングとアウトクロス
胚が成熟したら、エッグカップに超純水を加え、27°Cのインキュベーターに入れて孵化させます。通常、幼虫が3番目のインスターステージに成長するのに5〜7日かかります。最初に、蛍光を示したG0幼虫を事前にスクリーニングしましたが、他の変異株と比較して弱かったか部分的でした。これにより、マーカーが正しく発現し、プラスミドが胚に正常に注入されることが保証されました。
次に、これらの幼虫を野生型(WT)蚊と交配しました。その後のスクリーニングでは、より強く完全な蛍光発現を示したG1幼虫を選抜して保持し、飼育、系統ストックの拡大、およびさらなる実験を行いました。G1蛍光幼虫は、レポータープロモーターと蛍光タンパク質の組み合わせによって提供される蛍光タンパク質発現のパターンに基づいて同定することができます:プロモーターHr5Ie1-eGFP(図7A)は、全身全体で緑色蛍光タンパク質の発現を促進します。Hr5Ie1-DsRed(図7B)は、体内で赤色蛍光を発現させます。OpIE2-CFP(図7C)は、体の特定の部分のみにシアン(青色)蛍光タンパク質の発現を誘導します。3xP3-TdTomatoの組み合わせを使用すると、赤色蛍光タンパク質が眼に特異的に発現します(図7D)。このトランスジェニック蚊の特定の系統には、全身でCFPの発現を促進するOpIE2プロモーターも含まれています(図7D)。
ノックアウト変異体のためのホモ接合体確立の特別なプロセス
ラインのメンテナンスを簡素化し、すべての蚊が変異体であることを確認するために、非蛍光性ホモ接合体変異蚊を生成するパイプラインを実装しました。プロトコルステップ15で説明したように、WT蚊と交配した後、G6変異蚊を同じケージに入れて交配しました(図9A)。交配後、雌を産卵のために個々のバイアルに集めました(図9B)。このステップにより、異なる遺伝子型を持つ女性を分離することができました。
G7を得るために卵を採取した後、G6雌からゲノムDNAを抽出し、続いてPCRおよび制限酵素で処理したまたは未処理のPCRフラグメントのゲル電気泳動を行った(図9C)。消化されたPCRフラグメントのみを示す、突然変異がないことを示す行は破棄した。一方、突然変異の存在、ひいてはヘテロ接合性の雌の存在を示す二重PCRバンドを示すこれらの系統は保持され、孵化されました(図9D)。その後、G7蚊を成熟させ、すべての成虫を同じケージに入れて、ホモ接合線を確立する可能性を高めるために、別の交配ラウンドを行いました(図9E)。血液を与えられたG7の雌は、G8卵の採集のために再び個別に収集され、3つの潜在的な遺伝子型を表しました(図9F)。ゲノムDNAをG7雌から抽出し、PCRと続いてゲル電気泳動を行い、ホモ接合体を決定しました(図9G)。非変異体またはヘテロ接合性の雌からの卵子は廃棄し、ホモ接合性の結果を持つもののみを保持しました(図9H)。ステップHのG8卵を孵化させ、得られた成体を別のケージで交配しました(図9I)。各ケージから、10人の成人男性を無作為に選択し、ゲル電気泳動およびシーケンシングを行いました(図9J)。このステップにより、ホモ接合体の個体が生まれ、それがストックとして維持されました(図9K)。
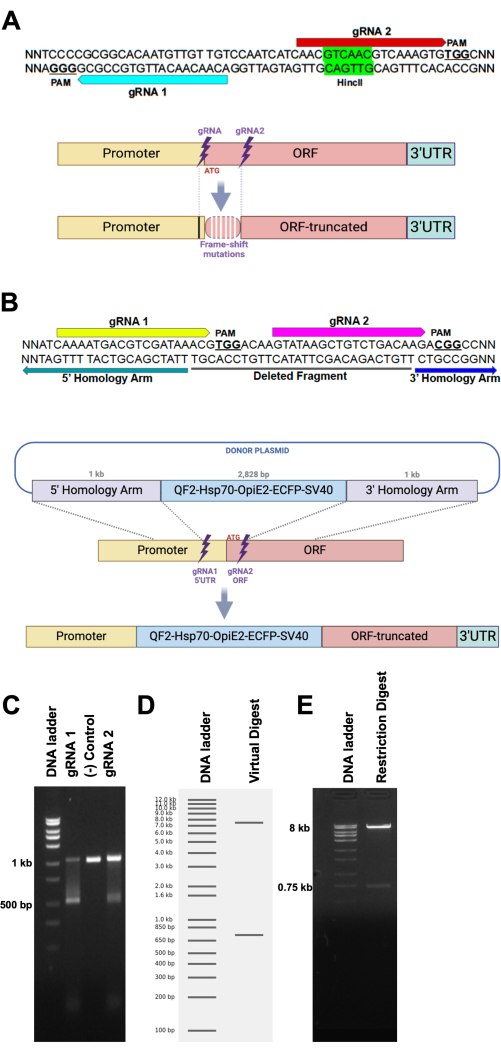
図1:gRNAおよびHDRドナープラスミド設計の概略図(A、上)2つのgRNAの仮想的なDNA配列標的配列(青と赤の矢印とそれらに関連するPAM部位、および制限酵素標的部位(HincII)。(A、下)NHEJを投与された標的部位の模式図で、小さな配列の欠失または挿入によるフレームシフト変異(白い縦棒)の出現につながります。突然変異は、gRNAの切断部位(黒の縦棒)を超えて広がることがあります。(B、上)gRNA 1(黄色の矢印)とgRNA 2(マゼンタの矢印)の標的部位と、続いてPAM部位(NGG、太字、下線部)の標的部位を示す架空のDNA配列。両方のgRNA間で欠落している可能性のあるDNA配列は、黒いバー(欠失フラグメント)で下線が引かれています。緑と青の矢印は、それぞれ5'相同性アームの最も多くの3'配列と3'ホモロジーアームの最も多くの5'配列を強調しています。(B、下)プラスミドからゲノムの相同部位へのDNAカセットのHDR組換えの概略図。(C)体外切断アッセイ。1 kb の DNA フラグメントのインキュベーションは、gRNA 1 (左レーン) または gRNA 2 (右レーン) のいずれかをロードした Cas9 によって消化され、500 bp のフラグメントが得られます。中央のレーンは、gRNAを欠いたネガティブコントロール反応を示しています。(D)プラスミド(8.75 kb)を2つのシングルカット制限酵素で仮想消化し、仮想ゲル上に実質的に2つのバンド(8 kbおよび750 bp)を形成。(E)プラスミドの制限酵素消化反応を充填したアガロースゲルで、仮想消化物によって提供される期待される結果を示しています。この図の拡大版を表示するには、ここをクリックしてください。

図2:マイクロインジェクションの針形状 (A)針先の代表画像。(B)蚊の卵のマイクロインジェクションのための針先の理想的な形状。(C)避けるべき針先形状。 この図の拡大版を表示するには、ここをクリックしてください。

図3:胚の採取 (A)注射用の薄い灰色および(A1)均質ではないが(A2)不均一な胚の収集。スケールバー=500 μm. (B) 整列した胚の画像;スケールバー=500 μm. この図の拡大版を見るには、ここをクリックしてください。
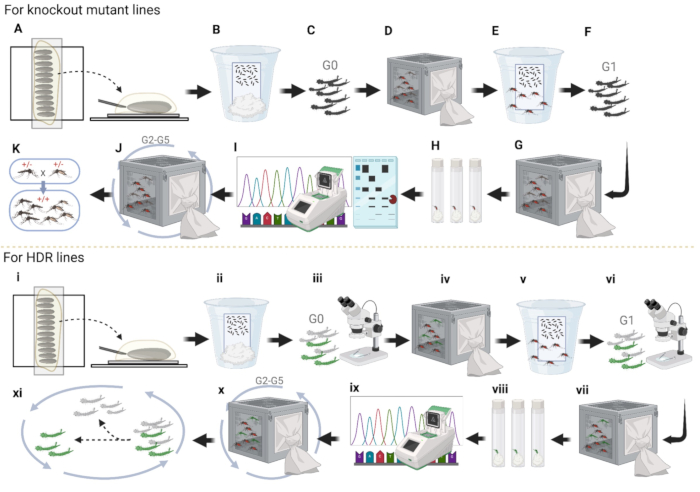
図4:蚊の胚マイクロインジェクションのパイプライン。 (A-K)ノックアウトラインインジェクション。(A)蚊の胚を整列させ、プラスミドを後端に注入します。(B)湿った紙でカップに胚保存を注入し、(C)G0幼虫が孵化し、雌雄に分離します。(D)別々のケージでWTに渡る大人の両性。(E)エッグカップにメスの卵を集める。(F)G1幼虫が孵化し、雌雄に分離します。(G) 別々のケージでWTに渡る成体の両性。(H)G1の雌の卵をプラスチックのバイアルに個別に集め、卵を孵化させます。(I)PCRを行い、酵素処理フラグメントでゲルを走らせ、卵子の孵化に成功したG1単一雌のシーケンシングを行います。(J)G2とWTをさらに3世代にわたって交配し、G1の場合と同じ方法で蚊の配列を決定します。(K)交差によりホモ接合体を確立する。(I-XI)HDRラインインジェクション。(i)胚のアライメント。(ii)湿った紙でカップに胚保存を注入します。(iii)蛍光のためのG0幼虫の事前スクリーニング。(iv)蛍光幼虫を成虫に成長させ、ケージ内でWTと交配します。(v)エッグカップにG1卵を集める。(vi)蛍光G1幼虫のスクリーニングと選択。(vii)G1蛍光成体とWTを交配させた(viii)プラスチックバイアルに個別に卵を産む血液栄養蛍光G1成体雌。(ix)蛍光G1成虫の個々の収集とシーケンシング。(x)さらに3世代にわたって野生型と交配し、(xi)ホモ接合体を確立するために蛍光なしで幼虫を処分する。 この図の拡大版を表示するには、ここをクリックしてください。

図5:胚の注入。 蚊の胚にプラスミドを注入する瞬間。挿入図:胚内の濁った液体として視覚化された注入混合物(黄色の点線)。スケールバー=200 μm. この図の拡大版を見るには、ここをクリックしてください。
図6:胚コレクション。 (A)注入後の胚を採取し、湿った濾紙に移します。スケールバー=7.5 mm. (B) 注入した胚をカップに保持する。スケールバー=25 mm. (C) 注入後の暗い胚;スケールバー=500 μm. この図の拡大版を見るには、ここをクリックしてください。
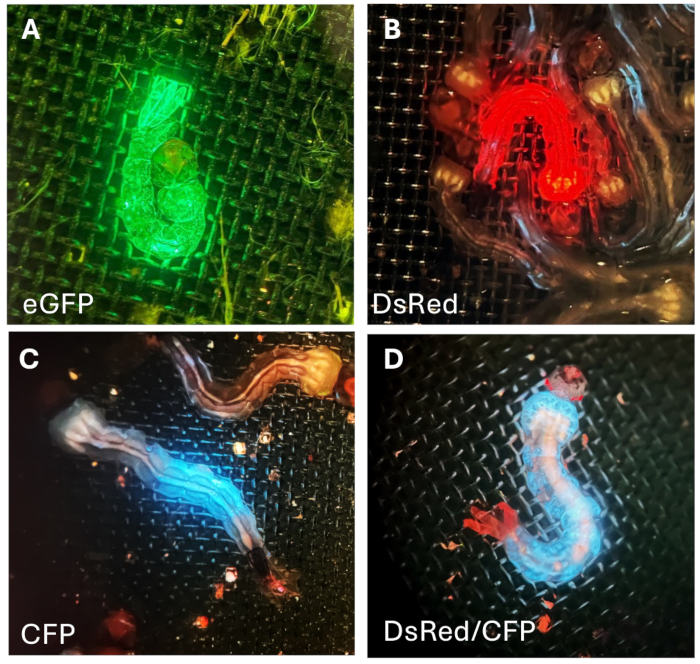
図7:G1幼虫の蛍光。 (A)蚊の幼虫の体上の緑色、(B)赤色、および(C)青色の蛍光タンパク質。(D)眼特有の赤色蛍光と体特異的な青色蛍光を示す幼虫。この図の拡大版を表示するには、ここをクリックしてください。

図8:蛹の形態における性的二型 (A1,A2) 雄の蛹は、体のサイズが小さく、性器葉が目立ち、尖った卵子があり、尾端に幅の広いパドルがあることを特徴としています。(B1、B2)雌の蛹は、体のサイズが大きく、性器葉が小さく、目立たず、尾端のパドルが狭くなっています。 この図の拡大版を表示するには、ここをクリックしてください。
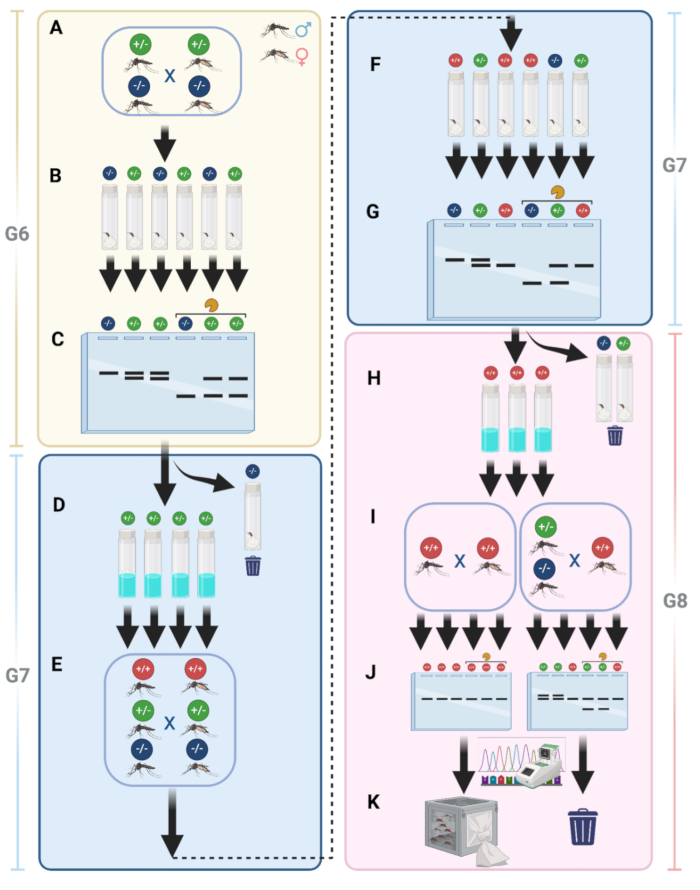
図9:ノックアウト変異株のホモ接合体確立の詳細。 (A)変異蚊は、WTと異種交配した後、同じケージ内で交配されます。(B)交配後、個々の雌を産卵用のバイアルに集めます。(C)G6雌からゲノムDNAを抽出し、続いて酵素処理フラグメントと未処理フラグメントを用いてPCRおよびゲル電気泳動を行います。(D)PCR結果が陽性のG7卵(ヘテロ接合体)が孵化します。(E)G7成体を再び交配して、ホモ接合個体を得る可能性を高める。(F)血液を与えられたG7の雌は、3つの遺伝子型を代表するG8卵子収集のために個別に収集されます。(G)ゲノムDNA抽出、PCR、およびゲル電気泳動をG7女性に対して行い、ホモ接合体個体を同定する。(H)非変異体またはヘテロ接合性の女性からの卵子は廃棄されます。ホモ接合卵は保持されます。(I)G8の卵は孵化し、別のケージで交配されます。(J)各ケージから10匹の成人男性を無作為に選択し、ゲル電気泳動およびシーケンシングを行う。(K)ホモ接合性の蚊は、ストックとして識別され、維持されています。この図の拡大版を表示するには、ここをクリックしてください。
| インジェクションコンストラクト | 最終濃度 |
| Cas9タンパク質 | 100-300 ng /μL |
| gRNAの | 50-100 ng/μL |
| ドナープラスミド | 100-500 ng/μL |
表 1:各注入コンストラクトの推奨最終濃度範囲。
ディスカッション
CRISPR-Cas技術は、染色体1の標的特異的な変化を促進することにより、ゲノム編集の状況を変えました。トランスポーザブルエレメントは、最初のトランスジェニック蚊の生成に不可欠であったにもかかわらず、それらの挿入部位は幾分ランダムであり、カーゴコンストラクト(プロモーター+遺伝子)の発現は、ゲノムの位置効果(すなわち、挿入部位)のために実際の遺伝子の発現プロファイルに対応しない可能性があり、これは通常、異所性発現につながる21.CRISPR-Cas9分子ハサミの出現に先立ち、特定の標的部位でのDNA二本鎖切断を促進する技術が開発され、TALEN22やZinc Finger23などのFokI制限酵素にタグ付けされた特定のタンパク質ドメインのDNA結合特性に依存していました。後者の技術は蚊の遺伝子工学にうまく利用されましたが、そのようなドメインのエンジニアリングには高いコストがかかり、オフターゲット切断の可能性がそのような技術の中止につながりました。それ以来、CRISPR-Cas9技術は、11番の選択肢となる分子ハサミとなりました。
蚊の遺伝子工学は、ほとんどの場合、形質転換混合物を胚に注入することによって行われてきた26,27。蚊の胚マイクロインジェクションを作るには、集中力、忍耐力、そして細部への注意が必要です。産卵後2時間の間に、これらの胚は、その発生を妨げないように、できるだけ注意深く並べ、注入し、洗浄する必要があります。フォーカスは、ラインナップ中に卵子が損傷したり、胚が乾燥したりする可能性を減らすために不可欠なスキルです。針の目詰まり、先端の折れ、注射剤の漏れなど、Cas9タンパク質が混ざっている場合によく起こる予期せぬ事態に直面した場合、忍耐力はこれらの状況を管理するためのもう一つの重要なスキルです。針先が大きすぎたり、注入液が漏れすぎたりした場合に胚を突くと、胚に損傷を与えるだけです。最善の解決策は、針を交換して注射を続けることです。細部へのこだわりも同様に重要です。若い雌の蚊(生後6〜10日)は、年配の蚊よりも多くの卵を産むため、健康な胚を収穫するのに役立ちます。マイクロインジェクション後の胚の保存には、脱イオン水または超純水と茶色(無漂白)のペーパータオルを選択してください。これは、汚染された水や漂白された表面と接触すると、胚の死亡率が大幅に増加する可能性があるためです。薄い灰色(<1時間齢)の胚はより柔らかく、注入手順を容易にし、突然変異株を得る可能性を高めます。さらに、注入された胚は、完全な回復と治癒を可能にするために、孵化する前に少なくとも5日間加湿された濾紙で休む必要があります。
注入液の品質と純度は、胚の生存の基本です。プラスミド抽出キットやgRNA合成キットのバッファー、塩、アルコールは、インジェクションミックスに持ち越さないでください。高価ではありますが、gRNAを注文するオプションにより、RNAが適切にフォールディングされる可能性が高くなる可能性があります。また、組換えバージョンはすでにロードされており、二本鎖切断を行う準備ができていると考えているため、転写および翻訳され、DNAに作用を開始するためにgRNAがロードされるプラスミドソースと比較して、組換えCas9タンパク質をCas9をコードするプラスミド上にリコンビナントCas9タンパク質を注入することをお勧めします。
蚊A. aegyptiのゲノム編集にCRISPR-Cas9技術を使用することについては、私たちの手で最も効果的なプロトコルについて説明しましたが、必要に応じて代替品としても使用できる代替試薬が文献に記載されています。例えば、すべての試薬を懸濁し、注入液を超純水で希釈します。あるいは、注入混合物を5mM KClおよび0.1M NaPO4(pH = 6.8)からなる注入緩衝液で希釈することもできる26,28。胚を並べた後の乾燥から保護するためにハロカーボンオイル700を使用するほか、ハロカーボンオイル2729、および27と70030の混合物も同様の保護を提供します。上述の注入緩衝液の10倍濃縮バージョンは、胚の乾燥を防止するために代替的に使用されてきた26。上述したように、Cas9をコードするプラスミドは市販されており(addgene)、蚊におけるCRISPR−Cas9媒介ゲノム編集に用いられてきた31。
蚊のトランスジェニック系統を得るための戦略の中には、NHEJを介したノックアウトアプローチとHDRを介したノックインアプローチがあります。前者の成功率は後者の32,33よりもはるかに高いが、図4に示すように、交差スキームとスクリーニングプロセスはノックインラインよりもノックアウトを得るのにはるかに時間がかかる。複雑な交差スキームを経ずにノックアウトラインを得るための代替アプローチとして、蛍光マーカーをコードする遺伝子カセットのHDR媒介ノックインは、遺伝子のコード配列を破壊し、蛍光マーカー14,34を用いて蚊のトランスジェニックラインを選択する可能性を提供する。
このプロトコルは、蚊の遺伝子形質転換の基本的なステップにのみ焦点を当てていますが、HDR用のドナープラスミドの設計は、検討が必要な重要なステップです。3xP3、OpIE2、Hr5-IE1、Pub 6,35,36,37,38などのプロモーターを使用することが重要です。3xP3は、昆虫の神経系や眼の発現を促進するために一般的に使用される合成プロモーターです。OpIE2およびHr5-IE1は、昆虫細胞で強力な発現を促進するために使用されるバキュロウイルス由来のプロモーターです。パブのプロモーターは、全身の表現を促進します。U6プロモーターはgRNAの発現に使用されます。dsRed(およびTdTomato)、eGFP、CFP、YFPなどの蛍光マーカーも、HDRのドナープラスミドに使用されます。これらは、それぞれ赤色、緑色、シアン、黄色の蛍光を発する蛍光タンパク質です。3' UTRの場合、p1039およびSV4040は、導入遺伝子のmRNAの安定性と翻訳効率を高めるためによく使用されます。
マイクロインジェクション手順の要約として、ノックアウト変異株については、まず、蚊の胚を両面テープ上に並べて注射します(図4A)。次に、胚の生存率を促進するためにハロカーボンオイルの大部分を取り除き、両面テープからきれいな湿った濾紙に胚を移します。超純水を入れたカップに入れて数日間高水分を維持し、胚が完全に発達するのを待ちます(図4B)。第三に、注入した胚を孵化させ、蛹を両性に分けて、それらを異なるケージに入れます(図4C)。第四に、すべての蛍光オスを1つのケージに入れて、生後5〜7日のWTメスと交配し、 その逆も同様です (図4D)。第五に、交配したWTの雌からG1の卵を集めて、最初に蚊の数を増やします(図4E)。第六に、G1卵を孵化させ、蛹を両性に分けます(図4F)。第七に、G1成虫は再びWTと交配します(図4G)。第八に、G1成体雌をバイアルに集めて個々のG2卵を採取し、綿球またはペーパータオルを使用してバイアルの底に超純水を数日間維持します(図4H)。第九に、G1雌が産卵した後にG1雌を採取し、G1雌からゲノムDNAを抽出してPCRとシーケンシングを行います(図4I)。第10に、ラインキーピングのために正しいG2卵を孵化させ、さらに3世代にわたってWTと交配して、ほとんどの非連結背景変異を除去する(図4J)。第11に、PCR、制限酵素アッセイ、ゲル電気泳動、および突然変異をスクリーニングするためのシーケンシングを使用して、ホモ接合性の蚊の系統を確立します(図4K)。
HDRラインを確立するには、蚊の胚を両面テープに並べて胚マイクロインジェクションを行います(図4i)。次に、注射後、ハロカーボンオイルの大部分を取り除き、超純水を入れたカップに胚を保管して数日間水分を維持し、胚が成熟するのを待ちます(図4ii)。第三に、ハッチに胚を注入し、G0幼虫をスクリーニングして、蛍光の有無にかかわらず胚を選別します(図4iii)。蛍光を発する幼虫は、プラスミドが蚊の胚に首尾よく注入されたことを意味します。第四に、蛍光幼虫を成虫に成熟させ、すべての蛍光オスを1つのケージに入れてWTメスと交配させ、 その逆も同様 にして蚊の数を増やします(図4iv)。第五に、交配したWTの雌から卵を収集します(図4v)。第六に、G1蛍光幼虫を孵化、選別、選別し、蛍光なしで幼虫を処分します(図4vi)。第七に、G1蛍光成体をWTと交差させる(図4vii)。第八に、G2卵をプラスチックバイアルに個々に集めて、蚊のプラスミドの異なる挿入部位を分離します(図4viii)。第九に、卵を孵化させ、蚊の健康状態をチェックし、シーケンシングを実行します(図4ix)。第10に、表現型が強い2系統を選択し、さらに3世代にわたってWTと交配します(図4x)。第11に、蛍光を持つ幼虫を選択してさらに2世代交配し、さらなる実験のためのホモ接合系統を確立します(図4xi)。
ここでは、複数のトランスジェニック株を作製するために使用したCRISPR-Cas9による A. aegypti ゲノム編集の最も包括的なプロトコルを提供しました。このプロトコルは、他の研究者の出発点として使用することをお勧めします。彼らは、マイクロインジェクション試験で得られた経験に基づいて変更を加える必要があります。常に異なる試薬と条件をテストする必要があります。蚊の遺伝子編集の成功率はまだ完全に最適化されておらず、CRISPR-Cas9を介してトランスジェニック蚊を作製できた研究室はほとんどありません。
開示事項
O.S.A.は、Agragene, Inc.およびSynvect, Inc.の株式持分を持つ創設者です。この取り決めの条件は、カリフォルニア大学サンディエゴ校の利益相反ポリシーに従って見直され、承認されています。残りの著者は、競合する利益を宣言しません。
謝辞
著者は、蚊の飼育を支援してくれたジュディ・イシカワとエヴァ・スティーブンソンに感謝します。この作業は、O.S.A.に授与されたNIH賞(R01AI151004、RO1AI148300、RO1AI175152)からの資金提供によって支援されました。K22AI166268からN.H.R.フィギュアはBioRenderを使用して作成されました。
資料
| Name | Company | Catalog Number | Comments |
| 10x Cas9 reaction buffer | PNA Bio | CB01 | |
| Benchling software | Benchling | N/A | www.benchling.com |
| Cas9 dilution buffer | PNA Bio | CB03 | |
| Cas9 protein | PNA Bio | CP01-50 | |
| DH5α E. coli Competent Cells | New England Biolabs | C2987 | |
| Double-sided sticky tape | Scotch Permanent | 3136 | |
| Drosophila vials | Genesee Scientific | 32-109 | |
| Filter papers | GE Healthcare Life Science | 1450-042 | |
| Fish food | Tetra | B00025Z6YI | goldfish flakes |
| Flugs | Genesee Scientific | AS273 | |
| Fluorescent microscope | Leica Microsystems | M165 FC | |
| Gene fragment | Integrated DNA Technologies | N/A | |
| gRNA | Synthego | N/A | |
| Halocarbon oil 700 | Sigma-Aldrich | H8898 | |
| Injection microscope | Leica Microsystems | DM2000 | |
| JM109 E. coli Competent Cells | Zymo Research | T3005 | |
| Microinjector | Eppendorf | FemtoJet 4x | |
| Microloader Tips for Filling Femtotips | Eppendorf | E5242956003 | |
| Micromanipulator | Eppendorf | TransferMan 4r | |
| Micropipette Pullers | Sutter Instrument | P-2000 | |
| Microscope Cover Glass | Fisherbrand | 12-542-B | |
| Microscope slide | Eisco | 12-550-A3 | |
| Mouse blood (live mice used for feeding) | University of California | IACUC, Animal Use Protocol #S17187 | Used for mosquito blood feeding; details comply with animal ethics protocols |
| NEB Q5 High-Fidelity DNA polymerase | New England Biolabs | M0491S | |
| PCR Purification Kit | Qiagen | 28004 | |
| Plasmid Miniprep Kit | Zymo Research | D4036 | |
| Quartz filament | Sutter Instruments | QF100-70-10 | |
| Transcription Clean-Up Kit | Fisher Scientific | AM1908 | |
| Ultra-pure water | Life Technologies | 10977-023 |
参考文献
- Anzalone, A. V., Koblan, L. W., Liu, D. R. Genome editing with CRISPR-Cas nucleases, base editors, transposases and prime editors. Nat Biotechnol. 38 (7), 824-844 (2020).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Shen, H., Li, Z. DNA double-strand break repairs and their application in plant DNA integration. Genes (Basel). 13 (2), 322 (2022).
- Vinauger, C., et al. Modulation of host learning in Aedes aegypti mosquitoes. Curr Biol. 28 (3), 333-344.e8 (2018).
- Li, M., Bui, M., Yang, T., Bowman, C. S., White, B. J., Akbari, O. S. Germline Cas9 expression yields highly efficient genome engineering in a major worldwide disease vector. Proc Natl Acad Sci U S A. 114 (49), E10540-E10549 (2017).
- Chaverra-Rodriguez, D., et al. Targeted delivery of CRISPR-Cas9 ribonucleoprotein into arthropod ovaries for heritable germline gene editing. Nat Commun. 9 (1), 3008 (2018).
- Rouyar, A., et al. Transgenic line for characterizing GABA-receptor expression to study the neural basis of olfaction in the yellow-fever mosquito. Front Physiol. 15, 1381164 (2024).
- Ang, J. X. D., et al. Considerations for homology-based DNA repair in mosquitoes: Impact of sequence heterology and donor template source. PLoS Genet. 18 (2), e1010060 (2022).
- Zhang, J. -. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biol. 18 (1), 35 (2017).
- Coutinho-Abreu, I. V., Akbari, O. S. Technological advances in mosquito olfaction neurogenetics. Trends Genet. 39 (2), 154-166 (2023).
- Li, M., et al. Targeting sex determination to suppress mosquito populations. eLife. 12, RP90199 (2024).
- Zhan, Y., Alonso San Alberto, D., Rusch, C., Riffell, J. A., Montell, C. Elimination of vision-guided target attraction in Aedes aegypti using CRISPR. Current Biol. 31 (18), 4180-4187.e6 (2021).
- Greppi, C., et al. Mosquito heat seeking is driven by an ancestral cooling receptor. Science. 367 (6478), 681-684 (2020).
- Potter, C. J., Tasic, B., Russler, E. V., Liang, L., Luo, L. The Q system: a repressible binary system for transgene expression, lineage tracing, and mosaic analysis. Cell. 141 (3), 536-548 (2010).
- Riabinina, O., et al. Improved and expanded Q-system reagents for genetic manipulations. Nat Methods. 12 (3), 219-222 (2015).
- Labun, K., et al. CHOPCHOP v3: expanding the CRISPR web toolbox beyond genome editing. Nucleic Acids Res. 47 (W1), W171-W174 (2019).
- Concordet, J. -. P., Haeussler, M. CRISPOR: intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Res. 46 (W1), W242-W245 (2018).
- Williams, A. E., et al. Quantifying fitness costs in transgenic Aedes aegypti mosquitoes. J Vis Exp. , e65136 (2023).
- Bassett, A. R., Tibbit, C., Ponting, C. P., Liu, J. -. L. Highly efficient targeted mutagenesis of Drosophila with the CRISPR/Cas9 system. Cell Rep. 4 (1), 220-228 (2013).
- Coutinho-Abreu, I. V., Zhu, K. Y., Ramalho-Ortigao, M. Transgenesis and paratransgenesis to control insect-borne diseases: current status and future challenges. Parasitol Int. 59 (1), 1-8 (2010).
- Christian, M., et al. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics. 186 (2), 757-761 (2010).
- Kim, Y. G., Cha, J., Chandrasegaran, S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain. Proc Natl Acad Sci U S A. 93 (3), 1156-1160 (1996).
- DeGennaro, M., et al. orco mutant mosquitoes lose strong preference for humans and are not repelled by volatile DEET. Nature. 498 (7455), 487-491 (2013).
- McMeniman, C. J., Corfas, R. A., Matthews, B. J., Ritchie, S. A., Vosshall, L. B. Multimodal integration of carbon dioxide and other sensory cues drives mosquito attraction to humans. Cell. 156 (5), 1060-1071 (2014).
- Lobo, N. F., Clayton, J. R., Fraser, M. J., Kafatos, F. C., Collins, F. H. High efficiency germ-line transformation of mosquitoes. Nat Protoc. 1 (3), 1312-1317 (2006).
- Kistler, K. E., Vosshall, L. B., Matthews, B. J. Genome engineering with CRISPR-Cas9 in the mosquito Aedes aegypti. Cell Rep. 11 (1), 51-60 (2015).
- Handler, A. M., Harrell, R. A. 2nd Transformation of the Caribbean fruit fly, Anastrephasuspensa, with a piggyBac vector marked with polyubiquitin-regulated GFP. Insect Biochem Mol Biol. 31 (2), 199-205 (2001).
- Harrell, R. A. . 2nd Mosquito embryo microinjection under halocarbon oil or in aqueous solution. 2024 (7), (2024).
- Sun, R., Raban, R., Akbari, O. S. Generating mutant strains with transgenic Cas9. Cold Spring Harb Protoc. 2023 (9), 671-678 (2023).
- Giraldo, D., et al. An expanded neurogenetic toolkit to decode olfaction in the African malaria mosquito Anopheles gambiae. Cell Rep Methods. 4 (2), 100714 (2024).
- Liu, G., Lin, Q., Jin, S., Gao, C. The CRISPR-Cas toolbox and gene editing technologies. Mol Cell. 82 (2), 333-347 (2022).
- Chu, V. T., et al. Increasing the efficiency of homology-directed repair for CRISPR-Cas9-induced precise gene editing in mammalian cells. Nat Biotechnol. 33 (5), 543-548 (2015).
- Laursen, W. J., et al. Humidity sensors that alert mosquitoes to nearby hosts and egg-laying sites. Neuron. 111 (6), 874-887.e8 (2023).
- Weng, S. -. C., Antoshechkin, I., Marois, E., Akbari, O. S. Efficient sex separation by exploiting differential alternative splicing of a dominant marker in Aedes aegypti. PLoS Genet. 19 (11), e1011065 (2023).
- Li, M., et al. Development of a confinable gene drive system in the human disease vector. eLife. 9, e51701 (2020).
- Dalla Benetta, E., et al. Engineered Antiviral Sensor Targets Infected Mosquitoes. The CRISPR journal. 6 (6), 543-556 (2023).
- Li, H. -. H., et al. C-Type lectins link immunological and reproductive processes in Aedes aegypti. iScience. 23 (9), 101486 (2020).
- van Oers, M. M., Vlak, J. M., Voorma, H. O., Thomas, A. A. M. Role of the 3' untranslated region of baculovirus p10 mRNA in high-level expression of foreign genes. J Gen Virol. 80 (Pt 8), 2253-2262 (1999).
- Salem, T. Z., et al. The influence of SV40 polyA on gene expression of baculovirus expression vector systems. PloS One. 10 (12), e0145019 (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved