Method Article
Édition du génome du moustique de la fièvre jaune Aedes aegypti à l’aide de CRISPR-Cas9
Dans cet article
Résumé
Nous décrivons ici un protocole détaillé d’édition du génome par micro-injection embryonnaire chez le moustique A. aegypti à l’aide de la technologie CRISPR-Cas9.
Résumé
L’émergence de la technologie CRISPR-Cas9 a révolutionné le domaine du génie génétique et ouvert les portes à l’édition précise du génome chez de multiples espèces, y compris des organismes non modèles. Chez le moustique Aedes aegypti, des mutations de perte de fonction et des insertions d’ADN ont été réalisées avec cette technologie. Ici, nous décrivons un protocole détaillé pour l’édition du génome par micro-injection embryonnaire chez le moustique A. aegypti à l’aide de la technologie CRISPR-Cas9, en se concentrant à la fois sur la génération de lignées de knock-out et de knockin. Dans ce protocole, les aiguilles de quartz sont remplies d’un mélange d’ARN guide, de Cas9 recombinant et d’un plasmide contenant une cassette d’ADN codant pour un gène pour un marqueur fluorescent, si l’on souhaite une knock-in génique. Les embryons au stade préblastoderme sont alignés sur une bande de ruban adhésif double face placée sur une lamelle, qui est ensuite montée sur une lame de verre. À l’aide d’un micro-injecteur, les aiguilles sont insérées doucement dans l’extrémité postérieure des embryons et un petit volume du mélange CRISPR est distribué. Lorsque les embryons éclosent, les larves sont vérifiées sous la lunette fluorescente, et les pupes sont triées par sexe et séparées dans différentes cages. Une fois que les adultes émergent, ceux-ci sont croisés réciproquement avec des individus de type sauvage, nourris au sang et placés pour la ponte. Une fois ces œufs éclos, les larves fluorescentes collectées représentent des individus avec une insertion stable de la cassette d’ADN dans leur génome. Ces larves sont ensuite cultivées jusqu’au stade adulte, croisées avec des individus de type sauvage, puis évaluées davantage à l’aide de techniques moléculaires pour confirmer que la séquence exacte de la cassette d’ADN est présente au site souhaité du génome du moustique. Des lignées homozygotes peuvent également être obtenues en suivant le pipeline fourni de schéma de croisement et de criblage moléculaire des mutations.
Introduction
L’édition précise du génome est sans doute devenue plus facile, mais possible, avec la mise en place des technologies CRISPR-Cas des ciseaux moléculaires1. Ces technologies tirent parti d’un mécanisme que le système immunitaire procaryote utilise pour lutter contre les phagues2. Parmi ces systèmes, les répétitions palindromiques courtes (CRISPR) groupées, régulièrement intercalées et la nucléase Cas9 reposent généralement sur 20 ARN à paires de bases, les ARN guides (ARNg), avec des séquences homologues à l’ADN ciblé, qui sont suivies d’un motif adjacent au protospacer NGG (PAM) séquence3. Les ARNg chargés sur le Cas9 guident précisément ces nucléases vers des sites cibles spécifiques du génome, déclenchant des cassures de l’ADN double brin3.
Les cassures double brin de l’ADN induisent les mécanismes de réparation pour patcher la double hélice4. Alors que toute réparation de l’ADN est censée être précise, il existe des mécanismes de réparation de l’ADN moins précis qui peuvent laisser des cicatrices de séquence et, à leur tour, des mutations de perte de fonction4. Parmi les mécanismes de réparation de l’ADN sujets aux erreurs, le NHEJ (Non-Homologous End-Joindre) peut provoquer des mutations par décalage de trame, y compris de petites délétions et insertions et des modifications nucléotidiques (SNP), qui peuvent entraîner une mutation de perte de fonction. Le mécanisme de réparation dirigée par homologie (HDR), quant à lui, s’appuie sur le chromosome homologue comme modèle pour copier la séquence exacte de l’allèle non endommagé et effectuer une réparation parfaite de la séquence d’ADN ciblée4.
Sur la base de ces connaissances, la technologie CRISPR-Cas9 a été développée pour modifier précisément les génomes, sans doute au niveau de n’importe quelle séquence contenant un site PAM3. Chez les moustiques, la technologie CRISPR-Cas9 a été utilisée pour éliminer une variété de gènes, par micro-injection embryonnaire d’un mélange de Cas9 et d’ARNg, en profitant du mécanisme de réparation NHEJ 5,6. Une mutagenèse germinale similaire est obtenue avec l’injection d’un mélange ARNg + Cas9 dans l’hémolymphe de moustiques femelles adultes7. Cette technologie a été inventée sous le nom de contrôle ReMOT et repose sur une version modifiée d’un Cas9 marqué avec un peptide qui est absorbé par les ovaires par endocytose pendant le processus de développement de l’ovule (vitellogenèse)7. L’insertion de cassettes de gènes spécifiques dans un génome n’est possible que par micro-injection embryonnaire d’un mélange d’ARNg et de Cas9 (ou d’un plasmide exprimant ces molécules) ainsi que d’un plasmide codant pour une cassette d’ADNsouhaitée 8. Tirant parti du mécanisme HDR, le plasmide contenant la cassette d’ADN d’intérêt flanquée de séquences homologues (500-1 000 bp)9,10 en amont et en aval du site cible est utilisé comme modèle pour réécrire la cassure double brin, en copiant également la cassette d’ADN dans la séquence cible9.
La technologie CRISPR-Cas9 a été utilisée pour éliminer plusieurs gènes principalement impliqués dans les systèmes sensoriels du moustique Aedes aegypti11, mais aussi des gènes associés à la fertilité masculine et à la viabilité féminine (PgSIT) pour le contrôle de la population12. L’inactivation des gènes cibles a également été accomplie en introduisant des gènes codant pour des marqueurs fluorescents dans les séquences codantes de gènes spécifiques13,14. Cette stratégie a l’avantage non seulement d’induire des mutations de décalage de cadre mais aussi de permettre l’utilisation de la lumière fluorescente pour trier les individus de la nouvelle lignée knock-out13,14. Le génome d’A. aegypti a également été modifié avec des séquences de systèmes d’expression binaires, tels que le système Q (QF-QUAS)11. L’insertion du gène codant pour le transactivateur QF en aval vers un promoteur d’un gène spécifique assure une expression spatio-temporelle définie du transactivateur 15,16. Une fois qu’une lignée de moustique exprimant QF est croisée avec une autre lignée de moustiques contenant les sites de liaison (QUAS) pour QF, ce dernier se lie à celle-ci et déclenche l’expression des gènes en aval de la séquence QUAS15,16. Dans l’ensemble, ce système permet l’expression tissulaire et temporelle de ces gènes effecteurs, qui peuvent être des marqueurs fluorescents utilisés pour la localisation cellulaire ou la détection de l’activité neuronale, et même des nucléases Cas9 pour perturber les gènes dans des tissus spécifiques (c’est-à-dire l’inactivation somatique)11.
Compte tenu de toutes les informations disponibles pour la transformation génétique d’A. aegypti , nous fournissons ici un protocole détaillé avec des instructions étape par étape pour effectuer l’édition du génome avec le système CRISPR-Cas9 par micro-injection embryonnaire. Les stratégies pour générer à la fois l’inactivation, par le biais de mutations et de délétions par décalage de cadre médiées par NHEJ, et les lignes d’knockin, par l’insertion de cassettes de gènes médiées par HDR, sont discutées.
Protocole
Les détails relatifs à l’équipement et aux réactifs utilisés dans ce protocole sont énumérés dans la table des matières. Tous les animaux ont été manipulés conformément au Guide pour le soin et l’utilisation des animaux de laboratoire, tel que recommandé par les National Institutes of Health. Les procédures ont été approuvées par le Comité institutionnel de l’UCSD sur le soin et l’utilisation des animaux (IACUC, Animal Use Protocol #S17187) et l’Autorisation d’utilisation biologique de l’UCSD (BUA #R2401).
1. Conception des ARNg et du plasmide donneur
- Pour fabriquer des mutants knock-out, concevez deux ARNg espacés de ~20 à 100 pb (Figure 1A).
- Concevez des ARNg de 20 pb pour CRISPR-Cas9, à l’exclusion de la séquence PAM (NGG) à l’aide d’un outil en ligne (Figure 1A), tel que CHOPCHOP ( https://chopchop.cbu.uib.no/)17ou Benchling (benchling.com) ou CRISPOR (http://crispor.gi.ucsc.edu/)18. Sélectionnez les ARNg les plus spécifiques et sans cible pour les expériences, comme suggéré par l’outil en ligne.
- Assurez-vous qu’un site de coupe d’enzyme de restriction efficace est inclus dans la séquence entre les sites de coupe de l’ARNg (Figure 1A). Inclure un site d’enzyme de restriction entre les sites de coupe de l’ARNg pour permettre une confirmation visuelle rapide des modifications réussies.
REMARQUE : Lorsque la délétion se produit, le site de restriction est supprimé, de sorte que l’enzyme ne coupera pas la séquence, produisant une seule bande non coupée sur un gel pour confirmer la délétion.
- Pour les insertions de cassettes de gènes médiées par HDR, concevez plusieurs ARNg à l’aide des outils mentionnés pour la fabrication de mutants knock-out et sélectionnez le plus efficace après une évaluation ultérieure.
- Concevez des plasmides donneurs pour la réparation dirigée par homologie (HDR) contenant des bras d’homologie de site cible, la séquence d’ADN cargo, un marqueur fluorescent, l’origine de la réplication et un gène de résistance aux antibiotiques (Figure 1B).
- Choisissez les bras d’homologie dans les régions en amont et en aval du site cible, chacun couvrant 500-1 000 pb10 (Figure 1B).
- Sélectionnez la séquence cargo, qui peut inclure un marqueur fluorescent, un gène d’intérêt ou un élément régulateur (p. ex., QF2).
- Sélectionnez un marqueur fluorescent. Les marqueurs fluorescents courants utilisés pour A. aegypti sont inclus dans une cassette d’ADN contenant un promoteur, un gène codant pour un marqueur fluorescent et une séquence UTR 3'. Voir la discussion pour plus de détails.
2. Préparation du mélange d’injection
- Diluez la protéine Cas9 avec le tampon de dilution Cas9 à 1 μg/μL.
REMARQUE : Ne décongelez pas et ne congelez pas Cas9 plus de 2 fois. Il est conseillé de faire des aliquotes de la protéine Cas9. - Achetez les ARNg ou produisez-les en interne.
REMARQUE : Pour la production en interne, utilisez des pratiques standard de décontamination de l’ARN et des matériaux sans ARNse.- Concevez 4 à 6 ARNg et choisissez les deux meilleurs ARNg pour l’injection (voir le test de clivage in vitro ci-dessous).
- Concevez une amorce directe qui inclut la séquence du promoteur T7 en amont de la séquence de l’ARNg. Utilisez l’amorce inverse universelle de l’ARNg pour les réactions PCR non matricielles, comme décrit ci-dessous. La base de séquence qui se chevauche s’apparie à la séquence correspondante dans l’amorce inverse universelle (en gras et soulignée), créant ainsi un modèle pour l’ADN polymérase afin d’amplifier leurs séquences.
REMARQUE : Pour ces réactions de PCR, l’amorce directe doit contenir le promoteur T7 (en gras), suivi de deux guanines (importantes pour l’initiation de la transcription via l’ARN polymérase T7), de la séquence à 20 nucléotides de l’ARNg (N20 ; sans la séquence PAM) et de la séquence de chevauchement de l’amorce (soulignée).
Amorce vers l’avant : 5'- GAAATTAATACGACTCACTATAGGN20 GTTTTAGAGCTAGAAATAGC- 3'
Amorce revers : 5''-AAAAGCACCGACTCGGTGCCACTTTTT CAAGTTGATAACGGACTAGC
CTTATT TTAACTT GCTATTTCTAGCTCTAAAAC - 3' - Synthétisez le modèle d’ADN par PCR non modèle. Mettez en place plusieurs réactions (chacune contenant 12,5 μL du mélange maître 2x, 1,25 μL de la solution 10 μM d’amorce directe, 1,25 μL de la solution 10 μM d’amorce inverse et 10 μL d’eau ultrapure) de sorte qu’il y ait suffisamment de produit PCR (300 ng) disponible pour la réaction de transcription in vitro . Utiliser les conditions de PCR suivantes : dénaturation initiale pendant 30 s à 98 °C ; 35 cycles d’amplification pendant 10 s à 98 °C, 10 s à 62 °C et 10 s à 72 °C ; extension finale à 72 °C pendant 2 min ; et stockage à 4 °C.
- Confirmer l’amplification d’un seul fragment d’ADN (122 paires de bases) sur un gel d’agarose (2 %).
- Nettoyez le modèle PCR à l’aide d’un kit de purification PCR, en suivant les recommandations du fabricant.
- Effectuez une réaction de transcription in vitro avec un kit de transcription T7. Mélangez 2 μL de tampon réactionnel 10x, 2 μL de nucléotides (ATP, CTP, UTP et GTP), 2 μL d’ARN polymérase T7, 3 μL d’ADN matrice (100 ng/μL) et 5 μL d’eau ultrapure. Incuber le mélange à 37 °C pendant au moins 2 h (pas plus de 16 h) jusqu’à toute la nuit (12 h).
REMARQUE : Dans cette réaction, l’ARN polymérase T7 se lie au promoteur T7 inclus dans la séquence directe de l’amorce, ce qui conduit à la transcription de l’ARNg. - Traitez la réaction de transcription avec la DNase en ajoutant 1 μL de DNase (bien mélanger) et en incubant à 37 °C pendant 15 min.
- Purifiez les ARNsg synthétisés à l’aide d’un kit de nettoyage de transcription en suivant les recommandations du fabricant.
- Effectuer un test de clivage in vitro pour évaluer l’efficacité de coupe des ARNg sélectionnés (Figure 1C).
- Concevez des amorces pour amplifier un fragment d’ADN (500-1 000 pb) flanquant le site de coupe de l’ARNg.
- Configurez les réactions de PCR comme suit : 12,5 μL du mélange maître 2x, 1,25 μL de la solution 10 μM d’amorce directe, 1,25 μL de la solution 10 μM d’amorce inverse, 9 μL d’eau ultrapure et 1 μL d’ADN matrice 5 ng/μL.
- Utiliser les conditions de PCR suivantes : conditions : dénaturation initiale pendant 30 s à 98 °C ; 35 cycles d’amplification pendant 10 s à 98 °C, 15-30 s à X °C (température de recuit idéale pour les amorces) et 35 s à 72 °C ; extension finale à 72 °C pendant 2 min ; et stockage final à long terme à 4 °C.
- Reportez-vous aux directives du fabricant pour connaître les meilleures températures d’amplification et la durée de l’amplification. Regrouper au moins cinq réactions ou augmenter les volumes des réactifs de manière à obtenir suffisamment de produit PCR (1,5 à 2 μg) après le nettoyage par PCR (bande unique) ou la purification en bande PCR sur gel d’agarose.
- Établissez des réactions de clivage de Cas9 avec du Cas9 recombinant en incubant le mélange suivant à 37 °C pendant 1 h : 1 μL de tampon réactionnel 10x Cas9, 0,35 μL de Cas9 recombinant (1 μg/μL), 1 μL d’ARNg (100 ng/μL), 6,65 μL d’eau ultrapure et 1 μL de 300 ng/μL d’ADN matrice.
- Mettez en place une réaction de contrôle négative sans aucun ARNg.
- Vérifiez l’efficacité du clivage en exécutant des réactions sur un gel d’agarose (1,5-2,0 % ; Figure 1C).
3. Assemblage du plasmide donneur
- PCR-amplifier les bras d’homologie à partir de l’ADN génomique d’A. aegypti.
- Pour la conception de l’amorce, reportez-vous aux instructions du fabricant du kit de clonage. Utilisez un outil en ligne (p. ex., Benchling), qui fournit un soutien pour la conception et l’assemblage de plasmides à l’aide de différentes stratégies de clonage (p. ex., Gibson, Golden Gate et clonage basé sur des enzymes de restriction).
- Amplifiez la séquence cargo de l’ADN génomique d’A. aegypti ou commandez un fragment de gène synthétisé commercialement.
- Amplifiez les marqueurs fluorescents des plasmides en interne.
- Comparez les fragments d’ADN des étapes 3.1.1 à 3.1.3 dans le squelette d’un plasmide existant par assemblage Gibson. Incuber un mélange de 10 μL de 2x master mix, X μL de tous les fragments d’ADN, 10-X μL d’eau ultrapure à 50 °C pendant 1 h.
REMARQUE : Reportez-vous aux directives du fabricant pour calculer le rapport idéal entre les inserts et le squelette plasmidique.- Calculez les concentrations de chaque fragment de clonage en pico mols : pmols = (poids en ng) × 1 000/(paires de bases × 650 daltons).
- Utilisez 50 à 100 ng de squelette plasmidique et 2 à 3 fois l’excès molaire de chaque insert.
- Si vous assemblez 2 à 3 fragments, ajoutez 0,02 à 0,5 mol de chaque fragment dans la réaction de Gibson. Si vous assemblez 4 à 6 fragments, ajoutez 0,2 à 1,0 pmols de chacun.
- Utilisez 3 à 5 μL de la réaction d’assemblage de Gibson pour transformer les cellules compétentes d’E. coli JM109.
REMARQUE : JM109 a été choisi pour l’assemblage Gibson en raison de son génotype recA- , qui réduit les événements de recombinaison indésirables et empêche le transfert de nucléases pendant la récolte cellulaire, garantissant ainsi l’intégrité des fragments d’ADN assemblés
Pour les plasmides de plus de 10 kb, nous recommandons la transformation des cellules compétentes DH5α à l’aide du protocole étendu, en suivant les recommandations du fabricant. - Développez les bactéries transformées et purifiez le plasmide à l’aide d’un kit miniprep.
- Pour confirmer l’assemblage correct du plasmide, exécutez une digestion d’enzyme de restriction diagnostique avec l’ADN plasmidique purifié et visualisez par électrophorèse sur gel d’agarose (Figure 1D,E).
REMARQUE : Nous suggérons l’utilisation d’un outil en ligne pour la sélection de quelques enzymes de restriction qui peuvent couper le plasmide sur un seul site. Un logiciel comme Benchling dispose d’un outil intégré qui effectue la digestion virtuelle des séquences plasmidiques avec les enzymes de restriction sélectionnées, en affichant le motif de bande d’ADN attendu sur un gel d’électrophorèse virtuel (Figure 1D).- Effectuer la digestion de l’enzyme de restriction plasmidique. Incuber un mélange de 1 μL de tampon de digestion de restriction 10x, 0,5 μL d’enzyme de restriction 1, 0,5 μL d’enzyme de restriction 2, 1 μL d’ADN plasmidique (300 ng/mL) et 7 μL d’eau ultrapure à 37 °C pendant 2 h.
- Exécutez les réactions de digestion de restriction sur un gel d’agarose (1,5 %).
REMARQUE : Le motif de bande d’ADN (Figure 1E) doit ressembler au motif de bande de digestion virtuelle (Figure 1D). Les plasmides peuvent également être envoyés pour le séquençage de plasmides entiers si le service est disponible.
- Cultivez le clone plasmidique dans 150 mL de milieu LB.
- Effectuez le plasmide maxiprep, en suivant les directives du fabricant.
- Mettez le plasmide en suspension dans de l’eau ultrapure.
4. Mélange de la construction d’injection
REMARQUE : Les plages de concentration finale suggérées pour chaque construction sont fournies dans le tableau 1. Commencez par un rapport de 2:1:2 (ng Cas9 : ng sgRNA : ng plasmide donneur) et ajustez si nécessaire pour optimiser l’efficacité du HDR. Le suivi des résultats aidera à déterminer la combinaison la plus efficace.
- Pour fabriquer des mutants knock-out,
- Diluer l’aliquote de la protéine Cas9 à la concentration souhaitée à l’aide du tampon de dilution Cas9 et diluer l’aliquote de l’ARNg avec de l’eau ultrapure.
- Prémélangez la protéine Cas9 avec chaque ARNg pour former un complexe de ribonucléoprotéines (RNP). Combinez les solutions prémélangées.
- Pour les insertions de cassettes de gènes médiées par HDR,
- Diluez et mélangez la protéine Cas9 et les ARNg comme suggéré pour fabriquer des mutants knock-out.
- Diluez le plasmide donneur avec de l’eau ultra-pure.
- Combinez toutes les constructions.
5. Tirer et charger les aiguilles de micro-injection
- Utilisez du filament de quartz pour tirer les aiguilles d’injection (Figure 2). Assurez-vous que le filament mesure 10 cm de long, avec un diamètre extérieur de 1 mm et un diamètre intérieur de 0,7 mm.
- Tirez les aiguilles à l’aide d’un extracteur de micropipette laser avec l’un des deux programmes suivants :
Programme 1 : Heat 805, Filament 4, Velocity 50, Delay 145, Pull 145
Programme 2 : Heat 650, Filament 4, Velocity 40, Delay 150, Pull 156
REMARQUE : Le programme 1 permet d’obtenir une pointe d’aiguille plus fine mais plus longue, ce qui le rend adapté aux injections avec des constructions à faible concentration, où une pointe plus fine est bénéfique pour la précision. Le programme 2 permet d’obtenir une pointe d’aiguille plus courte mais plus épaisse, idéale pour les injections à plus forte concentration, car la construction est plus épaisse et nécessite une aiguille plus robuste.
6. Prélèvement d’embryons
- Préparez un aspirateur électrique ou manuel pour recueillir les moustiques et utilisez des flacons en plastique pour la collecte et la récolte des embryons.
- Humidifiez les papiers-filtres à cercle blanc et placez-les sur la paroi intérieure ou sur du coton humide dans le collecteur.
- Placez 5 à 10 moustiques femelles qui ont été nourries de sang il y a 5 à 10 jours dans le collecteur. Placez le collecteur dans l’obscurité pendant 45 min.
- Sortez les papiers-filtres pour le prélèvement d’embryons.
7. Alignement des embryons
- Utiliser une souche sauvage (Liverpool) d’A. aegypti pour l’injection d’embryons.
- Sélectionnez les embryons au stade préblastodermique, en particulier ceux de couleur gris clair, sur le papier de récolte (Figure 3).
- Transférez quelques embryons avec une brosse mouillée sur le ruban adhésif double face placé sur une lamelle. Alignez les embryons en parallèle pendant que leur entourage est encore humide, en vous assurant qu’ils sont côte à côte, avec toutes les extrémités postérieures tournées vers l’avant.
- Une fois correctement positionné, laissez l’environnement sécher légèrement pour fixer les embryons en place.
- Ajoutez de l’huile d’halocarbure 700 sur les embryons pendant l’alignement pour éviter la dessiccation.
REMARQUE : Assurez-vous que les embryons ne sont pas entourés d’eau lors de l’ajout de l’huile, car cela ferait flotter les embryons dans l’huile.
8. Micro-injection d’embryons
REMARQUE : L’injection est effectuée à température ambiante ou à 18 °C. La température de 18°C est recommandée pour des raisons pratiques, car elle retarde le développement de l’embryon.
- Mettre en place un micro-injecteur avec les paramètres suivants : pression de compensation (Pc) 300 hPa, pression d’injection (Pi) 500 hPa. Ajustez ces conditions si nécessaire.
- Chargez 3 μL de la construction mixte dans une aiguille à l’aide d’un microchargeur.
- Placez le verre de protection avec les embryons alignés sur une lame de microscope (Figure 3). Positionnez le verre de protection et la lame sous le microscope pour l’injection. Assurez-vous que l’extrémité postérieure de l’embryon est positionnée vers l’aiguille.
- Placez une aiguille dans un porte-aiguille avec un micromanipulateur et positionnez l’aiguille à un angle de 10° vers l’extrémité postérieure des embryons alignés, en la maintenant immobile (Figure 4A). Ouvrez doucement l’aiguille sous un microscope et touchez-la légèrement avec le bord d’une lamelle.
REMARQUE : Une autre option consiste à créer une petite ouverture dans l’aiguille en la tapotant avec un embryon. Cependant, une ouverture de l’aiguille légèrement plus large est recommandée, car l’adhérence de la protéine Cas9 peut obstruer l’aiguille. - Déplacez le verre de la lame vers l’aiguille d’injection (Figure 5).
9. Suivi de l’embryon injecté
- Utilisez des lingettes jetables non pelucheuses pour enlever l’huile entourant les embryons.
- Ajoutez de l’eau déminéralisée pour rincer les embryons et déplacez-les sur un papier filtre humide. Placez le papier filtre humide sur un mouchoir humide à l’intérieur de gobelets de 9 oz de Carat (Figure 4B et Figure 6A,B).
- Placez du coton humide au fond de la tasse pour maintenir l’humidité (Figure 6B).
- Conservez les embryons injectés sur du papier filtre humide.
10. Éclosion d’embryons et dépistage des larves G0
- Au moins 4 jours après l’injection, transférez le papier filtre contenant des embryons dans environ 3 L d’eau déminéralisée dans des casseroles Sterilite de 6 pintes pour l’éclosion.
REMARQUE : Les œufs éclosent généralement plus efficacement dans les 2 semaines suivant l’injection. Assurez-vous que les œufs restent humides pendant cette période. Le papier filtre ne doit pas être trop humide, car une humidité excessive peut entraîner une éclosion précoce des embryons. Bien qu’une conservation plus longue puisse améliorer les taux d’éclosion, ne dépassez pas un mois pour des résultats optimaux. L’utilisation d’eau désoxygénée peut également aider à l’éclosion. - Une fois que les larves G0 éclosent, ajoutez de la nourriture pour poissons mélangée à de l’eau dans la casserole comme nourriture.
- Criblez les larves G0 pour détecter le marqueur fluorescent au stade larvaire du 3e au 4e stade (figure 7).
- Conservez les larves séparément en fonction de leur statut fluorescent, les larves fluorescentes positives et négatives fluorescentes étant conservées dans des bacs séparés.
11. Tri sexuel des pupes et croisement avec le type sauvage
- Séparez les moustiques injectés par sexe lorsqu’ils se nymphosent, en utilisant des structures spécifiques à la taille et au sexe au niveau du lobe génital pour l’identification :
- Mâles : De plus petite taille, un lobe génital plus proéminent et pointu, et des pagaies plus larges (structures à l’extrémité de la queue des nymphes ; Figure 8A 1,A2).
- Femelles : De plus grande taille, un lobe génital plus petit et moins prononcé, et des pagaies plus étroites (figures 8B 1,B2).
- Pool de moustiques fluorescents positifs ou fluorescents négatifs de chaque sexe.
- Croisez les moustiques regroupés avec des moustiques du sexe opposé de la souche Liverpool. Utilisez un rapport de 3:1 à 5:1 entre le type sauvage et le moustique G0.
- Croisez les moustiques pendant 4 jours.
- Donnez aux femelles un repas de sang après le croisement.
12. Dépistage G1
- Trois jours après l’alimentation sanguine, fournissez des coquetiers en plaçant une serviette en papier à l’intérieur de la paroi des tasses de 9 oz de Karat et en ajoutant environ 3 oz d’eau déminéralisée.
- Après 3-4 jours, récoltez et faites éclore les embryons G1. Criblez les larves G1 pour détecter la présence du marqueur fluorescent aux stades 3 et 4 (figure 7).
- Recueillir les larves fluorescentes, en suggérant une insertion HDR. Lorsque les individus fluorescents G1 atteignent le stade de nymphe, séparez-les par sexe et placez chaque sexe dans des cages séparées.
- Croisez les moustiques fluorescents G1 avec des individus du sexe opposé de la souche Liverpool.
13. Confirmation du site d’insertion G1
- Après avoir fourni le repas de sang, préparez les femelles G1 pour la ponte d’un seul œuf femelle en plaçant un petit morceau de serviette en papier dans des flacons individuels de drosophile et en ajoutant 3 ml d’eau désionisée.
- Pour les mutants knock-out :
- Après la ponte, l’eau s’évapore et la serviette en papier sèche, faites éclore les œufs des femelles qui présentent clairement des mutations knock-out et obtenez la génération G2.
- Collectez le corps des mères G1 qui ont réussi à faire éclore la progéniture G2 et extrayez l’ADN.
- Utilisez l’ADN comme modèle pour la PCR afin d’amplifier la séquence couvrant le site cible. Assurez-vous que les amorces amplifient un fragment d’ADN de ~200 pb. Préparez les réactions (chacune contenant 12,5 μL du mélange maître 2x, 1,25 μL de la solution 10 μM d’amorce directe, 1,25 μL de la solution 10 μM d’amorce inverse, 9 μL d’eau ultrapure et 1 μL d’ADN matrice [5 ng/μL]). Utiliser les conditions de PCR suivantes : dénaturation initiale pendant 30 s à 98 °C ; 35 cycles d’amplification pendant 10 s à 98 °C, 15-30 s à X °C (température de recuit idéale des amorces) et 10 s à 72 °C ; extension finale à 72 °C pendant 2 min ; et stockage final à long terme à 4 °C.
- Purifiez les fragments de PCR et effectuez la digestion enzymatique de restriction des fragments de PCR en incubant un mélange de 1 μL de tampon de digestion de restriction 10x, 0,5 μL d’enzyme de restriction 1, 0,5 μL d’enzyme de restriction 2, 1 μL de fragment de PCR (200 ng/mL) et 7 μL d’eau ultrapure à 37 °C pendant 30 min.
REMARQUE : Certaines enzymes de restriction peuvent être utilisées dans le mélange PCR sans purification préalable. - Visualisez la digestion des fragments de PCR par électrophorèse sur gel pour confirmer s’il y a eu clivage.
- Séquencez le fragment de PCR non digéré pour identifier les mutations knock-out.
- Pour les insertions médiées par HDR, après la ponte :
- Collectez le corps des mères G1 et extrayez l’ADN.
- Utilisez l’ADN comme modèle pour la PCR afin d’amplifier la séquence couvrant le site cible.
- Effectuez le séquençage du fragment d’ADN amplifié pour confirmer l’intégrité de la cassette d’ADN insérée.
- Faites éclore l’œuf et obtenez la génération G2.
14. Élargir les nouvelles gammes CRISPR
- Pour les lignées mutantes knock-out :
- Configurez les femelles G2 pour la ponte, avec 20 femelles par G1. Chaque G1 représente une ligne.
- Après la ponte, confirmer les mutations knock-out de la même manière que pour G1.
- Poursuivez avec cinq lignées G1 qui présentent des mutations clairement identifiées dans G1 et G2. Faites éclore les œufs collectés dans G2 de chaque lignée, donnant naissance à la génération G3.
- Parmi les cinq lignées croisées, sélectionnez deux lignées présentant des mutations clairement identifiées et une valeur adaptative élevée ou le phénotype souhaité.
- Croisez les femelles G3 de chaque lignée sélectionnée avec les mâles de la souche Liverpool.
- Installez 50 femelles seules pour la ponte à partir de chaque ligne. Confirmez les mutations knock-out comme cela a été fait précédemment pour G1.
REMARQUE : Augmentez le nombre de femmes dans la configuration pour maintenir la diversité. - Répétez les procédures pour deux générations supplémentaires avec les deux lignes (Figure 4J).
- Pour les nouvelles lignes d’insertions médiées par HDR :
- Vérifier la présence d’individus fluorescents dans la génération G2.
- Croisez des moustiques G2, de chaque individu G1, avec la souche Liverpool. Chaque individu G1 représente une ligne.
- Poursuivez avec trois autres générations de croisement. Vérifiez continuellement le marqueur fluorescent et observez la valeur adaptative19 de chaque ligne (Figure 4x).
- Sélectionnez une ligne avec des insertions correctes et une forme physique élevée et le phénotype souhaité.
15. Faites des lignes homozygotes
- Pour les lignées mutantes knock-out :
- Génération G6 :
- Faites éclore les œufs produits par G5 et obtenez la génération G6.
- Croiser les adultes G6 à l’intérieur de leurs lignes respectives (figure 4K et figure 9A).
- Installez 50 femelles G6 simples pour la ponte par ligne et faites éclore les œufs comme décrit précédemment (figure 9B).
- Prélever les femelles G6 qui ont réussi à faire éclore la progéniture G7, extraire l’ADN et effectuer une PCR et une digestion enzymatique de restriction comme pour G1 (figure 9C).
REMARQUE : Un tampon d’écrasement (SB) simple et peu coûteux peut être utilisé à cette étape pour l’extraction de l’ADN.- À l’aide d’un broyeur à main, faites macérer un seul moustique dans 100 μL de 1x SB (10 mM de Tris-HCl pH 8, 1 mM d’EDTA et 25 mM de NaCl). Ajouter la protéinase K (200 μg/mL) et incuber à 37 °C pendant 30 min, puis à 95 °C pendant 2 min. Faites tourner à 10 000 × g pendant 5 min et récupérez le surnageant20.
- Confirmez la mutation en visualisant les résultats sur un gel.
- Jetez les œufs produits par les femelles sans mutations (figure 9D).
- Conserver les larves des femelles présentant des mutations confirmées afin d’établir plusieurs lignées G7, et jeter les couvées sans mutations (figure 9D).
- Génération G7 :
- Croisement des femelles et des mâles au sein de chaque lignée G7 (figure 9E).
- Installez 10 femelles G7 simples pour la ponte par ligne et faites éclore les œufs comme décrit précédemment (figure 9F).
- Collectez les femelles G7 qui ont réussi à faire éclore la progéniture G7, extrayez l’ADN et effectuez une PCR et une digestion par enzyme de restriction (Figure 9G).
- Détectez les mutations knock-out sur les deux allèles ou sur un seul allèle en visualisant les résultats sur un gel.
- Maintenir les larves des femelles G7 dont il a été confirmé qu’elles ont des knock-out dans les deux allèles, produisant la génération G8 (figure 9H). Jetez les couvées sans mutations (figure 9H).
REMARQUE : Cette étape permet de s’assurer que les femelles sont homozygotes. Au G8, tester l’homozygotie de la descendance permet de confirmer si les pères sont également homozygotes.
- Génération G8 :
- Croisez des adultes G8 pour chaque lignée féminine G7 (figure 9I).
- Choisissez au hasard cinq mâles G8, extrayez l’ADN et effectuez une PCR et une digestion par enzyme de restriction (Figure 9J).
- Fournissez un repas de sang et faites éclore les œufs des femelles G8 qui ont été croisées avec des mâles G8 dont il a été confirmé qu’ils avaient des knock-out dans les deux allèles.
- Continuez avec les lignées G7 où tous les mâles détectés ont des knock-out dans les deux allèles, indiquant que ces lignées sont homozygotes.
- Génération G6 :
- Pour les lignes d’insertion médiées par HDR :
- Faites éclore les œufs produits par G5 et obtenez la génération G6. Criblez les larves au stade G6 pour les marqueurs fluorescents.
- Jeter les larves sans marqueurs fluorescents (figure 4xi).
- Croisez des moustiques qui présentent des marqueurs fluorescents.
- Poursuivre cette procédure pour chaque génération subséquente jusqu’à ce qu’aucune larve non fluorescente ne soit observée. À ce stade, la ligne de moustiques sera homozygote.
Résultats
Conception et validation du ciblage génique médié par l’ARNg pour la recombinaison par homologie HDR
Pour s’assurer que le gène souhaité est ciblé avec précision, nous recommandons de sélectionner quelques ARNg et de positionner les bras d’homologie 5' et 3' près du site de coupe pour la recombinaison homologue médiée par HDR (Figure 1A). Par exemple, nous avons conçu deux ARNg pour cibler les deux côtés du codon de départ du gène d’intérêt et avons utilisé la cassette QF2-Hsp70-OpIE2-ECFP-SV40 comme marqueur, en l’insérant dans le gène ciblé via des bras d’homologie de 1 kb via le mécanisme HDR (Figure 1B). Les cassures double brin entraînent la délétion d’un court fragment d’ADN entre les sites de coupe des ARNg (Figure 1A).
L’efficacité du clivage des ARNg et leur capacité à couper le gène cible ont été testées par un test de clivage Cas9 in vitro. Les fragments de PCR du gène cible ont été clivés en deux fragments par Cas9, chargés avec les ARNg (Figure 1C). De plus, des enzymes de restriction peuvent être utilisées pour vérifier l’intégrité du plasmide donneur avant de passer aux étapes en aval du protocole. Les résultats de la digestion plasmidique doivent afficher une ou deux bandes sur le gel, la présence d’une petite bande indiquant une digestion réussie en un court fragment (Figure 1D,E).
Préparation des aiguilles de micro-injection
Avant de commencer le processus d’injection, il est important de préparer des aiguilles de micro-injection fraîches. Les aiguilles doivent avoir une forme protubérante avec une légère courbure. Environ 2/3 de l’aiguille doit être étroit en haut, tandis que le 1/3 restant doit être plus large. L’extrémité avant de la pointe de l’aiguille ne doit pas dépasser 0,5 cm de longueur entre la partie étroite et la partie large (figure 2A). Après avoir utilisé le bord de la lamelle pour ouvrir l’aiguille, une aiguille à pointe biseautée est préférable pour la microinjection, car elle est plus facile à utiliser après avoir cassé l’aiguille en touchant doucement la surface de l’embryon de moustique noir (figure 2B). D’autre part, une aiguille à pointe émoussée peut rendre difficile la perforation des embryons gris clair lors de la micro-injection (Figure 2C). Les aiguilles ne doivent être ni trop molles ni trop dures pour pénétrer efficacement les embryons de moustiques afin d’éviter de casser l’aiguille.
Injection d’embryons
Pour assurer une injection réussie, commencez par récolter les embryons de moustiques gris clair et alignez-les de manière à ce que le pôle postérieur soit orienté dans la même direction (Figure 3A). Cet alignement directionnel permet d’injecter le plasmide près des cellules germinales (pôle postérieur), ce qui est crucial pour produire des mutants qui peuvent être hérités par la génération suivante. Choisissez des embryons frais, uniformes et gris clair provenant de femelles nourries au sang 5 à 10 jours auparavant (figure 3A1). Évitez d’utiliser des embryons qui sont inégalement gris ou bruns (figure 3A2), car ils pourraient ne pas se développer correctement.
Alignez les embryons de moustiques un par un sur le ruban adhésif double face pour faciliter une injection précise et efficace. Appliquez de l’huile d’halocarbure avant que les œufs ne se dessèchent et ne deviennent gris moyen ou foncé (figure 3B).
Lors de l’injection, concentrez-vous sur la surface du pôle postérieur de chaque embryon. La face antérieure de l’embryon (côté droit sur la figure 5), en revanche, porte une surface plane et dure (micropyle). À l’aide d’une aiguille biseautée contenant le mélange d’injection, percez l’embryon (figure 5). La surface de l’embryon s’enfonce et rebondit, indiquant que l’aiguille a pénétré l’embryon. Une fois l’injection réussie, la couleur de l’embryon s’éclaircit en quelques secondes (courbe jaune sur la figure 5, en haut à droite).
Après l’injection, utilisez la brosse avec précaution pour prélever et transférer les embryons sur un autre papier filtre humide à l’aide d’une lingette de laboratoire pour enlever l’excès d’huile (figure 6A). Conservez les embryons sur un essuie-tout humide pendant plusieurs jours à la température d’élevage (figure 6B). L’excès d’huile peut réduire le taux d’éclosion des embryons de moustiques. Utilisez des boules de coton avec de l’eau ultra-pure pour maintenir l’hydratation. Il est peu probable que les embryons qui restent gris ou bruns (flèche jaune) après l’injection éclosent (figure 6C).
Criblage et croisement
Une fois que les embryons ont mûri, ajoutez de l’eau ultrapure dans le coquetier et placez-le dans un incubateur à 27 °C pour l’éclosion. Il faut généralement 5 à 7 jours pour que les larves se développent jusqu’au 3e stade. Initialement, nous avons présélectionné les larves G0 qui présentaient une fluorescence, bien qu’elle soit plus faible ou partielle par rapport aux autres lignées mutantes. Cela a permis de s’assurer que le marqueur était exprimé correctement et que les plasmides étaient injectés avec succès dans les embryons.
Ces larves ont ensuite été croisées avec des moustiques de type sauvage (WT). Au cours des criblages subséquents, nous avons sélectionné et conservé les larves G1 qui présentaient une expression de fluorescence plus forte et complète pour l’élevage, l’expansion du stock de lignées et la réalisation d’autres expériences. Les larves fluorescentes G1 peuvent être identifiées sur la base du modèle d’expression de la protéine de fluorescence fourni par la combinaison du promoteur rapporteur et de la protéine fluorescente : le promoteur Hr5Ie1-eGFP (Figure 7A) entraîne l’expression de la protéine fluorescente verte dans tout le corps ; Hr5Ie1-DsRed (Figure 7B) conduit à l’expression de la fluorescence rouge dans le corps ; OpIE2-CFP (Figure 7C) induit l’expression de la protéine de fluorescence cyan (bleu) uniquement dans une partie spécifique du corps. Lorsque l’association 3xP3-TdTomato est utilisée, la protéine fluorescente rouge est spécifiquement exprimée dans les yeux (Figure 7D). Cette lignée spécifique de moustiques transgéniques contient également le promoteur OpIE2 qui détermine l’expression de la CFP dans l’ensemble du corps (Figure 7D).
Procédé spécial d’établissement homozygote pour les mutants knock-out
Pour simplifier la maintenance de la ligne et s’assurer que tous les moustiques sont mutants, nous avons mis en place un pipeline pour générer des moustiques mutants homozygotes non fluorescents. Comme décrit à l’étape 15 du protocole, après croisement avec des moustiques WT, nous avons placé les moustiques mutants G6 dans la même cage pour le croisement (Figure 9A). Après le croisement, les femelles ont été rassemblées dans des flacons individuels pour la ponte (figure 9B). Cette étape a permis de séparer les femelles ayant des génotypes différents.
Après le prélèvement des œufs pour obtenir G7, l’ADN génomique a été extrait des femelles G6, suivi d’une PCR et d’une électrophorèse sur gel de fragments de PCR, traités ou non avec une enzyme de restriction (Figure 9C). Les lignes ne montrant que des fragments de PCR digérés, indiquant l’absence de mutations, ont été éliminées. Tandis que les lignes montrant des bandes double-PCR, indiquant la présence de mutations et, à leur tour, de femelles hétérozygotes, ont été conservées et hachurées (Figure 9D). On a ensuite laissé les moustiques G7 arriver à maturité, et tous les adultes ont été placés dans la même cage pour une autre série de croisements afin d’augmenter la probabilité d’établir une lignée homozygote (figure 9E). Les femelles G7 nourries de sang ont de nouveau été prélevées individuellement pour la collecte d’œufs G8, représentant trois génotypes potentiels (figure 9F). L’ADN génomique a été extrait des femelles G7, et une PCR suivie d’une électrophorèse sur gel a été effectuée pour déterminer lesquelles étaient homozygotes (figure 9G). Les œufs de femelles non mutantes ou hétérozygotes ont été jetés, et seuls ceux qui avaient des résultats homozygotes ont été conservés (figure 9H). Les œufs G8 de l’étape H ont éclos, et les adultes résultants ont été croisés dans une cage distincte (figure 9I). Dans chaque cage, 10 mâles adultes ont été sélectionnés au hasard pour l’électrophorèse sur gel et le séquençage (figure 9J). Cette étape devrait permettre de trouver des individus homozygotes, qui ont ensuite été maintenus comme stock (figure 9K).
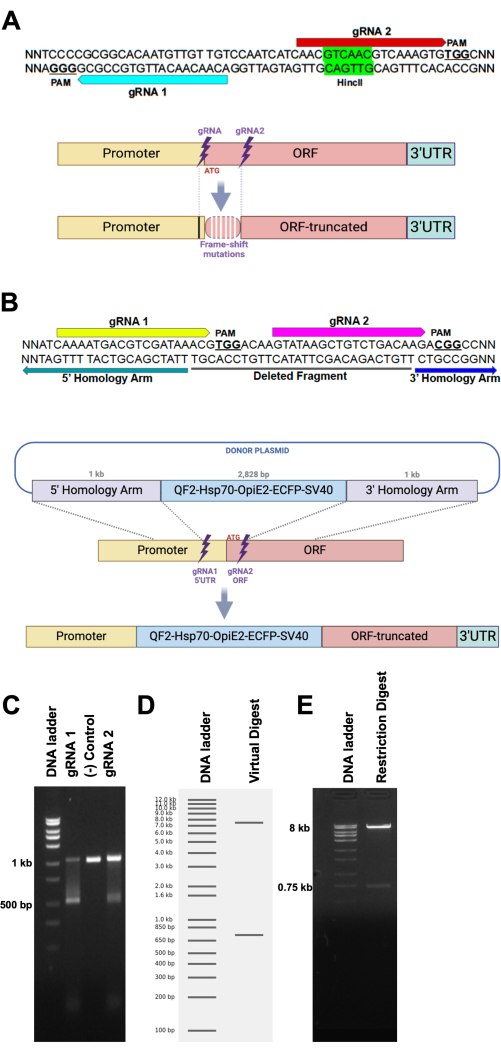
Figure 1 : Schéma de la conception du plasmide donneur d’ARNg et de HDR. (A, en haut) Séquence cible hypothétique de séquence d’ADN pour deux ARNg (flèches bleue et rouge et leurs sites PAM associés ainsi qu’un site cible d’enzyme de restriction (HincII). (A, en bas) Représentation schématique d’un site cible soumis à NHEJ, conduisant à l’émergence de mutations par décalage de cadre (barres verticales blanches) dues à la suppression ou à l’insertion d’une petite séquence. Les mutations peuvent s’étendre au-delà du site de clivage des ARNg (barre verticale noire). (B, haut) Séquence d’ADN hypothétique décrivant les sites cibles de l’ARNg 1 (flèche jaune) et de l’ARNg 2 (flèche magenta), suivis des sites PAM (NGG, gras et souligné). La séquence d’ADN potentiellement supprimée entre les deux ARNg est soulignée par une barre noire (fragment supprimé). Les flèches vertes et bleues mettent en évidence la séquence la plus importante de 3' du bras d’homologie 5' et la séquence la plus importante de 5' du bras d’homologie 3', respectivement. (B, en bas) Représentation schématique d’une recombinaison HDR d’une cassette d’ADN d’un plasmide à un site homologue dans le génome. (C) Essai de clivage in vitro. L’incubation d’un fragment d’ADN de 1 kb est digérée par Cas9 chargé soit avec l’ARNg 1 (voie gauche), soit l’ARNg 2 (voie droite), ce qui donne un fragment de 500 pb. La voie centrale montre la réaction de contrôle négative, qui était dépourvue de tout ARNg. (D) Digestion virtuelle d’un plasmide (8,75 kb) avec deux enzymes de restriction à coupe unique, résultant en pratiquement deux bandes (8 kb et 750 pb) sur un gel hypothétique. (E) Un gel d’agarose chargé de la réaction de digestion de restriction d’un plasmide, décrivant les résultats attendus fournis par la digestion virtuelle. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Forme de l’aiguille de micro-injection. (A) Image représentative d’une pointe d’aiguille. (B) Forme idéale de la pointe de l’aiguille pour la micro-injection d’œufs de moustiques. (C) Forme de la pointe de l’aiguille à éviter. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Récolte d’embryons. (A) Collecte d’embryons gris clair et (A1) homogènes mais pas (A2) inégaux pour injections ; Barre d’échelle = 500 μm. (B) Image pour les embryons alignés ; Barre d’échelle = 500 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
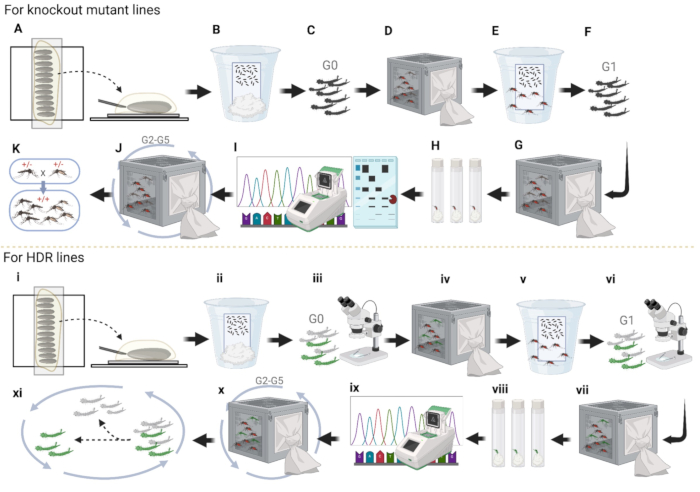
Figure 4 : Pipeline de micro-injection d’embryons de moustiques. (A-K) Injections de la ligne d’éjection. (A) Alignez les embryons de moustiques et injectez le plasmide dans l’extrémité postérieure. (B) Stockage d’embryons injecté dans le gobelet avec du papier humide ; (C) Les larves G0 éclosent et se séparent pour les deux sexes. (D) Les adultes des deux sexes traversent la rivière WT dans des cages séparées ; (E) la collecte des œufs des femelles dans le coquetier. (F) Larves G1 éclosent et se séparent entre les deux sexes. (G) Les adultes des deux sexes traversent la rivière WT dans des cages séparées. (H) Recueillir les œufs femelles G1 individuellement dans des flacons en plastique et faire éclore les œufs. (I) PCR, analyse du gel avec des fragments traités aux enzymes et séquençage pour les femelles uniques G1 qui ont réussi à faire éclore des œufs. (J) Croiser G2 avec WT pendant trois générations supplémentaires et séquencer les moustiques de la même manière que pour G1. (K) Établir l’homozygote par croisement. (i-xi) Injections de ligne HDR. (i) Alignement des embryons. (ii) Stockage d’embryons injecté dans le gobelet avec du papier humide. (iii) Prédépistage de la fluorescence des larves G0. (iv) Développer des larves fluorescentes jusqu’à l’âge adulte et les croiser avec des WT dans une cage. (v) Collecte des œufs G1 dans le coquetier. (vi) Dépistage et sélection des larves fluorescentes G1. (vii) Adultes fluorescents G1 croisés avec WT. (viii) Femelles adultes fluorescentes G1 nourries de sang pondant des œufs individuellement dans des flacons en plastique. (ix) Collecte et séquençage d’adultes G1 fluorescents individuellement. (x) Croisement avec le type sauvage pour trois générations supplémentaires et (xi) élimination des larves sans fluorescence pour établir des homozygotes. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Injection d’embryons. Le moment de l’injection du plasmide dans les embryons de moustiques. Encadré : mélange d’injection visualisé sous la forme d’un liquide trouble à l’intérieur de l’embryon (ligne pointillée jaune) ; Barre d’échelle = 200 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
Figure 6 : Collection d’embryons. (A) Récolte des embryons après injection et transfert sur un papier filtre humide. Barre d’échelle = 7,5 mm. (B) Maintien des embryons injectés dans une tasse. Barre d’échelle = 25 mm. (C) Embryons foncés après injection ; Barre d’échelle = 500 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
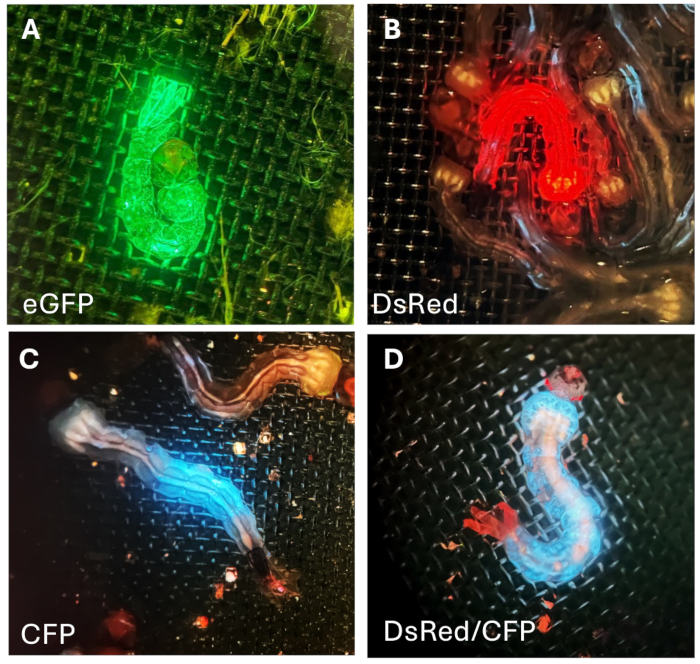
Figure 7 : Fluorescence chez les larves G1. (A) Protéines fluorescentes vertes, (B) rouges et (C) bleuessur le corps de la larve de moustique. (D) Larve présentant une fluorescence rouge spécifique à l’œil et une fluorescence bleue spécifique au corps. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 8 : Dimorphisme sexuel dans la morphologie des nymphes. (A1,A2) Les pupes mâles se caractérisent par une taille corporelle plus petite, un lobe génital proéminent et pointu et des palettes plus larges à l’extrémité de la queue. (B1,B2) Les pupes femelles présentent une taille corporelle plus grande, un lobe génital plus petit et moins prononcé et des pagaies plus étroites à l’extrémité de la queue. Veuillez cliquer ici pour voir une version agrandie de cette figure.
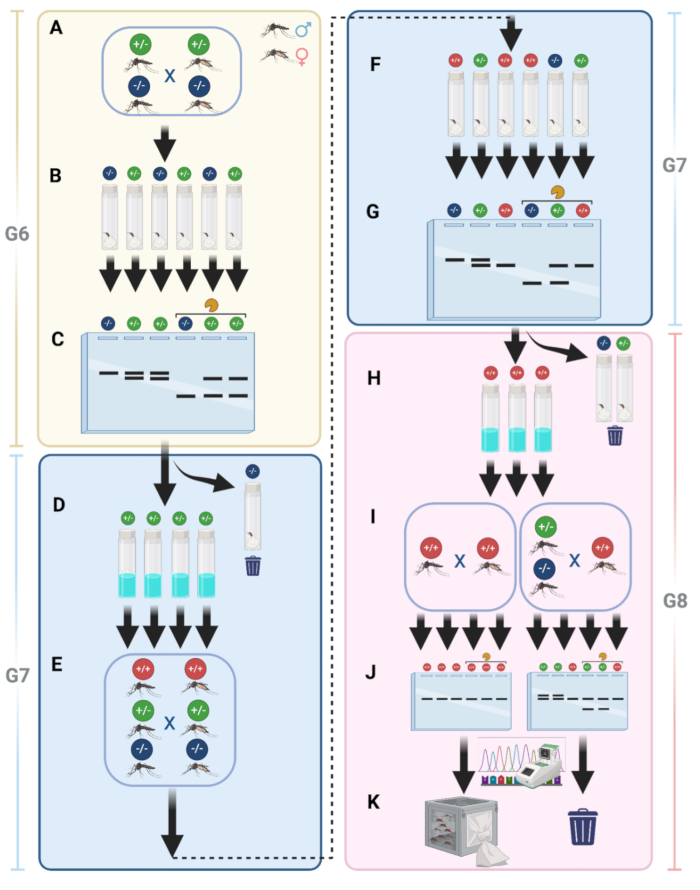
Figure 9 : Détails de l’établissement homozygote pour les lignées mutantes knock-out. (A) Les moustiques mutants sont croisés dans la même cage après s’être croisés avec WT. (B) Après l’intersection, les femelles individuelles sont collectées dans des flacons pour la ponte. (C) L’ADN génomique est extrait des femelles G6, suivi d’une PCR et d’une électrophorèse sur gel avec des fragments traités et non traités par enzyme. (D) Des œufs G7 avec des résultats positifs à la PCR (hétérozygotes) sont éclos. (E) Les adultes G7 sont à nouveau croisés pour augmenter les chances d’obtenir des individus homozygotes. (F) Les femelles G7 nourries de sang sont prélevées individuellement pour la collecte d’œufs G8, représentant trois génotypes. (G) L’extraction de l’ADN génomique, la PCR et l’électrophorèse sur gel sont effectuées sur les femelles G7 pour identifier les individus homozygotes. (H) Les œufs de femelles non mutantes ou hétérozygotes sont jetés ; Des œufs homozygotes sont conservés. (I) Les œufs G8 sont éclos et entrecroisés dans une cage séparée. (J) Dix mâles adultes sont sélectionnés au hasard dans chaque cage pour l’électrophorèse sur gel et le séquençage. (K) Les moustiques homozygotes sont identifiés et maintenus en tant que stock. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Construction d’injection | Concentration finale |
| Protéine Cas9 | 100 à 300 ng/μL |
| ARNg | 50 à 100 ng/μL |
| plasmide donneur | 100 à 500 ng/μL |
Tableau 1 : Plages de concentration finale suggérées pour chaque construction d’injection.
Discussion
La technologie CRISPR-Cas a changé le paysage de l’édition du génome en favorisant des changements spécifiques à la cible dans les chromosomes1. Même si les éléments transposables étaient essentiels à la génération des premiers moustiques transgéniques, leurs sites d’insertion sont quelque peu aléatoires et l’expression de la construction cargo (promoteur + gène) peut ne pas correspondre au profil d’expression du gène réel en raison d’un effet de position du génome (c’est-à-dire le site d’insertion), qui conduit généralement à l’expression ectopique21. Avant l’émergence des ciseaux moléculaires CRISPR-Cas9, des technologies qui favorisent les cassures double brin de l’ADN à des sites cibles spécifiques ont été développées et reposaient sur les propriétés de liaison à l’ADN de domaines protéiques spécifiques marqués à l’enzyme de restriction FokI, tels que TALEN22 et Zinc Finger23. Bien que cette dernière technologie ait été utilisée avec succès pour le génie génétique des moustiques24,25, le coût élevé de l’ingénierie de ces domaines et le risque de clivage hors cible ont conduit à l’abandon de ces technologies. Depuis, la technologie CRISPR-Cas9 est devenue les ciseaux moléculaires de choix11.
Le génie génétique des moustiques a été, pour l’essentiel, effectué par injection du mélange de transformation dans les embryons 26,27. La fabrication d’une micro-injection embryonnaire de moustique nécessite de la concentration, de la patience et une attention aux détails. Pendant les 2 h qui suivent la ponte, ces embryons doivent être alignés, injectés et lavés le plus soigneusement possible afin de ne pas perturber leur développement. La concentration est une compétence essentielle pour réduire la possibilité que les ovules soient endommagés ou que les embryons se dessèchent pendant la file d’attente. Face à des incidents inattendus tels que le colmatage de l’aiguille, la rupture de l’embout et la fuite du mélange d’injection, qui se produisent assez souvent lorsque la protéine Cas9 est dans le mélange, la patience est une autre compétence essentielle pour gérer ces situations. Le fait de piquer les embryons lorsque la pointe de l’aiguille est trop grande ou que trop de mélange d’injection fuit ne peut qu’endommager l’embryon. La meilleure solution est de remplacer l’aiguille et de poursuivre les injections. L’attention portée aux détails n’est pas moins importante. Les moustiques femelles plus jeunes (6 à 10 jours) pondent plus d’œufs que les plus âgés, ce qui est utile pour récolter des embryons sains. Choisissez de l’eau déminéralisée ou ultra-pure et des serviettes en papier brunes (non blanchies) pour stocker les embryons après la microinjection, car tout contact avec de l’eau contaminée ou des surfaces blanchies peut augmenter considérablement la mortalité embryonnaire. Les embryons gris clair (âgés de <1 h) sont plus mous, ce qui facilite la procédure d’injection, augmentant les chances d’obtenir la lignée mutante. De plus, les embryons injectés doivent reposer dans du papier filtre humidifié pendant au moins 5 jours avant d’être éclos pour permettre une récupération et une guérison complètes.
La qualité et la pureté du mélange d’injection sont fondamentales pour la survie de l’embryon. Ni les tampons, les sels ni les alcools des kits d’extraction de plasmides ou des kits de synthèse d’ARNg ne doivent être transférés dans les mélanges d’injection. Même si elle est coûteuse, la possibilité de commander les ARNg peut augmenter la probabilité d’un bon repliement de l’ARN. Nous recommandons également d’injecter la protéine recombinante Cas9 sur un plasmide codant pour Cas9, car nous pensons que la version recombinante est déjà chargée et prête à effectuer des cassures double brin par rapport à une source plasmidique qui a besoin d’être transcrite et traduite, puis chargée d’ARNg pour commencer à agir sur l’ADN.
Bien que nous ayons décrit le protocole qui fonctionne le mieux entre nos mains en ce qui concerne l’utilisation de la technologie CRISPR-Cas9 pour l’édition du génome chez le moustique A. aegypti, des réactifs alternatifs ont été décrits dans la littérature qui peuvent également être utilisés en remplacement si nécessaire. Par exemple, nous suspendons tous les réactifs et diluons les mélanges d’injection avec de l’eau ultrapure. Alternativement, les mélanges d’injection peuvent être dilués dans un tampon d’injection composé de 5 mM de KCl et de 0,1 M de NaPO4 (pH = 6,8)26,28. Outre l’utilisation de l’huile d’halocarbure 700 pour protéger les embryons de la dessiccation après les avoir alignés, l’huile d’halocarbure 2729 ainsi qu’un mélange de 27 et 70030 offrent une protection similaire. La version 10x concentrée du tampon d’injection décrite ci-dessus a été utilisée alternativement pour prévenir la dessiccation de l’embryon26. Comme mentionné ci-dessus, les plasmides codant pour Cas9 sont disponibles dans le commerce (addgene) et ont été utilisés pour l’édition du génome médiée par CRISPR-Cas9 chez les moustiques31.
Parmi les stratégies permettant d’obtenir une lignée transgénique de moustique, il existe des approches de knock-out médié par NHEJ et de knock-in médié par HDR. Alors que le taux de réussite du premier est beaucoup plus élevé que celui du second32,33, le schéma de croisement et le processus de dépistage prennent beaucoup plus de temps pour obtenir un knockout qu’une ligne knock-in, comme décrit à la figure 4. En tant qu’approche alternative pour obtenir une lignée knock-out sans passer par des schémas de croisement compliqués, la knockin médiée par HDR d’une cassette de gènes codant pour un marqueur fluorescent offre la possibilité de perturber la séquence codante d’un gène et de sélectionner la lignée transgénique de mosquito à l’aide du marqueur fluorescent14,34.
Bien que ce protocole ne se concentre que sur les étapes de base de la transformation génétique des moustiques, la conception de plasmides donneurs pour l’HDR est une étape essentielle qui doit être prise en compte. Il est important d’utiliser des promoteurs tels que 3xP3, OpIE2, Hr5-IE1 et Pub 6,35,36,37,38. 3xP3 est un promoteur synthétique couramment utilisé pour stimuler l’expression dans le système nerveux et les yeux des insectes. OpIE2 et Hr5-IE1 sont des promoteurs dérivés du baculovirus qui sont utilisés pour favoriser une forte expression dans les cellules d’insectes. Le promoteur de pub stimule l’expression dans tout le corps. Le promoteur U6 est utilisé pour l’expression des ARNg. Des marqueurs fluorescents tels que dsRed (et TdTomato), eGFP, CFP et YFP sont également utilisés pour les plasmides donneurs pour HDR. Ce sont des protéines fluorescentes qui émettent respectivement une fluorescence rouge, verte, cyan et jaune. Pour les UTR 3', p1039 et SV4040 sont souvent utilisés pour améliorer la stabilité de l’ARNm et l’efficacité de la traduction dans les transgènes.
En résumé de la procédure de micro-injection, pour les lignes mutantes knock-out, alignez d’abord les embryons de moustiques sur du ruban adhésif double face côte à côte pour l’injection (Figure 4A). Deuxièmement, retirez la majeure partie de l’huile d’halocarbure pour faciliter le taux de survie des embryons et transférez les embryons du ruban adhésif double face sur un papier filtre propre et humide. Conservez-les dans une tasse avec de l’eau ultra-pure pour maintenir une humidité élevée pendant quelques jours, en attendant que les embryons se développent complètement (Figure 4B). Troisièmement, faites éclore les embryons injectés, séparez les pupes des deux sexes et placez-les dans des cages différentes (figure 4C). Quatrièmement, placez tous les mâles fluorescents dans une cage pour les croiser avec des femelles WT âgées de 5 à 7 jours, et vice versa (figure 4D). Cinquièmement, prélever des œufs G1 sur les femelles WT croisées pour augmenter le nombre de moustiques au début (figure 4E). Sixièmement, l’éclosion des œufs G1 et la séparation des pupes entre les deux sexes (figure 4F). Septièmement, les adultes G1 se croisent de nouveau avec WT (figure 4G). Huitièmement, prélever les femelles adultes G1 dans des flacons pour recueillir les œufs individuels de G2 et maintenir de l’eau ultra-pure au fond des flacons à l’aide de boules de coton ou d’un essuie-tout pendant quelques jours (figure 4H). Neuvièmement, prélever les femelles G1 après qu’elles aient pondu et extraire l’ADN génomique des femelles G1 pour la PCR et le séquençage (figure 4I). Dixièmement, l’éclosion d’œufs G2 corrects pour le maintien de la ligne et des croisements avec WT pendant trois générations supplémentaires pour éliminer la plupart des mutations de fond non liées (figure 4J). Onzièmement, l’établissement d’une lignée de moustiques homozygotes à l’aide de la PCR, du dosage des enzymes de restriction, de l’électrophorèse sur gel et du séquençage pour dépister les mutations (figure 4K).
Pour l’établissement des lignes HDR, alignez les embryons de moustiques sur du ruban adhésif double face côte à côte pour la microinjection d’embryons (Figure 4i). Deuxièmement, après les injections, retirez la majeure partie de l’huile d’halocarbure et conservez les embryons dans une tasse avec de l’eau ultra-pure pour maintenir l’humidité pendant quelques jours et attendre que les embryons mûrissent (Figure 4ii). Troisièmement, faire éclore des embryons injectés et cribler les larves G0 pour trier celles avec ou sans fluorescence (figure 4iii) ; Les larves avec fluorescence signifient que le plasmide a été injecté avec succès dans des embryons de moustiques. Quatrièmement, laissez les larves fluorescentes devenir adultes et placez tous les mâles fluorescents dans une cage pour les croiser avec les femelles WT, et vice versa, pour augmenter le nombre de moustiques (figure 4iv). Cinquièmement, prélever les œufs des femelles WT croisées (figure 4v). Sixièmement, faire éclore, filtrer et trier les larves en fluorescence G1 et éliminer les larves sans fluorescence (figure 4vi). Septièmement, croiser des adultes en fluorescence G1 avec WT (Figure 4vii). Huitièmement, prélever les œufs G2 dans des flacons en plastique individuellement pour séparer les différents sites d’insertion du plasmide chez les moustiques (Figure 4viii). Neuvièmement, faites éclore les œufs, vérifiez la condition physique des moustiques et effectuez le séquençage (Figure 4ix). Dixièmement, sélectionnez deux lignées avec un phénotype plus fort et croisez-les avec WT pour trois générations supplémentaires (figure 4x). Onzièmement, sélection des larves avec fluorescence pour s’entrecroiser pendant deux générations supplémentaires afin d’établir une lignée homozygote pour d’autres expériences (figure 4xi).
Ici, nous avons fourni le protocole le plus complet pour l’édition du génome d’A. aegypti avec CRISPR-Cas9 que nous avons utilisé pour générer plusieurs lignées transgéniques. Nous suggérons que ce protocole soit utilisé comme point de départ pour d’autres chercheurs, qui devraient apporter des changements en fonction de l’expérience acquise avec leurs essais de micro-injection. Des réactifs et des conditions différents doivent toujours être testés. Le taux de réussite de l’édition génomique chez les moustiques n’a pas encore été entièrement optimisé et très peu de laboratoires ont été en mesure de fabriquer des moustiques transgéniques via CRISPR-Cas9.
Déclarations de divulgation
O.S.A. est l’un des fondateurs d’Agragene, Inc. et de Synvect, Inc. avec une participation au capital. Les termes de cet arrangement ont été examinés et approuvés par l’Université de Californie à San Diego, conformément à ses politiques en matière de conflits d’intérêts. Les autres auteurs ne déclarent aucun intérêt concurrent.
Remerciements
Les auteurs remercient Judy Ishikawa et Ava Stevenson pour leur aide dans l’élevage des moustiques. Ce travail a été soutenu par le financement des NIH (R01AI151004, RO1AI148300 RO1AI175152) décernés à O.S.A. et K22AI166268 à N.H.R. Les figures ont été créées à l’aide de BioRender.
matériels
| Name | Company | Catalog Number | Comments |
| 10x Cas9 reaction buffer | PNA Bio | CB01 | |
| Benchling software | Benchling | N/A | www.benchling.com |
| Cas9 dilution buffer | PNA Bio | CB03 | |
| Cas9 protein | PNA Bio | CP01-50 | |
| DH5α E. coli Competent Cells | New England Biolabs | C2987 | |
| Double-sided sticky tape | Scotch Permanent | 3136 | |
| Drosophila vials | Genesee Scientific | 32-109 | |
| Filter papers | GE Healthcare Life Science | 1450-042 | |
| Fish food | Tetra | B00025Z6YI | goldfish flakes |
| Flugs | Genesee Scientific | AS273 | |
| Fluorescent microscope | Leica Microsystems | M165 FC | |
| Gene fragment | Integrated DNA Technologies | N/A | |
| gRNA | Synthego | N/A | |
| Halocarbon oil 700 | Sigma-Aldrich | H8898 | |
| Injection microscope | Leica Microsystems | DM2000 | |
| JM109 E. coli Competent Cells | Zymo Research | T3005 | |
| Microinjector | Eppendorf | FemtoJet 4x | |
| Microloader Tips for Filling Femtotips | Eppendorf | E5242956003 | |
| Micromanipulator | Eppendorf | TransferMan 4r | |
| Micropipette Pullers | Sutter Instrument | P-2000 | |
| Microscope Cover Glass | Fisherbrand | 12-542-B | |
| Microscope slide | Eisco | 12-550-A3 | |
| Mouse blood (live mice used for feeding) | University of California | IACUC, Animal Use Protocol #S17187 | Used for mosquito blood feeding; details comply with animal ethics protocols |
| NEB Q5 High-Fidelity DNA polymerase | New England Biolabs | M0491S | |
| PCR Purification Kit | Qiagen | 28004 | |
| Plasmid Miniprep Kit | Zymo Research | D4036 | |
| Quartz filament | Sutter Instruments | QF100-70-10 | |
| Transcription Clean-Up Kit | Fisher Scientific | AM1908 | |
| Ultra-pure water | Life Technologies | 10977-023 |
Références
- Anzalone, A. V., Koblan, L. W., Liu, D. R. Genome editing with CRISPR-Cas nucleases, base editors, transposases and prime editors. Nat Biotechnol. 38 (7), 824-844 (2020).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Shen, H., Li, Z. DNA double-strand break repairs and their application in plant DNA integration. Genes (Basel). 13 (2), 322 (2022).
- Vinauger, C., et al. Modulation of host learning in Aedes aegypti mosquitoes. Curr Biol. 28 (3), 333-344.e8 (2018).
- Li, M., Bui, M., Yang, T., Bowman, C. S., White, B. J., Akbari, O. S. Germline Cas9 expression yields highly efficient genome engineering in a major worldwide disease vector. Proc Natl Acad Sci U S A. 114 (49), E10540-E10549 (2017).
- Chaverra-Rodriguez, D., et al. Targeted delivery of CRISPR-Cas9 ribonucleoprotein into arthropod ovaries for heritable germline gene editing. Nat Commun. 9 (1), 3008 (2018).
- Rouyar, A., et al. Transgenic line for characterizing GABA-receptor expression to study the neural basis of olfaction in the yellow-fever mosquito. Front Physiol. 15, 1381164 (2024).
- Ang, J. X. D., et al. Considerations for homology-based DNA repair in mosquitoes: Impact of sequence heterology and donor template source. PLoS Genet. 18 (2), e1010060 (2022).
- Zhang, J. -. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biol. 18 (1), 35 (2017).
- Coutinho-Abreu, I. V., Akbari, O. S. Technological advances in mosquito olfaction neurogenetics. Trends Genet. 39 (2), 154-166 (2023).
- Li, M., et al. Targeting sex determination to suppress mosquito populations. eLife. 12, RP90199 (2024).
- Zhan, Y., Alonso San Alberto, D., Rusch, C., Riffell, J. A., Montell, C. Elimination of vision-guided target attraction in Aedes aegypti using CRISPR. Current Biol. 31 (18), 4180-4187.e6 (2021).
- Greppi, C., et al. Mosquito heat seeking is driven by an ancestral cooling receptor. Science. 367 (6478), 681-684 (2020).
- Potter, C. J., Tasic, B., Russler, E. V., Liang, L., Luo, L. The Q system: a repressible binary system for transgene expression, lineage tracing, and mosaic analysis. Cell. 141 (3), 536-548 (2010).
- Riabinina, O., et al. Improved and expanded Q-system reagents for genetic manipulations. Nat Methods. 12 (3), 219-222 (2015).
- Labun, K., et al. CHOPCHOP v3: expanding the CRISPR web toolbox beyond genome editing. Nucleic Acids Res. 47 (W1), W171-W174 (2019).
- Concordet, J. -. P., Haeussler, M. CRISPOR: intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Res. 46 (W1), W242-W245 (2018).
- Williams, A. E., et al. Quantifying fitness costs in transgenic Aedes aegypti mosquitoes. J Vis Exp. , e65136 (2023).
- Bassett, A. R., Tibbit, C., Ponting, C. P., Liu, J. -. L. Highly efficient targeted mutagenesis of Drosophila with the CRISPR/Cas9 system. Cell Rep. 4 (1), 220-228 (2013).
- Coutinho-Abreu, I. V., Zhu, K. Y., Ramalho-Ortigao, M. Transgenesis and paratransgenesis to control insect-borne diseases: current status and future challenges. Parasitol Int. 59 (1), 1-8 (2010).
- Christian, M., et al. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics. 186 (2), 757-761 (2010).
- Kim, Y. G., Cha, J., Chandrasegaran, S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain. Proc Natl Acad Sci U S A. 93 (3), 1156-1160 (1996).
- DeGennaro, M., et al. orco mutant mosquitoes lose strong preference for humans and are not repelled by volatile DEET. Nature. 498 (7455), 487-491 (2013).
- McMeniman, C. J., Corfas, R. A., Matthews, B. J., Ritchie, S. A., Vosshall, L. B. Multimodal integration of carbon dioxide and other sensory cues drives mosquito attraction to humans. Cell. 156 (5), 1060-1071 (2014).
- Lobo, N. F., Clayton, J. R., Fraser, M. J., Kafatos, F. C., Collins, F. H. High efficiency germ-line transformation of mosquitoes. Nat Protoc. 1 (3), 1312-1317 (2006).
- Kistler, K. E., Vosshall, L. B., Matthews, B. J. Genome engineering with CRISPR-Cas9 in the mosquito Aedes aegypti. Cell Rep. 11 (1), 51-60 (2015).
- Handler, A. M., Harrell, R. A. 2nd Transformation of the Caribbean fruit fly, Anastrephasuspensa, with a piggyBac vector marked with polyubiquitin-regulated GFP. Insect Biochem Mol Biol. 31 (2), 199-205 (2001).
- Harrell, R. A. . 2nd Mosquito embryo microinjection under halocarbon oil or in aqueous solution. 2024 (7), (2024).
- Sun, R., Raban, R., Akbari, O. S. Generating mutant strains with transgenic Cas9. Cold Spring Harb Protoc. 2023 (9), 671-678 (2023).
- Giraldo, D., et al. An expanded neurogenetic toolkit to decode olfaction in the African malaria mosquito Anopheles gambiae. Cell Rep Methods. 4 (2), 100714 (2024).
- Liu, G., Lin, Q., Jin, S., Gao, C. The CRISPR-Cas toolbox and gene editing technologies. Mol Cell. 82 (2), 333-347 (2022).
- Chu, V. T., et al. Increasing the efficiency of homology-directed repair for CRISPR-Cas9-induced precise gene editing in mammalian cells. Nat Biotechnol. 33 (5), 543-548 (2015).
- Laursen, W. J., et al. Humidity sensors that alert mosquitoes to nearby hosts and egg-laying sites. Neuron. 111 (6), 874-887.e8 (2023).
- Weng, S. -. C., Antoshechkin, I., Marois, E., Akbari, O. S. Efficient sex separation by exploiting differential alternative splicing of a dominant marker in Aedes aegypti. PLoS Genet. 19 (11), e1011065 (2023).
- Li, M., et al. Development of a confinable gene drive system in the human disease vector. eLife. 9, e51701 (2020).
- Dalla Benetta, E., et al. Engineered Antiviral Sensor Targets Infected Mosquitoes. The CRISPR journal. 6 (6), 543-556 (2023).
- Li, H. -. H., et al. C-Type lectins link immunological and reproductive processes in Aedes aegypti. iScience. 23 (9), 101486 (2020).
- van Oers, M. M., Vlak, J. M., Voorma, H. O., Thomas, A. A. M. Role of the 3' untranslated region of baculovirus p10 mRNA in high-level expression of foreign genes. J Gen Virol. 80 (Pt 8), 2253-2262 (1999).
- Salem, T. Z., et al. The influence of SV40 polyA on gene expression of baculovirus expression vector systems. PloS One. 10 (12), e0145019 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon