Method Article
Editing del genoma nella zanzara della febbre gialla Aedes aegypti utilizzando CRISPR-Cas9
In questo articolo
Riepilogo
Qui, descriviamo un protocollo dettagliato per l'editing del genoma attraverso la microiniezione embrionale nella zanzara A. aegypti utilizzando la tecnologia CRISPR-Cas9.
Abstract
L'emergere della tecnologia CRISPR-Cas9 (clustered, regularly intersranged, short palindromic repeats)-Cas9 ha rivoluzionato il campo dell'ingegneria genetica e ha aperto le porte all'editing preciso del genoma in più specie, compresi gli organismi non modello. Nella zanzara Aedes aegypti, con questa tecnologia sono state realizzate mutazioni con perdita di funzione e inserzioni di DNA. Qui, descriviamo un protocollo dettagliato per l'editing del genoma attraverso la microiniezione embrionale nella zanzara A. aegypti utilizzando la tecnologia CRISPR-Cas9, concentrandosi sia sulla generazione di knockout genico che sulle linee knockin. In questo protocollo, gli aghi di quarzo sono riempiti con una miscela di RNA guida, Cas9 ricombinante e un plasmide contenente una cassetta di DNA che codifica un gene per un marcatore fluorescente, se si desidera il knockin genico. Gli embrioni allo stadio di preblastoderma vengono allineati su una striscia di nastro biadesivo posizionata su un vetrino, che viene successivamente montato su un vetrino. Con l'aiuto di un microiniettore, gli aghi vengono inseriti delicatamente nell'estremità posteriore degli embrioni e viene erogato un piccolo volume della miscela CRISPR. Quando gli embrioni si schiudono, le larve vengono controllate sotto il cannocchiale fluorescente e le pupe vengono selezionate per sesso e separate in gabbie diverse. Una volta che gli adulti emergono, questi vengono reciprocamente incrociati con individui di tipo selvatico, nutriti con sangue e posti per la deposizione delle uova. Una volta che queste uova si sono schiuse, le larve fluorescenti raccolte rappresentano individui con inserimento stabile della cassetta del DNA nel loro genoma. Queste larve vengono poi cresciute fino allo stadio adulto, incrociate con individui di tipo selvatico e quindi ulteriormente valutate attraverso tecniche molecolari per confermare che l'esatta sequenza della cassetta del DNA sia presente nel sito desiderato del genoma della zanzara. Le linee omozigoti possono essere ottenute anche seguendo la pipeline fornita di schema di incrocio e screening molecolare delle mutazioni.
Introduzione
L'editing preciso del genoma è diventato probabilmente più facile, ma possibile, con l'istituzione delle tecnologie CRISPR-Cas delle forbici molecolari1. Queste tecnologie sfruttano un meccanismo che il sistema immunitario procariotico utilizza per combattere le infezioni da fagi2. Tra questi sistemi, le ripetizioni palindromiche brevi (CRISPR) raggruppate, regolarmente intervallate, insieme alla nucleasi Cas9 di solito si basano su 20 RNA di coppie di basi, gli RNA guida (gRNA), con sequenze omologhe al DNA bersaglio, che sono seguite da una sequenza3 del motivo adiacente al protospaziatore NGG (PAM). I gRNA caricati sul Cas9 guidano queste nucleasi con precisione verso specifici siti bersaglio nel genoma, innescando rotture del DNA a doppio filamento3.
Le rotture del doppio filamento del DNA inducono i meccanismi di riparazione a rattoppare la doppia elica4. Mentre ci si aspetta che qualsiasi riparazione del DNA sia precisa, esistono meccanismi di riparazione del DNA meno accurati che possono lasciare cicatrici di sequenza e, a loro volta, mutazioni con perdita di funzione4. Tra i meccanismi di riparazione del DNA soggetti a errori, l'unione terminale non omologa (NHEJ) può causare mutazioni frame-shift, tra cui piccole delezioni, inserzioni e cambiamenti nucleotidici (SNP), che possono provocare mutazioni con perdita di funzione. Il meccanismo di riparazione diretta per omologia (HDR), d'altra parte, si basa sul cromosoma omologo come modello per copiare l'esatta sequenza dell'allele non danneggiato e realizzare una riparazione perfetta della sequenza di DNA mirata4.
Sulla base di queste conoscenze, la tecnologia CRISPR-Cas9 è stata sviluppata per modificare con precisione i genomi, probabilmente in qualsiasi sequenza contenente un sito PAM3. Nelle zanzare, la tecnologia CRISPR-Cas9 è stata utilizzata per eliminare una varietà di geni, attraverso la microiniezione embrionale di una miscela di Cas9 e gRNA, sfruttando il meccanismo di riparazione NHEJ 5,6. Una mutagenesi germinale simile si ottiene con l'iniezione di una miscela di gRNA + Cas9 nell'emolinfa di zanzare femmine adulte7. Questa tecnologia è stata coniata come controllo ReMOT e si basa su una versione modificata di un Cas9 marcato con un peptide che viene assorbito dalle ovaie attraverso l'endocitosi durante il processo di sviluppo dell'uovo (vitellogenesi)7. L'inserimento di specifiche cassette geniche in un genoma è possibile solo attraverso la microiniezione embrionale di una miscela di gRNA e Cas9 (o di un plasmide che esprime tali molecole) insieme a un plasmide che codifica una cassetta di DNA desiderata8. Sfruttando il meccanismo HDR, il plasmide contenente la cassetta di DNA di interesse affiancato da sequenze omologhe (500-1.000bp)9,10 a monte e a valle del sito bersaglio viene utilizzato come stampo per riscrivere la rottura del doppio filamento, copiando anche la cassetta di DNA nella sequenza bersaglio9.
La tecnologia CRISPR-Cas9 è stata utilizzata per eliminare diversi geni coinvolti principalmente con i sistemi sensoriali nella zanzara Aedes aegypti11, ma anche geni associati alla fertilità maschile e alla vitalità femminile (PgSIT) per il controllo della popolazione12. Il knocking out di geni bersaglio è stato ottenuto anche inserendo geni che codificano marcatori fluorescenti nelle sequenze codificanti di geni specifici13,14. Questa strategia ha il vantaggio non solo di indurre mutazioni frame-shift, ma anche di consentire l'uso della luce fluorescente per selezionare gli individui della nuova linea knockout13,14. Il genoma di A. aegypti è stato anche modificato con sequenze di sistemi di espressione binaria, come il sistema Q (QF-QUAS)11. L'inserimento del gene che codifica per il transattivatore QF a valle di un promotore di un gene specifico assicura un'espressione spazio-temporale definita del transattivatore15,16. Una volta che una linea di zanzara che esprime QF viene incrociata con un'altra linea di zanzara contenente i siti di legame (QUAS) per QF, quest'ultima si lega ad essa e innesca l'espressione dei geni a valle della sequenza QUAS15,16. Questo sistema, nel complesso, consente l'espressione tessuto-specifica e tempo-specifica di tali geni effettori, che possono essere marcatori fluorescenti utilizzati per la localizzazione cellulare o il rilevamento dell'attività neuronale, e persino nucleasi Cas9 per l'interruzione di geni in tessuti specifici (ad esempio, knockout somatico)11.
Date tutte le informazioni disponibili per la trasformazione genetica di A. aegypti , forniamo qui un protocollo dettagliato con indicazioni dettagliate per eseguire l'editing del genoma con il sistema CRISPR-Cas9 attraverso la microiniezione embrionale. Vengono discusse le strategie per generare sia knockout, attraverso mutazioni frame-shift e delezioni mediate da NHEJ, sia linee knockin, mediante inserzioni di cassette geniche mediate da HDR.
Protocollo
I dettagli relativi alle apparecchiature e ai reagenti utilizzati in questo protocollo sono elencati nella Tabella dei materiali. Tutti gli animali sono stati gestiti seguendo la Guida per la cura e l'uso degli animali da laboratorio, come raccomandato dal National Institutes of Health. Le procedure sono state approvate dall'UCSD Institutional Animal Care and Use Committee (IACUC, Animal Use Protocol #S17187) e dall'UCSD Biological Use Authorization (BUA #R2401).
1. gRNA e progettazione di plasmidi donatori
- Per creare mutanti knockout, progettare due gRNA distanziati di ~20-100 bp (Figura 1A).
- Progettare gRNA da 20 bp per CRISPR-Cas9, escludendo la sequenza PAM (NGG) utilizzando uno strumento online (Figura 1A), come CHOPCHOP ( https://chopchop.cbu.uib.no/)17o Benchling (benchling.com) o CRISPOR (http://crispor.gi.ucsc.edu/)18. Seleziona i gRNA più specifici e privi di bersagli per gli esperimenti, come suggerito dallo strumento online.
- Assicurarsi che un sito di taglio dell'enzima di restrizione efficiente sia incluso nella sequenza tra i siti di taglio del gRNA (Figura 1A). Includere un sito enzimatico di restrizione tra i siti di taglio del gRNA per consentire una rapida conferma visiva delle modifiche riuscite.
NOTA: Quando si verifica la delezione, il sito di restrizione viene rimosso, quindi l'enzima non taglierà la sequenza, producendo una singola banda non tagliata su un gel per confermare la delezione.
- Per gli inserimenti di cassette geniche mediate da HDR, progettare più gRNA utilizzando gli strumenti menzionati per creare mutanti knockout e selezionare quello più efficace dopo una valutazione successiva.
- Progettare plasmidi donatori per la riparazione diretta dall'omologia (HDR) contenenti bracci di omologia del sito bersaglio, la sequenza del DNA cargo, un marcatore fluorescente, l'origine della replicazione e un gene di resistenza agli antibiotici (Figura 1B).
- Scegliere i bracci di omologia dalle regioni a monte e a valle del sito target, ciascuno dei quali si estende a 500-1.000 bp10 (Figura 1B).
- Seleziona la sequenza del carico, che può includere un marcatore fluorescente, un gene di interesse o un elemento regolatorio (ad esempio, QF2).
- Seleziona un marcatore fluorescente. I marcatori fluorescenti comuni utilizzati per A. aegypti sono inclusi in una cassetta di DNA contenente un promotore, un gene codificante un marcatore fluorescente e una sequenza UTR 3'. Per ulteriori dettagli, vedere la discussione.
2. Preparazione della miscela per iniezione
- Diluire la proteina Cas9 con il tampone di diluizione Cas9 a 1 μg/μL.
NOTA: Non scongelare e congelare Cas9 più di 2 volte. Si consiglia di fare aliquote della proteina Cas9. - Acquista i gRNA o producili internamente.
NOTA: Per la produzione interna, utilizzare pratiche standard di decontaminazione dell'RNA e materiali privi di RNAsi.- Progettare 4-6 gRNA e scegliere i due migliori gRNA per l'iniezione (vedere il saggio di scissione in vitro di seguito).
- Progettare un primer diretto che includa la sequenza del promotore T7 a monte della sequenza di gRNA. Utilizzare il primer inverso universale al gRNA per le reazioni PCR non stampo, come descritto di seguito. La base della sequenza sovrapposta si accoppia alla sequenza corrispondente nel primer inverso universale (grassetto e sottolineato), creando un modello per la DNA polimerasi per amplificare le loro sequenze.
NOTA: Per queste reazioni di PCR, il primer diretto deve contenere il promotore T7 (grassetto), seguito da due guanine (importanti per l'inizio della trascrizione tramite l'RNA polimerasi T7), la sequenza di 20 nucleotidi del gRNA (N20; senza la sequenza PAM) e la sequenza di sovrapposizione del primer (sottolineata).
Innesco in avanti: 5'- GAAATTAATACGACTCACTATAGGN20 GTTTTAGAGCTAGAAATAGC- 3'
Innesco inverso: 5''-AAAAGCACCGACTCGGTGCCACTTTTT CAAGTTGATAACGGACTAGC
CTTATT TTAACTT GCTATTTCTAGCTCTAAAAC - 3' - Sintetizzare il modello di DNA mediante PCR non modello. Impostare reazioni multiple (ciascuna contenente 12,5 μl della miscela master 2x, 1,25 μl della soluzione 10 μM di primer diretto, 1,25 μl della soluzione 10 μM di primer inverso e 10 μl di acqua ultrapura) in modo che sia disponibile una quantità sufficiente di prodotto PCR (300 ng) per la reazione di trascrizione in vitro . Utilizzare le seguenti condizioni PCR: denaturazione iniziale per 30 s a 98 °C; 35 cicli di amplificazione per 10 s a 98 °C, 10 s a 62 °C e 10 s a 72 °C; prolungamento finale a 72 °C per 2 min; e stoccaggio a 4 °C.
- Confermare l'amplificazione di un singolo frammento di DNA (122 paia di basi) su un gel di agarosio (2%).
- Pulisci il modello PCR utilizzando un kit di purificazione PCR, seguendo le raccomandazioni del produttore.
- Eseguire una reazione di trascrizione in vitro con un kit di trascrizione T7. Miscelare 2 μL di tampone di reazione 10x, 2 μL ciascuno di nucleotidi (ATP, CTP, UTP e GTP), 2 μL di RNA polimerasi T7, 3 μL di DNA stampo (100 ng/μL) e 5 μL di acqua ultrapura. Incubare la miscela a 37 °C per almeno 2 ore (non più di 16 ore) fino a una notte (12 ore).
NOTA: In questa reazione, l'RNA polimerasi T7 si lega al promotore T7 incluso nella sequenza di primer in avanti, che porta alla trascrizione del gRNA. - Trattare la reazione di trascrizione con DNasi aggiungendo 1 μL di DNasi (mescolare bene) e incubando a 37 °C per 15 minuti.
- Purificare gli sgRNA sintetizzati con un kit di pulizia della trascrizione seguendo le raccomandazioni del produttore.
- Eseguire un saggio di scissione in vitro per valutare l'efficienza di taglio dei gRNA selezionati (Figura 1C).
- Progettare primer per amplificare un frammento di DNA (500-1.000 bp) che fiancheggia il sito di taglio del gRNA.
- Impostare le reazioni PCR come segue: 12,5 μL della miscela master 2x, 1,25 μL della soluzione 10 μM di primer diretto, 1,25 μL della soluzione 10 μM di primer inverso, 9 μL di acqua ultrapura e 1 μL di DNA stampo da 5 ng/μL.
- Utilizzare le seguenti condizioni PCR: condizioni: denaturazione iniziale per 30 s a 98 °C; 35 cicli di amplificazione per 10 s a 98 °C, 15-30 s a X °C (temperatura di ricottura ideale per i primer) e 35 s a 72 °C; prolungamento finale a 72 °C per 2 min; e stoccaggio finale a lungo termine a 4 °C.
- Fare riferimento alle linee guida del produttore per il miglior amptemperature di amplificazione e intervallo di tempo. Raggruppare almeno cinque reazioni o aumentare i volumi dei reagenti in modo da ottenere una quantità sufficiente di prodotto PCR (1,5-2 μg) dopo la pulizia PCR (banda singola) o la purificazione della banda PCR su gel di agarosio.
- Impostare le reazioni di scissione di Cas9 con Cas9 ricombinante incubando la seguente miscela a 37 °C per 1 ora: 1 μL di tampone di reazione Cas9 10x, 0,35 μL di Cas9 ricombinante (1 μg/μL), 1 μL di gRNA (100 ng/μL), 6,65 μL di acqua ultrapura e 1 μL di 300 ng/μL di DNA stampo.
- Impostare una reazione di controllo negativo senza alcun gRNA.
- Verificare l'efficienza della scissione eseguendo reazioni su un gel di agarosio (1,5-2,0%; Figura 1C).
3. Assemblaggio del plasmide donatore
- PCR-amplificare i bracci di omologia dal DNA genomico di A. aegypti.
- Per la progettazione del primer, fare riferimento alle linee guida del produttore del kit di clonazione. Utilizzare uno strumento online (ad esempio, Benchling), che fornisce supporto per la progettazione e l'assemblaggio di plasmidi utilizzando diverse strategie di clonazione (ad esempio, Gibson, Golden Gate e clonazione basata su enzimi di restrizione).
- Amplifica la sequenza cargo dal DNA genomico di A. aegypti o ordina un frammento genico sintetizzato commercialmente.
- Amplifica internamente i marcatori fluorescenti dei plasmidi.
- Frammenti di DNA ligate dai passaggi 3.1.1 a 3.1.3 nella spina dorsale di un plasmide esistente mediante assemblaggio di Gibson. Incubare una miscela di 10 μL di 2x master mix, X μL di tutti i frammenti di DNA, 10-X μL di acqua ultrapura a 50 °C per 1 ora.
NOTA: Fare riferimento alle linee guida del produttore per il calcolo del rapporto ideale tra inserti e dorsale plasmidica.- Calcola le concentrazioni di ogni frammento di clonazione in pico mol: pmol = (peso in ng) × 1.000/(paia di basi × 650 dalton).
- Utilizzare 50-100 ng di spina dorsale plasmidica e un eccesso molare di 2-3 volte di ciascun inserto.
- Se si assemblano 2-3 frammenti, aggiungere 0,02-0,5 pmol di ciascun frammento nella reazione di Gibson. Se si assemblano 4-6 frammenti, aggiungere 0,2-1,0 pmol di ciascuno.
- Utilizzare 3-5 μl della reazione di assemblaggio di Gibson per trasformare le cellule competenti di E. coli JM109.
NOTA: JM109 è stato scelto per l'assemblaggio di Gibson grazie al suo genotipo recA- , che riduce gli eventi di ricombinazione indesiderati e previene il carryover della nucleasi durante la raccolta delle cellule, garantendo l'integrità dei frammenti di DNA assemblati
Per plasmidi di dimensioni superiori a 10 kb, si consiglia la trasformazione di cellule competenti per DH5α utilizzando il protocollo esteso, seguendo le raccomandazioni del produttore. - Espandi i batteri trasformati e purifica il plasmide utilizzando un kit di miniprep.
- Per confermare il corretto assemblaggio del plasmide, eseguire un digest dell'enzima di restrizione diagnostica con il DNA plasmidico purificato e visualizzare mediante elettroforesi su gel di agarosio (Figura 1D, E).
NOTA: Suggeriamo l'uso di uno strumento online per la selezione di un paio di enzimi di restrizione in grado di tagliare il plasmide su un singolo sito. Software come Benchling hanno uno strumento integrato che esegue la digestione virtuale delle sequenze plasmidiche con gli enzimi di restrizione selezionati, visualizzando il modello di bande di DNA previsto su un gel di elettroforesi virtuale (Figura 1D).- Effettuare la digestione dell'enzima di restrizione plasmidica. Incubare una miscela di 1 μL di tampone digest di restrizione 10x, 0,5 μL di enzima di restrizione 1, 0,5 μL di enzima di restrizione 2, 1 μL di DNA plasmidico (300 ng/mL) e 7 μL di acqua ultrapura a 37 °C per 2 ore.
- Eseguire le reazioni digest di restrizione su un gel di agarosio (1,5%).
NOTA: Il modello di banda del DNA (Figura 1E) dovrebbe assomigliare al modello di banda del digest virtuale (Figura 1D). I plasmidi possono anche essere inviati per il sequenziamento dell'intero plasmide, se il servizio è disponibile.
- Coltura del clone plasmidico in 150 mL di terreno LB.
- Eseguire il plasmide maxiprep, seguendo le linee guida del produttore.
- Sospendi il plasmide in acqua ultrapura.
4. Miscelazione del costrutto di iniezione
NOTA: Gli intervalli di concentrazione finali suggeriti per ciascun costrutto sono forniti nella Tabella 1. Inizia con un rapporto di 2:1:2 (ng Cas9: ng sgRNA: ng plasmide donatore) e regola se necessario per ottimizzare l'efficienza HDR. Il monitoraggio dei risultati aiuterà a identificare la combinazione più efficace.
- Per fare mutanti knockout,
- Diluire l'aliquota della proteina Cas9 alla concentrazione desiderata utilizzando il tampone di diluizione Cas9 e diluire l'aliquota di gRNA con acqua ultrapura.
- Premiscelare la proteina Cas9 con ciascun gRNA per formare un complesso ribonucleoproteico (RNP). Unire le soluzioni premiscelate.
- Per l'inserimento di cassette geniche mediate da HDR,
- Diluire e mescolare la proteina Cas9 e i gRNA come suggerito per la produzione di mutanti knockout.
- Diluire il plasmide donatore con acqua ultrapura.
- Combina tutti i costrutti.
5. Estrazione e caricamento degli aghi per microiniezione
- Utilizzare il filamento di quarzo per tirare gli aghi di iniezione (Figura 2). Assicurati che il filamento sia lungo 10 cm, con un diametro esterno di 1 mm e un diametro interno di 0,7 mm.
- Tirare gli aghi utilizzando un estrattore laser per micropipette con uno dei due programmi seguenti:
Programma 1: Calore 805, Filamento 4, Velocità 50, Ritardo 145, Tiro 145
Programma 2: Calore 650, Filamento 4, Velocità 40, Ritardo 150, Tiro 156
NOTA: Il programma 1 si traduce in una punta dell'ago più sottile ma più lunga, che lo rende adatto per iniezioni con costrutti a concentrazione inferiore, dove una punta più fine è vantaggiosa per la precisione. Il programma 2 produce una punta dell'ago più corta ma più spessa, ideale per iniezioni ad alta concentrazione, poiché il costrutto è più spesso e richiede un ago più robusto.
6. Prelievo di embrioni
- Preparare un aspiratore elettrico o manuale per raccogliere le zanzare e utilizzare fiale di plastica per la raccolta e la raccolta degli embrioni.
- Inumidire le carte da filtro bianche circolari e posizionarle sulla parete interna o sopra il cotone umido nel raccoglitore.
- Metti 5-10 zanzare femmine che sono state nutrite con sangue 5-10 giorni fa nel raccoglitore. Posizionare il raccoglitore al buio per 45 min.
- Estrarre la carta da filtro per la raccolta degli embrioni.
7. Allineamento degli embrioni
- Utilizzare un ceppo wildtype (Liverpool) di A. aegypti per l'iniezione di embrioni.
- Selezionare gli embrioni allo stadio di preblastoderma, in particolare quelli di colore grigio chiaro, dalla carta da raccolta (Figura 3).
- Trasferisci alcuni embrioni con un pennello bagnato sul nastro biadesivo posizionato sopra un vetrino coprioggetti. Allinea gli embrioni in parallelo mentre i loro bordi sono ancora bagnati assicurandoti che siano fianco a fianco, con tutte le estremità posteriori rivolte in avanti.
- Una volta posizionato correttamente, lasciare asciugare leggermente l'ambiente per fissare gli embrioni in posizione.
- Aggiungere l'olio di alocarburi 700 sugli embrioni durante l'allineamento per evitare l'essiccazione.
NOTA: Assicurati che gli embrioni non siano circondati dall'acqua quando aggiungi l'olio, poiché ciò farebbe galleggiare gli embrioni nell'olio.
8. Microiniezione di embrioni
NOTA: L'iniezione viene condotta a temperatura ambiente o a 18 °C. La temperatura di 18°C è consigliata per motivi pratici, in quanto ritarda lo sviluppo dell'embrione.
- Impostare un microiniettore con i seguenti parametri: pressione di compensazione (Pc) 300 hPa, pressione di iniezione (Pi) 500 hPa. Regolare queste condizioni ogni volta che è necessario.
- Caricare 3 μl del costrutto misto in un ago utilizzando un microcaricatore.
- Posizionare il vetro di copertura con gli embrioni allineati sopra un vetrino da microscopio (Figura 3). Posizionare il vetro di copertura e farlo scorrere sotto il microscopio per l'iniezione. Assicurati che l'estremità posteriore dell'embrione sia posizionata verso l'ago.
- Posizionare un ago in un portaaghi insieme a un micromanipolatore e posizionare l'ago con un angolo di 10° verso l'estremità posteriore degli embrioni allineati, mantenendolo fermo (Figura 4A). Aprire delicatamente l'ago al microscopio e toccarlo leggermente con il bordo di un vetrino coprioggetti.
NOTA: Un'altra opzione è quella di creare una piccola apertura nell'ago picchiettandolo con un embrione. Tuttavia, si consiglia un'apertura dell'ago leggermente più ampia, poiché la viscosità della proteina Cas9 può ostruire l'ago. - Spostare il vetrino del vetrino verso l'ago per l'iniezione (Figura 5).
9. Cura dell'embrione iniettato
- Usa salviette usa e getta prive di lanugine per rimuovere l'olio che circonda gli embrioni.
- Aggiungere acqua deionizzata per sciacquare gli embrioni e spostare gli embrioni su una carta da filtro bagnata. Posizionare la carta da filtro bagnata su un fazzoletto umido all'interno di tazze Karat da 9 once (Figura 4B e Figura 6A, B).
- Posizionare del cotone bagnato sul fondo della tazza per mantenere l'umidità (Figura 6B).
- Conservare gli embrioni iniettati su carta da filtro bagnata.
10. Schiusa degli embrioni e screening delle larve G0
- Almeno 4 giorni dopo l'iniezione, trasferire la carta da filtro con gli embrioni in circa 3 L di acqua deionizzata in pentole Sterilite da 6 quarti per la schiusa.
NOTA: Le uova in genere si schiudono in modo più efficiente entro 2 settimane dall'iniezione. Assicurati che le uova rimangano umide durante questo periodo. La carta da filtro non deve essere troppo bagnata, poiché un'umidità eccessiva può portare a una schiusa precoce degli embrioni. Sebbene una conservazione più lunga possa migliorare i tassi di schiusa, non superare il mese per ottenere risultati ottimali. L'uso di acqua deossigenata può anche aiutare con la schiusa. - Una volta che le larve G0 si schiudono, aggiungi cibo per pesci mescolato con acqua nella padella come cibo.
- Esaminare le larve G0 per il marcatore fluorescente allo stadio larvale dal 3° al 4° stadio (Figura 7).
- Mantenere le larve separatamente in base al loro stato di fluorescenza, con le larve fluorescenti positive e fluorescenti negative tenute in vasche separate.
11. Selezione del sesso delle pupe e incrocio con il tipo selvatico
- Separare le zanzare iniettate in base al sesso quando si impupano, utilizzando le dimensioni e le strutture specifiche del sesso nel lobo genitale per l'identificazione:
- Maschi: di dimensioni più piccole, un lobo genitale più prominente e appuntito e pale più larghe (strutture all'estremità della coda delle pupe; Figura 8A 1,A2).
- Femmine: dimensioni maggiori, lobo genitale più piccolo e meno pronunciato e pale più strette (Figura 8B1, B2).
- Pool di zanzare fluorescenti positive o fluorescenti negative di ogni sesso.
- Incrocia le zanzare raggruppate con zanzare del sesso opposto del ceppo Liverpool. Utilizzare un rapporto da 3:1 a 5:1 tra zanzara selvatica e zanzara G0.
- Incrocia le zanzare per 4 giorni.
- Fornisci alle femmine un pasto di sangue dopo l'attraversamento.
12. Vagliatura G1
- Tre giorni dopo l'alimentazione del sangue, fornire portauova posizionando un tovagliolo di carta all'interno del muro di tazze da 9 once di Karat e aggiungendo circa 3 once di acqua deionizzata.
- Dopo 3-4 giorni, raccogli e schiudi gli embrioni G1. Screening delle larve G1 per il marcatore fluorescente al 3° e 4° stadio (Figura 7).
- Raccogli le larve fluorescenti, suggerendo un inserimento HDR. Quando gli individui fluorescenti G1 raggiungono lo stadio pupale, separali per sesso e metti ogni sesso in gabbie separate.
- Incrocia le zanzare fluorescenti G1 con individui del sesso opposto del ceppo Liverpool.
13. Conferma del sito di inserimento G1
- Dopo aver fornito il pasto di sangue, impostare le femmine G1 per la deposizione di un singolo ovulo femminile mettendo un piccolo pezzo di carta assorbente in singole fiale di Drosophila e aggiungendo 3 ml di acqua deionizzata.
- Per i mutanti knockout:
- Dopo la deposizione delle uova, l'acqua evapora e il tovagliolo di carta si asciuga, schiude le uova dalle femmine che mostrano chiaramente mutazioni knockout e ottengono la generazione G2.
- Raccogli il corpo delle madri G1 che hanno schiuso con successo la prole G2 ed estrai il DNA.
- Utilizzare il DNA come modello per la PCR per amplificare la sequenza che copre il sito target. Assicurati che i primer amplifichino un frammento di DNA di ~200 bp. Impostare le reazioni (ciascuna contenente 12,5 μl della miscela master 2x, 1,25 μl della soluzione 10 μM di primer diretto, 1,25 μl della soluzione 10 μM di primer inverso, 9 μl di acqua ultrapura e 1 μl di DNA stampo [5 ng/μl]). Utilizzare le seguenti condizioni PCR: denaturazione iniziale per 30 s a 98 °C; 35 cicli di amplificazione per 10 s a 98 °C, 15-30 s a X °C (temperatura di ricottura ideale dei primer) e 10 s a 72 °C; prolungamento finale a 72 °C per 2 min; e stoccaggio finale a lungo termine a 4 °C.
- Purificare i frammenti di PCR ed eseguire la digestione con l'enzima di restrizione dei frammenti di PCR incubando una miscela di 1 μL di tampone digest di restrizione 10x, 0,5 μL di enzima di restrizione 1, 0,5 μL di enzima di restrizione 2, 1 μL di frammento PCR (200 ng/mL) e 7 μL di acqua ultrapura a 37 °C per 30 minuti.
NOTA: Alcuni enzimi di restrizione possono essere utilizzati nella miscela PCR senza previa purificazione. - Visualizzare la digestione dei frammenti PCR mediante elettroforesi su gel per confermare se si è verificata la scissione.
- Sequenziare il frammento di PCR non digerito per identificare le mutazioni knockout.
- Per gli inserimenti mediati da HDR, dopo la deposizione delle uova:
- Raccogli il corpo delle madri G1 ed estrai il DNA.
- Utilizzare il DNA come modello per la PCR per amplificare la sequenza che copre il sito target.
- Eseguire il sequenziamento del frammento di DNA amplificato per confermare l'integrità della cassetta di DNA inserita.
- Schiudi l'uovo e ottieni la generazione G2.
14. Ampliamento delle nuove linee CRISPR
- Per le linee mutanti knockout:
- Prepara le femmine G2 per la deposizione delle uova, con 20 femmine per G1. Ogni G1 rappresenta una riga.
- Dopo la deposizione delle uova, confermare le mutazioni knockout allo stesso modo di G1.
- Procedere con cinque linee G1 che mostrano mutazioni chiaramente identificate sia in G1 che in G2. Schiudi le uova raccolte da G2 di ogni linea, dando origine alla generazione G3.
- Dalle cinque linee incrociate, selezionare due linee con mutazioni chiaramente identificate e alta fitness o il fenotipo desiderato.
- Incrocia le femmine G3 di ogni linea selezionata con i maschi del ceppo Liverpool.
- Prepara 50 femmine singole per la deposizione delle uova da ogni linea. Confermare le mutazioni knockout come fatto in precedenza per G1.
NOTA: Aumenta il numero di femmine nella configurazione per mantenere la diversità. - Ripetere le procedure per altre due generazioni con le due linee (Figura 4J).
- Per le nuove linee di inserimento mediate da HDR:
- Verificare la presenza di individui fluorescenti nella generazione G2.
- Incrocia le zanzare G2, di ogni individuo G1, con il ceppo Liverpool. Ogni individuo G1 rappresenta una linea.
- Procedi con altre tre generazioni di outcrossing. Controllare continuamente il marcatore fluorescente e osservare la fitness19 di ciascuna linea (Figura 4x).
- Selezionare una linea con inserzioni corrette e alta fitness e il fenotipo desiderato.
15. Crea linee omozigoti
- Per le linee mutanti knockout:
- Generazione G6:
- Schiudi le uova prodotte da G5 e ottieni la generazione G6.
- Incrociare gli adulti G6 all'interno delle rispettive linee (Figura 4K e Figura 9A).
- Prepara 50 femmine G6 singole per la deposizione delle uova per linea e fai schiudere le uova come descritto in precedenza (Figura 9B).
- Raccogliere le femmine G6 che hanno schiuso con successo la prole G7, estrarre il DNA ed eseguire la PCR e la digestione con enzimi di restrizione come fatto per G1 (Figura 9C).
NOTA: In questa fase è possibile utilizzare un tampone di schiacciamento (SB) semplice ed economico per l'estrazione del DNA.- Utilizzando un macinatore portatile, macerare una singola zanzara in 100 μl di 1x SB (10 mM Tris-HCl pH 8, 1 mM EDTA e 25 mM NaCl). Aggiungere la proteinasi K (200 μg/mL) e incubare a 37 °C per 30 minuti, quindi a 95 °C per 2 minuti. Centrifugare a 10.000 × g per 5 minuti e raccogliere il surnatante20.
- Confermare la mutazione visualizzando i risultati su un gel.
- Scartare le uova prodotte dalle femmine senza mutazioni (Figura 9D).
- Mantenere le larve delle femmine con mutazioni confermate per stabilire diversi lignaggi G7 e scartare le covate senza mutazioni (Figura 9D).
- Generazione G7:
- Incrociare femmine e maschi all'interno di ciascun lignaggio G7 (Figura 9E).
- Prepara 10 femmine G7 singole per la deposizione delle uova per linea e schiudi le uova come descritto in precedenza (Figura 9F).
- Raccogli le femmine G7 che hanno schiuso con successo la prole G7, estrai il DNA ed esegui la PCR e la digestione con enzimi di restrizione (Figura 9G).
- Rileva le mutazioni knockout su entrambi gli alleli o su un singolo allele visualizzando i risultati su un gel.
- Mantenere le larve delle femmine G7 che hanno confermato di avere knockout in entrambi gli alleli, producendo la generazione G8 (Figura 9H). Scartare le covate senza mutazioni (Figura 9H).
NOTA: Questo passaggio garantisce che le femmine siano omozigoti. In G8, testare l'omozigosi della progenie conferma se anche i padri sono omozigoti".
- Generazione G8:
- Incrociare gli adulti G8 per ogni lignaggio femminile G7 (Figura 9I).
- Scegli a caso cinque maschi G8, estrai il DNA ed esegui la PCR e la digestione con enzimi di restrizione (Figura 9J).
- Fornire un pasto di sangue e far schiudere le uova dalle femmine G8 che sono state incrociate con maschi G8 che hanno confermato di avere knockout in entrambi gli alleli.
- Continua con i lignaggi G7 in cui tutti i maschi rilevati hanno knockout in entrambi gli alleli, indicando che questi lignaggi sono omozigoti.
- Generazione G6:
- Per le linee di inserimento mediate da HDR:
- Schiudi le uova prodotte da G5 e ottieni la generazione G6. Screening delle larve allo stadio G6 per marcatori fluorescenti.
- Scartare le larve senza marcatori fluorescenti (Figura 4xi).
- Zanzare incrociate che mostrano marcatori fluorescenti.
- Continuare questa procedura per ogni generazione successiva fino a quando non si osservano larve non fluorescenti. A questo punto, la linea della zanzara sarà omozigote.
Risultati
Progettazione e validazione del gene targeting mediato da gRNA per la ricombinazione di omologia HDR
Per garantire che il gene desiderato sia mirato in modo accurato, si consiglia di selezionare un paio di gRNA e di posizionare i bracci di omologia 5' e 3' vicino al sito di taglio per la ricombinazione omologa mediata da HDR (Figura 1A). Ad esempio, abbiamo progettato due gRNA per colpire entrambi i lati del codone iniziale del gene di interesse e abbiamo utilizzato la cassetta QF2-Hsp70-OpIE2-ECFP-SV40 come marcatore, inserendola nel gene bersaglio tramite bracci di omologia da 1 kb attraverso il meccanismo HDR (Figura 1B). Le rotture a doppio filamento portano alla delezione di un breve frammento di DNA tra i siti di taglio dei gRNA (Figura 1A).
L'efficienza di scissione dei gRNA e la loro capacità di tagliare il gene bersaglio sono state testate mediante saggio di scissione Cas9 in vitro . I frammenti di PCR del gene bersaglio sono stati scissi in due frammenti da Cas9, caricati con i gRNA (Figura 1C). Inoltre, gli enzimi di restrizione possono essere utilizzati per verificare l'integrità del plasmide donatore prima di passare alle fasi a valle del protocollo. I risultati della digestione plasmidica dovrebbero mostrare una o due bande sul gel, con la presenza di una piccola banda che indica il successo della digestione in un breve frammento (Figura 1D, E).
Preparazione degli aghi per microiniezione
Prima di iniziare il processo di iniezione, è importante preparare nuovi aghi per microiniezione. Gli aghi devono avere una forma sporgente con una leggera curvatura. Circa 2/3 dell'ago dovrebbero essere stretti nella parte superiore, mentre il restante 1/3 più largo. L'estremità anteriore della punta dell'ago non deve superare i 0,5 cm di lunghezza dalla parte stretta a quella larga (Figura 2A). Dopo aver utilizzato il bordo del vetrino coprioggetti per aprire l'ago, per la microiniezione è preferibile un ago con punta smussata, poiché è più facile da usare dopo aver rotto l'ago toccando delicatamente la superficie dell'embrione di zanzara nera (Figura 2B). D'altra parte, un ago a punta smussata può rendere difficile perforare gli embrioni grigio chiaro durante la microiniezione (Figura 2C). Gli aghi non devono essere né troppo morbidi né troppo duri per penetrare efficacemente negli embrioni di zanzara per evitare di rompere l'ago.
Iniezione di embrioni
Per garantire il successo dell'iniezione, iniziare raccogliendo embrioni di zanzara grigio chiaro e allineandoli in modo che il polo posteriore sia rivolto nella stessa direzione (Figura 3A). Questo allineamento direzionale aiuta a iniettare il plasmide vicino alle cellule germinali (polo posteriore), che è fondamentale per la produzione di mutanti che possono essere ereditati dalla generazione successiva. Selezionare embrioni freschi, uniformi e di colore grigio chiaro da femmine che sono state nutrite con sangue 5-10 giorni prima (Figura 3A1). Evitare di utilizzare embrioni di colore grigio o marrone in modo non uniforme (Figura 3A2), poiché potrebbero non svilupparsi correttamente.
Allinea gli embrioni di zanzara uno per uno sul nastro biadesivo per facilitare un'iniezione accurata ed efficiente. Applicare olio di alocarburi prima che le uova si secchino e si scuriscano in un colore grigio medio o scuro (Figura 3B).
Durante l'iniezione, concentrarsi sulla superficie del polo posteriore di ciascun embrione. Il lato anteriore dell'embrione (lato destro nella Figura 5), invece, presenta una superficie piana e dura (micropilo). Utilizzare delicatamente un ago smussato contenente la miscela per iniezione per perforare l'embrione (Figura 5). La superficie dell'embrione si deprime e rimbalza, indicando che l'ago è penetrato nell'embrione. Una volta completata l'iniezione, il colore dell'embrione si schiarisce in pochi secondi (curva gialla nella Figura 5, in alto a destra).
Dopo l'iniezione, utilizzare con cautela lo spazzolino per raccogliere e trasferire gli embrioni su un'altra carta da filtro bagnata utilizzando un panno da laboratorio per rimuovere l'olio in eccesso (Figura 6A). Conservare gli embrioni su un tovagliolo di carta bagnato per diversi giorni alla temperatura di allevamento (Figura 6B). L'eccesso di olio può ridurre il tasso di schiusa degli embrioni di zanzara. Usa batuffoli di cotone con acqua ultra pura per mantenere l'idratazione. È improbabile che gli embrioni che rimangono grigi o marroni (freccia gialla) dopo 2 ore dall'iniezione si schiudano (Figura 6C).
Schermatura e outcrossing
Una volta che gli embrioni sono maturati, aggiungere acqua ultrapura al portauovo e metterlo in un'incubatrice a 27 °C per la schiusa. In genere ci vogliono 5-7 giorni perché le larve si sviluppino fino al 3° stadio di stadio. Inizialmente, abbiamo preselezionato le larve G0 che mostravano fluorescenza, sebbene fosse più debole o parziale rispetto ad altre linee mutanti. Ciò ha garantito che il marcatore fosse espresso correttamente e che i plasmidi fossero iniettati con successo negli embrioni.
Queste larve sono state poi incrociate con zanzare di tipo selvatico (WT). Durante gli screening successivi, abbiamo selezionato e conservato le larve G1 che mostravano un'espressione di fluorescenza più forte e completa per l'allevamento, l'espansione del line stock e la conduzione di ulteriori esperimenti. Le larve fluorescenti G1 possono essere identificate in base al pattern di espressione della proteina fluorescente fornito dalla combinazione di promotore reporter e proteina fluorescente: il promotore Hr5Ie1-eGFP (Figura 7A) guida l'espressione della proteina fluorescente verde in tutto il corpo; Hr5Ie1-DsRed (Figura 7B) porta all'espressione della fluorescenza rossa nel corpo; OpIE2-CFP (Figura 7C) induce l'espressione della proteina di fluorescenza ciano (blu) solo in una parte specifica del corpo. Quando si utilizza la combinazione 3xP3-TdTomato, la proteina fluorescente rossa è espressa in modo specifico negli occhi (Figura 7D). Questa specifica linea di zanzara transgenica contiene anche il promotore OpIE2 che guida l'espressione di CFP in tutto il corpo (Figura 7D).
Processo speciale di insediamento omozigote per mutanti knockout
Per semplificare la manutenzione della linea e garantire che tutte le zanzare siano mutanti, abbiamo implementato una pipeline per generare zanzare mutanti omozigoti non fluorescenti. Come descritto nella fase 15 del protocollo, dopo l'incrocio con le zanzare WT, abbiamo posizionato le zanzare mutanti G6 nella stessa gabbia per l'incrocio (Figura 9A). Dopo l'incrocio, le femmine sono state raccolte in fiale individuali per la deposizione delle uova (Figura 9B). Questo passaggio ha permesso la separazione di femmine con genotipi diversi.
Dopo aver raccolto gli ovuli per ottenere G7, il DNA genomico è stato estratto dalle femmine G6, seguito da PCR ed elettroforesi su gel di frammenti di PCR, trattati o non trattati con un enzima di restrizione (Figura 9C). Le righe che mostravano solo frammenti di PCR digeriti, indicando l'assenza di mutazioni, sono state scartate. Mentre quelle linee che mostravano bande a doppia PCR, indicanti la presenza di mutazioni e, a loro volta, femmine eterozigoti, sono state mantenute e covate (Figura 9D). Le zanzare G7 sono state quindi lasciate maturare e tutti gli adulti sono stati messi nella stessa gabbia per un altro giro di incroci per aumentare la probabilità di stabilire una linea omozigote (Figura 9E). Le femmine G7 alimentate con sangue sono state nuovamente raccolte individualmente per la raccolta degli ovuli G8, rappresentando tre potenziali genotipi (Figura 9F). Il DNA genomico è stato estratto dalle femmine G7 ed è stata eseguita la PCR seguita da elettroforesi su gel per determinare quali fossero omozigoti (Figura 9G). Le uova di femmine non mutanti o eterozigoti sono state scartate e sono state conservate solo quelle con risultati omozigoti (Figura 9H). Le uova G8 della fase H sono state covate e gli adulti risultanti sono stati incrociati in una gabbia separata (Figura 9I). Da ogni gabbia, 10 maschi adulti sono stati selezionati in modo casuale per l'elettroforesi su gel e il sequenziamento (Figura 9J). Questo passaggio dovrebbe produrre individui omozigoti, che sono stati poi mantenuti come ceppo (Figura 9K).
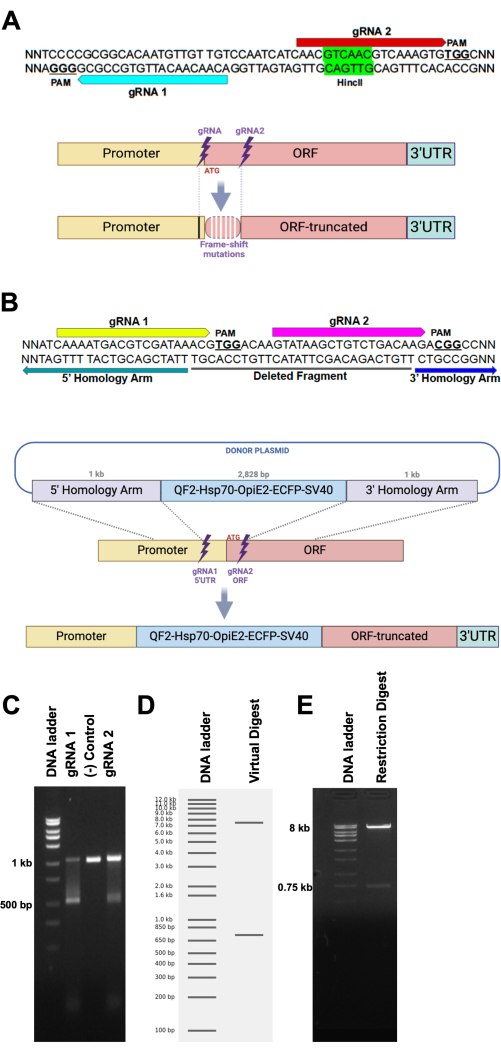
Figura 1: Schema del disegno del gRNA e del plasmide donatore HDR. (A, in alto) Sequenza di DNA ipotetica per due gRNA (frecce blu e rosse e i loro siti PAM associati insieme a un sito bersaglio dell'enzima di restrizione (HincII). (A, in basso) Rappresentazione schematica di un sito bersaglio sottoposto a NHEJ, che porta all'emergere di mutazioni frame-shift (barre verticali bianche) dovute a una piccola delezione o inserzione di sequenza. Le mutazioni possono estendersi oltre il sito di scissione dei gRNA (barra verticale nera). (B, in alto) Sequenza ipotetica di DNA che raffigura i siti bersaglio per il gRNA 1 (freccia gialla) e il gRNA 2 (freccia magenta), seguita dai siti PAM (NGG, grassetto e sottolineato). La sequenza di DNA potenzialmente eliminata tra i due gRNA è sottolineata da una barra nera (frammento eliminato). Le frecce verdi e blu evidenziano rispettivamente la sequenza più 3' del braccio di omologia 5' e la maggior parte della sequenza 5' del braccio di omologia 3'. (B, in basso) Rappresentazione schematica di una ricombinazione HDR di una cassetta di DNA da un plasmide a un sito omologo nel genoma. (C) Saggio di scissione in vitro . L'incubazione di un frammento di DNA di 1 kb viene digerito da Cas9 caricato con gRNA 1 (corsia sinistra) o gRNA 2 (corsia destra), ottenendo un frammento di 500 bp. La corsia centrale mostra la reazione di controllo negativo, che era priva di gRNA. (D) Digerire virtualmente un plasmide (8,75 kb) con due enzimi di restrizione single-cut, risultante in due bande (8 kb e 750 bp) su un ipotetico gel. (E) Un gel di agarosio caricato con la reazione di digestione di restrizione di un plasmide, raffigurante i risultati attesi forniti dal digest virtuale. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Forma dell'ago per microiniezione. (A) Immagine rappresentativa della punta di un ago. (B) Forma ideale della punta dell'ago per la microiniezione di uova di zanzara. (C) Forma della punta dell'ago che dovrebbe essere evitata. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Raccolta di embrioni. (A) Raccolta di embrioni grigio chiaro e (A1) omogenei ma non (A2) disomogenei per iniezioni; Barra della scala = 500 μm. (B) Immagine per gli embrioni allineati; Barra di scala=500 μm. Clicca qui per visualizzare una versione più grande di questa figura.
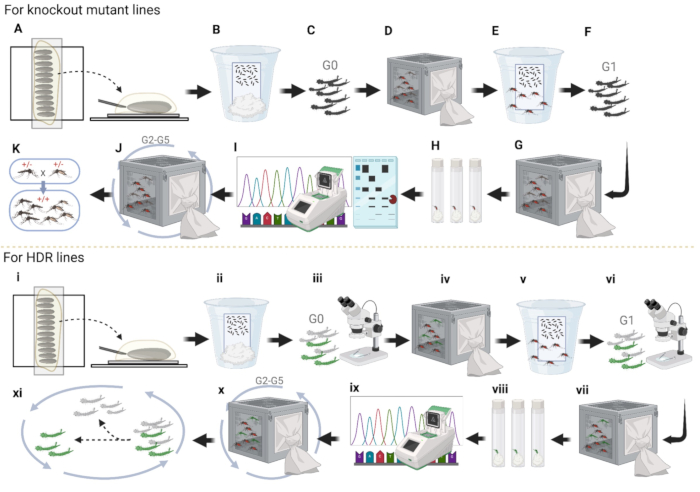
Figura 4: Pipeline di microiniezione di embrioni di zanzara. (A-K) Iniezioni di Knockout Line. (A) Allineare gli embrioni di zanzara e iniettare il plasmide nell'estremità posteriore. (B) Conservazione degli embrioni iniettati nella tazza con carta bagnata; (C) Larve G0 che si schiudono e si separano in entrambi i sessi. (D) entrambi i sessi di adulti che attraversano il WT in gabbie separate; (E) raccogliere le uova delle femmine nel portauovo. (F) Larve G1 che si schiudono e si separano in entrambi i sessi. (G) Entrambi i sessi degli adulti che attraversano il WT in gabbie separate. (H) Raccogliere le uova femminili G1 singolarmente in fiale di plastica e far schiudere le uova. (I) PCR, eseguire il gel con frammenti trattati con enzimi e sequenziamento per femmine singole G1 che hanno covato con successo le uova. (J) Incrociare G2 con WT per altre tre generazioni e sequenziare le zanzare nello stesso modo in cui è stato fatto per G1. (K) Stabilire omozigoti mediante incrocio. (i-xi) Iniezioni di linea HDR. (i) Allineamento degli embrioni. (ii) Conservazione degli embrioni iniettati nella tazza con carta bagnata. (iii) Prescreening delle larve G0 per la fluorescenza. (iv) Far crescere larve fluorescenti in adulti e incrociarle con WT in una gabbia. (v) Raccolta di uova G1 nel portauovo. (vi) Screening e selezione di larve fluorescenti G1. (vii) Adulti fluorescenti G1 incrociati con WT. (viii) Femmine adulte fluorescenti G1 alimentate con sangue che depongono le uova individualmente in fiale di plastica. (ix) Raccolta e sequenziamento di adulti fluorescenti G1 individualmente. (x) Incrocio con il tipo selvatico per altre tre generazioni aggiuntive e (xi) eliminazione delle larve senza fluorescenza per stabilire omozigoti. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: Iniezione di embrioni. Il momento dell'iniezione del plasmide negli embrioni di zanzara. Riquadro: miscela per iniezione visualizzata come un liquido torbido all'interno dell'embrione (linea tratteggiata gialla); Barra di scala=200 μm. Clicca qui per visualizzare una versione più grande di questa figura.
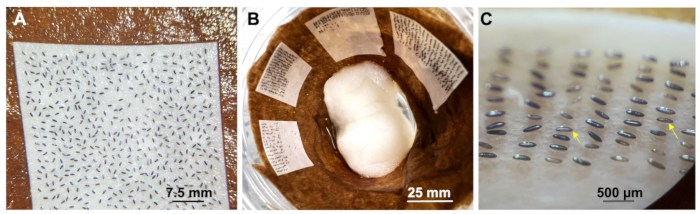
Figura 6: Raccolta di embrioni. (A) Raccolta di embrioni dopo l'iniezione e trasferimento su una carta da filtro bagnata. Barra della scala = 7,5 mm. (B) Mantenimento degli embrioni iniettati in una tazza. Barra della scala = 25 mm. (C) Embrioni scuri dopo l'iniezione; Barra di scala=500 μm. Clicca qui per visualizzare una versione più grande di questa figura.
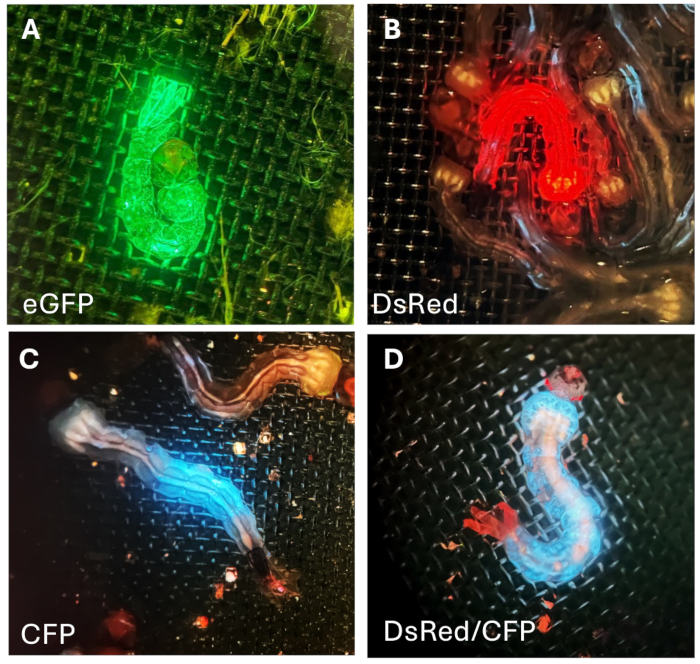
Figura 7: Fluorescenza nelle larve G1. (A) Proteine fluorescenti verdi, (B) rosse e (C) blusul corpo della larva di zanzara. (D) Larva che mostra fluorescenza rossa specifica per l'occhio e fluorescenza blu specifica per il corpo. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 8: Dimorfismo sessuale nella morfologia delle pupe. (A1,A2) Le pupe maschili sono caratterizzate da una corporatura più piccola, un lobo genitale prominente e appuntito e pale più larghe all'estremità della coda. (B1,B2) Le pupe femmine mostrano una dimensione corporea più grande, un lobo genitale più piccolo e meno pronunciato e pale posteriori più strette. Clicca qui per visualizzare una versione più grande di questa figura.
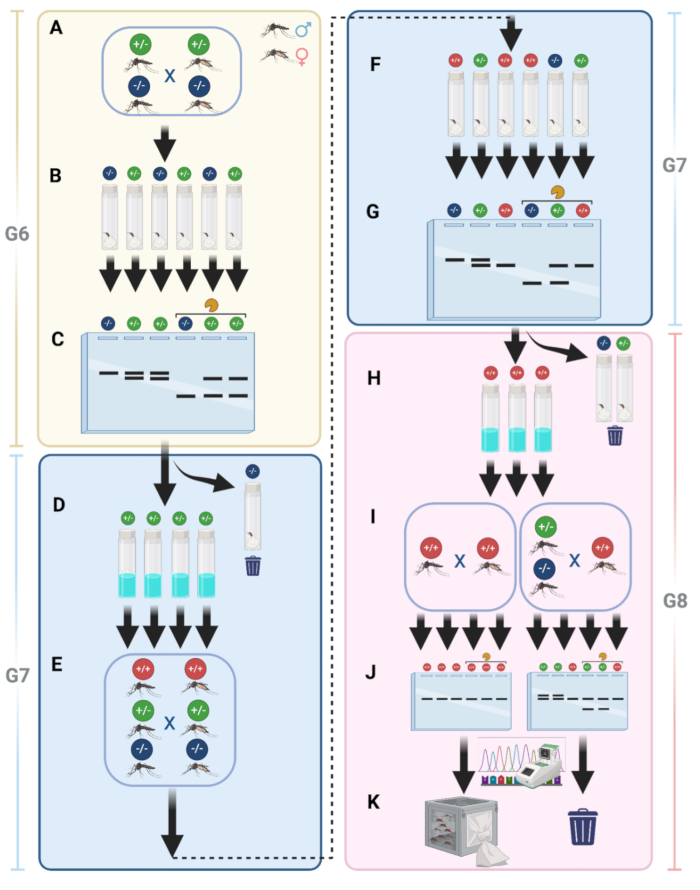
Figura 9: Dettagli dell'insediamento omozigote per le linee mutanti knockout. (A) Le zanzare mutanti vengono incrociate nella stessa gabbia dopo l'incrocio con WT. (B) Dopo l'incrocio, le singole femmine vengono raccolte in fiale per la deposizione delle uova. (C) Il DNA genomico viene estratto da femmine G6, seguito da PCR ed elettroforesi su gel con frammenti trattati con enzimi e non trattati. (D) Le uova G7 con risultati positivi alla PCR (eterozigoti) vengono schiuse. (E) Gli adulti G7 vengono nuovamente incrociati per aumentare le possibilità di ottenere individui omozigoti. (F) Le femmine G7 alimentate con sangue vengono raccolte individualmente per la raccolta degli ovuli G8, che rappresentano tre genotipi. (G) L'estrazione genomica del DNA, la PCR e l'elettroforesi su gel vengono eseguite su femmine G7 per identificare individui omozigoti. H) le uova di femmine non mutanti o eterozigoti siano scartate; Le uova omozigoti vengono conservate. (I) Le uova G8 sono covate e incrociate in una gabbia separata. (J) Dieci maschi adulti vengono selezionati casualmente da ogni gabbia per l'elettroforesi e il sequenziamento su gel. (K) Le zanzare omozigoti sono identificate e mantenute come ceppi. Clicca qui per visualizzare una versione più grande di questa figura.
| Costrutto di iniezione | Concentrazione finale |
| Proteina Cas9 | 100-300 ng/μl |
| gRNA | 50-100 ng/μl |
| plasmide donatore | 100-500 ng/μL |
Tabella 1: Gli intervalli di concentrazione finali suggeriti per ciascun costrutto di iniezione.
Discussione
La tecnologia CRISPR-Cas ha cambiato il panorama dell'editing del genoma promuovendo cambiamenti specifici del bersaglio nei cromosomi1. Anche se gli elementi trasponibili sono stati essenziali per la generazione delle prime zanzare transgeniche, i loro siti di inserzione sono in qualche modo casuali e l'espressione del costrutto di carico (promotore + gene) potrebbe non corrispondere al profilo di espressione del gene effettivo a causa di un effetto posizionale del genoma (cioè sito di inserzione), che di solito porta all'espressione ectopica21. Prima dell'emergere delle forbici molecolari CRISPR-Cas9, sono state sviluppate tecnologie che promuovono la rottura del doppio filamento del DNA in siti bersaglio specifici e si sono basate sulle proprietà di legame del DNA di specifici domini proteici marcati con l'enzima di restrizione FokI, come TALEN22 e Zinc Finger23. Sebbene quest'ultima tecnologia sia stata utilizzata con successo per l'ingegneria genetica delle zanzare24,25, l'alto costo dell'ingegnerizzazione di tali domini e il potenziale di scissione fuori bersaglio hanno portato all'interruzione di tali tecnologie. Da allora, la tecnologia CRISPR-Cas9 è diventata le forbici molecolari di scelta11.
L'ingegneria genetica delle zanzare è stata, per la maggior parte, effettuata mediante iniezione della miscela di trasformazione in embrioni26,27. La realizzazione di microiniezioni embrionali di zanzara richiede concentrazione, pazienza e attenzione ai dettagli. Durante le 2 ore successive alla deposizione degli ovociti, questi embrioni devono essere allineati, iniettati e lavati il più accuratamente possibile per non interrompere il loro sviluppo. La concentrazione è un'abilità essenziale per ridurre la possibilità che gli ovuli vengano danneggiati o che gli embrioni si secchino durante l'allineamento. Di fronte a incidenti imprevisti come l'intasamento dell'ago, la rottura della punta e la perdita della miscela per iniezione, che si verificano abbastanza spesso quando la proteina Cas9 è nella miscela, la pazienza è un'altra abilità essenziale per gestire queste situazioni. Punzecchiare gli embrioni quando la punta dell'ago è troppo grande o fuoriesce troppo mix per iniezione può solo danneggiare l'embrione. La soluzione migliore è sostituire l'ago e continuare con le iniezioni. Non meno importante è l'attenzione ai dettagli. Le zanzare femmine più giovani (6-10 giorni) depongono più uova di quelle più anziane e questo è utile per raccogliere embrioni sani. Scegli acqua deionizzata o ultrapura e tovaglioli di carta marroni (non sbiancati) per conservare gli embrioni dopo la microiniezione, poiché qualsiasi contatto con acqua contaminata o superfici sbiancate può aumentare significativamente la mortalità degli embrioni. Gli embrioni grigio chiaro (<1 h) sono più morbidi, il che facilita la procedura di iniezione, aumentando le possibilità di ottenere la linea mutante. Inoltre, gli embrioni iniettati devono riposare in carta da filtro umidificata per almeno 5 giorni prima di essere schiusi per consentire il pieno recupero e la guarigione.
La qualità e la purezza della miscela per iniezione sono fondamentali per la sopravvivenza dell'embrione. Né tamponi, sali né alcoli provenienti da kit per l'estrazione di plasmidi o da kit per la sintesi di gRNA devono essere trasferiti nelle miscele per iniezione. Anche se costosa, l'opzione di ordinare i gRNA può aumentare la probabilità di un corretto ripiegamento dell'RNA. Raccomandiamo anche di iniettare la proteina ricombinante Cas9 su un plasmide che codifica Cas9, poiché riteniamo che la versione ricombinante sia già caricata e pronta per eseguire rotture a doppio filamento rispetto a una fonte plasmidica che deve essere trascritta e tradotta e quindi caricata con gRNA per iniziare ad agire sul DNA.
Sebbene abbiamo descritto il protocollo che funziona meglio nelle nostre mani per quanto riguarda l'uso della tecnologia CRISPR-Cas9 per l'editing del genoma nella zanzara A. aegypti, in letteratura sono stati descritti reagenti alternativi che possono essere utilizzati anche in sostituzione se necessario. Ad esempio, sospendiamo tutti i reagenti e diluiamo le miscele per iniezione con acqua ultrapura. In alternativa, le miscele per iniezione possono essere diluite in un tampone per iniezione composto da 5 mM di KCl e 0,1 M di NaPO4 (pH = 6,8)26,28. Oltre all'uso dell'olio di alocarburi 700 per proteggere gli embrioni dall'essiccazione dopo averli allineati, l'olio di alocarburi 2729 e una miscela di 27 e 70030 forniscono una protezione simile. La versione concentrata 10x del tampone di iniezione sopra descritta è stata utilizzata in alternativa per prevenire l'essiccazione dell'embrione26. Come accennato in precedenza, i plasmidi che codificano Cas9 sono disponibili in commercio (addgene) e sono stati utilizzati per l'editing del genoma mediato da CRISPR-Cas9 nelle zanzare31.
Tra le strategie per ottenere una linea transgenica di zanzara, sono disponibili approcci knockout mediati da NHEJ e knockin mediati da HDR. Mentre il tasso di successo del primo è molto più alto di quello del secondo32,33, lo schema di attraversamento e il processo di screening richiedono molto più tempo per ottenere un knockout rispetto a una knockin line, come descritto nella Figura 4. Come approccio alternativo per ottenere una linea knockout senza passare attraverso complicati schemi di incrocio, il knockin mediato da HDR di una cassetta genica che codifica un marcatore fluorescente offre la possibilità di interrompere la sequenza codificante di un gene e selezionare la linea transgenica della zanzara utilizzando il marcatore fluorescente14,34.
Sebbene questo protocollo si concentri solo sui passaggi di base per la trasformazione genetica delle zanzare, la progettazione di plasmidi donatori per l'HDR è un passo essenziale che deve essere preso in considerazione. È importante utilizzare promotori come 3xP3, OpIE2, Hr5-IE1 e Pub 6,35,36,37,38. 3xP3 è un promotore sintetico comunemente usato per guidare l'espressione nel sistema nervoso e negli occhi degli insetti. OpIE2 e Hr5-IE1 sono promotori derivati dal baculovirus che vengono utilizzati per guidare una forte espressione nelle cellule di insetti. Il promotore del pub guida l'espressione in tutto il corpo. Il promotore U6 è utilizzato per l'espressione dei gRNA. I marcatori fluorescenti come dsRed (e TdTomato), eGFP, CFP e YFP vengono utilizzati anche per i plasmidi donatori per HDR. Si tratta di proteine fluorescenti che emettono rispettivamente fluorescenza rossa, verde, ciano e gialla. Per gli UTR 3', p1039 e SV4040 sono spesso utilizzati per migliorare la stabilità dell'mRNA e l'efficienza della traduzione nei transgeni.
Come riassunto della procedura di microiniezione, per le linee mutanti knockout, in primo luogo, allineare gli embrioni di zanzara su nastro biadesivo fianco a fianco per l'iniezione (Figura 4A). In secondo luogo, rimuovere la maggior parte dell'olio di alocarburi per facilitare il tasso di sopravvivenza degli embrioni e trasferire gli embrioni dal nastro biadesivo a una carta da filtro bagnata pulita. Conservali in una tazza con acqua ultrapura per mantenere un'elevata umidità per alcuni giorni, aspettando che gli embrioni si sviluppino completamente (Figura 4B). In terzo luogo, schiudere gli embrioni iniettati, separare le pupe in entrambi i sessi e metterle in gabbie diverse (Figura 4C). In quarto luogo, mettere tutti i maschi fluorescenti in una gabbia per incrociarli con femmine WT di 5-7 giorni e viceversa (Figura 4D). Quinto, raccogli le uova G1 dalle femmine WT incrociate per espandere il numero di zanzare all'inizio (Figura 4E). Sesto, schiusa delle uova G1 e separazione delle pupe in entrambi i sessi (Figura 4F). Settimo, gli adulti G1 si incrociano nuovamente con WT (Figura 4G). In ottavo luogo, raccogliere le femmine adulte G1 in fiale per la raccolta individuale di ovuli G2 e mantenere l'acqua ultrapura sul fondo delle fiale utilizzando batuffoli di cotone o un tovagliolo di carta per alcuni giorni (Figura 4H). Nono, raccogliere le femmine G1 dopo che hanno deposto le uova ed estrarre il DNA genomico dalle femmine G1 per la PCR e il sequenziamento (Figura 4I). Decimo, schiusa di uova G2 corrette per il mantenimento della linea e incroci con WT per altre tre generazioni per rimuovere la maggior parte delle mutazioni di fondo non collegate (Figura 4J). Undicesimo, stabilire una linea omozigote di zanzara utilizzando la PCR, il saggio enzimatico di restrizione, l'elettroforesi su gel e il sequenziamento per lo screening delle mutazioni (Figura 4K).
Per la creazione di linee HDR, allineare gli embrioni di zanzara su nastro biadesivo fianco a fianco per la microiniezione di embrioni (Figura 4i). In secondo luogo, dopo le iniezioni, rimuovere la maggior parte dell'olio di alocarburi e conservare gli embrioni in una tazza con acqua ultrapura per mantenere l'umidità per alcuni giorni e attendere che gli embrioni maturino (Figura 4ii). In terzo luogo, schiudere gli embrioni iniettati e selezionare le larve G0 per selezionare quelle con o senza fluorescenza (Figura 4iii); Larve con fluorescenza significa che il plasmide è stato iniettato con successo negli embrioni di zanzara. In quarto luogo, lasciare che le larve fluorescenti maturino in adulti e posizionare tutti i maschi fluorescenti in una gabbia per incrociarli con le femmine WT e, viceversa, per espandere il numero di zanzare (Figura 4iv). Quinto, raccogli le uova dalle femmine WT incrociate (Figura 4v). Sesto, schiudere, vagliare e selezionare le larve di fluorescenza G1 e smaltire le larve senza fluorescenza (Figura 4vi). Settimo, incrociare adulti a fluorescenza G1 con WT (Figura 4vii). Ottavo, raccogliere le uova G2 in fiale di plastica singolarmente per separare i diversi siti di inserzione del plasmide nelle zanzare (Figura 4viii). Nono, far schiudere le uova, controllare l'idoneità delle zanzare ed eseguire il sequenziamento (Figura 4ix). Decimo, selezionare due linee con fenotipo più forte e incrociarle con WT per altre tre generazioni (Figura 4x). Undicesimo, selezione di larve con fluorescenza da incrociare per altre due generazioni per stabilire una linea omozigote per ulteriori esperimenti (Figura 4xi).
Qui abbiamo fornito il protocollo più completo per l'editing del genoma di A. aegypti con CRISPR-Cas9 che abbiamo utilizzato per generare più linee transgeniche. Suggeriamo che questo protocollo venga utilizzato come punto di partenza per altri ricercatori, che dovrebbero apportare modifiche in base all'esperienza ottenuta con i loro studi di microiniezione. Devono sempre essere testati reagenti e condizioni diverse. Il tasso di successo dell'editing genetico nelle zanzare deve ancora essere completamente ottimizzato e pochissimi laboratori sono stati in grado di produrre zanzare transgeniche tramite CRISPR-Cas9.
Divulgazioni
O.S.A. è uno dei fondatori di Agragene, Inc. e Synvect, Inc. con una partecipazione azionaria. I termini di questo accordo sono stati esaminati e approvati dall'Università della California, San Diego, in conformità con le sue politiche sul conflitto di interessi. Gli altri autori dichiarano di non avere interessi concorrenti.
Riconoscimenti
Gli autori ringraziano Judy Ishikawa e Ava Stevenson per aver aiutato con l'allevamento delle zanzare. Questo lavoro è stato supportato dai finanziamenti dei premi NIH (R01AI151004, RO1AI148300, RO1AI175152) assegnati a O.S.A. e K22AI166268 a N.H.R. Le figure sono state create utilizzando BioRender.
Materiali
| Name | Company | Catalog Number | Comments |
| 10x Cas9 reaction buffer | PNA Bio | CB01 | |
| Benchling software | Benchling | N/A | www.benchling.com |
| Cas9 dilution buffer | PNA Bio | CB03 | |
| Cas9 protein | PNA Bio | CP01-50 | |
| DH5α E. coli Competent Cells | New England Biolabs | C2987 | |
| Double-sided sticky tape | Scotch Permanent | 3136 | |
| Drosophila vials | Genesee Scientific | 32-109 | |
| Filter papers | GE Healthcare Life Science | 1450-042 | |
| Fish food | Tetra | B00025Z6YI | goldfish flakes |
| Flugs | Genesee Scientific | AS273 | |
| Fluorescent microscope | Leica Microsystems | M165 FC | |
| Gene fragment | Integrated DNA Technologies | N/A | |
| gRNA | Synthego | N/A | |
| Halocarbon oil 700 | Sigma-Aldrich | H8898 | |
| Injection microscope | Leica Microsystems | DM2000 | |
| JM109 E. coli Competent Cells | Zymo Research | T3005 | |
| Microinjector | Eppendorf | FemtoJet 4x | |
| Microloader Tips for Filling Femtotips | Eppendorf | E5242956003 | |
| Micromanipulator | Eppendorf | TransferMan 4r | |
| Micropipette Pullers | Sutter Instrument | P-2000 | |
| Microscope Cover Glass | Fisherbrand | 12-542-B | |
| Microscope slide | Eisco | 12-550-A3 | |
| Mouse blood (live mice used for feeding) | University of California | IACUC, Animal Use Protocol #S17187 | Used for mosquito blood feeding; details comply with animal ethics protocols |
| NEB Q5 High-Fidelity DNA polymerase | New England Biolabs | M0491S | |
| PCR Purification Kit | Qiagen | 28004 | |
| Plasmid Miniprep Kit | Zymo Research | D4036 | |
| Quartz filament | Sutter Instruments | QF100-70-10 | |
| Transcription Clean-Up Kit | Fisher Scientific | AM1908 | |
| Ultra-pure water | Life Technologies | 10977-023 |
Riferimenti
- Anzalone, A. V., Koblan, L. W., Liu, D. R. Genome editing with CRISPR-Cas nucleases, base editors, transposases and prime editors. Nat Biotechnol. 38 (7), 824-844 (2020).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Shen, H., Li, Z. DNA double-strand break repairs and their application in plant DNA integration. Genes (Basel). 13 (2), 322 (2022).
- Vinauger, C., et al. Modulation of host learning in Aedes aegypti mosquitoes. Curr Biol. 28 (3), 333-344.e8 (2018).
- Li, M., Bui, M., Yang, T., Bowman, C. S., White, B. J., Akbari, O. S. Germline Cas9 expression yields highly efficient genome engineering in a major worldwide disease vector. Proc Natl Acad Sci U S A. 114 (49), E10540-E10549 (2017).
- Chaverra-Rodriguez, D., et al. Targeted delivery of CRISPR-Cas9 ribonucleoprotein into arthropod ovaries for heritable germline gene editing. Nat Commun. 9 (1), 3008 (2018).
- Rouyar, A., et al. Transgenic line for characterizing GABA-receptor expression to study the neural basis of olfaction in the yellow-fever mosquito. Front Physiol. 15, 1381164 (2024).
- Ang, J. X. D., et al. Considerations for homology-based DNA repair in mosquitoes: Impact of sequence heterology and donor template source. PLoS Genet. 18 (2), e1010060 (2022).
- Zhang, J. -. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biol. 18 (1), 35 (2017).
- Coutinho-Abreu, I. V., Akbari, O. S. Technological advances in mosquito olfaction neurogenetics. Trends Genet. 39 (2), 154-166 (2023).
- Li, M., et al. Targeting sex determination to suppress mosquito populations. eLife. 12, RP90199 (2024).
- Zhan, Y., Alonso San Alberto, D., Rusch, C., Riffell, J. A., Montell, C. Elimination of vision-guided target attraction in Aedes aegypti using CRISPR. Current Biol. 31 (18), 4180-4187.e6 (2021).
- Greppi, C., et al. Mosquito heat seeking is driven by an ancestral cooling receptor. Science. 367 (6478), 681-684 (2020).
- Potter, C. J., Tasic, B., Russler, E. V., Liang, L., Luo, L. The Q system: a repressible binary system for transgene expression, lineage tracing, and mosaic analysis. Cell. 141 (3), 536-548 (2010).
- Riabinina, O., et al. Improved and expanded Q-system reagents for genetic manipulations. Nat Methods. 12 (3), 219-222 (2015).
- Labun, K., et al. CHOPCHOP v3: expanding the CRISPR web toolbox beyond genome editing. Nucleic Acids Res. 47 (W1), W171-W174 (2019).
- Concordet, J. -. P., Haeussler, M. CRISPOR: intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Res. 46 (W1), W242-W245 (2018).
- Williams, A. E., et al. Quantifying fitness costs in transgenic Aedes aegypti mosquitoes. J Vis Exp. , e65136 (2023).
- Bassett, A. R., Tibbit, C., Ponting, C. P., Liu, J. -. L. Highly efficient targeted mutagenesis of Drosophila with the CRISPR/Cas9 system. Cell Rep. 4 (1), 220-228 (2013).
- Coutinho-Abreu, I. V., Zhu, K. Y., Ramalho-Ortigao, M. Transgenesis and paratransgenesis to control insect-borne diseases: current status and future challenges. Parasitol Int. 59 (1), 1-8 (2010).
- Christian, M., et al. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics. 186 (2), 757-761 (2010).
- Kim, Y. G., Cha, J., Chandrasegaran, S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain. Proc Natl Acad Sci U S A. 93 (3), 1156-1160 (1996).
- DeGennaro, M., et al. orco mutant mosquitoes lose strong preference for humans and are not repelled by volatile DEET. Nature. 498 (7455), 487-491 (2013).
- McMeniman, C. J., Corfas, R. A., Matthews, B. J., Ritchie, S. A., Vosshall, L. B. Multimodal integration of carbon dioxide and other sensory cues drives mosquito attraction to humans. Cell. 156 (5), 1060-1071 (2014).
- Lobo, N. F., Clayton, J. R., Fraser, M. J., Kafatos, F. C., Collins, F. H. High efficiency germ-line transformation of mosquitoes. Nat Protoc. 1 (3), 1312-1317 (2006).
- Kistler, K. E., Vosshall, L. B., Matthews, B. J. Genome engineering with CRISPR-Cas9 in the mosquito Aedes aegypti. Cell Rep. 11 (1), 51-60 (2015).
- Handler, A. M., Harrell, R. A. 2nd Transformation of the Caribbean fruit fly, Anastrephasuspensa, with a piggyBac vector marked with polyubiquitin-regulated GFP. Insect Biochem Mol Biol. 31 (2), 199-205 (2001).
- Harrell, R. A. . 2nd Mosquito embryo microinjection under halocarbon oil or in aqueous solution. 2024 (7), (2024).
- Sun, R., Raban, R., Akbari, O. S. Generating mutant strains with transgenic Cas9. Cold Spring Harb Protoc. 2023 (9), 671-678 (2023).
- Giraldo, D., et al. An expanded neurogenetic toolkit to decode olfaction in the African malaria mosquito Anopheles gambiae. Cell Rep Methods. 4 (2), 100714 (2024).
- Liu, G., Lin, Q., Jin, S., Gao, C. The CRISPR-Cas toolbox and gene editing technologies. Mol Cell. 82 (2), 333-347 (2022).
- Chu, V. T., et al. Increasing the efficiency of homology-directed repair for CRISPR-Cas9-induced precise gene editing in mammalian cells. Nat Biotechnol. 33 (5), 543-548 (2015).
- Laursen, W. J., et al. Humidity sensors that alert mosquitoes to nearby hosts and egg-laying sites. Neuron. 111 (6), 874-887.e8 (2023).
- Weng, S. -. C., Antoshechkin, I., Marois, E., Akbari, O. S. Efficient sex separation by exploiting differential alternative splicing of a dominant marker in Aedes aegypti. PLoS Genet. 19 (11), e1011065 (2023).
- Li, M., et al. Development of a confinable gene drive system in the human disease vector. eLife. 9, e51701 (2020).
- Dalla Benetta, E., et al. Engineered Antiviral Sensor Targets Infected Mosquitoes. The CRISPR journal. 6 (6), 543-556 (2023).
- Li, H. -. H., et al. C-Type lectins link immunological and reproductive processes in Aedes aegypti. iScience. 23 (9), 101486 (2020).
- van Oers, M. M., Vlak, J. M., Voorma, H. O., Thomas, A. A. M. Role of the 3' untranslated region of baculovirus p10 mRNA in high-level expression of foreign genes. J Gen Virol. 80 (Pt 8), 2253-2262 (1999).
- Salem, T. Z., et al. The influence of SV40 polyA on gene expression of baculovirus expression vector systems. PloS One. 10 (12), e0145019 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon