Method Article
CRISPR-Cas9을 이용한 황열병 모기 이집트숲모기(Mosquito Aedes aegypti )의 게놈 편집
요약
여기에서는 CRISPR-Cas9 기술을 사용하여 모기 A. aegypti 에서 배아 미세주입을 통한 게놈 편집을 위한 자세한 프로토콜에 대해 설명합니다.
초록
CRISPR(Clustered, regularly interspers, 단회문 반복)-Cas9 기술의 출현은 유전 공학 분야에 혁명을 일으켰으며 비모델 유기체를 포함한 여러 종에서 정밀한 게놈 편집의 문을 열었습니다. 이집트숲모기( Aedes aegypti)에서는 이 기술로 기능 상실 돌연변이와 DNA 삽입이 이루어졌습니다. 여기에서는 CRISPR-Cas9 기술을 사용하여 모기 A. aegypti 에서 배아 미세주입을 통한 게놈 편집을 위한 자세한 프로토콜에 대해 설명하며, 유전자 knockout 및 knockin line의 생성에 중점을 둡니다. 이 프로토콜에서 석영 바늘은 가이드 RNA, 재조합 Cas9 및 유전자 knockin이 필요한 경우 형광 마커용 유전자를 인코딩하는 DNA 카세트를 포함하는 플라스미드의 혼합물로 채워집니다. 배배엽 전 단계의 배아는 커버슬립에 놓인 양면 접착 테이프 스트립에 정렬된 다음 유리 슬라이드에 장착됩니다. 마이크로인젝터(microinjector)를 사용하여 바늘을 배아의 뒤쪽 끝에 부드럽게 삽입하고 소량의 CRISPR 혼합물을 분주합니다. 배아가 부화하면 형광등 아래에서 유충을 확인하고 번데기는 성별을 분류하여 다른 우리에서 분리합니다. 일단 성체가 나오면, 이들은 야생형 개체와 상호 교배되어 피를 먹고 알을 낳기 위해 배치됩니다. 이 알이 부화하면 수집된 형광 유충은 DNA 카세트가 게놈에 안정적으로 삽입된 개체를 나타냅니다. 그런 다음 이 유충은 성충 단계로 성장하여 야생형 개체로 교배된 다음 분자 기술을 통해 추가로 평가하여 DNA 카세트의 정확한 염기서열이 모기 게놈의 원하는 위치에 존재하는지 확인합니다. 동형접합 라인은 또한 제공된 교차 스키마 파이프라인과 돌연변이의 분자 스크리닝을 따라 얻을 수 있습니다.
서문
정확한 게놈 편집은 분자 가위의 CRISPR-Cas 기술이 확립됨에 따라 틀림없이 쉬워졌지만 가능해졌습니다1. 이러한 기술은 원핵 면역 체계가 파지 감염과 싸우는 데 사용하는 메커니즘을 이용합니다2. 이러한 시스템 중에서 Cas9 nuclease와 함께 clustered, regularly interspers, short palindromic repeats (CRISPR)는 일반적으로 20개의 염기쌍 RNA, 가이드 RNA(gRNA)에 의존하며, 표적 DNA와 상동적인 염기서열은 NGG protospacer adjacent motif (PAM) 염기서열3에 이어집니다. Cas9에 로드된 gRNA는 이러한 nuclease를 게놈의 특정 타겟 부위로 정확하게 안내하여 double-strand DNA break3를 유발합니다.
DNA 이중 가닥 절단은 복구 메커니즘이 이중 나선4를 패치하도록 유도합니다. 모든 DNA 복구는 정확해야 하지만, 염기서열 흉터를 남기고 결과적으로 기능 상실 돌연변이를 남길 수 있는 덜 정확한 DNA 복구 메커니즘이 존재합니다4. 오류가 발생하기 쉬운 DNA 복구 메커니즘 중 NHEJ(Non-Homologous End-Joining)는 작은 결실, 삽입 및 뉴클레오티드 변화(SNP)를 포함한 프레임 이동 돌연변이를 유발할 수 있으며, 이는 기능 상실 돌연변이를 초래할 수 있습니다. 반면에 HDR(Homology Directed Repair) 메커니즘은 상동 염색체를 템플릿으로 사용하여 손상되지 않은 대립유전자의 정확한 염기서열을 복사하고 표적 DNA 염기서열4을 완벽하게 복구합니다.
이러한 지식을 바탕으로 CRISPR-Cas9 기술은 PAM site3을 포함하는 모든 염기서열에서 게놈을 정밀하게 편집하기 위해 개발되었습니다. 모기의 경우, CRISPR-Cas9 기술은 NHEJ 복구 메커니즘 5,6을 활용하여 Cas9과 gRNA 혼합물의 배아 미세주입을 통해 다양한 유전자를 제거하는 데 사용되었습니다. 유사한 생식계열 돌연변이 유발은 성체 암컷 모기의 혈림프에 gRNA + Cas9 혼합물을 주입하여 얻을 수 있습니다7. 이 기술은 ReMOT control이라는 용어로 만들어졌으며 난자 발달(유리체 형성) 과정에서 세포내이입(endocytosis)을 통해 난소에서 흡수되는 펩타이드로 태그된 Cas9의 수정된 버전에 의존합니다7. 특정 유전자 카세트를 게놈에 knocking하는 것은 원하는 DNA 카세트8을 인코딩하는 플라스미드와 함께 gRNA와 Cas9(또는 이러한 분자를 발현하는 플라스미드)의 혼합물의 배아 미세주입을 통해서만 가능합니다. HDR 메카스테릭을 이용하여, 타겟 부위의 상류 및 다운스트림(homologous sequences, 500-1,000bp)9,10 측면에 있는 관심 DNA 카세트를 포함하는 플라스미드(plasmid)를 주형(template)으로 사용하여 이중 가닥 절단(double-strand break)을 다시 작성하고, DNA 카세트도 타겟 서열(9)에 복사합니다.
CRISPR-Cas9 기술은 주로 이집트숲모기(Aedes aegypti)의 감각 시스템과 관련된 여러 유전자를 제거하는 데 사용되었으며, 11 뿐만 아니라 개체군 조절을 위한 수컷 생식력 및 암컷 생존력(PgSIT)과 관련된 유전자도 제거했습니다12. 표적 유전자를 knocking out은 또한 형광 마커를 인코딩하는 유전자를 특정 유전자의 암호화 서열로 knocking in 함으로써 이루어졌습니다13,14. 이 전략은 프레임 시프트 돌연변이를 유도할 뿐만 아니라 형광등을 사용하여 새로운 녹아웃 라인13,14의 개체를 분류할 수 있는 이점이 있습니다. A. aegypti 게놈은 또한 Q-시스템(QF-QUAS)11과 같은 이진 발현 시스템의 염기서열로 편집되었습니다. QF 트랜스 액티베이터를 인코딩하는 유전자를 특정 유전자의 프로모터로 노크하면 트랜스 액티베이터의 정의 된 시공간 발현이 보장됩니다15, 16. QF 발현 모기 계통이 QF에 대한 결합 부위 (QUAS)를 포함하는 다른 모기 계통으로 교차하면, 후자는 이에 결합하고 QUAS 서열15,16으로 다운스트림에서 유전자 발현을 유발합니다. 이 시스템은 전반적으로 세포 국소화 또는 신경 세포 활동의 검출에 사용되는 형광 마커가 될 수 있는 이러한 효과기 유전자의 조직 및 시간별 발현을 허용하며, 특정 조직(즉, 체세포 녹아웃)의 유전자를 파괴하기 위한 Cas9 뉴클레아제도 될 수 있습니다11.
A. aegypti 유전자 형질전환에 대한 모든 정보를 감안하여, 배아 미세주입을 통해 CRISPR-Cas9 시스템으로 유전체 편집을 수행하기 위한 단계별 지침이 포함된 자세한 프로토콜을 제공합니다. NHEJ에 의해 매개되는 프레임 시프트 돌연변이 및 결실을 통한 knockout과 HDR 매개 유전자 카세트 삽입에 의한 knockin lines를 모두 생성하는 전략에 대해 논의합니다.
프로토콜
이 프로토콜에 사용되는 장비 및 시약과 관련된 세부 정보는 재료 표에 나열되어 있습니다. 모든 동물은 미국 국립보건원(National Institutes of Health)의 권고에 따라 실험실 동물의 관리 및 사용에 대한 가이드에 따라 취급되었습니다. 이 절차는 UCSD 기관 동물 관리 및 사용 위원회(IACUC, Animal Use Protocol #S17187) 및 UCSD 생물학적 사용 승인(BUA #R2401)의 승인을 받았습니다.
1. gRNA 및 donor plasmid design
- knockout mutant를 만들기 위해 ~20-100 bp 간격으로 두 개의 gRNA를 설계하십시오(그림 1A).
- CHOPCHOP ( https://chopchop.cbu.uib.no/)17또는 Benchling (benchling.com) 또는 CRISPOR (http://crispor.gi.ucsc.edu/)18과 같은 온라인 도구(그림 1A)를 사용하여 PAM 염기서열(NGG)을 제외한 CRISPR-Cas9용 20 bp gRNA를 설계합니다. 온라인 도구에서 제안한 대로 실험을 위해 가장 특이적이고 off-target-free gRNA를 선택합니다.
- 효율적인 제한 효소 절단 부위가 gRNA 절단 부위 사이의 염기서열 내에 포함되어 있는지 확인합니다(그림 1A). gRNA cut 부위 사이에 제한 효소 부위를 포함하여 성공적인 편집을 시각적으로 빠르게 확인할 수 있습니다.
참고: 결실이 발생하면 제한 부위가 제거되므로 효소는 염기서열을 절단하지 않고 결실을 확인하기 위해 겔에 절단되지 않은 단일 밴드를 생성합니다.
- HDR 매개 유전자 카세트 삽입의 경우, knockout mutant를 만들기 위해 언급된 도구를 사용하여 여러 gRNA를 설계하고 추후 평가 후 가장 효과적인 gRNA를 선택합니다.
- 표적 부위 상동성군, 화물 DNA 염기서열, 형광 마커, 복제 기원 및 항생제 내성 유전자를 포함하는 HDR(Homology-Directed Repair)을 위한 donor plasmid를 설계합니다(그림 1B).
- 각각 500-1,000 bp10 에 걸쳐 있는 타겟 부위의 업스트림 및 다운스트림 영역에서 상동성 암을 선택합니다(그림 1B).
- 형광 마커, 관심 유전자 또는 조절 요소(예: QF2)를 포함할 수 있는 화물 염기서열을 선택합니다.
- 형광 마커를 선택합니다. A. aegypti 에 사용되는 일반적인 형광 마커는 promoter, 형광 마커 인코딩 유전자 및 3' UTR 서열을 포함하는 DNA cassette에 포함되어 있습니다. 자세한 내용은 토론을 참조하십시오.
2. 주입 혼합 준비
- Cas9 희석 완충액으로 Cas9 단백질을 1μg/μL로 희석합니다.
알림: Cas9를 2회 이상 해동 및 동결하지 마십시오. Cas9 단백질의 부분 표본을 만드는 것이 좋습니다. - gRNA를 구매하거나 사내에서 생산할 수 있습니다.
참고: 자체 생산의 경우 표준 RNA 오염 제거 관행 및 RNAse-free 재료를 사용하십시오.- 4-6개의 gRNA를 설계하고 주입에 가장 적합한 2개의 gRNA를 선택합니다(아래 in vitro cleavage assay 참조).
- gRNA 염기서열의 업스트림에 T7 promoter 염기서열을 포함하는 forward primer를 설계합니다. 아래와 같이 non-template PCR 반응을 위해 범용 gRNA reverse primer를 사용합니다. 겹치는 염기서는 범용 리버스 프라이머(굵게 및 밑줄이 그어짐)의 해당 염기서열과 쌍을 이루어 DNA 중합효소가 염기서열을 증폭할 수 있는 주형을 생성합니다.
참고: 이러한 PCR 반응의 경우, 전방 프라이머에는 T7 프로모터(굵은 글씨체)와 두 개의 구아닌(T7 RNA 중합효소를 통한 전사 개시에 중요), gRNA의 20-뉴클레오티드 서열(N20, PAM 서열 없음) 및 프라이머 중첩 서열(밑줄 표시)이 포함되어야 합니다.
프라이머 포워드: 5'- GAAATTAATACGACTCACTATAGGN20 GTTTTAGAGCTAGAAATAGC- 3'
프라이머 리버스 : 5 ''-AAAAGCACCGACTCGGTGCCACTTTTT CAAGTTGATAACGGACTAGC
CTTTATT TTAACTT GCTATTTTAGCTCTAAAAC - 3' - non-template PCR로 DNA 주형을 합성합니다. in vitro transcription 반응에 충분한 PCR 산물(300ng)을 사용할 수 있도록 다중 반응(각각 2x 마스터 믹스 12.5 μL, 10 μM 용액 10 μM 용액 1.25 μL, 10 μL의 초순수 포함)을 설정합니다. 다음 PCR 조건을 사용하십시오 : 98 ° C에서 30 초 동안 초기 변성; 98 ° C에서 10 초, 62 ° C에서 10 초, 72 ° C에서 10 초 동안 35 사이클의 증폭; 72°C에서 2분 동안 최종 확장; 4 °C에서 보관하십시오.
- 아가로스 겔(2%)에서 단일 DNA 단편(122 염기쌍)의 증폭을 확인합니다.
- 제조업체의 권장 사항에 따라 PCR 정제 키트를 사용하여 PCR 템플릿을 정리합니다.
- T7 Transcription Kit를 사용하여 in vitro transcription 반응을 수행합니다. 10x 반응 완충액 2 μL, 뉴클레오티드(ATP, CTP, UTP 및 GTP) 각각 2 μL, T7 RNA 중합효소 2 μL, 템플릿 DNA(100 ng/μL) 3 μL 및 초순수 5 μL를 혼합합니다. 혼합물을 37 ° C에서 최소 2 시간 (16 시간 이하)에서 하룻밤 (12 시간) 동안 배양합니다.
참고: 이 반응에서 T7 RNA 중합효소는 프라이머 순방향 염기서열에 포함된 T7 promoter에 결합하여 gRNA의 transcription을 유도합니다. - 1 μL의 DNase(잘 혼합)를 첨가하고 37 °C에서 15분 동안 배양하여 DNase로 전사 반응을 처리합니다.
- 제조업체의 권장 사항에 따라 transcription cleanup kit로 합성된 sgRNA를 정제합니다.
- in vitro cleavage assay를 수행하여 선택한 gRNA의 절단 효율을 평가합니다(그림 1C).
- gRNA 절단 부위 옆에 있는 DNA 단편(500-1,000bp)을 증폭하기 위한 프라이머를 설계합니다.
- PCR 반응을 다음과 같이 설정합니다: 2x 마스터 믹스 12.5 μL, 10 μM 포워드 프라이머 용액 1.25 μL, 10 μM 리버스 프라이머 용액 1.25 μL, 초순수 9 μL, 5 ng/μL 템플릿 DNA 1 μL.
- 다음 PCR 조건을 사용하십시오 : 조건 : 98 ° C에서 30 초 동안 초기 변성; 98 ° C에서 10 초, X ° C (프라이머에 이상적인 어닐링 온도)에서 15-30 초, 72 ° C에서 35 초 동안 35 사이클의 증폭; 72°C에서 2분 동안 최종 확장; 그리고 4 °C에 마지막 장기 저장.
- 최상의 amplification 온도 및 시간 범위에 대해서는 제조업체의 지침을 참조하십시오. PCR cleanup(단일 밴드) 또는 agarose gel PCR band 정제 후 충분한 PCR 산물(1.5-2 μg)을 얻을 수 있도록 최소 5개의 반응을 풀링하거나 시약의 부피를 확장하십시오.
- 다음 혼합물을 37°C에서 1시간 동안 배양하여 재조합 Cas9로 Cas9 절단 반응을 설정합니다: 10x Cas9 반응 완충액 1μL, 재조합 Cas9 0.35μL(1μg/μL), gRNA(100ng/μL) 1μL, 초순수 6.65μL, 템플릿 DNA 300ng/μL 1μL.
- gRNA 없이 negative control 반응을 설정합니다.
- 아가로스 겔(1.5-2.0%; 그림 1C).
3. 공여체 플라스미드 조립
- A. aegypti의 게놈 DNA에서 상동성을 PCR-증폭합니다.
- 프라이머 설계는 클로닝 키트 제조업체의 지침을 참조하십시오. 다양한 클로닝 전략(예: Gibson, Golden Gate 및 제한 효소 기반 클로닝)을 사용하여 플라스미드 설계 및 조립을 지원하는 온라인 도구(예: Benchling)를 사용하십시오.
- A. aegypti 게놈 DNA의 화물 염기서열을 증폭하거나 상업적으로 합성된 유전자 단편을 주문할 수 있습니다.
- 사내에서 플라스미드의 형광 마커를 증폭합니다.
- 3.1.1 단계에서 3.1.3 단계의 DNA 단편을 Gibson 어셈블리에 의해 기존 플라스미드의 골격으로 접합합니다. 10 μL의 2x 마스터 믹스, X μL의 모든 DNA 단편, 10-X μL의 초순수의 혼합물을 50 °C에서 1 시간 동안 배양합니다.
참고: 플라스미드 백본에 대한 인서트의 이상적인 비율을 계산하기 위한 제조업체의 지침을 참조하십시오.- 각 클로닝 단편의 농도를 피코 몰 단위로 계산합니다: pmols = (무게(ng)) × 1,000/(염기쌍 × 650 달톤).
- 50-100 ng의 플라스미드 골격과 각 삽입물의 2-3배 여유량의 몰을 사용합니다.
- 2-3개의 단편을 조립하는 경우 각 단편의 0.02-0.5pmol을 Gibson 반응에 추가합니다. 4-6 개의 단편을 조립하는 경우 각 단편을 0.2-1.0 pmols를 첨가하십시오.
- 3-5 μL의 Gibson 어셈블리 반응을 사용하여 E. coli competent cells JM109를 형질전환시킵니다.
참고: JM109는 바람직하지 않은 재조합 이벤트를 줄이고 세포 수확 중 뉴클레아제 캐리오버를 방지하여 조립된 DNA 단편의 무결성을 보장하는 recA- 유전자형으로 인해 Gibson 조립에 선택되었습니다
10kb보다 큰 플라스미드의 경우, 제조업체의 권장 사항에 따라 확장 프로토콜을 사용하여 DH5α 수용 세포를 형질전환할 것을 권장합니다. - 형질전환된 박테리아를 확장하고 miniprep 키트를 사용하여 플라스미드를 정제합니다.
- 올바른 플라스미드 조립을 확인하기 위해 정제된 플라스미드 DNA로 진단 제한 효소 분해를 실행하고 아가로스 겔 전기영동으로 시각화합니다(그림 1D,E).
참고: 단일 부위에서 플라스미드를 절단할 수 있는 몇 가지 제한 효소를 선택하기 위해 온라인 도구를 사용하는 것이 좋습니다. Benchling과 같은 소프트웨어에는 선택된 제한 효소를 사용하여 플라스미드 염기서열의 가상 분해를 수행하는 도구가 내장되어 있어 가상 전기영동 겔에 예상되는 DNA 밴드 패턴을 표시합니다(그림 1D).- 플라스미드 제한 효소 분해를 수행합니다. 1 μL의 10x 제한 분해 완충액, 0.5 μL의 제한 효소 1, 0.5 μL의 제한 효소 2, 1 μL의 플라스미드 DNA(300 ng/mL) 및 7 μL의 초순수의 혼합물을 37 °C에서 2 시간 동안 배양합니다.
- 아가로스 겔(1.5%)에서 제한 분해 반응을 실행합니다.
참고: DNA 밴드 패턴(그림 1E)은 가상 다이제스트 밴드 패턴(그림 1D)과 유사해야 합니다. 서비스가 가능한 경우 전체 플라스미드 염기서열분석을 위해 플라스미드를 보낼 수도 있습니다.
- 플라스미드 클론을 150mL의 LB 배지로 배양합니다.
- 제조업체의 지침에 따라 plasmid maxiprep을 수행합니다.
- 플라스미드를 초순수에 현탁시킵니다.
4. 주입 구조체의 혼합
참고: 각 구성에 대해 제안된 최종 농도 범위는 표 1에 나와 있습니다. 2:1:2(ng Cas9: ng sgRNA: ng donor plasmid)의 비율로 시작하여 HDR 효율성을 최적화하기 위해 필요에 따라 조정합니다. 결과를 모니터링하면 가장 효과적인 조합을 식별하는 데 도움이 됩니다.
- 녹아웃 돌연변이를 만들기 위해,
- Cas9 희석 완충액을 사용하여 Cas9 단백질의 분취량을 원하는 농도로 희석하고 gRNA의 분취량을 초순수로 희석합니다.
- Cas9 단백질을 각 gRNA와 프리믹스하여 리보핵단백질(RNP) 복합체를 형성합니다. 미리 혼합된 용액을 결합합니다.
- HDR 매개 유전자 카세트 삽입의 경우,
- knockout mutant를 만들기 위해 제안된 대로 Cas9 단백질과 gRNA를 희석하고 혼합합니다.
- 공여체 플라스미드를 초순수로 희석합니다.
- 모든 구문을 결합합니다.
5. microinjection 바늘 당기고 적재하기
- 석영 필라멘트를 사용하여 주삿바늘을 당깁니다(그림 2). 필라멘트의 길이가 10cm이고 외경이 1mm, 내경이 0.7mm인지 확인하십시오.
- 다음 두 프로그램 중 하나와 함께 레이저 마이크로피펫 풀러를 사용하여 바늘을 당깁니다.
프로그램 1: 히트 805, 필라멘트 4, 속도 50, 지연 145, 풀 145
프로그램 2: 히트 650, 필라멘트 4, 속도 40, 지연 150, 풀 156
참고: 프로그램 1은 바늘 끝이 더 얇지만 길어지므로 더 미세한 팁이 정밀도에 유리한 저농도 구조의 주입에 적합합니다. 프로그램 2는 더 짧지만 더 두꺼운 바늘 끝을 생성하며, 구조가 더 두껍고 더 견고한 바늘이 필요하기 때문에 더 높은 농도의 주입에 이상적입니다.
6. 배아 적출
- 모기를 채취하기 위해 전기 또는 수동 흡인기와 채취 및 배아 채취를 위해 플라스틱 바이알을 준비합니다.
- 흰색 원형 여과지를 적셔 내벽이나 수집기의 촉촉한 면 위에 놓습니다.
- 5-10일 전에 피를 먹인 5-10마리의 암컷 모기를 수집기에 넣습니다. 수집기를 45분 동안 어두운 곳에 두십시오.
- 배아 적출을 위한 여과지를 꺼냅니다.
7. 배아 라인업
- 배아 주입을 위해 A. aegypti 의 야생형 균주(Liverpool)를 사용합니다.
- 채취지에서 preblastoderm stage 배아, 특히 밝은 회색의 배아를 선택합니다(그림 3).
- 젖은 브러시로 배아 몇 개를 커버슬립 위에 붙인 양면 접착 테이프로 옮깁니다. 배아의 주변이 아직 젖어 있는 동안 배아를 평행하게 정렬하여 배아가 나란히 있고 모든 뒤쪽 끝이 앞쪽을 향하도록 합니다.
- 올바르게 배치되면 배아를 제자리에 고정하기 위해 환경을 약간 건조시킵니다.
- 건조를 방지하기 위해 정렬하는 동안 배아에 할로카본 오일 700을 첨가합니다.
알림: 기름을 추가할 때 배아가 기름에 떠 있을 수 있으므로 배아가 물로 둘러싸여 있지 않은지 확인하십시오.
8. 배아 미세주입
참고: 주입은 실온 또는 18°C에서 수행됩니다. 18°C 온도는 배아 발달을 지연시키기 때문에 실용적인 이유로 권장됩니다.
- 보상 압력(Pc) 300hPa, 분사 압력(Pi) 500hPa 매개변수로 마이크로 인젝터를 설정합니다. 필요할 때마다 이러한 조건을 조정합니다.
- 3 μL의 혼합 구조체를 마이크로로더를 사용하여 니들에 로드합니다.
- 배아가 정렬된 커버 유리를 현미경 슬라이드 위에 놓습니다(그림 3). 커버 유리를 놓고 주입을 위해 현미경 아래로 밀어 넣습니다. 배아의 뒤쪽 끝이 바늘 쪽에 위치하는지 확인하십시오.
- 마이크로 매니퓰레이터와 함께 바늘 하나를 바늘 홀더에 넣고 바늘을 정렬된 배아의 뒤쪽 끝을 향해 10° 각도로 배치하여 고정된 상태로 유지합니다(그림 4A). 현미경으로 바늘을 부드럽게 열고 커버슬립의 가장자리로 가볍게 만집니다.
참고: 또 다른 옵션은 배아로 바늘을 두드려 바늘에 작은 구멍을 만드는 것입니다. 그러나 Cas9 단백질의 끈적임으로 인해 바늘이 막힐 수 있으므로 바늘 입구를 약간 넓히는 것이 좋습니다. - 주입할 바늘 쪽으로 슬라이드 유리를 이동합니다(그림 5).
9. 주입된 배아 사후 관리
- 보푸라기가 없는 일회용 물티슈를 사용하여 배아를 둘러싼 기름을 제거합니다.
- 탈이온수를 넣어 배아를 헹구고 배아를 젖은 여과지로 옮깁니다. Karat 9oz 컵(그림 4B 및 그림 6A,B) 안의 젖은 티슈에 젖은 여과지를 놓습니다.
- 수분을 유지하기 위해 컵 바닥에 젖은 솜을 놓습니다(그림 6B).
- 주입된 배아를 젖은 여과지에 보존합니다.
10. 배아 부화 및 G0 유충 스크리닝
- 주입 후 최소 4일 후, 부화를 위해 배아가 있는 여과지를 Sterilite 3쿼트 팬에 있는 약 6L의 탈이온수로 옮깁니다.
참고: 난자는 일반적으로 주입 후 2주 이내에 더 효율적으로 부화합니다. 이 기간 동안 계란이 촉촉한 상태를 유지하는지 확인하십시오. 여과지는 과도한 수분으로 인해 배아가 일찍 부화할 수 있으므로 너무 젖지 않아야 합니다. 더 오래 보존하면 부화율이 향상될 수 있지만 최적의 결과를 위해 한 달을 초과하지 마십시오. 탈산소화된 물을 사용하는 것도 부화에 도움이 될 수 있습니다. - G0 유충이 부화하면 팬에 물과 섞인 생선 먹이를 음식으로 추가합니다.
- G0 유충을 3번째에서 4번째 instar 유충 단계에서 형광 마커로 스크리닝합니다(그림 7).
- 유충은 형광 상태에 따라 별도로 유지하며, 형광 양성 유충과 형광 음성 유충은 별도의 팬에 보관합니다.
11. 번데기를 분류하고 야생형으로 교배하는 성
- 주입된 모기가 번데기가 될 때 성별별로 분리하고, 식별을 위해 생식엽의 크기와 성별에 따른 구조를 사용합니다.
- 수컷: 더 작은 크기, 더 두드러지고 뾰족한 생식기, 더 넓은 패들(번데기의 꼬리 끝에 있는 구조; 그림 8A, 1, A2).
- 암컷: 더 큰 크기, 더 작고 덜 뚜렷한 생식기, 더 좁은 패들(그림 8B1, B2).
- 각 성별의 수영장 형광 양성 또는 형광 음성 모기.
- 웅덩이 모기를 리버풀 균주에서 온 이성의 모기로 교배하십시오. 3:1에서 5:1 비율의 야생형과 G0 모기를 사용합니다.
- 4일 동안 모기를 건너십시오.
- 암컷에게 횡단 후 혈액 식사를 제공하십시오.
12. G1 스크리닝
- 혈액 수유 3일 후 Karat 9oz 컵 벽 내부에 종이 타월을 놓고 약 3oz의 탈이온수를 추가하여 계란 컵을 제공합니다.
- 3-4일 후에 G1 배아를 수확하여 부화시킵니다. G1 유충을 선별하여 3-4번째 instar 단계에서 형광 마커를 확인합니다(그림 7).
- 형광 유충을 수집하여 HDR 삽입을 제안합니다. 형광 G1 개체가 번데기 단계에 도달하면 성별별로 분리하고 각 성별을 별도의 케이지에 넣습니다.
- 형광 G1 모기를 리버풀 균주의 이성 개체로 교배하십시오.
13. G1 삽입 부위 확인
- 혈액 식사를 제공한 후, 개별 초파리 바이알에 작은 종이 타월 조각을 놓고 탈이온수 3mL를 추가하여 G1 암컷이 단일 암컷 알을 낳을 수 있도록 설정합니다.
- 녹아웃 돌연변이의 경우:
- 알을 낳은 후 물이 증발하고 종이 타월이 마르면 분명히 녹아웃 돌연변이를 보이는 암컷의 알을 부화시키고 G2 세대를 얻습니다.
- 성공적으로 부화한 G1 어미의 몸을 채취하여 G2 자손을 채취하고 DNA를 추출합니다.
- DNA를 PCR의 주형으로 사용하여 표적 부위를 덮고 있는 염기서열을 증폭합니다. 프라이머가 ~200bp의 DNA 단편을 증폭하는지 확인합니다. 반응을 설정합니다(각각 2x 마스터 믹스 12.5μL, 순방향 프라이머 10μM 용액 1.25μL, 역방향 프라이머 10μM 용액 1.25μL, 초순수 9μL 및 템플릿 DNA 1μL[5ng/μL]) 포함). 다음 PCR 조건을 사용하십시오 : 98 ° C에서 30 초 동안 초기 변성; 98 °C에서 10 초, X ° C (프라이머의 이상적인 어닐링 온도)에서 15-30 초, 72 ° C에서 10 초 동안 35 사이클의 증폭; 72°C에서 2분 동안 최종 확장; 그리고 4 °C에 마지막 장기 저장.
- 1 μL의 10x 제한효소 완충액, 0.5 μL의 제한효소 1, 0.5 μL의 제한효소 2, 1 μL의 PCR 절편(200 ng/mL) 및 7 μL의 초순수의 혼합물을 37°C에서 30분 동안 배양하여 PCR 단편을 정제하고 제한효소 절단을 수행합니다.
참고: 특정 제한 효소는 사전 정제 없이 PCR 혼합물에 사용할 수 있습니다. - 겔 전기영동에 의한 PCR 단편 분해를 시각화하여 절단이 발생했는지 확인합니다.
- 녹아웃 돌연변이를 식별하기 위해 분해되지 않은 PCR 단편을 염기서열분석합니다.
- HDR 매개 삽입의 경우, 알을 낳은 후:
- G1 어미의 시신을 채취하여 DNA를 추출합니다.
- DNA를 PCR의 템플릿으로 사용하여 타겟 부위를 덮고 있는 염기서열을 증폭합니다.
- 삽입된 DNA 카세트의 무결성을 확인하기 위해 증폭된 DNA 단편의 염기서열분석을 수행합니다.
- 알을 부화시키고 G2 세대를 얻습니다.
14. 새로운 CRISPR 라인 확장
- 녹아웃 돌연변이 라인의 경우:
- 알을 낳기 위해 G2 암컷을 G1당 20마리의 암컷으로 설정합니다. 각 G1은 하나의 선을 나타냅니다.
- 알을 낳은 후 G1과 동일한 방식으로 녹아웃 돌연변이를 확인합니다.
- G1과 G2 모두에서 명확하게 확인된 돌연변이를 보이는 5개의 G1 라인을 진행합니다. 각 계통의 G2에서 수집한 알을 부화시켜 G3 세대를 생성합니다.
- 5개의 outcrossed line에서 명확하게 식별된 돌연변이와 높은 적합성 또는 원하는 표현형을 가진 2개의 line을 선택합니다.
- 선택한 각 라인에서 G3 암컷을 리버풀 균주의 수컷과 교배합니다.
- 각 라인에서 알을 낳기 위해 50마리의 독신 암컷을 설정합니다. G1에 대해 이전에 했던 것처럼 녹아웃 돌연변이를 확인합니다.
참고: 다양성을 유지하기 위해 설정에서 여성의 수를 늘리십시오. - 두 개의 선을 사용하여 두 개의 추가 세대에 대해 절차를 반복합니다(그림 4J).
- 새로운 HDR 매개 삽입 라인의 경우:
- G2 세대에서 형광 개체의 존재를 확인합니다.
- 각 G1 개체에서 G2 모기를 리버풀 균주와 교배합니다. 각 G1 개인은 하나의 라인을 나타냅니다.
- 3 세대의 교배를 더 진행하십시오. 형광 마커를 지속적으로 확인하고 각 라인의 적합성19 를 관찰합니다(그림 4x).
- 올바른 삽입과 높은 적합성 및 원하는 표현형을 가진 하나의 라인을 선택합니다.
15. 동형접합 선 만들기
- 녹아웃 돌연변이 라인의 경우:
- G6 세대:
- G5에서 생산된 알을 부화시키고 G6 세대를 얻습니다.
- 해당 라인 내의 Intercross G6 성인(그림 4K 및 그림 9A).
- 라인당 알을 낳기 위해 50마리의 단일 G6 암컷을 설정하고 앞서 설명한 대로 알을 부화시킵니다(그림 9B).
- G7 자손을 성공적으로 부화시킨 G6 암컷을 수집하고, DNA를 추출하고, G1과 마찬가지로 PCR 및 제한 효소 분해를 수행합니다(그림 9C).
참고: 이 단계에서는 DNA 추출을 위해 간단하고 저렴한 스퀴싱 버퍼(SB)를 사용할 수 있습니다.- 휴대용 그라인더를 사용하여 100μL의 1x SB(10mM Tris-HCl pH 8, 1mM EDTA 및 25mM NaCl)에 모기 한 마리를 침용합니다. Proteinase K(200 μg/mL)를 넣고 37°C에서 30분 동안 배양한 다음 95°C에서 2분 동안 배양합니다. 10,000 ×g에서 5 분 동안 스핀 다운하고 상층액20을 수집합니다.
- 겔에 결과를 시각화하여 돌연변이를 확인합니다.
- 돌연변이가 없는 암컷이 생산한 난자는 버린다(그림 9D).
- 여러 G7 계통을 확립하기 위해 돌연변이가 확인된 암컷의 유충을 유지하고 돌연변이가 없는 새끼를 버립니다(그림 9D).
- G7 세대:
- 각 G7 계통 내의 교배 암컷과 수컷(그림 9E).
- 라인당 알을 낳기 위해 10마리의 단일 G7 암컷을 설정하고 앞서 설명한 대로 알을 부화시킵니다(그림 9F).
- G7 자손을 성공적으로 부화시킨 G7 암컷을 수집하고, DNA를 추출하고, PCR 및 제한 효소 분해를 수행합니다(그림 9G).
- 두 대립유전자 또는 단일 대립유전자에서 결과를 겔에 시각화하여 knockout 돌연변이를 검출합니다.
- 두 대립유전자 모두에 녹아웃이 있는 것으로 확인된 G7 암컷의 유충을 유지하여 G8 세대를 생성합니다(그림 9H). 돌연변이가 없는 새끼를 버립니다(그림 9H).
참고: 이 단계는 암컷이 동형접합적임을 확인합니다. G8에서는 자손의 동형접합성을 테스트하여 아버지도 동형접합적인지 여부를 확인합니다."
- G8 세대:
- 각 G7 여성 혈통당 G8 성인을 교차합니다(그림 9I).
- 5명의 G8 수컷을 무작위로 선택하여 DNA를 추출하고 PCR 및 제한 효소 분해를 수행합니다(그림 9J).
- 혈액 식사를 제공하고 G8 수컷과 교배된 G8 암컷의 난자를 부화시켜 두 대립유전자에 녹아웃이 있는 것으로 확인되었습니다.
- G7 계통을 계속 진행하면 검출된 모든 수컷이 두 대립유전자에 녹아웃이 있어 이러한 혈통이 동형접합적임을 나타냅니다.
- G6 세대:
- HDR 매개 삽입 줄의 경우:
- G5에서 생산된 알을 부화시키고 G6 세대를 얻습니다. 형광 마커를 위해 G6 단계에서 유충을 선별합니다.
- 형광 마커가 없는 유충은 버립니다(그림 4xi).
- 형광 마커를 나타내는 교배 모기.
- 비형광 유충이 관찰되지 않을 때까지 각 후속 세대에 대해 이 절차를 계속합니다. 이 시점에서 모기 계통은 동형접합적이 됩니다.
결과
HDR 상동성 재조합을 위한 gRNA 매개 유전자 표적화의 설계 및 검증
원하는 유전자가 정확하게 표적화되었는지 확인하기 위해 몇 개의 gRNA를 선택하고 HDR 매개 상동 재조합을 위해 5' 및 3' 상동성 암을 절단 부위에 가깝게 배치하는 것이 좋습니다(그림 1A). 예를 들어, 관심 유전자의 시작 코돈의 양쪽을 타겟으로 하는 두 개의 gRNA를 설계하고 QF2-Hsp70-OpIE2-ECFP-SV40 카세트를 마커로 사용하여 HDR 메커니즘을 통해 1kb 상동성 암을 통해 타겟 유전자에 삽입했습니다(그림 1B). 이중 가닥 절단은 gRNA의 절단 부위 사이의 짧은 DNA 단편의 결실로 이어집니다(그림 1A).
gRNA의 절단 효율과 타겟 유전자를 절단하는 능력은 in-vitro Cas9 절단 분석으로 테스트되었습니다. 타겟 유전자의 PCR 단편은 gRNA가 적재된 Cas9에 의해 두 개의 단편으로 절단되었습니다(그림 1C). 또한, 제한 효소는 프로토콜의 downstream 단계로 이동하기 전에 donor plasmid의 무결성을 확인하는 데 사용할 수 있습니다. 플라스미드 분해 결과는 겔에 하나 또는 두 개의 밴드를 표시해야 하며, 작은 밴드가 있으면 짧은 단편으로의 성공적인 절단을 나타냅니다(그림 1D, E).
마이크로 주사 바늘의 준비
주사 과정을 시작하기 전에 새로운 마이크로 주사 바늘을 준비하는 것이 중요합니다. 바늘은 약간의 곡선이 있는 돌출된 모양이어야 합니다. 바늘의 약 2/3는 위쪽이 좁아야 하고 나머지 1/3은 더 넓어야 합니다. 바늘 끝의 앞쪽 끝은 좁은 부분에서 넓은 부분까지 길이가 0.5cm를 초과해서는 안 됩니다(그림 2A). 커버슬립의 가장자리를 사용하여 바늘을 연 후, 검은 모기 배아의 표면을 부드럽게 만져 바늘을 부러뜨린 후 사용하기 쉽기 때문에 미세주입에는 끝이 비스듬한 바늘이 바람직합니다(그림 2B). 반면에, 끝이 뭉툭한 바늘은 미세주입 중에 밝은 회색 배아를 뚫는 것을 어렵게 만들 수 있습니다(그림 2C). 바늘은 바늘이 부러지지 않도록 모기 배아에 효과적으로 침투할 수 있을 만큼 너무 부드럽거나 너무 단단하지 않아야 합니다.
배아 주입
성공적인 주입을 위해서는 밝은 회색 모기 배아를 채취하여 후극이 같은 방향을 향하도록 정렬하는 것으로 시작합니다(그림 3A). 이러한 방향성 정렬은 플라스미드를 생식세포(후극)에 가깝게 주입하는 데 도움이 되며, 이는 다음 세대에 유전될 수 있는 돌연변이를 생성하는 데 중요합니다. 5-10일 전에 혈액을 공급받은 암컷의 신선하고 균일한 밝은 회색 배아를 선택합니다(그림 3A1). 고르지 않은 회색 또는 갈색(그림 3A2)인 배아는 제대로 발달하지 않을 수 있으므로 사용하지 마십시오.
정확하고 효율적인 주입을 용이하게 하기 위해 양면 테이프에 모기 배아를 하나씩 정렬합니다. 알이 건조되고 중간 또는 짙은 회색으로 어두워지기 전에 할로카본 오일을 바르십시오(그림 3B).
주입할 때는 각 배아의 후극 표면에 초점을 맞춥니다. 반면에 배아의 앞쪽( 그림 5의 오른쪽)은 평평하고 단단한 표면(마이크로파일)을 가지고 있습니다. 주입 혼합물이 들어 있는 비스듬한 바늘을 사용하여 배아를 관통합니다(그림 5). 배아의 표면이 눌렸다가 다시 튀어 오르는데, 이는 바늘이 배아를 관통했음을 나타냅니다. 성공적으로 주입되면 몇 초 내에 배아의 색이 밝아집니다( 오른쪽 상단 그림 5의 노란색 곡선).
주입 후 브러시를 조심스럽게 사용하여 배아를 집어 실험실용 물티슈를 사용하여 다른 젖은 여과지로 옮겨 과도한 오일을 제거합니다(그림 6A). 배아를 젖은 종이 타월에 올려 사육 온도에서 며칠 동안 보관합니다(그림 6B). 과도한 기름은 모기 배아의 부화 속도를 감소시킬 수 있습니다. 수분을 유지하기 위해 초순수에 화장솜을 사용하십시오. 주입 후 2시간 후에도 회색 또는 갈색(노란색 화살표)으로 남아 있는 배아는 부화할 가능성이 낮습니다(그림 6C).
스크리닝 및 아웃크로싱
배아가 성숙하면 난자에 초순수를 넣고 27°C의 인큐베이터에 넣어 부화시킵니다. 유충이 3번째 인스타 단계로 발달하는 데 일반적으로 5-7일이 걸립니다. 초기에는 형광을 보이는 G0 유충을 사전 선별했지만, 다른 돌연변이 계통에 비해 약하거나 부분적이었습니다. 이를 통해 마커가 올바르게 발현되고 플라스미드가 배아에 성공적으로 주입되었음을 확인할 수 있었습니다.
그런 다음 이 유충을 야생형(WT) 모기와 교배했습니다. 후속 스크리닝에서 우리는 사육, 라인 스톡 확장 및 추가 실험을 수행하기 위해 더 강력하고 완전한 형광 발현을 보이는 G1 유충을 선택하고 유지했습니다. G1 형광 유충은 리포터 프로모터와 형광 단백질의 조합에 의해 제공되는 형광 단백질 발현 패턴에 따라 식별될 수 있습니다: 프로모터 Hr5Ie1-eGFP(그림 7A)는 전신에 걸쳐 녹색 형광 단백질의 발현을 주도합니다. Hr5Ie1-DsRed(그림 7B)는 체내에서 적색 형광의 발현을 유도합니다. OpIE2-CFP(그림 7C)는 신체의 특정 부위에서만 청록색(청색) 형광 단백질의 발현을 유도합니다. 3xP3-Td토마토 조합을 사용하면 적색 형광 단백질이 눈에서 특이적으로 발현됩니다(그림 7D). 이 특정 형질전환 모기 계통에는 전신에서 CFP 발현을 유도하는 OpIE2 프로모터도 포함되어 있습니다(그림 7D).
녹아웃 돌연변이체에 대한 동형접합 설립의 특수 과정
라인 유지 관리를 단순화하고 모든 모기가 돌연변이가 되도록 하기 위해 우리는 비형광 동형접합 돌연변이 모기를 생성하는 파이프라인을 구현했습니다. 프로토콜 단계 15에서 설명한 바와 같이, WT 모기와 교배한 후, G6 돌연변이 모기를 교배를 위해 동일한 케이지에 배치했습니다(그림 9A). 교배 후, 암컷은 알을 낳기 위해 개별 바이알에 수집되었습니다(그림 9B). 이 단계를 통해 유전자형이 다른 암컷을 분리 할 수있었습니다.
G7을 얻기 위해 난자를 채취한 후, G6 암컷에서 게놈 DNA를 추출한 후, 제한 효소로 처리하거나 처리하지 않은 PCR 단편의 PCR 및 겔 전기영동을 수행했습니다(그림 9C). 돌연변이가 없음을 나타내는 소화된 PCR 단편만 보여주는 라인은 폐기되었습니다. 돌연변이의 존재를 나타내는 이중 PCR 밴드를 보여주는 선과 이형접합 암컷은 유지되고 부화되었습니다(그림 9D). 그런 다음 G7 모기를 성숙하게 하고 모든 성체를 같은 우리에 넣어 동형접합 계통을 형성할 가능성을 높이기 위해 또 다른 교배를 진행했습니다(그림 9E). 혈액을 공급받은 G7 암컷은 G8 난자 채취를 위해 다시 개별적으로 수집되었으며, 이는 세 가지 잠재적인 유전자형을 나타냅니다(그림 9F). G7 암컷에서 게놈 DNA를 추출하고, PCR에 이어 겔 전기영동을 수행하여 동형접합성을 확인했습니다(그림 9G). 돌연변이가 없거나 이형접합적인 암컷의 난자는 버리고 동형접합 결과를 가진 난자만 보관했습니다(그림 9H). H 단계의 G8 알을 부화시켰고, 그 결과로 생긴 성충을 별도의 우리에서 교배시켰다(그림 9I). 각 케이지에서 10명의 성인 수컷을 무작위로 선택하여 겔 전기영동 및 염기서열분석을 수행했습니다(그림 9J). 이 단계에서는 동형접합 개체가 생성되어야 하며, 이는 스톡으로 유지되었습니다(그림 9K).
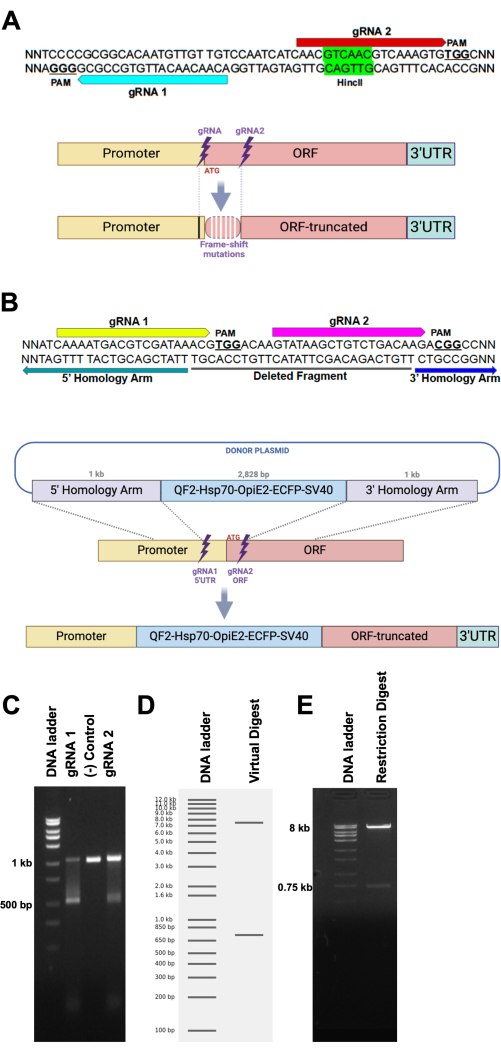
그림 1: gRNA 및 HDR 공여체 플라스미드 설계의 개략도. (A, 상단) 두 개의 gRNA에 대한 가상의 DNA 염기서열 표적 염기서열(파란색 및 빨간색 화살표 및 관련 PAM 부위와 제한 효소 표적 부위(HincII)). (A, 아래) NHEJ에 종속된 타겟 부위의 개략적 표현으로, 작은 염기서열 삭제 또는 삽입으로 인해 프레임 시프트 돌연변이(흰색 수직 막대)가 출현합니다. 돌연변이는 gRNA의 절단 부위(검은색 수직 막대)를 넘어 확장될 수 있습니다. (B, 위) gRNA 1(노란색 화살표) 및 gRNA 2(자홍색 화살표)의 타겟 부위를 묘사한 가상의 DNA 염기서열과 PAM 부위(NGG, 굵게 및 밑줄이 그어진 표시)를 보여줍니다. 두 gRNA 사이의 잠재적으로 삭제된 DNA 염기서열은 검은색 막대(deleted fragment)로 밑줄이 그어집니다. 녹색 및 파란색 화살표는 각각 5' 상동성 암의 최대 3' 시퀀스와 3' 상동성 암의 최대 5' 시퀀스를 강조 표시합니다. (B, 아래) 플라스미드에서 게놈의 상동 부위까지 DNA 카세트의 HDR 재조합에 대한 개략도. (C) 체외 절단 분석. 1 kb DNA 단편의 배양은 gRNA 1 (좌측 차선) 또는 gRNA 2 (우측 차선)가 적재된 Cas9에 의해 절단되어 500 bp의 단편이 됩니다. 중앙 차선은 gRNA가 없는 negative control 반응을 보여줍니다. (D) 두 개의 단일 절단 제한 효소가 있는 플라스미드(8.75 kb)의 가상 분해로 가상 겔에 사실상 두 개의 밴드(8 kb 및 750 bp)가 생성됩니다. (E) 플라스미드의 제한 분해 반응이 로드된 아가로스 겔로, 가상 분해에 의해 제공되는 예상 결과를 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2: 미세주입 바늘 모양. (A) 바늘 끝의 대표 이미지. (B) 모기 알의 미세 주입을 위한 이상적인 바늘 끝 모양. (C) 피해야 할 바늘 끝 모양. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 3: 배아 채취. (A) 주사를 위해 밝은 회색 및 (A1) 균질하지만 (A2) 고르지 않은 배아를 수집하는 단계; 눈금 막대 = 500 μm. (B) 정렬 된 배아에 대한 이미지; 스케일 바=500 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
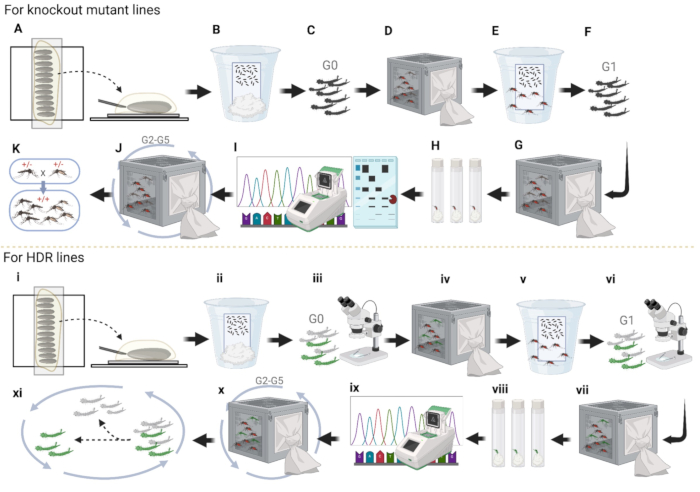
그림 4: 모기 배아 미세주입 파이프라인. (일대) 녹아웃 라인 주입. (A) 모기 배아를 정렬하고 플라스미드를 뒤쪽 끝에 주입합니다. (B) 젖은 종이와 함께 컵에 주입된 배아 저장고; (C) G0 유충이 부화하여 암수 모두로 분리됩니다. (D) 별도의 케이지에서 WT로 건너가는 성인의 남녀 모두; (E) 알 컵에 암컷의 알을 모으는 것. (F) G1 유충이 부화하여 암수 모두로 분리됩니다. (G) 별도의 케이지에서 WT로 건너가는 성인의 남녀 모두. (H) 플라스틱 바이알에 G1 암컷 난자를 개별적으로 모아 알을 부화시킵니다. (I) PCR, 효소 처리된 단편으로 겔을 실행하고 성공적으로 난자를 부화한 G1 단일 암컷에 대한 염기서열분석을 수행합니다. (J) G2를 WT와 3세대 더 교배하고 G1에 대해 했던 것과 동일한 방식으로 모기의 염기서열을 분석한다. (K) 교배를 통해 동형접합체를 확립합니다. (나-시) HDR 라인 주입. (i) 배아의 정렬. (ii) 젖은 종이와 함께 컵에 배아 저장을 주입했습니다. (iii) 형광을 위한 G0 유충의 사전 스크리닝. (iv) 형광 유충을 성충으로 성장시키고 케이지에서 WT와 교배합니다. (v) 달걀 컵에 G1 달걀 수집. (vi) 형광 G1 유충의 선별 및 선택. (vii) WT와 교배한 G1 형광 성체.(viii) 플라스틱 바이알에 개별적으로 알을 낳는 혈액 공급 형광 G1 성체 암컷. (ix) 형광 G1 성인의 개별 수집 및 염기서열분석. (x) 3 세대 더 야생형과 교배하고 (xi) 동형 접합체를 형성하기 위해 형광이없는 유충을 처분합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 5: 배아 주입. 모기 배아에 플라스미드가 주입되는 순간. 삽입 사진: 배아 내부에서 흐린 액체로 시각화된 주입 혼합물(노란색 점선); 눈금 막대 = 200 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
그림 6: 배아 채취. (A) 주입 후 배아를 채취하여 젖은 여과지에 이식하는 단계. 눈금 막대 = 7.5mm. (B) 주입 된 배아를 컵에 유지합니다. 눈금 막대 = 25mm. (C) 주입 후 어두운 배아; 스케일 바=500 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
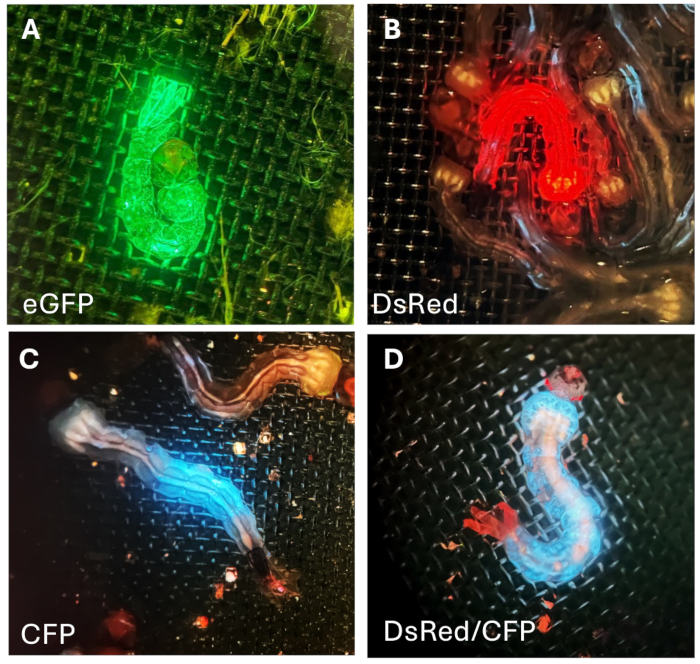
그림 7: G1 유충의 형광. (A) 녹색, (B) 빨간색, (C)파란색 형광 단백질이 모기 유충의 몸에 있습니다. (D) 눈 특이적 적색 형광과 신체 특이적 청색 형광을 보이는 유충. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 8: 번데기 형태학의 성적 이형성. (A1,A2) 수컷 번데기는 더 작은 몸 크기, 두드러지고 뾰족한 생식엽, 꼬리 끝의 더 넓은 패들이 특징입니다. (지하 1층, 지하 2층) 암컷 번데기는 더 큰 몸 크기, 더 작고 덜 뚜렷한 생식기, 더 좁은 꼬리 끝 패들을 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
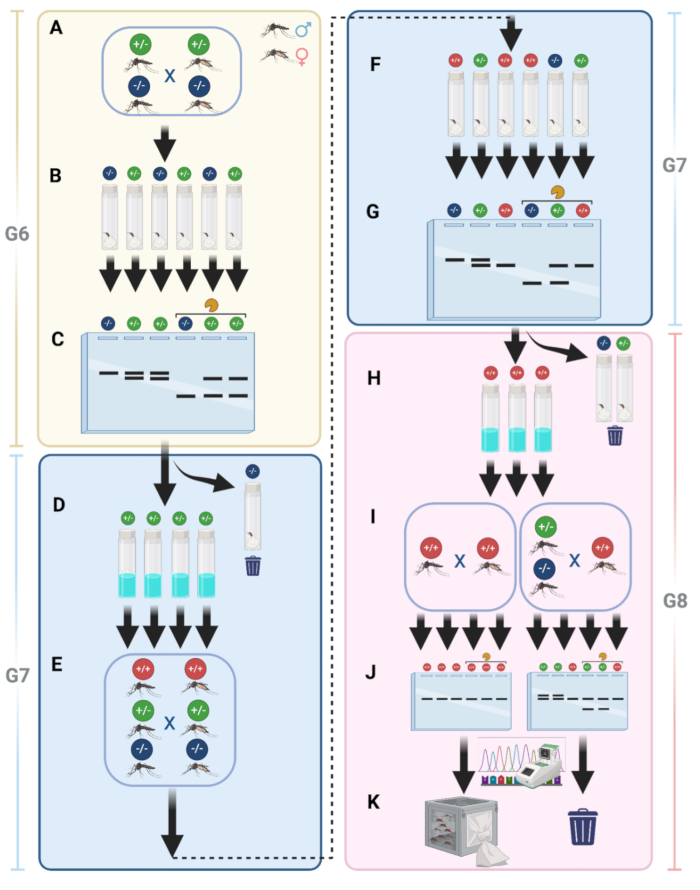
그림 9: 녹아웃 돌연변이 계통에 대한 동형접합 확립에 대한 세부 정보. (A) 돌연변이 모기는 WT와 교배한 후 같은 우리에서 교배합니다.(B) 교배 후 개별 암컷은 알을 낳기 위해 유리병에 모입니다. (C) G6 암컷에서 게놈 DNA를 추출한 후 효소 처리 및 처리되지 않은 단편을 사용하여 PCR 및 겔 전기영동을 수행합니다. (D) PCR 결과가 양성인 G7 난자(이형접합적)가 부화합니다. (E) G7 성체는 동형접합 개체를 얻을 가능성을 높이기 위해 다시 교배됩니다. (F) 혈액을 공급받은 G7 암컷은 G8 난자 채취를 위해 개별적으로 수집되며, 이는 세 가지 유전자형을 나타냅니다. (G) G7 암컷에서 게놈 DNA 추출, PCR 및 겔 전기영동을 수행하여 동형접합 개체를 식별합니다. (H) 돌연변이가 없거나 이형접합적인 암컷의 난자는 버려진다. 동형접합 난자는 보관됩니다. (I) G8 알은 별도의 우리에서 부화하여 교배됩니다. (J) 겔 전기영동과 염기서열분석을 위해 각 케이지에서 10마리의 성인 수컷을 무작위로 선택합니다. (K) 동형접합 모기는 재고로 확인되고 유지됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 주입 시공 | 최종 집중 |
| Cas9 단백질 | 100-300 ng/μL |
| 지RNA | 50-100 ng/μL |
| 공여체 플라스미드 | 100-500 ng/μL |
표 1: 각 주입 구조체에 대해 제안된 최종 농도 범위.
토론
CRISPR-Cas 기술은 염색체1의 표적 특이적 변화를 촉진하여 게놈 편집의 지형을 변화시켰습니다. 비록 transposable elements가 최초의 형질전환 모기의 생성에 필수적이었음에도 불구하고, 그들의 삽입 부위는 다소 무작위적이며, cargo construct (promoter + gene)의 발현은 일반적으로 이소성 발현을 유발하는 게놈 위치 효과(즉, 삽입 부위)로 인해 실제 유전자의 발현 프로파일과 일치하지 않을 수 있습니다21. CRISPR-Cas9 분자 가위가 등장하기 전, 특정 표적 부위에서 DNA 이중 가닥 절단을 촉진하는 기술이 개발되었으며 TALEN22 및 Zinc Finger23과 같은 FokI 제한 효소에 태그된 특정 단백질 도메인의 DNA 결합 특성에 의존했습니다. 후자의 기술은 모기 유전 공학에 성공적으로 활용되었지만, 24,25, 이러한 영역을 엔지니어링하는 데 드는 높은 비용과 off-target cleavage의 가능성으로 인해 이러한 기술이 중단되었습니다. 그 이후로 CRISPR-Cas9 기술은11의 분자 가위가 되었습니다.
모기의 유전 공학은 대부분 배아에 형질전환 혼합물을 주입하여 수행되었습니다26,27. 모기 배아 미세주입을 위해서는 집중력, 인내심, 그리고 세심한 주의가 필요합니다. 알을 낳은 후 2시간 동안 이러한 배아는 발달을 방해하지 않도록 가능한 한 조심스럽게 정렬하고, 주입하고, 세척해야 합니다. 집중은 라인업 중에 난자가 손상되거나 배아가 건조될 가능성을 줄이기 위한 필수 기술입니다. Cas9 단백질이 혼합되어 있을 때 매우 자주 발생하는 바늘 막힘, 팁 파손 및 주입 혼합물 누출과 같은 예기치 않은 사고에 직면하여 인내심은 이러한 상황을 관리하는 데 필수적인 또 다른 기술입니다. 바늘 끝이 너무 크거나 주입 혼합물이 너무 많이 누출될 때 배아를 찌르는 것은 배아를 손상시킬 수 있습니다. 가장 좋은 해결책은 바늘을 교체하고 주사를 계속하는 것입니다. 그에 못지 않게 중요한 것은 세부 사항에 대한 관심입니다. 어린 암컷 모기(생후 6-10일)는 나이 든 모기보다 더 많은 알을 낳는데, 이는 건강한 배아를 수확하는 데 도움이 됩니다. 오염된 물이나 표백된 표면과 접촉하면 배아 사망률이 크게 증가할 수 있으므로 미세 주입 후 배아를 보관하기 위해 탈이온수 또는 초순수와 갈색(표백되지 않은) 종이 타월을 선택하십시오. 밝은 회색(<1시간 된) 배아는 더 부드러워 주입 절차를 용이하게 하여 돌연변이 계통을 얻을 가능성을 높입니다. 또한, 주입된 배아는 완전한 회복과 치유를 위해 부화하기 전에 최소 5일 동안 가습된 여과지에 보관해야 합니다.
주입 혼합물의 품질과 순도는 배아 생존에 필수적입니다. plasmid extraction kit 또는 gRNA synthesis kit의 buffers, salt 또는 alcohol은 주입 mix로 전달되어서는 안 됩니다. 비용이 많이 들더라도 gRNA를 정렬하는 옵션은 적절한 RNA folding의 가능성을 높일 수 있습니다. 또한 Cas9을 인코딩하는 플라스미드 위에 재조합 Cas9 단백질을 주입하는 것이 좋은데, 이는 DNA에 작용하기 위해 전사 및 번역된 후 gRNA를 로드해야 하는 플라스미드 소스와 비교하여 재조합 버전이 이미 로드되어 이중 가닥 절단을 수행할 준비가 되어 있다고 믿기 때문입니다.
모기 A. aegypti의 게놈 편집을 위한 CRISPR-Cas9 기술의 사용에 관한 한 우리 손에 가장 잘 맞는 프로토콜에 대해 설명했지만, 필요한 경우 대체품으로도 사용할 수 있는 대체 시약이 문헌에 설명되어 있습니다. 예를 들어, 모든 시약을 현탁하고 주입 혼합물을 초순수로 희석합니다. 대안적으로, 주입 혼합물은 5 mM KCl 및 0.1 M NaPO4 (pH = 6.8) (pH = 6.8) 26 , 28로 만들어진 주입 버퍼에 희석 될 수 있습니다. 배아를 정렬한 후 배아가 건조되는 것을 방지하기 위해 할로카본 오일 700을 사용하는 것 외에도, 할로카본 오일 27(29) 과 27 및 700(30)의 혼합물이 유사한 보호를 제공합니다. 상기된 주입 완충액의 10x 농축 버전은 배아 건조를 방지하기 위해 대안적으로 사용되어 왔다(26). 위에서 언급한 바와 같이, Cas9를 인코딩하는 플라스미드는 상업적으로 이용 가능하고(addgene) 모기에서 CRISPR-Cas9 매개 유전체 편집에 사용되어 왔다31.
모기 형질전환 계통을 얻기 위한 전략 중 NHEJ 매개 knockout 및 HDR 매개 knockin 접근법을 사용할 수 있습니다. 전자의 성공률이 후자보다 훨씬 높지만(32,33), 그림 4에서 설명한 것처럼 교차 계획과 스크리닝 프로세스는 녹인 라인보다 녹아웃을 얻는 데 훨씬 더 많은 시간이 소요됩니다. 복잡한 교차 계획을 거치지 않고 녹아웃 라인을 얻기 위한 대안적인 접근 방식으로, 형광 마커를 암호화하는 유전자 카세트의 HDR 매개 노킨은 유전자의 암호화 서열을 방해하고 형광 마커14,34를 사용하여 모기의 형질전환 라인을 선택할 수 있는 가능성을 제공합니다.
이 프로토콜은 모기의 유전자 변형을 위한 기본 단계에만 초점을 맞추고 있지만, HDR을 위한 공여 플라스미드를 설계하는 것은 고려해야 할 필수 단계입니다. 3xP3, OpIE2, Hr5-IE1 및 Pub 6,35,36,37,38과 같은 프로모터를 사용하는 것이 중요합니다. 3xP3는 곤충의 신경계와 눈에서 발현을 촉진하는 데 일반적으로 사용되는 합성 프로모터입니다. OpIE2 및 Hr5-IE1은 곤충 세포에서 강력한 발현을 유도하는 데 사용되는 바큘로바이러스에서 유래한 프로모터입니다. 펍 프로모터는 몸 전체의 표현을 촉진합니다. U6 promoter는 gRNA의 발현에 사용됩니다. dsRed(및 TdTomato), eGFP, CFP 및 YFP와 같은 형광 마커도 HDR용 공여 플라스미드에 사용됩니다. 이들은 각각 빨간색, 녹색, 청록색 및 노란색 형광을 방출하는 형광 단백질입니다. 3' UTR의 경우, p1039 및 SV4040은 전이유전자에서 mRNA 안정성과 번역 효율을 향상시키기 위해 자주 사용됩니다.
미세주입 절차를 요약하자면, 녹아웃 돌연변이 세포주의 경우 먼저 주입을 위해 양면 테이프에 모기 배아를 나란히 정렬합니다(그림 4A). 둘째, 배아의 생존율을 촉진하기 위해 대부분의 할로카본 오일을 제거하고 배아를 양면 테이프에서 깨끗하고 젖은 여과지로 옮깁니다. 배아가 완전히 발달할 때까지 며칠 동안 높은 수분을 유지하기 위해 초순수가 담긴 컵에 보관합니다(그림 4B). 셋째, 주입된 배아를 부화시키고 번데기를 암수로 분리한 다음 다른 우리에 넣습니다(그림 4C). 넷째, 모든 형광 수컷을 하나의 케이지에 넣어 5-7일 된 WT 암컷과 교배하거나 그 반대의 경우도 마찬가지입니다 (그림 4D). 다섯째, 교배된 WT 암컷에서 G1 알을 수집하여 처음에 모기 수를 확장합니다(그림 4E). 여섯째, G1 알을 부화시키고 번데기를 암수 두 개로 분리하는 것입니다(그림 4F). 일곱째, G1 성인은 WT와 다시 교차합니다(그림 4G). 여덟째, 개별 G2 난자 채취를 위해 G1 성인 암컷을 바이알에 수집하고 면봉이나 종이 타월을 사용하여 며칠 동안 바이알 바닥에 초순수를 유지합니다(그림 4H). 아홉째, 알을 낳은 G1 암컷을 수집하고 PCR 및 염기서열분석을 위해 G1 암컷에서 게놈 DNA를 추출합니다(그림 4I). 열째, 계통 유지를 위해 올바른 G2 알을 부화시키고 대부분의 연결되지 않은 배경 돌연변이를 제거하기 위해 3세대 동안 WT와 교배합니다(그림 4J). 열한 번째, PCR, 제한 효소 분석, 겔 전기영동 및 염기서열분석을 사용하여 돌연변이를 스크리닝하여 동형접합 모기 계통을 확립합니다(그림 4K).
HDR 라인을 구축하기 위해 배아 미세주입을 위해 양면 테이프에 모기 배아를 나란히 정렬합니다(그림 4i). 둘째, 주사 후 대부분의 할로카본 오일을 제거하고 배아를 초순수가 담긴 컵에 보관하여 며칠 동안 수분을 유지하고 배아가 성숙할 때까지 기다립니다(그림 4ii). 셋째, 부화하여 주입된 배아를 만들고 G0 유충을 선별하여 형광이 있거나 없는 배아를 분류합니다(그림 4iii). 형광이 있는 유충은 플라스미드가 모기 배아에 성공적으로 주입되었음을 의미합니다. 넷째, 형광 유충이 성충으로 성숙하도록 하고 모든 형광 수컷을 하나의 우리에 넣어 WT 암컷과 교배하거나 그 반대로 모기 수를 확장하도록 합니다(그림 4iv). 다섯째, 교배된 WT 암컷의 난자를 수집합니다(그림 4v). 여섯째, G1 형광 유충을 부화, 선별 및 분류하고 형광이 없는 유충을 처분합니다(그림 4vi). 일곱째, WT를 가진 교차 G1 형광 성인(그림 4vii). 여덟째, 플라스틱 바이알에 G2 알을 개별적으로 수집하여 모기에서 플라스미드의 다른 삽입 부위를 분리합니다(그림 4viii). 아홉째, 알을 부화시키고, 모기의 적합성을 확인하고, 염기서열분석을 수행합니다(그림 4ix). 열째, 표현형이 더 강한 두 라인을 선택하고 3세대 더 WT와 교배합니다(그림 4x). 열한 번째, 형광을 가진 유충을 선택하여 추가 실험을 위한 동형접합 계통을 확립하기 위해 2세대 더 교배합니다(그림 4xi).
여기에서 우리는 여러 형질전환 세포주를 생성하는 데 사용한 CRISPR-Cas9을 사용한 A. aegypti 게놈 편집을 위한 가장 포괄적인 프로토콜을 제공했습니다. 우리는 이 프로토콜을 마이크로주입 시험에서 얻은 경험을 바탕으로 변경해야 하는 다른 연구자들을 위한 출발점으로 사용할 것을 제안합니다. 항상 다양한 시약과 조건을 테스트해야 합니다. 모기의 유전자 편집 성공률은 아직 완전히 최적화되지 않았으며 CRISPR-Cas9을 통해 형질전환 모기를 만들 수 있는 실험실은 거의 없습니다.
공개
O.S.A.는 Agragene, Inc.와 Synvect, Inc.의 창립자입니다. 이 합의의 조건은 이해 상충 정책에 따라 University of California, San Diego에서 검토 및 승인했습니다. 나머지 저자는 경쟁 이해관계가 없음을 선언합니다.
감사의 말
저자는 모기 사육을 도와준 Judy Ishikawa와 Ava Stevenson에게 감사를 표합니다. 이 작업은 O.S.A.에 수여된 NIH 상(R01AI151004, RO1AI148300, RO1AI175152)과 N.H.R.에 대한 K22AI166268의 자금 지원으로 이루어졌습니다. 피규어는 BioRender를 사용하여 제작되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| 10x Cas9 reaction buffer | PNA Bio | CB01 | |
| Benchling software | Benchling | N/A | www.benchling.com |
| Cas9 dilution buffer | PNA Bio | CB03 | |
| Cas9 protein | PNA Bio | CP01-50 | |
| DH5α E. coli Competent Cells | New England Biolabs | C2987 | |
| Double-sided sticky tape | Scotch Permanent | 3136 | |
| Drosophila vials | Genesee Scientific | 32-109 | |
| Filter papers | GE Healthcare Life Science | 1450-042 | |
| Fish food | Tetra | B00025Z6YI | goldfish flakes |
| Flugs | Genesee Scientific | AS273 | |
| Fluorescent microscope | Leica Microsystems | M165 FC | |
| Gene fragment | Integrated DNA Technologies | N/A | |
| gRNA | Synthego | N/A | |
| Halocarbon oil 700 | Sigma-Aldrich | H8898 | |
| Injection microscope | Leica Microsystems | DM2000 | |
| JM109 E. coli Competent Cells | Zymo Research | T3005 | |
| Microinjector | Eppendorf | FemtoJet 4x | |
| Microloader Tips for Filling Femtotips | Eppendorf | E5242956003 | |
| Micromanipulator | Eppendorf | TransferMan 4r | |
| Micropipette Pullers | Sutter Instrument | P-2000 | |
| Microscope Cover Glass | Fisherbrand | 12-542-B | |
| Microscope slide | Eisco | 12-550-A3 | |
| Mouse blood (live mice used for feeding) | University of California | IACUC, Animal Use Protocol #S17187 | Used for mosquito blood feeding; details comply with animal ethics protocols |
| NEB Q5 High-Fidelity DNA polymerase | New England Biolabs | M0491S | |
| PCR Purification Kit | Qiagen | 28004 | |
| Plasmid Miniprep Kit | Zymo Research | D4036 | |
| Quartz filament | Sutter Instruments | QF100-70-10 | |
| Transcription Clean-Up Kit | Fisher Scientific | AM1908 | |
| Ultra-pure water | Life Technologies | 10977-023 |
참고문헌
- Anzalone, A. V., Koblan, L. W., Liu, D. R. Genome editing with CRISPR-Cas nucleases, base editors, transposases and prime editors. Nat Biotechnol. 38 (7), 824-844 (2020).
- Garneau, J. E., et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA. Nature. 468 (7320), 67-71 (2010).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Shen, H., Li, Z. DNA double-strand break repairs and their application in plant DNA integration. Genes (Basel). 13 (2), 322 (2022).
- Vinauger, C., et al. Modulation of host learning in Aedes aegypti mosquitoes. Curr Biol. 28 (3), 333-344.e8 (2018).
- Li, M., Bui, M., Yang, T., Bowman, C. S., White, B. J., Akbari, O. S. Germline Cas9 expression yields highly efficient genome engineering in a major worldwide disease vector. Proc Natl Acad Sci U S A. 114 (49), E10540-E10549 (2017).
- Chaverra-Rodriguez, D., et al. Targeted delivery of CRISPR-Cas9 ribonucleoprotein into arthropod ovaries for heritable germline gene editing. Nat Commun. 9 (1), 3008 (2018).
- Rouyar, A., et al. Transgenic line for characterizing GABA-receptor expression to study the neural basis of olfaction in the yellow-fever mosquito. Front Physiol. 15, 1381164 (2024).
- Ang, J. X. D., et al. Considerations for homology-based DNA repair in mosquitoes: Impact of sequence heterology and donor template source. PLoS Genet. 18 (2), e1010060 (2022).
- Zhang, J. -. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biol. 18 (1), 35 (2017).
- Coutinho-Abreu, I. V., Akbari, O. S. Technological advances in mosquito olfaction neurogenetics. Trends Genet. 39 (2), 154-166 (2023).
- Li, M., et al. Targeting sex determination to suppress mosquito populations. eLife. 12, RP90199 (2024).
- Zhan, Y., Alonso San Alberto, D., Rusch, C., Riffell, J. A., Montell, C. Elimination of vision-guided target attraction in Aedes aegypti using CRISPR. Current Biol. 31 (18), 4180-4187.e6 (2021).
- Greppi, C., et al. Mosquito heat seeking is driven by an ancestral cooling receptor. Science. 367 (6478), 681-684 (2020).
- Potter, C. J., Tasic, B., Russler, E. V., Liang, L., Luo, L. The Q system: a repressible binary system for transgene expression, lineage tracing, and mosaic analysis. Cell. 141 (3), 536-548 (2010).
- Riabinina, O., et al. Improved and expanded Q-system reagents for genetic manipulations. Nat Methods. 12 (3), 219-222 (2015).
- Labun, K., et al. CHOPCHOP v3: expanding the CRISPR web toolbox beyond genome editing. Nucleic Acids Res. 47 (W1), W171-W174 (2019).
- Concordet, J. -. P., Haeussler, M. CRISPOR: intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Res. 46 (W1), W242-W245 (2018).
- Williams, A. E., et al. Quantifying fitness costs in transgenic Aedes aegypti mosquitoes. J Vis Exp. , e65136 (2023).
- Bassett, A. R., Tibbit, C., Ponting, C. P., Liu, J. -. L. Highly efficient targeted mutagenesis of Drosophila with the CRISPR/Cas9 system. Cell Rep. 4 (1), 220-228 (2013).
- Coutinho-Abreu, I. V., Zhu, K. Y., Ramalho-Ortigao, M. Transgenesis and paratransgenesis to control insect-borne diseases: current status and future challenges. Parasitol Int. 59 (1), 1-8 (2010).
- Christian, M., et al. Targeting DNA double-strand breaks with TAL effector nucleases. Genetics. 186 (2), 757-761 (2010).
- Kim, Y. G., Cha, J., Chandrasegaran, S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain. Proc Natl Acad Sci U S A. 93 (3), 1156-1160 (1996).
- DeGennaro, M., et al. orco mutant mosquitoes lose strong preference for humans and are not repelled by volatile DEET. Nature. 498 (7455), 487-491 (2013).
- McMeniman, C. J., Corfas, R. A., Matthews, B. J., Ritchie, S. A., Vosshall, L. B. Multimodal integration of carbon dioxide and other sensory cues drives mosquito attraction to humans. Cell. 156 (5), 1060-1071 (2014).
- Lobo, N. F., Clayton, J. R., Fraser, M. J., Kafatos, F. C., Collins, F. H. High efficiency germ-line transformation of mosquitoes. Nat Protoc. 1 (3), 1312-1317 (2006).
- Kistler, K. E., Vosshall, L. B., Matthews, B. J. Genome engineering with CRISPR-Cas9 in the mosquito Aedes aegypti. Cell Rep. 11 (1), 51-60 (2015).
- Handler, A. M., Harrell, R. A. 2nd Transformation of the Caribbean fruit fly, Anastrephasuspensa, with a piggyBac vector marked with polyubiquitin-regulated GFP. Insect Biochem Mol Biol. 31 (2), 199-205 (2001).
- Harrell, R. A. . 2nd Mosquito embryo microinjection under halocarbon oil or in aqueous solution. 2024 (7), (2024).
- Sun, R., Raban, R., Akbari, O. S. Generating mutant strains with transgenic Cas9. Cold Spring Harb Protoc. 2023 (9), 671-678 (2023).
- Giraldo, D., et al. An expanded neurogenetic toolkit to decode olfaction in the African malaria mosquito Anopheles gambiae. Cell Rep Methods. 4 (2), 100714 (2024).
- Liu, G., Lin, Q., Jin, S., Gao, C. The CRISPR-Cas toolbox and gene editing technologies. Mol Cell. 82 (2), 333-347 (2022).
- Chu, V. T., et al. Increasing the efficiency of homology-directed repair for CRISPR-Cas9-induced precise gene editing in mammalian cells. Nat Biotechnol. 33 (5), 543-548 (2015).
- Laursen, W. J., et al. Humidity sensors that alert mosquitoes to nearby hosts and egg-laying sites. Neuron. 111 (6), 874-887.e8 (2023).
- Weng, S. -. C., Antoshechkin, I., Marois, E., Akbari, O. S. Efficient sex separation by exploiting differential alternative splicing of a dominant marker in Aedes aegypti. PLoS Genet. 19 (11), e1011065 (2023).
- Li, M., et al. Development of a confinable gene drive system in the human disease vector. eLife. 9, e51701 (2020).
- Dalla Benetta, E., et al. Engineered Antiviral Sensor Targets Infected Mosquitoes. The CRISPR journal. 6 (6), 543-556 (2023).
- Li, H. -. H., et al. C-Type lectins link immunological and reproductive processes in Aedes aegypti. iScience. 23 (9), 101486 (2020).
- van Oers, M. M., Vlak, J. M., Voorma, H. O., Thomas, A. A. M. Role of the 3' untranslated region of baculovirus p10 mRNA in high-level expression of foreign genes. J Gen Virol. 80 (Pt 8), 2253-2262 (1999).
- Salem, T. Z., et al. The influence of SV40 polyA on gene expression of baculovirus expression vector systems. PloS One. 10 (12), e0145019 (2015).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유