Method Article
Análise quantitativa da estabilização da membrana do retículo endoplasmático (MAM) associada às mitocôndrias em um modelo neural da doença de Alzheimer (DA)
Neste Artigo
Resumo
Aqui, descrevemos a medição da taxa de transporte axonal de estabilizadores constitutivos de membranas do retículo endoplasmático (ER) associadas às mitocôndrias (MAMs), aumentando ou mantendo a geração de β-amilóide (Aβ) neurotóxico a partir de neurônios da doença de Alzheimer (DA) em tempo real para servir como uma métrica direta e quantitativa para medir a estabilização do MAM e auxiliar no desenvolvimento da terapêutica da DA.
Resumo
Um método para quantificar a estabilização das membranas do retículo endoplasmático associadas às mitocôndrias (MAMs) em um modelo neural tridimensional (3D) da doença de Alzheimer (DA) é apresentado aqui. Para começar, células ReN neuroprogenitoras humanas frescas que expressam proteína precursora de β-amilóide (APP) contendo doença de Alzheimer familiar (FAD) ou células ReN virgens são cultivadas em placas finas (1:100) de cultura de tecidos revestidas com Matrigel. Depois que as células atingem a confluência, elas são eletroporadas com plasmídeos de expressão que codificam a sequência de ligação às mitocôndrias conjugada com proteína de fluorescência vermelha (RFP) de AKAP1 (34-63) (Mito-RFP) que detecta mitocôndrias ou estabilizadores constitutivos de MAM MAM 1X ou MAM 9X que estabilizam MAMs apertados (6 nm ± 1 nm de largura de lacuna) ou soltos (24 nm ± 3 nm de largura de lacuna), respectivamente. Após 16-24 h, as células são colhidas e enriquecidas por um classificador de células ativadas por fluorescência (FACS). Um número igual de células enriquecidas com FACS são semeadas na matriz tridimensional (Matrigel 1:1) e podem se diferenciar em neurônios maduros por 10 dias. As imagens de células vivas das células diferenciadas de 10 dias que expressam os estabilizadores MAM conjugados com RFP são capturadas sob um microscópio fluorescente equipado com uma câmara de cultura de imagem de células vivas mantendo o CO2 (5%), temperatura (37 ° C) e umidade (~ 90%). Para esse fim, realizamos imagens de células vivas e análises quimográficas para medir a motilidade das mitocôndrias livres marcadas com mito-RFP ou mitocôndrias ligadas a ER de larguras de gap estreitas ou soltas estabilizadas por MAM 1X ou MAM 9X, respectivamente, no processo neuronal mais estendido de cada neurônio ReN GA que tem pelo menos 500 nm de comprimento, considerando-os como axônios.
Introdução
Evidências emergentes sugerem que os contatos de retículo endoplasmático associados a mitocôndrias (MERCs) especializados, colhidos bioquimicamente como membranas ER associadas a mitocôndrias, muitas vezes chamados de MAMs 1,2, desempenham um papel em várias doenças neurodegenerativas, incluindo AD 3,4. Esses MAMs são compostos de microdomínios lipídicos ricos em colesterol no RE e na membrana externa das mitocôndrias amarrados por uma série de proteínas que criam diversidades estruturais e funcionais entre os MAMs 5,6,7. A hipótese MAM recentemente cunhada postula que o aumento de MAMs leva ao aumento da produção de Aβ e à cascata patogênica da DA, incluindo formação de emaranhado neurofibrilar (NFT), dishomeostase de cálcio e neuroinflamação 3,8. Cerca de 5% a 20% das mitocôndrias fazem contato físico com o RE para formar MAMs9. A largura da folga dos MAMs é determinada pelo ER suave e áspero (sER e rER, respectivamente). A largura variável da lacuna entre sER-mitocôndrias (10-50 nm) e rER-mitocôndrias (50-80 nm) sugere que a largura da lacuna dos MAMs tem um longo espectro que varia entre apertado (~ 10 nm) a solto (~ 80 nm) 10 , 11 , 12 , 13 . A largura da lacuna da MAM determina as funções da MAM, como a homeostase do cálcio e o transporte lipídico 1,14. Um relatório recente mostrou que os MAMs formados entre ER e mitocôndrias fortemente conectados (~ 10 nm), chamados MAMs completos, são apoptóticos. Em contraste, os MAMs formados entre o ER fracamente conectado (~ 25 nm) e as mitocôndrias, denominados MAMs defeituosos ou médios, são antiapoptóticos14 , 15 , 16 . A estabilização de MAMs com uma largura de lacuna de 6 nm ± 1 nm aumentou a geração de Aβ a partir de um novo modelo de cultura neural tridimensional (3D) de DA. Em contraste, a estabilização de MAMs com largura de lacuna de 24 nm ± 3 nm não tem efeito na geração de Aβ17. Essa descoberta sugere pela primeira vez que regular o grau de estabilização do MAM, mas não desestabilizar os MAMs, é a chave para regular a geração de Aβ. Uma tentativa de desestabilizar completamente os MAMs pode ter consequências indesejadas porque os MAMs mantêm vários eventos celulares críticos para a sobrevivência celular12.
A modulação de MAMs é uma área emergente de pesquisa com implicações potenciais para vários distúrbios, incluindo câncer, distúrbios metabólicos e doenças neurodegenerativas18. Apesar da disponibilidade de muitos moduladores de MAM, nenhuma grande tentativa foi feita até agora para testar suas habilidades de desestabilizar MAMs e diminuir a patologia da DA, principalmente porque as diversidades estruturais dos MAMs os tornam um sistema altamente complexo para a descoberta de medicamentos. Mas, a farmacologia de sistemas estruturais recém-desenvolvida, que considera as propriedades específicas dos alvos de drogas e seu ambiente18,19 deve superar as dificuldades e desenvolver drogas altamente potentes direcionadas a MAMs ou proteínas associadas a MAM na DA. No entanto, a busca por um modulador eficaz da estabilização do MAM requer métodos para quantificar o grau de estabilização do MAM com precisão. Técnicas tradicionais como microscopia eletrônica (EM) ou microscopia de super-resolução têm limitações na determinação da estabilização do MAM. Superar esses desafios provavelmente exigiria o desenvolvimento de novas técnicas de imagem mais dinâmicas ou ensaios bioquímicos que possam fornecer medidas quantitativas de estabilização de MAM em células vivas. A microscopia eletrônica de varredura por feixe de íons focalizados (FIB-SEM) de neurônios primários revelou que o ER tende a formar uma rede ao redor das mitocôndrias que provavelmente limitará a motilidade mitocondrial20,21. A interrupção dos sistemas de transporte mitocondrial, retrógrado, anterógrado ou ambos, teve um impacto profundo na função sináptica e neuronal22. Assim, a nova imagem de células vivas e a análise baseada em quimografia da velocidade axonal de mitocôndrias ligadas ao ER descritas aqui como uma métrica para medir quantitativamente a estabilização do MAM facilitarão a identificação do (s) modulador (es) do MAM que podem mudar o limiar de estabilização do MAM para um que mantém ou possivelmente diminui em oposição ao aumento da geração de Aβ.
Protocolo
Modelos de cultura neural AD: Este estudo utilizou neurônios derivados de células ReN progenitoras neurais humanas [ReN ingênuo (Millipore)] ou células ReN expressando mutações familiares de DA (fAD) no gene da proteína precursora de amilóide (APP) (APPSwe / Lon), células ReN GA. O sistema de cultura tridimensional (3D) ReN-GA recapitula a patologia da DA, ou seja, emaranhados neurofibrilares (NFTs) acionados por oligômero Aβ 23,24. Células ReN virgens estão disponíveis comercialmente. As linhas ReN GA foram obtidas do Dr. Doo Y. Kim, Professor Associado, Massachusetts General Hospital (MGH)23,24,25.
Plasmídeos de expressão: AKP1 (34-63) e sequência direcionada a ER de proteínas Ubc 6 (283-303) ligadas diretamente com RFP (Mito-RFP-ER denotado como MAM 1X) ou contêm um ligante de 9 aminoácidos (Mito-9X-RFP-ER denotado como MAM 9X) projetado para estabilizar MAMs de 6 nm ± 1 nm ou 24 nm + 3 nm larguras de lacuna, respectivamente15,26 (Figura 1A).
1. Eletroporação
- Transfecção das células com estabilizadores MAM (1-2 h)
NOTA: Siga o protocolo descrito pelo fabricante do kit nucleofector (Tabela de Materiais). Antes de começar, prepare placas de cultura com fundo de vidro de 6 poços pré-revestidas com DMEM/F12 contendo 1% de Matrigel (doravante referida como matriz de membrana basal [BMM]). Use 2 mL de BMM para revestir cada poço. Incubar durante pelo menos 1 h a 37 °C. A BMM, o meio, as pontas das pipetas e as pipetas devem ser pré-arrefecidos antes de serem misturados para evitar que a BMM se solidifique.- Aspirar a mistura BMM. Substitua por 2 mL de DMEM/F12 à temperatura ambiente (RT) em cada poço.
- Combine o Nucleofector com o Suplemento 1 em uma proporção de 4,5: 1 (82 μL: 18 μL de relação Nucleofector/Suplemento para 100 μL de solução). Para cada transfecção desejada, são necessários 100 μL de Nucleofector
- DNA de vórtice. Em seguida, adicione 1-5 μg de DNA (por tratamento) em tubos de microcentrífuga de 1 mL para cada transfecção.
- Use a acustase para colher uma placa de células ReN-GA saudáveis.
NOTA: Células da placa em meio de expansão (Tabela 1) em placas de cultura de tecidos de 100 mm e aguarde até que a confluência atinja 70-80%.- Aspire o meio existente e lave uma vez com 10 mL de PBS.
- Adicione 1 mL de acutase diretamente às células e incube por 5-10 min a 37 °C.
- Desaloje as células do prato batendo suavemente na lateral do prato. Verifique ao microscópio se as células estão soltas e fluindo livremente.
- Neutralize a acutase com 10 mL de DMEM/F12 e transfira para um tubo limpo de 15 mL.
- Centrifugue o número necessário de células a 274 x g durante 5 min e aspire o sobrenadante para remover as células mortas. Em seguida, ressuspenda o pellet em 10 mL de DMEM/F12.
- Conte as células para determinar a densidade.
NOTA: Neste estudo, um contador de células automatizado foi usado para contar o número de células. Por eletroporação, são necessárias 3-5 X 106 células (por exemplo, 5 tratamentos exigiriam 15-25 X 106 células).- Pegue 10 μL das células suspensas e adicione ao lado A de uma câmara de contagem de células. Em seguida, adicione 10 μL ao lado B.
NOTA: Adicione a suspensão celular inclinando a pipeta para o lado. Evite bolhas não empurrando além do primeiro batente no êmbolo da pipeta. - Leve a câmara de contagem cheia para o contador de células e insira o lado A no slot principal na frente.
- Depois de pressionar Medir, anote o número de células por mL.
- Retire a câmara, vire para o lado B e repita as etapas 1.1.5.2-1.1.5.3.
- Some esses dois números, divida por 4 (se não estiver usando Trypan Blue) e multiplique o número pelos mililitros de líquido em que as células estão suspensas para obter o número total de células na suspensão.
- Pegue 10 μL das células suspensas e adicione ao lado A de uma câmara de contagem de células. Em seguida, adicione 10 μL ao lado B.
- Centrifugue o número necessário de células a 274 x g por 5 min e aspire o sobrenadante.
- Ressuspenda o pellet em 100 μL de mistura de Nucleofector por eletroporação. (por exemplo, 500 μL para 5 tratamentos).
NOTA: Deixar as células na solução de Nucleofector por mais de 15 minutos pode reduzir a viabilidade celular e a eficácia geral. - Adicionar 100 μL da suspensão celular a um dos tubos contendo DNA e misturar por pipetagem.
- Transfira a mistura de DNA para uma cubeta de eletroporação e sele com a tampa fornecida. Para evitar a criação de bolhas, incline a cubeta para baixo e pipete lentamente.
- Selecione o programa Nucleofector apropriado para o dispositivo que está sendo usado. Para o dispositivo usado neste estudo, use o Programa A-033 para transfecção. Para otimizar, experimente todos os 5 programas Nucleofector para determinar o mais apropriado para cada tipo de célula.
NOTA: Uma confirmação de uma eletroporação bem-sucedida é a espuma visível na parte superior da mistura. - Imediatamente, usando as pipetas estéreis fornecidas, adicione ~ 500 μL do DMEM da placa pré-cheia de 6 poços na cubeta. Misture delicadamente uma vez e transfira as células eletroporadas e o meio para o poço correspondente.
NOTA: Após a eletroporação, as células são extremamente sensíveis, portanto, transferir o meio rapidamente e pipetar com cuidado é essencial. - Repita as etapas 1.1.8 a 1.1.11 para todos os tratamentos de DNA restantes.
- Incubar as células a 37 °C na presença de CO2 (5%) durante a noite.
- No dia seguinte, troque o meio por meio de diferenciação fresco (Tabela 2) e deixe as células se diferenciarem por 10 dias. A cada 2-3 dias, substitua por mídia nova.
2. Imagem de células vivas
- Preparação de células para microscopia de células vivas (30 min)
- Preparação da câmara de células vivas (antes de mover as células para a câmara)
- Certifique-se de que o tanque de CO2 e o umidificador estejam conectados à câmara, as válvulas estejam abertas e os tanques cheios.
- Defina a temperatura para 37 °C, o CO2 para 5% e a umidade para 95%. (Isso pode levar algum tempo para que os níveis se equilibrem).
- Coloque uma placa de 6 poços contendo células na câmara e ajuste o foco do microscópio até que as células se tornem visíveis.
- Ligue o laser (Tabela de Materiais).
- Para capturar o sinal RFP, excite o fluoróforo usando um laser de 594 nm e use a emissão de 570-640 nm. Para GFP, use um laser de 488 nm para excitação e emissão de 510-540 nm.
- Usando o filtro fluorescente embutido, ajuste a intensidade do sinal até que o sinal de fundo se dissipe (deve ser quase preto sólido).
- Captura de vídeo ao vivo de axônios (10 h-15 h no total)
NOTA: Um microscópio confocal invertido Nikon C2 Eclipse Ti2 foi usado para capturar imagens fluorescentes usando o software NIS Element AR. Use ampliação de 60x em uma resolução de 512 pixels, gravando vídeos a 1 quadro por segundo por 3 minutos produziu quimógrafos mais limpos.- Encontre uma célula que expresse biossensores RFP. Exporte uma imagem da célula para referência posterior antes de gravar um vídeo.
- Corte a área de varredura para caber ao redor do axônio. O uso de uma área de varredura menor reduz o tempo de processamento do microscópio e torna a geração de quimografia muito mais fácil.
- Desligue todos os lasers para ajudar o software a funcionar com mais facilidade. Observe que, quando o programa é executado com todos os lasers ativos, nem todos os quadros são capturados.
NOTA: O sinal fluorescente vermelho do soma é muito mais brilhante do que no axônio. Portanto, o soma é excluído para aumentar a intensidade geral do sinal no axônio. Vá para a guia Medição de tempo . - Defina o intervalo para 1 quadro por segundo e defina o tempo geral em 181 s.
- Clique em Executar.
- Salve este arquivo como um arquivo .nds (devidamente rotulado) e repita o processo ~ 10 vezes por estabilizadores MAM (MAM 1X ou MAM 9X).
NOTA: A força do laser às vezes produzia um efeito de branqueamento perceptível no sinal RFP se escaneado por muito tempo. É importante considerar trabalhar rapidamente para capturar vídeos.
3. Pós-processamento (7 dias)
NOTA: Para analisar o transporte e gerar quimógrafos, foram utilizadas macros Fiji ImageJ. As vesículas que se deslocaram menos de 0,1 mm/s foram categorizadas como estacionárias. A frequência de movimento das partículas foi calculada dividindo-se o número de partículas que se movem em uma determinada direção (anterógrada, retrógrada) ou não se movem (estacionária) pelo número total de partículas analisadas no quimógrafo. O tempo que cada vesícula passou pausando ou se movendo foi calculado pela média da porcentagem de tempo gasto em cada condição para todas as vesículas em cada neurônio analisado. A distribuição de frequência para velocidade e comprimento de corrida foi calculada usando apenas vesículas móveis para cada condição experimental. A análise foi realizada em tratos axonais de 100 mm por 3 min.
- Gerando um quimógrafo
- Abra o arquivo .nds no Fiji ImageJ.
- Vá para a guia Imagem e clique em Propriedades. Registre a proporção de pixel para mícron. Isso é necessário para o cálculo posterior.
- Clique em Arquivo > Salvar como > Tiff para salvar o arquivo como um .tiff em sua pasta (a macro salvará automaticamente tudo o que for gerado nesta pasta).
- Arraste a macro Kymo para ImageJ. O código é fornecido no Arquivo de Codificação Suplementar 1. Clique em Executar.
- Não clique em OK. Alterne para a janela MAX_raw. Clique manualmente com o botão esquerdo ao longo do axônio e clique duas vezes para encerrar o rastreamento.
NOTA: Certifique-se de rastrear o axônio do soma para o terminal do axônio. Desta forma, os cálculos anterógrados e retrógrados estarão corretos. - Pressione o comando T ou Adicionar no gerenciador de ROI.
- Agora clique em OK. Um quimógrafo será gerado na pasta feita anteriormente. (O eixo X é o comprimento do axônio em mícrons e o eixo Y é o tempo em segundos).
- Rastreando o quimógrafo
- Arraste o quimógrafo para Fiji Image J (deve ser colocado automaticamente na pasta feita anteriormente).
- Arraste a macro Trilha para a Imagem J.
- No prompt a seguir, clique em Ok. Se estiver retomando o trabalho de antes, insira o ponto numérico para continuar e pressione Ok.
- Usando a ferramenta Retângulo no painel Selecionar , selecione uma área próxima ao meio do quimógrafo que tenha 60 pixels de altura e sempre 100 μm de diâmetro. Para fazer isso, divida 100 pela proporção de pixel para micro que foi registrada na etapa 3.1.2.
- Depois de selecionar uma área, pressione Ctrl T para adicionar i t ao ROI e salve o ROI na pasta que está sendo trabalhada (pressione Mais no menu ROI e depois Salvar).
NOTA: Inverter a área selecionada pressionando Ctrl+Shift+I facilitará o rastreamento. - Para rastrear o movimento de uma vesícula individual, segure Ctrl enquanto clica com o botão esquerdo de cima para baixo.
- Para ser feito, segure Ctrl enquanto clica com o botão direito do mouse e uma janela aparecerá mostrando onde os axônios são rastreados. Se isso parecer bom, pressione Ok.
- Para continuar e selecionar outra vesícula, clique em Sim e repita o processo das etapas 3.2.6 a 3.2.8. Depois que todas as vesículas visíveis forem rastreadas, pressione Não. Isso salvará automaticamente a sobreposição rastreada manualmente na mesma pasta.
- Medindo os dados do quimógrafo
- Crie uma nova pasta. Arraste todos os arquivos de texto gerados para a pasta.
- Abra o arquivo do quimógrafo e registre as dimensões.
- Arraste a macro Medida para a Imagem J.
- Insira a proporção de mícron para pixel registrada anteriormente em PixelScale.
- Altere o limite de velocidade baixo para 0,1. (As vesículas que se movem abaixo deste limite são consideradas estacionárias).
- Insira as dimensões do quimógrafo registradas anteriormente. O arquivo de resumo será gerado automaticamente na pasta de texto criada. Cada preenchimento de resumo conterá %Tempo Percorrido, Velocidade Geral (μm/s), Distância Total, Segmento Médio Percorrido, Número de Vezes Parado e Número de Vezes Revertido. O código para gerar, rastrear e medir os dados do quimógrafo é fornecido como Arquivo de Codificação Suplementar 1.
Resultados
Imagens de células vivas e análises quimográficas foram realizadas para medir a motilidade de mitocôndrias livres marcadas com mito-RFP ou mitocôndrias ligadas a ER de larguras de contato apertadas (6 nm ± 1 nm) ou soltas (24 nm ± 3 nm) estabilizadas por MAM 1X ou MAM 9X, respectivamente, no processo neuronal mais longo de cada neurônio ReN GA (AD) ou ReN (ingênuo) com pelo menos 500 nm de comprimento, considerando-o como um axônio (Figura 1 e Figura 2). As frequências de movimentos (geral, retrógrado e anterógrado) foram calculadas dividindo-se o número de pontos marcados com RFP móveis ou estacionários (MAMs) pelo número total nos quimógrafos (Figura 1A-E). A velocidade axonal geral das mitocôndrias ligadas a ER marcadas com MAM 1X foi drasticamente diminuída em ~ 50% em comparação com as mitocôndrias livres de ER marcadas com Mito-RFP ou mitocôndrias ligadas a ER marcadas com MAM 9X ( Figura 1B ). A análise quantitativa também revelou diferenças dramáticas entre os movimentos gerais e retrógrados das mitocôndrias ligadas ao ER estabilizadas com MAM 1X em comparação com as mitocôndrias livres (Mito-RFP) ou estabilizadas com MAM 9X ligadas ao ER. Enquanto 53,82% ± 3,3% das mitocôndrias livres de ER (Mito-RFP) eram móveis, apenas 26,6% ± 3,4% das mitocôndrias ligadas ao ER marcadas com MAM 1X eram móveis, sugerindo que a estabilização dos MAMs reduziu significativamente a mobilidade axonal geral das mitocôndrias, fortemente associadas ao ER, em comparação com as mitocôndrias não ligadas ou fracamente ligadas ao RE (44,79% ± 2,6% do MAM 9X versus 53,82% ± 3,3% do Mito RFP, respectivamente) (Figura 1C). Consistentemente, os movimentos retrógrados e anterógrados das mitocôndrias ligadas ao ER marcadas com MAM 1X foram significativamente menores em comparação com as mitocôndrias livres ou marcadas com MAM 9X (Mito-RFP) (retrógrado: 12,33% ± 2,55% para MAM 1X versus 25,78% ± 2,31% para Mito RFP; anterógrado: 14,27% ± 2,81% para MAM 1X versus 28,04% ± 2,48% para Mito RFP) (Figura 1D e E). A Tabela 3 fornece as velocidades axonais precisas das mitocôndrias livres ou aquelas fortemente ou frouxamente ligadas ao RE. Esses valores podem ser usados como um meio quantitativo notável para avaliar o grau de estabilização do MAM variando entre os MAMs apertados e soltos, levando à redução da geração de Ab. As taxas de transporte axonal mitocondrial após a estabilização dos MAMs apertados e soltos em células ReN virgens espelharam os padrões de transporte observados nos neurônios ReN GA (Figura 1F-G). Os resultados consistentes entre neurônios ReN ingênuos e neurônios ReN GA AD que expressam APPSwe/Lon sugerem que o efeito no transporte axonal é predominantemente atribuído ao estado de estabilização do MAM, independente da presença de APPSwe/Lon ou da produção de Aβ resultante.
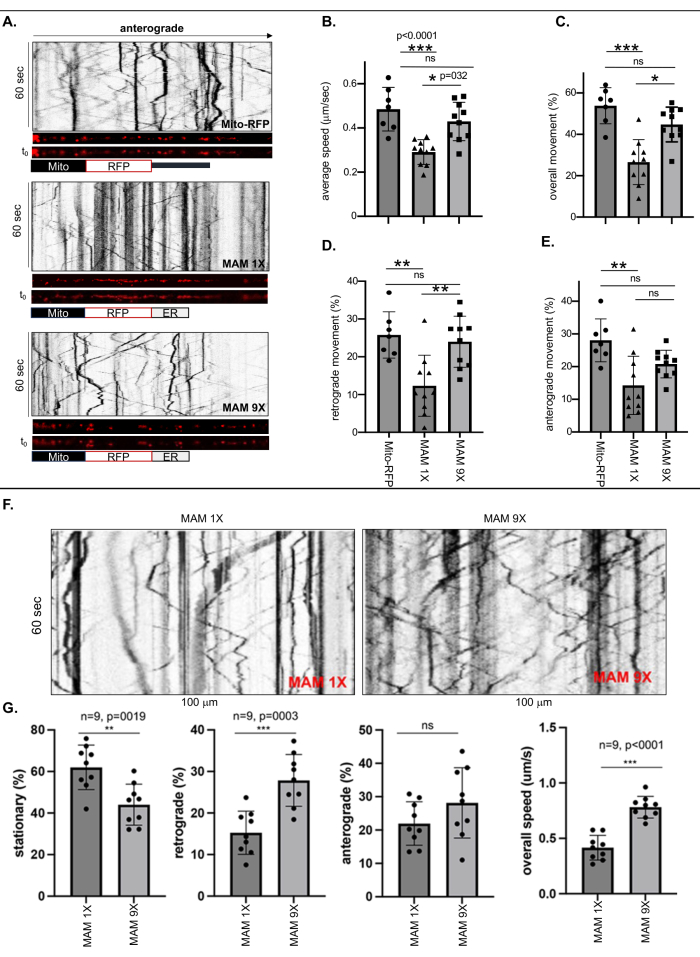
Figura 1: A estabilização de MAMs por MAM 1X reduziu a velocidade média e o movimento (geral, retrógrado e anterógrado) das mitocôndrias ligadas ao ER nos axônios de células ReN GA diferenciadas e ReN virgens. (A) Quimógrafos representativos dos pontos marcados com RFP representando mitocôndrias livres (Mito-RFP) ou mitocôndrias ligadas a ER estabilizadas por MAM 1X (MAMs apertados, largura de contato de 6 nm ± 1 nm) ou MAM 9X (MAMs soltos, largura de contato de 24 nm ± 3 nm) dentro dos axônios (~ 100 nm). (BE) Análise quantitativa da (B) velocidade média e movimento [(C) geral, (D) retrógrado e (E) anterógrado] de Mito-RFP, MAM 1X ou MAM 9X dentro de axônios de células Ren-GA diferenciadas de 10 dias. n>7; Foi realizada ANOVA de duas vias. *p < 0,05, **p < 0,001. Representante de três experimentos independentes. (F) Quimógrafos representativos do movimento de MAM 1X ou MAM 9X dentro de axônios de células ReN virgens diferenciadas de 10 dias. (G) Análise quantitativa do movimento percentual (%) (estacionário, retrógrado e anterógrado) e velocidade geral (micrômetro/segundo; μm/s) de MAMs estabilizados com MAM 1X ou MAM 9X dentro dos axônios de células ReN virgens. n = 9; Foi realizada ANOVA de duas vias. p < 0,0001. Essa figura foi adaptada com permissão de Zellmer et al.17. Clique aqui para ver uma versão maior desta figura.

Figura 2: Imagens de células vivas de axônios de células ReN virgens expressando MAM 1X ou MAM 9X. Imagens de vídeo representativas de células vivas exibindo os movimentos dos MAMs estabilizados por MAM 1X ou MAM 9X dentro de axônios de 100 μm de comprimento de células ReN diferenciadas que expressam GFP de 10 dias. n > 10 imagens de experimentos duplicados. As setas indicam transporte anterógrado. Barra de escala: 100 μm. Clique aqui para ver uma versão maior desta figura.
| Reagente | Concentração Final | Quantidade |
| DMEM com L-glutamina | N/A | 500 mL |
| Heparina | 2 μg/ml | 0,5 mL |
| B27 | 1x | 10 mL |
| bFGF | 20 ng/mL | 0,4 mL |
| FEG | 20 ng/mL | 0,5 mL |
| Penicilina/estreptomicina | 100 unidades/mL | 5 ml |
| Total | 516,4 mL | |
| Filtrar o meio antes de adicionar penicilina/estreptomicina. Conservar a 4 °C até 1 mês. | ||
Tabela 1: Composição dos meios de expansão.
| Reagente | Concentração Final | Quantidade |
| DMEM com L-glutamina | N/A | 500 mL |
| Heparina | 2 μg/ml | 0,5 mL |
| B27 | 1x | 10 mL |
| Penicilliion/estreptomicina | 100 unidades/mL | 5 ml |
| Total | 515,5 mL | |
| Filtrar o meio antes de adicionar penicilina/estreptomicina. Conservar a 4 °C até 1 mês | ||
Tabela 2: Composição dos meios de diferenciação.
| ReN GA | ReN (ingênuo) | ReN GA (3D) | |||||
| Geral (%) | Retrógrado (%) | Anterógrado (%) | Velocidade média (mm/s) | Ab40 (pM) | Ab42 (pM) | ||
| Mito-RFP | 53.82 ± 3.3% | 25.78 ± 2.31% | 28.04 ± 2.48% | 0,66 ± 0,03 | 0,69 ± 0,07 | 241,7 ± 26,74 | 13,77 ± 1,52 |
| MAM 1X | 26,6 ± 3,4% *** | 12,33 ± 2,5% *** | 14,27 ± 2,81% *** | 0,3 ± 0,02*** | 0,43 ± 0,04*** | 377,2 ± 76,87* | 26,62 ± 3,86* |
| MAM 9X | 44,79 ± 2,6% ns | 23,99 ± 2,17%ns | 20,80 ± 1,33%ns | 0,59 ± 0,02 ns | 0,62 ± 0,02 ns | 158,8 ± 3,27* | 17,01 ± 2,02* |
Tabela 3: Análise quantitativa. Imagem de células vivas e análise quantitativa baseada em quimografia da velocidade média (velocidade) e movimentos axonais (geral, retrógrado e anterógrado) de Mito-RFP, MAM 9X e MAM 1X. A ANOVA de duas vias foi realizada para velocidade ou movimento axonal (%). n = 9. Para Aβ, foi realizada ANOVA ordinária de uma via; n = 3, três experimentos independentes. A significância é medida em relação às células ReN GA não transfectadas (controle). **p < 0,0001; *p < 0,05; não significativo (ns). Esta tabela foi adaptada com permissão de Zellmer et al.17.
Arquivo de codificação suplementar 1: O código para gerar, rastrear e medir os dados do quimiograma. Clique aqui para baixar este arquivo.
Discussão
A inibição do receptor sigma-1 (S1R) regulou negativamente a estabilização do MAM nos processos neuronais e reduziu drasticamente (~ 90%) a geração de Aβ a partir de axônios, mas não de soma de um sistema de cultura tridimensional (3D) de células progenitoras neurais humanas (ReN) expressando mutações familiares de DA [FAD] no gene da proteína precursora de amilóide [APP] (ReN GA) 23 , 24 , 25 , 27. Os estabilizadores MAM constitutivos marcados com RFP (MAM 1X e MAM 9X) projetados para estabilizar MAMs apertados (6 nm ± 1 nm) e soltos (24 nm ± 3 nm)15,26 são ferramentas notáveis para medir quantitativamente a estabilização MAM. Ambos os estabilizadores não apenas exibem expressão igual e estável em células ReN GA diferenciadas em matriz 3D por ~ 10 dias, mas também detectaram MAMs em pontos discretos em soma e axônios. Mais importante ainda, enquanto a expressão estável de MAM 1X em 3D ReN GA enriquecido com FACS aumentou significativamente a geração de Aβ, a expressão de MAM 9X não teve efeito17. Também testamos o efeito de um estabilizador constitutivo de MAM contendo 18 ligantes de aminoácidos (MAM 18X) que detecta e estabiliza MAMs >25 nm. Ao contrário do MAM 1X ou MAM 9X, o MAM 18X é exclusivamente rotulado como MAMs somais. Neurônios ReN GA que expressam MAM 18X enriquecidos com FACS, reduziram a geração de Aβ17. Esses achados sugeriram a possibilidade de um limiar de estabilidade de MAM determinado por sua largura de lacuna que varia entre MAMs apertados patogênicos (aumento da geração de Aβ) e MAMs soltos não patogênicos (mantendo ou reduzindo a geração de Aβ). geração no cérebro.
Três abordagens diferentes foram empregadas para desenvolver moduladores de MAM: (1) moduladores que têm como alvo proteínas de ligação MAM, (2) moduladores que alteram os níveis de expressão de proteínas residentes em MAM e (3) moduladores de estruturas MAM18. Apesar dessas abordagens, o principal obstáculo para encontrar moduladores eficazes da estabilização do MAM é a falta de métodos para medir quantitativamente o grau de estabilização do MAM. Técnicas tradicionais como microscopia eletrônica (EM) ou microscopia de super-resolução têm limitações na captura de mudanças em tempo real ou no fornecimento de detalhes suficientes para avaliar a estabilização do MAM (revisado em28).
O método descrito aqui superará o obstáculo e fornecerá informações importantes sobre a relação entre a estabilização do MAM e a produção de Aβ. Os resultados mostram que MAMs com espessura de 6 nm ± 1 nm, exibindo um movimento geral de 26,6% ± 3,4% (Tabela 3), estão associados à geração de Aβ. Por outro lado, MAMs com espessura de 24 nm ± 3 nm, que exibem um movimento geral de 44,79% ± 2,6% (Tabela 3), não influenciam a geração de Aβ. O movimento geral das mitocôndrias (Mito-RFP) foi de 53,82% ± 3,3%. Dado que a espessura do MAM normalmente varia entre 6 nm e 80 nm, esses achados delineiam os limites superior e inferior da estabilização do MAM em relação à produção de Aβ. Consequentemente, este método pode orientar a identificação e otimização de um modulador (s) para estabilização de MAM. O objetivo seria alterar o movimento geral dos MAMs de 26,6% ± 3,4% para 53,82% ± 3,3%, ou sua velocidade média de  0,4 μm/s para 0,7 μm/s (Tabela 3), posicionando esse(s) modulador(es) como potenciais agentes terapêuticos contra a
0,4 μm/s para 0,7 μm/s (Tabela 3), posicionando esse(s) modulador(es) como potenciais agentes terapêuticos contra a  produção de Aβ.
produção de Aβ.
O uso de moduladores MAM constitutivos contendo ligantes sintéticos de comprimentos crescentes (0-18 aminoácidos) é um método poderoso para determinar quantitativamente o limiar de estabilização MAM para mudar a estabilização MAM para um que mantenha ou possivelmente diminua em oposição a aumentar a geração de Aβ. No entanto, para avaliar a eficiência ou eficácia dos moduladores de MAM, serão necessários estabilizadores de MAM induzíveis. Estão disponíveis estabilizadores MAM baseados em microscopia de ressonância de Förster/fluorescência (FRET/FLIM) induzíveis que são plasmídeos de expressão que codificam a sequência de mAKAP1 (34-63) e a fosfatase Sac1 (521-587) de mAKAP1 (521-587) fundida com ER fundida por CFP. Além disso, os estabilizadores constitutivos podem não representar os MAMs fisiológicos, enquanto os estabilizadores FRET/FLIM MAM, por outro lado, detectarão os MAMs fisiológicos. As sondas GFP divididas onde a GFP é dividida em dois fragmentos não fluorescentes ligados ao ER residente ou às proteínas mitocondriais ER-GFP (1-10) e Mito-GFP11 que geram complementação de fluorescência biomolecular (BiFC) após a formação de MAMs27, também podem ser usadas. Embora os fragmentos de GFP sejam propensos à montagem espontânea, o BiFC tem a leitura mais simples, o sinal mais claro e a análise menos associada ao ruído. Além disso, a interação entre a GFP dividida é altamente reversível28, portanto, suas vantagens superam as desvantagens e tornam o método BiFC adequado para identificar moduladores da estabilização do MAM.
Agradecimentos
Agradecemos ao Dr. György Hajnóczky, Professor da Thomas Jefferson University, Filadélfia, por generosamente nos fornecer plasmídeos de expressão que codificam RFP-Mito, MAM 1X, MAM 9X e MAM 18X. Um agradecimento especial ao Dr. Lai Ding, Cientista Sênior de Imagens, Brigham and Women's Hospital por nos ajudar a escrever o código para gerar, rastrear e medir os dados do quimiograma. Este estudo foi apoiado pelo Cure Alzheimer's Fund para RB e NIH grant 5R01NS045860-20 para RET.
Materiais
| Name | Company | Catalog Number | Comments |
| 6 Well Glass Bottom Plate | Cellvis | P06-1.5H-N | |
| B-27 Supplement (50X), serum free | Gibco/Thermo Fisher Scientific | 17504044 | |
| bFGF | R&D System | 233-FB | |
| BSA | Fisher Scientific | 501781532 | |
| Countess Cell Counting Chamber Slides | Invitrogen | C10283 | |
| DMEM/F12 with L-glutamine | Gibco/Thermo Fisher Scientific | 11320-033 | |
| EDTA | Life Technologies | 41116134 | |
| EGF | Sigma-Aldrich | 92090408 | |
| Falcon 6 Well Plates | VWR International | 41122107 | |
| GAPDH Polyclonal Antibody | Thermo Fisher Scientific | PA1-988 | |
| Gelatin | VWR International | 9000-70-8 | |
| Graphpad Prism N/A | Prism 9, version 9.5.0 | N/A | |
| Heparin | Sigma-Aldrich | H0200000 | |
| ImageJ Software | ImageJ 1.53a | N/A | |
| Matrigel Basement Membrane Matrix | Corning | 356234 | |
| mCherry Polyclonal Antibody | Invitrogen | PA5-34974 | |
| MS Excel | Microsoft Excel, version 2302 | N/A | |
| Multi-array electrochemiluminescence assay kit | Meso Scale Diagnostics (MSD) | K15200E-2 | V-PLEX Aβ Peptide Panel 1 (6E10) kit |
| NaCl | Fisher Scientific | 7647145 | |
| NuPAGE 4–12% Bis-Tris gel | Invitrogen | NP0321BOX | |
| Penicillin/Streptomycin/Amphotericin B | Lonza | 17-745E | |
| Photoshop | Adobe Photoshop CC 20.0.10 | N/A | |
| Rat Neuron Nucleofector Kit | Lonza | VPG-1003 | |
| StemPro Accutase | Gibco | A1110501 | |
| Tris-HCL, pH 7.6 | Boston BioProducts | 42000000 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween 20 | Fisher Scientific | 501657287 |
Referências
- Giacomello, M., Pellegrini, L. The coming of age of the mitochondria-ER contact: a matter of thickness. Cell Death Differ. 23 (9), 1417-1427 (2016).
- Degechisa, S. T., Dabi, Y. T., Gizaw, S. T. The mitochondrial associated endoplasmic reticulum membranes: A platform for the pathogenesis of inflammation-mediated metabolic diseases. Immun Inflamm Dis. 10 (7), e647 (2022).
- Schon, E. A., Area-Gomez, E. Mitochondria-associated ER membranes in Alzheimer disease. Mol Cell Neurosci. 55, 26-36 (2013).
- Erpapazoglou, Z., Mouton-Liger, F., Corti, O. From dysfunctional endoplasmic reticulum-mitochondria coupling to neurodegeneration. Neurochem Int. 109, 171-183 (2017).
- Sala-Vila, A., et al. Interplay between hepatic mitochondria-associated membranes, lipid metabolism and caveolin-1 in mice. Sci Rep. 6, 27351 (2016).
- Fujimoto, M., Hayashi, T. New insights into the role of mitochondria-associated endoplasmic reticulum membrane. Int Rev Cell Mol Biol. 292, 73-117 (2011).
- Hung, V., et al. Proteomic mapping of cytosol-facing outer mitochondrial and ER membranes in living human cells by proximity biotinylation. eLife. 6, e24463 (2017).
- Area-Gomez, E., Schon, E. A. On the pathogenesis of Alzheimer's disease: The MAM hypothesis. FASEB J. 31 (3), 864-867 (2017).
- Rizzuto, R., et al. Close contacts with the endoplasmic reticulum as determinants of mitochondrial Ca2+ responses. Science. 280 (5370), 1763-1766 (1998).
- Sukhorukov, V. S., et al. Molecular mechanisms of interactions between mitochondria and the endoplasmic reticulum: A new look at how important cell functions are supported. Mol Biol. 56 (1), 59-71 (2022).
- Zhang, P., Konja, D., Zhang, Y., Wang, Y. Communications between Mitochondria and endoplasmic reticulum in the regulation of metabolic homeostasis. Cells. 10 (9), 2195 (2021).
- Ziegler, D. V., Martin, N., Bernard, D. Cellular senescence links mitochondria-ER contacts and aging. Commun Biol. 4 (1), 1323 (2021).
- Csordas, G., et al. Structural and functional features and significance of the physical linkage between ER and mitochondria. J Cell Biol. 174 (7), 915-921 (2006).
- Cieri, D., et al. SPLICS: a split green fluorescent protein-based contact site sensor for narrow and wide heterotypic organelle juxtaposition. Cell Death Differ. 25 (6), 1131-1145 (2018).
- Carpio, M. A., et al. BOK controls apoptosis by Ca(2+) transfer through ER-mitochondrial contact sites. Cell Rep. 34 (10), 108827 (2021).
- Prudent, J., et al. MAPL SUMOylation of Drp1 stabilizes an ER/mitochondrial platform required for cell death. Mol Cell. 59 (6), 941-955 (2015).
- Zellmer, J. C., Tarantino, M. B., et al. Stabilization of mitochondria-associated endoplasmic reticulum membranes regulates Abeta generation in a three-dimensional neural model of Alzheimer’s disease. Alzheimer’s Dement. , 1-20 (2024).
- Magalhaes Rebelo, A. P., et al. Chemical modulation of mitochondria-endoplasmic reticulum contact sites. Cells. 9 (7), 1637 (2020).
- Berger, S. I., Iyengar, R. Role of systems pharmacology in understanding drug adverse events. Wiley Interdiscip Rev Syst Biol Med. 3 (2), 129-135 (2011).
- Friedman, J. R., Webster, B. M., Mastronarde, D. N., Verhey, K. J., Voeltz, G. K. ER sliding dynamics and ER-mitochondrial contacts occur on acetylated microtubules. J Cell Biol. 190 (3), 363-375 (2010).
- Wu, Y., et al. Contacts between the endoplasmic reticulum and other membranes in neurons. Proc Natl Acad Sci U S A. 114 (24), E4859-E4867 (2017).
- Cagin, U., et al. Mitochondrial retrograde signaling regulates neuronal function. Proc Natl Acad Sci U S A. 112 (44), E6000-E6009 (2015).
- Choi, S. H., et al. A three-dimensional human neural cell culture model of Alzheimer's disease. Nature. 515 (7526), 274-278 (2014).
- Kim, Y. H., et al. A 3D human neural cell culture system for modeling Alzheimer's disease. Nat Protoc. 10 (7), 985-1006 (2015).
- Kwak, S. S., et al. Amyloid-beta42/40 ratio drives tau pathology in 3D human neural cell culture models of Alzheimer's disease. Nat Commun. 11 (1), 1377 (2020).
- Csordas, G., et al. Imaging interorganelle contacts and local calcium dynamics at the ER-mitochondrial interface. Mol Cell. 39 (1), 121-132 (2010).
- Bhattacharyya, R., et al. Axonal generation of amyloid-beta from palmitoylated APP in mitochondria-associated endoplasmic reticulum membranes. Cell Rep. 35 (7), 109134 (2021).
- Tebo, A. G., Gautier, A. A split fluorescent reporter with rapid and reversible complementation. Nat Commun. 10 (1), 2822 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados