Method Article
Analyse quantitative de la stabilisation de la membrane endoplasmique du réticulum (MAM) associée aux mitochondries dans un modèle neuronal de la maladie d’Alzheimer (MA)
Dans cet article
Résumé
Ici, nous décrivons la mesure du taux de transport axonal des stabilisateurs constitutifs des membranes du réticulum endoplasmique (RE) associées aux mitochondries (MAM) en augmentant ou en maintenant la génération de β-amyloïde (Aβ) neurotoxiques à partir des neurones de la maladie d’Alzheimer (MA) en temps réel pour servir de mesure directe et quantitative pour mesurer la stabilisation de la MAM et aider au développement de traitements contre la MA.
Résumé
Une méthode de quantification de la stabilisation des membranes du réticulum endoplasmique (MAM) associées aux mitochondries dans un modèle neuronal tridimensionnel (3D) de la maladie d’Alzheimer (MA) est présentée ici. Pour commencer, des cellules ReN neuroprogénitrices humaines fraîches exprimant la protéine précurseur β-amyloïde (APP) contenant la maladie d’Alzheimer familiale (FAD) ou des cellules ReN naïves sont cultivées dans de fines plaques de culture tissulaire (1:100) recouvertes de Matrigel. Une fois que les cellules ont atteint la confluence, celles-ci sont électroporées avec des plasmides d’expression codant pour la séquence de liaison aux mitochondries conjuguée à la protéine de fluorescence rouge (RFP) de AKAP1(34-63) (Mito-RFP) qui détecte les mitochondries ou les stabilisateurs MAM constitutifs MAM 1X ou MAM 9X qui stabilisent respectivement les MAM serrés (6 nm ± 1 nm de largeur d’espace) ou lâches (24 nm ± 3 nm de largeur d’espace). Après 16 à 24 h, les cellules sont récoltées et enrichies par un trieur de cellules activé par fluorescence (FACS). Un nombre égal de cellules enrichies en FACS sont ensemencées dans la matrice tridimensionnelle (Matrigel 1:1) et laissées se différencier en neurones matures pendant 10 jours. Des images de cellules vivantes de cellules différenciées de 10 jours exprimant les stabilisateurs MAM conjugués à la RFP sont capturées sous un microscope fluorescent équipé d’une chambre de culture d’imagerie de cellules vivantes maintenant le CO2 (5 %), la température (37 °C) et l’humidité (~90 %). À cette fin, nous avons effectué des analyses d’imagerie et de kymographie de cellules vivantes pour mesurer la motilité des mitochondries libres marquées avec Mito-RFP ou des mitochondries liées à ER de largeurs d’espace serrées ou lâches stabilisées par MAM 1X ou MAM 9X, respectivement, dans le processus neuronal le plus étendu de chaque neurone ReN GA d’au moins 500 nm de long, en les considérant comme des axones.
Introduction
De nouvelles preuves suggèrent que les contacts du réticulum endoplasmique (MERCs) spécialisés associés aux mitochondries, récoltés biochimiquement sous forme de membranes du RE associées aux mitochondries, souvent appelées MAMs 1,2, jouent un rôle dans plusieurs maladies neurodégénératives, y compris la MA 3,4. Ces MAM sont composés de microdomaines riches en cholestérol dans le RE et la membrane externe des mitochondries attachés par une série de protéines qui créent des diversités structurelles et fonctionnelles parmi les MAM 5,6,7. L’hypothèse récemment inventée de la MAM postule que l’augmentation des MAM conduit à une production accrue d’Aβ et à la cascade pathogène de la MA, y compris la formation d’un enchevêtrement neurofibrillaire (NFT), la dyshoméostasie calcique et la neuroinflammation 3,8. Environ 5 à 20 % des mitochondries entrent en contact physique avec le RE pourformer des MAM9. La largeur de l’espace des MAM est déterminée par le RE lisse et rugueux (sER et rER, respectivement). La largeur variable de l’écart entre les mitochondries sER-mitochondries (10-50 nm) et les mitochondries rER-r (50-80 nm) suggère que la largeur de l’écart des MAM a un long spectre qui varie de serré (~10 nm) à lâche (~80 nm)10,11,12,13. La largeur de l’espace MAM détermine les fonctions MAM, telles que l’homéostasie calcique et le transport des lipides 1,14. Un rapport récent a montré que les MAM formées entre le RE étroitement connecté (~10 nm) et les mitochondries, appelées MAM complètes, sont apoptotiques. En revanche, les MAM formées entre le RE faiblement connecté (~25 nm) et les mitochondries, appelées MAM défectueuses ou moyennes, sont anti-apoptotiques 14,15,16. La stabilisation des MAM avec une largeur d’écart de 6 nm ± une augmentation de la génération d’Aβ à partir d’un nouveau modèle de culture neuronale tridimensionnelle (3D) de la MA. En revanche, la stabilisation des MAM avec une largeur d’écart de 24 nm ± 3 nm n’a aucun effet sur la génération Aβ17. Cette constatation suggère pour la première fois que la régulation du degré de stabilisation des MAM, mais non la déstabilisation des MAM, est la clé de la régulation de la génération Aβ. Une tentative de déstabilisation complète des MAM peut avoir des conséquences indésirables, car les MAM maintiennent plusieurs événements cellulaires essentiels à la survie cellulaire12.
La modulation des MAM est un domaine de recherche émergent avec des implications potentielles pour divers troubles, notamment le cancer, les troubles métaboliques et les maladies neurodégénératives18. Malgré la disponibilité de nombreux modulateurs MAM, aucune tentative majeure n’a été entreprise jusqu’à présent pour tester leurs capacités à déstabiliser les MAM et à réduire la pathologie de la MA, principalement parce que la diversité structurelle des MAM en fait un système très complexe à cibler pour la découverte de médicaments. Mais la pharmacologie des systèmes structurels nouvellement développée, qui prend en compte les propriétés spécifiques des cibles médicamenteuses et de leur environnement18,19, devrait surmonter les difficultés et développer des médicaments très puissants ciblant les MAM ou les protéines associées à la MAM dans la MA. Cependant, la recherche d’un modulateur efficace de la stabilisation MAM nécessite des méthodes permettant de quantifier précisément le degré de stabilisation MAM. Les techniques traditionnelles telles que la microscopie électronique (EM) ou la microscopie à super-résolution ont des limites dans la détermination de la stabilisation MAM. Pour surmonter ces défis, il faudrait probablement mettre au point de nouvelles techniques d’imagerie plus dynamiques ou d’essais biochimiques capables de fournir des mesures quantitatives de la stabilisation de la MAM dans les cellules vivantes. La microscopie électronique à balayage par faisceau d’ions focalisés (FIB-SEM) des neurones primaires a révélé que le RE a tendance à former un réseau autour des mitochondries susceptible de limiter la motilité mitochondriale20,21. La perturbation des systèmes de transport mitochondriaux, rétrogrades, antérogrades ou les deux, a eu un impact profond sur la fonction synaptique et neuronale22. Ainsi, la nouvelle imagerie de cellules vivantes et l’analyse basée sur la kymographie de la vitesse axonale des mitochondries liées au RE décrites ici comme une mesure pour mesurer quantitativement la stabilisation de la MAM faciliteront l’identification du ou des modulateurs de la MAM qui peuvent faire passer le seuil de stabilisation de la MAM à un seuil qui maintient ou éventuellement abaisse au lieu d’augmenter la génération d’Aβ.
Protocole
Modèles de culture neuronale de la maladie d’Alzheimer : Cette étude a utilisé des neurones dérivés de cellules ReN neurales humaines [naïve ReN (Millipore)] ou de cellules ReN exprimant des mutations familiales de la MA (fAD) dans le gène de la protéine précurseur de l’amyloïde (APPSwe/Lon), cellules ReN GA. Le système de culture tridimensionnel (3D) ReN-GA récapitule la pathologie de la MA, à savoir les enchevêtrements neurofibrillaires (NFT) induits par l’oligomère Aβ 23,24. Des cellules ReN naïves sont disponibles dans le commerce. Les lignées ReN GA ont été obtenues auprès du Dr Doo Y. Kim, professeur agrégé au Massachusetts General Hospital (MGH)23,24,25.
Plasmides d’expression : AKP1 (34-63) et la séquence ciblant le RE des protéines Ubc 6 (283-303) sont liées directement à la RFP (Mito-RFP-ER désignée MAM 1X) ou contiennent un linker à 9 acides aminés (Mito-9X-RFP-ER noté MAM 9X) conçu pour stabiliser les MAM de 6 nm ± 1 nm ou 24 nm + 3 nm de largeurs d’espace, respectivement15,26 (Figure 1A).
1. Électroporation
- Transfection des cellules avec des stabilisateurs MAM (1-2 h)
REMARQUE : Suivez le protocole décrit par le fabricant du kit nucleofector (Table des matériaux). Avant de commencer, préparez des boîtes de culture à fond de verre à 6 puits pré-enrobées de DMEM/F12 contenant 1 % de Matrigel (ci-après appelé matrice à membrane basale [BMM]). Utilisez 2 ml de BMM pour bien enrober chaque puits. Incuber pendant au moins 1 h à 37 °C. Le BMM, le support, les pointes de pipette et les pipettes doivent tous être pré-refroidis avant d’être mélangés pour éviter que le BMM ne se solidifie.- Aspirez le mélange BMM. Remplacer par 2 mL de DMEM/F12 à température ambiante (RT) dans chaque puits.
- Combiner Nucleofector avec le supplément 1 dans un rapport de 4,5:1 (82 μL :18 μL de rapport Nucleofector/Supplement pour 100 μL de solution). Pour chaque transfection souhaitée, 100 μL de Nucleofector sont nécessaires.
- L’ADN du vortex. Ensuite, ajoutez 1 à 5 μg d’ADN (par traitement) dans des tubes de microcentrifugation de 1 mL pour chaque transfection.
- Utilisez l’acustase pour récolter une plaque de cellules ReN-GA saines.
REMARQUE : Placer les cellules sur plaque dans un milieu d’expansion (Tableau 1) dans des boîtes de culture tissulaire de 100 mm et attendre que la confluence atteigne 70-80 %.- Aspirez le support existant et lavez-le une fois avec 10 mL de PBS.
- Ajouter 1 mL d’acutase directement dans les cellules et incuber pendant 5 à 10 min à 37 °C.
- Délogez les cellules de la parabole en tapotant doucement le côté de la parabole. Vérifiez au microscope pour vous assurer que les cellules sont desserrées et qu’elles s’écoulent librement.
- Neutralisez l’acutase avec 10 ml de DMEM/F12 et transférez-la dans un tube propre de 15 ml.
- Centrifuger le nombre requis de cellules à 274 x g pendant 5 min et aspirer le surnageant pour éliminer les cellules mortes. Ensuite, remettre la pastille en suspension dans 10 mL de DMEM/F12.
- Comptez les cellules pour déterminer la densité.
REMARQUE : Dans cette étude, un compteur de cellules automatisé a été utilisé pour compter le nombre de cellules. Par électroporation, 3-5 X 106 cellules sont nécessaires (par exemple, 5 traitements nécessiteraient 15-25 X 106 cellules).- Prélever 10 μL des cellules en suspension et les ajouter au côté A d’une chambre de comptage de cellules. Ajoutez ensuite 10 μL sur la face B.
REMARQUE : Ajoutez la suspension cellulaire en inclinant la pipette sur le côté. Évitez les bulles en ne poussant pas au-delà de la première butée sur le piston de la pipette. - Apportez la chambre de comptage remplie au compteur de cellules et insérez le côté A dans la fente principale à l’avant.
- Après avoir appuyé sur Mesurer, notez le nombre de cellules par ml.
- Sortez la chambre, retournez-la du côté B et répétez les étapes 1.1.5.2-1.1.5.3.
- Additionnez ces deux nombres, puis divisez par 4 (si vous n’utilisez pas de Trypan Blue), puis multipliez le nombre par les millilitres de liquide dans lesquels les cellules sont en suspension pour obtenir le nombre total de cellules dans la suspension.
- Prélever 10 μL des cellules en suspension et les ajouter au côté A d’une chambre de comptage de cellules. Ajoutez ensuite 10 μL sur la face B.
- Centrifuger le nombre requis de cellules à 274 x g pendant 5 min et aspirer le surnageant.
- Mettre les pastilles en suspension dans 100 μL de mélange Nucleofector par électroporation. (par exemple, 500 μL pour 5 traitements).
REMARQUE : Laisser les cellules dans la solution de Nucleofector pendant plus de 15 minutes pourrait réduire la viabilité cellulaire et l’efficacité globale. - Ajouter 100 μL de suspension cellulaire dans l’un des tubes contenant de l’ADN et mélanger par pipetage.
- Transférez le mélange d’ADN dans une cuvette d’électroporation et fermez-la avec le couvercle fourni. Pour éviter de créer des bulles, inclinez la cuvette vers le bas et pipetez lentement.
- Sélectionnez le programme Nucleofector approprié pour l’appareil utilisé. Pour l’appareil utilisé dans cette étude, utilisez le programme A-033 pour la transfection. Pour optimiser, essayez les 5 programmes Nucleofector afin de déterminer celui qui convient le mieux à chaque type de cellule.
REMARQUE : Une confirmation d’une électroporation réussie est une mousse visible au sommet du mélange. - Immédiatement, à l’aide des pipettes stériles fournies, ajoutez ~500 μL de DMEM de la plaque préremplie à 6 puits dans la cuvette. Mélangez doucement une fois, puis transférez les cellules électroporées et le milieu dans le puits correspondant.
REMARQUE : Après l’électroporation, les cellules sont extrêmement sensibles, il est donc essentiel de transférer rapidement le milieu et de le pipeter avec soin. - Répétez les étapes 1.1.8 à 1.1.11 pour tous les traitements d’ADN restants.
- Incuber les cellules à 37 °C en présence de CO2 (5 %) pendant la nuit.
- Le lendemain, échangez le milieu avec un milieu de différenciation frais (tableau 2) et laissez les cellules se différencier pendant 10 jours. Tous les 2-3 jours, remplacez-le par un support frais.
2. Imagerie de cellules vivantes
- Préparation des cellules pour la microscopie sur cellules vivantes (30 min)
- Préparation de la chambre de cellules vivantes (avant de déplacer les cellules dans la chambre)
- Assurez-vous que le réservoir de CO2 et l’humidificateur sont fixés à la chambre, que les vannes sont ouvertes et que les réservoirs sont remplis.
- Réglez la température à 37 °C, le COà 2 à 5 % et l’humidité à 95 %. (Cela peut prendre un certain temps pour que les niveaux s’équilibrent).
- Placez une plaque à 6 puits contenant des cellules dans la chambre et ajustez la mise au point du microscope jusqu’à ce que les cellules deviennent visibles.
- Allumez le laser (Table des matériaux).
- Pour capturer le signal RFP, excitez le fluorophore à l’aide d’un laser de 594 nm et utilisez une émission de 570-640 nm. Pour la GFP, utilisez un laser de 488 nm pour l’excitation et l’émission de 510-540 nm.
- À l’aide du filtre fluorescent intégré, ajustez l’intensité du signal jusqu’à ce que le signal d’arrière-plan se soit dissipé (doit être presque noir fixe).
- Capture vidéo en direct des axones (10 h-15 h au total)
REMARQUE : Un microscope confocal inversé Nikon C2 Eclipse Ti2 a été utilisé pour capturer des images fluorescentes à l’aide du logiciel NIS Element AR. Utilisez un grossissement 60x à une résolution de 512 pixels, en prenant des vidéos à 1 image par seconde pendant 3 minutes, vous obtiendrez des kymographes plus propres.- Trouvez une cellule exprimant des biocapteurs RFP. Exportez une image de la cellule pour référence ultérieure avant de prendre une vidéo.
- Recadrez la zone de balayage pour l’adapter autour de l’axone. L’utilisation d’une zone de balayage plus petite réduit le temps de traitement du microscope et facilite grandement la génération de la kymographie.
- Éteignez tous les lasers pour faciliter le fonctionnement du logiciel. Veuillez noter que lorsque le programme est exécuté avec tous les lasers actifs, toutes les images ne sont pas capturées.
REMARQUE : Le signal fluorescent rouge du soma est beaucoup plus brillant que dans l’axone. Par conséquent, le soma est exclu pour augmenter l’intensité globale du signal dans l’axone. Passez à l’onglet Mesure du temps . - Réglez l’intervalle sur 1 image par seconde et réglez le temps total sur 181 s.
- Cliquez sur Exécuter.
- Enregistrez ce fichier en tant que fichier .nds (correctement étiqueté) et répétez le processus ~10 fois par stabilisateurs MAM (MAM 1X ou MAM 9X).
REMARQUE : La puissance du laser produisait parfois un effet de blanchiment notable sur le signal RFP s’il était balayé trop longtemps. Il est important de travailler rapidement pour capturer des vidéos.
3. Post-traitement (7 jours)
REMARQUE : Pour analyser le transport et générer des kymographes, des macros Fiji ImageJ ont été utilisées. Les vésicules qui se déplaçaient de moins de 0,1 mm/s ont été classées comme stationnaires. La fréquence du mouvement des particules a été calculée en divisant le nombre de particules se déplaçant dans une direction donnée (antérograde, rétrograde) ou ne bougeant pas (stationnaire) par le nombre total de particules analysées dans le kymographe. Le temps passé par chaque vésicule à faire une pause ou à se déplacer a été calculé en faisant la moyenne du pourcentage de temps passé dans chaque condition pour toutes les vésicules de chaque neurone analysé. La distribution de fréquence pour la vitesse et la durée de course a été calculée en utilisant uniquement des vésicules mobiles pour chaque condition expérimentale. L’analyse a été réalisée sur des faisceaux axonaux de 100 mm pendant 3 min.
- Génération d’un kymographe
- Ouvrez le fichier .nds dans Fiji ImageJ.
- Allez dans l’onglet Image et cliquez sur Propriétés. Enregistrez le rapport pixel/micron. Ceci est nécessaire pour le calcul ultérieur.
- Cliquez sur Fichier > Enregistrer sous > Tiff pour enregistrer le fichier en tant que .tiff dans son dossier (la macro enregistrera automatiquement tout ce qui a été généré dans ce dossier).
- Faites glisser la macro Kymo dans ImageJ. Le code est fourni dans le fichier de codage supplémentaire 1. Cliquez sur Exécuter.
- N’appuyez pas sur OK. Passez à la fenêtre MAX_raw. Faites un clic gauche manuel le long de l’axone et double-cliquez pour terminer le suivi.
REMARQUE : Assurez-vous de suivre l’axone à partir du soma donc de la terminaison axonale. De cette façon, les calculs antérogrades et rétrogrades seront corrects. - Appuyez sur la commande T ou Ajouter dans le gestionnaire de retour sur investissement.
- Cliquez maintenant sur OK. Un kymographe sera généré dans le dossier créé précédemment. (L’axe X correspond à la longueur de l’axone en microns et l’axe Y est le temps en secondes).
- Suivi du kymographe
- Faites glisser le kymographe dans Fiji Image J (il devrait être automatiquement placé dans le dossier créé précédemment).
- Faites glisser la macro Piste dans l’image J.
- À l’invite suivante, cliquez sur OK. Si vous reprenez le travail à partir d’avant, entrez le point numérique à partir duquel continuer, puis appuyez sur OK.
- À l’aide de l’outil Rectangle du panneau Sélectionner , sélectionnez une zone près du milieu du kymographe qui a une hauteur de 60 pixels et toujours 100 μm de diamètre. Pour ce faire, divisez 100 par le rapport pixel/micro enregistré à l’étape 3.1.2.
- Après avoir sélectionné une zone, appuyez sur Ctrl T pour l’ajouter au retour d’intérêt et enregistrez le retour d’intérêt dans le dossier en cours de travail (appuyez sur Plus dans le menu ROI, puis sur Enregistrer).
REMARQUE : Inverser la zone sélectionnée en appuyant sur Ctrl + Maj + I facilitera le suivi. - Pour suivre le mouvement d’une vésicule individuelle, maintenez la touche Ctrl enfoncée tout en cliquant avec le bouton gauche de haut en bas.
- Pour ce faire, maintenez la touche Ctrl enfoncée tout en faisant un clic droit et une fenêtre apparaîtra indiquant où les axones sont suivis. Si cela semble bon, appuyez sur OK.
- Pour continuer et sélectionner une autre vésicule, cliquez sur Oui et répétez le processus à partir des étapes 3.2.6 à 3.2.8. Une fois que chaque vésicule visible a été suivie, appuyez sur Non. Cela enregistrera automatiquement la superposition suivie à la main dans le même dossier.
- Mesure des données du kymographe
- Créez un nouveau dossier. Faites glisser chaque fichier texte généré dans le dossier.
- Ouvrez le fichier kymographe et enregistrez les dimensions.
- Faites glisser la macro Mesure dans l’image J.
- Entrez le rapport micron/pixel enregistré précédemment dans PixelScale.
- Réglez la limite de vitesse basse sur 0,1. (Les vésicules se déplaçant en dessous de cette limite sont considérées comme stationnaires).
- Entrez les dimensions du kymographe précédemment enregistrées. Le fichier récapitulatif sera automatiquement généré dans le dossier de texte créé. Chaque remplissage récapitulatif contiendra le % de temps parcouru, la vitesse globale (μm/s), la distance totale, le segment moyen parcouru, le nombre de fois où il s’est arrêté et le nombre de fois où il a été inversé. Le code pour la génération, le suivi et la mesure des données du kymographe est fourni dans le fichier de codage supplémentaire 1.
Résultats
Des analyses d’imagerie et de kymographie de cellules vivantes ont été réalisées pour mesurer la motilité des mitochondries libres marquées avec Mito-RFP ou des mitochondries liées à RE de largeurs de contact serrées (6 nm ± 1 nm) ou lâches (24 nm ± 3 nm) stabilisées par MAM 1X ou MAM 9X, respectivement, dans le processus neuronal le plus long de chaque neurone ReN GA (AD) ou ReN (naïf) d’au moins 500 nm de long, en le considérant comme un axone (Figure 1 et Figure 2). Les fréquences des mouvements (globaux, rétrogrades et antérogrades) ont été calculées en divisant le nombre de points marqués RFP (MAM) en mouvement ou stationnaires par le nombre total dans les kymographes (figure 1A-E). La vitesse axonale globale des mitochondries liées au RE marquées MAM 1X a été considérablement diminuée de ~50 % par rapport aux mitochondries sans RE marquées au MITO-RFP ou aux mitochondries liées au RE marquées au MAM 9X (Figure 1B). L’analyse quantitative a également révélé des différences spectaculaires entre les mouvements globaux et rétrogrades des mitochondries liées au RE stabilisées MAM 1X par rapport aux mitochondries libres (Mito-RFP) ou stabilisées au RE MAM 9X. Alors que 53,82 % ± 3,3 % des mitochondries sans RE (Mito-RFP) étaient mobiles, seulement 26,6 % ± 3,4 % des mitochondries liées au RE marquées MAM 1X étaient mobiles, ce qui suggère que la stabilisation des MAMs réduisait significativement la mobilité axonale globale des mitochondries, étroitement associées au RE, par rapport aux mitochondries non liées ou faiblement liées au RE (44,79 % ± 2,6 % du MAM 9X contre 53,82 % ± 3,3 % du Mito RFP, respectivement) (Figure 1C). De manière constante, les mouvements rétrogrades et antérogrades des mitochondries liées au RE marquées MAM 1X étaient significativement plus faibles par rapport aux mitochondries marquées MAM 9X ou libres (Mito-RFP) (rétrogrades : 12,33 % ± 2,55 % pour MAM 1X contre 25,78 % ± 2,31 % pour Mito RFP ; antérogrades : 14,27 % ± 2,81 % pour MAM 1X contre 28,04 % ± 2,48 % pour Mito RFP) (figures 1D et E). Le tableau 3 fournit les vitesses axonales précises des mitochondries libres ou de celles étroitement ou faiblement liées au RE. Ces valeurs peuvent être utilisées comme un moyen quantitatif remarquable pour évaluer le degré de stabilisation de la MAM entre les MAM serrées et lâches, conduisant à la réduction de la génération d’Ab. Les taux de transport axonal mitochondrial lors de la stabilisation des MAM serrés et lâches dans les cellules ReN naïves reflétaient les modèles de transport observés dans les neurones ReN GA (Figure 1F-G). Les résultats cohérents entre les neurones ReN naïfs et les neurones ReN GA AD exprimant APPSwe/Lon suggèrent que l’effet sur le transport axonal est principalement attribué à l’état de stabilisation MAM, indépendamment de la présence d’APPSwe/Lon ou de la production Aβ résultante.
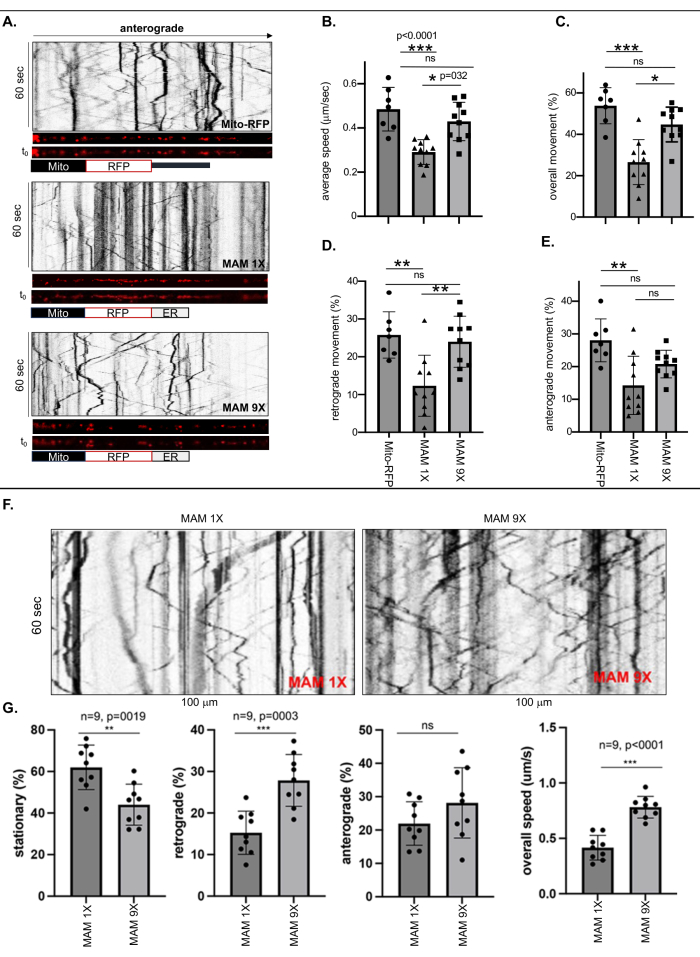
Figure 1 : La stabilisation des MAM par MAM 1X a réduit la vitesse et le mouvement moyens (global, rétrograde et antérograde) des mitochondries liées au RE dans les axones des cellules ReN GA différenciées et des cellules ReN naïves. (A) Kymographes représentatifs des puncta marqués RFP représentant des mitochondries libres (Mito-RFP) ou des mitochondries liées au RE stabilisées par MAM 1X (MAM serrées, largeur de contact de 6 nm ± 1 nm) ou MAM 9X (MAM libres, largeur de contact de 24 nm ± 3 nm) à l’intérieur des axones (~100 nm). (B-E) Analyse quantitative de la vitesse et du mouvement moyens (B) [(C) globalement, (D) rétrograde et (E) antérograde] de Mito-RFP, MAM 1X ou MAM 9X à l’intérieur des axones des cellules Ren-GA différenciées de 10 jours. n>7 ; Une ANOVA bidirectionnelle a été réalisée. *p < 0,05, **p < 0,001. Représentatif de trois expériences indépendantes. (F) Kymographes représentatifs du mouvement de MAM 1X ou MAM 9X à l’intérieur des axones des cellules ReN naïves différenciées de 10 jours. (G) Analyse quantitative du pourcentage ( %) de mouvement (stationnaire, rétrograde et antérograde) et de la vitesse globale (micromètre/seconde ; μm/s) des MAM stabilisées MAM 1X ou MAM 9X à l’intérieur des axones des cellules ReN naïves. n = 9 ; Une ANOVA bidirectionnelle a été réalisée. p < 0,0001. Cette figure a été adaptée avec la permission de Zellmer et al.17. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Images cellulaires en direct d’axones de cellules ReN naïves exprimant MAM 1X ou MAM 9X. Images vidéo représentatives de cellules vivantes montrant les mouvements des MAMs stabilisés par MAM 1X ou MAM 9X à l’intérieur d’axones de 100 μm de long de cellules ReN différenciées exprimant la GFP pendant 10 jours. n > 10 images issues d’expériences dupliquées. Les flèches indiquent le transport antérograde. Barre d’échelle : 100 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Réactif | Concentration finale | Quantité |
| DMEM avec L-glutamine | N/A | 500 ml |
| Héparine | 2 μg/mL | 0,5 mL |
| B27 | 1x | 10 ml |
| bFGF | 20 ng/mL | 0,4 mL |
| FEM | 20 ng/mL | 0,5 mL |
| Pénicilline/streptomycine | 100 unités/ml | 5 mL |
| Total | 516,4 ml | |
| Filtrer le média avant d’ajouter la pénicilline/streptomycine. Conserver à 4 °C jusqu’à 1 mois. | ||
Tableau 1 : Composition des supports de dilatation.
| Réactif | Concentration finale | Quantité |
| DMEM avec L-glutamine | N/A | 500 ml |
| Héparine | 2 μg/mL | 0,5 mL |
| B27 | 1x | 10 ml |
| Pénicilliion/Streptomycine | 100 unités/ml | 5 mL |
| Total | 515,5 ml | |
| Filtrer le média avant d’ajouter la pénicilline/streptomycine. Conserver à 4 °C jusqu’à 1 mois | ||
Tableau 2 : Composition des milieux de différenciation.
| ReN GA | ReN (naïf) | ReN GA (3D) | |||||
| Globalement ( %) | Rétrograde ( %) | Antérogrades ( %) | Vitesse moyenne (mm/s) | Ab40 (pM) | Ab42 (pM) | ||
| Mito-RFP | 53,82 ± 3,3 % | 25,78 ± 2,31 % | 28,04 ± 2,48 % | 0,66 ± 0,03 | 0,69 ± 0,07 | 241,7 ± 26,74 | 13,77 ± 1,52 |
| MAM 1X | 26,6 ± 3,4 % *** | 12,33 ± 2,5 % *** | 14,27 ± 2,81 % *** | 0,3 ± 0,02*** | 0,43 ± 0,04*** | 377,2 ± 76,87* | 26,62 ± 3,86* |
| MAM 9X | 44,79 ± 2,6 % N.-É. | 23,99 ± 2,17 % ns | 20,80 ± 1,33 % ns | 0,59 ± 0,02 ns | 0,62 ± 0,02 ns | 158,8 ± 3,27* | 17.01 ± 2.02* |
Tableau 3 : Analyse quantitative. Analyse quantitative basée sur l’imagerie de cellules vivantes et la kymographie de la vitesse moyenne (vitesse) et des mouvements axonaux (globaux, rétrogrades et antérogrades) de Mito-RFP, MAM 9X et MAM 1X. Une ANOVA bidirectionnelle a été réalisée pour la vitesse axonale ou le mouvement ( %). n = 9. Pour Aβ, une ANOVA unifacteur ordinaire a été réalisée ; n = 3, trois expériences indépendantes. La signification est mesurée par rapport aux cellules ReN GA non transfectées (contrôles). **P < 0,0001 ; *p < 0,05 ; Non significatif (NS). Ce tableau a été adapté avec la permission de Zellmer et al.17.
Fichier de codage supplémentaire 1 : Code de génération, de suivi et de mesure des données du kymographe. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
L’inhibition du récepteur sigma-1 (S1R) a régulé à la baisse la stabilisation de la MAM dans les processus neuronaux et a considérablement réduit (~90 %) la génération d’Aβ à partir des axones mais pas du soma d’un système de culture tridimensionnel (3D) de cellules progénitrices neurales humaines (ReN) exprimant des mutations familiales de la MA [FAD] dans le gène de la protéine précurseur de l’amyloïde [APP] (ReN GA)23,24,25,27. Les stabilisateurs MAM constitutifs marqués RFP (MAM1X et MAM 9X) conçus pour stabiliser les MAM 15,26 serrés (6 nm ± 1 nm) et lâches (24 nm ± 3 nm) sont des outils remarquables pour mesurer quantitativement la stabilisation MAM. Les deux stabilisateurs présentent non seulement une expression égale et stable dans les cellules ReN GA différenciées dans la matrice 3D pendant ~10 jours, mais ont également détecté des MAM dans des points discrets dans le soma et les axones. Plus important encore, alors que l’expression stable de MAM 1X dans le ReN GA 3D enrichi en FACS augmentait significativement la génération d’Aβ, l’expression de MAM 9X n’avait aucun effet17. Nous avons également testé l’effet d’un stabilisateur constitutif de MAM contenant 18 acides aminés (MAM 18X) qui détecte et stabilise les MAM >25 nm. Contrairement à MAM 1X ou MAM 9X, MAM 18X a exclusivement étiqueté les MAM somaux. Neurones ReN GA exprimant MAM 18X enrichis en FACS, génération Aβ réduite17. Ces résultats ont suggéré la possibilité d’un seuil de stabilité des MAM déterminé par la largeur de leur écart qui varie entre les MAM serrées pathogènes (augmentation de la génération d’Aβ) et les MAM lâches non pathogènes (maintien ou réduction de la génération d’Aβ).La découverte d’un modulateur MAM efficace et de sa concentration optimale qui peut atteindre la stabilisation optimale de la MAM requise pour franchir le seuil des MAM pathogènes et non pathogènes révélera une voie thérapeutique remarquable pour abaisser l’Aβ axonal ou neuronal génération dans le cerveau.
Trois approches différentes ont été employées pour développer des modulateurs MAM : (1) des modulateurs qui ciblent les protéines de connexion MAM, (2) des modulateurs qui modifient les niveaux d’expression des protéines résidentes de MAM, et (3) des modulateurs des structures MAM18. Malgré ces approches, le principal obstacle à la recherche de modulateurs efficaces de la stabilisation de la MAM est le manque de méthodes permettant de mesurer quantitativement le degré de stabilisation de la MAM. Les techniques traditionnelles comme la microscopie électronique (EM) ou la microscopie à super-résolution ont des limites pour capturer les changements en temps réel ou fournir suffisamment de détails pour évaluer la stabilisation de la MAM (examiné dans28).
La méthode décrite ici permettra de surmonter cet obstacle et de fournir des informations clés sur la relation entre la stabilisation de la MAM et la production d’Aβ. Les résultats montrent que les MAM d’une épaisseur de 6 nm ± 1 nm, affichant un mouvement global de 26,6 % ± 3,4 % (Tableau 3), sont associées à la génération d’Aβ. En revanche, les MAM d’une épaisseur de 24 nm ± 3 nm, qui présentent un mouvement global de 44,79 % ± 2,6 % (tableau 3), n’influencent pas la génération d’Aβ. Le mouvement global des mitochondries (Mito-RFP) était de 53,82 % ± 3,3 %. Étant donné que l’épaisseur de la MAM varie généralement entre 6 nm et 80 nm, ces résultats délimitent les limites supérieure et inférieure de la stabilisation de la MAM par rapport à la production d’Aβ. Par conséquent, cette méthode peut guider l’identification et l’optimisation d’un ou plusieurs modulateurs pour la stabilisation MAM. L’objectif serait de modifier le mouvement global des MAM de 26,6 % ± 3,4 % à 53,82 % ± 3,3 %, ou leur vitesse moyenne de  0,4 μm/s à
0,4 μm/s à  0,7 μm/s (tableau 3), positionnant ces modulateurs comme des agents thérapeutiques potentiels contre la production d’Aβ.
0,7 μm/s (tableau 3), positionnant ces modulateurs comme des agents thérapeutiques potentiels contre la production d’Aβ.
L’utilisation de modulateurs MAM constitutifs contenant des linkers synthétiques de longueurs croissantes (0-18 acides aminés) est une méthode puissante pour déterminer quantitativement le seuil de stabilisation MAM afin de passer à une stabilisation MAM qui maintient ou éventuellement diminue au lieu d’augmenter la génération d’Aβ. Cependant, pour évaluer l’efficacité ou l’efficience des modulateurs MAM, des stabilisateurs MAM inductibles seront nécessaires. Il existe des stabilisateurs MAM inductibles basés sur la microscopie d’imagerie à vie de résonance de Förster (FRET/FLIM) qui sont des plasmides d’expression codant pour la séquence de ciblage OMM de mAKAP1 (34-63) et la phosphatase Sac1 (521-587) fusionnée au CFP (521-587). De plus, les stabilisants constitutifs peuvent ne pas représenter les MAM physiologiques, tandis que les stabilisants FRET/FLIM MAM, en revanche, détecteront les MAM physiologiques. Les sondes GFP divisées où la GFP est divisée en deux fragments non fluorescents attachés soit aux protéines résidentes ER ou mitochondriales ER-GFP (1-10) et Mito-GFP11 qui génèrent une complémentation de fluorescence biomoléculaire (BiFC) lors de la formation de MAMs27, peuvent également être utilisées. Bien que les fragments GFP soient sujets à l’assemblage spontané, BiFC a la lecture la plus simple, le signal le plus clair et le moins d’analyse associée au bruit. De plus, l’interaction entre les GFP divisés est hautement réversible28, de sorte que leurs avantages l’emportent sur les inconvénients et rendent la méthode BiFC adaptée à l’identification des modulateurs de stabilisation MAM.
Remerciements
Nous remercions le Dr György Hajnóczky, professeur à l’Université Thomas Jefferson, à Philadelphie, de nous avoir généreusement fourni des plasmides d’expression codant pour RFP-Mito, MAM 1X, MAM 9X et MAM 18X. Nous remercions tout particulièrement la Dre Lai Ding, scientifique principale en imagerie, Brigham and Women’s Hospital, de nous avoir aidés à écrire le code pour générer, suivre et mesurer les données du kymographe. Cette étude a été financée par le Cure Alzheimer’s Fund à RB et la subvention NIH 5R01NS045860-20 à RET.
matériels
| Name | Company | Catalog Number | Comments |
| 6 Well Glass Bottom Plate | Cellvis | P06-1.5H-N | |
| B-27 Supplement (50X), serum free | Gibco/Thermo Fisher Scientific | 17504044 | |
| bFGF | R&D System | 233-FB | |
| BSA | Fisher Scientific | 501781532 | |
| Countess Cell Counting Chamber Slides | Invitrogen | C10283 | |
| DMEM/F12 with L-glutamine | Gibco/Thermo Fisher Scientific | 11320-033 | |
| EDTA | Life Technologies | 41116134 | |
| EGF | Sigma-Aldrich | 92090408 | |
| Falcon 6 Well Plates | VWR International | 41122107 | |
| GAPDH Polyclonal Antibody | Thermo Fisher Scientific | PA1-988 | |
| Gelatin | VWR International | 9000-70-8 | |
| Graphpad Prism N/A | Prism 9, version 9.5.0 | N/A | |
| Heparin | Sigma-Aldrich | H0200000 | |
| ImageJ Software | ImageJ 1.53a | N/A | |
| Matrigel Basement Membrane Matrix | Corning | 356234 | |
| mCherry Polyclonal Antibody | Invitrogen | PA5-34974 | |
| MS Excel | Microsoft Excel, version 2302 | N/A | |
| Multi-array electrochemiluminescence assay kit | Meso Scale Diagnostics (MSD) | K15200E-2 | V-PLEX Aβ Peptide Panel 1 (6E10) kit |
| NaCl | Fisher Scientific | 7647145 | |
| NuPAGE 4–12% Bis-Tris gel | Invitrogen | NP0321BOX | |
| Penicillin/Streptomycin/Amphotericin B | Lonza | 17-745E | |
| Photoshop | Adobe Photoshop CC 20.0.10 | N/A | |
| Rat Neuron Nucleofector Kit | Lonza | VPG-1003 | |
| StemPro Accutase | Gibco | A1110501 | |
| Tris-HCL, pH 7.6 | Boston BioProducts | 42000000 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween 20 | Fisher Scientific | 501657287 |
Références
- Giacomello, M., Pellegrini, L. The coming of age of the mitochondria-ER contact: a matter of thickness. Cell Death Differ. 23 (9), 1417-1427 (2016).
- Degechisa, S. T., Dabi, Y. T., Gizaw, S. T. The mitochondrial associated endoplasmic reticulum membranes: A platform for the pathogenesis of inflammation-mediated metabolic diseases. Immun Inflamm Dis. 10 (7), e647 (2022).
- Schon, E. A., Area-Gomez, E. Mitochondria-associated ER membranes in Alzheimer disease. Mol Cell Neurosci. 55, 26-36 (2013).
- Erpapazoglou, Z., Mouton-Liger, F., Corti, O. From dysfunctional endoplasmic reticulum-mitochondria coupling to neurodegeneration. Neurochem Int. 109, 171-183 (2017).
- Sala-Vila, A., et al. Interplay between hepatic mitochondria-associated membranes, lipid metabolism and caveolin-1 in mice. Sci Rep. 6, 27351 (2016).
- Fujimoto, M., Hayashi, T. New insights into the role of mitochondria-associated endoplasmic reticulum membrane. Int Rev Cell Mol Biol. 292, 73-117 (2011).
- Hung, V., et al. Proteomic mapping of cytosol-facing outer mitochondrial and ER membranes in living human cells by proximity biotinylation. eLife. 6, e24463 (2017).
- Area-Gomez, E., Schon, E. A. On the pathogenesis of Alzheimer's disease: The MAM hypothesis. FASEB J. 31 (3), 864-867 (2017).
- Rizzuto, R., et al. Close contacts with the endoplasmic reticulum as determinants of mitochondrial Ca2+ responses. Science. 280 (5370), 1763-1766 (1998).
- Sukhorukov, V. S., et al. Molecular mechanisms of interactions between mitochondria and the endoplasmic reticulum: A new look at how important cell functions are supported. Mol Biol. 56 (1), 59-71 (2022).
- Zhang, P., Konja, D., Zhang, Y., Wang, Y. Communications between Mitochondria and endoplasmic reticulum in the regulation of metabolic homeostasis. Cells. 10 (9), 2195 (2021).
- Ziegler, D. V., Martin, N., Bernard, D. Cellular senescence links mitochondria-ER contacts and aging. Commun Biol. 4 (1), 1323 (2021).
- Csordas, G., et al. Structural and functional features and significance of the physical linkage between ER and mitochondria. J Cell Biol. 174 (7), 915-921 (2006).
- Cieri, D., et al. SPLICS: a split green fluorescent protein-based contact site sensor for narrow and wide heterotypic organelle juxtaposition. Cell Death Differ. 25 (6), 1131-1145 (2018).
- Carpio, M. A., et al. BOK controls apoptosis by Ca(2+) transfer through ER-mitochondrial contact sites. Cell Rep. 34 (10), 108827 (2021).
- Prudent, J., et al. MAPL SUMOylation of Drp1 stabilizes an ER/mitochondrial platform required for cell death. Mol Cell. 59 (6), 941-955 (2015).
- Zellmer, J. C., Tarantino, M. B., et al. Stabilization of mitochondria-associated endoplasmic reticulum membranes regulates Abeta generation in a three-dimensional neural model of Alzheimer’s disease. Alzheimer’s Dement. , 1-20 (2024).
- Magalhaes Rebelo, A. P., et al. Chemical modulation of mitochondria-endoplasmic reticulum contact sites. Cells. 9 (7), 1637 (2020).
- Berger, S. I., Iyengar, R. Role of systems pharmacology in understanding drug adverse events. Wiley Interdiscip Rev Syst Biol Med. 3 (2), 129-135 (2011).
- Friedman, J. R., Webster, B. M., Mastronarde, D. N., Verhey, K. J., Voeltz, G. K. ER sliding dynamics and ER-mitochondrial contacts occur on acetylated microtubules. J Cell Biol. 190 (3), 363-375 (2010).
- Wu, Y., et al. Contacts between the endoplasmic reticulum and other membranes in neurons. Proc Natl Acad Sci U S A. 114 (24), E4859-E4867 (2017).
- Cagin, U., et al. Mitochondrial retrograde signaling regulates neuronal function. Proc Natl Acad Sci U S A. 112 (44), E6000-E6009 (2015).
- Choi, S. H., et al. A three-dimensional human neural cell culture model of Alzheimer's disease. Nature. 515 (7526), 274-278 (2014).
- Kim, Y. H., et al. A 3D human neural cell culture system for modeling Alzheimer's disease. Nat Protoc. 10 (7), 985-1006 (2015).
- Kwak, S. S., et al. Amyloid-beta42/40 ratio drives tau pathology in 3D human neural cell culture models of Alzheimer's disease. Nat Commun. 11 (1), 1377 (2020).
- Csordas, G., et al. Imaging interorganelle contacts and local calcium dynamics at the ER-mitochondrial interface. Mol Cell. 39 (1), 121-132 (2010).
- Bhattacharyya, R., et al. Axonal generation of amyloid-beta from palmitoylated APP in mitochondria-associated endoplasmic reticulum membranes. Cell Rep. 35 (7), 109134 (2021).
- Tebo, A. G., Gautier, A. A split fluorescent reporter with rapid and reversible complementation. Nat Commun. 10 (1), 2822 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon