Method Article
Análisis cuantitativo de la estabilización de la membrana del retículo endoplásmico (MAM) asociada a mitocondrias en un modelo neural de la enfermedad de Alzheimer (EA)
En este artículo
Resumen
Aquí, describimos la medición de la tasa de transporte axonal de los estabilizadores constitutivos de las membranas del retículo endoplásmico (RE) (MAM) asociadas a las mitocondrias mediante el aumento o el mantenimiento de la generación de β-amiloide (Aβ) neurotóxica de las neuronas de la enfermedad de Alzheimer (EA) en tiempo real para que sirva como una métrica directa y cuantitativa para medir la estabilización de MAM y ayudar al desarrollo de terapias para la EA.
Resumen
Aquí se presenta un método para cuantificar la estabilización de las membranas del retículo endoplásmico (MAM) asociadas a las mitocondrias en un modelo neuronal tridimensional (3D) de la enfermedad de Alzheimer (EA). Para empezar, las células ReN neuroprogenitoras humanas frescas que expresan β-proteína precursora de amiloide (APP) que contiene enfermedad de Alzheimer familiar (FAD) o células ReN ingenuas se cultivan en placas de cultivo de tejidos delgadas (1:100) recubiertas de Matrigel. Una vez que las células alcanzan la confluencia, estas se electroporan con plásmidos de expresión que codifican la secuencia de unión a mitocondrias conjugadas con la proteína de fluorescencia roja (RFP) de AKAP1(34-63) (Mito-RFP) que detecta mitocondrias o estabilizadores MAM constitutivos MAM 1X o MAM 9X que estabilizan MAMs apretados (6 nm ± 1 nm de ancho de espacio) o sueltos (24 nm ± 3 nm de ancho de brecha), respectivamente. Después de 16-24 h, las células se cosechan y enriquecen mediante un clasificador celular activado por fluorescencia (FACS). Un número igual de células enriquecidas con FACS se siembran en la matriz tridimensional (1:1 Matrigel) y se les permite diferenciarse en neuronas maduras durante 10 días. Las imágenes de células vivas de las células diferenciadas de 10 días que expresan los estabilizadores MAM conjugados con RFP se capturan bajo un microscopio fluorescente equipado con una cámara de cultivo de imágenes de células vivas que mantiene el CO2 (5%), la temperatura (37 °C) y la humedad (~90%). Con este fin, realizamos imágenes de células vivas y análisis cimográficos para medir la motilidad de las mitocondrias libres marcadas con Mito-RFP o mitocondrias unidas a ER de anchos de espacio estrechos o sueltos estabilizadas por MAM 1X o MAM 9X, respectivamente, en el proceso neuronal más extendido de cada neurona ReN GA que tiene al menos 500 nm de largo, considerándolos como axones.
Introducción
La evidencia emergente sugiere que los contactos especializados del retículo endoplásmico (MERCs) asociados a las mitocondrias, recolectados bioquímicamente como membranas RE asociadas a las mitocondrias, a menudo denominadas MAMs 1,2 desempeñan un papel en varias enfermedades neurodegenerativas, incluida la EA 3,4. Estos MAMs están compuestos por microdominios lipídicos ricos en colesterol en el RE y en la membrana externa de las mitocondrias unidos por una serie de proteínas que crean diversidades estructurales y funcionales entre los MAMs 5,6,7. La hipótesis de la MAM recientemente acuñada postula que el aumento de las MAM conduce a una mayor producción de Aβ y a la cascada patogénica de la EA, incluida la formación de ovillos neurofibrilares (NFT), la dishomeostasis del calcio y la neuroinflamación 3,8. Alrededor del 5%-20% de las mitocondrias entran en contacto físico con el RE para formar MAMs9. El ancho del espacio de los MAM está determinado por el ER suave y rugoso (sER y rER, respectivamente). La anchura de brecha variable entre sER-mitocondrias (10-50 nm) y rER-mitocondrias (50-80 nm) sugiere que la anchura de brecha de los MAM tiene un espectro largo que oscila entre estrecho (~10 nm) y suelto (~80 nm)10,11,12,13. La anchura del espacio MAM determina las funciones de MAM, como la homeostasis del calcio y el transporte de lípidos 1,14. Un informe reciente ha demostrado que los MAMs formados entre el RE estrechamente conectado (~10 nm) y las mitocondrias, llamados MAMs completos, son apoptóticos. Por el contrario, los MAMs formados entre el RE débilmente conectado (~25 nm) y las mitocondrias, denominados MAMs defectuosos o medianos, son antiapoptóticos 14,15,16. La estabilización de MAMs con un ancho de separación de 6 nm ± 1 nm aumentó la generación de Aβ a partir de un nuevo modelo de cultivo neuronal tridimensional (3D) de AD. Por el contrario, la estabilización de MAMs con un ancho de separación de 24 nm ± 3 nm no tiene ningún efecto sobre la generación Aβ17. Este hallazgo sugiere por primera vez que regular el grado de estabilización de MAM, pero no desestabilizar los MAM, es la clave para regular la generación de Aβ. Un intento de desestabilizar completamente los MAMs puede tener consecuencias no deseadas porque los MAMs mantienen varios eventos celulares críticos para la supervivencia celular12.
La modulación de los MAMs es un área emergente de investigación con implicaciones potenciales para diversos trastornos, incluyendo el cáncer, los trastornos metabólicos y las enfermedades neurodegenerativas18. A pesar de la disponibilidad de muchos moduladores de MAM, hasta ahora no se ha hecho ningún intento importante para probar su capacidad para desestabilizar los MAMs y reducir la patología de la EA, principalmente porque las diversidades estructurales de los MAMs los convierten en un sistema muy complejo para el descubrimiento de fármacos. Sin embargo, la farmacología de sistemas estructurales recientemente desarrollada, que considera las propiedades específicas de las dianas farmacológicas y su entorno18,19, debería superar las dificultades y desarrollar fármacos altamente potentes dirigidos a los MAM o a las proteínas asociadas a MAM en la EA. Sin embargo, la búsqueda de un modulador eficaz de la estabilización de MAM requiere métodos para cuantificar con precisión el grado de estabilización de MAM. Las técnicas tradicionales como la microscopía electrónica (EM) o la microscopía de superresolución tienen limitaciones para determinar la estabilización de MAM. Superar estos desafíos probablemente requeriría el desarrollo de técnicas de imagen novedosas y más dinámicas o ensayos bioquímicos que puedan proporcionar medidas cuantitativas de la estabilización de MAM en células vivas. La microscopía electrónica de barrido por haz de iones focalizada (FIB-SEM) de neuronas primarias reveló que el RE tiende a formar una red alrededor de las mitocondrias que probablemente limite la motilidad mitocondrial20,21. La interrupción de los sistemas de transporte mitocondrial, ya sea retrógrado, anterógrado o ambos, tuvo un profundo impacto en la función sináptica y neuronal22. Por lo tanto, el novedoso análisis basado en imágenes de células vivas y en la cimografía de la velocidad axonal de las mitocondrias unidas a RE descrito aquí como una métrica para medir cuantitativamente la estabilización de MAM facilitará la identificación de moduladores de MAM que pueden cambiar el umbral de estabilización de MAM a uno que mantenga o posiblemente disminuya en lugar de aumentar la generación de Aβ.
Protocolo
Modelos de cultivo neuronal de EA: En este estudio se utilizaron neuronas derivadas de células ReN progenitoras neurales humanas [ReN naïve (Millipore)] o células ReN que expresan mutaciones familiares de la EA (fAD) en el gen de la proteína precursora de amiloide (APP) (APPSwe/Lon), células ReN GA. El sistema de cultivo tridimensional (3D) ReN-GA recapitula la patología de la EA, a saber, los oligómeros Aβ oligómeros (NFT) 23,24. Las células ReN ingenuas están disponibles comercialmente. Las líneas ReN GA se obtuvieron del Dr. Doo Y. Kim, Profesor Asociado, Hospital General de Massachusetts (MGH)23,24,25.
Plásmidos de expresión: AKP1 (34-63) y la secuencia de proteínas Ubc 6 (283-303) enlazadas directamente con RFP (Mito-RFP-ER denotada como MAM 1X) o contienen un enlazador de 9 aminoácidos (Mito-9X-RFP-ER denotado como MAM 9X) diseñado para estabilizar MAMs de 6 nm ± 1 nm o 24 nm + 3 nm anchos de espacio, respectivamente 15,26 (Figura 1A).
1. Electroporación
- Transfectación de las células con estabilizadores MAM (1-2 h)
NOTA: Siga el protocolo descrito por el fabricante del kit de nucleofactor (Tabla de materiales). Antes de comenzar, prepare placas de cultivo de 6 pocillos con fondo de vidrio pre-recubiertas con DMEM/F12 que contengan un 1% de Matrigel (en adelante, matriz de membrana basal [BMM]). Use 2 mL de BMM para cubrir cada pocillo. Incubar durante al menos 1 h a 37 °C. El BMM, los medios, las puntas de pipeta y las pipetas deben enfriarse previamente antes de mezclar para evitar que el BMM se solidifique.- Aspire la mezcla de BMM. Reemplace con 2 mL de DMEM/F12 a temperatura ambiente (RT) en cada pocillo.
- Combine Nucleofector con Suplemento 1 en una proporción de 4,5:1 (82 μL:18 μL de proporción Nucleofector/Suplemento para 100 μL de solución). Para cada transfección deseada, se necesitan 100 μL de Nucleofector.
- ADN de vórtice. A continuación, añadir 1-5 μg de ADN (por tratamiento) en tubos de microcentrífuga de 1 mL para cada transfección.
- Use acustase para recolectar una placa de células ReN-GA sanas.
NOTA: Coloque las células en placas en medios de expansión (Tabla 1) en placas de cultivo de tejidos de 100 mm y espere hasta que la confluencia alcance el 70-80%.- Aspire el medio existente y lávese una vez con 10 mL de PBS.
- Añadir 1 mL de acutasa directamente a las células e incubar durante 5-10 min a 37 °C.
- Desaloje las células del plato golpeando suavemente el costado del plato. Revise bajo un microscopio para asegurarse de que las células estén aflojadas y fluyan libremente.
- Neutralice la acutasa con 10 mL de DMEM/F12 y transfiérala a un tubo limpio de 15 mL.
- Centrifugar el número requerido de células a 274 x g durante 5 min y aspirar el sobrenadante para eliminar las células muertas. A continuación, vuelva a suspender el pellet en 10 mL de DMEM/F12.
- Cuente las celdas para determinar la densidad.
NOTA: En este estudio, se utilizó un contador de células automatizado para contar el número de células. Por electroporación, se necesitan 3-5 X 106 celdas (por ejemplo, 5 tratamientos requerirían 15-25 X 106 celdas).- Tome 10 μL de las células suspendidas y añádalas al lado A de una cámara de recuento de células. A continuación, añada 10 μL al lado B.
NOTA: Añada la suspensión de células inclinando la pipeta hacia un lado. Evite las burbujas al no empujar más allá del primer tope en el émbolo de la pipeta. - Lleve la cámara de conteo llena al contador de celdas e inserte el lado A en la ranura principal de la parte delantera.
- Después de presionar Medir, anote el número de celdas por mL.
- Saque la cámara, voltee al lado B y repita los pasos 1.1.5.2-1.1.5.3.
- Suma estos dos números, luego divídelos por 4 (si no usas Trypan Blue), luego multiplica el número por los mililitros de líquido en los que están suspendidas las celdas para obtener el número total de celdas en la suspensión.
- Tome 10 μL de las células suspendidas y añádalas al lado A de una cámara de recuento de células. A continuación, añada 10 μL al lado B.
- Centrifugar el número requerido de células a 274 x g durante 5 min y aspirar el sobrenadante.
- Resuspender el pellet en 100 μL de mezcla Nucleofector por electroporación. (p. ej., 500 μL para 5 tratamientos).
NOTA: Dejar las células en la solución Nucleofector durante más de 15 minutos podría reducir la viabilidad celular y la eficacia general. - Añadir 100 μL de la suspensión celular a uno de los tubos que contienen ADN y mezclar mediante pipeteo.
- Transfiera la mezcla de ADN a una cubeta de electroporación y selle con la tapa provista. Para evitar la creación de burbujas, incline la cubeta hacia abajo y pipetee lentamente.
- Seleccione el programa Nucleofector apropiado para el dispositivo que se está utilizando. Para el dispositivo utilizado en este estudio, utilice el Programa A-033 para la transfección. Para optimizar, pruebe los 5 programas de Nucleofector para determinar el más apropiado para cada tipo de célula.
NOTA: Una confirmación de una electroporación exitosa es la espuma visible en la parte superior de la mezcla. - Inmediatamente, utilizando las pipetas estériles suministradas, añada ~500 μL de DMEM de la placa de 6 pocillos precargada a la cubeta. Mezcle suavemente una vez y luego transfiera las celdas electroporadas y el medio al pocillo correspondiente.
NOTA: Después de la electroporación, las celdas son extremadamente sensibles, por lo que es esencial transferir los medios rápidamente y pipetear con cuidado. - Repita los pasos 1.1.8-1.1.11 para todos los tratamientos de ADN restantes.
- Incubar las células a 37 °C en presencia de CO2 (5%) durante la noche.
- Al día siguiente, intercambie los medios con medios de diferenciación nuevos (Tabla 2) y permita que las células se diferencien durante 10 días. Cada 2-3 días, reemplácelo con medios nuevos.
2. Imágenes de células vivas
- Preparación de células para microscopía de células vivas (30 min)
- Preparación de la cámara de células vivas (antes de mover las células a la cámara)
- Asegúrese de que el tanque deCO2 y el humidificador estén conectados a la cámara, que las válvulas estén abiertas y que los tanques estén llenos.
- Ajuste la temperatura a 37 °C, el CO2 al 5% y la humedad al 95%. (Esto puede llevar algún tiempo para que los niveles se equilibren).
- Coloque una placa de 6 pocillos que contenga células en la cámara y ajuste el enfoque del microscopio hasta que las células se vuelvan visibles.
- Encienda el láser (Tabla de Materiales).
- Para capturar la señal RFP, excite el fluoróforo con un láser de 594 nm y use una emisión de 570-640 nm. Para GFP, use un láser de 488 nm para la excitación y la emisión de 510-540 nm.
- Con el filtro fluorescente incorporado, ajuste la intensidad de la señal hasta que la señal de fondo se haya disipado (debe ser casi negra sólida).
- Captura de vídeo en directo de los axones (10 h-15 h en total)
NOTA: Se utilizó un microscopio confocal invertido Nikon C2 Eclipse Ti2 para capturar imágenes fluorescentes utilizando el software NIS Element AR. Utilice un aumento de 60x a una resolución de 512 píxeles, tomando vídeos a 1 fotograma por segundo durante 3 minutos produciendo kymographs más limpios.- Encuentre una célula que exprese biosensores RFP. Exporte una imagen de la celda para referencia posterior antes de tomar un video.
- Recorte el área de escaneo para que quepa alrededor del axón. El uso de un área de escaneo más pequeña reduce el tiempo de procesamiento del microscopio y facilita mucho la generación de la cimografía.
- Apague todos los láseres para ayudar a que el software funcione sin problemas. Tenga en cuenta que cuando el programa se ejecuta con todos los láseres activos, no se capturan todos los fotogramas.
NOTA: La señal fluorescente roja del soma es mucho más brillante que la del axón. Por lo tanto, se excluye el soma para aumentar la intensidad general de la señal en el axón. Vaya a la pestaña Medición de tiempo . - Establezca el intervalo en 1 fotograma por segundo y establezca el tiempo total en 181 s.
- Haga clic en Ejecutar.
- Guarde este archivo como un archivo .nds (debidamente etiquetado) y repita el proceso ~ 10 veces por estabilizadores MAM (MAM 1X o MAM 9X).
NOTA: La potencia del láser a veces produce un efecto de blanqueo notable en la señal RFP si se escanea durante demasiado tiempo. Es importante tener en cuenta el trabajo rápido para capturar videos.
3. Post-procesamiento (7 días)
NOTA: Para analizar el transporte y generar kymographs, se utilizaron macros Fiji ImageJ. Las vesículas que se movieron menos de 0,1 mm/s se clasificaron como estacionarias. La frecuencia del movimiento de las partículas se calculó dividiendo el número de partículas que se mueven en una dirección determinada (anterógrada, retrógrada) o que no se mueven (estacionarias) por el número total de partículas analizadas en el quimograma. El tiempo que cada vesícula pasó en pausa o en movimiento se calculó promediando el porcentaje de tiempo pasado en cada condición para todas las vesículas en cada neurona analizada. La distribución de frecuencias para la velocidad y la longitud de la carrera se calculó utilizando solo vesículas móviles para cada condición experimental. El análisis se realizó en tractos axonales de 100 mm durante 3 min.
- Generación de un kymograph
- Abra el archivo .nds en Fiji ImageJ.
- Vaya a la pestaña Imagen y haga clic en Propiedades. Registre la relación píxel a micra. Esto es necesario para el cálculo posterior.
- Haga clic en Archivo > Guardar como > Tiff para guardar el archivo como un .tiff en su carpeta (la macro guardará automáticamente todo lo generado en esta carpeta).
- Arrastre la macro Kymo a ImageJ. El código se proporciona en el Archivo de codificación suplementaria 1. Haga clic en Ejecutar.
- No presione OK. Cambie a la ventana de MAX_raw. Haga clic con el botón izquierdo manualmente a lo largo del axón y haga doble clic para finalizar el seguimiento.
NOTA: Asegúrese de seguir el axón desde el soma hasta el terminal del axón. De esta manera los cálculos anterógrados y retrógrados serán correctos. - Presione el comando T o Agregar en el administrador de ROI.
- Ahora haga clic en Aceptar. Se generará un kymograph en la carpeta realizada anteriormente. (El eje X es la longitud del axón en micras y el eje Y es el tiempo en segundos).
- Seguimiento del quimógrafo
- Arrastre el kymograph a la imagen J de Fiji (debería colocarse automáticamente en la carpeta realizada anteriormente).
- Arrastre la macro Pista a la imagen J.
- En el siguiente mensaje, haga clic en Aceptar. Si reanuda el trabajo desde antes, introduzca el punto numérico desde el que desea continuar y, a continuación, pulse Aceptar.
- Con la herramienta Rectángulo del panel Seleccionar , seleccione un área cerca del centro del cimógrafo que tenga 60 píxeles de altura y siempre 100 μm de ancho. Para ello, divida 100 por la relación píxel-micro que se registró en el paso 3.1.2.
- Después de seleccionar un área, presione Ctrl T para agregar i t al ROI y guardar el ROI en la carpeta en la que se está trabajando (presione Más en el menú ROI y luego Guardar).
NOTA: Invertir el área seleccionada presionando Ctrl + Shift + I facilitará el seguimiento. - Para seguir el movimiento de una vesícula individual, mantenga presionada la tecla Ctrl mientras hace clic con el botón izquierdo de arriba a abajo.
- Para hacerlo, mantenga presionada la tecla Ctrl mientras hace clic con el botón derecho y aparecerá una ventana que muestra dónde se rastrean los axones. Si esto se ve bien, presione Aceptar.
- Para continuar y seleccionar otra vesícula, haga clic en Sí y repita el proceso de los pasos 3.2.6-3.2.8. Una vez que se haya rastreado cada vesícula visible, presione No. Esto guardará automáticamente la superposición con seguimiento manual en la misma carpeta.
- Medición de los datos del cimograma
- Cree una nueva carpeta. Arrastre todos los archivos de texto generados a la carpeta.
- Abra el archivo de quimógrafo y registre las dimensiones.
- Arrastre la macro Medir a la imagen J.
- Introduzca la relación entre micras y píxeles registrada anteriormente en PixelScale.
- Cambie el límite de velocidad bajo a 0.1. (Las vesículas que se mueven por debajo de este límite se consideran estacionarias).
- Introduzca las dimensiones del kymograph registradas anteriormente. El archivo de resumen se generará automáticamente en la carpeta de texto creada. Cada relleno de resumen contendrá el % de tiempo recorrido, la velocidad total (μm/s), la distancia total, el segmento medio recorrido, el número de veces que se detuvo y el número de veces que se invirtió. El código para generar, rastrear y medir los datos del kymograph se proporciona como Archivo de codificación suplementario 1.
Resultados
Se realizaron imágenes de células vivas y análisis cimográficos para medir la motilidad de las mitocondrias libres marcadas con Mito-RFP o mitocondrias unidas a ER de anchos de contacto estrechos (6 nm ± 1 nm) o sueltos (24 nm ± 3 nm) estabilizados por MAM 1X o MAM 9X, respectivamente, en el proceso neuronal más largo de cada neurona ReN GA (AD) o ReN (naïve) que tenga al menos 500 nm de largo, considerándolo como un axón (Figura 1 y Figura 2). Las frecuencias de los movimientos (generales, retrógrados y anterógrados) se calcularon dividiendo el número de puntos marcados con RFP (MAM) en movimiento o estacionarios por el número total en los kymographs (Figura 1A-E). La velocidad axonal general de las mitocondrias unidas a RE marcadas con MAM 1X disminuyó drásticamente en ~50% en comparación con las mitocondrias libres de RE marcadas con Mito-RFP o las mitocondrias unidas a ER marcadas con MAM 9X (Figura 1B). El análisis cuantitativo también reveló diferencias dramáticas entre los movimientos generales y retrógrados de las mitocondrias unidas a RE estabilizadas con MAM 1X en comparación con las mitocondrias libres (Mito-RFP) o estabilizadas con MAM 9X unidas a ER. Mientras que el 53,82 % ± el 3,3 % de las mitocondrias libres de RE (Mito-RFP) eran móviles, solo el 26,6 % ± el 3,4 % de las mitocondrias unidas a RE marcadas con MAM 1X eran móviles, lo que sugiere que la estabilización de los MAM redujo significativamente la movilidad axonal general de las mitocondrias, estrechamente asociadas con el RE, en comparación con las mitocondrias no unidas o débilmente unidas al RE (44,79 % ± 2,6 % de MAM 9X frente al 53,82 % ± 3,3 % de las Mito RFP, respectivamente) (Figura 1C). Consistentemente, tanto los movimientos retrógrados como los anterógrados de las mitocondrias unidas a ER marcadas con MAM 1X fueron significativamente más bajos en comparación con las mitocondrias libres o marcadas con MAM 9X (Mito-RFP) (retrógradas: 12,33 % ± 2,55 % para MAM 1X frente a 25,78 % ± 2,31 % para Mito RFP; anterógradas: 14,27 % ± 2,81 % para MAM 1X frente a 28,04 % ± 2,48 % para Mito RFP) (Figura 1D y E). La Tabla 3 proporciona las velocidades axonales precisas de las mitocondrias libres o de aquellas que están unidas fuerte o débilmente al RE. Estos valores se pueden utilizar como un medio cuantitativo notable para evaluar el grado de estabilización de MAM que oscila entre los MAM apretados y sueltos, lo que conduce a la reducción de la generación de Ab. Las tasas de transporte axonal mitocondrial tras la estabilización de los MAMs apretados y sueltos en las células ReN naïve reflejaron los patrones de transporte observados en las neuronas ReN GA (Figura 1F-G). Los resultados consistentes entre las neuronas ReN ingenuas y las neuronas ReN GA AD que expresan APP Swe/Lon sugieren que el efecto sobre el transporte axonal se atribuye predominantemente al estado de estabilización de MAM, independientemente de la presencia de APPSwe/Lon o de la producción de Aβ resultante.
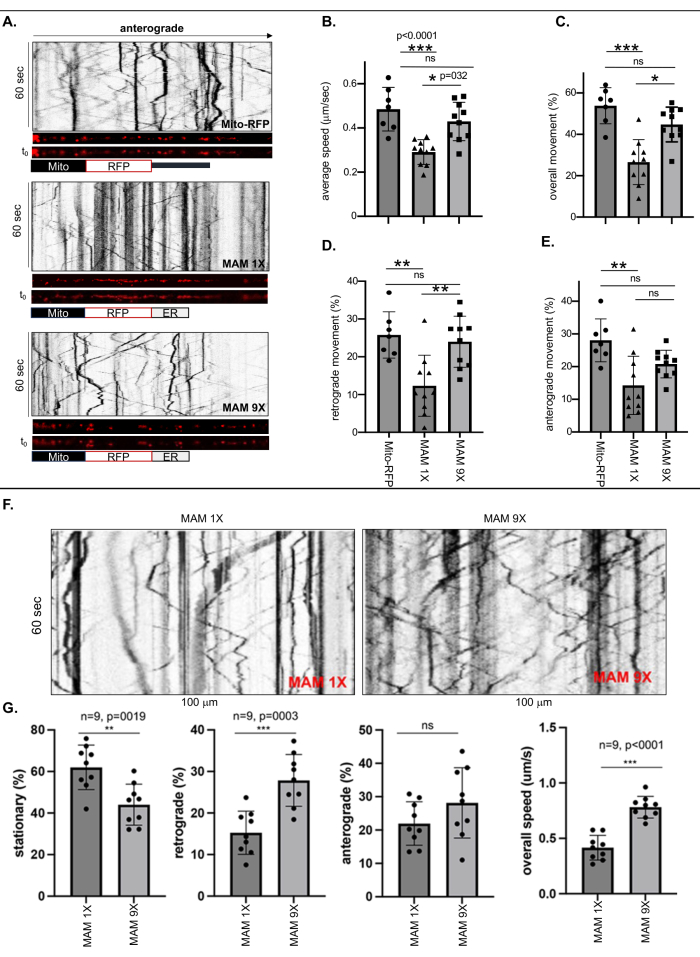
Figura 1: La estabilización de MAMs por MAM 1X redujo la velocidad media y el movimiento (general, retrógrado y anterógrado) de las mitocondrias unidas a ER en los axones de las células ReN GA diferenciadas y las células ReN naïve. (A) Quimogramas representativos de los puntos marcados con RFP que representan mitocondrias libres (Mito-RFP) o mitocondrias unidas a ER estabilizadas por MAM 1X (MAM ajustados, 6 nm ± 1 nm de ancho de contacto) o MAM 9X (MAMs sueltos, 24 nm ± 3 nm de ancho de contacto) dentro de los axones (~100 nm). (B-E) Análisis cuantitativo de la (B) velocidad y movimiento promedio [(C) general, (D) retrógrado y (E) anterógrado] de Mito-RFP, MAM 1X o MAM 9X dentro de los axones de células Ren-GA diferenciadas de 10 días. n>7; Se realizó un ANOVA de dos vías. *p < 0,05, **p < 0,001. Representante de tres experimentos independientes. (F) Quimógrafos representativos del movimiento de MAM 1X o MAM 9X dentro de los axones de células ReN vírgenes diferenciadas de 10 días. (G) Análisis cuantitativo del porcentaje (%) de movimiento (estacionario, retrógrado y anterógrado) y la velocidad general (micrómetro/segundo; μm/s) de MAM 1X o MAM 9X estabilizados dentro de los axones de células ReN vírgenes. n = 9; Se realizó un ANOVA de dos vías. p < 0,0001. Esta figura ha sido adaptada con permiso de Zellmer et al.17. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Imágenes de células vivas de axones de células ReN vírgenes que expresan MAM 1X o MAM 9X. Imágenes de vídeo representativas de células vivas que muestran los movimientos de los MAMs estabilizados por MAM 1X o MAM 9X dentro de axones de 100 μm de largo de células ReN diferenciadas que expresan GFP durante 10 días. n > 10 imágenes de experimentos duplicados. Las flechas indican el transporte anterógrado. Barra de escala: 100 μm. Haga clic aquí para ver una versión más grande de esta figura.
| Reactivo | Concentración final | Importe |
| DMEM con L-glutamina | N/A | 500 mL |
| Heparina | 2 μg/mL | 0,5 ml |
| B27 | 1 vez | 10 mL |
| bFGF | 20 ng/mL | 0,4 mL |
| FEAG | 20 ng/mL | 0,5 ml |
| Penicilina/Estreptomicina | 100 unidades/mL | 5 mL |
| Total | 516.4 mL | |
| Filtre el medio antes de agregar penicilina/estreptomicina. Almacenar a 4 °C hasta por 1 mes. | ||
Tabla 1: Composición de los medios de expansión.
| Reactivo | Concentración final | Importe |
| DMEM con L-glutamina | N/A | 500 mL |
| Heparina | 2 μg/mL | 0,5 ml |
| B27 | 1 vez | 10 mL |
| Penicilion/Estreptomicina | 100 unidades/mL | 5 mL |
| Total | 515.5 mL | |
| Filtre el medio antes de agregar penicilina/estreptomicina. Almacenar a 4 °C hasta 1 mes | ||
Tabla 2: Composición de los medios de diferenciación.
| ReN GA | ReN (ingenuo) | ReN GA (3D) | |||||
| En general (%) | Retrógrado (%) | Anterógrado (%) | Velocidad media (mm/s) | Ab40 (pM) | Ab42 (pM) | ||
| Mito-RFP | 53,82 ± 3,3% | 25,78 ± 2,31% | 28.04 ± 2.48% | 0,66 ± 0,03 | 0,69 ± 0,07 | 241,7 ± 26,74 | 13,77 ± 1,52 |
| MAM 1X | 26,6 ± 3,4% *** | 12,33 ± 2,5% *** | 14,27 ± 2,81% *** | 0,3 ± 0,02*** | 0,43 ± 0,04*** | 377,2 ± 76,87* | 26,62 ± 3,86* |
| MAM 9X | 44.79 ± 2.6% ns | 23.99 ± 2.17%ns | 20.80 ± 1.33%ns | 0,59 ± 0,02 ns | 0,62 ± 0,02 ns | 158,8 ± 3,27* | 17,01 ± 2,02* |
Tabla 3: Análisis cuantitativo. Análisis cuantitativo basado en imágenes de células vivas y cinografía de la velocidad media (velocidad) y los movimientos axonales (general, retrógrado y anterógrado) de Mito-RFP, MAM 9X y MAM 1X. Se realizó un ANOVA de dos vías para la velocidad o el movimiento axonal (%). n = 9. Para Aβ, se realizó un ANOVA ordinario de un factor; n = 3, tres experimentos independientes. La significación se mide con respecto a las células ReN GA no transfectadas (control). **p < 0,0001; *p < 0,05; No significativo (ns). Esta tabla ha sido adaptada con permiso de Zellmer et al.17.
Fichero de codificación suplementario 1: El código para generar, rastrear y medir los datos del kymograph. Haga clic aquí para descargar este archivo.
Discusión
La inhibición del receptor sigma-1 (S1R) disminuyó la estabilización de MAM en los procesos neuronales y redujo drásticamente (~90%) la generación de Aβ a partir de axones pero no de soma de un sistema de cultivo tridimensional (3D) de células progenitoras neurales humanas (ReN) que expresan mutaciones familiares de AD [FAD] en el gen de la proteína precursora de amiloide [APP] (ReN GA)23,24,25,27. Los estabilizadores MAM constitutivos marcados con RFP (MAM 1X y MAM 9X) diseñados para estabilizar MAMs apretados (6 nm ± 1 nm) y sueltos (24 nm ± 3 nm)15,26 son herramientas notables para medir cuantitativamente la estabilización de MAM. Ambos estabilizadores no solo exhiben una expresión igual y estable en las células ReN GA diferenciadas en matriz 3D durante ~ 10 días, sino que también detectaron MAMs en puntos discretos en soma y axones. Y lo que es más importante, mientras que la expresión estable de MAM 1X en 3D ReN GA enriquecido con FACS aumentó significativamente la generación de Aβ, la expresión de MAM 9X no tuvo ningún efecto17. También probamos el efecto de un estabilizador MAM constitutivo que contiene 18 aminoácidos enlazadores (MAM 18X) que detecta y estabiliza los MAMs >25 nm. A diferencia de MAM 1X o MAM 9X, MAM 18X está etiquetado exclusivamente como MAM somales. Las neuronas GA ReN enriquecidas con MAM 18X enriquecidas con FACS reducen la generación de Aβ17. Estos hallazgos sugirieron la posibilidad de un umbral de estabilidad de MAM determinado por su ancho de brecha que oscila entre los MAM apretados patógenos (aumento de la generación de Aβ) y los MAM sueltos no patógenos (mantenimiento o reducción de la generación de Aβ). Encontrar un modulador de MAM eficaz y su concentración óptima que pueda lograr la estabilización óptima de MAM necesaria para cruzar el umbral de los MAM patógenos y no patógenos revelará una vía terapéutica notable para reducir el Aβ axonal o neuronal generación en el cerebro.
Se han empleado tres enfoques diferentes para desarrollar moduladores de MAM: (1) Moduladores que se dirigen a las proteínas de anclaje de MAM, (2) Moduladores que alteran los niveles de expresión de las proteínas residentes en MAM y (3) Moduladores de las estructuras de MAM18. A pesar de estos enfoques, el principal obstáculo para encontrar moduladores eficaces de la estabilización de MAM es la falta de métodos para medir cuantitativamente el grado de estabilización de MAM. Las técnicas tradicionales, como la microscopía electrónica (EM) o la microscopía de superresolución, tienen limitaciones para capturar cambios en tiempo real o proporcionar suficientes detalles para evaluar la estabilización de MAM (revisado en28).
El método descrito aquí superará el obstáculo y proporcionará información clave sobre la relación entre la estabilización de MAM y la producción de Aβ. Los resultados muestran que los MAMs con un espesor de 6 nm ± 1 nm, mostrando un movimiento total de 26.6% ± 3.4% (Tabla 3), se asocian con la generación de Aβ. Por el contrario, los MAMs con un espesor de 24 nm ± 3 nm, que exhiben un movimiento total de 44,79% ± 2,6% (Tabla 3), no influyen en la generación de Aβ. El movimiento total de las mitocondrias (Mito-RFP) fue del 53,82% ± del 3,3%. Dado que el espesor de MAM suele variar entre 6 nm y 80 nm, estos hallazgos delinean los límites superior e inferior de la estabilización de MAM en relación con la producción de Aβ. En consecuencia, este método puede guiar la identificación y optimización de uno o varios moduladores para la estabilización de MAM. El objetivo sería alterar el movimiento general de los MAMs del 26,6% ± 3,4% al 53,82% ± 3,3%, o su velocidad media de 0,4 μm/s a 0,7 μm/s (Tabla 3), posicionando a dichos moduladores como potenciales agentes terapéuticos frente a  la producción de
la producción de  Aβ.
Aβ.
El uso de moduladores MAM constitutivos que contienen enlazadores sintéticos de longitudes crecientes (0-18 aminoácidos) es un método poderoso para determinar cuantitativamente el umbral de estabilización de MAM para cambiar la estabilización de MAM a una que mantenga o posiblemente disminuya en lugar de aumentar la generación de Aβ. Sin embargo, para evaluar la eficiencia o eficacia de los moduladores MAM, se necesitarán estabilizadores MAM inducibles. Existen estabilizadores MAM inducibles basados en microscopía de resonancia de Förster, transferencia de energía/fluorescencia por vida útil (FRET/FLIM), que son plásmidos de expresión que codifican la secuencia de mAKAP1 (34-63) y la fosfatasa Sac1 de RE fusionada con CFP (521-587). Además, es posible que los estabilizadores constitutivos no representen las MAMs fisiológicas, mientras que los estabilizadores MAM FRIT/FLIM, por otro lado, detectarán las MAMs fisiológicas. También se pueden utilizar las sondas de GFP divididas en las que la GFP se divide en dos fragmentos no fluorescentes unidos a las proteínas RE residentes o mitocondriales ER-GFP (1-10) y Mito-GFP11 que generan complementación de fluorescencia biomolecular (BiFC) tras la formación de MAMs27. Aunque los fragmentos de GFP son propensos al ensamblaje espontáneo, BiFC tiene la lectura más simple, la señal más clara y el análisis menos asociado al ruido. Además, la interacción entre la GFP dividida es altamente reversible28, por lo que sus ventajas superan los inconvenientes y hacen que el método BiFC sea adecuado para identificar moduladores de la estabilización MAM.
Agradecimientos
Agradecemos al Dr. György Hajnóczky, Profesor de la Universidad Thomas Jefferson, Filadelfia, por proporcionarnos generosamente plásmidos de expresión que codifican RFP-Mito, MAM 1X, MAM 9X y MAM 18X. Un agradecimiento especial al Dr. Lai Ding, científico sénior de imágenes del Brigham and Women's Hospital, por ayudarnos a escribir el código para generar, rastrear y medir los datos del kymograph. Este estudio contó con el apoyo del Cure Alzheimer's Fund para RB y la subvención de los NIH 5R01NS045860-20 para RET.
Materiales
| Name | Company | Catalog Number | Comments |
| 6 Well Glass Bottom Plate | Cellvis | P06-1.5H-N | |
| B-27 Supplement (50X), serum free | Gibco/Thermo Fisher Scientific | 17504044 | |
| bFGF | R&D System | 233-FB | |
| BSA | Fisher Scientific | 501781532 | |
| Countess Cell Counting Chamber Slides | Invitrogen | C10283 | |
| DMEM/F12 with L-glutamine | Gibco/Thermo Fisher Scientific | 11320-033 | |
| EDTA | Life Technologies | 41116134 | |
| EGF | Sigma-Aldrich | 92090408 | |
| Falcon 6 Well Plates | VWR International | 41122107 | |
| GAPDH Polyclonal Antibody | Thermo Fisher Scientific | PA1-988 | |
| Gelatin | VWR International | 9000-70-8 | |
| Graphpad Prism N/A | Prism 9, version 9.5.0 | N/A | |
| Heparin | Sigma-Aldrich | H0200000 | |
| ImageJ Software | ImageJ 1.53a | N/A | |
| Matrigel Basement Membrane Matrix | Corning | 356234 | |
| mCherry Polyclonal Antibody | Invitrogen | PA5-34974 | |
| MS Excel | Microsoft Excel, version 2302 | N/A | |
| Multi-array electrochemiluminescence assay kit | Meso Scale Diagnostics (MSD) | K15200E-2 | V-PLEX Aβ Peptide Panel 1 (6E10) kit |
| NaCl | Fisher Scientific | 7647145 | |
| NuPAGE 4–12% Bis-Tris gel | Invitrogen | NP0321BOX | |
| Penicillin/Streptomycin/Amphotericin B | Lonza | 17-745E | |
| Photoshop | Adobe Photoshop CC 20.0.10 | N/A | |
| Rat Neuron Nucleofector Kit | Lonza | VPG-1003 | |
| StemPro Accutase | Gibco | A1110501 | |
| Tris-HCL, pH 7.6 | Boston BioProducts | 42000000 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween 20 | Fisher Scientific | 501657287 |
Referencias
- Giacomello, M., Pellegrini, L. The coming of age of the mitochondria-ER contact: a matter of thickness. Cell Death Differ. 23 (9), 1417-1427 (2016).
- Degechisa, S. T., Dabi, Y. T., Gizaw, S. T. The mitochondrial associated endoplasmic reticulum membranes: A platform for the pathogenesis of inflammation-mediated metabolic diseases. Immun Inflamm Dis. 10 (7), e647 (2022).
- Schon, E. A., Area-Gomez, E. Mitochondria-associated ER membranes in Alzheimer disease. Mol Cell Neurosci. 55, 26-36 (2013).
- Erpapazoglou, Z., Mouton-Liger, F., Corti, O. From dysfunctional endoplasmic reticulum-mitochondria coupling to neurodegeneration. Neurochem Int. 109, 171-183 (2017).
- Sala-Vila, A., et al. Interplay between hepatic mitochondria-associated membranes, lipid metabolism and caveolin-1 in mice. Sci Rep. 6, 27351 (2016).
- Fujimoto, M., Hayashi, T. New insights into the role of mitochondria-associated endoplasmic reticulum membrane. Int Rev Cell Mol Biol. 292, 73-117 (2011).
- Hung, V., et al. Proteomic mapping of cytosol-facing outer mitochondrial and ER membranes in living human cells by proximity biotinylation. eLife. 6, e24463 (2017).
- Area-Gomez, E., Schon, E. A. On the pathogenesis of Alzheimer's disease: The MAM hypothesis. FASEB J. 31 (3), 864-867 (2017).
- Rizzuto, R., et al. Close contacts with the endoplasmic reticulum as determinants of mitochondrial Ca2+ responses. Science. 280 (5370), 1763-1766 (1998).
- Sukhorukov, V. S., et al. Molecular mechanisms of interactions between mitochondria and the endoplasmic reticulum: A new look at how important cell functions are supported. Mol Biol. 56 (1), 59-71 (2022).
- Zhang, P., Konja, D., Zhang, Y., Wang, Y. Communications between Mitochondria and endoplasmic reticulum in the regulation of metabolic homeostasis. Cells. 10 (9), 2195 (2021).
- Ziegler, D. V., Martin, N., Bernard, D. Cellular senescence links mitochondria-ER contacts and aging. Commun Biol. 4 (1), 1323 (2021).
- Csordas, G., et al. Structural and functional features and significance of the physical linkage between ER and mitochondria. J Cell Biol. 174 (7), 915-921 (2006).
- Cieri, D., et al. SPLICS: a split green fluorescent protein-based contact site sensor for narrow and wide heterotypic organelle juxtaposition. Cell Death Differ. 25 (6), 1131-1145 (2018).
- Carpio, M. A., et al. BOK controls apoptosis by Ca(2+) transfer through ER-mitochondrial contact sites. Cell Rep. 34 (10), 108827 (2021).
- Prudent, J., et al. MAPL SUMOylation of Drp1 stabilizes an ER/mitochondrial platform required for cell death. Mol Cell. 59 (6), 941-955 (2015).
- Zellmer, J. C., Tarantino, M. B., et al. Stabilization of mitochondria-associated endoplasmic reticulum membranes regulates Abeta generation in a three-dimensional neural model of Alzheimer’s disease. Alzheimer’s Dement. , 1-20 (2024).
- Magalhaes Rebelo, A. P., et al. Chemical modulation of mitochondria-endoplasmic reticulum contact sites. Cells. 9 (7), 1637 (2020).
- Berger, S. I., Iyengar, R. Role of systems pharmacology in understanding drug adverse events. Wiley Interdiscip Rev Syst Biol Med. 3 (2), 129-135 (2011).
- Friedman, J. R., Webster, B. M., Mastronarde, D. N., Verhey, K. J., Voeltz, G. K. ER sliding dynamics and ER-mitochondrial contacts occur on acetylated microtubules. J Cell Biol. 190 (3), 363-375 (2010).
- Wu, Y., et al. Contacts between the endoplasmic reticulum and other membranes in neurons. Proc Natl Acad Sci U S A. 114 (24), E4859-E4867 (2017).
- Cagin, U., et al. Mitochondrial retrograde signaling regulates neuronal function. Proc Natl Acad Sci U S A. 112 (44), E6000-E6009 (2015).
- Choi, S. H., et al. A three-dimensional human neural cell culture model of Alzheimer's disease. Nature. 515 (7526), 274-278 (2014).
- Kim, Y. H., et al. A 3D human neural cell culture system for modeling Alzheimer's disease. Nat Protoc. 10 (7), 985-1006 (2015).
- Kwak, S. S., et al. Amyloid-beta42/40 ratio drives tau pathology in 3D human neural cell culture models of Alzheimer's disease. Nat Commun. 11 (1), 1377 (2020).
- Csordas, G., et al. Imaging interorganelle contacts and local calcium dynamics at the ER-mitochondrial interface. Mol Cell. 39 (1), 121-132 (2010).
- Bhattacharyya, R., et al. Axonal generation of amyloid-beta from palmitoylated APP in mitochondria-associated endoplasmic reticulum membranes. Cell Rep. 35 (7), 109134 (2021).
- Tebo, A. G., Gautier, A. A split fluorescent reporter with rapid and reversible complementation. Nat Commun. 10 (1), 2822 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados