Method Article
アルツハイマー病(AD)の神経モデルにおけるミトコンドリア関連小胞体膜(MAM)安定化の定量的解析
要約
ここでは、アルツハイマー病(AD)ニューロンからの神経毒性β-アミロイド(Aβ)の生成をリアルタイムで増加または維持することにより、ミトコンドリア関連小胞体(ER)膜(MAM)の構成的安定剤の軸索輸送速度を測定することについて説明し、MAMの安定化を測定し、AD治療薬の開発を支援するための直接的かつ定量的な指標として機能します。
要約
ここでは、アルツハイマー病(AD)の3次元(3D)神経モデルにおけるミトコンドリア関連小胞体膜(MAM)の安定化を定量化する方法を示します。まず、家族性アルツハイマー病(FAD)またはナイーブなReN細胞を含むβ-アミロイド前駆体タンパク質(APP)を発現する新鮮なヒト神経前駆細胞ReN細胞を、薄い(1:100)マトリゲル被覆組織培養プレートで増殖させます。細胞がコンフルエントに達した後、ミトコンドリアを検出するAKAP1(34-63)(Mito-RFP)の赤色蛍光タンパク質(RFP)結合ミトコンドリア結合配列をコードする発現プラスミド、またはタイト(6 nm ± 1 nmギャップ幅)またはルーズ(24 nm±3 nm)のMAMをそれぞれ安定化する構成的MAM安定剤MAM 1XまたはMAM 9Xをエレクトロポレーションします。16〜24時間後、細胞を回収し、蛍光活性化セルソーター(FACS)で濃縮します。同数のFACSに富んだ細胞を3次元マトリックス(1:1 Matrigel)に播種し、10日間成熟ニューロンに分化させます。RFP標識MAM安定剤を発現する10日間の分化細胞の生細胞画像を、CO2 (5%)、温度(37°C)、湿度(~90%)を維持した生細胞イメージング培養チャンバーを備えた蛍光顕微鏡で撮影します。この目的のために、私たちは、Mito-RFPで標識された遊離ミトコンドリアまたはMAM 1XまたはMAM 9Xによって安定化されたタイトまたはルーズギャップ幅のER結合ミトコンドリアの遊離ミトコンドリアの運動性を測定するために、少なくとも500 nmの長さの各ReN GAニューロンの最も拡張されたニューロンプロセスで、これらを軸索と見なして運動性を測定するために、生細胞イメージングおよびキモグラフィー分析を行いました。
概要
新たな証拠は、ミトコンドリア関連ER膜として生化学的に採取された特殊なミトコンドリア関連小胞体接触(MERC)が、しばしばMAMs1,2と呼ばれ、AD3,4を含むいくつかの神経変性疾患で役割を果たしていることを示唆しています。これらのMAMは、小胞体とミトコンドリアの外膜にあるコレステロールに富む脂質ラフト様のマイクロドメインで構成されており、MAM5,6,7の間に構造的および機能的多様性を生み出す一連のタンパク質によってつながれています。最近提唱されたMAM仮説では、MAMの増加がAβ産生の増強と、神経原線維もつれ(NFT)形成、カルシウム恒常性障害、神経炎症などのADの病原性カスケードにつながると仮定しています3,8。ミトコンドリアの約5%〜20%がERと物理的に接触してMAMを形成します9。MAMのギャップ幅は、滑らかなERと粗いER(それぞれsERとrER)によって決まります。sER-ミトコンドリア(10-50nm)とrER-ミトコンドリア(50-80nm)の間の可変ギャップ幅は、MAMのギャップ幅がタイト(~10nm)からルーズ(~80nm)までの範囲の長いスペクトルを有することを示唆している10,11,12,13。MAMギャップ幅は、カルシウム恒常性や脂質輸送などのMAM機能を決定する1,14。最近の報告では、密接に接続された(~10 nm)ERとミトコンドリアの間に形成されるMAM(フルMAMと呼ばれる)はアポトーシスであることが示されています。対照的に、緩やかに結合した(~25 nm)ERとミトコンドリアの間に形成されたMAMは、欠陥性MAMまたは中型MAMと呼ばれ、抗アポトーシス性を示します14,15,16。ギャップ幅が6 nm±1 nmのMAMの安定化は、ADの新規3次元(3D)神経培養モデルからのAβ生成を増加させました。対照的に、ギャップ幅が24 nm±3 nmのMAMの安定化は、Aβ世代17に影響を与えません。この知見は、MAMの安定化の度合いを調節すること、しかしMAMを不安定化させないことが、Aβの生成を調節する鍵であることを初めて示唆しています。MAMを完全に不安定化させようとする試みは、MAMが細胞の生存に重要ないくつかの細胞イベントを維持するため、望ましくない結果をもたらす可能性がある12。
MAMの調節は、がん、代謝障害、神経変性疾患など、さまざまな疾患に影響を与える可能性のある新たな研究分野です18。多くのMAMモジュレーターが利用可能であるにもかかわらず、MAMの構造的多様性が創薬の標的となる非常に複雑なシステムであるため、MAMを不安定化し、ADの病理を低下させる能力をテストするための主要な試みはこれまで行われていませんでした。しかし、新たに開発された構造系薬理学は、創薬標的の特異的な性質やその環境を考慮した18,19ことで、この困難を克服し、ADのMAMやMAM関連タンパク質を標的とした高薬理学を開発できるはずである。しかし、MAM安定化の効果的な変調器を探すためには、MAM安定化の程度を正確に定量化する方法が必要です。電子顕微鏡(EM)や超解像顕微鏡などの従来の手法では、MAMの安定化を決定するには限界があります。これらの課題を克服するには、生細胞におけるMAM安定化を定量的に測定できる、よりダイナミックな新規イメージング技術や生化学的アッセイの開発が必要になる可能性があります。初代ニューロンの集束イオンビーム走査型電子顕微鏡(FIB-SEM)により、ERはミトコンドリアの周囲にネットワークを形成する傾向があり、ミトコンドリアの運動性を制限する可能性が高いことが明らかになった20,21。ミトコンドリア輸送システムの混乱は、逆行性、順行性、またはその両方であり、シナプスおよびニューロン機能に大きな影響を与えました22。したがって、MAM安定化を定量的に測定するための指標としてここで説明するER結合ミトコンドリアの軸索速度の新しい生細胞イメージングおよびキモグラフィーベースの分析は、MAM安定化閾値をAβ生成を増加させるのではなく、維持または低下する可能性のある閾値に切り替えることができるMAMモジュレーターの同定を容易にします。
プロトコル
ADニューラルカルチャーモデル: 本研究では、ヒト神経前駆細胞ReN細胞[ナイーブReN(Millipore)]またはアミロイド前駆体タンパク質(APP遺伝子)の家族性AD(fAD)変異を発現するReN細胞(APPSwe/Lon)、ReN GA細胞に由来するニューロンを使用しました。ReN-GAの3次元(3D)培養系は、ADの病理、すなわちAβオリゴマー駆動型神経原線維変化(NFT)を再現する23,24。ナイーブReN細胞は市販されています。ReN GA系統は、マサチューセッツ総合病院(MGH)の准教授であるDoo Y. Kim博士から入手しました23,24,25。
発現プラスミド:RFPと直接結合したUbc 6(283-303)タンパク質のAKP1(34-63)およびER標的配列(Mito-RFP-ERはMAM 1Xと表記)または9アミノ酸リンカー(Mito-9X-RFP-ERはMAM 9Xと表記)を含み、それぞれ6 nm±1 nmまたは24 nm + 3 nmのギャップ幅15,26のMAMを安定化するように設計されています(図1A)。
1.エレクトロポレーション
- MAM安定剤による細胞へのトランスフェクション(1-2時間)
注:ヌクレオフェクターキットの製造元が説明したプロトコルに従ってください(資料表).開始する前に、1% マトリゲルを含むDMEM/F12(以下、基底膜マトリックス[BMM])でプレコートした6ウェルガラス底培養皿を準備します。2 mLのBMMを使用して各ウェルをコーティングします。BMM、培地、ピペットチップ、およびピペットは、BMMが固まらないように、混合する前にすべて予冷する必要があります。- BMM混合物を吸引します。各ウェルに2 mLの室温(RT)DMEM/F12と交換します。
- Nucleofector と Supplement 1 を 4.5:1 の比率で組み合わせます (82 μL:18 μL の Nucleofector/100 μL の溶液に対する Supplement の比率)。必要なトランスフェクションごとに、100 μLのNucleofectorが必要です。
- ボルテックスDNA。次に、トランスフェクションごとに1 mLの微量遠心チューブに1〜5 μgのDNA(処理あたり)を加えます。
- アカスターゼを使用して、健康なReN-GA細胞のプレートを回収します。
注:100 mm組織培養皿内の増殖培地(表1)中の細胞をプレートし、コンフルエント度が70〜80%に達するまで待ちます。- 既存の培地を吸引し、10mLのPBSで1回洗浄します。
- 1 mLのアクターゼを直接細胞に加え、37°Cで5〜10分間インキュベートします。
- 皿の側面を軽くたたいて、皿から細胞を取り除きます。顕微鏡で観察して、細胞がほぐれて自由に流れていることを確認します。
- 10 mLのDMEM/F12でアクターゼを中和し、清潔な15 mLチューブに移します。
- 必要な数の細胞を274 x g で5分間遠心分離し、上清を吸引して死んだ細胞を取り除きます。次に、ペレットを10 mLのDMEM/F12に再懸濁します。
- セルをカウントして密度を決定します。
注:この研究では、自動セルカウンターを使用して細胞の数をカウントしました。エレクトロポレーションごとに、3-5 X 106 細胞が必要です(例えば、5回の治療には15-25 X 106 細胞が必要です)。- 懸濁した細胞10μLを取り出し、細胞計数チャンバーのA面に添加します。次に、B面に10μLを加えます。
注意: ピペットを横に傾けて、セル懸濁液を追加します。ピペットのプランジャーの最初のストップを超えて押し込まないようにして、気泡を避けてください。 - 充填された計数チャンバーをセルカウンターに持っていき、側面Aを前面のメインスロットに挿入します。
- Measureを押した後、mLあたりの細胞数をメモします。
- チャンバーを取り出し、サイドBに反転して、手順1.1.5.2-1.1.5.3を繰り返します。
- これら2つの数値を合計し、次に4で割り(トリパンブルーを使用しない場合)、次にその数値に細胞が懸濁している液体のミリリットルを掛けて、懸濁液中の細胞の総数を求めます。
- 懸濁した細胞10μLを取り出し、細胞計数チャンバーのA面に添加します。次に、B面に10μLを加えます。
- 必要な数の細胞を274 x g で5分間遠心分離し、上清を吸引します。
- エレクトロポレーションごとにペレットを100 μLのNucleofector Mixに再懸濁します。(例:5回の治療で500μL)。
注:細胞をNucleofetor溶液に15分以上放置すると、細胞の生存率と全体的な有効性が低下する可能性があります。 - DNAを含むチューブの1つに細胞懸濁液100μLを加え、ピペッティングで混合します。
- DNA混合物をエレクトロポレーションキュベットに移し、付属の蓋で密封します。気泡が発生しないように、キュベットを下向きに傾け、ゆっくりとピペットで動かします。
- 使用するデバイスに適したNucleofectorプログラムを選択します。この試験で使用したデバイスには、トランスフェクション用のプログラム A-033 を使用してください。最適化するには、5つのNucleofetorプログラムすべてを試して、各細胞タイプに最も適したプログラムを決定します。
注:エレクトロポレーションが成功したことを確認するのは、混合物の上部に見える泡です。 - すぐに、付属の滅菌ピペットを使用して、プレフィルド6ウェルプレートから~500μLのDMEMをキュベットに加えます。一度穏やかに混合し、エレクトロポレーションした細胞と培地を対応するウェルに移します。
注:エレクトロポレーション後、細胞は非常に敏感であるため、培地を迅速に移し、慎重にピペッティングすることが不可欠です。 - 残りのすべてのDNA処理について、手順1.1.8〜1.1.11を繰り返します。
- 細胞をCO2 (5%)の存在下で37°Cで一晩インキュベートします。
- 翌日、培地を新鮮な分化培地(表2)と交換し、細胞を10日間分化させます。2〜3日ごとに、新しいメディアと交換します。
2. ライブセルイメージング
- 生細胞顕微鏡用細胞の調製 (30分)
- 生細胞チャンバーの準備(細胞をチャンバーに移動する前)
- CO2タンクと加湿器がチャンバーに取り付けられ、バルブが開いており、タンクが満たされていることを確認します。
- 温度を37°C、COを2 〜5%、湿度を95%に設定します。(これは、レベルが平衡化するのに時間がかかる場合があります)。
- 細胞を含む6ウェルプレートをチャンバーに置き、細胞が見えるまで顕微鏡の焦点を調整します。
- レーザーをオンにします(材料の表)。
- RFPシグナルを捕捉するには、594 nmレーザーを使用して蛍光色素を励起し、570〜640 nmの発光を使用します。GFPの場合、励起には488nmのレーザーを使用し、発光には510〜540nmのレーザーを使用します。
- 内蔵の蛍光フィルターを使用して、バックグラウンドシグナルが消散するまで信号強度を調整します(ほぼ黒く点灯します)。
- 軸索のライブビデオのキャプチャ(合計10時間から15時間)
注:Nikon C2 Eclipse Ti2倒立共焦点顕微鏡を使用して、NIS Element ARソフトウェアを使用して蛍光画像を撮影しました。512ピクセルの解像度で60倍の倍率を使用し、1秒あたり1フレームでビデオを3分間撮影すると、よりクリーンなキモグラフが生成されました。- RFPバイオセンサーを発現する細胞を見つけます。ビデオを撮る前に、後で参照できるようにセルの画像をエクスポートします。
- 軸索の周りに合うようにスキャン領域を切り抜きます。スキャン領域を小さくすることで、顕微鏡の処理時間が短縮され、キモグラフィーの生成がはるかに容易になります。
- ソフトウェアがよりスムーズに実行されるように、すべてのレーザーをオフにします。すべてのレーザーをアクティブにしてプログラムを実行すると、すべてのフレームがキャプチャされるわけではないことに注意してください。
注:体細胞からの赤色蛍光シグナルは、軸索よりもはるかに明るいです。したがって、体細胞は、軸索の全体的な信号強度を増加させるために除外されます。 [Time Measurement ]タブに移動します。 - 間隔を 1 フレーム/秒に設定し、全体の時間を 181 秒に設定します。
- 「実行」をクリックします。
- このファイルを .nds ファイル (適切にラベル付けされたファイル) として保存し、MAM スタビライザー (MAM 1X または MAM 9X) ごとにこのプロセスを ~10 回繰り返します。
注意: レーザーの強度により、スキャンが長すぎるとRFP信号に顕著な漂白効果が生じることがありました。ビデオをキャプチャするために迅速に作業することは、考慮することが重要です。
3. 後処理(7日間)
注:輸送を分析し、キモグラフを生成するために、Fiji ImageJマクロが使用されました。0.1 mm/s未満の移動量を持つ小胞は静止していると分類されました。粒子の移動頻度は、特定の方向(順行性、逆行性)に移動する粒子の数、または移動しない(静止する)粒子の数を、キモグラフで分析された粒子の総数で割ることによって計算されました。各小胞が一時停止または移動に費やした時間は、分析された各ニューロンのすべての小胞について、各条件で費やされた時間の割合を平均することによって計算されました。速度とランレングスの度数分布は、各実験条件の移動小胞のみを使用して計算しました。分析は、100 mmの軸索路で3分間行われました。
- キモグラフの生成
- Fiji ImageJで.ndsファイルを開きます。
- [画像]タブに移動し、[プロパティ]をクリックします。ピクセルとミクロンの比率を記録します。これは、後で計算するために必要になります。
- [ ファイル] > [Tiff として保存] をクリックして> ファイルを.tiffとしてそのフォルダーに保存します (マクロは、生成されたすべてのものをこのフォルダーに自動的に保存します)。
- Kymo マクロを ImageJ にドラッグします。コードは、Supplementary Coding File 1 で提供されています。「実行」をクリックします。
- [OK] をヒットしません。 MAX_raw ウィンドウに切り替えます。軸索に沿って手動で左クリックし、ダブルクリックしてトラッキングを終了します。
注:軸索終末のように体細胞から軸索を追跡してください。このようにして、順行性と逆行性の計算は正確になります。 - ROIマネージャーでコマンドTまたはAddを押します。
- 次に、[ OK]をクリックします。キモグラフは、以前に作成したフォルダに生成されます。(X軸はミクロン単位の軸索長、Y軸は秒単位の時間です)。
- キモグラフの追跡
- キモグラフをFiji Image Jにドラッグします(以前に作成したフォルダに自動的に配置されます)。
- Track マクロを画像 J にドラッグします。
- 次のプロンプトで、[OK]をクリックします。前から作業を再開する場合は、続行する番号ポイントを入力してからOKを押してください。
- [選択] パネルの [矩形] ツールを使用して、高さ 60 ピクセル、直径が常に 100 μm のキモグラフの中央付近の領域を選択します。これを行うには、100 を手順 3.1.2 で記録したピクセルとマイクロの比率で割ります。
- 領域を選択したら、 Ctrl T を押して i t を ROI に追加し、作業中のフォルダに ROI を保存します (ROI メニューの [その他] を押してから [保存] をクリックします)。
注: Ctrl + Shift + I を押して選択した領域を反転すると、追跡しやすくなります。 - 個々の小胞の動きを追跡するには、 Ctrl キーを押しながら上から下に左クリックします。
- これを行うには、 Ctrl キーを押しながら右クリックすると、軸索が追跡されている場所を示すウィンドウがポップアップ表示されます。これが良さそうに見える場合は、[ OK]を押します。
- 続行して別の小胞を選択するには、[ はい]をクリックし、手順3.2.6〜3.2.8のプロセスを繰り返します。目に見えるすべての小胞が追跡されたら、 Noを押します。これにより、ハンド トラッキング オーバーレイが同じフォルダーに自動的に保存されます。
- キモグラフデータの測定
- 新しいフォルダを作成します。生成されたすべてのテキストファイルをフォルダにドラッグします。
- キモグラフファイルを開き、寸法を記録します。
- Measure マクロを画像 J にドラッグします。
- 以前に記録したミクロンとピクセルの比率を PixelScale に入力します。
- [制限速度] を 0.1 に変更します。(この制限を下回る小胞は静止していると見なされます)。
- 以前に記録したキモグラフの次元を入力します。作成したテキストフォルダにサマリーファイルが自動的に生成されます。各サマリーフィルには、移動時間 (%)、全体の速度 (μm/s)、合計距離、平均移動セグメント、停止回数、逆転回数が含まれます。キモグラフデータの生成、追跡、および測定のためのコードは、 Supplementary Coding File 1として提供されています。
結果
生細胞イメージングおよびキモグラフィー分析を行い、MAM 1XまたはMAM 9Xで安定化されたタイト(6 nm ± 1 nm)またはルース(24 nm± 3 nm)の接触幅を、少なくとも500 nmの長さの各ReN GA(AD)ニューロンまたはReN(ナイーブ)ニューロンの最長ニューロンプロセスで、それぞれタイト(6 nm 1 nm)またはルース(24 nm 3 nm)の接触幅で標識した遊離ミトコンドリアの運動性を測定しました。 これを軸索と見なします(図1および図2)。移動の頻度(全体、逆行性、および前行性)は、移動または静止したRFP標識点(MAM)の数をキモグラフの総数で割ることによって計算されました(図1A-E)。MAM 1X標識ER結合ミトコンドリアの全体的な軸索速度は、Mito-RFP標識ERフリーミトコンドリアまたはMAM 9X標識ER結合ミトコンドリアと比較して、~50%劇的に低下しました(図1B)。定量分析により、MAM 1X安定化ER結合ミトコンドリアの全体的な動きと逆行性運動と、遊離ミトコンドリア(Mito-RFP)またはMAM 9X安定化ER結合ミトコンドリアとの間に劇的な違いがあることも明らかになりました。ERフリーミトコンドリア(Mito-RFP)の53.82%±3.3%が可動性であった一方で、MAM 1X標識ER結合ミトコンドリアは26.6%±3.4%にとどまり、MAMの安定化は、ERに結合していない、または緩く結合したミトコンドリアと比較して、ERと密接に関連するミトコンドリアの全体的な軸索移動性を有意に低下させたことを示唆しています(MAM 9Xの44.79%±2.6%に対し、Mito RFPの53.82%±3.3%)。 それぞれ)(図1C)。一貫して、MAM 1X標識ER結合ミトコンドリアの逆行性および順行性運動は、MAM 9X標識または遊離ミトコンドリア(Mito-RFP)と比較して有意に低かった(逆行性:MAM 1X±12.33%±2.55%対Mito RFPの25.78%2.31%、前向性:MAM 1Xの14.27%±2.81%対Mito RFPの28.04%±2.48%)(図1DおよびE)。表3は、遊離ミトコンドリア、またはERに密接に結合または緩く結合しているミトコンドリアの正確な軸索速度を示しています。これらの値は、タイトなMAMと緩いMAMの間のMAMの安定化の程度を評価するための注目すべき定量的手段として使用でき、Ab生成の減少につながります。ナイーブReN細胞のタイトMAMとルーズMAMの安定化によるミトコンドリア軸索輸送速度は、ReN GAニューロンで観察された輸送パターンを反映していました(図1F-G)。ナイーブなReNニューロンとAPPSwe/Lonを発現するReN GA ADニューロンとの間の一貫した結果は、軸索輸送への影響が主にMAM安定化の状態に起因することを示唆しています。これは、APPSwe/Lonの存在または結果として生じるAβ産生とは無関係です。
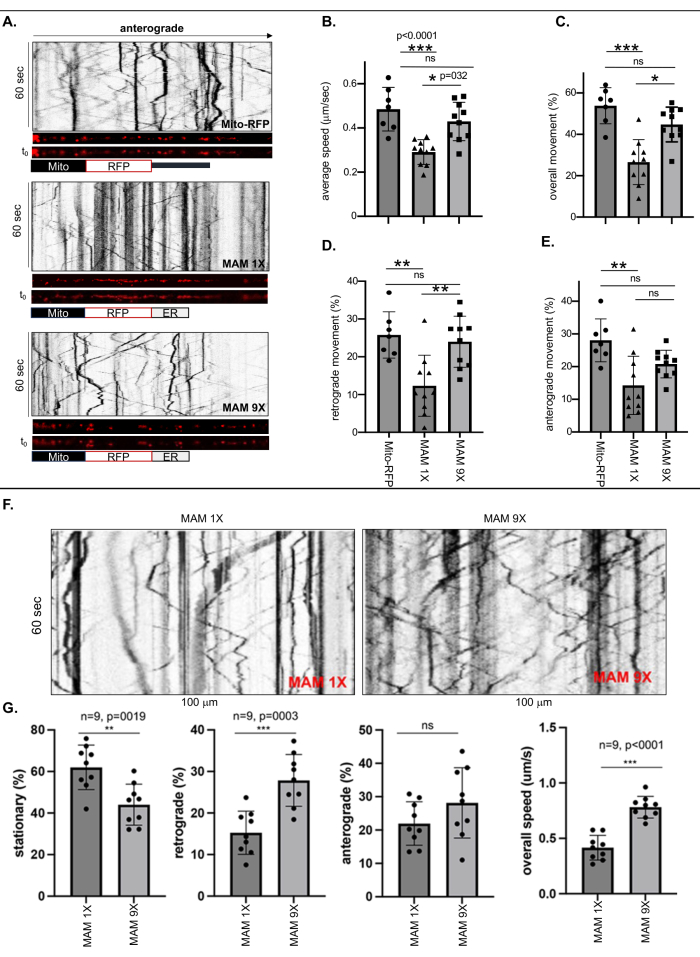
図1:MAM 1XによるMAMの安定化は、分化したReN GA細胞とナイーブReN細胞の軸索におけるER結合ミトコンドリアの平均速度と動き(全体的、逆行性、および前向性)を減少させた。 (A)遊離ミトコンドリアを表すRFP標識点の代表的なキモグラフ(Mito-RFP)またはMAM 1X(タイトMAM、6nm±1nmの接触幅)またはMAM 9X(ルーズMAM、24nm±3nmの接触幅)で安定化された軸索(~100nm)。(B-E)10日間分化したRen-GA細胞の軸索内のMito-RFP、MAM 1X、またはMAM 9Xの(B)平均速度と動き[(C)全体、(D)逆行性、および(E)前向性]の定量的分析。n>7;二元配置分散分析を実行しました。*p < 0.05、**p < 0.001。3つの独立した実験の代表。(F)10日間分化したナイーブReN細胞の軸索内のMAM 1XまたはMAM 9Xの動きの代表的なキモグラフ。(G)ナイーブReN細胞の軸索内のMAM 1XまたはMAM 9X安定化MAMの運動率(静止、逆行性、および順行性)および全体的な速度(マイクロメートル/秒;μm/s)の定量的分析。n = 9;二元配置分散分析を実行しました。p < 0.0001 です。この図は、Zellmer et al.17の許可を得て改作したものです。この図の拡大版を表示するには、ここをクリックしてください。

図2:MAM 1XまたはMAM 9Xを発現するナイーブReN細胞の軸索の生細胞画像。 10日間分化したGFP発現ReN細胞の長100μm軸索内でMAM 1XまたはMAM 9Xによって安定化されたMAMの動きを示す代表的な生細胞ビデオ画像。n > 重複実験から 10 枚の画像。矢印は順行性輸送を示しています。スケールバー:100μm。 この図の拡大版を表示するには、ここをクリックしてください。
| 試薬 | 最終濃度 | 量 |
| L-グルタミンを含むDMEM | 該当なし | 500ミリリットル |
| ヘパリン | 2 μg/mL | 0.5ミリリットル |
| B27 さん | 1倍 | 10ミリリットル |
| bFGFの | 20 ng/mL | 0.4ミリリットル |
| EGFの | 20 ng/mL | 0.5ミリリットル |
| ペニシリン/ストレプトマイシン | 100単位/mL | 5ミリリットル |
| トータル | 516.4ミリリットル | |
| ペニシリン/ストレプトマイシンを追加する前にメディアをろ過します。.4°Cで最大1か月間保管してください。 | ||
表1:拡張メディアの構成。
| 試薬 | 最終濃度 | 量 |
| L-グルタミンを含むDMEM | 該当なし | 500ミリリットル |
| ヘパリン | 2 μg/mL | 0.5ミリリットル |
| B27 さん | 1倍 | 10ミリリットル |
| ペニシリオン/ストレプトマイシン | 100単位/mL | 5ミリリットル |
| トータル | 515.5ミリリットル | |
| ペニシリン/ストレプトマイシンを追加する前にメディアをろ過します。.4°Cで最大1か月間保存 | ||
表2:分化媒体の組成。
| ReN GA | ReN(ナイーブ) | ReN GA (3D) | |||||
| 全体 (%) | 逆行性 (%) | 前行性 (%) | 平均速度(mm / s) | AB40 (pM) | AB42 (pM) | ||
| 水戸-RFP | 53.82±3.3% | 25.78±2.31% | 28.04±2.48% | 0.66 ± 0.03 | 0.69 ± 0.07 | 241.7±26.74 | 13.77 ± 1.52 |
| MAMの1X | 26.6 ± 3.4% *** | 12.33 ± 2.5% *** | 14.27 ± 2.81% *** | 0.3 ± 0.02*** | 0.43 ± 0.04*** | 377.2 ± 76.87* | 26.62±3.86* |
| MAMの9X | 44.79 ± 2.6% ns | 23.99±2.17%ns | 20.80 ± 1.33%ns | 0.59 ± 0.02 ns | 0.62 ± 0.02 ns | 158.8±3.27* | 17.01±2.02* |
表3:定量分析。 Mito-RFP、MAM 9X、およびMAM 1Xの平均速度(速度)および軸索運動(全体、逆行性、および前行性)の生細胞イメージングおよびキモグラフィーベースの定量分析。二元配置ANOVAは、軸索の速度または動き(%)に対して実行されました。n = 9 です。Aβについては、通常の一元配置ANOVAを行った。n = 3、3つの独立した実験。有意性は、トランスフェクションされていない(コントロールされた)ReN GA細胞に対して測定されます。**p < 0.0001;*p < 0.05;有意ではない (ns)。この表は、Zellmer et al.17の許可を得て改作したものです。
補足コーディングファイル1: キモグラフデータを生成、追跡、測定するためのコード。 このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
シグマ-1受容体(S1R)の阻害は、ニューロンプロセスにおけるMAM安定化をダウンレギュレートし、アミロイド前駆体タンパク質[APP]遺伝子(ReN GA)の家族性AD[FAD]変異を発現するヒト神経前駆細胞(ReN)細胞の3次元(3D)培養系からではなく、軸索からのAβ生成を劇的に減少(~90%)した)23,24,25,27.タイト(6 nm ± 1 nm)およびルーズ(24 nm ± 3 nm)のMAM15,26を安定化するように設計されたRFP標識構成的MAMスタビライザー(MAM 1XおよびMAM 9X)は、MAMの安定化を定量的に測定するための優れたツールです。どちらの安定剤も、3Dマトリックスで~10日間分化したReN GA細胞で同等かつ安定な発現を示すだけでなく、体細胞と軸索の離散的な点状にMAMを検出しました。最も重要なことは、FACSに富んだ3D ReN GAにおけるMAM 1Xの安定発現はAβ生成を有意に増加させたが、MAM 9X発現は効果がなかったことである17。また、18アミノ酸リンカーを含む構成的MAM安定剤(MAM 18X)がMAMを検出し、MAMを安定化させる効果も試験しました>25nm。MAM 1XまたはMAM 9Xとは異なり、MAM 18XはソマルMAMのみを標識しました。FACSに富んだMAM 18X発現ReN GAニューロンは、Aβ生成を減少させた17。これらの知見は、病原性(Aβ生成の増加)タイトMAMから非病原性(Aβ生成の維持または減少)緩いMAMまでのギャップ幅によって決定されるMAM安定性閾値の可能性を示唆しました.効果的なMAMモジュレーターとその最適な濃度を見つけることは、病原性および非病原性MAMからの閾値を超えるために必要な最適なMAM安定化を達成できるものであり、軸索またはニューロンのAβを低下させる顕著な治療法を明らかにします脳内の生成。
MAMモジュレーターの開発には、(1)MAMテザリングタンパク質を標的とするモジュレーター、(2)MAM常在タンパク質の発現レベルを変化させるモジュレーター、(3)MAM構造のモジュレーター、の3つの異なるアプローチが採用されています18。これらのアプローチにもかかわらず、MAM安定化の効果的なモジュレーターを見つける上での主な障害は、MAM安定化の程度を定量的に測定する方法がないことです。電子顕微鏡(EM)や超解像顕微鏡のような従来の手法では、MAMの安定化を評価するためのリアルタイムな変化を捉えたり、十分な詳細を提供したりすることに限界がある(28の文献で総説)。
ここで説明する方法は、この障害を克服し、MAM安定化とAβ産生の関係に関する重要な洞察を提供します。その結果、厚さ6 nm±1 nmのMAMは、全体の動きが26.6%±3.4%(表3)を示しており、Aβの生成と関連していることがわかりました。対照的に、厚さが24 nm±3 nmのMAMは、全体の動きが44.79%±2.6%(表3)であり、Aβの生成に影響を与えません。ミトコンドリア全体の動き(Mito-RFP)は53.82%±3.3%でした。MAMの厚さは通常6 nmから80 nmの間で変化することを考えると、これらの知見は、Aβ産生に関連するMAM安定化の上限と下限を示しています。したがって、この方法は、MAM安定化のための変調器の同定と最適化を導くことができます。目標は、MAMの全体的な動きを26.6%±3.4%から53.82%±3.3%に変更するか、平均速度を  0.4 μm/sから
0.4 μm/sから  0.7 μm/sに変更し(表3)、そのようなモジュレーターをAβ産生に対する潜在的な治療薬として位置付けることです。
0.7 μm/sに変更し(表3)、そのようなモジュレーターをAβ産生に対する潜在的な治療薬として位置付けることです。
長さが長くなる(0〜18アミノ酸)合成リンカーを含む構成的MAMモジュレーターの使用は、MAM安定化閾値を定量的に決定し、MAM安定化をAβ生成の増加とは対照的に維持または低下するものに切り替えるための強力な方法です。ただし、MAMモジュレーターの効率または有効性を評価するには、誘導性MAM安定剤が必要になります。YFP融合OMM標的配列のmAKAP1(34-63)およびCFP融合ER標的Sac1ホスファターゼ(521-587)をコードする発現プラスミドである誘導性フェルスター共鳴エネルギー移動/蛍光寿命イメージング顕微鏡(FRET/FLIM)ベースのMAM安定剤が利用可能です。さらに、構成的安定剤は生理学的MAMを表さない場合がありますが、一方、FRET/FLIM MAM安定剤は生理学的MAMを検出します。GFPが常在するERまたはミトコンドリアタンパク質ER−GFP(1−10)およびMito−GFP11のいずれかにつながれた2つの非蛍光フラグメントに分割される分割GFPプローブは、MAMの形成時に生体分子蛍光相補(BiFC)を生成する27も使用され得る。GFPフラグメントは自発的に組み立てられる傾向がありますが、BiFCは最も単純な読み出し、最も明瞭なシグナル、および最小限のノイズ関連分析を備えています。さらに、分割されたGFP間の相互作用は非常に可逆的であり28、したがって、それらの利点は欠点を上回り、BiFC法をMAM安定化のモジュレーターの同定に適したものにする。
謝辞
フィラデルフィアのトーマス・ジェファーソン大学教授であるGyörgy Hajnóczky博士には、RFP-Mito、MAM 1X、MAM 9X、およびMAM 18Xをコードする発現プラスミドを寛大に提供していただいたことに感謝します。特に、Brigham and Women's HospitalのシニアイメージングサイエンティストであるLai Ding博士には、キモグラフデータの生成、追跡、測定のためのコードの作成にご協力いただき、誠にありがとうございます。この研究は、Cure Alzheimer's Fund to RBおよびNIH助成金5R01NS045860-20からRETの支援を受けました。
資料
| Name | Company | Catalog Number | Comments |
| 6 Well Glass Bottom Plate | Cellvis | P06-1.5H-N | |
| B-27 Supplement (50X), serum free | Gibco/Thermo Fisher Scientific | 17504044 | |
| bFGF | R&D System | 233-FB | |
| BSA | Fisher Scientific | 501781532 | |
| Countess Cell Counting Chamber Slides | Invitrogen | C10283 | |
| DMEM/F12 with L-glutamine | Gibco/Thermo Fisher Scientific | 11320-033 | |
| EDTA | Life Technologies | 41116134 | |
| EGF | Sigma-Aldrich | 92090408 | |
| Falcon 6 Well Plates | VWR International | 41122107 | |
| GAPDH Polyclonal Antibody | Thermo Fisher Scientific | PA1-988 | |
| Gelatin | VWR International | 9000-70-8 | |
| Graphpad Prism N/A | Prism 9, version 9.5.0 | N/A | |
| Heparin | Sigma-Aldrich | H0200000 | |
| ImageJ Software | ImageJ 1.53a | N/A | |
| Matrigel Basement Membrane Matrix | Corning | 356234 | |
| mCherry Polyclonal Antibody | Invitrogen | PA5-34974 | |
| MS Excel | Microsoft Excel, version 2302 | N/A | |
| Multi-array electrochemiluminescence assay kit | Meso Scale Diagnostics (MSD) | K15200E-2 | V-PLEX Aβ Peptide Panel 1 (6E10) kit |
| NaCl | Fisher Scientific | 7647145 | |
| NuPAGE 4–12% Bis-Tris gel | Invitrogen | NP0321BOX | |
| Penicillin/Streptomycin/Amphotericin B | Lonza | 17-745E | |
| Photoshop | Adobe Photoshop CC 20.0.10 | N/A | |
| Rat Neuron Nucleofector Kit | Lonza | VPG-1003 | |
| StemPro Accutase | Gibco | A1110501 | |
| Tris-HCL, pH 7.6 | Boston BioProducts | 42000000 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween 20 | Fisher Scientific | 501657287 |
参考文献
- Giacomello, M., Pellegrini, L. The coming of age of the mitochondria-ER contact: a matter of thickness. Cell Death Differ. 23 (9), 1417-1427 (2016).
- Degechisa, S. T., Dabi, Y. T., Gizaw, S. T. The mitochondrial associated endoplasmic reticulum membranes: A platform for the pathogenesis of inflammation-mediated metabolic diseases. Immun Inflamm Dis. 10 (7), e647 (2022).
- Schon, E. A., Area-Gomez, E. Mitochondria-associated ER membranes in Alzheimer disease. Mol Cell Neurosci. 55, 26-36 (2013).
- Erpapazoglou, Z., Mouton-Liger, F., Corti, O. From dysfunctional endoplasmic reticulum-mitochondria coupling to neurodegeneration. Neurochem Int. 109, 171-183 (2017).
- Sala-Vila, A., et al. Interplay between hepatic mitochondria-associated membranes, lipid metabolism and caveolin-1 in mice. Sci Rep. 6, 27351 (2016).
- Fujimoto, M., Hayashi, T. New insights into the role of mitochondria-associated endoplasmic reticulum membrane. Int Rev Cell Mol Biol. 292, 73-117 (2011).
- Hung, V., et al. Proteomic mapping of cytosol-facing outer mitochondrial and ER membranes in living human cells by proximity biotinylation. eLife. 6, e24463 (2017).
- Area-Gomez, E., Schon, E. A. On the pathogenesis of Alzheimer's disease: The MAM hypothesis. FASEB J. 31 (3), 864-867 (2017).
- Rizzuto, R., et al. Close contacts with the endoplasmic reticulum as determinants of mitochondrial Ca2+ responses. Science. 280 (5370), 1763-1766 (1998).
- Sukhorukov, V. S., et al. Molecular mechanisms of interactions between mitochondria and the endoplasmic reticulum: A new look at how important cell functions are supported. Mol Biol. 56 (1), 59-71 (2022).
- Zhang, P., Konja, D., Zhang, Y., Wang, Y. Communications between Mitochondria and endoplasmic reticulum in the regulation of metabolic homeostasis. Cells. 10 (9), 2195 (2021).
- Ziegler, D. V., Martin, N., Bernard, D. Cellular senescence links mitochondria-ER contacts and aging. Commun Biol. 4 (1), 1323 (2021).
- Csordas, G., et al. Structural and functional features and significance of the physical linkage between ER and mitochondria. J Cell Biol. 174 (7), 915-921 (2006).
- Cieri, D., et al. SPLICS: a split green fluorescent protein-based contact site sensor for narrow and wide heterotypic organelle juxtaposition. Cell Death Differ. 25 (6), 1131-1145 (2018).
- Carpio, M. A., et al. BOK controls apoptosis by Ca(2+) transfer through ER-mitochondrial contact sites. Cell Rep. 34 (10), 108827 (2021).
- Prudent, J., et al. MAPL SUMOylation of Drp1 stabilizes an ER/mitochondrial platform required for cell death. Mol Cell. 59 (6), 941-955 (2015).
- Zellmer, J. C., Tarantino, M. B., et al. Stabilization of mitochondria-associated endoplasmic reticulum membranes regulates Abeta generation in a three-dimensional neural model of Alzheimer’s disease. Alzheimer’s Dement. , 1-20 (2024).
- Magalhaes Rebelo, A. P., et al. Chemical modulation of mitochondria-endoplasmic reticulum contact sites. Cells. 9 (7), 1637 (2020).
- Berger, S. I., Iyengar, R. Role of systems pharmacology in understanding drug adverse events. Wiley Interdiscip Rev Syst Biol Med. 3 (2), 129-135 (2011).
- Friedman, J. R., Webster, B. M., Mastronarde, D. N., Verhey, K. J., Voeltz, G. K. ER sliding dynamics and ER-mitochondrial contacts occur on acetylated microtubules. J Cell Biol. 190 (3), 363-375 (2010).
- Wu, Y., et al. Contacts between the endoplasmic reticulum and other membranes in neurons. Proc Natl Acad Sci U S A. 114 (24), E4859-E4867 (2017).
- Cagin, U., et al. Mitochondrial retrograde signaling regulates neuronal function. Proc Natl Acad Sci U S A. 112 (44), E6000-E6009 (2015).
- Choi, S. H., et al. A three-dimensional human neural cell culture model of Alzheimer's disease. Nature. 515 (7526), 274-278 (2014).
- Kim, Y. H., et al. A 3D human neural cell culture system for modeling Alzheimer's disease. Nat Protoc. 10 (7), 985-1006 (2015).
- Kwak, S. S., et al. Amyloid-beta42/40 ratio drives tau pathology in 3D human neural cell culture models of Alzheimer's disease. Nat Commun. 11 (1), 1377 (2020).
- Csordas, G., et al. Imaging interorganelle contacts and local calcium dynamics at the ER-mitochondrial interface. Mol Cell. 39 (1), 121-132 (2010).
- Bhattacharyya, R., et al. Axonal generation of amyloid-beta from palmitoylated APP in mitochondria-associated endoplasmic reticulum membranes. Cell Rep. 35 (7), 109134 (2021).
- Tebo, A. G., Gautier, A. A split fluorescent reporter with rapid and reversible complementation. Nat Commun. 10 (1), 2822 (2019).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved