Method Article
Isolamento de RNA Total de Pseudomonas aeruginosa em Biofilmes para Medição da Expressão Gênica
Neste Artigo
Resumo
Este protocolo apresenta um método para isolar RNA de biofilmes de Pseudomonas aeruginosa cultivados em lâminas de câmara para sequenciamento de alto rendimento.
Resumo
Pseudomonas aeruginosa é um patógeno bacteriano oportunista que causa infecções nas vias aéreas de pacientes com fibrose cística (FC). P. aeruginosa é conhecida por sua capacidade de formar biofilmes protegidos por uma matriz de exopolissacarídeos. Essa matriz permite que os microrganismos sejam mais resistentes a fatores externos, incluindo o tratamento com antibióticos. Um dos métodos mais comuns de crescimento de biofilme para pesquisa é em placas de microtitulação ou lâminas com câmara. A vantagem desses sistemas é que eles permitem o teste de múltiplas condições de crescimento, mas sua desvantagem é que eles produzem quantidades limitadas de biofilme para extração de RNA. O objetivo deste artigo é fornecer um protocolo detalhado, passo a passo, sobre como extrair RNA total de pequenas quantidades de biofilme de qualidade e quantidade suficientes para sequenciamento de alto rendimento. Este protocolo permite o estudo da expressão gênica dentro desses sistemas de biofilme.
Introdução
A maioria das infecções bacterianas crônicas, como infecções pulmonares em pacientes com fibrose cística (FC) e infecções relacionadas a próteses, são caracterizadas pelo crescimento de organismos dentro de biofilmes. Os biofilmes1 são comunidades de bactérias envoltas em uma matriz composta principalmente de polissacarídeos2. As bactérias dentro dos biofilmes podem ser de crescimento lento, metabolicamente dormentes e em condições anaeróbicas e hipóxicas. Os biofilmes são mais resistentes aos antibióticos devido a fatores como diminuição da penetração de antibióticos, aumento da expressão de bombas de efluxo de medicamentos e diminuição da divisão celular3. Por essas e outras razões, elas são de grande interesse de pesquisa.
Para estudar com precisão infecções persistentes, como infecções crônicas por Pseudomonas aeruginosa em pacientes com FC, as condições de crescimento observadas com a formação de biofilme precisam ser refletidas com precisão in vitro. Um método comum e de alto rendimento é cultivá-los em lâminas de câmara ou placas de microtitulação e monitorar a formação de biofilme por microscopia confocal4. Sabe-se que um regulador chave na transição de um estilo de vida bacteriano planctônico, ou flutuante, para o biofilme é o mensageiro secundário, cíclico-di-GMP5. O aumento dos níveis de di-GMP cíclico aumenta a expressão de genes específicos que promovem o crescimento do biofilme. Pequenos RNAs reguladores não codificantes e detecção de quorum também desempenham papéis importantes na regulação da formação de biofilme5. Medir a expressão gênica do biofilme por meio do sequenciamento do RNA bacteriano extraído pode ser um desafio. A P. aeruginosa, por exemplo, produz três exopolissacarídeos (Psl, Pel e alginato), que são produzidos em quantidades significativas em biofilmes 6,7. Esses polissacarídeos podem interferir na extração e purificação do RNA, levando a preparações impuras contendo baixos níveis de mRNA bacteriano8. Os kits de extração de RNA disponíveis comercialmente são capazes de produzir RNA de alta qualidade a partir de culturas bacterianas planctônicas, mas podem não funcionar tão bem com culturas de biofilme 9,10,11. Existem alguns kits comerciais de extração de RNA que afirmam funcionar para biofilmes, um dos quais usamos com esse método.
Neste manuscrito, descrevemos os procedimentos para o cultivo de biofilmes de P. aeruginosa em lâminas de câmara e extração de mRNA bacteriano para sequenciamento de alto rendimento12,13. Utilizando isolados clínicos coletados de amostras de escarro de pacientes com FC, demonstramos que esses métodos podem ser usados para isolados com características de crescimento variadas. Em comparação com publicações anteriores, este protocolo é descrito em detalhes para permitir melhor sucesso no estudo da expressão gênica do biofilme bacteriano 11,14,15,16.
Protocolo
O Comitê de Ética em Pesquisa (REB) é necessário para a coleta e processamento de amostras de escarro de seres humanos. Este estudo foi aprovado pelo Hospital for Sick Children (REB#1000019444). O Conselho de Ética em Pesquisa (REB) é obrigado a coletar e armazenar amostras de escarro de seres humanos. Esses estudos foram aprovados pelo Hospital for Sick Children REB#1000058579.
1. Formação de biofilme
- Cultive isolados de Pseudomonas aeruginosa obtidos a partir de amostras de escarro de pacientes com FC usadas neste estudo em placas de ágar Luria Broth (LB) em uma incubadora a 37 ° C durante a noite.
NOTA: A técnica adequada de estrias é importante para a obtenção de colônias bacterianas únicas. Estriar enquanto gira a placa diluirá as células bacterianas o suficiente para que colônias únicas possam crescer.- Listre as bactérias usando um loop de inoculação em zigue-zague na extremidade superior de uma placa LB fresca até que cerca de 1/4 da placa esteja coberta.
- Gire a placa cerca de 60°. Pegue um novo loop de inoculação e passe-o uma vez pela área listrada e em uma segunda área limpa da placa, repetindo o padrão em zigue-zague.
- Repita a etapa 1.1.2 com um novo laço em uma terceira área da placa. Recoloque a tampa e inverta a placa ao colocá-la na incubadora.
- Use uma alça de inoculação estéril para pegar uma única colônia bacteriana de uma placa de ágar (contendo uma única cepa bacteriana) e inocule um tubo de cultura cheio de 5 mL de meio LB estéril. Usando uma nova alça a cada vez, inocule dois tubos de cultura adicionais cheios de 5 mL de meio LB da mesma placa.
- Repita o mesmo procedimento de inoculação para as diferentes cepas bacterianas. Cultive as culturas durante a noite a 37 °C com agitação a 220 RPM.
NOTA: Cada placa de ágar contendo uma única cepa bacteriana é usada para inocular 3 tubos de cultura independentes. Os três tubos representam réplicas biológicas em triplicata para uma cepa e são tratados como amostras separadas. Isso é diferente das réplicas técnicas que implicariam a extração de RNA 3 vezes de um único tubo de cultura. - Prepare diluições de 1:100 das culturas noturnas transferindo 50 μL de uma cultura noturna para um novo tubo de cultura contendo 4,95 mL de novo meio LB.
- Cultive as culturas diluídas por mais 3 h a 37 ° C com agitação a 220 RPM ou até que o OD600 seja 0,1 ou superior. Meça as densidades celulares em um espectrofotômetro em OD600.
- Em um novo tubo de microfuga de 1,7 mL, ajuste o OD600 para 0,1 (fase logarítmica inicial) em um volume total de 1,5 mL com LB fresco.
- Misture delicadamente por inversão. Transfira 300 μL de cada cultura ajustada para 4 poços de uma lâmina de câmara de 8 poços para obter 2 amostras diferentes por lâmina (Figura 1).
- Colocar as lâminas, sem perturbações, numa incubadora a 37 °C durante a noite durante 24 h. Para evitar a evaporação, coloque as lâminas em cima de uma toalha de papel úmida em uma pequena bandeja de plástico.
2. Recuperação de biofilme
NOTA: Cada lâmina de vidro contém oito poços separados. Uma única amostra consiste em quatro poços com biofilmes que serão agrupados17. Este protocolo de extração é para 1 amostra (4 poços) onde os biofilmes são recuperados de 2 poços de cada vez. As extrações de RNA são realizadas usando um kit comercial de extração de RNA que inclui uma etapa de batimento de esferas e uma limpeza baseada em coluna, com modificações. Siga as instruções do fabricante para a preparação do reagente.
- Em uma capela de fluxo laminar, remova lentamente o meio de 2 dos 4 poços usando uma ponta de pipeta. Incline a lâmina em um ângulo de 45° e pipete o meio para fora do canto inferior dos poços para evitar o desprendimento dos biofilmes.
- Mantendo a lâmina inclinada, lave as células planctônicas pipetando suavemente 300 μL de água livre de RNase no canto inferior dos dois poços vazios. Remova a água pipetando-a suavemente, conforme descrito na etapa 2.1. Repita a etapa de lavagem removendo o máximo de água possível.
- Adicione 300 μL de um reagente de proteção de RNA (consulte a Tabela de Materiais) a cada um dos dois poços esvaziados com biofilmes em sua base. Coloque a lâmina da câmara em uma placa de vidro para evitar que os poços se quebrem e, em seguida, raspe os biofilmes nos 2 poços com uma pequena espátula de metal estéril sem nuclease para ressuspender as bactérias do biofilme. Deixe descansar até que os biofilmes sejam recuperados dos 2 poços intocados restantes da mesma amostra.
NOTA: A adição de um reagente de proteção de RNA garante a estabilidade das amostras de biofilme nos poços raspados à temperatura ambiente durante o processamento dos dois poços restantes da mesma amostra. O reagente de proteção de RNA lisa células e inativa nucleases e agentes infecciosos, resultando na preservação do RNA. - Para recuperar biofilmes dos 2 poços restantes para uma amostra (lembrete: uma amostra é composta por 4 poços), remova o meio LB dos 2 novos poços restantes da mesma forma descrita na etapa 2.1. Repita a etapa 2.2 para lavar as células planctônicas com água livre de RNase de ambos os novos poços, como antes.
- Volte para os primeiros 2 poços com biofilmes raspados em reagente de proteção, gerados no final da etapa 2.3, e pipete lentamente para misturar os 300 μL de biofilme ressuspenso de um poço raspado, tentando não criar muitas bolhas.
- Transfira todo o conteúdo do poço para um dos poços recém-lavados e esvaziados. Misture e transfira 300 μL de biofilme ressuspenso do segundo poço raspado no poço restante, recém-lavado e esvaziado.
NOTA: Em vez de adicionar um novo reagente de proteção de RNA aos 2 poços recém-lavados com biofilmes, transfira os biofilmes previamente raspados já no reagente de proteção para esses poços recém-lavados. Isso manterá o volume de amostra combinado baixo o suficiente para atender aos requisitos de entrada para o kit comercial de extração de RNA. Consulte a Figura 1 para obter um esquema.
- Transfira todo o conteúdo do poço para um dos poços recém-lavados e esvaziados. Misture e transfira 300 μL de biofilme ressuspenso do segundo poço raspado no poço restante, recém-lavado e esvaziado.
- Repita a raspagem dos biofilmes nos novos poços como na etapa 2.3, colocando a lâmina da câmara em uma placa de vidro e raspando os biofilmes nos 2 novos poços com uma pequena espátula de metal estéril sem nuclease para ressuspender as bactérias do biofilme.
- Combine todo o biofilme ressuspenso dos 2 novos poços em um único tubo de microcentrífuga de 1,5 mL sem RNase, de baixa ligação. Meça o volume, que deve ser ~ 500 - 600 μL no total.
NOTA: A suspensão combinada de biofilme deste segundo par de novos poços conterá todo o material de biofilme dos 4 poços originais de uma amostra.
3. Isolamento total de RNA e avaliação da qualidade
NOTA: A extração de RNA é realizada usando um kit comercial de extração de RNA que afirma funcionar em biofilmes. Os componentes individuais estão incluídos na Tabela de Materiais, se possível. Explicações sobre os mecanismos por trás de cada etapa de purificação são fornecidas quando possível.
- Adicione reagente de proteção de RNA suficiente para totalizar 750 μL no tubo. Transfira todo o volume para um tubo de lise de 2 mL contendo 0,1 e 0,5 mm (consulte a Tabela de Materiais). Bata por 2 1/2 min em um batedor de contas na velocidade máxima.
NOTA: A combinação de grânulos de alta densidade de 0,1 mm e 0,5 mm mais mistura de alta velocidade em um batedor de grânulos garante a homogeneização completa das paredes celulares microbianas. - Centrifugue a 16.000 x g por 1 min para pellet as contas. Transfira o sobrenadante para um novo tubo de microcentrífuga, minimizando a transferência de quaisquer esferas, o que facilitará a etapa 3.3. Meça o volume.
- Adicione um volume igual de tampão de lise de RNA (~ 450 μL) e misture bem. Transfira até 800 μL de amostra, evitando a transferência de quaisquer grânulos, para uma coluna de sílica em um tubo de coleta e centrifugue a 16.000 x g por 30 s. Salve o fluxo, pois ele contém o RNA enquanto o DNA está ligado à coluna.
NOTA: O tampão de lise de RNA contém tiocianato de guanidínio e o detergente N-lauroilsarcossina para lisar as células. O tiocianato de guanidínio é um agente caotrópico que também inativa nucleases e, na presença de sílica, encontrada na coluna de spin, promove a ligação do DNA à sílica18. A ausência de etanol permite a ligação preferencial do DNA e não do RNA à coluna de spin da sílica19. O objetivo da etapa 3.3 é ligar e remover o DNA genômico. Queremos reter o RNA, que está contido na porção de fluxo. - Se restar mais amostra, transfira a coluna para um novo tubo de coleta e recarregue com o restante da amostra. Centrifugue a 16.000 x g por 30 s. Manter o fluxo com o ARN e combinar com a primeira alíquota.
- Medir o volume de escoamento combinado e adicionar um volume igual de etanol a 100 % e homogeneizar bem. Transferir até 800 μL da solução para uma nova segunda coluna de centrifugação de sílica em um tubo de coleta e centrifugar a 16.000 x g por 30 s. Descarte o fluxo.
NOTA: A adição de etanol à solução de sal caotrópico contendo o RNA aumenta a ligação do RNA à coluna de rotação da sílica19. - Para soluções > 800 μL, recarregar a coluna de rotação e centrifugar até que toda a solução esteja girada. Descarte o fluxo após cada rodada.
- Adicione 400 μL de tampão de lavagem à coluna e centrifugue a 16.000 x g por 30 s para remover alguns dos sais. Descarte o fluxo.
- Preparar a mistura de reação DNase I de acordo com as instruções do fabricante e realizar o tratamento DNase na coluna para remover qualquer ADN residual.
- Ressuspenda a DNase I liofilizada em 275 μL de água livre de RNase para fazer uma solução de 1 U/μL. Misture por inversão suave.
- Combine 5 μL de DNase I diluída com 75 μL do tampão de digestão DNase fornecido. Misture delicadamente por inversão.
- Adicione 80 μL da solução preparada diretamente na matriz da coluna. Incubar à temperatura ambiente durante 20 min.
- Adicione 400 μL de tampão de preparação de RNA à coluna e centrifugue a 16.000 x g por 30 s. Descarte o fluxo.
- Adicione 700 μL de tampão de lavagem de RNA à coluna e repita a centrifugação. Descarte o fluxo.
- Adicione 400 μL de tampão de lavagem de RNA e centrifugue a coluna por 2 min para remover completamente qualquer tampão residual.
NOTA: Existem 2 etapas de lavagem diferentes para remover as impurezas da coluna. O tampão de preparação contém um sal caotrópico fraco misturado com etanol para remover proteínas residuais. Em seguida, o tampão de lavagem é usado para realizar lavagens com etanol para remover sais. Qualquer etanol restante deve ser removido para permitir a eluição eficiente do RNA19,20. - Transfira cuidadosamente a coluna para um novo tubo de microcentrífuga de baixa ligação.
- Adicione 50 μL de água livre de RNase diretamente à matriz da coluna e incube por 5 min. Centrifugue a 16.000 x g por 1 min para eluir o RNA.
- Para limpeza adicional para remover os inibidores de PCR, coloque um filtro inibidor de PCR em um novo tubo de coleta. Adicione 600 μL da solução de preparação do inibidor fornecida (consulte a Tabela de Materiais).
- Centrifugue a 8.000 x g por 3 min para lavar o filtro. Transfira o filtro lavado para um novo tubo de microcentrífuga de baixa ligação.
- Transfira o RNA eluído da etapa 3.13 para o filtro lavado e centrifugue a 16.000 x g por 3 min.
NOTA: O RNA pode ser usado imediatamente ou armazenado a -80 °C. - Determinar a concentração do RNA usando um sistema fluorométrico de alta sensibilidade21.
NOTA: Esses sistemas permitem a quantificação sensível de uma pequena quantidade de RNA em solução específica para o alvo de interesse.- Avalie a qualidade do RNA usando um sistema de eletroforese automatizado que pode fornecer um RIN (número de integridade do RNA), que é uma medida da qualidade do RNA22,23.
4. Depleção de RNA ribossômico e sequenciamento de alto rendimento
- Envie RNAs para o centro de genoma do Centro de Análise da Evolução e Função do Genoma (CAGEF) da Universidade de Toronto (Toronto, Canadá) (https://www.cagef.utoronto.ca/) para depleção de rRNA bacteriano e preparação da biblioteca direcional de RNA (ver Tabela de Materiais).
NOTA: A depleção de rRNA bacteriano tem como alvo os rRNAs 5S, 16S e 23S para remoção24. - Esgote os rRNAs usando um kit comercial de depleção bacteriana de rRNA. Siga o protocolo para entrada de 10 ng - 1 μg de RNA total intacto ou parcialmente degradado.
- Construa bibliotecas de sequenciamento de RNA usando um kit de preparação de biblioteca direcional de RNA com diferentes índices anexados a cada biblioteca.
- Agrupe quantidades equimolares de cada biblioteca de RNA e execute sequenciamento de alto rendimento com leituras de extremidade emparelhada de 100 bases25.
5. Avaliação da qualidade das leituras de sequenciamento
NOTA: Verifique a qualidade das leituras de sequenciamento usando o programa disponível gratuitamente, FastQC26, disponível através da plataforma gratuita e de código aberto, Galaxy27.
- Vá para https://usegalaxy.org/. Clique no menu Login ou Registrar e faça login com credenciais ou crie uma conta.
- Clique no link Carregar dados no canto superior esquerdo da página, no menu Ferramentas, e carregue os arquivos de sequenciamento fastq.gz. Aguarde até que os nomes dos arquivos apareçam no lado direito da página, no painel Histórico.
- Selecione Controle de qualidade FASTQ no menu Ferramentas para revelar uma lista de programas. Selecione FastQC, que preencherá o painel do meio da tela.
- Em Dados de leitura curta do seu histórico atual, selecione os arquivos de fastq.gz carregados no menu suspenso.
- Selecione Executar para executar o programa.
- Exiba os resultados no painel Histórico (Tabela 2).
NOTA: Para obter instruções mais detalhadas sobre como usar o Galaxy, visite a página de suporte em https://galaxyproject.org/support/.
6. Mapeamento de leituras de sequenciamento
NOTA: Listado está um pipeline básico para corte de adaptador e mapeamento de leitura para dados de RNA-seq. As sequências do adaptador são cortadas das leituras usando o Trimmomatic28. As leituras aparadas são mapeadas para o genoma de referência PAO1 de P. aeruginosa (NC_002516.2), obtido do NCBI (https://www.ncbi.nlm.nih.gov/)29usando BWA30 e Samtools31. Para simplificar, um par de leituras é chamado de PA_1.fq e PA_2.fq; O arquivo de leitura do adaptador a ser cortado é chamado de adapter.fa; e a sequência de referência PAO1 é chamada PAO1.fasta. Todas as ferramentas são de código aberto e executadas em um ambiente UNIX/LINUX. É altamente recomendável que você se familiarize com os fundamentos do UNIX/LINUX para executar esses comandos.
- Abra uma janela no UNIX/LINUX.
- Instale Java, Trimmomatic, BWA e Samtools.
- Navegue até a pasta onde o trimmomatic-0.39.jar de arquivos reside.
- Corte todas as sequências de adaptador das leituras digitando o comando:
Java -jar PA_1.fq PA_2.fq PA_1_paired.fq PA_1_unpaired.fq PA_2_paired.fq PA_2_unpaired.fq ILLUMINACLIP: adapters.fa
NOTA: Somente as sequências do adaptador são removidas. As leituras não foram cortadas para qualidade32. - Mova o arquivo de referência PAO1.fasta para a mesma pasta.
- Indexe a referência usando BWA com o comando:
Índice Bwa PA01.fasta - Mapeie as leituras emparelhadas para o genoma de referência digitando os 4 comandos a seguir. Digite cada comando após a conclusão do anterior.
Bwa -mem PA01.fasta PA_1_paired.fq PA2_2_paired.fq > PA_R1R2_map.mem.sam
Samtools view -S -b PA_R1R2_map.mem.sam > PA_R1R2.bam
Samtools sort PA_R1R2.bam -o PA_R1R2_sorted.bam
Índice Samtools PA288_Rep1_R1R2_sorted.bam - Exiba as estatísticas de mapeamento digitando o comando:
Samtools flagstat PA_R1R2_sorted.bam
NOTA: A3ª linha das estatísticas relata a proporção das leituras que mapeiam para o genoma de referência. - Calcule a profundidade média de leitura e a amplitude da cobertura com os 2 comandos33 a seguir, respectivamente:
Samtools Depth -A PA_R1R2_sorted.bam | awk '{c++; s+=$3}END{print s/c}'
Samtools Profundidade -A PA288_Rep1_R1R2_sorted.bam | awk '{c++; if($3>0) total+=1}END{print (total/c)*100}'
Resultados
A visão geral do método é mostrada na Figura 1. Anteriormente, usamos lâminas de câmara de 8 poços para cultivar biofilmes de P. aeruginosa e expô-los a antibióticos antes de examiná-los por microscopia confocal em diferentes momentos12,13. Este método pode ser usado para extrair RNA total diretamente de biofilmes cultivados neste sistema, a fim de estudar alterações na expressão gênica pós-tratamento. Este protocolo foi otimizado para P. aeruginosa, mas pode ser facilmente adaptado para outras espécies bacterianas.
Pode ser um desafio extrair uma quantidade suficiente de RNA de boa qualidade de pequenas quantidades de biofilme para depleção de rRNA e sequenciamento de alto rendimento. Usando este protocolo, o RNA total é extraído com sucesso de 17 diferentes isolados de biofilme de P. aeruginosa em triplicata, totalizando 51 amostras separadas. As quantidades de RNAs extraídos representando altos e baixos rendimentos são mostradas na Tabela 1. Os RNAs variam em concentração de 3,4 ng/μL (mais baixo) a 49,6 ng/μL (mais alto), com uma concentração média de 14 ng/μL e uma mediana de 13,7 ng/μL. Concentrações de RNA intactas abaixo de 10 ng/μL são consideradas amostras de baixa abundância para depleção de rRNA e sequenciamento de próxima geração, mas amostras de RNA de baixa abundância de biofilmes podem resultar em dados de sequenciamento de qualidade ainda pior em comparação com amostras mais concentradas34, 35,36,37. As qualidades dos RNAs são mostradas na Tabela 1 pelo RIN e na Figura 2 pelos eletroferogramas de RNA correspondentes para as amostras concentradas baixas (PA565-3) e altas (PA288-1); as duas amostras restantes (PA375-3 e PA921-1) são representativas da maioria das amostras. Como visto na Figura 2, o RNA extraído dessas quantidades de biofilme sempre contém algum RNA degradado, o que afeta seus valores de RIN. Portanto, a confirmação visual dos picos de rRNA procariótico 16S e 23S é usada para determinar a qualidade do RNA quando um RIN não é relatado. Usando esses critérios, todas as 51 amostras de RNA são escolhidas para prosseguir com a depleção e sequenciamento de rRNA, com a previsão de que a amostra de pior qualidade, PA565-3, pode falhar. Das amostras de RNA enviadas para sequenciamento, bibliotecas bem-sucedidas são geradas e sequenciadas para 49 amostras, com PA565-3 falhando, conforme previsto.
O número de leituras de sequenciamento geradas para a amostra de alta concentração com um bom RIN (PA288-1) e a amostra de menor concentração sem um RIN (PA375-3) estão listados na Tabela 2. As estatísticas básicas incluem o número total de leituras, a duração da leitura e o conteúdo do GC. As estatísticas resumidas para ambas as amostras mostram um alto número de leituras geradas e indicam que nenhuma é sinalizada como de baixa qualidade, sugerindo bons dados de sequenciamento. O número médio de leituras por amostra é de aproximadamente 48 milhões, o que é considerado um bom rendimento. Uma ferramenta padrão usada para avaliar a qualidade dos dados brutos de sequenciamento é o FastQC26. Este programa é usado para realizar verificações de controle de qualidade nos arquivos de sequenciamento brutos para determinar se a qualidade é suficiente para análises posteriores ou se há problemas ou vieses nos dados gerados pelo próprio sequenciador ou pelas bibliotecas de RNA de entrada. A Figura 3 apresenta as métricas de controle de qualidade para os dados de sequenciamento de PA288-1 e PA375-3, representando uma amostra de RNA de alta qualidade e uma amostra de RNA típica de qualidade inferior, respectivamente. Um dos gráficos mais informativos do FastQC é o gráfico de qualidade de sequência por base. Bons dados de sequenciamento mostrarão uma alta pontuação de qualidade mediana (>30) para cada posição em todas as leituras, com uma queda na pontuação de qualidade média ao longo da leitura. Uma pontuação de qualidade de 30 representa uma taxa de erro de 1 em 1000, correspondendo a uma precisão de chamada de base de 99,9%. A grande maioria das bases na Figura 3 tem pontuações médias de qualidade ≥ 35 em toda a duração da leitura para ambas as amostras, o que é indicativo de dados de sequenciamento de qualidade excepcionalmente boa. Isso fornece fortes evidências de que o protocolo de extração de RNA apresentado aqui é bem-sucedido.
Para mostrar que nosso método pode recuperar transcritos de P. aeruginosa, as leituras de sequenciamento de alta qualidade para PA288-1 e PA375-3 são mapeadas para o genoma de referência PAO1 de P. aeruginosa (NCBI NC_002516.2). As leituras são cortadas apenas para adaptador de sequenciamento e não para qualidade, mantendo todas as bases antes do mapeamento32. As estatísticas de mapeamento são apresentadas na Tabela 3. A porcentagem de leituras mapeadas é uma medida importante da precisão do sequenciamento. Simplificando, quanto mais leituras se alinharem à sua sequência de referência, melhor. Para PA288-1 e PA375-3, 84% e 91% das leituras, respectivamente, são mapeadas para o genoma de referência. A faixa esperada para o mapeamento de leituras padrão de RNA-seq fica entre 70 e 90%, portanto, esses valores são muito bons, especialmente quando bases de baixa qualidade não são removidas38. A profundidade média de leitura é um bom indicador do número médio de leituras que se alinham em cada posição base na sequência de referência. Quanto maior a profundidade em cada posição de base, mais precisa é a chamada de base em cada posição. É calculado dividindo a soma das profundidades de leitura mapeadas em cada posição no genoma de referência pelo número total de bases na referência. PA288-1 e PA375-3 têm profundidades médias de leitura de 400 ou mais, o que é bom para análise de expressão gênica a jusante39,40. A amplitude da cobertura informa a porcentagem do comprimento do genoma de referência que é coberto pelo sequenciamento41. As leituras alinhadas de PA288-1 e PA375-3 cobrem 96% do genoma de referência PAO1 de P. aeruginosa. Isso sugere que a maior parte do genoma de P. aeruginosa está representada nos dados de sequenciamento e não apenas em trechos curtos. As estatísticas de mapeamento para PA288-1 e PA375-3 mostram que este método pode recuperar transcritos que se alinham com boa cobertura e distribuição para o genoma de P. aeruginosa, apoiando ainda mais um protocolo de extração bem-sucedido.
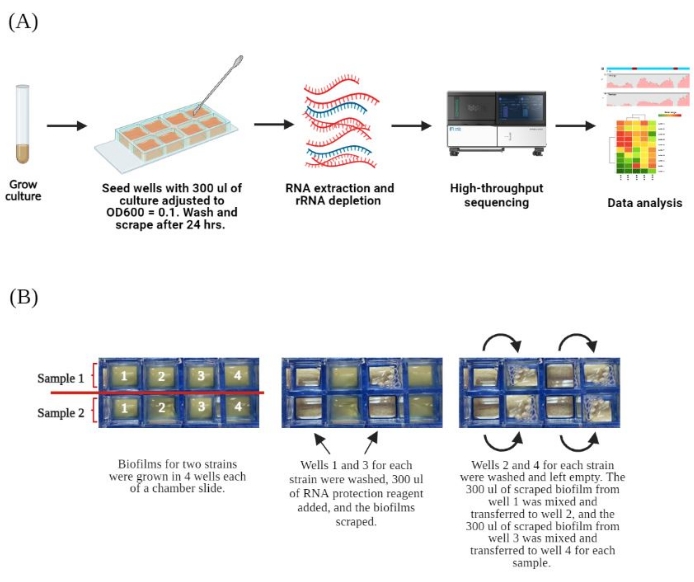
Figura 1. Visão geral do protocolo. Um. Esquema do fluxo de trabalho experimental. As culturas planctônicas foram cultivadas durante a noite a 37 °C, diluídas a 1:100 com meio fresco no dia seguinte e cultivadas por mais 3 h. As culturas foram ajustadas para uma DO600 de 0,1 e 300 μL inoculadas em 4 poços de lâmina de 8 câmaras para gerar biofilmes. Após 24 h, os biofilmes foram lavados para remoção das células planctônicas; O reagente de proteção de RNA foi adicionado e as células rasparam os poços. O RNA total foi extraído, depletado de RNAs ribossômicos e sequenciado. B. Fluxo de trabalho detalhado do crescimento e remoção de biofilme de uma lâmina de 8 câmaras. Duas linhagens foram cultivadas por lâmina na orientação mostrada à esquerda. A disposição de cada amostra em quatro poços da lâmina é mostrada. Para cada amostra independente, os poços 1 e 3 foram processados primeiro, terminando com 300 μL de células raspadas em reagente de proteção de RNA em cada poço. Um exemplo é representado pelo slide do meio. Em seguida, os poços 2 e 4 foram lavados e o líquido removido. As células ressuspensas dos poços 1 e 3 são transferidas para os poços 2 e 4, respectivamente, mostrados no slide à direita. Após a raspagem, as células ressuspensas nos poços 2 e 4 são combinadas em um único tubo de microcentrífuga. Esta figura foi criada com BioRender.com. Clique aqui para ver uma versão maior desta figura.

Figura 2. Exemplos de eletroferogramas de RNA de amostras de RNA extraídas de alta a baixa qualidade. Os picos ribossômicos 16S e 23S são rotulados na base de seus picos. O RNA degradado é representado por picos de pequeno porte, indicados pelas setas e uma linha de base acidentada acima de zero. Amostras de RNA de qualidade aceitável são mostradas nos painéis A, B e D. O painel C mostra uma amostra de RNA de má qualidade onde faltam os picos ribossômicos e a concentração era de abundância muito baixa, indicada pela escala no eixo Y. FU, unidades de fluorescência; NT, nucleotídeo. Clique aqui para ver uma versão maior desta figura.

Figura 3. Gráfico de qualidade de sequência FastQC Per Base para dados de sequência PA288-1 e PA375-3. Um. O gráfico de qualidade para a amostra de alta qualidade PA288-1. B. O gráfico de qualidade para a amostra típica PA375-3. Os gráficos mostram a pontuação de qualidade agregada para cada posição base para todas as leituras no arquivo. A linha azul representa a pontuação de qualidade média em cada posição base. A linha vermelha dentro da caixa amarela representa a pontuação de qualidade mediana em cada posição, e a caixa amarela mostra o intervalo de quartis paraos percentis 25a 75. Clique aqui para ver uma versão maior desta figura.
| Isolar | Qubit | RIN | Sequenciado |
| PA288-1 | 26 ng/μl | 7.5 | Sim |
| PA375-3 | 4,07 ng/μl | NA | Sim |
| PA565-3 | 3,4 ng/μl | NA | Não |
| PA921-1 | 9,11 ng/μl | 6.6 | Sim |
Tabela 1. Métricas de qualidade de RNAs extraídos de amostras representativas.
| PA288-1 | PA375-3 | |
| Total de leituras | 8,59,57,720 | 3,18,49,575 |
| Leituras sinalizadas como de baixa qualidade | 0 | 0 |
| Comprimento de leitura | 100 | 100 |
| %GC | 61 | 60 |
Tabela 2. PA288-1 e PA375-3 FastQC Resumo Estatísticas
| Isolar | % de leituras mapeadas para a referência | Profundidade média de leitura | Amplitude de cobertura |
| PA288-1 | 83.93% | 404 | 96.68% |
| PA375-3 | 91.2% | 578 | 96.97% |
Tabela 3. Estatísticas de mapeamento PA288-1 e PA375-3
Discussão
O RNA total é extraído com sucesso de 17 amostras diferentes de biofilme bacteriano em triplicata, resultando em um total de 51 amostras. As quarenta e nove bibliotecas de RNA são agrupadas e sequenciadas com sucesso. No geral, isso valida nossos critérios de qualidade com uma taxa de sucesso de 96%, embora mais da metade das amostras sejam consideradas de baixa abundância e de qualidade abaixo do ideal 34,35,36,37.
Significado
Este protocolo de extração de RNA é único em sua explicação detalhada para extrair RNA de quantidades limitadas de biofilme que foram cultivadas em lâminas de câmara. O crescimento de biofilmes em uma lâmina de câmara de 8 poços é um sistema útil para estudar os efeitos de fatores adicionados exogenamente aos biofilmes ou efeitos de interações microbianas na formação de biofilme por microscopia confocal 12,13,42. Com o objetivo de examinar a expressão de genes envolvidos na formação de biofilme neste sistema, apresentamos um método detalhado para extrair RNA intacto de qualidade e quantidade suficientes para sequenciamento de RNA. Outros estudos relataram extração bem-sucedida de RNA de quantidades limitadas de biofilme, mas a maioria cultiva biofilmes em uma área de superfície maior e geralmente por 48 h em vez de 24 h. Eles também carecem de detalhes suficientes para garantir o sucesso 4,15,16,43,44,46.
Além disso, este protocolo evita o uso de produtos químicos perigosos (como fenol) ou equipamentos especializados (como um sonicador). O protocolo clássico de extração de fenol-clorofórmio tiocianato de guanidínio47 não é usado para este sistema porque, embora produza aproximadamente 2x mais RNA em comparação com o kit comercial, em nossas mãos resulta consistentemente em RNA totalmente degradado, conforme avaliado em um sistema de eletroforese automatizado. Além disso, o uso de um kit de extração comercial resulta em um protocolo amigável que produz resultados consistentes 15,16,45.
Etapas críticas
Há uma série de etapas críticas neste protocolo que aumentam a probabilidade de extração de RNA que pode ser sequenciado com sucesso. Primeiro, é importante inocular pelo menos 4 poços da lâmina de câmara com a mesma cepa para obter quantidades suficientes de RNA para sequenciar. Os biofilmes dos 4 poços são agrupados para uma única extração, o que tem a vantagem adicional de reduzir a variabilidade na análise de expressão gênica a jusante17. A extração de RNA de menos de 4 poços geralmente leva a rendimentos muito baixos para serem detectados em um sistema fluorométrico de alta sensibilidade. A extração de RNA de 8 poços ou de uma lâmina inteira produzirá maiores quantidades de RNA de qualidade semelhante à obtida ao usar 4 poços, mas deve-se considerar se o tempo, esforço e custo adicionais para a extração valem o aumento do rendimento. A pipetagem de 300 μL de OD600 = 0,1 cultura diluída para semear cada poço em vez dos 200 μL padrão melhora a recuperação do material de biofilme raspado de cada poço. Lavar suavemente o biofilme duas vezes com água livre de nuclease antes de aplicar o reagente de proteção de RNA é fundamental para remover o maior número possível de células mortas e/ou planctônicas. O uso do reagente protetor de RNA é importante para evitar a degradação do RNA devido ao tempo necessário para processar cada poço. Além disso, em vez de raspar com pontas de pipeta, preferimos usar espátulas de metal com uma extremidade plana que entra em contato com uma área de superfície maior do que uma ponta de pipeta e que sejam pequenas o suficiente para caber em um poço. A raspagem usando uma ponta de pipeta de 1000 μL funcionará, mas é menos eficiente e leva mais tempo para raspar todo o biofilme em comparação com a espátula de metal. Em nossa experiência, o uso de espátulas de metal economiza tempo e esforço. Ao raspar, certifique-se de colocar a corrediça da câmara em cima da placa de vidro para evitar rachaduras no fundo dos poços. A sonicação não é eficaz, pois resulta consistentemente em RNA altamente degradado devido ao pequeno volume de amostra e muita produção de calor. Após a coleta do material de biofilme, a amostra é inserida em um kit comercial para extrações de RNA mais confiáveis e reprodutíveis. Finalmente, devido às fontes potenciais de variação neste método, é muito importante incluir a replicação experimental, além do agrupamento de amostras17,48. Este protocolo descreve a replicação biológica triplicata de cada amostra.
Limitações
Há uma variedade de técnicas usadas para cultivar biofilmes para estudo, a mais comum das quais é a formação em placas de microtitulação4. Estas placas estão disponíveis com diferentes tamanhos e números de poços. Os slides da câmara se enquadram nesta categoria. A capacidade de extrair RNA puro e intacto é importante para todos os sistemas de biofilme, mas esse sistema é limitado pelos baixos rendimentos de RNA. As concentrações são baixas o suficiente para que sua qualidade e quantidade sejam avaliadas por instrumentos Qubit e Bioanalyzer usando kits de RNA de alta sensibilidade. Uma nanogota também pode ser usada, mas tem sensibilidade mais limitada em comparação com o Qubit e não consegue distinguir o DNA contaminante do RNA49. Se possível, um nanodrop deve ser usado para obter proporções de pureza A260 / A280 e A260 / A230 . Essas proporções são úteis, especialmente porque a qualidade do RNA desse sistema de biofilme não é tão boa quanto a de grandes quantidades de material de partida. Isso provavelmente se deve ao baixo rendimento de RNA e à presença de células mortas nos biofilmes e/ou RNase que podem degradar o RNA. Outra limitação desse método é que ele não pode separar diferentes tipos de células dentro de um biofilme heterogêneo, mas só pode extrair as células como uma população inteira, potencialmente obscurecendo a expressão gênica em quaisquer subpopulações sub-representadas.
Aplicações potenciais
O protocolo aqui apresentado foi otimizado para biofilmes de P. aeruginosa a partir de isolados clínicos cultivados em lâminas de câmara. Este método pode ser usado para estudar mudanças na expressão gênica durante a formação do biofilme sob diferentes condições de crescimento observadas por microscopia confocal. O protocolo também pode ser otimizado para outras espécies bacterianas geradoras de biofilme. Com este método, também é possível extrair DNA genômico, pois um kit de extração dupla pode ser usado. Dessa forma, a expressão gênica do biofilme bacteriano em isolados clínicos causadores de infecções em pacientes pode ser estudada com maior precisão, levando a resultados que podem orientar futuras estratégias de tratamento.
Divulgações
Os autores não têm divulgações a declarar.
Agradecimentos
Contribuições dos autores: P.W., Y.Y. e V.W estiveram envolvidos na conceituação do estudo. K.G., L.J., A.M. e P.W. otimizaram os protocolos de laboratório. O financiamento para K.G. foi apoiado pelo subsídio do Programa de Colocação de Trabalho do Estudante através da BioTalent Canada.
Materiais
| Name | Company | Catalog Number | Comments |
| Agilent 2100 Bioanalyzer | Agilent | G2939BA | Automated electrophoresis of biomolecules |
| Agilent RNA 6000 pico kit | Agilent | 5067-1513 | High sensitivity RNA electrophoresis chip to generate a RIN |
| DNA/RNA Lysis Buffer | Zymo Research | D7001-1-50 | A guanidinium thiocyanate and N-Lauroylsarcosine-based lysis buffer sold as part of a nucleic acid purification kit |
| DNA/RNA Prep Buffer | Zymo Research | D7010-2-10 | A guanidine HCl and ethanol buffer used for purification of DNA and RNA |
| DNA/RNA Shield | Zymo Research | R1100-50 | DNA and RNA preservation/protection reagent |
| DNA/RNA Wash Buffer | Zymo Research | D7010-3-6 | A salt and ethanol buffer used for purification of DNA and RNA |
| DNBSEQ G-400RS | MGI | G-400RS | High throughput sequencer |
| MGIEasy RNA Directional Library Prep Set | MGI | 1000006386 | Generate libraries for MGI high-throughput sequencing platforms from total RNA. |
| Mini-Beadbeater-96 | BioSpec | 1001 | A high energy, high throughput cell disrupter |
| NEBNext rRNA Depletion Kit (bacteria) | New England Biolabs | E7850X | Efficient and specific depletion of bacterial rRNA (5S, 16S, 23S) |
| Nunc Lab-Tek II chamber slide system | Thermo Fisher Scientific | 154534 | 8-well chamber slide with removable wells |
| Qubit Fluorometer | Thermo Fisher Scientific | Q33238 | Fluorometer for DNA, RNA and proteins |
| Qubit RNA HS Assay Kit | Thermo Fisher Scientific | Q32852 | High sensitivity fluorometric assay to measure RNA concentration |
| Spin-Away Filters | Zymo Research | C1006-50-F | Silica-based spin column primarily used to bind or remove genomic DNA |
| Sterile inoculation loops, 1 uL | Sarstedt | 86.1567.050 | Sterile, disposable inoculation loops for manipulation of microorganisms |
| ZR BashingBead Lysis tubes | Zymo Research | S6003-50 | 2 mL tubes containing 0.1 and 0.5 mm bead lysis matrix for homogenizing biological samples |
| Zymo Spin IIICG Columns | Zymo Research | C1006-50-G | Silica-based spin column for purification of DNA and RNA |
| Zymo-Spin III-HRC Filters | Zymo Research | C1058-50 | Remove inhibitors such as polyphenolic compounds, humic/fulvic acids, tannins, melanin, etc. |
| Zymobiomics DNA/RNA Miniprep kit | Zymo Research | R2002 | DNA and RNA dual extraction kit |
| Zymobiomics HRC Prep solution | Zymo Research | D4300-7-30 | To be used with Zymo-Spin III-HRC Filters to remove PCR inhibitors |
Referências
- Beaudoin, T., Waters, V. Infections With Biofilm Formation: Selection of Antimicrobials and Role of Prolonged Antibiotic Therapy. The Pediatric Infectious Disease Journal. 35 (6), 695-697 (2016).
- Bjarnsholt, T., et al. The in vivo biofilm. Trends in Microbiology. 21 (9), 466-474 (2013).
- Folsom, J. P., et al. Physiology of Pseudomonas aeruginosa in biofilms as revealed by transcriptome analysis. BMC Microbiology. 10, 294(2010).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43 (3), 313-351 (2017).
- Bjarnsholt, T., Ciofu, O., Molin, S., Givskov, M., Hoiby, N. Applying insights from biofilm biology to drug development - can a new approach be developed. Nature Reviews Drug Discovery. 12 (10), 791-808 (2013).
- Colvin, K. M., et al. The Pel and Psl polysaccharides provide Pseudomonas aeruginosa structural redundancy within the biofilm matrix. Environmental Microbiology. 14 (8), 1913-1928 (2012).
- Hentzer, M., et al. Alginate overproduction affects Pseudomonas aeruginosa biofilm structure and function. Journal of Bacteriology. 183 (18), 5395-5401 (2001).
- Cury, J. A., Koo, H. Extraction and purification of total RNA from Streptococcus mutans biofilms. Analytical Biochemistry. 365 (2), 208-214 (2007).
- Francavilla, M., et al. Extraction, characterization and in vivo neuromodulatory activity of phytosterols from microalga Dunaliella tertiolecta. Current Medicinal Chemistry. 19 (18), 3058-3067 (2012).
- Atshan, S. S., et al. Improved method for the isolation of RNA from bacteria refractory to disruption, including S. aureus producing biofilm. Gene. 494 (2), 219-224 (2012).
- Franca, A., Melo, L. D., Cerca, N. Comparison of RNA extraction methods from biofilm samples of Staphylococcus epidermidis. BMC Research Notes. 4, 572(2011).
- Jurcisek, J. A., Dickson, A. C., Bruggeman, M. E., Bakaletz, L. O. In vitro biofilm formation in an 8-well chamber slide. The Journal of Visusalized Experiments. (47), e2481(2011).
- Beaudoin, T., Kennedy, S., Yau, Y., Waters, V. Visualizing the effects of sputum on biofilm development using a chambered coverglass model. The Journal of Visusalized Experiments. (118), e54819(2016).
- Cockeran, R., et al. Biofilm formation and induction of stress response genes is a common response of several serotypes of the pneumococcus to cigarette smoke condensate. The Journal of Infection. 80 (2), 204-209 (2020).
- Bisht, K., Moore, J. L., Caprioli, R. M., Skaar, E. P., Wakeman, C. A. Impact of temperature-dependent phage expression on Pseudomonas aeruginosa biofilm formation. npj Biofilmsand Microbiomes. 7 (22), (2021).
- Harrison, A., et al. Reprioritization of biofilm metabolism is associated with nutrient adaptation and long-term survival of Haemophilus influenzae. NPJ Biofilms and Microbiomes. 5 (1), 33(2019).
- Sousa, C., Franca, A., Cerca, N. Assessing and reducing sources of gene expression variability in Staphylococcus epidermidis biofilms. BioTechniques. 57, 295-301 (2014).
- Boom, R. Rapid and simple method for purification of nucleic acids. Journal of Clinical Microbiology. 28 (3), 495-503 (1990).
- Coppin, C. Re: How do silica based RNA spin columns only bind RNA and not DNA. , Available from: https://www.researchgate.net/post/How_do_silica_based_RNA_spin_columns_only_bind_RNA_and_not_DNA/60b017bffa5c4151cac1c/citation/download (2021).
- Kennedy, S. A complete guide to how nucleic extraction kits work. , Available from: https://bitesizebio.com/13516/how-dna-extraction-rna-miniprep-kits-work/ (2021).
- Qubit RNA HS Assay Kit User Guide. Thermo Fisher Scientific. , Available from: https://www.thermofisher.com/document-connect/document-connect.html?url=https%3A%2F%2Fassets.thermofisher.com%2FTFS-Assets%2FLSG%2Fmanuals%2FQubit_RNA_HS_Assay_UG.pdf&title=VXNlciBHdWlkZTogUXViaXQgUk5BIEhTIEFzc2F5IEtpdHM (2015).
- Mueller, O., Lightfoot, S., Schroeder, A. RNA Integrity Number (RIN) - Standardization of RNA Quality Control (Application report # 5989-1165EN). , Available from: https://www.agilent.com/cd/library/applications/5989-1165EN.pdf (2016).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 3(2006).
- Culviner, P. H., Guegler, C. K., Laub, M. T. A Simple, Cost-Effective, and Robust Method for rRNA Depletion in RNA-Sequencing Studies. mBio. 11 (2), (2020).
- MGIEasy RNA Directional Library Prep Set User Manual verA2. MGI Tech Co. , Available from: https://en.mgi-tech.com/products/reagents_info/14/ (2020).
- Andrews, S. FastQC: A Quality Control Tool for High Throughput Sequence Data. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina Sequence Data. Bioinformatics. , (2014).
- NCBI Resource Coordinators. Database resources of the National Center for Biotechnology Information. Nucleic Acids Research. 44 (1), (2016).
- Li, H. Aligning sequence reads, clone sequences and assembly contigs with BWA-MEM. arXiv. , (2013).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), (2009).
- Liao, Y., Shi, W. Read trimming is not required for mapping and quantification of RNA-seq reads at the gene level. NAR Genomics and Bioinformatics. 2 (3), (2020).
- Penir, S. Calculating Mapping Statistics from a SAM/BAM file using SAMtools and awk. , Available from: https://sarahpenir.github.io/bioinformatics/awk/calculating-mapping-stats-from-a-bam-file-using-samtools-and-awk/ (2019).
- Haile, S., et al. Evaluation of protocols for rRNA depletion based RNA sequencing of nanogram inputs of mammalian total RNA. PLoS ONE. 14 (10), 0224578(2019).
- Schuierer, S., et al. A comprehensive assessment of RNA-seq protocols for degraded and low-quantity samples. BMC Genomics. 18 (442), (2017).
- Shanker, S., et al. Evaluation of Commercially Available RNA Amplification Kits for RNA Sequencing Using Very Low Input Amounts of Total RNA. Journal of Biomolecular Techniques. 26 (1), (2015).
- Adiconis, X., et al. Comparative analysis of RNA sequencing methods for degraded or low-input samples. Nature Methods. 10, 623-629 (2013).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (13), (2016).
- Illumina, Inc. Coverage depth recommendations. , Available from: https://www.illumina.com/science/technology/next-generation-sequencing/plan-experiments/coverage.html (2021).
- What is a good sequencing depth for bulk RNA-Seq. ECSEQ Bioinformatics. , Available from: https://www.ecseq.com/support/ngs/what-is-a-good-sequencing-death-for-bulk-rna-seq (2019).
- Bedre, R. Sequencing coverage and breadth of coverage. , Available from: https://www.reneshbedre.com/blog/sequencing-coverage.html (2021).
- Dotsch, A., et al. The Pseudomonas aeruginosa transcriptome in planktonic cultures and static biofilms using RNA sequencing. PLoS One. 7 (2), 31092(2012).
- Chen, Y., et al. Population dynamics and transcriptomic responses of Pseudomonas aeruginosa in a complex laboratory microbial community. npj Biofilms and Microbiomes. 5 (1), (2019).
- Thoming, J. G., et al. Parallel evolutionary paths to produce more than one Pseudomonas aeruginosa biofilm phenotype. NPJ Biofilms and Microbiomes. 6, 2(2020).
- Soares, A., et al. Understanding ciprofloxacin failure in Pseudomonas aeruginosa biofilm: persister cells survive matrix disruption. Frontiers in Microbiology. 10, 2603(2019).
- Whiteley, M., et al. Gene expression in Pseudomonas aeruginosa biofilms. Nature. 413, (2001).
- Chomczynski, P., Sacchi, N. The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: twenty-something years on. Nature Protocols. 1, (2006).
- Liu, Y., Zhou, J., White, K. P. RNA-seq differential expression studies: more sequence or more replication. Bioinformatics. 30 (3), (2014).
- Wieczorek, D., Delauriere, L., Schagat, T. Methods of RNA Quality Assessment. , Available from: https://www.promega.ca/resources/pubhub/methods-of-rna-quality-assessment/ (2021).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados