Method Article
عزل الحمض النووي الريبي الكلي عن الزائفة الزنجارية داخل الأغشية الحيوية لقياس التعبير الجيني
In This Article
Summary
يقدم هذا البروتوكول طريقة لعزل الحمض النووي الريبي من الأغشية الحيوية الزائفة الزنجارية المزروعة في شرائح الغرفة لتسلسل عالي الإنتاجية.
Abstract
الزائفة الزنجارية هي مسببات الأمراض البكتيرية الانتهازية التي تسبب التهابات في الشعب الهوائية لمرضى التليف الكيسي (CF). من المعروف أن المتصورة الزنجارية بقدرتها على تكوين أغشية حيوية محمية بواسطة مصفوفة من عديدات السكاريد الخارجية. تسمح هذه المصفوفة للكائنات الحية الدقيقة بأن تكون أكثر مرونة في مواجهة العوامل الخارجية ، بما في ذلك العلاج بالمضادات الحيوية. واحدة من أكثر الطرق شيوعا لنمو الأغشية الحيوية للبحث هي في ألواح الميكروتيتر أو الشرائح ذات الغرفة. تتمثل ميزة هذه الأنظمة في أنها تسمح باختبار ظروف النمو المتعددة ، لكن عيبها هو أنها تنتج كميات محدودة من الأغشية الحيوية لاستخراج الحمض النووي الريبي. الغرض من هذه المقالة هو توفير بروتوكول مفصل خطوة بخطوة حول كيفية استخراج إجمالي الحمض النووي الريبي من كميات صغيرة من الأغشية الحيوية ذات الجودة والكمية الكافية لتسلسل عالي الإنتاجية. يسمح هذا البروتوكول بدراسة التعبير الجيني داخل أنظمة الأغشية الحيوية هذه.
Introduction
تتميز معظم الالتهابات البكتيرية المزمنة ، مثل الالتهابات الرئوية لدى مرضى التليف الكيسي (CF) والالتهابات المرتبطة بالأطراف الاصطناعية ، بنمو الكائنات الحية داخل الأغشية الحيوية. الأغشية الحيوية1 عبارة عن مجتمعات من البكتيريا المغلفة في مصفوفة تتكون بشكل أساسي من السكريات2. يمكن أن تكون البكتيريا الموجودة في الأغشية الحيوية بطيئة النمو ، ونائمة في التمثيل الغذائي ، وفي الظروف اللاهوائية ونقص الأكسجة. تعتبر الأغشية الحيوية أكثر مقاومة للمضادات الحيوية بسبب عوامل مثل انخفاض اختراق المضادات الحيوية ، وزيادة التعبير عن مضخات تدفق الأدوية ، وانخفاض انقسام الخلايا3. لهذه الأسباب وغيرها ، فهي ذات أهمية بحثية كبيرة.
من أجل دراسة العدوى المستمرة بدقة مثل عدوى الزائفة الزنجارية المزمنة في مرضى التليف الكيسي ، يجب أن تنعكس ظروف النمو التي تظهر مع تكوين الأغشية الحيوية بدقة في المختبر. تتمثل إحدى الطرق الشائعة ذات الإنتاجية العالية في زراعتها في شرائح الغرفة أو ألواح الميكروتيتر ومراقبة تكوين الأغشية الحيوية عن طريق الفحص المجهري متحد البؤر4. من المعروف أن المنظم الرئيسي في الانتقال من نمط الحياة البكتيري العوالق ، أو الطفو الحر ، إلى نمط الحياة البكتيري الحيوي هو الرسول الثانوي ، cyclic-di-GMP5. زيادة مستويات دورية ثنائي ممارسات التصنيع الجيدة تزيد من التعبير عن جينات معينة تعزز نمو الأغشية الحيوية. تلعب الحمض النووي الريبي التنظيمي الصغير غير المشفر واستشعار النصاب أيضا أدوارا مهمة في تنظيم تكوين الأغشيةالحيوية 5. قد يكون قياس التعبير الجيني للأغشية الحيوية عن طريق تسلسل الحمض النووي الريبي البكتيري المستخرج أمرا صعبا. P. aeruginosa ، على سبيل المثال ، ينتج ثلاثة عديد السكاريد الخارجي (Psl و Pel و alginate) ، والتي يتم إنتاجها بكميات كبيرة في الأغشية الحيوية6،7. يمكن أن تتداخل السكريات هذه مع استخراج الحمض النووي الريبي وتنقيته مما يؤدي إلى مستحضرات غير نقية تحتوي على مستويات منخفضة من الرناالمرسال البكتيري 8. مجموعات استخراج الحمض النووي الريبي المتوفرة تجاريا قادرة على إنتاج الحمض النووي الريبي عالي الجودة من المزارع البكتيرية العوالق ولكنها قد لا تعمل بشكل جيد مع مزارع الأغشية الحيوية9،10،11. هناك عدد قليل من مجموعات استخراج الحمض النووي الريبي التجارية التي تدعي أنها تعمل مع الأغشية الحيوية ، والتي نستخدمها بهذه الطريقة.
في هذه المخطوطة ، نصف إجراءات زراعة P. aeruginosa الأغشية الحيوية في شرائح الغرفة واستخراج mRNA البكتيري لتسلسل عالي الإنتاجية12،13. باستخدام العزلات السريرية التي تم جمعها من عينات البلغم من مرضى التليف الكيسي ، نوضح أنه يمكن استخدام هذه الطرق للعزلات ذات خصائص النمو المختلفة. بالمقارنة مع المنشورات السابقة ، تم وصف هذا البروتوكول بالتفصيل لتمكين نجاح أفضل في دراسة التعبير الجيني للأغشية الحيوية البكتيرية11،14،15،16.
Protocol
مجلس أخلاقيات البحث (REB) مطلوب لجمع ومعالجة عينات البلغم من البشر. تمت الموافقة على هذه الدراسة من قبل مستشفى الأطفال المرضى (REB # 1000019444). يطلب من مجلس أخلاقيات البحث (REB) جمع عينات البلغم وتخزينها من البشر. تمت الموافقة على هذه الدراسات من قبل مستشفى الأطفال المرضى REB # 1000058579.
1. تكوين الأغشية الحيوية
- تنمو عزلات Pseudomonas aeruginosa التي تم الحصول عليها من عينات البلغم لمرضى التليف الكيسي المستخدمة في هذه الدراسة على ألواح أجار Luria Broth (LB) في حاضنة 37 درجة مئوية بين عشية وضحاها.
ملاحظة: تقنية الخطوط المناسبة مهمة للحصول على مستعمرات بكتيرية مفردة. سيؤدي الخطوط أثناء تدوير اللوحة إلى تخفيف خلايا البكتيريا بشكل كاف بحيث يمكن أن تنمو المستعمرات الفردية.- قم بإزالة البكتيريا الخطية باستخدام حلقة تلقيح بنمط متعرج في الطرف العلوي من لوحة LB جديدة حتى يتم تغطية حوالي 1/4 من اللوحة.
- قم بتدوير اللوحة حوالي 60 درجة. خذ حلقة تلقيح جديدة وقم بتمريرها مرة واحدة عبر المنطقة المخططة وإلى منطقة ثانية نظيفة من اللوحة ، مع تكرار النمط المتعرج.
- كرر الخطوة 1.1.2 بحلقة جديدة في منطقة ثالثة من اللوحة. استبدل الغطاء واقلب اللوحة عند وضعها في الحاضنة.
- استخدم حلقة تلقيح معقمة لاختيار مستعمرة بكتيرية واحدة من صفيحة أجار (تحتوي على سلالة بكتيرية واحدة) وتلقيح أنبوب مزرعة مملوء ب 5 مل من وسائط LB المعقمة. باستخدام حلقة جديدة في كل مرة ، قم بتلقيح أنبوبين إضافيين مملوءين ب 5 مل من وسائط LB من نفس اللوحة.
- كرر نفس إجراء التلقيح للسلالات البكتيرية المختلفة. قم بزراعة الثقافات بين عشية وضحاها عند 37 درجة مئوية مع الاهتزاز عند 220 دورة في الدقيقة.
ملاحظة: يتم استخدام كل صفيحة أجار تحتوي على سلالة بكتيرية واحدة لتلقيح 3 أنابيب استزراع مستقلة. تمثل الأنابيب الثلاثة تكرارات بيولوجية في ثلاث نسخ لسلالة واحدة ويتم التعامل معها كعينات منفصلة. هذا يختلف عن التكرارات التقنية التي قد تستلزم استخراج الحمض النووي الريبي 3 مرات من أنبوب استزراع واحد. - قم بإعداد 1: 100 تخفيفات من المزارع الليلية عن طريق نقل 50 ميكرولتر من المزرعة الليلية إلى أنبوب استزراع جديد يحتوي على 4.95 مل من وسائط LB الجديدة.
- قم بزراعة المزارع المخففة لمدة 3 ساعات أخرى عند 37 درجة مئوية مع الاهتزاز عند 220 دورة في الدقيقة أو حتى يصبح OD600 0.1 أو أعلى. قم بقياس كثافة الخلايا على مقياس الطيف الضوئي عند OD600.
- في أنبوب جديد سعة 1.7 مل ، اضبطOD600 إلى 0.1 (مرحلة السجل المبكر) بحجم إجمالي قدره 1.5 مل مع LB الطازج.
- تخلط بلطف عن طريق الانعكاس. انقل 300 ميكرولتر من كل ثقافة معدلة إلى 4 آبار من شريحة غرفة مكونة من 8 آبار لينتهي بها الأمر بعينتين مختلفتين لكل شريحة (الشكل 1).
- ضع الشرائح ، دون إزعاج ، في حاضنة 37 درجة مئوية طوال الليل لمدة 24 ساعة. لمنع التبخر ، ضع الشرائح فوق منشفة ورقية مبللة في صينية بلاستيكية صغيرة.
2. استعادة الأغشية الحيوية
ملاحظة: تحتوي كل شريحة زجاجية على ثمانية آبار منفصلة. تتكون عينة واحدة من أربعة آبار بها أغشية حيوية سيتم تجميعها17. بروتوكول الاستخراج هذا مخصص لعينة واحدة (4 آبار) حيث يتم استرداد الأغشية الحيوية من بئرين في المرة الواحدة. يتم إجراء عمليات استخراج الحمض النووي الريبي باستخدام مجموعة استخراج الحمض النووي الريبي التجارية التي تتضمن خطوة ضرب الخرزة والتنظيف القائم على العمود ، مع التعديلات. اتبع تعليمات الشركة المصنعة لإعداد الكاشف.
- في غطاء التدفق الرقائقي، قم بإزالة الوسائط ببطء من 2 من أصل 4 آبار باستخدام طرف الماصة. قم بإمالة الشريحة بزاوية 45 درجة وقم بإخراج الوسائط من الزاوية السفلية للآبار لمنع انفصال الأغشية الحيوية.
- مع الحفاظ على الشريحة مائلة ، اغسل الخلايا العوالق عن طريق سحب 300 ميكرولتر من الماء الخالي من RNase برفق في الزاوية السفلية من البئرين الفارغين. قم بإزالة الماء عن طريق إخراجه برفق، كما هو موضح في الخطوة 2.1. كرر خطوة الغسيل مع إزالة أكبر قدر ممكن من الماء.
- أضف 300 ميكرولتر من كاشف حماية الحمض النووي الريبي (انظر جدول المواد) إلى كل من البئرين الفارغين مع وجود أغشية حيوية في قاعدتها. ضع شريحة الغرفة على صفيحة زجاجية لمنع الآبار من الانكسار ، ثم كشط الأغشية الحيوية في 2 الآبار بملعقة معدنية صغيرة خالية من النوكلياز ومعقمة لإعادة تعليق بكتيريا الأغشية الحيوية. اتركيه حتى يتم استرداد الأغشية الحيوية من الآبار المتبقية التي لم تمسها من نفس العينة.
ملاحظة: تضمن إضافة كاشف حماية الحمض النووي الريبي استقرار عينات الأغشية الحيوية في الآبار المكشطة عند درجة الحرارة المحيطة أثناء معالجة البئرين المتبقيين من نفس العينة. يعمل كاشف حماية الحمض النووي الريبي على تحلل الخلايا وتعطيل النوكليازات والعوامل المعدية ، مما يؤدي إلى الحفاظ على الحمض النووي الريبي. - لاستعادة الأغشية الحيوية من الآبار المتبقية لعينة (تذكير: تتكون عينة واحدة من 4 آبار) ، قم بإزالة وسائط LB من الآبار الجديدة المتبقية بنفس الطريقة الموضحة في الخطوة 2.1. كرر الخطوة 2.2 لغسل الخلايا العوالق بمياه خالية من RNase من كلا البئرين الجديدين ، كما كان من قبل.
- ارجع إلى أول 2 بئر مع أغشية حيوية مكشطة في كاشف الحماية ، تم إنشاؤها في نهاية الخطوة 2.3 ، وقم بسحب الأنابيب ببطء لخلط 300 ميكرولتر من الأغشية الحيوية المعاد تعليقها من بئر واحد مكشط ، في محاولة لعدم تكوين الكثير من الفقاعات.
- انقل جميع المحتويات من البئر إلى أحد الآبار التي تم غسلها حديثا وتفريغها. امزج وانقل 300 ميكرولتر من الأغشية الحيوية المعاد تعليقها من البئر الثاني الذي تم كشطه جيدا في الباقي ، المغسول حديثا ، المفرغ جيدا.
ملاحظة: بدلا من إضافة كاشف حماية جديد من الحمض النووي الريبي إلى 2 من الآبار المغسولة حديثا مع الأغشية الحيوية ، انقل الأغشية الحيوية التي تم كشطها مسبقا الموجودة بالفعل في كاشف الحماية إلى هذه الآبار المغسولة حديثا. سيؤدي ذلك إلى الحفاظ على حجم العينة المركب منخفضا بما يكفي لتلبية متطلبات الإدخال لمجموعة استخراج الحمض النووي الريبي التجارية. انظر الشكل 1 للحصول على مخطط.
- انقل جميع المحتويات من البئر إلى أحد الآبار التي تم غسلها حديثا وتفريغها. امزج وانقل 300 ميكرولتر من الأغشية الحيوية المعاد تعليقها من البئر الثاني الذي تم كشطه جيدا في الباقي ، المغسول حديثا ، المفرغ جيدا.
- كرر كشط الأغشية الحيوية في الآبار الجديدة كما في الخطوة 2.3 ، عن طريق وضع شريحة الغرفة على صفيحة زجاجية وكشط الأغشية الحيوية في الآئرين الجديدين باستخدام ملعقة معدنية صغيرة خالية من النوكلياز ومعقمة لإعادة تعليق بكتيريا الأغشية الحيوية.
- اجمع كل الأغشية الحيوية المعاد تعليقها من 2 بئر جديدين في أنبوب واحد خال من RNase ، منخفض الارتباط ، 1.5 مل من أنبوب الطرد المركزي الدقيق. قم بقياس الحجم ، والذي يجب أن يكون ~ 500 - 600 ميكرولتر إجمالا.
ملاحظة: سيحتوي تعليق الأغشية الحيوية المدمج من هذا الزوج الثاني من الآبار الجديدة على جميع مواد الأغشية الحيوية من الآبار الأربعة الأصلية للعينة.
3. العزل الكلي للحمض النووي الريبي وتقييم الجودة
ملاحظة: يتم إجراء استخراج الحمض النووي الريبي باستخدام مجموعة استخراج الحمض النووي الريبي التجارية التي تدعي أنها تعمل على الأغشية الحيوية. يتم تضمين المكونات الفردية في جدول المواد ، إن أمكن. يتم تقديم تفسيرات للآليات الكامنة وراء كل خطوة من خطوات التنقية عندما يكون ذلك ممكنا.
- أضف ما يكفي من كاشف حماية الحمض النووي الريبي لإجمالي 750 ميكرولتر في الأنبوب. انقل الحجم بالكامل إلى أنبوب تحلل خفق حبة سعة 2 مل يحتوي على حبات 0.1 و 0.5 مم (انظر جدول المواد). اضرب لمدة 2 1/2 دقيقة في مضرب حبة بأقصى سرعة.
ملاحظة: يضمن الجمع بين الخرز عالي الكثافة 0.1 مم و 0.5 مم بالإضافة إلى الخلط عالي السرعة على مضرب الخرز تجانسا شاملا لجدران الخلايا الميكروبية. - جهاز طرد مركزي عند 16,000 × جم لمدة 1 دقيقة لحبيبات الخرز. انقل المادة الطافية إلى أنبوب طرد مركزي دقيق جديد ، مما يقلل من نقل أي حبات ، مما يجعل الخطوة 3.3 أسهل. قياس الحجم.
- أضف حجما متساويا من المخزن المؤقت لتحلل الحمض النووي الريبي (~ 450 ميكرولتر) واخلطه جيدا. انقل ما يصل إلى 800 ميكرولتر من العينة ، مع تجنب نقل أي حبات ، إلى عمود السيليكا في أنبوب تجميع وجهاز طرد مركزي عند 16,000 × جم لمدة 30 ثانية. احفظ التدفق لأنه يحتوي على الحمض النووي الريبي بينما يرتبط الحمض النووي بالعمود.
ملاحظة: يحتوي المخزن المؤقت لتحلل الحمض النووي الريبي على غوانيدينيوم ثيوسيانات والمنظف N-lauroylsarcosine لتحلل الخلايا. ثيوسيانات Guanidinium هو عامل فوضوي يعطل أيضا النوكليازات ، وفي وجود السيليكا ، الموجودة في عمود الدوران ، تعزز ارتباط الحمض النووي بالسيليكا18. يسمح غياب الإيثانول بالارتباط التفضيلي للحمض النووي وليس الحمض النووي الريبي بعمود دوران السيليكا19. الغرض من الخطوة 3.3 هو ربط وإزالة الحمض النووي الجيني. نريد الاحتفاظ بالحمض النووي الريبي الموجود في جزء التدفق. - إذا بقي المزيد من العينة ، فقم بنقل العمود إلى أنبوب تجميع جديد وأعد تحميله ببقية العينة. جهاز طرد مركزي عند 16,000 × جم لمدة 30 ثانية. حافظ على التدفق مع الحمض النووي الريبي وادمجه مع الكمية الأولى.
- قم بقياس حجم التدفق المجمعة وأضف حجما متساويا من الإيثانول بنسبة 100٪ واخلطه جيدا. انقل ما يصل إلى 800 ميكرولتر من المحلول إلى عمود دوران جديد ثان من السيليكا في أنبوب تجميع وجهاز طرد مركزي عند 16,000 × جم لمدة 30 ثانية. تجاهل التدفق.
ملاحظة: إن إضافة الإيثانول إلى محلول الملح الفوضوي الذي يحتوي على الحمض النووي الريبي يعزز ارتباط الحمض النووي الريبي بعمود دوران السيليكا19. - بالنسبة للمحاليل > 800 ميكرولتر ، أعد تحميل عمود الدوران وجهاز الطرد المركزي حتى يتم تدوير المحلول بالكامل. تجاهل التدفق بعد كل دورة.
- أضف 400 ميكرولتر من محلول الغسيل إلى العمود وجهاز الطرد المركزي عند 16,000 × جم لمدة 30 ثانية لإزالة بعض الأملاح. تجاهل التدفق.
- قم بإعداد مزيج تفاعل DNase I وفقا لتعليمات الشركة المصنعة وقم بإجراء معالجة DNase في العمود لإزالة أي حمض نووي متبقي.
- أعد تعليق DNase I المجفف بالتجميد في 275 ميكرولتر من الماء الخالي من RNase لعمل محلول 1 U / μL. امزج عن طريق الانعكاس اللطيف.
- امزج 5 ميكرولتر من DNase I المخفف مع 75 ميكرولتر من المخزن المؤقت لهضم DNase المقدم. تخلط بلطف عن طريق الانعكاس.
- أضف 80 ميكرولتر من المحلول المحضر مباشرة إلى مصفوفة العمود. احتضن في درجة حرارة الغرفة لمدة 20 دقيقة.
- أضف 400 ميكرولتر من المخزن المؤقت لإعداد الحمض النووي الريبي إلى العمود وجهاز الطرد المركزي عند 16,000 × جم لمدة 30 ثانية. تجاهل التدفق.
- أضف 700 ميكرولتر من مخزن غسيل الحمض النووي الريبي إلى العمود وكرر الطرد المركزي. تجاهل التدفق.
- أضف 400 ميكرولتر من مخزن غسيل الحمض النووي الريبي وقم بالطرد المركزي للعمود لمدة دقيقتين لإزالة أي مخزن مؤقت متبقي تماما.
ملاحظة: هناك 2 خطوات غسيل مختلفة لإزالة الشوائب على العمود. يحتوي المخزن المؤقت التحضيري على ملح ضعيف من الفوضى ممزوج بالإيثانول من أجل إزالة البروتينات المتبقية. بعد ذلك ، يتم استخدام مخزن الغسيل المؤقت لإجراء غسل الإيثانول لإزالة الأملاح. يجب إزالة أي إيثانول متبقي للسماح بالشطف الفعال للحمض النووي الريبي19،20. - انقل العمود بعناية إلى أنبوب طرد مركزي دقيق جديد منخفض الربط.
- أضف 50 ميكرولتر من الماء الخالي من RNase مباشرة إلى مصفوفة العمود واحتضنه لمدة 5 دقائق. جهاز طرد مركزي عند 16,000 × جم لمدة 1 دقيقة لإزالة الحمض النووي الريبي.
- لتنظيف إضافي لإزالة مثبطات تفاعل البوليميراز المتسلسل ، ضع مرشح مثبط تفاعل البوليميراز المتسلسل في أنبوب تجميع جديد. أضف 600 ميكرولتر من محلول تحضير المثبط المقدم (انظر جدول المواد).
- جهاز طرد مركزي عند 8,000 × جم لمدة 3 دقائق لغسل الفلتر. انقل الفلتر المغسول إلى أنبوب طرد مركزي دقيق جديد منخفض الربط.
- انقل الحمض النووي الريبي المغطى من الخطوة 3.13 إلى الفلتر المغسول ، وجهاز الطرد المركزي عند 16,000 × جم لمدة 3 دقائق.
ملاحظة: يمكن استخدام الحمض النووي الريبي على الفور أو تخزينه عند -80 درجة مئوية. - تحديد تركيز الحمض النووي الريبي باستخدام نظام فلورومتري عاليالحساسية 21.
ملاحظة: تسمح هذه الأنظمة بالقياس الكمي الحساس لكمية صغيرة من الحمض النووي الريبي في محلول خاص بالهدف محل الاهتمام.- قم بتقييم جودة الحمض النووي الريبي باستخدام نظام الرحلان الكهربائي الآلي الذي يمكن أن يوفر RIN (رقم سلامة الحمض النووي الريبي) ، وهو مقياس لجودة الحمض النوويالريبي 22،23.
4. استنفاد الحمض النووي الريبي الريبوسومي وتسلسل عالي الإنتاجية
- إرسال الحمض النووي الريبي إلى مركز جينوم مركز تحليل تطور الجينوم ووظيفته (CAGEF) في جامعة تورنتو (تورنتو ، كندا) (https://www.cagef.utoronto.ca/) لاستنفاد الحمض النووي الريبي البكتيري وإعداد مكتبة اتجاه الحمض النووي الريبي (انظر جدول المواد).
ملاحظة: يستهدف استنفاد الحمض النووي الريبي البكتيري 5S و 16S و 23S rRNAs للإزالة24. - استنفاد الحمض النووي الريبي الريبي باستخدام مجموعة استنفاد بكتيريا الحمض النووي الريبي التجاري. اتبع بروتوكول إدخال 10 نانوغرام - 1 ميكروغرام من إجمالي الحمض النووي الريبي السليم أو المتدهور جزئيا.
- قم ببناء مكتبات تسلسل الحمض النووي الريبي باستخدام مجموعة إعداد مكتبة اتجاهية للحمض النووي الريبي مع فهارس مختلفة مرفقة بكل مكتبة.
- تجمع كميات متساوية المولار لكل مكتبة RNA وقم بإجراء تسلسل عالي الإنتاجية معقراءات 25 مقترنة ذات 100 قاعدة.
5. تقييم جودة قراءات التسلسل
ملاحظة : تحقق من جودة قراءات التسلسل باستخدام البرنامج المتاح مجانا ، FastQC26 ، المتوفر من خلال النظام الأساسي المجاني مفتوح المصدر ، Galaxy27.
- انتقل إلى https://usegalaxy.org/. انقر فوق قائمة تسجيل الدخول أو التسجيل وقم بتسجيل الدخول باستخدام بيانات الاعتماد أو قم بإنشاء حساب.
- انقر فوق الرابط تحميل البيانات في الجزء العلوي الأيسر من الصفحة ، ضمن قائمة الأدوات ، وقم بتحميل ملفات تسلسل fastq.gz. انتظر حتى تظهر أسماء الملفات على الجانب الأيسر من الصفحة، ضمن لوحة المحفوظات.
- حدد FASTQ Quality Control ضمن قائمة الأدوات للكشف عن قائمة البرامج. حدد FastQC ، والذي سيملأ اللوحة الوسطى للشاشة.
- ضمن بيانات القراءة القصيرة من السجل الحالي، حدد ملفات fastq.gz التي تم تحميلها من القائمة المنسدلة.
- حدد تنفيذ لتشغيل البرنامج.
- عرض النتائج ضمن لوحة History (الجدول 2).
ملاحظة: للحصول على إرشادات أكثر تفصيلا حول كيفية استخدام Galaxy، تفضل بزيارة صفحة الدعم على https://galaxyproject.org/support/.
6. رسم خرائط قراءات التسلسل
ملاحظة: المدرج هو خط أنابيب أساسي لقص المحول وقراءة الخرائط لبيانات تسلسل الحمض النووي الريبي. يتم اقتطاع تسلسلات المحول من القراءات باستخدام Trimmomatic28. يتم تعيين القراءات المشذبة إلى P. aeruginosa الجينوم المرجعي PAO1 (NC_002516.2) ، الذي تم الحصول عليه من NCBI (https://www.ncbi.nlm.nih.gov/) 29باستخدام BWA30 و Samtools31. من أجل البساطة ، يطلق على زوج من القراءات اسم PA_1.fq و PA_2.fq. يسمى ملف قراءة المحول المراد قصه Adapter.fa ؛ ويسمى التسلسل المرجعي PAO1 PAO1.fasta. جميع الأدوات مفتوحة المصدر وتعمل في بيئة UNIX / LINUX. ينصح بشدة بالتعرف على أساسيات UNIX / LINUX من أجل تنفيذ هذه الأوامر.
- افتح نافذة في UNIX / LINUX.
- قم بتثبيت Java و Trimmomatic و BWA و Samtools.
- انتقل إلى المجلد حيث يوجد trimmomatic-0.39.jar الملف.
- قم بقص أي تسلسلات محول من القراءات عن طريق كتابة الأمر:
Java -jar PA_1.fq PA_2.fq PA_1_paired.fq PA_1_unpaired.fq PA_2_paired.fq PA_2_unpaired.fq ILLUMINACLIP: adapters.fa
ملاحظة: تتم إزالة تسلسلات المحول فقط. لم يتم قطع القراءات للجودة32. - انقل الملف المرجعي PAO1.fasta إلى نفس المجلد.
- قم بفهرسة المرجع باستخدام BWA باستخدام الأمر:
مؤشر BWA PA01.fasta - قم بتعيين القراءات المقترنة إلى الجينوم المرجعي عن طريق كتابة الأوامر الأربعة التالية. اكتب كل أمر بعد انتهاء الأمر السابق.
Bwa -mem PA01.fasta PA_1_paired.fq PA2_2_paired.fq > PA_R1R2_map.mem.sam
عرض Samtools -S -b PA_R1R2_map.mem.sam > PA_R1R2.bam
Samtools فرز PA_R1R2.bam -o PA_R1R2_sorted.bam
فهرس Samtools PA288_Rep1_R1R2_sorted.bam - عرض إحصائيات التعيين عن طريق كتابة الأمر:
Samtools flagstat PA_R1R2_sorted.bam
ملاحظة:يشير السطر الثالث من الإحصائيات إلى نسبة القراءات التي يتم تعيينها إلى الجينوم المرجعي. - احسب متوسط عمق القراءة واتساع التغطية باستخدام الأمرين التاليين33 ، على التوالي:
عمق Samtools - PA_R1R2_sorted.BAM | awk '{c ++ ; s +=$3}END{print s / c}'
عمق Samtools - A PA288_Rep1_R1R2_sorted.BAM | awk '{c ++ ; if ($3>0) الإجمالي +=1}END{print (Total / c) * 100}'
النتائج
تظهر نظرة عامة على الطريقة في الشكل 1. استخدمنا سابقا شرائح غرفة 8 آبار لزراعة P. aeruginosa الأغشية الحيوية وتعريضها للمضادات الحيوية قبل فحصها عن طريق الفحص المجهري متحد البؤر في نقاط زمنية مختلفة12،13. يمكن استخدام هذه الطريقة لاستخراج الحمض النووي الريبي الكلي مباشرة من الأغشية الحيوية المزروعة في هذا النظام من أجل دراسة تغيرات التعبير الجيني بعد العلاج. تم تحسين هذا البروتوكول ل P. aeruginosa ولكن يمكن تكييفه بسهولة مع الأنواع البكتيرية الأخرى.
قد يكون من الصعب استخراج كمية كافية من الحمض النووي الريبي عالي الجودة من كميات صغيرة من الأغشية الحيوية لاستنفاد الحمض النووي الريبي وتسلسل الإنتاجية العالية. باستخدام هذا البروتوكول ، يتم استخراج الحمض النووي الريبي الكلي بنجاح من 17 عزلة مختلفة من الأغشية الحيوية P. aeruginosa في ثلاث نسخ ، بإجمالي 51 عينة منفصلة. يتم عرض كميات الحمض النووي الريبي المستخرج التي تمثل عوائد عالية ومنخفضة في الجدول 1. يتراوح تركيز الحمض النووي الريبي من 3.4 نانوغرام / ميكرولتر (أدنى) إلى 49.6 نانوغرام / ميكرولتر (أعلى) ، بمتوسط تركيز 14 نانوغرام / ميكرولتر ومتوسط 13.7 نانوغرام / ميكرولتر تعتبر تركيزات الحمض النووي الريبي السليمة التي تقل عن 10 نانوغرام / ميكرولتر عينات منخفضة الوفرة لاستنفاد الحمض النووي الريبي وتسلسل الجيل التالي ، لكن عينات الحمض النووي الريبي منخفضة الوفرة من الأغشية الحيوية يمكن أن تؤدي إلى بيانات تسلسل أقل جودة مقارنة بالعينات الأكثرتركيزا 34 ، 35،36،37. يتم عرض صفات الحمض النووي الريبي في الجدول 1 بواسطة RIN وفي الشكل 2 بواسطة مخططات الحمض النووي الريبي الكهربية المقابلة للعينات المركزة المنخفضة (PA565-3) والعالية (PA288-1). تمثل العينتان المتبقيتان (PA375-3 و PA921-1) غالبية العينات. كما هو موضح في الشكل 2 ، يحتوي الحمض النووي الريبي المستخرج من هذه الكميات من الأغشية الحيوية دائما على بعض الحمض النووي الريبي المتدهور ، مما يؤثر على قيم RIN الخاصة بهم. لذلك ، يتم استخدام التأكيد البصري لقمم الحمض النووي الريبي بدائية النواة 16S و 23S لتحديد جودة الحمض النووي الريبي عند عدم الإبلاغ عن RIN. باستخدام هذه المعايير ، يتم اختيار جميع عينات الحمض النووي الريبي البالغ عددها 51 للمضي قدما في استنفاد الحمض النووي الريبي وتسلسله ، مع التنبؤ بأن العينة ذات الجودة الرديئة ، PA565-3 ، قد تفشل. من بين عينات الحمض النووي الريبي المقدمة للتسلسل ، يتم إنشاء مكتبات ناجحة وتسلسلها ل 49 عينة ، مع فشل PA565-3 ، كما هو متوقع.
يتم سرد عدد قراءات التسلسل التي تم إنشاؤها لعينة التركيز العالي مع RIN جيد (PA288-1) وعينة التركيز المنخفض بدون RIN (PA375-3) في الجدول 2. تتضمن الإحصائيات الأساسية العدد الإجمالي للقراءات وطول القراءة ومحتوى GC. تظهر إحصائيات الموجز لكلتا العينتين عددا كبيرا من القراءات التي تم إنشاؤها وتشير إلى أنه لم يتم وضع علامة على أي منها على أنها ذات جودة رديئة ، مما يشير إلى بيانات تسلسل جيدة. يبلغ متوسط عدد القراءات لكل عينة حوالي 48 مليون ، وهو ما يعتبر عائدا جيدا. الأداة القياسية المستخدمة لتقييم جودة بيانات التسلسل الأولية هي FastQC26. يستخدم هذا البرنامج لإجراء فحوصات مراقبة الجودة على ملفات التسلسل الخام لتحديد ما إذا كانت الجودة كافية لمزيد من التحليلات ، أو ما إذا كانت هناك مشاكل أو تحيزات في البيانات التي تم إنشاؤها بواسطة جهاز التسلسل نفسه أو من مكتبات الحمض النووي الريبي المدخلة. يعرض الشكل 3 مقاييس مراقبة الجودة لبيانات تسلسل PA288-1 و PA375-3 ، والتي تمثل عينة RNA عالية الجودة وعينة RNA نموذجية منخفضة الجودة ، على التوالي. واحدة من أكثر المؤامرات إفادة من FastQC هي مخطط جودة التسلسل الأساسي. ستظهر بيانات التسلسل الجيد متوسط درجة جودة عالية (>30) لكل موضع في جميع القراءات ، مع انخفاض في متوسط درجة الجودة على طول القراءة. تمثل درجة الجودة 30 معدل خطأ 1 في 1000 ، وهو ما يعادل دقة نداء أساسية تبلغ 99.9٪. تحتوي الغالبية العظمى من القواعد في الشكل 3 على درجات جودة متوسطة ≥ 35 على طول القراءة بالكامل لكلتا العينتين ، مما يدل على بيانات تسلسل عالية الجودة بشكل استثنائي. يوفر هذا دليلا قويا على نجاح بروتوكول استخراج الحمض النووي الريبي المقدم هنا.
لإظهار أن طريقتنا يمكن أن تستعيد P. aeruginosa نصوص ، يتم تعيين قراءات التسلسل عالية الجودة ل PA288-1 و PA375-3 إلى P. aeruginosa الجينوم المرجعي PAO1 (NCBI NC_002516.2). يتم قطع القراءات فقط لمحول التسلسل وليس للجودة ، مع الاحتفاظ بجميع القواعد قبل تعيين32. وترد إحصاءات رسم الخرائط في الجدول 3. تعد النسبة المئوية للقراءات المعينة مقياسا مهما لدقة التسلسل. ببساطة ، كلما زاد عدد القراءات التي تتماشى مع التسلسل المرجعي الخاص بك كان ذلك أفضل. بالنسبة إلى PA288-1 و PA375-3 ، يتم تعيين 84٪ و 91٪ من القراءات ، على التوالي ، إلى الجينوم المرجعي. يتراوح النطاق المتوقع لتعيين قراءات تسلسل الحمض النووي الريبي القياسية بين 70 - 90٪ ، لذا فإن هذه القيم جيدة جدا ، خاصة عندما لا تتم إزالة القواعد ذات الجودة الرديئة38. يعد متوسط عمق القراءة مؤشرا جيدا لمتوسط عدد القراءات التي تتم محاذاة كل موضع أساسي في التسلسل المرجعي. كلما زاد العمق في كل موضع أساسي ، زادت دقة المكالمة الأساسية في كل موضع. يتم حسابه بقسمة مجموع أعماق القراءة المعينة في كل موضع في الجينوم المرجعي على العدد الإجمالي للقواعد في المرجع. يبلغ متوسط أعماق القراءة PA288-1 و PA375-3 400 أو أكثر ، وهو أمر جيد لتحليل التعبير الجيني في اتجاه مجرىالنهر 39،40. يخبرنا اتساع التغطية النسبة المئوية لطول الجينوم المرجعي الذي يغطيه التسلسل41. تغطي القراءات المحاذاة من كل من PA288-1 و PA375-3 96٪ من P. aeruginosa الجينوم المرجعي PAO1. يشير هذا إلى أن معظم P. aeruginosa الجينوم ممثل في بيانات التسلسل وليس فقط الامتدادات القصيرة. تظهر إحصائيات رسم الخرائط ل PA288-1 و PA375-3 أن هذه الطريقة يمكنها استعادة النصوص التي تتوافق مع التغطية والتوزيع الجيد ل P. aeruginosa الجينوم ، مما يدعم بروتوكول الاستخراج الناجح.
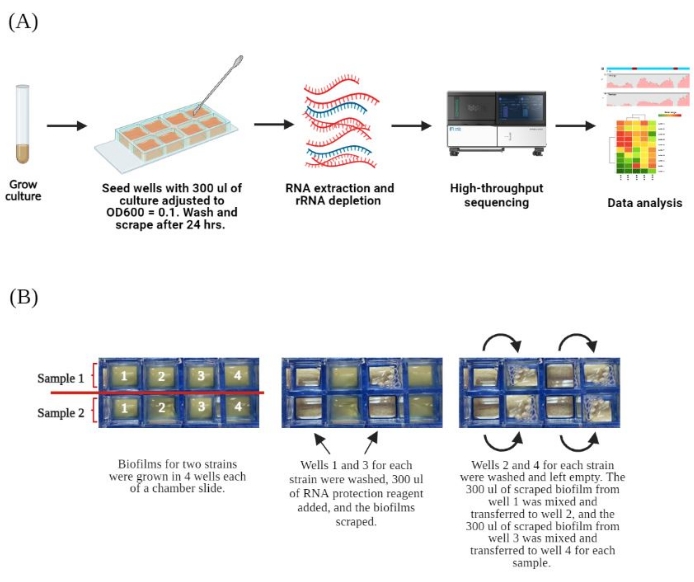
الشكل 1. نظرة عامة على البروتوكول. أ. تخطيطي لسير العمل التجريبي. نمت مزارع العوالق بين عشية وضحاها عند 37 درجة مئوية ، وتم تخفيفها بنسبة 1: 100 بوسائط جديدة في اليوم التالي ونمت لمدة 3 ساعات أخرى. تم تعديل الثقافات إلى OD600 من 0.1 و 300 ميكرولتر تم تلقيحها في 4 آبار من شريحة من 8 غرف لتوليد الأغشية الحيوية. بعد 24 ساعة ، تم غسل الأغشية الحيوية لإزالة الخلايا العوالق. تمت إضافة كاشف حماية الحمض النووي الريبي ، وكشطت الخلايا من الآبار. تم استخراج الحمض النووي الريبي الكلي ، واستنفاد الحمض النووي الريبي الريبوسومي وتسلسله. ب. سير عمل مفصل لنمو الأغشية الحيوية وإزالته من شريحة مكونة من 8 غرف. نمت سلالتان لكل شريحة في الاتجاه الموضح على اليسار. يتم عرض ترتيب كل عينة في أربعة آبار من الشريحة. لكل عينة مستقلة ، تمت معالجة الآبار 1 و 3 أولا ، وانتهت ب 300 ميكرولتر من الخلايا المكشطة في كاشف حماية الحمض النووي الريبي في كل بئر. مثال يتم تصويره بواسطة الشريحة الوسطى. بعد ذلك ، تم غسل الآبار 2 و 4 وإزالة السائل. يتم نقل الخلايا المعاد تعليقها من الآبار 1 و 3 إلى الآبار 2 و 4 ، على التوالي ، كما هو موضح في شريحة اليد اليمنى. بعد الكشط ، يتم دمج الخلايا المعاد تعليقها في الآبار 2 و 4 في أنبوب واحد للطرد المركزي الدقيق. تم إنشاء هذا الرقم مع BioRender.com. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 2. أمثلة على مخططات الحمض النووي الريبي الكهربائي من عينات الحمض النووي الريبي المستخرجة عالية إلى منخفضة الجودة. يتم تصنيف قمم الريبوسومات 16S و 23S عند قاعدة قممها. يتم تمثيل الحمض النووي الريبي المتدهور بقمم صغيرة الحجم ، يشار إليها بالأسهم وخط الأساس الوعر فوق الصفر. يتم عرض عينات الحمض النووي الريبي ذات الجودة المقبولة في اللوحات A و B و D. تظهر اللوحة C عينة من الحمض النووي الريبي ذات النوعية الرديئة حيث تكون قمم الريبوسومات مفقودة وكان التركيز منخفضا جدا ، كما يشار إليه بالمقياس الموجود على المحور Y. FU ، وحدات التألق ؛ NT ، نيوكليوتيدات. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 3. مخطط جودة FastQC لكل تسلسل أساسي لبيانات التسلسل PA288-1 و PA375-3. أ. مؤامرة الجودة للعينة عالية الجودة PA288-1. ب. مؤامرة الجودة للعينة النموذجية PA375-3. تعرض المخططات درجة الجودة المجمعة لكل موضع أساسي لجميع القراءات في الملف. يمثل الخط الأزرق متوسط درجة الجودة في كل موضع أساسي. يمثل الخط الأحمر داخل المربع الأصفر متوسط درجة الجودة في كل موضع ، ويظهر المربع الأصفر النطاق الرباعيللبنى المئوية من 25إلى 75. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
| عزل | كيوبت | رين | التسلسل |
| السلطة الفلسطينية288-1 | 26 نانوغرام / ميكرولتر | 7.5 | نعم |
| السلطة الفلسطينية 375-3 | 4.07 نانوغرام / ميكرولتر | غير أ | نعم |
| PA565-3 | 3.4 نانوغرام / ميكرولتر | غير أ | لا |
| PA921-1 | 9.11 نانوغرام / ميكرولتر | 6.6 | نعم |
الجدول 1. مقاييس جودة الحمض النووي الريبي المستخرج من عينات تمثيلية.
| السلطة الفلسطينية288-1 | السلطة الفلسطينية 375-3 | |
| إجمالي القراءات | 8,59,57,720 | 3,18,49,575 |
| يقرأ تم وضع علامة عليه على أنه جودة رديئة | 0 | 0 |
| طول القراءة | 100 | 100 |
| ٪ GC | 61 | 60 |
الجدول 2. إحصائيات ملخص PA288-1 و PA375-3 FastQC
| عزل | النسبة المئوية للقراءة المعينة إلى المرجع | متوسط عمق القراءة | اتساع نطاق التغطية |
| السلطة الفلسطينية288-1 | 83.93% | 404 | 96.68% |
| السلطة الفلسطينية 375-3 | 91.2% | 578 | 96.97% |
الجدول 3. إحصائيات رسم الخرائط PA288-1 و PA375-3
Discussion
تم استخراج إجمالي الحمض النووي الريبي بنجاح من 17 عينة مختلفة من الأغشية الحيوية البكتيرية في ثلاث نسخ ، مما ينتج عنه ما مجموعه 51 عينة. يتم تجميع مكتبات الحمض النووي الريبي التسع والأربعين وتسلسلها بنجاح. بشكل عام ، يثبت هذا صحة معايير الجودة لدينا بمعدل نجاح 96٪ على الرغم من أن أكثر من نصف العينات تعتبر منخفضة الوفرة وذات جودة دون المستوىالأمثل 34،35،36،37.
اهميه
يعد بروتوكول استخراج الحمض النووي الريبي فريدا من نوعه في شرحه التفصيلي لاستخراج الحمض النووي الريبي من كميات محدودة من الأغشية الحيوية التي نمت في شرائح الغرفة. يعد نمو الأغشية الحيوية في شريحة غرفة مكونة من 8 آبار نظاما مفيدا لدراسة تأثيرات العوامل المضافة خارجيا إلى الأغشية الحيوية أو تأثيرات التفاعلات الميكروبية في تكوين الأغشية الحيوية عن طريق الفحص المجهري متحد البؤر12،13،42. من أجل فحص التعبير عن الجينات المشاركة في تكوين الأغشية الحيوية في هذا النظام ، نقدم طريقة مفصلة لاستخراج الحمض النووي الريبي السليم بجودة وكمية كافية لتسلسل الحمض النووي الريبي. أفادت دراسات أخرى بنجاح استخراج الحمض النووي الريبي من كميات محدودة من الأغشية الحيوية ، لكن الغالبية تنمو الأغشية الحيوية على مساحة سطح أكبر وغالبا لمدة 48 ساعة بدلا من 24 ساعة. كما أنهم يفتقرون إلى التفاصيل الكافية لضمان النجاح4،15،16،43،44،46.
علاوة على ذلك ، يتجنب هذا البروتوكول استخدام المواد الكيميائية الخطرة (مثل الفينول) أو المعدات المتخصصة (مثل مكبر الصوت). لا يتم استخدام بروتوكول استخراج الفينول وكلوروفورم الكلاسيكي47 في هذا النظام لأنه على الرغم من أنه ينتج ما يقرب من 2 أضعاف الحمض النووي الريبي مقارنة بالمجموعة التجارية ، إلا أنه في أيدينا ينتج عنه باستمرار تحلل كامل في الحمض النووي الريبي ، كما تم تقييمه على نظام الرحلان الكهربائي الآلي. بالإضافة إلى ذلك ، يظهر أن استخدام مجموعة أدوات الاستخراج التجارية ينتج عنه بروتوكول سهل الاستخدام ينتج عنه نتائج متسقة15،16،45.
الخطوات الحاسمة
هناك عدد من الخطوات الحاسمة في هذا البروتوكول التي تزيد من احتمالية استخراج الحمض النووي الريبي الذي يمكن تسلسله بنجاح. أولا ، من المهم تلقيح ما لا يقل عن 4 آبار من شريحة الغرفة بنفس السلالة من أجل الحصول على كميات كافية من الحمض النووي الريبي للتسلسل. يتم تجميع الأغشية الحيوية من الآبار الأربعة لاستخراج واحد ، والذي له ميزة إضافية تتمثل في تقليل التباين في تحليل التعبير الجيني النهائي17. غالبا ما يؤدي استخراج الحمض النووي الريبي من أقل من 4 آبار إلى عوائد منخفضة جدا بحيث لا يمكن اكتشافها على نظام القياس الفلوري عالي الحساسية. سيؤدي استخراج الحمض النووي الريبي من 8 آبار أو شريحة كاملة إلى إنتاج كميات أعلى من الحمض النووي الريبي بجودة مماثلة لتلك التي تم الحصول عليها عند استخدام 4 آبار ، ولكن يجب على المرء أن يفكر فيما إذا كان الوقت والجهد والتكلفة الإضافية للاستخراج تستحق زيادة العائد. يؤدي سحب 300 ميكرولتر من OD600 = 0.1 مزرعة مخففة لبذر كل بئر بدلا من 200 ميكرولتر القياسي إلى تحسين استعادة مادة الأغشية الحيوية المكشطة من كل بئر. يعد غسل الأغشية الحيوية برفق مرتين بالماء الخالي من النوكلياز قبل تطبيق كاشف حماية الحمض النووي الريبي أمرا بالغ الأهمية لإزالة أكبر عدد ممكن من الخلايا الميتة و / أو العوالق. يعد استخدام الكاشف الواقي للحمض النووي الريبي أمرا مهما لمنع تدهور الحمض النووي الريبي بسبب الوقت اللازم لمعالجة كل بئر. علاوة على ذلك ، بدلا من الكشط بأطراف الماصة ، نفضل استخدام ملاعق وزن معدنية ذات نهاية مسطحة تلامس مساحة سطح أكبر من طرف الماصة ، وهي صغيرة بما يكفي لتناسب البئر. سيعمل الكشط باستخدام طرف ماصة سعة 1000 ميكرولتر ولكنه أقل كفاءة ويستغرق وقتا أطول لكشط الأغشية الحيوية بالكامل مقارنة بالملعقة المعدنية. في تجربتنا ، فإن استخدام ملاعق معدنية يوفر الوقت والجهد. عند الكشط ، تأكد من وضع شريحة الغرفة أعلى الصفيحة الزجاجية لمنع تكسير قاع الآبار. صوتنة ليست فعالة لأنها تؤدي باستمرار إلى تدهور شديد في الحمض النووي الريبي بسبب قلة حجم العينة وإنتاج الحرارة الزائدة. بعد جمع مادة الأغشية الحيوية ، يتم إدخال العينة في مجموعة تجارية لاستخراج الحمض النووي الريبي الأكثر موثوقية وقابلية للتكرار. أخيرا ، نظرا للمصادر المحتملة للتباين في هذه الطريقة ، من المهم جدا تضمين النسخ المتماثل التجريبي بالإضافة إلى تجميع العينات17،48. يصف هذا البروتوكول النسخ المتماثل البيولوجي ثلاث مرات لكل عينة.
القيود
هناك مجموعة متنوعة من التقنيات المستخدمة لزراعة الأغشية الحيوية للدراسة ، وأكثرها شيوعا هو التكوين في ألواح الميكروتيتر4. هذه الألواح متوفرة بأحجام وأعداد مختلفة من الآبار. تندرج شرائح الغرفة في هذه الفئة. تعد القدرة على استخراج الحمض النووي الريبي النقي السليم أمرا مهما لجميع أنظمة الأغشية الحيوية ، لكن هذا النظام محدود بانخفاض إنتاجية الحمض النووي الريبي. التركيزات منخفضة بما يكفي بحيث يجب تقييم جودتها وكمياتها بواسطة أدوات Qubit و Bioanalyzer باستخدام مجموعات الحمض النووي الريبي عالية الحساسية. يمكن أيضا استخدام قطرة نانوية ، لكن لها حساسية محدودة مقارنة ب Qubit ولا يمكنها التمييز بين الحمض النووي الملوث والحمض النوويالريبي 49. إذا أمكن ، يجب استخدام قطرة نانوية للحصول على نسبA 260 / A280 و A260 / A230 للنقاء. هذه النسب مفيدة ، خاصة وأن جودة الحمض النووي الريبي من نظام الأغشية الحيوية هذا ليست جيدة مثل كميات أكبر من المواد الأولية. ويرجع ذلك على الأرجح إلى انخفاض إنتاجية الحمض النووي الريبي ووجود الخلايا الميتة في الأغشية الحيوية و / أو RNase التي قد تؤدي إلى تحلل الحمض النووي الريبي. قيد آخر لهذه الطريقة هو أنه لا يمكنه فصل أنواع الخلايا المختلفة داخل غشاء حيوي غير متجانس ولكن يمكنه فقط استخراج الخلايا كمجموعة كاملة ، مما قد يحجب التعبير الجيني في أي مجموعات سكانية فرعية ممثلة تمثيلا ناقصا.
التطبيقات المحتملة
تم تحسين البروتوكول المقدم هنا ل P. aeruginosa الأغشية الحيوية من العزلات السريرية المزروعة في شرائح الغرفة. يمكن استخدام هذه الطريقة لدراسة التغيرات في التعبير الجيني أثناء تكوين الأغشية الحيوية في ظل ظروف نمو مختلفة لوحظت بواسطة الفحص المجهري متحد البؤر. يمكن أيضا تحسين البروتوكول للأنواع البكتيرية الأخرى التي تولد الأغشية الحيوية. باستخدام هذه الطريقة ، من الممكن أيضا استخراج الحمض النووي الجيني حيث يمكن استخدام مجموعة أدوات الاستخراج المزدوجة. بهذه الطريقة ، يمكن دراسة التعبير الجيني للأغشية الحيوية البكتيرية في العزلات السريرية المسببة للعدوى لدى المرضى بشكل أكثر دقة ، مما يؤدي إلى نتائج قد توجه استراتيجيات العلاج المستقبلية.
Disclosures
وليس لدى أصحاب البلاغ أي إفصاحات يعلنون عنها.
Acknowledgements
مساهمات المؤلفين: P.W. و YY و V.W شاركت في وضع تصور للدراسة. قام KG و LJ و AMو P.W. بتحسين بروتوكولات المختبر. تم دعم تمويل KG من خلال دعم برنامج التوظيف الطلابي من خلال BioTalent Canada.
Materials
| Name | Company | Catalog Number | Comments |
| Agilent 2100 Bioanalyzer | Agilent | G2939BA | Automated electrophoresis of biomolecules |
| Agilent RNA 6000 pico kit | Agilent | 5067-1513 | High sensitivity RNA electrophoresis chip to generate a RIN |
| DNA/RNA Lysis Buffer | Zymo Research | D7001-1-50 | A guanidinium thiocyanate and N-Lauroylsarcosine-based lysis buffer sold as part of a nucleic acid purification kit |
| DNA/RNA Prep Buffer | Zymo Research | D7010-2-10 | A guanidine HCl and ethanol buffer used for purification of DNA and RNA |
| DNA/RNA Shield | Zymo Research | R1100-50 | DNA and RNA preservation/protection reagent |
| DNA/RNA Wash Buffer | Zymo Research | D7010-3-6 | A salt and ethanol buffer used for purification of DNA and RNA |
| DNBSEQ G-400RS | MGI | G-400RS | High throughput sequencer |
| MGIEasy RNA Directional Library Prep Set | MGI | 1000006386 | Generate libraries for MGI high-throughput sequencing platforms from total RNA. |
| Mini-Beadbeater-96 | BioSpec | 1001 | A high energy, high throughput cell disrupter |
| NEBNext rRNA Depletion Kit (bacteria) | New England Biolabs | E7850X | Efficient and specific depletion of bacterial rRNA (5S, 16S, 23S) |
| Nunc Lab-Tek II chamber slide system | Thermo Fisher Scientific | 154534 | 8-well chamber slide with removable wells |
| Qubit Fluorometer | Thermo Fisher Scientific | Q33238 | Fluorometer for DNA, RNA and proteins |
| Qubit RNA HS Assay Kit | Thermo Fisher Scientific | Q32852 | High sensitivity fluorometric assay to measure RNA concentration |
| Spin-Away Filters | Zymo Research | C1006-50-F | Silica-based spin column primarily used to bind or remove genomic DNA |
| Sterile inoculation loops, 1 uL | Sarstedt | 86.1567.050 | Sterile, disposable inoculation loops for manipulation of microorganisms |
| ZR BashingBead Lysis tubes | Zymo Research | S6003-50 | 2 mL tubes containing 0.1 and 0.5 mm bead lysis matrix for homogenizing biological samples |
| Zymo Spin IIICG Columns | Zymo Research | C1006-50-G | Silica-based spin column for purification of DNA and RNA |
| Zymo-Spin III-HRC Filters | Zymo Research | C1058-50 | Remove inhibitors such as polyphenolic compounds, humic/fulvic acids, tannins, melanin, etc. |
| Zymobiomics DNA/RNA Miniprep kit | Zymo Research | R2002 | DNA and RNA dual extraction kit |
| Zymobiomics HRC Prep solution | Zymo Research | D4300-7-30 | To be used with Zymo-Spin III-HRC Filters to remove PCR inhibitors |
References
- Beaudoin, T., Waters, V. Infections With Biofilm Formation: Selection of Antimicrobials and Role of Prolonged Antibiotic Therapy. The Pediatric Infectious Disease Journal. 35 (6), 695-697 (2016).
- Bjarnsholt, T., et al. The in vivo biofilm. Trends in Microbiology. 21 (9), 466-474 (2013).
- Folsom, J. P., et al. Physiology of Pseudomonas aeruginosa in biofilms as revealed by transcriptome analysis. BMC Microbiology. 10, 294(2010).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43 (3), 313-351 (2017).
- Bjarnsholt, T., Ciofu, O., Molin, S., Givskov, M., Hoiby, N. Applying insights from biofilm biology to drug development - can a new approach be developed. Nature Reviews Drug Discovery. 12 (10), 791-808 (2013).
- Colvin, K. M., et al. The Pel and Psl polysaccharides provide Pseudomonas aeruginosa structural redundancy within the biofilm matrix. Environmental Microbiology. 14 (8), 1913-1928 (2012).
- Hentzer, M., et al. Alginate overproduction affects Pseudomonas aeruginosa biofilm structure and function. Journal of Bacteriology. 183 (18), 5395-5401 (2001).
- Cury, J. A., Koo, H. Extraction and purification of total RNA from Streptococcus mutans biofilms. Analytical Biochemistry. 365 (2), 208-214 (2007).
- Francavilla, M., et al. Extraction, characterization and in vivo neuromodulatory activity of phytosterols from microalga Dunaliella tertiolecta. Current Medicinal Chemistry. 19 (18), 3058-3067 (2012).
- Atshan, S. S., et al. Improved method for the isolation of RNA from bacteria refractory to disruption, including S. aureus producing biofilm. Gene. 494 (2), 219-224 (2012).
- Franca, A., Melo, L. D., Cerca, N. Comparison of RNA extraction methods from biofilm samples of Staphylococcus epidermidis. BMC Research Notes. 4, 572(2011).
- Jurcisek, J. A., Dickson, A. C., Bruggeman, M. E., Bakaletz, L. O. In vitro biofilm formation in an 8-well chamber slide. The Journal of Visusalized Experiments. (47), e2481(2011).
- Beaudoin, T., Kennedy, S., Yau, Y., Waters, V. Visualizing the effects of sputum on biofilm development using a chambered coverglass model. The Journal of Visusalized Experiments. (118), e54819(2016).
- Cockeran, R., et al. Biofilm formation and induction of stress response genes is a common response of several serotypes of the pneumococcus to cigarette smoke condensate. The Journal of Infection. 80 (2), 204-209 (2020).
- Bisht, K., Moore, J. L., Caprioli, R. M., Skaar, E. P., Wakeman, C. A. Impact of temperature-dependent phage expression on Pseudomonas aeruginosa biofilm formation. npj Biofilmsand Microbiomes. 7 (22), (2021).
- Harrison, A., et al. Reprioritization of biofilm metabolism is associated with nutrient adaptation and long-term survival of Haemophilus influenzae. NPJ Biofilms and Microbiomes. 5 (1), 33(2019).
- Sousa, C., Franca, A., Cerca, N. Assessing and reducing sources of gene expression variability in Staphylococcus epidermidis biofilms. BioTechniques. 57, 295-301 (2014).
- Boom, R. Rapid and simple method for purification of nucleic acids. Journal of Clinical Microbiology. 28 (3), 495-503 (1990).
- Coppin, C. Re: How do silica based RNA spin columns only bind RNA and not DNA. , Available from: https://www.researchgate.net/post/How_do_silica_based_RNA_spin_columns_only_bind_RNA_and_not_DNA/60b017bffa5c4151cac1c/citation/download (2021).
- Kennedy, S. A complete guide to how nucleic extraction kits work. , Available from: https://bitesizebio.com/13516/how-dna-extraction-rna-miniprep-kits-work/ (2021).
- Qubit RNA HS Assay Kit User Guide. Thermo Fisher Scientific. , Available from: https://www.thermofisher.com/document-connect/document-connect.html?url=https%3A%2F%2Fassets.thermofisher.com%2FTFS-Assets%2FLSG%2Fmanuals%2FQubit_RNA_HS_Assay_UG.pdf&title=VXNlciBHdWlkZTogUXViaXQgUk5BIEhTIEFzc2F5IEtpdHM (2015).
- Mueller, O., Lightfoot, S., Schroeder, A. RNA Integrity Number (RIN) - Standardization of RNA Quality Control (Application report # 5989-1165EN). , Available from: https://www.agilent.com/cd/library/applications/5989-1165EN.pdf (2016).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 3(2006).
- Culviner, P. H., Guegler, C. K., Laub, M. T. A Simple, Cost-Effective, and Robust Method for rRNA Depletion in RNA-Sequencing Studies. mBio. 11 (2), (2020).
- MGIEasy RNA Directional Library Prep Set User Manual verA2. MGI Tech Co. , Available from: https://en.mgi-tech.com/products/reagents_info/14/ (2020).
- Andrews, S. FastQC: A Quality Control Tool for High Throughput Sequence Data. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina Sequence Data. Bioinformatics. , (2014).
- NCBI Resource Coordinators. Database resources of the National Center for Biotechnology Information. Nucleic Acids Research. 44 (1), (2016).
- Li, H. Aligning sequence reads, clone sequences and assembly contigs with BWA-MEM. arXiv. , (2013).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), (2009).
- Liao, Y., Shi, W. Read trimming is not required for mapping and quantification of RNA-seq reads at the gene level. NAR Genomics and Bioinformatics. 2 (3), (2020).
- Penir, S. Calculating Mapping Statistics from a SAM/BAM file using SAMtools and awk. , Available from: https://sarahpenir.github.io/bioinformatics/awk/calculating-mapping-stats-from-a-bam-file-using-samtools-and-awk/ (2019).
- Haile, S., et al. Evaluation of protocols for rRNA depletion based RNA sequencing of nanogram inputs of mammalian total RNA. PLoS ONE. 14 (10), 0224578(2019).
- Schuierer, S., et al. A comprehensive assessment of RNA-seq protocols for degraded and low-quantity samples. BMC Genomics. 18 (442), (2017).
- Shanker, S., et al. Evaluation of Commercially Available RNA Amplification Kits for RNA Sequencing Using Very Low Input Amounts of Total RNA. Journal of Biomolecular Techniques. 26 (1), (2015).
- Adiconis, X., et al. Comparative analysis of RNA sequencing methods for degraded or low-input samples. Nature Methods. 10, 623-629 (2013).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (13), (2016).
- Illumina, Inc. Coverage depth recommendations. , Available from: https://www.illumina.com/science/technology/next-generation-sequencing/plan-experiments/coverage.html (2021).
- What is a good sequencing depth for bulk RNA-Seq. ECSEQ Bioinformatics. , Available from: https://www.ecseq.com/support/ngs/what-is-a-good-sequencing-death-for-bulk-rna-seq (2019).
- Bedre, R. Sequencing coverage and breadth of coverage. , Available from: https://www.reneshbedre.com/blog/sequencing-coverage.html (2021).
- Dotsch, A., et al. The Pseudomonas aeruginosa transcriptome in planktonic cultures and static biofilms using RNA sequencing. PLoS One. 7 (2), 31092(2012).
- Chen, Y., et al. Population dynamics and transcriptomic responses of Pseudomonas aeruginosa in a complex laboratory microbial community. npj Biofilms and Microbiomes. 5 (1), (2019).
- Thoming, J. G., et al. Parallel evolutionary paths to produce more than one Pseudomonas aeruginosa biofilm phenotype. NPJ Biofilms and Microbiomes. 6, 2(2020).
- Soares, A., et al. Understanding ciprofloxacin failure in Pseudomonas aeruginosa biofilm: persister cells survive matrix disruption. Frontiers in Microbiology. 10, 2603(2019).
- Whiteley, M., et al. Gene expression in Pseudomonas aeruginosa biofilms. Nature. 413, (2001).
- Chomczynski, P., Sacchi, N. The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: twenty-something years on. Nature Protocols. 1, (2006).
- Liu, Y., Zhou, J., White, K. P. RNA-seq differential expression studies: more sequence or more replication. Bioinformatics. 30 (3), (2014).
- Wieczorek, D., Delauriere, L., Schagat, T. Methods of RNA Quality Assessment. , Available from: https://www.promega.ca/resources/pubhub/methods-of-rna-quality-assessment/ (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved