Method Article
בידוד של RNA כולל מ-Pseudomonas aeruginosa בתוך ביופילמים למדידת ביטוי גנים
In This Article
Summary
פרוטוקול זה מציג שיטה לבידוד RNA מביופילמים של Pseudomonas aeruginosa הגדלים בשקופיות תא לריצוף תפוקה גבוהה.
Abstract
Pseudomonas aeruginosa הוא פתוגן חיידקי אופורטוניסטי הגורם לזיהומים בדרכי הנשימה של חולי סיסטיק פיברוזיס (CF). P. aeruginosa ידוע ביכולתו ליצור ביופילמים המוגנים על ידי מטריצה של אקסופוליסכרידים. מטריצה זו מאפשרת למיקרואורגניזמים להיות עמידים יותר בפני גורמים חיצוניים, כולל טיפול אנטיביוטי. אחת השיטות הנפוצות ביותר לגידול ביופילם למחקר היא בלוחות מיקרוטיטר או בשקופיות תאיות. היתרון של מערכות אלו הוא בכך שהן מאפשרות בדיקה של תנאי גידול מרובים, אך החיסרון שלהן הוא שהן מייצרות כמויות מוגבלות של ביופילם למיצוי RNA. מטרת מאמר זה היא לספק פרוטוקול מפורט, צעד אחר צעד, כיצד לחלץ RNA כולל מכמויות קטנות של ביופילם באיכות ובכמות מספיקות לריצוף תפוקה גבוהה. פרוטוקול זה מאפשר לחקור את ביטוי הגנים בתוך מערכות ביופילם אלה.
Introduction
רוב הזיהומים החיידקיים הכרוניים, כגון זיהומים ריאתיים בחולי סיסטיק פיברוזיס (CF) וזיהומים הקשורים לתותבות, מאופיינים בגדילה של אורגניזמים בתוך ביופילמים. ביופילמים1 הם קהילות של חיידקים עטופים במטריצה המורכבת בעיקר מפוליסכרידים2. חיידקים בתוך ביופילמים יכולים להיות איטיים, רדומים מבחינה מטבולית ובתנאים אנאירוביים והיפוקסיים. ביופילמים עמידים יותר לאנטיביוטיקה בשל גורמים כמו ירידה בחדירת אנטיביוטיקה, ביטוי מוגבר של משאבות פליטת תרופות וירידה בחלוקת התאים3. מסיבות אלה ואחרות, הם מעוררים עניין מחקרי רב.
על מנת לחקור במדויק זיהומים מתמשכים כגון זיהומים כרוניים של Pseudomonas aeruginosa בחולי CF, תנאי הגידול הנראים עם היווצרות ביופילם צריכים להשתקף במדויק במבחנה. שיטה נפוצה בעלת תפוקה גבוהה היא לגדל אותם בשקופיות תא או בלוחות מיקרוטיטר ולנטר את היווצרות הביופילם על ידי מיקרוסקופיה קונפוקלית4. ידוע כי מווסת מפתח במעבר מפלנקטוני, או צף חופשי, לאורח חיים חיידקי ביופילם הוא השליח המשני, cyclic-di-GMP5. רמות מחזוריות מוגברות של GMP מגבירות את הביטוי של גנים ספציפיים המקדמים צמיחת ביופילם. רנ"א רגולטורי קטן שאינו מקודד וחישת מניין ממלאים גם תפקידים חשובים בוויסות היווצרות ביופילם5. מדידת ביטוי גנים של ביופילם על ידי ריצוף RNA חיידקי שחולץ יכולה להיות מאתגרת. P. aeruginosa, למשל, מייצר שלושה אקסופוליסכרידים (Psl, Pel ואלגינט), המיוצרים בכמויות משמעותיות בביופילמים 6,7. פוליסכרידים אלה יכולים להפריע למיצוי וטיהור RNA ולהוביל לתכשירים לא טהורים המכילים רמות נמוכות של mRNA8 חיידקי. ערכות מיצוי RNA זמינות מסחרית מסוגלות לייצר RNA באיכות גבוהה מתרביות חיידקים פלנקטוניות, אך עשויות שלא לעבוד טוב עם תרביות ביופילם 9,10,11. ישנן כמה ערכות מיצוי רנ"א מסחריות שמתיימרות לעבוד עבור ביופילמים, שאחת מהן אנו משתמשים בשיטה זו.
בכתב יד זה, אנו מתארים את ההליכים לגידול ביופילמים של P. aeruginosa בשקופיות תא וחילוץ mRNA חיידקי לריצוף תפוקה גבוהה12,13. תוך שימוש בבידוד קליני שנאסף מדגימות כיח מחולי CF, אנו מדגימים כי ניתן להשתמש בשיטות אלה עבור מבודדים בעלי מאפייני גדילה משתנים. בהשוואה לפרסומים קודמים, פרוטוקול זה מתואר בפירוט כדי לאפשר הצלחה טובה יותר בחקר ביטוי גנים של ביופילם חיידקי 11,14,15,16.
Protocol
מועצת האתיקה של המחקר (REB) נדרשת לאיסוף ועיבוד דגימות כיח מנבדקים אנושיים. מחקר זה אושר על ידי בית החולים לילדים חולים (REB#1000019444). מועצת האתיקה של המחקר (REB) נדרשת לאסוף ולאחסן דגימות ליחה מנבדקים אנושיים. מחקרים אלה אושרו על ידי בית החולים לילדים חולים REB#1000058579.
1. היווצרות ביופילם
- לגדל מבודדי Pseudomonas aeruginosa שהתקבלו מדגימות כיח של חולי CF ששימשו במחקר זה על צלחות אגר מרק לוריא (LB) באינקובטור של 37 מעלות צלזיוס למשך הלילה.
הערה: טכניקת פסים נכונה חשובה להשגת מושבות חיידקים בודדות. פסים תוך כדי סיבוב הצלחת ידללו את תאי החיידקים מספיק כדי שמושבות בודדות יוכלו לגדול.- פס חיידקים באמצעות לולאת חיסון בתבנית זיגזג בקצה העליון של צלחת LB טרייה עד לכסות כ-1/4 מהצלחת.
- סובב את הצלחת בערך 60°. קח לולאת חיסון חדשה והעביר אותה פעם אחת דרך האזור המפוספס לאזור שני ונקי של הצלחת, תוך חזרה על דפוס הזיגזג.
- חזור על שלב 1.1.2 עם לולאה טרייה לאזור שלישי של הצלחת. החזר את המכסה והפוך את הצלחת בעת הנחתו בחממה.
- השתמש בלולאת חיסון סטרילית כדי לבחור מושבת חיידקים בודדת מצלחת אגר (המכילה זן חיידקי יחיד) ולחסן צינור תרבית מלא ב-5 מ"ל של חומר LB סטרילי. באמצעות לולאה חדשה בכל פעם, חסנו שני צינורות תרבית נוספים מלאים ב-5 מ"ל של מדיה LB מאותה צלחת.
- חזור על אותו הליך חיסון עבור זני החיידקים השונים. יש לגדל את התרביות למשך הלילה בטמפרטורה של 37 מעלות צלזיוס עם ניעור בטמפרטורה של 220 סל"ד.
הערה: כל צלחת אגר המכילה זן חיידקי בודד משמשת לחיסון 3 צינורות תרבית עצמאיים. שלושת הצינורות מייצגים שכפולים ביולוגיים בשלושה עותקים עבור זן אחד ומטופלים כדגימות נפרדות. זה שונה משכפולים טכניים שהיו כרוכים בחילוץ RNA 3 פעמים מצינור תרבית יחיד. - הכן דילול של 1:100 של תרביות הלילה על ידי העברת 50 מיקרוליטר של תרבית לילה לתוך צינור תרבית חדש המכיל 4.95 מ"ל של מדיה חדשה של LB.
- גדל את התרביות המדוללות למשך 3 שעות נוספות ב-37 מעלות צלזיוס עם ניעור ב-220 סל"ד או עד שה-OD600 הוא 0.1 ומעלה. מדוד את צפיפות התאים בספקטרופוטומטר ב-OD600.
- בצינור מיקרופוגה חדש של 1.7 מ"ל, כוונן את ה-OD600 ל-0.1 (שלב יומן מוקדם) בנפח כולל של 1.5 מ"ל עם LB טרי.
- מערבבים בעדינות על ידי היפוך. העבירו 300 מיקרוליטר מכל תרבית מותאמת ל-4 בארות של מגלשת תא של 8 בארות כדי להגיע ל-2 דגימות שונות לכל שקופית (איור 1).
- הנח את המגלשות, ללא הפרעה, באינקובטור של 37 מעלות צלזיוס למשך הלילה למשך 24 שעות. כדי למנוע אידוי, הניחו את השקופיות על גבי מגבת נייר לחה במגש פלסטיק קטן.
2. שחזור ביופילם
הערה: כל מגלשת זכוכית מכילה שמונה בארות נפרדות. דגימה אחת מורכבת מארבע בארות עם ביופילמים שיאוגדו17. פרוטוקול מיצוי זה מיועד לדגימה אחת (4 בארות) שבה הביופילמים נשאבים מ-2 בארות בכל פעם. מיצוי RNA מבוצע באמצעות ערכת מיצוי RNA מסחרית הכוללת שלב הכאת חרוזים וניקוי מבוסס עמודות, עם שינויים. עקוב אחר הוראות היצרן להכנת ריאגנטים.
- במכסה זרימה למינרית, הסר לאט את המדיה מ-2 מתוך 4 בארות באמצעות קצה פיפטה. הטה את המגלשה בזווית של 45 מעלות והוציא את המדיה החוצה מהפינה התחתונה של הבארות כדי למנוע ניתוק של הביופילמים.
- תוך שמירה על המגלשה מוטה, שטפו את תאי הפלנקטון על ידי פיפטינג עדין של 300 מיקרוליטר מים נטולי RNase לפינה התחתונה של שתי הבארות המרוקנות. הסר את המים על ידי פיפטינג בעדינות החוצה, כמתואר בשלב 2.1. חזור על שלב הכביסה והסר כמה שיותר מים.
- הוסף 300 מיקרוליטר של מגיב הגנת RNA (ראה טבלת חומרים) לכל אחת משתי הבארות המרוקנות עם ביופילמים בבסיסן. הנח את מגלשת התא על צלחת זכוכית כדי למנוע את שבירת הבארות, ולאחר מכן גרד את הביופילמים ב-2 הבארות בעזרת מרית מתכת סטרילית קטנה ונטולת נוקלאז כדי להשעות מחדש את חיידקי הביופילם. הניחו לשבת עד שהביופילמים יתאוששו מ-2 הבארות הנותרות שלא נגעו בהן מאותה דגימה.
הערה: תוספת של מגיב הגנת RNA מבטיחה את יציבות דגימות הביופילם בבארות המגורדות בטמפרטורת הסביבה תוך עיבוד שתי הבארות הנותרות של אותה דגימה. מגיב הגנת ה-RNA משבית תאים ומנטרל נוקלאזות וגורמים זיהומיים, וכתוצאה מכך משמר את ה-RNA. - כדי לשחזר ביופילמים מ-2 הבארות הנותרות עבור דגימה (תזכורת: דגימה אחת מורכבת מ-4 בארות), הסר את מדיית ה-LB מ-2 הבארות החדשות הנותרות באותו אופן כמתואר בשלב 2.1. חזור על שלב 2.2 כדי לשטוף את התאים הפלנקטוניים במים נטולי RNase משתי הבארות החדשות, כמו קודם.
- חזור לשתי הבארות הראשונות עם ביופילמים מגורדים במגיב הגנה, שנוצר בסוף שלב 2.3, ולאט לאט לערבב את 300 מיקרוליטר של ביופילם מושעה מחדש מבאר אחת מגורדת, בניסיון לא ליצור יותר מדי בועות.
- העבירו את כל התכולה מהבאר לאחת הבארות שנשטפו לאחרונה והתרוקנו. מערבבים ומעבירים 300 מיקרוליטר של ביופילם מושעה מחדש מהבאר השנייה שנגרדה בבאר הנותרת, שנשטפה לאחרונה, מרוקנת.
הערה: במקום להוסיף מגיב הגנת RNA חדש ל-2 הבארות שנשטפו לאחרונה עם ביופילמים, העבירו את הביופילמים שכבר גרדו בעבר לבארות הטריות הללו. זה ישמור על נפח הדגימה המשולב נמוך מספיק כדי לעמוד בדרישות הקלט עבור ערכת מיצוי ה-RNA המסחרית. ראה איור 1 לסכמה.
- העבירו את כל התכולה מהבאר לאחת הבארות שנשטפו לאחרונה והתרוקנו. מערבבים ומעבירים 300 מיקרוליטר של ביופילם מושעה מחדש מהבאר השנייה שנגרדה בבאר הנותרת, שנשטפה לאחרונה, מרוקנת.
- חזור על גירוד הביופילמים בבארות החדשות כמו בשלב 2.3, על ידי הנחת מגלשת התא על צלחת זכוכית וגירוד הביופילמים ב-2 הבארות החדשות עם מרית מתכת סטרילית קטנה ונטולת נוקלאז כדי להשעות מחדש את חיידקי הביופילם.
- שלב את כל הביופילם המושעה מחדש מ-2 הבארות החדשות לתוך צינור מיקרו-צנטריפוגה יחיד נטול RNase וקשר נמוך בנפח 1.5 מ"ל. מדוד את עוצמת הקול, שצריכה להיות ~500 - 600 מיקרוליטר בסך הכל.
הערה: תרחיף הביופילם המשולב מזוג הבארות החדשות השני הזה יכיל את כל חומר הביופילם מ-4 הבארות המקוריות של דגימה.
3. בידוד RNA כולל והערכת איכות
הערה: מיצוי RNA מתבצע באמצעות ערכת מיצוי RNA מסחרית המתיימרת לעבוד על ביופילמים. הרכיבים הבודדים כלולים בטבלת החומרים, במידת האפשר. הסברים על המנגנונים מאחורי כל שלב טיהור ניתנים במידת האפשר.
- הוסף מספיק ריאגנט להגנת RNA כדי לסכם 750 מיקרוליטר בצינור. העבירו את כל הנפח לצינור ליזה מכה חרוזים של 2 מ"ל המכיל חרוזים של 0.1 ו-0.5 מ"מ (ראה טבלת חומרים). מקציפים 2 1/2 דקות במקצף חרוזים במהירות מרבית.
הערה: השילוב של חרוזים בצפיפות גבוהה של 0.1 מ"מ ו-0.5 מ"מ בתוספת ערבוב מהיר על מקצף חרוזים מבטיח הומוגניזציה יסודית של דפנות התאים המיקרוביאליים. - צנטריפוגה ב 16,000 x גרם למשך דקה אחת לכדור החרוזים. העבירו את הסופרנטנט לצינור מיקרו-צנטריפוגה חדש, תוך מזעור העברת כל החרוזים, מה שיקל על שלב 3.3. מדוד את עוצמת הקול.
- הוסף נפח שווה של מאגר ליזה RNA (~450 מיקרוליטר) וערבב היטב. העבירו עד 800 מיקרוליטר של דגימה, הימנעו מהעברת חרוזים כלשהם, לעמוד סיליקה בצינור איסוף וצנטריפוגה ב-16,000 x גרם למשך 30 שניות. שמור את הזרימה מכיוון שהיא מכילה את ה-RNA בזמן שה-DNA קשור לעמודה.
הערה: מאגר הליזה של RNA מכיל גואנידיניום תיוציאנט ואת חומר הניקוי N-lauroylsarcosine לתאי ליז. גואנידיניום תיוציאנט הוא חומר כאוטרופי שגם משבית נוקלאזות, ובנוכחות סיליקה, שנמצא בעמוד הספין, מקדם את קשירת ה- DNA לסיליקה18. היעדר אתנול מאפשר קשירה מועדפת של DNA ולא RNA לעמודת ספין הסיליקה19. מטרת שלב 3.3 היא לקשור ולהסיר DNA גנומי. אנו רוצים לשמור על ה-RNA, הכלול בחלק הזרימה. - אם נותרה דגימה נוספת, העבירו את העמודה לצינור איסוף חדש וטענו מחדש עם שאר הדגימה. צנטריפוגה ב-16,000 x גרם למשך 30 שניות. שמור על הזרימה עם ה-RNA ושלב עם ה-aliquot הראשון.
- מדוד את נפח הזרימה המשולב והוסף נפח שווה של 100% אתנול וערבב היטב. העבירו עד 800 מיקרוליטר מהתמיסה לעמודת ספין סיליקה שנייה חדשה בצינור איסוף וצנטריפוגה ב-16,000 x גרם למשך 30 שניות. השליכו את הזרימה.
הערה: הוספת אתנול לתמיסת המלח הכאוטרופית המכילה את ה-RNA משפרת את קשירת ה-RNA לעמודת ספין הסיליקה19. - עבור פתרונות > 800 μL, טען מחדש את עמודת הספין והצנטריפוגה עד לסיבוב התמיסה כולה. השליכו את הזרימה לאחר כל סיבוב.
- הוסף 400 מיקרוליטר של מאגר כביסה לעמוד ולצנטריפוגה ב-16,000 x גרם למשך 30 שניות כדי להסיר חלק מהמלחים. השליכו את הזרימה.
- הכן את תערובת התגובה של DNase I בהתאם להוראות היצרן ובצע את הטיפול ב-DNase בעמודה כדי להסיר שאריות DNA.
- השעו מחדש את ה-DNase I הליופילי ב-275 מיקרוליטר של מים נטולי RNase כדי ליצור תמיסה של 1 U/μL. מערבבים על ידי היפוך עדין.
- שלב 5 מיקרוליטר של DNase I מדולל עם 75 מיקרוליטר של מאגר העיכול של DNase המסופק. מערבבים בעדינות על ידי היפוך.
- הוסף 80 מיקרוליטר מהתמיסה המוכנה ישירות על מטריצת העמודה. דגירה בטמפרטורת החדר למשך 20 דקות.
- הוסף 400 מיקרוליטר של מאגר הכנת RNA לעמודה ולצנטריפוגה ב-16,000 x g למשך 30 שניות. השליכו את הזרימה.
- הוסף 700 מיקרוליטר של מאגר שטיפת RNA לעמודה וחזור על הצנטריפוגה. השליכו את הזרימה.
- הוסף 400 מיקרוליטר של מאגר שטיפת RNA וצנטריפוגה את העמודה למשך 2 דקות כדי להסיר לחלוטין כל מאגר שיורי.
הערה: ישנם 2 שלבי כביסה שונים להסרת זיהומים על העמוד. מאגר ההכנה מכיל מלח כאוטרופי חלש מעורבב עם אתנול על מנת להסיר שאריות חלבונים. לאחר מכן, מאגר הכביסה משמש לביצוע שטיפות אתנול להסרת מלחים. יש להסיר את כל האתנול שנותר כדי לאפשר פליטה יעילה של ה-RNA19,20. - העבירו בזהירות את העמוד לצינור מיקרו-צנטריפוגה חדש בעל קשירה נמוכה.
- הוסף 50 מיקרוליטר מים נטולי RNase ישירות למטריצת העמודה ודגירה למשך 5 דקות. צנטריפוגה ב-16,000 x גרם למשך דקה אחת כדי לחסל את ה-RNA.
- לניקוי נוסף להסרת מעכבי PCR, הנח מסנן מעכב PCR בצינור איסוף חדש. הוסף 600 מיקרוליטר מתמיסת ההכנה למעכב המסופקת (ראה טבלת חומרים).
- צנטריפוגה ב-8,000 x גרם למשך 3 דקות לשטיפת המסנן. העבירו את המסנן השטוף לצינור מיקרו-צנטריפוגה חדש בעל קשירה נמוכה.
- העבירו את ה-RNA המנומק משלב 3.13 למסנן השטוף, וצנטריפוגה ב-16,000 x גרם למשך 3 דקות.
הערה: ניתן להשתמש ב-RNA באופן מיידי או לאחסן בטמפרטורה של -80 מעלות צלזיוס. - קבע את ריכוז ה-RNA באמצעות מערכת פלואורומטרית ברגישות גבוהה21.
הערה: מערכות אלו מאפשרות כימות רגיש של כמות קטנה של RNA בתמיסה הספציפית ליעד המבוקש.- העריכו את איכות ה-RNA באמצעות מערכת אלקטרופורזה אוטומטית שיכולה לספק RIN (מספר שלמות RNA), שהוא מדד לאיכות RNA 22,23.
4. דלדול RNA ריבוזומלי וריצוף תפוקה גבוהה
- הגש RNA למרכז לניתוח אבולוציה ותפקוד גנום (CAGEF) במרכז הגנום באוניברסיטת טורונטו (טורונטו, קנדה) (https://www.cagef.utoronto.ca/) לדלדול rRNA חיידקי והכנת ספריית RNA כיוונית RNA (ראה טבלת חומרים).
הערה: דלדול rRNA חיידקי מכוון ל-5S, 16S ו-23S rRNAs להסרה24. - לדלל rRNA באמצעות ערכת דלדול חיידקי rRNA מסחרית. עקוב אחר הפרוטוקול להזנת 10 ננוגרם - 1 מיקרוגרם RNA כולל שלם או מפורק חלקית.
- בנה ספריות ריצוף RNA באמצעות ערכת הכנת ספריית RNA כיוונית עם אינדקסים שונים המצורפים לכל ספרייה.
- אסוף כמויות שוות ערך של כל ספריית RNA ובצע רצף תפוקה גבוה עם קריאות קצה זוגיות של 100 בסיסים25.
5. הערכת איכות של קריאות רצף
הערה: בדוק את איכות קריאות הרצף באמצעות התוכנית הזמינה בחינם, FastQC26, הזמינה דרך פלטפורמת הקוד הפתוח החינמית, Galaxy27.
- עבור אל https://usegalaxy.org/. לחץ על תפריט התחברות או הרשמה והיכנס עם אישורים או צור חשבון.
- לחץ על הקישור העלה נתונים בפינה השמאלית העליונה של הדף, תחת תפריט כלים, והעלה את קבצי הרצף fastq.gz. המתן עד ששמות הקבצים יופיעו בצד ימין של הדף, תחת החלונית History.
- בחר בקרת איכות FASTQ בתפריט כלים כדי לחשוף רשימה של תוכניות. בחר FastQC, שיאכלס את הפאנל האמצעי של המסך.
- תחת נתוני קריאה קצרים מההיסטוריה הנוכחית שלך, בחר את קבצי fastq.gz שהועלו מהתפריט הנפתח.
- בחר Execute כדי להפעיל את התוכנית.
- הצג את התוצאות בחלונית History (טבלה 2).
הערה: להוראות מפורטות יותר על אופן השימוש ב-Galaxy, בקרו בדף התמיכה בכתובת https://galaxyproject.org/support/.
6. מיפוי קריאות רצף
הערה: רשום צינור בסיסי לחיתוך מתאם ומיפוי קריאה עבור נתוני RNA-seq. רצפי המתאמים נחתכים מהקריאות באמצעות Trimmomatic28. הקריאות החתוכות ממופות לגנום הייחוס P. aeruginosa PAO1 (NC_002516.2), שהתקבל מ-NCBI (https://www.ncbi.nlm.nih.gov/)29באמצעות BWA30 ו-Samtools31. לשם הפשטות, זוג קריאות נקרא PA_1.fq ו-PA_2.fq; קובץ הקריאה של המתאם שיש לחתוך נקרא adapter.fa; ורצף הייחוס PAO1 נקרא PAO1.fasta. כל הכלים הם קוד פתוח ופועלים בסביבת UNIX/LINUX. מומלץ בחום להכיר את היסודות של UNIX/LINUX על מנת לבצע פקודות אלה.
- פתח חלון ב-UNIX/LINUX.
- התקן את Java, Trimmomatic, BWA ו-Samtools.
- נווט אל התיקיה שבה נמצא trimmomatic-0.39.jar הקובץ.
- חתוך את כל רצפי המתאמים מהקריאות על-ידי הקלדת הפקודה:
Java -jar PA_1.fq PA_2.fq PA_1_paired.fq PA_1_unpaired.fq PA_2_paired.fq PA_2_unpaired.fq ILLUMINACLIP: adapters.fa
הערה: רק רצפי מתאמים מוסרים. הקריאות לא קוצצו לאיכות32. - העבר את קובץ ההפניה PAO1.fasta לאותה תיקיה.
- צור אינדקס של ההפניה באמצעות BWA עם הפקודה:
מדד Bwa PA01.fasta - מפה את הקריאות הזוגיות לגנום הייחוס על ידי הקלדת 4 הפקודות הבאות. הקלד כל פקודה לאחר סיום הפקודה הקודמת.
Bwa -mem PA01.fasta PA_1_paired.fq PA2_2_paired.fq > PA_R1R2_map.mem.sam
Samtools view -S -b PA_R1R2_map.mem.sam > PA_R1R2.bam
Samtools sort PA_R1R2.bam -o PA_R1R2_sorted.bam
אינדקס Samtools PA288_Rep1_R1R2_sorted.bam - הצג את סטטיסטיקת המיפוי על-ידי הקלדת הפקודה:
Samtools flagstat PA_R1R2_sorted.bam
הערה: השורההשלישית של הסטטיסטיקה מדווחת על שיעור הקריאות הממופות לגנום הייחוס. - חשב את עומק הקריאה הממוצע ואת רוחב הכיסוי עם 2 הפקודות הבאות33, בהתאמה:
Samtools עומק -A PA_R1R2_sorted.bam | awk '{C++; s+=$3}END{print s/c}'
Samtools עומק -A PA288_Rep1_R1R2_sorted.bam | awk '{c++; if($3>0) total+=1}END{print (total/c)*100}'
תוצאות
הסקירה הכללית של השיטה מוצגת באיור 1. בעבר השתמשנו במגלשות עם 8 בארות כדי לגדל ביופילמים של P. aeruginosa ולחשוף אותם לאנטיביוטיקה לפני שבדקנו אותם באמצעות מיקרוסקופיה קונפוקלית בנקודות זמן שונות12,13. ניתן להשתמש בשיטה זו כדי לחלץ את סך ה-RNA ישירות מביופילמים הגדלים במערכת זו על מנת לחקור שינויים בביטוי גנים לאחר הטיפול. פרוטוקול זה עבר אופטימיזציה עבור P. aeruginosa, אך ניתן להתאים אותו בקלות למיני חיידקים אחרים.
זה יכול להיות מאתגר לחלץ כמות מספקת של RNA באיכות טובה מכמויות קטנות של ביופילם לדלדול rRNA וריצוף תפוקה גבוהה. באמצעות פרוטוקול זה, ה-RNA הכולל מופק בהצלחה מ-17 מבודדי ביופילם שונים של P. aeruginosa בשלוש עותקים, בסך הכל 51 דגימות נפרדות. כמויות של RNA שחולצו המייצגות תשואות גבוהות ונמוכות מוצגות בטבלה 1. ריכוזי ה-RNA נעים בין 3.4 ננוגרם/מיקרוליטר (הנמוך ביותר) ל-49.6 ננוגרם/מיקרוליטר (הגבוה ביותר), עם ריכוז ממוצע של 14 ננוגרם/מיקרוליטר וחציון של 13.7 ננוגרם/מיקרוליטר. ריכוזי RNA שלמים מתחת ל-10 ננוגרם/מיקרוליטר נחשבים לדגימות בשפע נמוך לדלדול rRNA ולריצוף הדור הבא, אך דגימות RNA בשפע נמוך מביופילמים עלולות לגרום לנתוני ריצוף באיכות ירודה עוד יותר בהשוואה לדגימות מרוכזות יותר. 35,36,37. איכויות ה-RNA מוצגות בטבלה 1 על ידי ה-RIN ובאיור 2 על ידי אלקטרופרוגרמות ה-RNA המתאימות עבור הדגימות המרוכזות הנמוכות (PA565-3) והגבוהות (PA288-1); שתי הדגימות הנותרות (PA375-3 ו-PA921-1) מייצגות את רוב הדגימות. כפי שניתן לראות באיור 2, רנ"א שמופק מכמויות אלה של ביופילם תמיד מכיל רנ"א מפורק, מה שמשפיע על ערכי ה-RIN שלהם. לכן, אישור חזותי של שיאי ה-rRNA הפרוקריוטיים 16S ו-23S משמש לקביעת איכות ה-RNA כאשר לא מדווח על RIN. באמצעות קריטריונים אלה, כל 51 דגימות ה-RNA נבחרות להמשיך לדלדול וריצוף rRNA, עם תחזית שהמדגם באיכות הירודה ביותר, PA565-3, עלול להיכשל. מתוך דגימות ה-RNA שהוגשו לריצוף, ספריות מוצלחות נוצרות ומרוצפות עבור 49 דגימות, כאשר PA565-3 נכשל, כצפוי.
מספר קריאות הריצוף שנוצרו עבור דגימת הריכוז הגבוה עם RIN טוב (PA288-1) והדגימה בריכוז נמוך יותר ללא RIN (PA375-3) מפורטים בטבלה 2. הסטטיסטיקה הבסיסית כוללת את המספר הכולל של הקריאות, אורך הקריאה ותוכן ה-GC. הנתונים הסטטיסטיים המסכמים של שתי הדגימות מציגים מספר גבוה של קריאות שנוצרו ומצביעים על כך שאף אחד מהם לא סומן כבאיכות ירודה, מה שמרמז על נתוני ריצוף טובים. מספר הקריאות הממוצע לדגימה הוא כ-48 מיליון, מה שנחשב לתשואה טובה. כלי סטנדרטי המשמש להערכת איכות נתוני הריצוף הגולמיים הוא FastQC26. תוכנית זו משמשת לביצוע בדיקות בקרת איכות על קבצי הריצוף הגולמיים כדי לקבוע אם האיכות מספיקה לניתוחים נוספים, או אם יש בעיות או הטיות בנתונים שנוצרו על ידי הרצף עצמו או מספריות ה-RNA הקלט. איור 3 מציג את מדדי בקרת האיכות עבור נתוני ריצוף PA288-1 ו-PA375-3, המייצגים דגימת RNA באיכות גבוהה ודגימת RNA טיפוסית באיכות נמוכה יותר, בהתאמה. אחת העלילות האינפורמטיביות יותר מ-FastQC היא עלילת איכות רצף הבסיס. נתוני ריצוף טובים יציגו ציון איכות חציוני גבוה (>30) עבור כל מיקום בכל הקריאות, עם ירידה בציון האיכות הממוצע לאורך הקריאה. ציון איכות של 30 מייצג שיעור שגיאה של 1 ל-1000, המתאים לדיוק קריאת בסיס של 99.9%. לרוב המכריע של הבסיסים באיור 3 יש ציוני איכות ממוצעים ≥ 35 לאורך כל אורך הקריאה עבור שני המדגמים, המעידים על נתוני ריצוף באיכות טובה במיוחד. זה מספק ראיות חזקות לכך שפרוטוקול מיצוי ה-RNA המוצג כאן מוצלח.
כדי להראות שהשיטה שלנו יכולה לשחזר תעתיקי P. aeruginosa, קריאות הריצוף האיכותיות עבור PA288-1 ו-PA375-3 ממופות לגנום הייחוס של P. aeruginosa PAO1 (NCBI NC_002516.2). הקריאות נחתכות רק למתאם רצף ולא לאיכות, תוך שמירה על כל הבסיסים לפני מיפוי32. סטטיסטיקות המיפוי מוצגות בטבלה 3. אחוז הקריאות הממופות הוא מדד חשוב לדיוק הרצף. במילים פשוטות, ככל שיותר קריאות מתיישרות לרצף ההתייחסות שלך כך ייטב. עבור PA288-1 ו-PA375-3, 84% ו-91% מהקריאות, בהתאמה, ממופות לגנום הייחוס. הטווח הצפוי למיפוי קריאות RNA-seq סטנדרטיות נופל בין 70 - 90%, כך שערכים אלה טובים מאוד, במיוחד כאשר בסיסים באיכות ירודה אינם מוסרים38. עומק הקריאה הממוצע הוא אינדיקטור טוב למספר הממוצע של קריאות המתיישרות בכל מיקום בסיס ברצף הייחוס. ככל שהעומק גבוה יותר בכל עמדת בסיס, כך קריאת הבסיס מדויקת יותר בכל עמדה. הוא מחושב על ידי חלוקת סכום עומק הקריאה הממופה בכל מיקום בגנום הייחוס במספר הכולל של בסיסים בהפניה. ל-PA288-1 ו-PA375-3 יש עומק קריאה ממוצע של 400 ומעלה, וזה טוב לניתוח ביטוי גנים במורד הזרם39,40. רוחב הכיסוי אומר את אחוז האורך של גנום הייחוס המכוסה על ידי ריצוף41. הקריאות המיושרות הן מ-PA288-1 והן מ-PA375-3 מכסות 96% מגנום הייחוס של P. aeruginosa PAO1. זה מצביע על כך שרוב הגנום של P. aeruginosa מיוצג בנתוני הריצוף ולא רק בקטעים קצרים. סטטיסטיקת המיפוי עבור PA288-1 ו-PA375-3 מראה ששיטה זו יכולה לשחזר תעתיקים המתאימים לכיסוי והפצה טובים לגנום P. aeruginosa, ולתמוך עוד יותר בפרוטוקול מיצוי מוצלח.
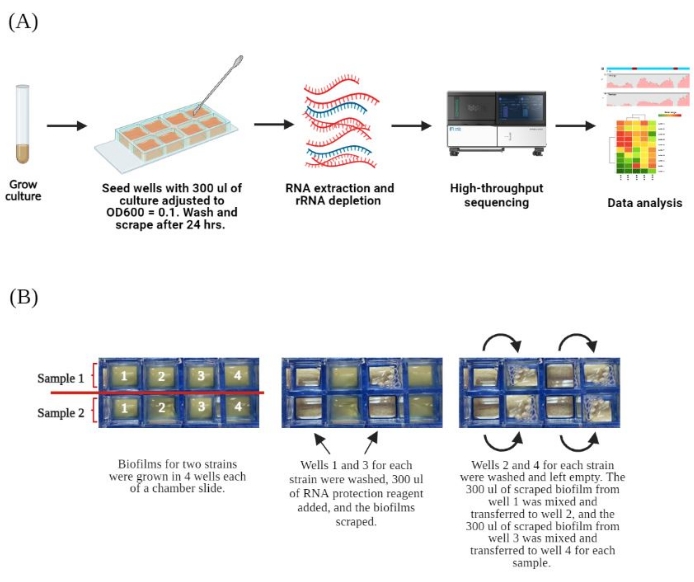
איור 1. סקירה כללית של הפרוטוקול. ת. סכמטי של זרימת עבודה ניסיונית. תרביות פלנקטוניות גודלו בן לילה בטמפרטורה של 37 מעלות צלזיוס, דוללו ב-1:100 במצע טרי למחרת וגודלו במשך 3 שעות נוספות. התרביות הותאמו ל-OD600 של 0.1 ו-300 מיקרוליטר שחוסנו ל-4 בארות של מגלשה בת 8 חדרים ליצירת ביופילמים. לאחר 24 שעות, הביופילמים נשטפו כדי להסיר תאים פלנקטוניים; נוסף ריאגנט להגנת RNA, ותאים נגרדו מהבארות. סך ה-RNA חולץ, התרוקן מ-RNA ריבוזומלי ורוצף. ב. זרימת עבודה מפורטת של גידול ביופילם והסרה ממגלשה בת 8 חדרים. שני זנים גדלו בכל מגלשה בכיוון המוצג משמאל. מוצג הסידור של כל דגימה בארבע בארות של השקופית. עבור כל דגימה עצמאית, בארות 1 ו-3 עובדו תחילה, והסתיימו ב-300 מיקרוליטר של תאים מגורדים במגיב הגנת RNA בכל באר. דוגמה מתוארת על ידי השקופית האמצעית. לאחר מכן, בארות 2 ו -4 נשטפו והנוזל הוסר. התאים התלויים מחדש מבארות 1 ו-3 מועברים לבארות 2 ו-4, בהתאמה, המוצגות בשקופית הימנית. לאחר הגירוד, התאים התלויים מחדש בבארות 2 ו -4 משולבים לצינור מיקרו-צנטריפוגה יחיד. דמות זו נוצרה עם BioRender.com. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 2. דוגמאות לאלקטרופרוגרמות RNA מדגימות RNA שחולצו באיכות גבוהה עד נמוכה. הפסגות הריבוזומליות 16S ו-23S מסומנות בבסיס הפסגות שלהן. RNA מושפל מיוצג על ידי פסגות קטנות, המסומנות על ידי החצים וקו בסיס גבשושי מעל האפס. דגימות RNA באיכות מקובלת מוצגות בפאנלים A, B ו-D. פאנל C מציג דגימת רנ"א באיכות ירודה שבה חסרות הפסגות הריבוזומליות והריכוז היה בשפע נמוך מאוד, כפי שמעיד קנה המידה על ציר ה-Y. FU, יחידות פלואורסצנטיות; NT, נוקלאוטיד. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 3. FastQC לכל תרשים איכות רצף בסיס עבור נתוני רצף PA288-1 ו-PA375-3. ת. העלילה האיכותית לדוגמא האיכותית PA288-1. ב. חלקת האיכות עבור מדגם טיפוסי PA375-3. התרשימים מציגים את ניקוד האיכות המצטבר עבור כל מיקום בסיס עבור כל הקריאות בקובץ. הקו הכחול מייצג את ציון האיכות הממוצע בכל עמדת בסיס. הקו האדום בתוך התיבה הצהובה מייצג את ציון האיכות החציוני בכל עמדה, והתיבה הצהובה מציגה את טווח הרביעונים עבורהאחוזונים ה-25 עד ה-75. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
| לבודד | קיוביט | רין | רצף |
| PA288-1 | 26 ננוגרם/מיקרוליטר | 7.5 | כן |
| PA375-3 | 4.07 ננוגרם/מיקרוליטר | נה | כן |
| PA565-3 | 3.4 ננוגרם/מיקרוליטר | נה | לא |
| PA921-1 | 9.11 ננוגרם/מיקרוליטר | 6.6 | כן |
טבלה 1. מדדי איכות של RNA שחולצו מדגימות מייצגות.
| PA288-1 | PA375-3 | |
| סה"כ קריאות | 8,59,57,720 | 3,18,49,575 |
| קריאות מסומנות כאיכות ירודה | 0 | 0 |
| אורך הקריאה | 100 | 100 |
| % GC | 61 | 60 |
טבלה 2. PA288-1 ו- PA375-3 סטטיסטיקת סיכום FastQC
| לבודד | % קורא ממופה להפניה | עומק קריאה ממוצע | רוחב הכיסוי |
| PA288-1 | 83.93% | 404 | 96.68% |
| PA375-3 | 91.2% | 578 | 96.97% |
טבלה 3. PA288-1 ו- PA375-3 סטטיסטיקות מיפוי
Discussion
סה"כ RNA מופק בהצלחה מ-17 דגימות ביופילם חיידקיות שונות בשלוש עותקים, ומניב בסך הכל 51 דגימות. ארבעים ותשע ספריות ה-RNA מאוגדות ומרוצפות בהצלחה. בסך הכל, זה מאמת את קריטריוני האיכות שלנו עם שיעור הצלחה של 96% למרות שיותר ממחצית הדגימות נחשבות לשפע נמוך ובאיכות תת-אופטימלית 34,35,36,37.
משמעות
פרוטוקול מיצוי RNA זה ייחודי בהסבר המפורט שלו למיצוי RNA מכמויות מוגבלות של ביופילם שגודלו בשקופיות תא. גידול ביופילמים בשקופית תא של 8 בארות הוא מערכת שימושית לחקר ההשפעות של גורמים שנוספו אקסוגנית לביופילם או השפעות של אינטראקציות מיקרוביאליות ביצירת ביופילם על ידי מיקרוסקופיה קונפוקלית 12,13,42. על מנת לבחון את ביטוי הגנים המעורבים ביצירת ביופילם במערכת זו, אנו מציגים שיטה מפורטת למיצוי RNA שלם באיכות ובכמות מספיקים לריצוף RNA. מחקרים אחרים דיווחו על מיצוי מוצלח של RNA מכמויות מוגבלות של ביופילם, אך רובם מגדלים ביופילמים על שטח פנים גדול יותר ולעתים קרובות במשך 48 שעות במקום 24 שעות. הם גם חסרים מספיק פרטים כדי להבטיח הצלחה 4,15,16,43,44,46.
יתר על כן, פרוטוקול זה נמנע משימוש בכימיקלים מסוכנים (כגון פנול) או ציוד מיוחד (כגון סוניקטור). פרוטוקול מיצוי פנול-כלורופורם הקלאסי של גואנידיניום תיוציאנט47 אינו משמש למערכת זו מכיוון שלמרות שהוא מניב בערך פי 2 יותר RNA בהשוואה לערכה המסחרית, בידינו הוא מביא באופן עקבי ל-RNA מפורק לחלוטין, כפי שהוערך במערכת אלקטרופורזה אוטומטית. בנוסף, הוכח כי השימוש בערכת מיצוי מסחרית מביא לפרוטוקול ידידותי למשתמש המניב תוצאות עקביות 15,16,45.
צעדים קריטיים
ישנם מספר שלבים קריטיים בפרוטוקול זה המגדילים את ההסתברות לחילוץ RNA שניתן לרצף בהצלחה. ראשית, חשוב לחסן לפחות 4 בארות של מגלשת התא באותו זן על מנת להשיג כמויות מספיקות של RNA לרצף. הביופילמים מ-4 הבארות מאוגדים למיצוי יחיד, שיש לו יתרון נוסף של הפחתת השונות בניתוח ביטוי הגנים במורד הזרם17. הפקת RNA מפחות מ-4 בארות מובילה לרוב לתשואות נמוכות מכדי לזהות במערכת פלואורומטרית ברגישות גבוהה. הפקת RNA מ-8 בארות או מגלשה שלמה תניב כמויות גבוהות יותר של RNA באיכות דומה לזו המתקבלת בעת שימוש ב-4 בארות, אך יש לקחת בחשבון אם הזמן, המאמץ והעלות הנוספים להפקת הגזע שווים את התשואה המוגברת. פיפטינג של 300 מיקרוליטר של תרבית מדוללת OD600 = 0.1 לזריעה של כל באר במקום 200 מיקרוליטר הסטנדרטי משפר את ההתאוששות של חומר ביופילם מגורד מכל באר. שטיפה עדינה של הביופילם פעמיים במים נטולי נוקלאז לפני מריחת מגיב הגנת ה-RNA היא קריטית להסרת כמה שיותר תאים מתים ו/או פלנקטוניים. השימוש במגיב המגן RNA חשוב למניעת פירוק RNA בשל הזמן הנדרש לעיבוד כל באר. יתר על כן, במקום לגרד עם קצות פיפט, אנו מעדיפים להשתמש במרית מתכת במשקל עם קצה שטוח המגע עם שטח פנים גדול יותר מאשר קצה פיפט, ושהם קטנים מספיק כדי להתאים לבאר. גירוד באמצעות קצה פיפט של 1000 מיקרוליטר יעבוד אך הוא פחות יעיל ולוקח יותר זמן לגרד את כל הביופילם בהשוואה למרית המתכת. מניסיוננו, שימוש במרית מתכת חוסך זמן ומאמץ. בעת גירוד, הקפד להניח את מגלשת החדר על גבי צלחת הזכוכית כדי למנוע סדקים בתחתית הבארות. סוניקציה אינה יעילה מכיוון שהיא גורמת באופן עקבי ל-RNA מפורק מאוד עקב נפח דגימה קטן מדי וייצור חום רב מדי. לאחר איסוף חומר הביופילם, הדגימה מוזנת לערכה מסחרית למיצוי RNA אמין יותר וניתן לשחזור. לבסוף, בגלל מקורות פוטנציאליים לשונות בשיטה זו, חשוב מאוד לכלול שכפול ניסיוני בנוסף לאיגום דגימות17,48. פרוטוקול זה מתאר שכפול ביולוגי משולש של כל דגימה.
מגבלות
ישנן מגוון טכניקות המשמשות לגידול ביופילמים למחקר, הנפוצה שבהן היא היווצרות בלוחות מיקרוטיטר4. צלחות אלה זמינות בגדלים ומספרים שונים של בארות. שקופיות קאמריות נכנסות לקטגוריה זו. היכולת לחלץ RNA שלם וטהור חשובה לכל מערכות הביופילם, אך מערכת זו מוגבלת על ידי התפוקות הנמוכות של RNA. הריכוזים נמוכים מספיק כדי להעריך את איכותם וכמותם על ידי מכשירי Qubit ו-Bioanalyzer באמצעות ערכות RNA בעלות רגישות גבוהה. ניתן להשתמש גם בננו-טיפה, אך יש לה רגישות מוגבלת יותר בהשוואה לקוביט ואינה יכולה להבחין בין DNA מזהם ל-RNA49. במידת האפשר, יש להשתמש בננו-טיפה כדי לקבל יחסי A260/A280 ו-A260/A230 לטוהר. יחסים אלה שימושיים, במיוחד מכיוון שאיכות ה-RNA ממערכת ביופילם זו אינה טובה כמו מכמויות גדולות יותר של חומר מוצא. סביר להניח שהסיבה לכך היא התפוקה הנמוכה של RNA ונוכחות של תאים מתים בביופילמים ו/או RNase שעלולים לפרק RNA. מגבלה נוספת של שיטה זו היא שהיא אינה יכולה להפריד בין סוגי תאים שונים בתוך ביופילם הטרוגני אלא יכולה רק לחלץ את התאים כאוכלוסייה שלמה, מה שעלול לטשטש את ביטוי הגנים בכל תת-אוכלוסייה שאינה מיוצגת.
יישומים פוטנציאליים
הפרוטוקול המוצג כאן עבר אופטימיזציה עבור ביופילמים של P. aeruginosa מבודדים קליניים שגדלו בשקופיות תא. ניתן להשתמש בשיטה זו כדי לחקור שינויים בביטוי גנים במהלך היווצרות ביופילם בתנאי גידול שונים שנצפו במיקרוסקופיה קונפוקלית. ניתן לבצע אופטימיזציה של הפרוטוקול גם עבור מיני חיידקים אחרים המייצרים ביופילם. בשיטה זו ניתן גם לחלץ DNA גנומי מכיוון שניתן להשתמש בערכת מיצוי כפולה. בדרך זו, ניתן לחקור בצורה מדויקת יותר את ביטוי הגנים של ביופילם חיידקי בבידוד קליני הגורם לזיהומים בחולים, מה שמוביל לתוצאות שעשויות להנחות אסטרטגיות טיפול עתידיות.
Disclosures
למחברים אין גילוי נאות להצהיר.
Acknowledgements
תרומת המחברים: P.W., Y.Y. ו-V.W היו מעורבים בהמשגת המחקר. K.G., L.J., A.M. ו-P.W. ביצעו אופטימיזציה של פרוטוקולי המעבדה. המימון ל-KG נתמך על ידי סבסוד תוכנית ההשמה לעבודה לסטודנטים באמצעות BioTalent Canada.
Materials
| Name | Company | Catalog Number | Comments |
| Agilent 2100 Bioanalyzer | Agilent | G2939BA | Automated electrophoresis of biomolecules |
| Agilent RNA 6000 pico kit | Agilent | 5067-1513 | High sensitivity RNA electrophoresis chip to generate a RIN |
| DNA/RNA Lysis Buffer | Zymo Research | D7001-1-50 | A guanidinium thiocyanate and N-Lauroylsarcosine-based lysis buffer sold as part of a nucleic acid purification kit |
| DNA/RNA Prep Buffer | Zymo Research | D7010-2-10 | A guanidine HCl and ethanol buffer used for purification of DNA and RNA |
| DNA/RNA Shield | Zymo Research | R1100-50 | DNA and RNA preservation/protection reagent |
| DNA/RNA Wash Buffer | Zymo Research | D7010-3-6 | A salt and ethanol buffer used for purification of DNA and RNA |
| DNBSEQ G-400RS | MGI | G-400RS | High throughput sequencer |
| MGIEasy RNA Directional Library Prep Set | MGI | 1000006386 | Generate libraries for MGI high-throughput sequencing platforms from total RNA. |
| Mini-Beadbeater-96 | BioSpec | 1001 | A high energy, high throughput cell disrupter |
| NEBNext rRNA Depletion Kit (bacteria) | New England Biolabs | E7850X | Efficient and specific depletion of bacterial rRNA (5S, 16S, 23S) |
| Nunc Lab-Tek II chamber slide system | Thermo Fisher Scientific | 154534 | 8-well chamber slide with removable wells |
| Qubit Fluorometer | Thermo Fisher Scientific | Q33238 | Fluorometer for DNA, RNA and proteins |
| Qubit RNA HS Assay Kit | Thermo Fisher Scientific | Q32852 | High sensitivity fluorometric assay to measure RNA concentration |
| Spin-Away Filters | Zymo Research | C1006-50-F | Silica-based spin column primarily used to bind or remove genomic DNA |
| Sterile inoculation loops, 1 uL | Sarstedt | 86.1567.050 | Sterile, disposable inoculation loops for manipulation of microorganisms |
| ZR BashingBead Lysis tubes | Zymo Research | S6003-50 | 2 mL tubes containing 0.1 and 0.5 mm bead lysis matrix for homogenizing biological samples |
| Zymo Spin IIICG Columns | Zymo Research | C1006-50-G | Silica-based spin column for purification of DNA and RNA |
| Zymo-Spin III-HRC Filters | Zymo Research | C1058-50 | Remove inhibitors such as polyphenolic compounds, humic/fulvic acids, tannins, melanin, etc. |
| Zymobiomics DNA/RNA Miniprep kit | Zymo Research | R2002 | DNA and RNA dual extraction kit |
| Zymobiomics HRC Prep solution | Zymo Research | D4300-7-30 | To be used with Zymo-Spin III-HRC Filters to remove PCR inhibitors |
References
- Beaudoin, T., Waters, V. Infections With Biofilm Formation: Selection of Antimicrobials and Role of Prolonged Antibiotic Therapy. The Pediatric Infectious Disease Journal. 35 (6), 695-697 (2016).
- Bjarnsholt, T., et al. The in vivo biofilm. Trends in Microbiology. 21 (9), 466-474 (2013).
- Folsom, J. P., et al. Physiology of Pseudomonas aeruginosa in biofilms as revealed by transcriptome analysis. BMC Microbiology. 10, 294(2010).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43 (3), 313-351 (2017).
- Bjarnsholt, T., Ciofu, O., Molin, S., Givskov, M., Hoiby, N. Applying insights from biofilm biology to drug development - can a new approach be developed. Nature Reviews Drug Discovery. 12 (10), 791-808 (2013).
- Colvin, K. M., et al. The Pel and Psl polysaccharides provide Pseudomonas aeruginosa structural redundancy within the biofilm matrix. Environmental Microbiology. 14 (8), 1913-1928 (2012).
- Hentzer, M., et al. Alginate overproduction affects Pseudomonas aeruginosa biofilm structure and function. Journal of Bacteriology. 183 (18), 5395-5401 (2001).
- Cury, J. A., Koo, H. Extraction and purification of total RNA from Streptococcus mutans biofilms. Analytical Biochemistry. 365 (2), 208-214 (2007).
- Francavilla, M., et al. Extraction, characterization and in vivo neuromodulatory activity of phytosterols from microalga Dunaliella tertiolecta. Current Medicinal Chemistry. 19 (18), 3058-3067 (2012).
- Atshan, S. S., et al. Improved method for the isolation of RNA from bacteria refractory to disruption, including S. aureus producing biofilm. Gene. 494 (2), 219-224 (2012).
- Franca, A., Melo, L. D., Cerca, N. Comparison of RNA extraction methods from biofilm samples of Staphylococcus epidermidis. BMC Research Notes. 4, 572(2011).
- Jurcisek, J. A., Dickson, A. C., Bruggeman, M. E., Bakaletz, L. O. In vitro biofilm formation in an 8-well chamber slide. The Journal of Visusalized Experiments. (47), e2481(2011).
- Beaudoin, T., Kennedy, S., Yau, Y., Waters, V. Visualizing the effects of sputum on biofilm development using a chambered coverglass model. The Journal of Visusalized Experiments. (118), e54819(2016).
- Cockeran, R., et al. Biofilm formation and induction of stress response genes is a common response of several serotypes of the pneumococcus to cigarette smoke condensate. The Journal of Infection. 80 (2), 204-209 (2020).
- Bisht, K., Moore, J. L., Caprioli, R. M., Skaar, E. P., Wakeman, C. A. Impact of temperature-dependent phage expression on Pseudomonas aeruginosa biofilm formation. npj Biofilmsand Microbiomes. 7 (22), (2021).
- Harrison, A., et al. Reprioritization of biofilm metabolism is associated with nutrient adaptation and long-term survival of Haemophilus influenzae. NPJ Biofilms and Microbiomes. 5 (1), 33(2019).
- Sousa, C., Franca, A., Cerca, N. Assessing and reducing sources of gene expression variability in Staphylococcus epidermidis biofilms. BioTechniques. 57, 295-301 (2014).
- Boom, R. Rapid and simple method for purification of nucleic acids. Journal of Clinical Microbiology. 28 (3), 495-503 (1990).
- Coppin, C. Re: How do silica based RNA spin columns only bind RNA and not DNA. , Available from: https://www.researchgate.net/post/How_do_silica_based_RNA_spin_columns_only_bind_RNA_and_not_DNA/60b017bffa5c4151cac1c/citation/download (2021).
- Kennedy, S. A complete guide to how nucleic extraction kits work. , Available from: https://bitesizebio.com/13516/how-dna-extraction-rna-miniprep-kits-work/ (2021).
- Qubit RNA HS Assay Kit User Guide. Thermo Fisher Scientific. , Available from: https://www.thermofisher.com/document-connect/document-connect.html?url=https%3A%2F%2Fassets.thermofisher.com%2FTFS-Assets%2FLSG%2Fmanuals%2FQubit_RNA_HS_Assay_UG.pdf&title=VXNlciBHdWlkZTogUXViaXQgUk5BIEhTIEFzc2F5IEtpdHM (2015).
- Mueller, O., Lightfoot, S., Schroeder, A. RNA Integrity Number (RIN) - Standardization of RNA Quality Control (Application report # 5989-1165EN). , Available from: https://www.agilent.com/cd/library/applications/5989-1165EN.pdf (2016).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 3(2006).
- Culviner, P. H., Guegler, C. K., Laub, M. T. A Simple, Cost-Effective, and Robust Method for rRNA Depletion in RNA-Sequencing Studies. mBio. 11 (2), (2020).
- MGIEasy RNA Directional Library Prep Set User Manual verA2. MGI Tech Co. , Available from: https://en.mgi-tech.com/products/reagents_info/14/ (2020).
- Andrews, S. FastQC: A Quality Control Tool for High Throughput Sequence Data. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina Sequence Data. Bioinformatics. , (2014).
- NCBI Resource Coordinators. Database resources of the National Center for Biotechnology Information. Nucleic Acids Research. 44 (1), (2016).
- Li, H. Aligning sequence reads, clone sequences and assembly contigs with BWA-MEM. arXiv. , (2013).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), (2009).
- Liao, Y., Shi, W. Read trimming is not required for mapping and quantification of RNA-seq reads at the gene level. NAR Genomics and Bioinformatics. 2 (3), (2020).
- Penir, S. Calculating Mapping Statistics from a SAM/BAM file using SAMtools and awk. , Available from: https://sarahpenir.github.io/bioinformatics/awk/calculating-mapping-stats-from-a-bam-file-using-samtools-and-awk/ (2019).
- Haile, S., et al. Evaluation of protocols for rRNA depletion based RNA sequencing of nanogram inputs of mammalian total RNA. PLoS ONE. 14 (10), 0224578(2019).
- Schuierer, S., et al. A comprehensive assessment of RNA-seq protocols for degraded and low-quantity samples. BMC Genomics. 18 (442), (2017).
- Shanker, S., et al. Evaluation of Commercially Available RNA Amplification Kits for RNA Sequencing Using Very Low Input Amounts of Total RNA. Journal of Biomolecular Techniques. 26 (1), (2015).
- Adiconis, X., et al. Comparative analysis of RNA sequencing methods for degraded or low-input samples. Nature Methods. 10, 623-629 (2013).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (13), (2016).
- Illumina, Inc. Coverage depth recommendations. , Available from: https://www.illumina.com/science/technology/next-generation-sequencing/plan-experiments/coverage.html (2021).
- What is a good sequencing depth for bulk RNA-Seq. ECSEQ Bioinformatics. , Available from: https://www.ecseq.com/support/ngs/what-is-a-good-sequencing-death-for-bulk-rna-seq (2019).
- Bedre, R. Sequencing coverage and breadth of coverage. , Available from: https://www.reneshbedre.com/blog/sequencing-coverage.html (2021).
- Dotsch, A., et al. The Pseudomonas aeruginosa transcriptome in planktonic cultures and static biofilms using RNA sequencing. PLoS One. 7 (2), 31092(2012).
- Chen, Y., et al. Population dynamics and transcriptomic responses of Pseudomonas aeruginosa in a complex laboratory microbial community. npj Biofilms and Microbiomes. 5 (1), (2019).
- Thoming, J. G., et al. Parallel evolutionary paths to produce more than one Pseudomonas aeruginosa biofilm phenotype. NPJ Biofilms and Microbiomes. 6, 2(2020).
- Soares, A., et al. Understanding ciprofloxacin failure in Pseudomonas aeruginosa biofilm: persister cells survive matrix disruption. Frontiers in Microbiology. 10, 2603(2019).
- Whiteley, M., et al. Gene expression in Pseudomonas aeruginosa biofilms. Nature. 413, (2001).
- Chomczynski, P., Sacchi, N. The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: twenty-something years on. Nature Protocols. 1, (2006).
- Liu, Y., Zhou, J., White, K. P. RNA-seq differential expression studies: more sequence or more replication. Bioinformatics. 30 (3), (2014).
- Wieczorek, D., Delauriere, L., Schagat, T. Methods of RNA Quality Assessment. , Available from: https://www.promega.ca/resources/pubhub/methods-of-rna-quality-assessment/ (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved