Method Article
遺伝子発現測定のためのバイオフィルム内緑 膿菌 からのTotal RNAの単離
要約
このプロトコルは、チャンバースライドで成長した 緑膿菌バイオ フィルムからRNAを単離し、ハイスループットシーケンシングを行う方法を提示します。
要約
緑膿菌 は、嚢胞性線維症(CF)患者の気道に感染症を引き起こす日和見性細菌性病原体です。 緑膿菌は 、エキソ多糖のマトリックスによって保護されるバイオフィルムを形成する能力で知られています。このマトリックスにより、微生物は抗生物質治療などの外的要因に対してより回復力を持つことができます。研究用のバイオフィルム成長の最も一般的な方法の1つは、マイクロタイタープレートまたはチャンバースライドです。これらのシステムの利点は、複数の増殖条件を試験できることですが、欠点は、RNA抽出のためのバイオフィルムの生成量が限られていることです。この記事の目的は、ハイスループットシーケンシングに十分な品質と量の少量のバイオフィルムから全RNAを抽出する方法について、詳細なステップバイステップのプロトコルを提供することです。このプロトコルにより、これらのバイオフィルムシステム内での遺伝子発現の研究が可能になります。
概要
嚢胞性線維症(CF)患者の肺感染症やプロテーゼ関連感染症など、ほとんどの慢性細菌感染症は、バイオフィルム内の生物の増殖を特徴としています。バイオフィルム1 は、主に多糖類2からなるマトリックスに包まれた細菌の群集である。バイオフィルム内の細菌は、成長が遅く、代謝的に休眠しており、嫌気性、低酸素状態にある可能性があります。バイオフィルムは、抗生物質の浸透性の低下、薬物排出ポンプの発現の増加、細胞分裂の減少などの要因により、抗生物質に対してより耐性があります3。これらの理由やその他の理由から、それらは非常に研究上の関心を集めています。
CF患者における慢性緑膿菌感染症などの持続性感染症を正確に研究するためには、バイオフィルム形成に見られる成長条件をin vitroで正確に反映する必要があります。一般的なハイスループットな方法は、チャンバースライドやマイクロタイタープレートでそれらを成長させ、共焦点顕微鏡4でバイオフィルム形成をモニターすることです。プランクトン性または自由浮遊性からバイオフィルム細菌生活への移行における主要な調節因子は、二次メッセンジャーである環状ジGMP5であることが知られています。サイクリックジGMPレベルが増加すると、バイオフィルムの成長を促進する特定の遺伝子の発現が増加します。また、低分子のノンコーディング制御RNAやクオラムセンシングも、バイオフィルム形成の制御に重要な役割を果たしています5。抽出された細菌RNAのシーケンシングによるバイオフィルム遺伝子発現の測定は困難な場合があります。例えば、P. aeruginosaは、3つのエキソ多糖類(Psl、Pelおよびアルギン酸塩)を産生し、これらはバイオフィルム6,7において大量に産生される。これらの多糖類は、RNAの抽出と精製を妨害し、低レベルの細菌mRNA8を含む不純な調製物につながる可能性があります。市販のRNA抽出キットは、プランクトン性細菌培養物から高品質のRNAを産生することができるが、バイオフィルム培養物ではうまく機能しない可能性がある9,10,11。バイオフィルムに効くと謳っている市販のRNA抽出キットがいくつかあり、そのうちの1つをこの方法で使用しています。
この原稿では、チャンバスライドで緑膿菌のバイオフィルムを増殖させ、ハイスループットシーケンシング12,13のための細菌mRNAを抽出する手順について説明します。CF患者の喀痰サンプルから採取した臨床分離株を利用して、これらの方法がさまざまな成長特性を持つ分離株に使用できることを実証します。以前の出版物と比較して、このプロトコルは、細菌のバイオフィルム遺伝子発現11,14,15,16の研究においてより良い成功を可能にするために詳細に説明されています。
プロトコル
研究倫理委員会(REB)は、ヒト被験者からの喀痰サンプルの収集と処理に必要です。この研究は、Hospital for Sick Children(REB#1000019444)によって承認されました。研究倫理委員会(REB)は、ヒトを対象とする喀痰サンプルを収集し、保存することが義務付けられています。これらの研究は、Hospital for Sick Children REB#1000058579によって承認されました。
1.バイオフィルム形成
- この研究で使用したCF患者の喀痰サンプルから得られた 緑膿菌 分離株を、37°Cのインキュベーター内のLuria Broth(LB)寒天プレートで一晩育てます。
注:適切なストリーキング技術は、単一の細菌コロニーを得るために重要です。プレートを回転させながらストリーキングをすると、バクテリア細胞が十分に希釈され、単一のコロニーが成長できるようになります。- 新鮮なLBプレートの上端にジグザグパターンの接種ループを使用して、プレートの約1/4が覆われるまでストリークバクテリア。
- プレートを約60°回転させます。新しい接種ループを取り、それを縞模様の領域に一度通し、プレートの2番目のきれいな領域に通し、ジグザグパターンを繰り返します。
- 手順1.1.2を繰り返し、プレートの3番目の領域に新しいループを入れます。インキュベーターに入れるときは、蓋を元に戻し、プレートを反転させます。
- 滅菌接種ループを使用して、寒天プレート(単一の細菌株を含む)から単一の細菌コロニーを取り出し、5 mLの滅菌LB培地を充填した培養チューブに接種します。毎回新しいループを使用して、同じプレートから5 mLのLB培地を充填した2本の培養チューブを追加で接種します。
- 異なる細菌株に対して同じ接種手順を繰り返します。培養物を37°Cで一晩培養し、220RPMで振とうします。
注:1つの細菌株を含む各寒天プレートは、3つの独立した培養チューブを接種するために使用されます。3本のチューブは、1つの菌株の生物学的複製を三重に表し、別々のサンプルとして扱われます。これは、1本の培養チューブからRNAを3回抽出するテクニカルリピートとは異なります。 - 50 μLのオーバーナイトカルチャーを4.95 mLの新しいLB培地を含む新しい培養チューブに移すことにより、オーバーナイト培養液を1:100希釈液で調製します。
- 希釈した培養物を37°Cでさらに3時間、220 RPMで振とうするか、OD600 が0.1以上になるまで培養します。OD600の分光光度計で細胞密度を測定します。
- 新しい1.7 mLマイクロチューブで、OD600 を0.1(初期対数期)に調整し、新鮮なLBで総容量1.5 mLにします。
- 反転させてやさしく混ぜます。調整した各培養物300μLを8ウェルチャンバースライドの4ウェルに移し、スライドごとに2つの異なるサンプルが得られます(図1)。
- スライドをそのまま、37°Cのインキュベーターに24時間一晩置きます。蒸発を防ぐために、広告の上にスライドを置きますamp 小さなプラスチックトレイのペーパータオル。
2. バイオフィルムの回復
注:各スライドガラスには、8つの別々のウェルが含まれています。1つのサンプルは、プールされるバイオフィルムを備えた4つのウェルで構成されています17。この抽出プロトコルは、1つのサンプル(4つのウェル)を対象としており、バイオフィルムは一度に2つのウェルから回収されます。RNA抽出は、ビーズビーティングステップとカラムベースのクリーンアップを含む市販のRNA抽出キットを使用して実行されます。試薬の調製については、製造元の指示に従ってください。
- 層流フードで、ピペットチップを使用して、4つのウェルのうち2つからメディアをゆっくりと取り出します。スライドを45°の角度で傾け、バイオフィルムの剥離を防ぐために、ウェルの下部の角から培地をピペットで取り出します。
- スライドを傾けたまま、空になった2つのウェルの底隅に300 μLのRNaseを含まない水を静かにピペッティングして、プランクトン細胞を洗い流します。手順2.1で説明したように、水を静かにピペッティングして水を取り除きます。できるだけ多くの水を取り除き、洗浄手順を繰り返します。
- 300 μLのRNA保護試薬( 材料表を参照)を、バイオフィルムを基部に持つ2つの空になったウェルのそれぞれに加えます。チャンバースライドをガラス板に載せてウェルが壊れないようにし、2つのウェルのバイオフィルムをヌクレアーゼフリーの滅菌金属ヘラでこすり落とし、バイオフィルム細菌を再懸濁します。バイオフィルムが同じサンプルから残っている2つの手つかずのウェルから回収されるまで、座ってみましょう。
注:RNA保護試薬を添加すると、同じサンプルの残りの2つのウェルを処理しながら、掻き取ったウェル内のバイオフィルムサンプルの周囲温度での安定性が保証されます。RNA保護試薬は、細胞を溶解し、ヌクレアーゼや感染性病原体を不活性化することで、RNAの保存につながります。 - サンプルの残りの 2 つのウェルからバイオフィルムを回収するには (注意: 1 つのサンプルは 4 つのウェルで構成されています)、ステップ 2.1 で説明したのと同じ方法で、残りの 2 つの新しいウェルから LB メディアを取り出します。ステップ2.2を繰り返して、以前と同様に、両方の新しいウェルからRNaseを含まない水でプランクトン細胞を洗い流します。
- ステップ2.3の最後に生成された保護試薬に掻き取りされたバイオフィルムがある最初の2つのウェルに戻り、掻き取った 1つの ウェルから300μLの再懸濁バイオフィルムをゆっくりとピペットで混合し、気泡が多すぎないようにします。
- ウェルからすべての内容物を、新しく洗浄して空にしたウェルの 1つ に移します。2番目に掻き取ったウェルから再懸濁したバイオフィルム300μLを、残りのウェルに新たに洗浄し、空にしたバイオフィルムに混合して移します。
注:バイオフィルムで新たに洗浄した2つのウェルに新しいRNA保護試薬を追加する代わりに、すでに保護試薬に掻き取ったバイオフィルムをこれらの新しく洗浄したウェルに移します。これにより、組み合わせたサンプル量を十分に低く抑え、市販のRNA抽出キットのインプット要件を満たすことができます。図 1 の概略図を参照してください。
- ウェルからすべての内容物を、新しく洗浄して空にしたウェルの 1つ に移します。2番目に掻き取ったウェルから再懸濁したバイオフィルム300μLを、残りのウェルに新たに洗浄し、空にしたバイオフィルムに混合して移します。
- ステップ2.3と同様に、チャンバースライドをガラス板に置き、ヌクレアーゼフリーの滅菌金属ヘラで2つの新しいウェルのバイオフィルムを掻き取り、バイオフィルム細菌を再懸濁することにより、新しいウェルのバイオフィルムのスクレイピングを繰り返します。
- 2つの新しいウェルから再懸濁した バイオフィルムをすべて 、RNaseフリーで低結合の1.5 mLマイクロ遠心チューブに結合します。容量を測定します(合計で~500 - 600 μL)。
注:この2番目の新しいウェルのペアから結合されたバイオフィルム懸濁液には、サンプルの元の4つのウェルからのすべてのバイオフィルム材料が含まれます。
3. Total RNAの単離と品質評価
注:RNA抽出は、バイオフィルムに効くと主張する市販のRNA抽出キットを使用して行われます。個々のコンポーネントは、可能であれば 、材料表に含まれています。各精製ステップの背後にあるメカニズムについては、可能な限り説明します。
- チューブ内に合計750μLのRNA保護試薬を十分に加えます。0.1 mmおよび0.5 mmのビーズが入った2 mLのビーズビート溶解チューブに全容量を移します( 材料の表を参照)。ビーズビーターで最高速度で2分半叩きます。
注:0.1 mmと0.5 mmの高密度ビーズの組み合わせとビーズビーターでの高速混合により、微生物細胞壁の徹底的な均質化が保証されます。 - 16,000 x g で1分間遠心分離し、ビーズをペレット化します。上清を新しい微量遠心チューブに移し、ビーズの移し替えを最小限に抑えます。これにより、ステップ3.3が容易になります。音量を測定します。
- 等量のRNA溶解バッファー(~450 μL)を添加し、よく混合します。ビーズの移し替えを避けながら、最大800 μLのサンプルをコレクションチューブ内のシリカカラムに移し、16,000 x g で30秒間遠心分離します。フロースルーにはRNAが含まれており、DNAはカラムに結合しているため、フロースルーを保存します。
注:RNA溶解バッファーには、細胞を溶解するためのチオシアン酸グアニジンと界面活性剤N-ラウロイルサルコシンが含まれています。チオシアン酸グアニジニウムは、ヌクレアーゼを不活性化し、スピンカラムに見られるシリカの存在下で、DNAのシリカへの結合を促進するカオトロピック剤である18。エタノールの不在は、シリカスピンカラム19へのRNAではなくDNAの優先的な結合を可能にする。ステップ3.3の目的は、ゲノムDNAを結合して除去することです。フロースルー部分に含まれるRNAを保持したいのです。 - さらにサンプルが残っている場合は、カラムを新しい収集チューブに移し、残りのサンプルと一緒に再ロードします。16,000 x g で30秒間遠心分離します。RNAとのフロースルーを維持し、最初のアリコートと結合します。
- 複合フロースルー体積を測定し、等量の100%エタノールを加えてよく混合します。最大800 μLの溶液をコレクションチューブ内の新しい2番目のシリカスピンカラムに移し、16,000 x g で30秒間遠心分離します。フロースルーを破棄します。
注:RNAを含有するカオトロピック塩溶液へのエタノールの添加は、シリカスピンカラム19へのRNAの結合を増強する。 - 800 μL>溶液の場合は、スピンカラムを再ロードし、溶液全体が遠心分離されるまで遠心分離します。各スピンの後にフロースルーを捨てます。
- 400 μL の洗浄バッファーをカラムに加え、16,000 x g で 30 秒間遠心分離して、一部の塩分を除去します。フロースルーを破棄します。
- DNase I反応混合物を製造元の指示に従って調製し、インカラムDNase処理を実施して残留DNAを除去します。
- 凍結乾燥したDNase Iを275 μLのRNaseフリー水に再懸濁して、1 U/μL溶液を作ります。穏やかに反転させて混ぜます。
- 希釈したDNase I5 μLと付属のDNase消化バッファー75 μLを組み合わせます。反転させてやさしく混ぜます。
- 調製した溶液80 μLをカラムマトリックスに直接加えます。室温で20分間インキュベートします。
- 400 μL の RNA 調製バッファーをカラムに加え、16,000 x g で 30 秒間遠心分離します。フロースルーを破棄します。
- 700 μL の RNA 洗浄バッファーをカラムに加え、遠心分離を繰り返します。フロースルーを破棄します。
- 400 μL の RNA 洗浄バッファーを添加し、カラムを 2 分間遠心分離して、残留バッファーを完全に除去します。
注:カラムの不純物を除去するには、2つの異なる洗浄ステップがあります。調製バッファーには、残留タンパク質を除去するために、エタノールと混合した弱なカオトロピック塩が含まれています。次に、洗浄バッファーを使用して、塩分を除去するためのエタノール洗浄を行います。RNA19,20の効率的な溶出を可能にするために、残っているエタノールを除去する必要があります。 - カラムを新しい低結合の微量遠心チューブに慎重に移します。
- 50 μL の RNase フリー水を直接カラムマトリックスに加え、5 分間インキュベートします。16,000 x gで1分間遠心分離し、RNAを溶出します。
- PCR阻害剤を除去するための追加のクリーンアップを行うには、PCR阻害剤フィルターを新しい収集チューブに入れます。付属の阻害剤調製液600 μLを加えます( 材料表を参照)。
- 8,000 x gで3分間遠心分離し、フィルターを洗浄します。洗浄したフィルターを新しい低結合微量遠心チューブに移します。
- ステップ3.13で溶出したRNAを洗浄したフィルターに移し、16,000 x gで3分間遠心分離します。
注:RNAはすぐに使用することも、-80°Cで保存することもできます。 - 高感度蛍光測定システム21を用いてRNAの濃度を決定する。
注:これらのシステムでは、目的のターゲットに特異的な溶液中の少量のRNAを高感度に定量できます。- RNAの品質の尺度であるRIN(RNA完全性番号)を提供できる自動電気泳動システムを使用して、RNAの品質を評価します22,23。
4. リボソームRNAの枯渇とハイスループットシーケンシング
- トロント大学(カナダ、トロント)のCentre for the Analysis of Genome Evolution and Function(CAGEF)ゲノムセンター(https://www.cagef.utoronto.ca/)にRNAを登録し、細菌のrRNAの枯渇とRNA指向性ライブラリーの調製を行います(資料表を参照)。
注:細菌のrRNAの枯渇は、5S、16S、および23SのrRNAを標的とし、除去されます24。 - 市販のrRNA細菌枯渇キットを使用してrRNAを枯渇させます。10 ng - 1 μgの無傷または部分的に分解された全RNAを入力するためのプロトコルに従ってください。
- 各ライブラリに異なるインデックスが付加されたRNA指向性ライブラリ調製キットを使用して、RNAシーケンシングライブラリを構築します。
- 各RNAライブラリーの等モル量をプールし、100塩基対末端リード25でハイスループットシーケンシングを実行する。
5. シーケンシングリードの品質評価
注:無料のオープンソースプラットフォームであるGalaxy27から入手できる無料のプログラムFastQC26を使用して、シーケンシングリードの品質を確認してください。
- https://usegalaxy.org/ に移動します。[ログイン]または[登録 ]メニューをクリックし、資格情報でログインするか、アカウントを作成します。
- ページの左上にある[ツール]メニューの下にある[ データのアップロード ]リンクをクリックし、fastq.gzシーケンシングファイルをアップロードします。ファイル名がページ右側の[履歴]パネルの下に表示されるのを待ちます。
- [ツール]メニューの[FASTQ品質管理]を選択して、プログラムのリストを表示します。FastQCを選択すると、画面の中央パネルに表示されます。
- [ 現在の履歴からデータを読み取る] で、プルダウン メニューからアップロードした fastq.gz ファイルを選択します。
- [実行] を選択してプログラムを実行します。
- [History]パネルで結果を表示します(表2)。
注:Galaxyの使用方法の詳細については、https://galaxyproject.org/support/ のサポートページを参照してください。
6. シーケンシングリードのマッピング
注:リストされているのは、RNA-seqデータのアダプタートリミングとリードマッピングの基本的なパイプラインです。アダプターシーケンスは、Trimmomatic28を使用してリードからトリミングされます。トリミングされたリードは、BWA30およびSamtools31を使用してNCBI(https://www.ncbi.nlm.nih.gov/)29から得られた緑膿菌PAO1参照ゲノム(NC_002516.2)にマッピングされます。わかりやすくするために、読み取りのペアは PA_1.fq と PA_2.fq と呼ばれます。トリミングされるアダプタ読み取りファイルの名前は adapter.fa;PAO1 参照配列は PAO1.fasta と呼ばれます。すべてのツールはオープンソースであり、UNIX / LINUX環境で実行されます。これらのコマンドを実行するには、UNIX/LINUX の基本をよく理解しておくことを強くお勧めします。
- UNIX/LINUXでウィンドウを開きます。
- Java、Trimmomatic、BWA、Samtoolsをインストールします。
- ファイルtrimmomatic-0.39.jarが存在するフォルダに移動します。
- 次のコマンドを入力して、読み取りからアダプター シーケンスをトリミングします。
Java -jar PA_1.fq PA_2.fq PA_1_paired.fq PA_1_unpaired.fq PA_2_paired.fq PA_2_unpaired.fq ILLUMINACLIP: adapters.fa
注: アダプター シーケンスのみが削除されます。読み取りは品質32 のためにトリミングされていません。 - PAO1.fasta 参照ファイルを同じフォルダに移動します。
- 次のコマンドで、BWA を使用して参照のインデックスを作成します。
BwaインデックスPA01.fasta - ペアの読み取りを参照ゲノムにマッピングするには、次の 4 つのコマンドを入力します。前のコマンドが終了した後に、各コマンドを入力します。
Bwa -mem PA01.fasta PA_1_paired.fq PA2_2_paired.fq > PA_R1R2_map.mem.sam
サムツールズビュー -S -b PA_R1R2_map.mem.sam > PA_R1R2.bam
サムツール ソート PA_R1R2.bam -o PA_R1R2_sorted.bam
SamtoolsインデックスPA288_Rep1_R1R2_sorted.bam - マッピング統計を表示するには、次のコマンドを入力します。
Samtools flagstat PA_R1R2_sorted.bam
注:統計の3行目 は、参照ゲノムにマッピングされるリードの割合を報告します。 - 平均読み取り深度とカバレッジの幅を、それぞれ以下の2つのコマンド33で計算する。
samtools depth -a PA_R1R2_sorted.bam |awk '{c ++;s+=$3}END{print s/c}'
samtools depth -a PA288_Rep1_R1R2_sorted.bam |awk '{c++; if($3>0) total+=1}END{print (total/c)*100}'
結果
このメソッドの一般的な概要を図 1 に示します。我々は以前、8ウェルチャンバースライドを使用して緑膿菌のバイオフィルムを増殖させ、それらを抗生物質に曝露した後、異なる時点で共焦点顕微鏡でそれらを調べていた12,13。この方法は、このシステムで成長したバイオフィルムから直接トータルRNAを抽出し、処理後の遺伝子発現変化を研究するために使用できます。このプロトコールは緑膿菌に最適化されていますが、他の細菌種にも簡単に適応できます。
少量のバイオフィルムから十分な量の良質なRNAを抽出することは、rRNAの枯渇とハイスループットシーケンシングのために困難な場合があります。このプロトコルを使用して、17種類の 緑膿菌 バイオフィルム分離株からトータルRNAを三重に抽出し、合計51の別々のサンプルに成功裏に抽出します。高収量と低収量を表す抽出RNAの量を 表1に示します。RNAの濃度は3.4 ng/μL(最低)から49.6 ng/μL(最高)の範囲で、平均濃度は14 ng/μL、中央値は13.7 ng/μLです。10 ng/μL未満のインタクトRNA濃度は、rRNA枯渇および次世代シーケンシングのための低存在量サンプルと見なされますが、バイオフィルムからの少量のRNAサンプルは、より濃縮されたサンプルと比較してさらに品質の低いシーケンシングデータをもたらす可能性があります34。35、36、37。RNAの品質は、 表1 にRINで示され、 図2 に低濃度(PA565-3)および高濃度(PA288-1)のサンプルに対応するRNAエレクトロフェログラムで示されています。残りの2つのサンプル(PA375-3およびPA921-1)は、サンプルの大部分を代表しています。 図2に示すように、これらの量のバイオフィルムから抽出されたRNAには、常に劣化したRNAが含まれており、これがRIN値に影響を与えます。したがって、16Sおよび23S原核生物のrRNAピークの目視確認は、RINが報告されない場合のRNA品質を決定するために使用されます。これらの基準を使用して、51 個の RNA サンプルすべてが選択され、rRNA の枯渇とシーケンシングに進むと、最も品質の低い PA565-3 のサンプルが失敗する可能性があると予測されます。シーケンシングのために提出されたRNAサンプルのうち、成功したライブラリが生成され、49のサンプルに対してシーケンシングが行われ、PA565-3は予測どおりに失敗しました。
良好なRIN(PA288-1)を持つ高濃度サンプルとRINを含まない低濃度サンプル(PA375-3)で生成されたシーケンシングリードの数を 表2に示します。基本的な統計には、読み取りの合計数、読み取りの長さ、GC コンテンツが含まれます。両方のサンプルのサマリー統計量は、生成されたリードの数が多く、品質が低いとフラグが立てられたものがないことを示しており、良好なシーケンシングデータを示唆しています。サンプルあたりの平均リード数は約4,800万回で、これは良好な収率と考えられています。生のシーケンシングデータの品質を評価するために使用される標準ツールはFastQC26です。このプログラムは、生のシーケンシングファイルに対して品質管理チェックを行い、さらなる解析に十分な品質であるかどうか、またはシーケンサー自体またはインプットRNAライブラリから生成されたデータに問題やバイアスがあるかどうかを判断するために使用されます。 図3 は、PA288-1およびPA375-3シーケンシングデータの品質管理指標を示しており、それぞれ高品質のRNAサンプルと典型的な低品質のRNAサンプルを表しています。FastQCのより有益なプロットの1つは、Per Base Sequence Qualityプロットです。良好なシーケンシング データでは、すべてのリードの各位置で高い品質スコアの中央値 (>30) が示され、リードの長さにわたって平均品質スコアが低下します。品質スコア 30 は、エラー率が 1000 人に 1 人で、基本コールの精度が 99.9 % であることに相当します。 図 3 の塩基の大部分は、両方のサンプルの読み取り全長にわたって平均品質スコアが 35 ≥ており、これは非常に高品質のシーケンシングデータを示しています。これは、ここで紹介したRNA抽出プロトコルが成功しているという強力な証拠を提供します。
私たちの方法が緑膿菌の転写産物を回復できることを示すために、PA288-1およびPA375-3の高品質シーケンシングリードを緑膿菌PAO1参照ゲノム(NCBI NC_002516.2)にマッピングします。リードは、シーケンシングアダプターのためだけにトリミングされ、品質のためにはトリミングされず、マッピング32の前にすべてのベースが保持されます。マッピング統計を表 3 に示します。マッピングされたリードの割合は、シーケンシングの精度の重要な尺度です。簡単に言えば、参照配列に整列するリードが多ければ多いほど良いのです。PA288-1とPA375-3では、それぞれリードの84%と91%がリファレンスゲノムにマッピングされます。標準的なRNA-seqリードのマッピングの予想範囲は70〜90%であるため、特に低品質の塩基が除去されていない場合、これらの値は非常に良好です38。平均読み取り深度は、参照シーケンスの各ベース位置に整列する平均読み取り数を示す優れた指標です。各ベース位置の深さが大きければ大きいほど、各位置でのベースコールの精度が高くなります。これは、参照ゲノムの各位置でマッピングされた読み取り深度の合計を、参照内の塩基の総数で割ることによって計算されます。PA288-1およびPA375-3は、平均読み取り深度が400以上であり、これは下流の遺伝子発現解析39,40に適している。カバレッジの幅は、シーケンシング41によってカバーされる参照ゲノムの長さのパーセンテージを示す。PA288-1とPA375-3の両方からのアラインメントされたリードは、緑膿菌PAO1参照ゲノムの96%をカバーしています。このことは、緑膿菌のゲノムの大部分がシーケンシングデータに表れており、短いストレッチだけではないことを示唆しています。PA288-1およびPA375-3のマッピング統計は、この方法が緑膿菌のゲノムへの良好なカバレッジと分布に一致する転写産物を回復できることを示しており、成功した抽出プロトコルをさらにサポートしています。
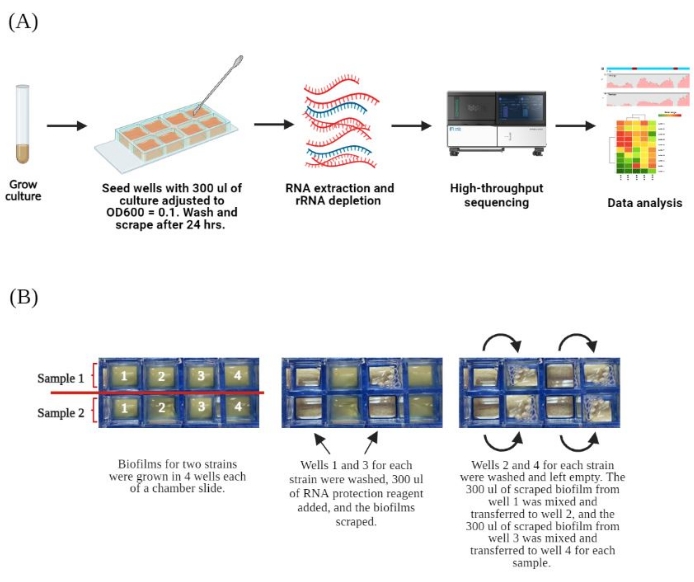
図 1.プロトコルの概要。ある。 実験ワークフローの概略図。プランクトン培養物を37°Cで一晩培養し、翌日新鮮な培地で1:100に希釈し、さらに3時間培養しました。培養物をOD600 0.1および300 μLに調整し、8チャンバースライドの4ウェルに接種してバイオフィルムを生成しました。24時間後、バイオフィルムを洗浄してプランクトン細胞を除去しました。RNA保護試薬を添加し、ウェルから細胞を掻き取りました。全RNAを抽出し、リボソームRNAを枯渇させ、配列を決定しました。 イ. 8チャンバースライドでのバイオフィルムの成長と除去の詳細なワークフロー。スライドごとに2つの株を左に示す向きで成長させました。スライドの4つのウェルにおける各サンプルの配置が示されています。各独立したサンプルについて、ウェル1と3を最初に処理し、最後に各ウェルのRNA保護試薬に300 μLの細胞を掻き取りました。この例は、中央のスライドに示されています。次に、ウェル2と4を洗浄し、液体を除去しました。ウェル1および3から再懸濁されたセルは、右側のスライドに示されているように、それぞれウェル2および4に移されます。掻き取り後、ウェル2および4に再懸濁した細胞を1本の微量遠心チューブに結合します。このフィギュアは BioRender.com で作成されました。 この図の拡大版を表示するには、ここをクリックしてください。

図 2.高品質の抽出された RNA サンプルから低品質の RNA サンプルまでの RNA エレクトロフェログラムの例。16Sおよび23Sリボソームピークは、ピークの基部にラベルが付けられています。分解されたRNAは、矢印で示される小さなサイズのピークと、ゼロより上のでこぼこしたベースラインで表されます。許容可能な品質の RNA サンプルをパネル A、B、D に示します。パネルCは、リボソームのピークが欠落しており、Y軸のスケールで示されているように濃度が非常に低かった品質のRNAサンプルを示しています。FU、蛍光ユニット;NT、ヌクレオチド。この図の拡大版を表示するには、ここをクリックしてください。

図 3.PA288-1およびPA375-3配列データのFastQC Per Base Sequence Qualityプロット。ある。高品質サンプルPA288-1の品質プロット。イ.一般的なサンプルPA375-3の品質プロット。プロットには、ファイル内のすべての読み取りについて、各ベース位置の集計品質スコアが表示されます。青い線は、各ベースポジションの平均品質スコアを表しています。黄色のボックス内の赤い線は各ポジションの品質スコアの中央値を表し、黄色のボックスは 25パーセンタイルから 75 パーセンタイルの四分位範囲を示しています。この図の拡大版を表示するには、ここをクリックしてください。
| 隔離する | 量子ビット | 鈴 | シーケンス |
| PA288-1 | 26 ng/μl | 7.5 | はい |
| PA375-3 | 4.07 ng/μl | NAの | はい |
| PA565-3 | 3.4 ng/μl | NAの | いいえ |
| PA921-1 | 9.11 ng/μl | 6.6 | はい |
テーブル 1. 代表的なサンプルから抽出されたRNAの品質指標。
| PA288-1 | PA375-3 | |
| 合計読み取り数 | 8,59,57,720 | 3,18,49,575 |
| 低品質としてフラグが付けられた読み取り | 0 | 0 |
| 読み取り長さ | 100 | 100 |
| %GCさん | 61 | 60 |
テーブル2. PA288-1 および PA375-3 FastQC サマリー統計量
| 隔離する | % 参照にマップされた読み取り | 平均読み取り深度 | カバレッジの幅広さ |
| PA288-1 | 83.93% | 404 | 96.68% |
| PA375-3 | 91.2% | 578 | 96.97% |
テーブル3。 PA288-1 および PA375-3 マッピング統計
ディスカッション
17種類の細菌バイオフィルムサンプルからトータルRNAを三重に抽出し、合計51のサンプルが得られます。49のRNAライブラリーがプールされ、成功裏に配列決定されました。全体として、これは、サンプルの半分以上が存在量が少なく、最適ではない品質であると考えられているにもかかわらず、96%の成功率で私たちの品質基準を検証しています34,35,36,37。
意味
このRNA抽出プロトコルは、チャンバースライドで成長した限られた量のバイオフィルムからRNAを抽出する詳細な説明においてユニークです。8ウェルチャンバースライドにおけるバイオフィルムの成長は、バイオフィルムへの外因的に添加された因子の影響やバイオフィルム形成における微生物相互作用の影響を共焦点顕微鏡法によって研究するための有用なシステムである12,13,42。本システムにおけるバイオフィルム形成に関与する遺伝子の発現を調べるために、RNAシーケンシングに十分な質と量のインタクトRNAを抽出するための詳細な方法を提示する。他の研究では、限られた量のバイオフィルムからRNA抽出が成功したと報告されていますが、大多数はより大きな表面積でバイオフィルムを成長させ、多くの場合、24時間ではなく48時間成長させます。また、成功を確実にするための十分な詳細も欠けています4,15,16,43,44,46。
さらに、このプロトコルは、危険な化学物質(フェノールなど)や特殊な機器(超音波処理器など)の使用を回避します。古典的なグアニジニウムチオシアン酸フェノール-クロロホルム抽出プロトコル47は、市販のキットと比較して約2倍のRNAを生成するにもかかわらず、私たちの手では、自動電気泳動システムで評価されるように、一貫して完全に分解されたRNAをもたらすため、このシステムでは使用されません。さらに、市販の抽出キットの使用は、一貫した結果をもたらすユーザーフレンドリーなプロトコルをもたらすことが示されている15,16,45。
クリティカルステップ
このプロトコルには、配列決定が成功するRNAを抽出する確率を高めるための重要なステップがいくつかあります。まず、配列決定に十分な量のRNAを得るためには、チャンバースライドの少なくとも4つのウェルに同じ株を接種することが重要です。4つのウェルからのバイオフィルムは、単一の抽出のためにプールされ、これには、下流の遺伝子発現解析17のばらつきを減らすという追加の利点があります。4ウェル未満のウェルからRNAを抽出すると、高感度の蛍光測定システムで検出するには低すぎる収量になることがよくあります。8ウェルまたはスライド全体からRNAを抽出すると、4ウェルを使用した場合と同様の品質のRNAが大量に得られますが、抽出のための追加の時間、労力、およびコストが増加した収率に見合うかどうかを考慮する必要があります。標準の200 μLではなく、300 μLのOD600 = 0.1希釈培養物を各ウェルに播種すると、各ウェルから掻き取りされたバイオフィルム材料の回収率が向上します。RNA保護試薬を塗布する前に、バイオフィルムをヌクレアーゼフリーの水で2回優しく洗浄することは、死細胞やプランクトン細胞をできるだけ多く除去するために重要です。RNA保護試薬の使用は、各ウェルの処理に時間がかかるため、RNAの分解を防ぐために重要です。さらに、ピペットチップで掻き取るのではなく、ピペットチップよりも大きな表面積に接触する平らな端を持つ金属製の計量スパチュラを使用することを好みます。1000 μLのピペットチップを使用したスクレイピングは機能しますが、金属製のスパチュラに比べてバイオフィルム全体をスクレイピングする効率が悪く、時間がかかります。私たちの経験では、金属製のヘラを使用すると、時間と労力を節約できます。こすり落とすときは、ウェルの底にひびが入らないように、チャンバースライドをガラスプレートの上に置いてください。超音波処理は、サンプル量が少なすぎると発熱が多すぎるため、一貫してRNAが高度に分解されるため、効果的ではありません。バイオフィルム材料を採取した後、サンプルを市販のキットにインプットして、より信頼性と再現性の高いRNA抽出を行います。最後に、この方法には潜在的な変動源があるため、サンプルプーリング17,48に加えて実験的複製を含めることが非常に重要です。このプロトコルは、各サンプルの三重の生物学的複製を説明しています。
制限
研究用のバイオフィルムを成長させるために使用されるさまざまな技術がありますが、その中で最も一般的なのはマイクロタイタープレート4での形成です。これらのプレートは、さまざまなサイズと数のウェルで利用できます。チャンバースライドはこのカテゴリに分類されます。無傷の純粋なRNAを抽出する能力は、すべてのバイオフィルムシステムにとって重要ですが、このシステムはRNAの低収率によって制限されます。濃度は十分に低いため、その品質と量は、高感度 RNA キットを使用して Qubit および Bioanalyzer 機器で評価する必要があります。ナノドロップも使用できますが、Qubitに比べて感度が限られており、汚染DNAとRNA49を区別することはできません。可能であれば、ナノドロップを使用して、純度のA260/A280 およびA260/A230 の比率を得る必要があります。これらの比率は、特にこのバイオフィルムシステムからのRNA品質が大量の出発物質から得られるものほど良くないため、有用です。これは、RNAの収量が低く、バイオフィルムやRNaseに死細胞が存在するため、RNAを分解する可能性があるためである可能性が最も高いです。この方法の別の制限は、不均一なバイオフィルム内で異なる細胞タイプを分離できず、細胞を集団全体としてのみ抽出できるため、過小評価されている亜集団の遺伝子発現が不明瞭になる可能性があることです。
潜在的なアプリケーション
ここで紹介するプロトコールは、チャンバースライドで増殖させた臨床分離株由来の 緑膿菌バイオ フィルムに最適化されています。この方法は、共焦点顕微鏡で観察されるさまざまな成長条件下でのバイオフィルム形成中の遺伝子発現の変化を研究するために使用できます。このプロトコルは、他のバイオフィルム生成細菌種にも最適化できます。この方法では、デュアル抽出キットを使用できるため、ゲノムDNAを抽出することも可能です。このようにして、患者の感染症を引き起こす臨床分離株における細菌バイオフィルム遺伝子発現をより正確に研究することができ、将来の治療戦略を導く可能性のある結果につながります。
開示事項
著者には、宣言する開示はありません。
謝辞
著者の貢献:P.W.、Y.Y.、V.W.が研究の概念化に関与しました。K.G.、L.J.、A.M.、P.W.は、ラボのプロトコルを最適化しました。K.G.の資金は、BioTalent CanadaによるStudent Work Placement Program補助金によって支えられました。
資料
| Name | Company | Catalog Number | Comments |
| Agilent 2100 Bioanalyzer | Agilent | G2939BA | Automated electrophoresis of biomolecules |
| Agilent RNA 6000 pico kit | Agilent | 5067-1513 | High sensitivity RNA electrophoresis chip to generate a RIN |
| DNA/RNA Lysis Buffer | Zymo Research | D7001-1-50 | A guanidinium thiocyanate and N-Lauroylsarcosine-based lysis buffer sold as part of a nucleic acid purification kit |
| DNA/RNA Prep Buffer | Zymo Research | D7010-2-10 | A guanidine HCl and ethanol buffer used for purification of DNA and RNA |
| DNA/RNA Shield | Zymo Research | R1100-50 | DNA and RNA preservation/protection reagent |
| DNA/RNA Wash Buffer | Zymo Research | D7010-3-6 | A salt and ethanol buffer used for purification of DNA and RNA |
| DNBSEQ G-400RS | MGI | G-400RS | High throughput sequencer |
| MGIEasy RNA Directional Library Prep Set | MGI | 1000006386 | Generate libraries for MGI high-throughput sequencing platforms from total RNA. |
| Mini-Beadbeater-96 | BioSpec | 1001 | A high energy, high throughput cell disrupter |
| NEBNext rRNA Depletion Kit (bacteria) | New England Biolabs | E7850X | Efficient and specific depletion of bacterial rRNA (5S, 16S, 23S) |
| Nunc Lab-Tek II chamber slide system | Thermo Fisher Scientific | 154534 | 8-well chamber slide with removable wells |
| Qubit Fluorometer | Thermo Fisher Scientific | Q33238 | Fluorometer for DNA, RNA and proteins |
| Qubit RNA HS Assay Kit | Thermo Fisher Scientific | Q32852 | High sensitivity fluorometric assay to measure RNA concentration |
| Spin-Away Filters | Zymo Research | C1006-50-F | Silica-based spin column primarily used to bind or remove genomic DNA |
| Sterile inoculation loops, 1 uL | Sarstedt | 86.1567.050 | Sterile, disposable inoculation loops for manipulation of microorganisms |
| ZR BashingBead Lysis tubes | Zymo Research | S6003-50 | 2 mL tubes containing 0.1 and 0.5 mm bead lysis matrix for homogenizing biological samples |
| Zymo Spin IIICG Columns | Zymo Research | C1006-50-G | Silica-based spin column for purification of DNA and RNA |
| Zymo-Spin III-HRC Filters | Zymo Research | C1058-50 | Remove inhibitors such as polyphenolic compounds, humic/fulvic acids, tannins, melanin, etc. |
| Zymobiomics DNA/RNA Miniprep kit | Zymo Research | R2002 | DNA and RNA dual extraction kit |
| Zymobiomics HRC Prep solution | Zymo Research | D4300-7-30 | To be used with Zymo-Spin III-HRC Filters to remove PCR inhibitors |
参考文献
- Beaudoin, T., Waters, V. Infections With Biofilm Formation: Selection of Antimicrobials and Role of Prolonged Antibiotic Therapy. The Pediatric Infectious Disease Journal. 35 (6), 695-697 (2016).
- Bjarnsholt, T., et al. The in vivo biofilm. Trends in Microbiology. 21 (9), 466-474 (2013).
- Folsom, J. P., et al. Physiology of Pseudomonas aeruginosa in biofilms as revealed by transcriptome analysis. BMC Microbiology. 10, 294(2010).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43 (3), 313-351 (2017).
- Bjarnsholt, T., Ciofu, O., Molin, S., Givskov, M., Hoiby, N. Applying insights from biofilm biology to drug development - can a new approach be developed. Nature Reviews Drug Discovery. 12 (10), 791-808 (2013).
- Colvin, K. M., et al. The Pel and Psl polysaccharides provide Pseudomonas aeruginosa structural redundancy within the biofilm matrix. Environmental Microbiology. 14 (8), 1913-1928 (2012).
- Hentzer, M., et al. Alginate overproduction affects Pseudomonas aeruginosa biofilm structure and function. Journal of Bacteriology. 183 (18), 5395-5401 (2001).
- Cury, J. A., Koo, H. Extraction and purification of total RNA from Streptococcus mutans biofilms. Analytical Biochemistry. 365 (2), 208-214 (2007).
- Francavilla, M., et al. Extraction, characterization and in vivo neuromodulatory activity of phytosterols from microalga Dunaliella tertiolecta. Current Medicinal Chemistry. 19 (18), 3058-3067 (2012).
- Atshan, S. S., et al. Improved method for the isolation of RNA from bacteria refractory to disruption, including S. aureus producing biofilm. Gene. 494 (2), 219-224 (2012).
- Franca, A., Melo, L. D., Cerca, N. Comparison of RNA extraction methods from biofilm samples of Staphylococcus epidermidis. BMC Research Notes. 4, 572(2011).
- Jurcisek, J. A., Dickson, A. C., Bruggeman, M. E., Bakaletz, L. O. In vitro biofilm formation in an 8-well chamber slide. The Journal of Visusalized Experiments. (47), e2481(2011).
- Beaudoin, T., Kennedy, S., Yau, Y., Waters, V. Visualizing the effects of sputum on biofilm development using a chambered coverglass model. The Journal of Visusalized Experiments. (118), e54819(2016).
- Cockeran, R., et al. Biofilm formation and induction of stress response genes is a common response of several serotypes of the pneumococcus to cigarette smoke condensate. The Journal of Infection. 80 (2), 204-209 (2020).
- Bisht, K., Moore, J. L., Caprioli, R. M., Skaar, E. P., Wakeman, C. A. Impact of temperature-dependent phage expression on Pseudomonas aeruginosa biofilm formation. npj Biofilmsand Microbiomes. 7 (22), (2021).
- Harrison, A., et al. Reprioritization of biofilm metabolism is associated with nutrient adaptation and long-term survival of Haemophilus influenzae. NPJ Biofilms and Microbiomes. 5 (1), 33(2019).
- Sousa, C., Franca, A., Cerca, N. Assessing and reducing sources of gene expression variability in Staphylococcus epidermidis biofilms. BioTechniques. 57, 295-301 (2014).
- Boom, R. Rapid and simple method for purification of nucleic acids. Journal of Clinical Microbiology. 28 (3), 495-503 (1990).
- Coppin, C. Re: How do silica based RNA spin columns only bind RNA and not DNA. , Available from: https://www.researchgate.net/post/How_do_silica_based_RNA_spin_columns_only_bind_RNA_and_not_DNA/60b017bffa5c4151cac1c/citation/download (2021).
- Kennedy, S. A complete guide to how nucleic extraction kits work. , Available from: https://bitesizebio.com/13516/how-dna-extraction-rna-miniprep-kits-work/ (2021).
- Qubit RNA HS Assay Kit User Guide. Thermo Fisher Scientific. , Available from: https://www.thermofisher.com/document-connect/document-connect.html?url=https%3A%2F%2Fassets.thermofisher.com%2FTFS-Assets%2FLSG%2Fmanuals%2FQubit_RNA_HS_Assay_UG.pdf&title=VXNlciBHdWlkZTogUXViaXQgUk5BIEhTIEFzc2F5IEtpdHM (2015).
- Mueller, O., Lightfoot, S., Schroeder, A. RNA Integrity Number (RIN) - Standardization of RNA Quality Control (Application report # 5989-1165EN). , Available from: https://www.agilent.com/cd/library/applications/5989-1165EN.pdf (2016).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 3(2006).
- Culviner, P. H., Guegler, C. K., Laub, M. T. A Simple, Cost-Effective, and Robust Method for rRNA Depletion in RNA-Sequencing Studies. mBio. 11 (2), (2020).
- MGIEasy RNA Directional Library Prep Set User Manual verA2. MGI Tech Co. , Available from: https://en.mgi-tech.com/products/reagents_info/14/ (2020).
- Andrews, S. FastQC: A Quality Control Tool for High Throughput Sequence Data. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina Sequence Data. Bioinformatics. , (2014).
- NCBI Resource Coordinators. Database resources of the National Center for Biotechnology Information. Nucleic Acids Research. 44 (1), (2016).
- Li, H. Aligning sequence reads, clone sequences and assembly contigs with BWA-MEM. arXiv. , (2013).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), (2009).
- Liao, Y., Shi, W. Read trimming is not required for mapping and quantification of RNA-seq reads at the gene level. NAR Genomics and Bioinformatics. 2 (3), (2020).
- Penir, S. Calculating Mapping Statistics from a SAM/BAM file using SAMtools and awk. , Available from: https://sarahpenir.github.io/bioinformatics/awk/calculating-mapping-stats-from-a-bam-file-using-samtools-and-awk/ (2019).
- Haile, S., et al. Evaluation of protocols for rRNA depletion based RNA sequencing of nanogram inputs of mammalian total RNA. PLoS ONE. 14 (10), 0224578(2019).
- Schuierer, S., et al. A comprehensive assessment of RNA-seq protocols for degraded and low-quantity samples. BMC Genomics. 18 (442), (2017).
- Shanker, S., et al. Evaluation of Commercially Available RNA Amplification Kits for RNA Sequencing Using Very Low Input Amounts of Total RNA. Journal of Biomolecular Techniques. 26 (1), (2015).
- Adiconis, X., et al. Comparative analysis of RNA sequencing methods for degraded or low-input samples. Nature Methods. 10, 623-629 (2013).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (13), (2016).
- Illumina, Inc. Coverage depth recommendations. , Available from: https://www.illumina.com/science/technology/next-generation-sequencing/plan-experiments/coverage.html (2021).
- What is a good sequencing depth for bulk RNA-Seq. ECSEQ Bioinformatics. , Available from: https://www.ecseq.com/support/ngs/what-is-a-good-sequencing-death-for-bulk-rna-seq (2019).
- Bedre, R. Sequencing coverage and breadth of coverage. , Available from: https://www.reneshbedre.com/blog/sequencing-coverage.html (2021).
- Dotsch, A., et al. The Pseudomonas aeruginosa transcriptome in planktonic cultures and static biofilms using RNA sequencing. PLoS One. 7 (2), 31092(2012).
- Chen, Y., et al. Population dynamics and transcriptomic responses of Pseudomonas aeruginosa in a complex laboratory microbial community. npj Biofilms and Microbiomes. 5 (1), (2019).
- Thoming, J. G., et al. Parallel evolutionary paths to produce more than one Pseudomonas aeruginosa biofilm phenotype. NPJ Biofilms and Microbiomes. 6, 2(2020).
- Soares, A., et al. Understanding ciprofloxacin failure in Pseudomonas aeruginosa biofilm: persister cells survive matrix disruption. Frontiers in Microbiology. 10, 2603(2019).
- Whiteley, M., et al. Gene expression in Pseudomonas aeruginosa biofilms. Nature. 413, (2001).
- Chomczynski, P., Sacchi, N. The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: twenty-something years on. Nature Protocols. 1, (2006).
- Liu, Y., Zhou, J., White, K. P. RNA-seq differential expression studies: more sequence or more replication. Bioinformatics. 30 (3), (2014).
- Wieczorek, D., Delauriere, L., Schagat, T. Methods of RNA Quality Assessment. , Available from: https://www.promega.ca/resources/pubhub/methods-of-rna-quality-assessment/ (2021).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved