Method Article
Isolement de l’ARN total de Pseudomonas aeruginosa dans des biofilms pour la mesure de l’expression génique
Dans cet article
Résumé
Ce protocole présente une méthode pour isoler l’ARN des biofilms de Pseudomonas aeruginosa cultivés dans des lames de chambre pour un séquençage à haut débit.
Résumé
Pseudomonas aeruginosa est un pathogène bactérien opportuniste qui provoque des infections des voies respiratoires des patients atteints de mucoviscidose (FK). P. aeruginosa est connu pour sa capacité à former des biofilms protégés par une matrice d’exopolysaccharides. Cette matrice permet aux micro-organismes d’être plus résistants aux facteurs externes, y compris le traitement antibiotique. L’une des méthodes les plus courantes de croissance du biofilm pour la recherche est l’utilisation de plaques de microtitration ou de lames chambrées. L’avantage de ces systèmes est qu’ils permettent de tester plusieurs conditions de croissance, mais leur inconvénient est qu’ils produisent des quantités limitées de biofilm pour l’extraction de l’ARN. Le but de cet article est de fournir un protocole détaillé, étape par étape, sur la façon d’extraire l’ARN total de petites quantités de biofilm de qualité et de quantité suffisantes pour le séquençage à haut débit. Ce protocole permet d’étudier l’expression des gènes au sein de ces systèmes de biofilms.
Introduction
La plupart des infections bactériennes chroniques, telles que les infections pulmonaires chez les patients atteints de mucoviscidose (FK) et les infections liées aux prothèses, sont caractérisées par la croissance d’organismes dans les biofilms. Les biofilms1 sont des communautés de bactéries enfermées dans une matrice composée principalement de polysaccharides2. Les bactéries présentes dans les biofilms peuvent être à croissance lente, métaboliquement dormantes et dans des conditions anaérobies et hypoxiques. Les biofilms sont plus résistants aux antibiotiques en raison de facteurs tels que la diminution de la pénétration des antibiotiques, l’augmentation de l’expression des pompes d’efflux de médicament et la diminution de la division cellulaire3. Pour ces raisons et d’autres, ils présentent un grand intérêt pour la recherche.
Afin d’étudier avec précision les infections persistantes telles que les infections chroniques à Pseudomonas aeruginosa chez les patients atteints de mucoviscidose, les conditions de croissance observées avec la formation de biofilms doivent être reflétées avec précision in vitro. Une méthode courante à haut débit consiste à les cultiver dans des lames de chambre ou des plaques de microtitration et à surveiller la formation de biofilms par microscopie confocale4. On sait qu’un régulateur clé dans la transition d’un mode de vie bactérien planctonique, ou flottant libre, à un mode de vie bactérien à biofilm est le messager secondaire, cyclique-di-GMP5. L’augmentation des niveaux cycliques de di-GMP augmente l’expression de gènes spécifiques qui favorisent la croissance du biofilm. Les petits ARN régulateurs non codants et la détection du quorum jouent également un rôle important dans la régulation de la formation du biofilm5. Mesurer l’expression génique d’un biofilm par séquençage de l’ARN bactérien extrait peut s’avérer difficile. P. aeruginosa, par exemple, produit trois exopolysaccharides (Psl, Pel et alginate), qui sont produits en quantités importantes dans les biofilms 6,7. Ces polysaccharides peuvent interférer avec l’extraction et la purification de l’ARN, conduisant à des préparations impures contenant de faibles niveaux d’ARNm 8 bactérien. Les kits d’extraction d’ARN disponibles dans le commerce sont capables de produire de l’ARN de haute qualité à partir de cultures bactériennes planctoniques, mais peuvent ne pas fonctionner aussi bien avec les cultures de biofilm 9,10,11. Il existe quelques kits commerciaux d’extraction d’ARN qui prétendent fonctionner pour les biofilms, dont l’un que nous utilisons avec cette méthode.
Dans ce manuscrit, nous décrivons les procédures de croissance des biofilms de P. aeruginosa dans des lames de chambre et d’extraction de l’ARNm bactérien pour le séquençage à haut débit12,13. À l’aide d’isolats cliniques prélevés sur des échantillons d’expectorations de patients atteints de mucoviscidose, nous démontrons que ces méthodes peuvent être utilisées pour des isolats présentant des caractéristiques de croissance variables. Par rapport aux publications précédentes, ce protocole est décrit en détail pour permettre un meilleur succès dans l’étude de l’expression génique du biofilm bactérien 11,14,15,16.
Protocole
Le comité d’éthique de la recherche (CER) est requis pour la collecte et le traitement d’échantillons d’expectorations provenant de sujets humains. Cette étude a été approuvée par l’Hôpital pour enfants malades (REB#1000019444). Le comité d’éthique de la recherche (CER) est tenu de prélever et d’entreposer des échantillons d’expectorations provenant de sujets humains. Ces études ont été approuvées par le CER#1000058579 de l’Hôpital pour enfants malades.
1. Formation du biofilm
- Cultivez pendant la nuit des isolats de Pseudomonas aeruginosa obtenus à partir d’échantillons d’expectorations de patients atteints de mucoviscidose utilisés dans cette étude sur des plaques de gélose Luria Broth (LB) dans un incubateur à 37 °C.
REMARQUE : Une bonne technique de stries est importante pour obtenir des colonies bactériennes uniques. Les stries pendant la rotation de la plaque dilueront suffisamment les cellules bactériennes pour que des colonies individuelles puissent se développer.- Striez les bactéries à l’aide d’une boucle d’inoculation en zigzag à l’extrémité supérieure d’une plaque LB fraîche jusqu’à ce qu’environ 1/4 de la plaque soit couverte.
- Faites pivoter la plaque d’environ 60°. Prenez une nouvelle boucle d’inoculation et passez-la une fois à travers la zone striée et dans une deuxième zone propre de la plaque, en répétant le motif en zigzag.
- Répétez l’étape 1.1.2 avec une boucle fraîche dans une troisième zone de la plaque. Remettez le couvercle et retournez la plaque lorsque vous la placez dans l’incubateur.
- À l’aide d’une boucle d’inoculation stérile, prélever une seule colonie bactérienne dans une plaque de gélose (contenant une seule souche bactérienne) et inoculer un tube de culture rempli de 5 mL de milieu stérile LB. À l’aide d’une nouvelle boucle à chaque fois, inoculer deux tubes de culture supplémentaires remplis de 5 mL de milieu LB à partir de la même plaque.
- Répéter la même procédure d’inoculation pour les différentes souches bactériennes. Faites pousser les cultures pendant la nuit à 37 °C en secouant à 220 tr/min.
REMARQUE : Chaque plaque de gélose contenant une seule souche bactérienne est utilisée pour inoculer 3 tubes de culture indépendants. Les trois tubes représentent des répétitions biologiques en triple exemplaire pour une souche et sont traités comme des échantillons distincts. Ceci est différent des réplicats techniques qui impliqueraient d’extraire 3 fois l’ARN d’un seul tube de culture. - Préparez des dilutions de 1:100 des cultures de nuit en transférant 50 μL d’une culture de nuit dans un nouveau tube de culture contenant 4,95 mL de nouveau milieu LB.
- Faites pousser les cultures diluées pendant encore 3 h à 37 °C en agitant à 220 tr/min ou jusqu’à ce que la DO600 soit de 0,1 ou plus. Mesurez les densités cellulaires sur un spectrophotomètre àOD 600.
- Dans un nouveau tube microfuge de 1,7 ml, ajustez l’OD600 à 0,1 (phase logarithmique précoce) dans un volume total de 1,5 mL avec LB frais.
- Mélanger délicatement par inversion. Transférez 300 μL de chaque culture ajustée dans 4 puits d’une lame de chambre à 8 puits pour obtenir 2 échantillons différents par lame (figure 1).
- Placez les lames, sans les déranger, dans un incubateur à 37 °C pendant la nuit pendant 24 h. Pour éviter l’évaporation, placez les lames sur une serviette en papier humide dans un petit plateau en plastique.
2. Récupération du biofilm
REMARQUE : Chaque lame de verre contient huit puits séparés. Un seul échantillon se compose de quatre puits avec des biofilms qui seront regroupésen 17. Ce protocole d’extraction porte sur 1 échantillon (4 puits) où les biofilms sont récupérés à partir de 2 puits à la fois. Les extractions d’ARN sont effectuées à l’aide d’un kit d’extraction d’ARN commercial qui comprend une étape de battage de billes et un nettoyage sur colonne, avec des modifications. Suivez les instructions du fabricant pour la préparation du réactif.
- Dans une hotte à flux laminaire, retirez lentement le support de 2 des 4 puits à l’aide d’une pointe de pipette. Inclinez la glissière à un angle de 45° et faites sortir le média à partir du coin inférieur des puits pour éviter le détachement des biofilms.
- En gardant la lame inclinée, lavez les cellules planctoniques en pipetant doucement 300 μL d’eau sans RNase dans le coin inférieur des deux puits vidés. Retirez l’eau en la pipetant doucement, comme décrit à l’étape 2.1. Répétez l’étape de lavage en enlevant autant d’eau que possible.
- Ajouter 300 μL d’un réactif de protection à ARN (voir le tableau des matériaux) dans chacun des deux puits vidés avec des biofilms à leur base. Placez la lame de la chambre sur une plaque de verre pour éviter que les puits ne se brisent, puis raclez les biofilms dans les 2 puits avec une petite spatule métallique stérile sans nucléase pour remettre en suspension les bactéries du biofilm. Laisser reposer jusqu’à ce que les biofilms soient récupérés des 2 puits intacts restants du même échantillon.
REMARQUE : L’ajout d’un réactif de protection à ARN assure la stabilité des échantillons de biofilm dans les puits raclés à température ambiante pendant le traitement des deux puits restants du même échantillon. Le réactif de protection de l’ARN lyse les cellules et inactive les nucléases et les agents infectieux, ce qui permet de préserver l’ARN. - Pour récupérer les biofilms des 2 puits restants pour un échantillon (rappel : un échantillon est composé de 4 puits), prélever le milieu LB des 2 nouveaux puits restants de la même manière que décrit à l’étape 2.1. Répétez l’étape 2.2 pour laver les cellules planctoniques avec de l’eau sans RNase des deux nouveaux puits, comme précédemment.
- Retournez aux 2 premiers puits avec des biofilms grattés dans un réactif de protection, généré à la fin de l’étape 2.3, et pipetez lentement pour mélanger les 300 μL de biofilm remis en suspension à partir d’un puits gratté, en essayant de ne pas créer trop de bulles.
- Transférez tout le contenu du puits dans l’un des puits nouvellement lavés et vidés. Mélanger et transférer 300 μL de biofilm remis en suspension du deuxième puits gratté dans le puits restant, nouvellement lavé et vidé.
REMARQUE : Au lieu d’ajouter un nouveau réactif de protection à ARN dans les 2 puits nouvellement lavés avec des biofilms, transférez les biofilms précédemment grattés déjà dans le réactif de protection dans ces puits fraîchement lavés. Cela permettra de maintenir le volume d’échantillon combiné suffisamment bas pour répondre aux exigences d’entrée du kit d’extraction d’ARN commercial. Voir la figure 1 pour un schéma.
- Transférez tout le contenu du puits dans l’un des puits nouvellement lavés et vidés. Mélanger et transférer 300 μL de biofilm remis en suspension du deuxième puits gratté dans le puits restant, nouvellement lavé et vidé.
- Répétez le grattage des biofilms dans les nouveaux puits comme à l’étape 2.3, en plaçant la lame de la chambre sur une plaque de verre et en grattant les biofilms dans les 2 nouveaux puits avec une petite spatule métallique stérile sans nucléases pour remettre en suspension les bactéries du biofilm.
- Combinez tout le biofilm remis en suspension des 2 nouveaux puits dans un seul tube de microcentrifugation de 1,5 mL sans RNase et à faible liaison. Mesurez le volume, qui doit être de ~500 à 600 μL au total.
REMARQUE : La suspension combinée du biofilm de cette deuxième paire de nouveaux puits contiendra tout le matériau du biofilm des 4 puits originaux d’un échantillon.
3. Isolement total de l’ARN et évaluation de la qualité
REMARQUE : L’extraction de l’ARN est effectuée à l’aide d’un kit d’extraction d’ARN commercial qui prétend fonctionner sur des biofilms. Les différents composants sont inclus dans la table des matériaux, si possible. Des explications sur les mécanismes à l’origine de chaque étape de purification sont fournies lorsque cela est possible.
- Ajouter suffisamment de réactif de protection à ARN pour totaliser 750 μL dans le tube. Transvasez tout le volume dans un tube de lyse de 2 mL contenant des billes de 0,1 et 0,5 mm (voir le tableau des matériaux). Battre pendant 2 1/2 min dans un batteur à perles à vitesse maximale.
REMARQUE : La combinaison de billes haute densité de 0,1 mm et 0,5 mm et d’un mélange à grande vitesse sur un batteur de billes assure une homogénéisation complète des parois cellulaires microbiennes. - Centrifuger à 16 000 x g pendant 1 min pour granuler les billes. Transférez le surnageant dans un nouveau tube de microcentrifugation, en minimisant le transfert de billes, ce qui facilitera l’étape 3.3. Mesurez le volume.
- Ajouter un volume égal de tampon de lyse de l’ARN (~450 μL) et bien mélanger. Transférez jusqu’à 800 μL d’échantillon, en évitant le transfert de billes, dans une colonne de silice dans un tube de collecte et centrifugez à 16 000 x g pendant 30 s. Conservez le flux continu car il contient l’ARN tandis que l’ADN est lié à la colonne.
REMARQUE : Le tampon de lyse de l’ARN contient du thiocyanate de guanidinium et le détergent N-lauroylsarcosine pour lyser les cellules. Le thiocyanate de guanidinium est un agent chaotrope qui inactive également les nucléases et, en présence de silice, présente dans la colonne de spin, favorise la liaison de l’ADN à la silice18. L’absence d’éthanol permet une liaison préférentielle de l’ADN et non de l’ARN à la colonne de spin de silice19. Le but de l’étape 3.3 est de lier et d’éliminer l’ADN génomique. Nous voulons conserver l’ARN, qui est contenu dans la partie accréditive. - S’il reste plus d’échantillon, transférez la colonne dans un nouveau tube de prélèvement et rechargez avec le reste de l’échantillon. Centrifugeuse à 16 000 x g pendant 30 s. Conservez le flux avec l’ARN et combinez avec la première aliquote.
- Mesurez le volume d’écoulement combiné et ajoutez un volume égal de 100 % d’éthanol et mélangez bien. Transférez jusqu’à 800 μL de la solution dans une nouvelle deuxième colonne de centrifugation de silice dans un tube de collecte et centrifugez à 16 000 x g pendant 30 s. Jetez le flux continu.
REMARQUE : L’ajout d’éthanol à la solution de sel chaotrope contenant l’ARN améliore la liaison de l’ARN à la colonne de spin de silice19. - Pour les solutions > 800 μL, rechargez la colonne de centrifugation et centrifugez jusqu’à ce que toute la solution soit essorée. Jetez le flux continu après chaque essorage.
- Ajouter 400 μL de tampon de lavage dans la colonne et centrifuger à 16 000 x g pendant 30 s pour éliminer une partie des sels. Jetez le flux continu.
- Préparez le mélange réactionnel DNase I selon les instructions du fabricant et effectuez le traitement DNase en colonne pour éliminer tout ADN résiduel.
- Remettre en suspension la DNase I lyophilisée dans 275 μL d’eau exempte de RNase pour obtenir une solution à 1 U/μL. Mélanger par inversion douce.
- Combinez 5 μL de DNase I diluée avec 75 μL du tampon de digestion DNase fourni. Mélanger délicatement par inversion.
- Ajouter 80 μL de la solution préparée directement sur la matrice de colonne. Incuber à température ambiante pendant 20 min.
- Ajouter 400 μL de tampon de préparation d’ARN dans la colonne et centrifuger à 16 000 x g pendant 30 s. Jetez le flux continu.
- Ajoutez 700 μL de tampon de lavage d’ARN dans la colonne et répétez la centrifugation. Jetez le flux continu.
- Ajoutez 400 μL de tampon de lavage d’ARN et centrifugez la colonne pendant 2 minutes pour éliminer complètement tout tampon résiduel.
REMARQUE : Il y a 2 étapes de lavage différentes pour éliminer les impuretés sur la colonne. Le tampon de préparation contient un sel chaotrope faible mélangé à de l’éthanol afin d’éliminer les protéines résiduelles. Ensuite, le tampon de lavage est utilisé pour effectuer des lavages à l’éthanol afin d’éliminer les sels. Tout éthanol restant doit être éliminé pour permettre une élution efficace de l’ARN19,20. - Transférez soigneusement la colonne dans un nouveau tube de microcentrifugation à faible liaison.
- Ajouter 50 μL d’eau exempte de RNase directement dans la matrice de la colonne et incuber pendant 5 min. Centrifuger à 16 000 x g pendant 1 min pour éluer l’ARN.
- Pour un nettoyage supplémentaire afin d’éliminer les inhibiteurs de PCR, placez un filtre inhibiteur de PCR dans un nouveau tube de collecte. Ajouter 600 μL de la solution de préparation d’inhibiteur fournie (voir le tableau des matériaux).
- Centrifuger à 8 000 x g pendant 3 min pour laver le filtre. Transférez le filtre lavé dans un nouveau tube de microcentrifugation à faible liaison.
- Transférez l’ARN élué de l’étape 3.13 dans le filtre lavé et centrifugez à 16 000 x g pendant 3 min.
REMARQUE : L’ARN peut être utilisé immédiatement ou stocké à -80 °C. - Déterminer la concentration de l’ARN à l’aide d’un système fluorométrique à haute sensibilité21.
REMARQUE : Ces systèmes permettent une quantification sensible d’une petite quantité d’ARN en solution spécifique à la cible d’intérêt.- Évaluez la qualité de l’ARN à l’aide d’un système d’électrophorèse automatisé qui peut fournir un RIN (RNA integrity number), qui est une mesure de la qualité de l’ARN22,23.
4. Déplétion de l’ARN ribosomique et séquençage à haut débit
- Soumettre des ARN au Centre d’analyse de l’évolution et de la fonction du génome (CAGEF) de l’Université de Toronto (Toronto, Canada) (https://www.cagef.utoronto.ca/) pour la déplétion de l’ARNr bactérien et la préparation de banques d’ARN directionnels (voir la table des matériaux).
REMARQUE : La déplétion de l’ARNr bactérien cible les ARNr 5S, 16S et 23S pour l’élimination24. - Épuisez les ARNr à l’aide d’un kit commercial de déplétion bactérienne d’ARNr. Suivez le protocole pour la saisie de 10 ng à 1 μg d’ARN total intact ou partiellement dégradé.
- Construisez des banques de séquençage d’ARN à l’aide d’un kit de préparation de bibliothèque directionnelle d’ARN avec différents index attachés à chaque bibliothèque.
- Regroupez les quantités équimolaires de chaque banque d’ARN et effectuez un séquençage à haut débit avec des lectures à 100 bases à extrémités appariées25.
5. Évaluation de la qualité des lectures de séquençage
REMARQUE : Vérifiez la qualité des lectures de séquençage à l’aide du programme disponible gratuitement, FastQC26, disponible via la plate-forme gratuite et open-source, Galaxy27.
- Allez à https://usegalaxy.org/. Cliquez sur le menu Connexion ou S’inscrire et connectez-vous avec vos identifiants ou créez un compte.
- Cliquez sur le lien Télécharger les données en haut à gauche de la page, sous le menu Outils, et téléchargez les fichiers de séquençage fastq.gz. Attendez que les noms de fichiers apparaissent sur le côté droit de la page, sous le panneau Historique.
- Sélectionnez Contrôle de qualité FASTQ dans le menu Outils pour afficher une liste de programmes. Sélectionnez FastQC, qui remplira le panneau central de l’écran.
- Sous Lire brièvement les données de votre historique actuel, sélectionnez les fichiers fastq.gz téléchargés dans le menu déroulant.
- Sélectionnez Exécuter pour exécuter le programme.
- Affichez les résultats dans le panneau Historique (Tableau 2).
REMARQUE : Pour des instructions plus détaillées sur l’utilisation de Galaxy, visitez la page d’assistance à l’adresse https://galaxyproject.org/support/.
6. Cartographie des lectures de séquençage
REMARQUE : Il s’agit d’un pipeline de base pour le découpage de l’adaptateur et le mappage de lecture des données de séquençage de l’ARN. Les séquences d’adaptateur sont coupées à partir des lectures à l’aide de Trimmomatic28. Les lectures tronquées sont cartographiées sur le génome de référence PAO1 de P. aeruginosa (NC_002516.2), obtenu du NCBI (https://www.ncbi.nlm.nih.gov/)29en utilisant BWA30 et Samtools31. Pour simplifier, une paire de lectures s’appelle PA_1.fq et PA_2.fq ; Le fichier de lecture de l’adaptateur à découper s’appelle adapter.fa ; et la séquence de référence PAO1 s’appelle PAO1.fasta. Tous les outils sont open source et fonctionnent dans un environnement UNIX/LINUX. Il est fortement conseillé de vous familiariser avec les fondamentaux d’UNIX/LINUX afin d’exécuter ces commandes.
- Ouvrez une fenêtre sous UNIX/LINUX.
- Installez Java, Trimmomatic, BWA et Samtools.
- Accédez au dossier dans lequel se trouve le fichier trimmomatic-0.39.jar.
- Coupez toutes les séquences d’adaptateur des lectures en tapant la commande :
Java -jar PA_1.fq PA_2.fq PA_1_paired.fq PA_1_unpaired.fq PA_2_paired.fq PA_2_unpaired.fq ILLUMINACLIP : adapters.fa
REMARQUE : Seules les séquences d’adaptateur sont supprimées. Les lectures n’ont pas été coupées pour la qualité32. - Déplacez le fichier de référence PAO1.fasta dans le même dossier.
- Indexez la référence à l’aide de BWA avec la commande :
Indice Bwa PA01.fasta - Mappez les lectures appariées au génome de référence en tapant les 4 commandes suivantes. Tapez chaque commande une fois la précédente terminée.
Bwa -mem PA01.fasta PA_1_paired.fq PA2_2_paired.fq > PA_R1R2_map.mem.sam
Samtools view -S -b PA_R1R2_map.mem.sam > PA_R1R2.bam
Samtools sort PA_R1R2.bam -o PA_R1R2_sorted.bam
Indice Samtools PA288_Rep1_R1R2_sorted.bam - Affichez les statistiques de mappage en tapant la commande :
Samtools flagstat PA_R1R2_sorted.bam
REMARQUE : La 3e ligne des statistiques indique la proportion des lectures qui correspondent au génome de référence. - Calculez la profondeur de lecture moyenne et l’étendue de la couverture à l’aide des 2 commandes suivantes33, respectivement :
Samtools Profondeur -A PA_R1R2_sorted.bam | awk '{c++ ; s+=$3}FIN{imprimer s/c}'
samtools profondeur -a PA288_Rep1_R1R2_sorted.bam | awk '{c++ ; if($3>0) total+=1}END{print (total/c)*100}'
Résultats
La figure 1 donne un aperçu général de la méthode. Nous avons précédemment utilisé des lames de chambre à 8 puits pour cultiver des biofilms de P. aeruginosa et les exposer à des antibiotiques avant de les examiner par microscopie confocale à différents moments12,13. Cette méthode peut être utilisée pour extraire l’ARN total directement des biofilms cultivés dans ce système afin d’étudier les changements d’expression génique après le traitement. Ce protocole a été optimisé pour P. aeruginosa mais peut facilement être adapté à d’autres espèces bactériennes.
Il peut être difficile d’extraire une quantité suffisante d’ARN de bonne qualité à partir de petites quantités de biofilm pour la déplétion de l’ARNr et le séquençage à haut débit. À l’aide de ce protocole, l’ARN total est extrait avec succès de 17 isolats différents du biofilm de P. aeruginosa en trois exemplaires, soit un total de 51 échantillons distincts. Les quantités d’ARN extraits représentant des rendements élevés et faibles sont indiquées dans le tableau 1. La concentration des ARN varie de 3,4 ng/μL (la plus faible) à 49,6 ng/μL (la plus élevée), avec une concentration moyenne de 14 ng/μL et une médiane de 13,7 ng/μL. Les concentrations d’ARN intact inférieures à 10 ng/μL sont considérées comme des échantillons de faible abondance pour la déplétion de l’ARNr et le séquençage de nouvelle génération, mais les échantillons d’ARN de faible abondance provenant de biofilms peuvent entraîner des données de séquençage de qualité encore plus médiocres par rapport aux échantillons plus concentrés34, 35, 36, 37. Les qualités des ARN sont indiquées dans le tableau 1 par le RIN et dans la figure 2 par les électrophérogrammes d’ARN correspondants pour les échantillons concentrés faibles (PA565-3) et élevés (PA288-1) ; les deux autres échantillons (PA375-3 et PA921-1) sont représentatifs de la majorité des échantillons. Comme le montre la figure 2, l’ARN extrait de ces quantités de biofilm contient toujours de l’ARN dégradé, ce qui affecte leurs valeurs RIN. Par conséquent, la confirmation visuelle des pics d’ARNr procaryotes 16S et 23S est utilisée pour déterminer la qualité de l’ARN lorsqu’un RIN n’est pas signalé. À l’aide de ces critères, les 51 échantillons d’ARN sont choisis pour procéder à la déplétion et au séquençage de l’ARNr, avec la prédiction que l’échantillon de la moins bonne qualité, PA565-3, pourrait échouer. À partir des échantillons d’ARN soumis pour le séquençage, des banques réussies sont générées et séquencées pour 49 échantillons, le PA565-3 étant défaillant, comme prévu.
Le tableau 2 indique le nombre de lectures de séquençage générées pour l’échantillon à forte concentration avec un bon RIN (PA288-1) et l’échantillon à faible concentration sans RIN (PA375-3). Les statistiques de base comprennent le nombre total de lectures, la durée de lecture et le contenu GC. Les statistiques récapitulatives des deux exemples montrent un nombre élevé de lectures générées et indiquent qu’aucune n’est signalée comme étant de mauvaise qualité, ce qui suggère de bonnes données de séquençage. Le nombre moyen de lectures par échantillon est d’environ 48 millions, ce qui est considéré comme un bon rendement. Un outil standard utilisé pour évaluer la qualité des données de séquençage brutes est FastQC26. Ce programme est utilisé pour effectuer des contrôles de qualité sur les fichiers de séquençage bruts afin de déterminer si la qualité est suffisante pour des analyses ultérieures, ou s’il existe des problèmes ou des biais dans les données générées par le séquenceur lui-même ou à partir des banques d’ARN d’entrée. La figure 3 présente les paramètres de contrôle de la qualité pour les données de séquençage PA288-1 et PA375-3, représentant respectivement un échantillon d’ARN de haute qualité et un échantillon d’ARN typique de qualité inférieure. L’un des graphiques les plus informatifs de FastQC est le graphique de qualité de séquence par base. De bonnes données de séquençage montreront un score de qualité médian élevé (>30) pour chaque position dans toutes les lectures, avec une baisse du score de qualité moyen sur la durée de la lecture. Un score de qualité de 30 représente un taux d’erreur de 1 sur 1000, ce qui correspond à une précision d’appel de base de 99,9 %. La grande majorité des bases de la figure 3 ont des scores de qualité moyens ≥ 35 sur toute la durée de la lecture pour les deux échantillons, ce qui indique des données de séquençage de qualité exceptionnelle. Cela fournit des preuves solides que le protocole d’extraction d’ARN présenté ici est efficace.
Pour montrer que notre méthode peut récupérer les transcrits de P. aeruginosa, les lectures de séquençage de haute qualité pour PA288-1 et PA375-3 sont cartographiées sur le génome de référence PAO1 de P. aeruginosa (NCBI NC_002516.2). Les lectures ne sont coupées que pour l’adaptateur de séquençage et non pour la qualité, en conservant toutes les bases avant le mappage32. Les statistiques de cartographie sont présentées dans le tableau 3. Le pourcentage de lectures mappées est une mesure importante de la précision du séquençage. En termes simples, plus il y a de lectures alignées sur votre séquence de référence, mieux c’est. Pour PA288-1 et PA375-3, 84 % et 91 % des lectures, respectivement, correspondent au génome de référence. La plage attendue pour la cartographie des lectures standard de RNA-seq se situe entre 70 et 90 %, donc ces valeurs sont très bonnes, surtout lorsque les bases de mauvaise qualité ne sont pas éliminées38. La profondeur de lecture moyenne est un bon indicateur du nombre moyen de lectures qui s’alignent à chaque position de base dans la séquence de référence. Plus la profondeur est élevée à chaque position de base, plus l’appel de base est précis à chaque position. Il est calculé en divisant la somme des profondeurs de lecture cartographiées à chaque position du génome de référence par le nombre total de bases dans le génome de référence. Les modèles PA288-1 et PA375-3 ont des profondeurs de lecture moyennes de 400 ou plus, ce qui est bon pour l’analyse de l’expression génique en aval39,40. L’étendue de la couverture indique le pourcentage de la longueur du génome de référence qui est couvert par le séquençage41. Les lectures alignées de PA288-1 et PA375-3 couvrent 96 % du génome de référence PAO1 de P. aeruginosa. Cela suggère que la majeure partie du génome de P. aeruginosa est représentée dans les données de séquençage et pas seulement de courts tronçons. Les statistiques de cartographie de PA288-1 et PA375-3 montrent que cette méthode peut récupérer des transcrits qui s’alignent avec une bonne couverture et une bonne distribution dans le génome de P. aeruginosa, ce qui soutient davantage la réussite du protocole d’extraction.
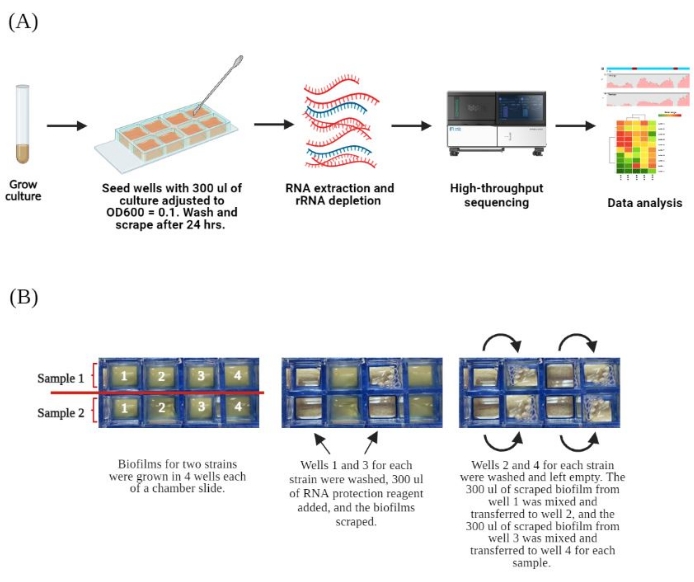
Graphique 1. Vue d’ensemble du protocole. Un. Schéma du flux de travail expérimental. Les cultures planctoniques ont été cultivées pendant la nuit à 37 °C, diluées à l’échelle 1:100 avec un milieu frais le lendemain et cultivées pendant 3 h supplémentaires. Les cultures ont été ajustées à une DOde 600 de 0,1 et 300 μL inoculées dans 4 puits d’une lame à 8 chambres pour générer des biofilms. Après 24 h, les biofilms ont été lavés pour éliminer les cellules planctoniques ; Un réactif de protection à ARN a été ajouté et les cellules ont gratté les puits. L’ARN total a été extrait, appauvri en ARN ribosomique et séquencé. B. Flux de travail détaillé de la croissance et du retrait du biofilm d’une lame à 8 chambres. Deux souches ont été cultivées par lame dans l’orientation indiquée à gauche. La disposition de chaque échantillon dans quatre puits de la lame est illustrée. Pour chaque échantillon indépendant, les puits 1 et 3 ont été traités en premier, ce qui a permis d’obtenir 300 μL de cellules grattées dans un réactif de protection contre l’ARN dans chaque puits. La diapositive du milieu en est un exemple. Ensuite, les puits 2 et 4 ont été lavés et le liquide a été retiré. Les cellules remises en suspension des puits 1 et 3 sont transférées aux puits 2 et 4, respectivement, comme le montre la diapositive de droite. Après le raclage, les cellules remises en suspension dans les puits 2 et 4 sont combinées en un seul tube de microcentrifugation. Cette figure a été créée avec BioRender.com. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Graphique 2. Exemples d’électrophérogrammes d’ARN à partir d’échantillons d’ARN extraits de haute à faible qualité. Les pics ribosomiques 16S et 23S sont marqués à la base de leurs pics. L’ARN dégradé est représenté par des pics de petite taille, indiqués par des flèches et une ligne de base bosselée au-dessus de zéro. Les échantillons d’ARN de qualité acceptable sont présentés dans les panneaux A, B et D. Le panneau C montre un échantillon d’ARN de mauvaise qualité où les pics ribosomiques sont manquants et la concentration était de très faible abondance, indiquée par l’échelle sur l’axe des Y. FU, unités de fluorescence ; nt, nucléotide. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Graphique 3. Graphique de qualité par séquence de base FastQC pour les données de séquence PA288-1 et PA375-3. Un. Le graphique de qualité pour l’échantillon de haute qualité PA288-1. B. Le graphique de qualité pour l’échantillon typique PA375-3. Les graphiques affichent le score de qualité agrégé pour chaque position de base pour toutes les lectures du fichier. La ligne bleue représente le score de qualité moyen à chaque position de base. La ligne rouge à l’intérieur de la boîte jaune représente la note de qualité médiane à chaque position, et la boîte jaune montre la plage quartile pour les 25e à 75e centiles. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Isoler | Qubit | RIN | Séquencé |
| PA288-1 | 26 ng/μl | 7.5 | Oui |
| PA375-3 | 4,07 ng/μl | NA | Oui |
| Réf. PA565-3 | 3,4 ng/μl | NA | Non |
| PA921-1 | 9,11 ng/μl | 6.6 | Oui |
Tableau 1. Métriques de qualité des ARN extraits d’échantillons représentatifs.
| PA288-1 | PA375-3 | |
| Nombre total de lectures | 8,59,57,720 | 3,18,49,575 |
| Lectures signalées comme étant de mauvaise qualité | 0 | 0 |
| Durée de lecture | 100 | 100 |
| %GC | 61 | 60 |
Tableau 2. PA288-1 et PA375-3 FastQC Statistiques sommaires
| Isoler | % de lectures mappées à la référence | Profondeur de lecture moyenne | Étendue de la couverture |
| PA288-1 | 83.93% | 404 | 96.68% |
| PA375-3 | 91.2% | 578 | 96.97% |
Tableau 3. Statistiques de cartographie des PA288-1 et PA375-3
Discussion
L’ARN total est extrait avec succès de 17 échantillons différents de biofilm bactérien en trois exemplaires, ce qui donne un total de 51 échantillons. Les quarante-neuf banques d’ARN sont regroupées et séquencées avec succès. Dans l’ensemble, cela valide nos critères de qualité avec un taux de réussite de 96 % même si plus de la moitié des échantillons sont considérés comme étant de faible abondance et de qualité sous-optimale 34,35,36,37.
Importance
Ce protocole d’extraction d’ARN est unique dans son explication détaillée pour extraire l’ARN de quantités limitées de biofilm qui ont été cultivées dans des lames de chambre. La croissance de biofilms dans une lame de chambre à 8 puits est un système utile pour étudier les effets de facteurs exogènes ajoutés aux biofilms ou les effets des interactions microbiennes dans la formation du biofilm par microscopie confocale 12,13,42. Afin d’examiner l’expression des gènes impliqués dans la formation du biofilm dans ce système, nous présentons une méthode détaillée pour extraire de l’ARN intact de qualité et de quantité suffisantes pour le séquençage de l’ARN. D’autres études ont rapporté une extraction réussie de l’ARN à partir de quantités limitées de biofilm, mais la majorité d’entre elles cultivent des biofilms sur une plus grande surface et souvent pendant 48 h au lieu de 24 h. Ils manquent également de suffisamment de détails pour assurer le succès 4,15,16,43,44,46.
De plus, ce protocole évite l’utilisation de produits chimiques dangereux (tels que le phénol) ou d’équipements spécialisés (tels qu’un sonicateur). Le protocole classique d’extraction du thiocyanate de guanidinium phénol-chloroforme47 n’est pas utilisé pour ce système car, même s’il produit environ 2 fois plus d’ARN par rapport au kit commercial, entre nos mains, il aboutit systématiquement à un ARN entièrement dégradé, tel qu’évalué par un système d’électrophorèse automatisé. De plus, il a été démontré que l’utilisation d’un kit d’extraction commercial permet d’obtenir un protocole convivial qui donne des résultats cohérents 15,16,45.
Étapes critiques
Il y a un certain nombre d’étapes critiques dans ce protocole qui augmentent la probabilité d’extraire de l’ARN qui peut être séquencé avec succès. Tout d’abord, il est important d’inoculer au moins 4 puits de la lame de chambre avec la même souche afin d’obtenir des quantités suffisantes d’ARN à séquencer. Les biofilms des 4 puits sont regroupés pour une seule extraction, ce qui présente l’avantage supplémentaire de réduire la variabilité de l’analyse de l’expression génique en aval17. L’extraction de l’ARN de moins de 4 puits conduit souvent à des rendements trop faibles pour être détectés sur un système fluorométrique à haute sensibilité. L’extraction de l’ARN de 8 puits ou d’une lame entière produira des quantités plus élevées d’ARN de qualité similaire à celles obtenues lors de l’utilisation de 4 puits, mais il faut se demander si le temps, les efforts et les coûts supplémentaires pour l’extraction valent le rendement accru. Le pipetage de 300 μL deOD 600 = 0,1 culture diluée pour ensemencer chaque puits au lieu des 200 μL standard améliore la récupération du matériau de biofilm raclé de chaque puits. Il est essentiel de laver doucement le biofilm deux fois avec de l’eau sans nucléase avant d’appliquer le réactif de protection à ARN pour éliminer autant de cellules mortes et/ou planctoniques que possible. L’utilisation du réactif protecteur de l’ARN est importante pour prévenir la dégradation de l’ARN en raison du temps nécessaire au traitement de chaque puits. De plus, au lieu de gratter avec des pointes de pipette, nous préférons utiliser des spatules de pesée en métal avec une extrémité plate qui touche une plus grande surface qu’une pointe de pipette et qui sont suffisamment petites pour s’insérer dans un puits. Le grattage à l’aide d’une pointe de pipette de 1000 μL fonctionnera, mais il est moins efficace et prend plus de temps pour gratter l’ensemble du biofilm par rapport à la spatule métallique. D’après notre expérience, l’utilisation de spatules métalliques permet d’économiser du temps et des efforts. Lors du grattage, assurez-vous de placer la glissière de la chambre sur le dessus de la plaque de verre pour éviter de fissurer le fond des puits. La sonication n’est pas efficace car elle entraîne constamment une forte dégradation de l’ARN en raison d’un volume d’échantillon trop faible et d’une production de chaleur trop importante. Après avoir collecté le matériau du biofilm, l’échantillon est introduit dans un kit commercial pour des extractions d’ARN plus fiables et reproductibles. Enfin, en raison des sources potentielles de variation dans cette méthode, il est très important d’inclure la réplication expérimentale en plus de la mise en commun des échantillons17,48. Ce protocole décrit la réplication biologique en trois exemplaires de chaque échantillon.
Limitations
Il existe une variété de techniques utilisées pour cultiver des biofilms à des fins d’étude, dont la plus courante est la formation dans des plaques de microtitration4. Ces plaques sont disponibles avec différentes tailles et nombres de puits. Les toboggans de chambre entrent dans cette catégorie. La capacité d’extraire de l’ARN intact et pur est importante pour tous les systèmes de biofilm, mais ce système est limité par les faibles rendements d’ARN. Les concentrations sont suffisamment faibles pour que leur qualité et leur quantité soient évaluées par des instruments Qubit et Bioanalyzer à l’aide de kits d’ARN à haute sensibilité. Une nanogoutte peut également être utilisée, mais elle a une sensibilité plus limitée que le Qubit et ne peut pas distinguer l’ADN contaminant de l’ARN49. Si possible, une nanogoutte doit être utilisée pour obtenir des rapports A260/A280 et A260/A230 pour la pureté. Ces ratios sont utiles, d’autant plus que la qualité de l’ARN de ce système de biofilm n’est pas aussi bonne qu’à partir de plus grandes quantités de matériel de départ. Cela est probablement dû au faible rendement en ARN et à la présence de cellules mortes dans les biofilms et/ou les RNases qui peuvent dégrader l’ARN. Une autre limitation de cette méthode est qu’elle ne peut pas séparer différents types de cellules au sein d’un biofilm hétérogène, mais ne peut extraire les cellules que dans une population entière, ce qui peut masquer l’expression des gènes dans les sous-populations sous-représentées.
Applications potentielles
Le protocole présenté ici a été optimisé pour les biofilms de P. aeruginosa à partir d’isolats cliniques cultivés dans des lames de chambre. Cette méthode peut être utilisée pour étudier les changements dans l’expression des gènes lors de la formation du biofilm dans différentes conditions de croissance observées par microscopie confocale. Le protocole peut également être optimisé pour d’autres espèces bactériennes génératrices de biofilm. Avec cette méthode, il est également possible d’extraire de l’ADN génomique puisqu’un kit d’extraction double peut être utilisé. De cette façon, l’expression des gènes du biofilm bactérien dans les isolats cliniques causant des infections chez les patients peut être étudiée avec plus de précision, ce qui conduit à des résultats qui pourraient guider les futures stratégies de traitement.
Déclarations de divulgation
Les auteurs n’ont aucune divulgation à déclarer.
Remerciements
Contributions des auteurs : P.W., Y.Y. et V.W ont participé à la conceptualisation de l’étude. K.G., L.J., A.M. et P.W. ont optimisé les protocoles de laboratoire. Le financement de K.G. a été financé par la subvention du Programme de stages pratiques pour étudiants par l’intermédiaire de BioTalent Canada.
matériels
| Name | Company | Catalog Number | Comments |
| Agilent 2100 Bioanalyzer | Agilent | G2939BA | Automated electrophoresis of biomolecules |
| Agilent RNA 6000 pico kit | Agilent | 5067-1513 | High sensitivity RNA electrophoresis chip to generate a RIN |
| DNA/RNA Lysis Buffer | Zymo Research | D7001-1-50 | A guanidinium thiocyanate and N-Lauroylsarcosine-based lysis buffer sold as part of a nucleic acid purification kit |
| DNA/RNA Prep Buffer | Zymo Research | D7010-2-10 | A guanidine HCl and ethanol buffer used for purification of DNA and RNA |
| DNA/RNA Shield | Zymo Research | R1100-50 | DNA and RNA preservation/protection reagent |
| DNA/RNA Wash Buffer | Zymo Research | D7010-3-6 | A salt and ethanol buffer used for purification of DNA and RNA |
| DNBSEQ G-400RS | MGI | G-400RS | High throughput sequencer |
| MGIEasy RNA Directional Library Prep Set | MGI | 1000006386 | Generate libraries for MGI high-throughput sequencing platforms from total RNA. |
| Mini-Beadbeater-96 | BioSpec | 1001 | A high energy, high throughput cell disrupter |
| NEBNext rRNA Depletion Kit (bacteria) | New England Biolabs | E7850X | Efficient and specific depletion of bacterial rRNA (5S, 16S, 23S) |
| Nunc Lab-Tek II chamber slide system | Thermo Fisher Scientific | 154534 | 8-well chamber slide with removable wells |
| Qubit Fluorometer | Thermo Fisher Scientific | Q33238 | Fluorometer for DNA, RNA and proteins |
| Qubit RNA HS Assay Kit | Thermo Fisher Scientific | Q32852 | High sensitivity fluorometric assay to measure RNA concentration |
| Spin-Away Filters | Zymo Research | C1006-50-F | Silica-based spin column primarily used to bind or remove genomic DNA |
| Sterile inoculation loops, 1 uL | Sarstedt | 86.1567.050 | Sterile, disposable inoculation loops for manipulation of microorganisms |
| ZR BashingBead Lysis tubes | Zymo Research | S6003-50 | 2 mL tubes containing 0.1 and 0.5 mm bead lysis matrix for homogenizing biological samples |
| Zymo Spin IIICG Columns | Zymo Research | C1006-50-G | Silica-based spin column for purification of DNA and RNA |
| Zymo-Spin III-HRC Filters | Zymo Research | C1058-50 | Remove inhibitors such as polyphenolic compounds, humic/fulvic acids, tannins, melanin, etc. |
| Zymobiomics DNA/RNA Miniprep kit | Zymo Research | R2002 | DNA and RNA dual extraction kit |
| Zymobiomics HRC Prep solution | Zymo Research | D4300-7-30 | To be used with Zymo-Spin III-HRC Filters to remove PCR inhibitors |
Références
- Beaudoin, T., Waters, V. Infections With Biofilm Formation: Selection of Antimicrobials and Role of Prolonged Antibiotic Therapy. The Pediatric Infectious Disease Journal. 35 (6), 695-697 (2016).
- Bjarnsholt, T., et al. The in vivo biofilm. Trends in Microbiology. 21 (9), 466-474 (2013).
- Folsom, J. P., et al. Physiology of Pseudomonas aeruginosa in biofilms as revealed by transcriptome analysis. BMC Microbiology. 10, 294(2010).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43 (3), 313-351 (2017).
- Bjarnsholt, T., Ciofu, O., Molin, S., Givskov, M., Hoiby, N. Applying insights from biofilm biology to drug development - can a new approach be developed. Nature Reviews Drug Discovery. 12 (10), 791-808 (2013).
- Colvin, K. M., et al. The Pel and Psl polysaccharides provide Pseudomonas aeruginosa structural redundancy within the biofilm matrix. Environmental Microbiology. 14 (8), 1913-1928 (2012).
- Hentzer, M., et al. Alginate overproduction affects Pseudomonas aeruginosa biofilm structure and function. Journal of Bacteriology. 183 (18), 5395-5401 (2001).
- Cury, J. A., Koo, H. Extraction and purification of total RNA from Streptococcus mutans biofilms. Analytical Biochemistry. 365 (2), 208-214 (2007).
- Francavilla, M., et al. Extraction, characterization and in vivo neuromodulatory activity of phytosterols from microalga Dunaliella tertiolecta. Current Medicinal Chemistry. 19 (18), 3058-3067 (2012).
- Atshan, S. S., et al. Improved method for the isolation of RNA from bacteria refractory to disruption, including S. aureus producing biofilm. Gene. 494 (2), 219-224 (2012).
- Franca, A., Melo, L. D., Cerca, N. Comparison of RNA extraction methods from biofilm samples of Staphylococcus epidermidis. BMC Research Notes. 4, 572(2011).
- Jurcisek, J. A., Dickson, A. C., Bruggeman, M. E., Bakaletz, L. O. In vitro biofilm formation in an 8-well chamber slide. The Journal of Visusalized Experiments. (47), e2481(2011).
- Beaudoin, T., Kennedy, S., Yau, Y., Waters, V. Visualizing the effects of sputum on biofilm development using a chambered coverglass model. The Journal of Visusalized Experiments. (118), e54819(2016).
- Cockeran, R., et al. Biofilm formation and induction of stress response genes is a common response of several serotypes of the pneumococcus to cigarette smoke condensate. The Journal of Infection. 80 (2), 204-209 (2020).
- Bisht, K., Moore, J. L., Caprioli, R. M., Skaar, E. P., Wakeman, C. A. Impact of temperature-dependent phage expression on Pseudomonas aeruginosa biofilm formation. npj Biofilmsand Microbiomes. 7 (22), (2021).
- Harrison, A., et al. Reprioritization of biofilm metabolism is associated with nutrient adaptation and long-term survival of Haemophilus influenzae. NPJ Biofilms and Microbiomes. 5 (1), 33(2019).
- Sousa, C., Franca, A., Cerca, N. Assessing and reducing sources of gene expression variability in Staphylococcus epidermidis biofilms. BioTechniques. 57, 295-301 (2014).
- Boom, R. Rapid and simple method for purification of nucleic acids. Journal of Clinical Microbiology. 28 (3), 495-503 (1990).
- Coppin, C. Re: How do silica based RNA spin columns only bind RNA and not DNA. , Available from: https://www.researchgate.net/post/How_do_silica_based_RNA_spin_columns_only_bind_RNA_and_not_DNA/60b017bffa5c4151cac1c/citation/download (2021).
- Kennedy, S. A complete guide to how nucleic extraction kits work. , Available from: https://bitesizebio.com/13516/how-dna-extraction-rna-miniprep-kits-work/ (2021).
- Qubit RNA HS Assay Kit User Guide. Thermo Fisher Scientific. , Available from: https://www.thermofisher.com/document-connect/document-connect.html?url=https%3A%2F%2Fassets.thermofisher.com%2FTFS-Assets%2FLSG%2Fmanuals%2FQubit_RNA_HS_Assay_UG.pdf&title=VXNlciBHdWlkZTogUXViaXQgUk5BIEhTIEFzc2F5IEtpdHM (2015).
- Mueller, O., Lightfoot, S., Schroeder, A. RNA Integrity Number (RIN) - Standardization of RNA Quality Control (Application report # 5989-1165EN). , Available from: https://www.agilent.com/cd/library/applications/5989-1165EN.pdf (2016).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 3(2006).
- Culviner, P. H., Guegler, C. K., Laub, M. T. A Simple, Cost-Effective, and Robust Method for rRNA Depletion in RNA-Sequencing Studies. mBio. 11 (2), (2020).
- MGIEasy RNA Directional Library Prep Set User Manual verA2. MGI Tech Co. , Available from: https://en.mgi-tech.com/products/reagents_info/14/ (2020).
- Andrews, S. FastQC: A Quality Control Tool for High Throughput Sequence Data. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina Sequence Data. Bioinformatics. , (2014).
- NCBI Resource Coordinators. Database resources of the National Center for Biotechnology Information. Nucleic Acids Research. 44 (1), (2016).
- Li, H. Aligning sequence reads, clone sequences and assembly contigs with BWA-MEM. arXiv. , (2013).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), (2009).
- Liao, Y., Shi, W. Read trimming is not required for mapping and quantification of RNA-seq reads at the gene level. NAR Genomics and Bioinformatics. 2 (3), (2020).
- Penir, S. Calculating Mapping Statistics from a SAM/BAM file using SAMtools and awk. , Available from: https://sarahpenir.github.io/bioinformatics/awk/calculating-mapping-stats-from-a-bam-file-using-samtools-and-awk/ (2019).
- Haile, S., et al. Evaluation of protocols for rRNA depletion based RNA sequencing of nanogram inputs of mammalian total RNA. PLoS ONE. 14 (10), 0224578(2019).
- Schuierer, S., et al. A comprehensive assessment of RNA-seq protocols for degraded and low-quantity samples. BMC Genomics. 18 (442), (2017).
- Shanker, S., et al. Evaluation of Commercially Available RNA Amplification Kits for RNA Sequencing Using Very Low Input Amounts of Total RNA. Journal of Biomolecular Techniques. 26 (1), (2015).
- Adiconis, X., et al. Comparative analysis of RNA sequencing methods for degraded or low-input samples. Nature Methods. 10, 623-629 (2013).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (13), (2016).
- Illumina, Inc. Coverage depth recommendations. , Available from: https://www.illumina.com/science/technology/next-generation-sequencing/plan-experiments/coverage.html (2021).
- What is a good sequencing depth for bulk RNA-Seq. ECSEQ Bioinformatics. , Available from: https://www.ecseq.com/support/ngs/what-is-a-good-sequencing-death-for-bulk-rna-seq (2019).
- Bedre, R. Sequencing coverage and breadth of coverage. , Available from: https://www.reneshbedre.com/blog/sequencing-coverage.html (2021).
- Dotsch, A., et al. The Pseudomonas aeruginosa transcriptome in planktonic cultures and static biofilms using RNA sequencing. PLoS One. 7 (2), 31092(2012).
- Chen, Y., et al. Population dynamics and transcriptomic responses of Pseudomonas aeruginosa in a complex laboratory microbial community. npj Biofilms and Microbiomes. 5 (1), (2019).
- Thoming, J. G., et al. Parallel evolutionary paths to produce more than one Pseudomonas aeruginosa biofilm phenotype. NPJ Biofilms and Microbiomes. 6, 2(2020).
- Soares, A., et al. Understanding ciprofloxacin failure in Pseudomonas aeruginosa biofilm: persister cells survive matrix disruption. Frontiers in Microbiology. 10, 2603(2019).
- Whiteley, M., et al. Gene expression in Pseudomonas aeruginosa biofilms. Nature. 413, (2001).
- Chomczynski, P., Sacchi, N. The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: twenty-something years on. Nature Protocols. 1, (2006).
- Liu, Y., Zhou, J., White, K. P. RNA-seq differential expression studies: more sequence or more replication. Bioinformatics. 30 (3), (2014).
- Wieczorek, D., Delauriere, L., Schagat, T. Methods of RNA Quality Assessment. , Available from: https://www.promega.ca/resources/pubhub/methods-of-rna-quality-assessment/ (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon