Method Article
从生物膜内的 铜绿假单胞 菌中分离总 RNA 以测量基因表达
摘要
该方案提出了一种从腔室载玻片中生长的 铜绿假单胞 菌生物膜中分离 RNA 以进行高通量测序的方法。
摘要
铜绿假单胞 菌是一种机会性细菌病原体,可引起囊性纤维化 (CF) 患者的气道感染。 铜绿假单 胞菌以其形成受胞外多糖基质保护的生物膜的能力而闻名。这种基质使微生物对外部因素(包括抗生素治疗)更具弹性。用于研究的生物膜生长最常见的方法之一是在微量滴定板或腔室载玻片中。这些系统的优点是它们允许测试多种生长条件,但它们的缺点是它们产生有限量的生物膜用于 RNA 提取。本文的目的是提供详细的分步方案,说明如何从少量质量和数量足够高的生物膜中提取总 RNA,以进行高通量测序。该协议允许研究这些生物膜系统内的基因表达。
引言
大多数慢性细菌感染,例如囊性纤维化 (CF) 患者的肺部感染和假体相关感染,其特征是生物膜内生物体的生长。生物膜1 是包裹在主要由多糖2 组成的基质中的细菌群落。生物膜内的细菌可以缓慢生长、代谢休眠,并且在厌氧、缺氧条件下。由于抗生素渗透性降低、药物外排泵表达增加和细胞分裂减少等因素,生物膜对抗生素的耐药性更强3。由于这些和其他原因,它们具有极大的研究兴趣。
为了准确研究 CF 患者的慢性铜绿假单胞菌感染等持续感染,需要在体外准确反映生物膜形成的生长条件。一种常见的高通量方法是在腔室载玻片或微量滴定板中培养它们,并通过共聚焦显微镜监测生物膜的形成4。众所周知,从浮游或自由漂浮到生物膜细菌生活方式转变的关键调节因子是第二信使 cyclic-di-GMP5。cyclic-di-GMP 水平升高会增加促进生物膜生长的特定基因的表达。小的非编码调节 RNA 和群体感应在生物膜形成的调节中也起着重要作用5。通过对提取的细菌 RNA 进行测序来测量生物膜基因表达可能具有挑战性。例如,铜绿假单胞菌产生三种胞外多糖(Psl、Pel 和藻酸盐),它们在生物膜中大量产生 6,7。这些多糖会干扰 RNA 提取和纯化,导致制备物含低水平细菌 mRNA8。市售的 RNA 提取试剂盒能够从浮游细菌培养物中产生高质量的 RNA,但可能无法很好地与生物膜培养物配合使用 9,10,11。有一些商业 RNA 提取试剂盒声称适用于生物膜,其中一种我们与这种方法一起使用。
在本手稿中,我们描述了在腔室载玻片中培养铜绿假单胞菌生物膜和提取细菌 mRNA 以进行高通量测序的程序12,13。利用从 CF 患者痰液样本中收集的临床分离株,我们证明这些方法可用于具有不同生长特征的分离株。与以前的出版物相比,详细描述了该协议,以便在研究细菌生物膜基因表达方面取得更大的成功 11,14,15,16。
研究方案
研究伦理委员会 (REB) 是收集和处理人类受试者痰液样本所必需的。这项研究得到了病童医院 (REB#1000019444) 的批准。研究伦理委员会 (REB) 需要收集和储存人类受试者的痰液样本。这些研究得到了病童医院 REB#1000058579 的批准。
1. 生物膜形成
- 在 37 °C 培养箱中,在 Luria 肉汤 (LB) 琼脂平板上培养从本研究中使用的 CF 患者痰液样本中获得的铜 绿假单胞 菌分离株过夜。
注意:正确的划线技术对于获得单个细菌菌落很重要。旋转板时划线会充分稀释细菌细胞,以便单个菌落可以生长。- 在新鲜 LB 板的顶端使用锯齿形图案的接种环对细菌进行划线,直到覆盖板的约 1/4。
- 将板旋转约 60°。取一个新的接种环,将其穿过条纹区域一次,然后进入板的第二个干净区域,重复锯齿形图案。
- 重复步骤 1.1.2 将新循环放入板的第三个区域。放入培养箱时,请装回盖子并倒置板。
- 使用无菌接种环从琼脂平板(包含单个细菌菌株)中挑选单个细菌菌落,并接种装有 5 mL 无菌 LB 培养基的培养管。每次使用新定量环,接种两个额外的培养管,其中填充有来自同一板的 5 mL LB 培养基。
- 对不同的细菌菌株重复相同的接种程序。将培养物在 37 °C 下以 220 RPM 振荡过夜。
注意:每个含有单个细菌菌株的琼脂板用于接种 3 个独立的培养管。这三个试管代表一个菌株的生物学重复,一式三份,并作为单独的样品处理。这与技术重复不同,后者需要从单个培养管中提取 RNA 3 次。 - 通过将 50 μL 过夜培养物转移到含有 4.95 mL 新 LB 培养基的新培养管中,制备 1:100 稀释的过夜培养物。
- 将稀释的培养物在 37 °C 下再培养 3 小时,以 220 RPM 摇动或直到 OD600 为 0.1 或更高。在分光光度计上以 OD600 测量细胞密度。
- 在新的 1.7 mL 微量离心管中,用新鲜 LB 将 OD600 调整至 0.1(早期对数期),总体积为 1.5 mL。
- 倒置轻轻混合。将 300 μL 每种调整后的培养物转移到 8 孔室载玻片的 4 个孔中,最终每张载玻片有 2 个不同的样品(图 1)。
- 将载玻片置于 37 °C 培养箱中过夜,不受干扰 24 小时。为防止蒸发,请将玻片放在小塑料托盘中的湿纸巾顶部。
2. 生物膜回收
注:每个载玻片包含八个独立的孔。单个样品由四个带有生物膜的孔组成,这些孔将被合并17.该提取方案适用于 1 个样品(4 个孔),其中生物膜一次从 2 个孔中回收。使用市售 RNA 提取试剂盒进行 RNA 提取,该试剂盒包括磁珠打纬步骤和基于柱的纯化,并进行了修饰。按照制造商的说明进行试剂制备。
- 在层流罩中,使用移液器吸头从 4 个孔中的 2 个孔中缓慢去除培养基。将载玻片倾斜 45° 角,并从孔的底角吸出培养基,以防止生物膜脱落。
- 保持载玻片倾斜,将 300 μL 不含 RNase 的水轻轻移液到两个空井的底角,洗掉浮游细胞。如步骤 2.1 中所述,轻轻移出水,将其移出。重复洗涤步骤,尽可能多地去除水。
- 将 300 μL RNA 保护试剂(参见 材料表)添加到两个空孔中,其底部有生物膜。将腔室载玻片放在玻璃板上以防止孔破裂,然后用小的、无核酸酶的无菌金属刮刀刮擦 2 个孔中的生物膜,以重新悬浮生物膜细菌。静置,直到从同一样品中剩余的 2 个未触及的孔中回收生物膜。
注:添加 RNA 保护试剂可确保在处理同一样品的其余两个孔时,刮取孔中生物膜样品在环境温度下的稳定性。RNA 保护试剂裂解细胞并灭活核酸酶和感染因子,从而保存 RNA。 - 要从样品的剩余 2 个孔中回收生物膜(提醒:一个样品由 4 个孔组成),请按照步骤 2.1 中描述的相同方式从剩余的 2 个新孔中去除 LB 培养基。像以前一样,重复步骤 2.2 用不含 RNase 的水从两个新孔中洗掉浮游细胞。
- 回到前 2 个孔,在步骤 2.3 结束时生成的保护试剂中刮取生物膜,然后缓慢移液以混合来自 一个 刮取的井的 300 μL 重新悬浮的生物膜,尽量不要产生太多气泡。
- 将井中的所有内容物转移到新清洗、清空的井 中 。将第二个刮得好的井中的 300 μL 重新悬浮的生物膜混合并转移到剩余的、新洗涤、清空的井中。
注:不要向 2 个带有生物膜的新洗涤的孔中添加新的 RNA 保护试剂,而是将保护试剂中已有的先前刮下的生物膜转移到这些新洗涤的孔中。这将使组合样品体积保持足够低,以满足市售 RNA 提取试剂盒的起始量要求。原理图见 图 1 。
- 将井中的所有内容物转移到新清洗、清空的井 中 。将第二个刮得好的井中的 300 μL 重新悬浮的生物膜混合并转移到剩余的、新洗涤、清空的井中。
- 如步骤 2.3 所示,将腔室载玻片放在玻璃板上,并用小的、无核酸酶的无菌金属刮刀刮擦 2 个新孔中的生物膜,以重新悬浮生物膜细菌。
- 将 2 个新孔中 所有 重悬的生物膜合并到一个无 RNase、低结合力的 1.5 mL 微量离心管中。测量体积,总体积应为 ~500 - 600 μL。
注:来自第二对新孔的混合生物膜悬浮液将包含来自样品原始 4 个孔的所有生物膜材料。
3. 总 RNA 分离和质量评估
注意:RNA 提取是使用声称适用于生物膜的商业 RNA 提取试剂盒进行的。如果可能,各个组件都包含在 材料表中。如果可能,将解释每个纯化步骤背后的机制。
- 加入足够的 RNA 保护试剂,使试管中总共 750 μL。将整个体积转移到含有 0.1 和 0.5 mm 珠子的 2 mL 珠子打浆裂解管中(参见 材料表)。在打珠器中以最大速度搅拌 2 1/2 分钟。
注:0.1 mm 和 0.5 mm 高密度微珠的组合加上微珠打浆机上的高速混合,可确保微生物细胞壁的彻底均质化。 - 以 16,000 x g 离心 1 分钟以沉淀珠子。将上清液转移到新的微量离心管中,最大限度地减少任何珠子的转移,这将使步骤 3.3 更容易。测量音量。
- 加入等体积的 RNA 裂解缓冲液 (~450 μL) 并充分混合。将多达 800 μL 的样品转移至收集管中的硅胶柱中,避免转移任何珠子,并以 16,000 x g 离心 30 秒。保存流出液,因为它含有 RNA,而 DNA 与色谱柱结合。
注:RNA 裂解缓冲液含有硫氰酸胍和去污剂 N-月桂酰肌氨酸,用于裂解细胞。硫氰酸鸟苷是一种离液剂,也可使核酸酶失活,并且在离心柱中发现的二氧化硅存在下,可促进 DNA 与二氧化硅的结合18。由于不含乙醇,因此 DNA 优先结合 DNA,而不是 RNA 与硅胶离心柱19。步骤 3.3 的目的是结合和去除基因组 DNA。我们想保留包含在流通部分的 RNA。 - 如果剩余更多样品,请将色谱柱转移到新的收集管中,然后重新装入其余样品。以 16,000 x g 离心 30 秒。保持 RNA 的流出液,并与第一等分试样混合。
- 测量组合流通体积,加入等体积的 100% 乙醇并充分混合。将多达 800 μL 的溶液转移到收集管中新的第二个硅胶离心柱中,并以 16,000 x g 离心 30 秒。丢弃流出物。
注:向含有 RNA 的离液盐溶液中添加乙醇可增强 RNA 与硅胶离心柱19 的结合。 - 对于 > 800 μL 的溶液,重新加载离心柱并离心,直到整个溶液离心。每次离心后丢弃流通液。
- 向色谱柱中加入 400 μL 洗涤缓冲液,并以 16,000 x g 离心 30 秒以去除一些盐。丢弃流出物。
- 根据制造商的说明制备 DNase I 反应混合物,并进行柱内 DNase 处理以去除任何残留的 DNA。
- 将冻干的 DNase I 重悬于 275 μL 不含 RNase 的水中,制成 1 U/μL 溶液。轻轻倒置混合。
- 将 5 μL 稀释的 DNase I 与 75 μL 提供的 DNase 消化缓冲液混合。倒置轻轻混合。
- 将 80 μL 制备好的溶液直接添加到柱基质上。在室温下孵育 20 分钟。
- 向色谱柱中加入 400 μL RNA 制备缓冲液,并以 16,000 x g 离心 30 秒。丢弃流出物。
- 向色谱柱中加入 700 μL RNA 洗涤缓冲液并重复离心。丢弃流出物。
- 加入 400 μL RNA 洗涤缓冲液,将色谱柱离心 2 分钟,以完全去除任何残留缓冲液。
注:有 2 种不同的清洗步骤可去除色谱柱上的杂质。制备缓冲液含有与乙醇混合的弱离液盐,以去除残留蛋白质。接下来,使用洗涤缓冲液进行乙醇洗涤以去除盐分。必须去除任何残留的乙醇,以便高效洗脱 RNA19,20。 - 小心地将色谱柱转移到新的低结合微量离心管中。
- 将 50 μL 不含 RNase 的水直接加入柱基质中,并孵育 5 分钟。以 16,000 x g 离心 1 分钟以洗脱 RNA。
- 如需进一步纯化以去除 PCR 抑制剂,请将 PCR 抑制剂过滤器放入新的收集管中。加入 600 μL 提供的抑制剂制备溶液(参见 材料表)。
- 以 8,000 x g 离心 3 分钟以洗涤过滤器。将洗涤后的过滤器转移到新的低结合微量离心管中。
- 将步骤 3.13 中洗脱的 RNA 转移到洗涤的过滤器中,并以 16,000 x g 离心 3 分钟。
注:RNA 可立即使用或储存在 -80 °C 下。 - 使用高灵敏度荧光系统测定 RNA 的浓度21.
注:这些系统允许对目标靶标特异性溶液中的少量 RNA 进行灵敏定量。- 使用可提供 RIN(RNA 完整性值)的自动电泳系统评估 RNA 的质量,该系统是衡量 RNA 质量的指标22,23。
4. 核糖体 RNA 耗竭和高通量测序
- 将 RNA 提交给多伦多大学(加拿大多伦多)(https://www.cagef.utoronto.ca/)的基因组进化和功能分析中心 (CAGEF) 基因组中心,用于细菌 rRNA 耗竭和 RNA 定向文库制备(参见材料表)。
注:细菌 rRNA 耗竭靶向 5S、16S 和 23S rRNA 以去除24。 - 使用市售 rRNA 细菌去除试剂盒去除 rRNA。按照方案输入 10 ng - 1 μg 完整或部分降解的总 RNA。
- 使用 RNA 定向文库制备试剂盒构建 RNA 测序文库,每个文库连接不同的索引。
- 汇集等摩尔量的每个 RNA 文库,并使用 100 个碱基的双端读数25 进行高通量测序。
5. 测序读长的质量评估
注意:使用免费提供的程序 FastQC26 检查测序读数的质量,该程序可通过免费的开源平台 Galaxy27 获得。
- 转到 https://usegalaxy.org/。单击 Login 或 Register 菜单,然后使用凭据登录或创建帐户。
- 单击页面左上角的 Upload Data 链接,在 Tools 菜单下,然后上传fastq.gz排序文件。等待文件名出现在页面右侧的 History 面板下。
- 在"工具"菜单下选择"FASTQ 质量控制"以显示程序列表。选择 FastQC,这将填充屏幕的中间面板。
- 在 Short read data from your current history (从当前历史记录中短读数据) 下,从下拉菜单中选择上传的 fastq.gz 文件。
- 选择 Execute (执行 ) 以运行程序。
- 在 History 面板下查看结果(表 2)。
注意:有关如何使用 Galaxy 的更多详细说明,请访问 https://galaxyproject.org/support/ 的支持页面。
6. 测序读长的映射
注意:列出的是 RNA-seq 数据的接头修剪和读取映射的基本管道。使用 Trimmomatic28 从读数中修剪接头序列。修剪的读数映射到铜绿假单胞菌 PAO1 参考基因组 (NC_002516.2),使用 BWA30 和 Samtools31 从 NCBI (https://www.ncbi.nlm.nih.gov/)29获得。为简单起见,一对读取称为 PA_1.fq 和 PA_2.fq;要修剪的适配器读取文件称为 adapter.fa;PAO1 参考序列称为 PAO1.fasta。所有工具都是开源的,并在 UNIX/LINUX 环境中运行。强烈建议您熟悉 UNIX/LINUX 的基础知识,以便执行这些命令。
- 在 UNIX/LINUX 中打开一个窗口。
- 安装 Java、Trimmomatic、BWA 和 Samtools。
- 导航到文件trimmomatic-0.39.jar所在的文件夹。
- 通过键入命令从读取中修剪掉任何适配器序列:
Java -jar PA_1.fq PA_2.fq PA_1_paired.fq PA_1_unpaired.fq PA_2_paired.fq PA_2_unpaired.fq ILLUMINACLIP:adapters.fa
注意: 仅删除适配器序列。读取尚未针对质量32 进行修剪。 - 将 PAO1.fasta 参考文件移动到同一文件夹中。
- 使用 BWA 和命令为引用编制索引:
Bwa 指数 PA01.fasta - 通过键入以下 4 个命令,将配对的读数映射到参考基因组。在前一个命令完成后键入每个命令。
Bwa -mem PA01.fasta PA_1_paired.fq PA2_2_paired.fq > PA_R1R2_map.mem.sam
Samtools 视图 -S -b PA_R1R2_map.mem.sam > PA_R1R2.bam
Samtools 排序 PA_R1R2.bam -o PA_R1R2_sorted.bam
Samtools 索引 PA288_Rep1_R1R2_sorted.bam - 通过键入命令查看映射统计信息:
Samtools flagstat PA_R1R2_sorted.bam
注意:统计数据的第 3 行报告了映射到参考基因组的读数的比例。 - 分别使用以下 2 个命令33 计算平均读取深度和覆盖范围广度:
SAMToos深度 -A PA_R1R2_sorted.bam |awk '{c++;s+=$3}END{print s/c}'
SAMTories 深度 -A PA288_Rep1_R1R2_sorted.bam |awk '{c++; if($3>0) total+=1}END{print (total/c)*100}'
结果
该方法的一般概述如图 1 所示。我们之前使用 8 孔室载玻片来生长铜绿假单胞菌生物膜并将它们暴露于抗生素中,然后在不同时间点通过共聚焦显微镜检查它们12,13。该方法可用于直接从该系统中生长的生物膜中提取总 RNA,以研究处理后的基因表达变化。该方案已针对铜绿假单胞菌进行了优化,但可以很容易地适应其他细菌种类。
从少量生物膜中提取足够数量的优质 RNA 用于 rRNA 去除和高通量测序可能具有挑战性。使用该方案,从 17 种不同的铜绿假单胞菌生物膜分离物中成功提取总 RNA,一式三份,总共 51 个单独的样品。代表高产量和低产量的提取 RNA 的数量如表 1 所示。RNA 的浓度范围为 3.4 ng/μL(最低)至 49.6 ng/μL(最高),平均浓度为 14 ng/μL,中位数为 13.7 ng/μL。低于 10 ng/μL 的完整 RNA 浓度被认为是 rRNA 耗竭和下一代测序的低丰度样品,但与浓度更高的样品相比,来自生物膜的低丰度 RNA 样品会导致测序数据质量更差34, 35,36,37。RNA 的质量在表 1 中由 RIN 表示,在图 2 中由低浓度 (PA565-3) 和高浓度 (PA288-1) 样品的相应 RNA 电泳图表示;其余两个样品 (PA375-3 和 PA921-1) 代表了大多数样品。如图 2 所示,从这些数量的生物膜中提取的 RNA 总是包含一些降解的 RNA,这会影响它们的 RIN 值。因此,当未报告 RIN 时,16S 和 23S 原核 rRNA 峰的目视确认用于确定 RNA 质量。使用这些标准,选择所有 51 个 RNA 样品进行 rRNA 去除和测序,并预测质量最差的样品 PA565-3 可能会失败。在提交测序的 RNA 样本中,生成了 49 个样本的成功文库并进行了测序,正如预测的那样,PA565-3 失败。
表 2 列出了为具有良好 RIN 的高浓度样品 (PA288-1) 和没有 RIN 的低浓度样品 (PA375-3) 生成的测序读数数量。基本统计信息包括读取总数、读取长度和 GC 内容。两个样品的汇总统计数据显示大量生成的读数,并表明没有读数被标记为质量差,表明测序数据良好。每个样本的平均读取数约为 4800 万次,这被认为是一个不错的产量。用于评估原始测序数据质量的标准工具是 FastQC26。该程序用于对原始测序文件进行质量控制检查,以确定质量是否足以进行进一步分析,或者测序仪本身或输入 RNA 文库生成的数据是否存在问题或偏差。图 3 显示了 PA288-1 和 PA375-3 测序数据的质量控制指标,分别代表高质量 RNA 样品和典型的低质量 RNA 样品。FastQC 中信息量更大的图之一是每个碱基序列质量图。良好的测序数据将显示所有 reads 中每个位置的中位质量评分 (>30),并且平均质量评分在 reads 的长度内下降。质量分数 30 表示错误率为 1/1000,对应于 99.9% 的基本调用准确率。图 3 中的绝大多数碱基在两个样品的整个读长长度上的平均质量评分≥ 35,这表明测序数据的质量非常好。这提供了强有力的证据,证明此处介绍的 RNA 提取方案是成功的。
为了证明我们的方法可以恢复铜绿假单胞菌转录本,将 PA288-1 和 PA375-3 的高质量测序读数映射到铜绿假单胞菌 PAO1 参考基因组 (NCBI NC_002516.2)。仅针对测序接头修剪读数,而不针对质量进行修剪,在定位32 之前保留所有碱基。映射统计数据如表 3 所示。映射读数的百分比是测序准确性的重要指标。简而言之,与参考序列对齐的 reads 越多越好。对于 PA288-1 和 PA375-3,分别 84% 和 91% 的读数映射到参考基因组。标测标准 RNA-seq 读数的预期范围在 70 - 90% 之间,因此这些值非常好,尤其是在未去除质量差的碱基时38。平均读深是参考序列中每个碱基位置对齐的平均读长数的良好指标。每个 base 位置的深度越高,每个位置的 base call 就越准确。计算方法是将参考基因组中每个位置的映射读取深度之和除以参考基因组中的碱基总数。PA288-1 和 PA375-3 的平均读取深度为 400 或更高,这有利于下游基因表达分析39,40。覆盖面的广度表示测序41 覆盖的参考基因组长度的百分比。来自 PA288-1 和 PA375-3 的比对读数覆盖了铜绿假单胞菌 PAO1 参考基因组的 96%。这表明大部分铜绿假单胞菌基因组都在测序数据中表示,而不仅仅是短片段。PA288-1 和 PA375-3 的定位统计表明,这种方法可以恢复与铜绿假单胞菌基因组的良好覆盖和分布一致的转录本,进一步支持成功的提取方案。
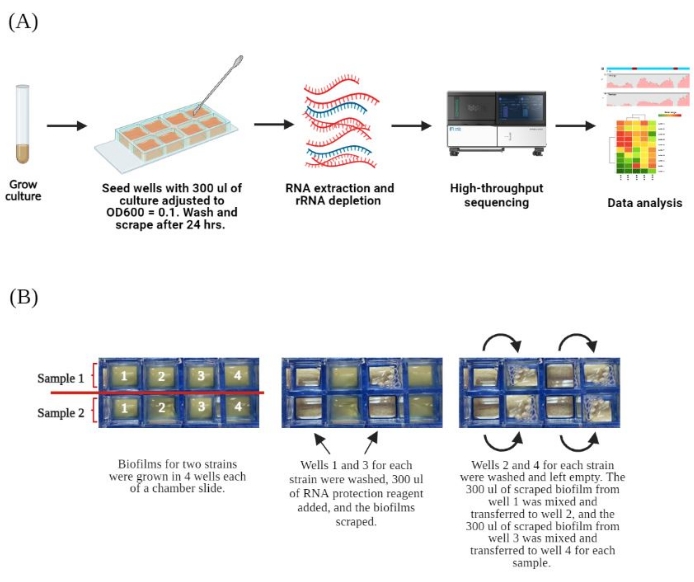
图 1.协议概述。一个。 实验工作流程示意图。浮游生物培养物在37°C下生长过夜,第二天用新鲜培养基1:100稀释,再生长3小时。将培养物调整至 OD600 为 0.1,并将 300 μL 接种到 8 室载玻片的 4 个孔中以产生生物膜。24 小时后,洗涤生物膜以去除浮游细胞;加入 RNA 保护试剂,从孔中刮下细胞。提取总 RNA,去除核糖体 RNA 并测序。 湾。 生物膜在 8 腔载玻片中生长和去除的详细工作流程。每张玻片沿左图所示的方向生长两个菌株。显示了载玻片四个孔中每个样品的排列。对于每个独立样品,首先处理孔 1 和 3,最后在每个孔中用 RNA 保护试剂刮取 300 μL 细胞。中间的幻灯片描绘了一个示例。接下来,洗涤 2 和 4 孔并去除液体。将来自孔 1 和 3 的重悬细胞分别转移到孔 2 和 4 中,如右幻灯片所示。刮擦后,将孔 2 和 4 中重悬的细胞合并成单个微量离心管。这个数字是用 BioRender.com 创建的。 请单击此处查看此图的较大版本。

图 2.从高质量到低质量提取的 RNA 样品的 RNA 电泳图示例。 16S 和 23S 核糖体峰标记在其峰的底部。降解的 RNA 由小峰表示,由箭头和高于零的凹凸基线表示。可接受质量的 RNA 样品如图 A、B 和 D 所示。图 C 显示质量较差的 RNA 样品,其中核糖体峰缺失,浓度丰度非常低,由 Y 轴上的刻度表示。FU,荧光单位;nt,核苷酸。 请单击此处查看此图的较大版本。

图 3.PA288-1 和 PA375-3 序列数据的 FastQC 每个碱基序列质量图。一个。 高质量样品 PA288-1 的质量图。 湾。 典型样品 PA375-3 的质量图。这些图显示了文件中所有 reads 的每个碱基位置的聚合质量分数。蓝线表示每个基本位置的平均质量得分。黄色框内的红线表示每个位置的质量得分中位数,黄色框显示第 25个 到第 75个 百分位数的四分位数范围。 请单击此处查看此图的较大版本。
| 隔离 | 量子比特 | 凛 | 测 序 |
| PA288-1 | 26 纳克/微升 | 7.5 | 是的 |
| PA375-3 | 4.07 纳克/微升 | 那 | 是的 |
| PA565-3 | 3.4 纳克/微升 | 那 | 不 |
| PA921-1 | 9.11 纳克/微升 | 6.6 | 是的 |
表 1. 从代表性样品中提取的 RNA 的质量指标。
| PA288-1 | PA375-3 | |
| 总读取数 | 8,59,57,720 | 3,18,49,575 |
| 读取标记为质量差 | 0 | 0 |
| 读取长度 | 100 | 100 |
| %气相色谱 | 61 | 60 |
表 2. PA288-1 和 PA375-3 FastQC 汇总统计
| 隔离 | 映射到参考的 % reads | 平均读取深度 | 覆盖范围广 |
| PA288-1 | 83.93% | 404 | 96.68% |
| PA375-3 | 91.2% | 578 | 96.97% |
表 3. PA288-1 和 PA375-3 映射统计
讨论
从 17 种不同的细菌生物膜样品中成功提取总 RNA,一式三份,总共得到 51 个样品。将 49 个 RNA 文库合并并成功测序。总体而言,这验证了我们的质量标准,成功率为 96%,尽管超过一半的样品被认为丰度较低且质量欠佳 34,35,36,37。
意义
该 RNA 提取方案的独特之处在于其详细解释从腔室载玻片中生长的有限量生物膜中提取 RNA。生物膜在 8 孔室载玻片中的生长是一种有用的系统,可通过共聚焦显微镜研究外源性添加因子对生物膜的影响或微生物相互作用对生物膜形成的影响 12,13,42。为了检查该系统中参与生物膜形成的基因的表达,我们提出了一种详细的方法来提取足够质量和数量的完整 RNA 用于 RNA 测序。其他研究报道了从有限量的生物膜中成功提取 RNA,但大多数生物膜在更大的表面积上生长,并且通常持续 48 小时而不是 24 小时。它们也缺乏足够的细节来确保成功 4,15,16,43,44,46。
此外,该协议避免使用危险化学品(如苯酚)或专用设备(如超声仪)。经典的硫氰酸胍苯酚-氯仿提取方案47 不用于该系统,因为尽管与商业试剂盒相比,它产生的 RNA 大约多 2 倍,但在我们手中,它始终会导致 RNA 完全降解,如在自动电泳系统上评估的那样。此外,使用市售提取试剂盒可产生用户友好的方案,可产生一致的结果15,16,45。
关键步骤
该方案中有许多关键步骤可以增加提取可成功测序的 RNA 的可能性。首先,重要的是用相同的菌株接种腔室载玻片的至少 4 个孔,以获得足够数量的 RNA 进行测序。将 4 个孔中的生物膜合并进行单次提取,这具有减少下游基因表达分析变异性的额外优势17。从少于 4 个孔中提取 RNA 通常会导致产量太低,无法在高灵敏度荧光系统上检测。从8个孔或整个玻片中提取RNA将产生比使用4个孔时获得的更大量质量相似的RNA,但必须考虑提取的额外时间、精力和成本是否值得增加的产量。将 300 μL OD600 = 0.1 稀释的培养物移液到每个孔中接种,而不是标准的 200 μL,可以提高从每个孔中刮下的生物膜材料的回收率。在使用 RNA 保护试剂之前,用不含核酸酶的水轻轻洗涤生物膜两次,这对于去除尽可能多的死亡和/或浮游细胞至关重要。由于 RNA 处理每个孔需要时间,因此使用 RNA 保护试剂对于防止 RNA 降解非常重要。此外,与其使用移液器吸头刮擦,我们更喜欢使用金属称量刮刀,其扁平端比移液器吸头接触的表面积更大,并且足够小,可以放入孔中。使用 1000 μL 移液器吸头进行刮擦可以,但与金属刮刀相比,刮擦整个生物膜的效率较低且需要更长的时间。根据我们的经验,使用金属刮刀可以节省时间和精力。刮擦时,确保将腔室载玻片放在玻璃板顶部,以防止孔底部开裂。超声处理效果不佳,因为样品量太少和产生的热量过多,它始终会导致 RNA 高度降解。收集生物膜材料后,将样品输入到商业试剂盒中,以实现更可靠和可重现的 RNA 提取。最后,由于这种方法存在潜在的变异来源,除了样本混合之外,还包括实验复制非常重要17,48。该协议描述了每个样品的一式三份生物复制。
局限性
有多种技术可用于生长用于研究的生物膜,其中最常见的是在微量滴定板中形成4。这些板具有不同的尺寸和孔数。腔室滑轨属于这一类。提取完整、纯 RNA 的能力对所有生物膜系统都很重要,但该系统受到 RNA 产量低的限制。浓度足够低,因此应使用高灵敏度 RNA 试剂盒通过 Qubit 和生物分析仪仪器评估其质量和数量。也可以使用 nanodrop,但与 Qubit 相比,它的灵敏度更有限,并且无法区分污染的 DNA和 RNA 49。如果可能,应使用 nanodrop 来获得 A260/A280 和 A260/A230 的纯度比例。这些比率很有用,特别是因为该生物膜系统的 RNA 质量不如大量起始材料的 RNA 质量。这很可能是由于 RNA 产量低以及生物膜和/或 RNase 中存在可能降解 RNA 的死细胞。这种方法的另一个局限性是它不能分离异质生物膜内的不同细胞类型,而只能提取整个细胞群中的细胞,这可能会掩盖任何代表性不足的亚群中的基因表达。
潜在应用
此处介绍的方案已针对来自腔室载玻片中生长的临床分离株的 铜绿假单 胞菌生物膜进行了优化。该方法可用于研究在共聚焦显微镜观察到的不同生长条件下生物膜形成过程中基因表达的变化。该方案还可以针对其他产生生物膜的细菌物种进行优化。使用这种方法,由于可以使用双重提取试剂盒,因此也可以提取基因组 DNA。通过这种方式,可以更准确地研究导致患者感染的临床分离株中的细菌生物膜基因表达,从而得出可能指导未来治疗策略的结果。
披露声明
作者没有需要声明的披露。
致谢
作者贡献:P.W.、Y.Y. 和 V.W 参与了这项研究的概念化。K.G.、L.J.、A.M. 和 P.W. 优化了实验室方案。K.G. 的资金得到了 BioTalent Canada 的学生工作安置计划补贴的支持。
材料
| Name | Company | Catalog Number | Comments |
| Agilent 2100 Bioanalyzer | Agilent | G2939BA | Automated electrophoresis of biomolecules |
| Agilent RNA 6000 pico kit | Agilent | 5067-1513 | High sensitivity RNA electrophoresis chip to generate a RIN |
| DNA/RNA Lysis Buffer | Zymo Research | D7001-1-50 | A guanidinium thiocyanate and N-Lauroylsarcosine-based lysis buffer sold as part of a nucleic acid purification kit |
| DNA/RNA Prep Buffer | Zymo Research | D7010-2-10 | A guanidine HCl and ethanol buffer used for purification of DNA and RNA |
| DNA/RNA Shield | Zymo Research | R1100-50 | DNA and RNA preservation/protection reagent |
| DNA/RNA Wash Buffer | Zymo Research | D7010-3-6 | A salt and ethanol buffer used for purification of DNA and RNA |
| DNBSEQ G-400RS | MGI | G-400RS | High throughput sequencer |
| MGIEasy RNA Directional Library Prep Set | MGI | 1000006386 | Generate libraries for MGI high-throughput sequencing platforms from total RNA. |
| Mini-Beadbeater-96 | BioSpec | 1001 | A high energy, high throughput cell disrupter |
| NEBNext rRNA Depletion Kit (bacteria) | New England Biolabs | E7850X | Efficient and specific depletion of bacterial rRNA (5S, 16S, 23S) |
| Nunc Lab-Tek II chamber slide system | Thermo Fisher Scientific | 154534 | 8-well chamber slide with removable wells |
| Qubit Fluorometer | Thermo Fisher Scientific | Q33238 | Fluorometer for DNA, RNA and proteins |
| Qubit RNA HS Assay Kit | Thermo Fisher Scientific | Q32852 | High sensitivity fluorometric assay to measure RNA concentration |
| Spin-Away Filters | Zymo Research | C1006-50-F | Silica-based spin column primarily used to bind or remove genomic DNA |
| Sterile inoculation loops, 1 uL | Sarstedt | 86.1567.050 | Sterile, disposable inoculation loops for manipulation of microorganisms |
| ZR BashingBead Lysis tubes | Zymo Research | S6003-50 | 2 mL tubes containing 0.1 and 0.5 mm bead lysis matrix for homogenizing biological samples |
| Zymo Spin IIICG Columns | Zymo Research | C1006-50-G | Silica-based spin column for purification of DNA and RNA |
| Zymo-Spin III-HRC Filters | Zymo Research | C1058-50 | Remove inhibitors such as polyphenolic compounds, humic/fulvic acids, tannins, melanin, etc. |
| Zymobiomics DNA/RNA Miniprep kit | Zymo Research | R2002 | DNA and RNA dual extraction kit |
| Zymobiomics HRC Prep solution | Zymo Research | D4300-7-30 | To be used with Zymo-Spin III-HRC Filters to remove PCR inhibitors |
参考文献
- Beaudoin, T., Waters, V. Infections With Biofilm Formation: Selection of Antimicrobials and Role of Prolonged Antibiotic Therapy. The Pediatric Infectious Disease Journal. 35 (6), 695-697 (2016).
- Bjarnsholt, T., et al. The in vivo biofilm. Trends in Microbiology. 21 (9), 466-474 (2013).
- Folsom, J. P., et al. Physiology of Pseudomonas aeruginosa in biofilms as revealed by transcriptome analysis. BMC Microbiology. 10, 294(2010).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43 (3), 313-351 (2017).
- Bjarnsholt, T., Ciofu, O., Molin, S., Givskov, M., Hoiby, N. Applying insights from biofilm biology to drug development - can a new approach be developed. Nature Reviews Drug Discovery. 12 (10), 791-808 (2013).
- Colvin, K. M., et al. The Pel and Psl polysaccharides provide Pseudomonas aeruginosa structural redundancy within the biofilm matrix. Environmental Microbiology. 14 (8), 1913-1928 (2012).
- Hentzer, M., et al. Alginate overproduction affects Pseudomonas aeruginosa biofilm structure and function. Journal of Bacteriology. 183 (18), 5395-5401 (2001).
- Cury, J. A., Koo, H. Extraction and purification of total RNA from Streptococcus mutans biofilms. Analytical Biochemistry. 365 (2), 208-214 (2007).
- Francavilla, M., et al. Extraction, characterization and in vivo neuromodulatory activity of phytosterols from microalga Dunaliella tertiolecta. Current Medicinal Chemistry. 19 (18), 3058-3067 (2012).
- Atshan, S. S., et al. Improved method for the isolation of RNA from bacteria refractory to disruption, including S. aureus producing biofilm. Gene. 494 (2), 219-224 (2012).
- Franca, A., Melo, L. D., Cerca, N. Comparison of RNA extraction methods from biofilm samples of Staphylococcus epidermidis. BMC Research Notes. 4, 572(2011).
- Jurcisek, J. A., Dickson, A. C., Bruggeman, M. E., Bakaletz, L. O. In vitro biofilm formation in an 8-well chamber slide. The Journal of Visusalized Experiments. (47), e2481(2011).
- Beaudoin, T., Kennedy, S., Yau, Y., Waters, V. Visualizing the effects of sputum on biofilm development using a chambered coverglass model. The Journal of Visusalized Experiments. (118), e54819(2016).
- Cockeran, R., et al. Biofilm formation and induction of stress response genes is a common response of several serotypes of the pneumococcus to cigarette smoke condensate. The Journal of Infection. 80 (2), 204-209 (2020).
- Bisht, K., Moore, J. L., Caprioli, R. M., Skaar, E. P., Wakeman, C. A. Impact of temperature-dependent phage expression on Pseudomonas aeruginosa biofilm formation. npj Biofilmsand Microbiomes. 7 (22), (2021).
- Harrison, A., et al. Reprioritization of biofilm metabolism is associated with nutrient adaptation and long-term survival of Haemophilus influenzae. NPJ Biofilms and Microbiomes. 5 (1), 33(2019).
- Sousa, C., Franca, A., Cerca, N. Assessing and reducing sources of gene expression variability in Staphylococcus epidermidis biofilms. BioTechniques. 57, 295-301 (2014).
- Boom, R. Rapid and simple method for purification of nucleic acids. Journal of Clinical Microbiology. 28 (3), 495-503 (1990).
- Coppin, C. Re: How do silica based RNA spin columns only bind RNA and not DNA. , Available from: https://www.researchgate.net/post/How_do_silica_based_RNA_spin_columns_only_bind_RNA_and_not_DNA/60b017bffa5c4151cac1c/citation/download (2021).
- Kennedy, S. A complete guide to how nucleic extraction kits work. , Available from: https://bitesizebio.com/13516/how-dna-extraction-rna-miniprep-kits-work/ (2021).
- Qubit RNA HS Assay Kit User Guide. Thermo Fisher Scientific. , Available from: https://www.thermofisher.com/document-connect/document-connect.html?url=https%3A%2F%2Fassets.thermofisher.com%2FTFS-Assets%2FLSG%2Fmanuals%2FQubit_RNA_HS_Assay_UG.pdf&title=VXNlciBHdWlkZTogUXViaXQgUk5BIEhTIEFzc2F5IEtpdHM (2015).
- Mueller, O., Lightfoot, S., Schroeder, A. RNA Integrity Number (RIN) - Standardization of RNA Quality Control (Application report # 5989-1165EN). , Available from: https://www.agilent.com/cd/library/applications/5989-1165EN.pdf (2016).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 3(2006).
- Culviner, P. H., Guegler, C. K., Laub, M. T. A Simple, Cost-Effective, and Robust Method for rRNA Depletion in RNA-Sequencing Studies. mBio. 11 (2), (2020).
- MGIEasy RNA Directional Library Prep Set User Manual verA2. MGI Tech Co. , Available from: https://en.mgi-tech.com/products/reagents_info/14/ (2020).
- Andrews, S. FastQC: A Quality Control Tool for High Throughput Sequence Data. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina Sequence Data. Bioinformatics. , (2014).
- NCBI Resource Coordinators. Database resources of the National Center for Biotechnology Information. Nucleic Acids Research. 44 (1), (2016).
- Li, H. Aligning sequence reads, clone sequences and assembly contigs with BWA-MEM. arXiv. , (2013).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), (2009).
- Liao, Y., Shi, W. Read trimming is not required for mapping and quantification of RNA-seq reads at the gene level. NAR Genomics and Bioinformatics. 2 (3), (2020).
- Penir, S. Calculating Mapping Statistics from a SAM/BAM file using SAMtools and awk. , Available from: https://sarahpenir.github.io/bioinformatics/awk/calculating-mapping-stats-from-a-bam-file-using-samtools-and-awk/ (2019).
- Haile, S., et al. Evaluation of protocols for rRNA depletion based RNA sequencing of nanogram inputs of mammalian total RNA. PLoS ONE. 14 (10), 0224578(2019).
- Schuierer, S., et al. A comprehensive assessment of RNA-seq protocols for degraded and low-quantity samples. BMC Genomics. 18 (442), (2017).
- Shanker, S., et al. Evaluation of Commercially Available RNA Amplification Kits for RNA Sequencing Using Very Low Input Amounts of Total RNA. Journal of Biomolecular Techniques. 26 (1), (2015).
- Adiconis, X., et al. Comparative analysis of RNA sequencing methods for degraded or low-input samples. Nature Methods. 10, 623-629 (2013).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (13), (2016).
- Illumina, Inc. Coverage depth recommendations. , Available from: https://www.illumina.com/science/technology/next-generation-sequencing/plan-experiments/coverage.html (2021).
- What is a good sequencing depth for bulk RNA-Seq. ECSEQ Bioinformatics. , Available from: https://www.ecseq.com/support/ngs/what-is-a-good-sequencing-death-for-bulk-rna-seq (2019).
- Bedre, R. Sequencing coverage and breadth of coverage. , Available from: https://www.reneshbedre.com/blog/sequencing-coverage.html (2021).
- Dotsch, A., et al. The Pseudomonas aeruginosa transcriptome in planktonic cultures and static biofilms using RNA sequencing. PLoS One. 7 (2), 31092(2012).
- Chen, Y., et al. Population dynamics and transcriptomic responses of Pseudomonas aeruginosa in a complex laboratory microbial community. npj Biofilms and Microbiomes. 5 (1), (2019).
- Thoming, J. G., et al. Parallel evolutionary paths to produce more than one Pseudomonas aeruginosa biofilm phenotype. NPJ Biofilms and Microbiomes. 6, 2(2020).
- Soares, A., et al. Understanding ciprofloxacin failure in Pseudomonas aeruginosa biofilm: persister cells survive matrix disruption. Frontiers in Microbiology. 10, 2603(2019).
- Whiteley, M., et al. Gene expression in Pseudomonas aeruginosa biofilms. Nature. 413, (2001).
- Chomczynski, P., Sacchi, N. The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: twenty-something years on. Nature Protocols. 1, (2006).
- Liu, Y., Zhou, J., White, K. P. RNA-seq differential expression studies: more sequence or more replication. Bioinformatics. 30 (3), (2014).
- Wieczorek, D., Delauriere, L., Schagat, T. Methods of RNA Quality Assessment. , Available from: https://www.promega.ca/resources/pubhub/methods-of-rna-quality-assessment/ (2021).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。