Method Article
Isolamento dell'RNA totale da Pseudomonas aeruginosa all'interno di biofilm per la misurazione dell'espressione genica
In questo articolo
Riepilogo
Questo protocollo presenta un metodo per isolare l'RNA dai biofilm di Pseudomonas aeruginosa cresciuti in vetrini da camera per il sequenziamento ad alto rendimento.
Abstract
Pseudomonas aeruginosa è un patogeno batterico opportunista che causa infezioni nelle vie aeree dei pazienti affetti da fibrosi cistica (FC). P. aeruginosa è noto per la sua capacità di formare biofilm protetti da una matrice di esopolisaccaridi. Questa matrice consente ai microrganismi di essere più resistenti ai fattori esterni, compreso il trattamento antibiotico. Uno dei metodi più comuni di crescita del biofilm per la ricerca è costituito da piastre per microtitolazione o vetrini a camera. Il vantaggio di questi sistemi è che consentono di testare più condizioni di crescita, ma il loro svantaggio è che producono quantità limitate di biofilm per l'estrazione dell'RNA. Lo scopo di questo articolo è quello di fornire un protocollo dettagliato e passo dopo passo su come estrarre l'RNA totale da piccole quantità di biofilm di qualità e quantità sufficienti per il sequenziamento ad alto rendimento. Questo protocollo consente lo studio dell'espressione genica all'interno di questi sistemi di biofilm.
Introduzione
La maggior parte delle infezioni batteriche croniche, come le infezioni polmonari nei pazienti con fibrosi cistica (FC) e le infezioni correlate alla protesi, sono caratterizzate dalla crescita di organismi all'interno dei biofilm. I biofilm1 sono comunità di batteri racchiusi in una matrice composta principalmente da polisaccaridi2. I batteri all'interno dei biofilm possono essere a crescita lenta, metabolicamente dormienti e in condizioni anaerobiche e ipossiche. I biofilm sono più resistenti agli antibiotici a causa di fattori quali la diminuzione della penetrazione degli antibiotici, l'aumento dell'espressione delle pompe di efflusso dei farmaci e la diminuzione della divisione cellulare3. Per questi e altri motivi, sono di grande interesse per la ricerca.
Al fine di studiare accuratamente le infezioni persistenti come le infezioni croniche da Pseudomonas aeruginosa nei pazienti con FC, le condizioni di crescita osservate con la formazione di biofilm devono essere accuratamente riflesse in vitro. Un metodo comune ad alto rendimento consiste nel coltivarli in vetrini da camera o piastre per microtitolazione e monitorare la formazione di biofilm mediante microscopia confocale4. È noto che un regolatore chiave nella transizione da uno stile di vita planctonico, o free-floating, a uno stile di vita batterico del biofilm è il messaggero secondario, ciclico-di-GMP5. L'aumento dei livelli di di-GMP ciclico aumenta l'espressione di geni specifici che promuovono la crescita del biofilm. Anche i piccoli RNA regolatori non codificanti e il quorum sensing svolgono un ruolo importante nella regolazione della formazione del biofilm5. Misurare l'espressione genica del biofilm sequenziando l'RNA batterico estratto può essere difficile. P. aeruginosa, ad esempio, produce tre esopolisaccaridi (Psl, Pel e alginato), che sono prodotti in quantità significative nei biofilm 6,7. Questi polisaccaridi possono interferire con l'estrazione e la purificazione dell'RNA, portando a preparazioni impure contenenti bassi livelli di mRNAbatterico 8. I kit di estrazione dell'RNA disponibili in commercio sono in grado di produrre RNA di alta qualità da colture batteriche planctoniche, ma potrebbero non funzionare altrettanto bene con le colture di biofilm 9,10,11. Esistono alcuni kit commerciali per l'estrazione dell'RNA che affermano di funzionare per i biofilm, uno dei quali viene utilizzato con questo metodo.
In questo manoscritto, descriviamo le procedure per la crescita di biofilm di P. aeruginosa in vetrini da camera e l'estrazione dell'mRNA batterico per il sequenziamento ad alto rendimento12,13. Utilizzando isolati clinici raccolti da campioni di espettorato di pazienti con FC, dimostriamo che questi metodi possono essere utilizzati per isolati con caratteristiche di crescita variabili. Rispetto alle pubblicazioni precedenti, questo protocollo è descritto in dettaglio per consentire un migliore successo nello studio dell'espressione genica del biofilm batterico 11,14,15,16.
Protocollo
Il Comitato Etico della Ricerca (REB) è necessario per la raccolta e l'elaborazione di campioni di espettorato da soggetti umani. Questo studio è stato approvato dall'Hospital for Sick Children (REB#1000019444). Il Research Ethics Board (REB) è tenuto a raccogliere e conservare campioni di espettorato da soggetti umani. Questi studi sono stati approvati dall'Hospital for Sick Children REB#1000058579.
1. Formazione del biofilm
- Coltiva gli isolati di Pseudomonas aeruginosa ottenuti dai campioni di espettorato di pazienti con FC utilizzati in questo studio su piastre di agar Luria Broth (LB) in un'incubatrice a 37 °C durante la notte.
NOTA: Una corretta tecnica di striature è importante per ottenere singole colonie batteriche. Le striature durante la rotazione della piastra diluiranno le cellule batteriche a sufficienza in modo che le singole colonie possano crescere.- Striare i batteri utilizzando un anello di inoculazione a zig-zag all'estremità superiore di una piastra LB fresca fino a coprire circa 1/4 della piastra.
- Ruotare la piastra di circa 60°. Prendi un nuovo anello di inoculazione e passalo una volta attraverso l'area striata e in una seconda area pulita della piastra, ripetendo lo schema a zig-zag.
- Ripetere il passaggio 1.1.2 con un nuovo anello in una terza area della piastra. Riposizionare il coperchio e capovolgere la piastra quando si inserisce nell'incubatrice.
- Utilizzare un ansa di inoculazione sterile per prelevare una singola colonia batterica da una piastra di agar (contenente un singolo ceppo batterico) e inoculare una provetta di coltura riempita con 5 ml di terreno sterile LB. Utilizzando ogni volta un nuovo ansa, inoculare due provette di coltura aggiuntive riempite con 5 mL di terreno LB dalla stessa piastra.
- Ripetere la stessa procedura di inoculazione per i diversi ceppi batterici. Coltivare le colture durante la notte a 37 °C con agitazione a 220 giri/min.
NOTA: Ogni piastra di agar contenente un singolo ceppo batterico viene utilizzata per inoculare 3 provette di coltura indipendenti. Le tre provette rappresentano repliche biologiche in triplicato per un ceppo e sono trattate come campioni separati. Questo è diverso dalle repliche tecniche che comporterebbero l'estrazione dell'RNA 3 volte da una singola provetta di coltura. - Preparare diluizioni 1:100 delle colture notturne trasferendo 50 μL di una coltura notturna in una nuova provetta contenente 4,95 mL di nuovo terreno LB.
- Far crescere le colture diluite per altre 3 ore a 37 °C agitando a 220 giri/min o fino a quando l'OD600 è 0,1 o superiore. Misurare le densità delle celle su uno spettrofotometro a OD600.
- In una nuova provetta per microfugo da 1,7 mL, regolare l'OD600 a 0,1 (fase di log iniziale) in un volume totale di 1,5 mL con LB fresco.
- Mescolare delicatamente per inversione. Trasferire 300 μl di ciascuna coltura regolata in 4 pozzetti di un vetrino a camera da 8 pozzetti per ottenere 2 campioni diversi per vetrino (Figura 1).
- Posizionare i vetrini, indisturbati, in un'incubatrice a 37 °C per una notte per 24 ore. Per evitare l'evaporazione, posizionare i vetrini sopra un tovagliolo di carta umido in un piccolo vassoio di plastica.
2. Recupero del biofilm
NOTA: Ogni vetrino contiene otto pozzetti separati. Un singolo campione è costituito da quattro pozzetti con biofilm che verranno raggruppati17. Questo protocollo di estrazione è per 1 campione (4 pozzetti) in cui i biofilm vengono recuperati da 2 pozzetti alla volta. Le estrazioni dell'RNA vengono eseguite utilizzando un kit di estrazione dell'RNA commerciale che include una fase di battitura delle perle e una pulizia basata su colonna, con modifiche. Seguire le istruzioni del produttore per la preparazione del reagente.
- In una cappa a flusso laminare, rimuovere lentamente il terreno da 2 dei 4 pozzetti utilizzando un puntale per pipetta. Inclinare il vetrino di un angolo di 45° e pipettare il terreno dall'angolo inferiore dei pozzetti per evitare il distacco dei biofilm.
- Mantenendo il vetrino inclinato, lavare via le cellule planctoniche pipettando delicatamente 300 μl di acqua priva di RNasi nell'angolo inferiore dei due pozzetti svuotati. Rimuovere l'acqua pipettandola delicatamente, come descritto al punto 2.1. Ripetere la fase di lavaggio rimuovendo quanta più acqua possibile.
- Aggiungere 300 μl di un reagente di protezione dell'RNA (vedi Tabella dei materiali) a ciascuno dei due pozzetti svuotati con biofilm alla base. Posizionare il vetrino della camera su una lastra di vetro per evitare che i pozzetti si rompano, quindi raschiare i biofilm nei 2 pozzetti con una piccola spatola metallica sterile priva di nucleasi per risospendere i batteri del biofilm. Lasciare riposare fino a quando i biofilm non vengono recuperati dai 2 pozzetti rimanenti intatti dello stesso campione.
NOTA: L'aggiunta di un reagente di protezione dell'RNA garantisce la stabilità dei campioni di biofilm nei pozzetti raschiati a temperatura ambiente durante l'elaborazione dei restanti due pozzetti dello stesso campione. Il reagente di protezione dell'RNA lisa le cellule e inattiva le nucleasi e gli agenti infettivi, con conseguente conservazione dell'RNA. - Per recuperare i biofilm dai restanti 2 pozzetti per un campione (promemoria: un campione è composto da 4 pozzetti), rimuovere il terreno LB dai 2 nuovi pozzetti rimanenti nello stesso modo descritto nel passaggio 2.1. Ripetere il passaggio 2.2 per lavare via le cellule planctoniche con acqua priva di RNasi da entrambi i nuovi pozzetti, come prima.
- Tornare ai primi 2 pozzetti con i biofilm raschiati nel reagente di protezione, generati alla fine del passaggio 2.3, e pipettare lentamente per miscelare i 300 μL di biofilm risospeso da un pozzetto raschiato, cercando di non creare troppe bolle.
- Trasferire tutto il contenuto dal pozzetto in uno dei pozzetti appena lavati e svuotati. Miscelare e trasferire 300 μl di biofilm risospeso dal secondo pozzetto raschiato, nel pozzetto rimanente, appena lavato e svuotato.
NOTA: Invece di aggiungere un nuovo reagente di protezione dell'RNA ai 2 pozzetti appena lavati con biofilm, trasferire i biofilm precedentemente raschiati già nel reagente di protezione in questi pozzetti appena lavati. In questo modo il volume del campione combinato rimarrà sufficientemente basso da soddisfare i requisiti di input per il kit di estrazione dell'RNA commerciale. Vedere la Figura 1 per uno schema.
- Trasferire tutto il contenuto dal pozzetto in uno dei pozzetti appena lavati e svuotati. Miscelare e trasferire 300 μl di biofilm risospeso dal secondo pozzetto raschiato, nel pozzetto rimanente, appena lavato e svuotato.
- Ripetere la raschiatura dei biofilm nei nuovi pozzetti come al punto 2.3, posizionando il vetrino della camera su una lastra di vetro e raschiando i biofilm nei 2 nuovi pozzetti con una piccola spatola metallica sterile priva di nucleasi per risospendere i batteri del biofilm.
- Combina tutto il biofilm risospeso dai 2 nuovi pozzetti in un'unica provetta per microcentrifuga da 1,5 mL, priva di RNasi, a basso legame. Misurare il volume, che dovrebbe essere ~500 - 600 μL totali.
NOTA: La sospensione combinata di biofilm di questa seconda coppia di nuovi pozzetti conterrà tutto il materiale del biofilm proveniente dai 4 pozzetti originali di un campione.
3. Isolamento dell'RNA totale e valutazione della qualità
NOTA: L'estrazione dell'RNA viene eseguita utilizzando un kit di estrazione dell'RNA commerciale che afferma di funzionare sui biofilm. I singoli componenti sono inclusi nella tabella dei materiali, se possibile. Quando possibile, vengono fornite spiegazioni sui meccanismi alla base di ogni fase di purificazione.
- Aggiungere una quantità sufficiente di reagente di protezione dell'RNA per un totale di 750 μl nella provetta. Trasferire l'intero volume in una provetta da 2 mL contenente perle da 0,1 e 0,5 mm (vedere la Tabella dei materiali). Sbattere per 2 1/2 min in una frusta per perline alla massima velocità.
NOTA: La combinazione di perle ad alta densità da 0,1 mm e 0,5 mm e la miscelazione ad alta velocità su un battitore di microsfere garantisce un'omogeneizzazione completa delle pareti cellulari microbiche. - Centrifugare a 16.000 x g per 1 minuto per pellettare le perline. Trasferire il surnatante in una nuova provetta da microcentrifuga, riducendo al minimo il trasferimento di eventuali perline, il che renderà più facile il passaggio 3.3. Misura il volume.
- Aggiungere un volume uguale di tampone di lisi dell'RNA (~450 μL) e mescolare bene. Trasferire fino a 800 μl di campione, evitando il trasferimento di perline, in una colonna di silice in una provetta di raccolta e centrifugare a 16.000 x g per 30 s. Salvare il flusso in quanto contiene l'RNA mentre il DNA è legato alla colonna.
NOTA: Il tampone di lisi dell'RNA contiene tiocianato di guanidinio e il detergente N-lauroilsarcosina per lisi le cellule. Il tiocianato di guanidinio è un agente caotropo che inattiva anche le nucleasi e, in presenza di silice, presente nella colonna di spin, favorisce il legame del DNA alla silice18. L'assenza di etanolo consente il legame preferenziale del DNA e non dell'RNA alla colonna di spin19 della silice. Lo scopo della fase 3.3 è quello di legare e rimuovere il DNA genomico. Vogliamo trattenere l'RNA, che è contenuto nella porzione a flusso continuo. - Se rimane più campione, trasferire la colonna in una nuova provetta di raccolta e ricaricare con il resto del campione. Centrifugare a 16.000 x g per 30 s. Mantenere il flusso con l'RNA e combinarlo con la prima aliquota.
- Misurare il volume di flusso combinato e aggiungere un volume uguale di etanolo al 100 % e mescolare bene. Trasferire fino a 800 μl della soluzione in una nuova seconda colonna di centrifuga di silice in una provetta di raccolta e centrifugare a 16.000 x g per 30 s. Scartare il flusso.
NOTA: L'aggiunta di etanolo alla soluzione salina caotropica contenente l'RNA migliora il legame dell'RNA alla colonna di spin della silice19. - Per soluzioni > 800 μl, ricaricare la colonna rotante e centrifugare fino a quando l'intera soluzione non è stata centrifugata. Scartare il flusso dopo ogni centrifuga.
- Aggiungere 400 μl di tampone di lavaggio alla colonna e centrifugare a 16.000 x g per 30 s per rimuovere alcuni sali. Scartare il flusso.
- Preparare la miscela di reazione DNasi I secondo le istruzioni del produttore ed eseguire il trattamento DNasi in colonna per rimuovere eventuali residui di DNA.
- Risospendere la DNasi I liofilizzata in 275 μL di acqua priva di RNasi per ottenere una soluzione da 1 U/μL. Mescolare per leggera inversione.
- Combinare 5 μl di DNasi I diluita con 75 μl del tampone di digestione DNasi fornito. Mescolare delicatamente per inversione.
- Aggiungere 80 μl della soluzione preparata direttamente sulla matrice della colonna. Incubare a temperatura ambiente per 20 min.
- Aggiungere 400 μl di tampone di preparazione dell'RNA alla colonna e centrifugare a 16.000 x g per 30 s. Scartare il flusso.
- Aggiungere 700 μL di tampone di lavaggio dell'RNA alla colonna e ripetere la centrifugazione. Scartare il flusso.
- Aggiungere 400 μl di tampone di lavaggio dell'RNA e centrifugare la colonna per 2 minuti per rimuovere completamente l'eventuale tampone residuo.
NOTA: Ci sono 2 diverse fasi di lavaggio per rimuovere le impurità sulla colonna. Il tampone di preparazione contiene un sale caotropico debole mescolato con etanolo per rimuovere le proteine residue. Successivamente, il tampone di lavaggio viene utilizzato per eseguire lavaggi con etanolo per rimuovere i sali. L'eventuale etanolo rimanente deve essere rimosso per consentire un'efficiente eluizione dell'RNA19,20. - Trasferire con cautela la colonna in una nuova provetta per microcentrifuga a basso legante.
- Aggiungere 50 μl di acqua priva di RNasi direttamente nella matrice della colonna e incubare per 5 minuti. Centrifugare a 16.000 x g per 1 minuto per eluire l'RNA.
- Per un'ulteriore pulizia per rimuovere gli inibitori della PCR, posizionare un filtro inibitore della PCR in una nuova provetta di raccolta. Aggiungere 600 μl della soluzione di preparazione dell'inibitore fornita (vedere la Tabella dei materiali).
- Centrifugare a 8.000 x g per 3 min per lavare il filtro. Trasferire il filtro lavato in una nuova provetta da microcentrifuga a basso legante.
- Trasferire l'RNA eluito dal passaggio 3.13 nel filtro lavato e centrifugare a 16.000 x g per 3 minuti.
NOTA: L'RNA può essere utilizzato immediatamente o conservato a -80 °C. - Determinare la concentrazione dell'RNA utilizzando un sistema fluorimetrico ad alta sensibilità21.
NOTA: Questi sistemi consentono la quantificazione sensibile di una piccola quantità di RNA in soluzione specifica per il bersaglio di interesse.- Valutare la qualità dell'RNA utilizzando un sistema di elettroforesi automatizzato in grado di fornire un RIN (numero di integrità dell'RNA), che è una misura della qualità dell'RNA22,23.
4. Deplezione dell'RNA ribosomiale e sequenziamento ad alto rendimento
- Inviare gli RNA al Centro per l'Analisi dell'Evoluzione e della Funzione del Genoma (CAGEF) presso l'Università di Toronto (Toronto, Canada) (https://www.cagef.utoronto.ca/) per la deplezione batterica dell'rRNA e la preparazione della libreria direzionale dell'RNA (vedi Tabella dei Materiali).
NOTA: La deplezione batterica dell'rRNA prende di mira gli rRNA 5S, 16S e 23S per la rimozione24. - Esaurire gli rRNA utilizzando un kit di deplezione batterica rRNA commerciale. Seguire il protocollo per l'immissione di 10 ng - 1 μg di RNA totale intatto o parzialmente degradato.
- Costruisci librerie di sequenziamento dell'RNA utilizzando un kit di preparazione della libreria direzionale dell'RNA con diversi indici collegati a ciascuna libreria.
- Raggruppare quantità equimolari di ciascuna libreria di RNA ed eseguire il sequenziamento ad alto rendimento con 100 letture di estremità accoppiate di 100 basi25.
5. Valutazione della qualità delle letture di sequenziamento
NOTA : Verificare la qualità delle letture di sequenziamento utilizzando il programma disponibile gratuitamente, FastQC26, disponibile attraverso la piattaforma gratuita e open source, Galaxy27.
- Vai a https://usegalaxy.org/. Clicca sul menu Accedi o Registrati ed effettua l'accesso con le credenziali o crea un account.
- Fare clic sul collegamento Carica dati in alto a sinistra della pagina, nel menu Strumenti, e caricare i file di sequenziamento fastq.gz. Attendi che i nomi dei file appaiano sul lato destro della pagina, sotto il pannello Cronologia.
- Selezionare Controllo qualità FASTQ nel menu Strumenti per visualizzare un elenco di programmi. Seleziona FastQC, che popolerà il pannello centrale dello schermo.
- In Lettura breve dei dati dalla cronologia corrente, selezionare i file di fastq.gz caricati dal menu a discesa.
- Selezionare Esegui per eseguire il programma.
- Visualizzare i risultati nel pannello Cronologia (Tabella 2).
NOTA: Per istruzioni più dettagliate su come utilizzare Galaxy, visitare la pagina di supporto all'indirizzo https://galaxyproject.org/support/.
6. Mappatura delle letture di sequenziamento
NOTA : Di seguito è elencata una pipeline di base per il taglio dell'adattatore e la mappatura della lettura per i dati RNA-seq. Le sequenze dell'adattatore vengono tagliate dalle letture utilizzando Trimmomatic28. Le reads tagliate sono mappate sul genoma di riferimento PAO1 di P. aeruginosa (NC_002516.2), ottenuto da NCBI (https://www.ncbi.nlm.nih.gov/)29utilizzando BWA30 e Samtools31. Per semplicità, una coppia di letture è chiamata PA_1.fq e PA_2.fq; Il file letto dell'adattatore da tagliare si chiama adapter.fa; e la sequenza di riferimento PAO1 è chiamata PAO1.fasta. Tutti gli strumenti sono open source e vengono eseguiti in ambiente UNIX/LINUX. Si consiglia vivamente di familiarizzare con i fondamenti di UNIX/LINUX per eseguire questi comandi.
- Aprire una finestra in UNIX/LINUX.
- Installa Java, Trimmomatic, BWA e Samtools.
- Passare alla cartella in cui risiede il file trimmomatic-0.39.jar.
- Taglia tutte le sequenze dell'adattatore dalle letture digitando il comando:
Java -jar PA_1.fq PA_2.fq PA_1_paired.fq PA_1_unpaired.fq PA_2_paired.fq PA_2_unpaired.fq ILLUMINACLIP: adattatori.fa
NOTA: Vengono rimosse solo le sequenze dell'adattatore. Le letture non sono state tagliate per la qualità32. - Spostare il file di riferimento PAO1.fasta nella stessa cartella.
- Indicizzare il riferimento utilizzando BWA con il comando:
Indice Bwa PA01.fasta - Mappare le letture accoppiate al genoma di riferimento digitando i seguenti 4 comandi. Digitare ogni comando al termine del precedente.
Bwa -mem PA01.fasta PA_1_paired.fq PA2_2_paired.fq > PA_R1R2_map.mem.sam
Samtools view -S -b PA_R1R2_map.mem.sam > PA_R1R2.bam
Samtools ordina PA_R1R2.bam -o PA_R1R2_sorted.bam
Indice Samtools PA288_Rep1_R1R2_sorted.bam - Visualizza le statistiche di mappatura digitando il comando:
Samtools flagstat PA_R1R2_sorted.bam
NOTA: La 3ariga delle statistiche riporta la proporzione delle letture che mappano il genoma di riferimento. - Calcola la profondità media di lettura e l'ampiezza della copertura con i seguenti 2 comandi33, rispettivamente:
Samtools profondità -A PA_R1R2_sorted.bam | awk '{c++; s+=$3}END{stampa s/c}'
Samtools profondità -A PA288_Rep1_R1R2_sorted.bam | awk '{c++; if($3>0) totale+=1}END{stampa (totale/c)*100}'
Risultati
La panoramica generale del metodo è illustrata nella Figura 1. In precedenza abbiamo utilizzato vetrini da camera a 8 pozzetti per far crescere biofilm di P. aeruginosa ed esporli ad antibiotici prima di esaminarli tramite microscopia confocale in diversi punti temporali12,13. Questo metodo può essere utilizzato per estrarre l'RNA totale direttamente dai biofilm cresciuti in questo sistema al fine di studiare i cambiamenti dell'espressione genica dopo il trattamento. Questo protocollo è stato ottimizzato per P. aeruginosa, ma può essere facilmente adattato per altre specie batteriche.
Può essere difficile estrarre una quantità sufficiente di RNA di buona qualità da piccole quantità di biofilm per la deplezione dell'rRNA e il sequenziamento ad alto rendimento. Utilizzando questo protocollo, l'RNA totale viene estratto con successo da 17 diversi isolati di biofilm di P. aeruginosa in triplicato, per un totale di 51 campioni separati. Le quantità di RNA estratti che rappresentano rese alte e basse sono mostrate nella Tabella 1. Gli RNA variano in concentrazione da 3,4 ng/μL (il più basso) a 49,6 ng/μL (il più alto), con una concentrazione media di 14 ng/μL e una mediana di 13,7 ng/μL. Concentrazioni di RNA intatto inferiori a 10 ng/μL sono considerate campioni a bassa abbondanza per la deplezione dell'rRNA e il sequenziamento di nuova generazione, ma i campioni di RNA a bassa abbondanza dai biofilm possono portare a dati di sequenziamento di qualità ancora più scadente rispetto a campioni più concentrati34, 35,36,37. Le qualità degli RNA sono mostrate nella Tabella 1 dal RIN e nella Figura 2 dai corrispondenti elettroferogrammi dell'RNA per i campioni concentrati a bassa (PA565-3) e alta (PA288-1); i due campioni rimanenti (PA375-3 e PA921-1) sono rappresentativi della maggior parte dei campioni. Come si vede nella Figura 2, l'RNA estratto da queste quantità di biofilm contiene sempre un po' di RNA degradato, che influisce sui loro valori RIN. Pertanto, la conferma visiva dei picchi di rRNA procariotico 16S e 23S viene utilizzata per determinare la qualità dell'RNA quando non viene riportato un RIN. Utilizzando questi criteri, tutti i 51 campioni di RNA vengono scelti per procedere alla deplezione e al sequenziamento dell'rRNA, con la previsione che il campione di qualità più scarsa, PA565-3, potrebbe fallire. Dei campioni di RNA inviati per il sequenziamento, le librerie di successo vengono generate e sequenziate per 49 campioni, con PA565-3 che fallisce, come previsto.
Il numero di letture di sequenziamento generate per il campione ad alta concentrazione con un buon RIN (PA288-1) e il campione a bassa concentrazione senza RIN (PA375-3) è elencato nella Tabella 2. Le statistiche di base includono il numero totale di letture, la lunghezza di lettura e il contenuto GC. Le statistiche di riepilogo per entrambi i campioni mostrano un numero elevato di letture generate e indicano che nessuna è contrassegnata come di scarsa qualità, suggerendo buoni dati di sequenziamento. Il numero medio di letture per campione è di circa 48 milioni, che è considerato un buon rendimento. Uno strumento standard utilizzato per valutare la qualità dei dati grezzi di sequenziamento è FastQC26. Questo programma viene utilizzato per eseguire controlli di qualità sui file di sequenziamento grezzi per determinare se la qualità è sufficiente per ulteriori analisi o se ci sono problemi o distorsioni nei dati generati dal sequenziatore stesso o dalle librerie di RNA in ingresso. La Figura 3 presenta le metriche di controllo della qualità per i dati di sequenziamento di PA288-1 e PA375-3, che rappresentano rispettivamente un campione di RNA di alta qualità e un campione di RNA tipico di qualità inferiore. Uno dei grafici più informativi di FastQC è il grafico di qualità per sequenza di base. Buoni dati di sequenziamento mostreranno un punteggio di qualità mediano elevato (>30) per ogni posizione in tutte le letture, con un calo del punteggio di qualità medio per tutta la durata della lettura. Un punteggio di qualità di 30 rappresenta un tasso di errore di 1 su 1000, corrispondente a una precisione di chiamata di base del 99,9%. La stragrande maggioranza delle basi nella Figura 3 ha punteggi di qualità medi ≥ 35 per l'intera lunghezza della lettura per entrambi i campioni, che sono indicativi di dati di sequenziamento di qualità eccezionalmente buona. Ciò fornisce una forte prova che il protocollo di estrazione dell'RNA qui presentato ha successo.
Per dimostrare che il nostro metodo è in grado di recuperare i trascritti di P. aeruginosa, le letture di sequenziamento di alta qualità per PA288-1 e PA375-3 sono mappate sul genoma di riferimento PAO1 di P. aeruginosa (NCBI NC_002516.2). Le letture vengono tagliate solo per l'adattatore di sequenziamento e non per la qualità, mantenendo tutte le basi prima della mappatura32. Le statistiche di mappatura sono presentate nella Tabella 3. La percentuale di letture mappate è una misura importante dell'accuratezza della sequenziazione. In poche parole, più letture si allineano alla sequenza di riferimento, meglio è. Per PA288-1 e PA375-3, l'84% e il 91% delle read, rispettivamente, mappano il genoma di riferimento. L'intervallo previsto per la mappatura delle letture standard di RNA-seq è compreso tra il 70 e il 90%, quindi questi valori sono molto buoni, soprattutto quando le basi di scarsa qualità non vengono rimosse38. La profondità media di lettura è un buon indicatore del numero medio di letture che si allineano in ogni posizione di base nella sequenza di riferimento. Maggiore è la profondità in ogni posizione di base, più accurata è la chiamata di base in ogni posizione. Viene calcolato dividendo la somma delle profondità di lettura mappate in ogni posizione nel genoma di riferimento per il numero totale di basi nel riferimento. PA288-1 e PA375-3 hanno profondità medie di lettura di 400 o superiori, il che è buono per l'analisi dell'espressione genica a valle39,40. L'ampiezza della copertura indica la percentuale della lunghezza del genoma di riferimento che è coperta dal sequenziamento41. Le letture allineate di PA288-1 e PA375-3 coprono il 96% del genoma di riferimento PAO1 di P. aeruginosa. Ciò suggerisce che la maggior parte del genoma di P. aeruginosa è rappresentata nei dati di sequenziamento e non solo in brevi tratti. Le statistiche di mappatura per PA288-1 e PA375-3 mostrano che questo metodo può recuperare trascrizioni che si allineano con una buona copertura e distribuzione al genoma di P. aeruginosa, supportando ulteriormente un protocollo di estrazione di successo.
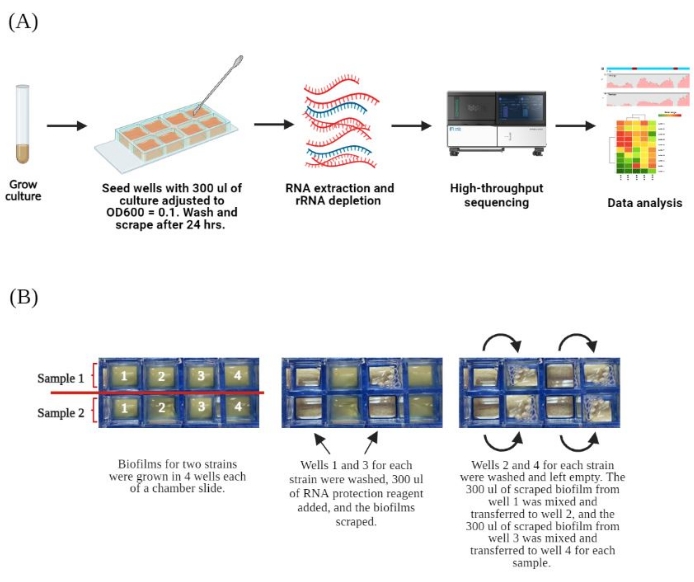
Figura 1. Panoramica del protocollo. Un. Schema del flusso di lavoro sperimentale. Le colture planctoniche sono state coltivate per una notte a 37 °C, diluite 1:100 con terreno fresco il giorno successivo e coltivate per altre 3 ore. Le colture sono state regolate con un OD600 di 0,1 e 300 μl inoculate in 4 pozzetti di un vetrino a 8 camere per generare biofilm. Dopo 24 ore, i biofilm sono stati lavati per rimuovere le cellule planctoniche; È stato aggiunto un reagente di protezione dell'RNA e le cellule sono state raschiate via dai pozzetti. L'RNA totale è stato estratto, impoverito di RNA ribosomiale e sequenziato. B. Flusso di lavoro dettagliato della crescita e della rimozione del biofilm da un vetrino a 8 camere. Sono stati coltivati due ceppi per vetrino nell'orientamento mostrato a sinistra. Viene mostrata la disposizione di ciascun campione in quattro pozzetti del vetrino. Per ogni campione indipendente, i pozzetti 1 e 3 sono stati processati per primi, terminando con 300 μl di cellule raschiate nel reagente di protezione dell'RNA in ciascun pozzetto. Un esempio è rappresentato dalla diapositiva centrale. Successivamente, i pozzetti 2 e 4 sono stati lavati e il liquido rimosso. Le celle ri-sospese dai pozzetti 1 e 3 vengono trasferite rispettivamente ai pozzetti 2 e 4, mostrati nella diapositiva di destra. Dopo la raschiatura, le celle risospese nei pozzetti 2 e 4 vengono combinate in un'unica provetta per microcentrifuga. Questa figura è stata creata con BioRender.com. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2. Esempi di elettroferogrammi di RNA da campioni di RNA estratti di alta e bassa qualità. I picchi ribosomiali 16S e 23S sono etichettati alla base dei loro picchi. L'RNA degradato è rappresentato da picchi di piccole dimensioni, indicati dalle frecce e da una linea di base irregolare sopra lo zero. I campioni di RNA di qualità accettabile sono mostrati nei pannelli A, B e D. Il pannello C mostra un campione di RNA di scarsa qualità in cui mancano i picchi ribosomiali e la concentrazione era di abbondanza molto bassa, indicata dalla scala sull'asse Y. FU, unità di fluorescenza; NT, nucleotide. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3. Grafico di qualità FastQC per sequenza di base per i dati delle sequenze PA288-1 e PA375-3. Un. Il grafico di qualità per il campione di alta qualità PA288-1. B. Il grafico di qualità per il campione tipico PA375-3. I grafici mostrano il punteggio di qualità aggregato per ogni posizione di base per tutte le letture nel file. La linea blu rappresenta il punteggio medio di qualità in ogni posizione di base. La linea rossa all'interno della casella gialla rappresenta il punteggio di qualità mediano in ogni posizione, mentre la casella gialla mostra l'intervallo dei quartili per il 25° e il 75° percentile. Clicca qui per visualizzare una versione più grande di questa figura.
| Isolare | Qubit | RIN | Sequenziato |
| PA288-1 | 26 ng/μl | 7.5 | Sì |
| PA375-3 | 4,07 ng/μl | NA | Sì |
| PA565-3 | 3,4 ng/μl | NA | No |
| PA921-1 | 9,11 ng/μl | 6.6 | Sì |
Tabella 1. Metriche di qualità degli RNA estratti da campioni rappresentativi.
| PA288-1 | PA375-3 | |
| Letture totali | 8,59,57,720 | 3,18,49,575 |
| Letture contrassegnate come di scarsa qualità | 0 | 0 |
| Lunghezza di lettura | 100 | 100 |
| % GC | 61 | 60 |
Tabella 2. Statistiche riassuntive FastQC PA288-1 e PA375-3
| Isolare | % letture mappate al riferimento | Profondità di lettura media | Ampiezza della copertura |
| PA288-1 | 83.93% | 404 | 96.68% |
| PA375-3 | 91.2% | 578 | 96.97% |
Tabella 3. Statistiche di mappatura PA288-1 e PA375-3
Discussione
L'RNA totale viene estratto con successo da 17 diversi campioni di biofilm batterico in triplicato, per un totale di 51 campioni. Le quarantanove librerie di RNA vengono raggruppate e sequenziate con successo. Nel complesso, ciò convalida i nostri criteri di qualità con un tasso di successo del 96%, anche se più della metà dei campioni è considerata di bassa abbondanza e di qualità non ottimale 34,35,36,37.
Significato
Questo protocollo di estrazione dell'RNA è unico nella sua spiegazione dettagliata per estrarre l'RNA da quantità limitate di biofilm che sono stati coltivati in vetrini da camera. La crescita di biofilm in un vetrino da camera a 8 pozzetti è un sistema utile per studiare gli effetti di fattori aggiunti esogenamente ai biofilm o gli effetti delle interazioni microbiche nella formazione di biofilm mediante microscopia confocale 12,13,42. Al fine di esaminare l'espressione di geni coinvolti nella formazione del biofilm in questo sistema, presentiamo un metodo dettagliato per estrarre RNA intatto di qualità e quantità sufficienti per il sequenziamento dell'RNA. Altri studi hanno riportato il successo dell'estrazione dell'RNA da quantità limitate di biofilm, ma la maggior parte fa crescere i biofilm su una superficie più ampia e spesso per 48 ore invece di 24 ore. Mancano anche di dettagli sufficienti per garantire il successo 4,15,16,43,44,46.
Inoltre, questo protocollo evita l'uso di sostanze chimiche pericolose (come il fenolo) o di attrezzature specializzate (come un sonicatore). Il classico protocollo di estrazione guanidinio tiocianato fenolo-cloroformio47 non viene utilizzato per questo sistema perché, anche se produce circa 2 volte più RNA rispetto al kit commerciale, nelle nostre mani risulta costantemente in RNA completamente degradato, come valutato su un sistema di elettroforesi automatizzato. Inoltre, è dimostrato che l'uso di un kit di estrazione commerciale si traduce in un protocollo di facile utilizzo che produce risultati coerenti 15,16,45.
Passaggi critici
Ci sono una serie di passaggi critici in questo protocollo che aumentano la probabilità di estrarre l'RNA che può essere sequenziato con successo. Innanzitutto, è importante inoculare almeno 4 pozzetti del vetrino della camera con lo stesso ceppo al fine di ottenere quantità sufficienti di RNA da sequenziare. I biofilm dei 4 pozzetti sono raggruppati per una singola estrazione, il che ha l'ulteriore vantaggio di ridurre la variabilità nell'analisi dell'espressione genica a valle17. L'estrazione di RNA da meno di 4 pozzetti spesso porta a rese troppo basse per essere rilevate su un sistema fluorimetrico ad alta sensibilità. L'estrazione dell'RNA da 8 pozzetti o da un intero vetrino produrrà quantità maggiori di RNA di qualità simile a quella ottenuta utilizzando 4 pozzetti, ma bisogna considerare se il tempo, lo sforzo e il costo aggiuntivi per l'estrazione valgono l'aumento della resa. Il pipettaggio di 300 μl di coltura diluita OD600 = 0,1 per seminare ogni pozzetto invece dei 200 μl standard migliora il recupero del biofilm raschiato da ciascun pozzetto. Lavare delicatamente il biofilm due volte con acqua priva di nucleasi prima di applicare il reagente di protezione dell'RNA è fondamentale per rimuovere il maggior numero possibile di cellule morte e/o planctoniche. L'uso del reagente protettivo dell'RNA è importante per prevenire la degradazione dell'RNA a causa del tempo necessario per elaborare ciascun pozzetto. Inoltre, invece di raschiare con i puntali delle pipette, preferiamo utilizzare spatole di pesatura in metallo con un'estremità piatta che entra in contatto con una superficie più ampia rispetto a una punta di pipetta e che sono abbastanza piccole da entrare in un pozzetto. La raschiatura con un puntale per pipetta da 1000 μl funzionerà, ma è meno efficiente e richiede più tempo per raschiare l'intero biofilm rispetto alla spatola metallica. Nella nostra esperienza, l'uso di spatole metalliche consente di risparmiare tempo e fatica. Durante la raschiatura, assicurarsi di posizionare il vetrino della camera sopra la lastra di vetro per evitare di rompere il fondo dei pozzetti. La sonicazione non è efficace in quanto provoca costantemente un RNA altamente degradato a causa di un volume di campione insufficiente e di un'eccessiva produzione di calore. Dopo aver raccolto il materiale del biofilm, il campione viene inserito in un kit commerciale per estrazioni di RNA più affidabili e riproducibili. Infine, a causa delle potenziali fonti di variazione in questo metodo, è molto importante includere la replicazione sperimentale oltre al pooling dei campioni17,48. Questo protocollo descrive la replicazione biologica triplicata di ciascun campione.
Limitazioni
Esistono diverse tecniche utilizzate per far crescere i biofilm per lo studio, la più comune delle quali è la formazione in piastre per microtitolazione4. Queste piastre sono disponibili con diverse dimensioni e numeri di pozzetti. I vetrini da camera rientrano in questa categoria. La capacità di estrarre RNA puro e intatto è importante per tutti i sistemi di biofilm, ma questo sistema è limitato dalle basse rese di RNA. Le concentrazioni sono sufficientemente basse da richiedere che la loro qualità e quantità debbano essere valutate da strumenti Qubit e Bioanalyzer utilizzando kit di RNA ad alta sensibilità. Può essere utilizzato anche un nanodrop, ma ha una sensibilità più limitata rispetto al Qubit e non è in grado di distinguere il DNA contaminante dall'RNA49. Se possibile, si dovrebbe utilizzare una nanogoccia per ottenere i rapporti di purezza A260/A280 e A260/A230 . Questi rapporti sono utili, soprattutto perché la qualità dell'RNA di questo sistema di biofilm non è buona come quella di grandi quantità di materiale di partenza. Ciò è molto probabilmente dovuto alla bassa resa di RNA e alla presenza di cellule morte nei biofilm e/o nell'RNasi che possono degradare l'RNA. Un'altra limitazione di questo metodo è che non può separare diversi tipi di cellule all'interno di un biofilm eterogeneo, ma può solo estrarre le cellule come un'intera popolazione, oscurando potenzialmente l'espressione genica in eventuali sottopopolazioni sottorappresentate.
Potenziali Applicazioni
Il protocollo qui presentato è stato ottimizzato per i biofilm di P. aeruginosa provenienti da isolati clinici cresciuti in vetrini da camera. Questo metodo può essere utilizzato per studiare i cambiamenti nell'espressione genica durante la formazione del biofilm in diverse condizioni di crescita osservate dalla microscopia confocale. Il protocollo può anche essere ottimizzato per altre specie batteriche che generano biofilm. Con questo metodo è anche possibile estrarre il DNA genomico poiché è possibile utilizzare un kit a doppia estrazione. In questo modo, l'espressione genica del biofilm batterico negli isolati clinici che causano infezioni nei pazienti può essere studiata in modo più accurato, portando a risultati che potrebbero guidare le future strategie di trattamento.
Divulgazioni
Gli autori non hanno divulgazioni da dichiarare.
Riconoscimenti
Contributi degli autori: P.W., Y.Y. e V.W sono stati coinvolti nella concettualizzazione dello studio. K.G., L.J., A.M. e P.W. hanno ottimizzato i protocolli di laboratorio. Il finanziamento di K.G. è stato sostenuto dal sussidio del programma di tirocinio studentesco attraverso BioTalent Canada.
Materiali
| Name | Company | Catalog Number | Comments |
| Agilent 2100 Bioanalyzer | Agilent | G2939BA | Automated electrophoresis of biomolecules |
| Agilent RNA 6000 pico kit | Agilent | 5067-1513 | High sensitivity RNA electrophoresis chip to generate a RIN |
| DNA/RNA Lysis Buffer | Zymo Research | D7001-1-50 | A guanidinium thiocyanate and N-Lauroylsarcosine-based lysis buffer sold as part of a nucleic acid purification kit |
| DNA/RNA Prep Buffer | Zymo Research | D7010-2-10 | A guanidine HCl and ethanol buffer used for purification of DNA and RNA |
| DNA/RNA Shield | Zymo Research | R1100-50 | DNA and RNA preservation/protection reagent |
| DNA/RNA Wash Buffer | Zymo Research | D7010-3-6 | A salt and ethanol buffer used for purification of DNA and RNA |
| DNBSEQ G-400RS | MGI | G-400RS | High throughput sequencer |
| MGIEasy RNA Directional Library Prep Set | MGI | 1000006386 | Generate libraries for MGI high-throughput sequencing platforms from total RNA. |
| Mini-Beadbeater-96 | BioSpec | 1001 | A high energy, high throughput cell disrupter |
| NEBNext rRNA Depletion Kit (bacteria) | New England Biolabs | E7850X | Efficient and specific depletion of bacterial rRNA (5S, 16S, 23S) |
| Nunc Lab-Tek II chamber slide system | Thermo Fisher Scientific | 154534 | 8-well chamber slide with removable wells |
| Qubit Fluorometer | Thermo Fisher Scientific | Q33238 | Fluorometer for DNA, RNA and proteins |
| Qubit RNA HS Assay Kit | Thermo Fisher Scientific | Q32852 | High sensitivity fluorometric assay to measure RNA concentration |
| Spin-Away Filters | Zymo Research | C1006-50-F | Silica-based spin column primarily used to bind or remove genomic DNA |
| Sterile inoculation loops, 1 uL | Sarstedt | 86.1567.050 | Sterile, disposable inoculation loops for manipulation of microorganisms |
| ZR BashingBead Lysis tubes | Zymo Research | S6003-50 | 2 mL tubes containing 0.1 and 0.5 mm bead lysis matrix for homogenizing biological samples |
| Zymo Spin IIICG Columns | Zymo Research | C1006-50-G | Silica-based spin column for purification of DNA and RNA |
| Zymo-Spin III-HRC Filters | Zymo Research | C1058-50 | Remove inhibitors such as polyphenolic compounds, humic/fulvic acids, tannins, melanin, etc. |
| Zymobiomics DNA/RNA Miniprep kit | Zymo Research | R2002 | DNA and RNA dual extraction kit |
| Zymobiomics HRC Prep solution | Zymo Research | D4300-7-30 | To be used with Zymo-Spin III-HRC Filters to remove PCR inhibitors |
Riferimenti
- Beaudoin, T., Waters, V. Infections With Biofilm Formation: Selection of Antimicrobials and Role of Prolonged Antibiotic Therapy. The Pediatric Infectious Disease Journal. 35 (6), 695-697 (2016).
- Bjarnsholt, T., et al. The in vivo biofilm. Trends in Microbiology. 21 (9), 466-474 (2013).
- Folsom, J. P., et al. Physiology of Pseudomonas aeruginosa in biofilms as revealed by transcriptome analysis. BMC Microbiology. 10, 294(2010).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43 (3), 313-351 (2017).
- Bjarnsholt, T., Ciofu, O., Molin, S., Givskov, M., Hoiby, N. Applying insights from biofilm biology to drug development - can a new approach be developed. Nature Reviews Drug Discovery. 12 (10), 791-808 (2013).
- Colvin, K. M., et al. The Pel and Psl polysaccharides provide Pseudomonas aeruginosa structural redundancy within the biofilm matrix. Environmental Microbiology. 14 (8), 1913-1928 (2012).
- Hentzer, M., et al. Alginate overproduction affects Pseudomonas aeruginosa biofilm structure and function. Journal of Bacteriology. 183 (18), 5395-5401 (2001).
- Cury, J. A., Koo, H. Extraction and purification of total RNA from Streptococcus mutans biofilms. Analytical Biochemistry. 365 (2), 208-214 (2007).
- Francavilla, M., et al. Extraction, characterization and in vivo neuromodulatory activity of phytosterols from microalga Dunaliella tertiolecta. Current Medicinal Chemistry. 19 (18), 3058-3067 (2012).
- Atshan, S. S., et al. Improved method for the isolation of RNA from bacteria refractory to disruption, including S. aureus producing biofilm. Gene. 494 (2), 219-224 (2012).
- Franca, A., Melo, L. D., Cerca, N. Comparison of RNA extraction methods from biofilm samples of Staphylococcus epidermidis. BMC Research Notes. 4, 572(2011).
- Jurcisek, J. A., Dickson, A. C., Bruggeman, M. E., Bakaletz, L. O. In vitro biofilm formation in an 8-well chamber slide. The Journal of Visusalized Experiments. (47), e2481(2011).
- Beaudoin, T., Kennedy, S., Yau, Y., Waters, V. Visualizing the effects of sputum on biofilm development using a chambered coverglass model. The Journal of Visusalized Experiments. (118), e54819(2016).
- Cockeran, R., et al. Biofilm formation and induction of stress response genes is a common response of several serotypes of the pneumococcus to cigarette smoke condensate. The Journal of Infection. 80 (2), 204-209 (2020).
- Bisht, K., Moore, J. L., Caprioli, R. M., Skaar, E. P., Wakeman, C. A. Impact of temperature-dependent phage expression on Pseudomonas aeruginosa biofilm formation. npj Biofilmsand Microbiomes. 7 (22), (2021).
- Harrison, A., et al. Reprioritization of biofilm metabolism is associated with nutrient adaptation and long-term survival of Haemophilus influenzae. NPJ Biofilms and Microbiomes. 5 (1), 33(2019).
- Sousa, C., Franca, A., Cerca, N. Assessing and reducing sources of gene expression variability in Staphylococcus epidermidis biofilms. BioTechniques. 57, 295-301 (2014).
- Boom, R. Rapid and simple method for purification of nucleic acids. Journal of Clinical Microbiology. 28 (3), 495-503 (1990).
- Coppin, C. Re: How do silica based RNA spin columns only bind RNA and not DNA. , Available from: https://www.researchgate.net/post/How_do_silica_based_RNA_spin_columns_only_bind_RNA_and_not_DNA/60b017bffa5c4151cac1c/citation/download (2021).
- Kennedy, S. A complete guide to how nucleic extraction kits work. , Available from: https://bitesizebio.com/13516/how-dna-extraction-rna-miniprep-kits-work/ (2021).
- Qubit RNA HS Assay Kit User Guide. Thermo Fisher Scientific. , Available from: https://www.thermofisher.com/document-connect/document-connect.html?url=https%3A%2F%2Fassets.thermofisher.com%2FTFS-Assets%2FLSG%2Fmanuals%2FQubit_RNA_HS_Assay_UG.pdf&title=VXNlciBHdWlkZTogUXViaXQgUk5BIEhTIEFzc2F5IEtpdHM (2015).
- Mueller, O., Lightfoot, S., Schroeder, A. RNA Integrity Number (RIN) - Standardization of RNA Quality Control (Application report # 5989-1165EN). , Available from: https://www.agilent.com/cd/library/applications/5989-1165EN.pdf (2016).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 3(2006).
- Culviner, P. H., Guegler, C. K., Laub, M. T. A Simple, Cost-Effective, and Robust Method for rRNA Depletion in RNA-Sequencing Studies. mBio. 11 (2), (2020).
- MGIEasy RNA Directional Library Prep Set User Manual verA2. MGI Tech Co. , Available from: https://en.mgi-tech.com/products/reagents_info/14/ (2020).
- Andrews, S. FastQC: A Quality Control Tool for High Throughput Sequence Data. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina Sequence Data. Bioinformatics. , (2014).
- NCBI Resource Coordinators. Database resources of the National Center for Biotechnology Information. Nucleic Acids Research. 44 (1), (2016).
- Li, H. Aligning sequence reads, clone sequences and assembly contigs with BWA-MEM. arXiv. , (2013).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), (2009).
- Liao, Y., Shi, W. Read trimming is not required for mapping and quantification of RNA-seq reads at the gene level. NAR Genomics and Bioinformatics. 2 (3), (2020).
- Penir, S. Calculating Mapping Statistics from a SAM/BAM file using SAMtools and awk. , Available from: https://sarahpenir.github.io/bioinformatics/awk/calculating-mapping-stats-from-a-bam-file-using-samtools-and-awk/ (2019).
- Haile, S., et al. Evaluation of protocols for rRNA depletion based RNA sequencing of nanogram inputs of mammalian total RNA. PLoS ONE. 14 (10), 0224578(2019).
- Schuierer, S., et al. A comprehensive assessment of RNA-seq protocols for degraded and low-quantity samples. BMC Genomics. 18 (442), (2017).
- Shanker, S., et al. Evaluation of Commercially Available RNA Amplification Kits for RNA Sequencing Using Very Low Input Amounts of Total RNA. Journal of Biomolecular Techniques. 26 (1), (2015).
- Adiconis, X., et al. Comparative analysis of RNA sequencing methods for degraded or low-input samples. Nature Methods. 10, 623-629 (2013).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (13), (2016).
- Illumina, Inc. Coverage depth recommendations. , Available from: https://www.illumina.com/science/technology/next-generation-sequencing/plan-experiments/coverage.html (2021).
- What is a good sequencing depth for bulk RNA-Seq. ECSEQ Bioinformatics. , Available from: https://www.ecseq.com/support/ngs/what-is-a-good-sequencing-death-for-bulk-rna-seq (2019).
- Bedre, R. Sequencing coverage and breadth of coverage. , Available from: https://www.reneshbedre.com/blog/sequencing-coverage.html (2021).
- Dotsch, A., et al. The Pseudomonas aeruginosa transcriptome in planktonic cultures and static biofilms using RNA sequencing. PLoS One. 7 (2), 31092(2012).
- Chen, Y., et al. Population dynamics and transcriptomic responses of Pseudomonas aeruginosa in a complex laboratory microbial community. npj Biofilms and Microbiomes. 5 (1), (2019).
- Thoming, J. G., et al. Parallel evolutionary paths to produce more than one Pseudomonas aeruginosa biofilm phenotype. NPJ Biofilms and Microbiomes. 6, 2(2020).
- Soares, A., et al. Understanding ciprofloxacin failure in Pseudomonas aeruginosa biofilm: persister cells survive matrix disruption. Frontiers in Microbiology. 10, 2603(2019).
- Whiteley, M., et al. Gene expression in Pseudomonas aeruginosa biofilms. Nature. 413, (2001).
- Chomczynski, P., Sacchi, N. The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: twenty-something years on. Nature Protocols. 1, (2006).
- Liu, Y., Zhou, J., White, K. P. RNA-seq differential expression studies: more sequence or more replication. Bioinformatics. 30 (3), (2014).
- Wieczorek, D., Delauriere, L., Schagat, T. Methods of RNA Quality Assessment. , Available from: https://www.promega.ca/resources/pubhub/methods-of-rna-quality-assessment/ (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon