Method Article
Triagem para inibidores da pirofosia ligada à membrana de maritoga
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Aqui apresentamos um método de triagem para inibidores de pirofosfatase ligado à membrana (da Thermotoga maritima)com base na reação azul molibdênio em um formato de placa de 96 poços.
Resumo
Pirofosfatases ligadas à membrana (mPPases) são enzimas diméricas que ocorrem em bactérias, arqueais, plantas e parasitas protistas. Estas proteínas acovardam o pirofosfato em duas moléculas de ortofosfato, que é acoplado com próton e / ou íon de sódio bombeamento através da membrana. Como não existem proteínas homólogas em animais e humanos, os mPPases são bons candidatos na concepção de potenciais alvos de drogas. Aqui apresentamos um protocolo detalhado para a tela para inibidores de mPase utilizando a reação azul molibdênio em um sistema de placa de 96 poços. Usamos mPase da bactéria termofílica Thermotoga maritima (TmPPase) como uma enzima modelo. Este protocolo é simples e barato, produzindo um resultado consistente e robusto. Leva apenas cerca de uma hora para concluir o protocolo de ensaio de atividade desde o início do ensaio até a medição de absorção. Uma vez que a cor azul produzida neste ensaio é estável por um longo período de tempo, ensaio (s) subseqüente pode ser realizado imediatamente após o lote anterior, e a absorção pode ser medida mais tarde para todos os lotes de uma só vez. A desvantagem deste protocolo é que ele é feito manualmente e, portanto, pode ser desgastante, bem como exigir boas habilidades de pipetting e tempo de manutenção. Além disso, a solução arsenite-citrate usada neste ensaio contém arsenita de sódio, que é tóxica e deve ser tratada com as precauções necessárias.
Introdução
Aproximadamente 25% do total de proteínas celulares são proteínas da membrana e cerca de 60% delas são alvos de drogas1,2. Um dos alvos potenciais da droga3, pirofosfofases ligados à membrana (mPPases), são enzimas diméricas que bombeiam próton e/ou íon de sódio através da membrana por hidróforadede de pirofosfato em dois ortopofosfatos4. mPPases podem ser encontrados em vários organismos5, como bactérias, archaea, plantas e parasitas protistas, com exceção de seres humanos e animais4. Em parasitas protistas, por exemplo Plasmodium falciparum, Toxoplasma gondii e Trypanosoma brucei, mPPases são essenciais para a virulência parasita6 e nocaute desta expressão nos parasitas levam à incapacidade de manter pH intracelular após a exposição ao pH básico externo7. Devido à sua importância e falta de proteína homóloga presente nos vertebrados, os mPPases podem ser considerados como alvos potenciais de medicamentos para doenças protistais3.
A triagem in vitro dos inibidores de mPase neste trabalho é baseada em um sistema modelo TmPPase. TmPPase é um bombeamento de íons de sódio e mPase dependente de íons de potássio de T. maritima e tem sua atividade ideal em 71 °C8. Os benefícios desta enzima são, por exemplo, a sua facilidade na produção e purificação, boa estabilidade térmica e alta atividade específica. TmPPase mostra alta semelhança, além da conservação completa da posição, bem como a identidade de todos os resíduos catalíticos para os mPPases protista3,9 e para a estrutura resolvida de Vigna radiata10 mPPase. As estruturas disponíveis de TmPPase em diferentes conformações também são úteis para experimentos de design de drogas baseados em estrutura (como triagem virtual e design de novo).
Aqui relatamos um protocolo detalhado para triagem de inibidores de TmPPase em um formato de placa de 96 poços (Figura 1). O protocolo é baseado no método colorimétrico da reação azul molibdênio, que foi desenvolvida pela primeira vez por Fiske e Subbarow11. Este método envolve a formação de ácido 12-fosphomolídico de ortopofosfato e molibdênio condições ácidas, que é então reduzida para dar a espécie de fosphomolybdenum de cor azul característica12.
Protocolo
1. Preparação de proteínas
NOTA: A expressão e purificação de TmPPase foi descrita em outros lugares13.
- Prepare 10 mL da solução tampão de reativação contendo 20 mM 2-(N-morpholino) ácido ethanesulfonic (MES) pH 6.5, 3.5% (v/v) glycerol, 2 mM dithiothreitol (DTT), e 0.05% dodecyl maltoside (DDM).
- Prepare 10 mL da mistura de reação contendo 200 mM Tris-Cl pH 8.0, 8.0 mM MgCl2,333 mM KCl, e 67 mM NaCl.
NOTA: Mg2+ é necessário para chelate o pirofosfato como o substrato de mPase, K+ é necessário para aumentar a atividade enzimática como TmPPase é um mPase dependente de potássio, e Na+ é necessário para a atividade enzimática durante a translocação de íons de sódio por TmPPase. - Prepare 30 mg/mL lipossomos para reativação enzimática.
- Adicione 10 mL de 20 mM Tris-HCl pH 8.0 com 1 mM DTT a 0.3 g de L-α-phosphatidylcholine de soja para 10 mL de 20 mM Tris-HCl pH 8.0 com 1 mM DTT.
- Coloque o lipossoma no gelo, e sonicate com intervalo de pulso de 1 segundo por 1 minuto, faça uma pausa por 1 minuto, e repita até que a solução se torne amarelo transparente.
- Aliquot os lipossomos, congelar em nitrogênio líquido e armazenar a -80 °C até usado.
- Reativar a enzima.
- Misture 40 μL da solução de lipossomos com 22,5 μL de 20% DDM.
- Aqueça a mistura em 55 °C por 15 min e deixe esfriar a temperatura ambiente.
- Adicione 36,5 μL da solução tampão de reativação, misture e adicione 1 μL de proteína concentrada (13 mg/mL) para fazer uma concentração total de 0,13 mg/mL.
NOTA: A proteína é geralmente congelada em alíquotas de 10 μL após a purificação e descongelada no gelo antes do uso.
- Tome 20 μL da enzima reativada e adicione a 1.480 μL da mistura de reação, em seguida, misture suavemente.
NOTA: A adição da enzima reativada à mistura de reação deve ser realizada pouco antes de ser usada.
2. Preparação composta
- Dissolva os compostos em dimetil sulfoxida (DMSO) para fazer soluções de estoque de 25 a 100 mM em 50-200 μL, com base na disponibilidade dos compostos.
NOTA: Todos os compostos usados aqui(Figura 2A)foram publicados anteriormente9. Se a solubilidade composta for baixa, a concentração de estoque pode ser ajustada em conformidade. - Prepare três concentrações diferentes de cada composto na água.
NOTA: As concentrações finais na mistura de reação serão 1, 5 e 50 micromolar ou 1, 5 e 20 micromolar para compostos solúveis e com moderação solúveis, respectivamente.- Diluir a solução de estoque com água para 1 mL em microtubos para dar 2 μM, 10 μM e 100 μM para compostos solúveis, ou alternativamente 2 μM, 10 μM e 40 μM para compostos parparingly solúveis.
- Vortex a solução composta instantaneamente após a diluição da solução de estoque para a mistura adequada.
- Verifique se há agregação composta usando um nefrômetro.
NOTA: Este foi estudado como triplicados em três concentrações (1 μM, 5 μM e 20 μM) e normalizado ao espaço em branco em uma placa de 96 poços.- Dispense 75 μL da mistura da reação em cada poço usando uma pipeta multichannel.
- Adicione 75 μL de cada composto (para o espaço em branco, use 75 μL de água em vez disso) e misture por pipetting cima e para baixo 5 ×.
- Medir cada poço em 300 V usando um nephelometer microplate.
3. Reagentes para a preparação do ensaio
- Prepare a solução arsenite-citrate.
- Pese 5 g de arsenita de sódio e 5 g de dihydrate de citrato trissódico.
CUIDADO: O arsenite do sódio é tóxico, assim use o equipamento protetor apropriado e segure com cuidado especial. Como precaução, não segure antes que todas as precauções de segurança necessárias tenham sido lidas e compreendidas. Segure apenas em um capô de fumaça, a fim de não inalar poeira / vapores do composto ou sua solução (s). Se inalado, mova-se para o ar fresco e obtenha atendimento médico. Use óculos de segurança químicos adequados, luvas de proteção e roupas para evitar a ingestão e contato olho/pele. Se engolido, ligue imediatamente para um centro de intoxicação ou médico/médico. Se ele fica na pele ou no olho (s), lave com muita água e obter atenção médica. - Dissolva-se em 100 mL de água.
- Adicione 5 mL de ácido acético glacial, misture e adicione água a 250 mL.
- Armazenar à temperatura ambiente protegido da luz.
NOTA: A solução é estável por mais de um ano.
- Pese 5 g de arsenita de sódio e 5 g de dihydrate de citrato trissódico.
- Prepare a solução A e a solução B.
- Para solução A, adicione 10 mL de gelo frio 0,5 M HCl para 0,3 g de ácido ascórbico. Dissolva o ácido ascórbico por vórtice.
- Para solução B, adicione 1 mL de água gelada a 70 mg de tetrahidrato e vórtice de amônio heptamolybdate para dissolver.
NOTA: Armazenar ambas as soluções no gelo até o uso. Para a consistência do resultado do ensaio, ambas as soluções podem ser armazenadas no gelo por um máximo de uma semana.
- Prepare o padrão de fosfato (Pi)com a concentração de 0 μM, 62,5 μM, 250 μM e 500 μM para calibração.
- Adicione 0 μL, 25 μL, 50 μL e 100 μL de 5 mM Na2HPO4 desidratar a quatro microtubos contendo 370 μL da mistura de reação.
- Cubra até 1 mL com água.
4. Ensaio de atividade para uma placa de 96 poços
NOTA: Veja a Figura 1 para o fluxo de trabalho esquemático do ensaio.
- Adicione 1 mL de solução B a 10 mL de solução A, misture vórtice e guarde a solução no gelo.
NOTA: Esta solução deve ser transparente e amarela. Mantenha a solução A + B no gelo por pelo menos 30 minutos antes do uso. No entanto, use a solução dentro de 3 h, pois vai mal depois de armazenamento a longo prazo. - Adicione 40 μL de 0 μM, 62,5 μM, 250 μM e 500 μM Pi padrão para as tiras de tubo em triplicado usando uma pipeta multicanal.
NOTA: A mistura de reação sem Pi adicionado será usado como um espaço em branco. - Adicione 25 μL de solução composta para as tiras de tubo usando uma pipeta multicanal.
NOTA: Cada composto tem três concentrações diferentes em triplicado, o que é suficiente para a estimativa inicial da concentração inibil máxima (IC50). Para uma determinação ic50 mais precisa, oito concentrações compostas diferentes podem ser usadas. Para a enzima desinibida, a solução composta é substituída por quantidade igual de água. Como controles positivos foram utilizados 2,5 μM, 25 μM e 250 μM de imidodifosfato (IDP). - Adicione 15 μL de mistura de solução mPPase para as tiras de tubo (exceto para os tubos contendo Pi padrão) usando uma pipeta multicanal.
- Selar as tiras do tubo com uma folha de vedação adesiva. Corte a folha de vedação para separar cada tira de tubo.
- Pré-incubar as amostras para 5 min a 71 °C. Coloque as amostras no bloco de aquecimento com intervalo de 20 s entre cada tira, a fim de minimizar o consumo de tempo durante as etapas subsequentes.
- Para cada tira, abra a vedação adesiva. Adicione 10 μL de 2 mM pirofosfato de sódio dibasic usando uma pipeta multicanal e misture por pipetting cima e para baixo para 5 ×. Selar a tira de tubo novamente usando a mesma vedação.
NOTA: Esta etapa pode inicialmente ser difícil de realizar em 20 s; no entanto, ele vai se tornar mais fácil depois de alguns ensaios. - Incubar a 71 °C por 5 min.
- Coloque as amostras no aparelho de resfriamento com intervalo de 20 s entre cada tira. Deixe esfriar por 10 min, mas centrífuga cada tira brevemente após 5 min de resfriamento, para decantar gotas de água a folha de vedação, em seguida, colocá-lo de volta para o aparelho de resfriamento e remover o vedação.
NOTA: O aparelho de refrigeração pode simplesmente ser feito colocando uma placa PCR de 96 poços em uma placa de Petri de poliestireno (tamanho 150 mm × 15 mm) cheia de água e congelada por pelo menos 1 h. O aparelho deve ser retirado do congelador cerca de 5 minutos antes do início do ensaio. Não tire o aparelho de resfriamento logo antes do resfriamento da amostra, pois congelará a mistura de reação e dificultará o desenvolvimento da cor. - Após 10 min de resfriamento, adicione 60 μL de solução A + B, misture por pipetting para cima e para baixo para 5 × e manter as tiras de tubo no aparelho de refrigeração por 10 min.
- Adicione 90 μL da solução arsenite-citrato e mantenha em temperatura ambiente por pelo menos 30 min para produzir uma cor azul estável.
CUIDADO: Devido à sua toxicidade todas as soluções que contenham arsenita de sódio devem ser tratadas com cuidado extra em todos os tempos. Assim, a adição de solução arsenite-citrato deve ser feita em um capô de fumaça. - Dispense 180 μL de cada mistura de reação em uma clara microplaca de poliestireno de 96 poços.
- Medir a absorção de cada poço em 860 nm usando um espectrômetro microplaca.
5. Análise de resultados
- Média dos triplicados de cada amostra e dospp P i. Em seguida, subtraia com o espaço em branco para eliminar o sinal de fundo.
- Faça uma curva de calibração traçando a absorção (A860)valores contra a quantidade de Pi padrão (nmol) e realizar uma regressão linear para obter a função de linha de tendência usando a seguinte fórmula:
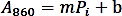
- Calcule a quantidade de fosfato (nmol) liberada da reação enzimática com base na fórmula de regressão linear acima.
- Calcule a atividade específica usando a seguinte fórmula:
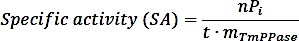
onde nPi é a quantidade de fosfato liberado da reação (nmol), t é o tempo de reação (min), e mTmPPase é a quantidade do TmPPase puro usado no ensaio (mg). - Calcule a atividade percentual para cada concentração inibidora usando a seguinte fórmula:
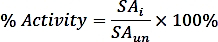
onde SAiisa atividade específica de uma amostra com inibidor e SAun é a atividade específica da amostra desinibida. - Calcule o logIC50 (estimativa) e IC50 (estimativa) com um ajuste de regressão não linear da curva de dose-resposta de quatro parâmetros usando a seguinte fórmula:
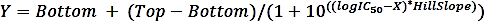
onde X é registro de concentração (μM), Y é atividade (%), Top e Bottom são planaltos na mesma unidade que Y (100% e 0%, respectivamente), logIC50 tem as mesmas unidades de registro como X, e HillSlope = fator de inclinação ou encosta, que é unitless.
NOTA: Software(Tabela de Materiais)é usado para a montagem. Use a concentração de 0,01 μM (em vez de 0,00 μM) para a amostra sem inibidor, pois o logarithm de zero não é definido.
Resultados
Neste protocolo, oito compostos (1-8) foram testados(Figura 2A)juntamente com o BID, inibidor comum de pirofosfatases, como controle positivo. Cada composto foi testado em três concentrações diferentes (1 μM, 5 μM e 20 μM) em triplicado. O fluxo de trabalho da triagem é representado na Figura 1,a partir da amostra e preparação do reagente até a medida de absorção em 860 nm.
Ao final deste protocolo, após a adição da solução A + B e arsenite-citrate, as soluções desenvolvem uma cor azul estável com a absorção máxima em 709 nm e 860 nm14 devido à complexa formação de íons de fosfato com molítoque que pode ser observado e mostra a ocorrência da reação enzimática. Para este experimento, usamos a absorção em 860 nm para a medição da quantidade Pi liberada, pois tem melhor limite de detecção e sensibilidade em comparação com a absorção em 709 nm15. A cor azul é totalmente desenvolvida em 30 min de incubação à temperatura ambiente e estável por pelo menos 5 h14. O ensaio tem a sensibilidade até pi concentração de 10 μM e a absorção é linear sobre uma faixa de concentração de 10-800 μM14. No resultado representativo aqui, os poços E1-E3(Figura 2C)contêm a mistura de reação sem inibidor e a solução azul pode ser observada no final do ensaio. Isso também pode ser observado em baixas concentrações compostas onde a inibição completa não foi alcançada, como nos poços F1-F3 para IDP e poços A4-A6 para composto 1 (ATC, um inibidor não competitivo recentemente conhecido de TmPPase9) na concentração de 2,5 μM e 1 μM, respectivamente. A maior concentração de DESLOCADO Ida e composto 1, menos a nenhuma cor azul pode ser observada (G1-G3 e H1-H3 para IDP e B4−B6 e C4−C6 para composto 1)indicando inibição da atividade enzimática. Todas as três concentrações de compostos não inibidores (2, 3e 8)apresentaram a mesma intensidade de cor azul, bem como E1-E3 sem qualquer inibidor (Figura 2C).
Após a medição de absorção em 860 nm, os dados podem ser processados e analisados (ver seção de protocolo 5). Figura 2D mostra o gráfico de calibração do padrão Pi com sua montagem linear (y = 0,0576x + 0,0019; r2 = 0,999). Figura 3 mostra o enredo da atividade enzimática (%) contra a concentração de cada composto testado. Para compostos com atividade de inibição, uma curva não linear também é mostrada. O IDP, usado como um controle positivo, mostra claramente uma diminuição na atividade em uma concentração mais elevada. O IC50 (estimativa) calculado com base em três concentrações diferentes é de 88,2 μM (Tabela 1),que é semelhante à medida anterior (80,0 μM) com oito pontos de concentração14. Os compostos 1, 4, 5, 6e 7 apresentaram tendência semelhante à do IDP, uma vez que a concentração aumentou com o IC50 (estimativa) de aproximadamente 1,3 μM, 7,4 μM, 19,0 μM, 37,4 μM e 156,1 μM, respectivamente (Tabela 1). Para compostos 2, 3,e 8 nenhuma redução na atividade ou inibição podem ser observadas nas concentrações do ensaio. Um ensaio adicional com oito pontos de concentração pode ser feito para gerar IC preciso50. A Figura 4 mostra a curva de inibição para compostos 1, 5, 6, 7 e 8 com IC50 de 1,7 μM, 21,4 μM, 58,8 μM, 239,0 μM e >500 μM, respectivamente9.
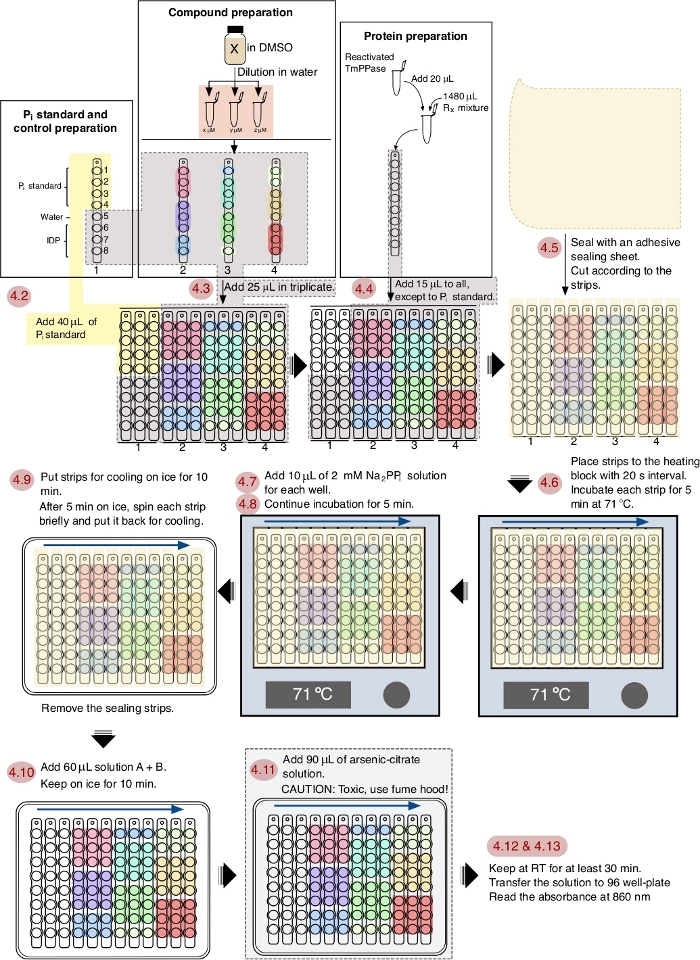
Figura 1: Um fluxo de trabalho esquemático do ensaio de inibição de TmPPase em um formato de placa de 96 poços. A numeração vermelha mostra os passos do ensaio de acordo com o protocolo e as setas azuis mostram a ordem de intervalo. Clique aqui para ver uma versão maior deste número.
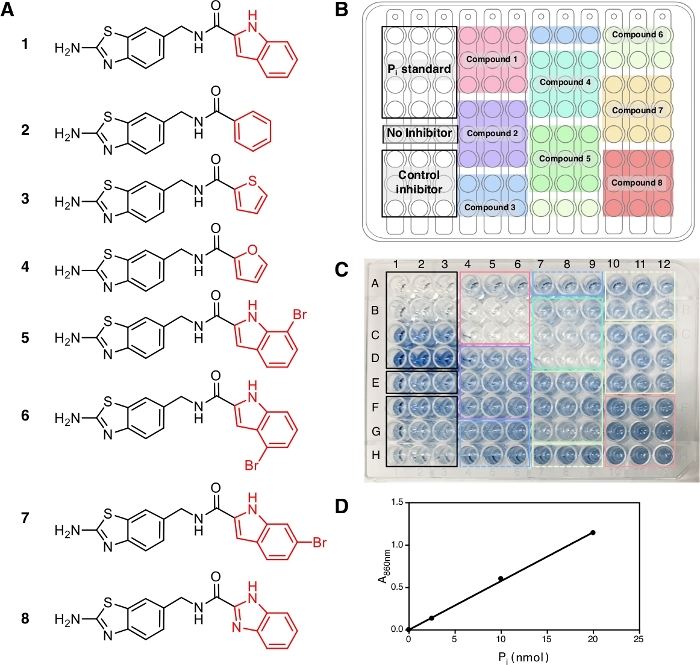
Figura 2: Amostras, seu arranjo e desenvolvimento de cores em uma placa de 96 poços. (A)As estruturas dos compostos 1-8 usado para o ensaio. A atividade de inibição destes compostos tem sido relatada em Vidilaseris et al.9. (B)Arranjo da amostra. (C)Desenvolvimento de cores, 30 min após a adição da solução arsenite-citrato. As concentrações de inibidor de controle (IDP) e amostras utilizadas, dispostas de cima para baixo, são de 2,5 μM, 25 μM e concentração de 250 μM e 1 μM, 5 μM e concentração de 20 μM, respectivamente. A intensidade da cor azul corresponde à quantidade de Pi lançado devido à reação enzimática e a falta de cor corresponde a nenhuma reação enzimática. (D)Curva de calibração para Pi padrão (nmol) contra A860 com montagem linear (y = 0,0576x + 0,0019; r2 = 0,999). Clique aqui para ver uma versão maior deste número.
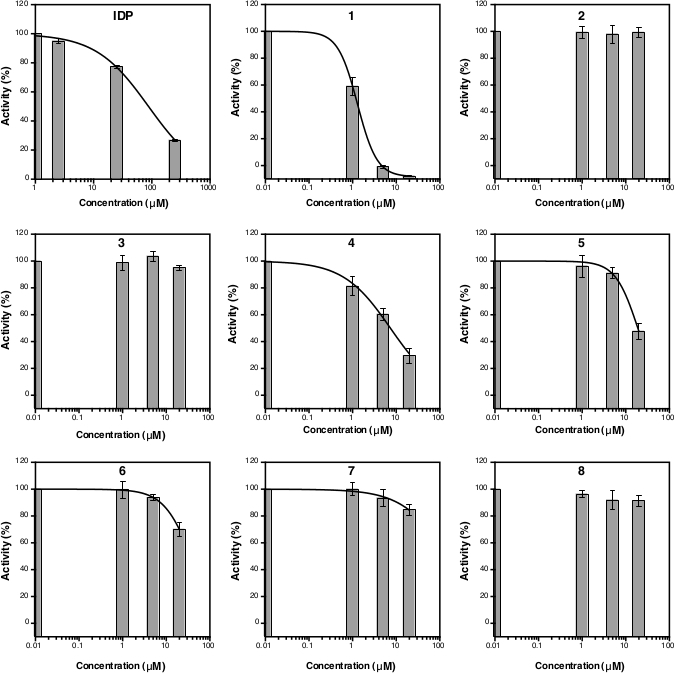
Figura 3: Curva da atividade percentual de TmPPase para três concentrações inibidoras diferentes. As curvas de regressão não lineares para calcular o IC50 (estimativa) são mostradas para o IDP, bem como para os compostos 1, 4, 5, 6 e 7, mas não para compostos 2, 3,e 8, pois não estavam inibindo a atividade de TmPPase nas concentrações de ensaio. O logIC50 e IC50 (estimativa) de cada composto é mostrado na Tabela 1. Todos os dados são mostrados como média ± SD com três replicações. Clique aqui para ver uma versão maior deste número.

Figura 4: Curva de inibição de oito pontos de concentração dos compostos 1, 5, 6, 7 e 8. Esta figura é tomada de Vidilaseris et al.9 com ligeira modificação. Todos os dados são mostrados como média ± SD com três replicações. Clique aqui para ver uma versão maior deste número.
| Amostra | LogIC 50 LogIC50 | IC50 (estimativa) (μM) |
| Idp | 1,95 ± 0,0142 | 87,9 ± 2,46 |
| 1 | 0,112 ± 0,0274 | 1,29 ± 0,0816 |
| 2 | − | sem inibição |
| 3 | − | sem inibição |
| 4 | 0,870 ± 0,0447 | 7,39 ± 0,760 |
| 5 | 1,28 ± 0,0296 | 19,0 ± 1,29 |
| 6 | 1,57 ± 0,0846 | 37,4 ± 7,29 |
| 7 | 2.19 ± 0,366 | 156 ± 131 |
| 8 | − | sem inibição |
Tabela 1: LogIC50 e IC50 (estimativa) do IDP e compostos 1-8 com base nos dados da Figura 3.
Discussão
Aqui relatamos um protocolo detalhado para triagem simples de inibidores para pirofosfatase ligada à membrana de T. maritima em um formato de placa de 96 poços baseado em Vidilaseris et al.14. Este protocolo é barato e baseado em ácido 12-fosphomolybdic, que é formado a partir de ortopofosfato e molibdênio em condições ácidas e reduzido a espécies de fosphomolybdenum com uma cor azul distinta12. Este método é preferido em relação a outros protocolos, como o ensaio verde malaquita mais sensível16,pois este método não mostra interferência na presença de alta concentração de fosfolípido que é necessária para a reativação tmppase14.
O fluxo de trabalho do protocolo de triagem é retratado na Figura 1 e esse processo pode ser totalmente realizado em 1h. Este protocolo é otimizado para TmPPase com a temperatura de trabalho ideal em 71 °C e um tempo de reação de 5 min. Como a água evaporará a essa temperatura a partir da mistura de reação, uma folha de vedação adesiva (cortada para caber e cobrir as tiras) é aplicada para evitar a evaporação14 e a água evaporada é simplesmente coletada com centrífuga. O tempo de incubação de 5 min é escolhido, pois ainda está na faixa linear do fosfato enzimaticamente liberado e suficiente para triagem confiável14. Neste protocolo, o tempo e as habilidades de pipetting são fatores importantes para obter um resultado bom e confiável. A adição de reagentes durante o ensaio com intervalo de 20 s entre tiras é uma opção de tempo otimizada para facilitar a realização das etapas subsequentes.
Para diferentes mPPases, a temperatura ideal e o tempo de incubação devem ser determinados separadamente antes do uso no ensaio inibitório. O protocolo de reativação de enzimas acima é otimizado para TmPPase e outros mPPases podem precisar de um protocolo de reativação diferente. Por exemplo, ddm não deve ser adicionado para reativação de mPase de pyrobaculum aerophilum como ele vai diminuir a sua atividade enzimática17. Como a enzima se tornará menos ativa se for preparada com bastante antecedência, a adição de enzima reativada deve ser adicionada à mistura de reação pouco antes do ensaio ser iniciado. Após a adição da solução arsenite-citrato o produto de reação é estável por pelo menos 5 h14. Portanto, o próximo lote do ensaio pode ser realizado imediatamente, e a medição de absorção pode ser feita mais tarde para todos os lotes de uma só vez.
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Este trabalho contou com o apoio das subvenções da Fundação Jane e Aatos Erkko e do BBSRC (BB/M021610) a Adrian Goldman, a Academia da Finlândia (nº 308105) para Keni Vidilaseris (nº 310297) a Henri Xhaard, e (nº 265481) a Jari Yli-Kauhaluoma, e os Fundos de Helsínquia da Universidade de Pesquisa a Gustav Boije af Gennäs. Os autores agradecem a Bernadette Gehl por sua ajuda técnica durante o projeto.
Materiais
| Name | Company | Catalog Number | Comments |
| Adhesive sealing sheet | Thermo Scientific | AB0558 | |
| Ammonium heptamolybdate tetrahydrate | Merck | F1412481 636 | |
| Ascorbic acid | Sigma-Aldrich | 95212-250G | |
| BioLite 96Well Multidish | Thermo Scientific | 130188 | |
| Dimethyl sulfoxide (DMSO) | Merck | 1167431000 | |
| 8-well PCR Tube Strips 0.2 ml without caps (120) | Nippon genetics | FG-028 | |
| Dodecyl maltoside (DDM) | Melford | B2010-100G | |
| Ethanol | Merck | 1009901001 | |
| Glacial acetic acid | Merck | 1000631011 | |
| Hydrochloric acid | Sigma-Aldrich | 258148-500ML | |
| Imidodiphosphate sodium salt | Sigma-Aldrich | I0631-1G | |
| L-α-Phosphatidyl choline from soybean lecithin | Sigma | 429415-100GM | |
| Magnesium chloride | Sigma-Aldrich | 8147330500 | |
| Multiplate 96-Well PCR Plates | Bio-Rad | MLL9651 | |
| MultiSkan Go | Thermo Scientific | 10680879 | |
| Nepheloskan Ascent (Type 750) | Labsystems | ||
| Polystyrene Petri dish (size 150 mm x 15 mm) | Sigma-Aldrich | P5981-100EA | |
| Potassium chloride | Merck | 104936 | |
| Prism 6 software | GraphPad | ||
| QBT2 Heating block | Grant Instruments | ||
| Sodium meta-arsenite | Fisher Chemical | 12897692 | |
| Sodium phosphate dibasic (Pi) | Sigma | S0876-1KG | |
| Sodium pyrophosphate dibasic | Fluka | 71501-100G | |
| Trisodium citrate dihydrate | Fluka | 71404-1KG |
Referências
- Terstappen, G. C., Reggiani, A. In silico research in drug discovery. Trends in Pharmacological Sciences. 22 (1), 23-26 (2001).
- Rask-Andersen, M., Almen, M. S., Schioth, H. B. Trends in the exploitation of novel drug targets. Nature Reviews Drug Discovery. 10 (8), 579-590 (2011).
- Shah, N. R., Vidilaseris, K., Xhaard, H., Goldman, A. Integral membrane pyrophosphatases: a novel drug target for human pathogens. AIMS Biophysics. 3 (1), 171-194 (2016).
- Baykov, A. A., Malinen, A. M., Luoto, H. H., Lahti, R. Pyrophosphate-Fueled Na+ and H+ Transport in Prokaryotes. Microbiology and Molecular Biology Reviews. 77 (2), 267-276 (2013).
- Serrano, A., Perez-Castineira, J. R., Baltscheffsky, M., Baltscheffsky, H. H+-PPases: yesterday, today and tomorrow. IUBMB Life. 59 (2), 76-83 (2007).
- Liu, J., et al. A vacuolar-H+-pyrophosphatase (TgVP1) is required for microneme secretion, host cell invasion, and extracellular survival of Toxoplasma gondii. Molecular Microbiology. 93 (4), 698-712 (2014).
- Lemercier, G., et al. A pyrophosphatase regulating polyphosphate metabolism in acidocalcisomes is essential for Trypanosoma brucei virulence in mice. Journal of Biological Chemistry. 279 (5), 3420-3425 (2004).
- Belogurov, G. A., et al. Membrane-bound pyrophosphatase of Thermotoga maritima requires sodium for activity. Biochemistry. 44 (6), 2088-2096 (2005).
- Vidilaseris, K., et al. Asymmetry in catalysis by Thermotoga maritima membrane-bound pyrophosphatase demonstrated by a nonphosphorus allosteric inhibitor. Science Advances. 5 (5), (2019).
- Lin, S. M., et al. Crystal structure of a membrane-embedded H+-translocating pyrophosphatase. Nature. 484 (7394), 399-403 (2012).
- Fiske, C. H., Subbarow, Y. The colorimetric determination of phosphorus. Journal of Biological Chemistry. 66 (2), 375-400 (1925).
- Nagul, E. A., McKelvie, I. D., Worsfold, P., Kolev, S. D. The molybdenum blue reaction for the determination of orthophosphate revisited: Opening the black box. Analytica Chimica Acta. 890, 60-82 (2015).
- Kellosalo, J., Kajander, T., Palmgren, M. G., Lopez-Marques, R. L., Goldman, A. Heterologous expression and purification of membrane-bound pyrophosphatases. Protein Expression and Purification. 79 (1), 25-34 (2011).
- Vidilaseris, K., Kellosalo, J., Goldman, A. A high-throughput method for orthophosphate determination of thermostable membrane-bound pyrophosphatase activity. Analytical Methods. 10 (6), 646-651 (2018).
- He, Z. Q., Honeycutt, C. W. A modified molybdenum blue method for orthophosphate determination suitable for investigating enzymatic hydrolysis of organic phosphates. Communications in Soil Science and Plant Analysis. 36 (9-10), 1373-1383 (2005).
- Martin, B., Pallen, C. J., Wang, J. H., Graves, D. J. Use of fluorinated tyrosine phosphates to probe the substrate specificity of the low molecular weight phosphatase activity of calcineurin. Journal of Biological Chemistry. 260 (28), 14932-14937 (1985).
- Strauss, J., Wilkinson, C., Vidilaseris, K., Harborne, S. P. D., Goldman, A. A simple strategy to determine the dependence of membrane-bound pyrophosphatases on K+ as a cofactor. Methods in Enzymology. 607, 131-156 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados